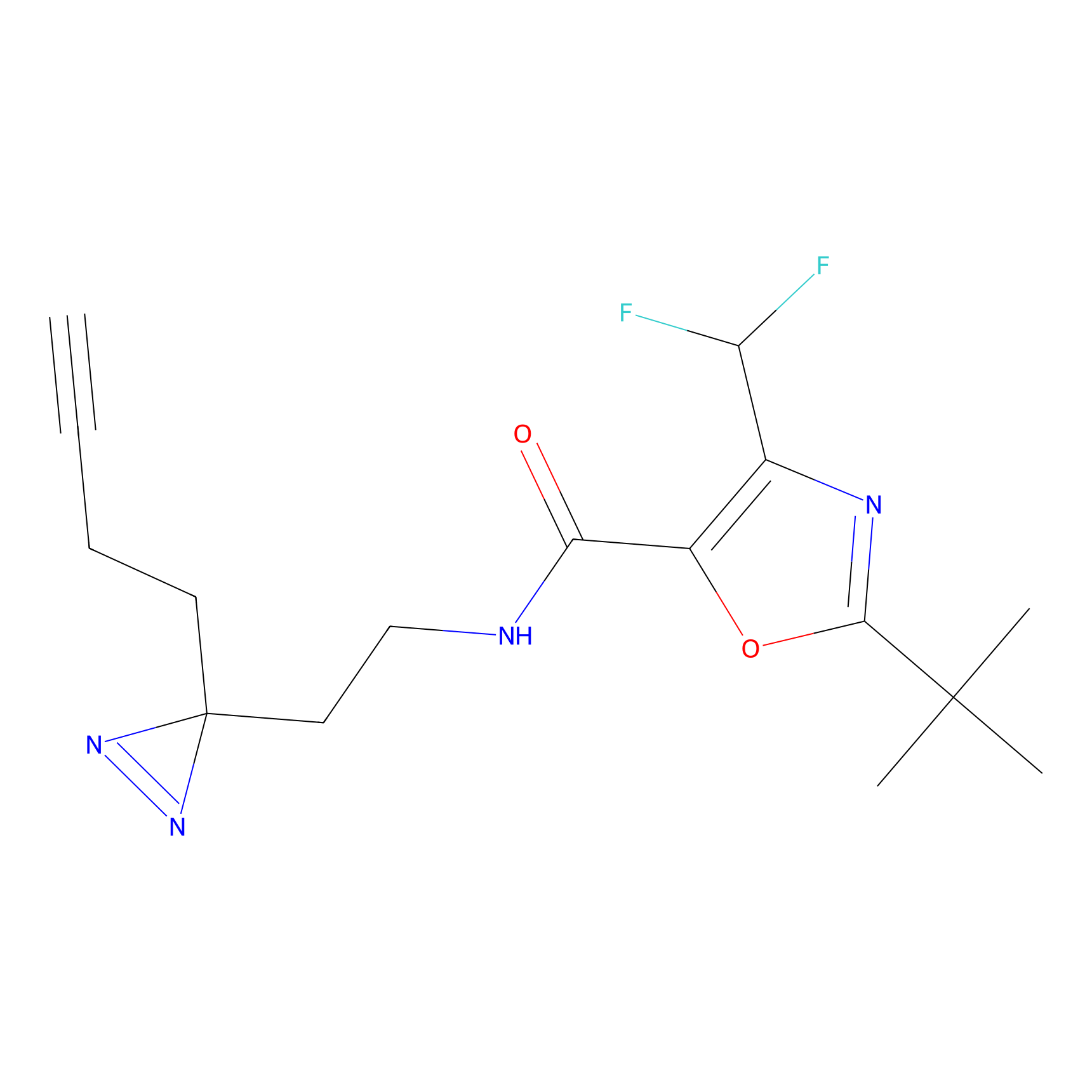
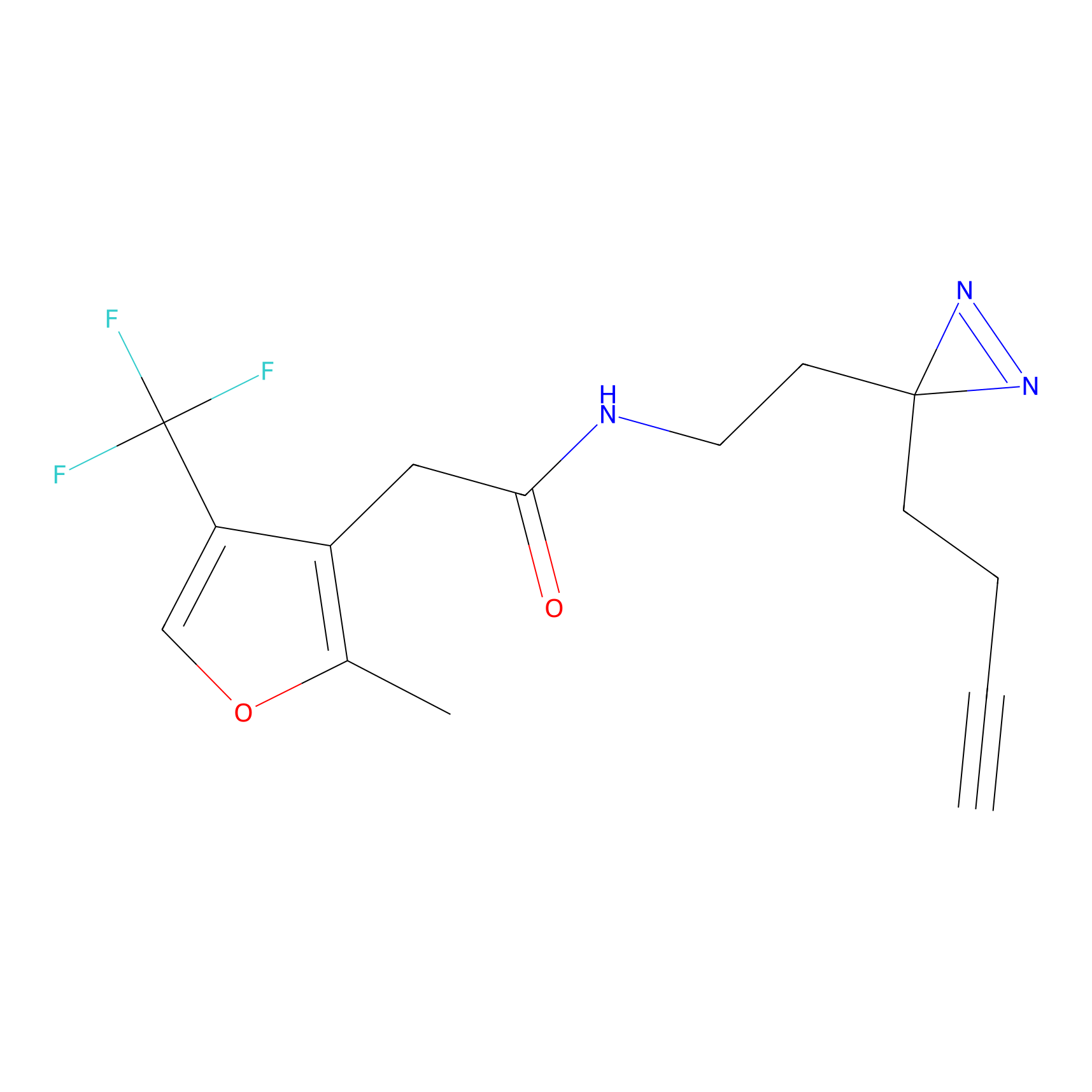
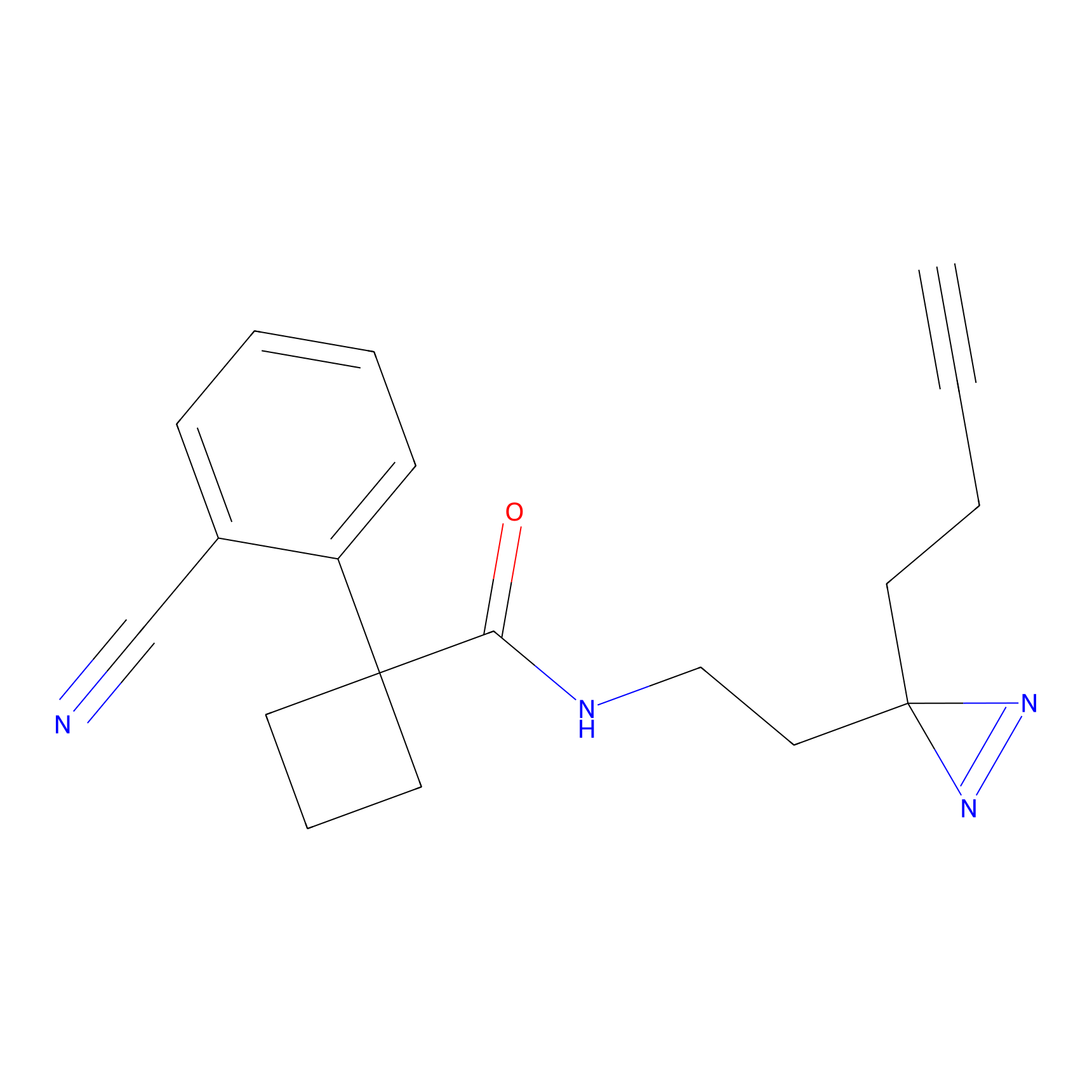
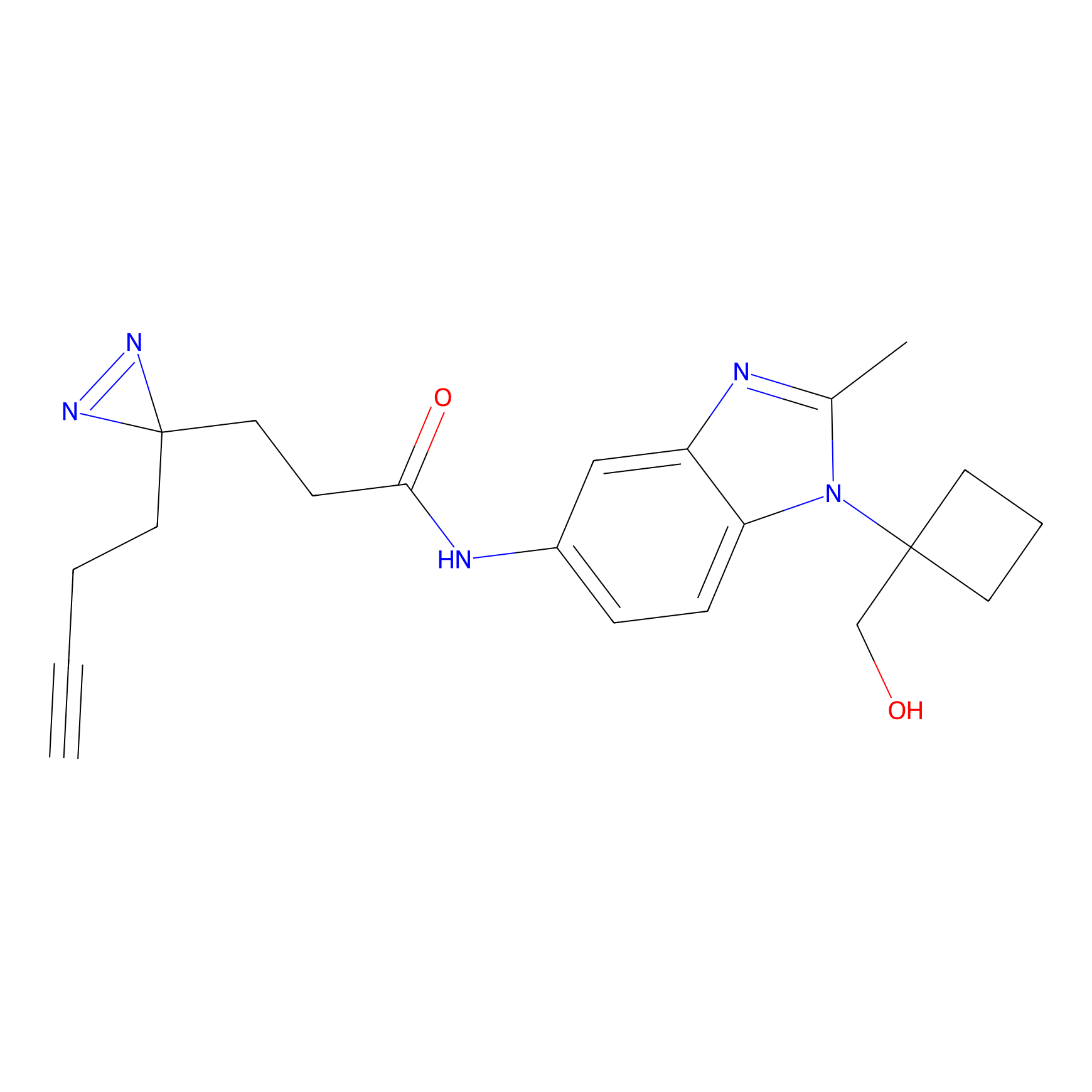
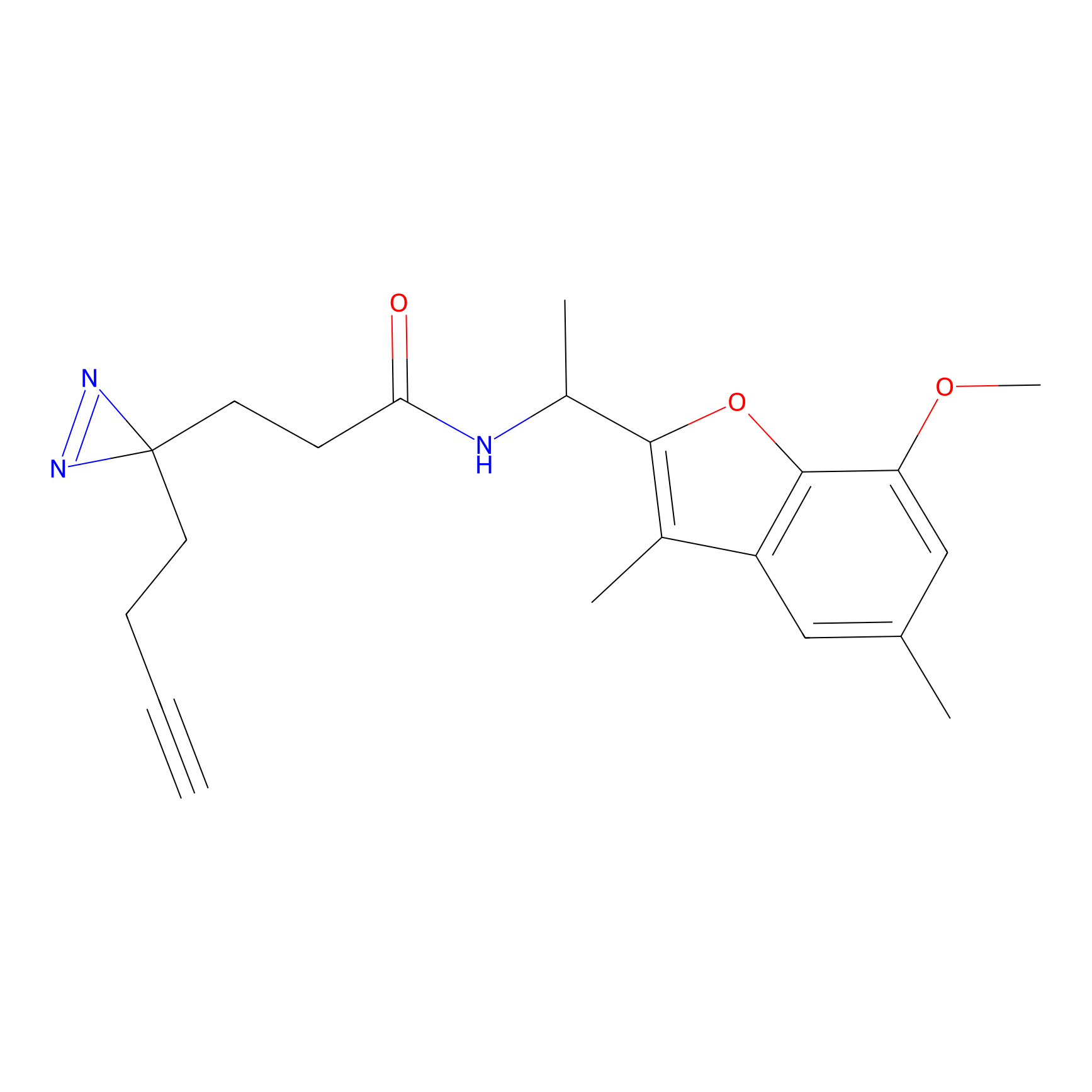
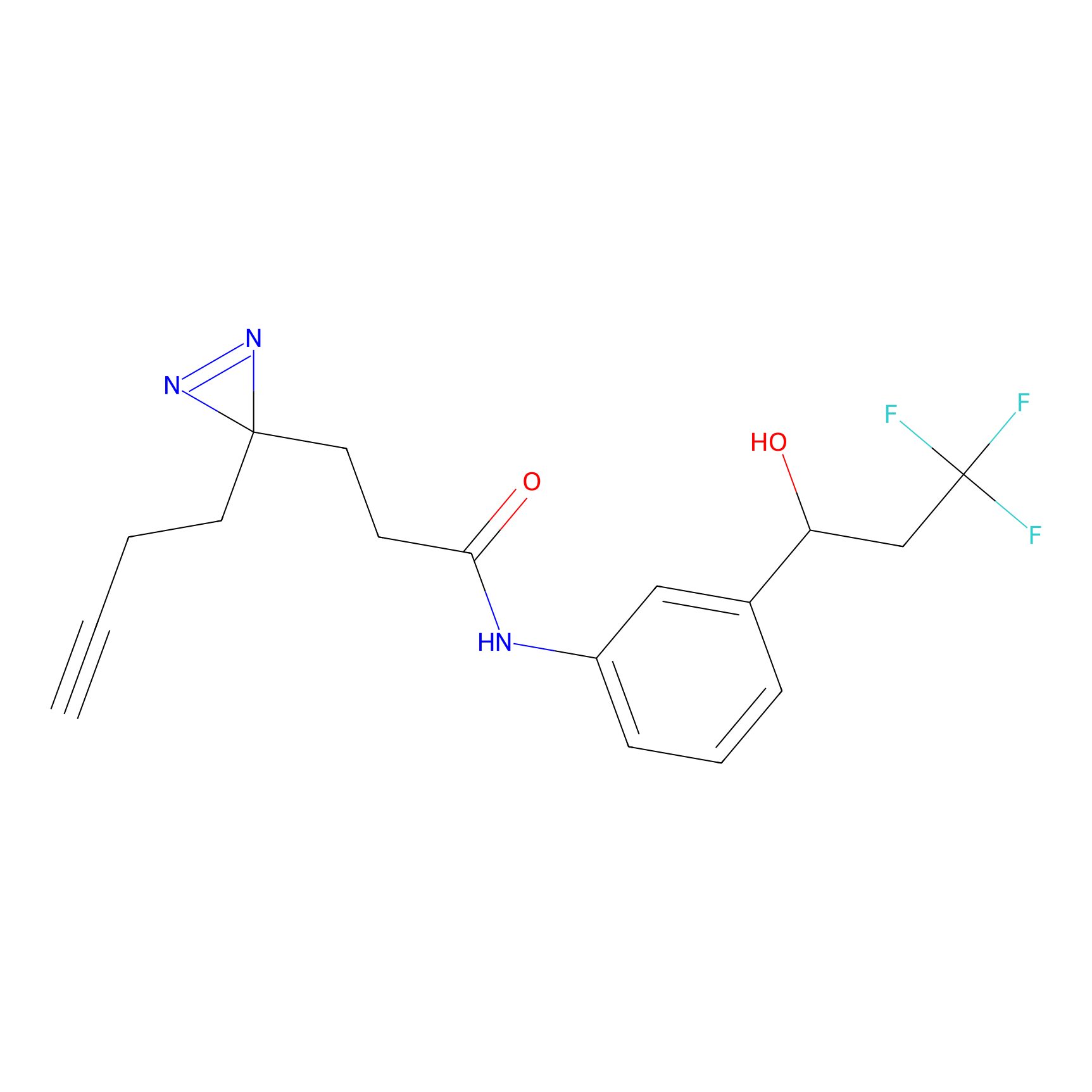
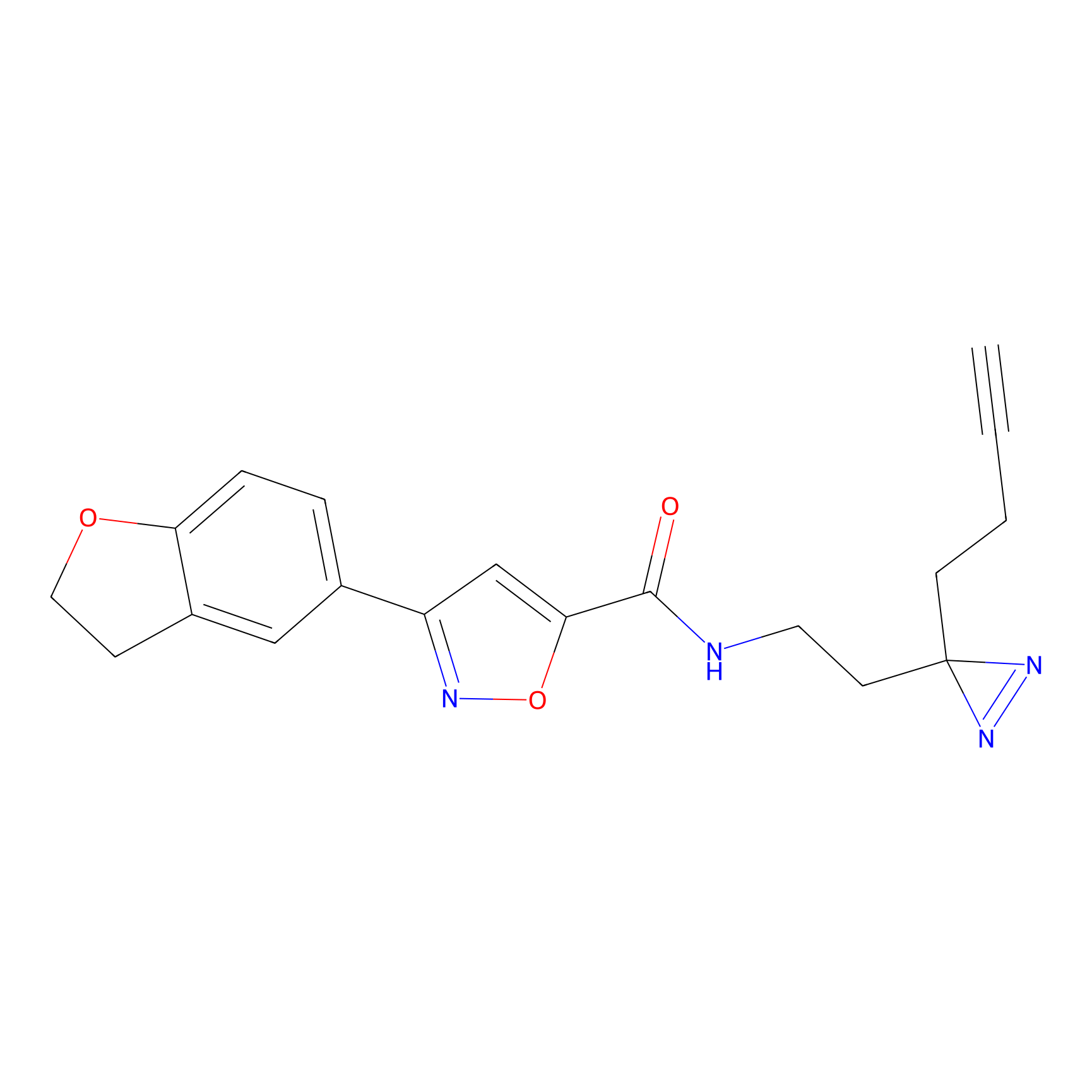
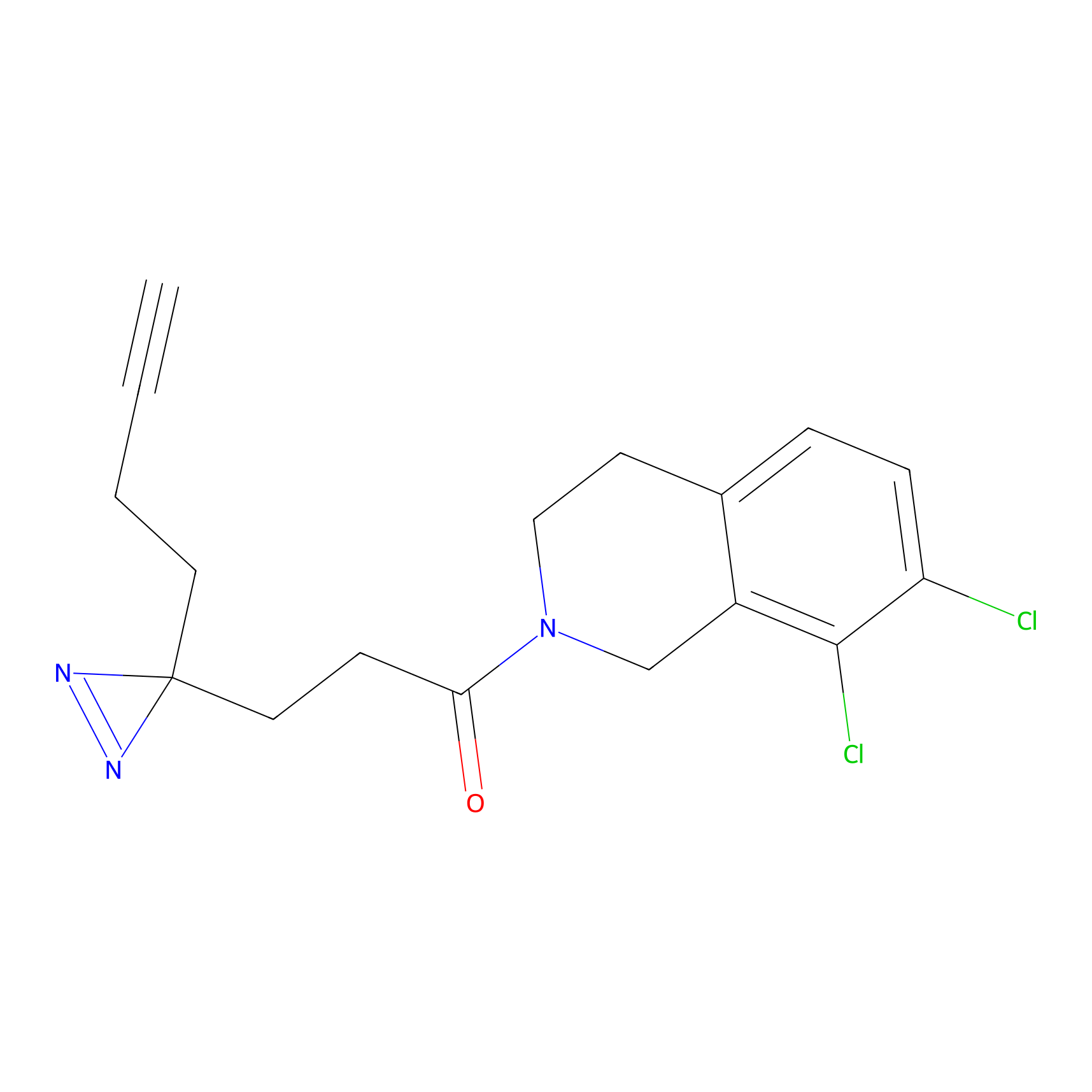
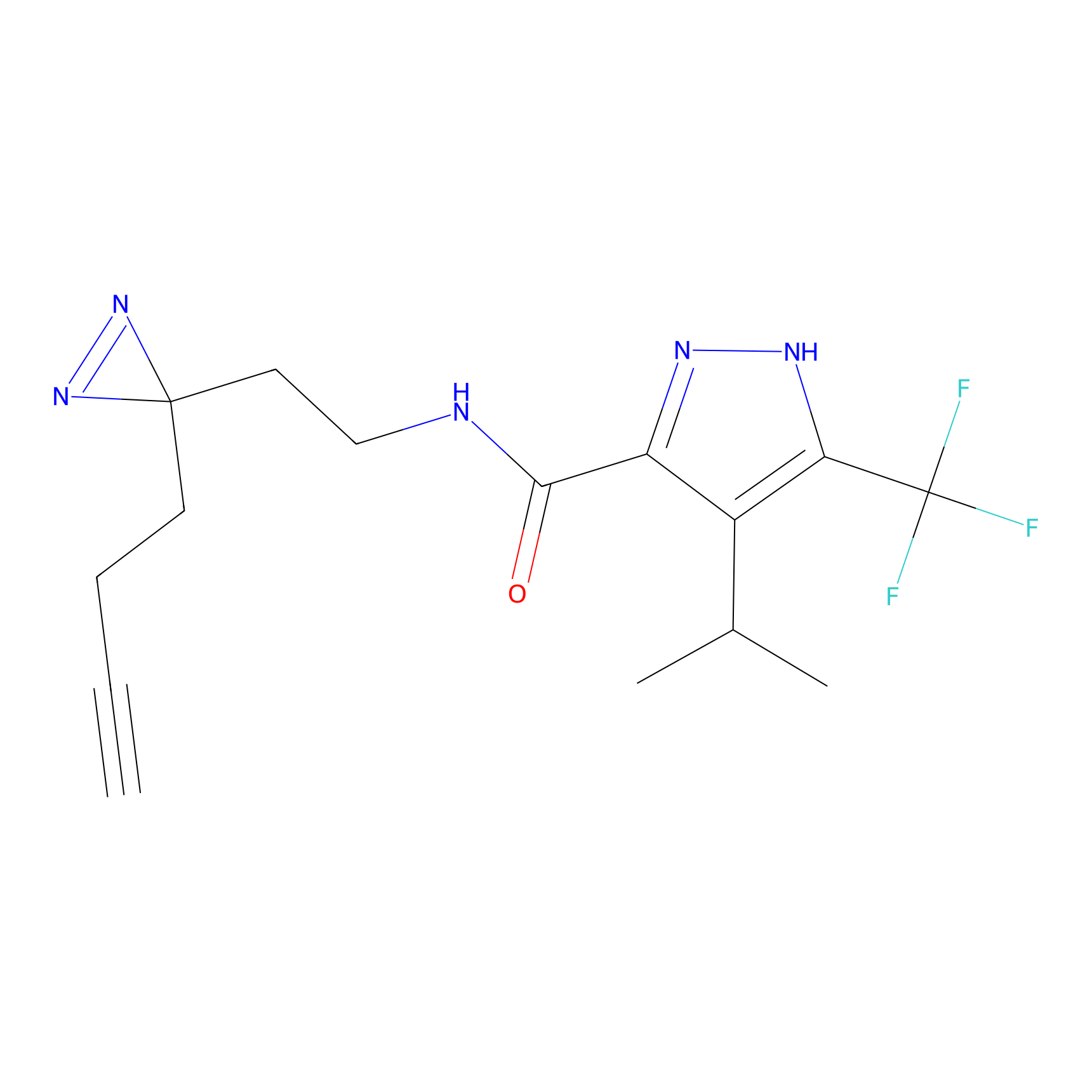
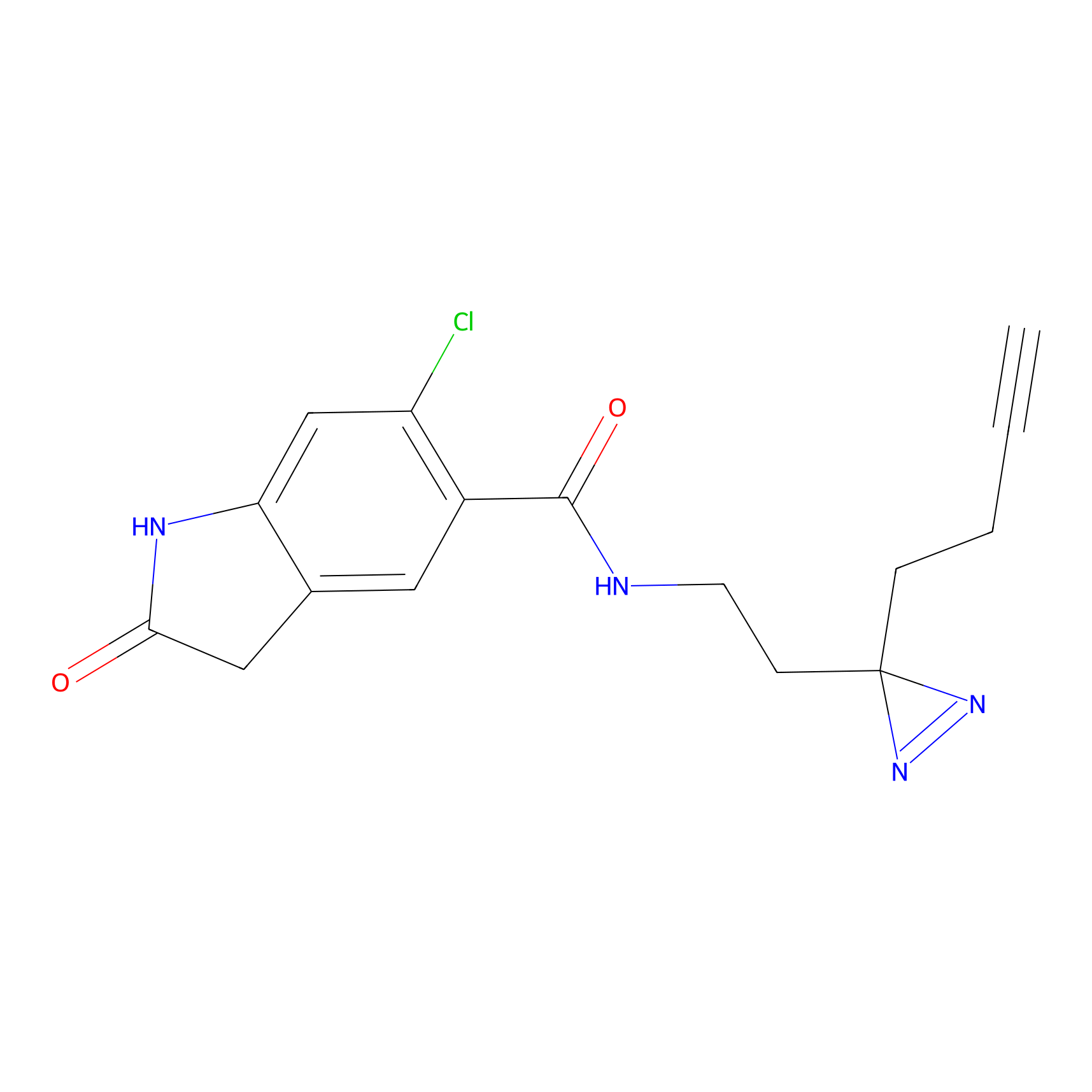
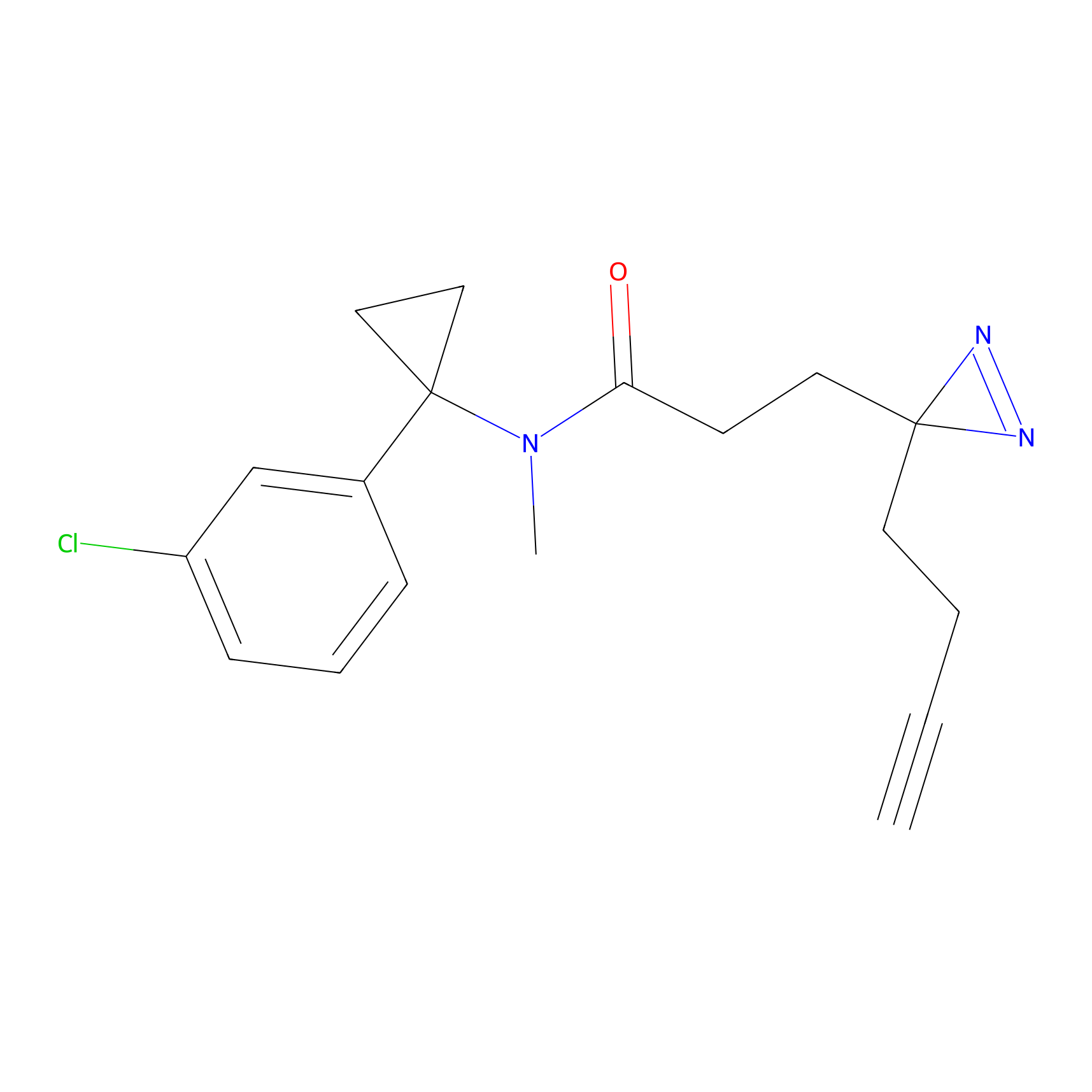
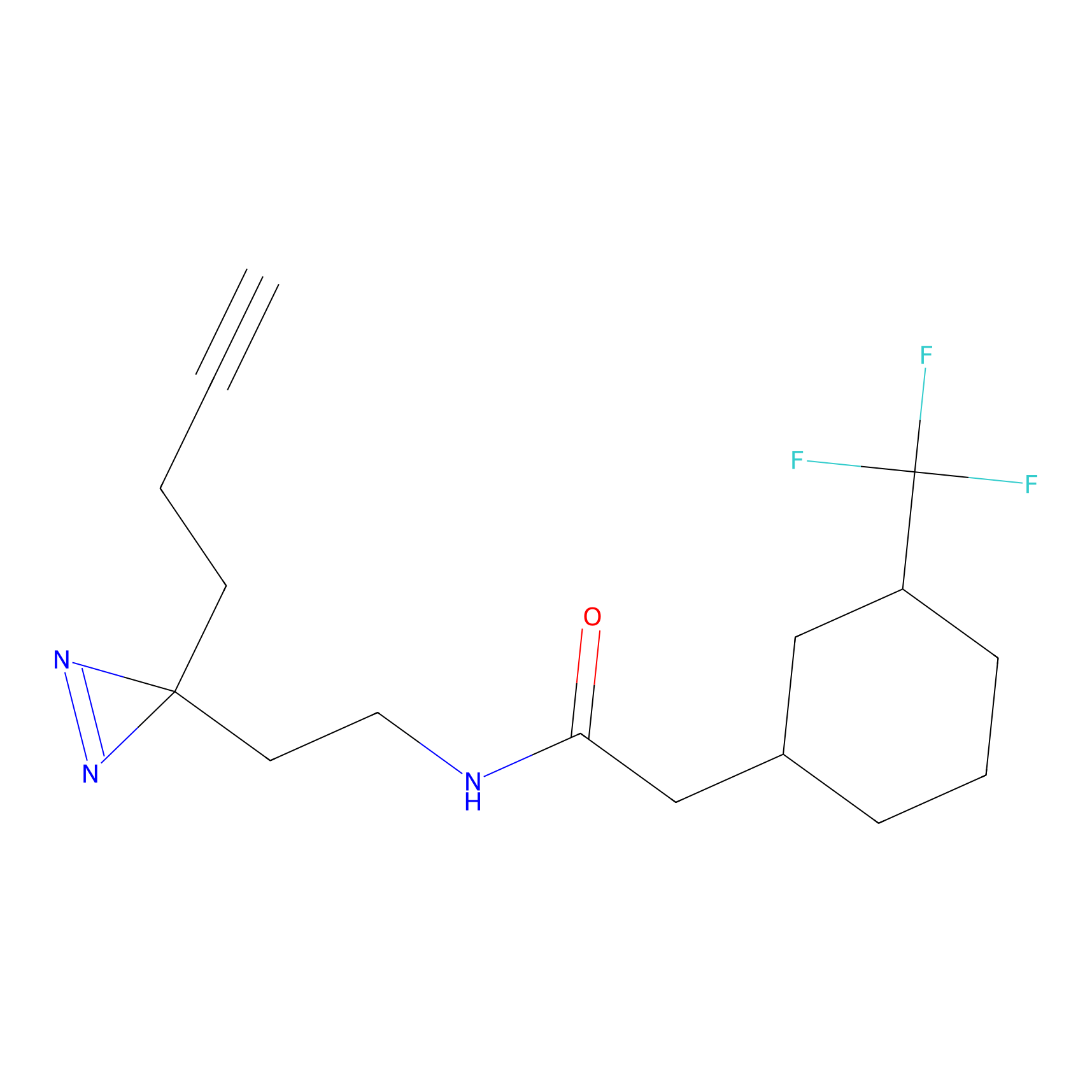
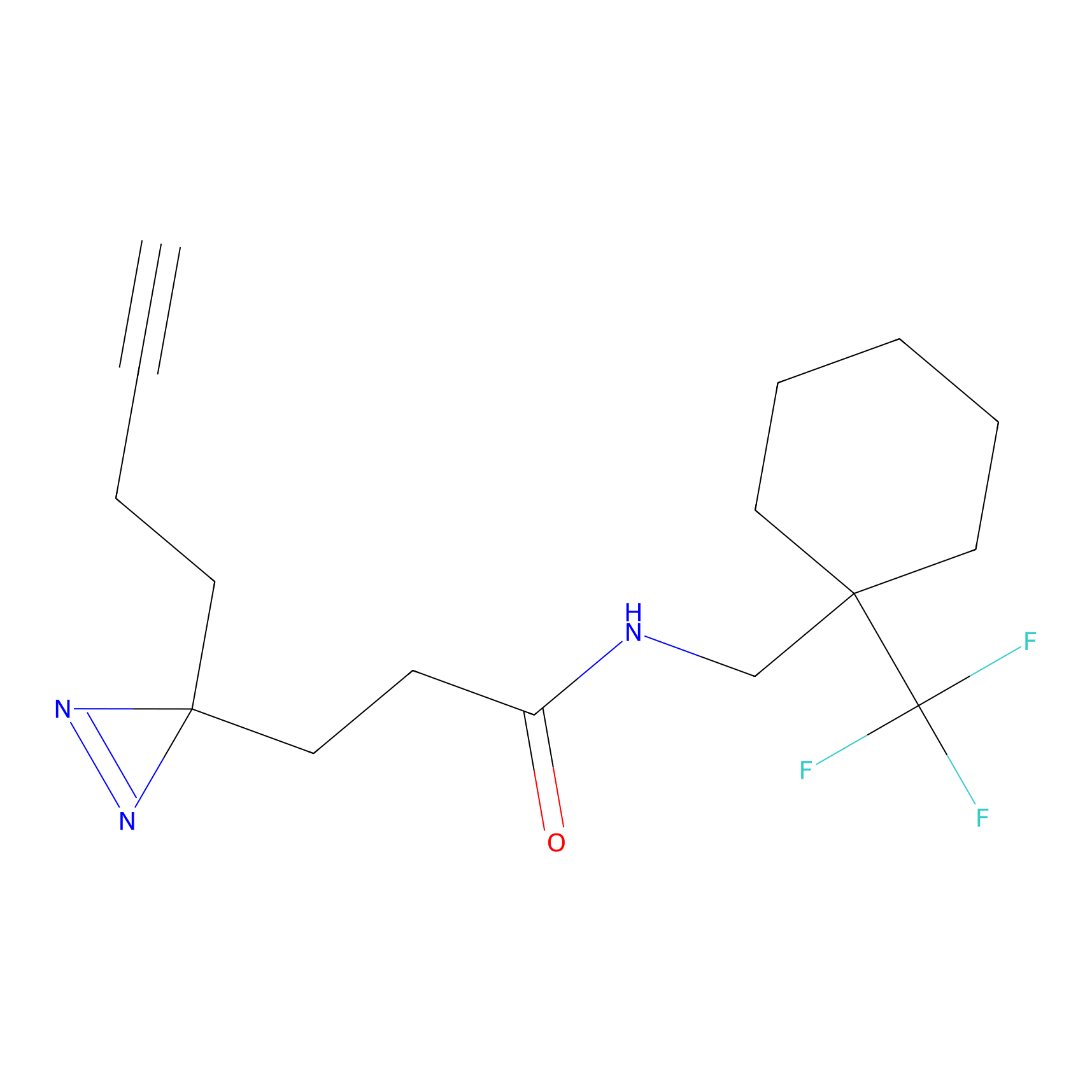
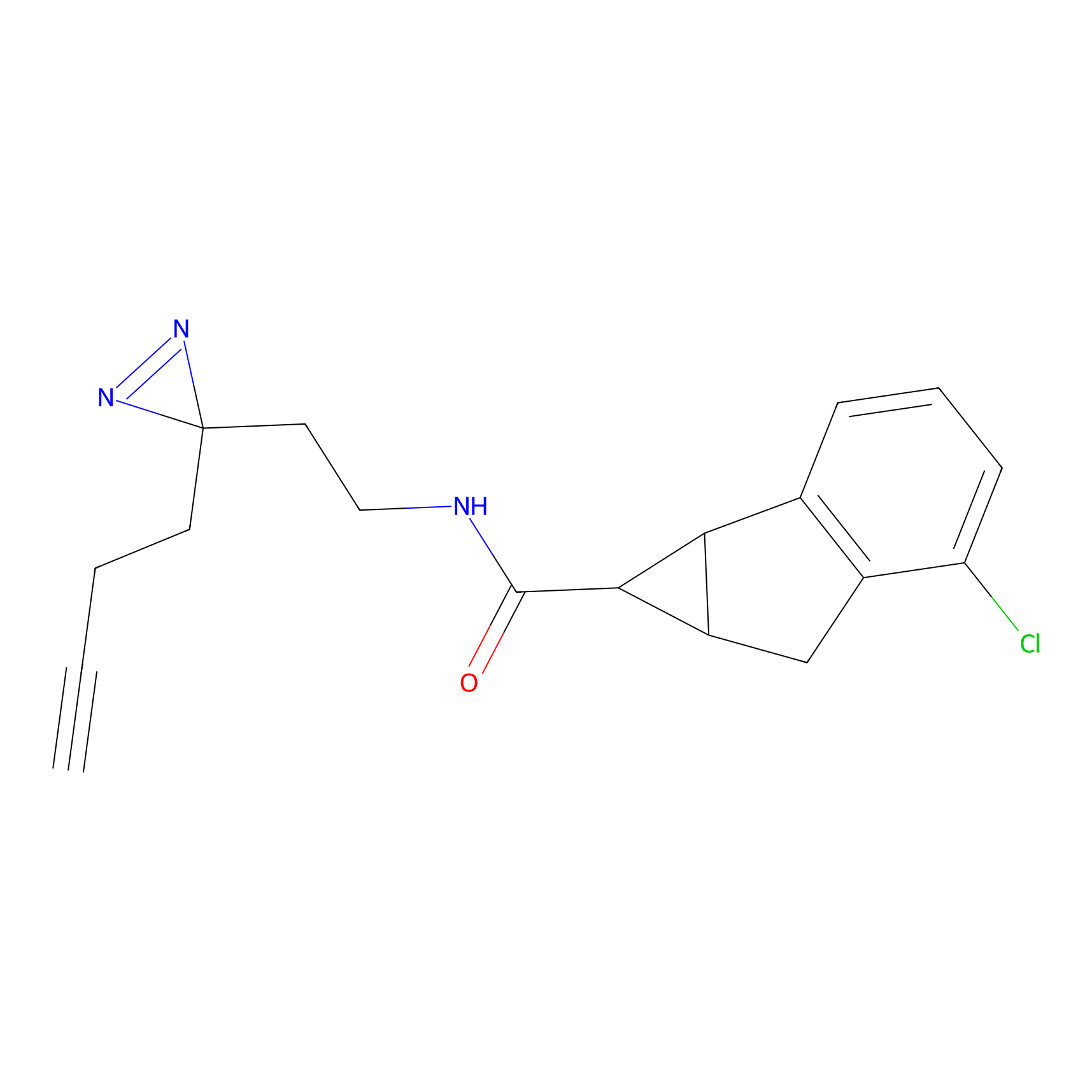
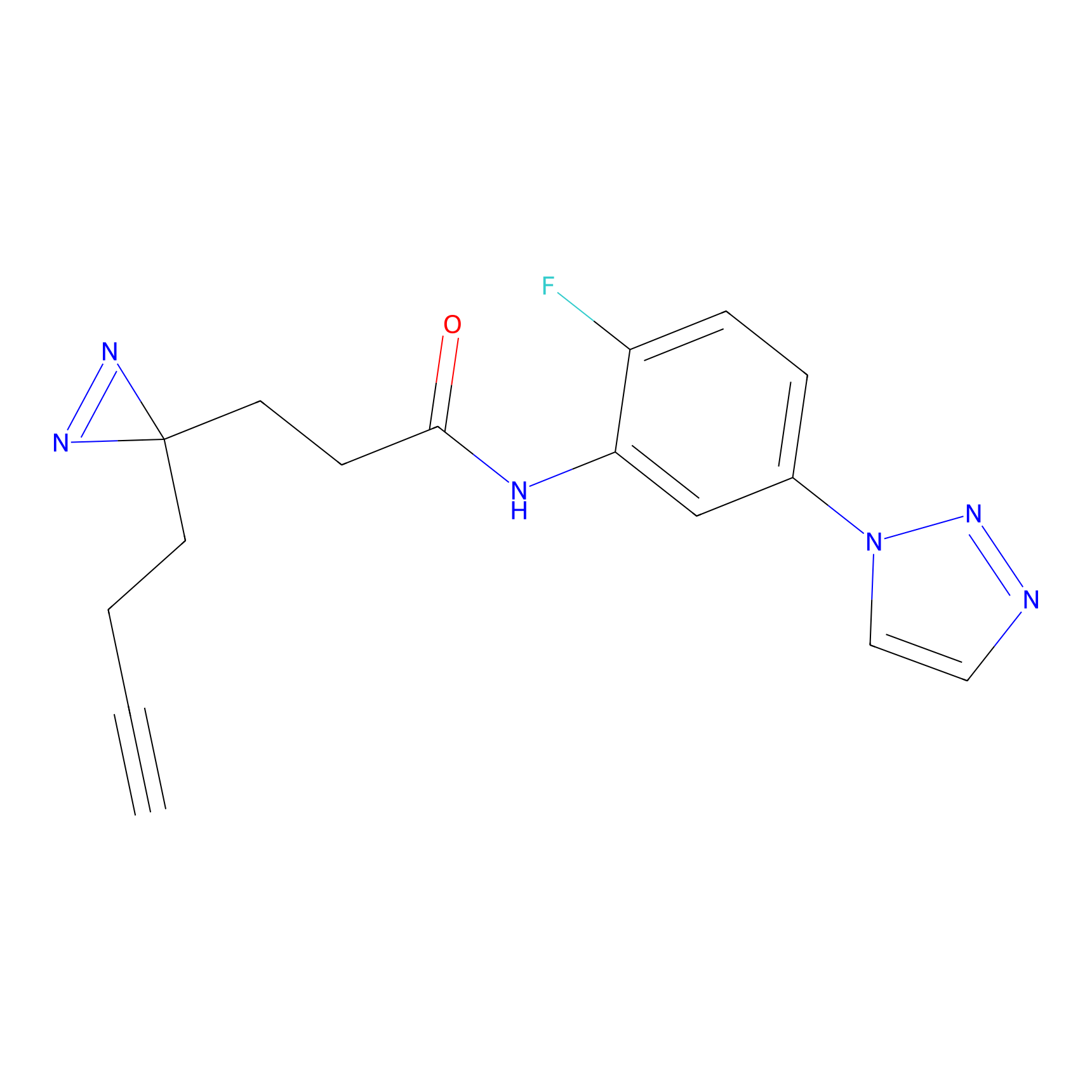
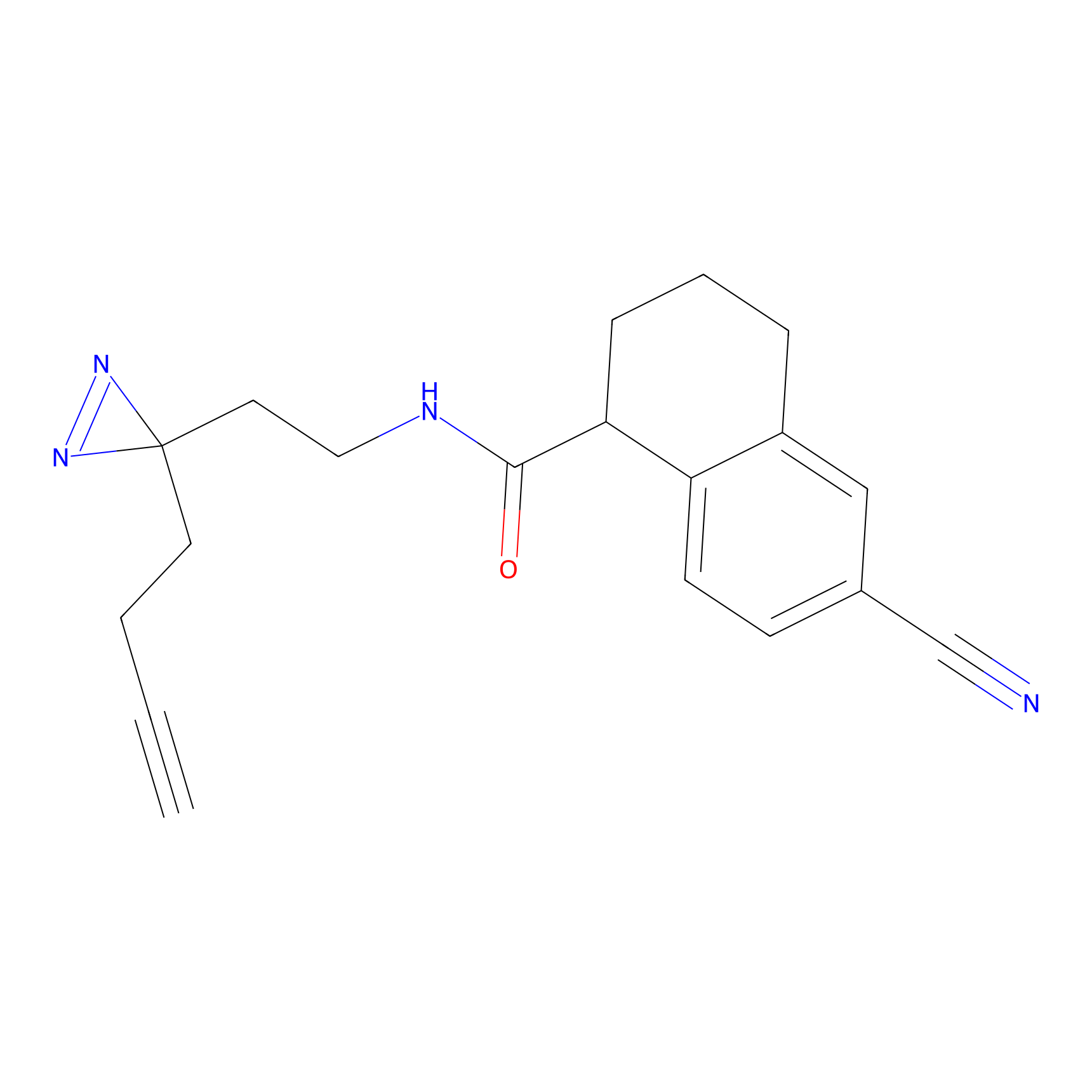
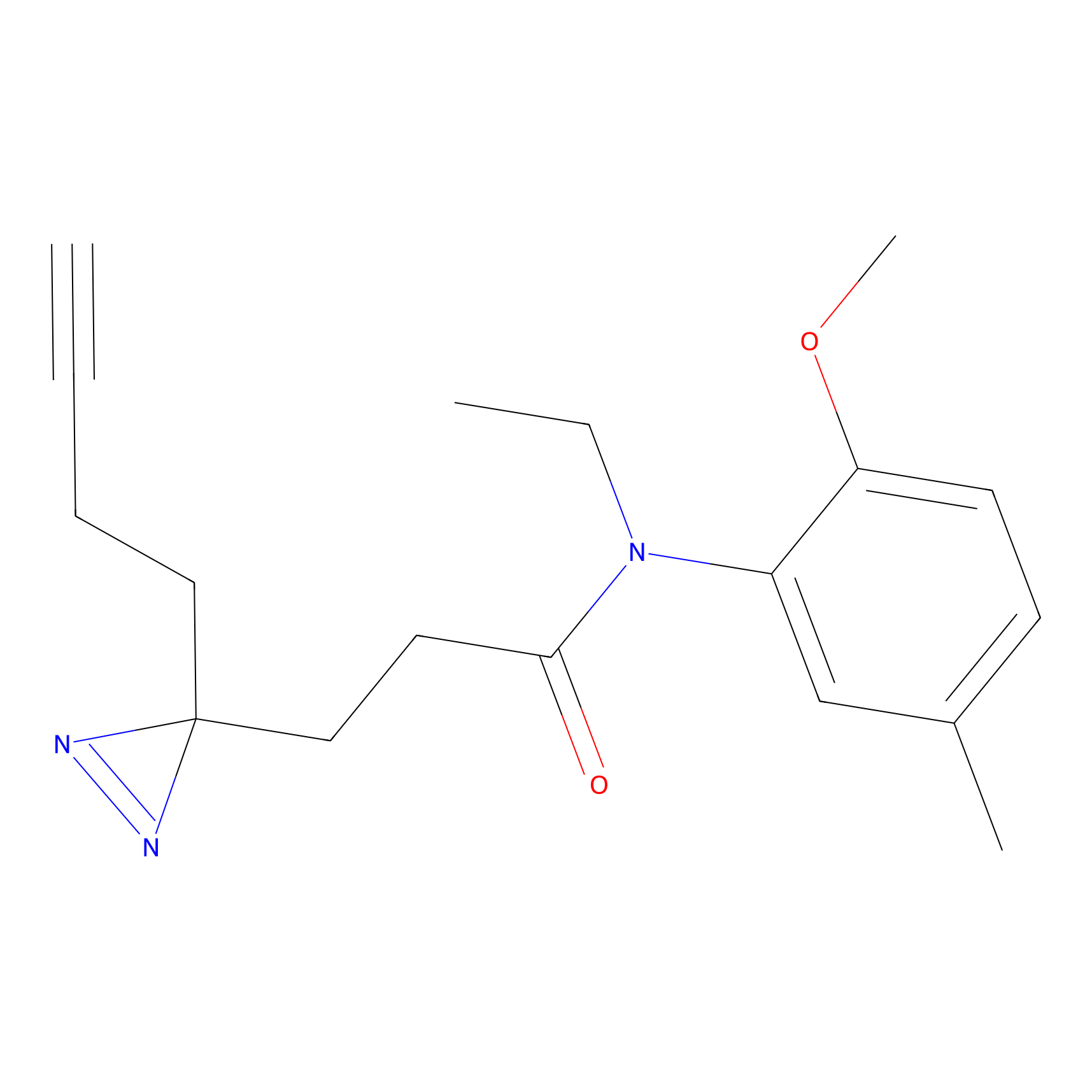
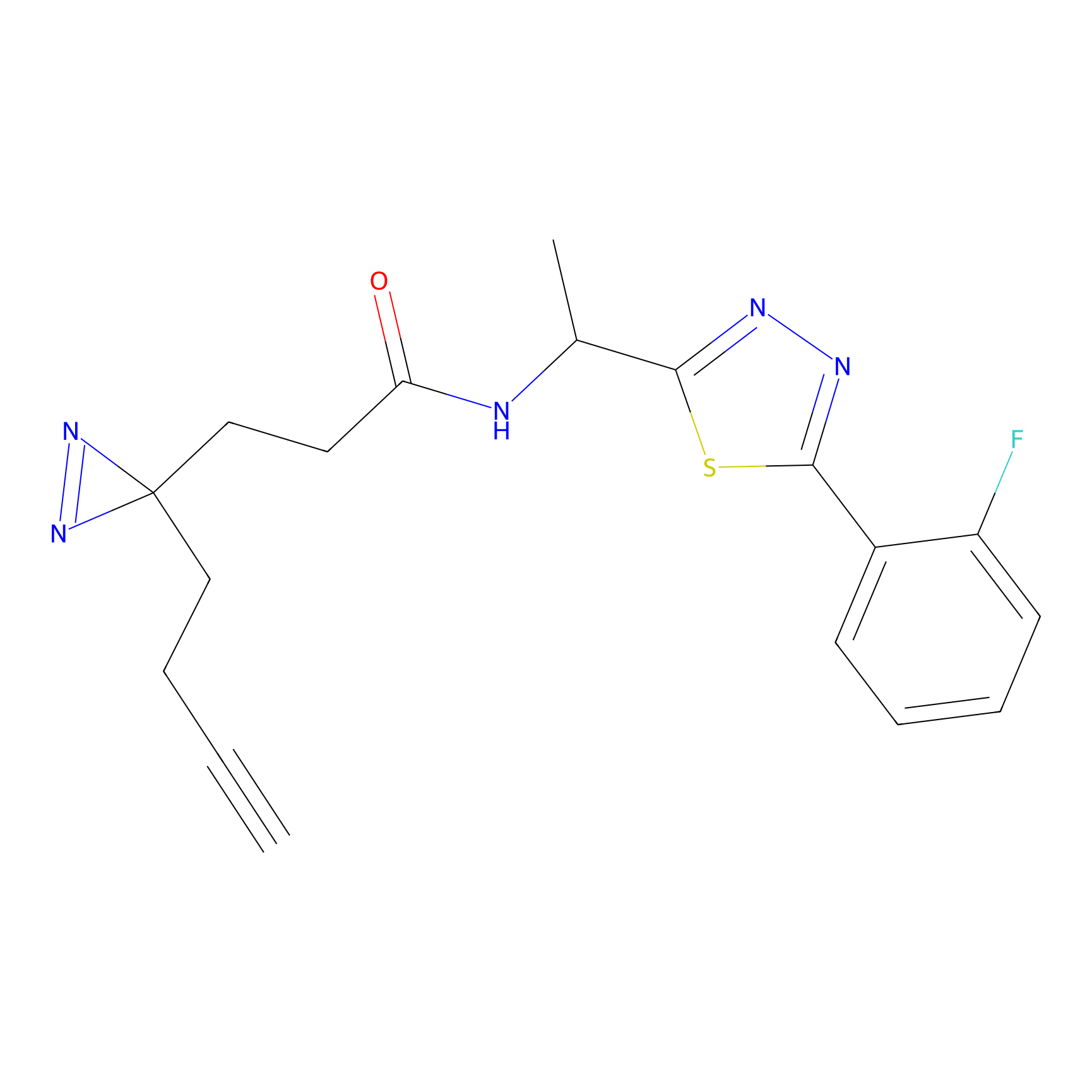
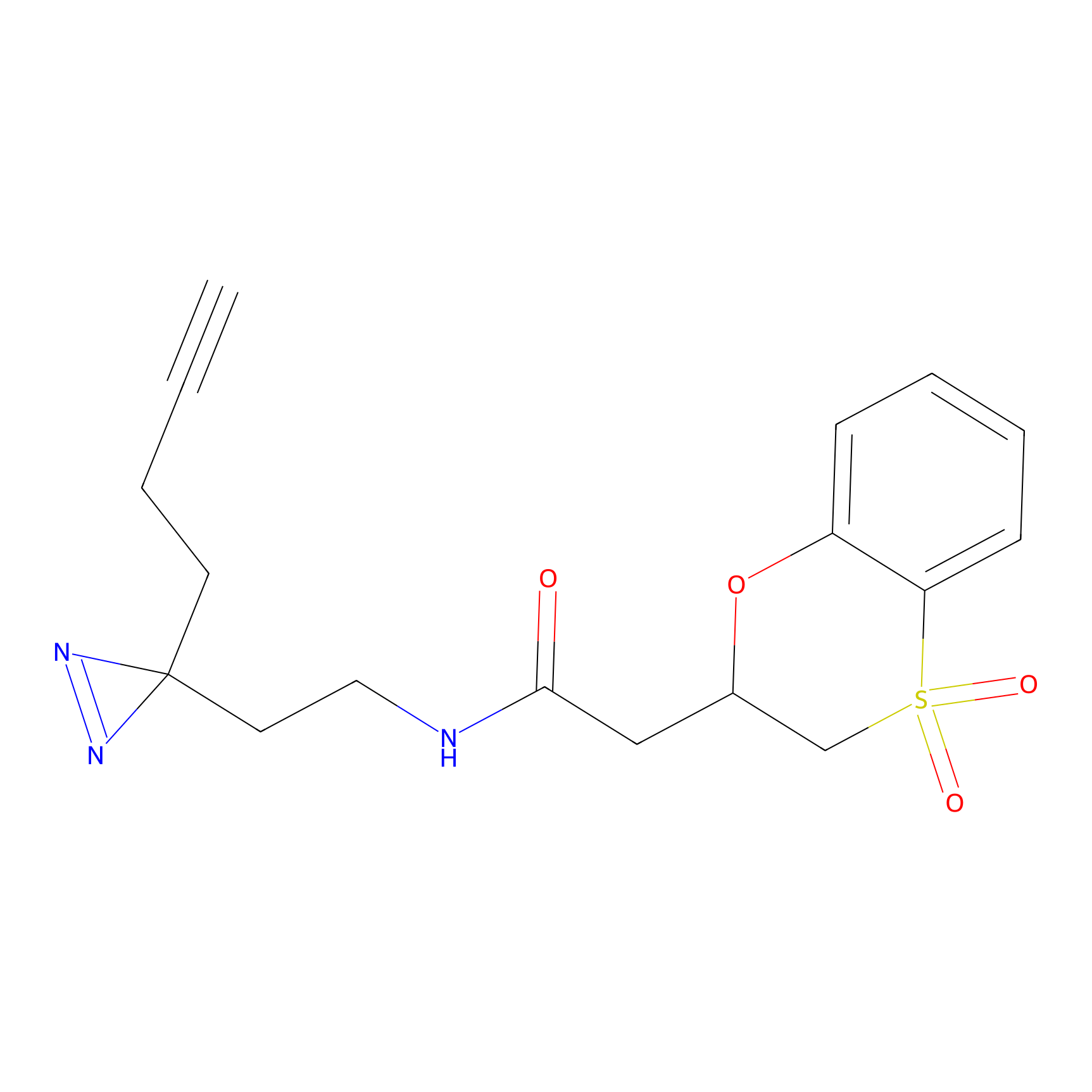
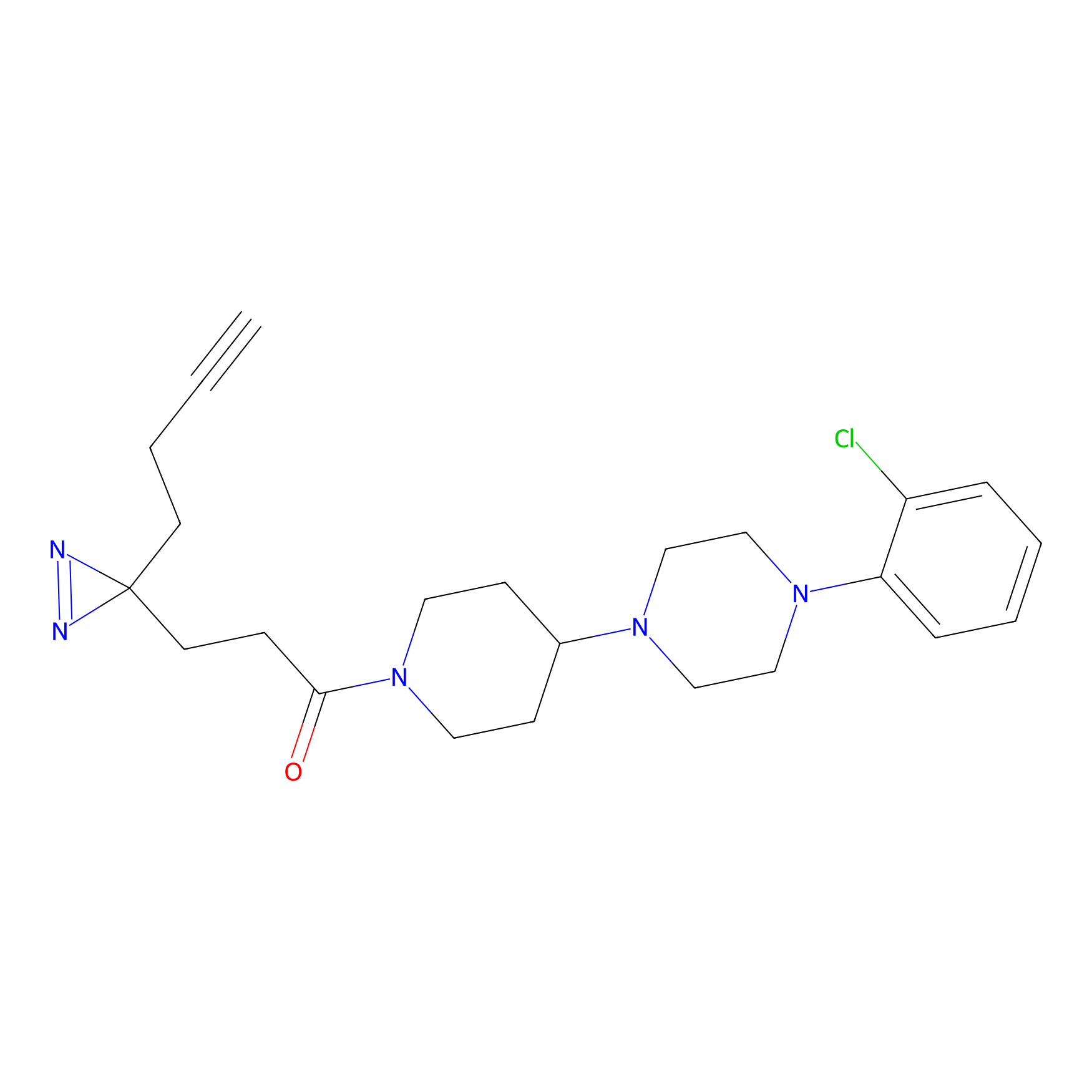
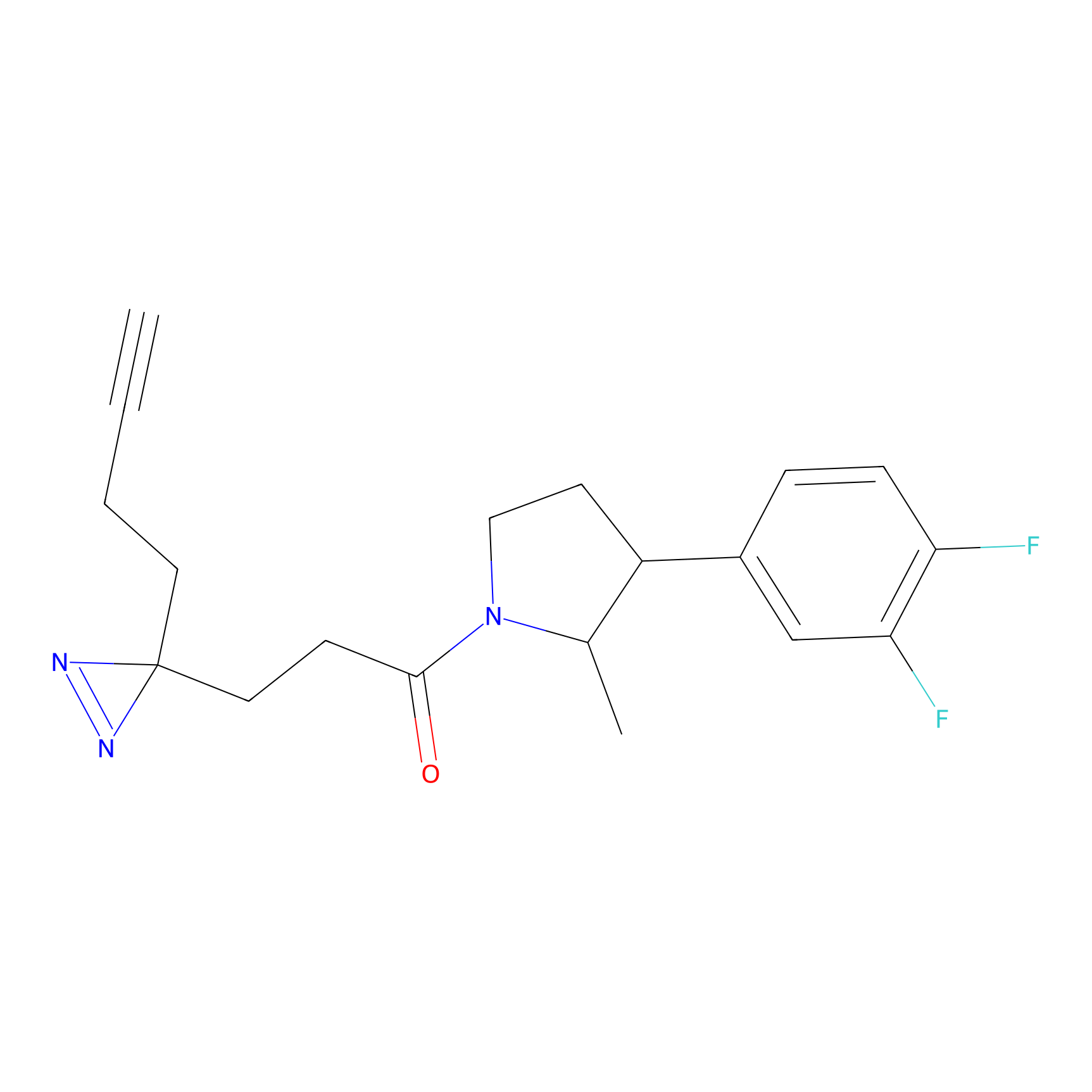
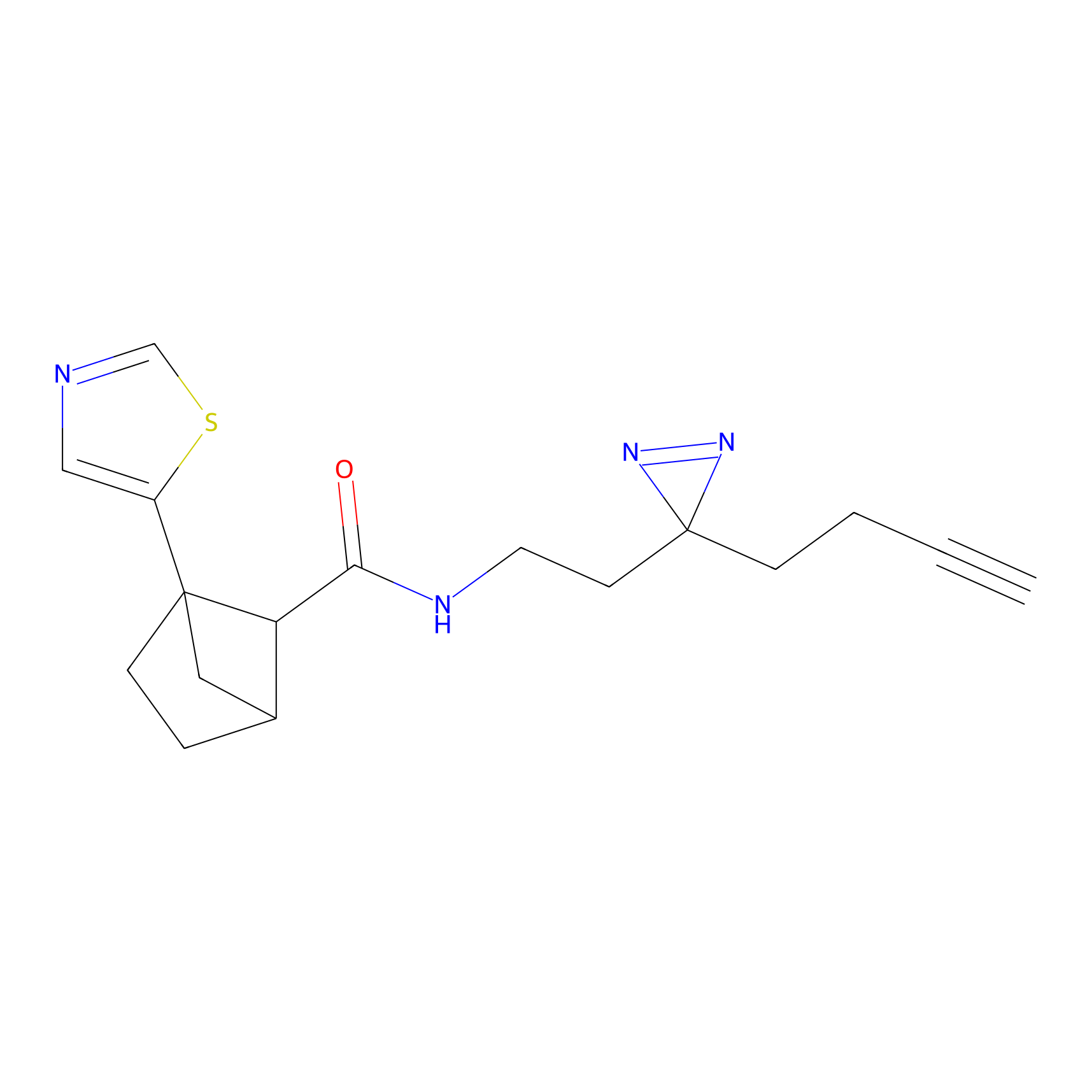
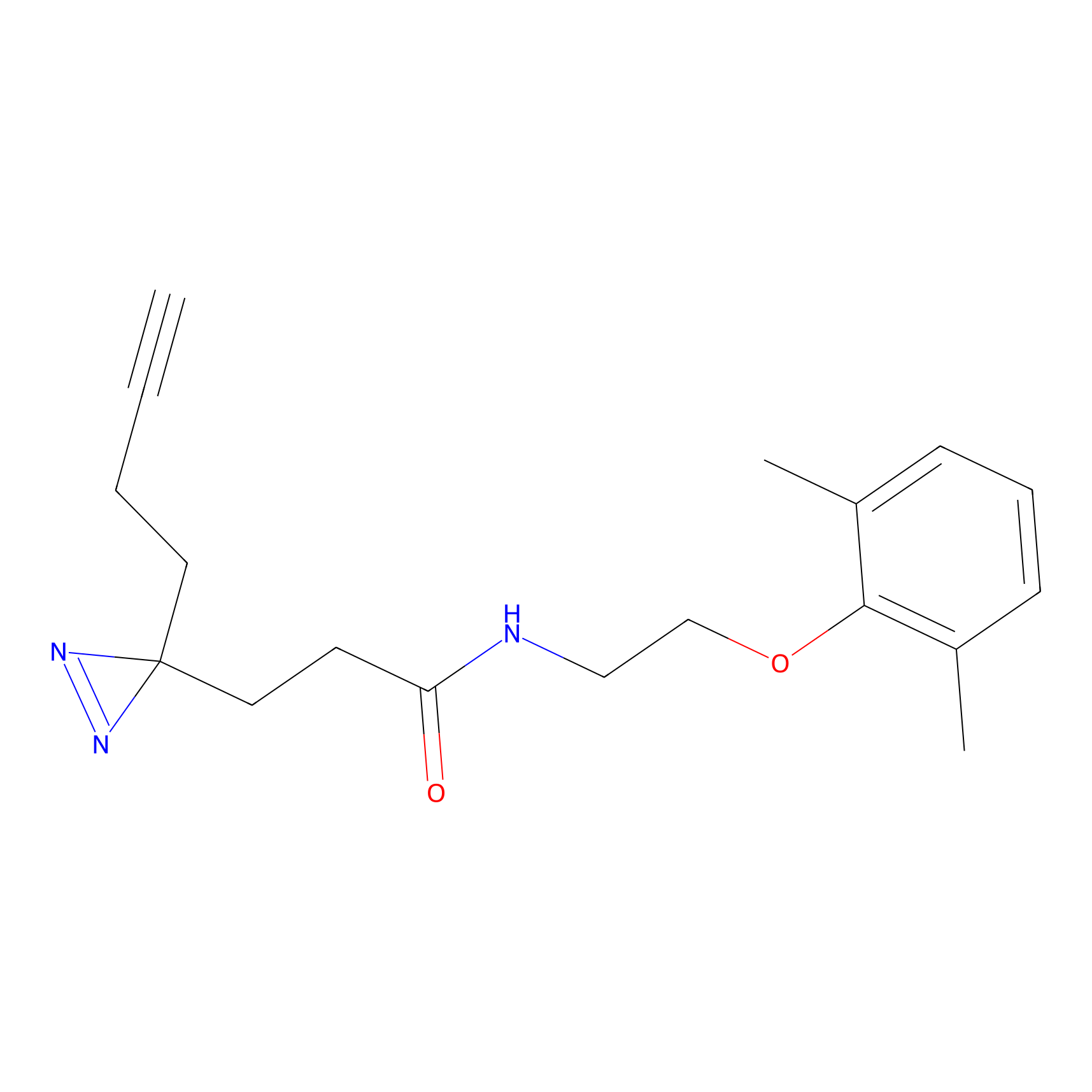
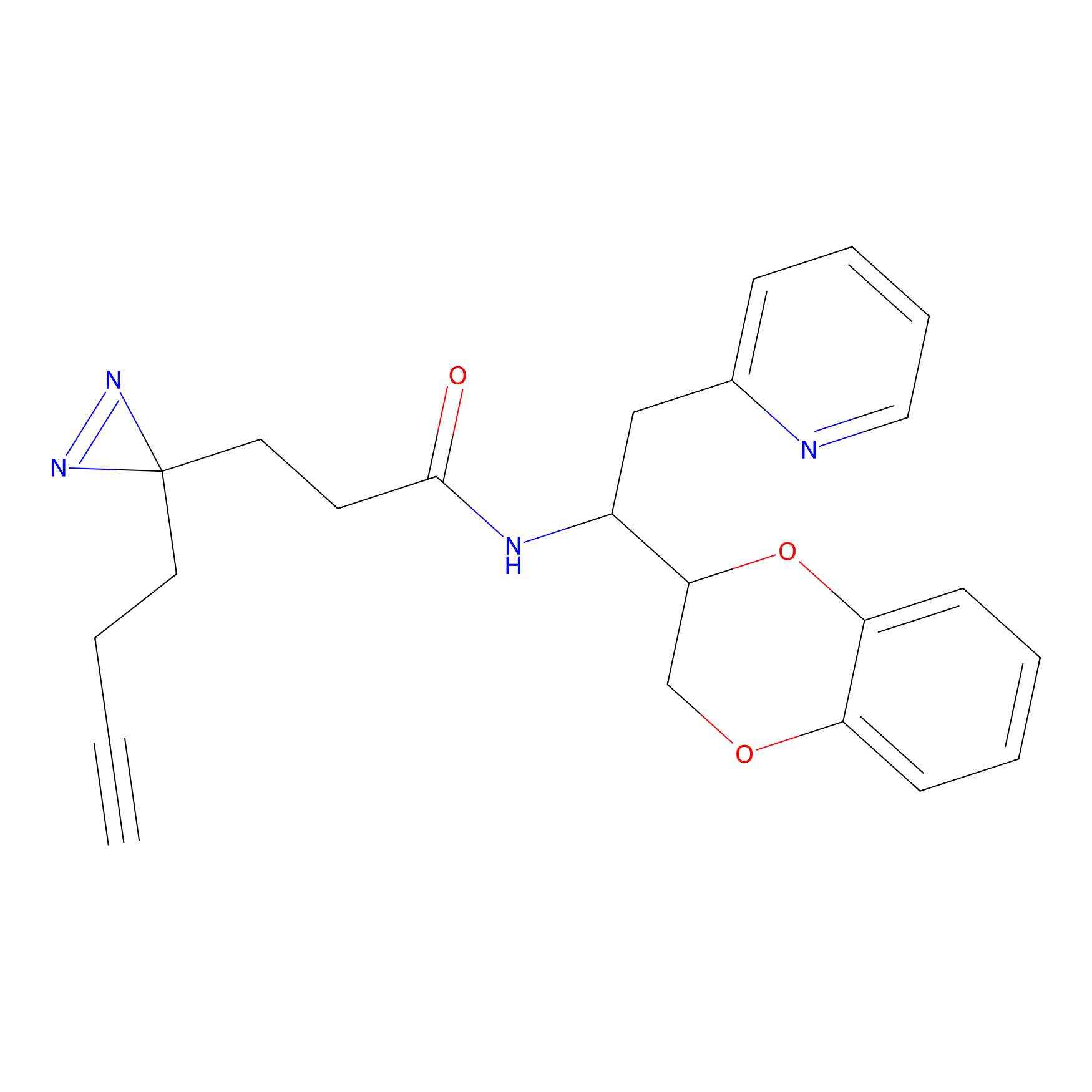
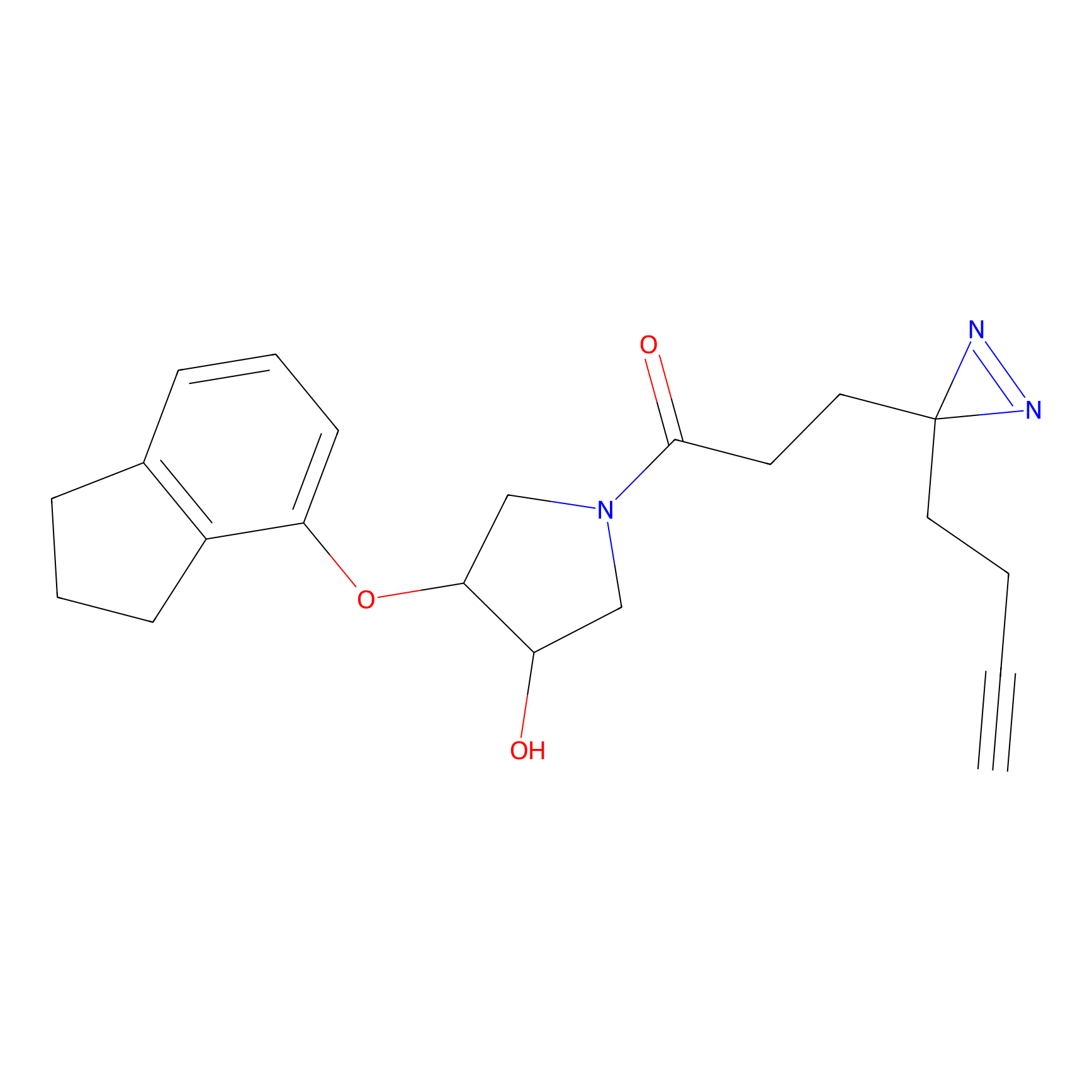
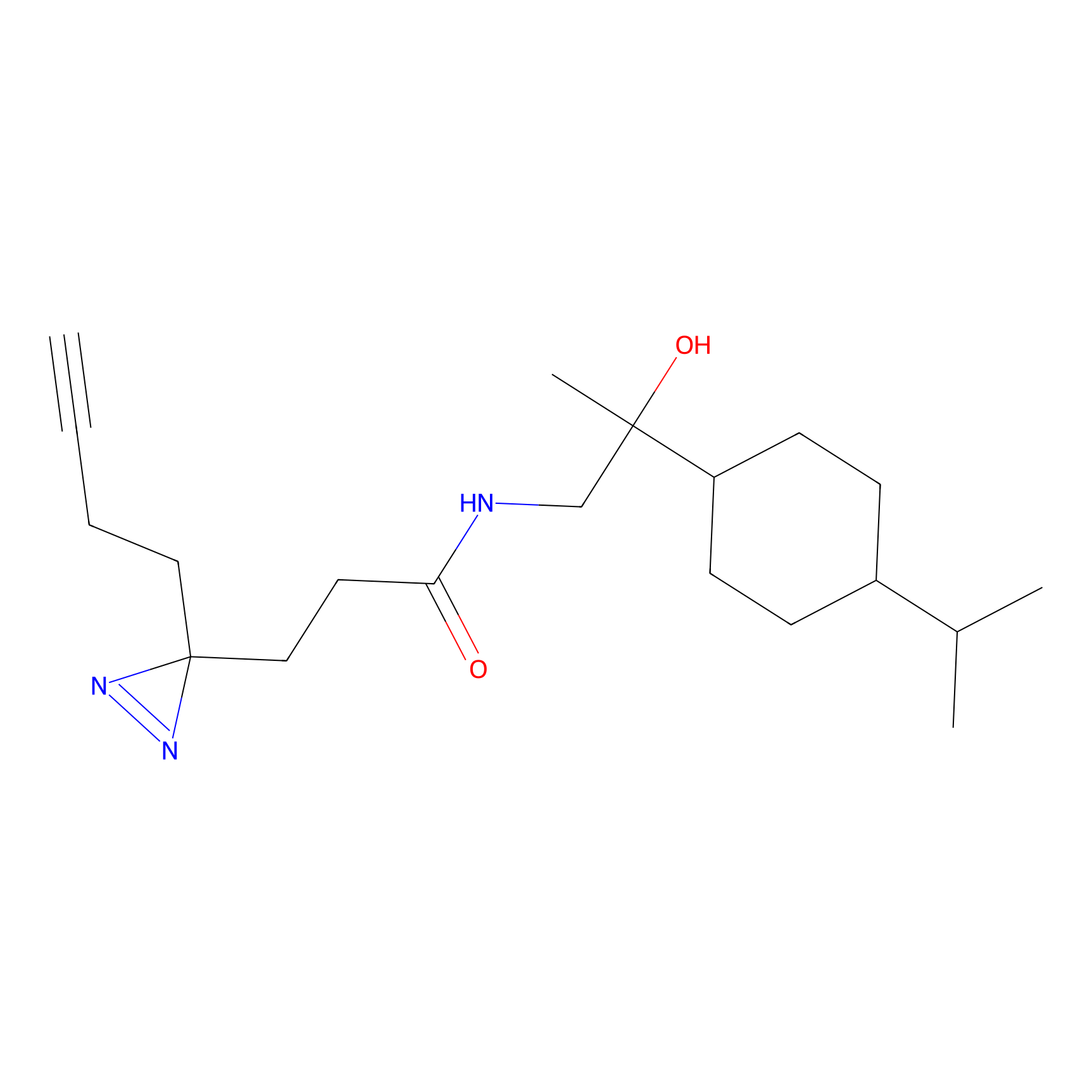
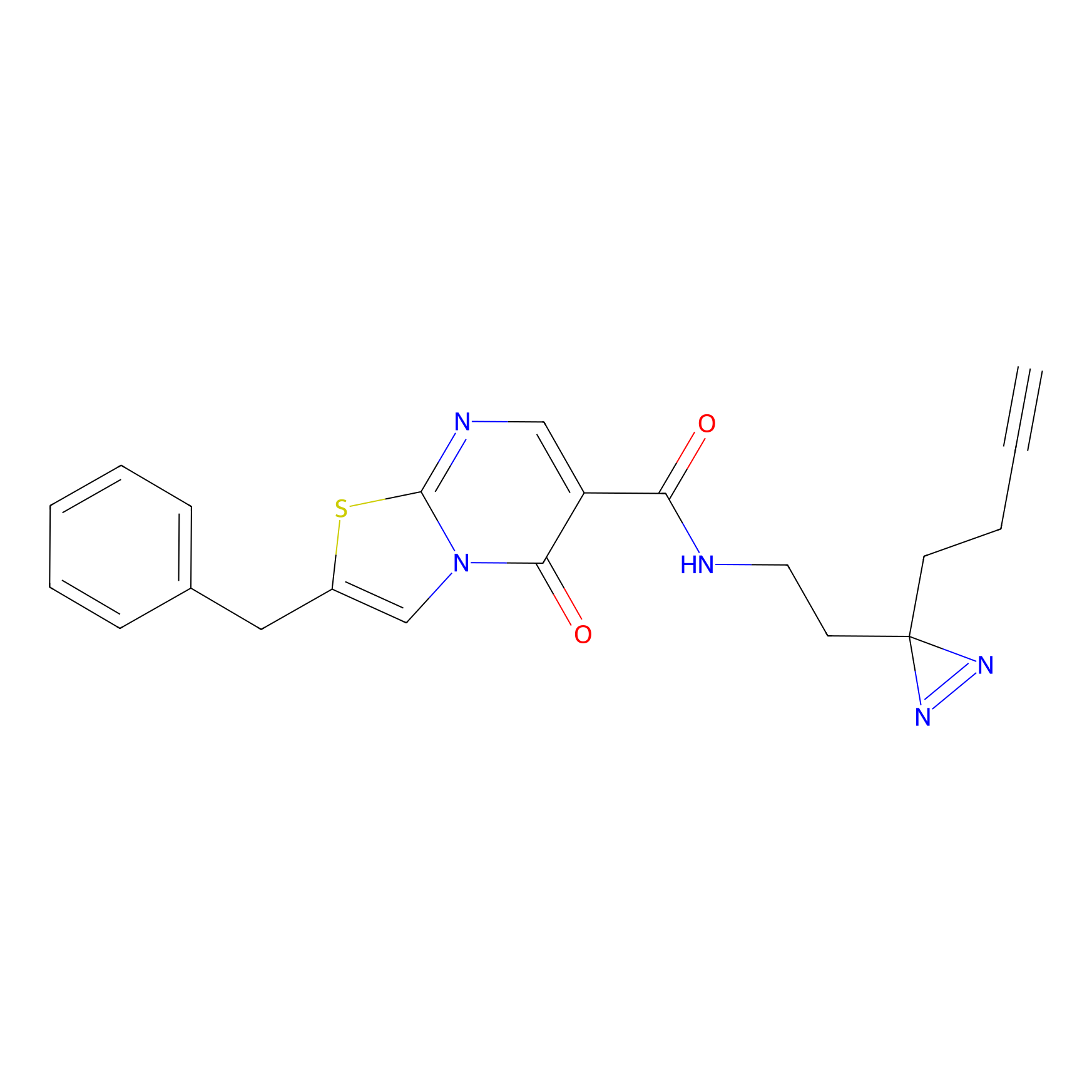
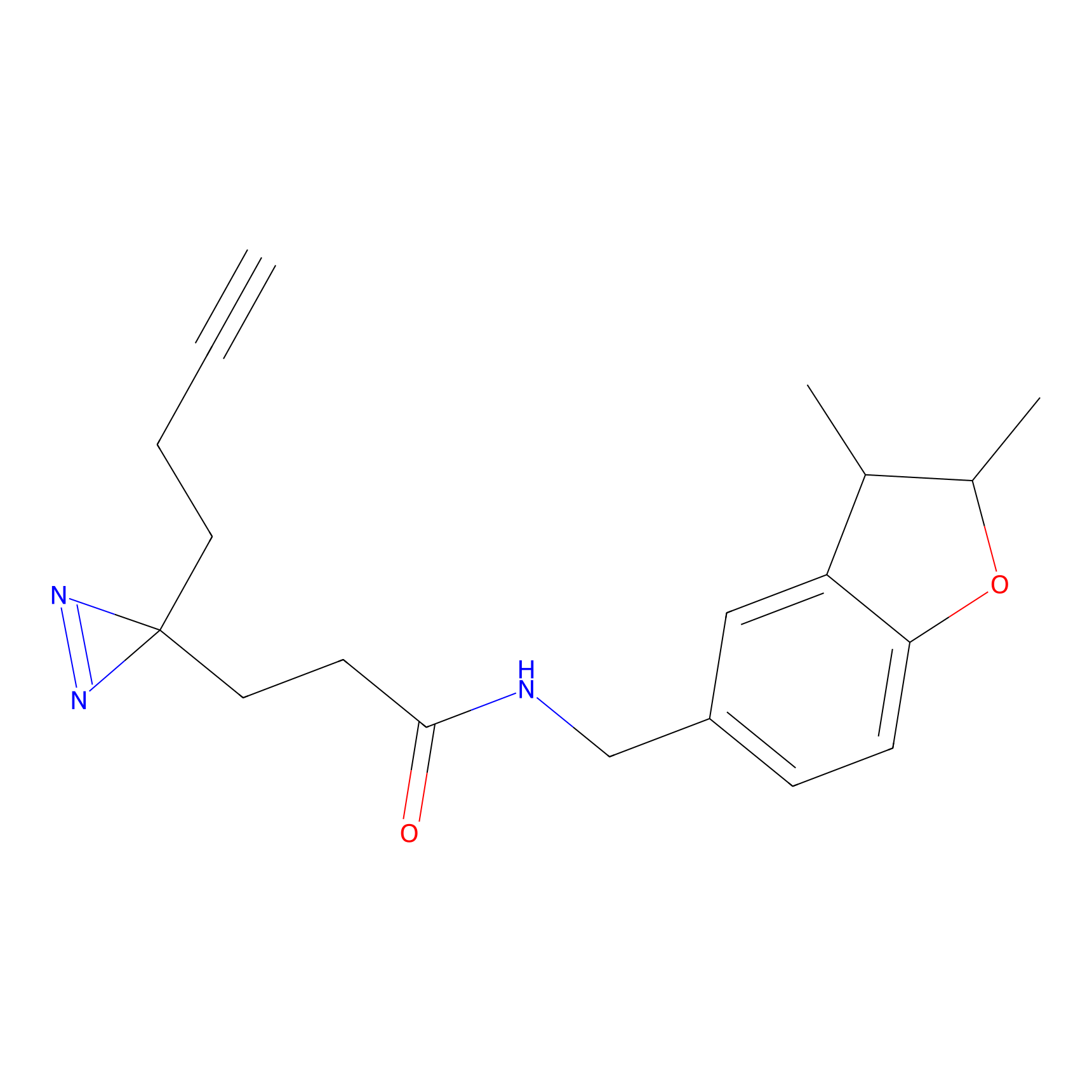
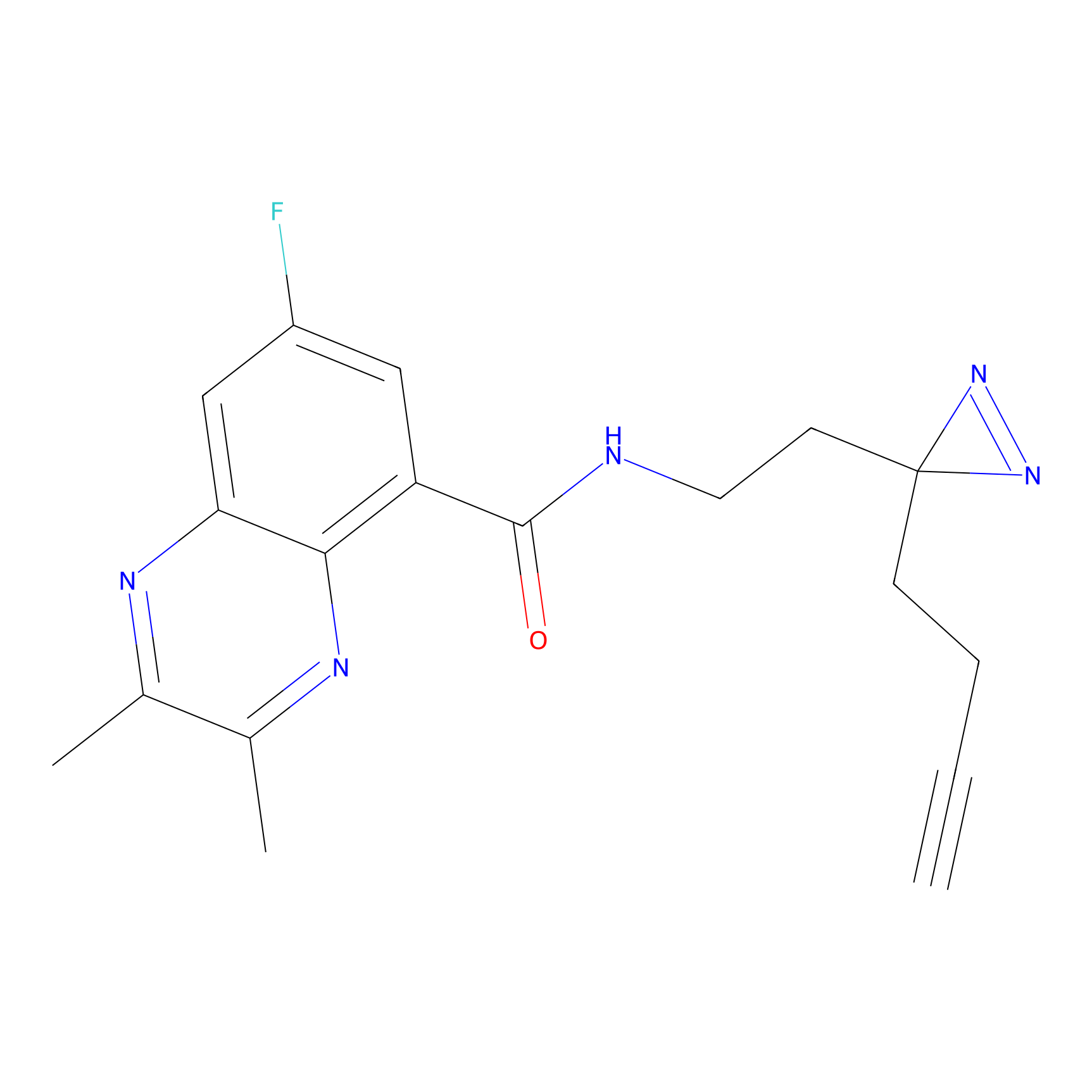
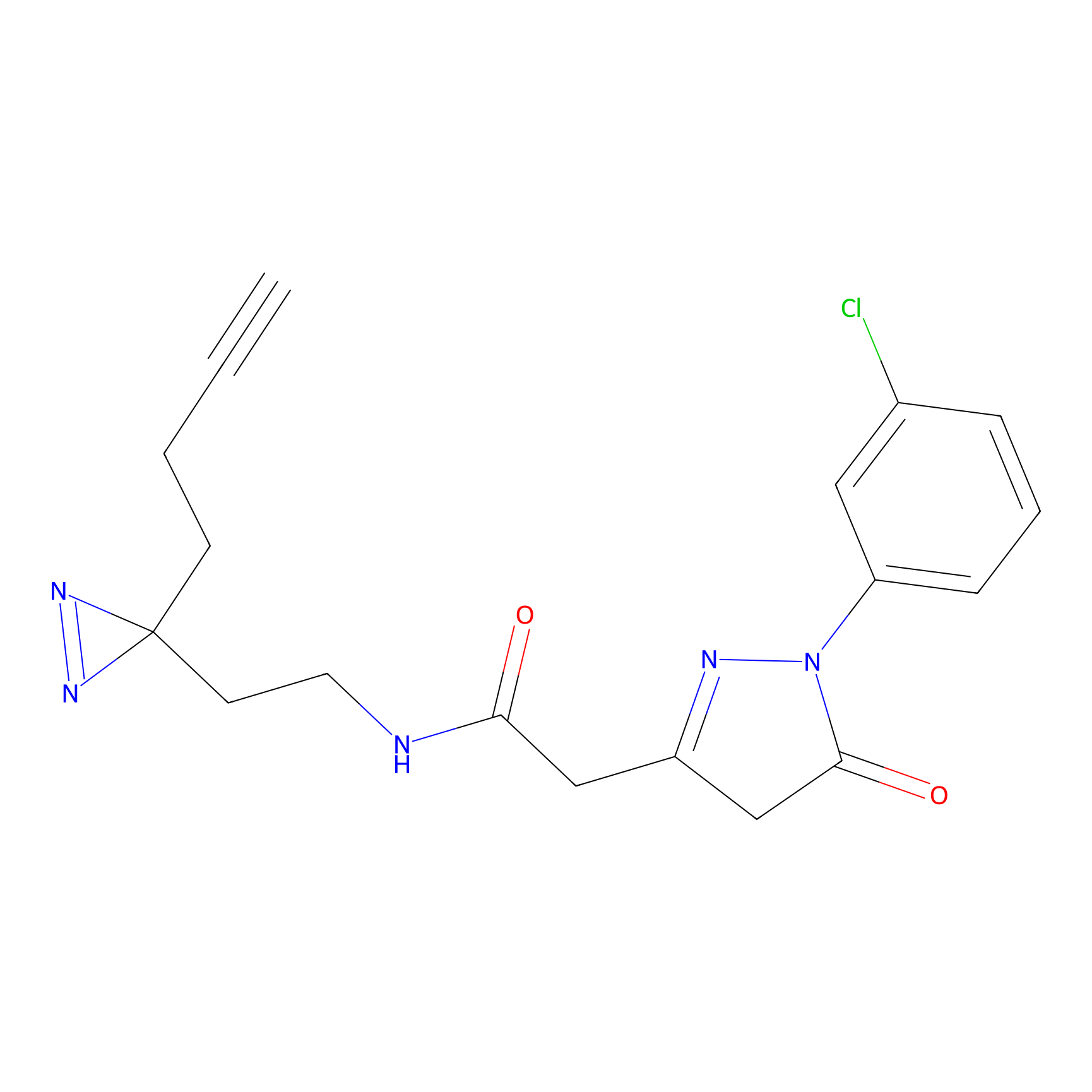
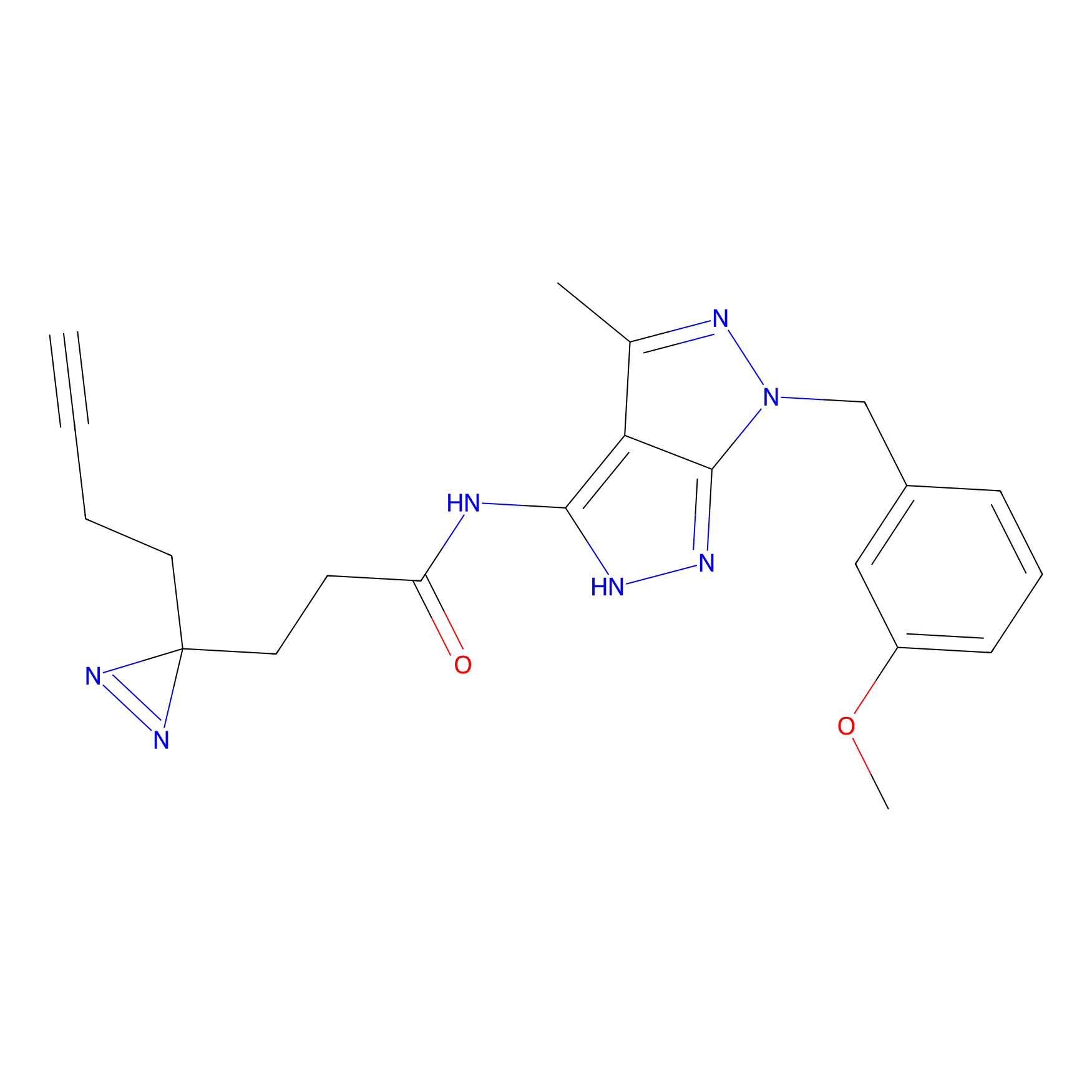
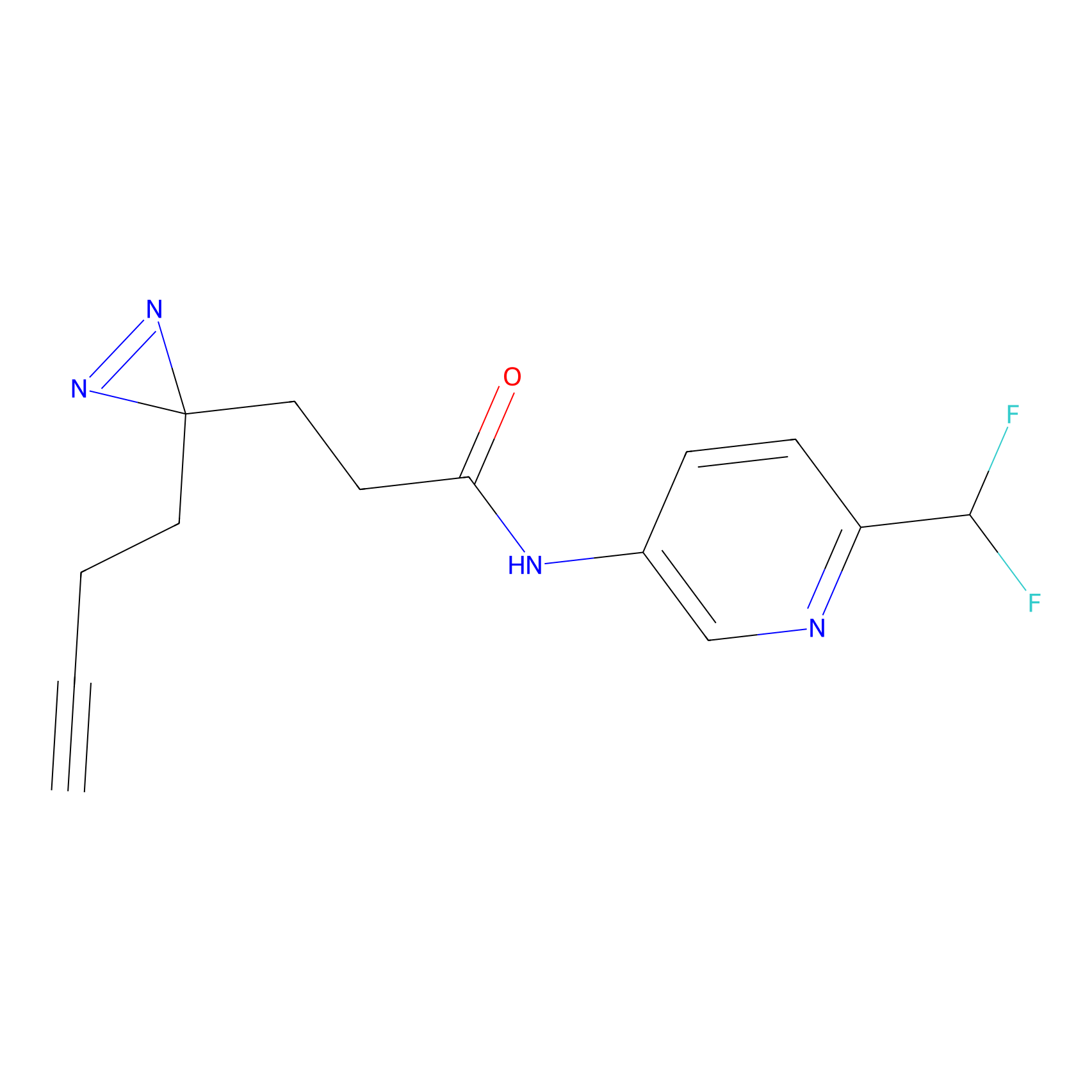
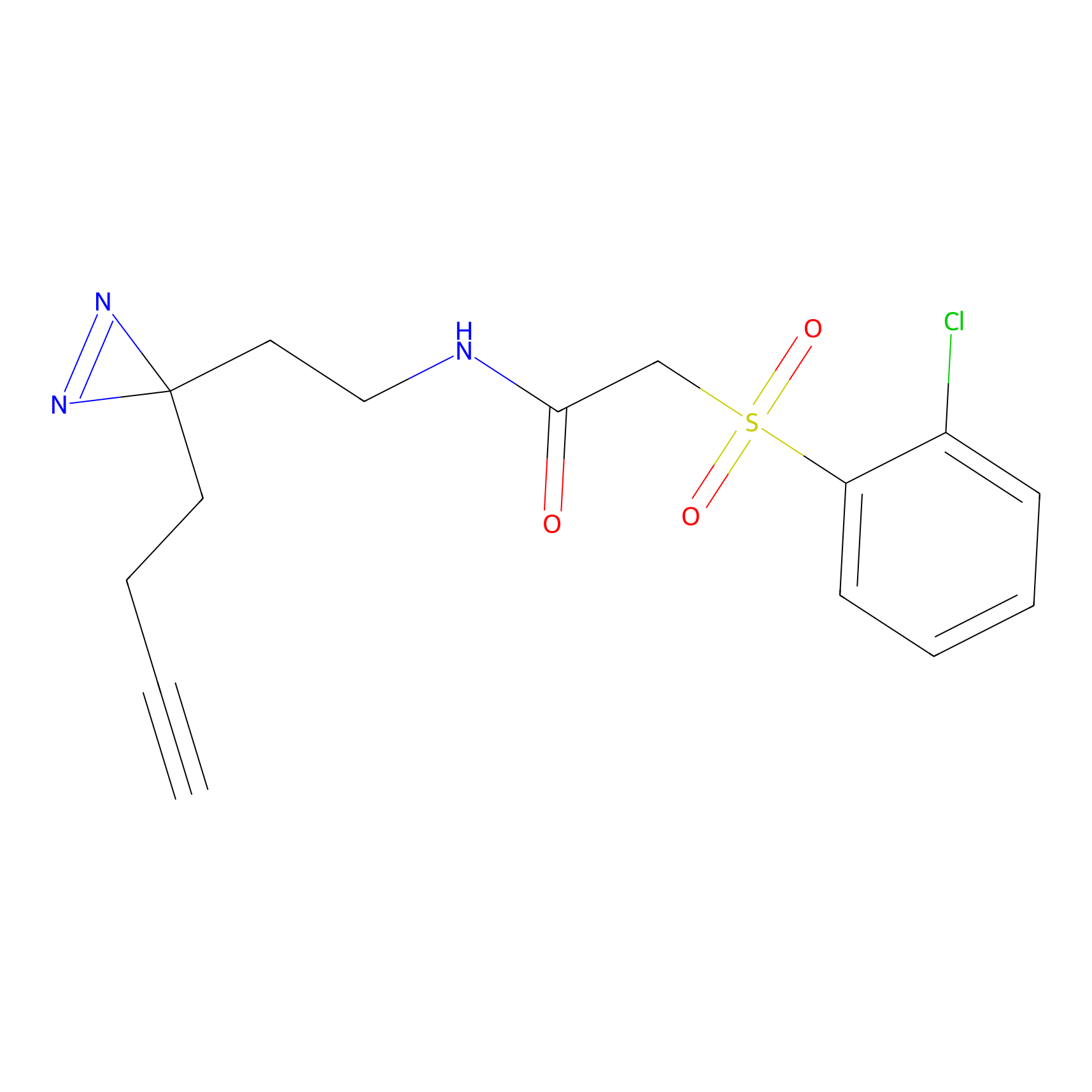
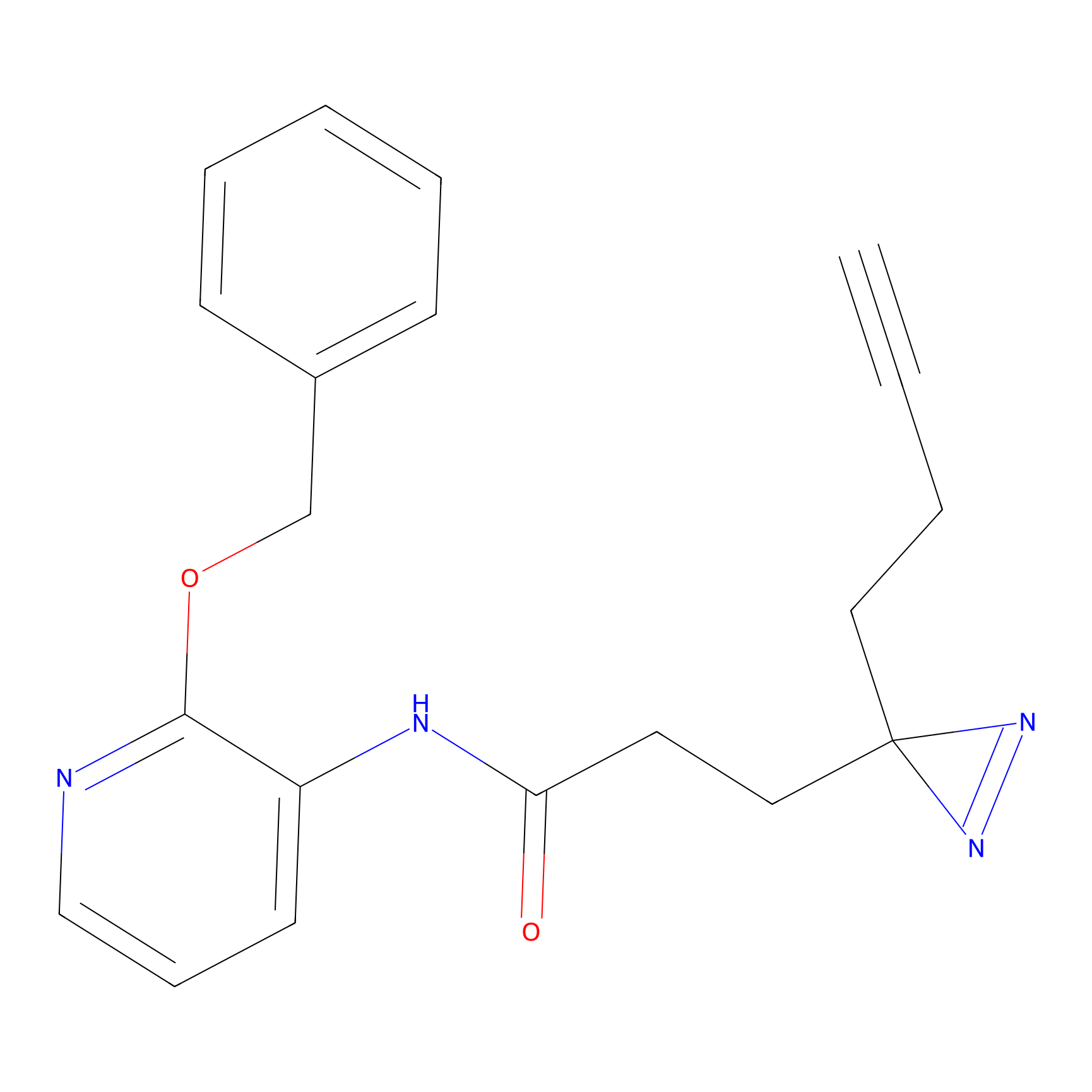
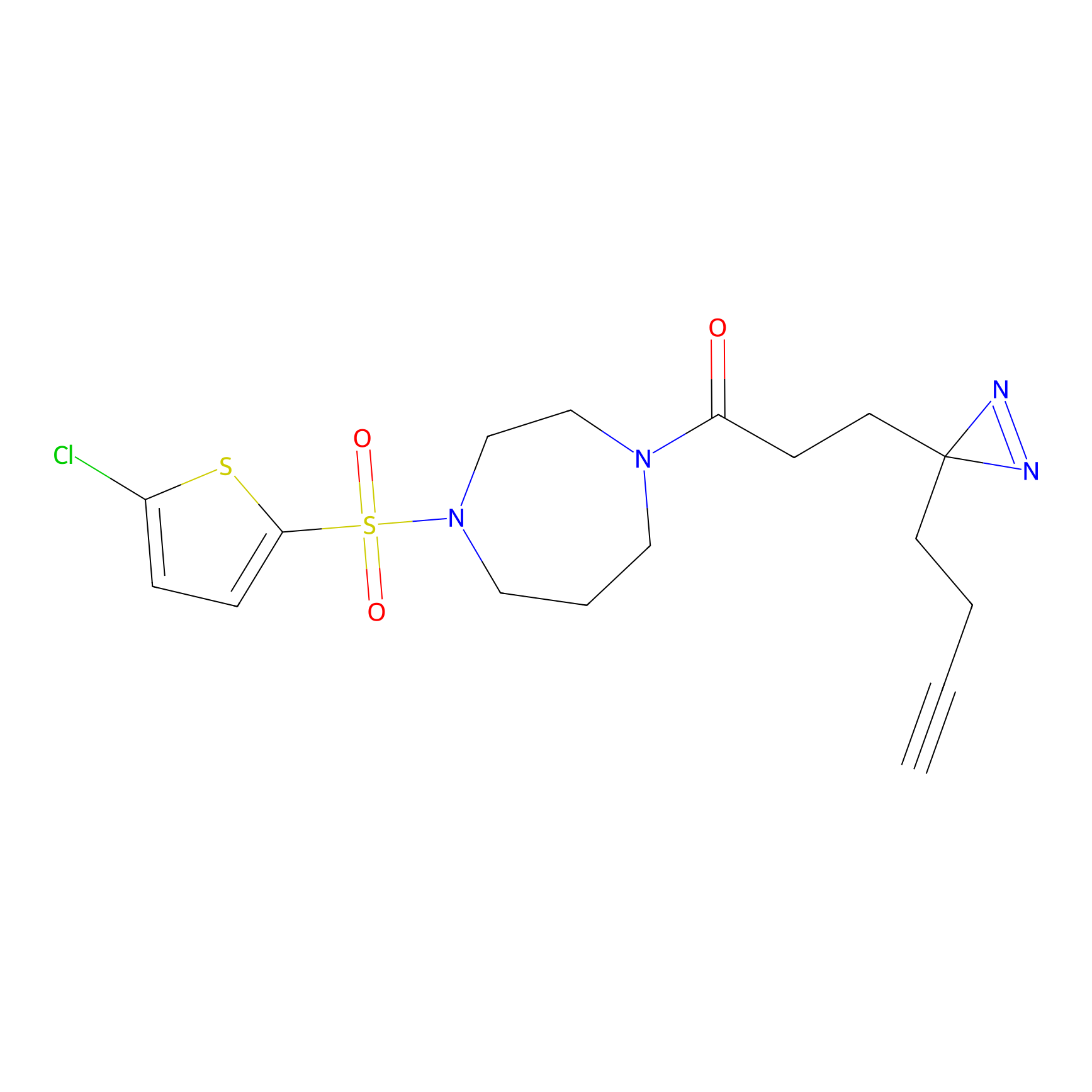
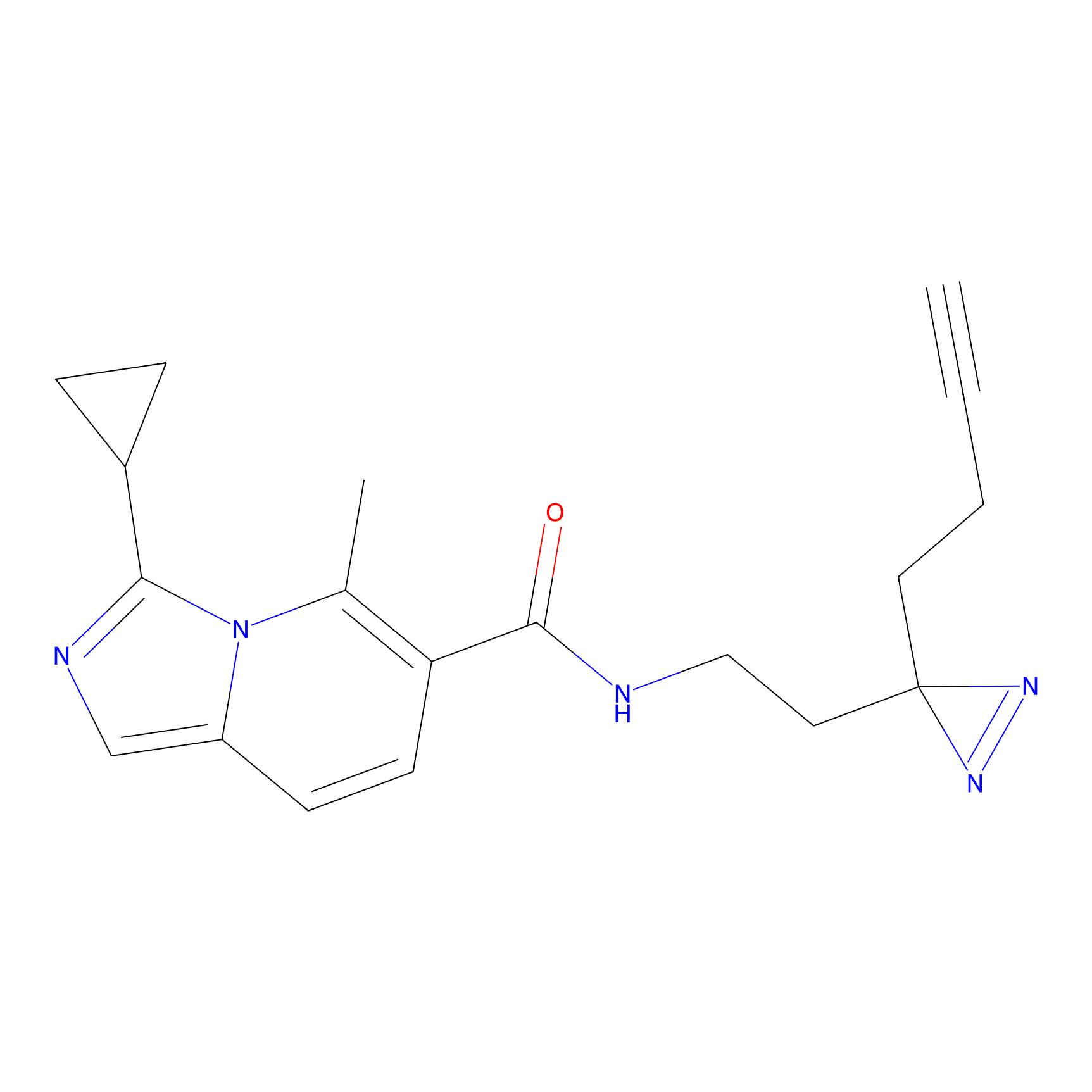
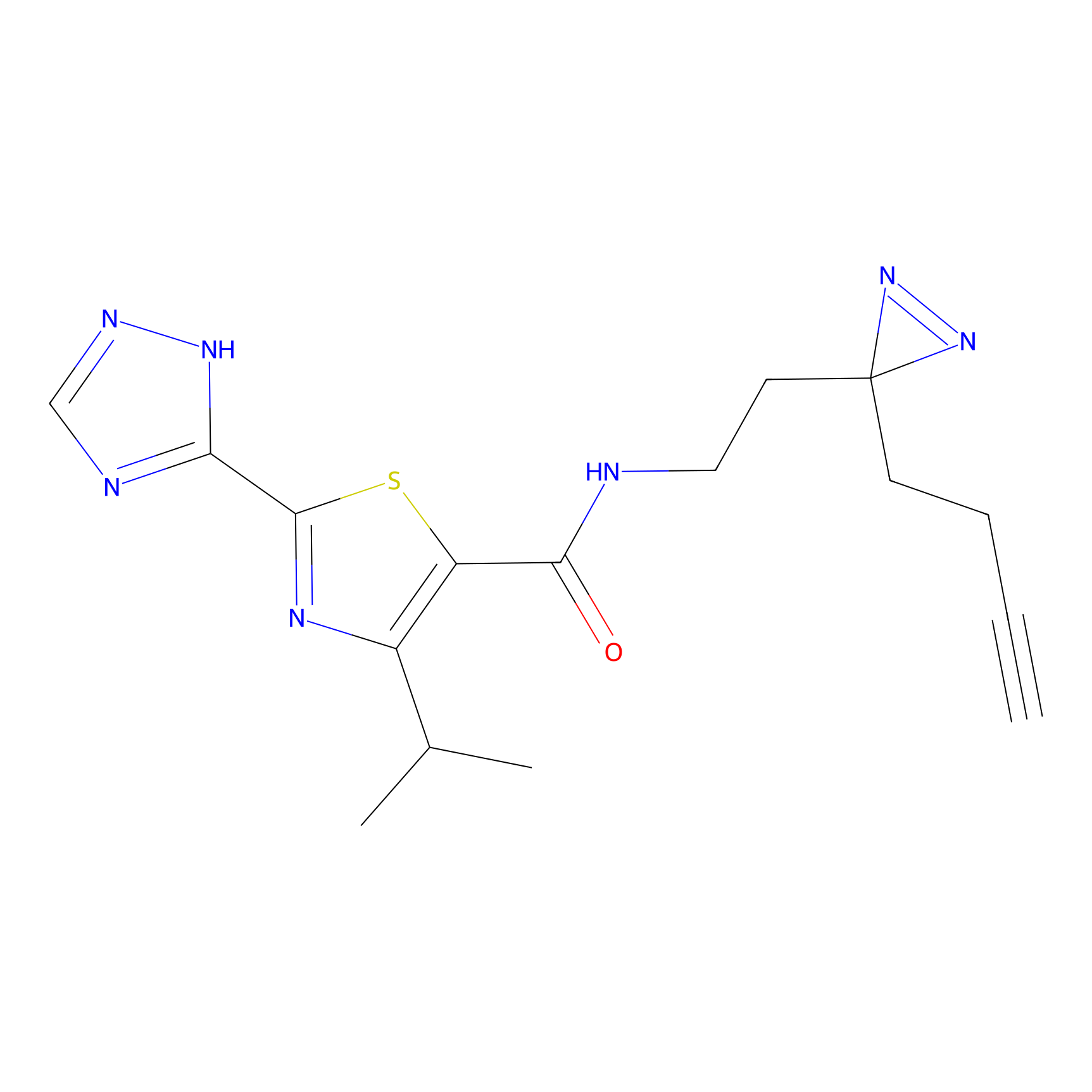
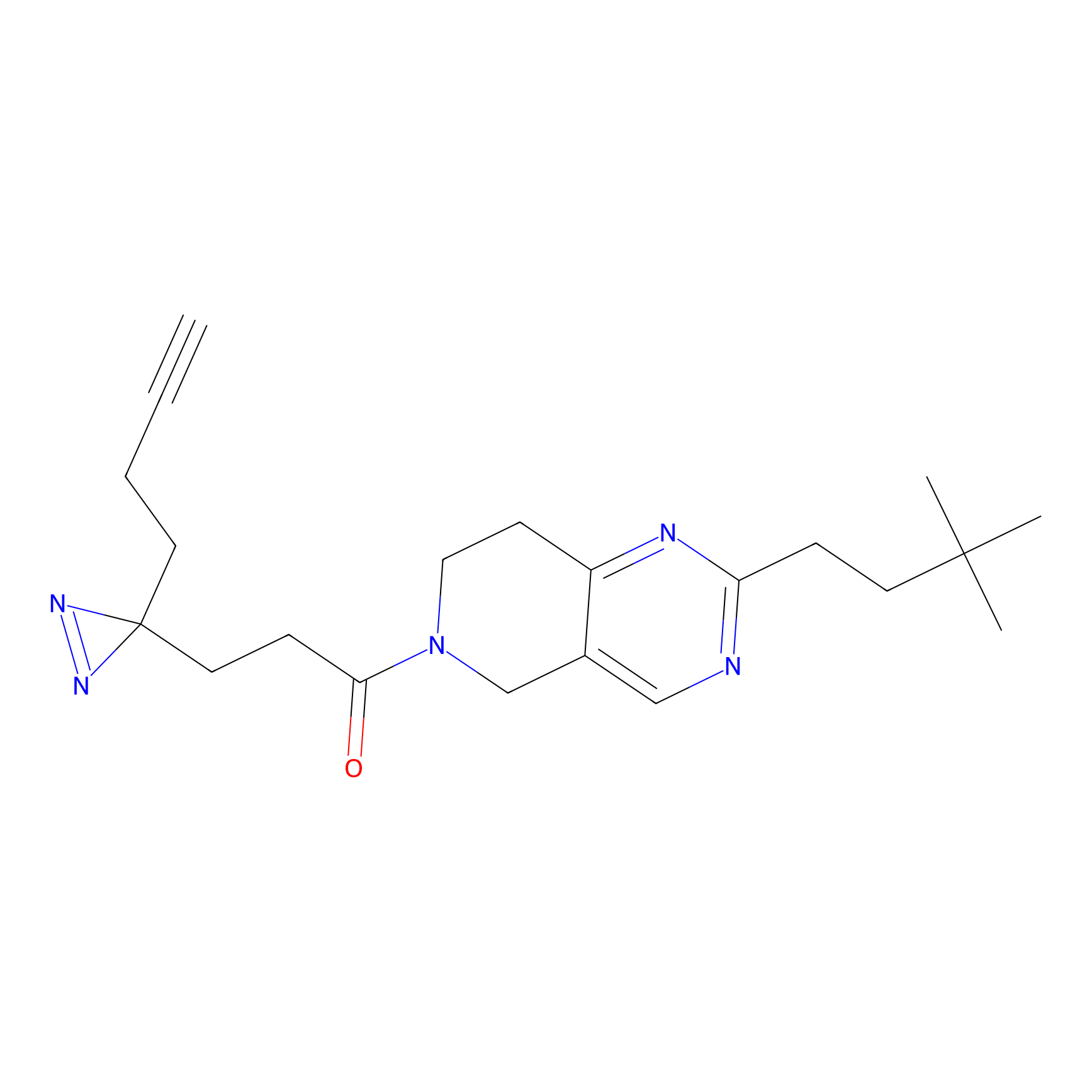
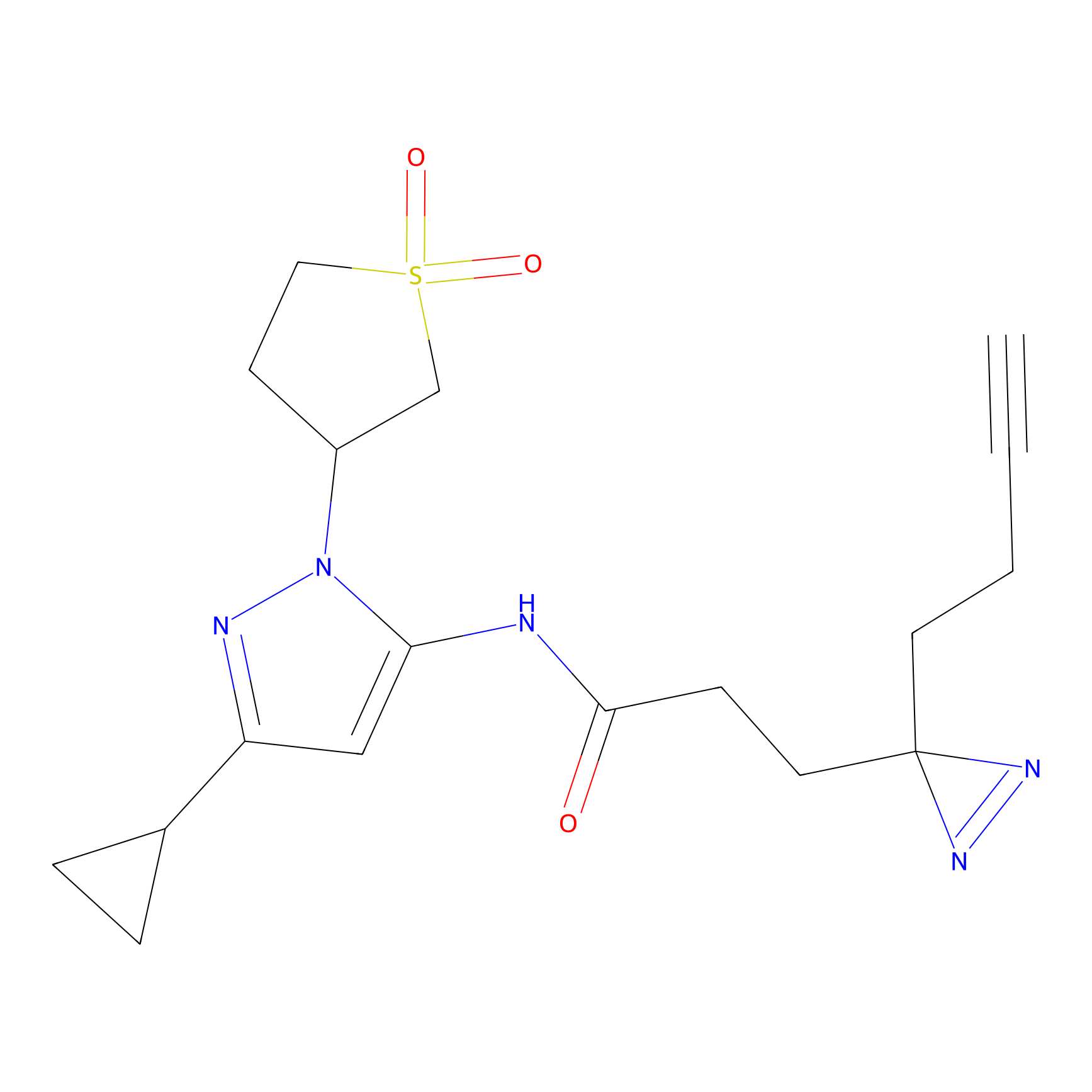
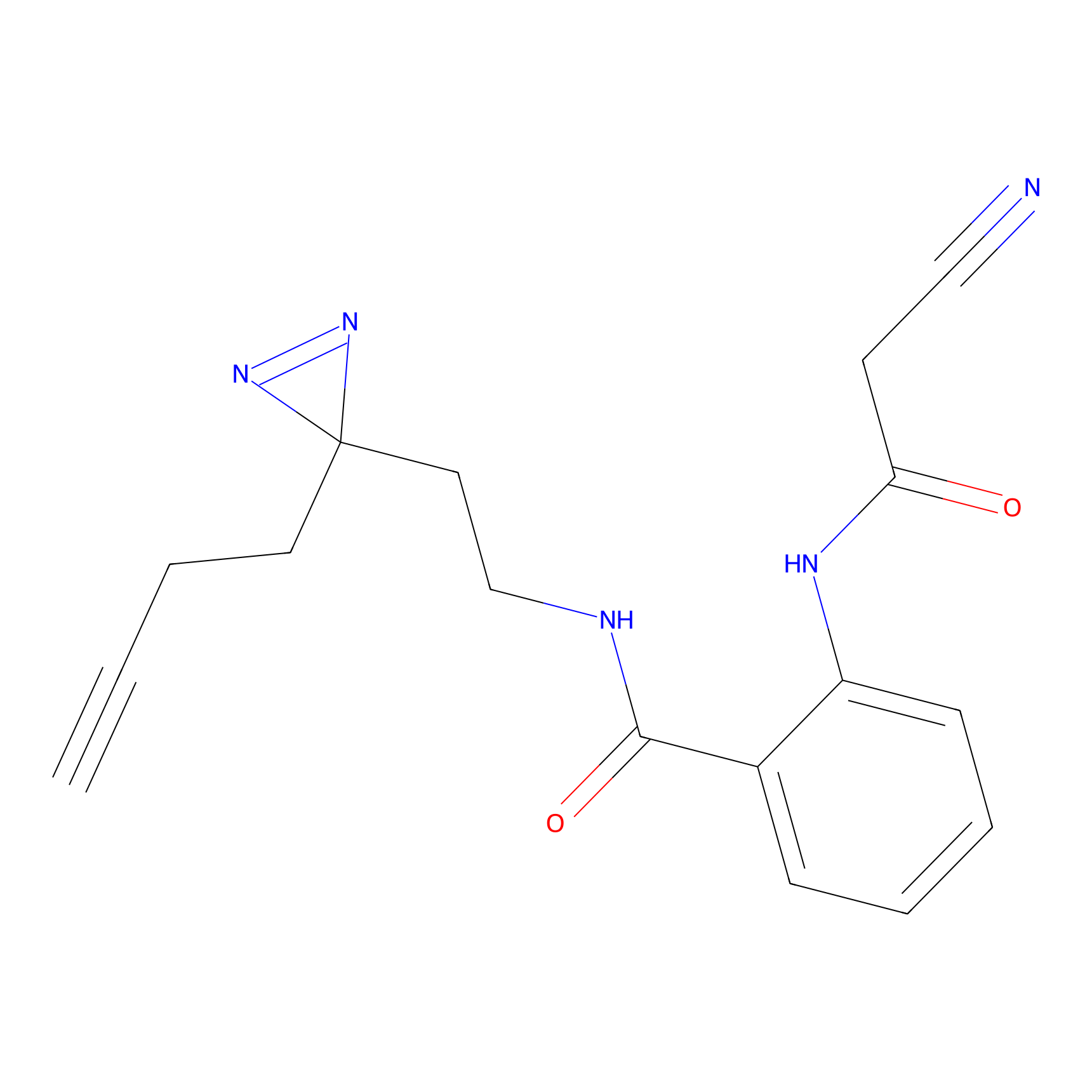
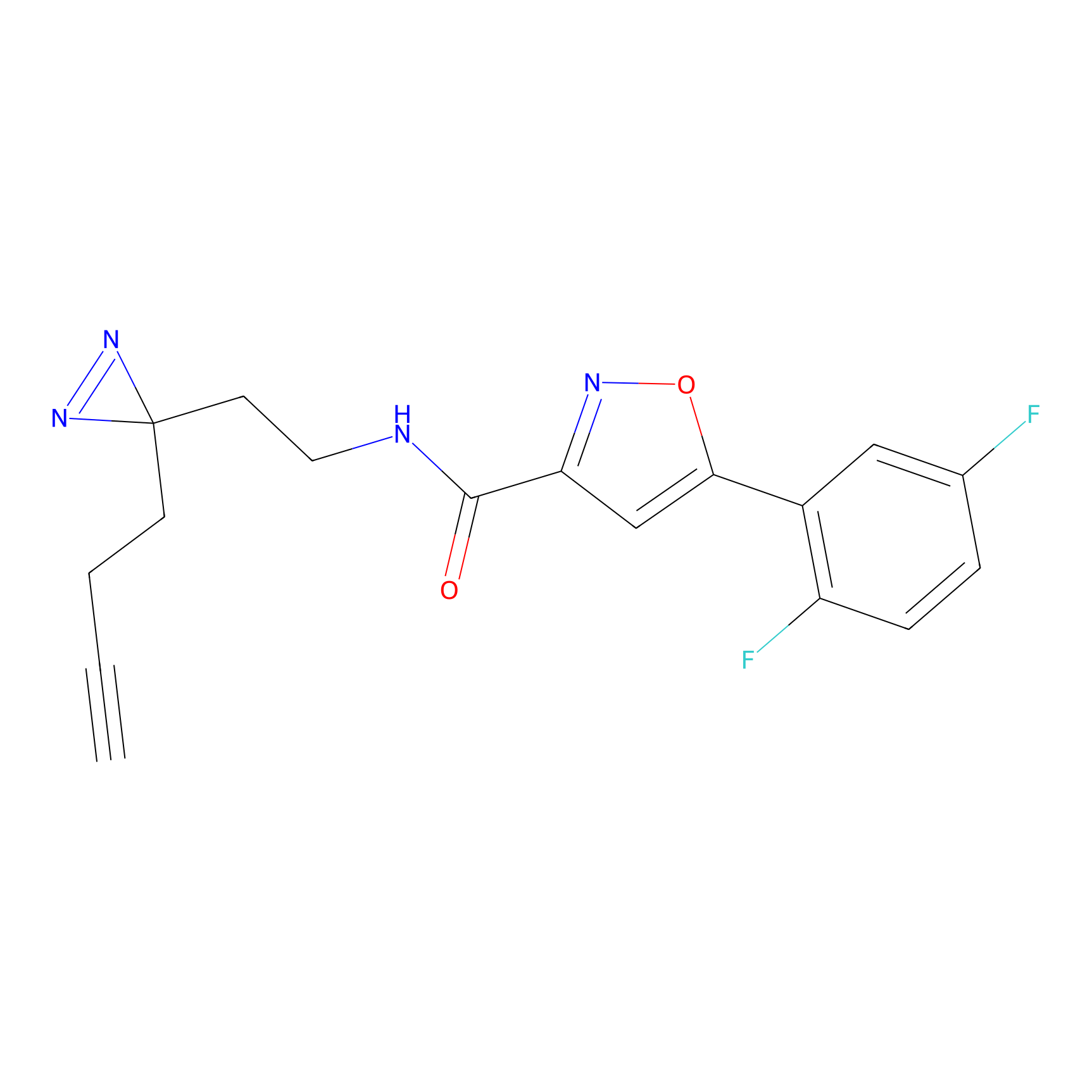
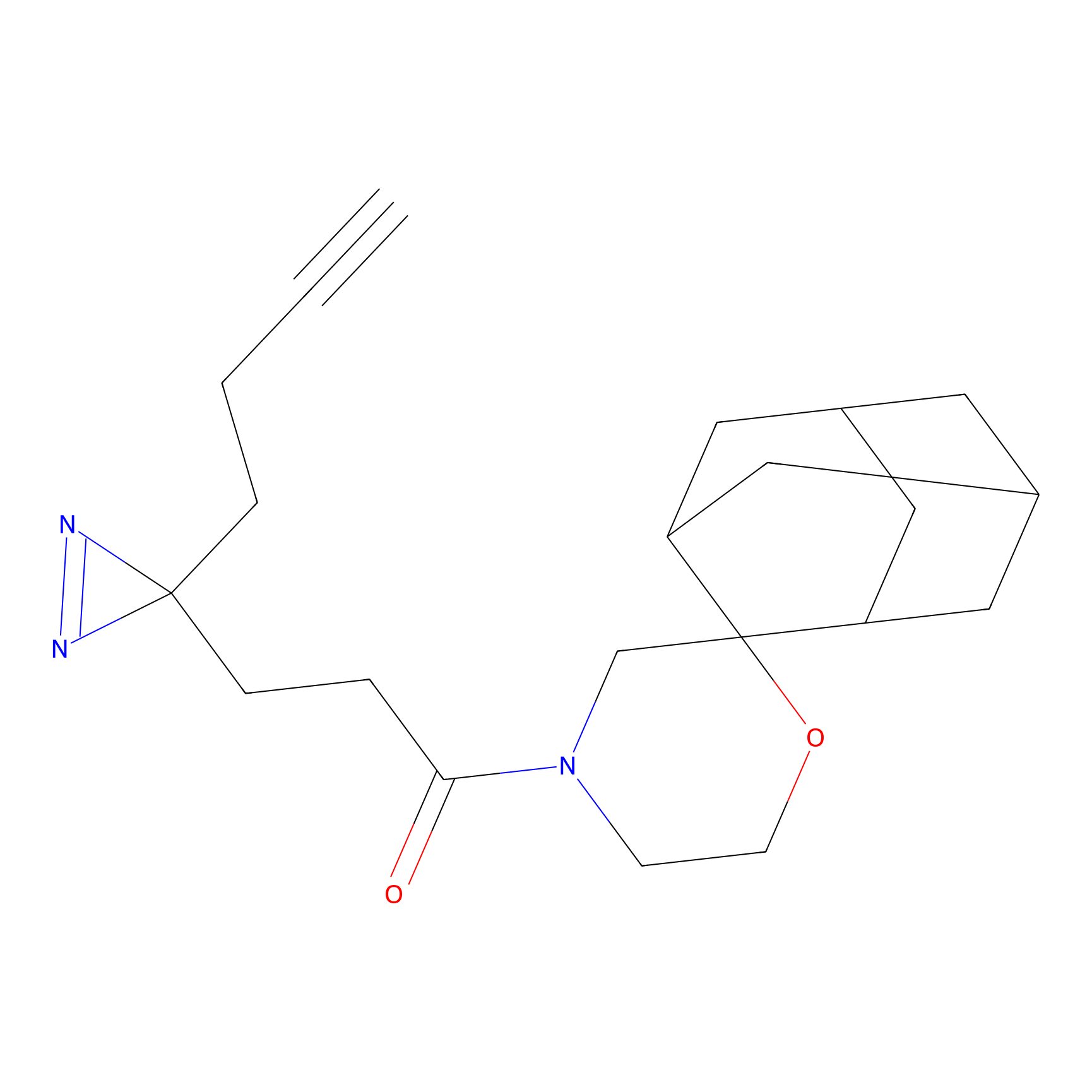
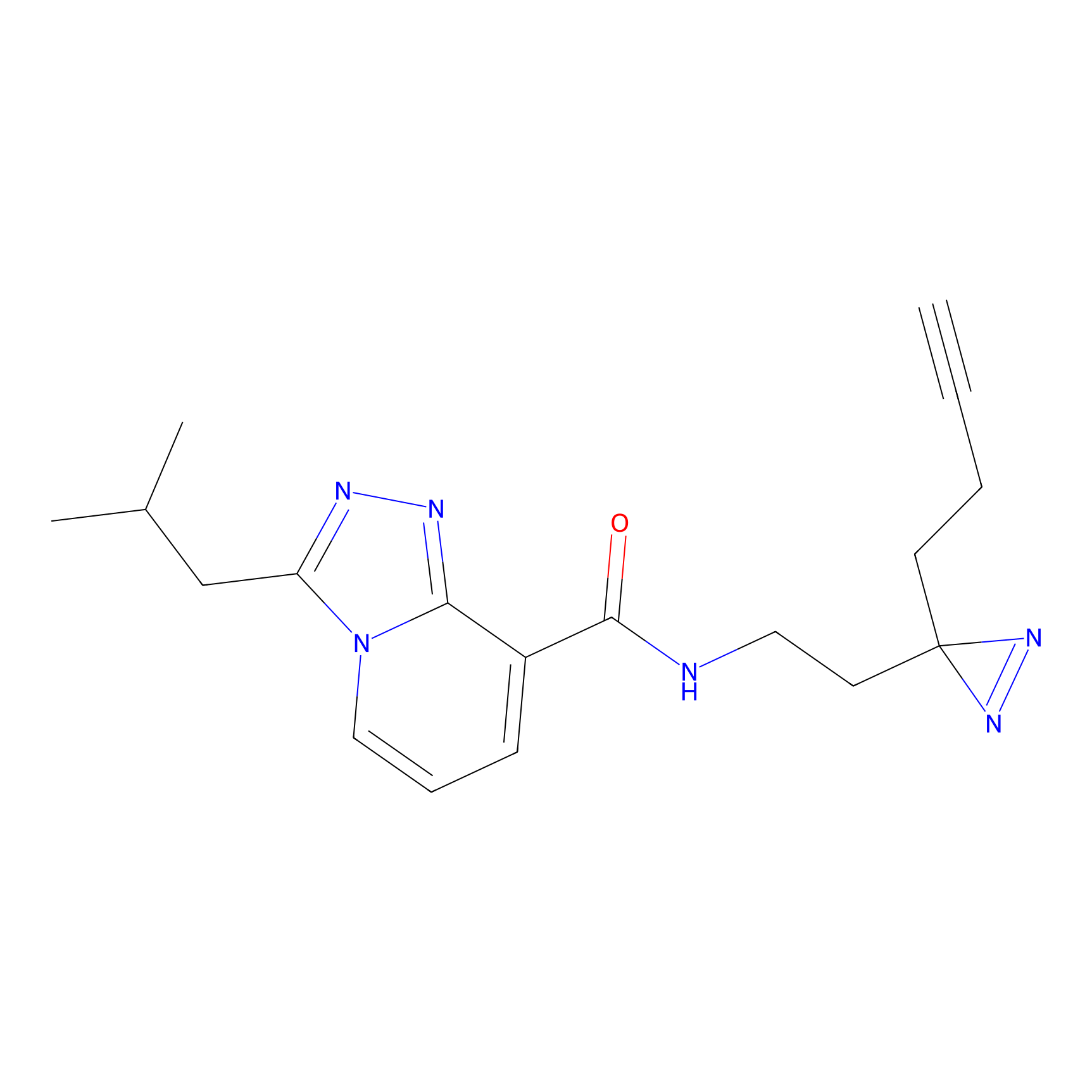
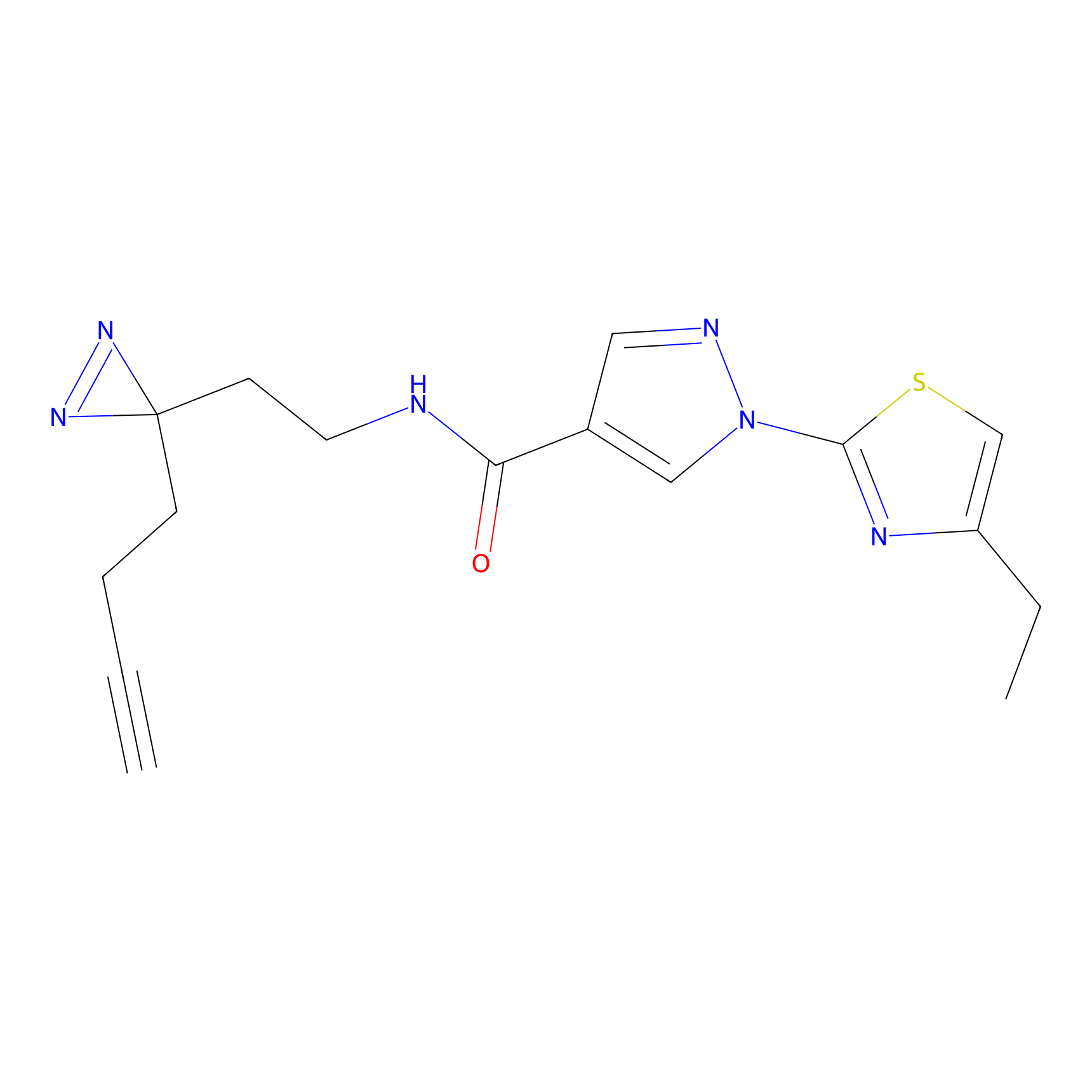
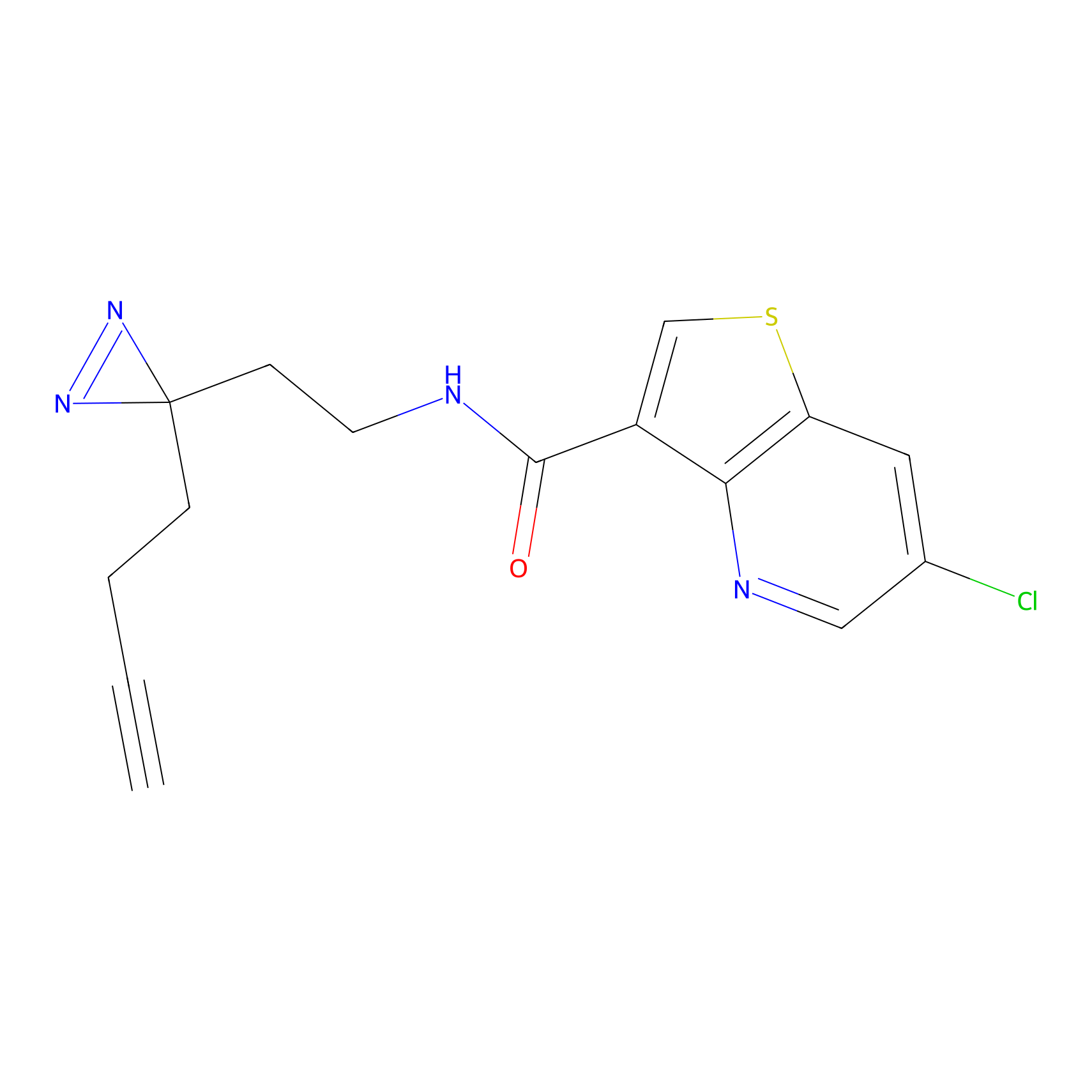
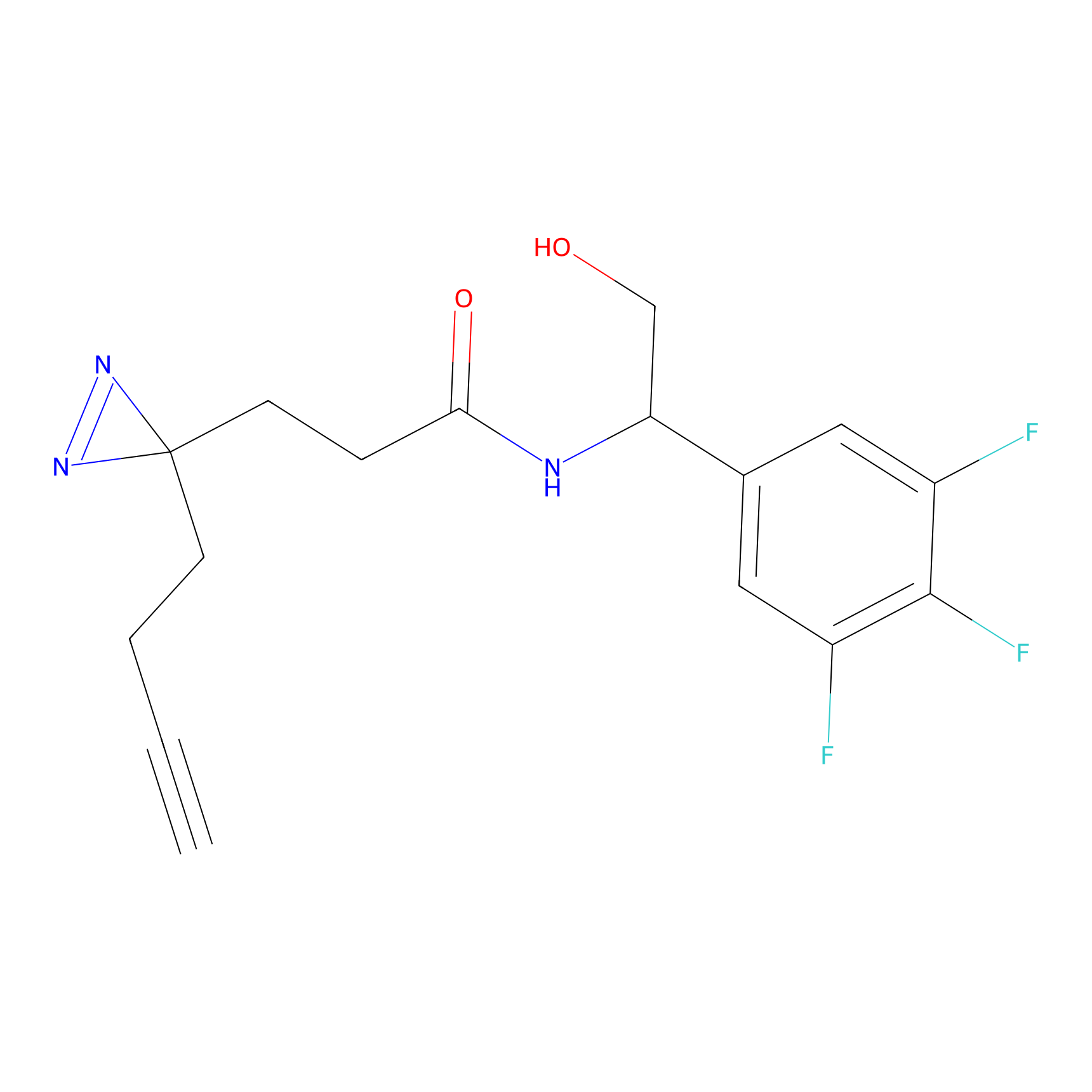
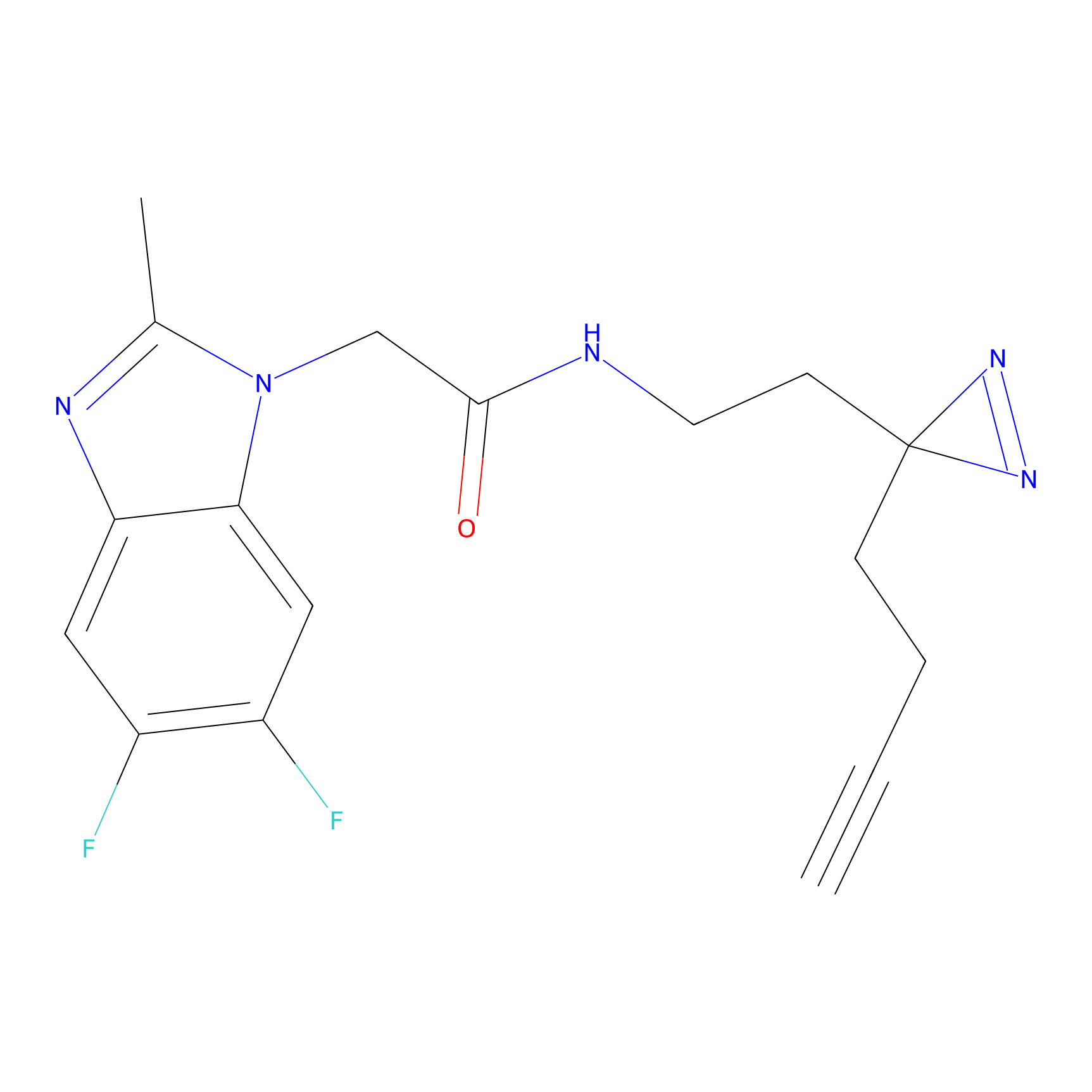
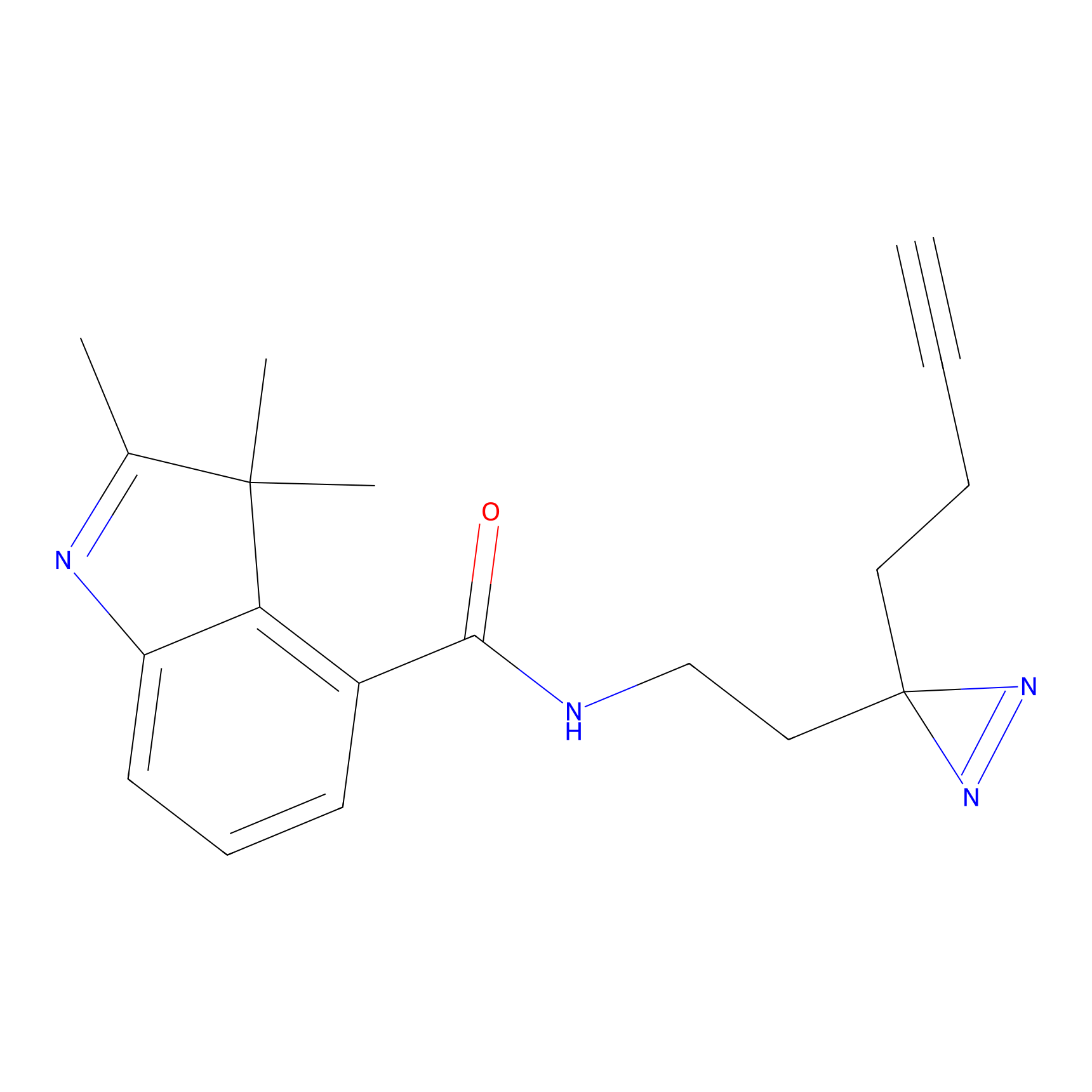
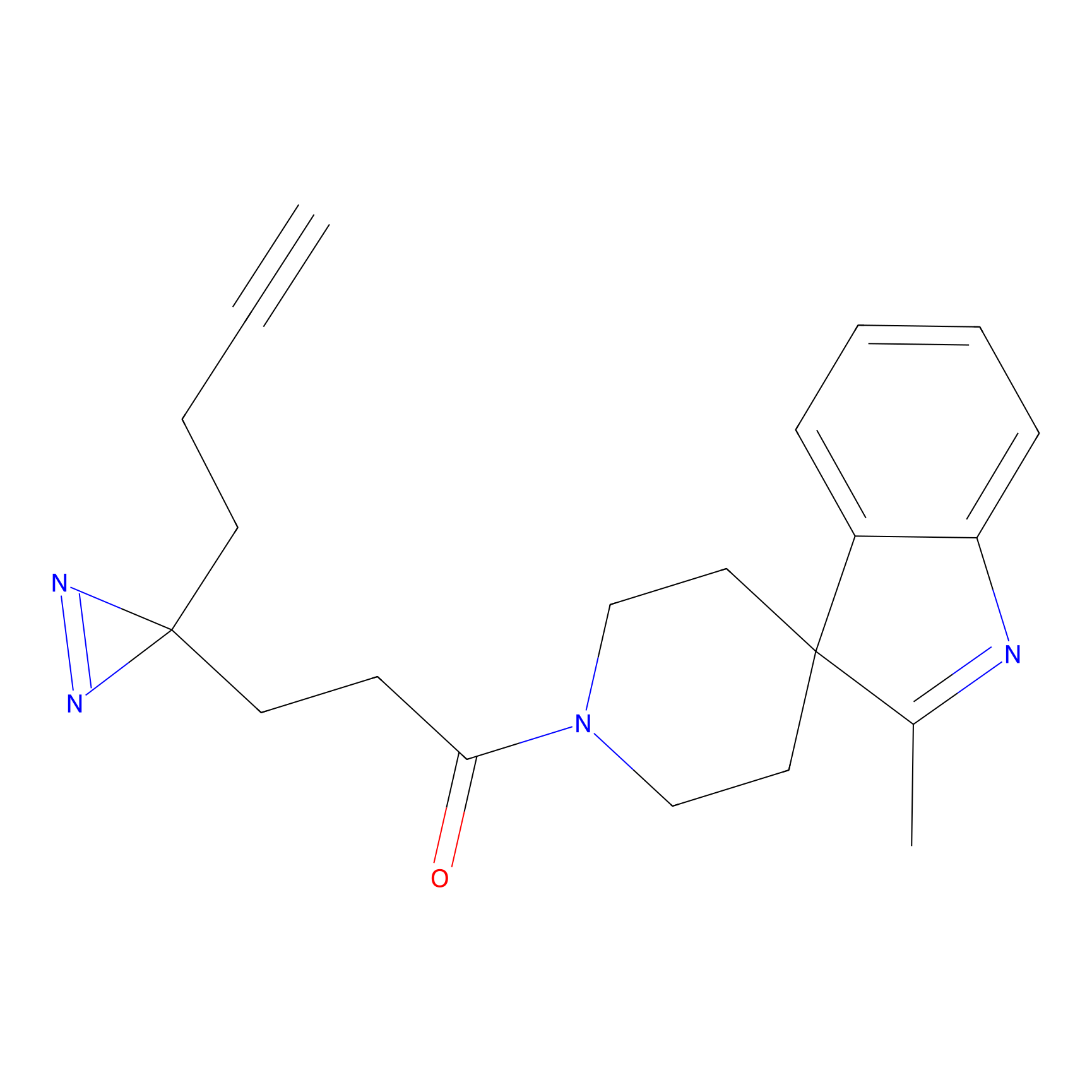
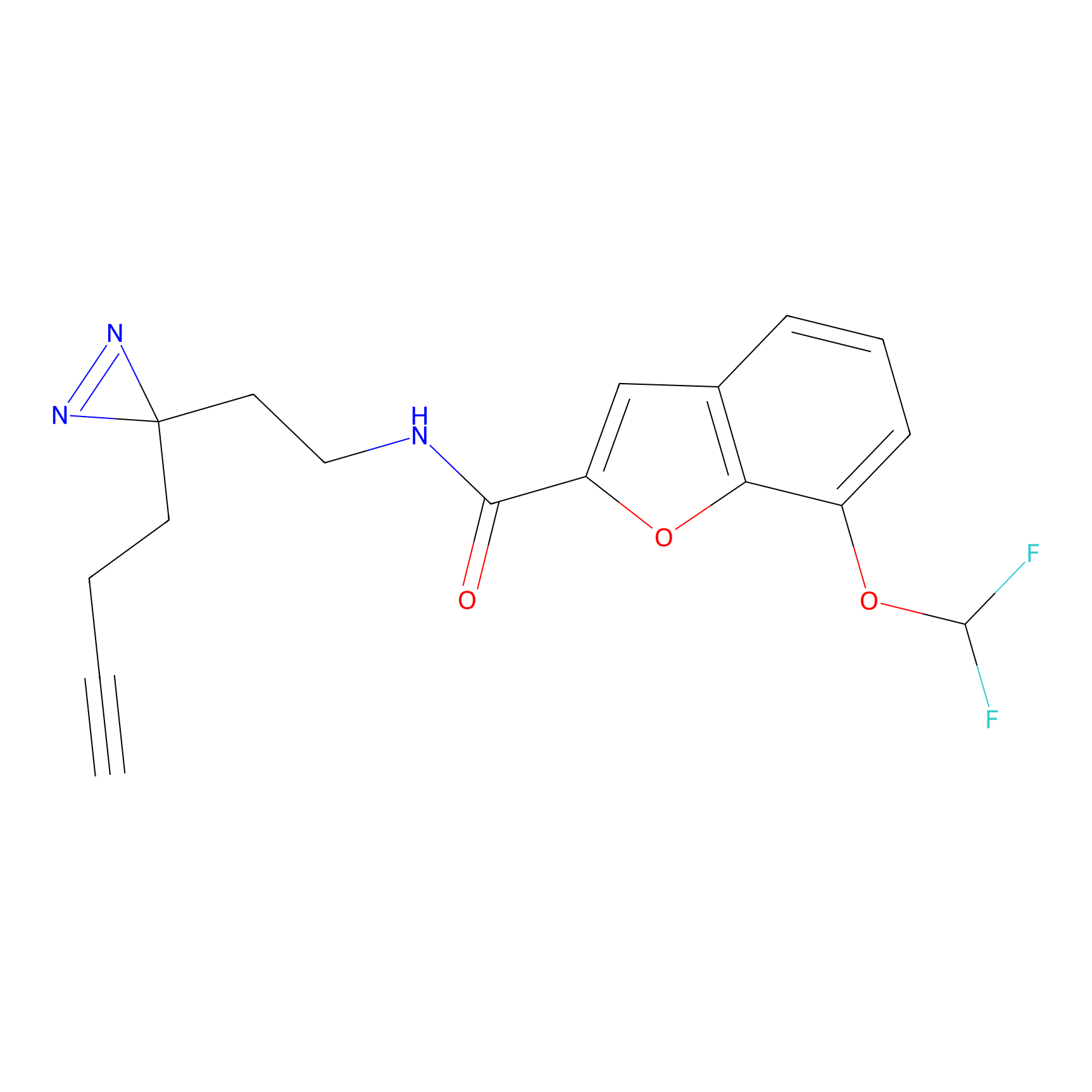
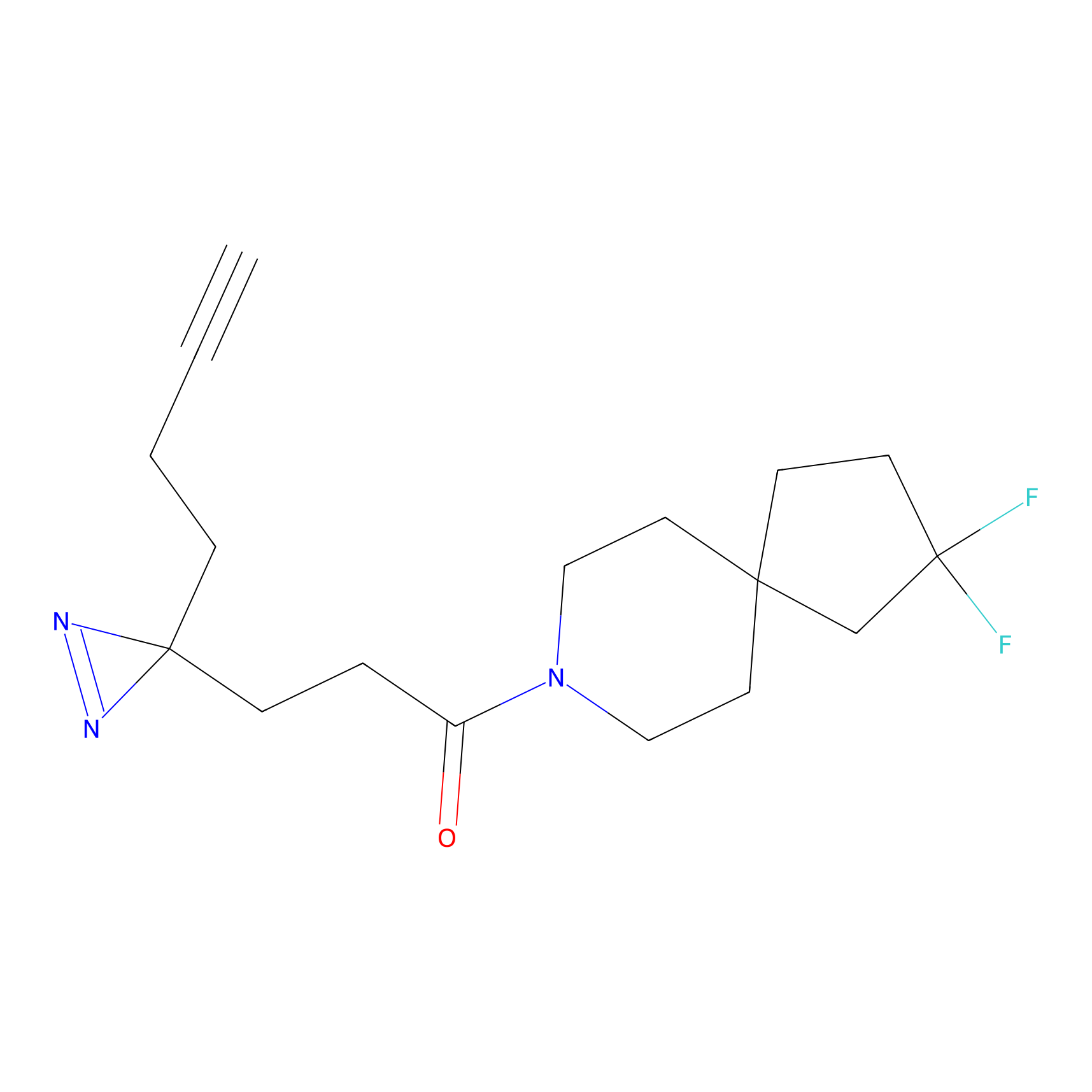
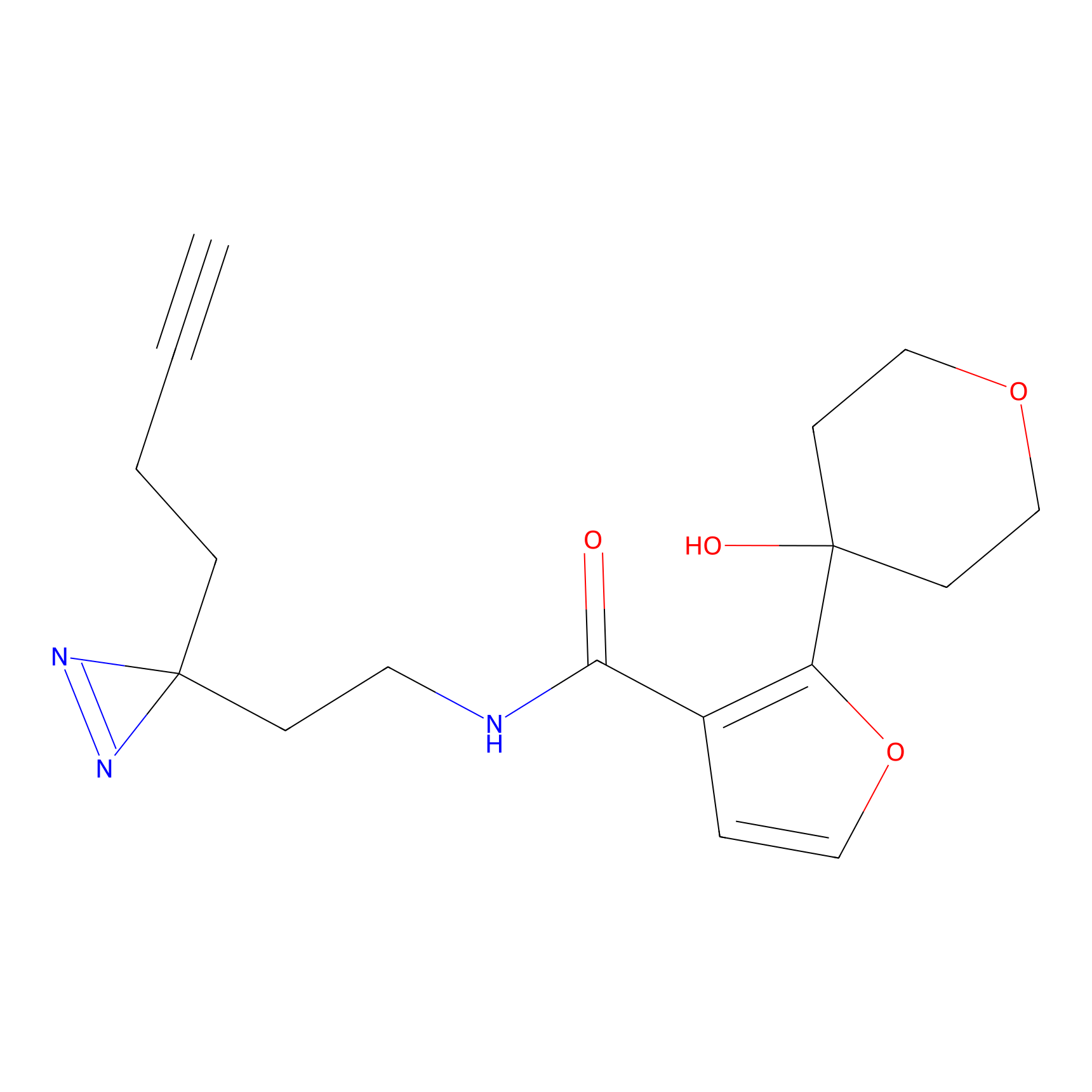
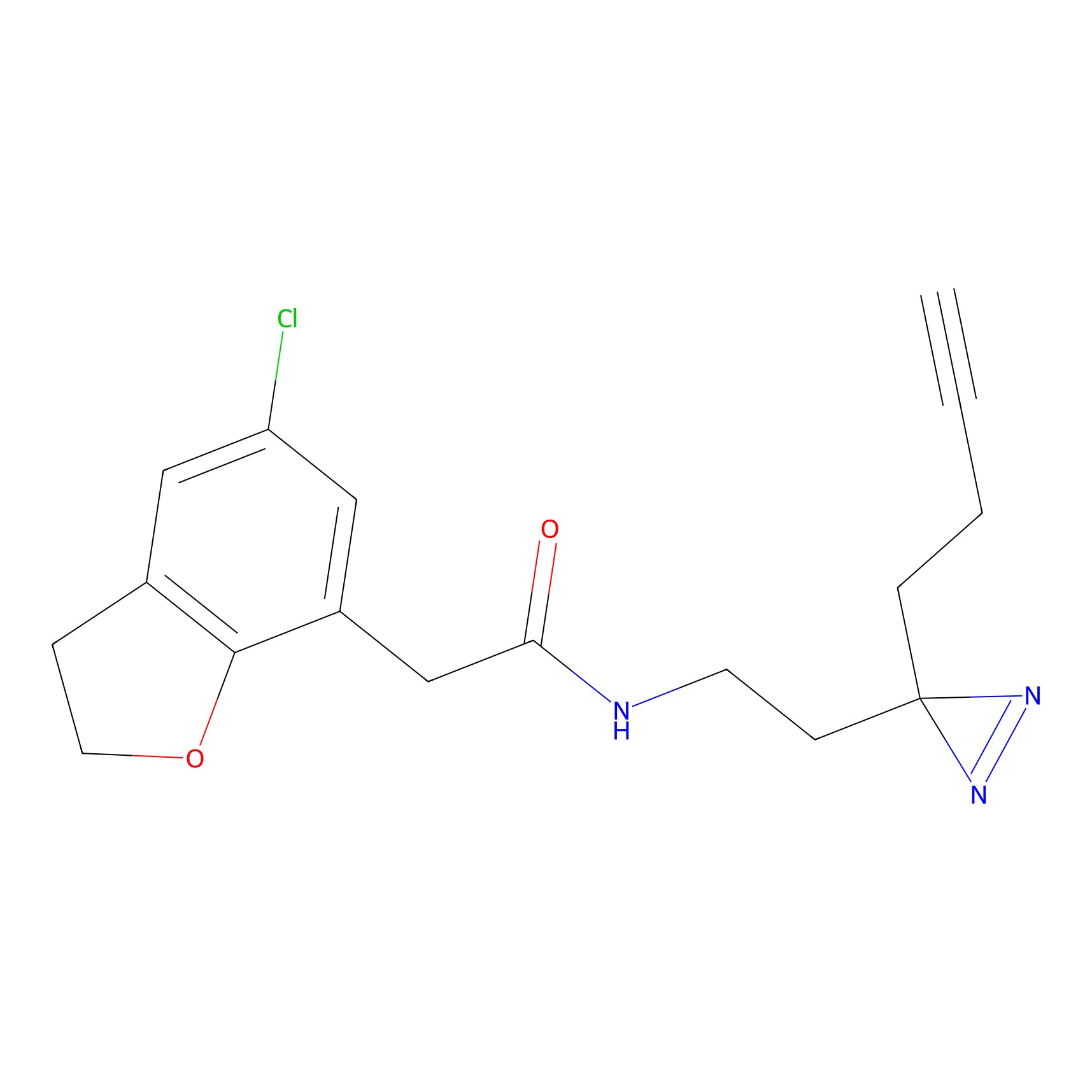
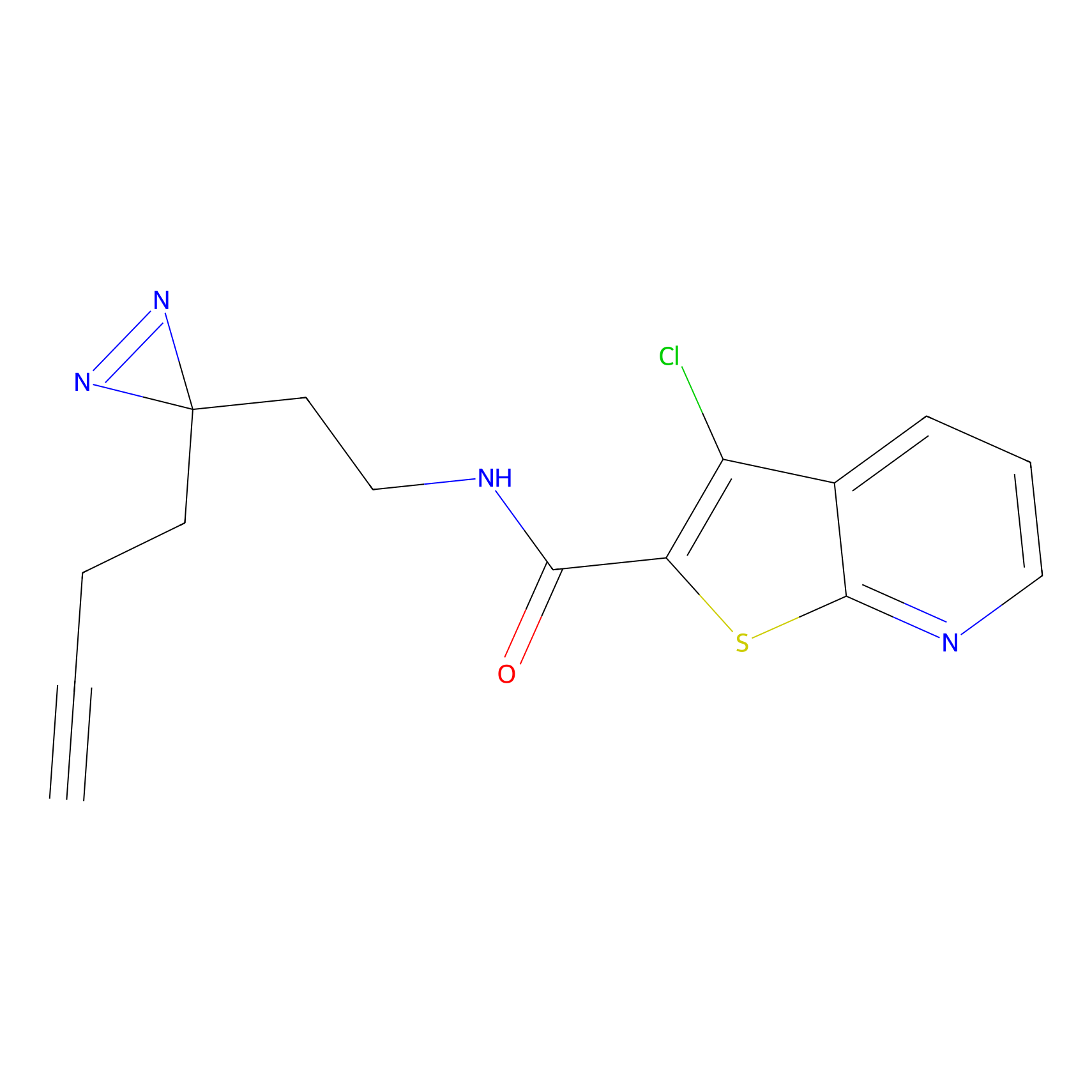
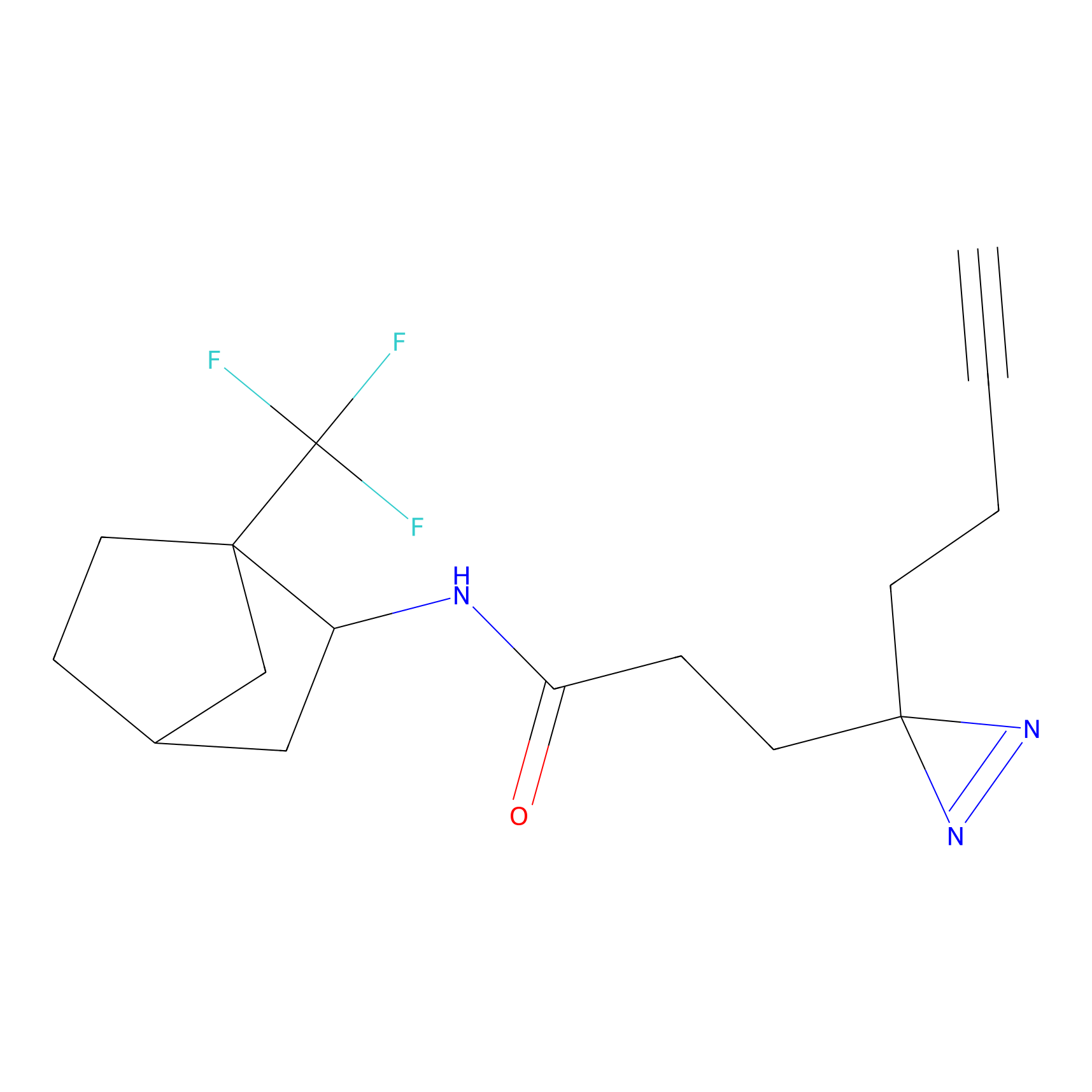
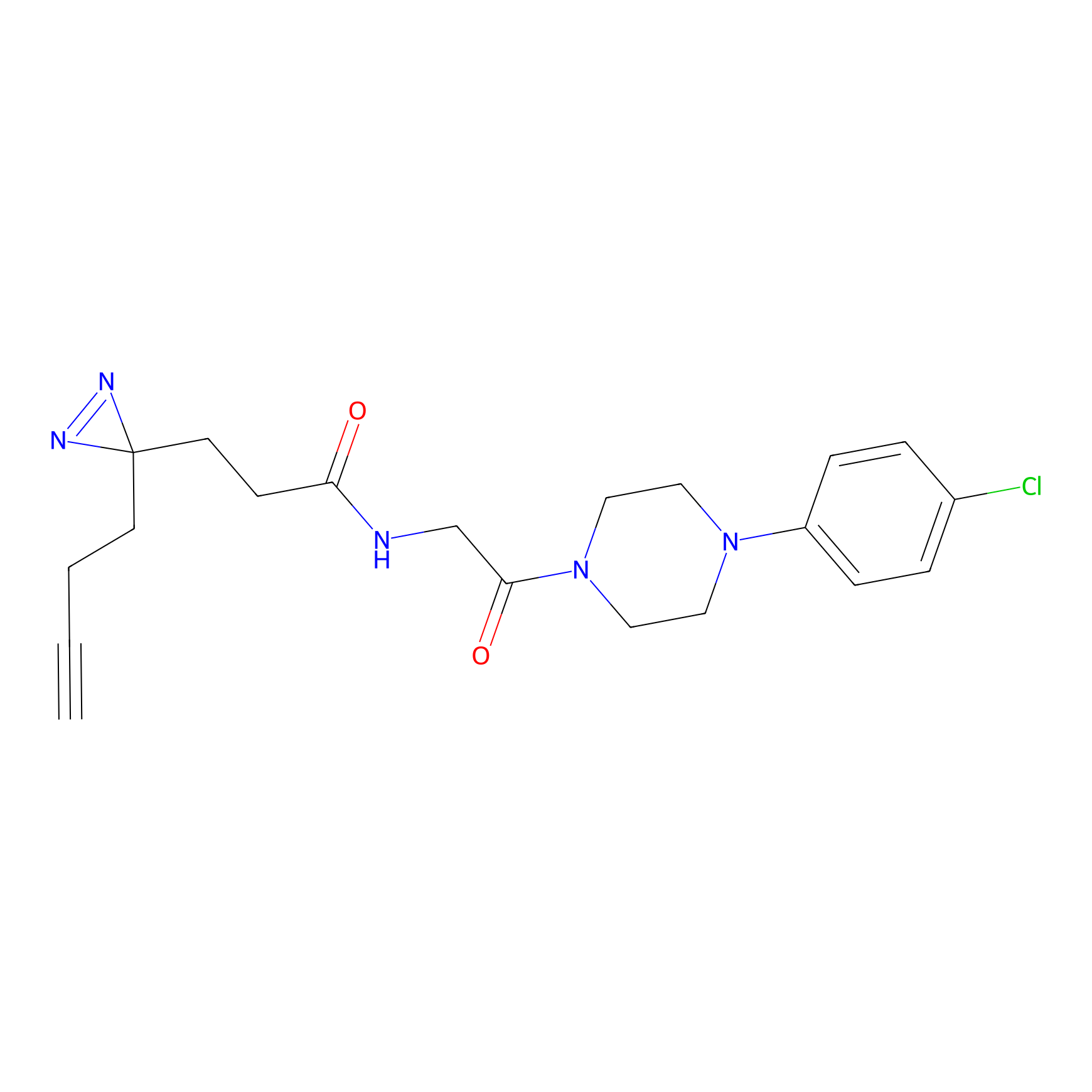
Details of the Target
General Information of Target
| Target ID | LDTP13282 | |||||
|---|---|---|---|---|---|---|
| Target Name | Prenylcysteine oxidase 1 (PCYOX1) | |||||
| Gene Name | PCYOX1 | |||||
| Gene ID | 51449 | |||||
| Synonyms |
KIAA0908; PCL1; Prenylcysteine oxidase 1; EC 1.8.3.5; Prenylcysteine lyase |
|||||
| 3D Structure | ||||||
| Sequence |
MAGSPLLWGPRAGGVGLLVLLLLGLFRPPPALCARPVKEPRGLSAASPPLAETGAPRRFR
RSVPRGEAAGAVQELARALAHLLEAERQERARAEAQEAEDQQARVLAQLLRVWGAPRNSD PALGLDDDPDAPAAQLARALLRARLDPAALAAQLVPAPVPAAALRPRPPVYDDGPAGPDA EEAGDETPDVDPELLRYLLGRILAGSADSEGVAAPRRLRRAADHDVGSELPPEGVLGALL RVKRLETPAPQVPARRLLPP |
|||||
| Target Bioclass |
Enzyme
|
|||||
| Family |
Prenylcysteine oxidase family
|
|||||
| Subcellular location |
Lysosome
|
|||||
| Function |
Prenylcysteine oxidase that cleaves the thioether bond of prenyl-L-cysteines, such as farnesylcysteine and geranylgeranylcysteine. Only active against free prenylcysteines and not prenylcysteine residues within prenylated proteins or peptides. Involved in the final step in the degradation of prenylated proteins, by degrading prenylcysteines after the protein has been degraded.
|
|||||
| Uniprot ID | ||||||
| Ensemble ID | ||||||
| HGNC ID | ||||||
Target Site Mutations in Different Cell Lines
| Cell line | Mutation details | Probe for labeling this protein in this cell | |||
|---|---|---|---|---|---|
| ABC1 | SNV: p.E67K | DBIA Probe Info | |||
| HCC44 | SNV: p.V4F | DBIA Probe Info | |||
| HCT116 | SNV: p.L17M | . | |||
| HGC27 | SNV: p.Q204K | DBIA Probe Info | |||
| OVKATE | SNV: p.R197Ter | DBIA Probe Info | |||
| SNU478 | Substitution: p.A28F | DBIA Probe Info | |||
| SUPT1 | SNV: p.A478T | DBIA Probe Info | |||
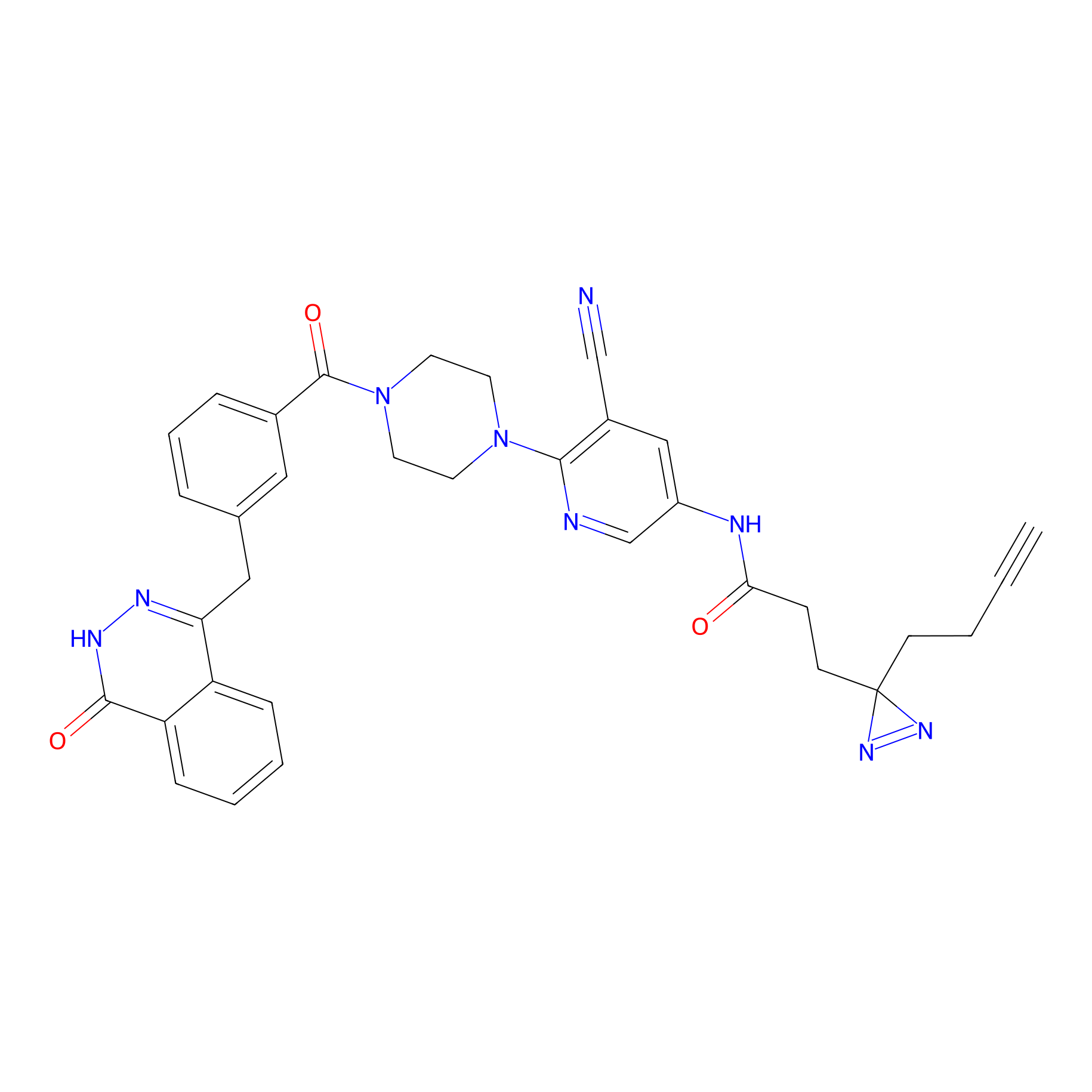
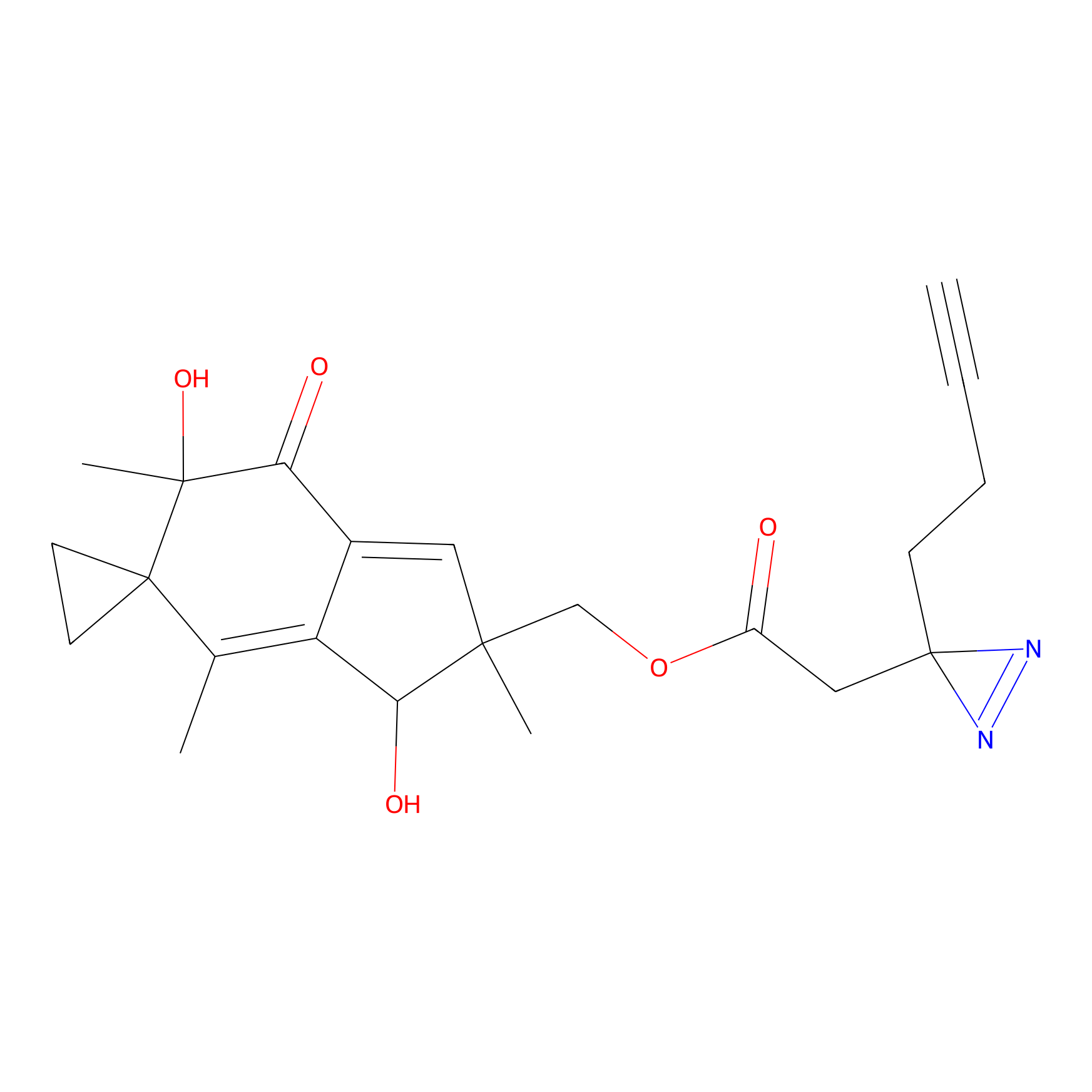
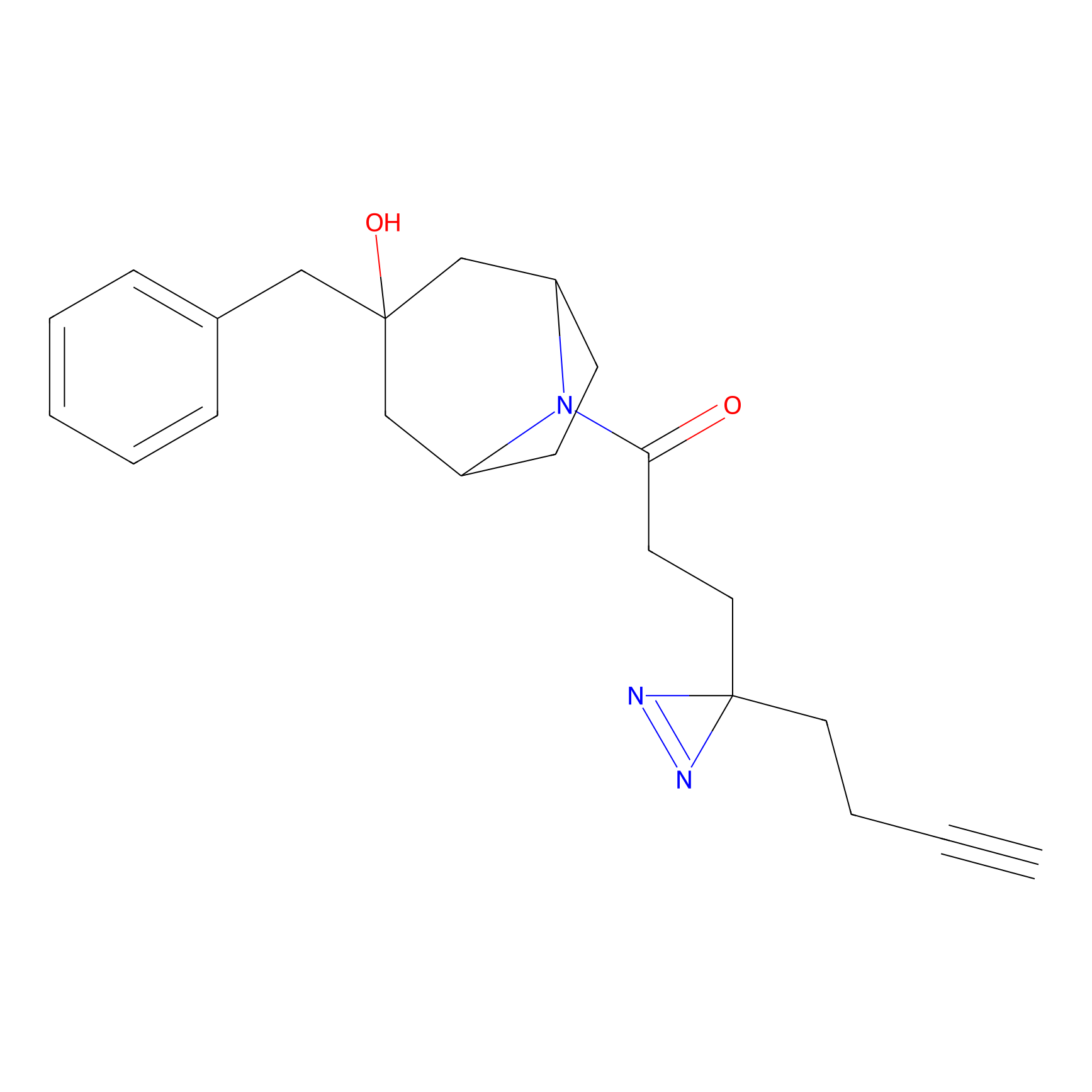
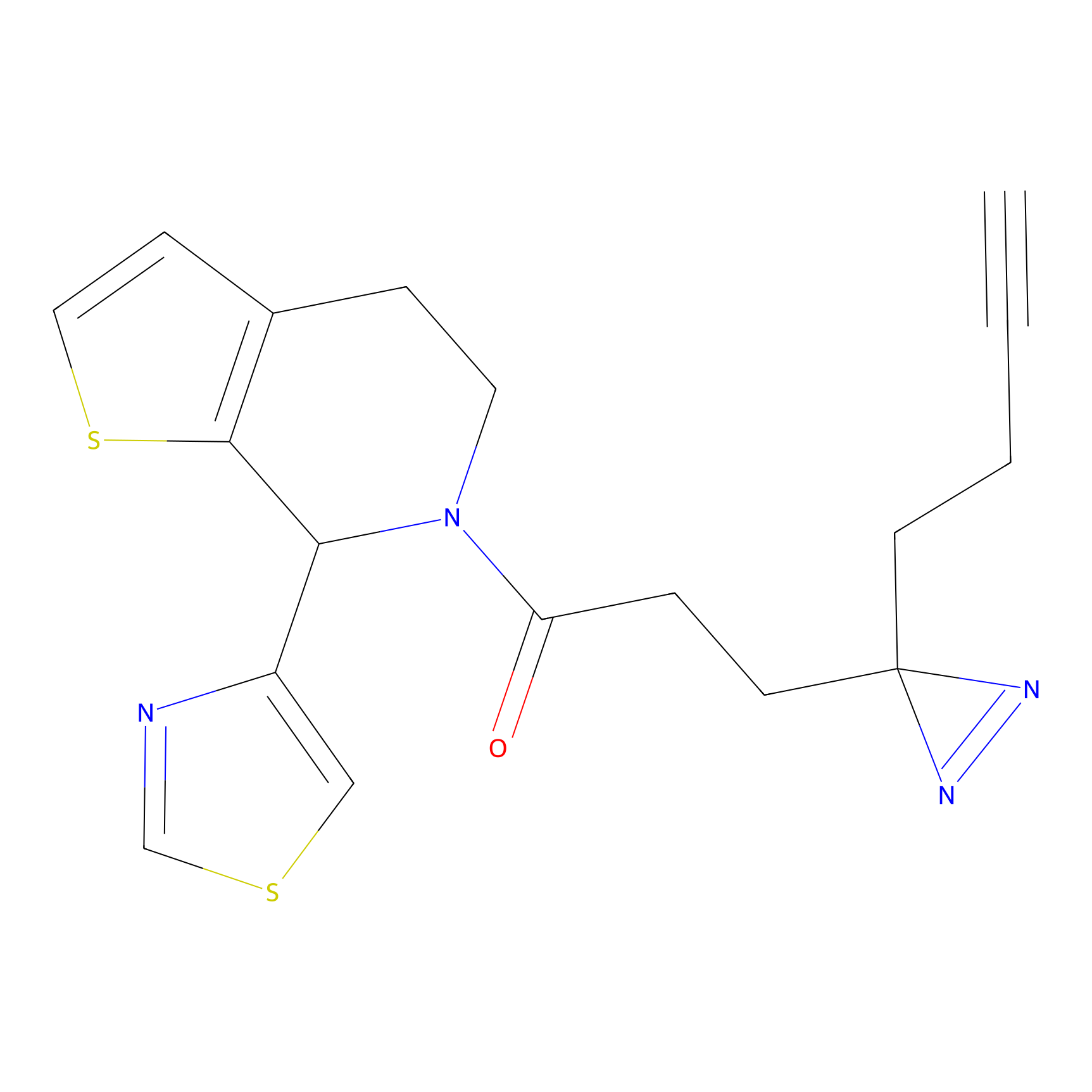
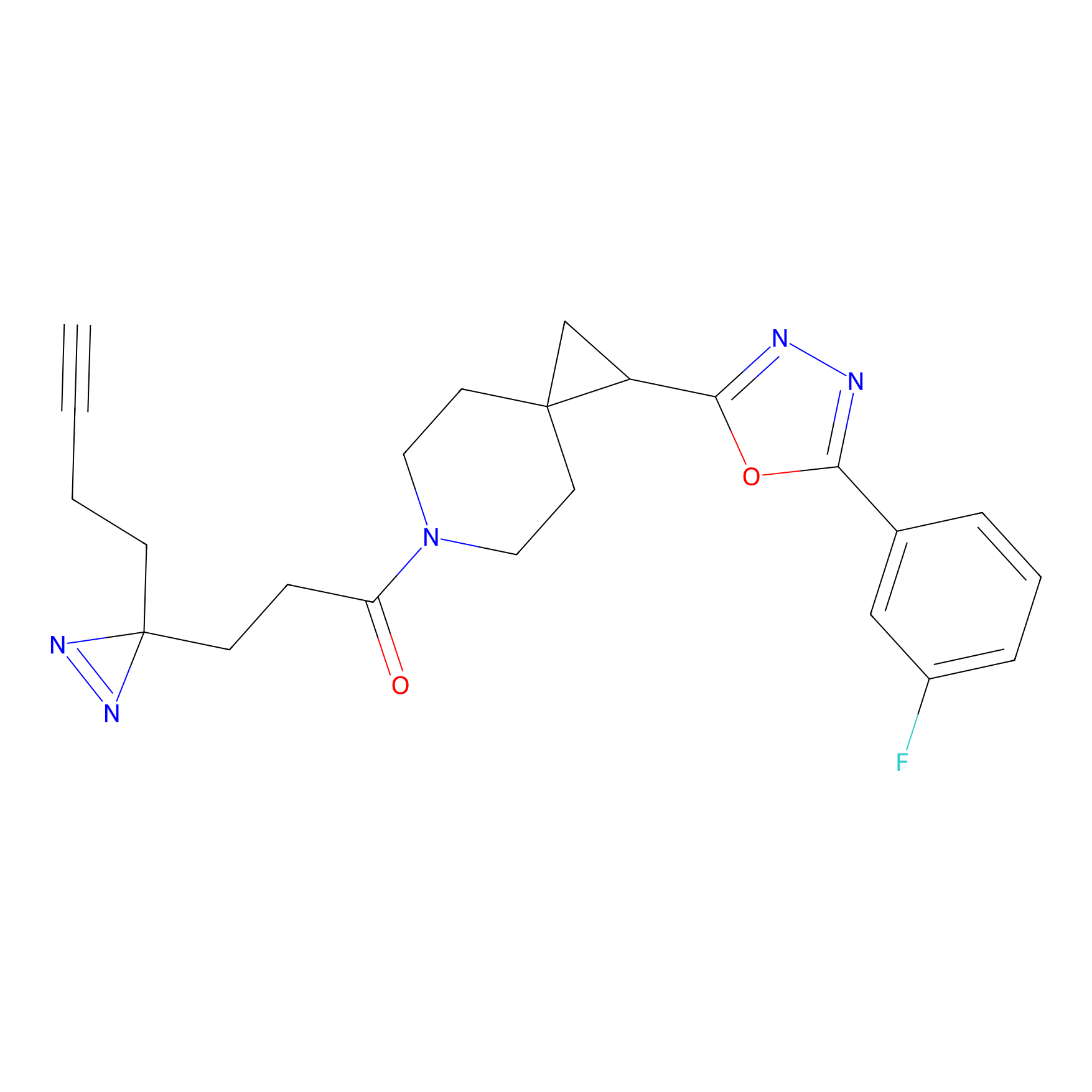
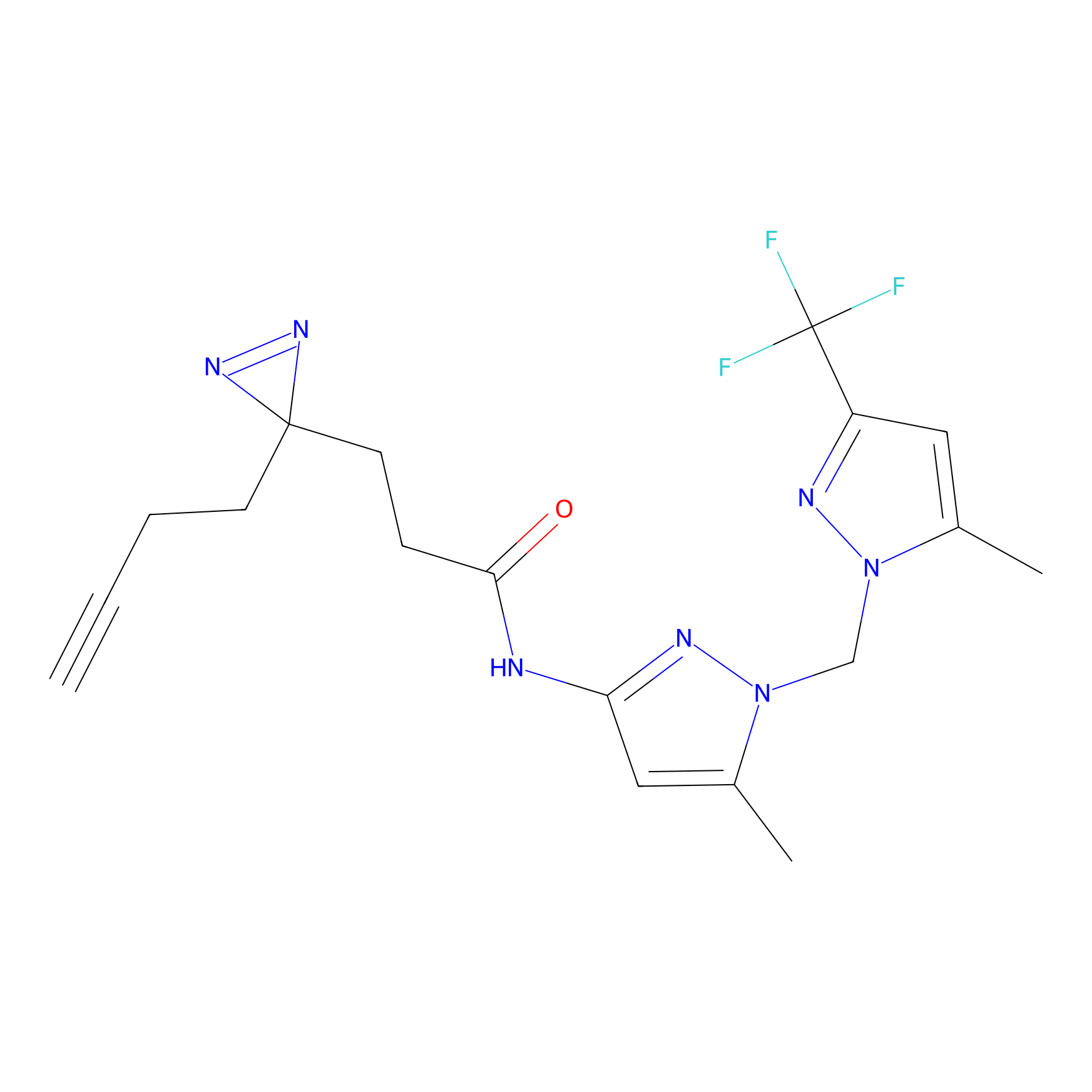
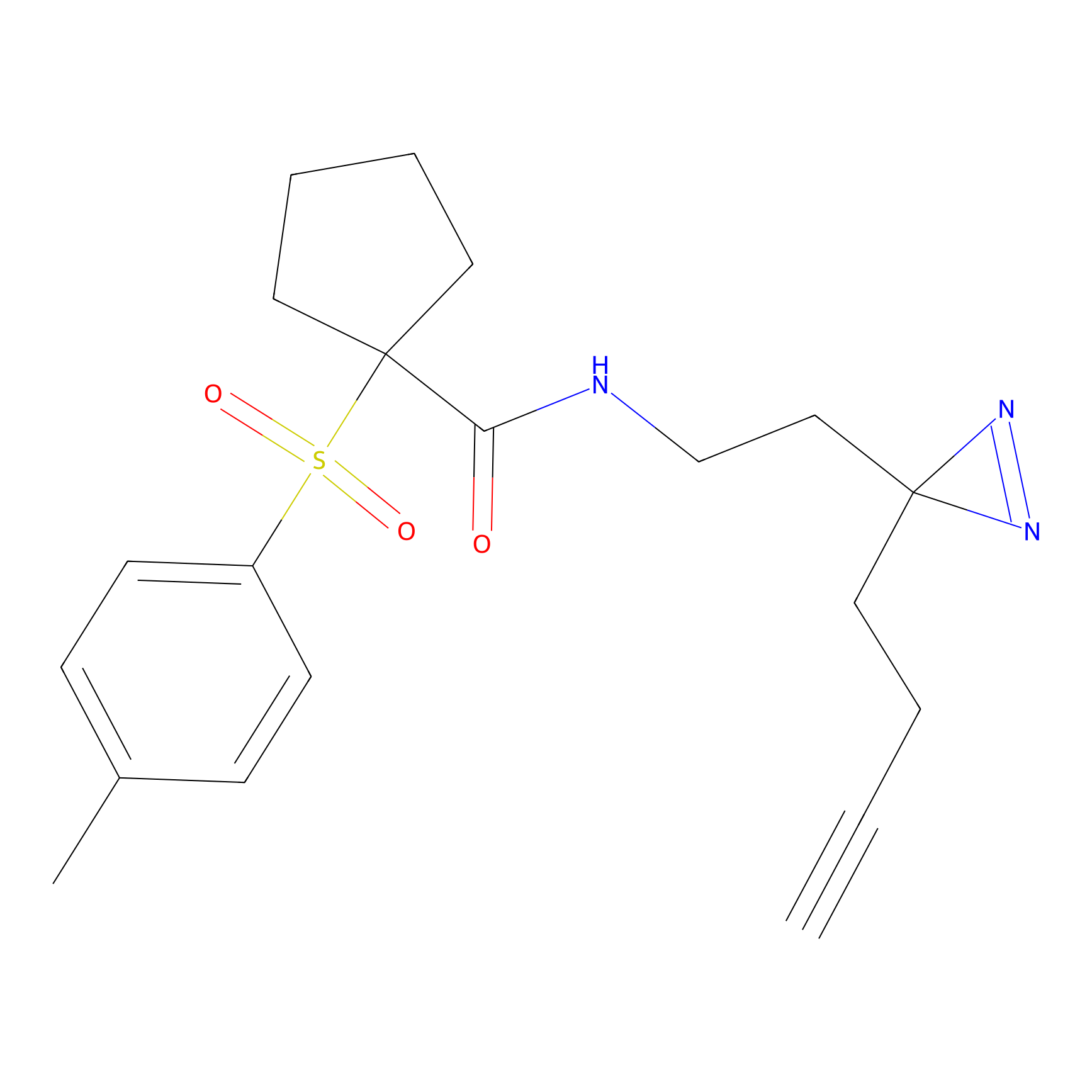
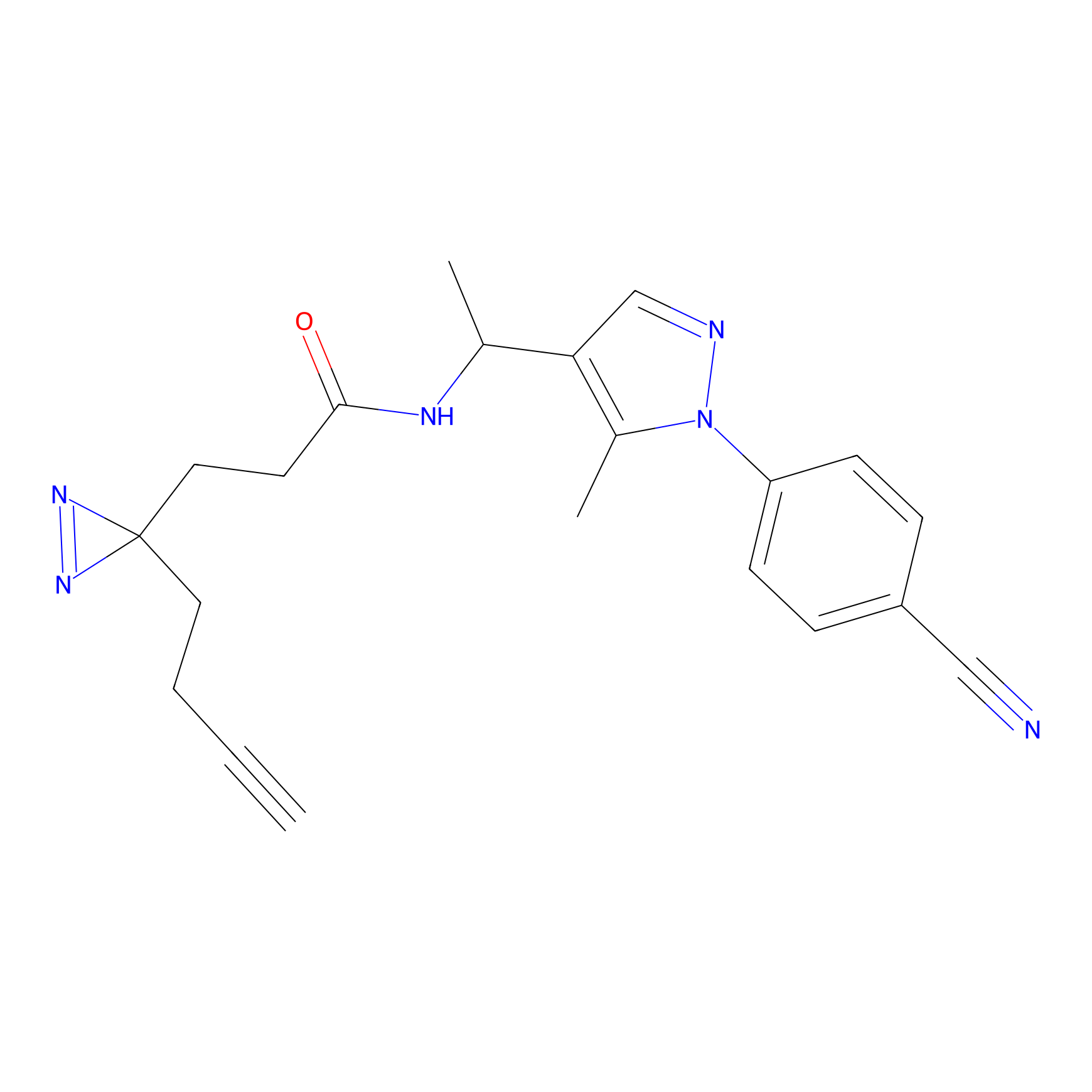
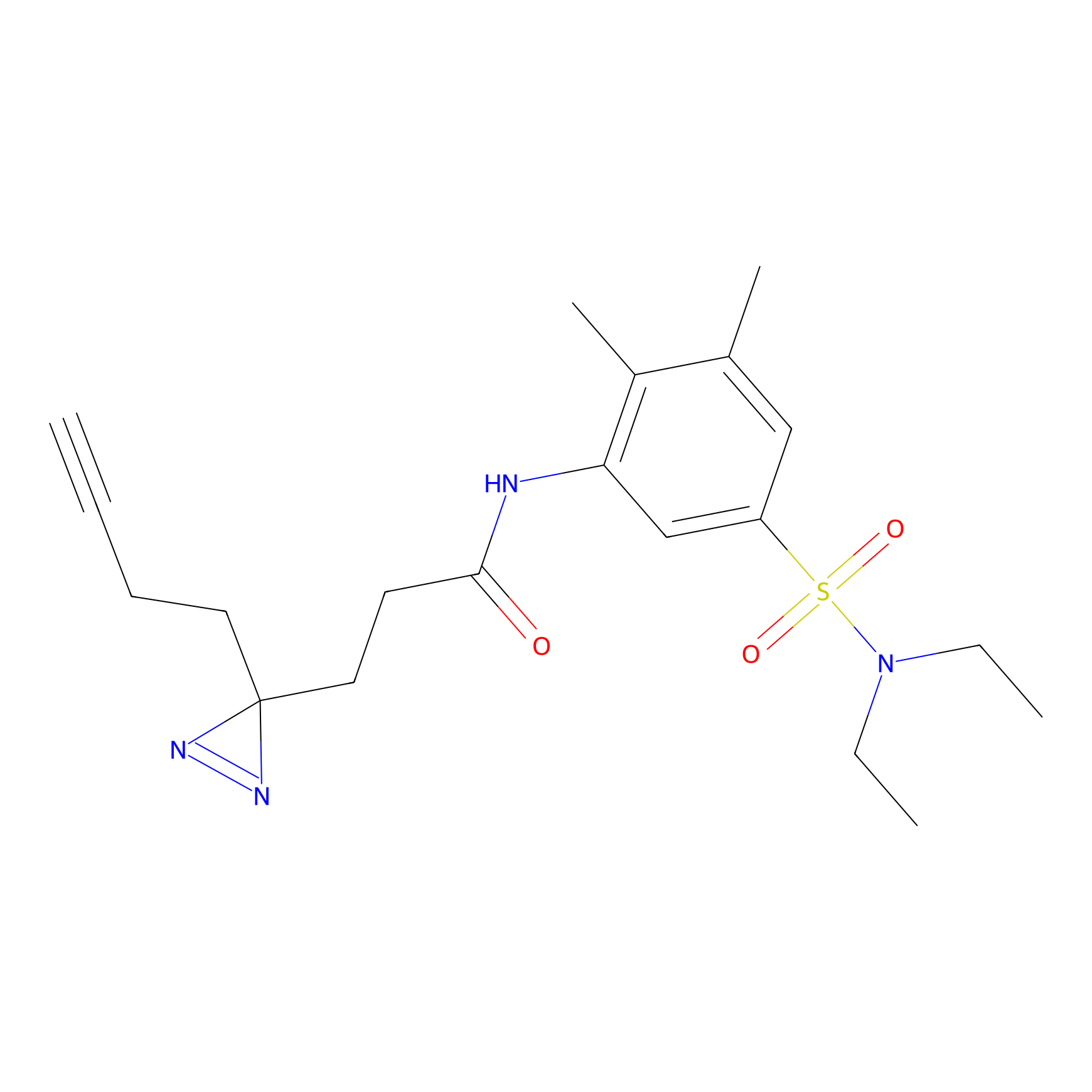
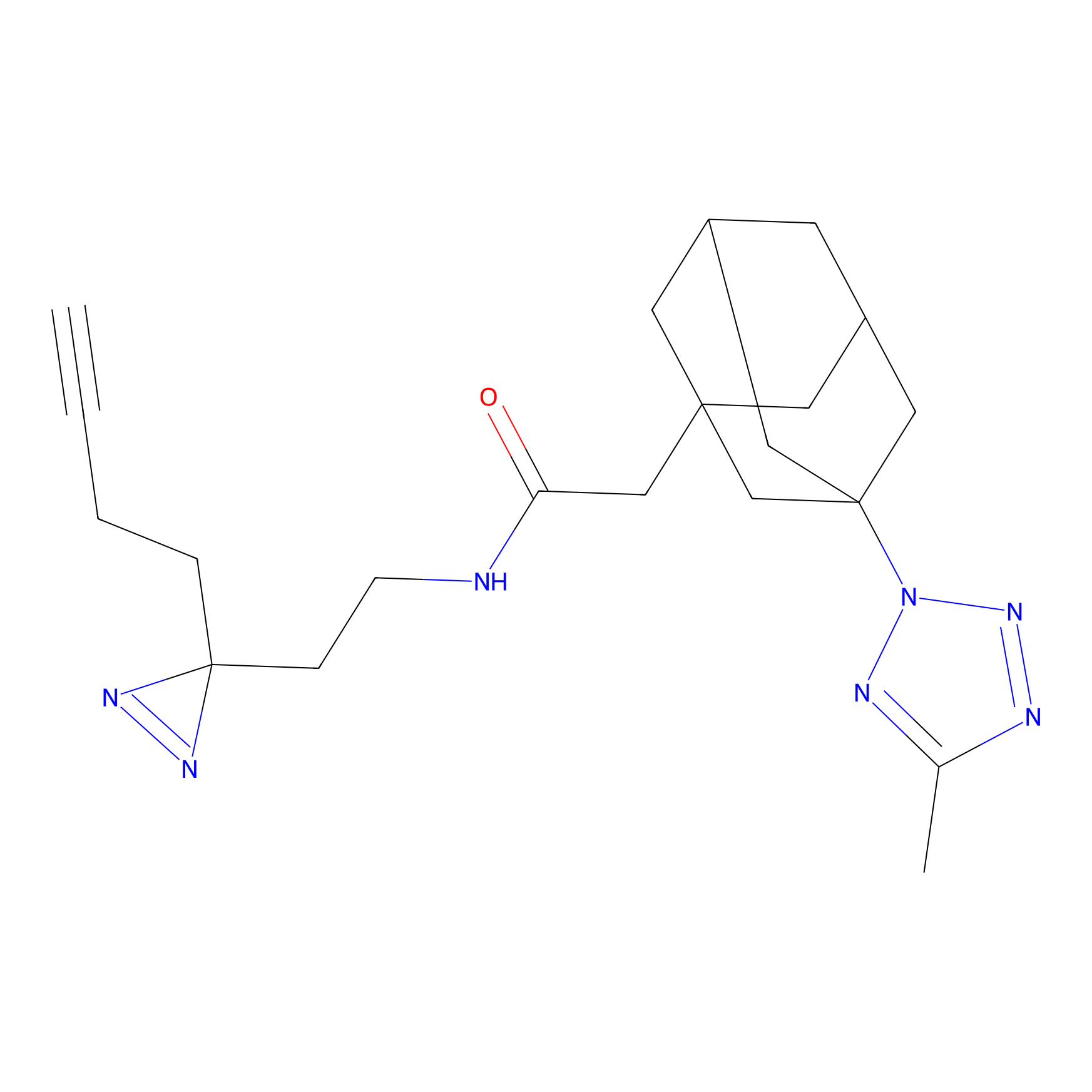
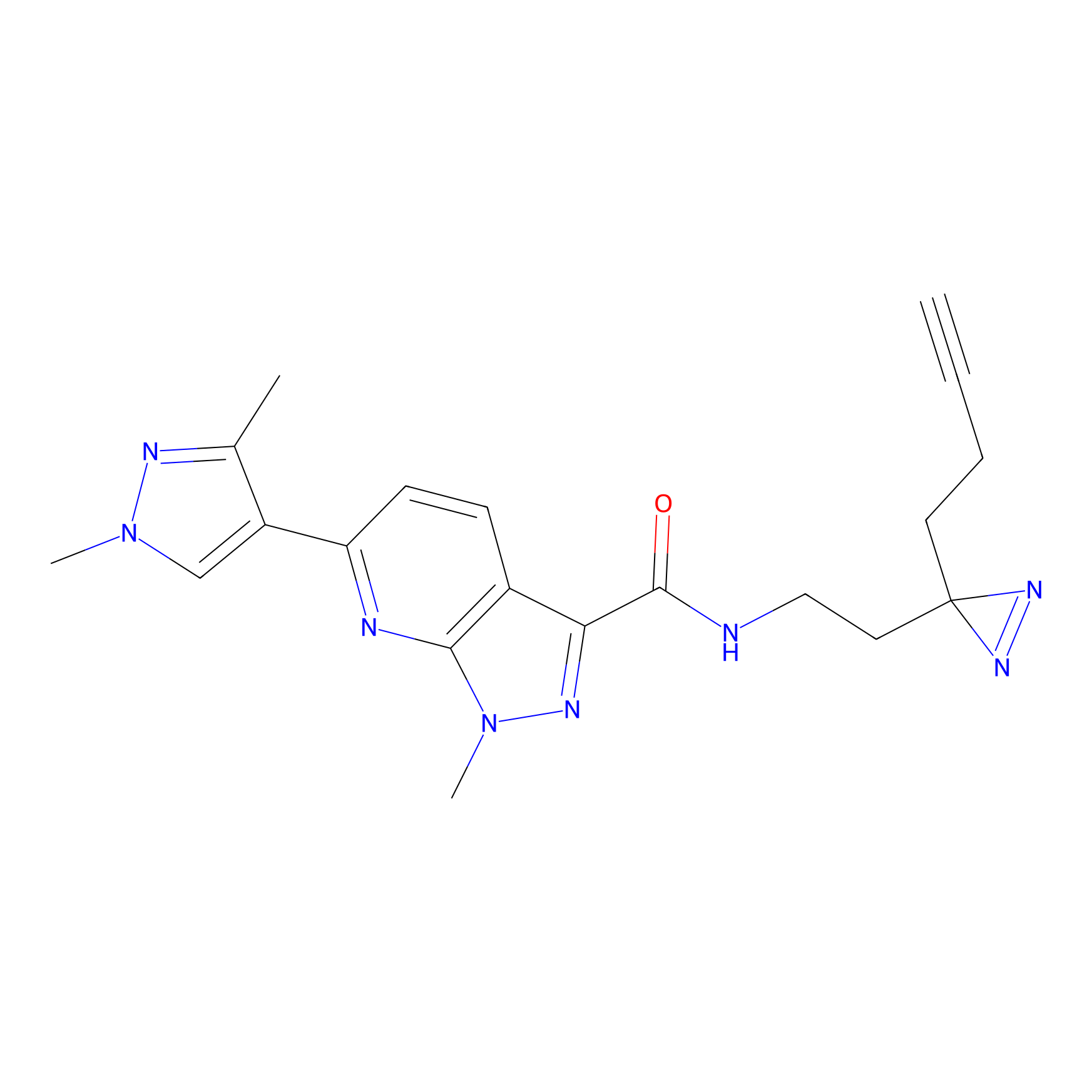
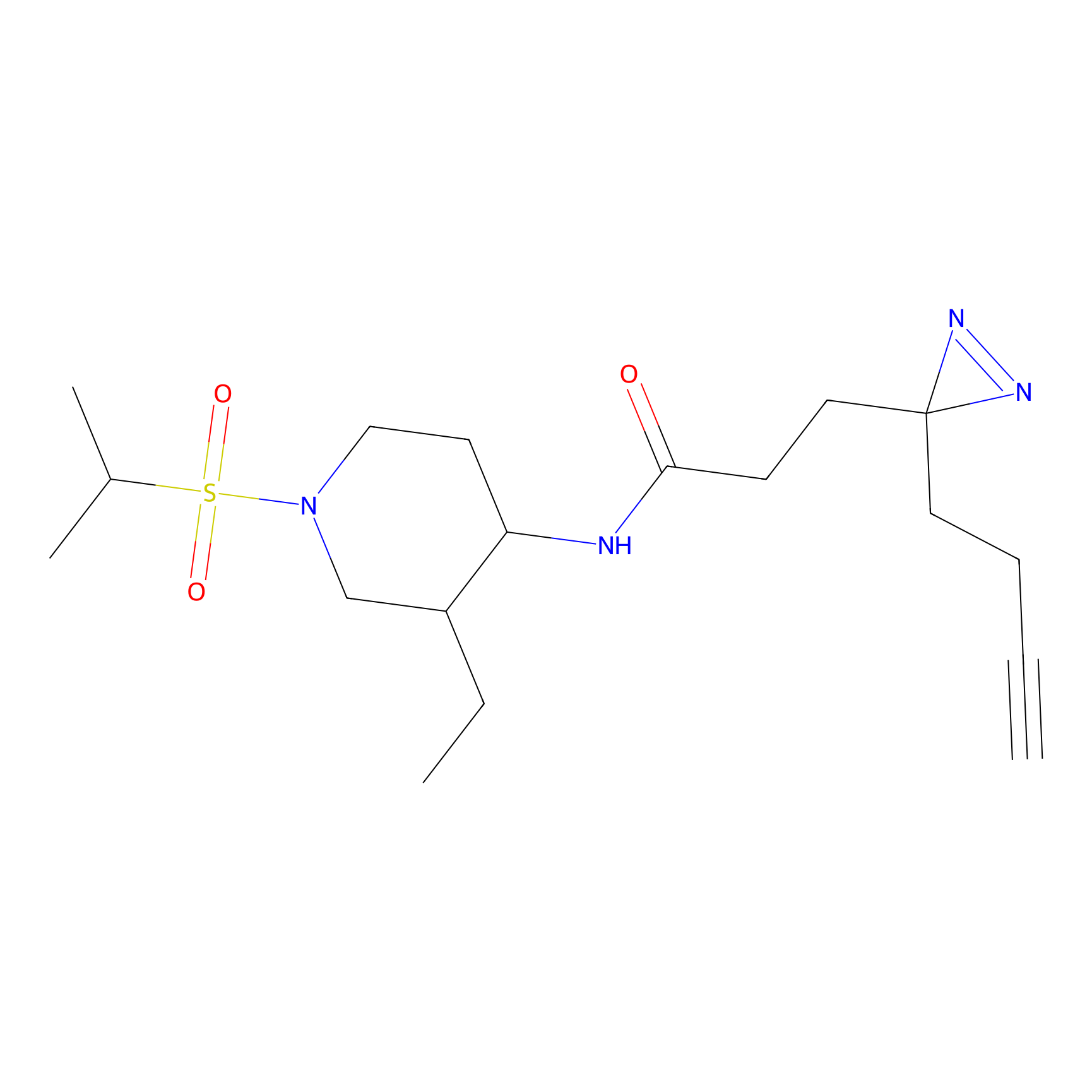
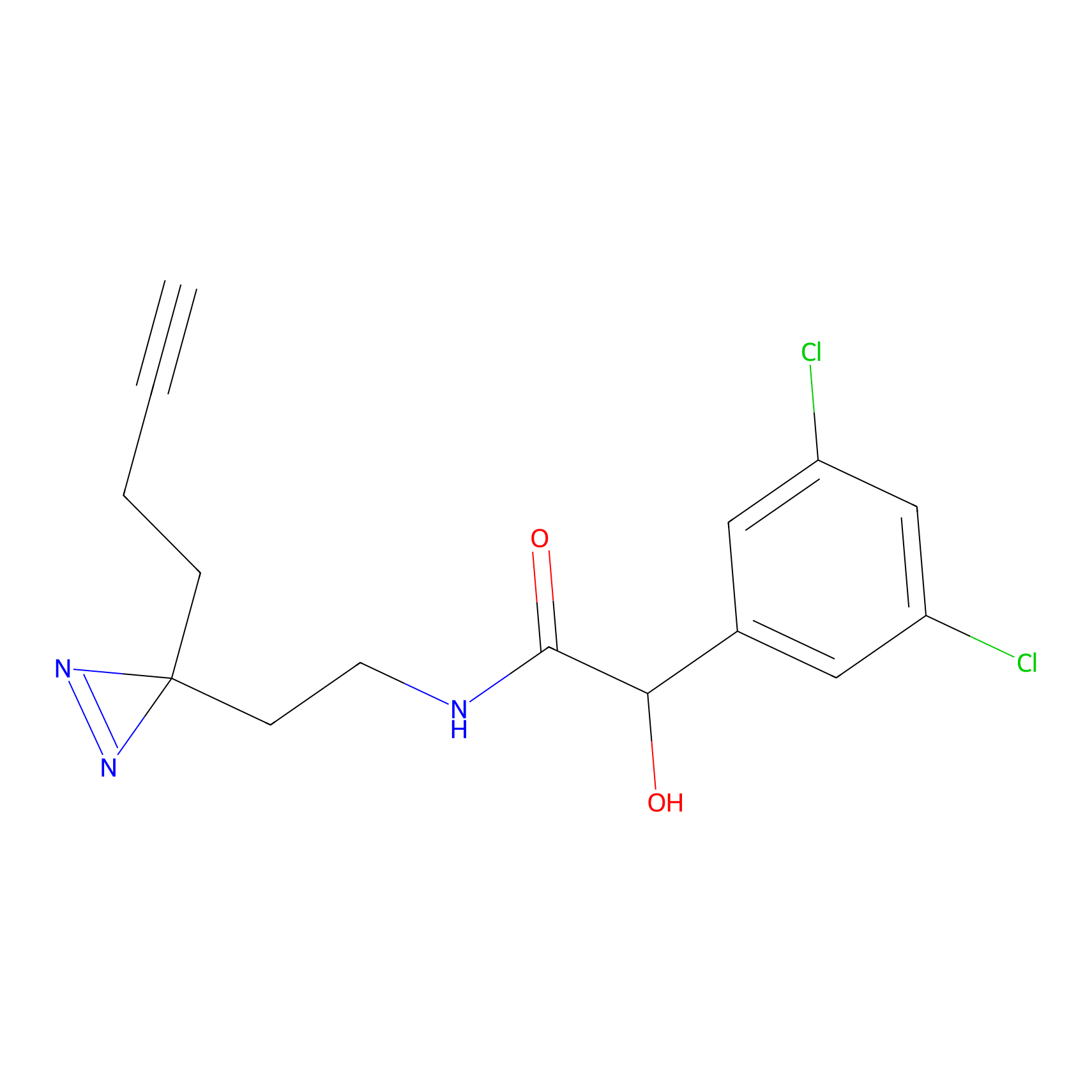
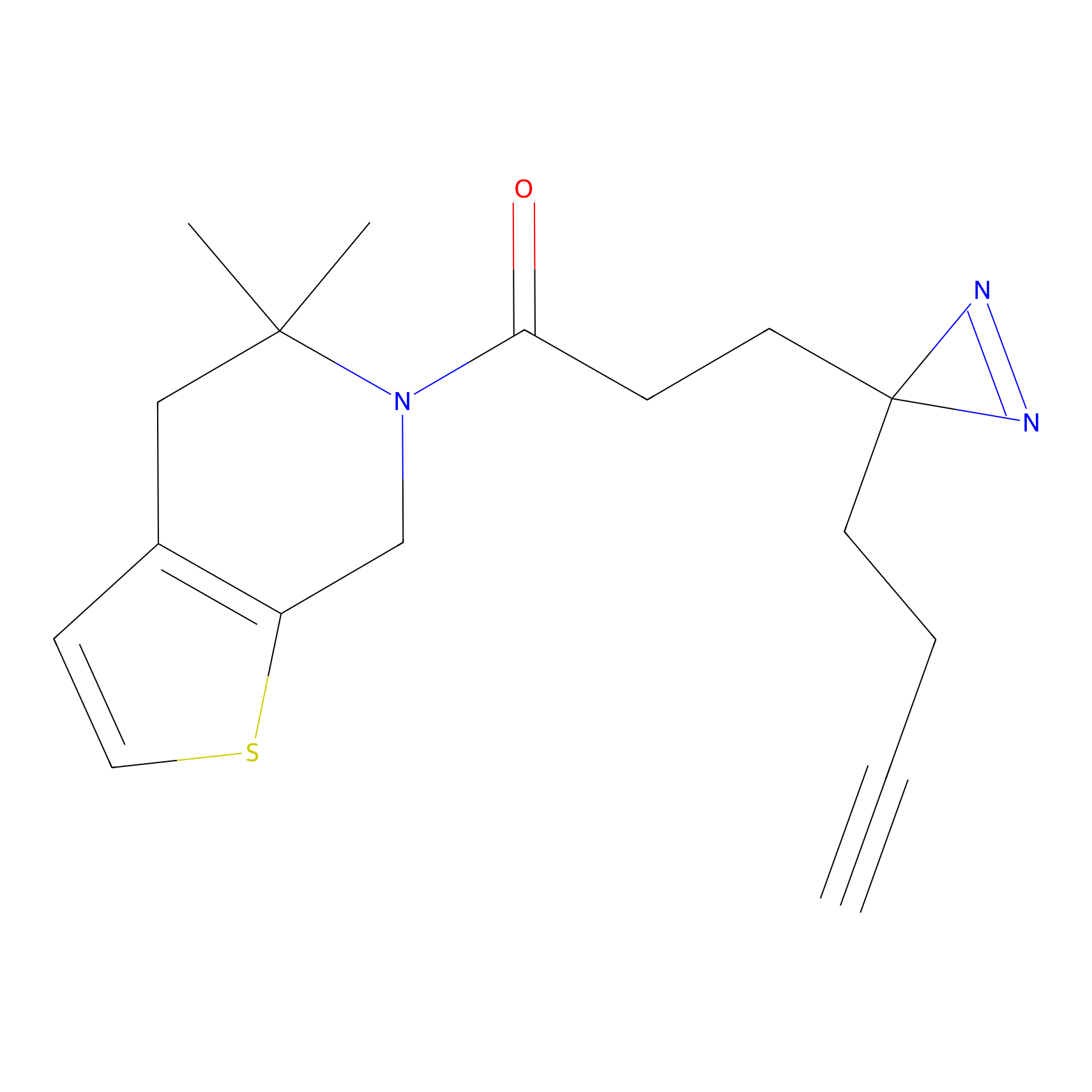
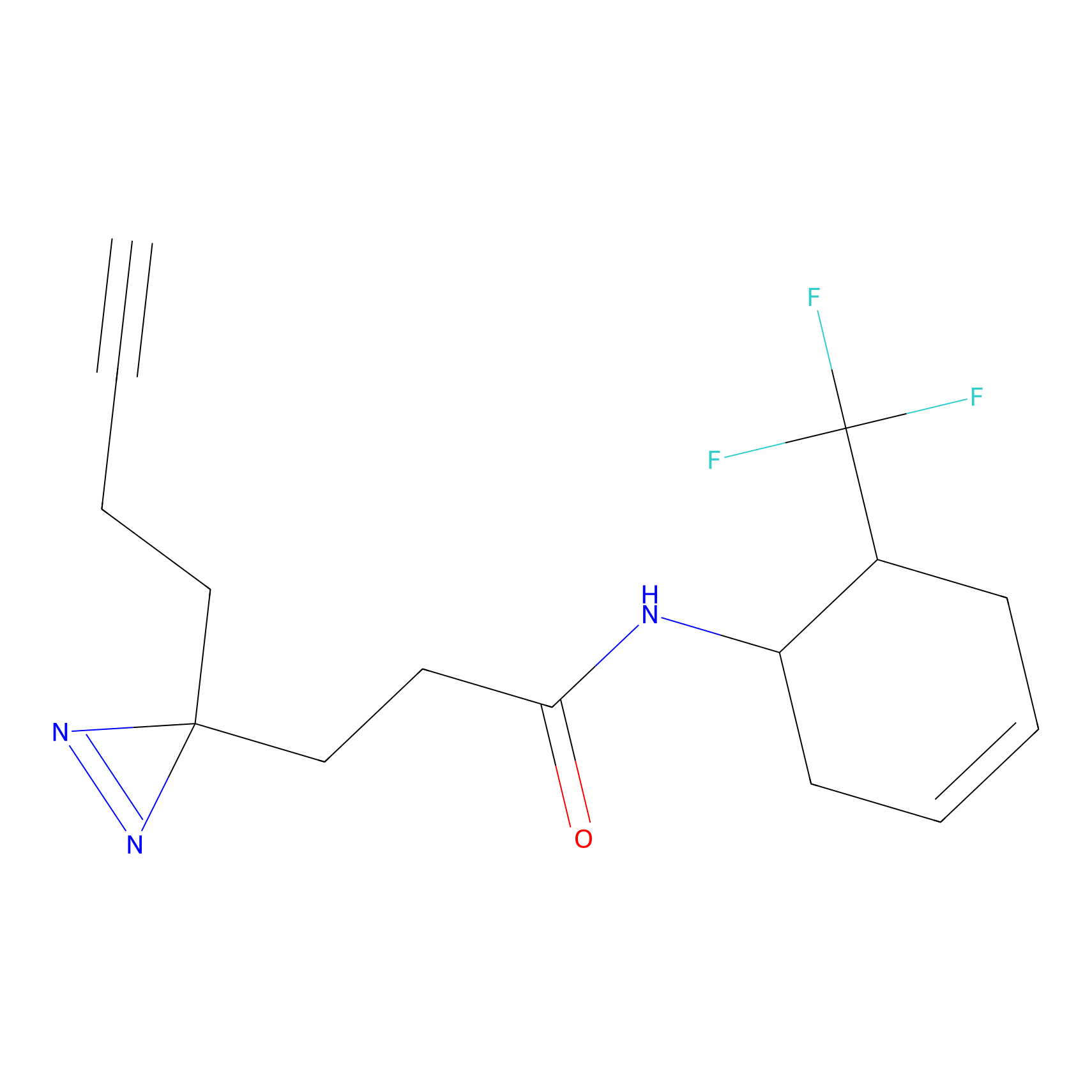
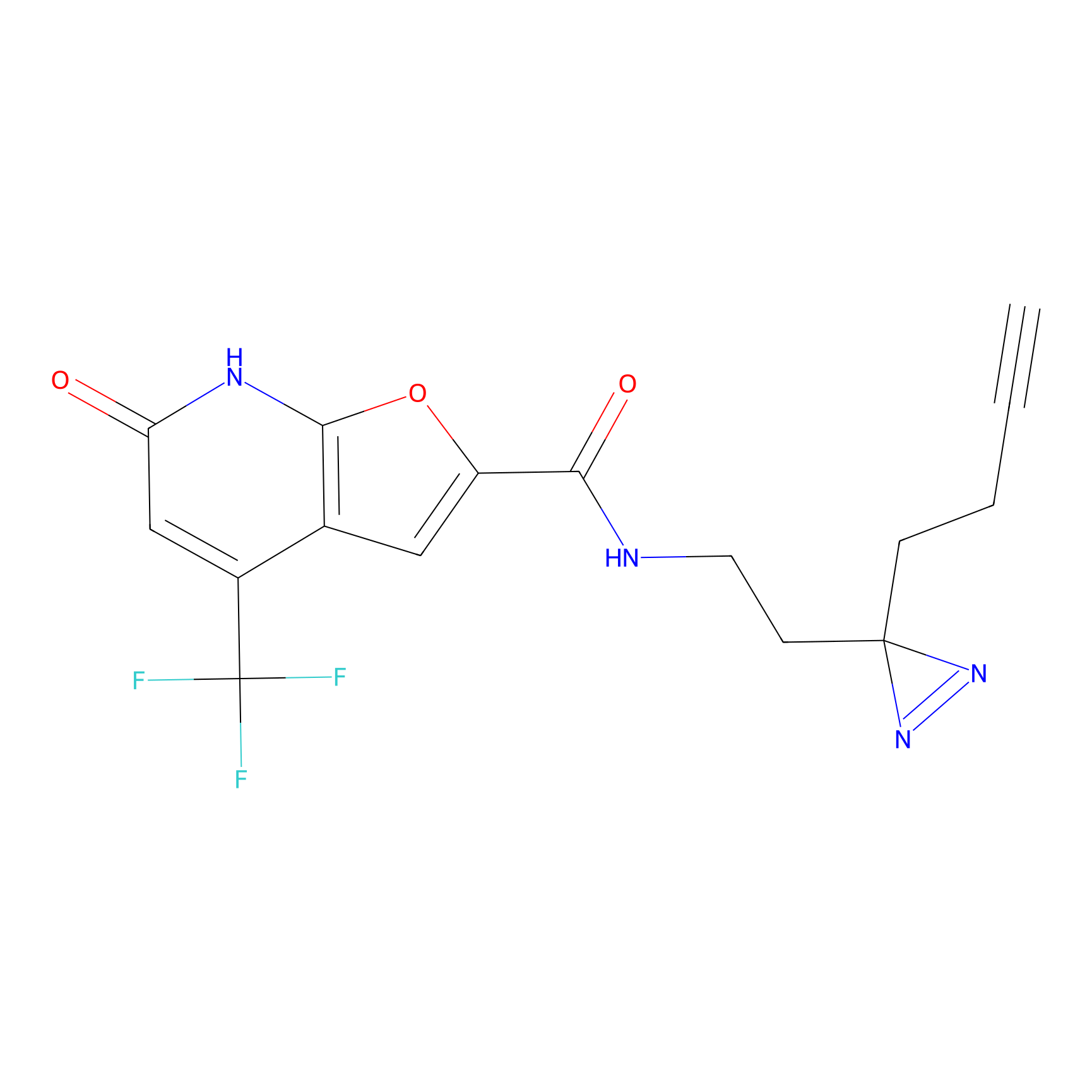
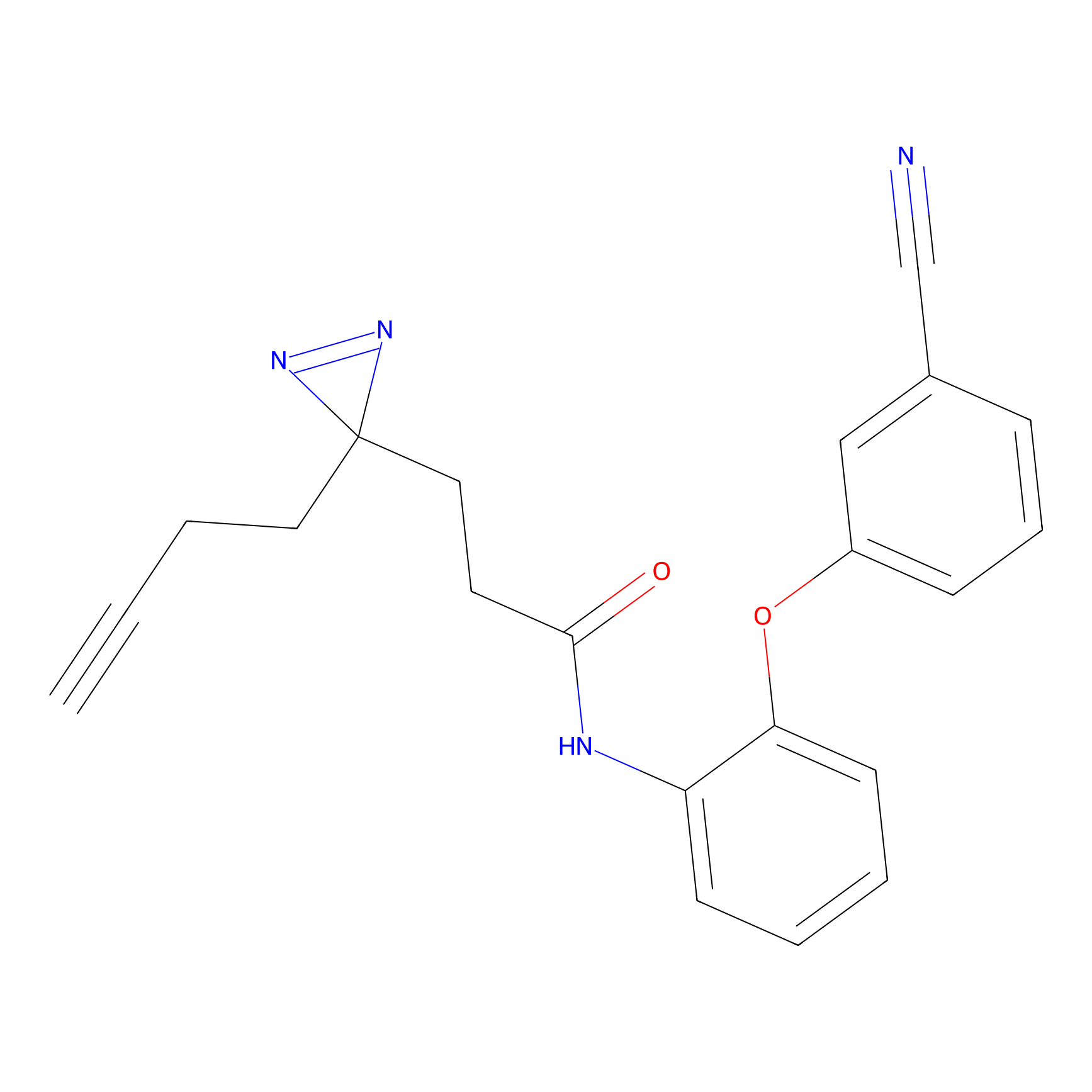
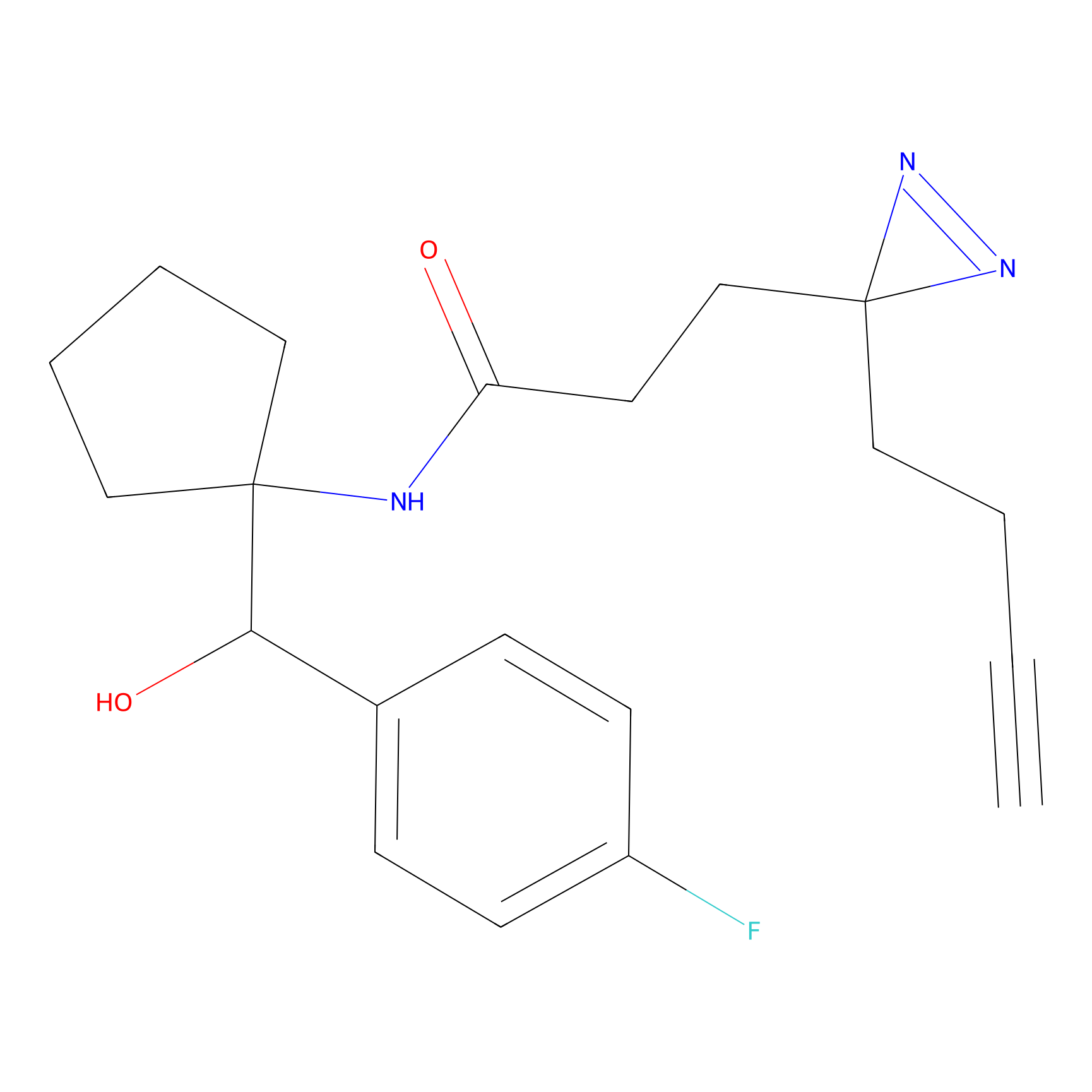
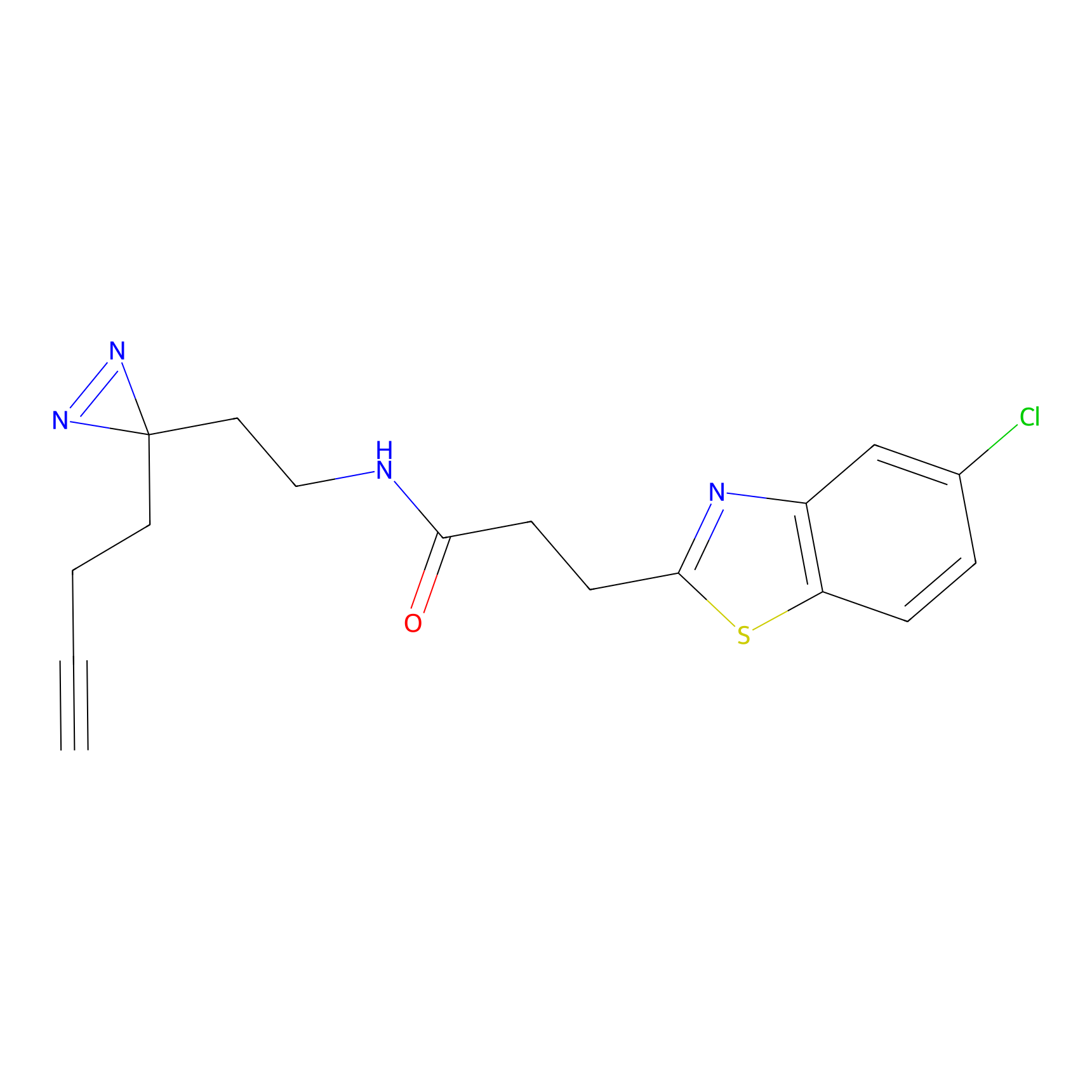
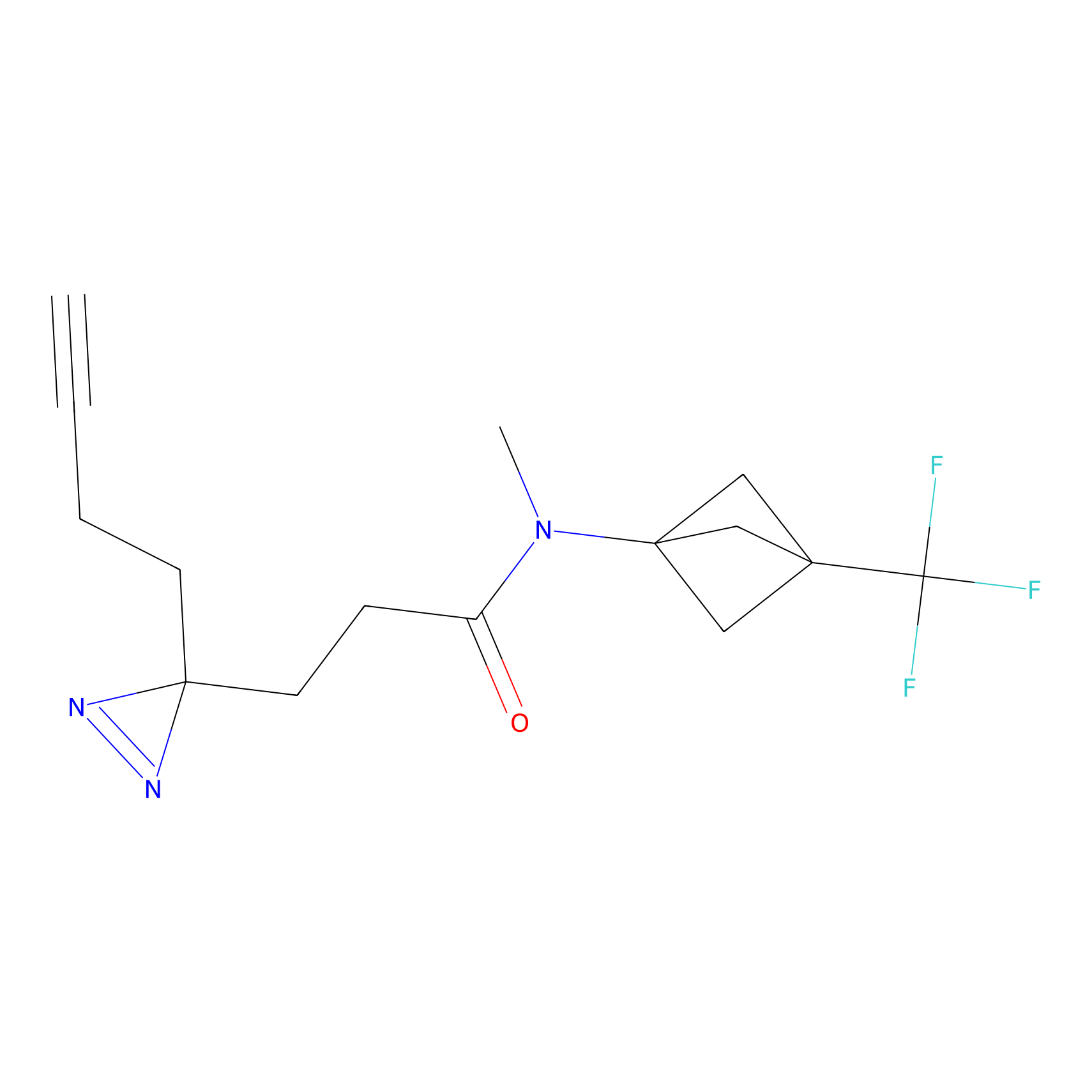
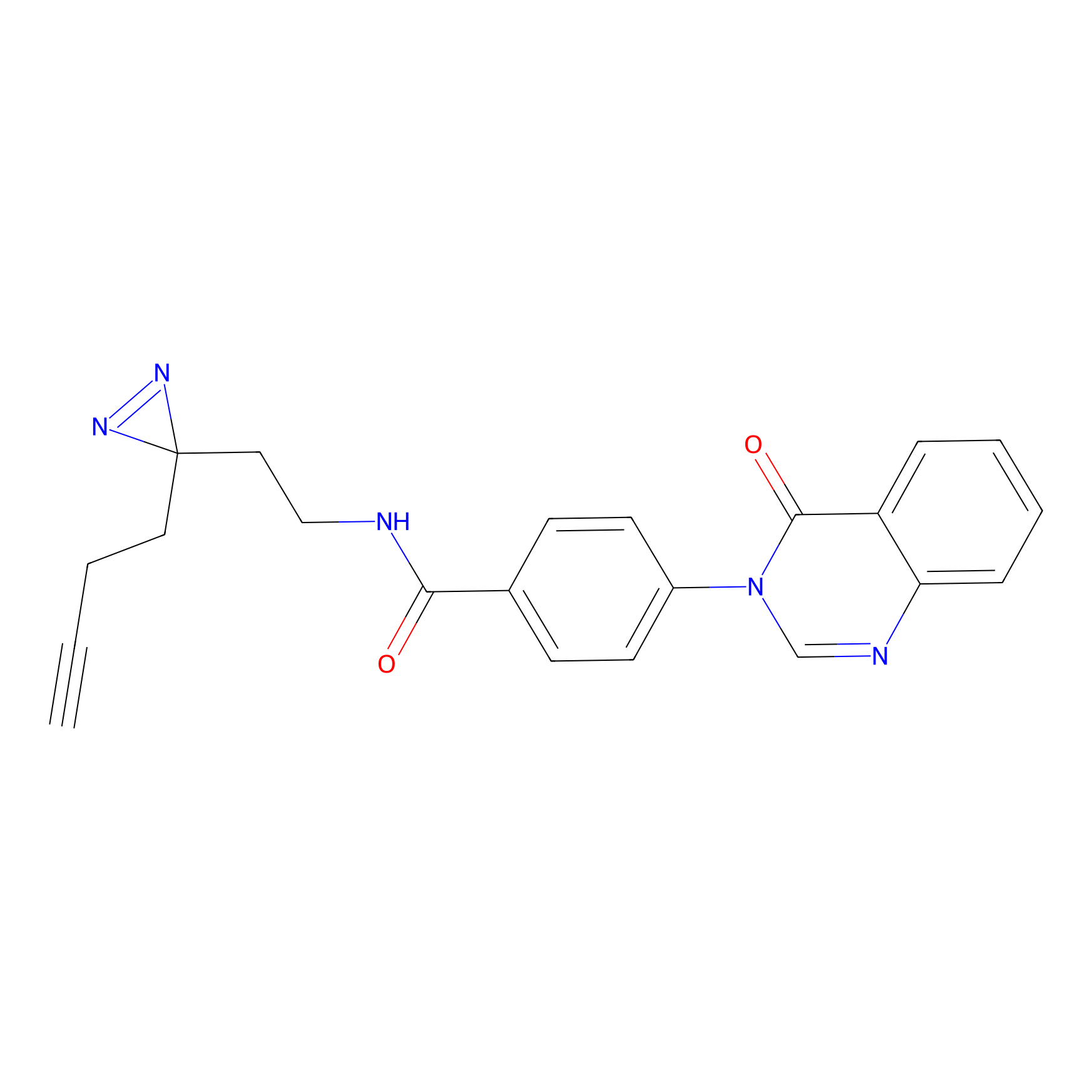
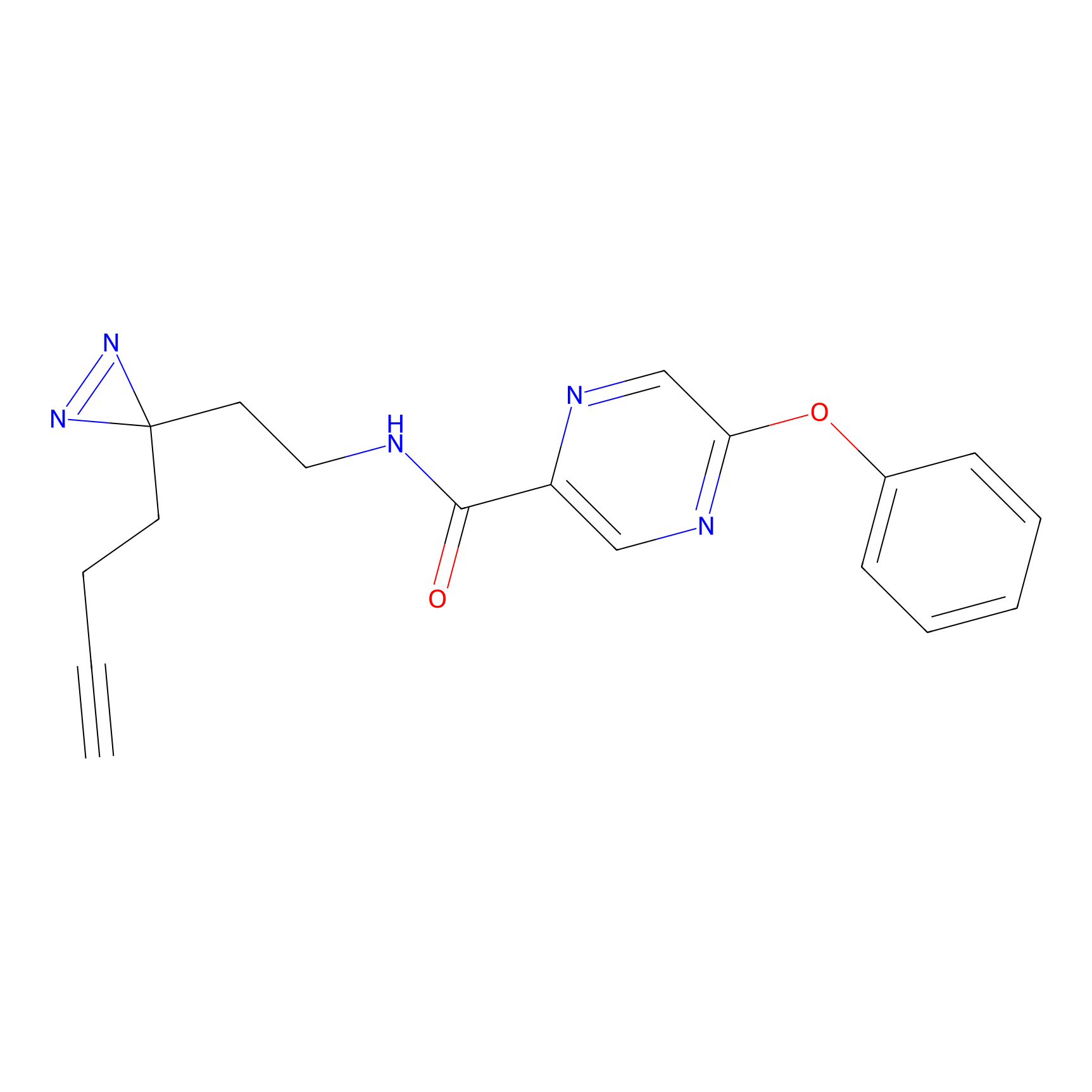
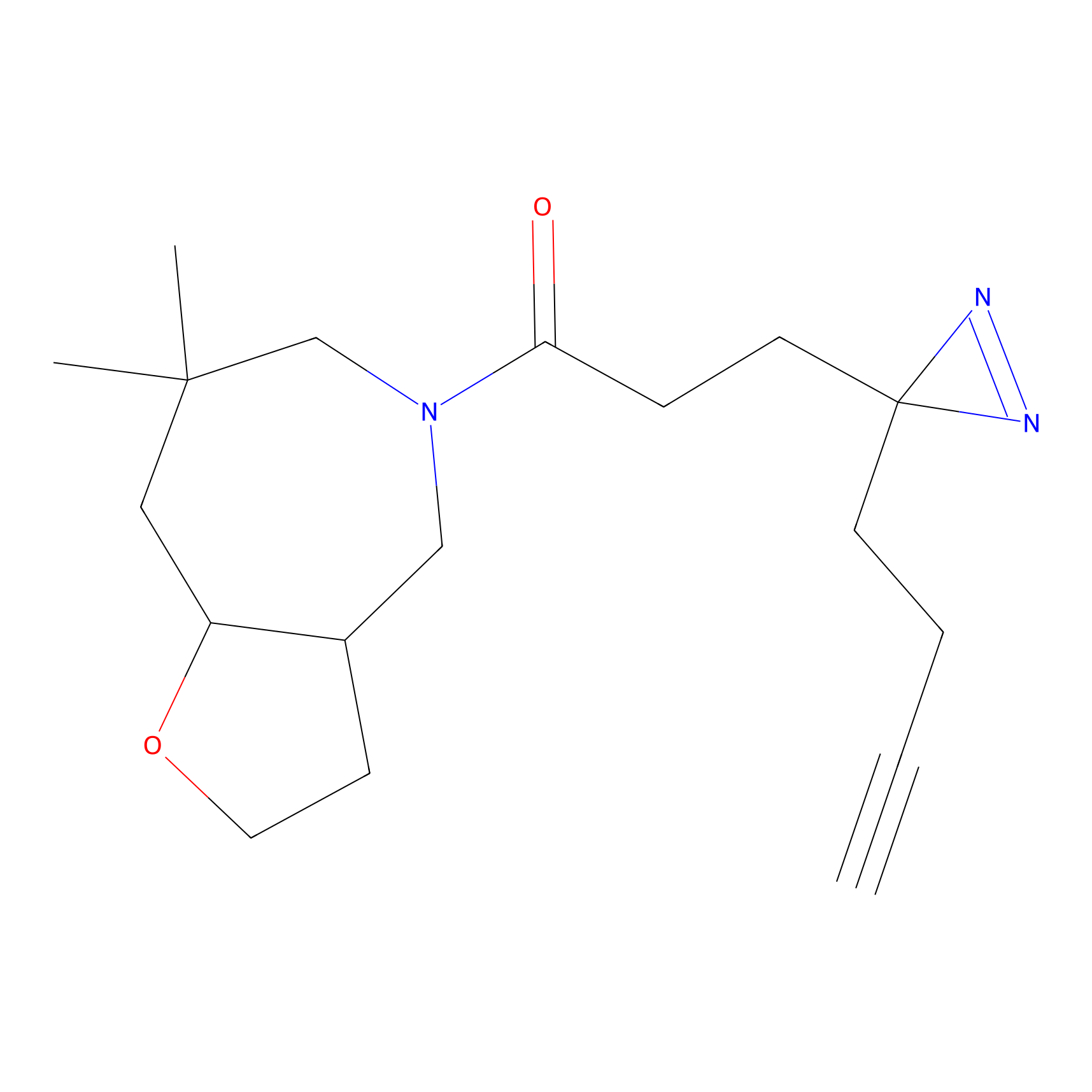
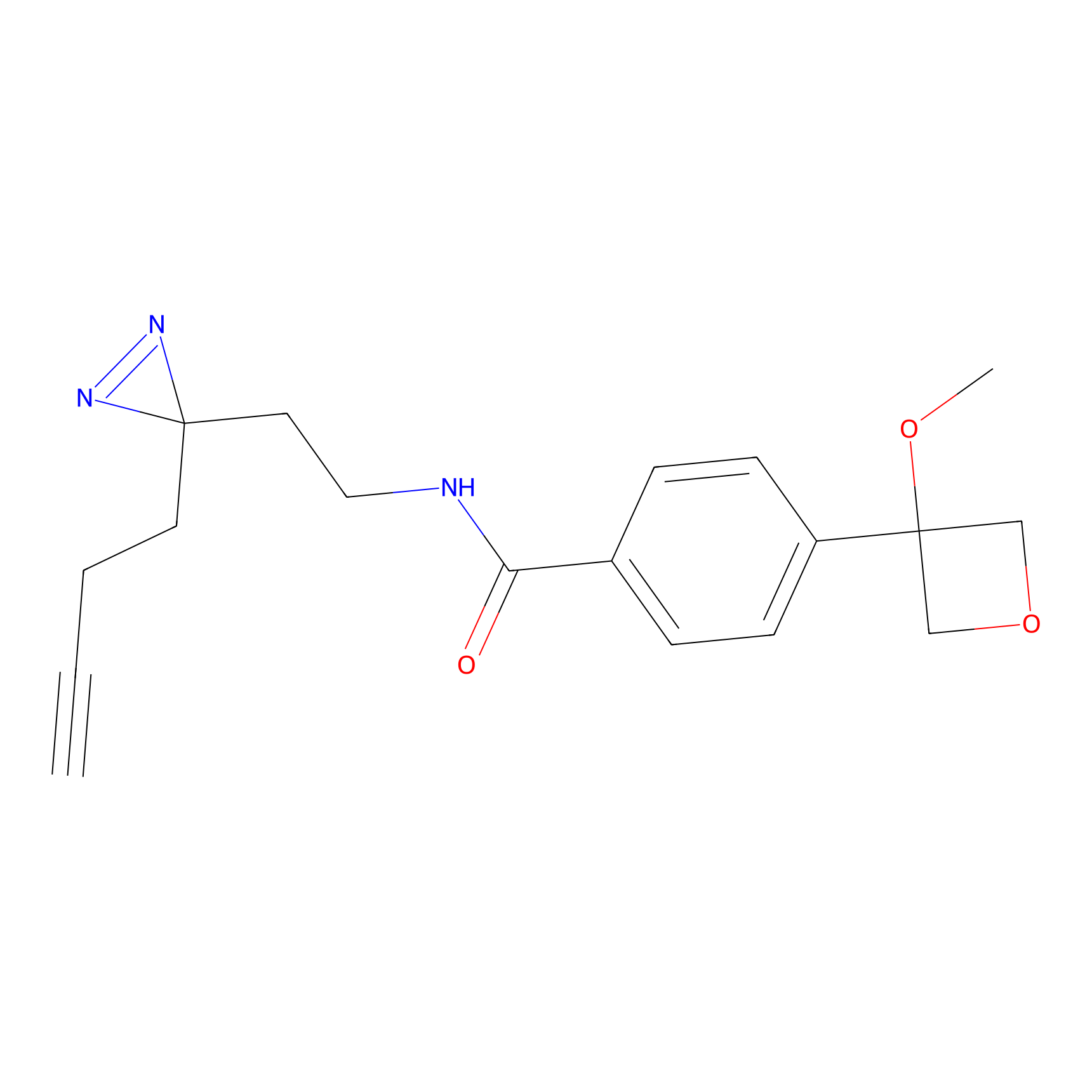
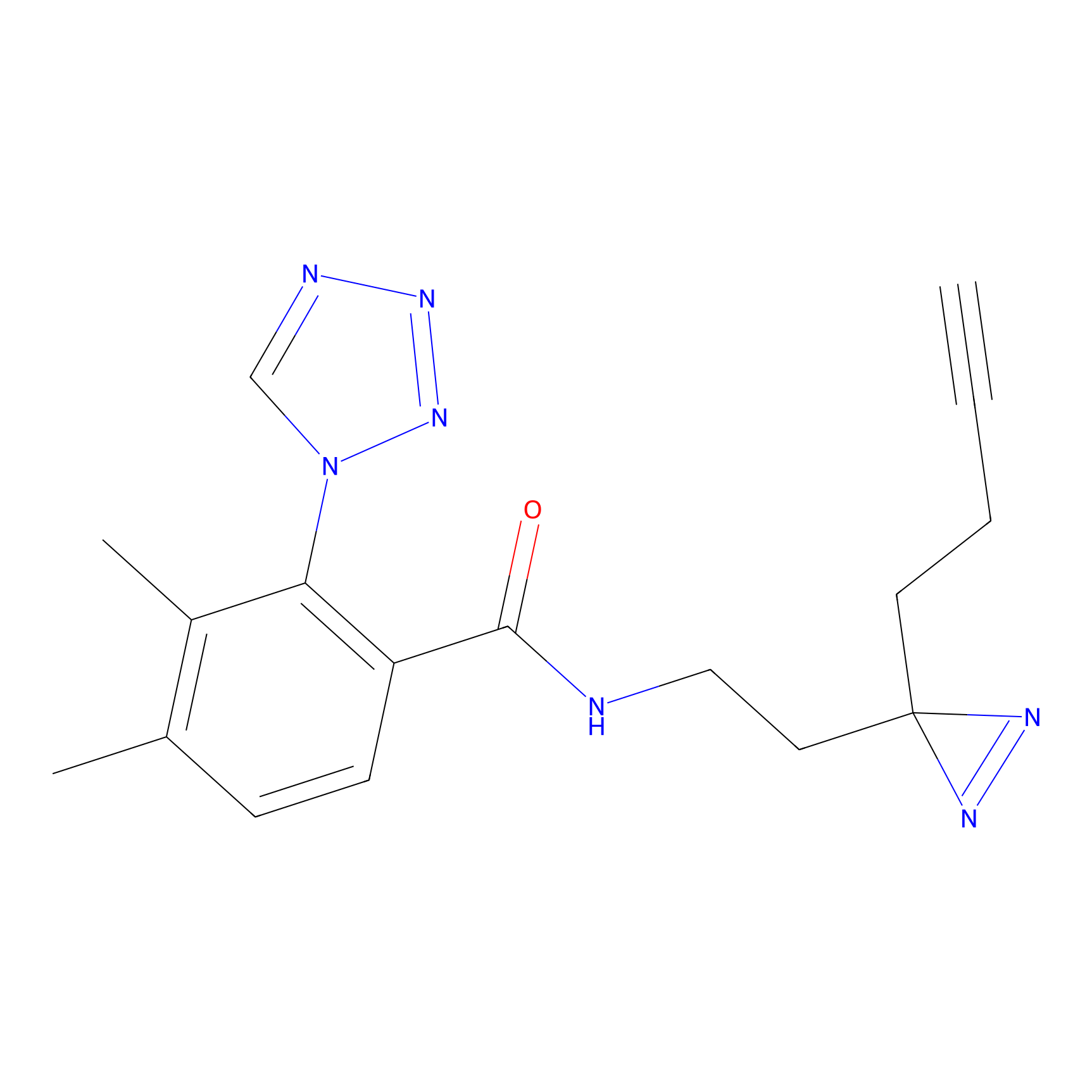
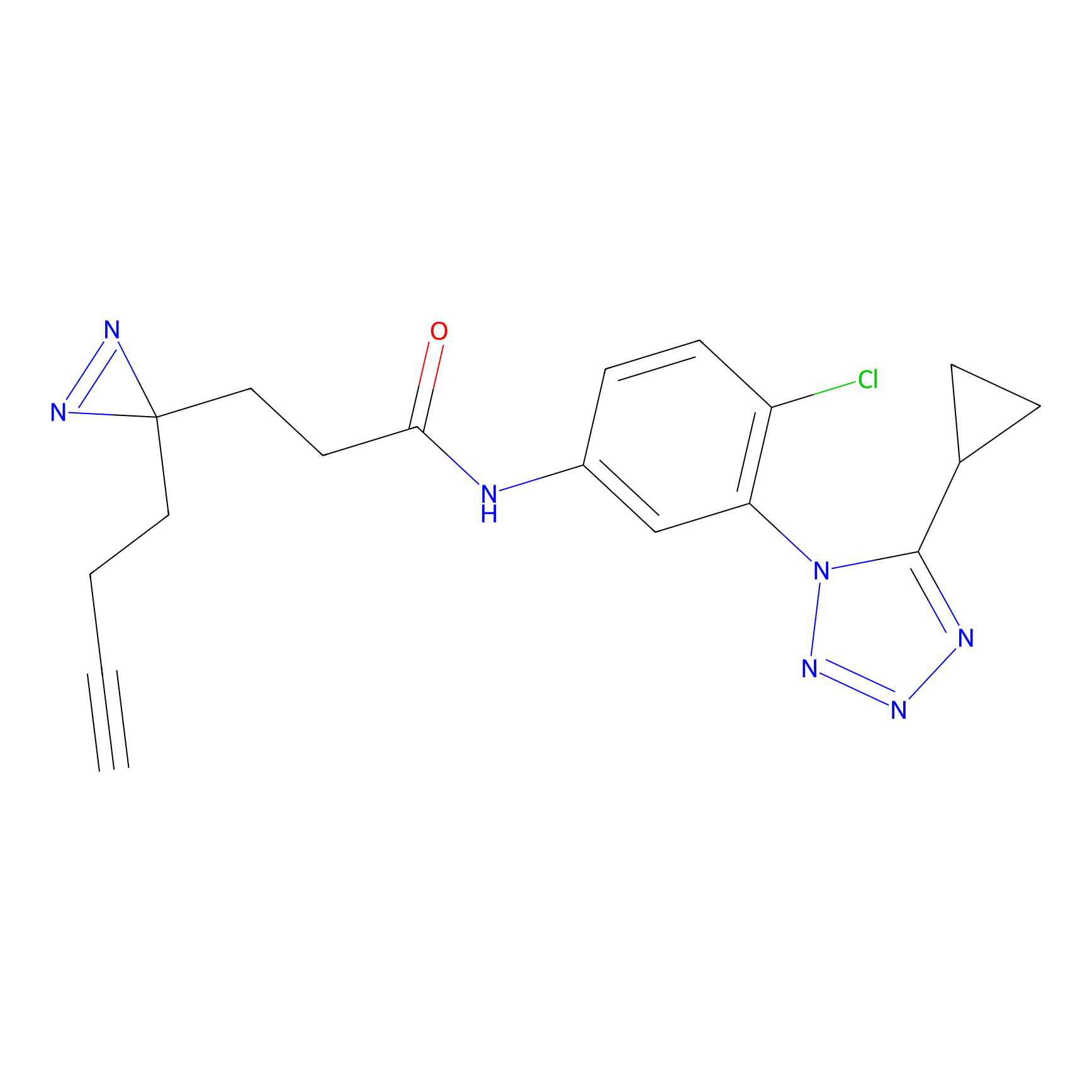
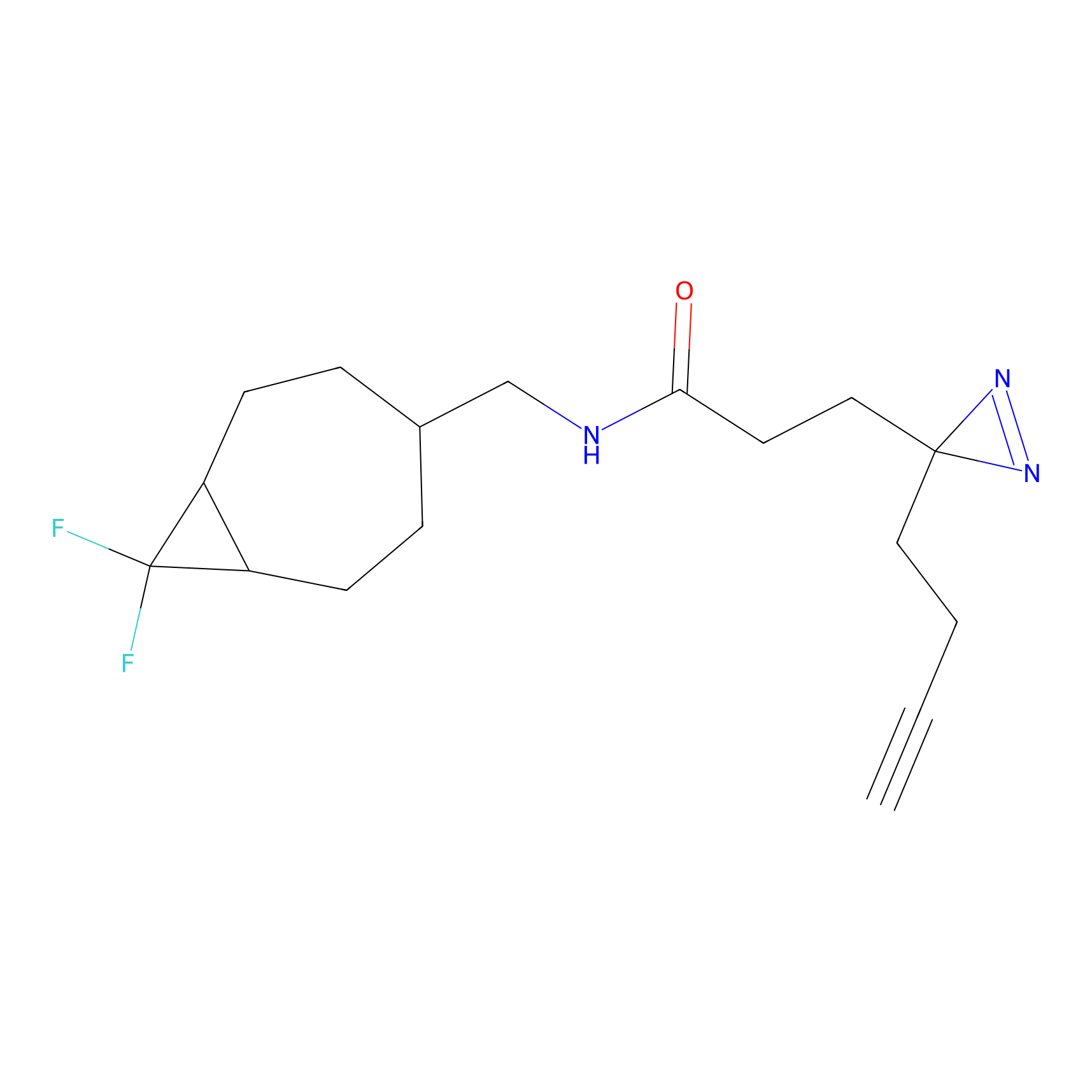
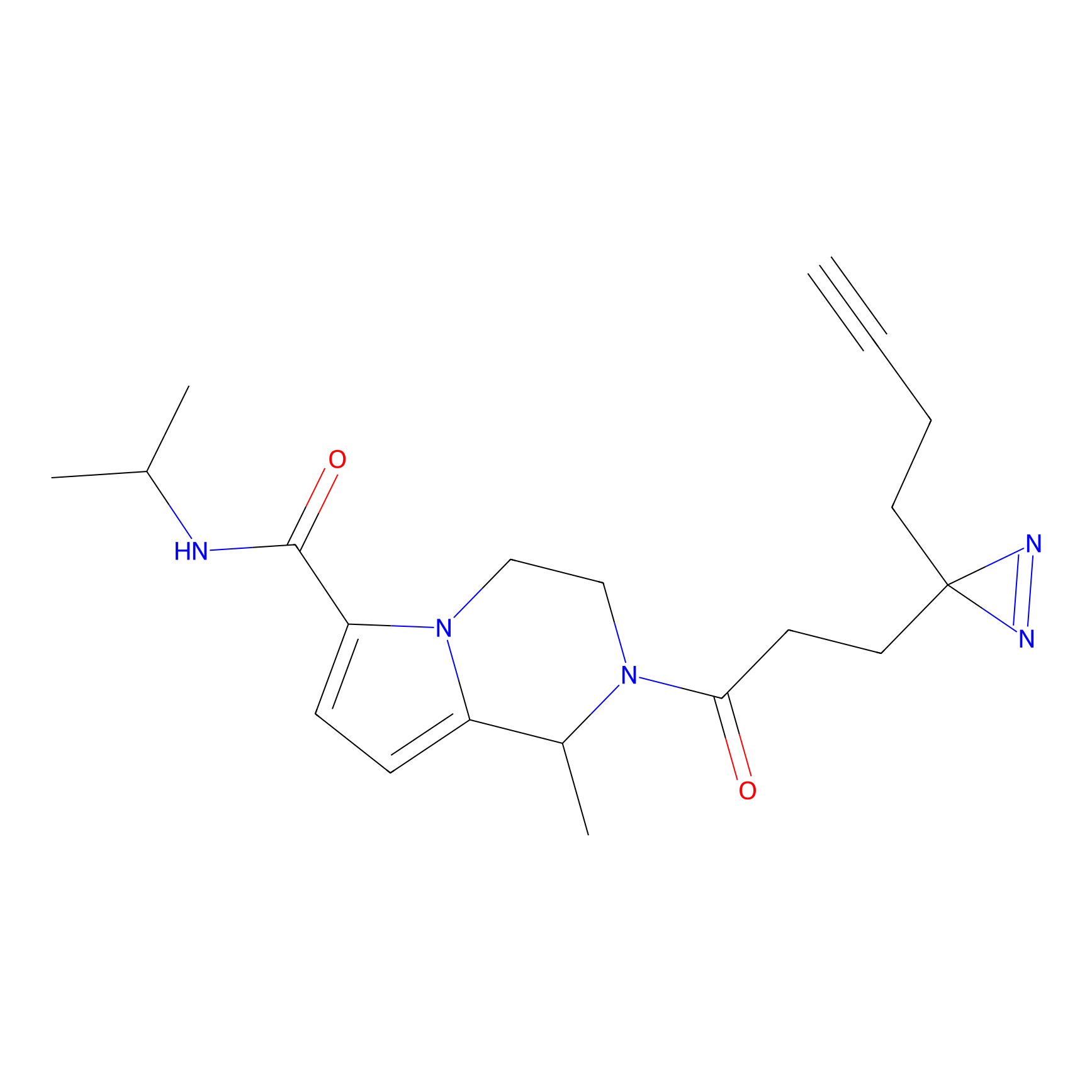
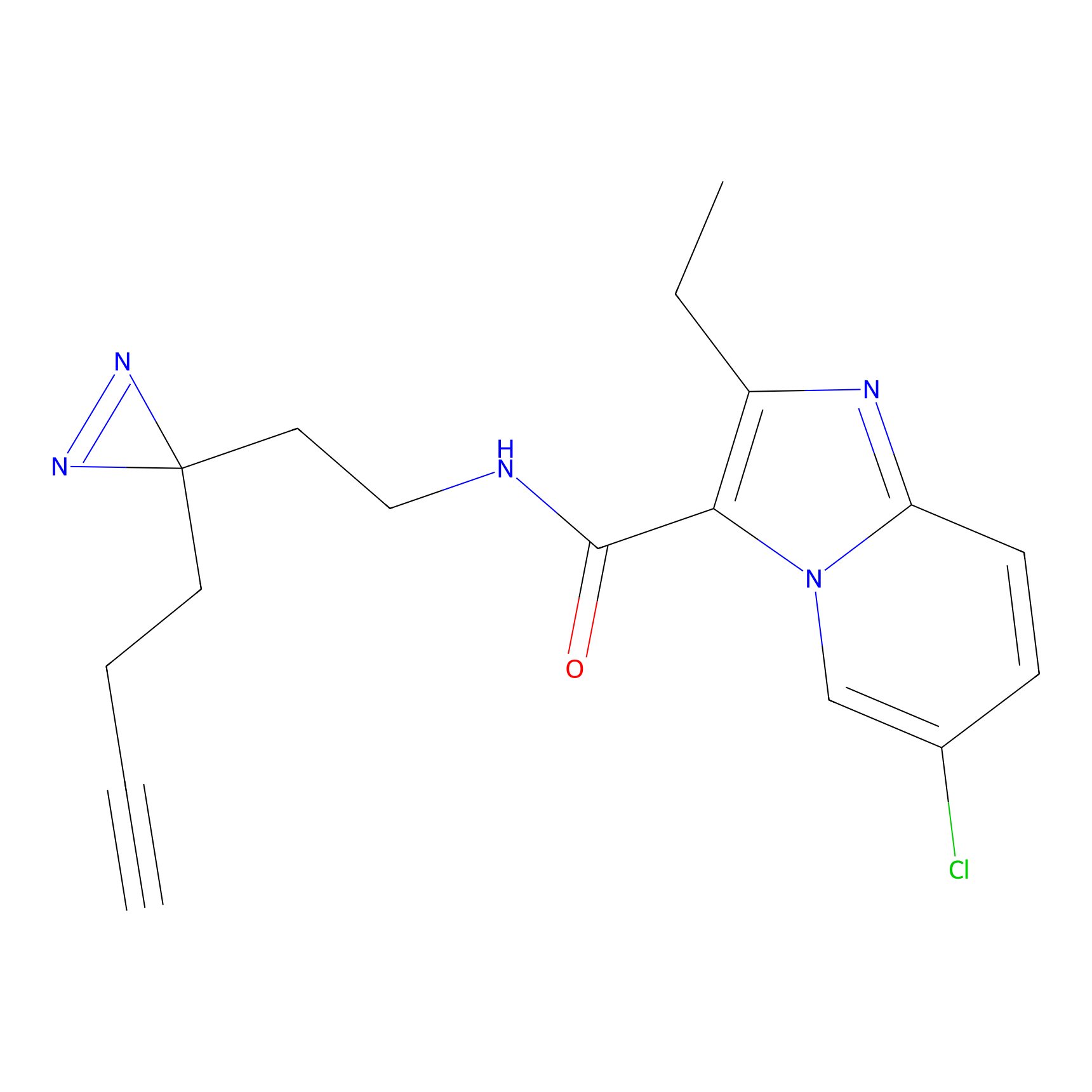
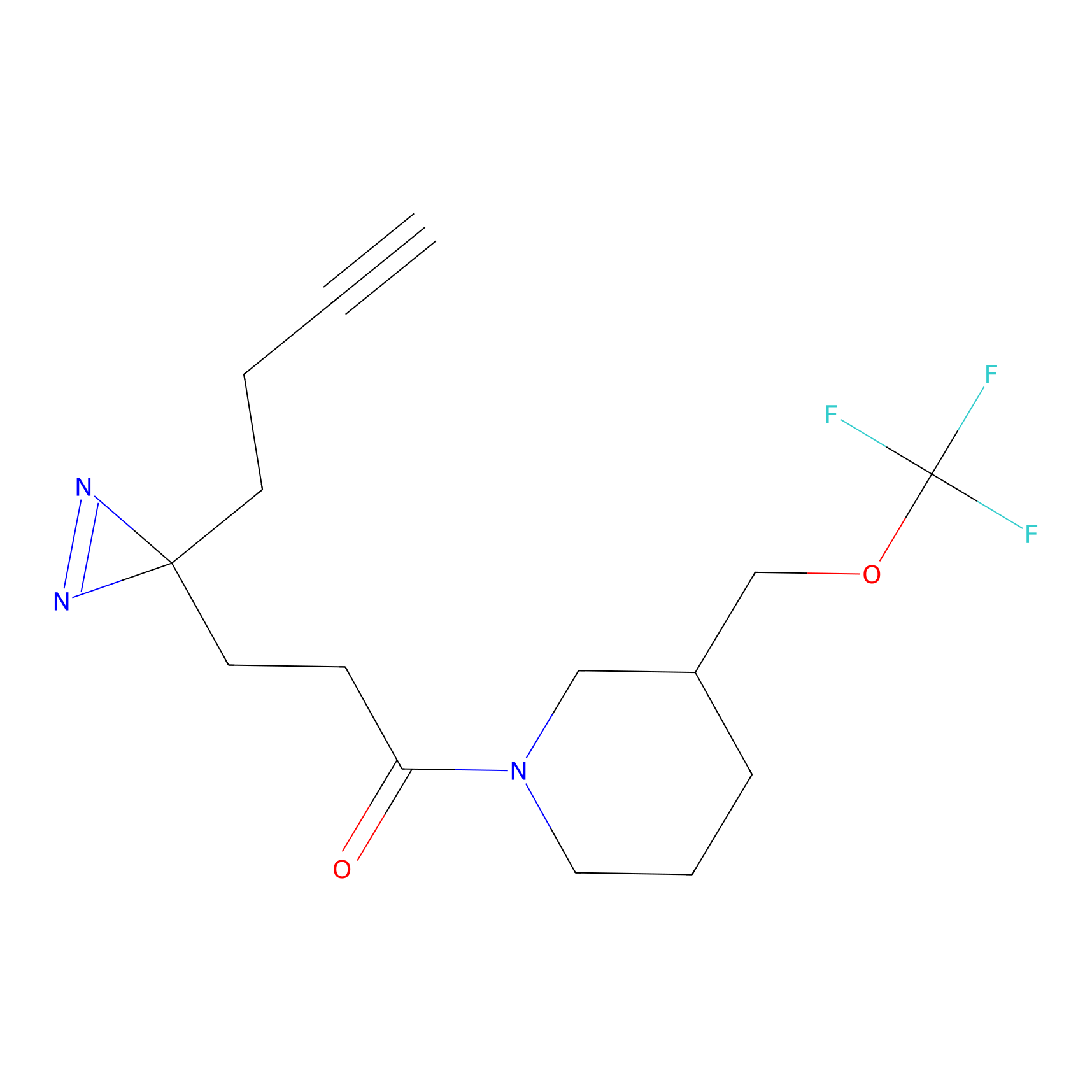
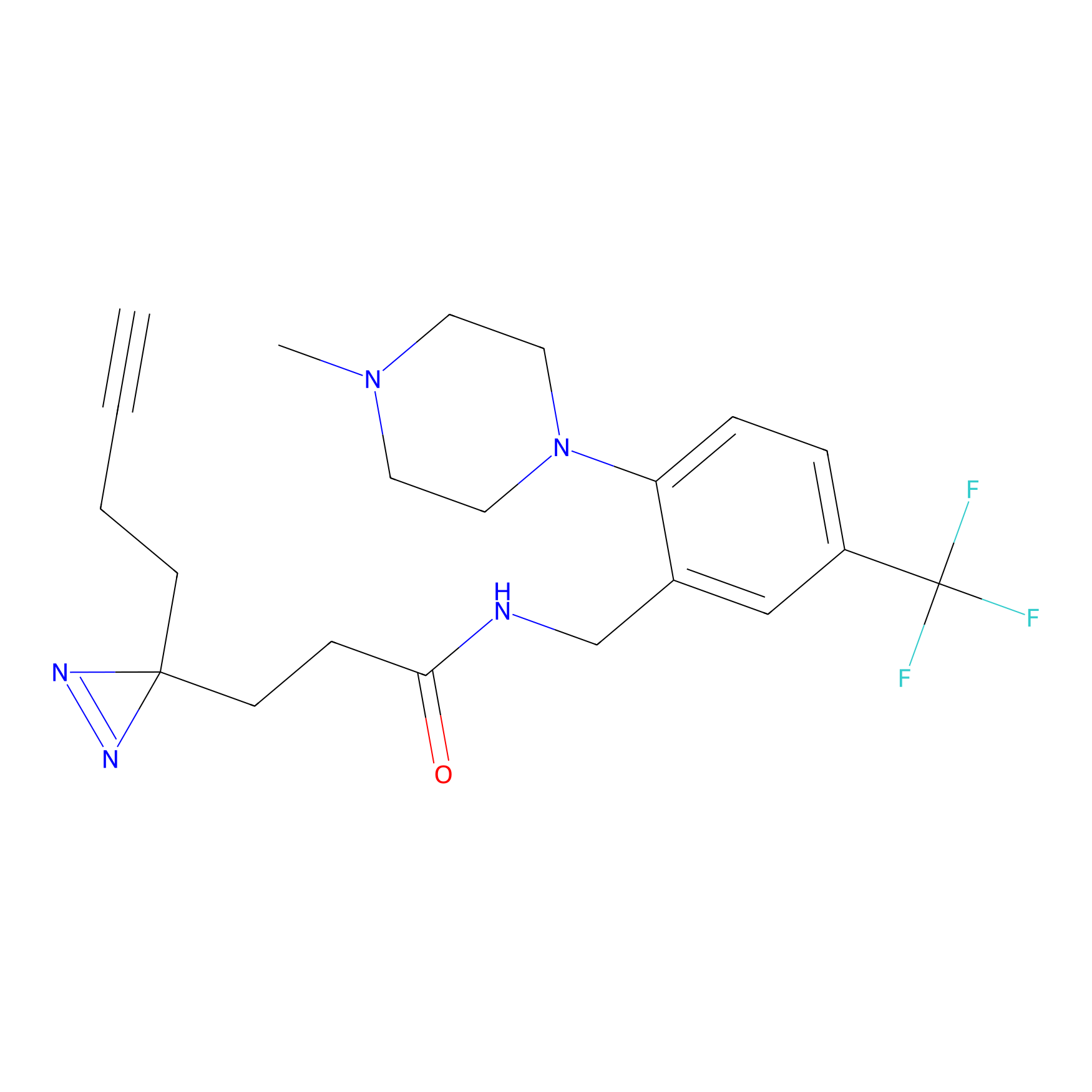
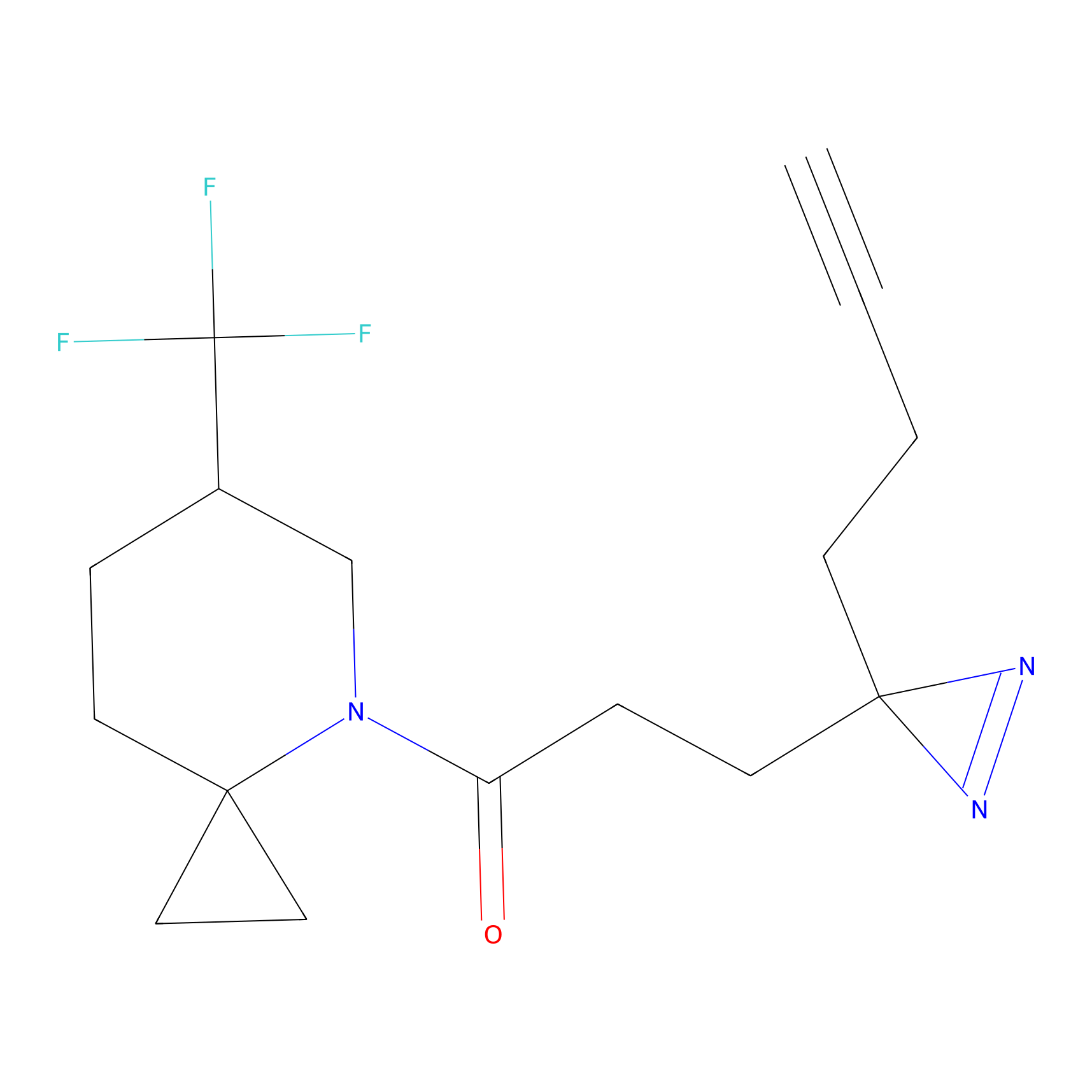
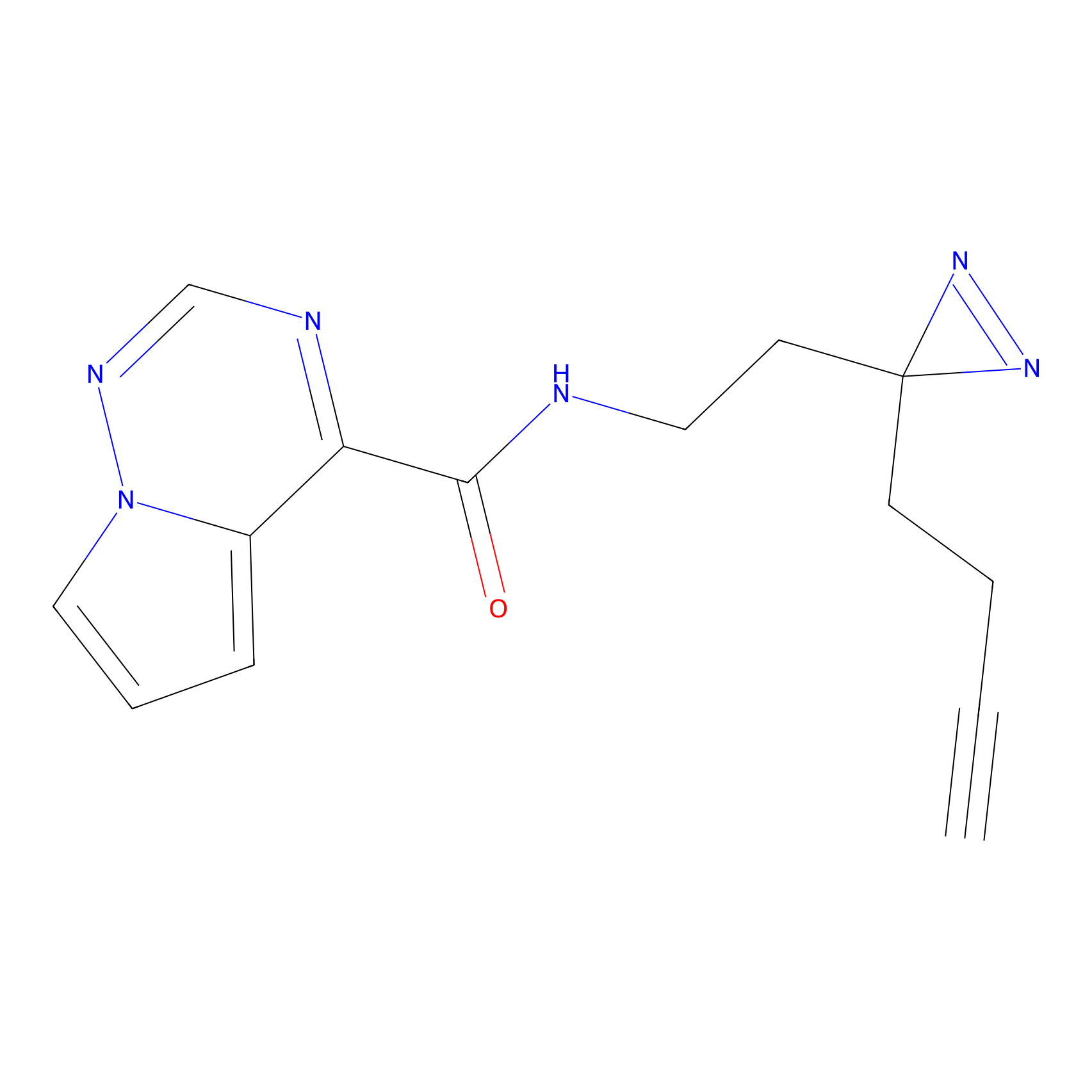
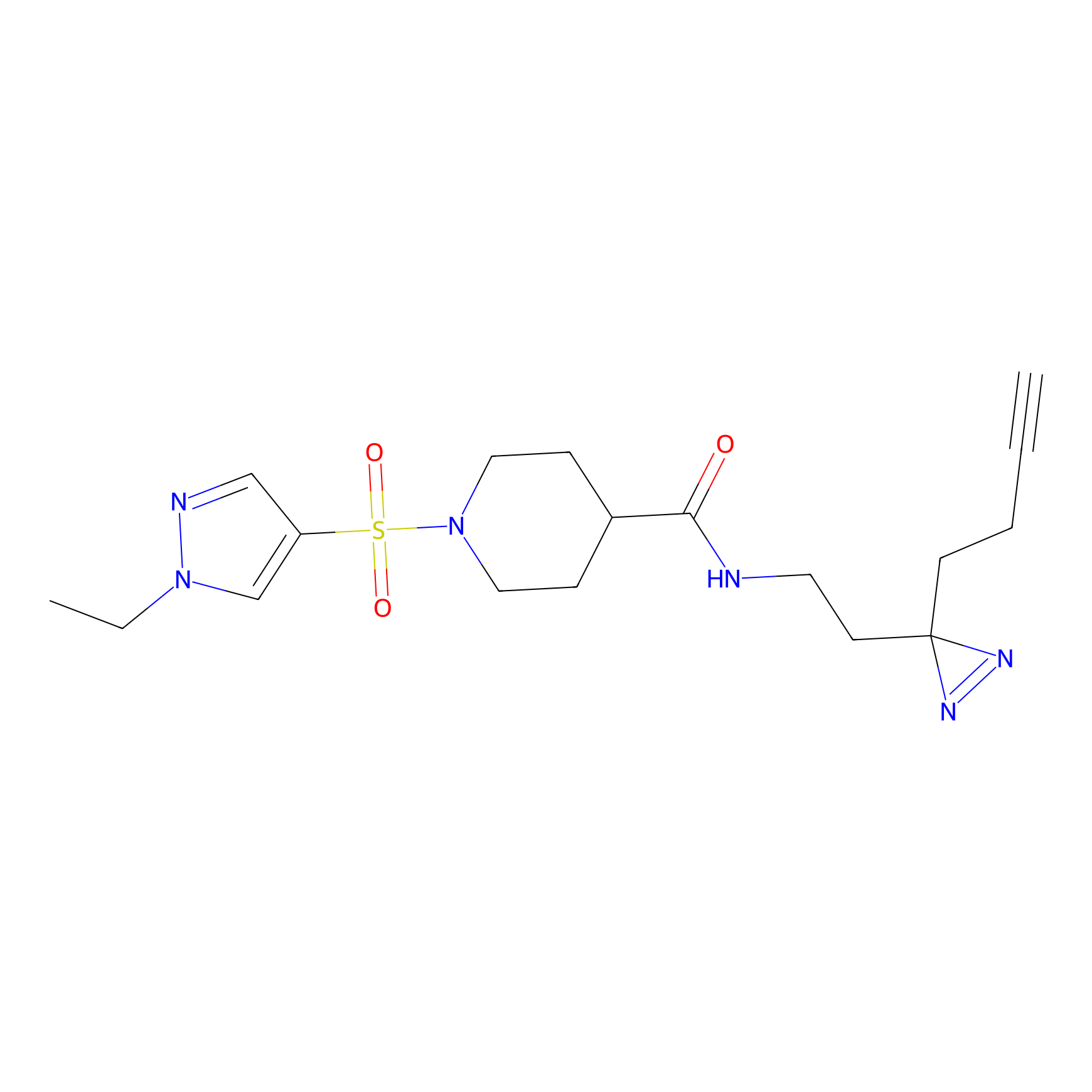
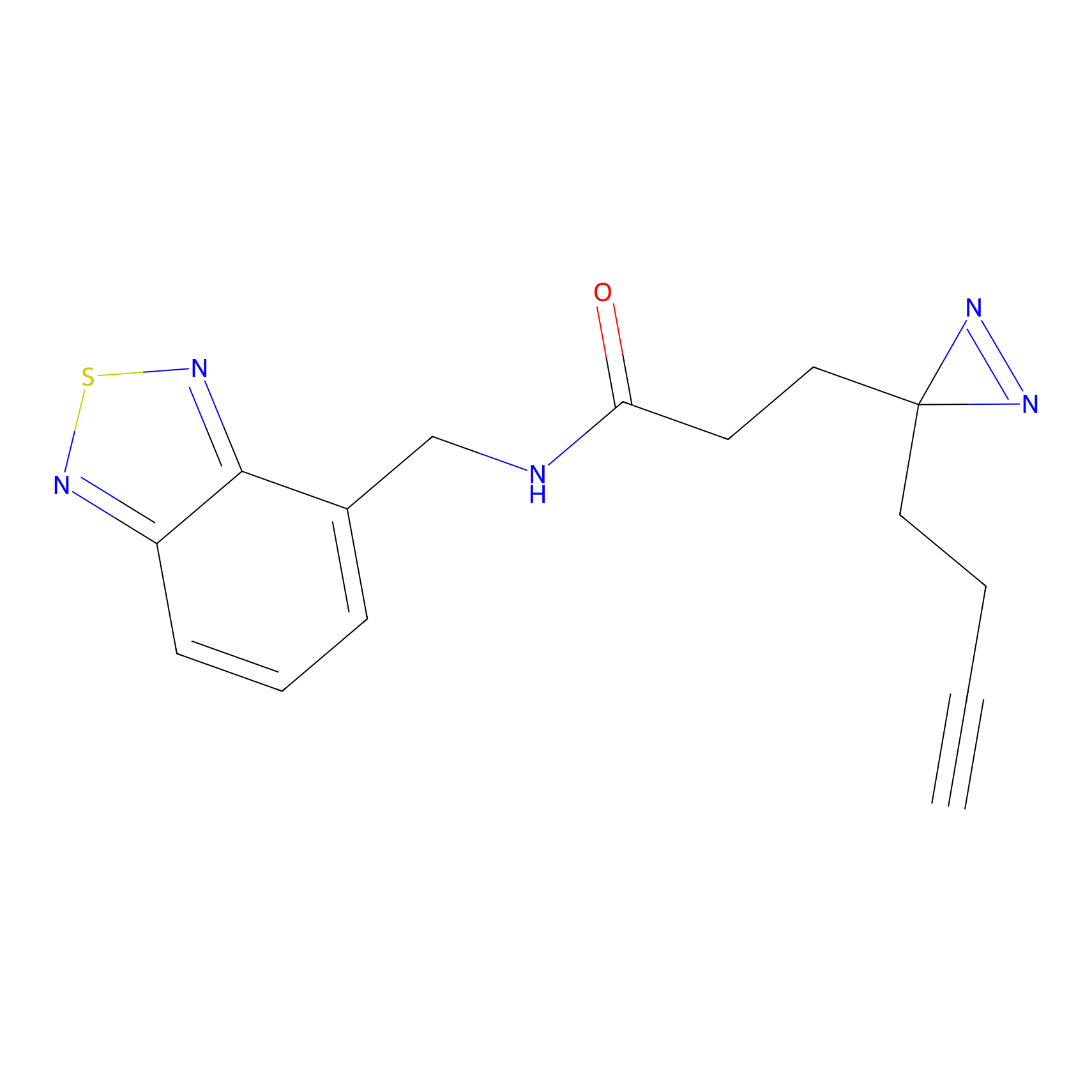
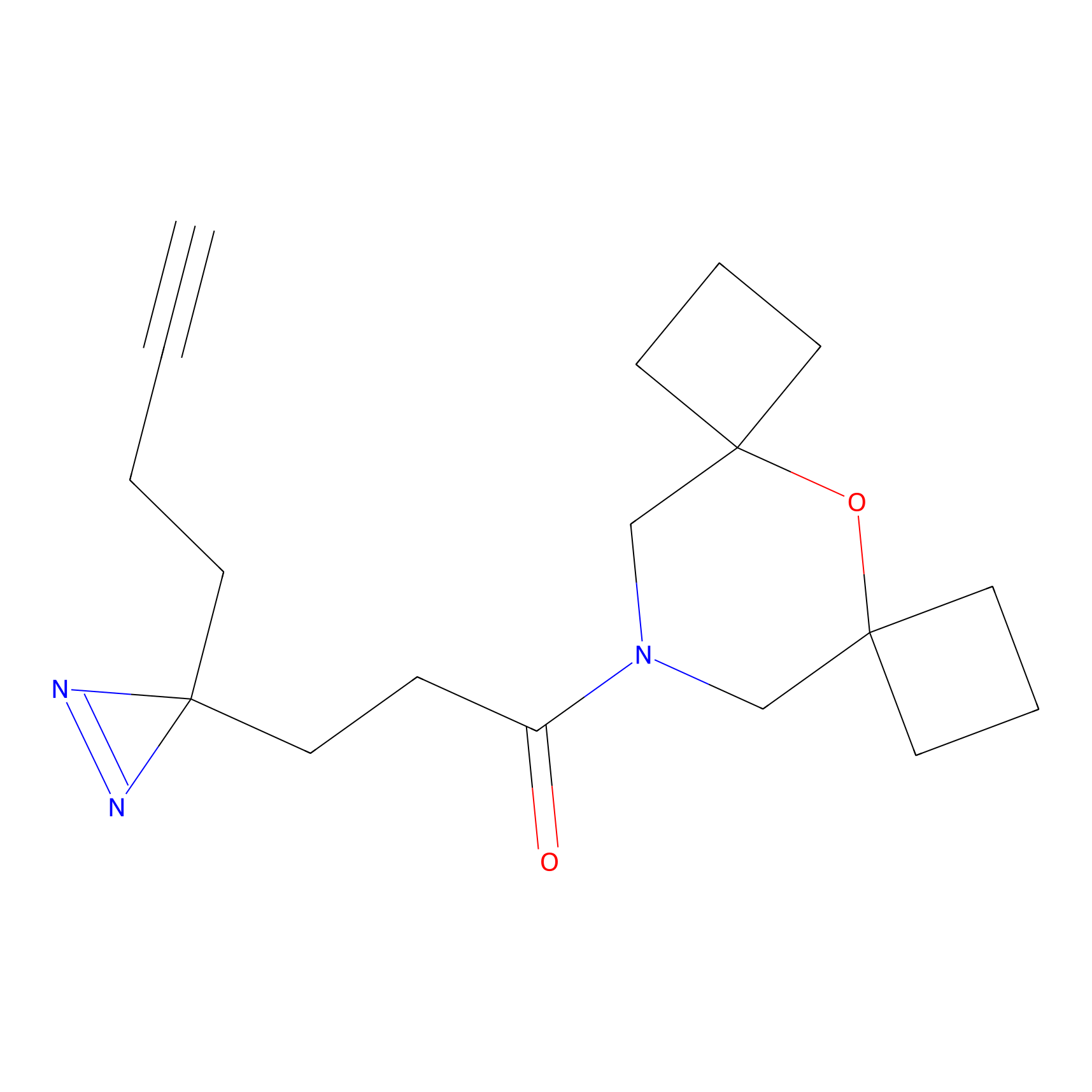
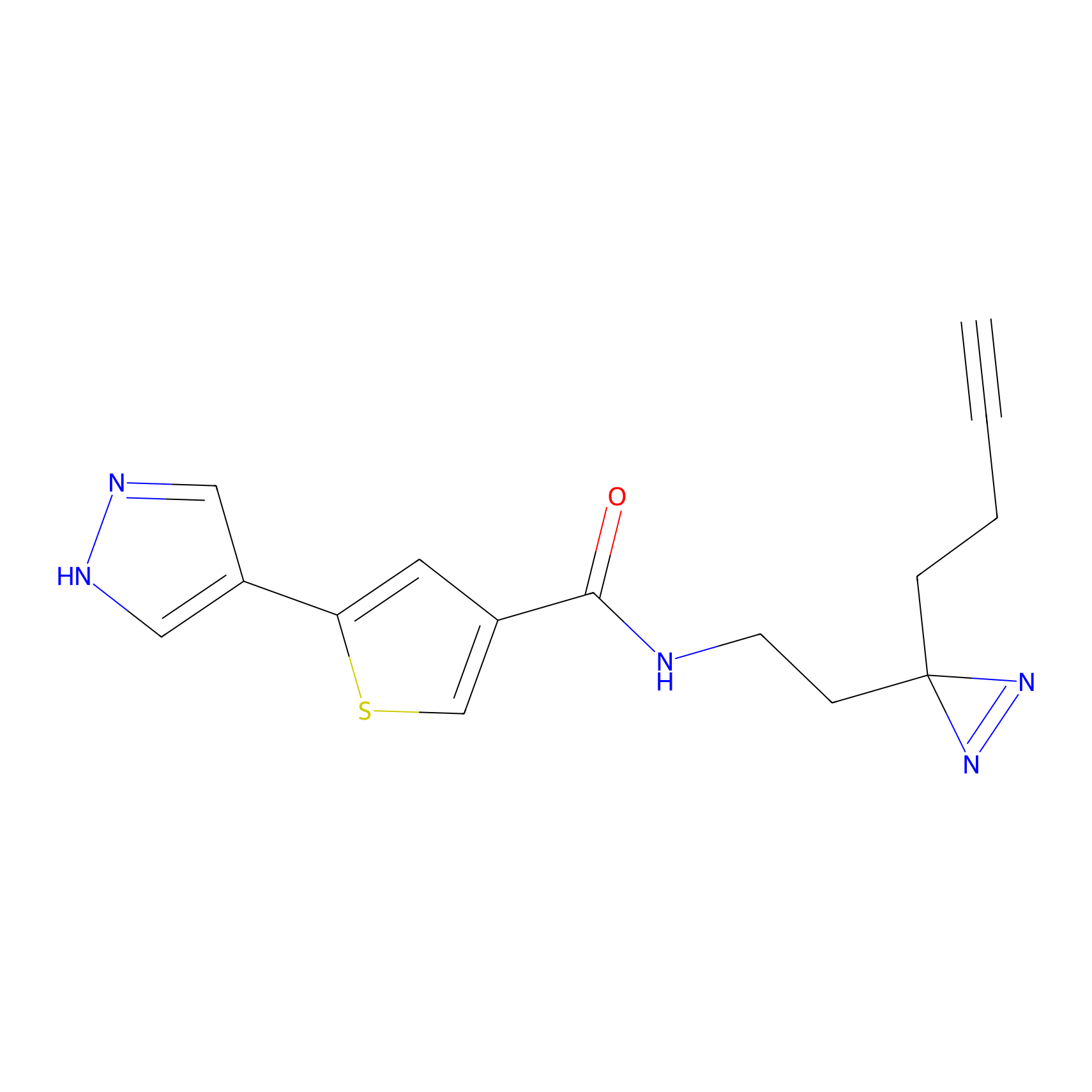
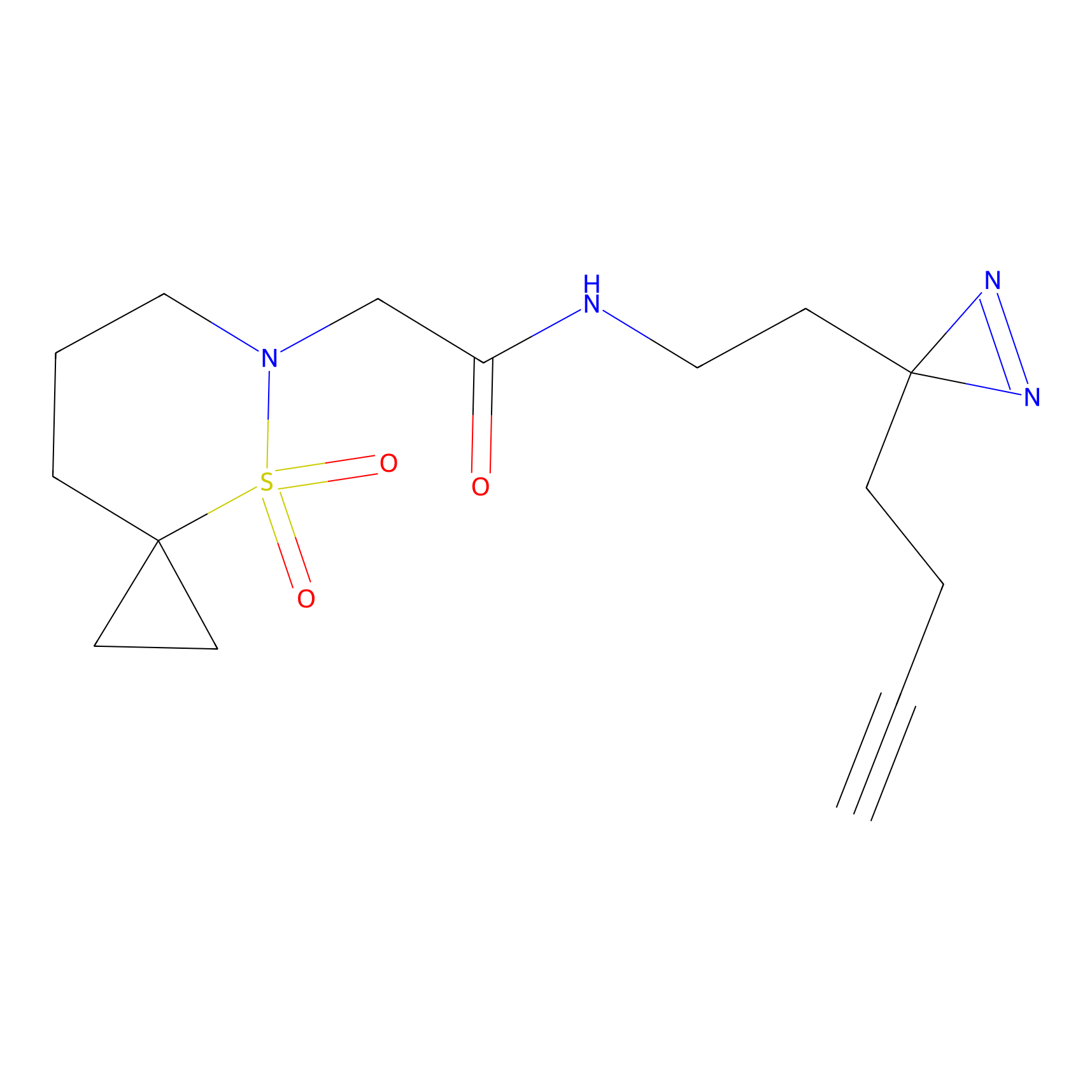
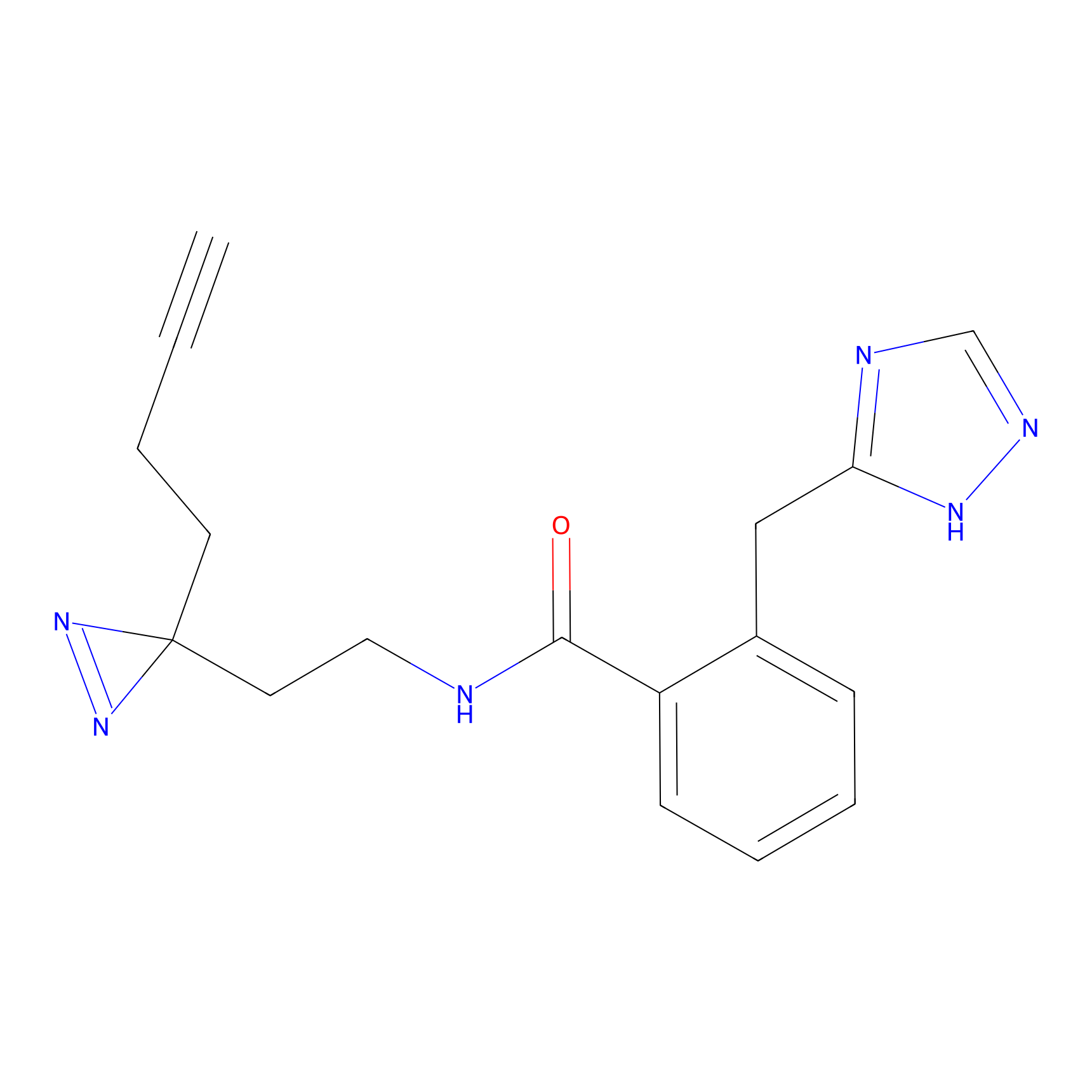
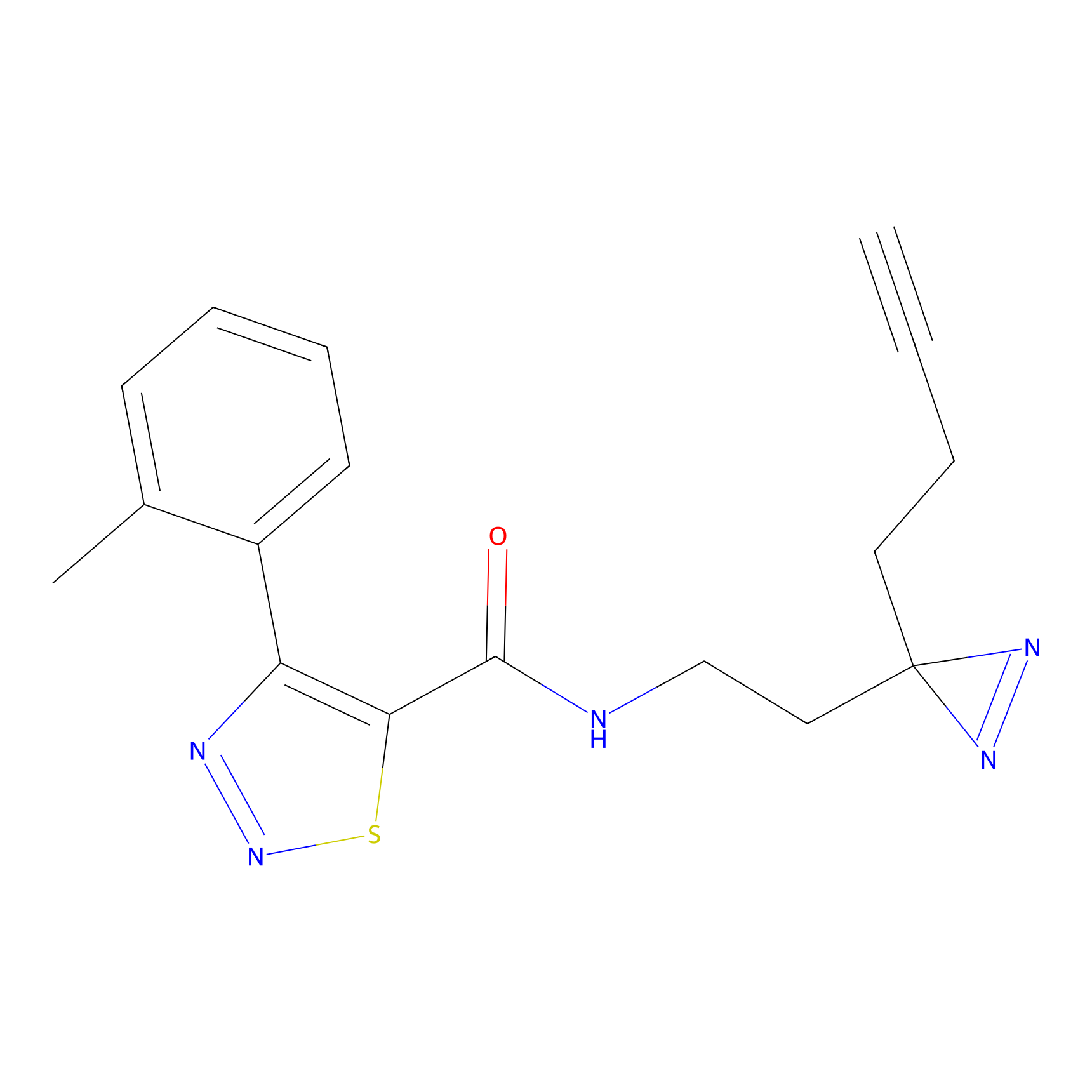
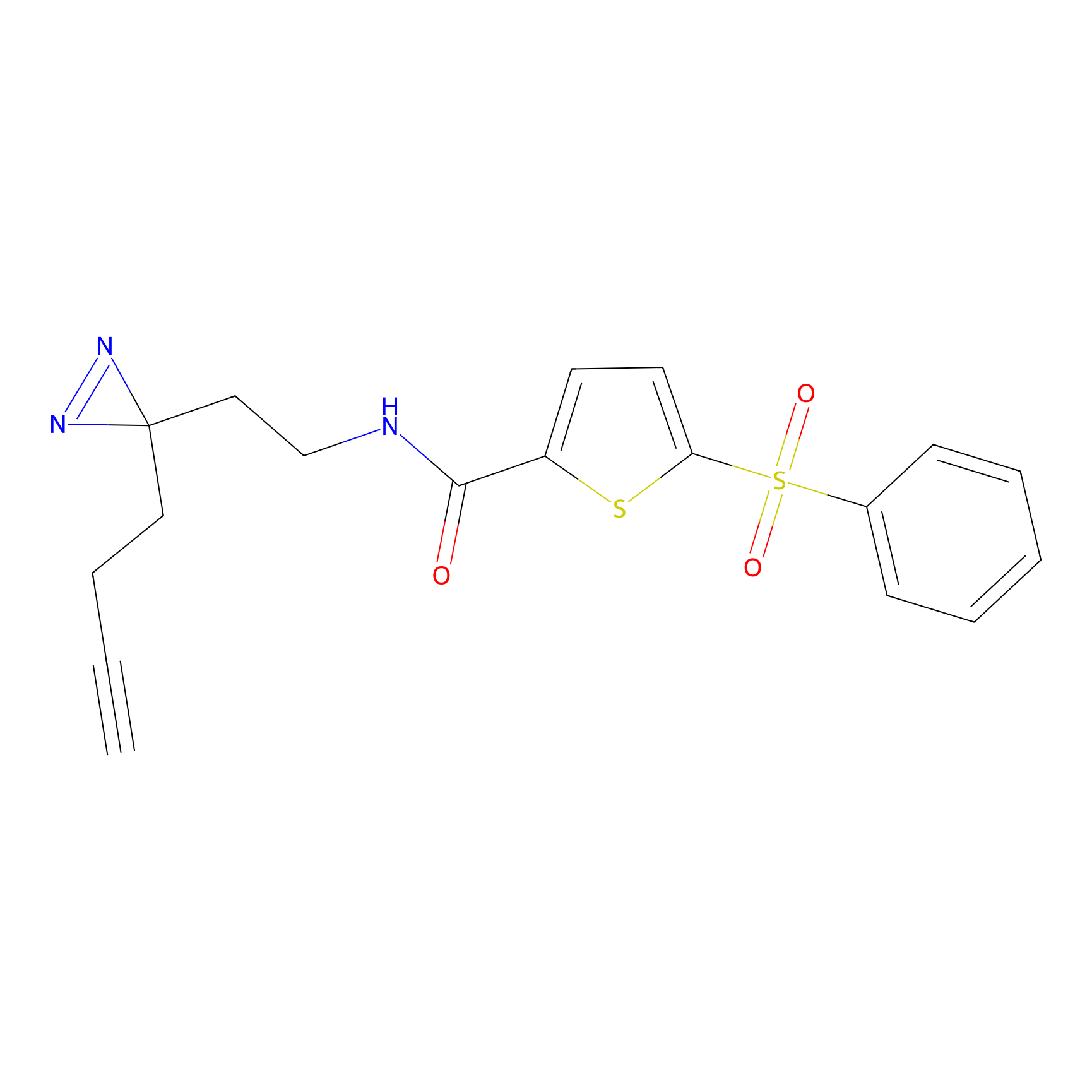
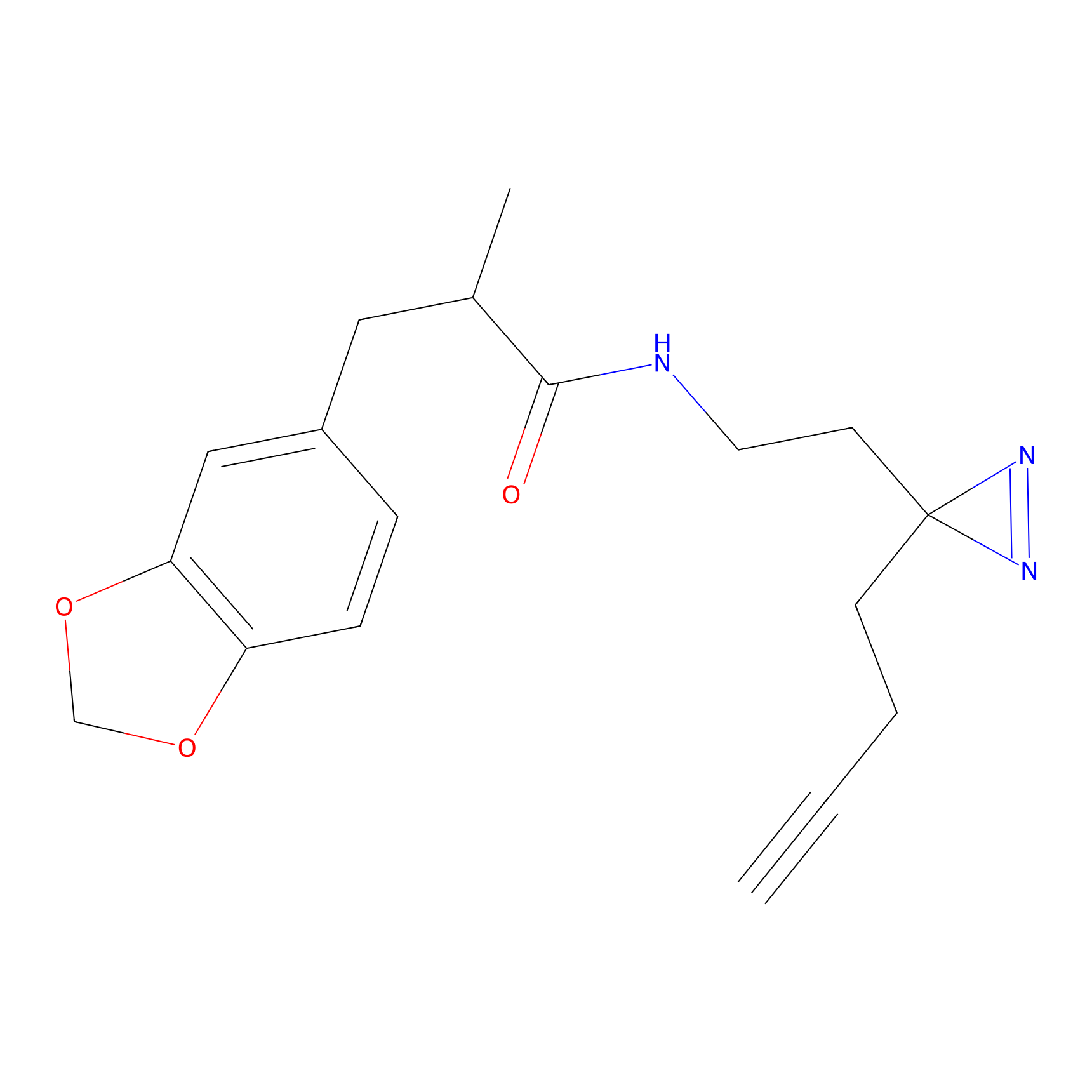
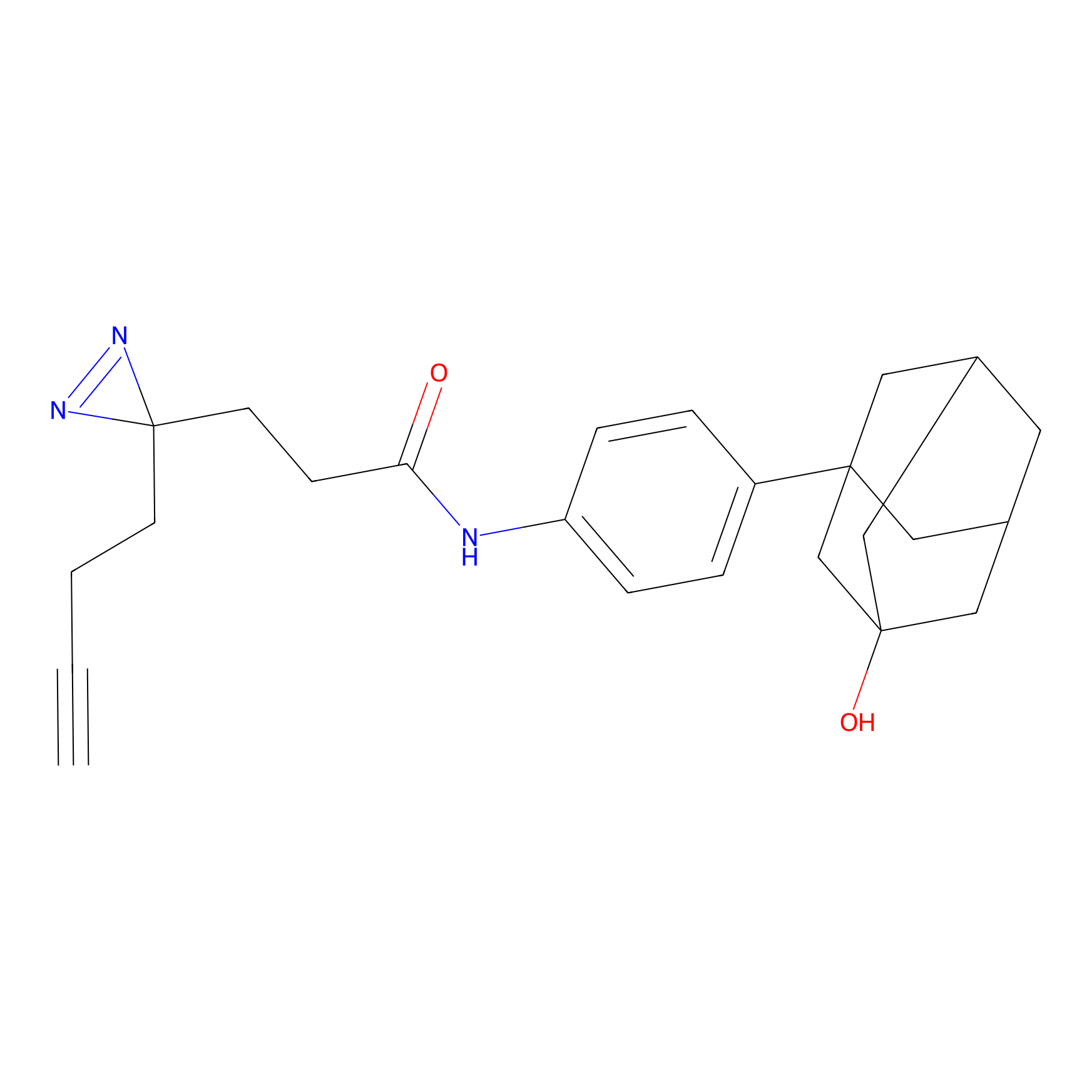
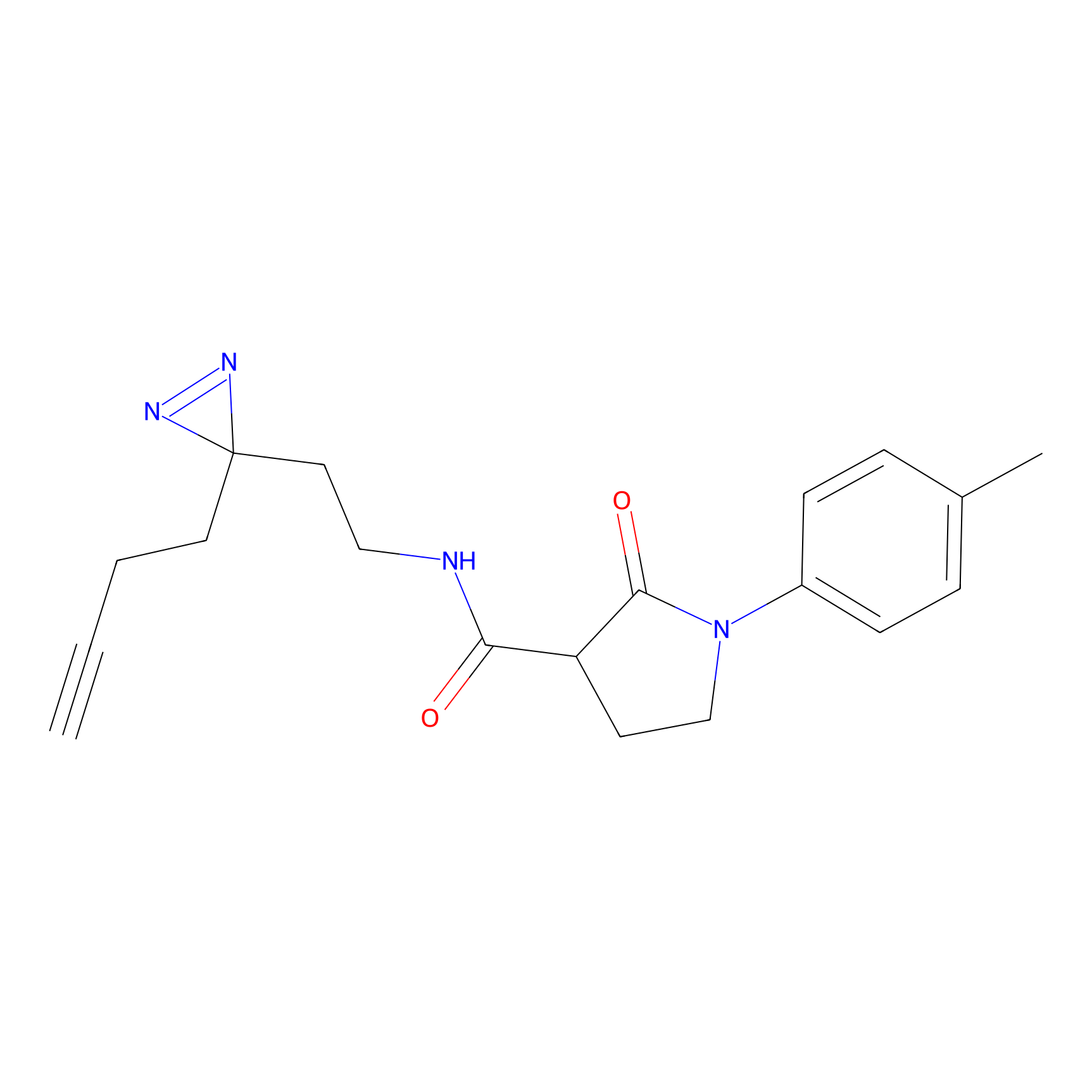
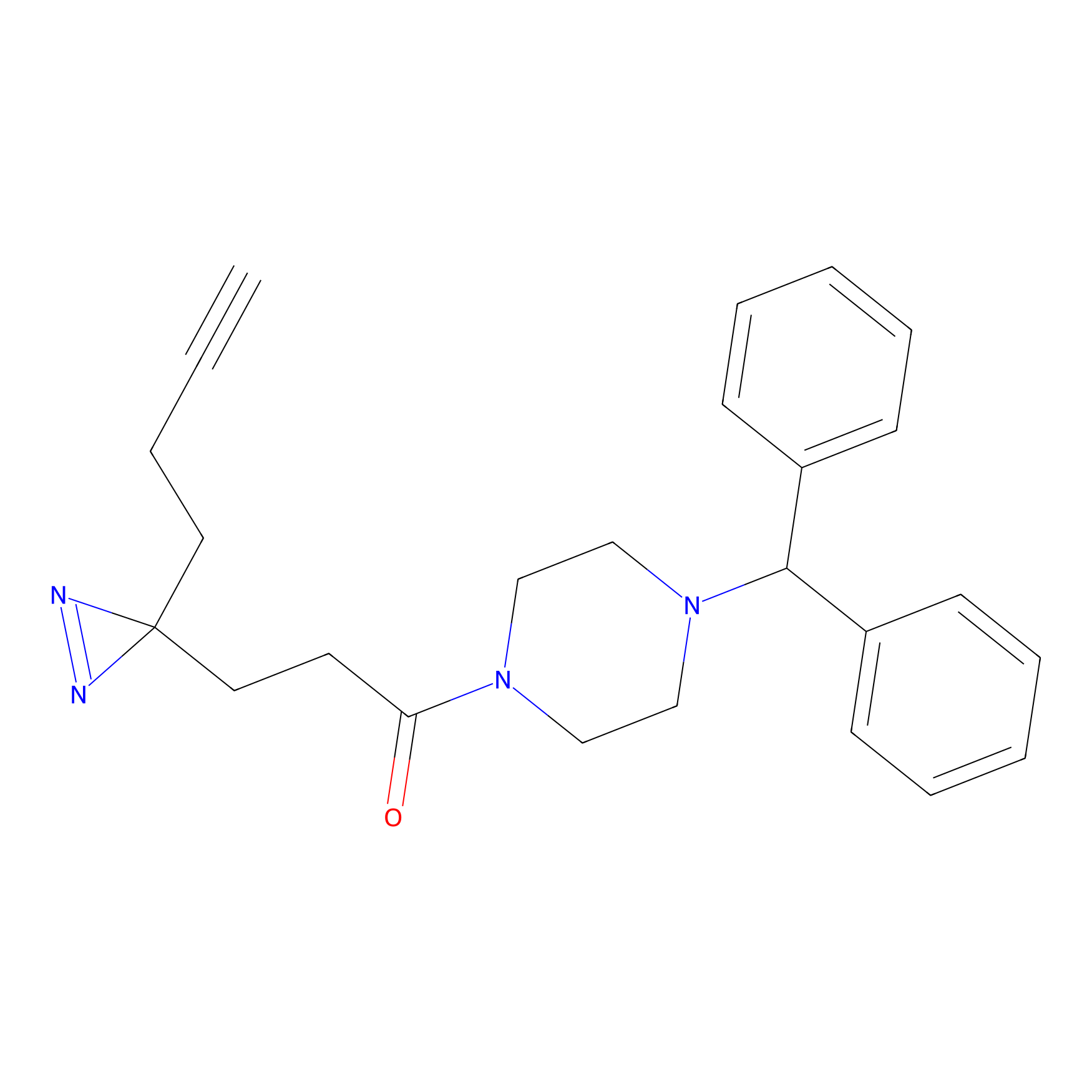
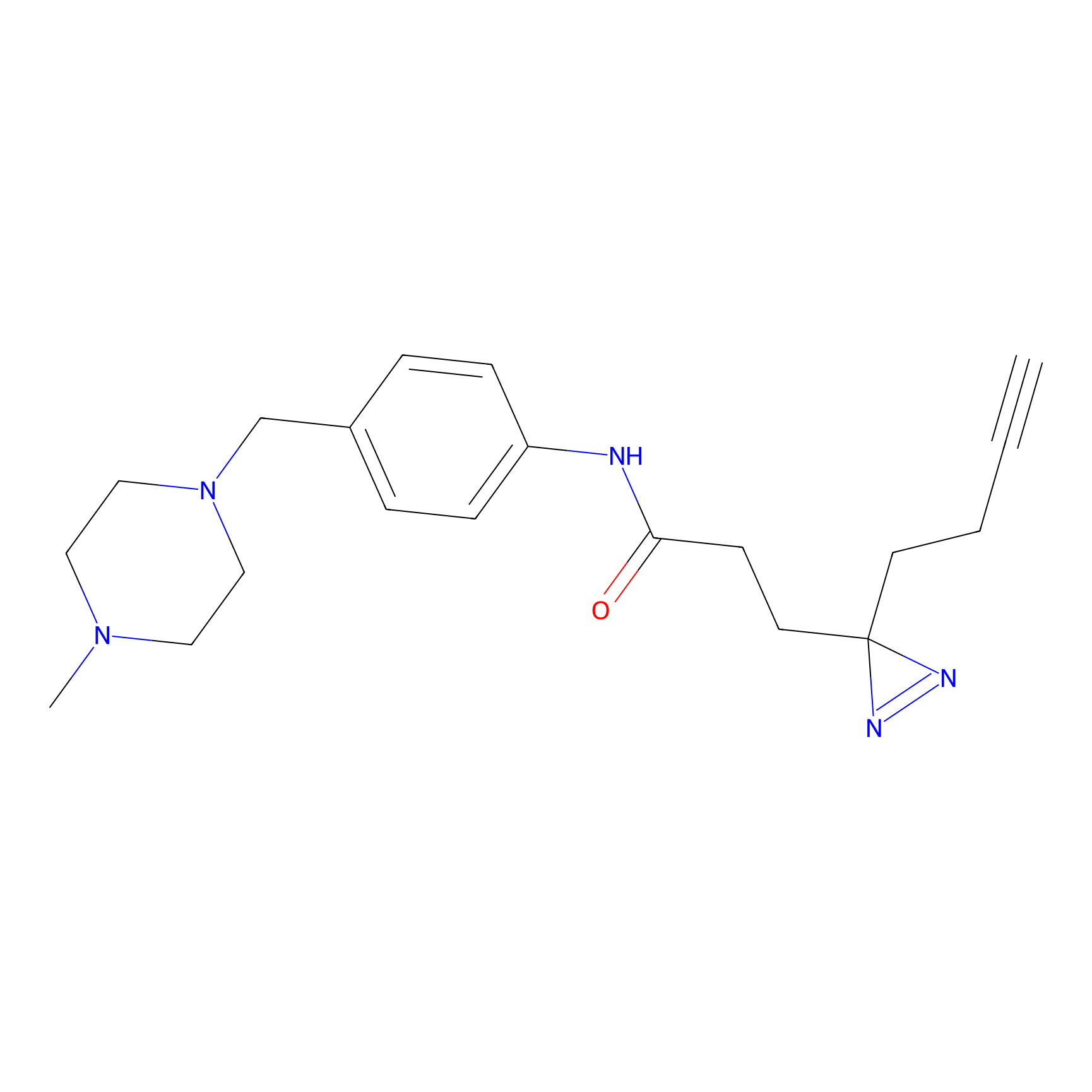
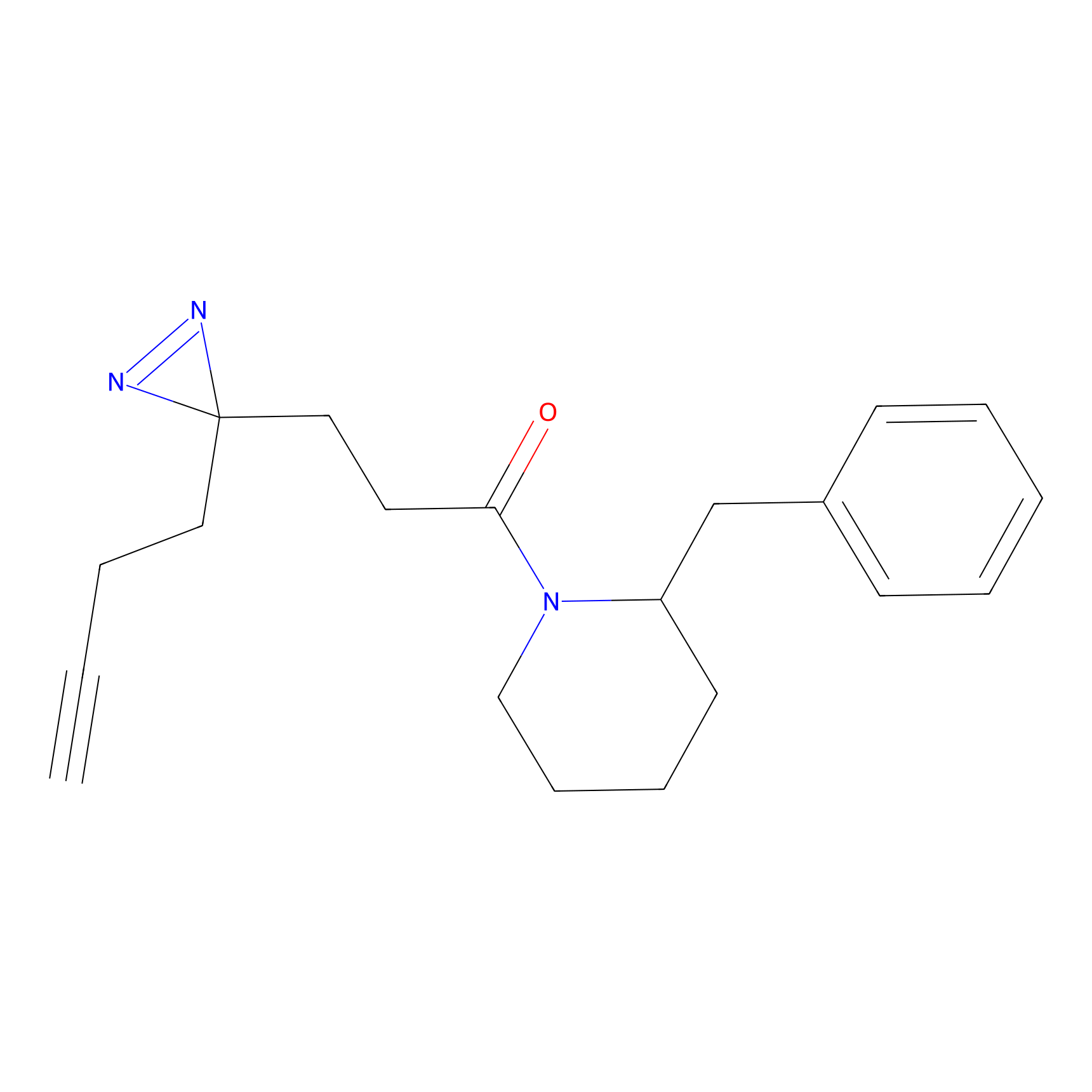
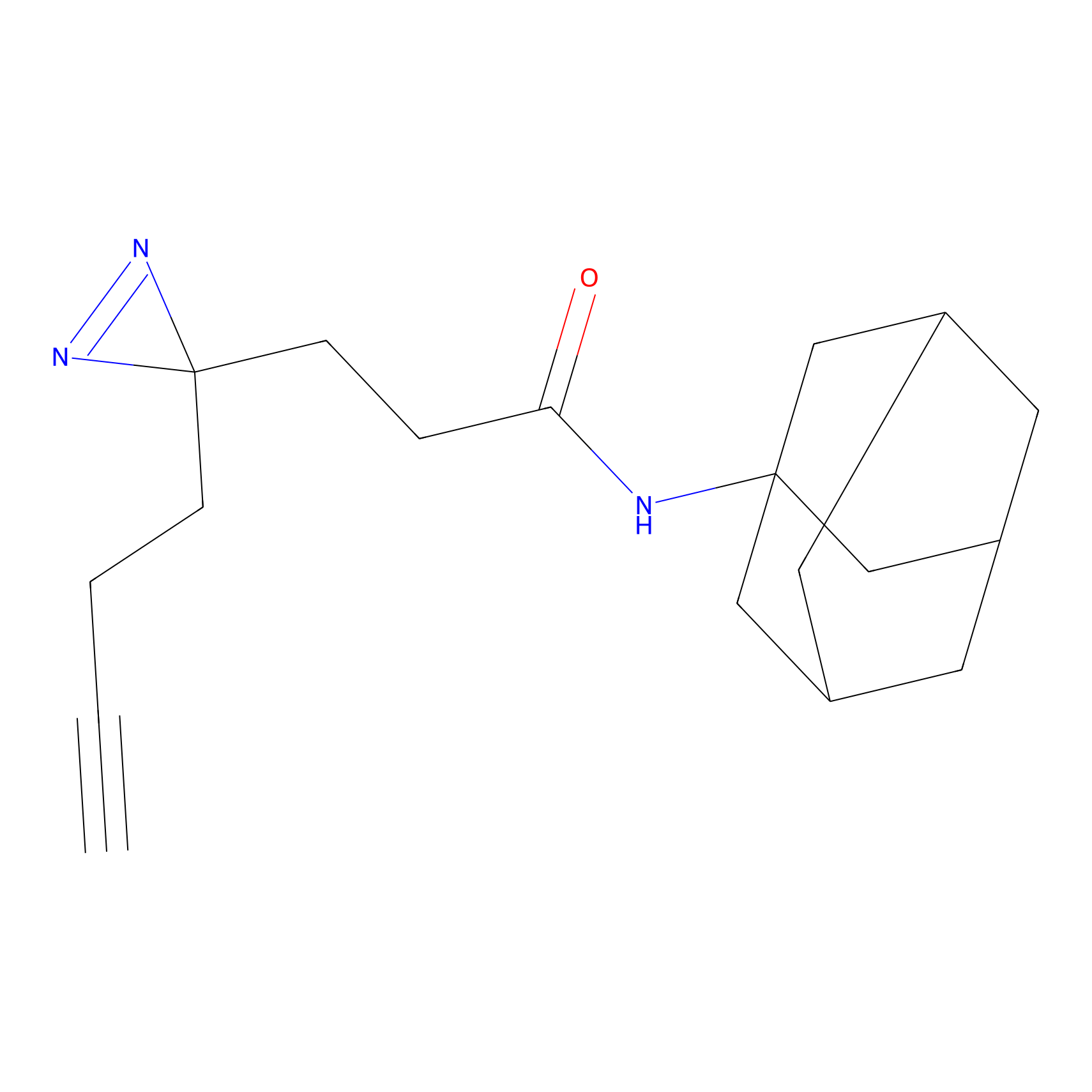
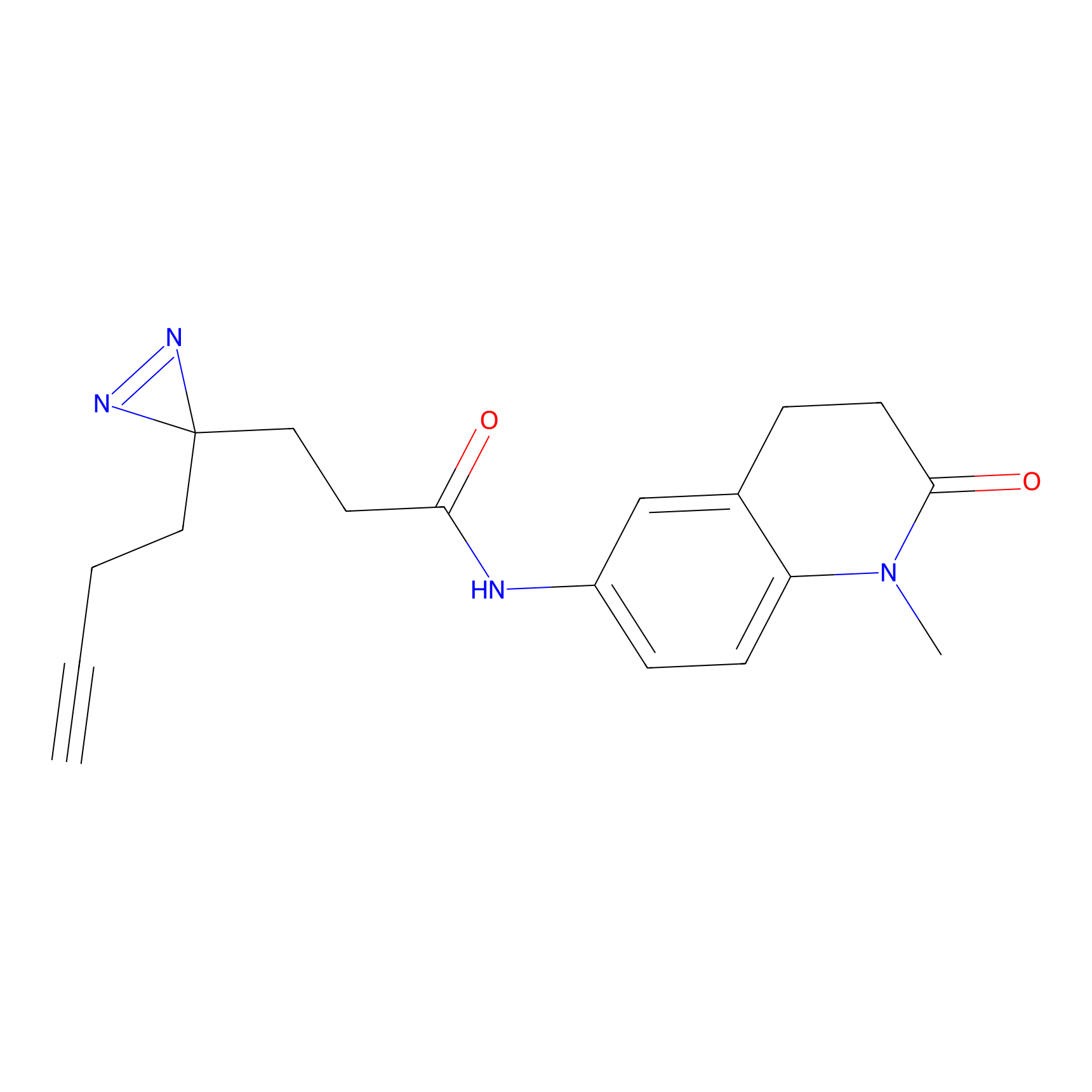
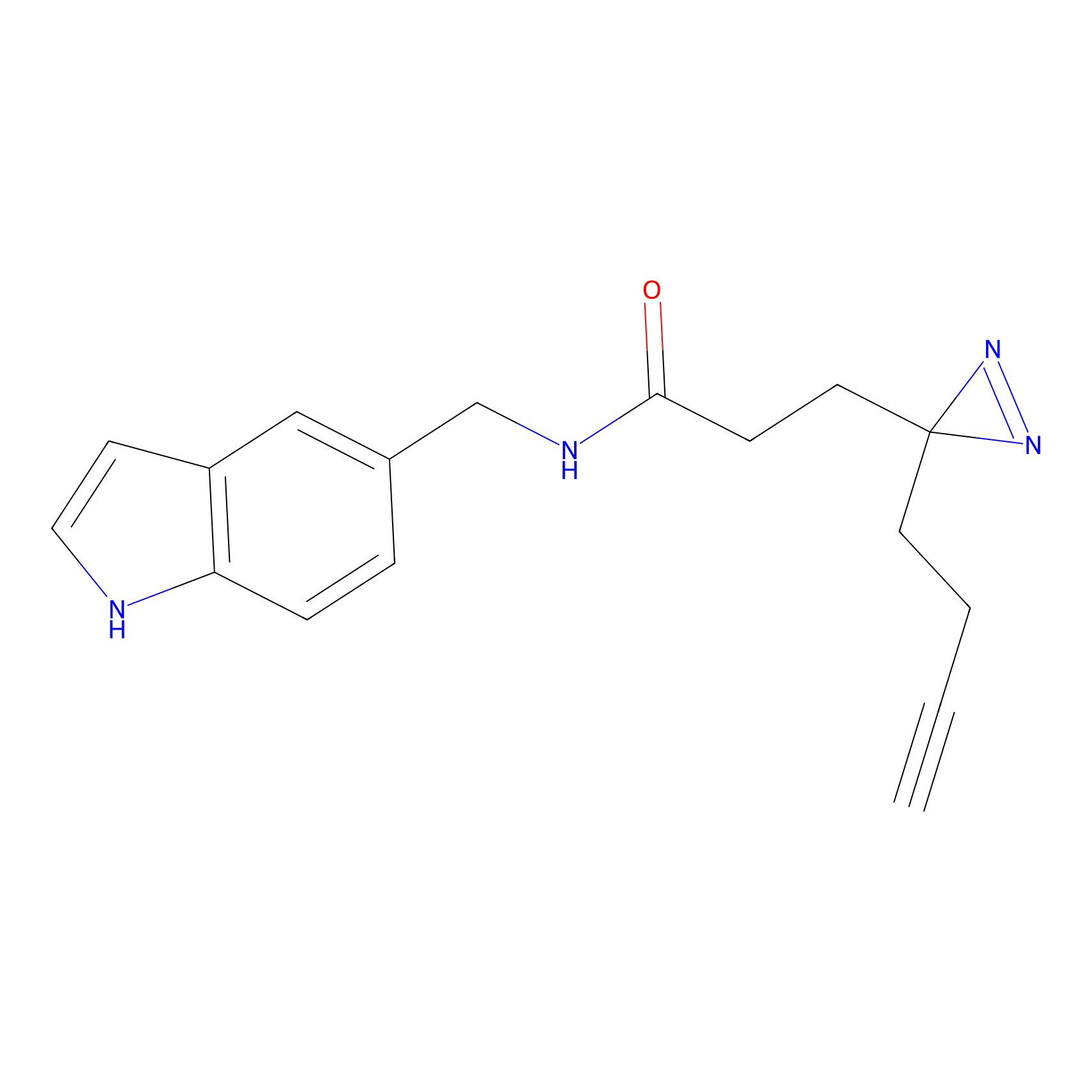
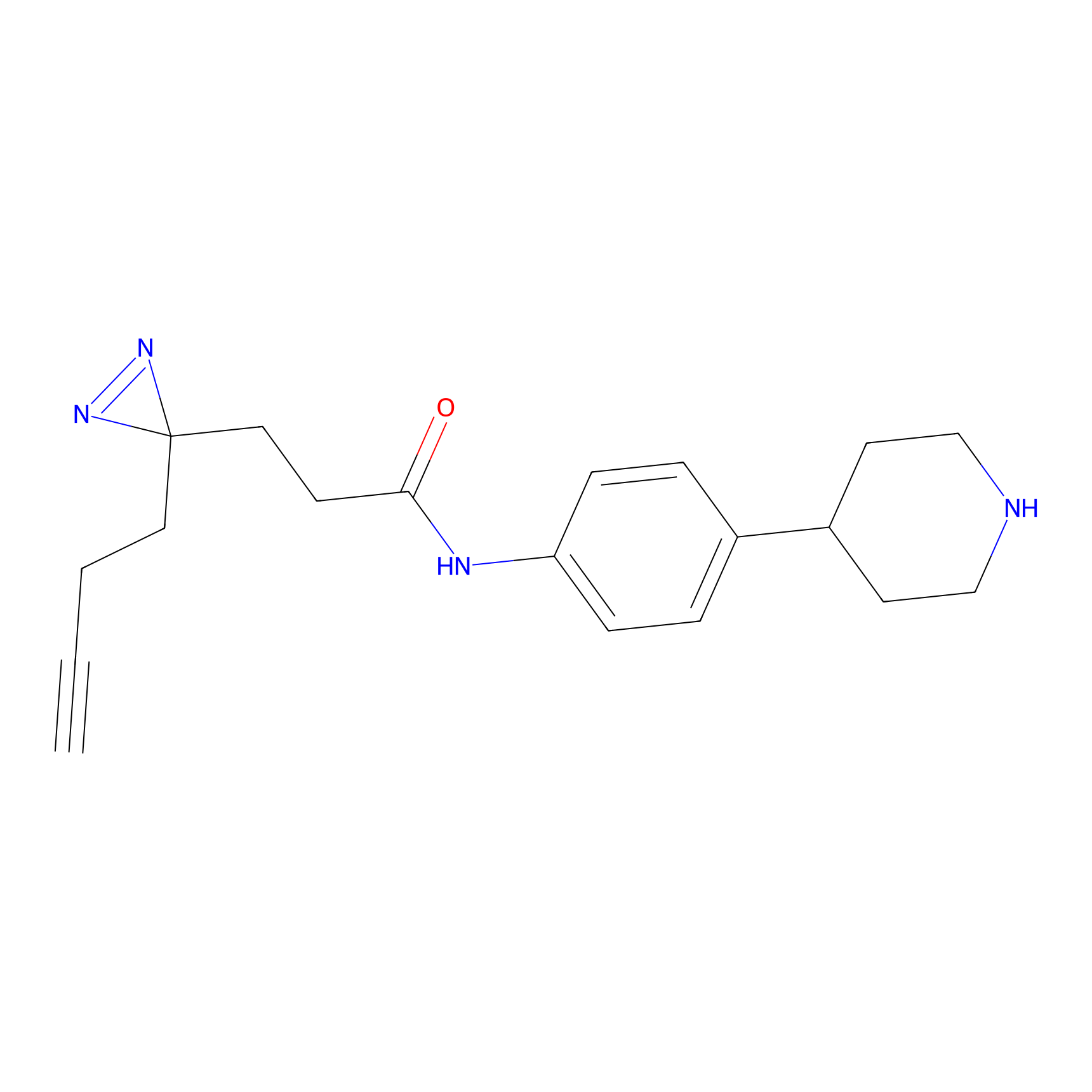
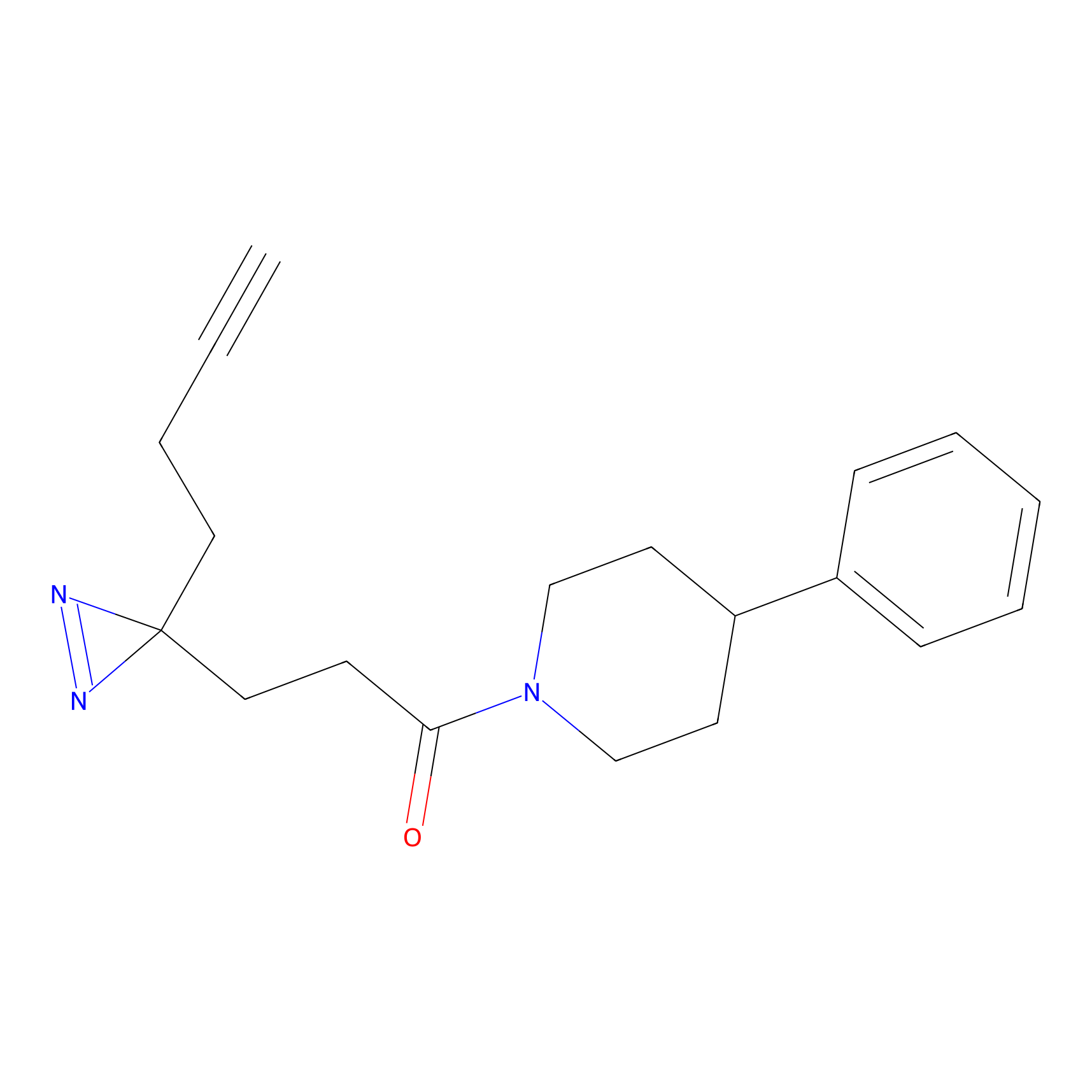
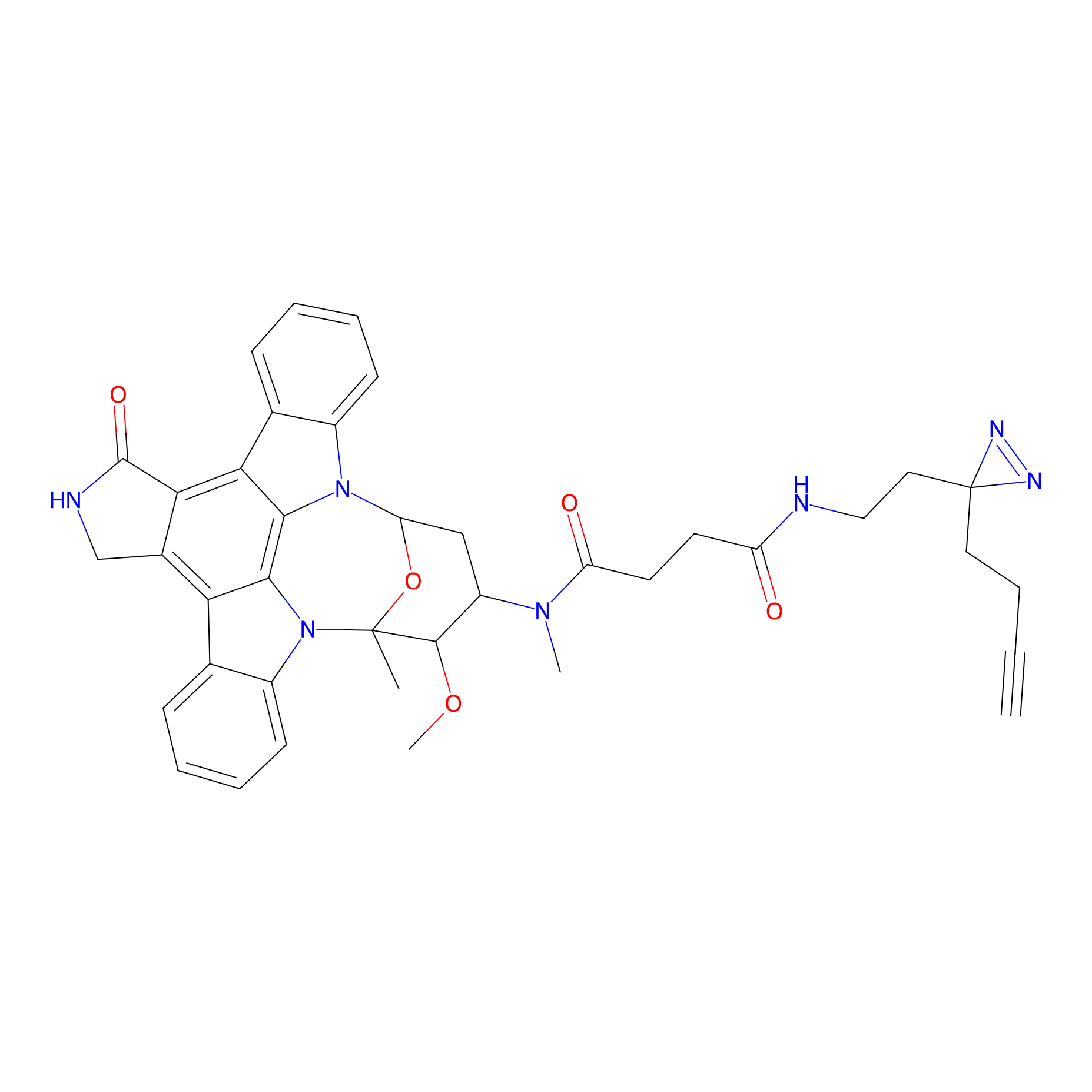
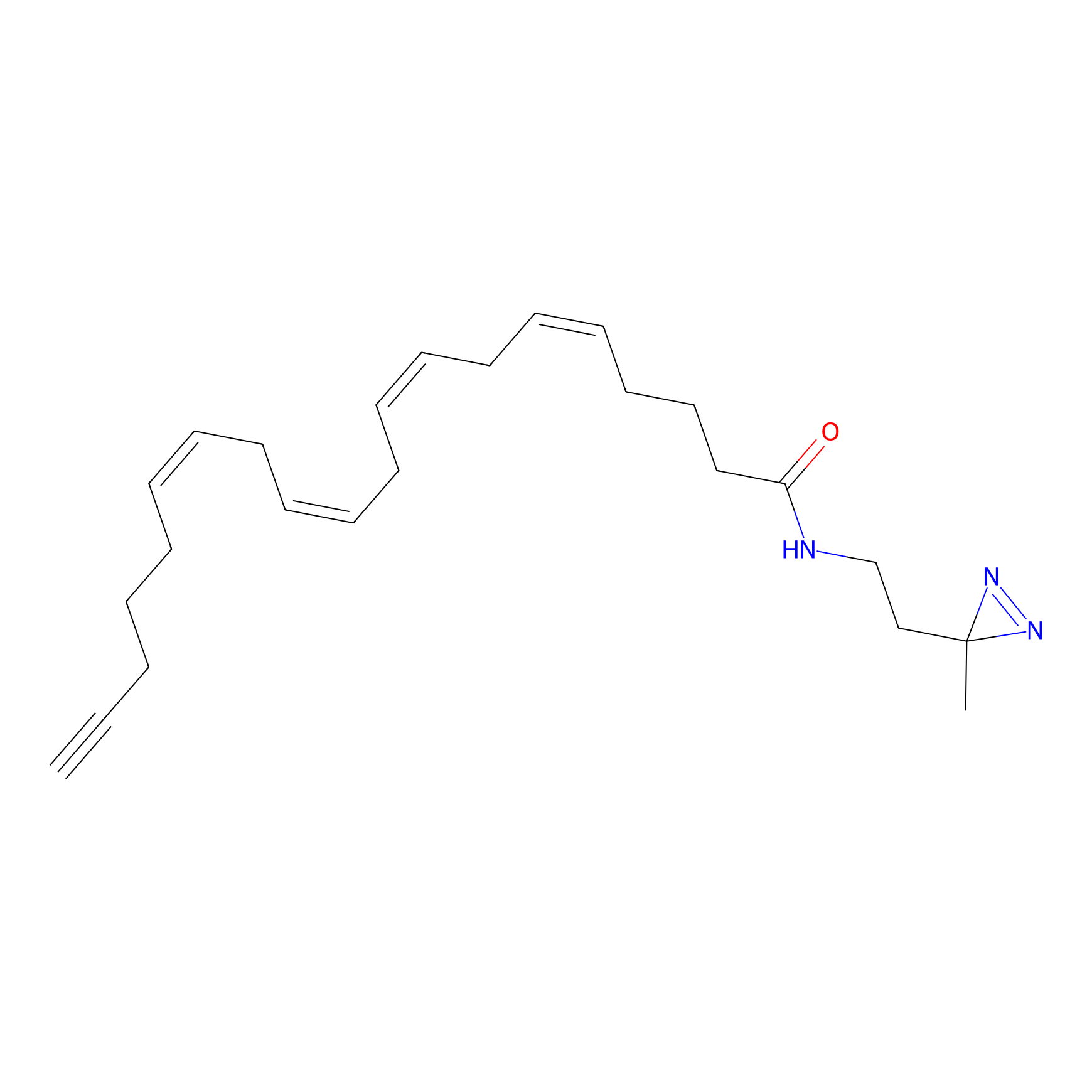
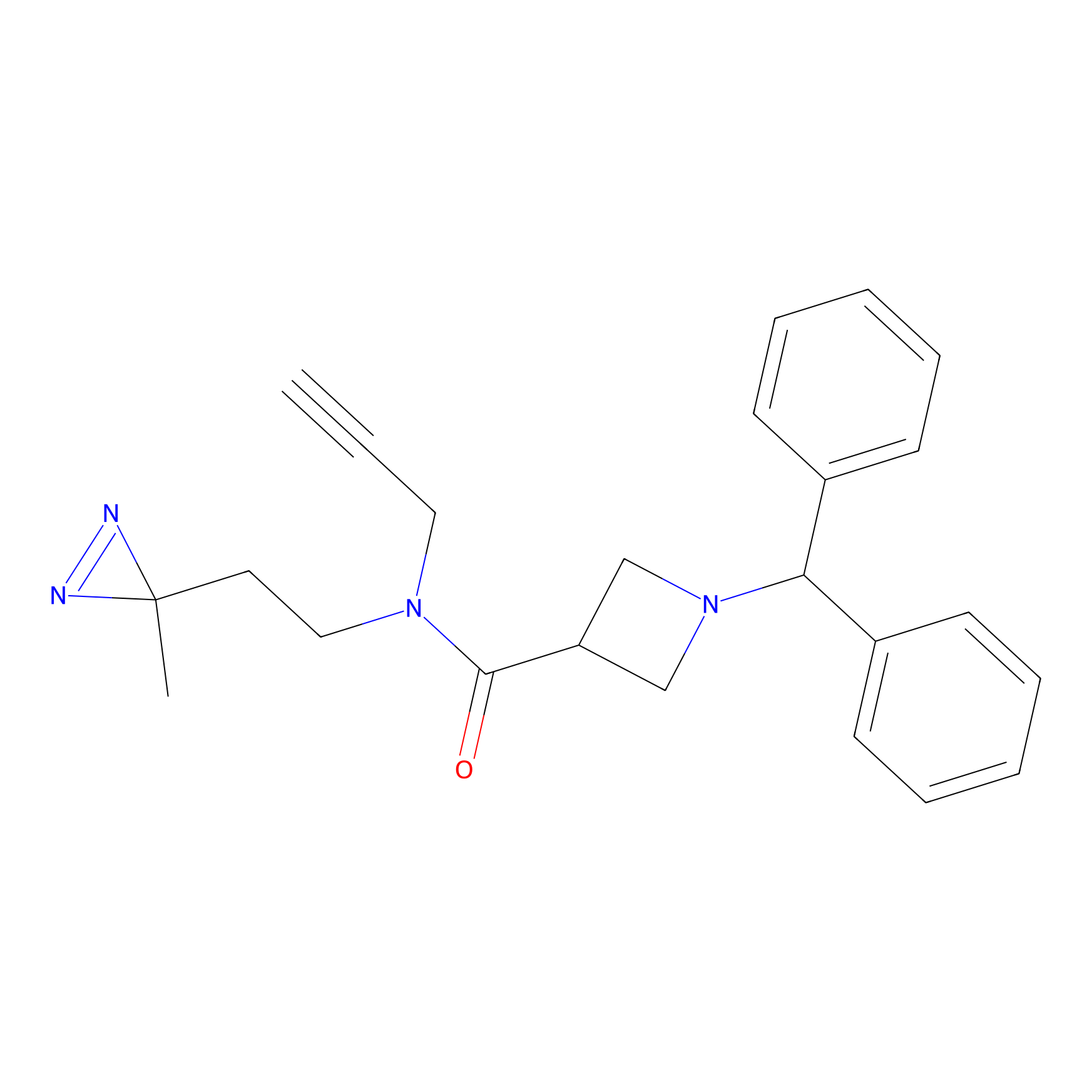
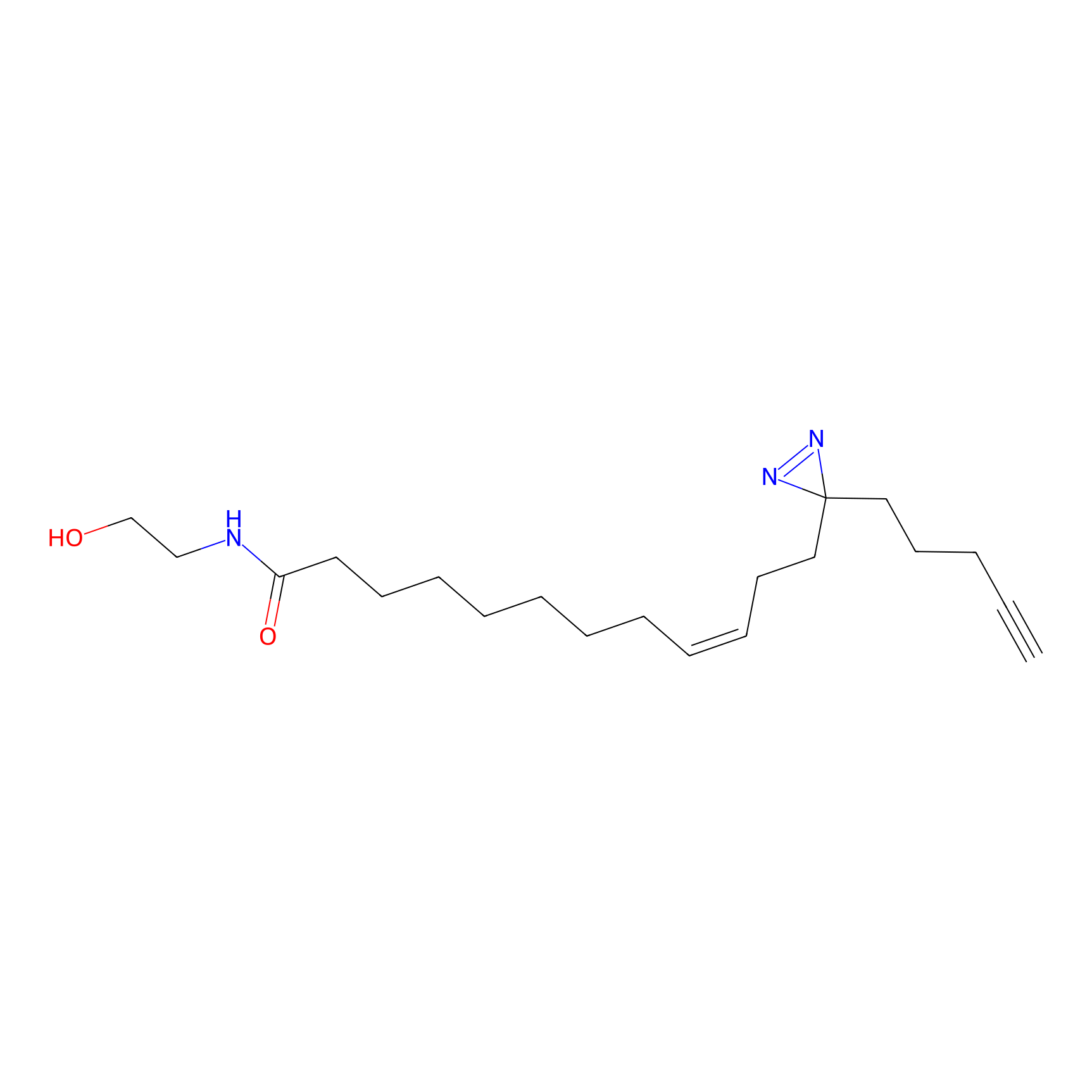
Probe(s) Labeling This Target
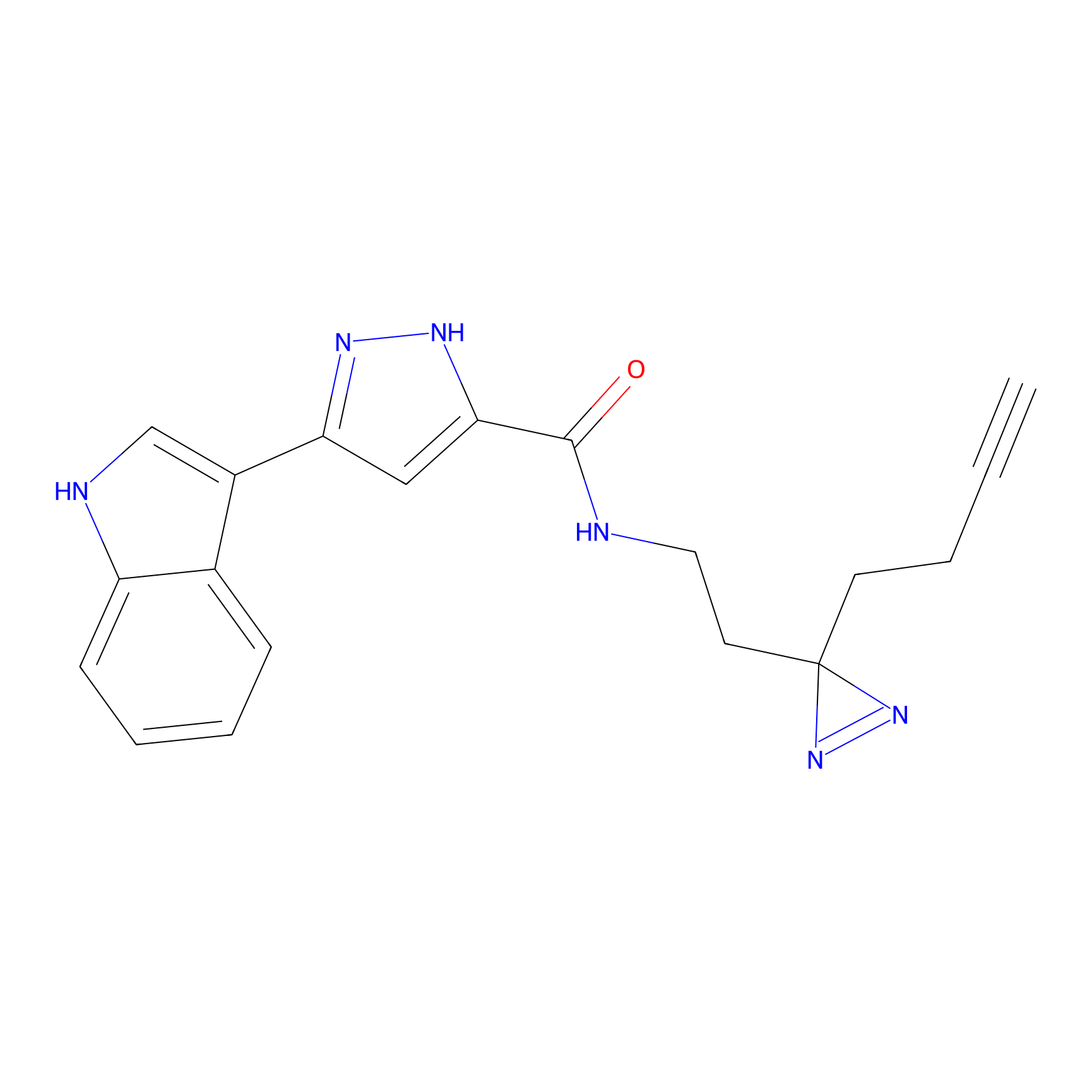
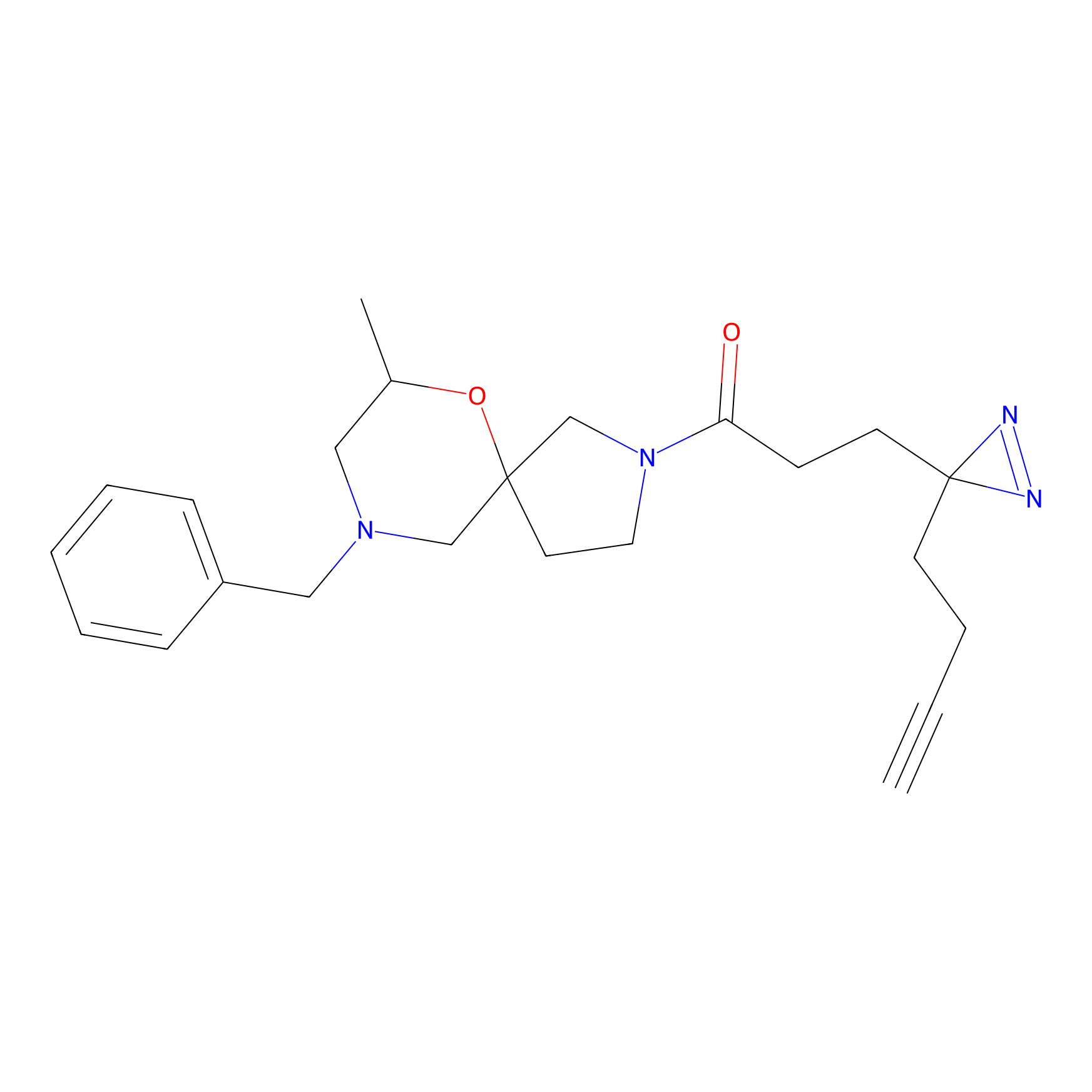
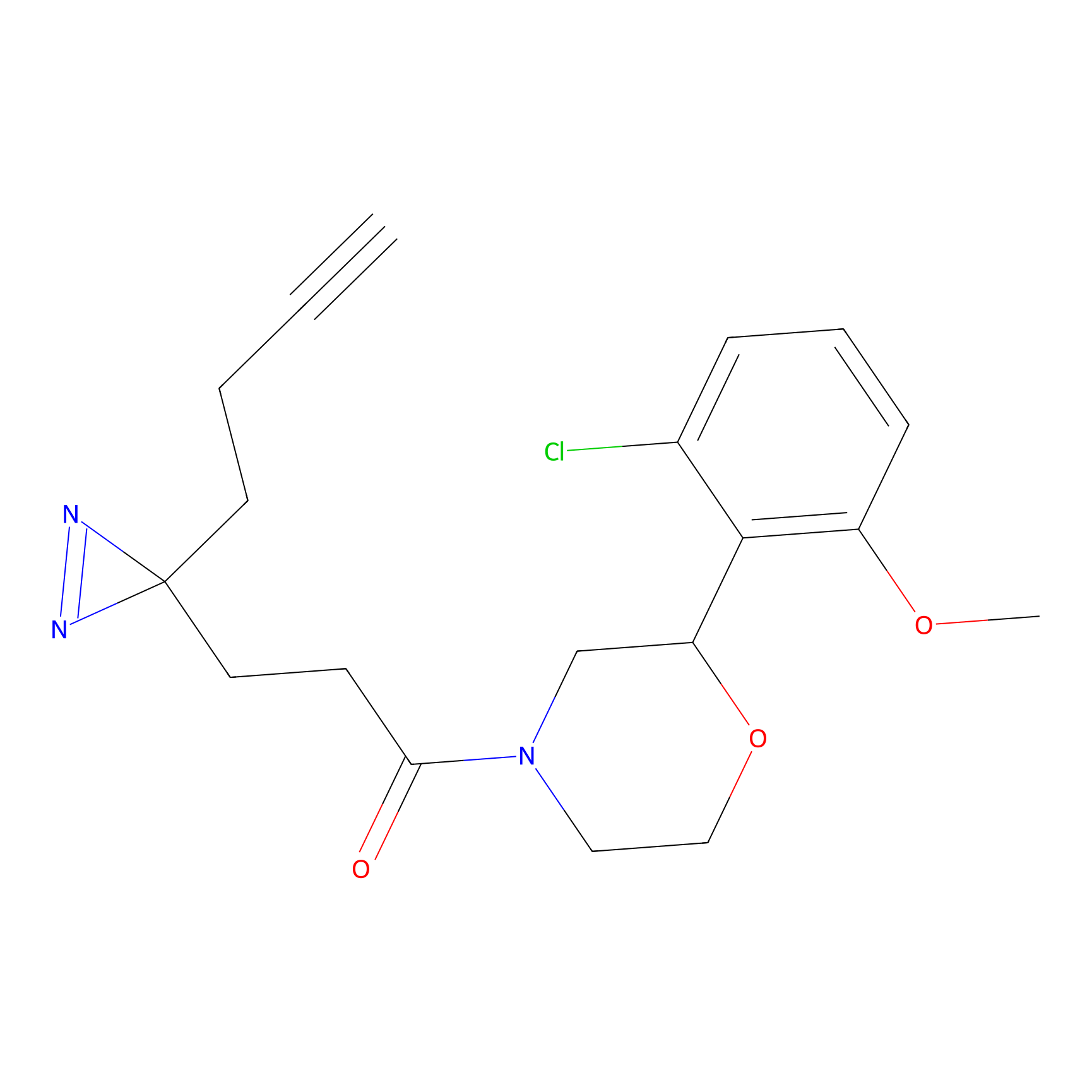
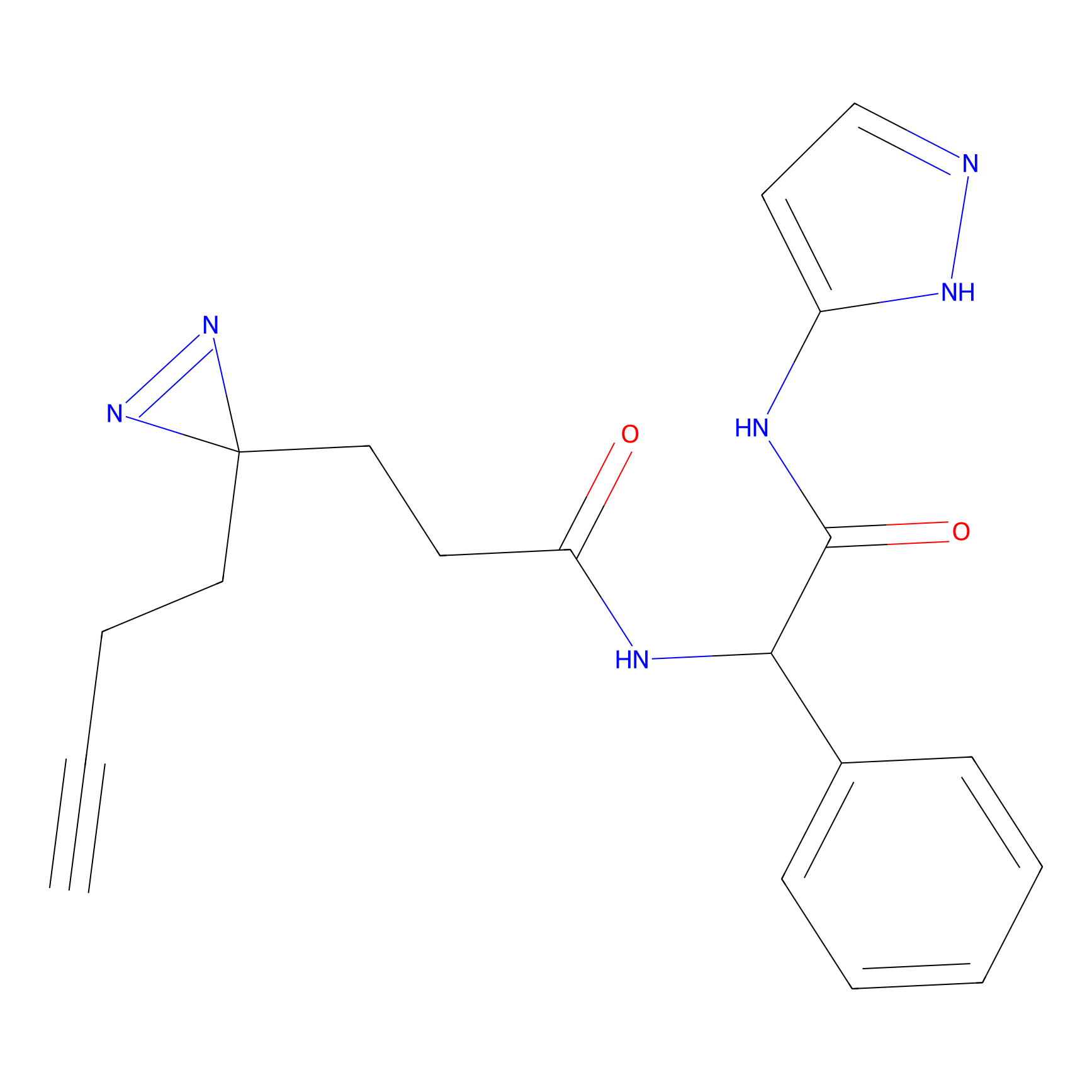
ABPP Probe
| Probe name | Structure | Binding Site(Ratio) | Interaction ID | Ref | |
|---|---|---|---|---|---|
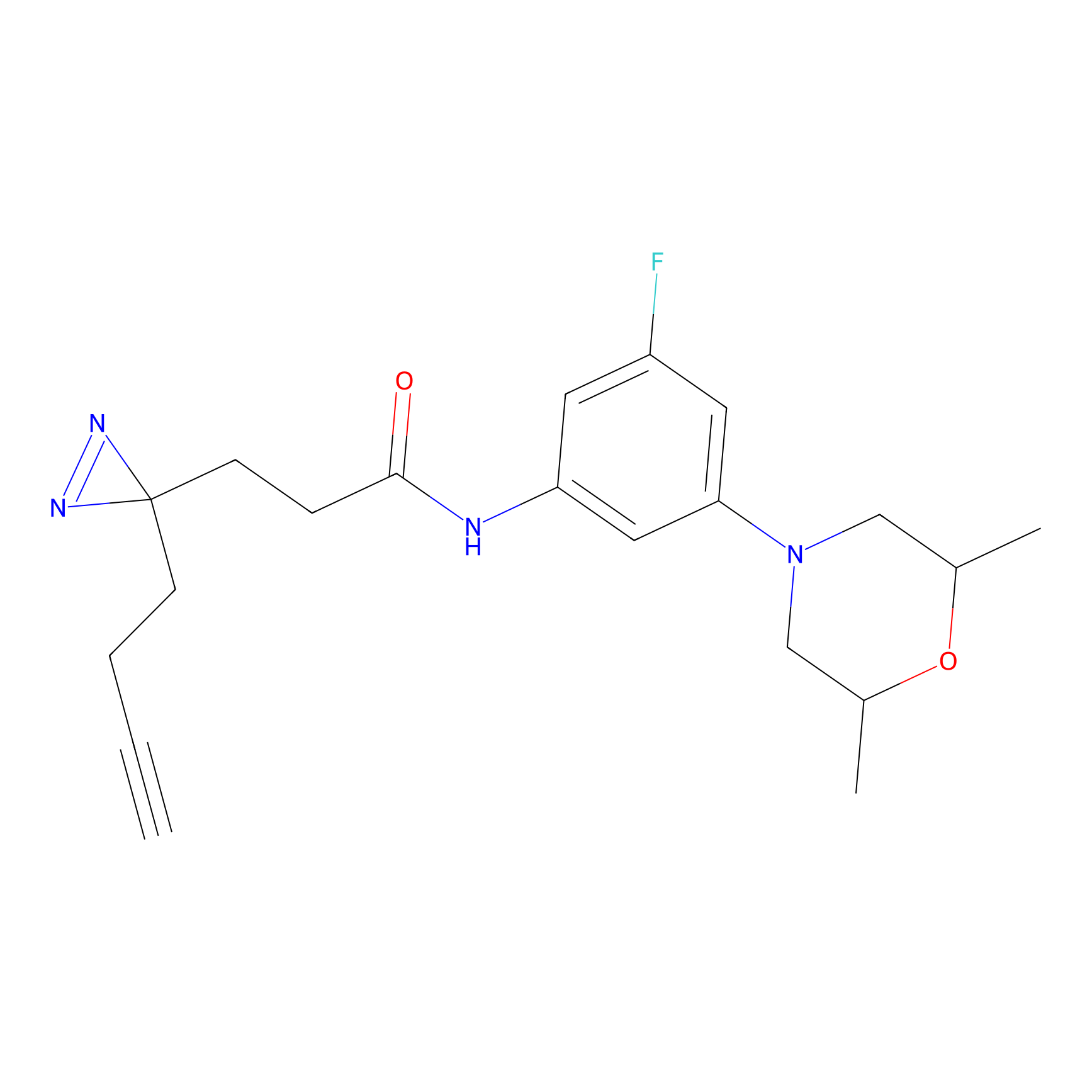
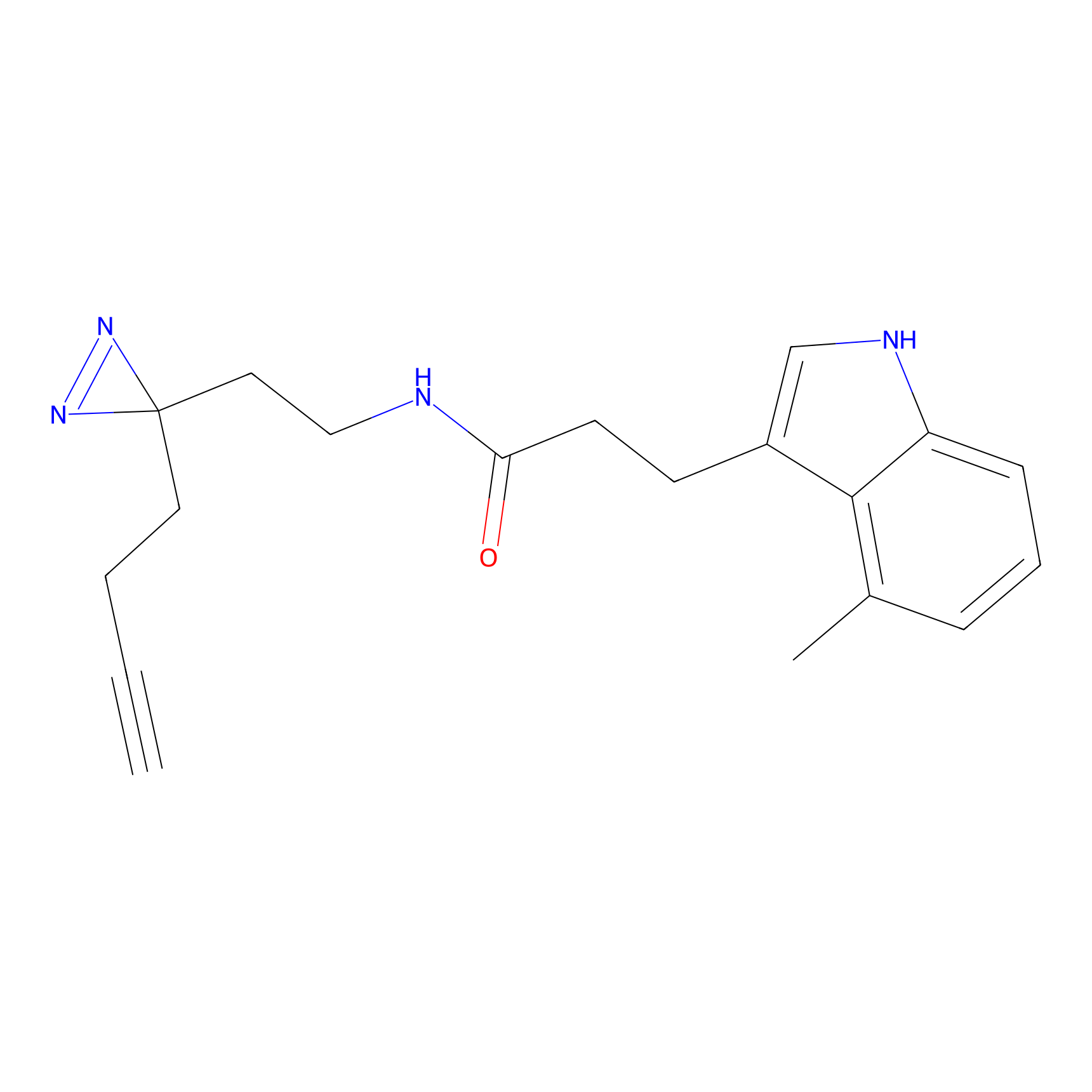
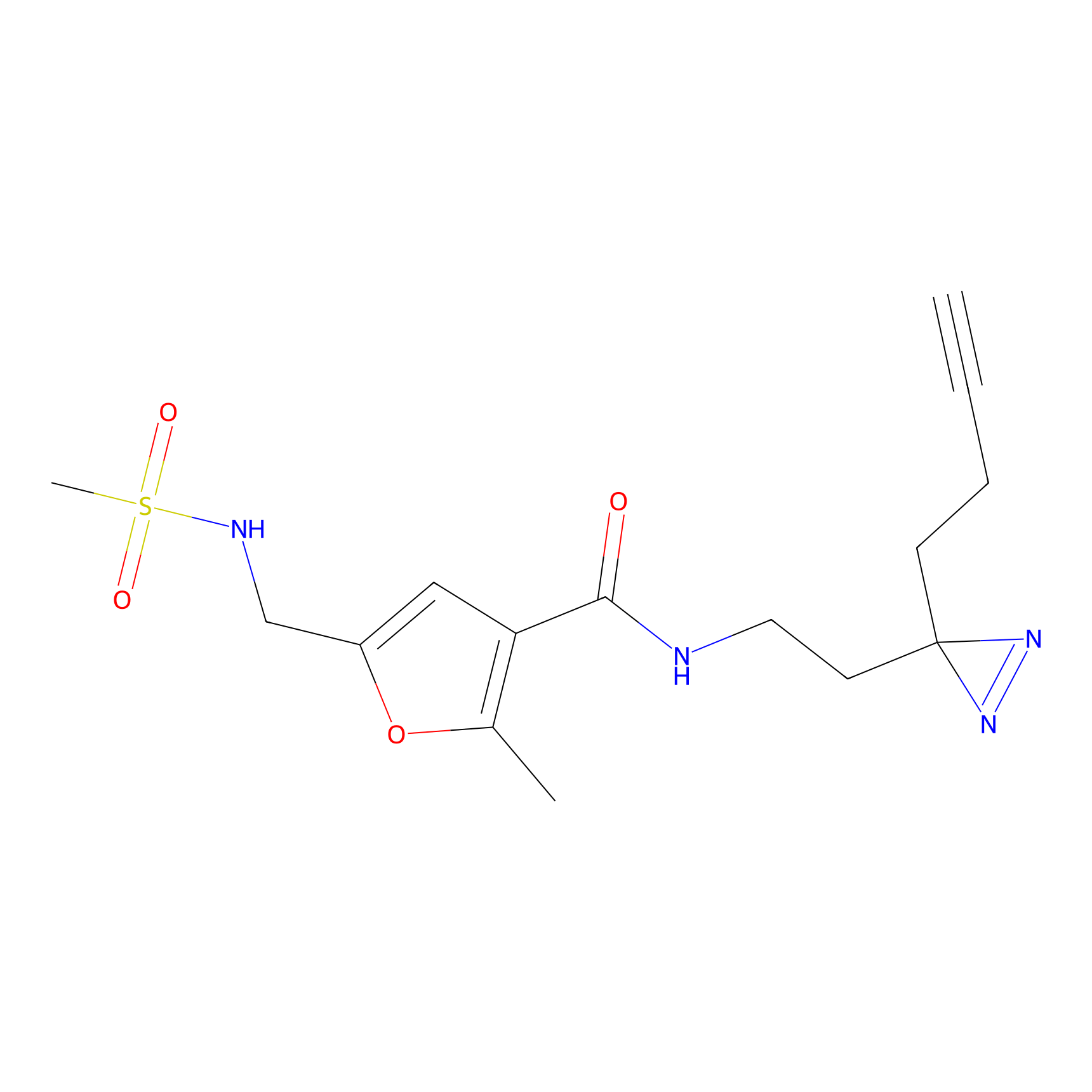
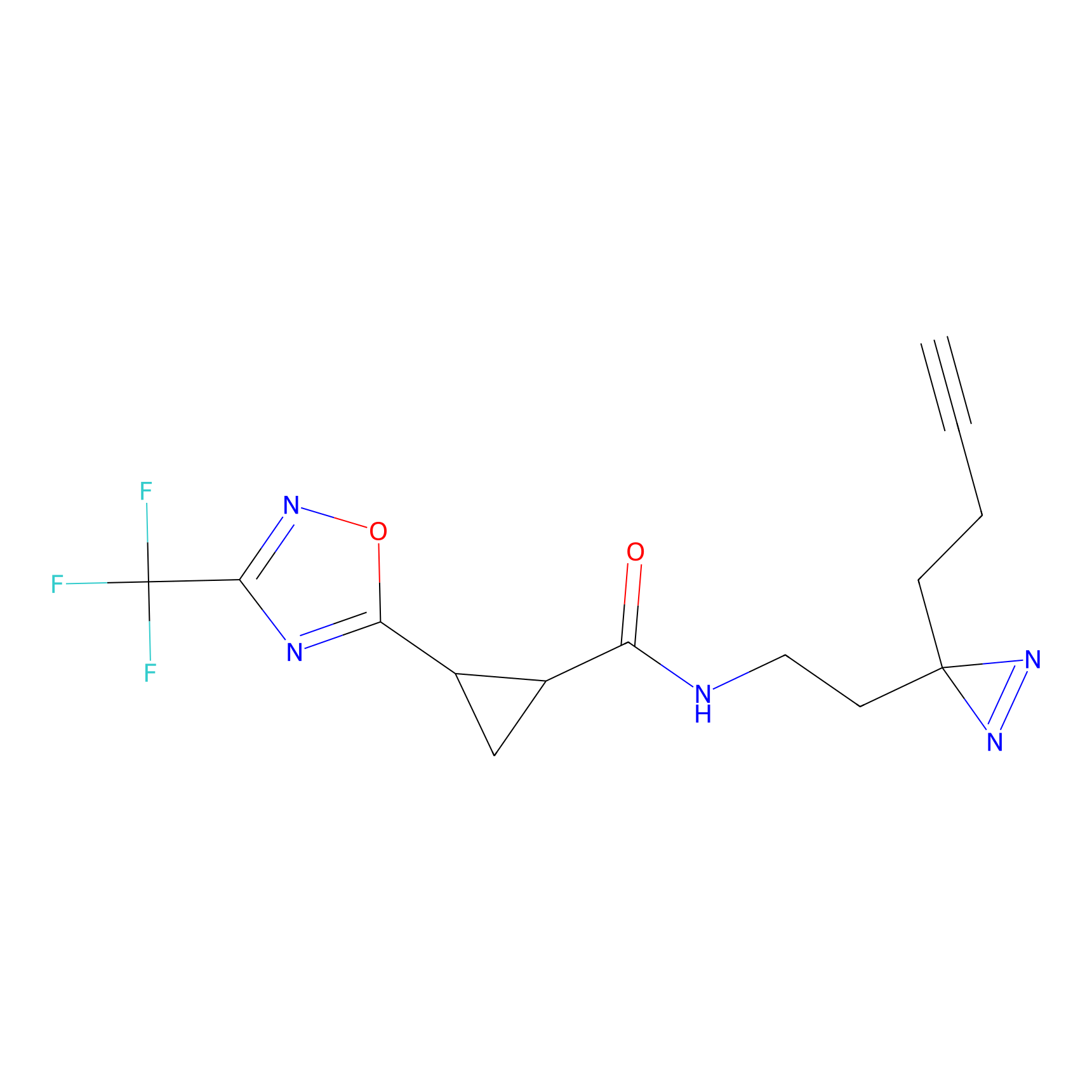
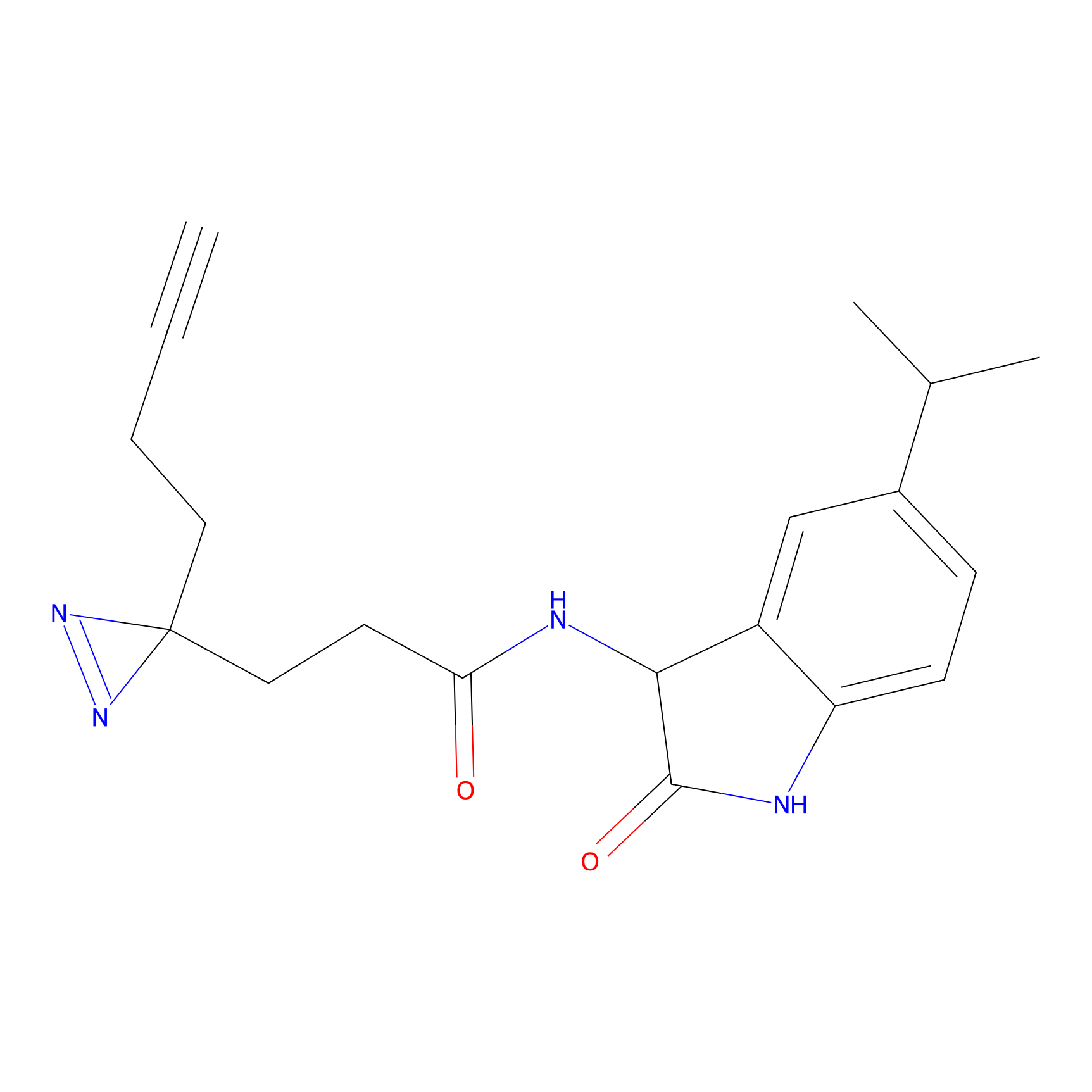
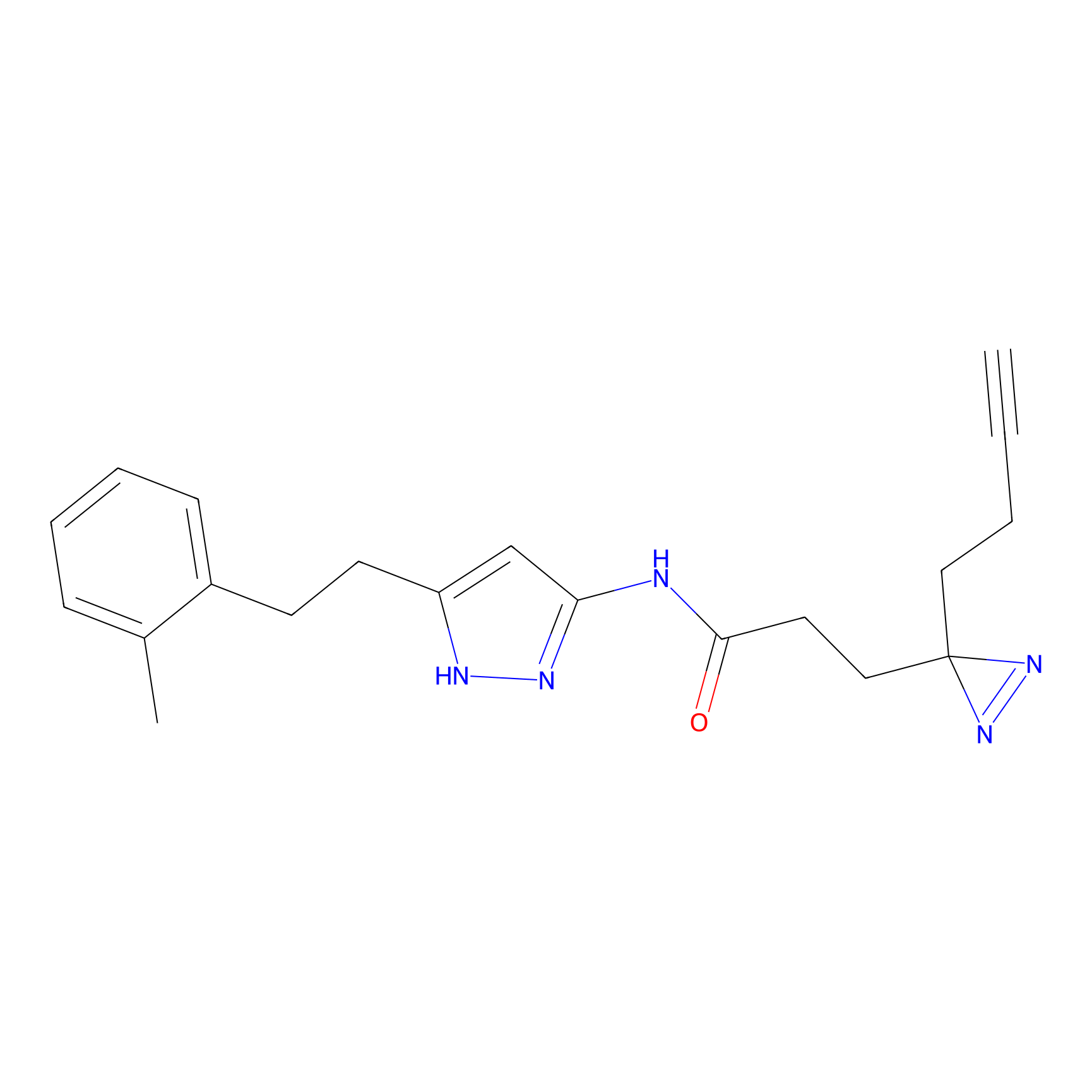
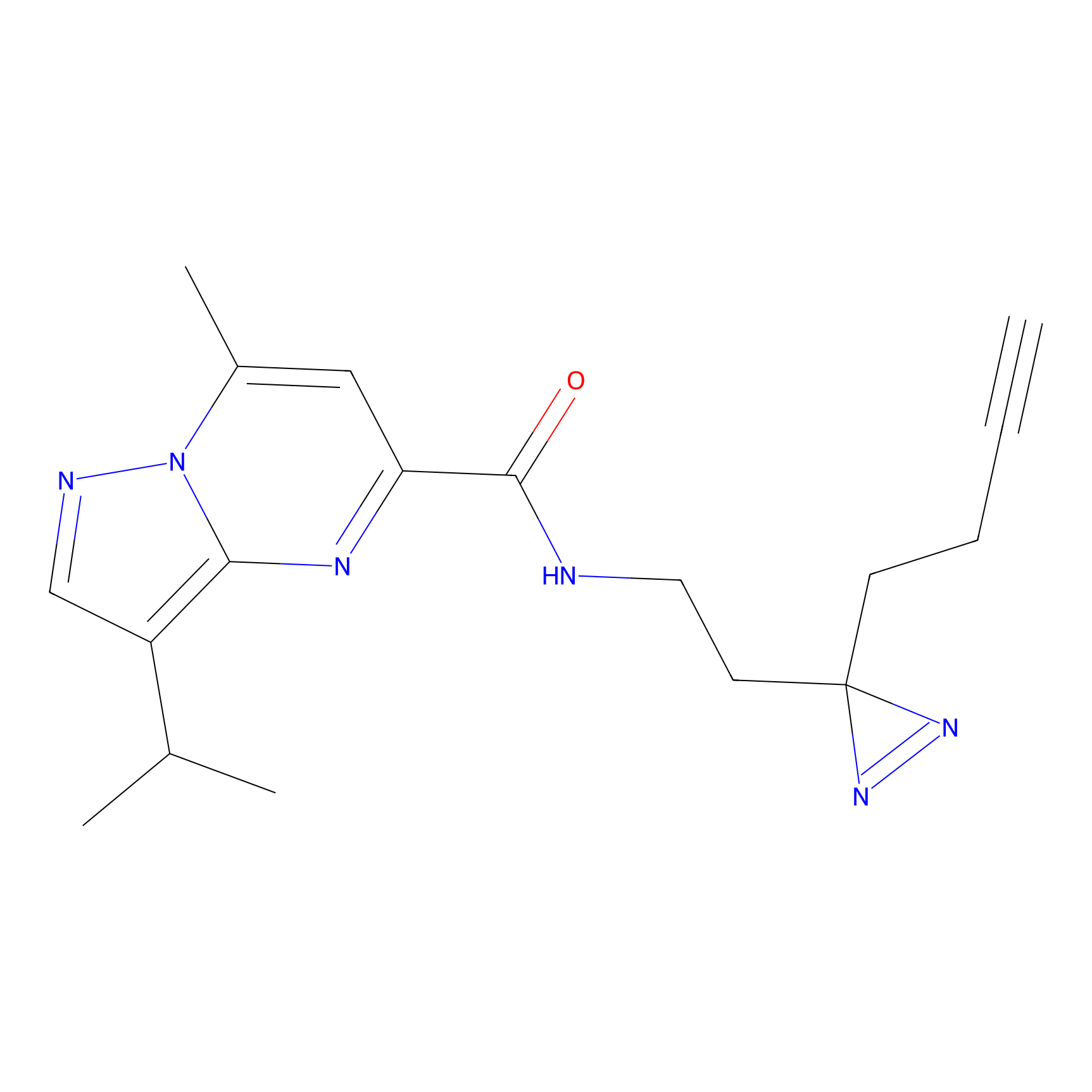
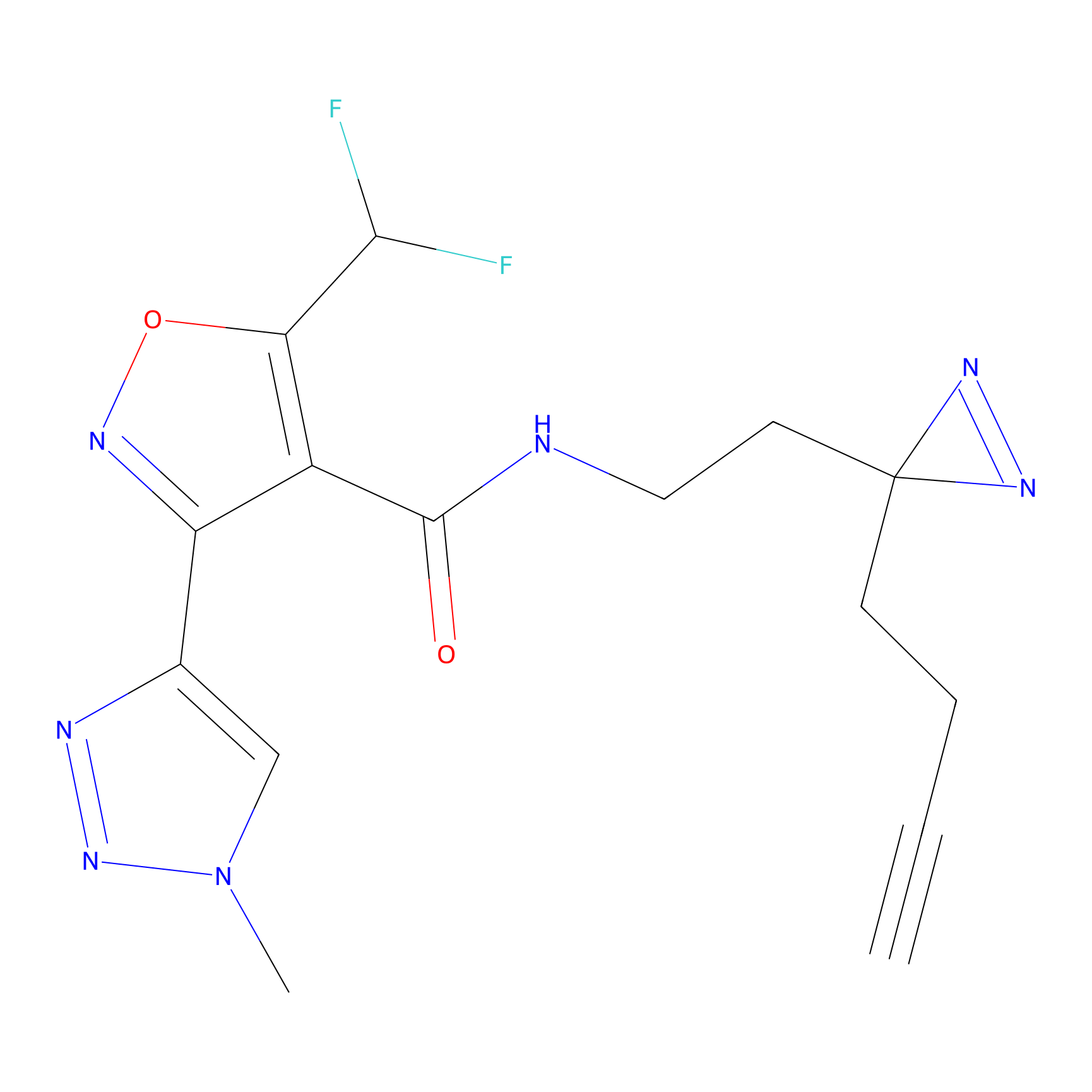
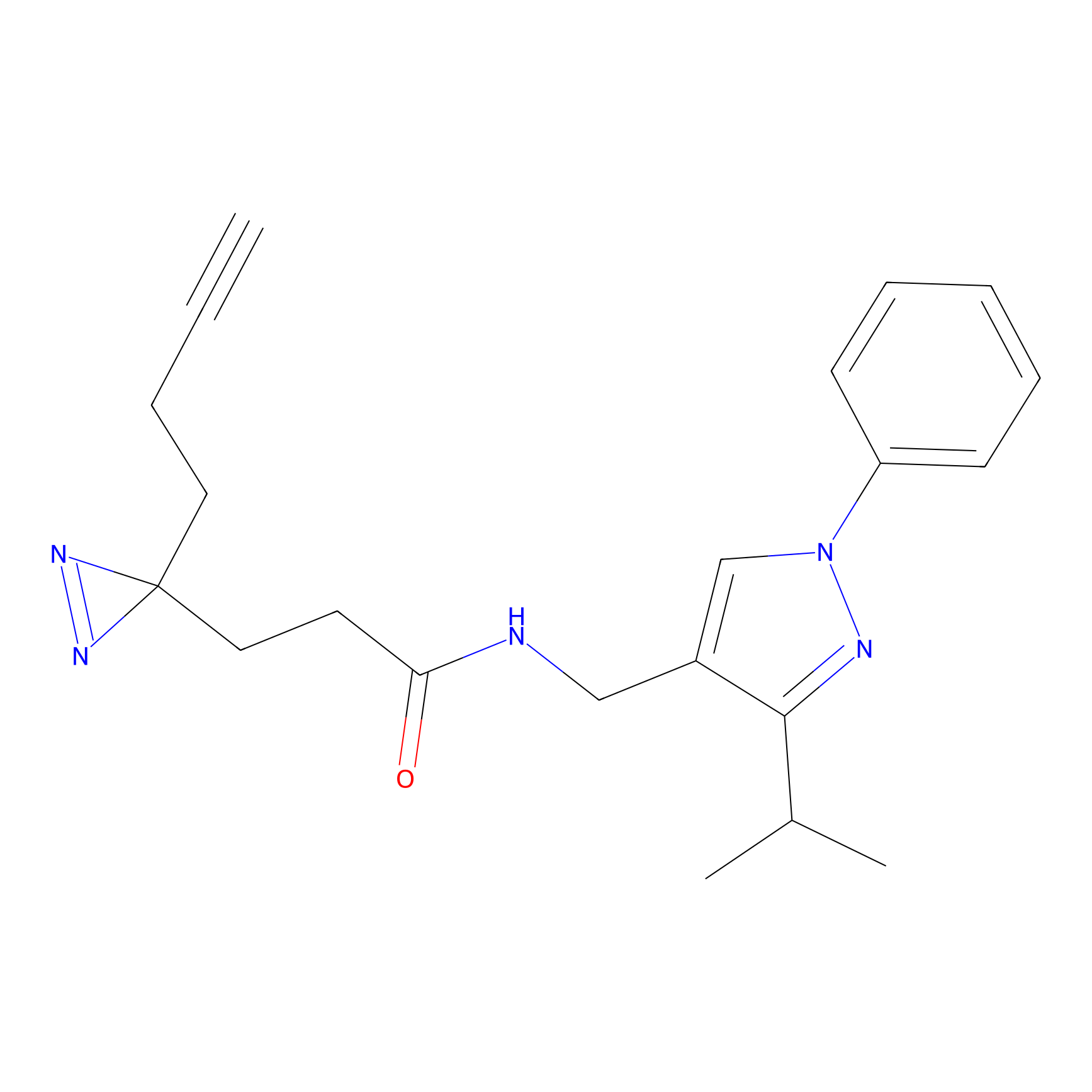
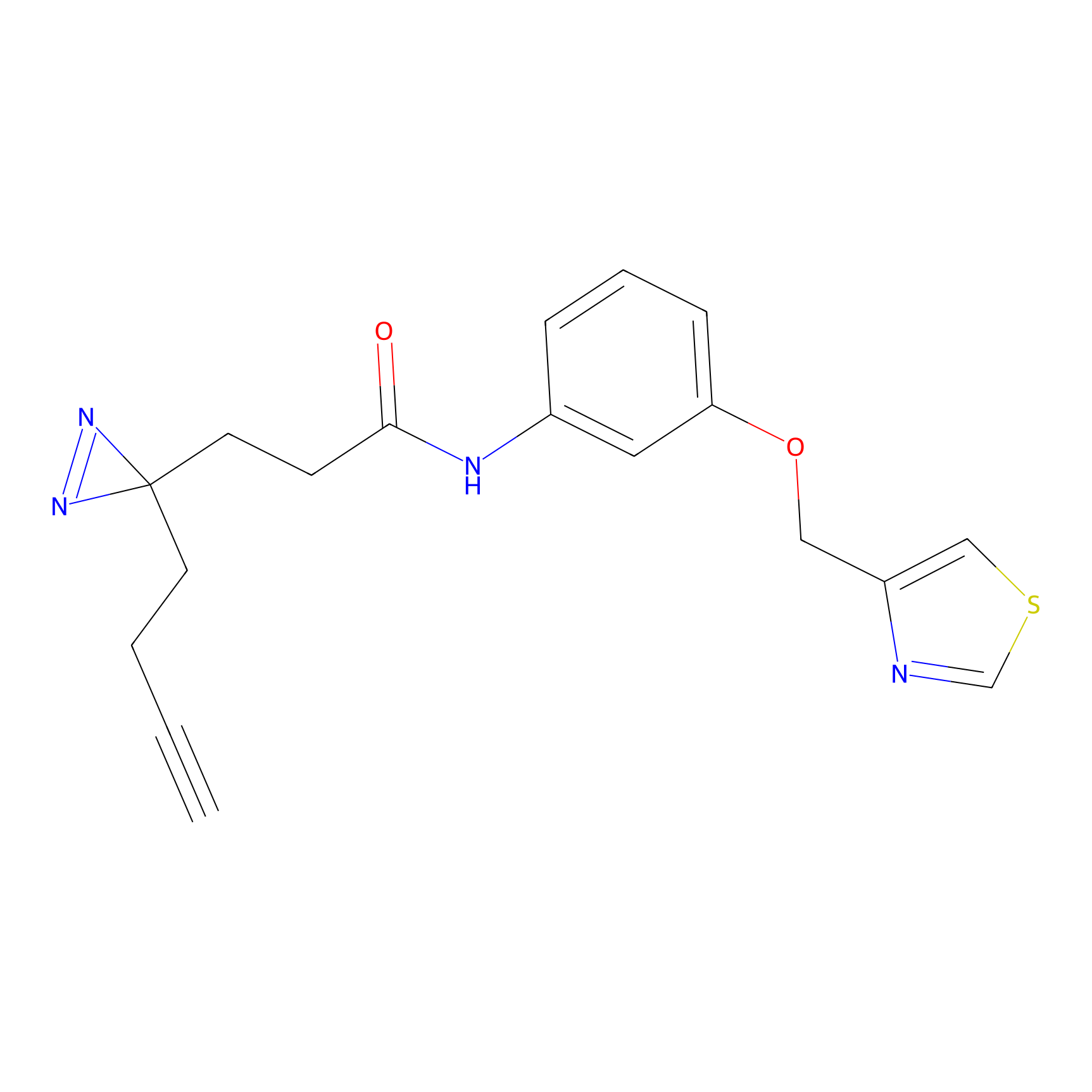
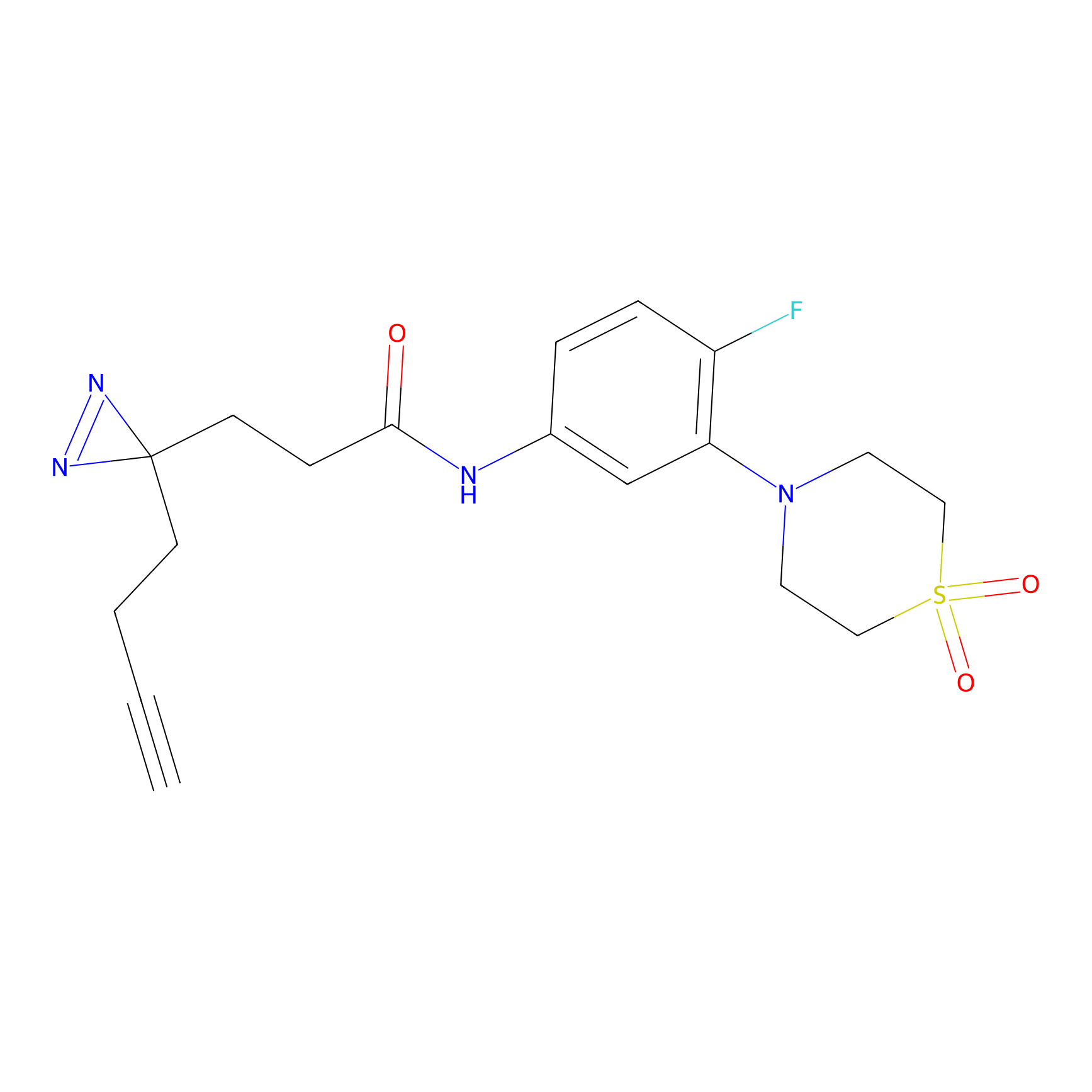
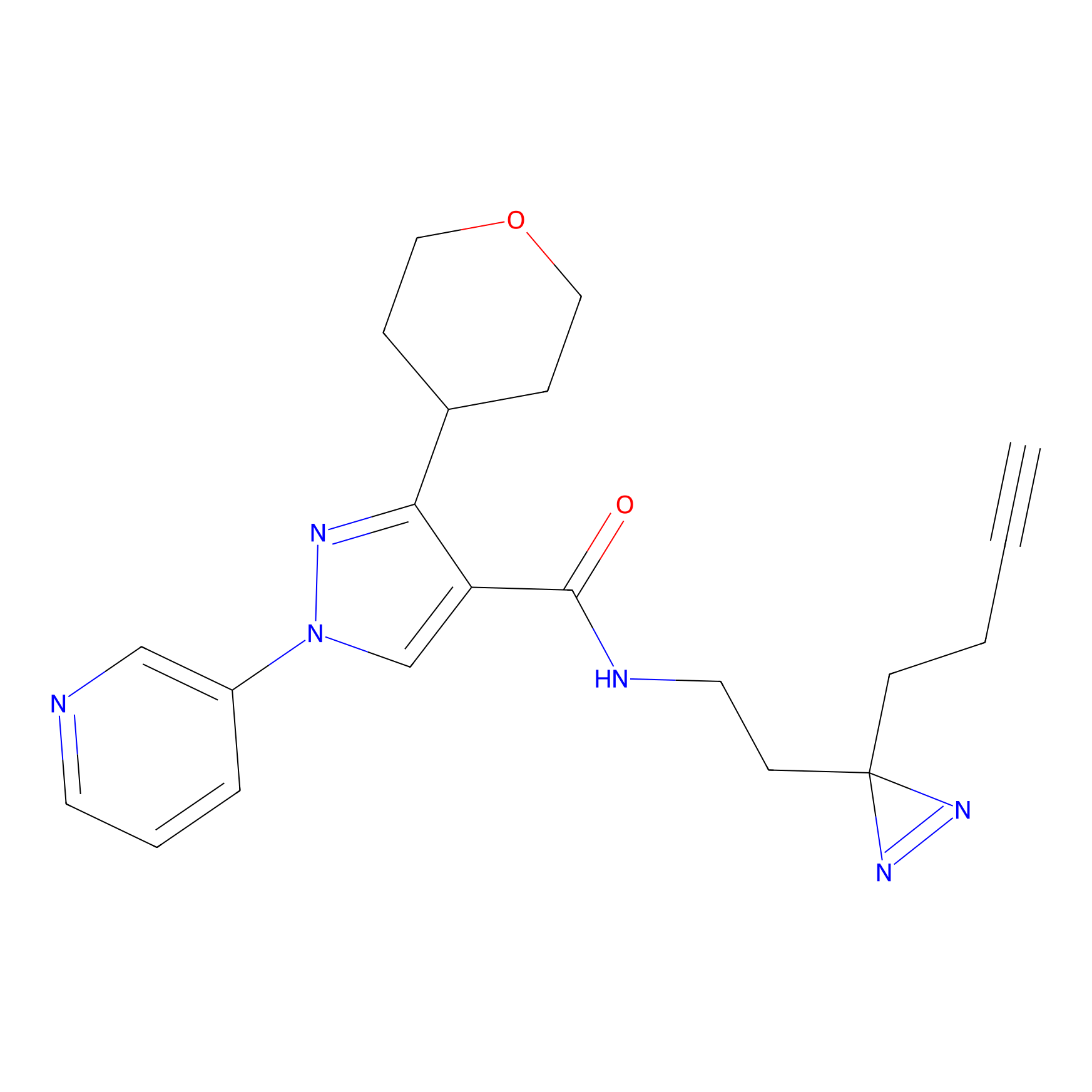
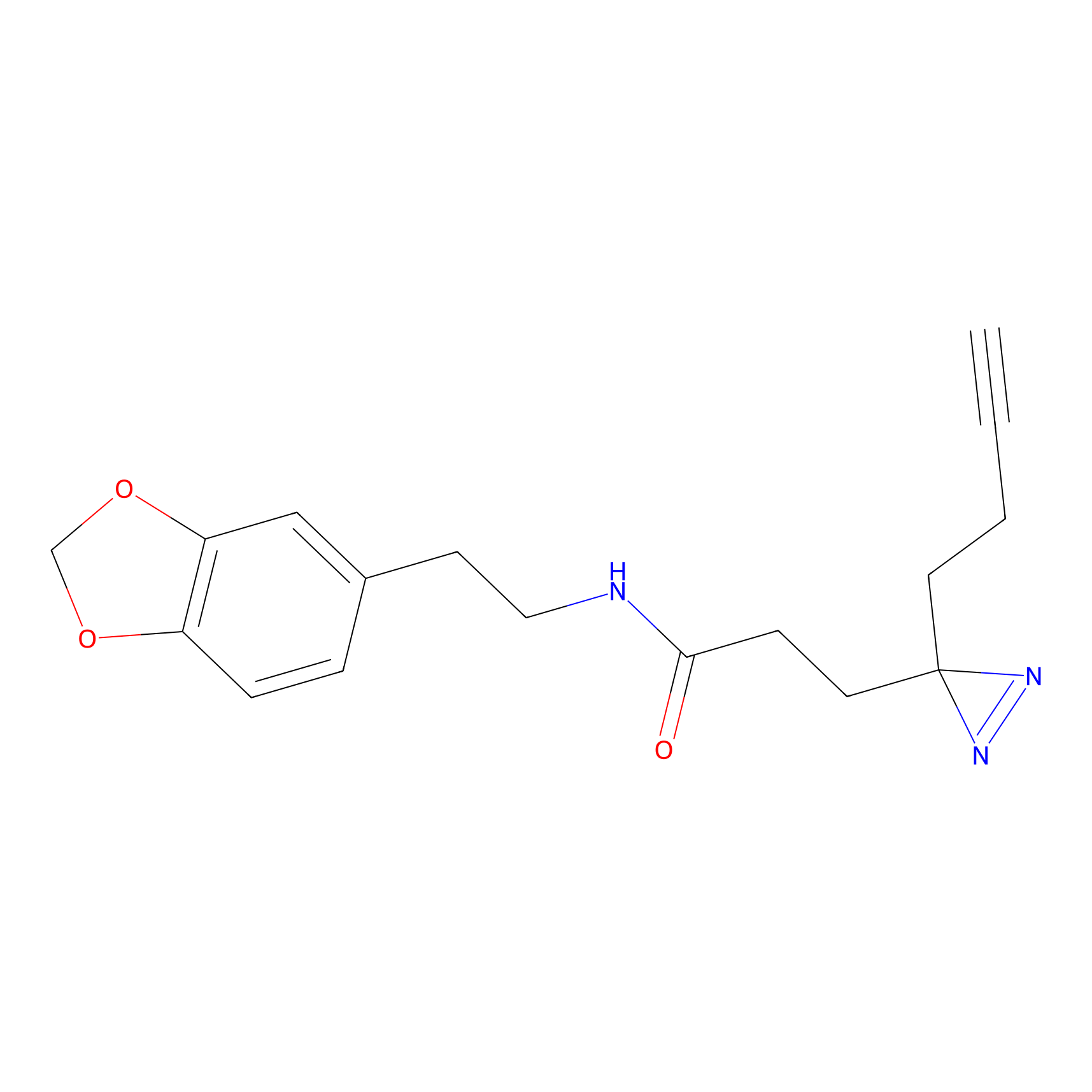
|
ILS-2 Probe Info |
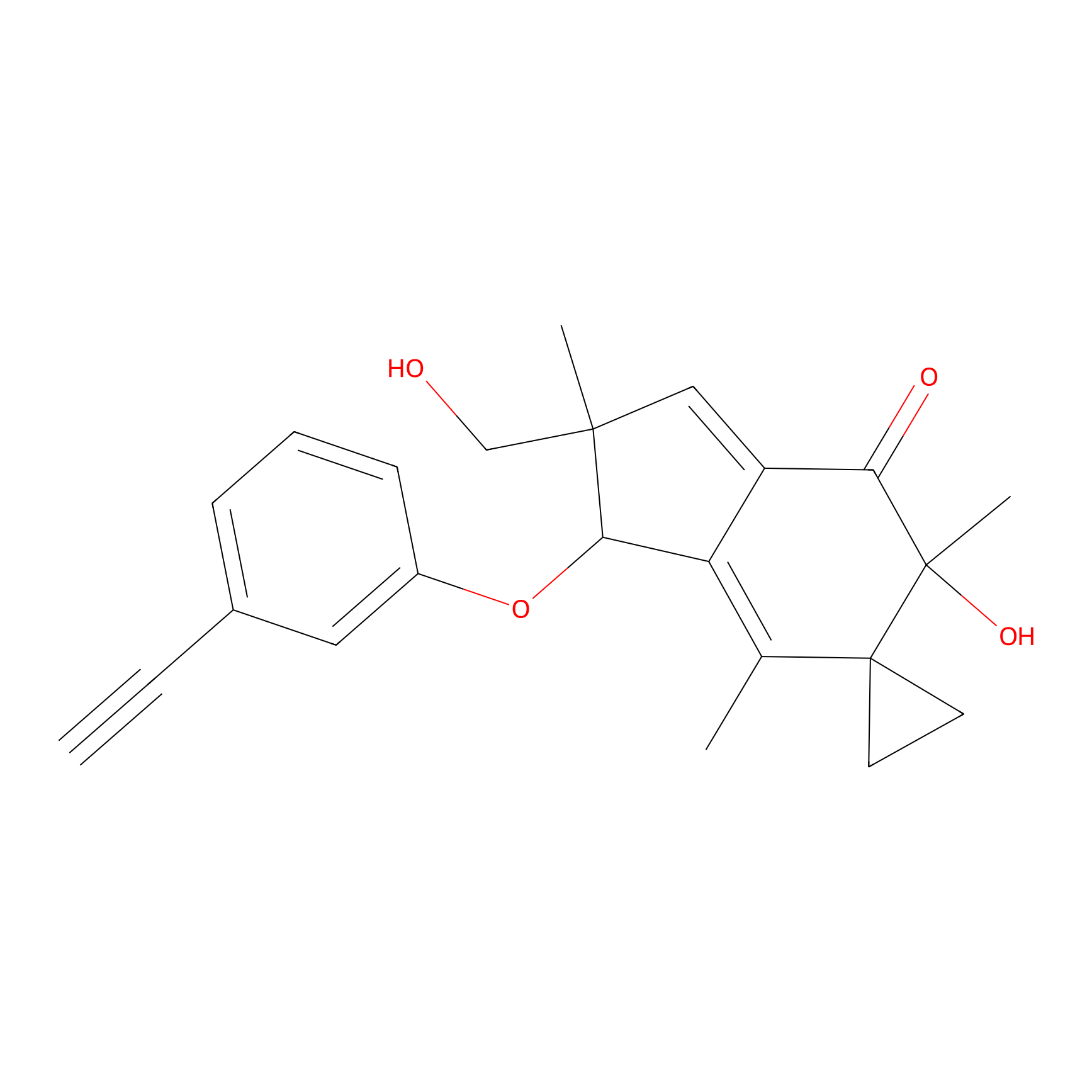 |
2.44 | LDD0416 | [2] | |
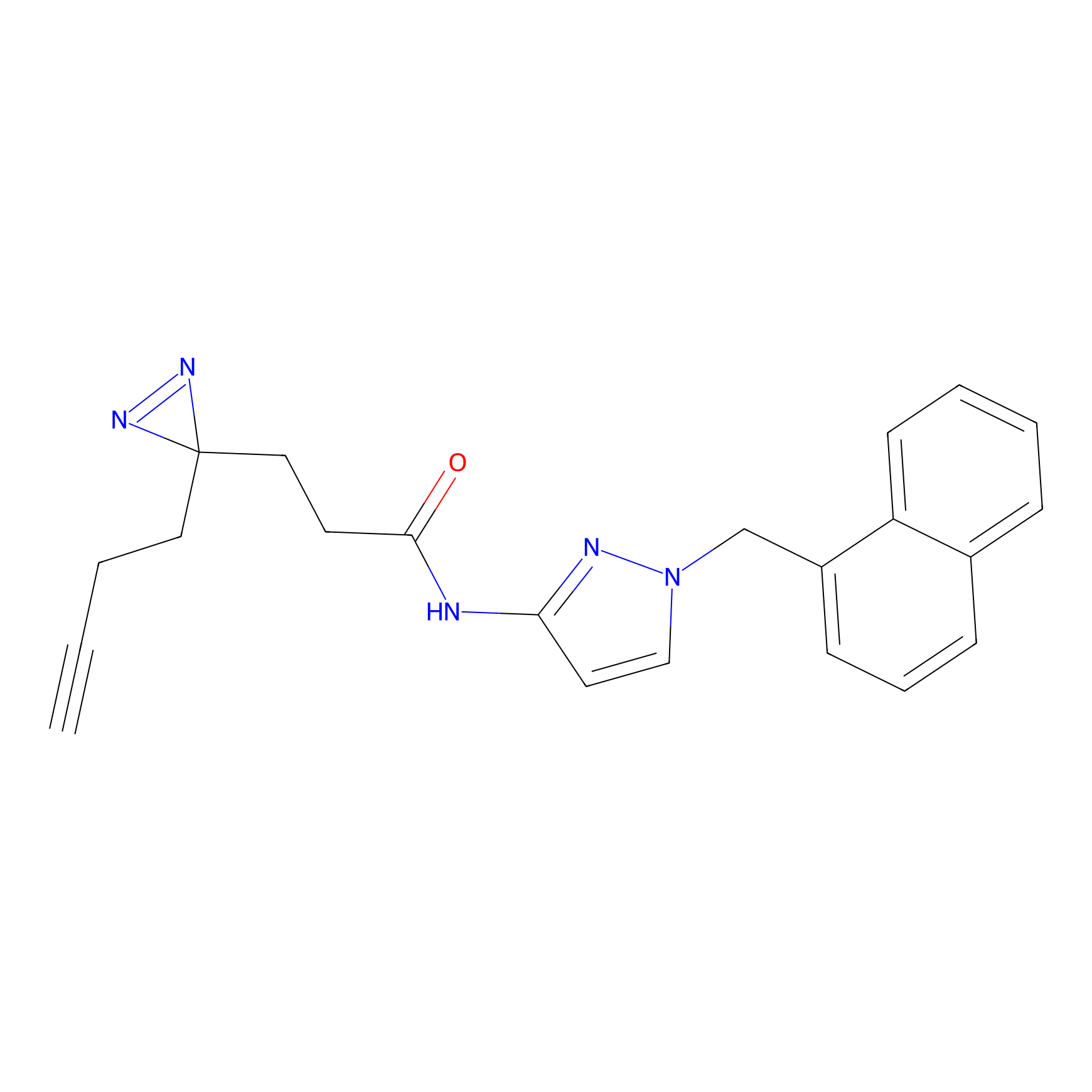
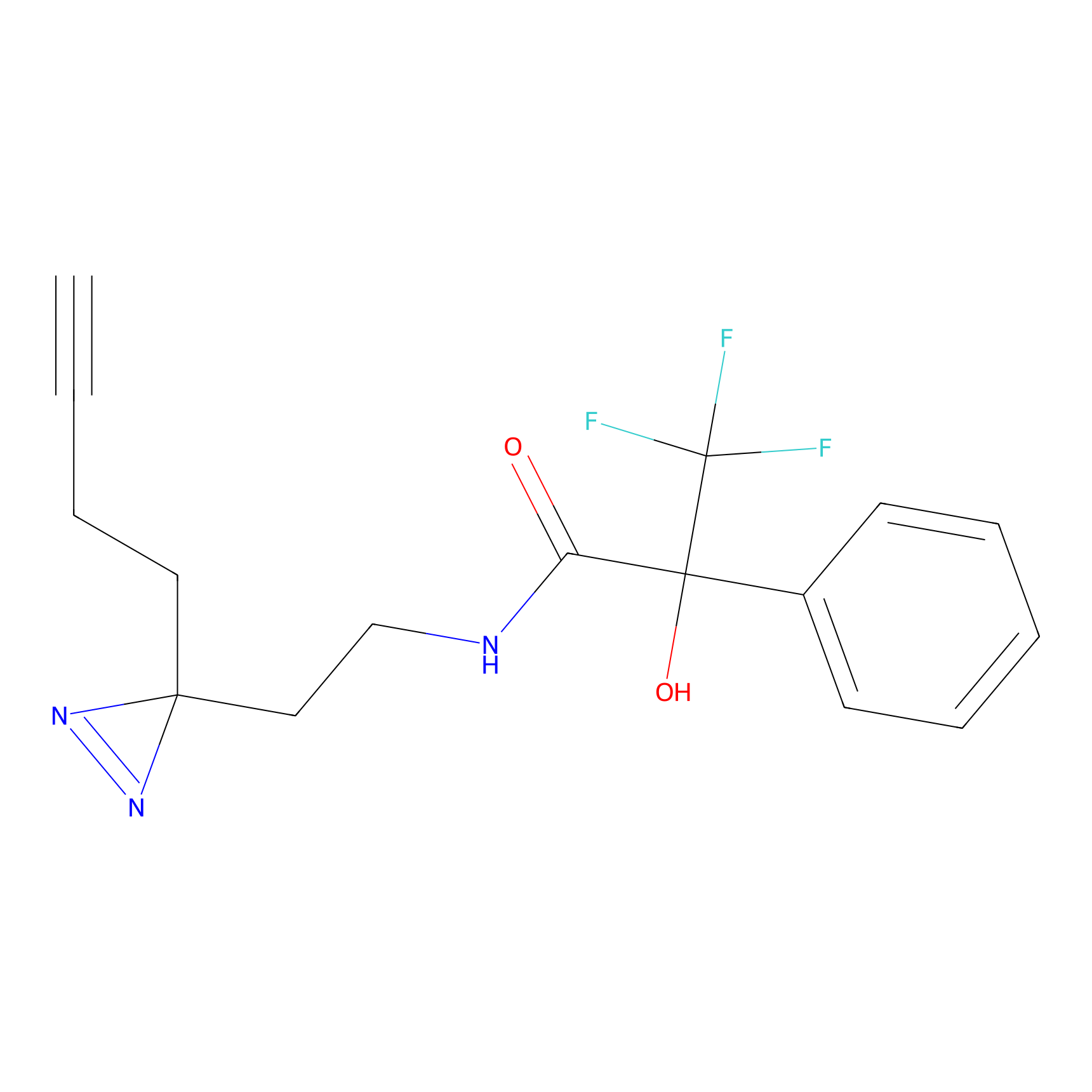
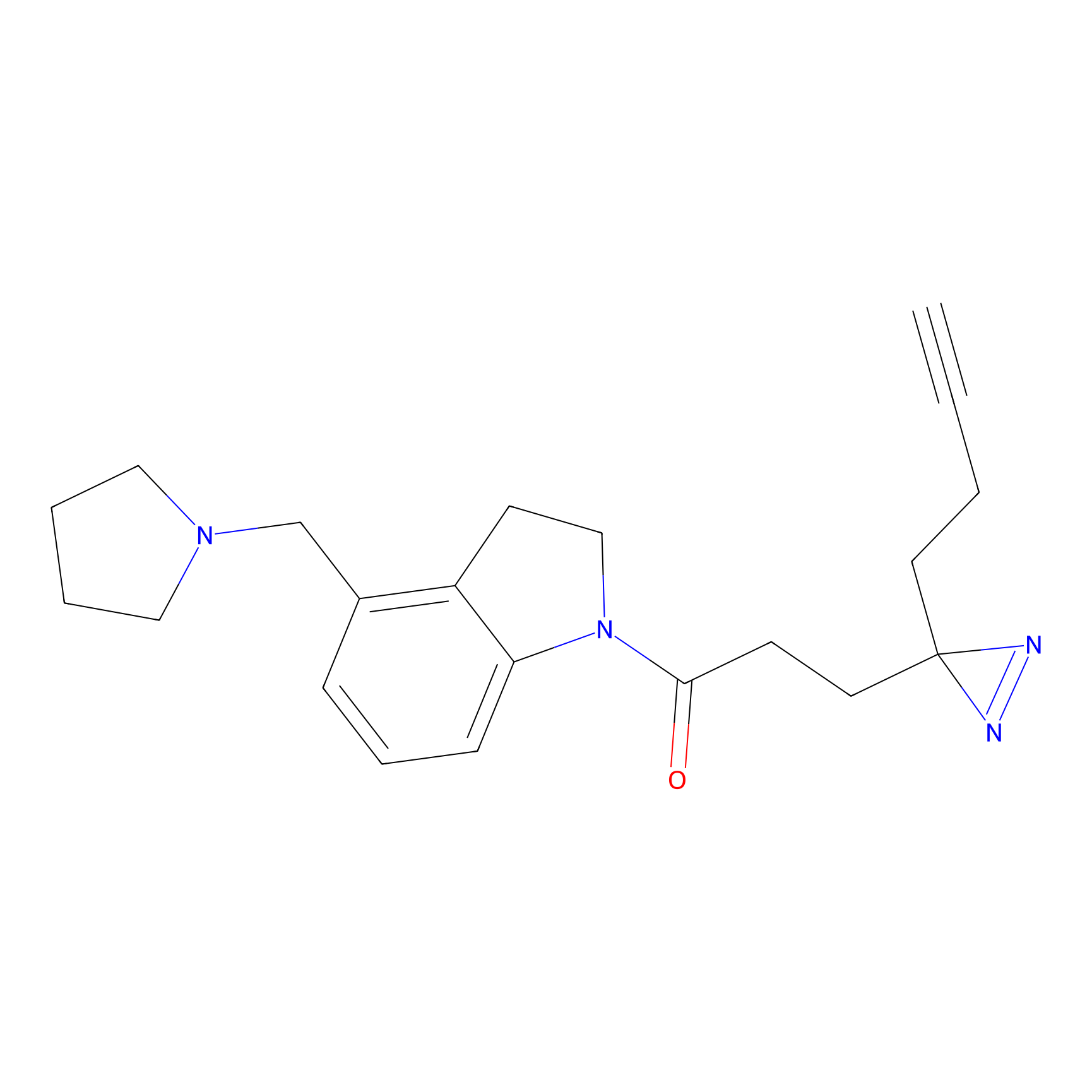
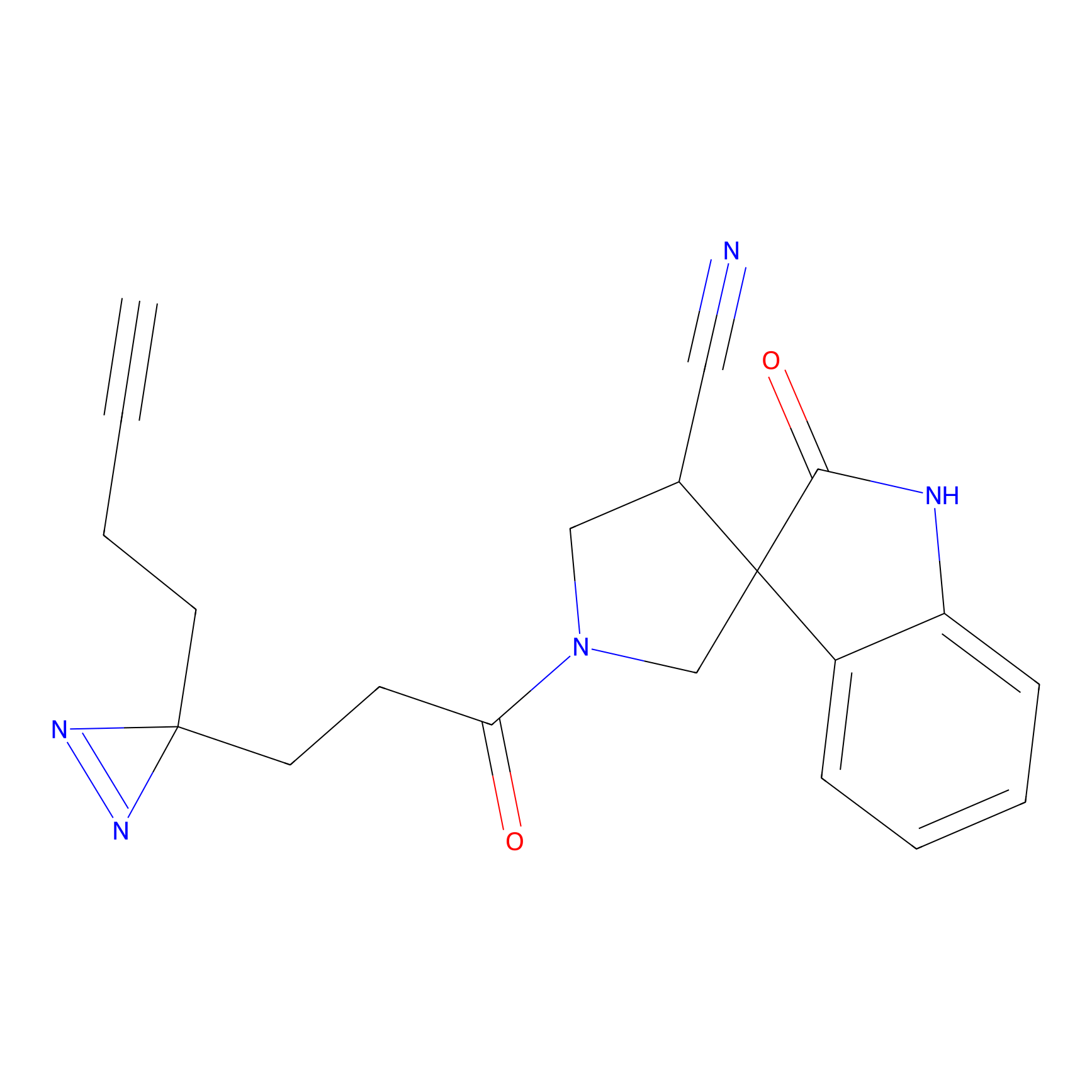
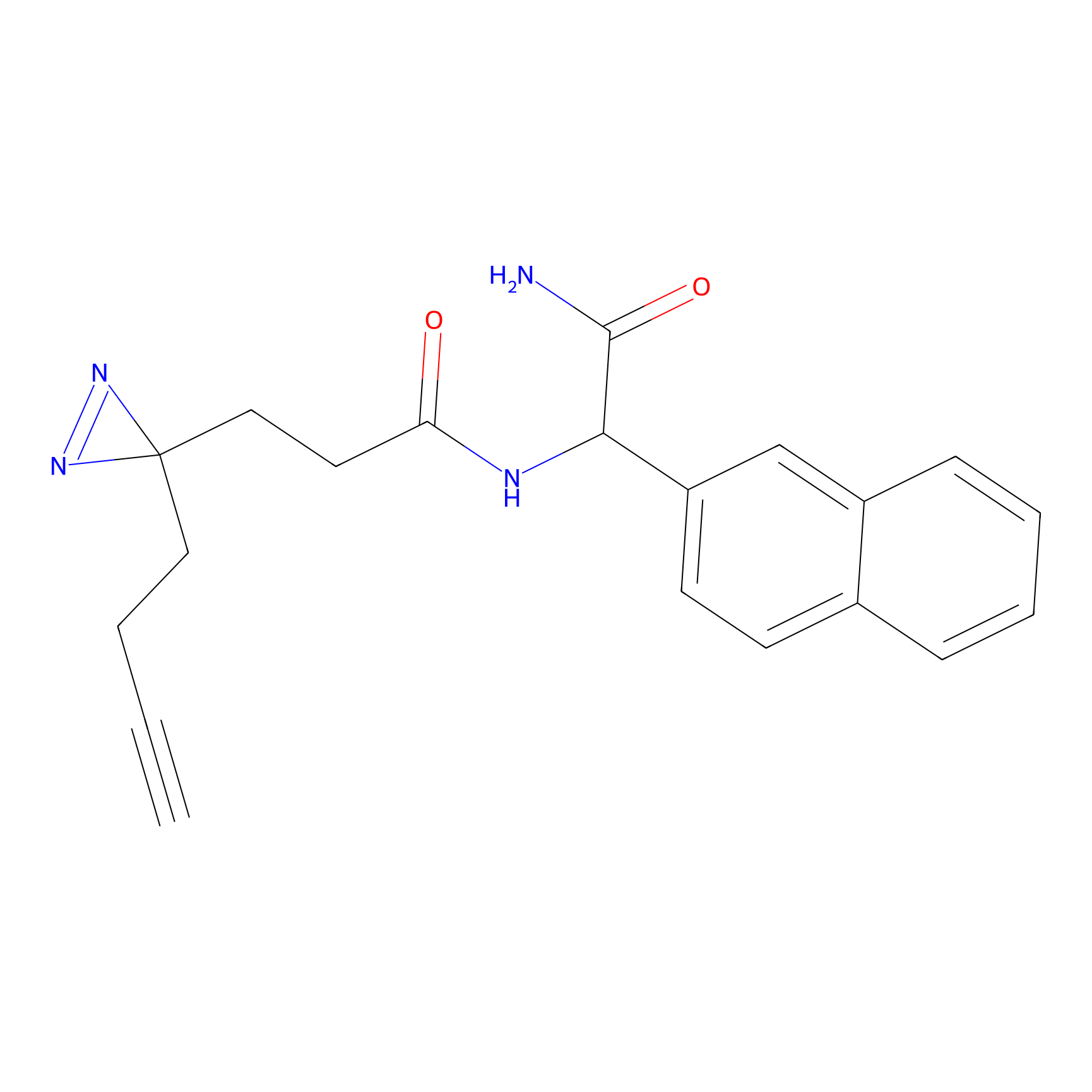
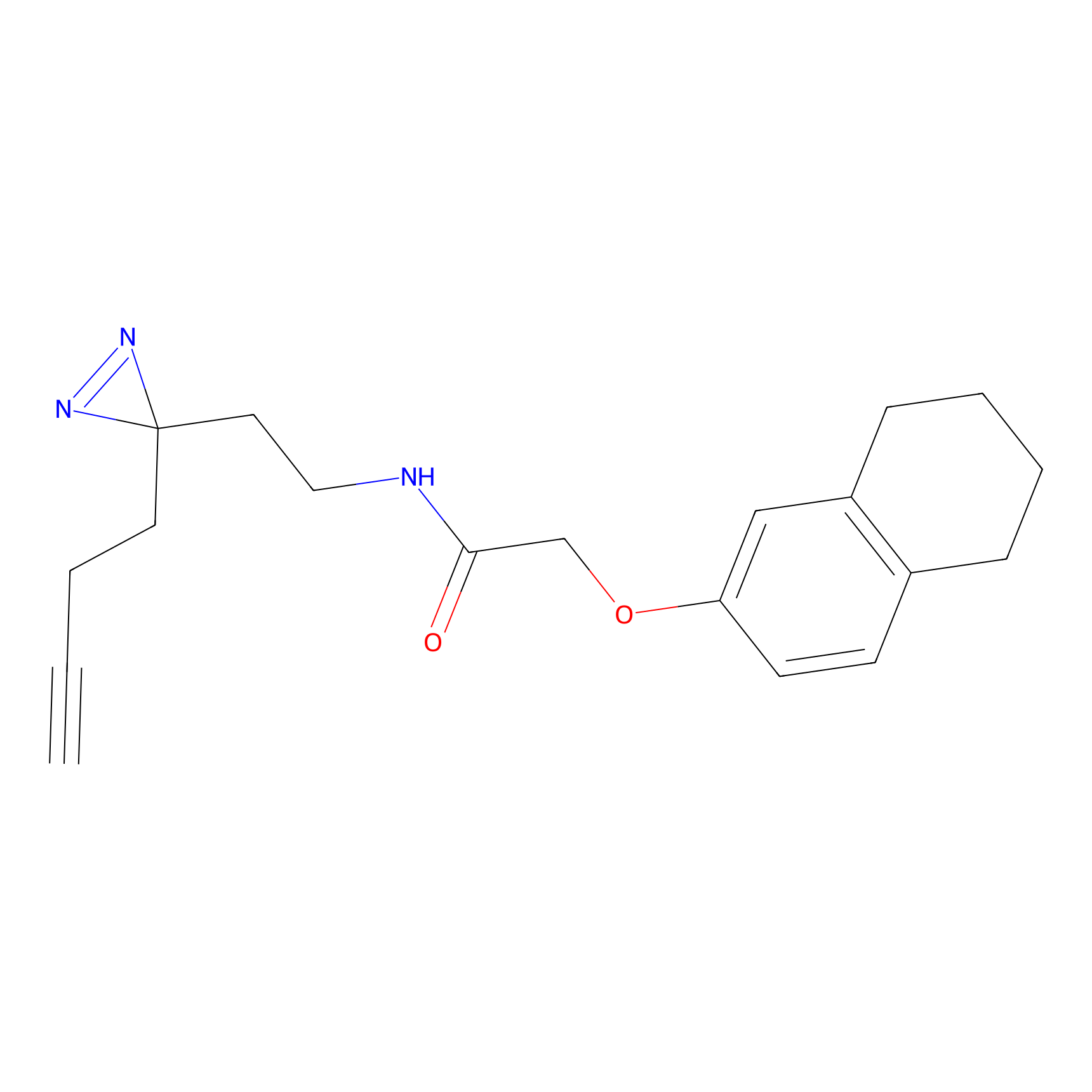
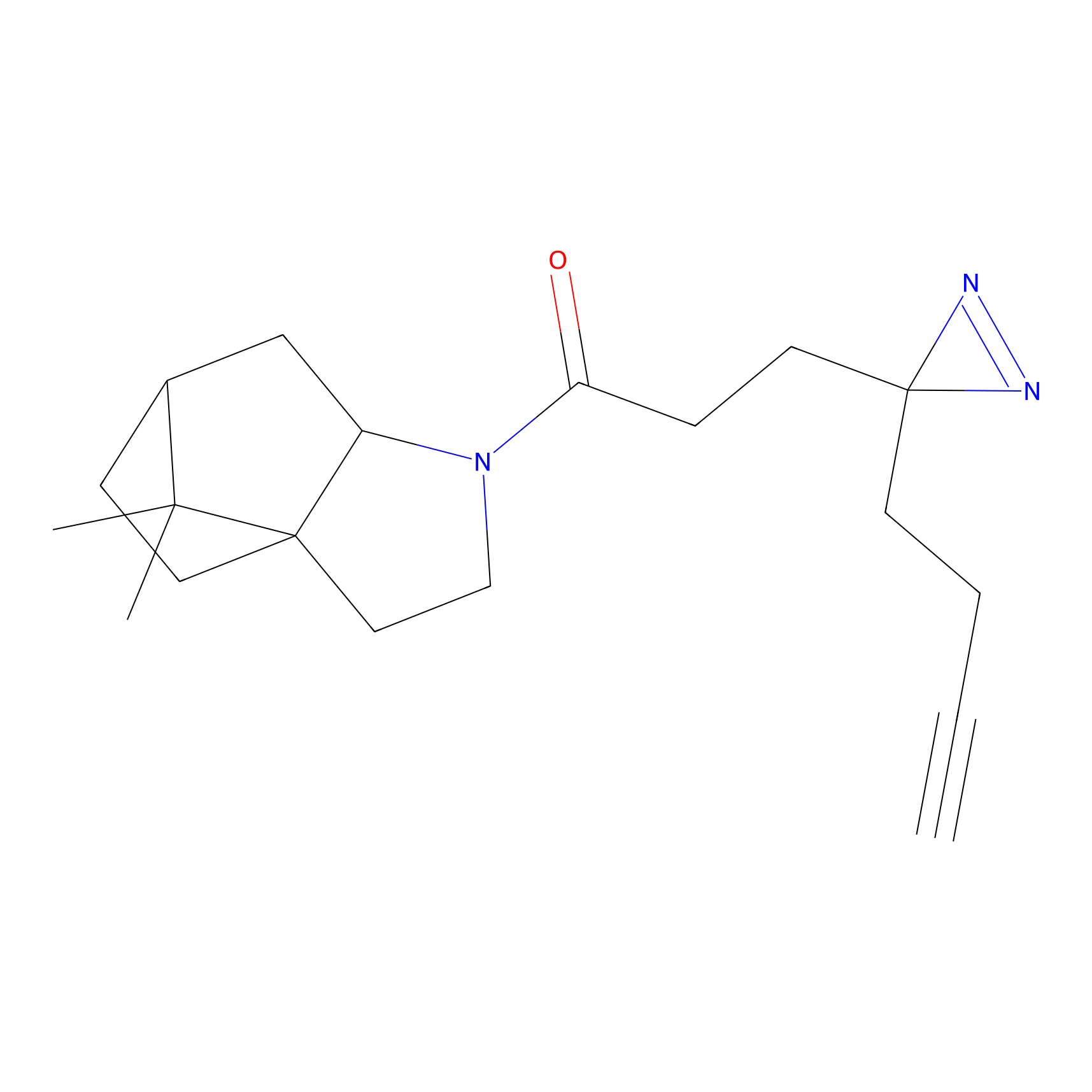
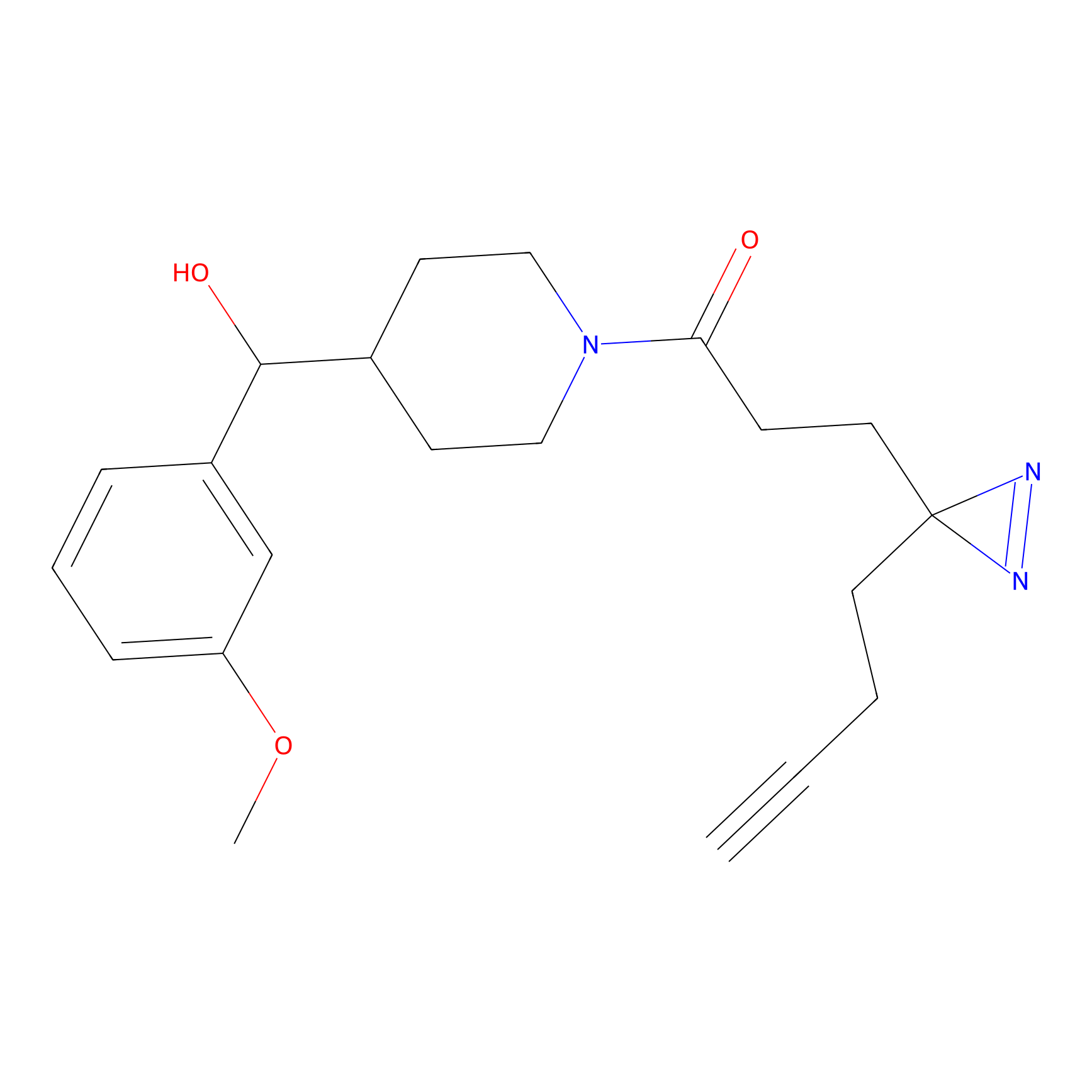
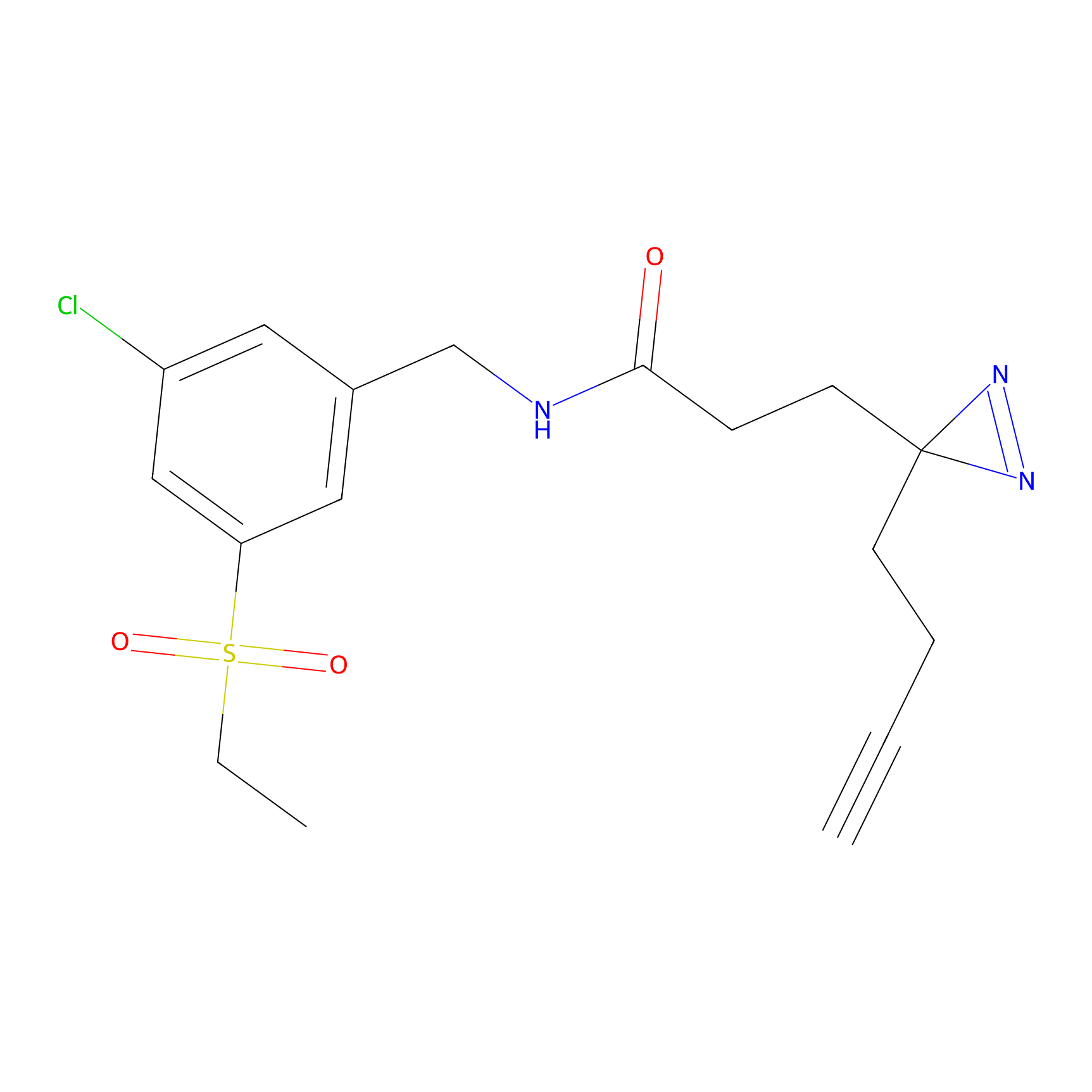
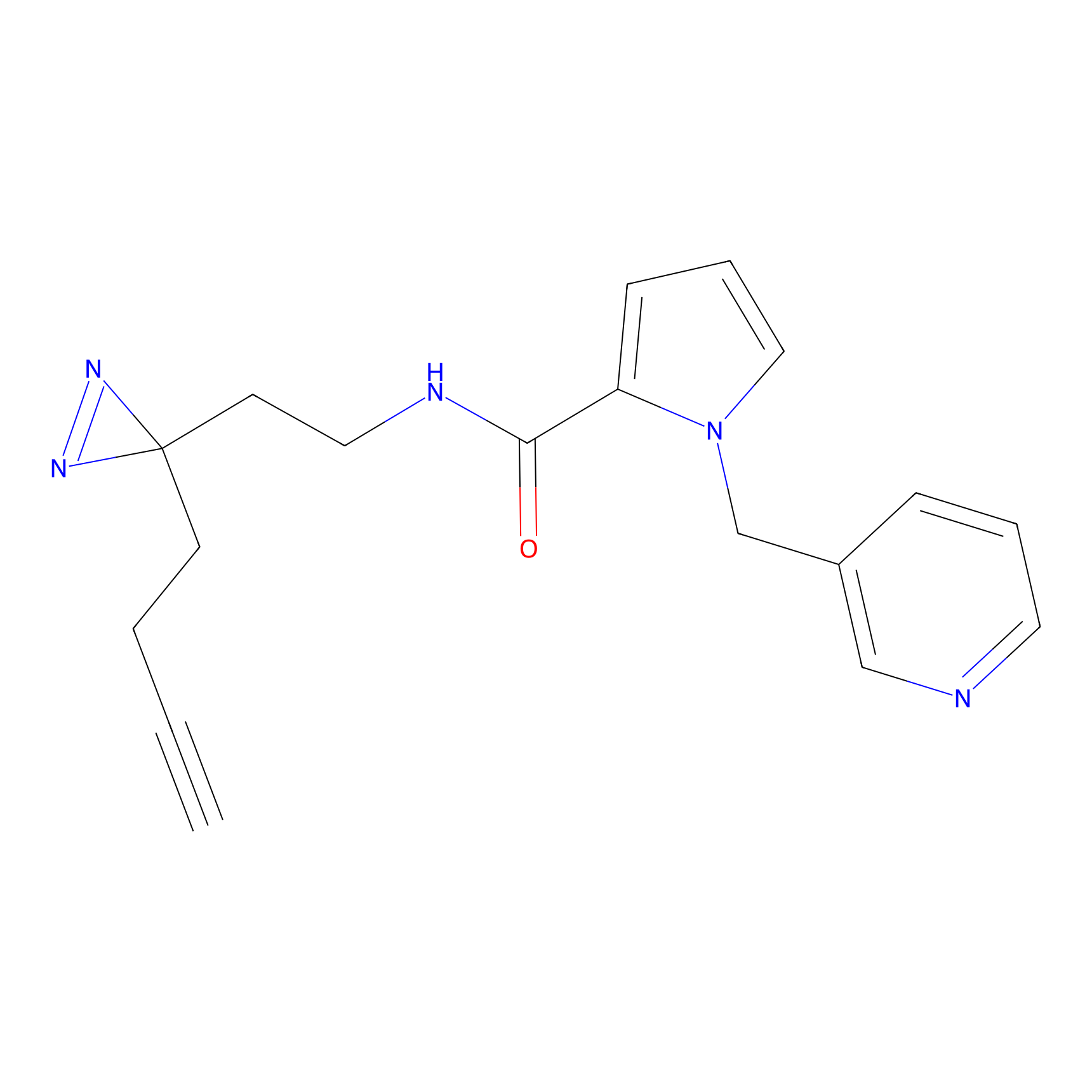
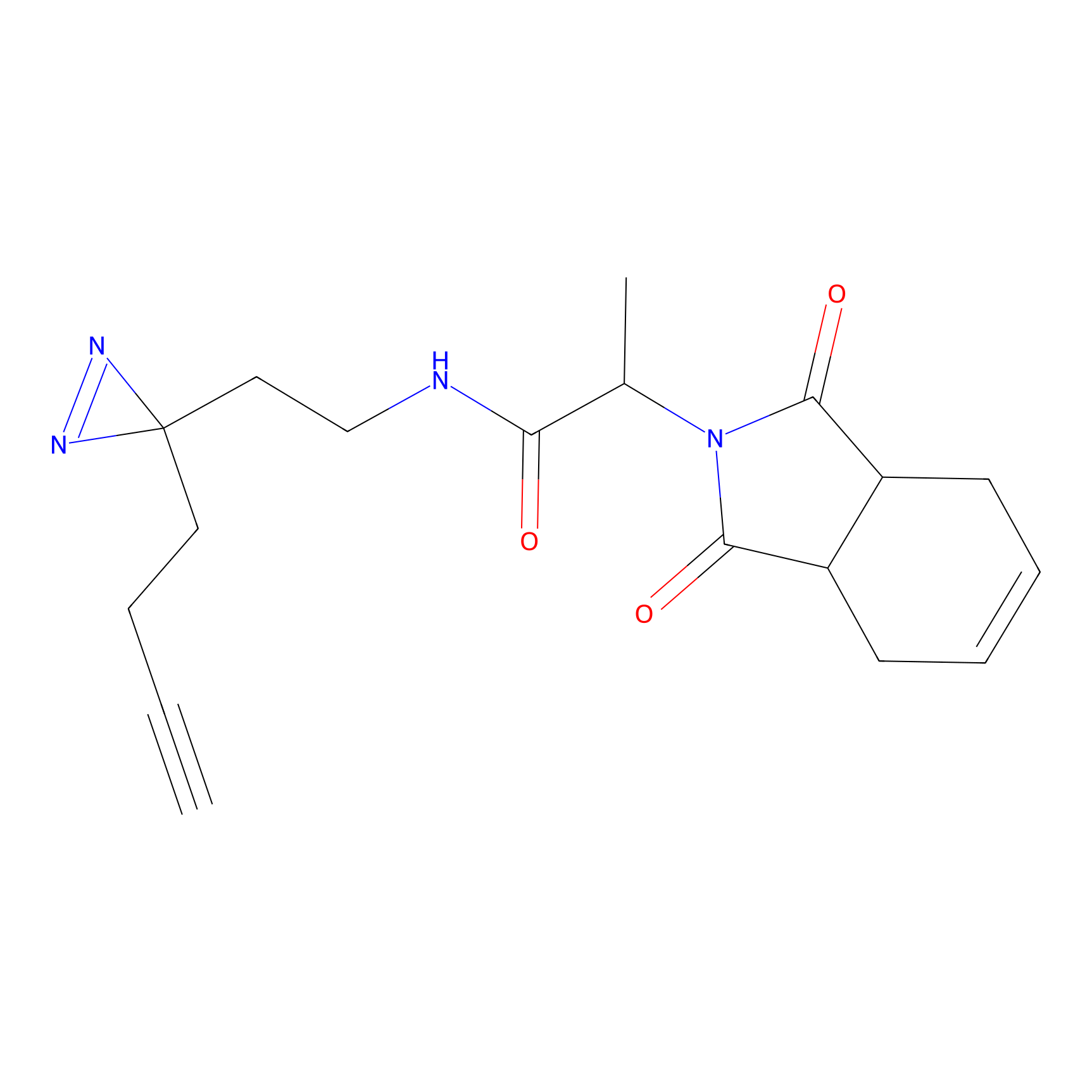
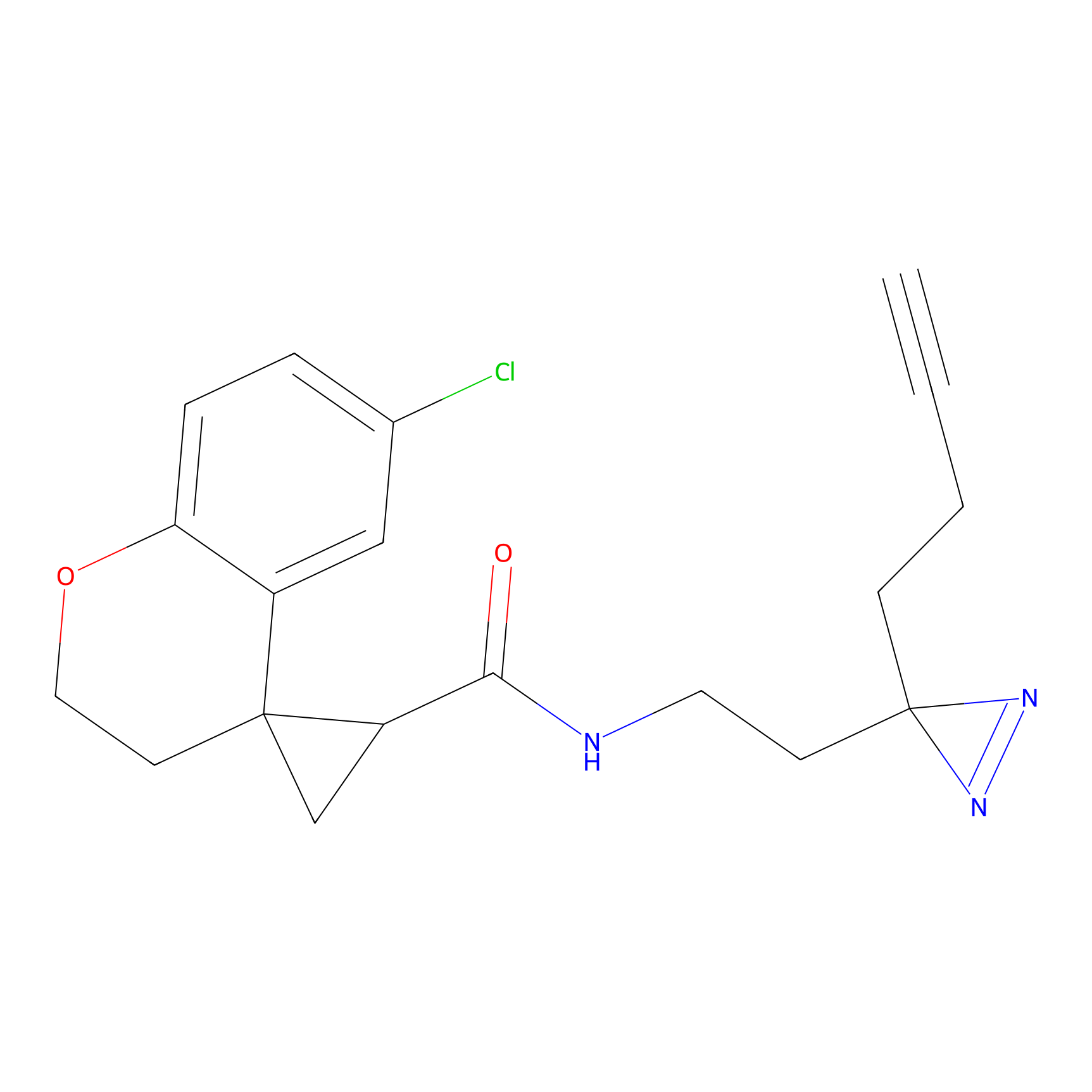
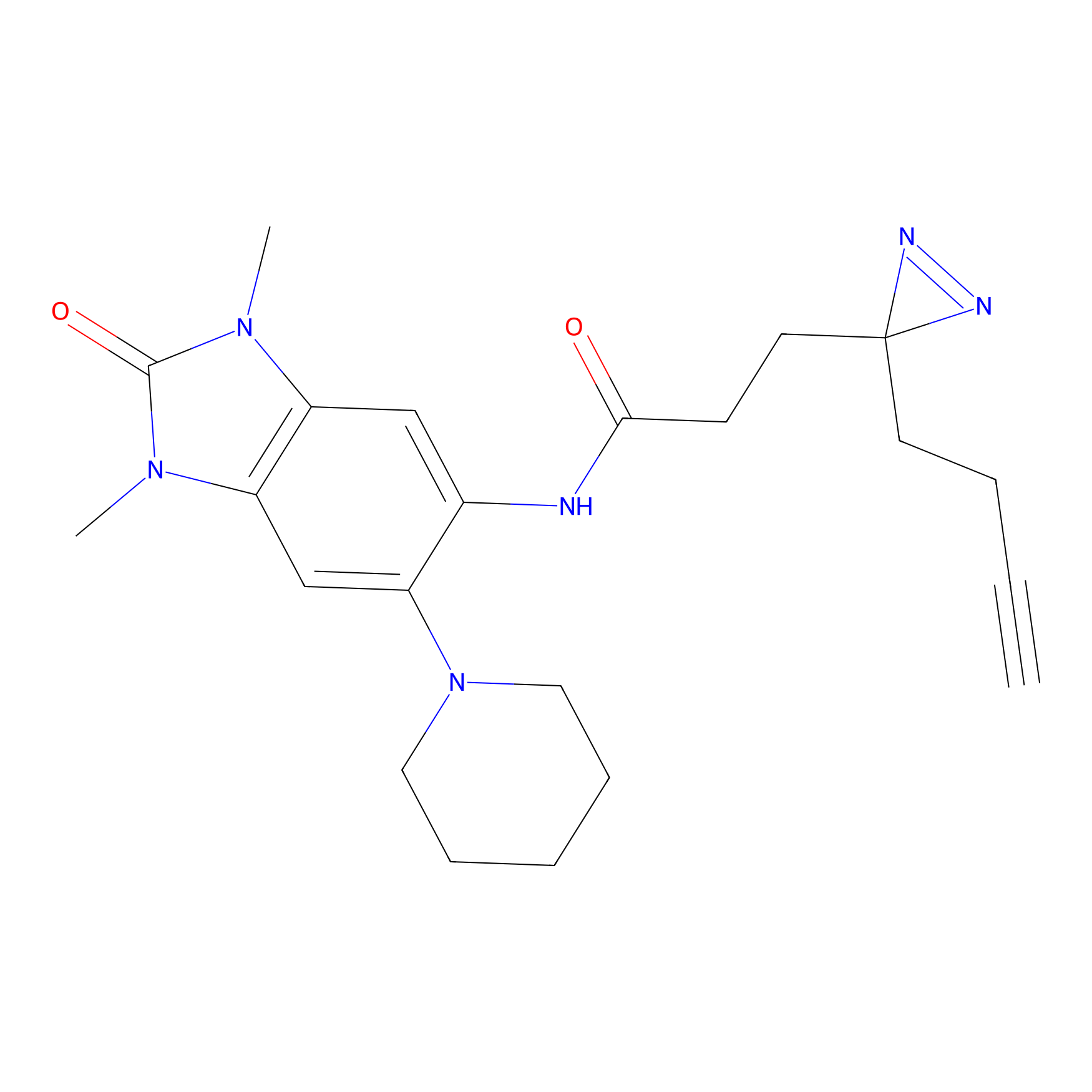
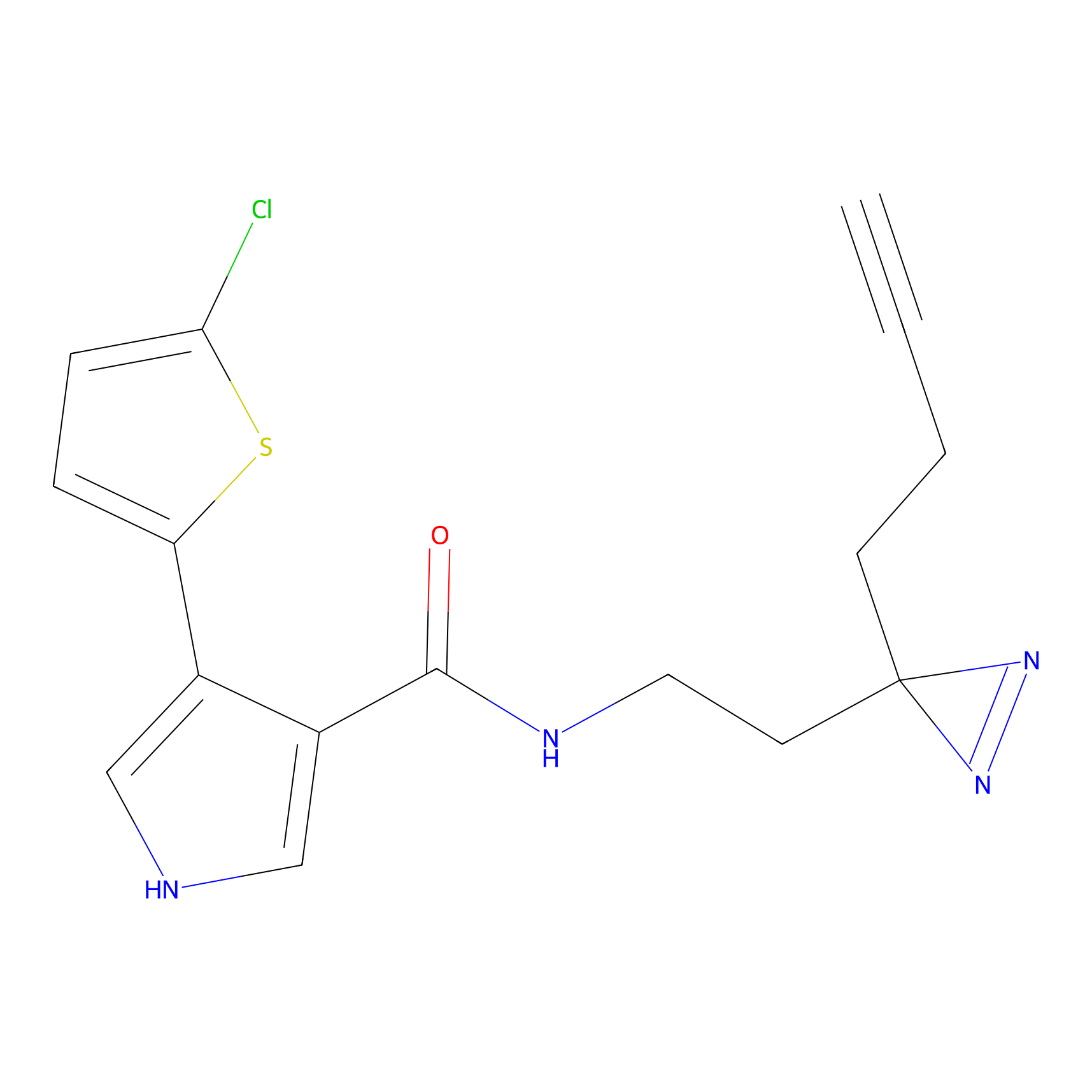
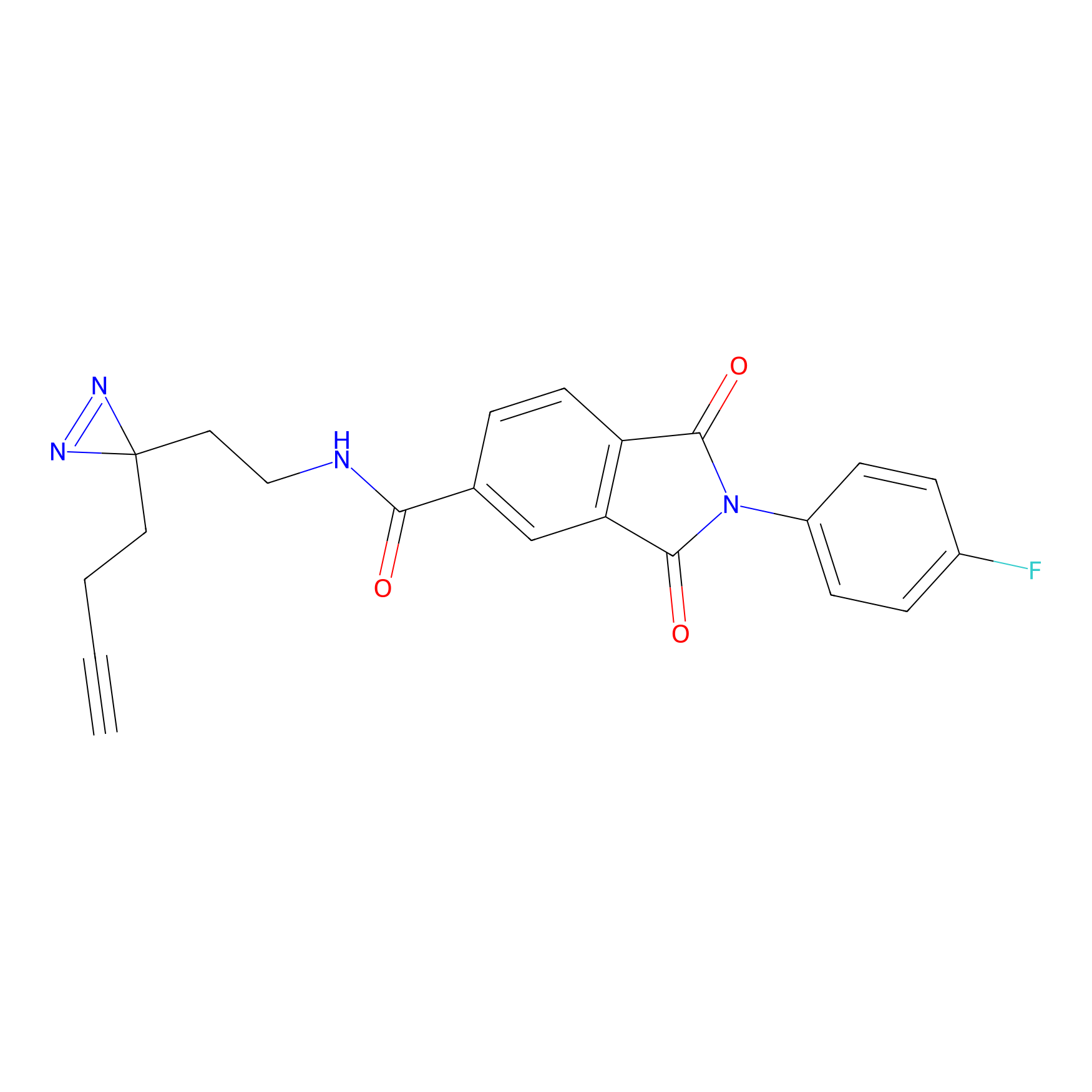
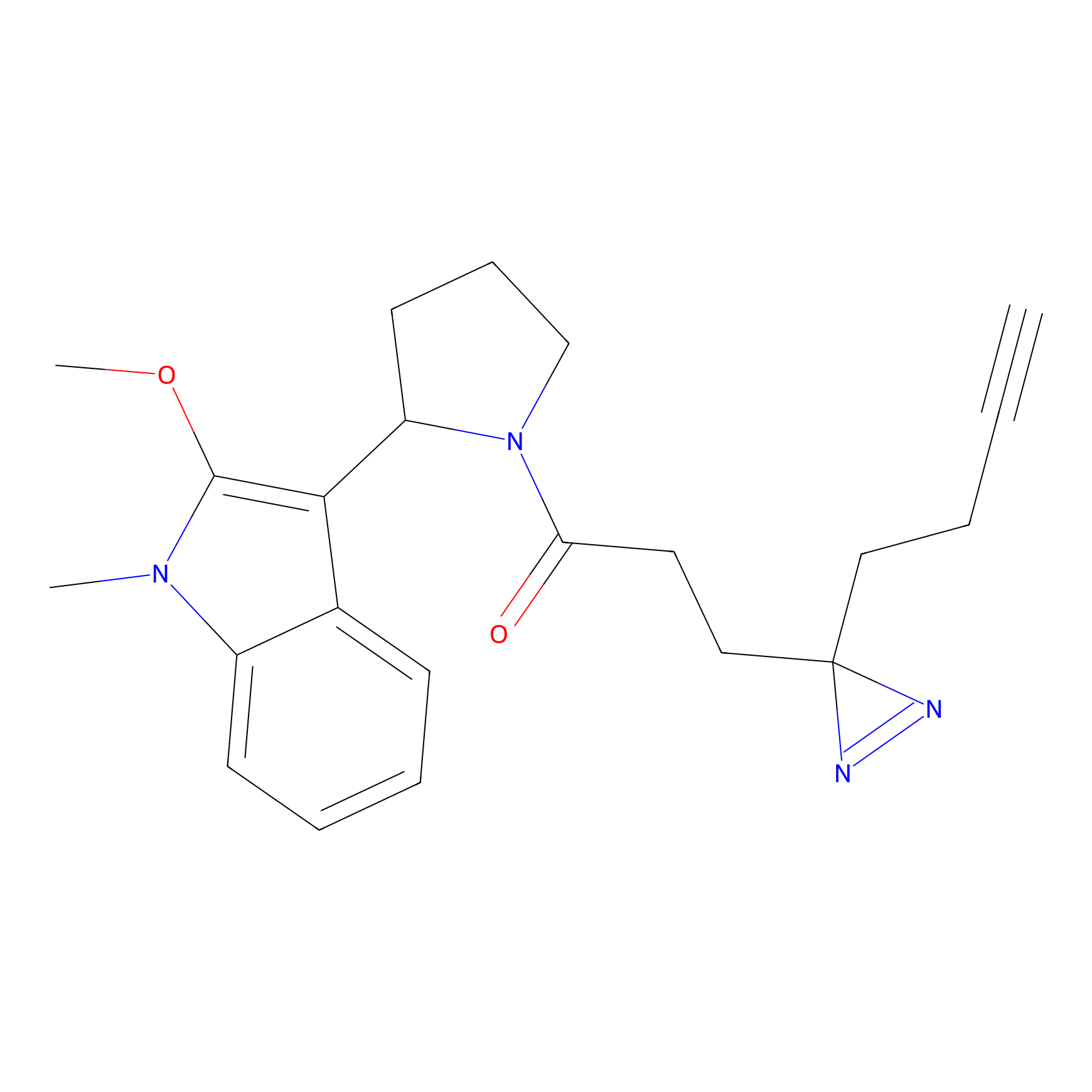
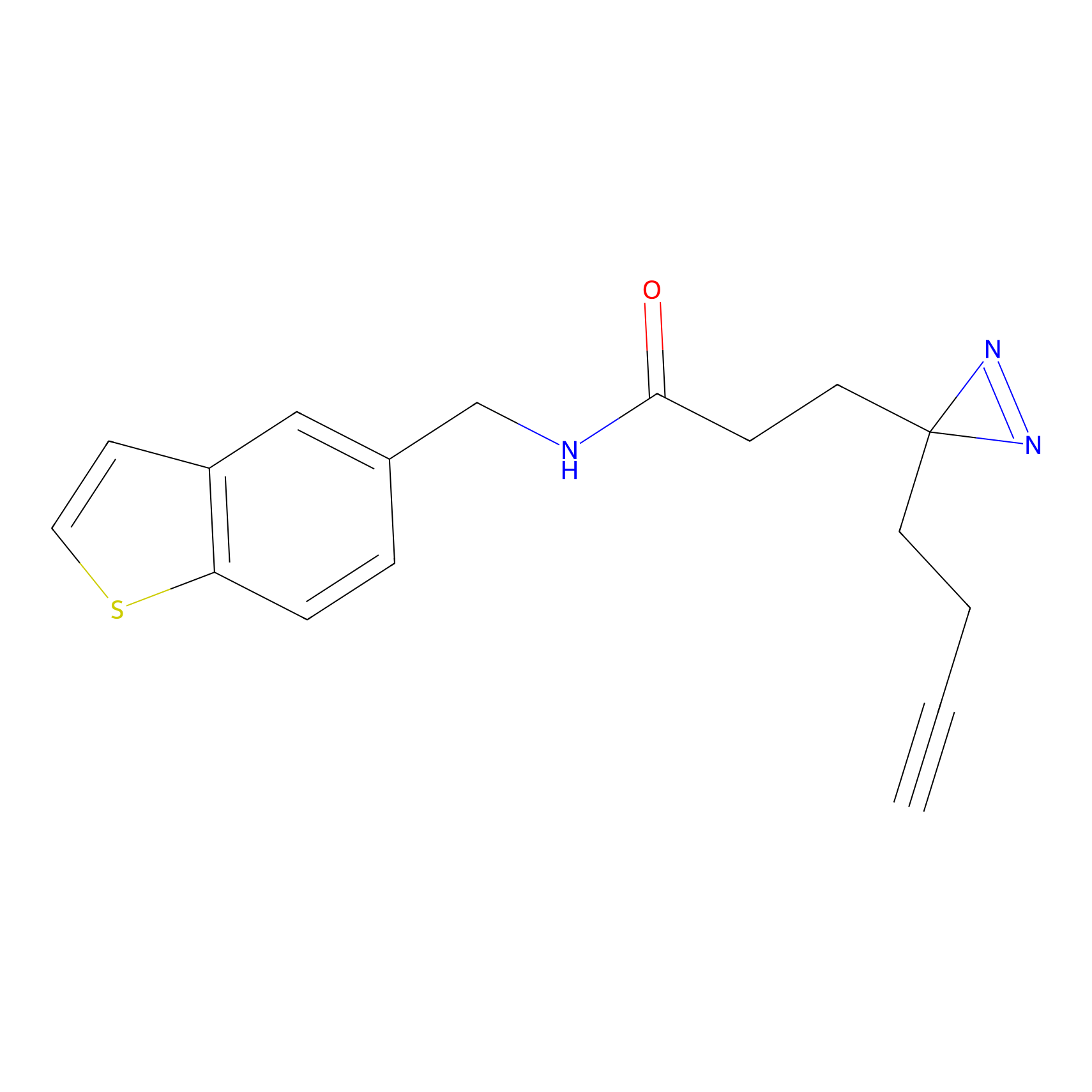
|
FBPP2 Probe Info |
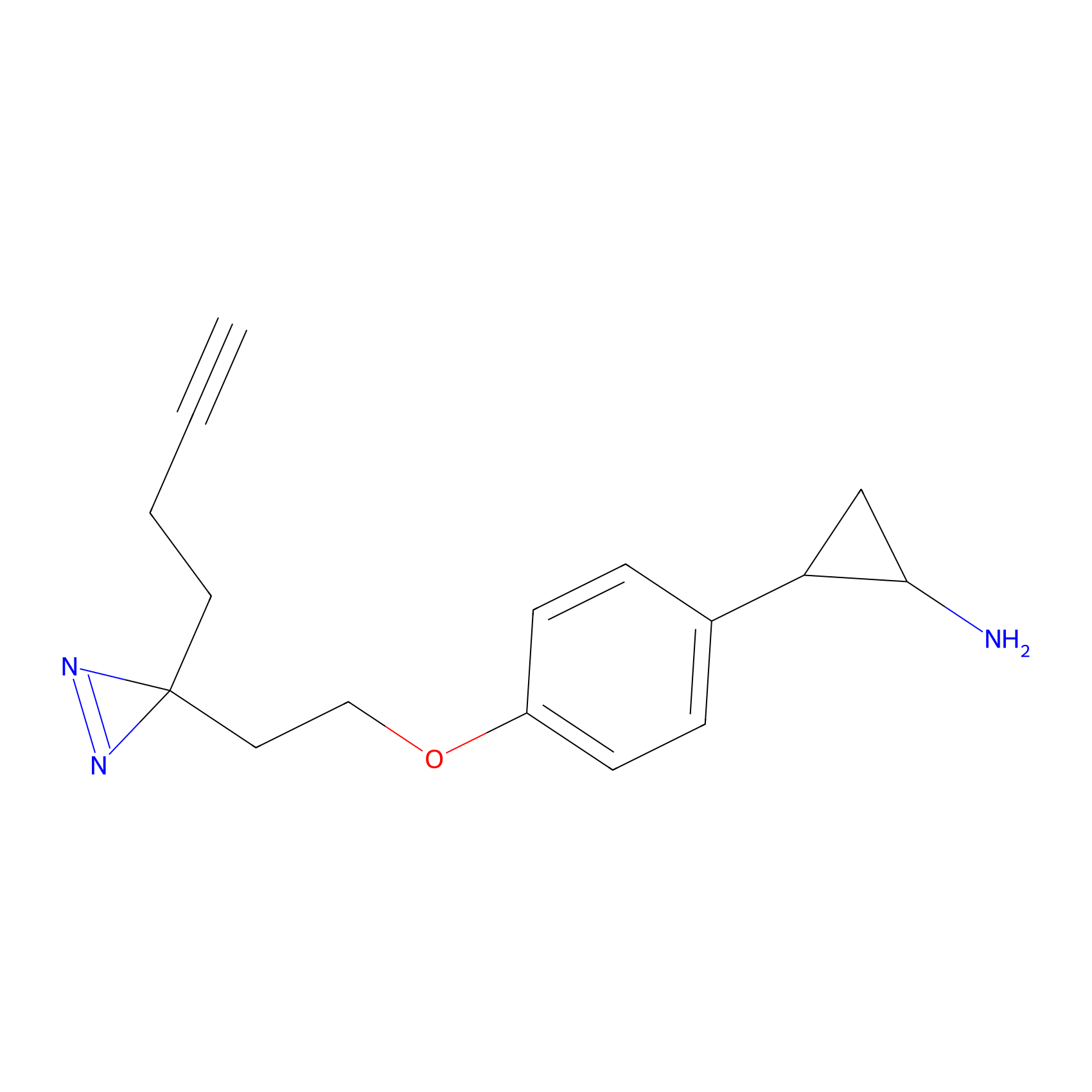 |
2.05 | LDD0318 | [3] | |
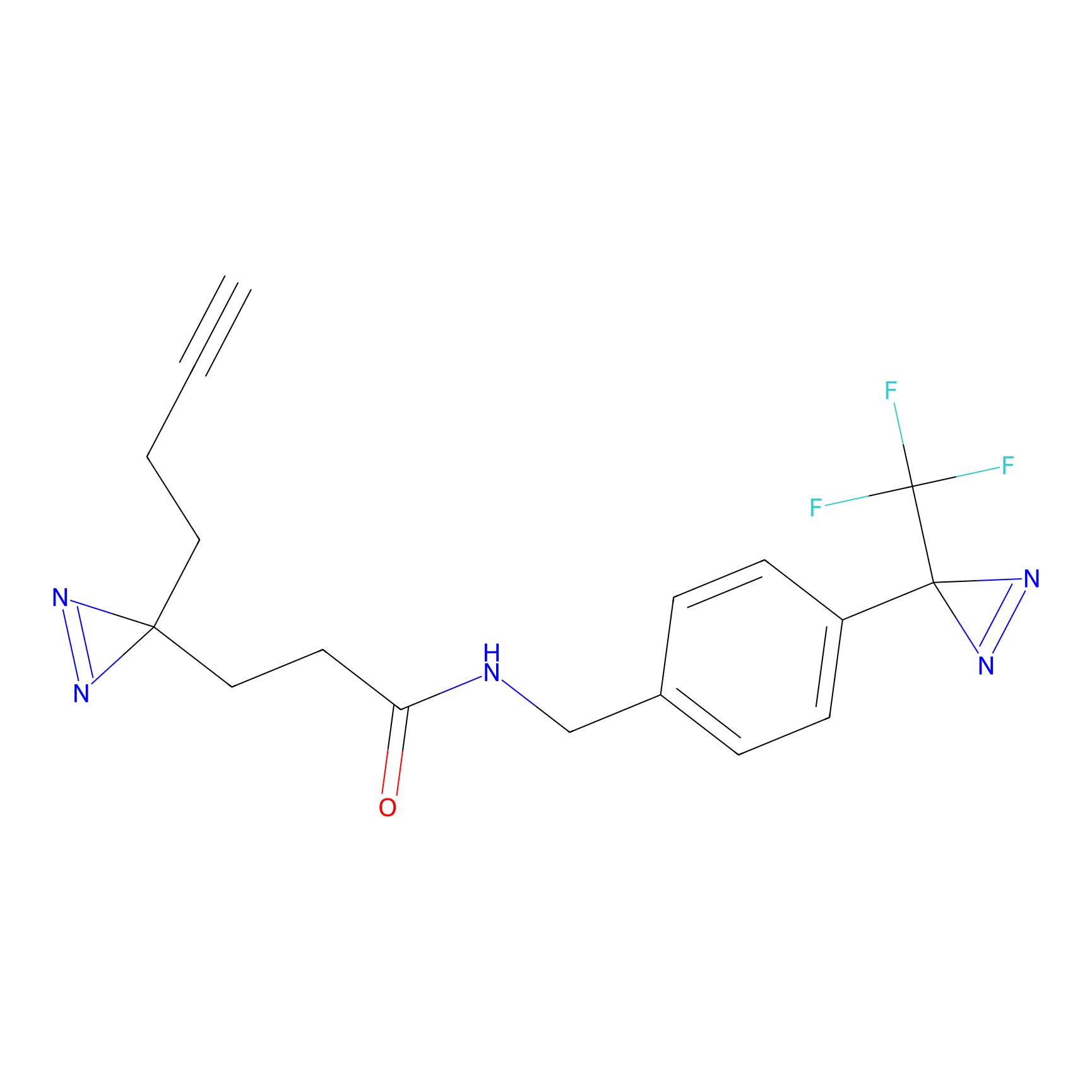
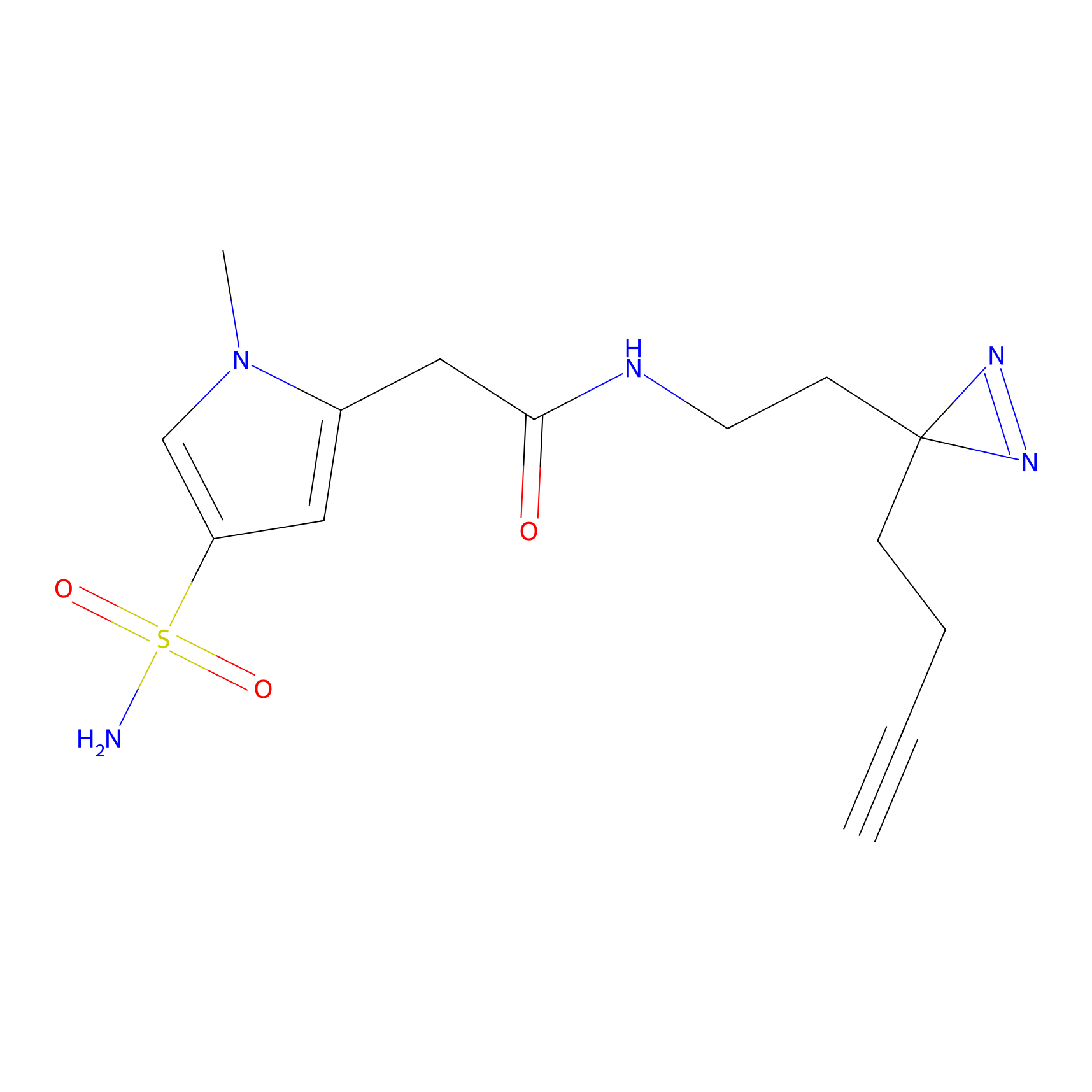
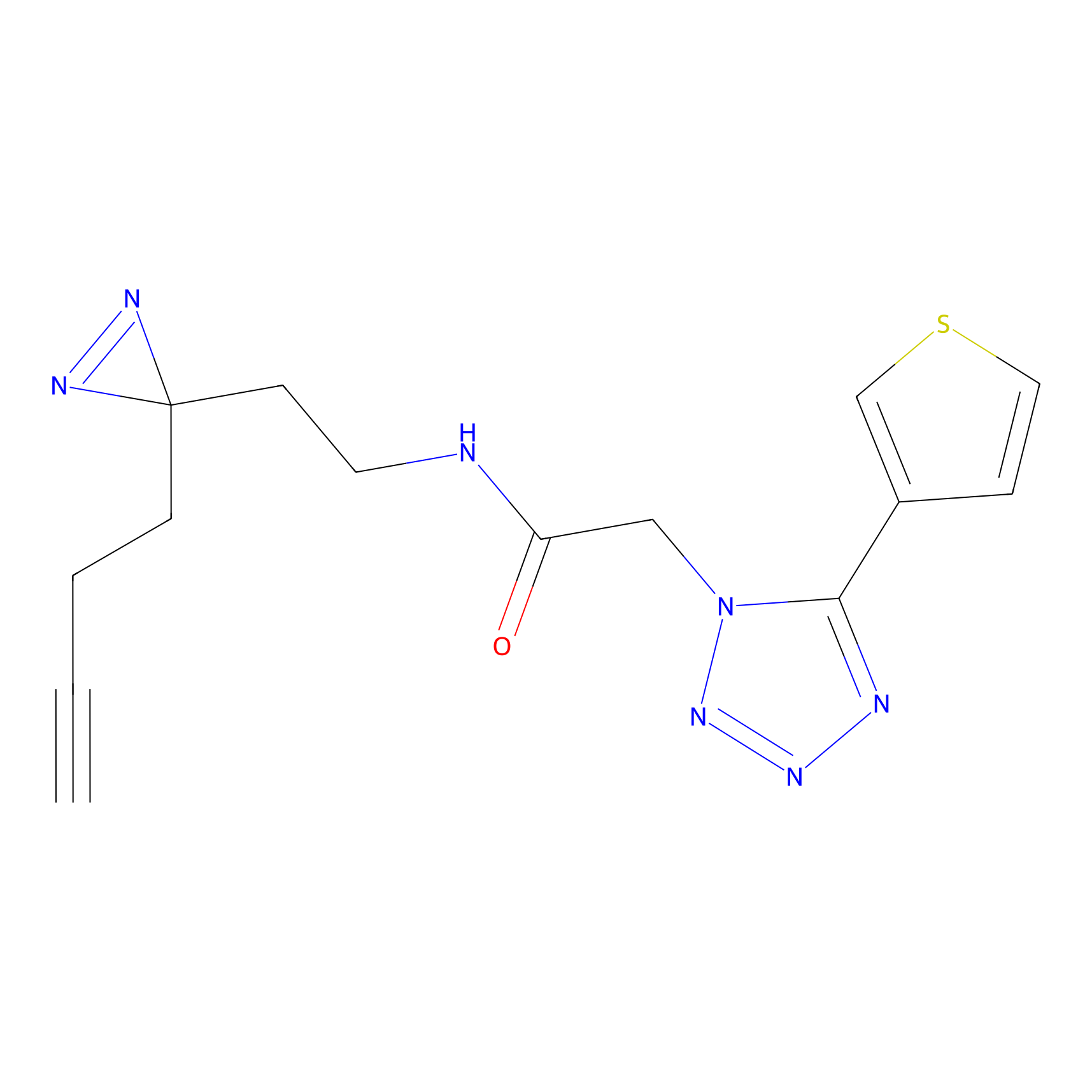
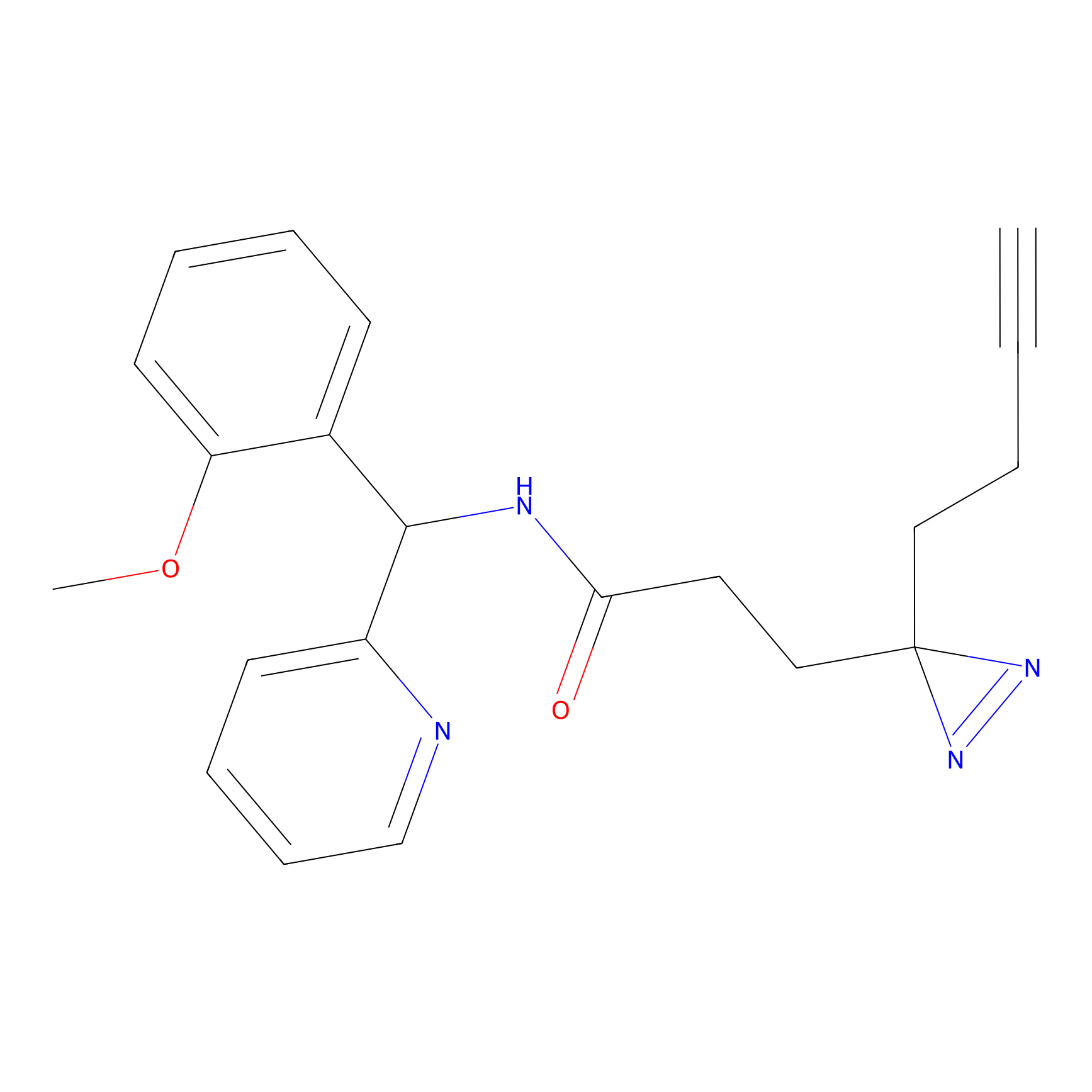
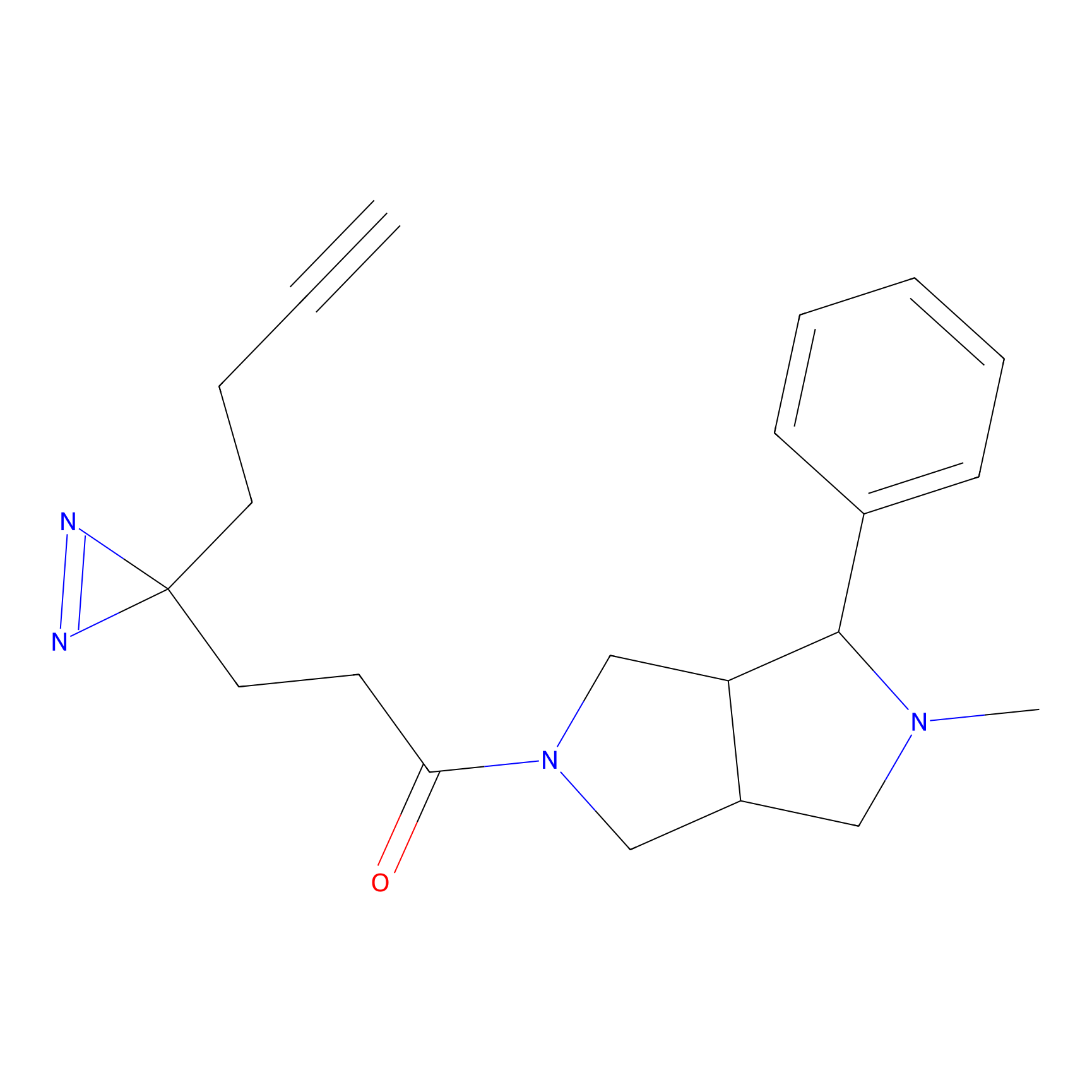
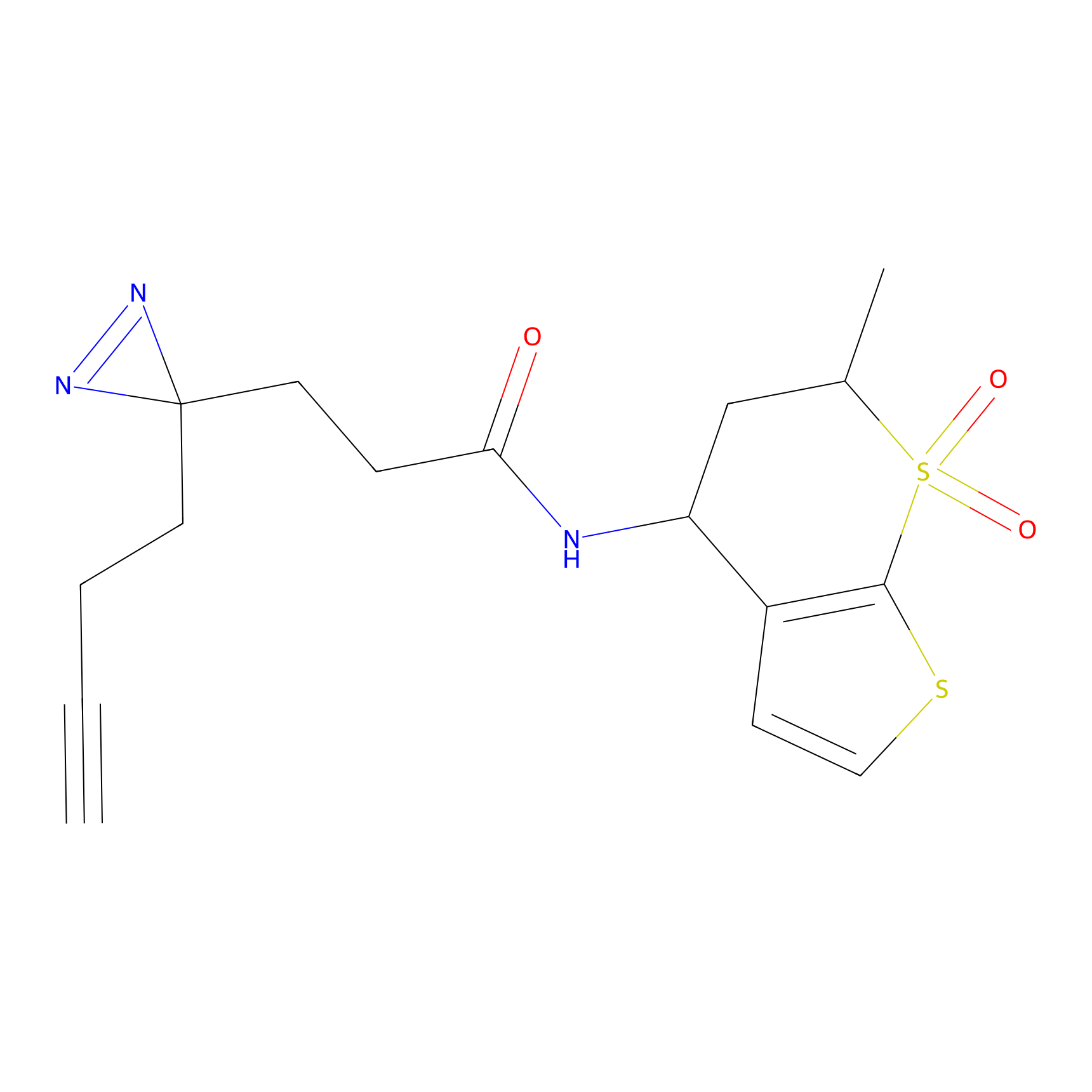
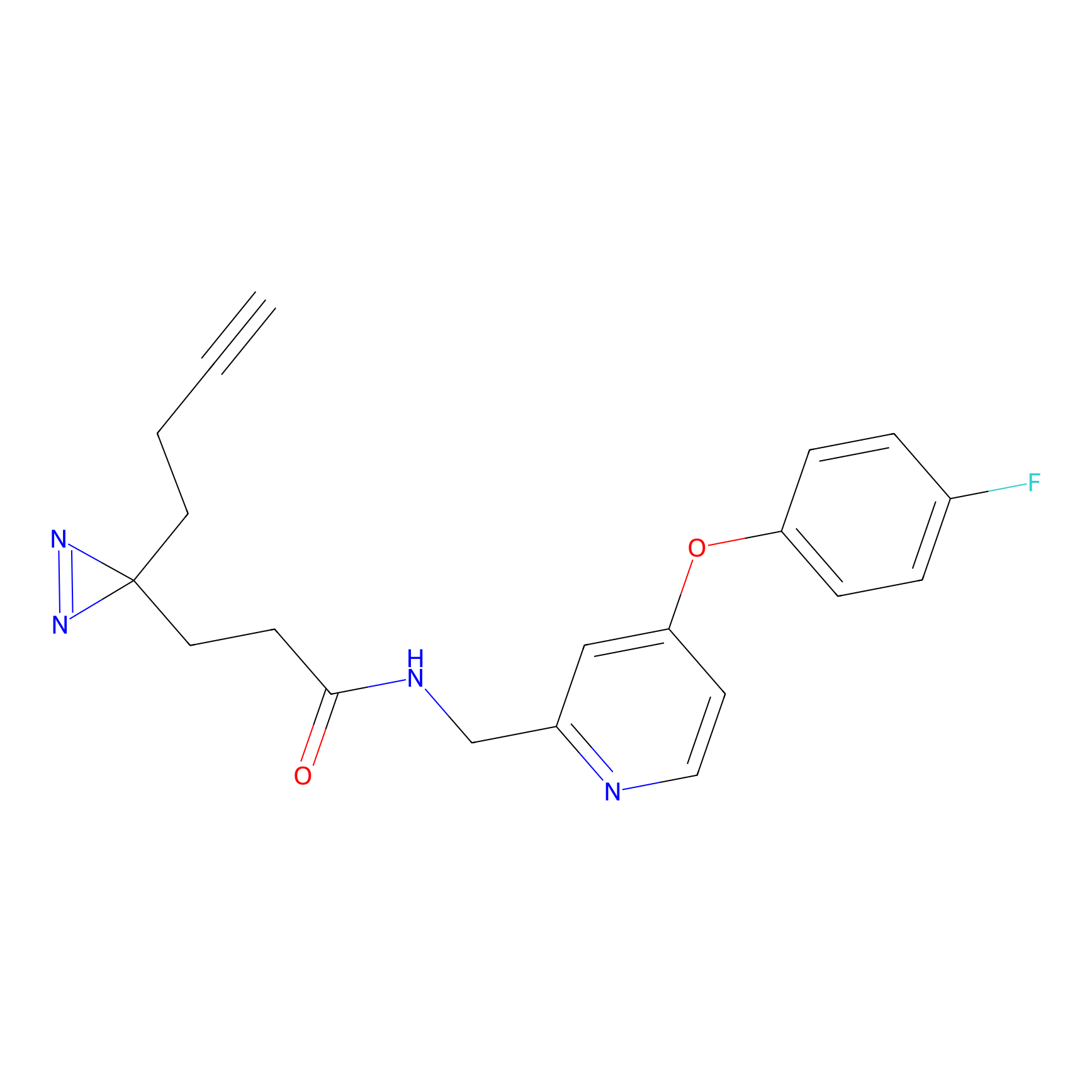
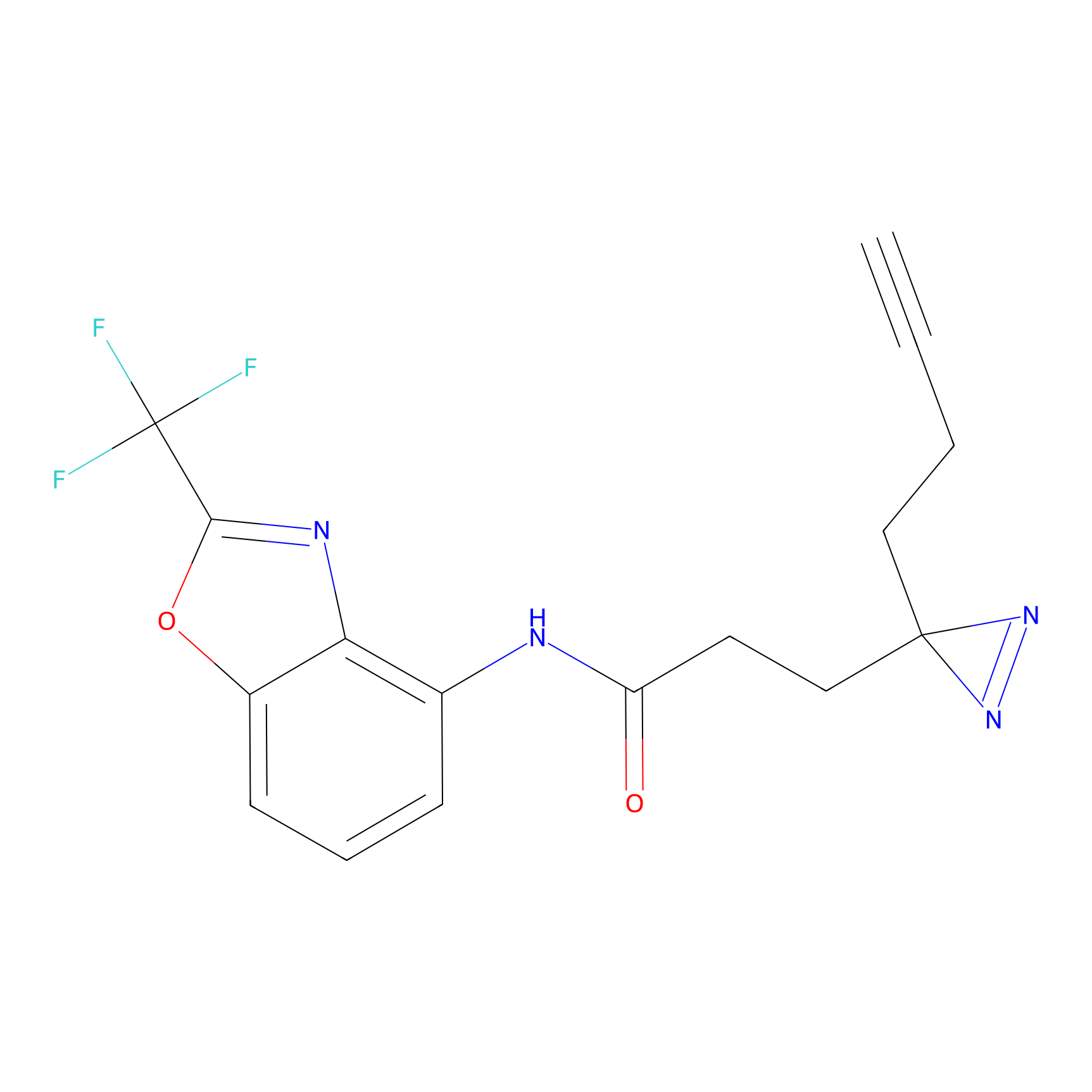
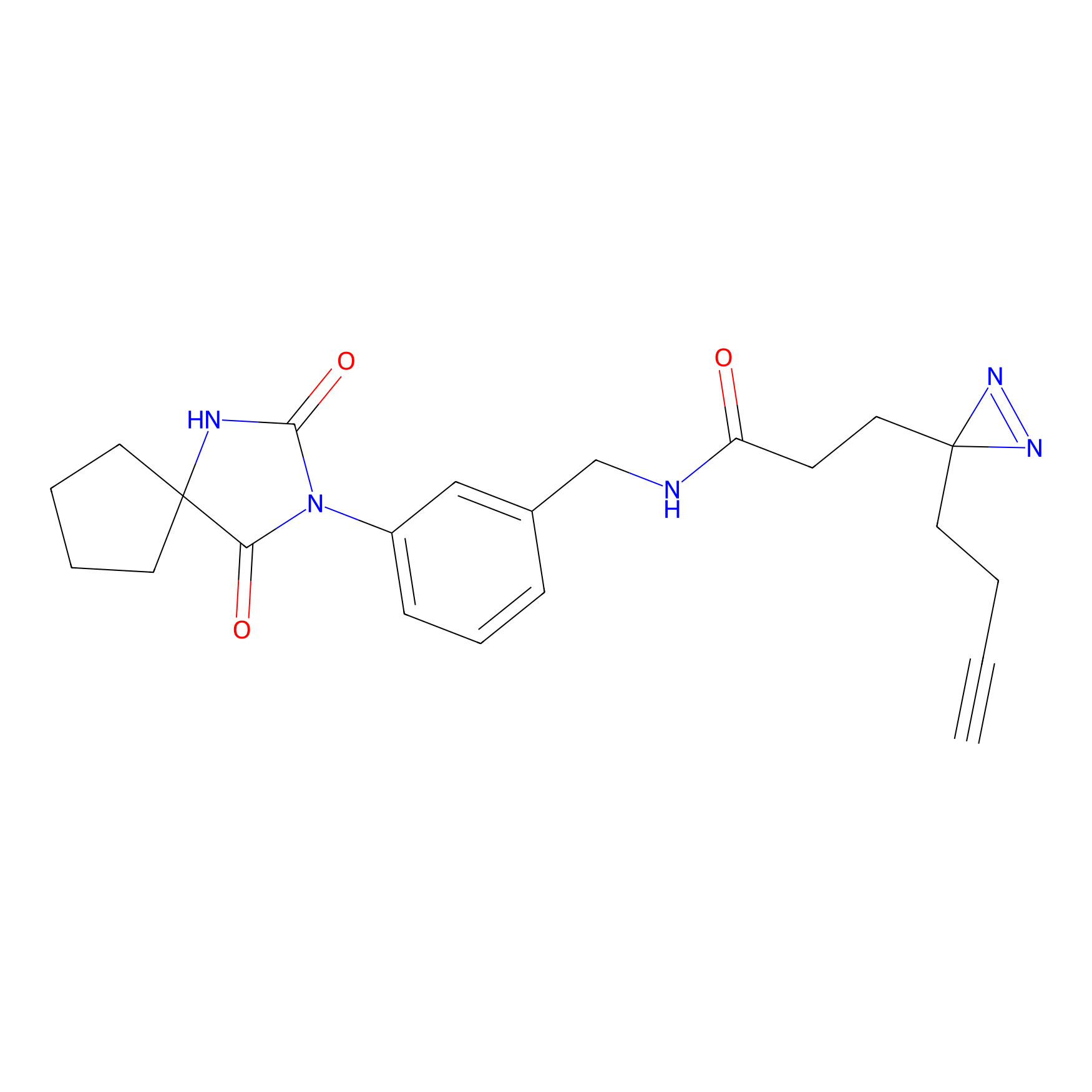
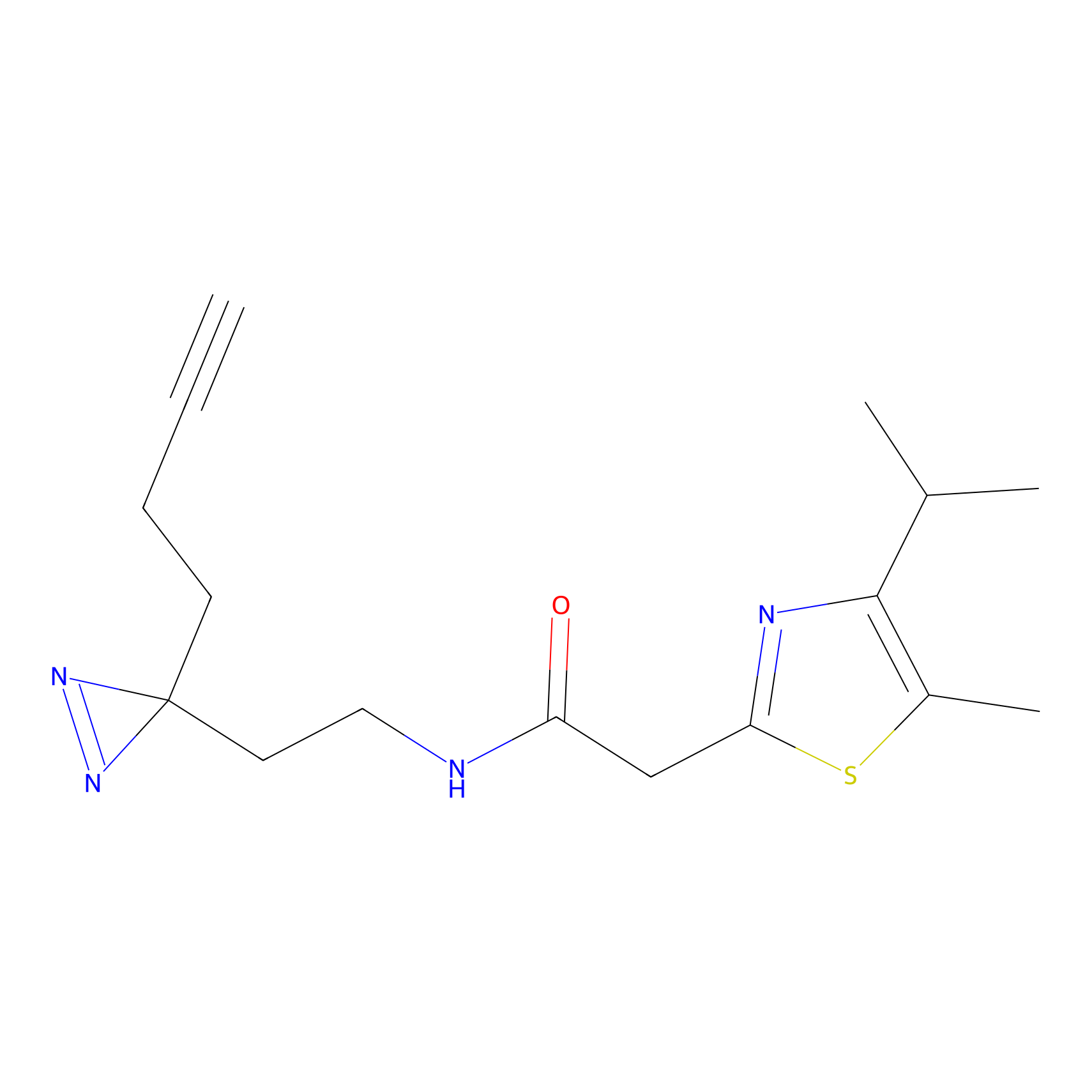
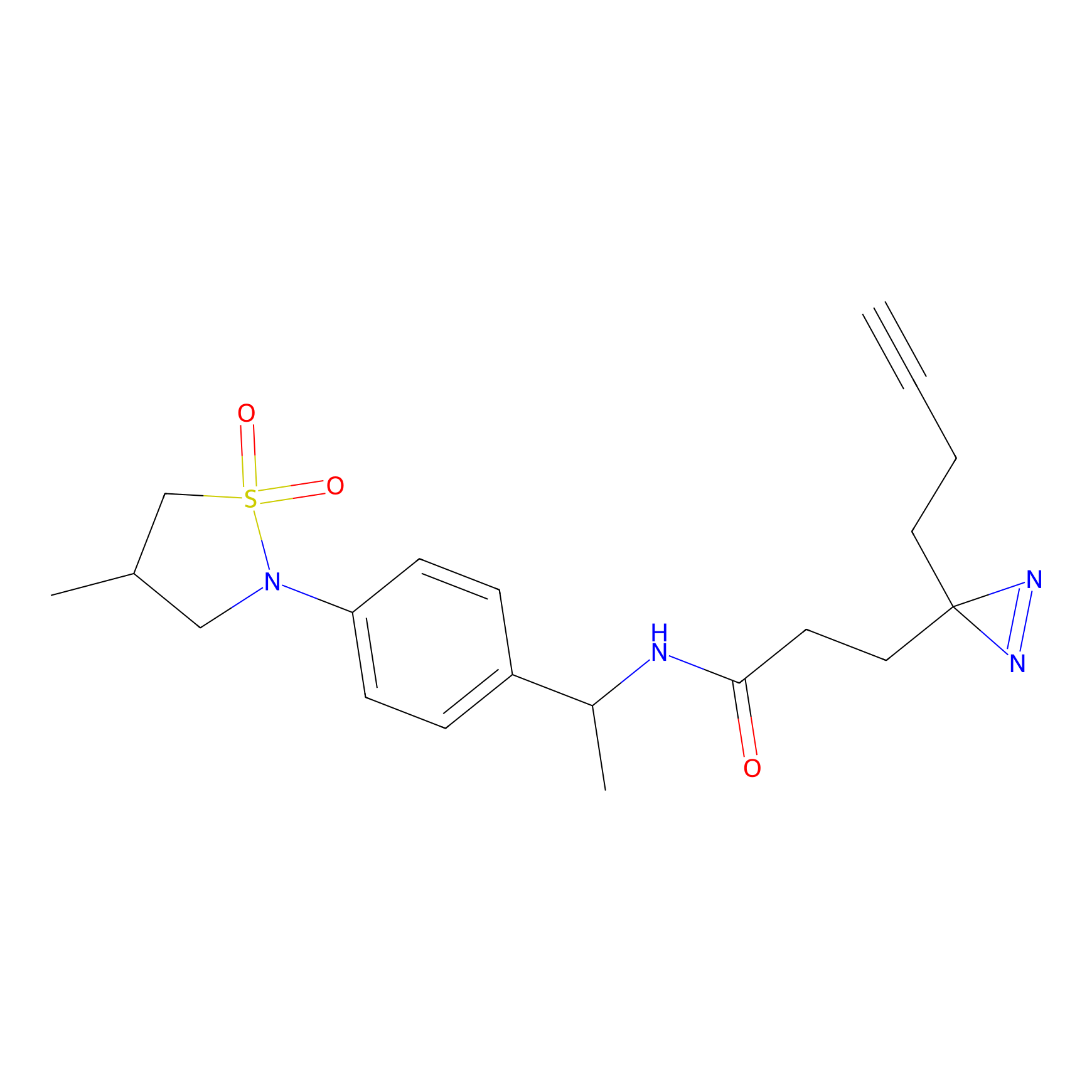
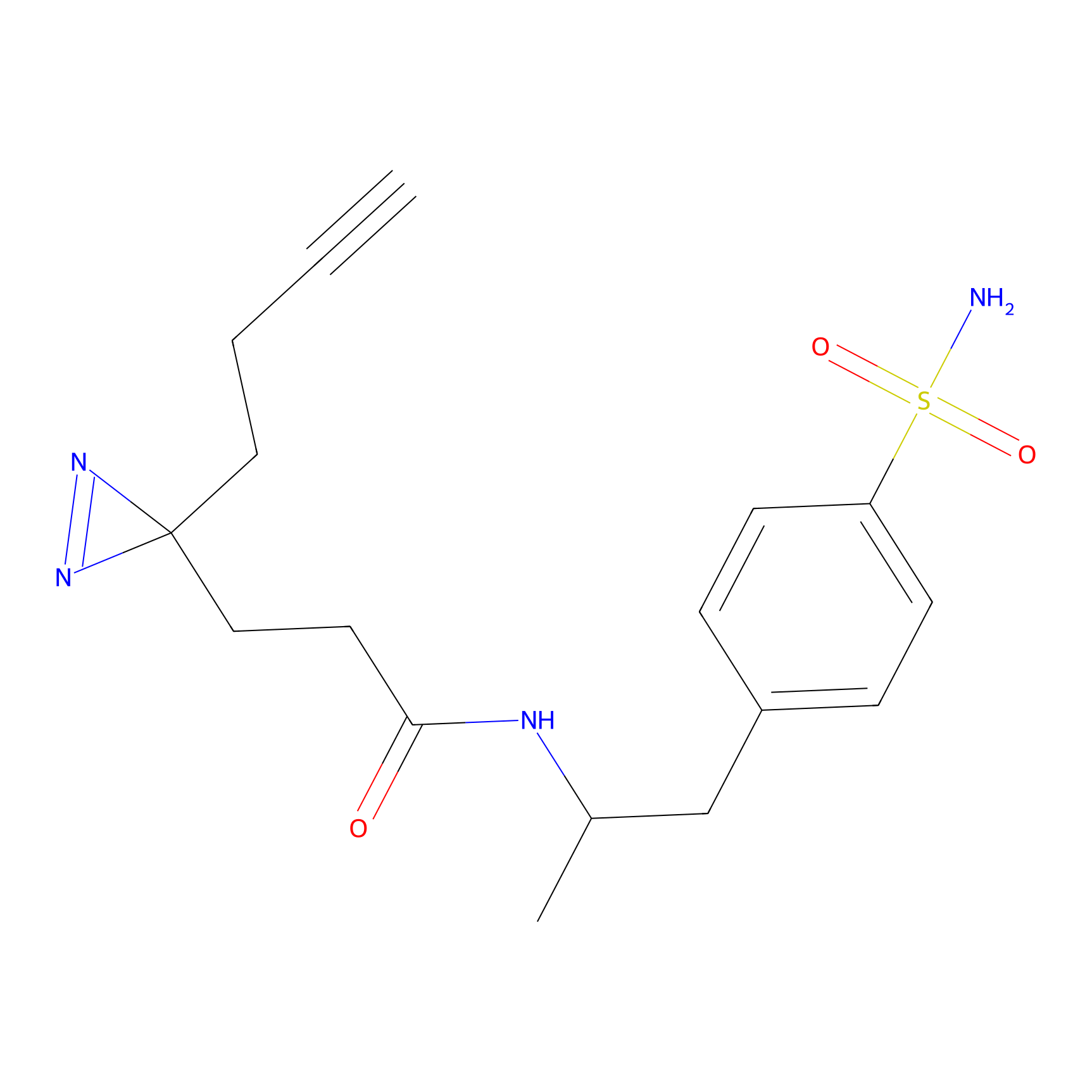
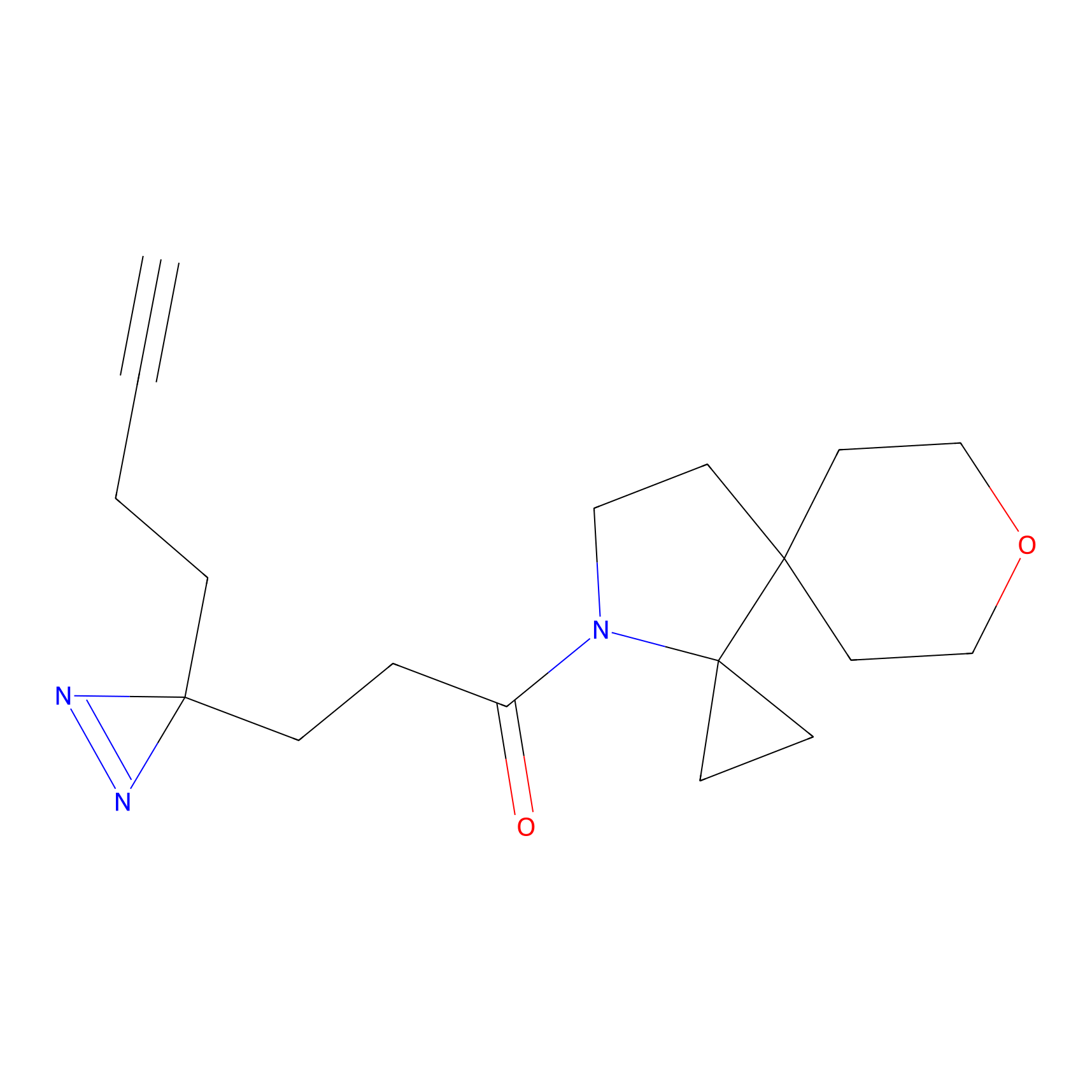
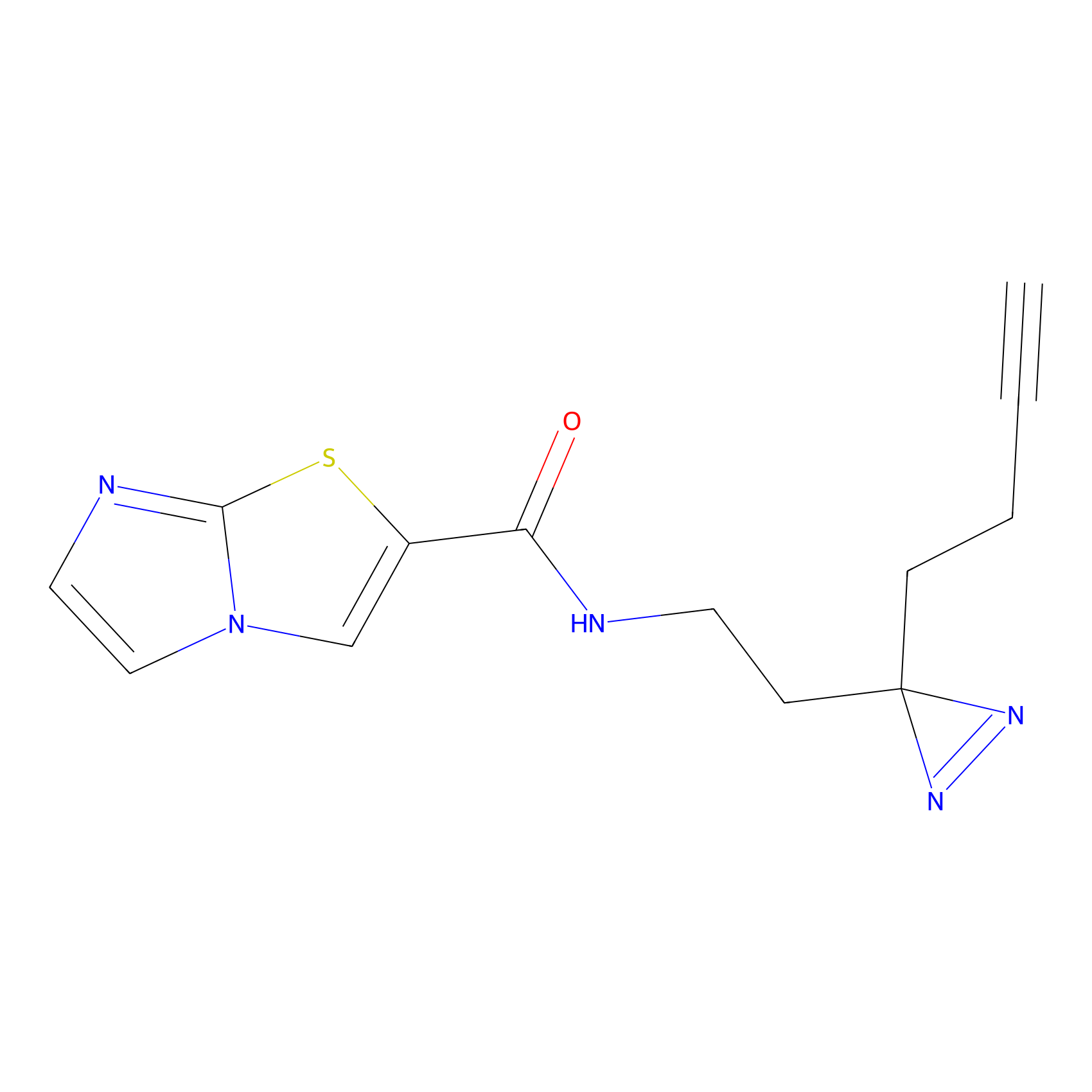
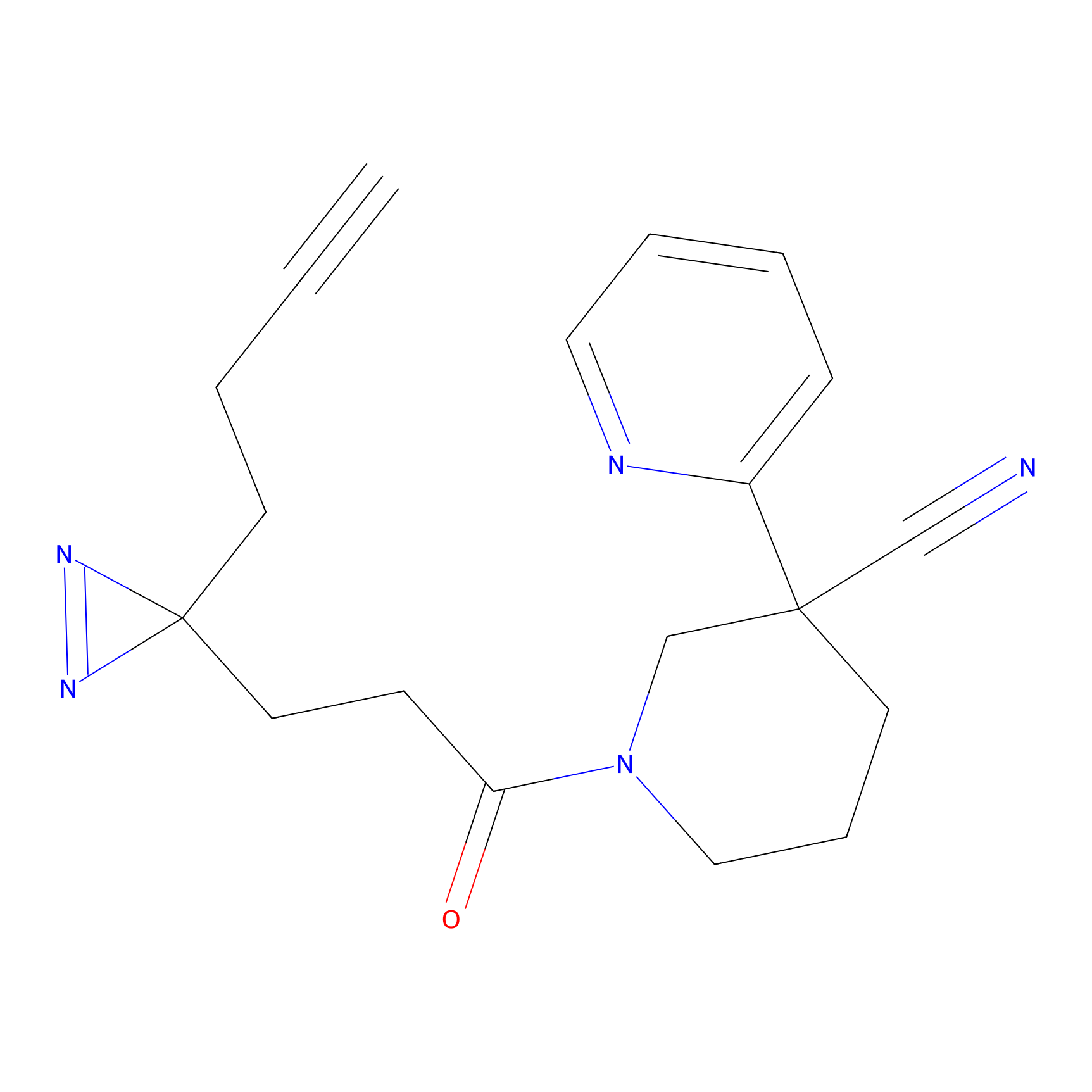
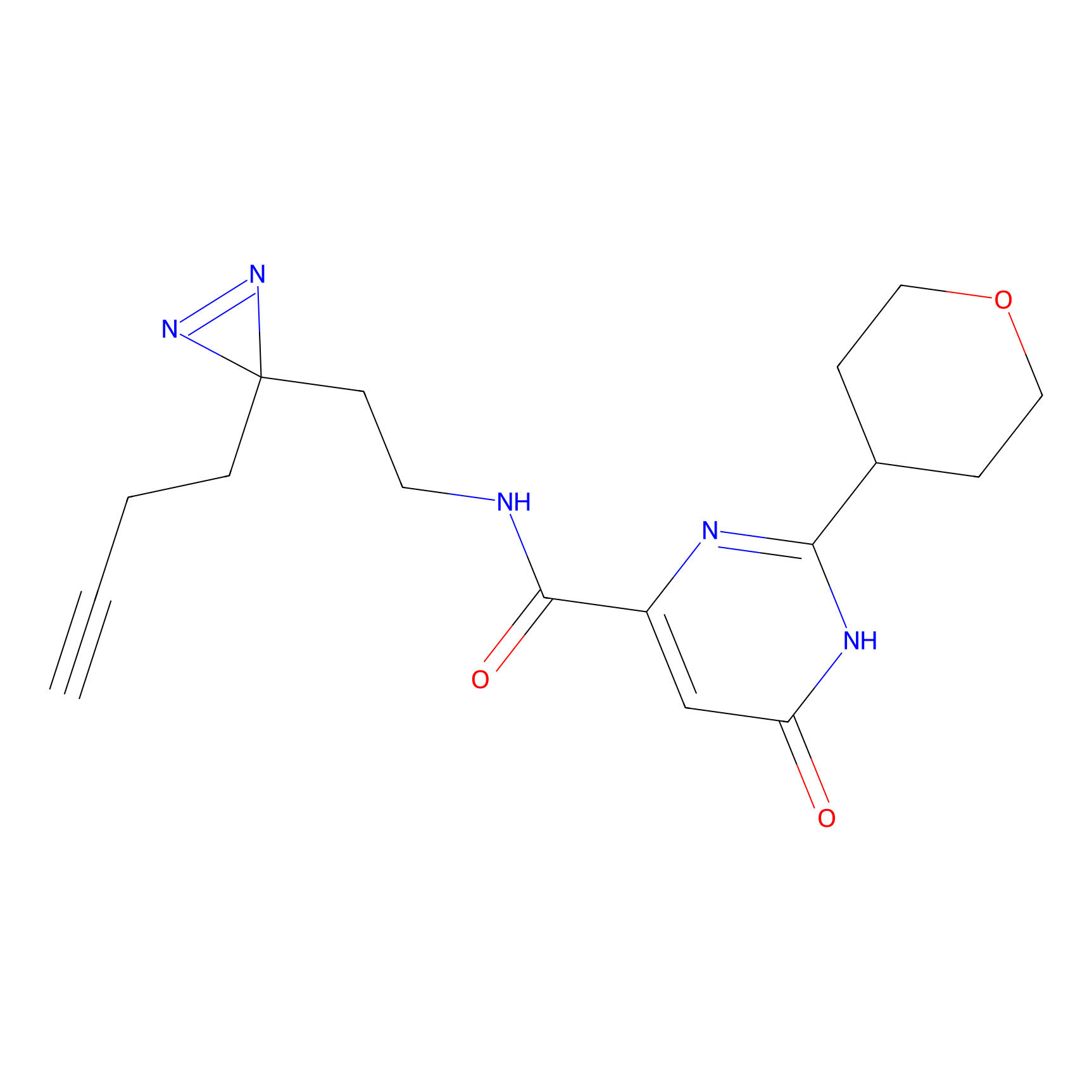
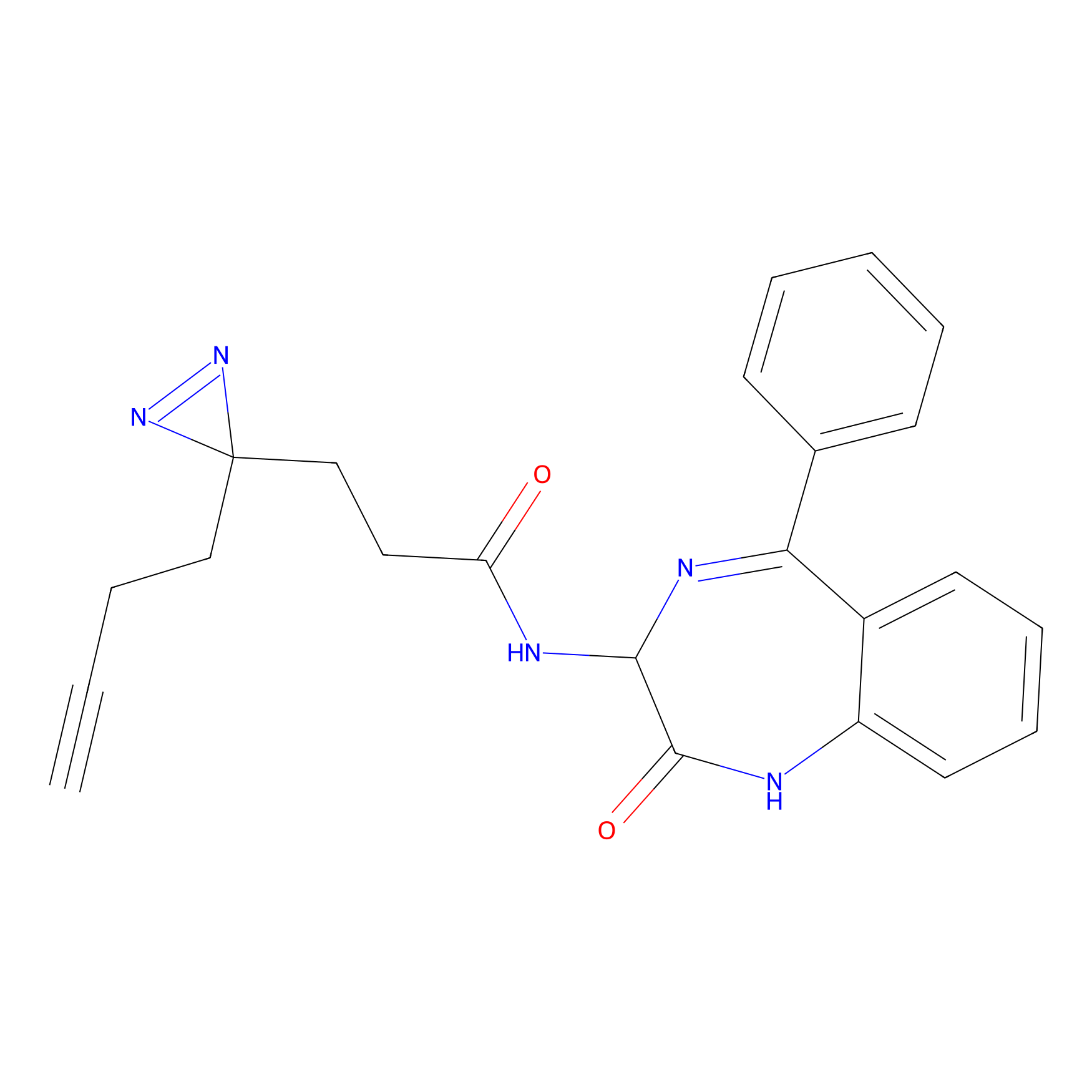
|
TG42 Probe Info |
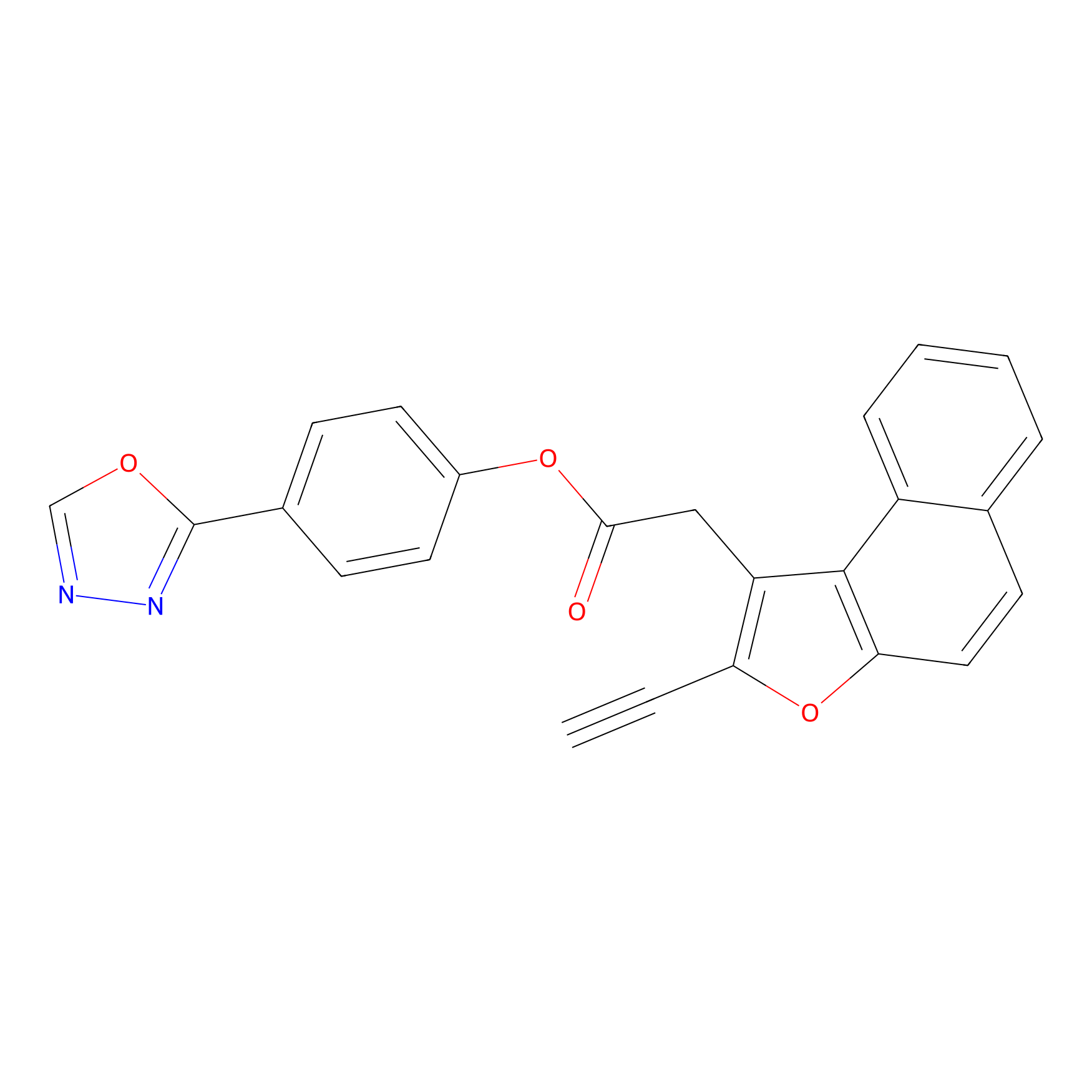 |
4.23 | LDD0326 | [4] | |
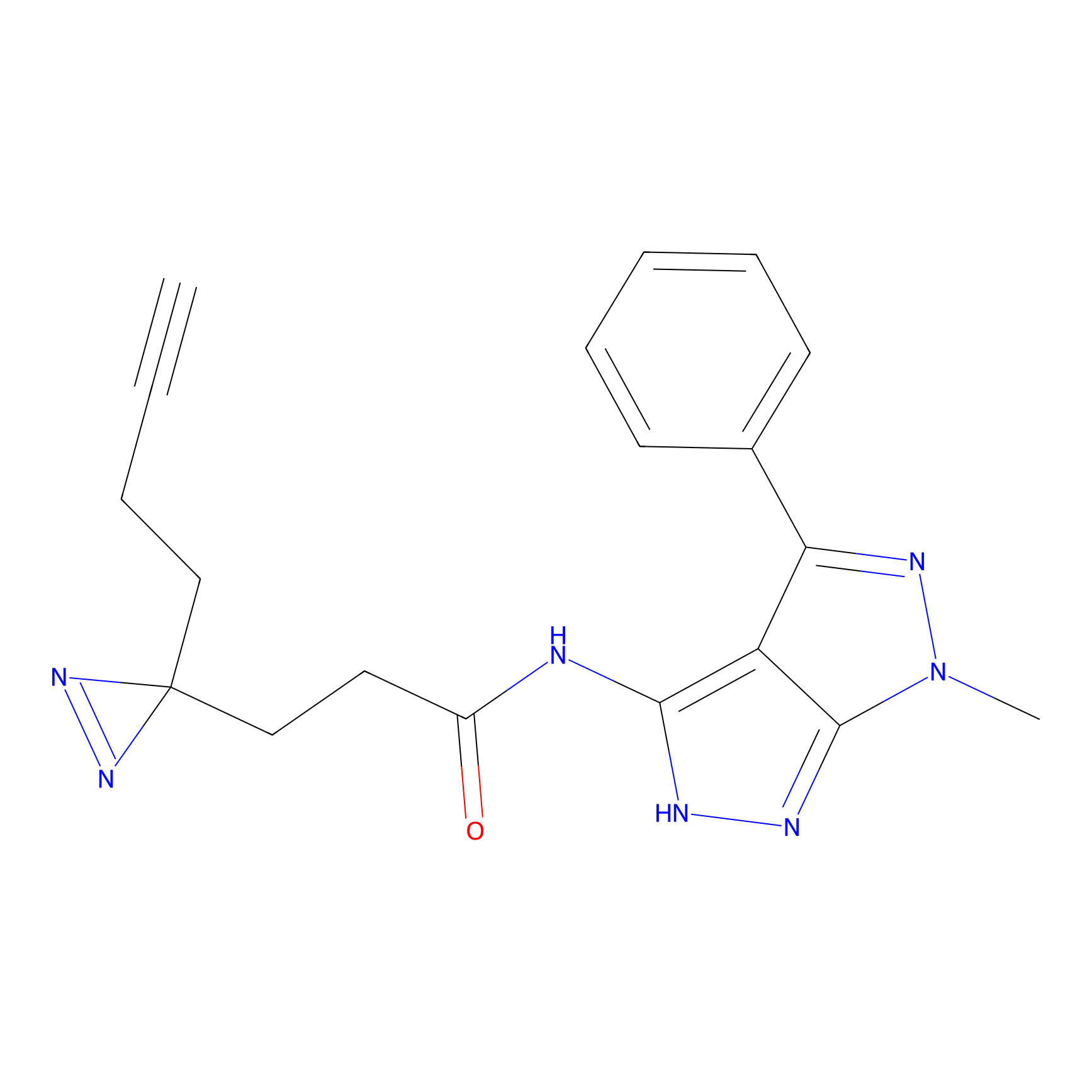
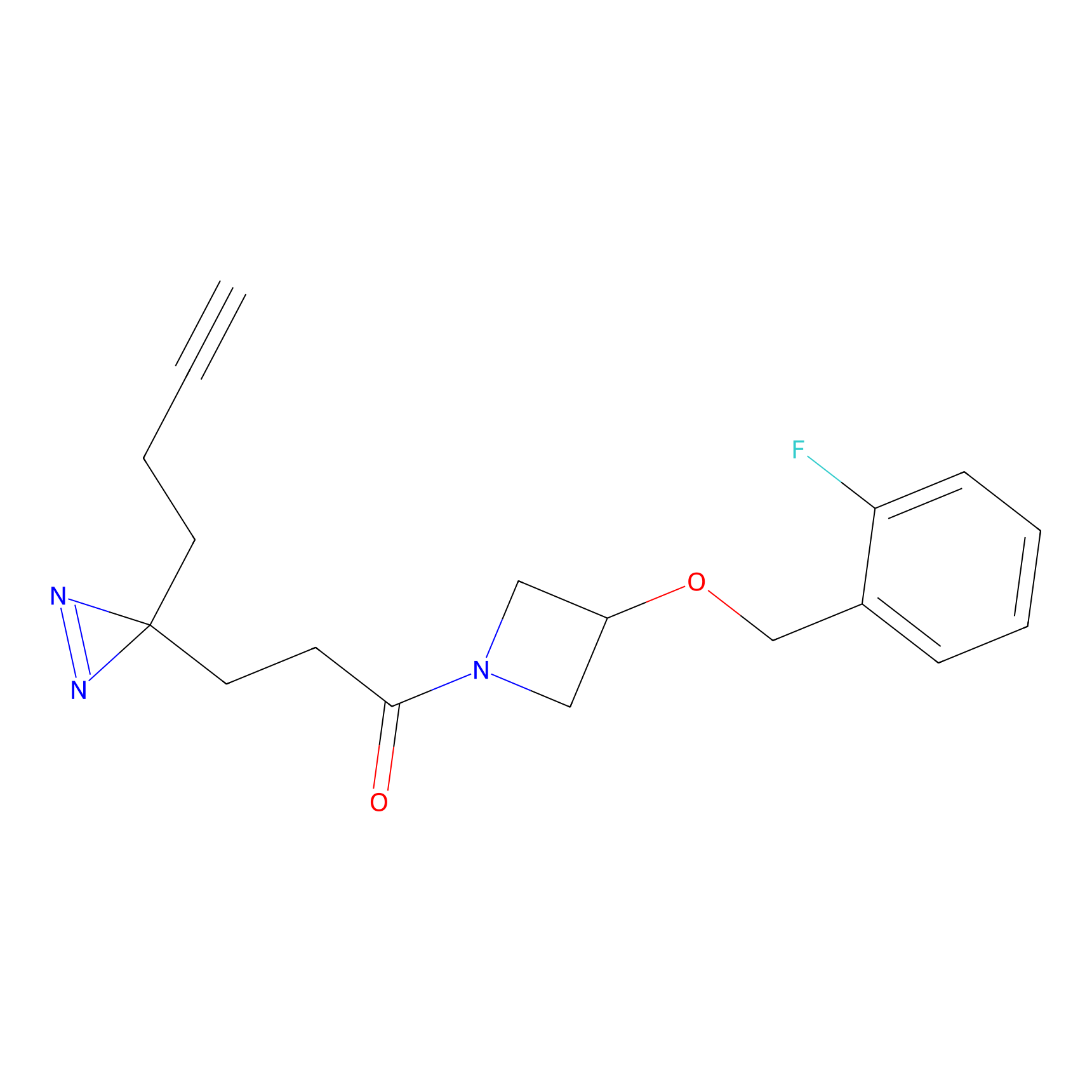
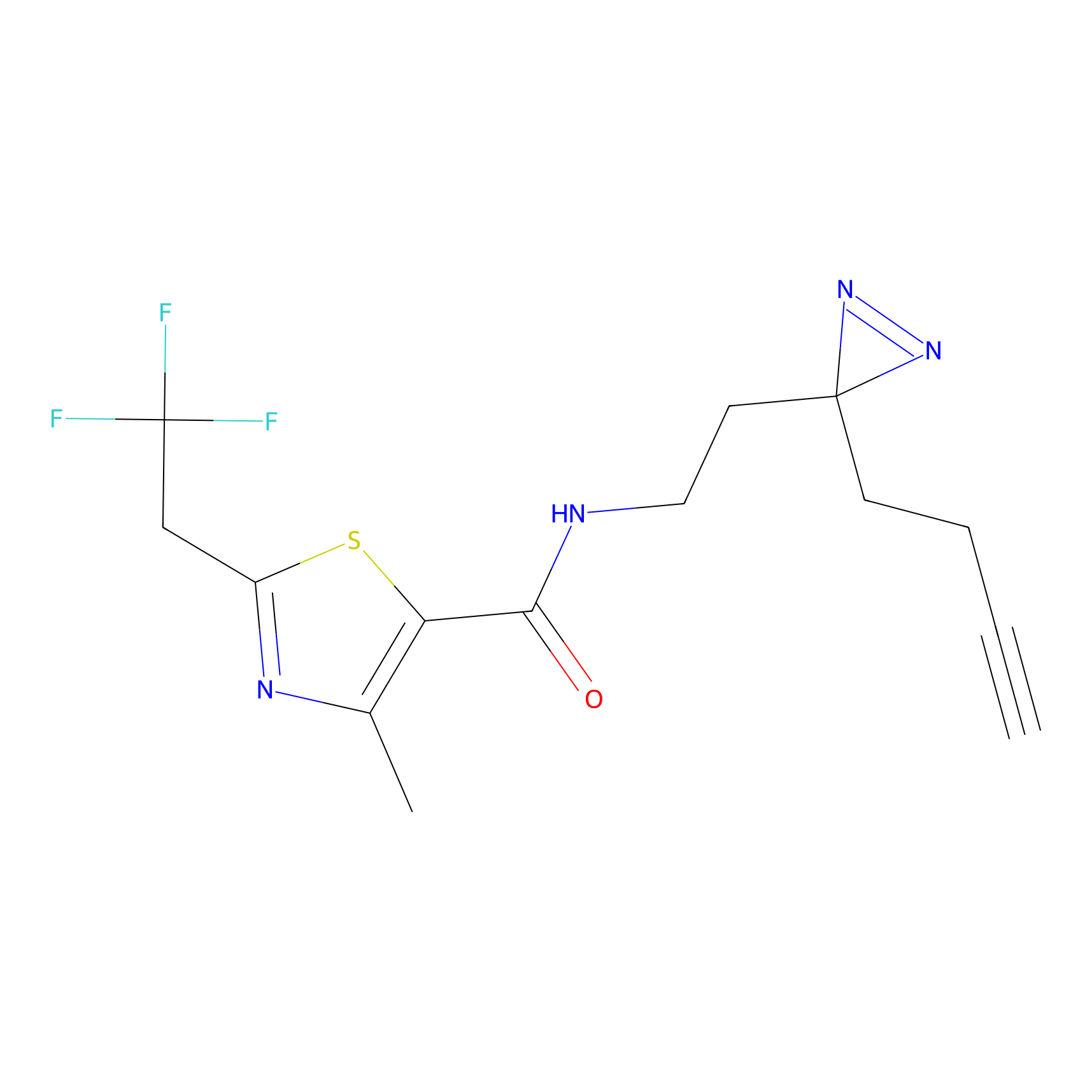
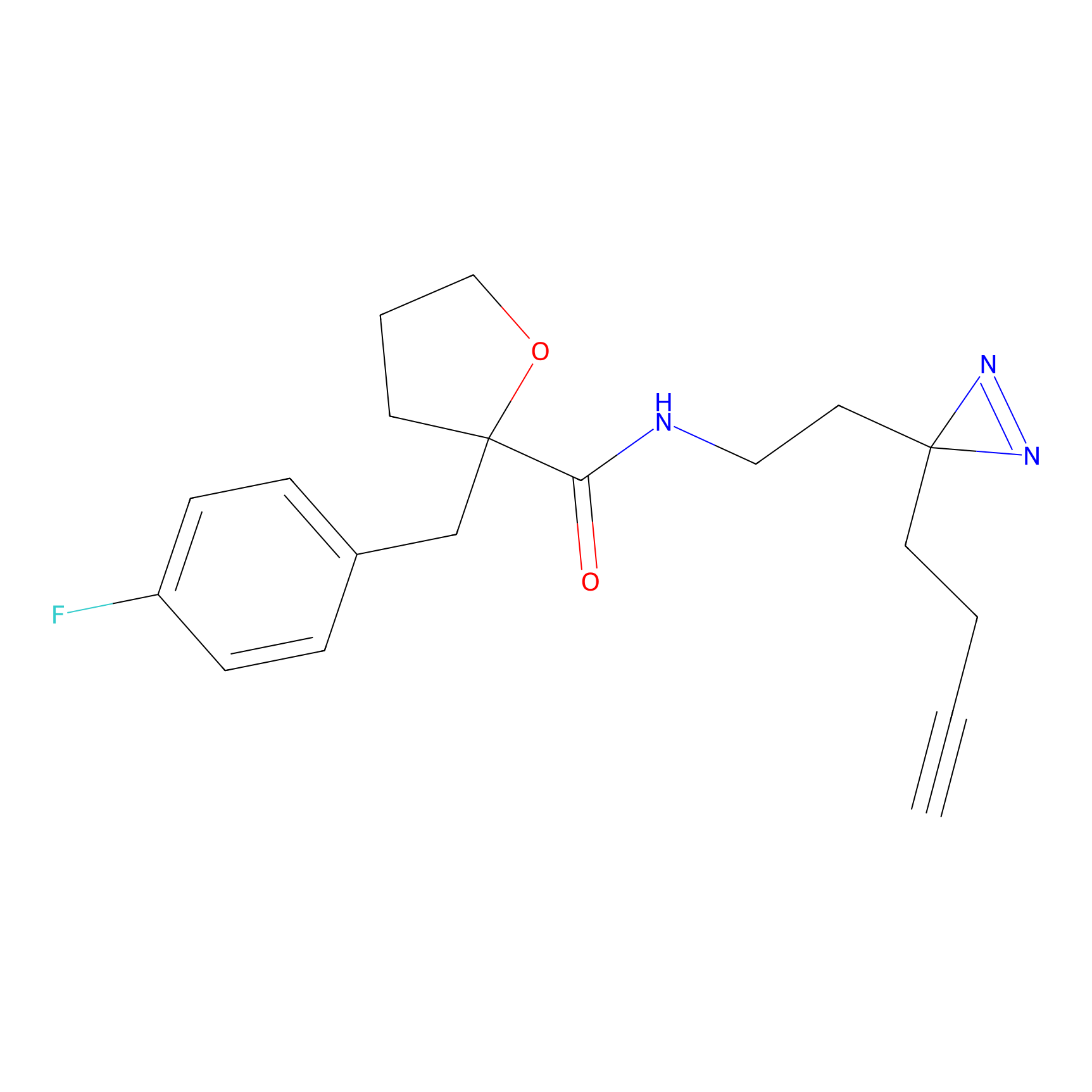
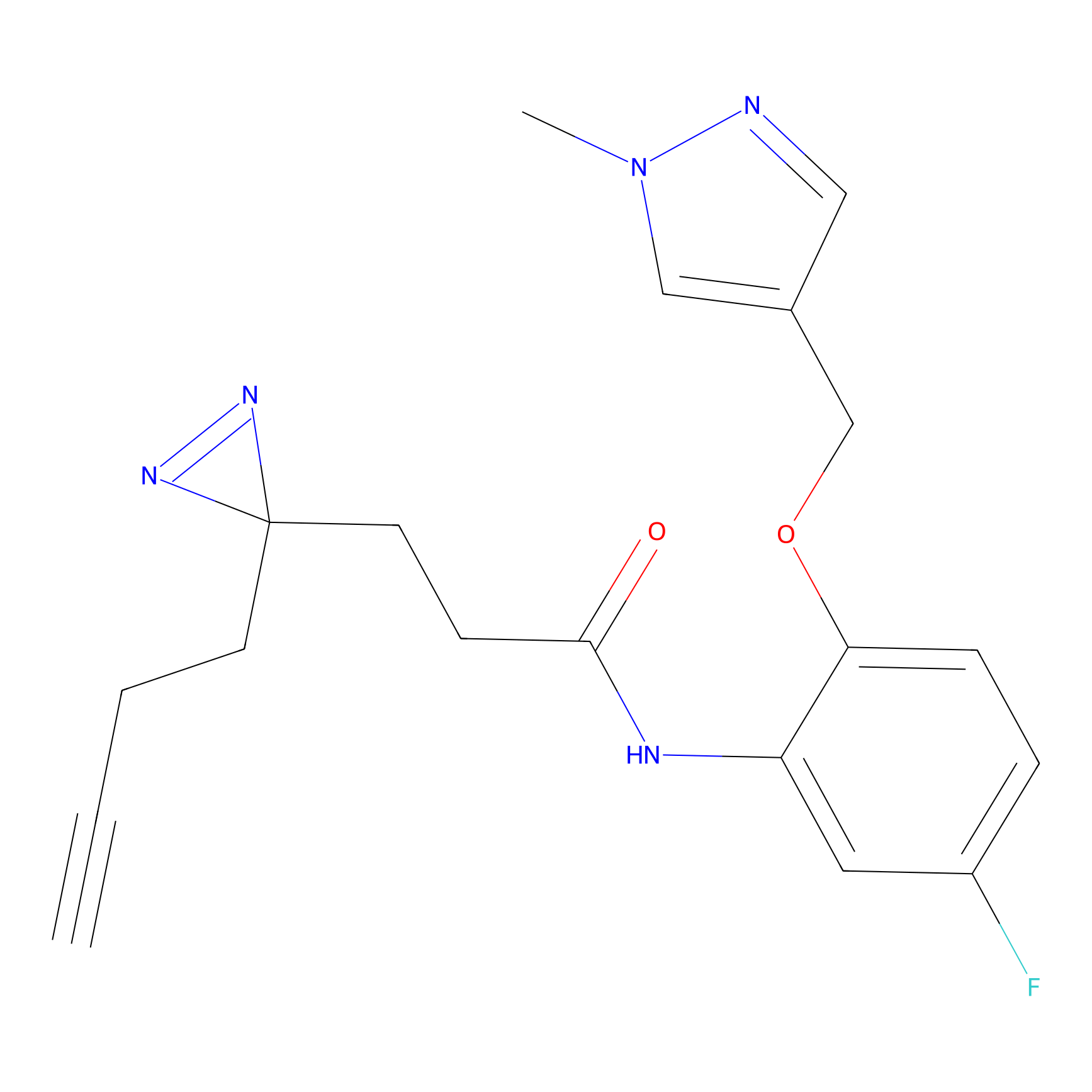
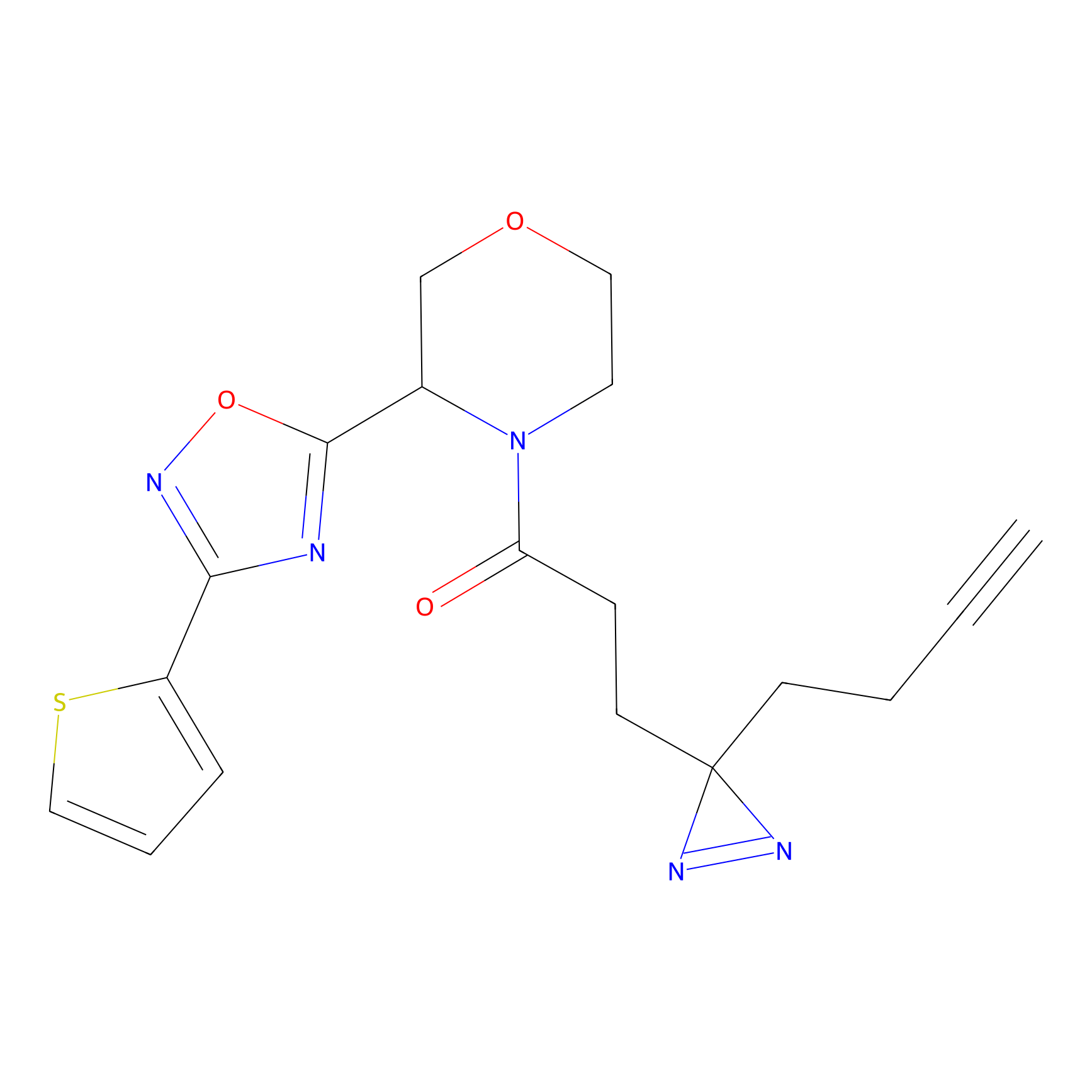
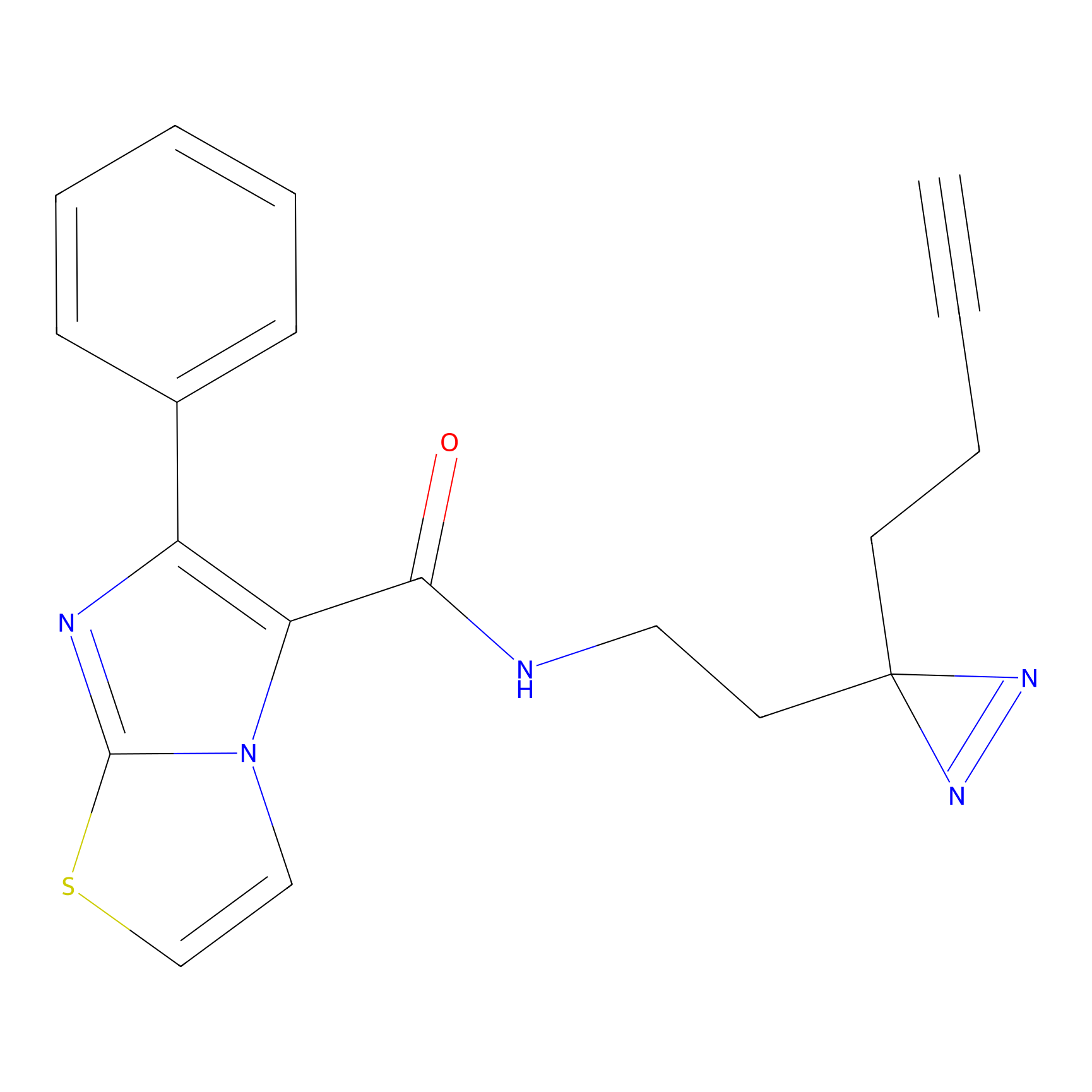
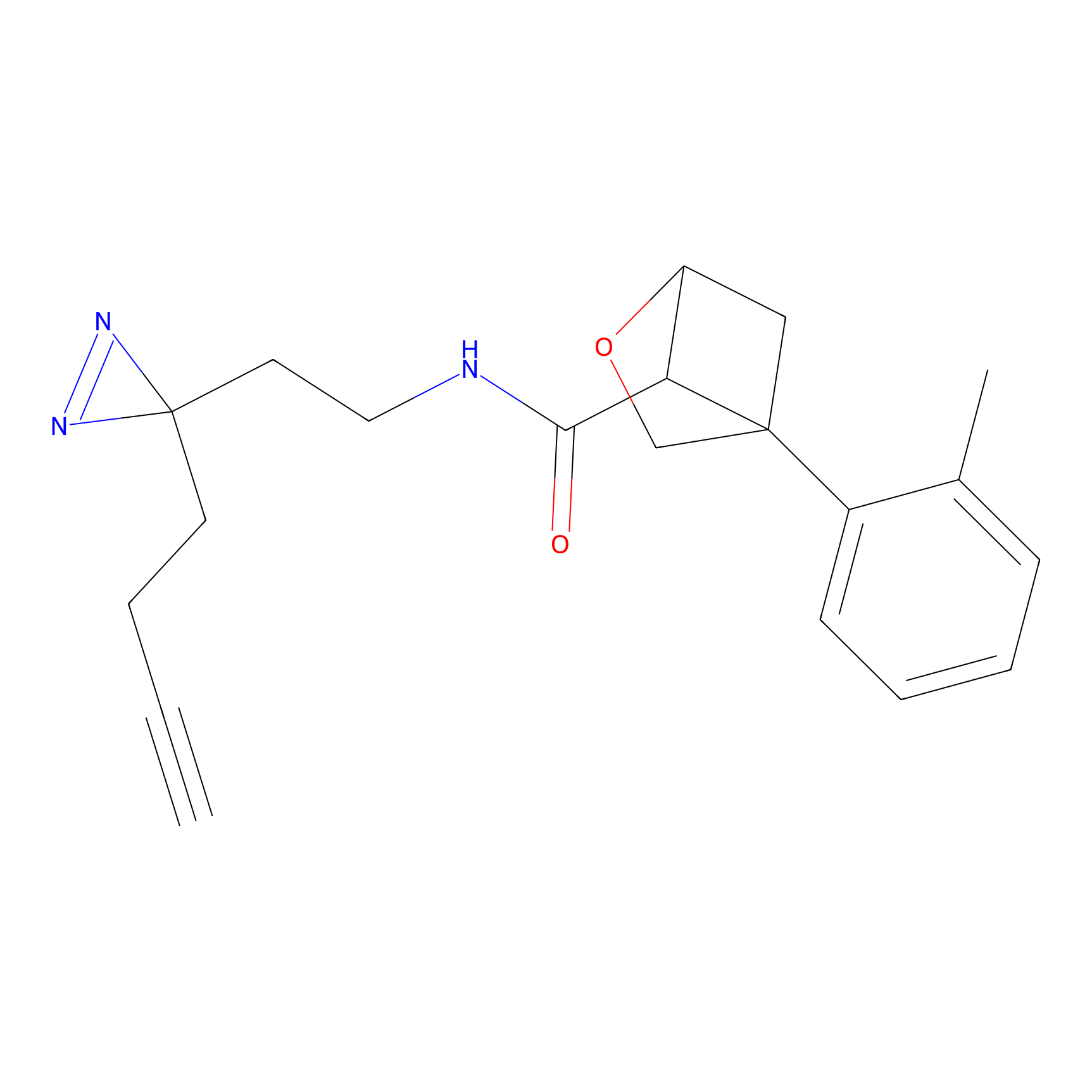
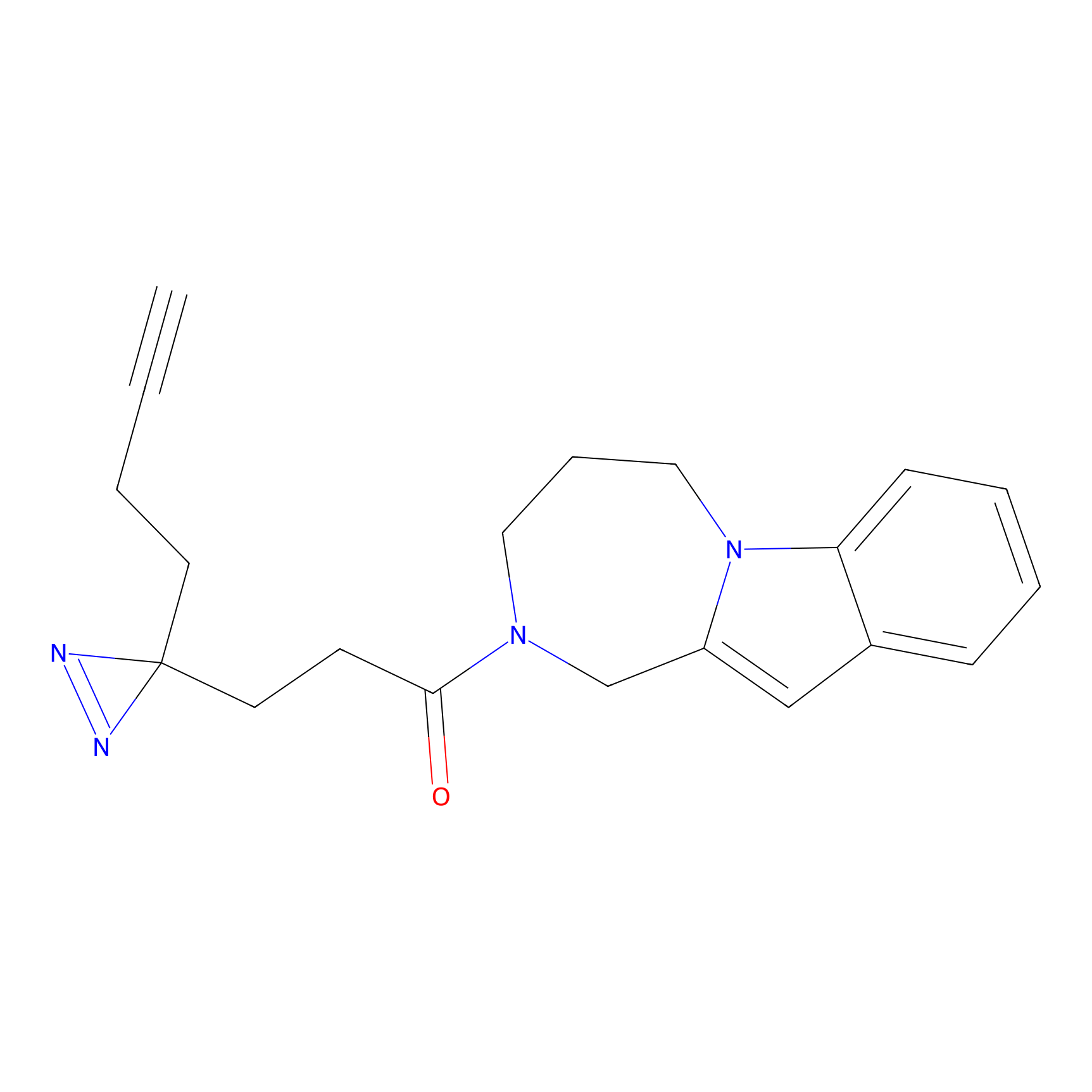
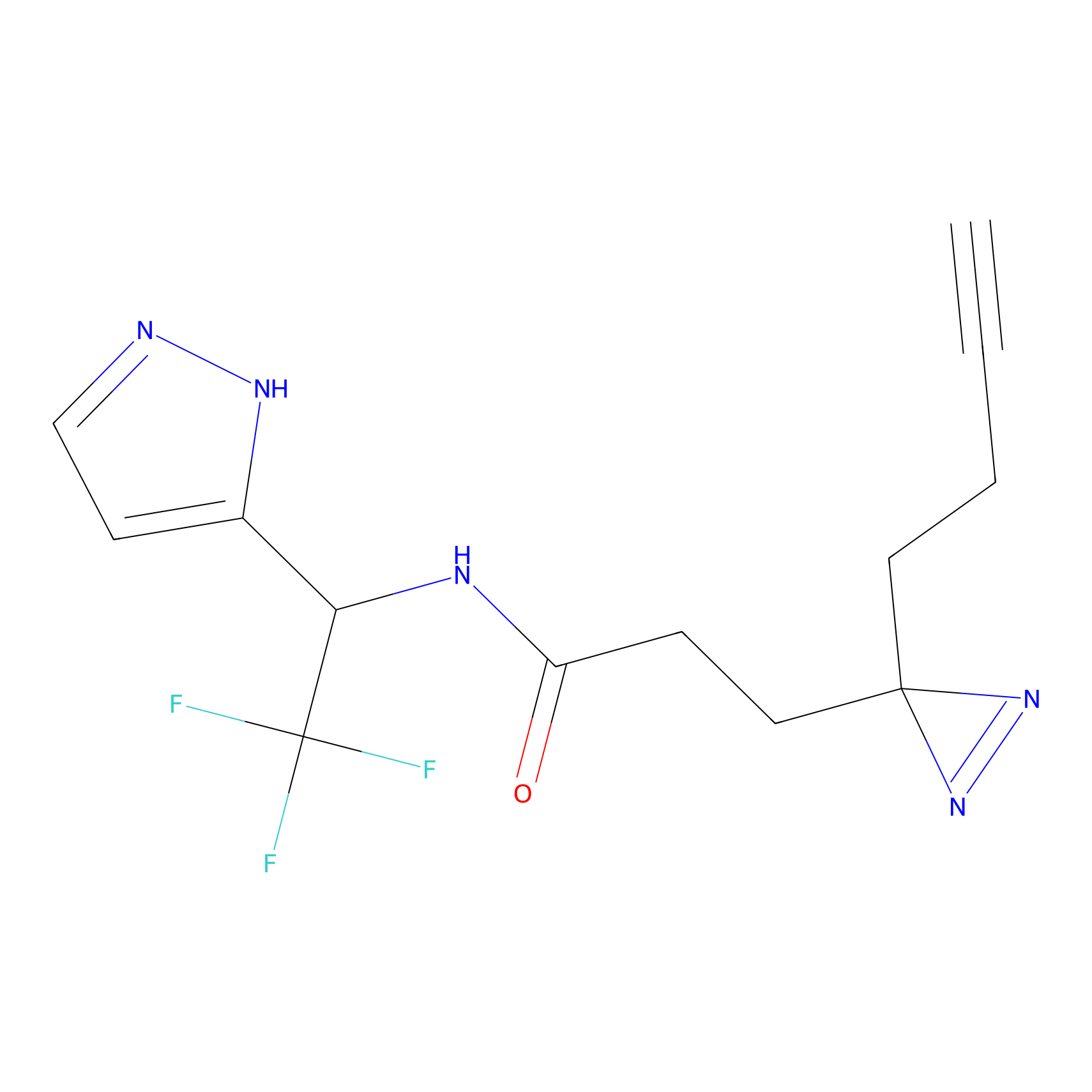
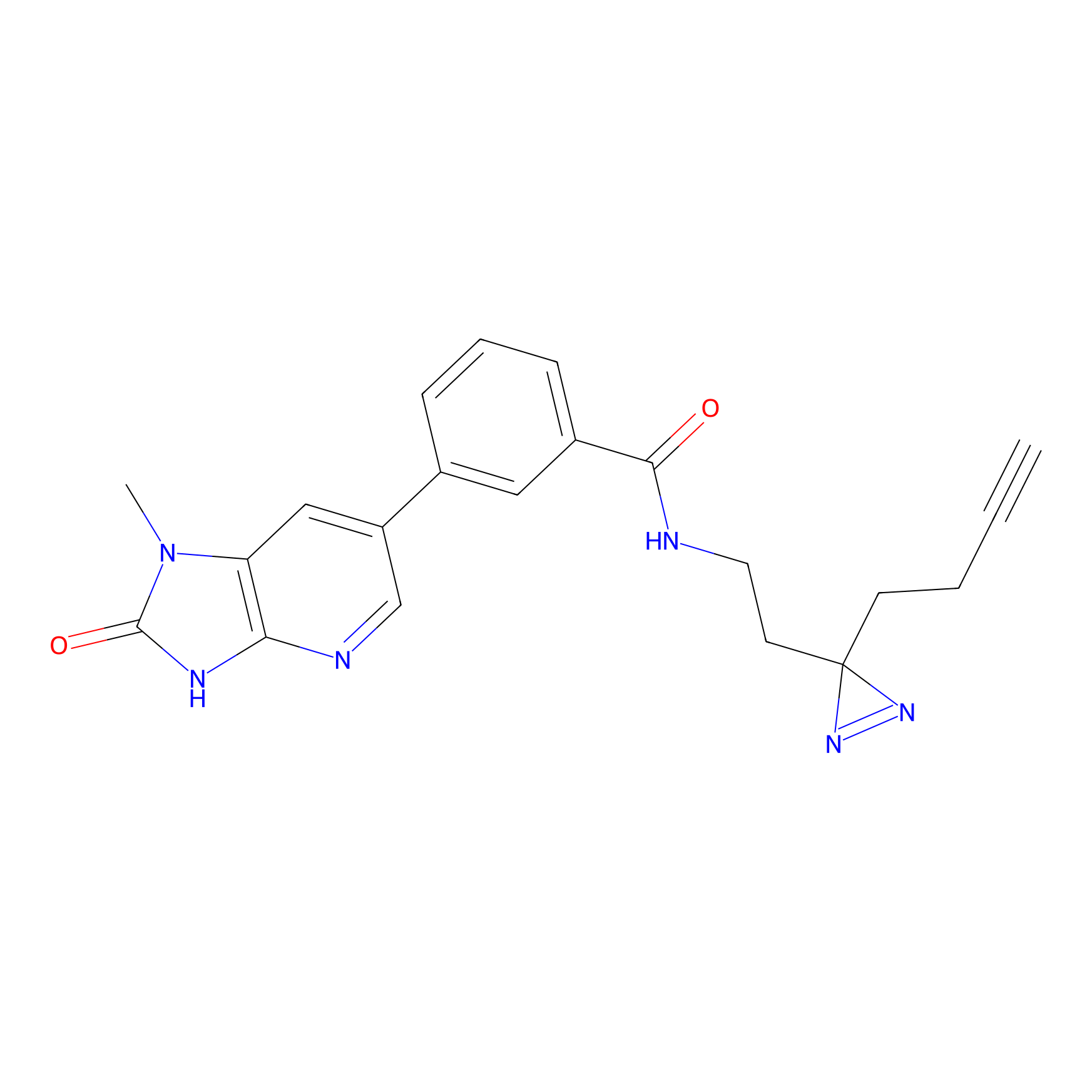
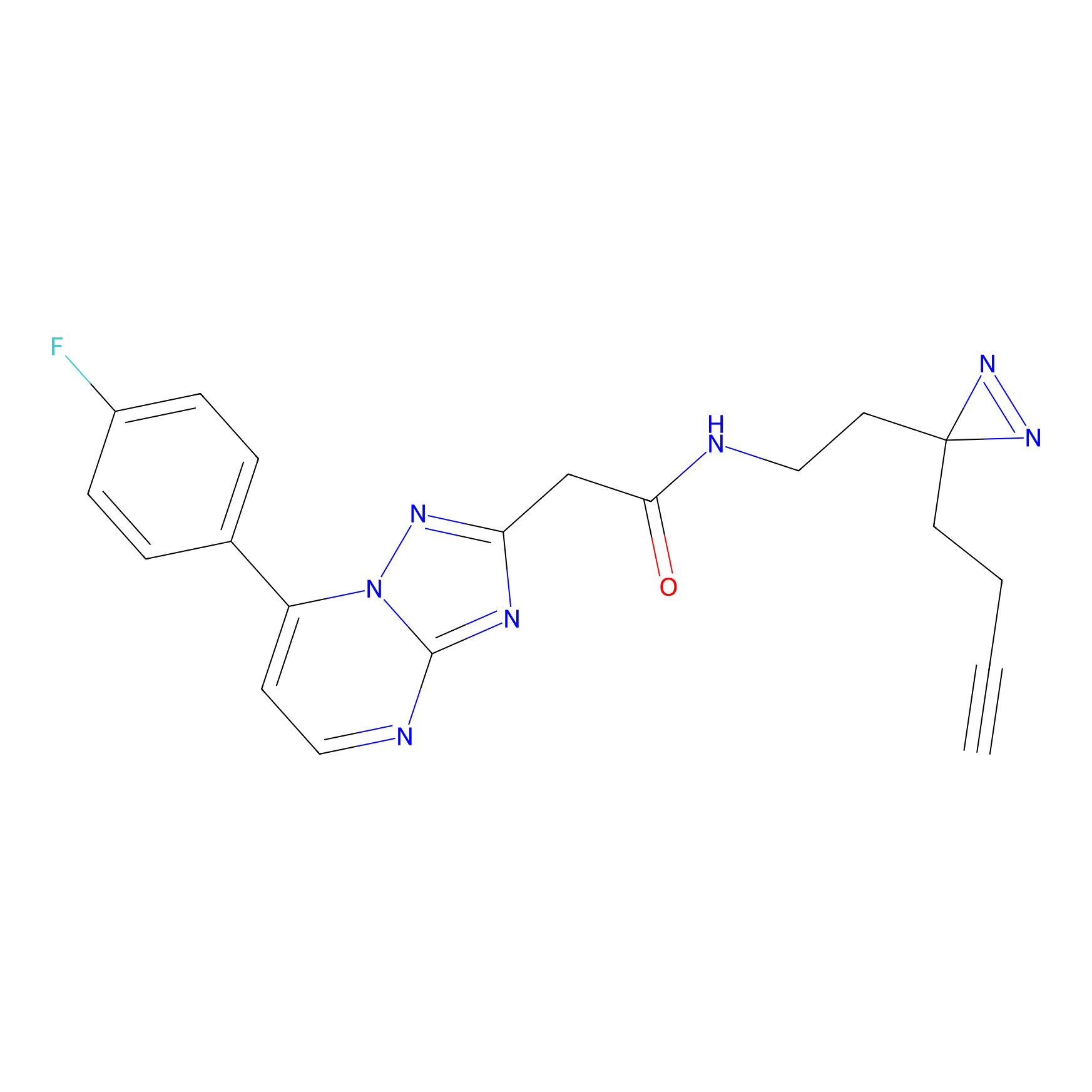
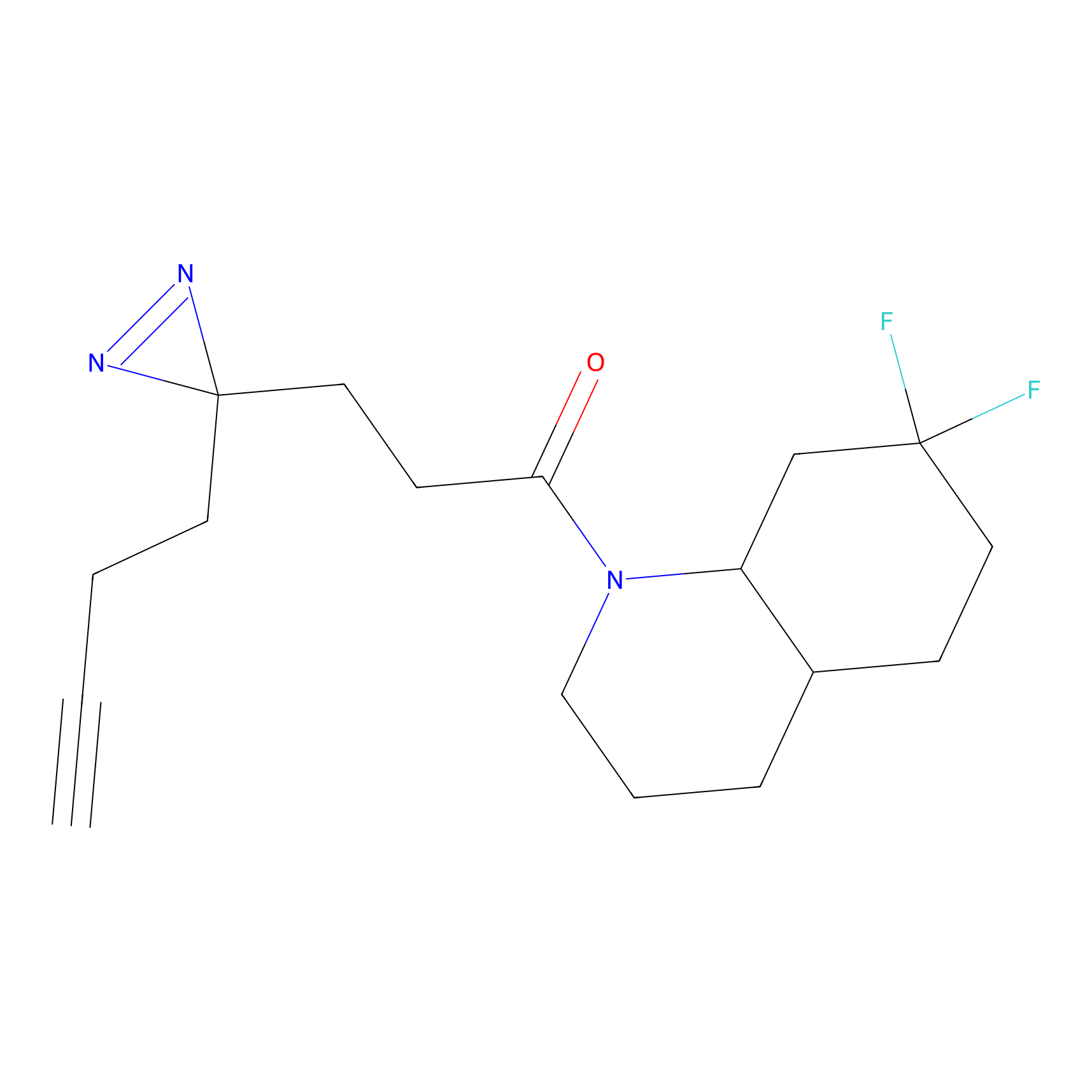
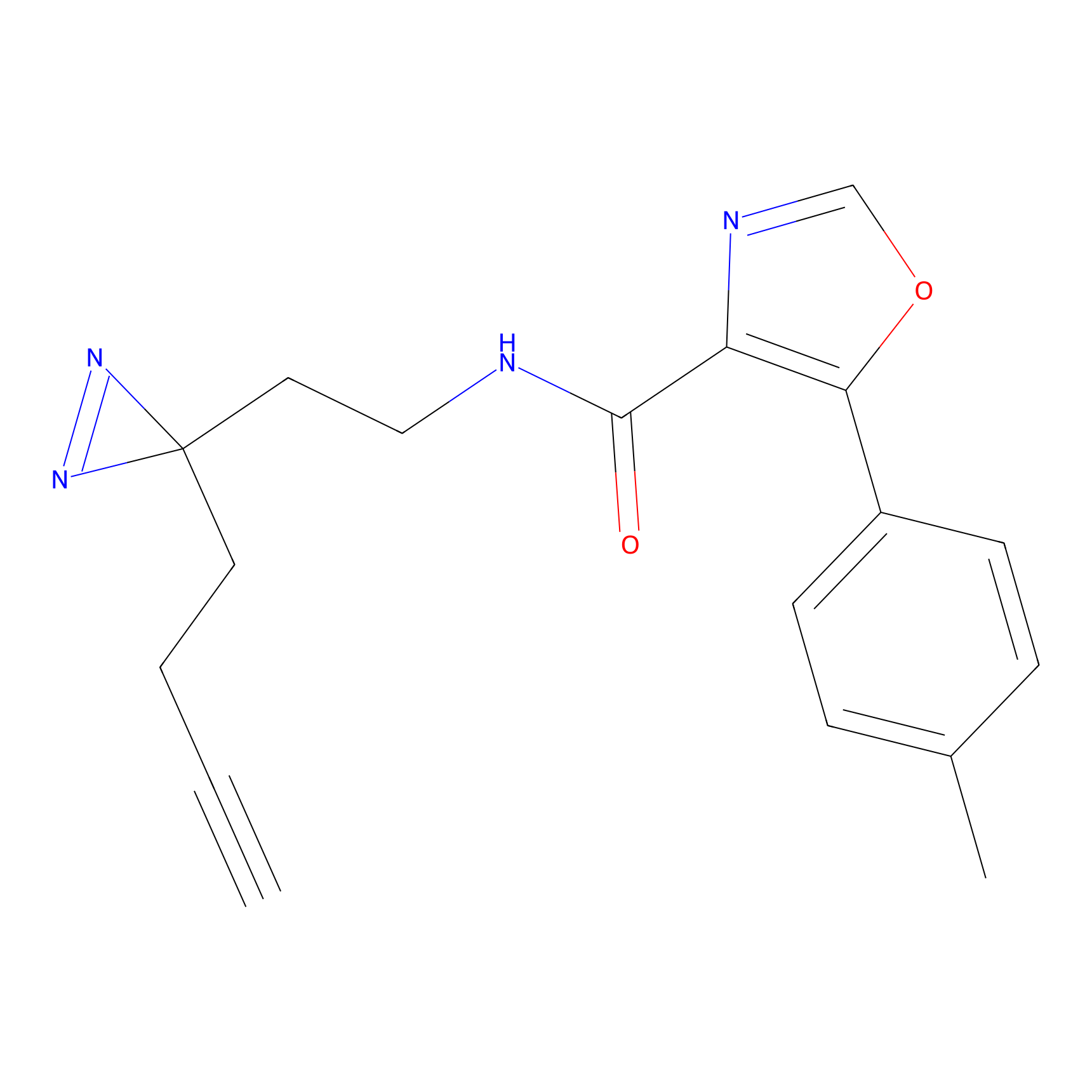
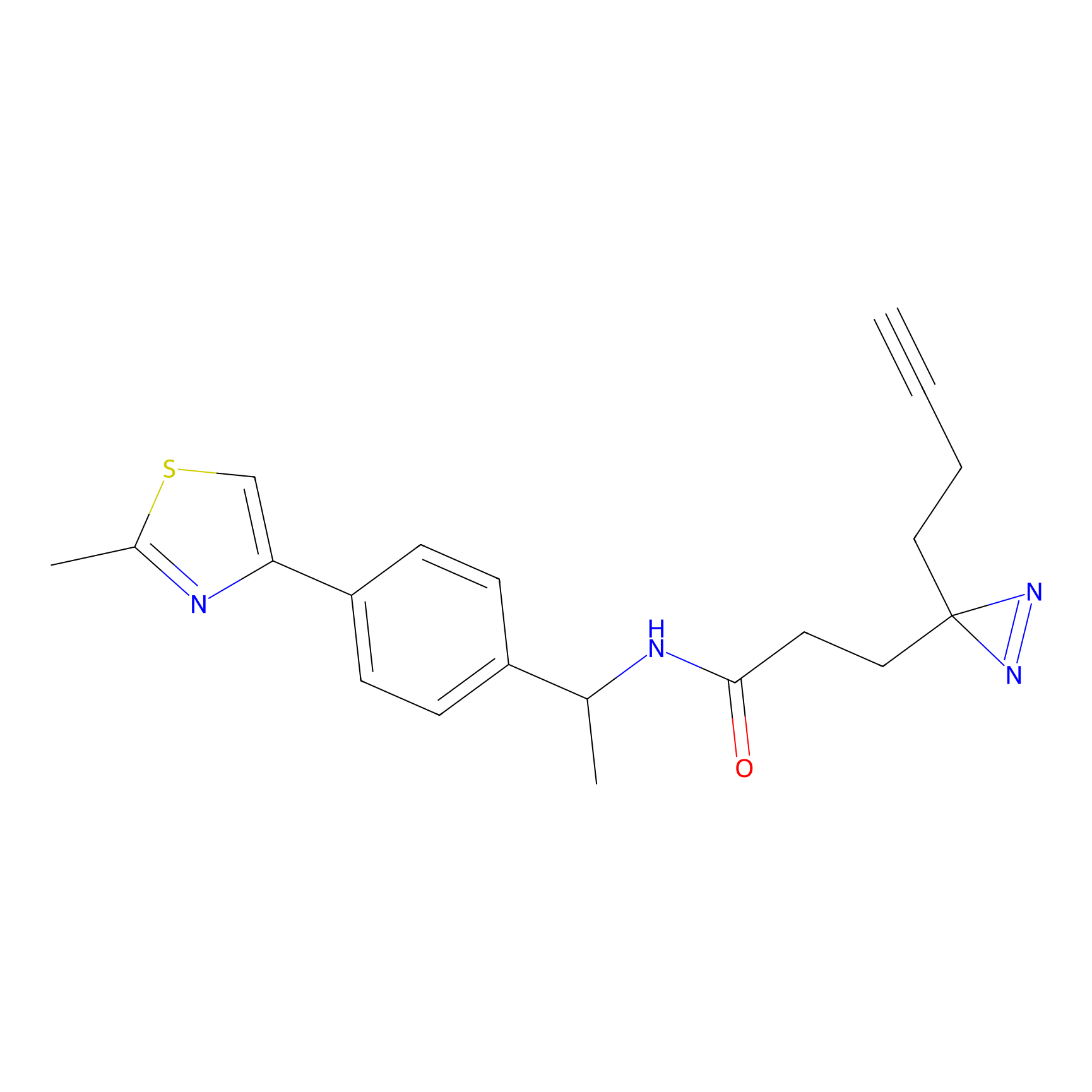
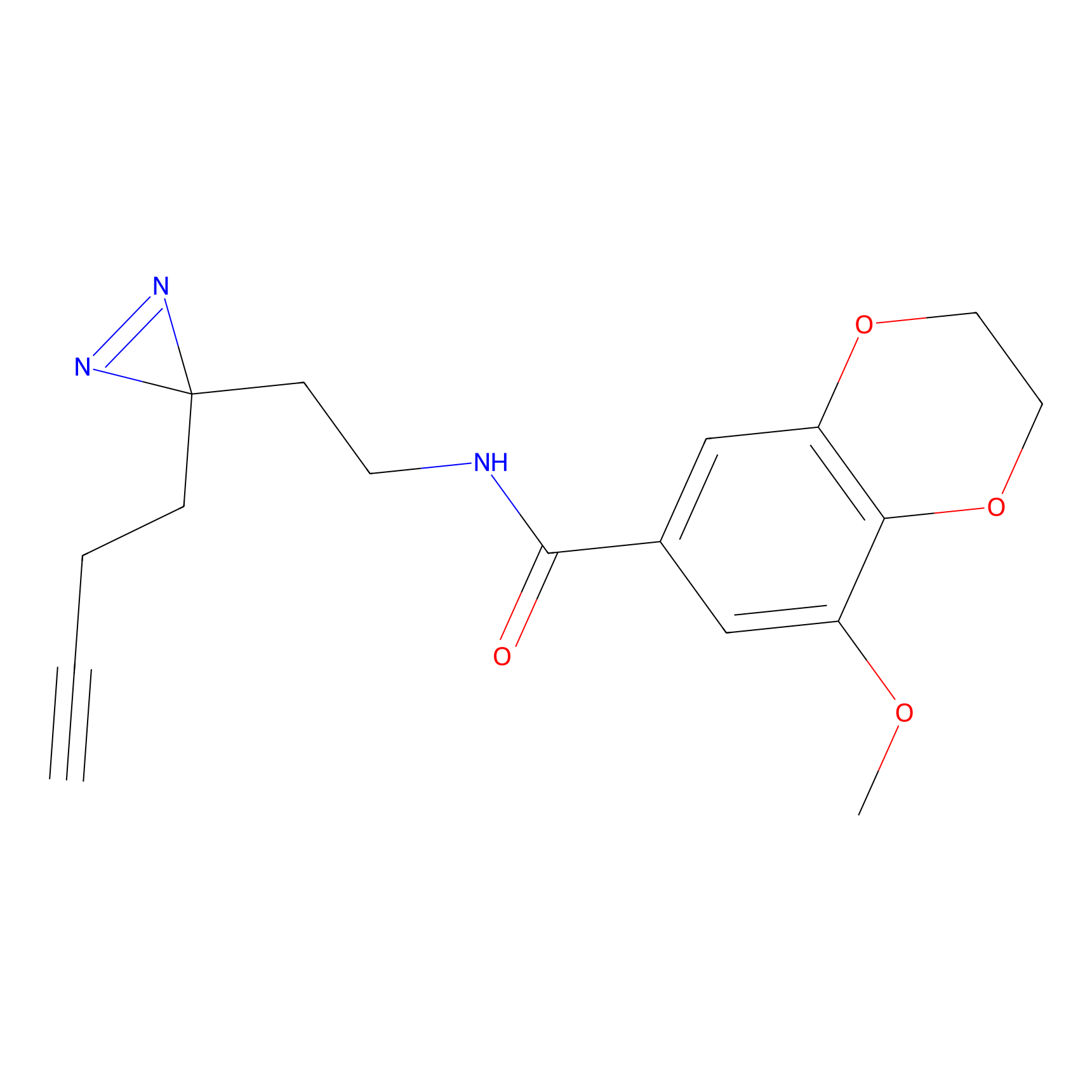
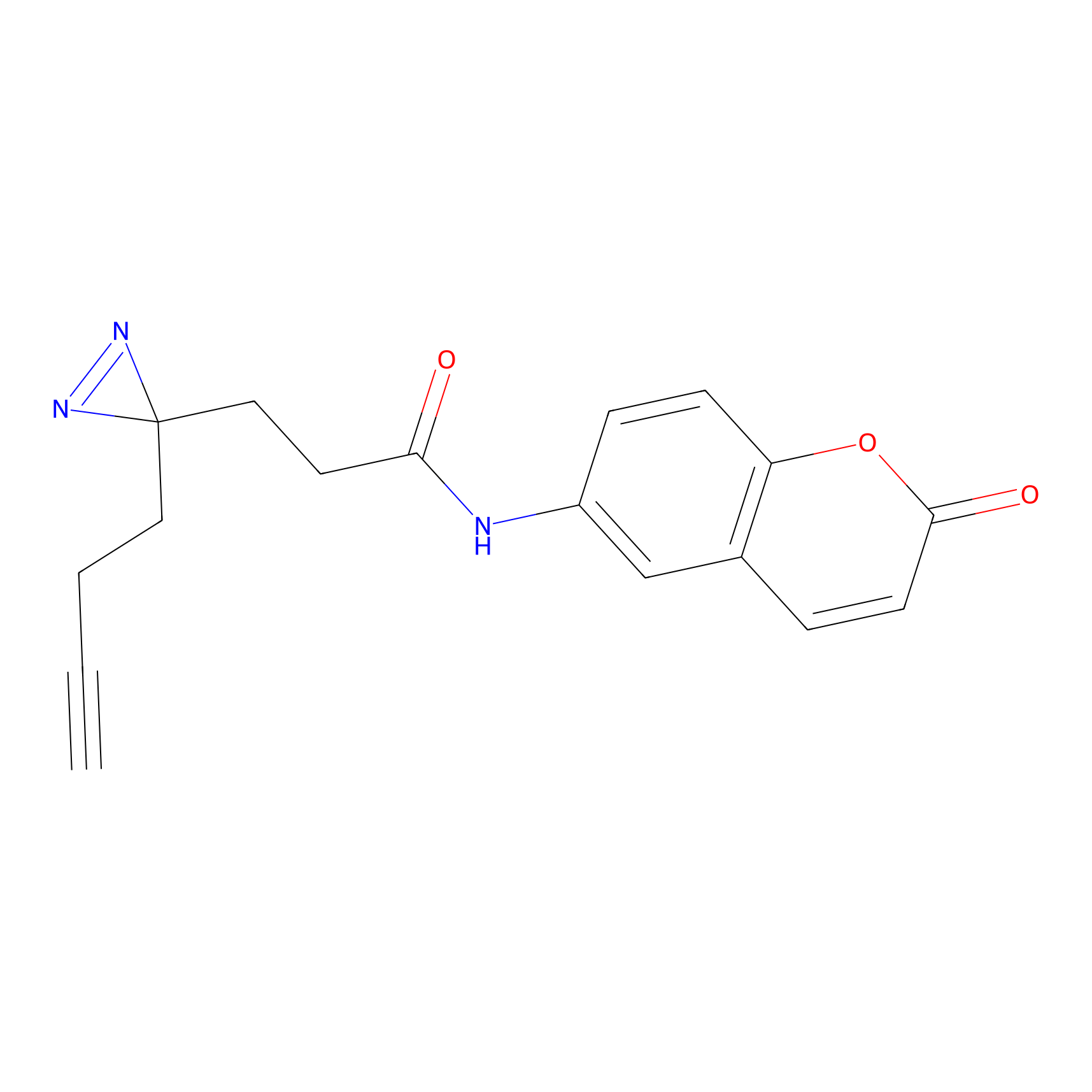
|
C-Sul Probe Info |
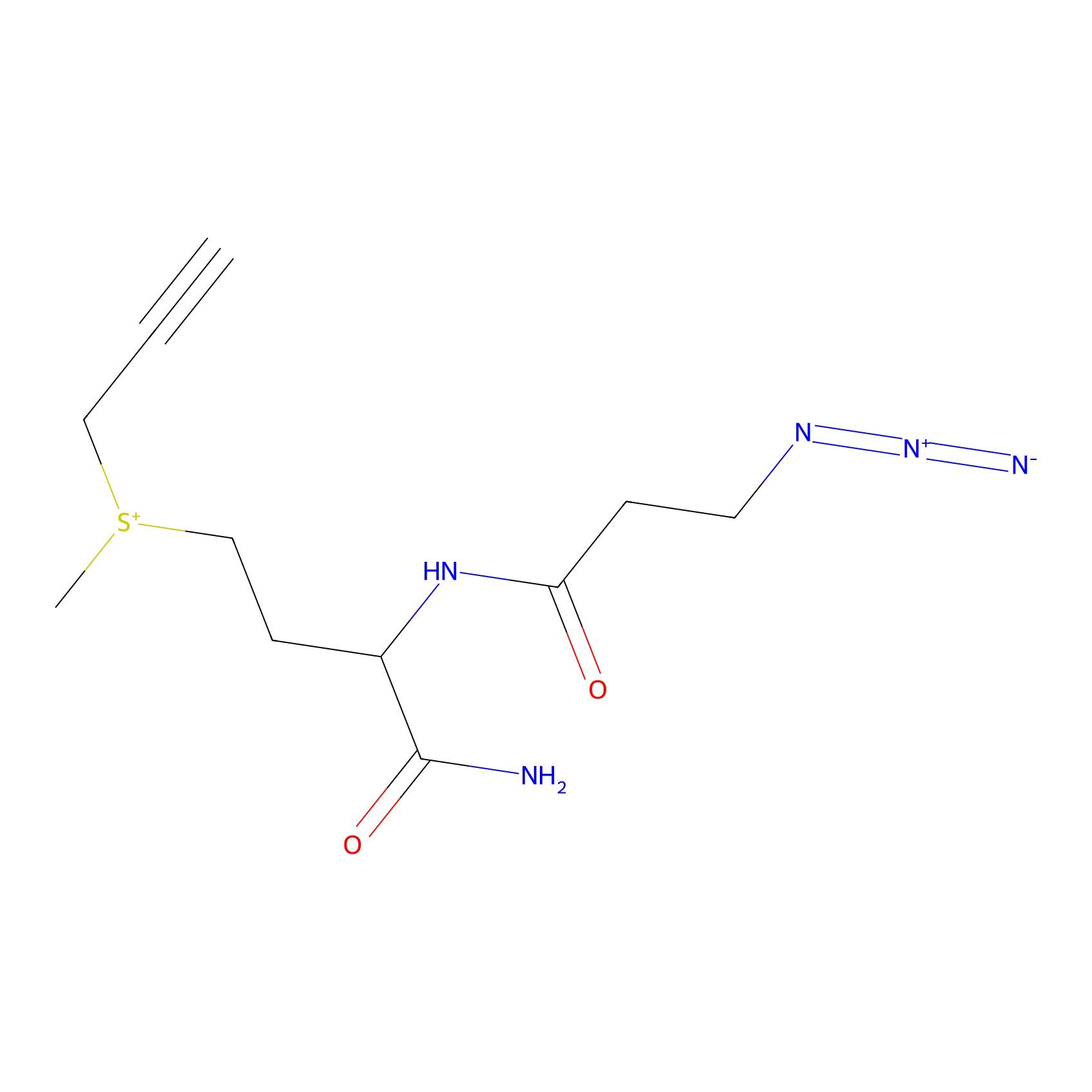 |
3.75 | LDD0066 | [5] | |
|
YN-1 Probe Info |
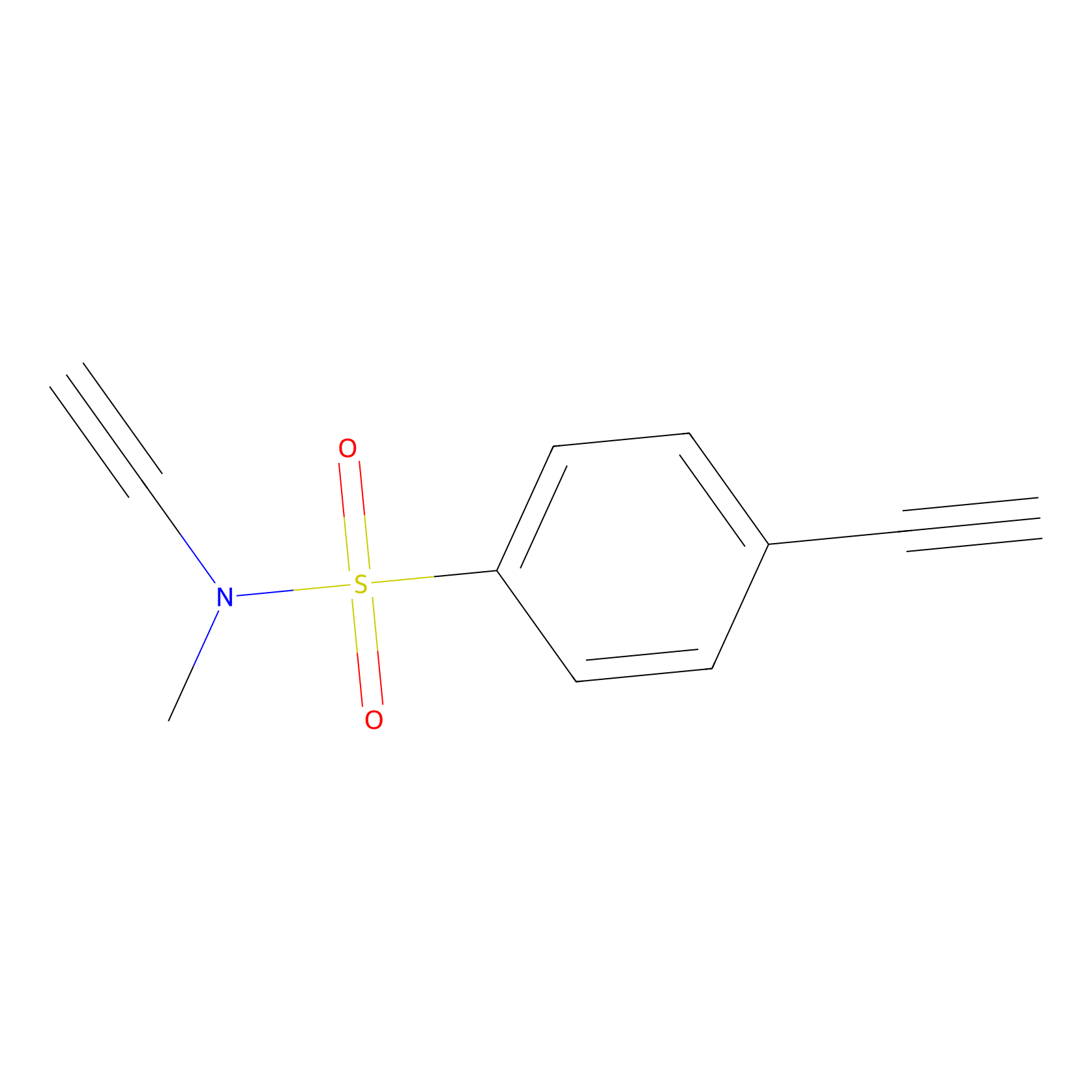 |
100.00 | LDD0444 | [6] | |
|
YN-4 Probe Info |
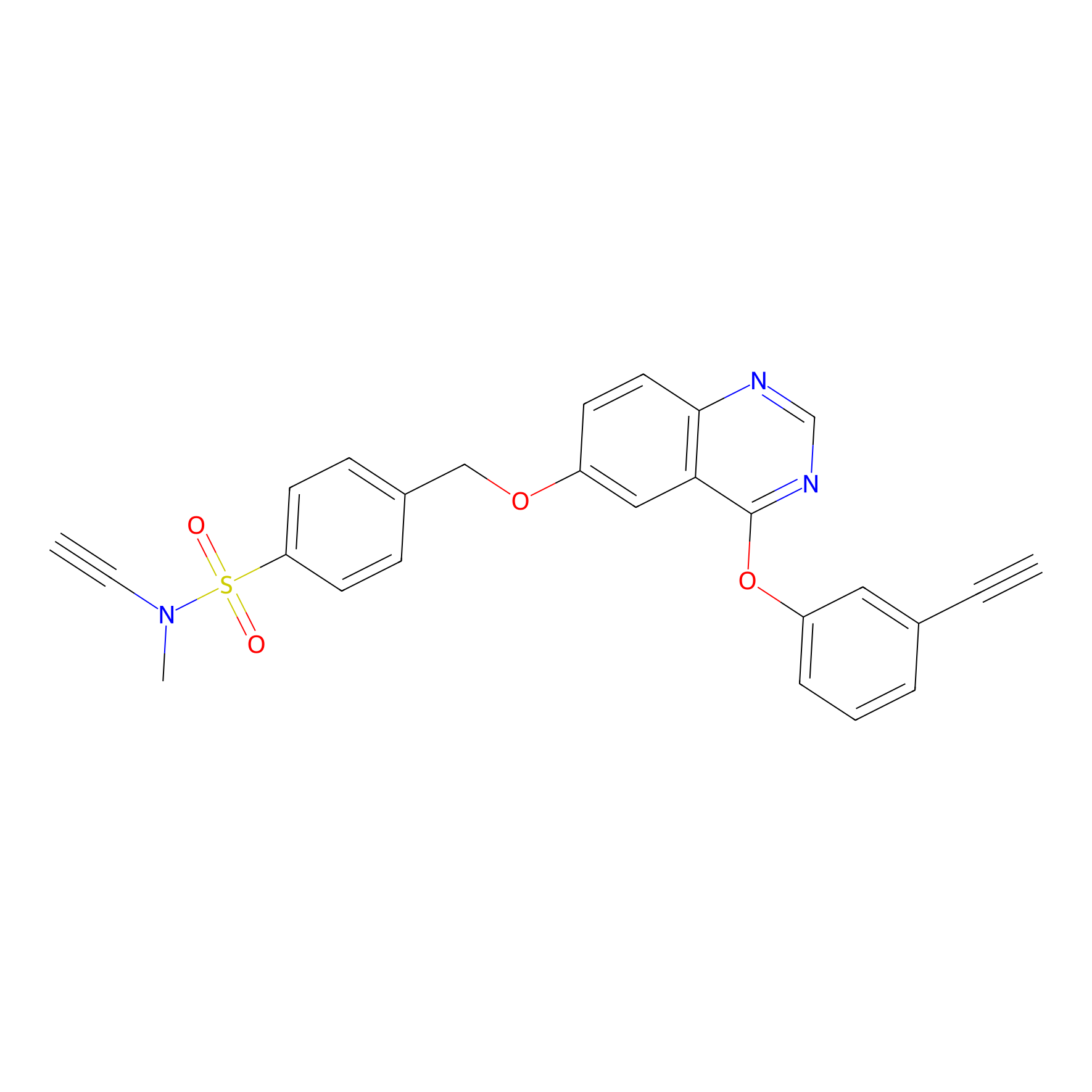 |
100.00 | LDD0445 | [6] | |
|
STPyne Probe Info |
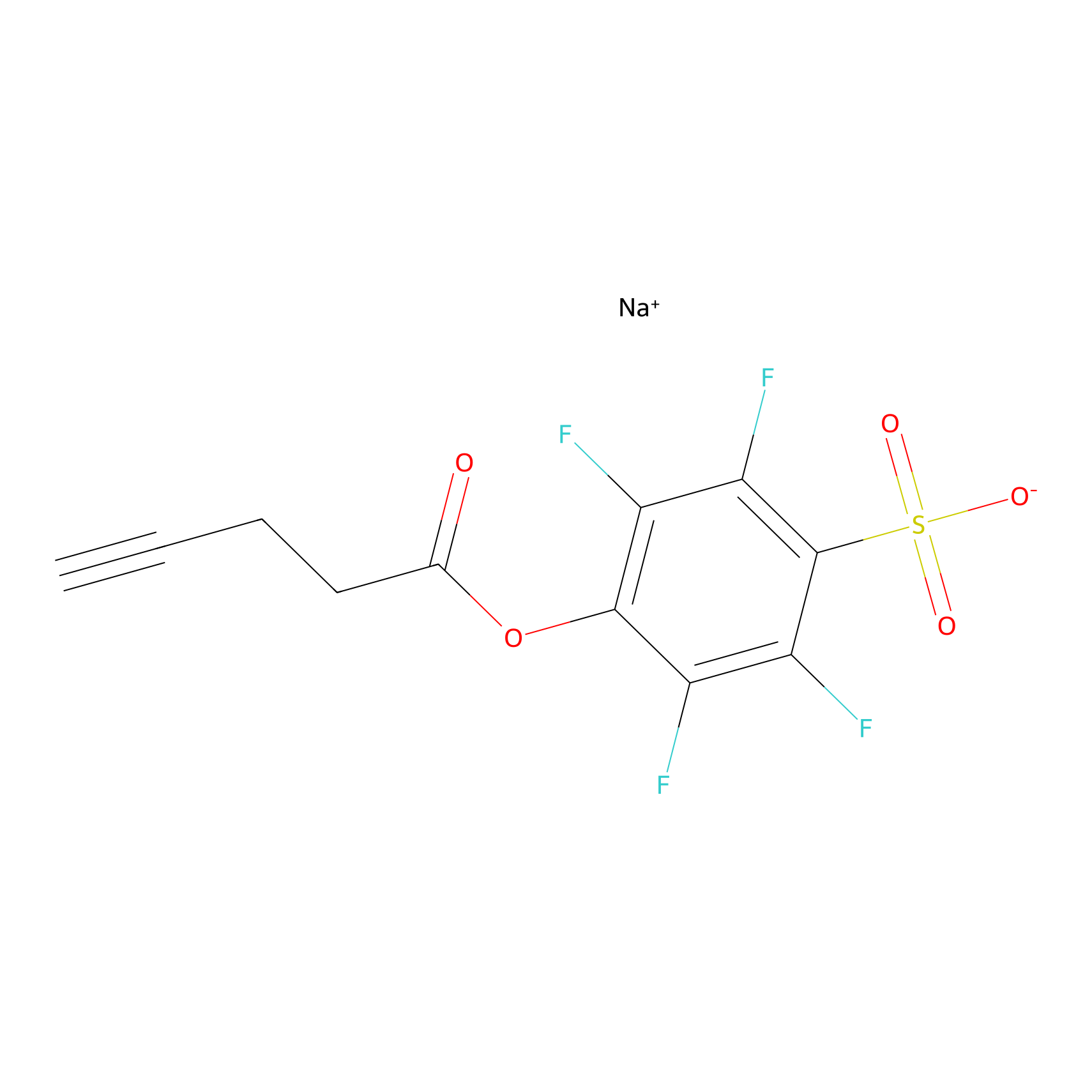 |
K415(6.94); K62(10.00) | LDD0277 | [7] | |
|
IPM Probe Info |
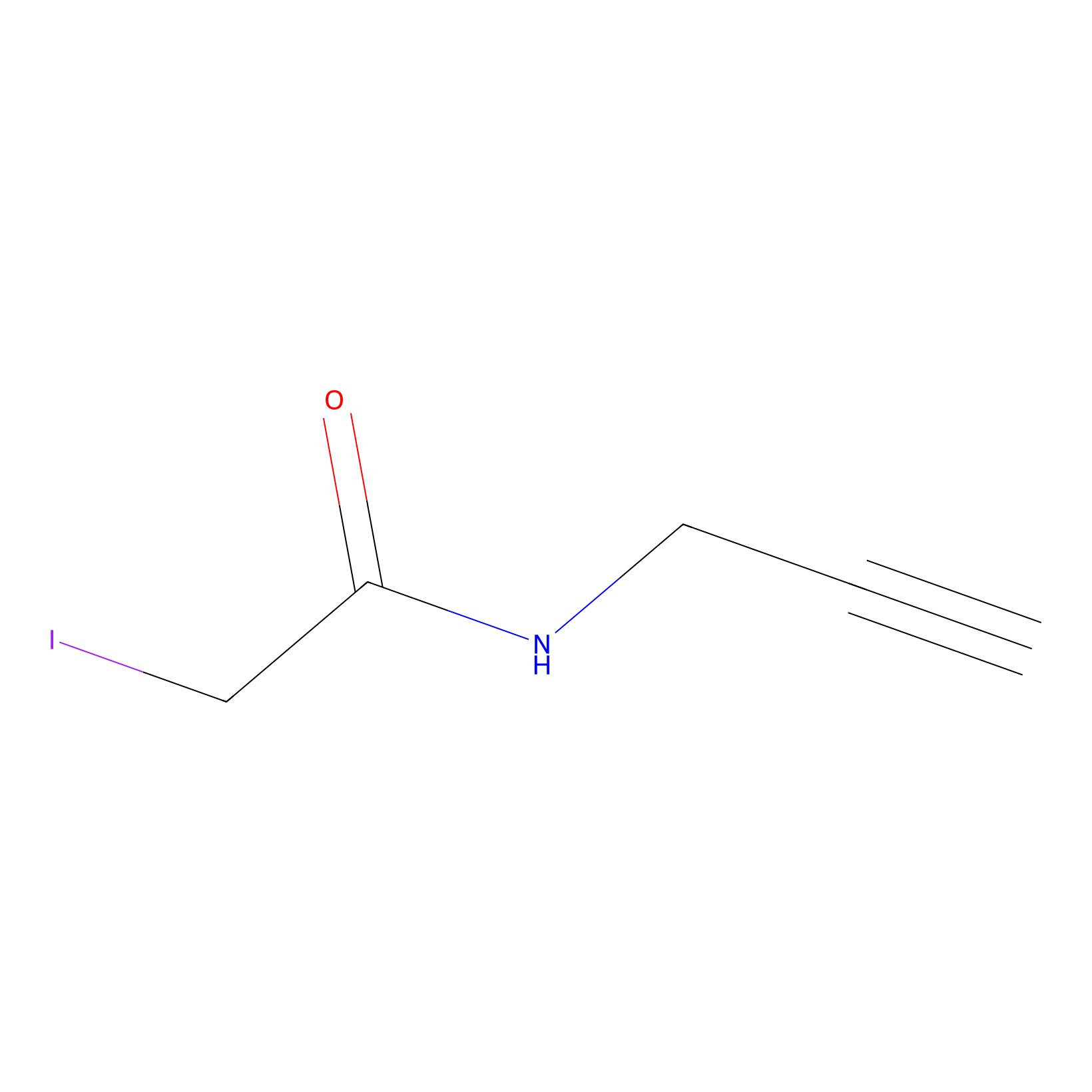 |
N.A. | LDD0241 | [8] | |
|
Probe 1 Probe Info |
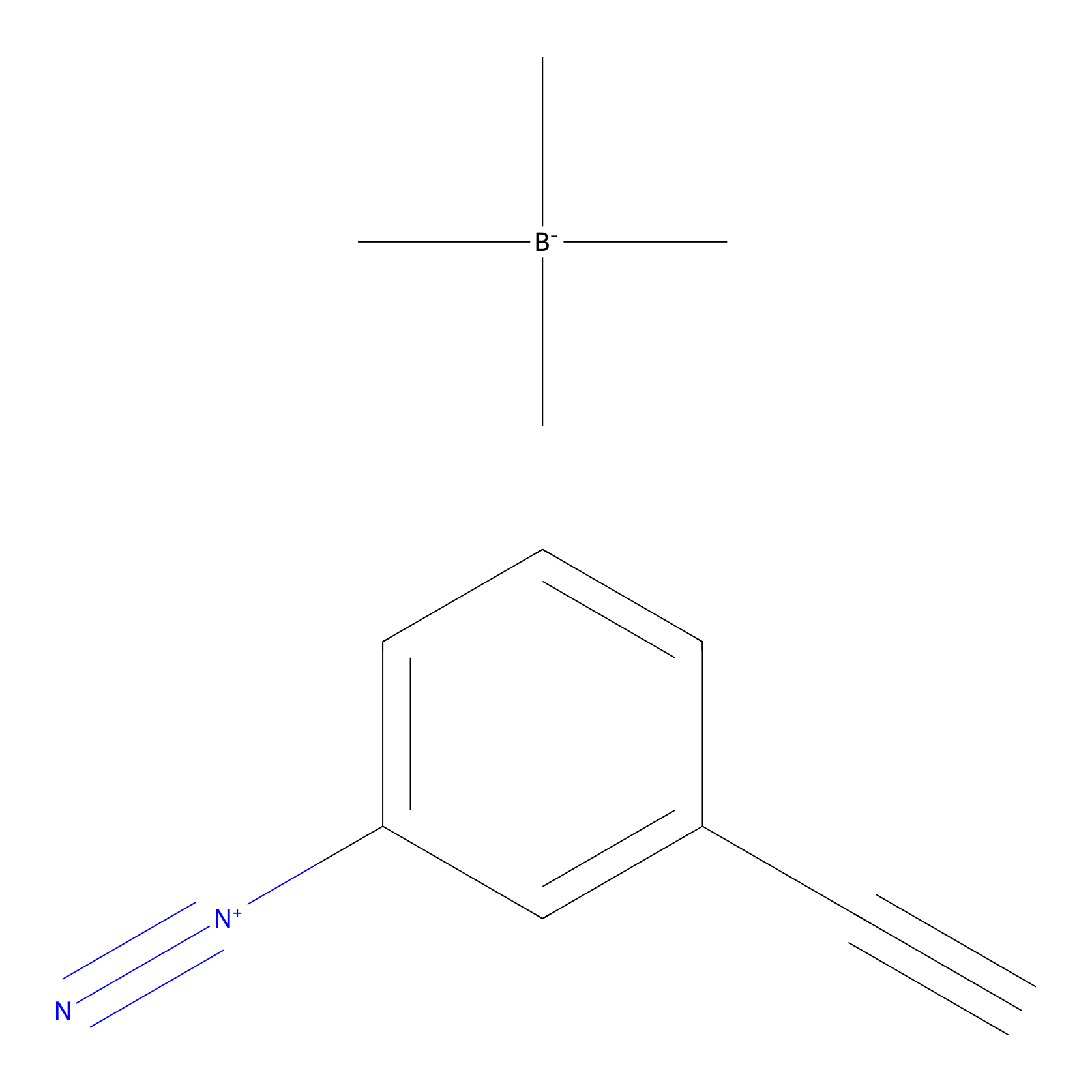 |
Y174(9.64); Y285(95.37); Y403(19.11) | LDD3495 | [9] | |
|
Alkylaryl probe 3 Probe Info |
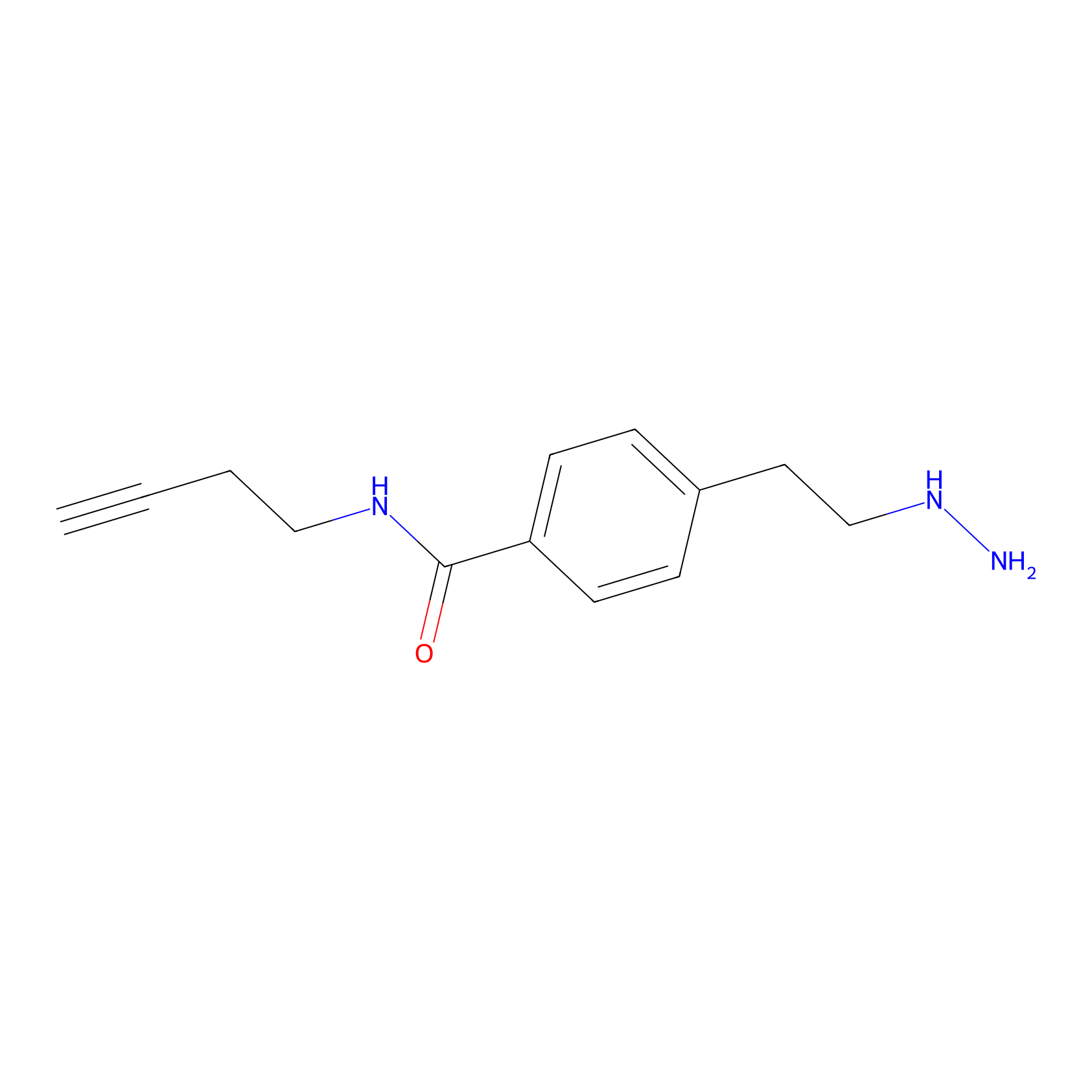 |
7.69 | LDD0283 | [10] | |
|
BTD Probe Info |
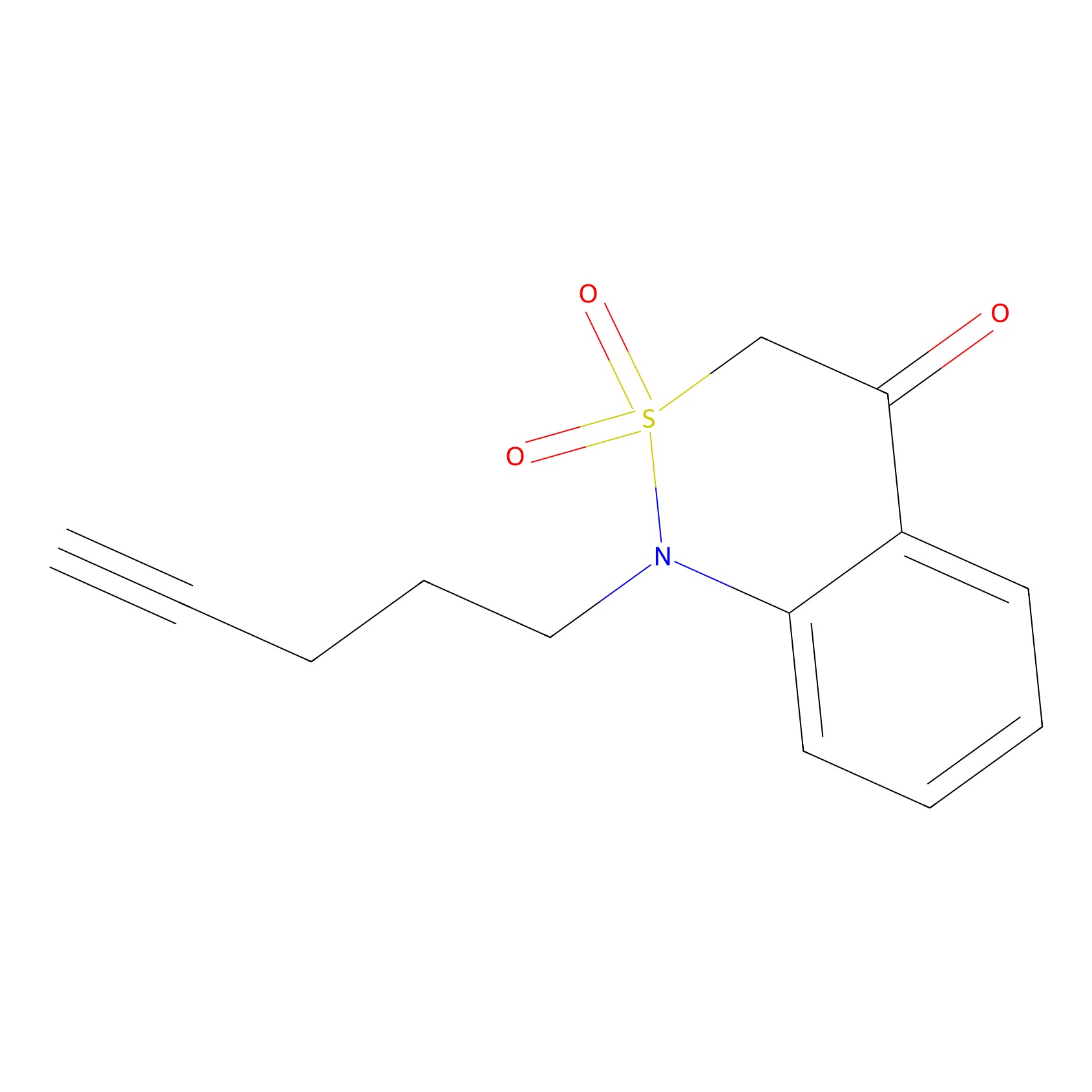 |
C258(1.01) | LDD2093 | [11] | |
|
Curcusone 37 Probe Info |
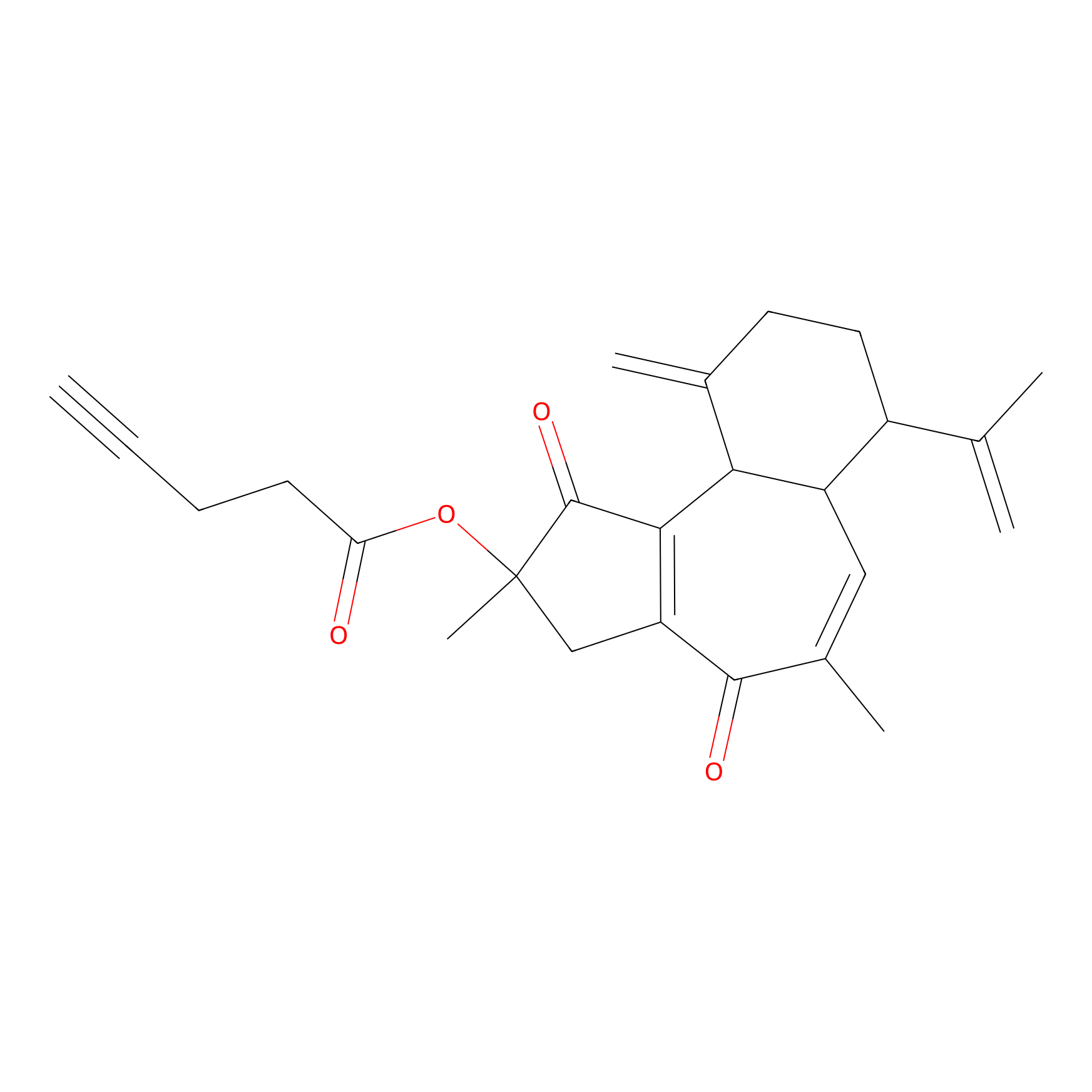 |
2.19 | LDD0188 | [12] | |
|
HPAP Probe Info |
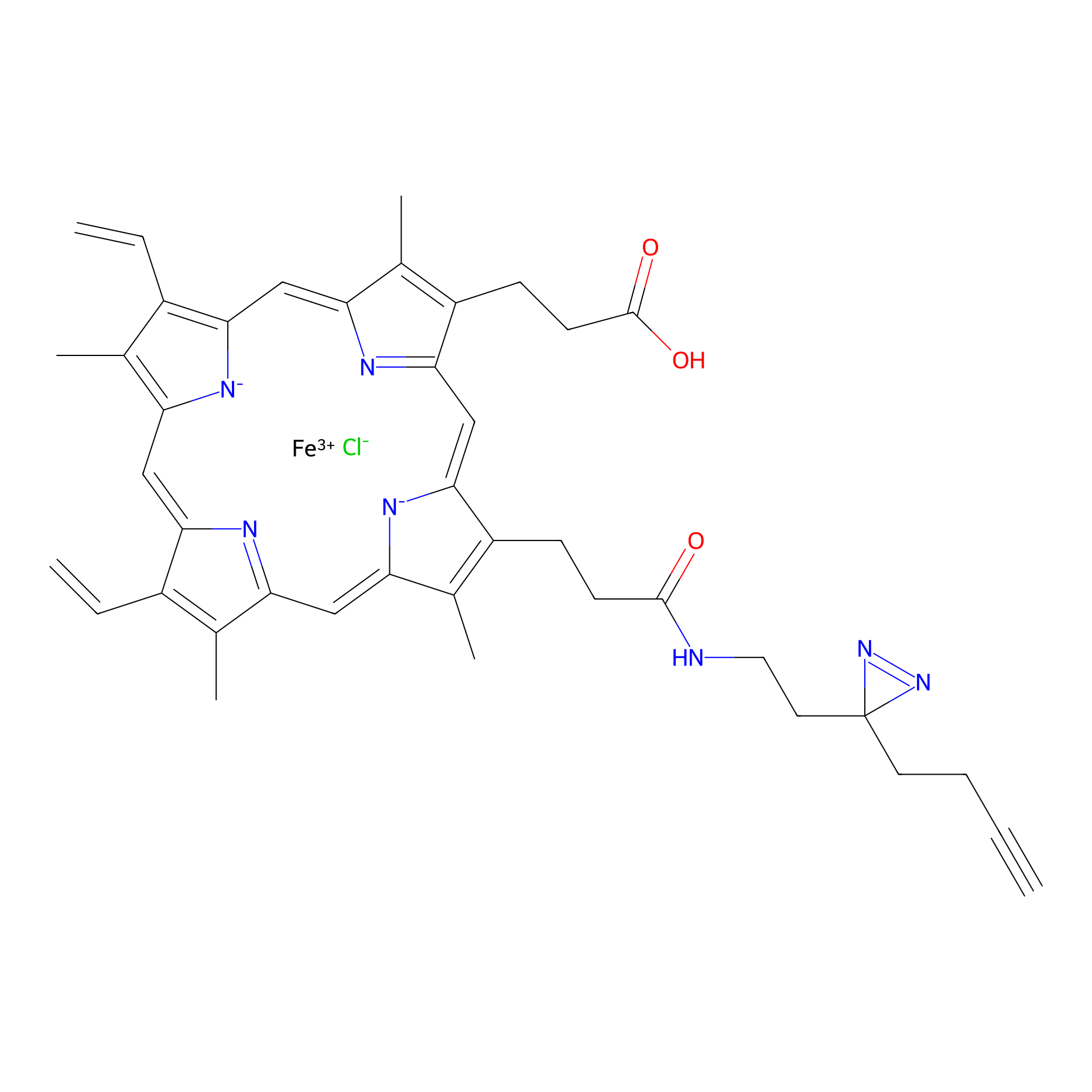 |
3.00 | LDD0063 | [13] | |
|
Alkyne-RA190 Probe Info |
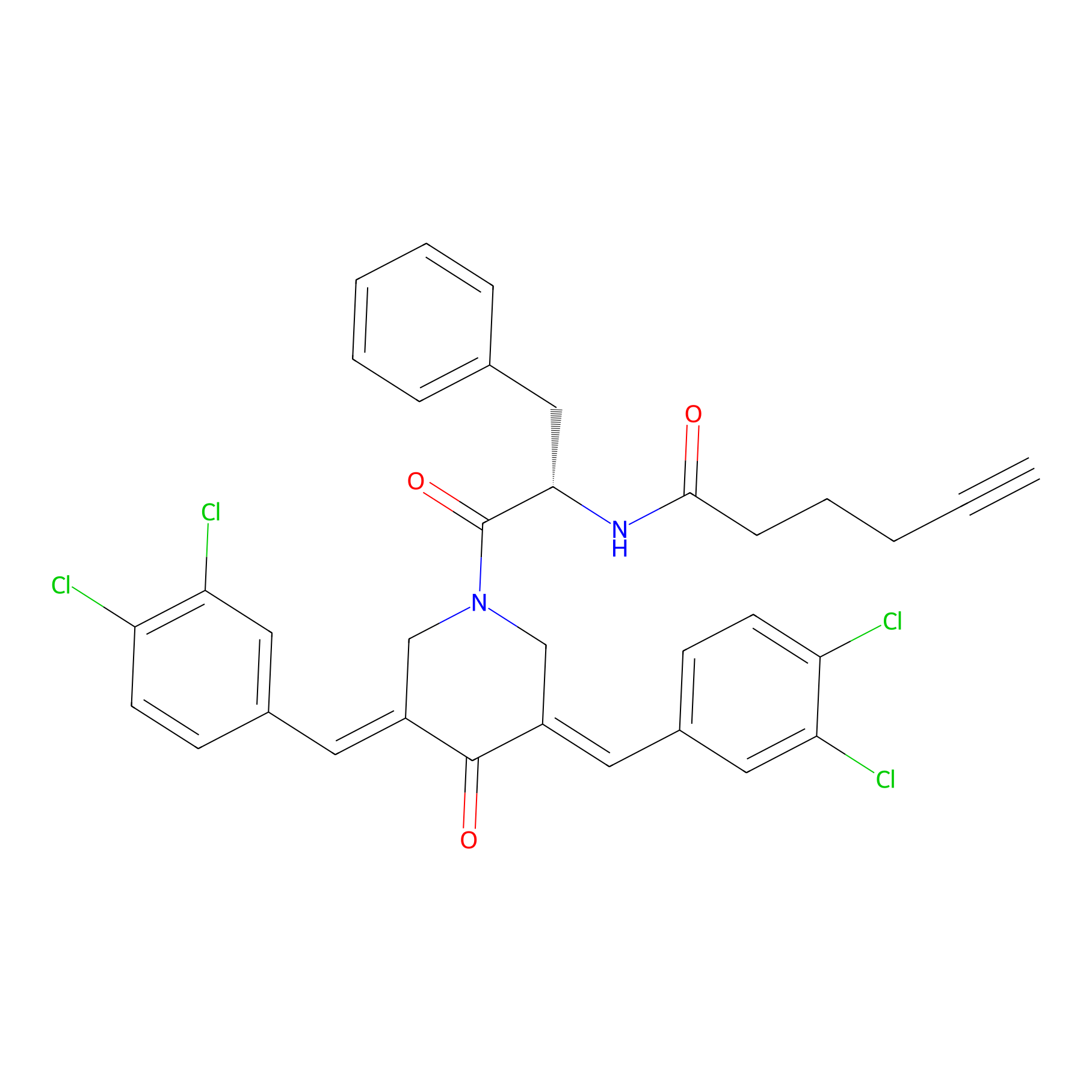 |
3.43 | LDD0299 | [14] | |
|
DBIA Probe Info |
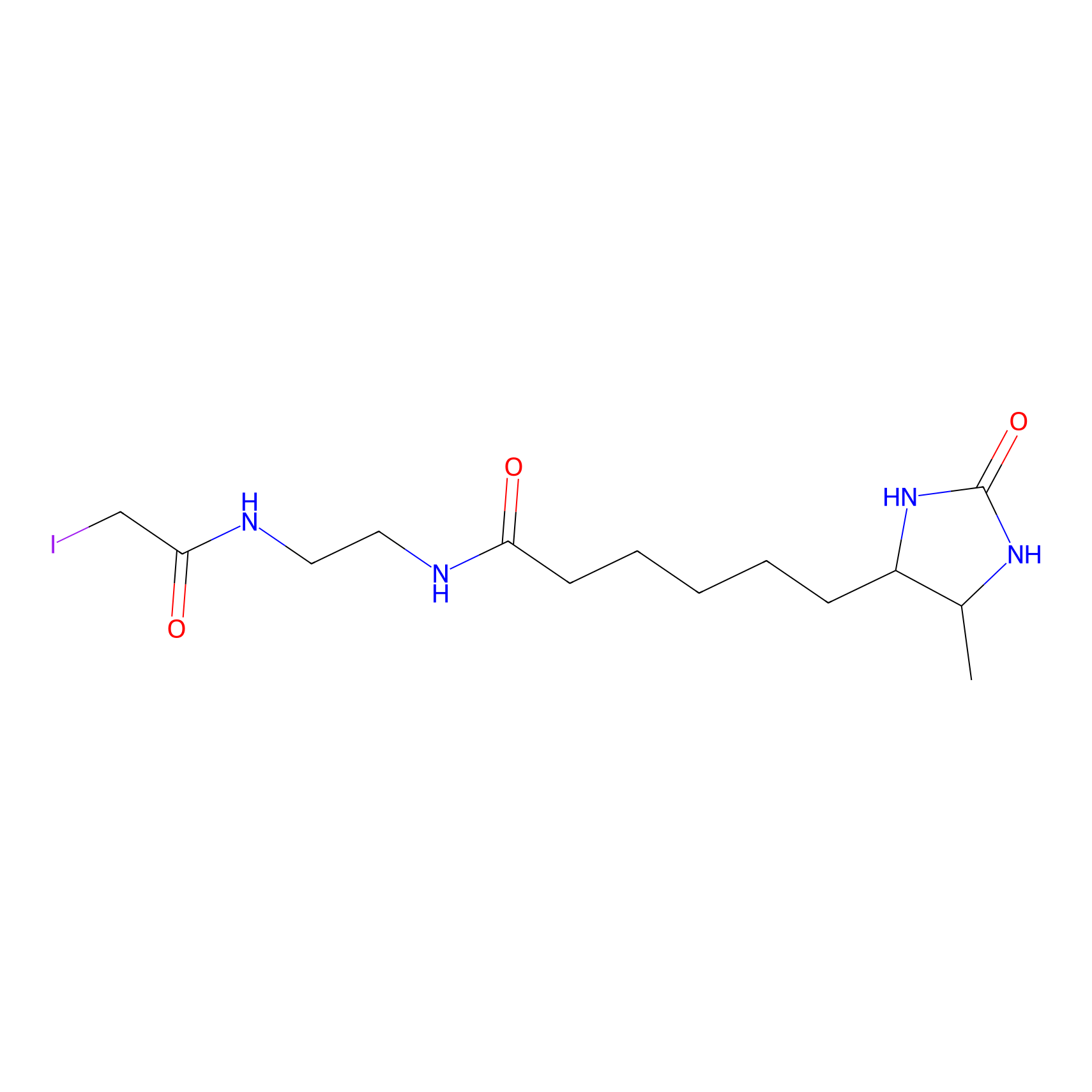 |
C242(0.92) | LDD0547 | [15] | |
|
5E-2FA Probe Info |
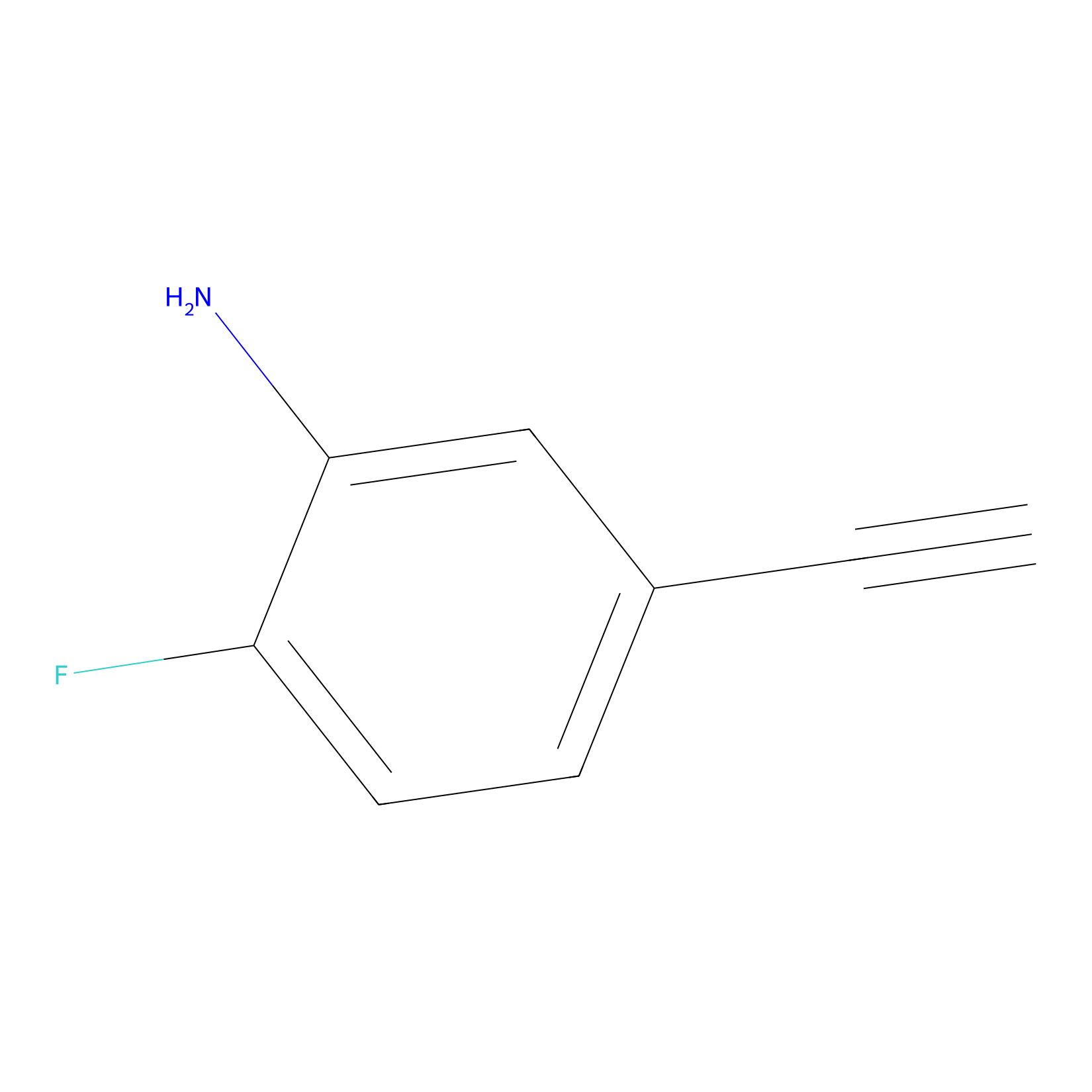 |
N.A. | LDD2235 | [16] | |
|
ATP probe Probe Info |
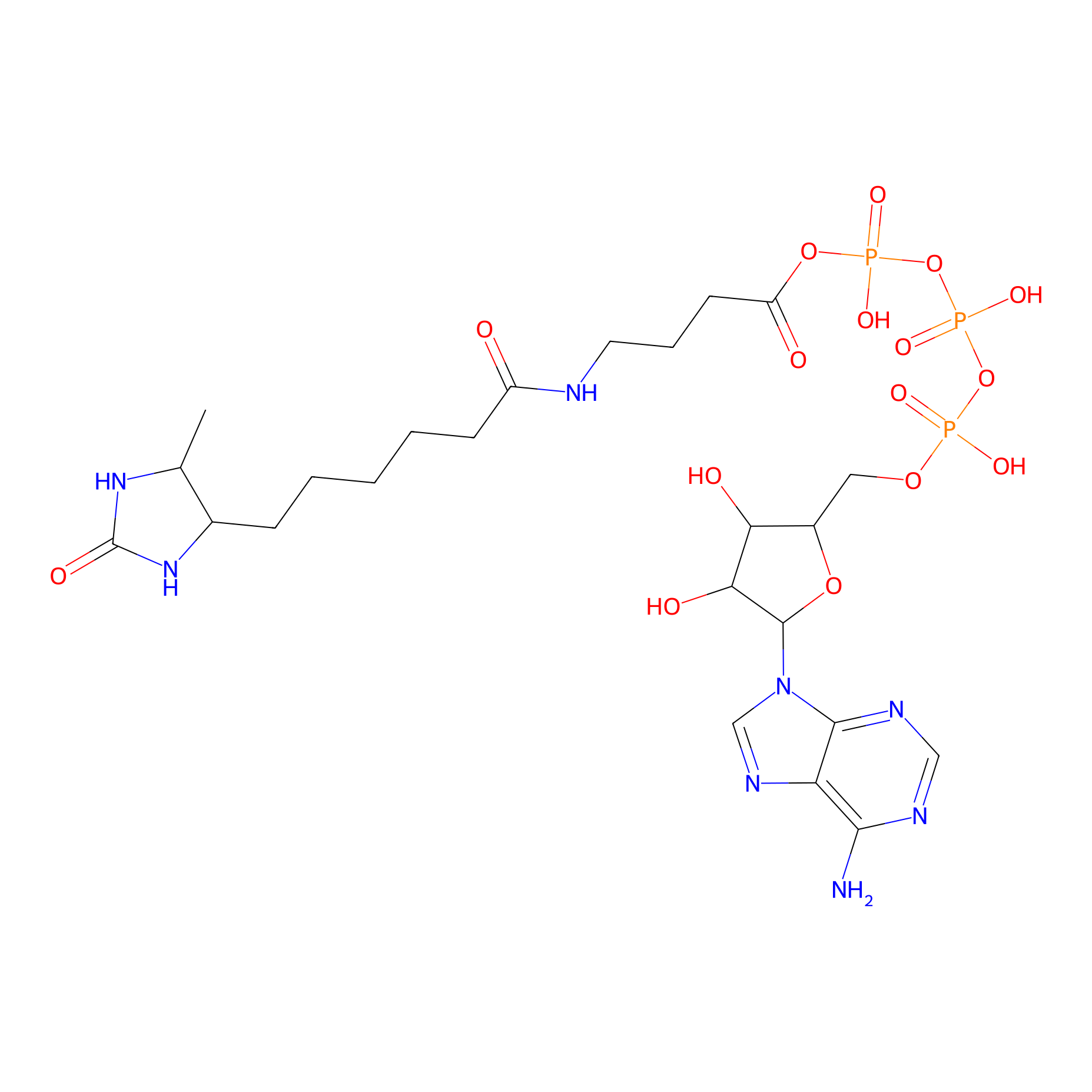 |
N.A. | LDD0199 | [17] | |
|
m-APA Probe Info |
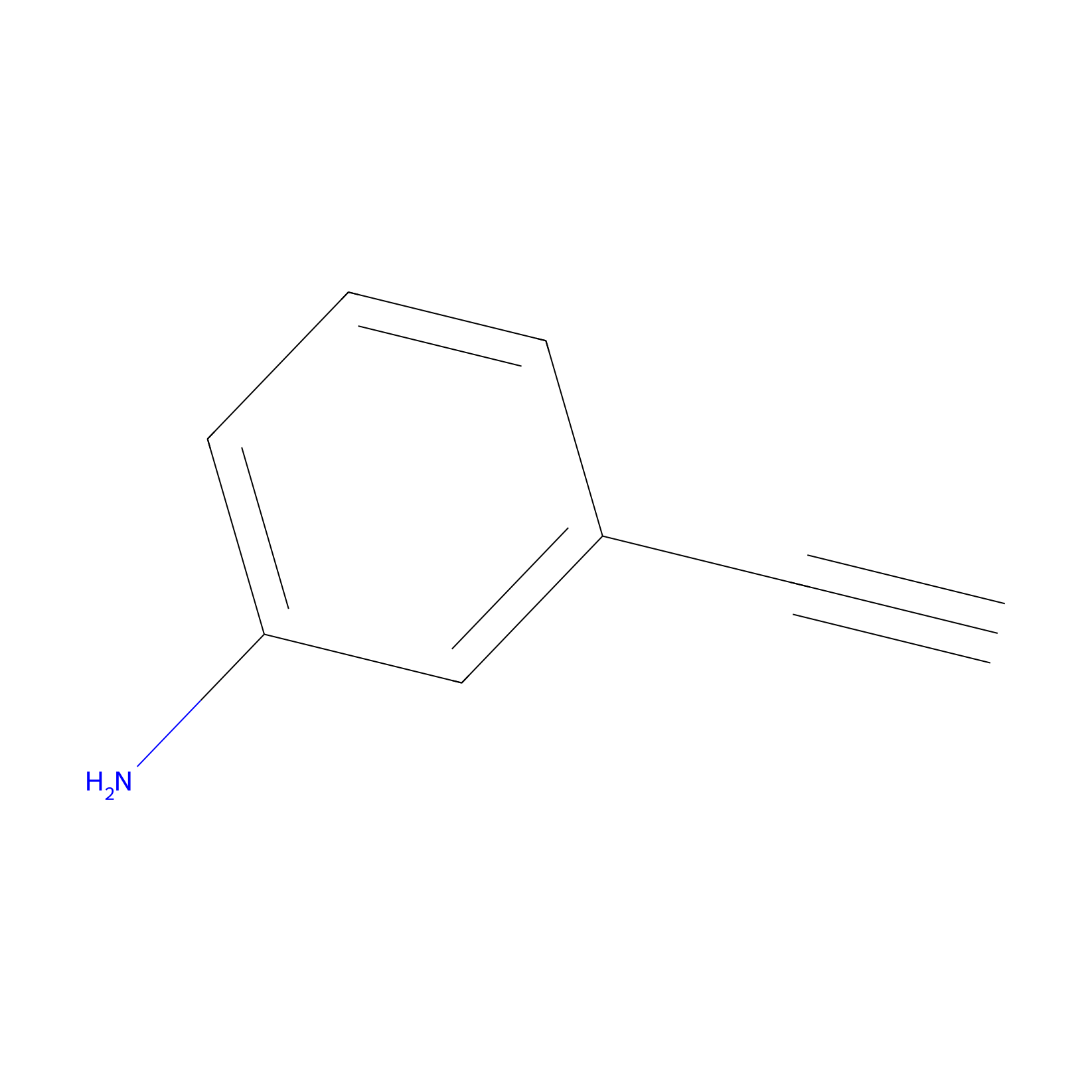 |
N.A. | LDD2231 | [16] | |
|
4-Iodoacetamidophenylacetylene Probe Info |
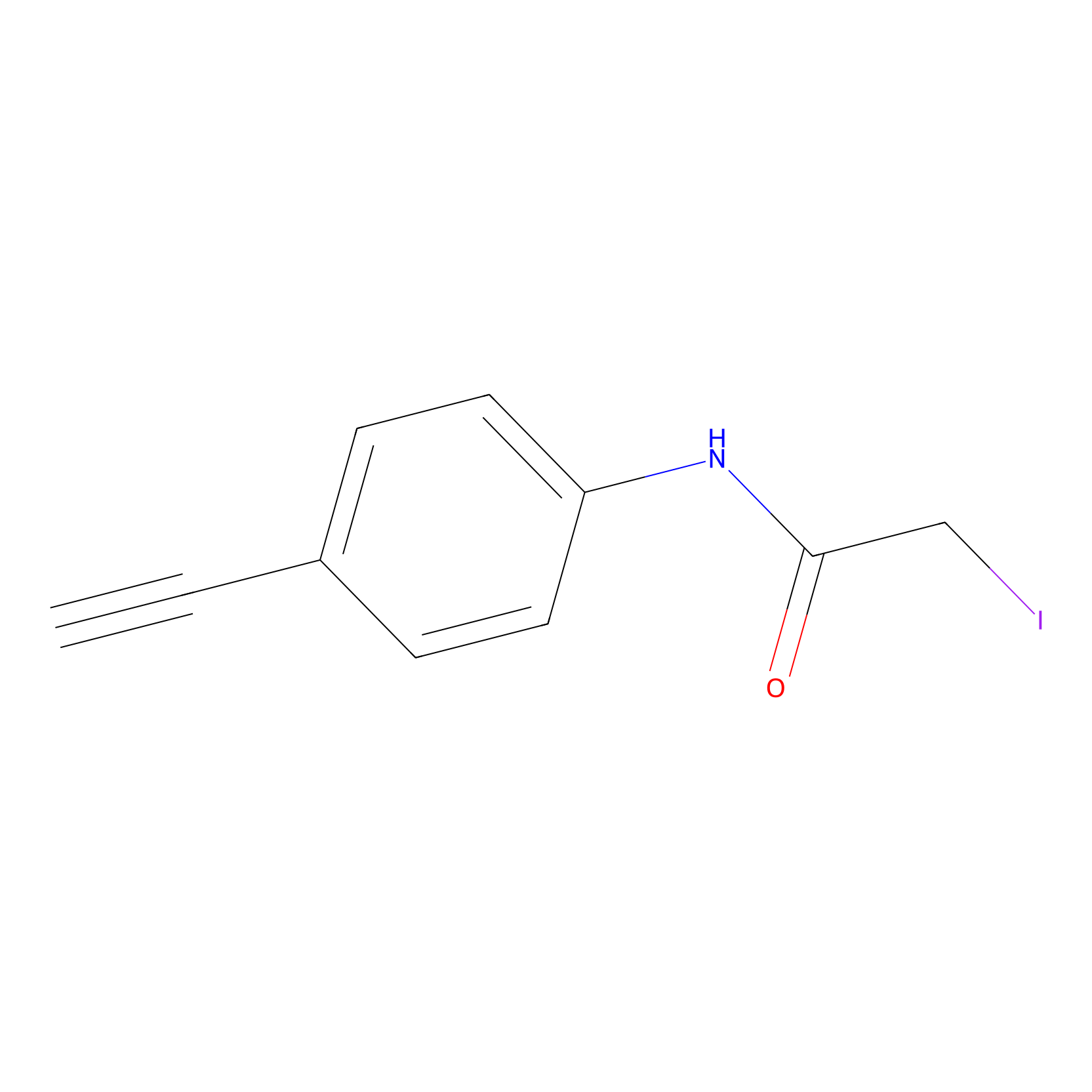 |
C445(0.00); C462(0.00); C258(0.00) | LDD0038 | [18] | |
|
IA-alkyne Probe Info |
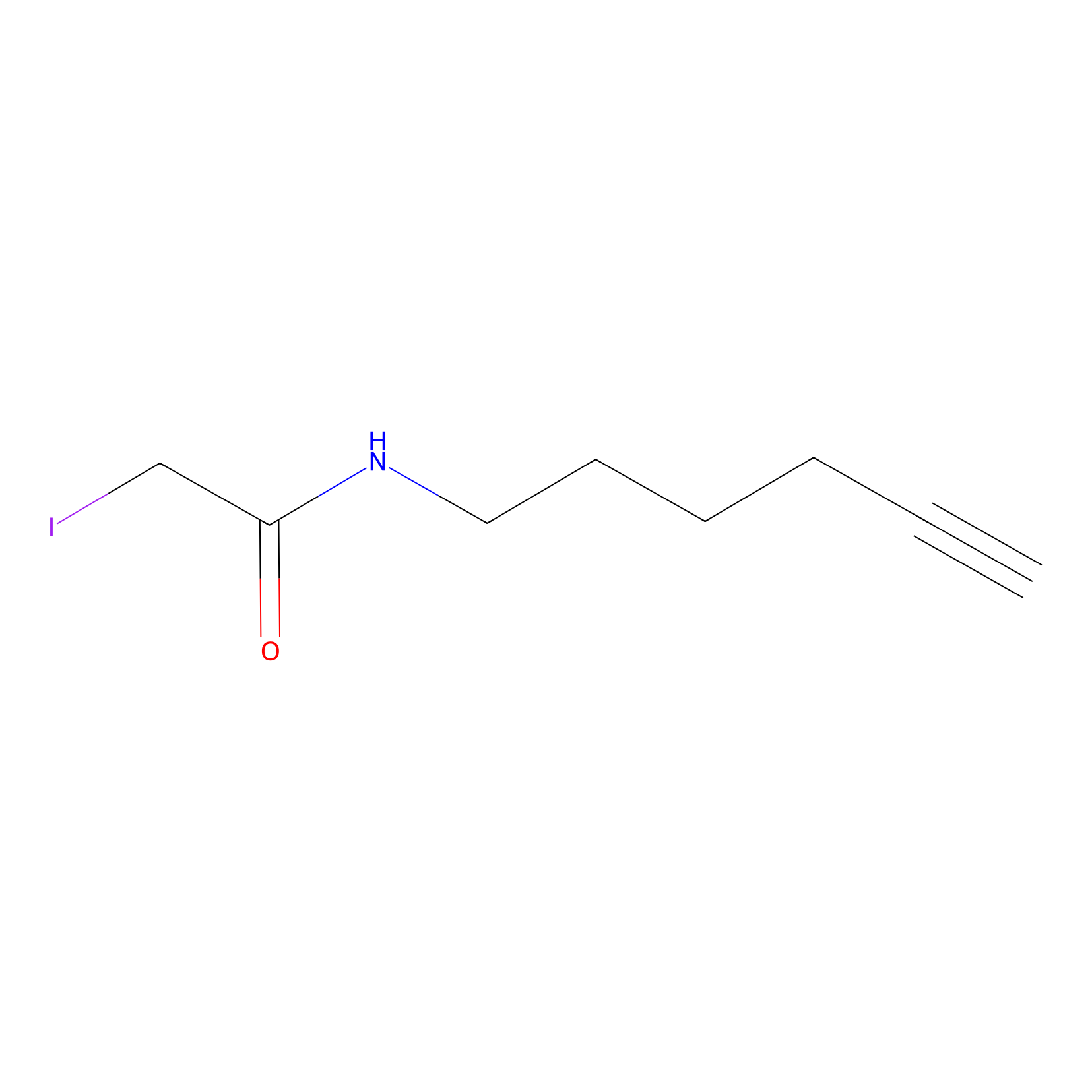 |
C462(0.00); C445(0.00); C258(0.00) | LDD0036 | [18] | |
|
Lodoacetamide azide Probe Info |
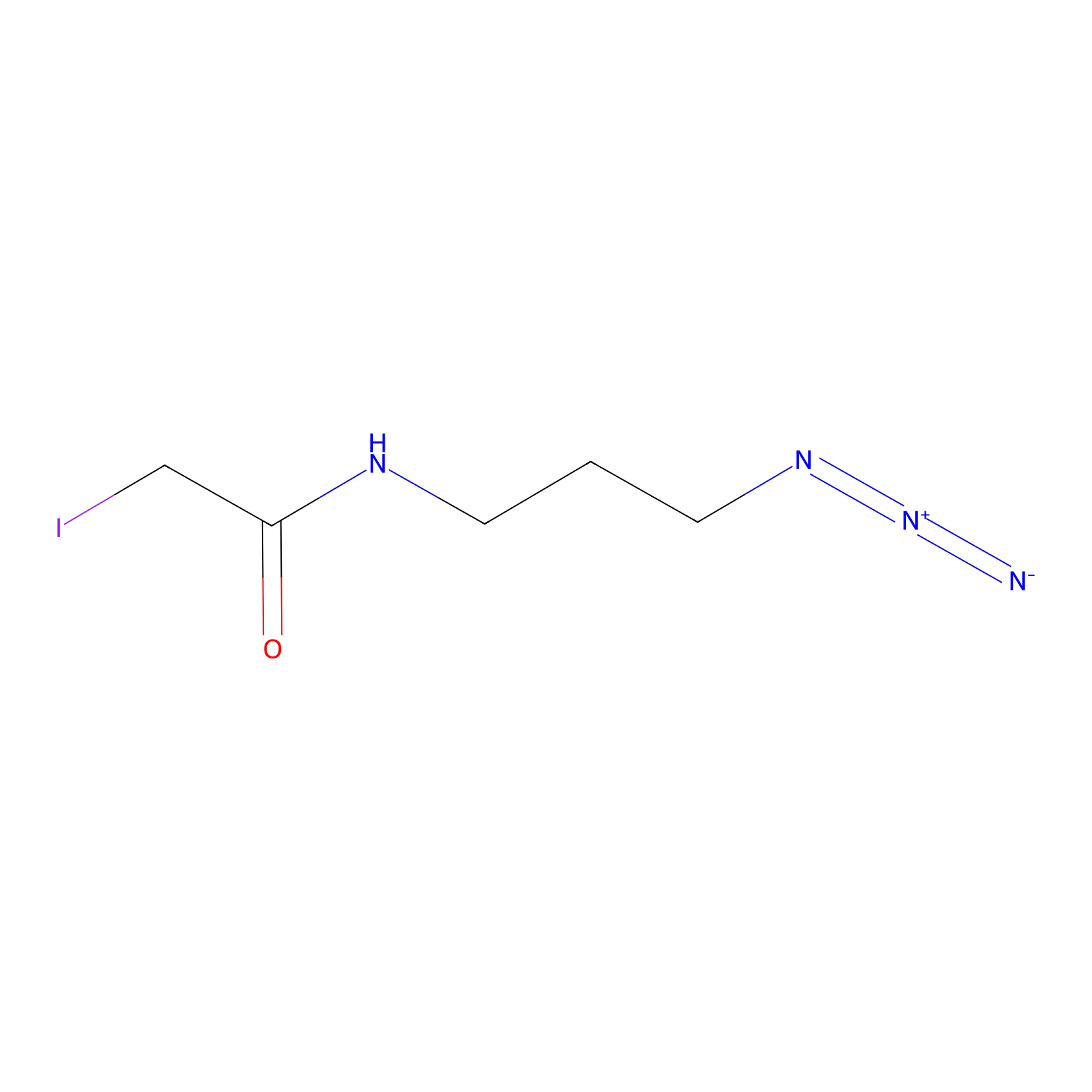 |
C445(0.00); C258(0.00); C462(0.00) | LDD0037 | [18] | |
|
WYneN Probe Info |
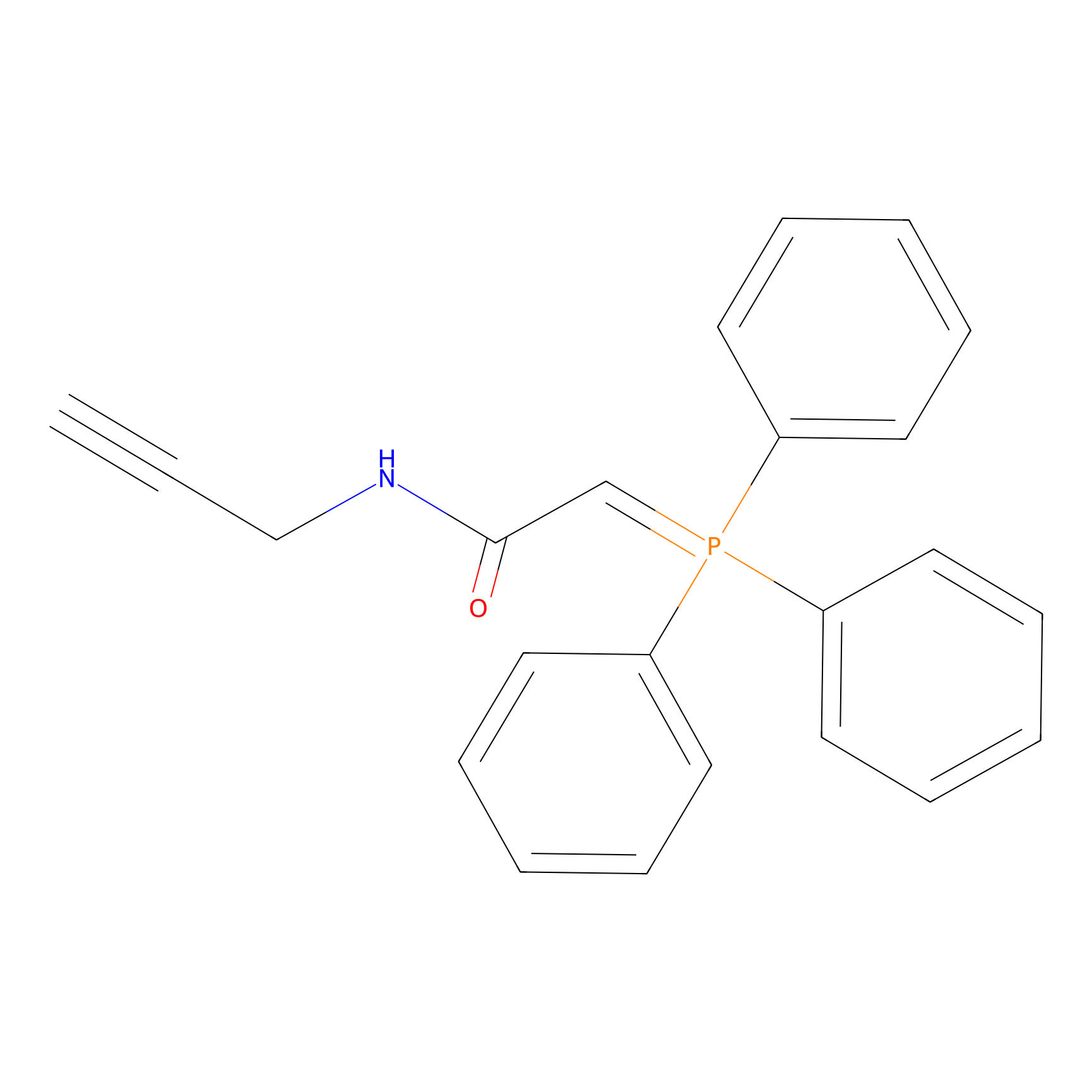 |
N.A. | LDD0021 | [19] | |
|
WYneO Probe Info |
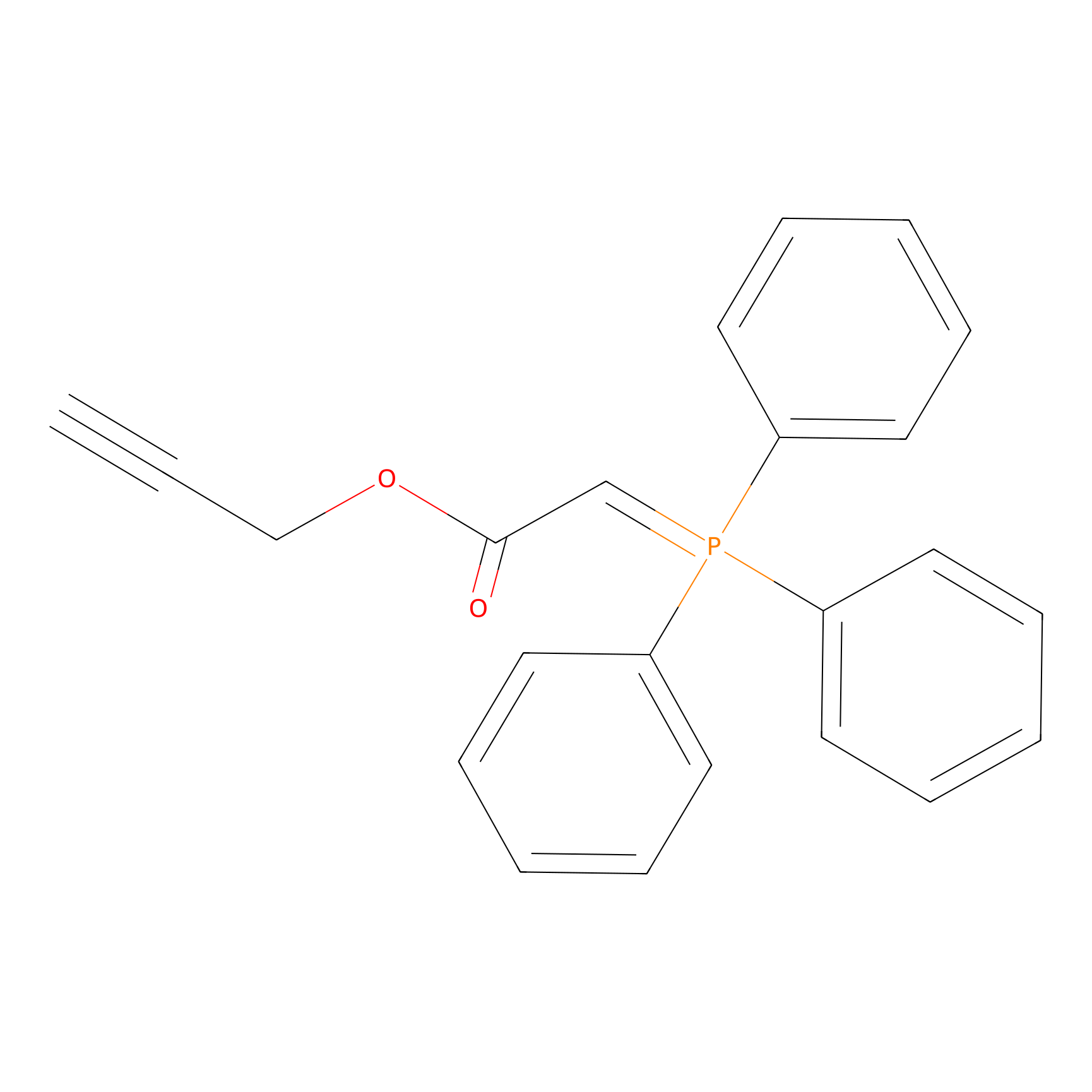 |
N.A. | LDD0022 | [19] | |
|
NHS Probe Info |
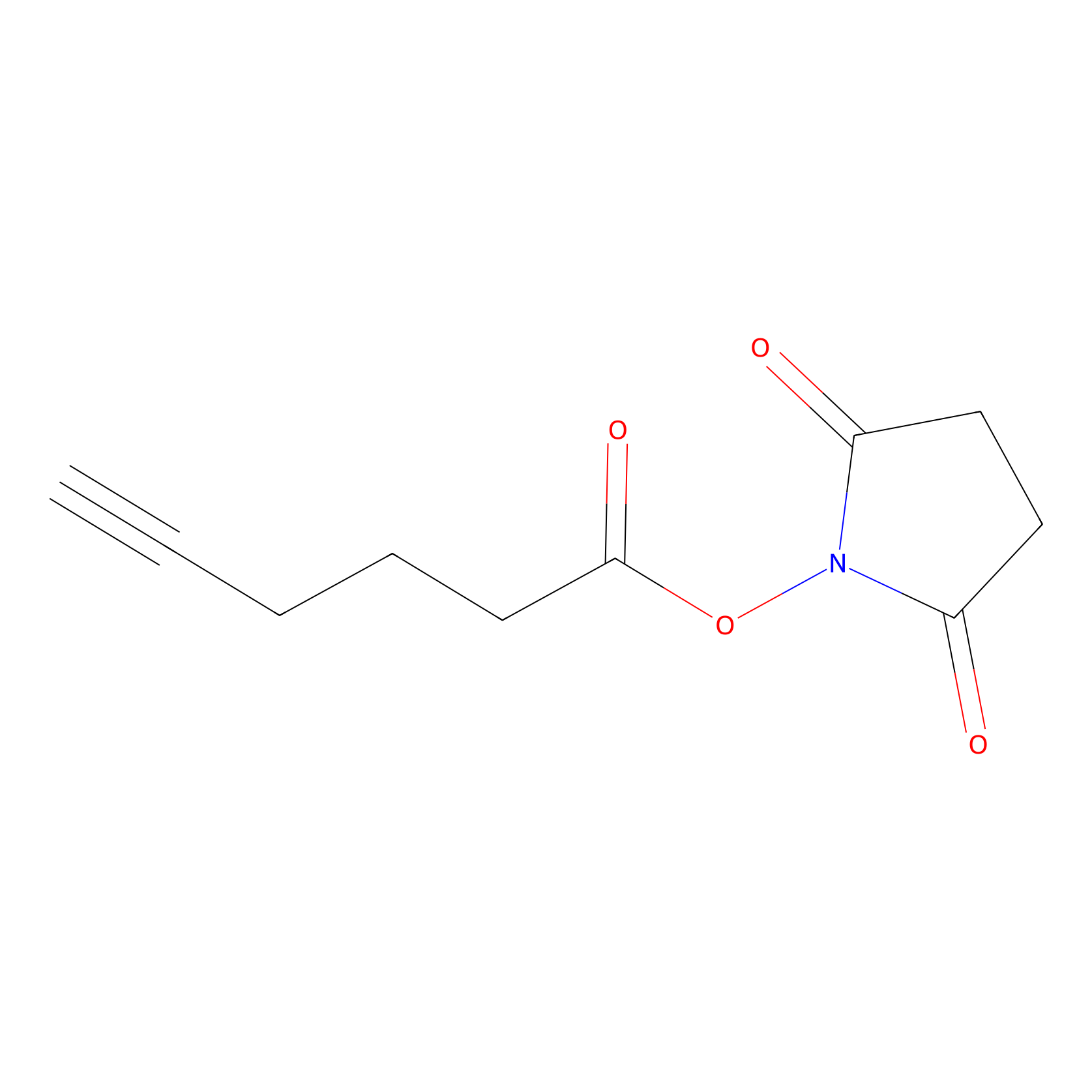 |
K415(0.00); K205(0.00) | LDD0010 | [19] | |
|
Acrolein Probe Info |
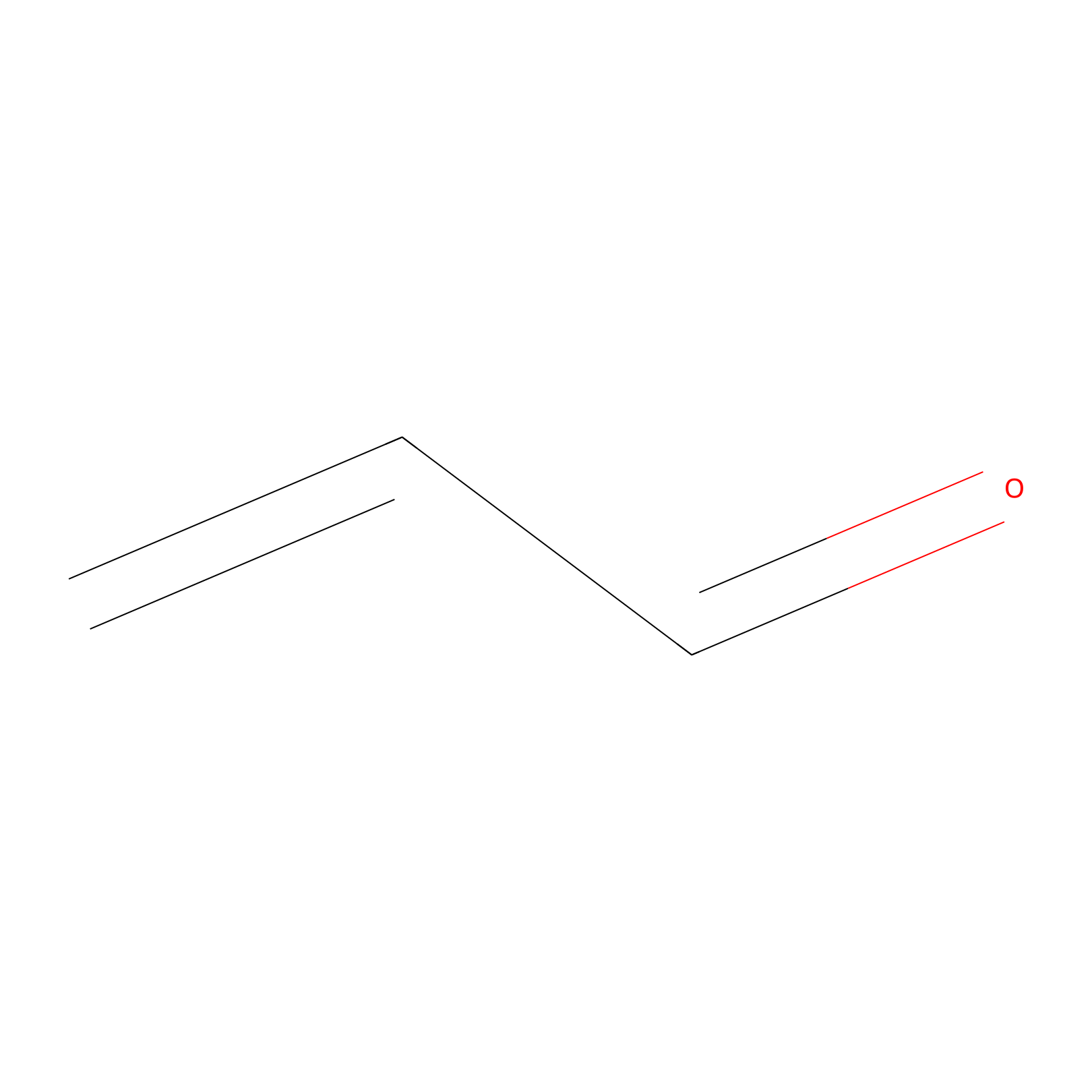 |
N.A. | LDD0217 | [20] | |
|
Crotonaldehyde Probe Info |
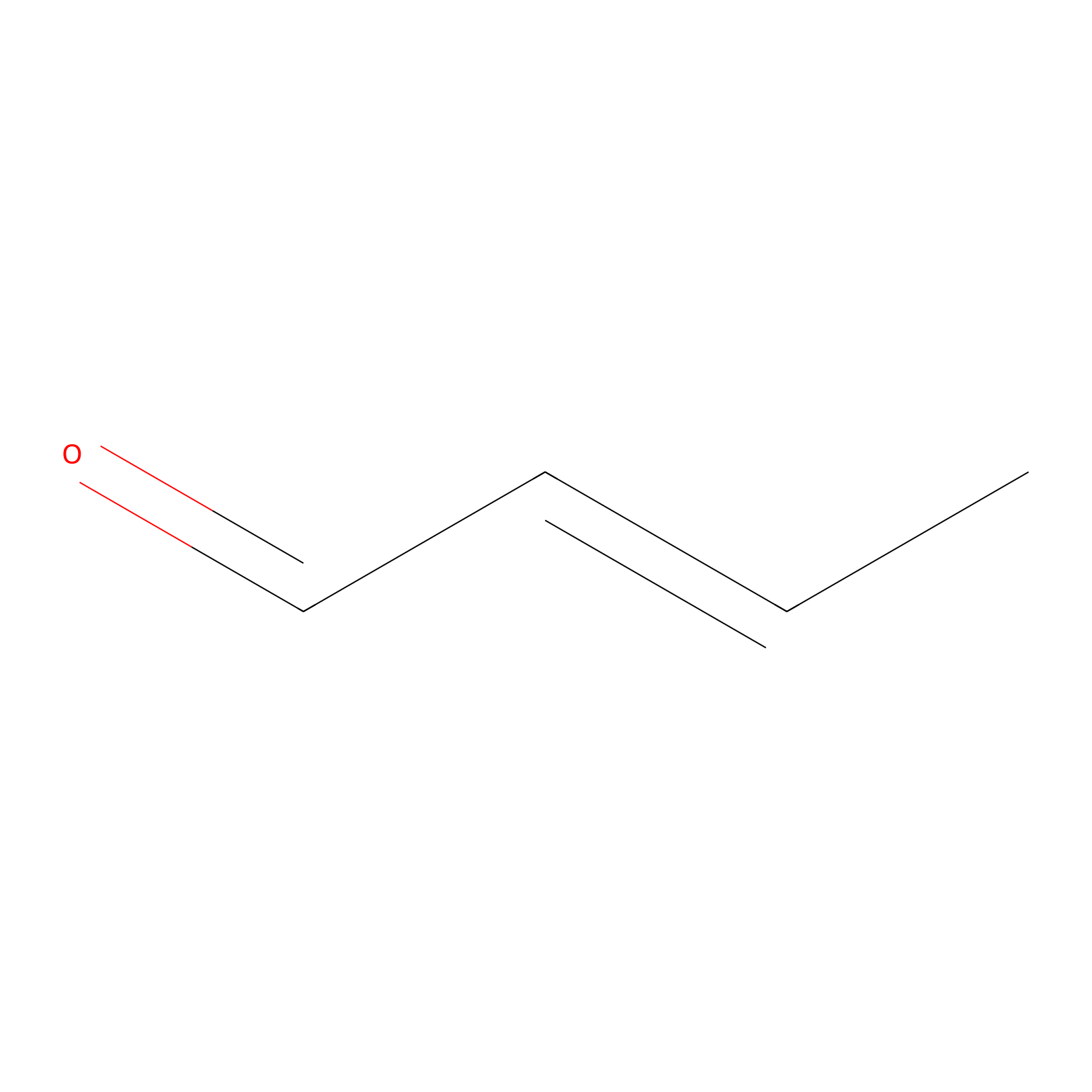 |
N.A. | LDD0219 | [20] | |
|
Methacrolein Probe Info |
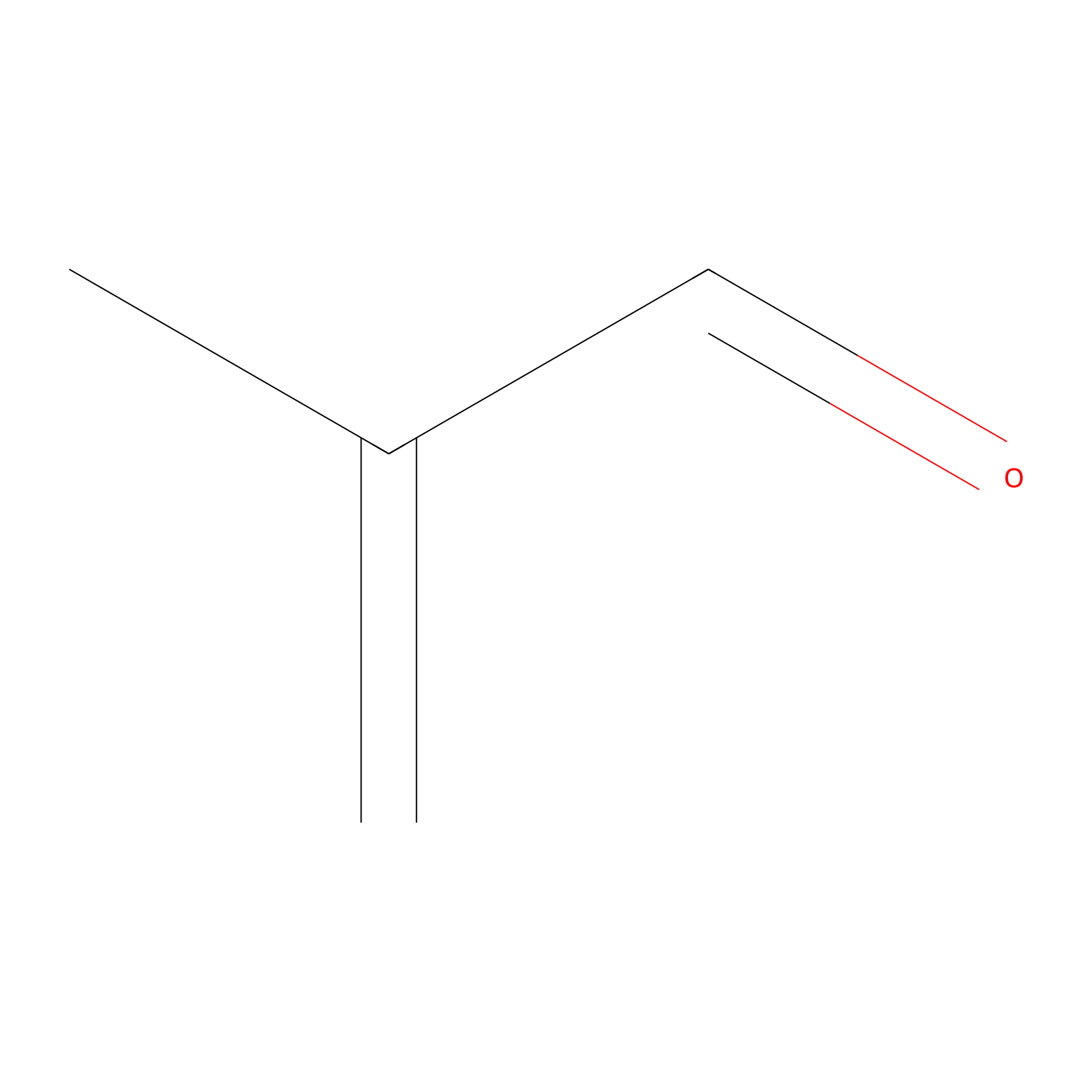 |
N.A. | LDD0218 | [20] | |
|
W1 Probe Info |
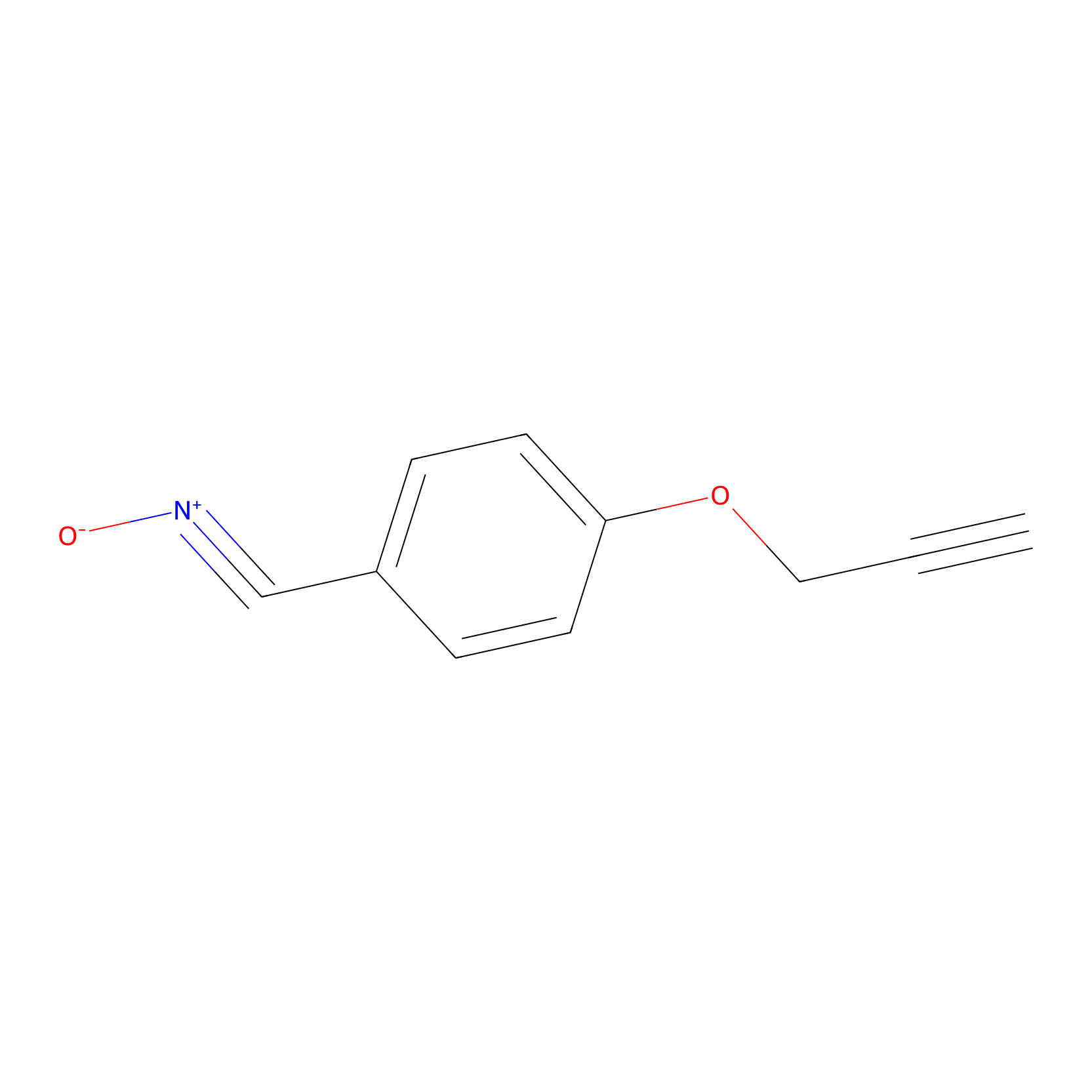 |
W155(0.00); H153(0.00) | LDD0236 | [8] | |
|
AOyne Probe Info |
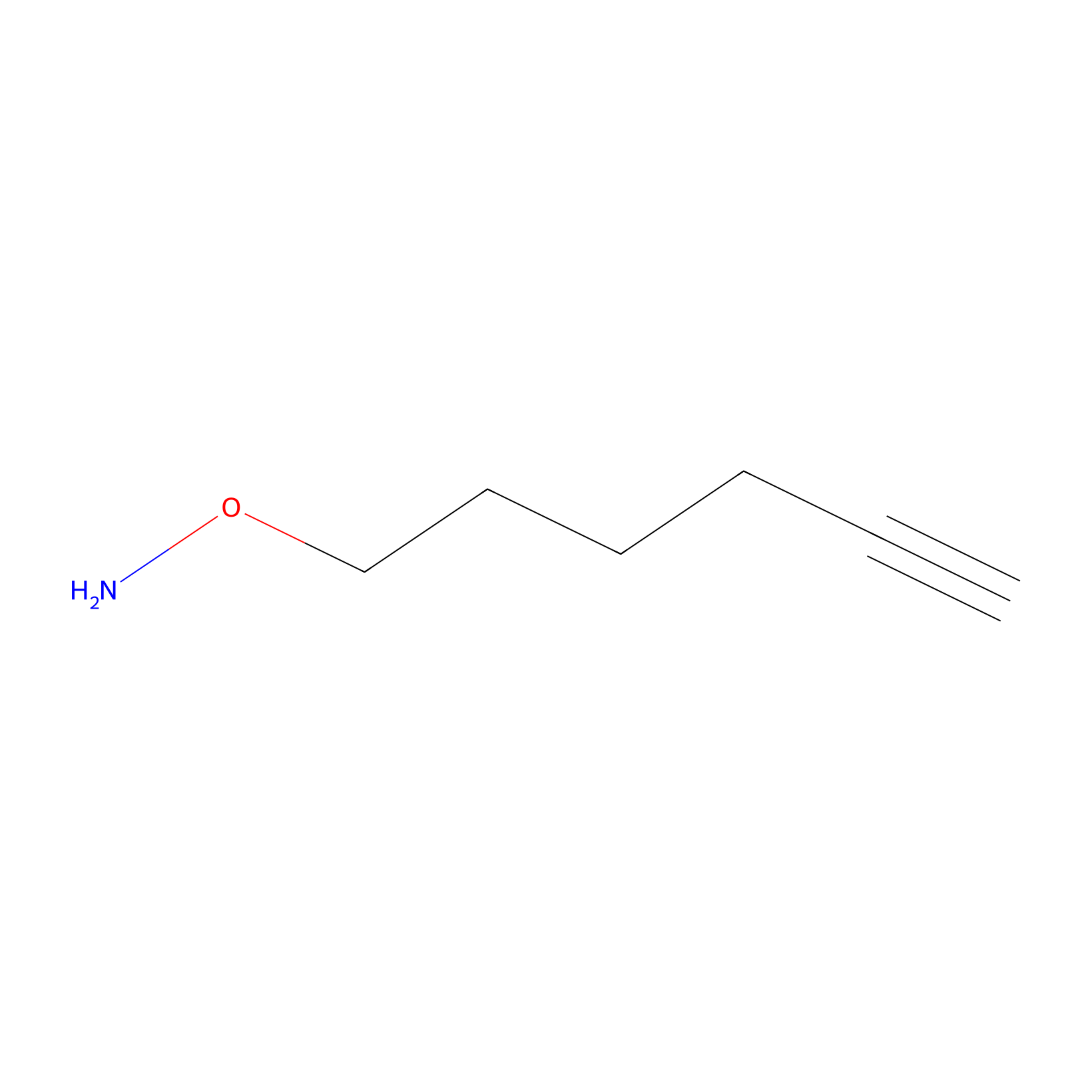 |
10.60 | LDD0443 | [21] | |
|
MPP-AC Probe Info |
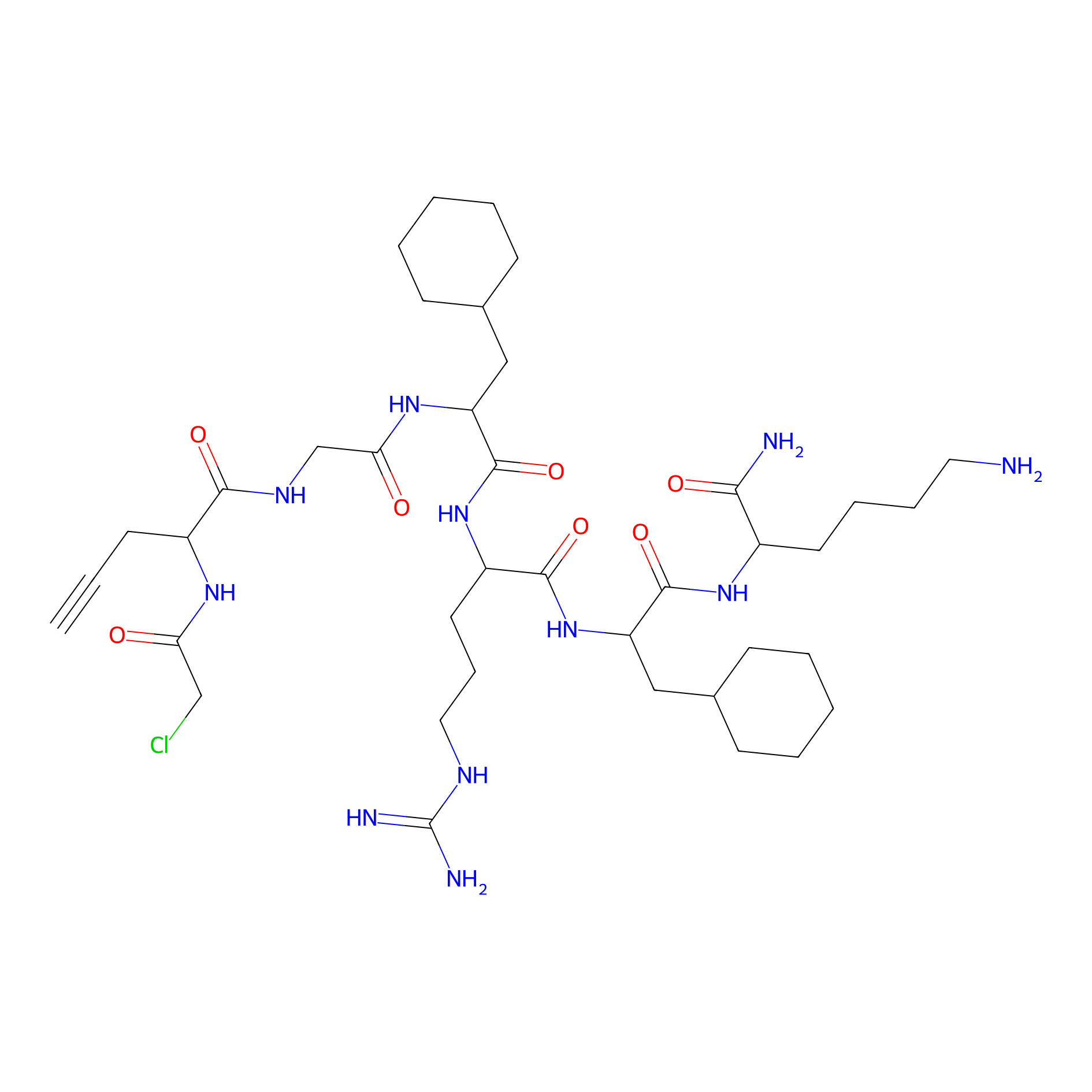 |
N.A. | LDD0428 | [22] | |
|
NAIA_5 Probe Info |
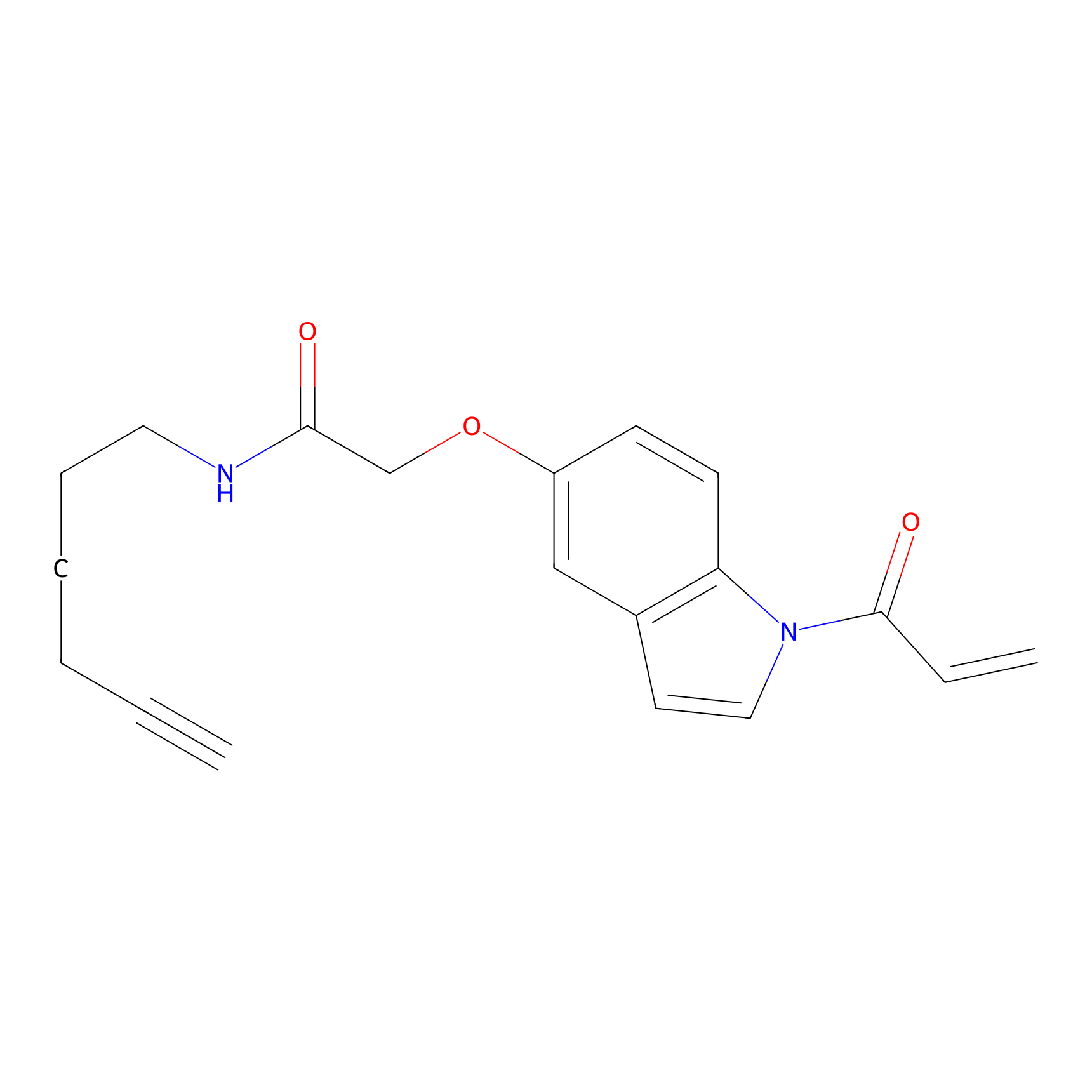 |
N.A. | LDD2223 | [23] | |
|
TER-AC Probe Info |
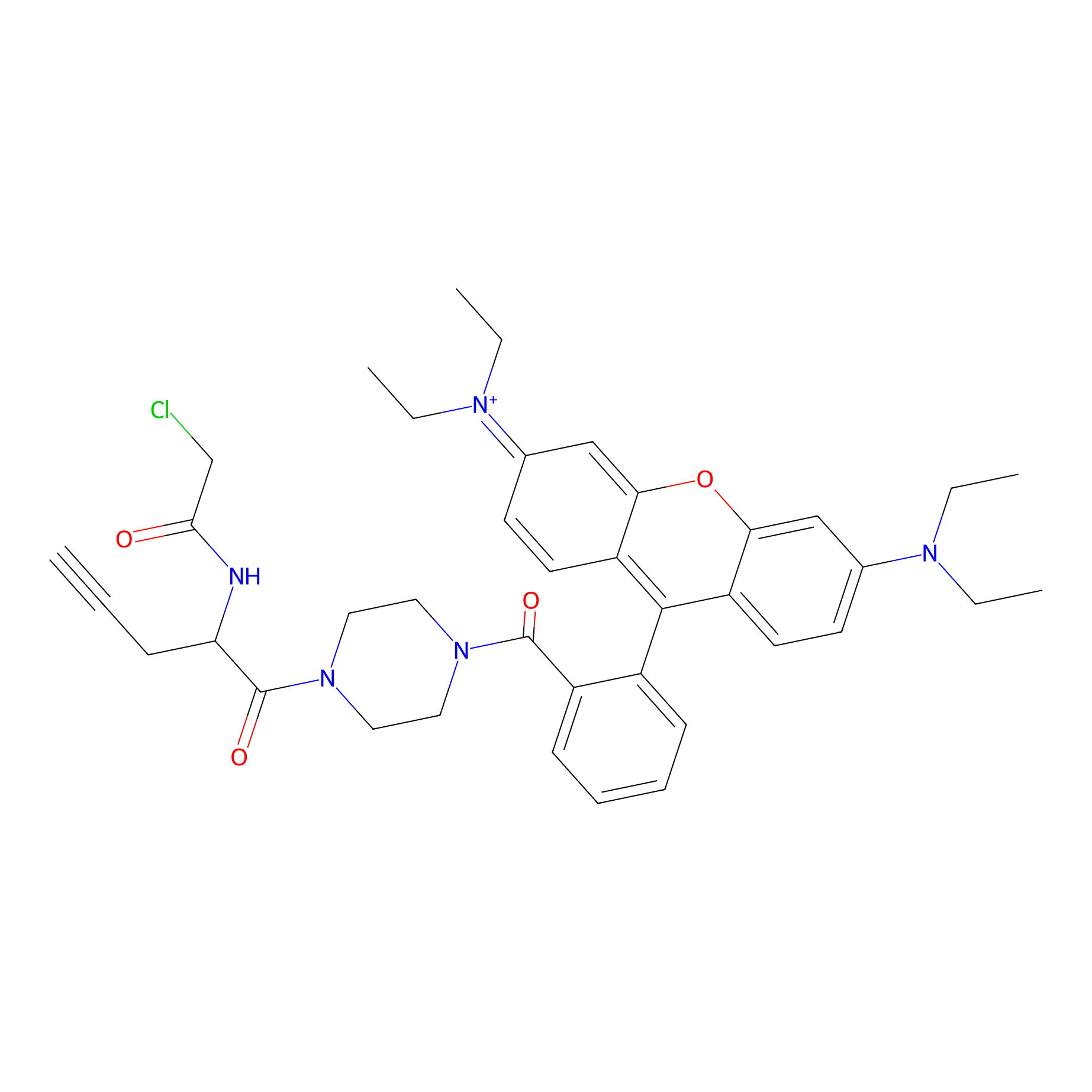 |
N.A. | LDD0426 | [22] | |
|
TPP-AC Probe Info |
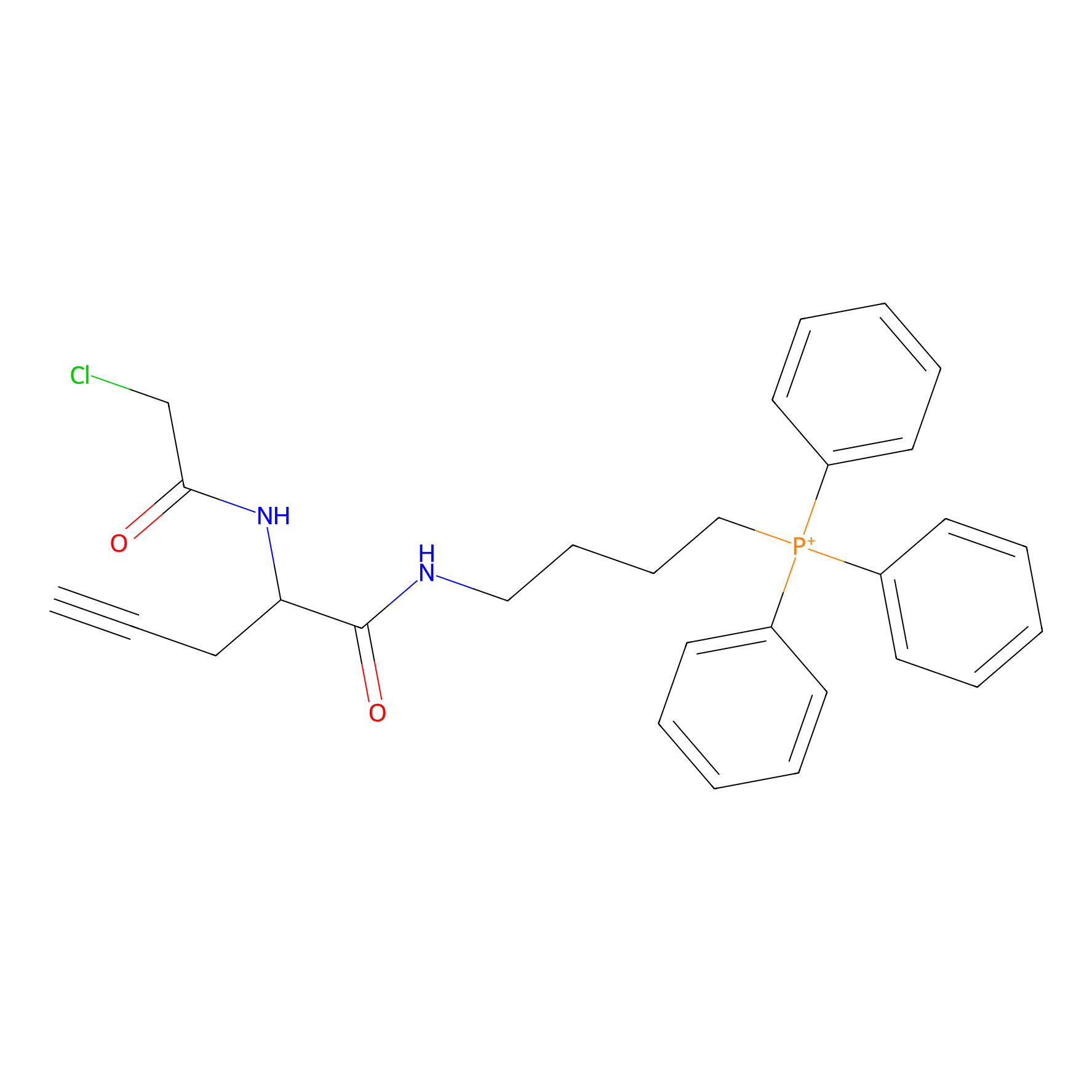 |
N.A. | LDD0427 | [22] | |
PAL-AfBPP Probe
Competitor(s) Related to This Target
| Competitor ID | Name | Cell line | Binding Site(Ratio) | Interaction ID | Ref |
|---|---|---|---|---|---|
| LDCM0558 | 2-Cyano-N-phenylacetamide | MDA-MB-231 | C258(1.44) | LDD2152 | [11] |
| LDCM0214 | AC1 | HEK-293T | C445(1.04); C242(1.00); C258(0.99) | LDD1507 | [31] |
| LDCM0215 | AC10 | HEK-293T | C445(0.89); C242(1.03) | LDD1508 | [31] |
| LDCM0216 | AC100 | HEK-293T | C242(0.85) | LDD0814 | [15] |
| LDCM0217 | AC101 | HEK-293T | C242(0.79) | LDD0815 | [15] |
| LDCM0218 | AC102 | HEK-293T | C242(0.73) | LDD0816 | [15] |
| LDCM0219 | AC103 | HEK-293T | C242(0.77) | LDD0817 | [15] |
| LDCM0220 | AC104 | HEK-293T | C242(0.93) | LDD0818 | [15] |
| LDCM0221 | AC105 | HEK-293T | C242(0.78) | LDD0819 | [15] |
| LDCM0222 | AC106 | HEK-293T | C242(0.96) | LDD0820 | [15] |
| LDCM0223 | AC107 | HEK-293T | C242(0.83) | LDD0821 | [15] |
| LDCM0224 | AC108 | HEK-293T | C242(1.17) | LDD0822 | [15] |
| LDCM0225 | AC109 | HEK-293T | C242(1.04) | LDD0823 | [15] |
| LDCM0226 | AC11 | HEK-293T | C445(0.87); C242(0.90); C258(1.02) | LDD1509 | [31] |
| LDCM0227 | AC110 | HEK-293T | C242(0.81) | LDD0825 | [15] |
| LDCM0228 | AC111 | HEK-293T | C242(0.81) | LDD0826 | [15] |
| LDCM0229 | AC112 | HEK-293T | C242(0.96) | LDD0827 | [15] |
| LDCM0230 | AC113 | HCT 116 | C242(0.92) | LDD0547 | [15] |
| LDCM0231 | AC114 | HCT 116 | C242(1.03) | LDD0548 | [15] |
| LDCM0232 | AC115 | HCT 116 | C242(1.20) | LDD0549 | [15] |
| LDCM0233 | AC116 | HCT 116 | C242(1.06) | LDD0550 | [15] |
| LDCM0234 | AC117 | HCT 116 | C242(1.21) | LDD0551 | [15] |
| LDCM0235 | AC118 | HCT 116 | C242(1.14) | LDD0552 | [15] |
| LDCM0236 | AC119 | HCT 116 | C242(0.95) | LDD0553 | [15] |
| LDCM0237 | AC12 | HEK-293T | C445(0.84); C242(1.04); C258(1.05) | LDD1510 | [31] |
| LDCM0238 | AC120 | HCT 116 | C242(1.08) | LDD0555 | [15] |
| LDCM0239 | AC121 | HCT 116 | C242(0.98) | LDD0556 | [15] |
| LDCM0240 | AC122 | HCT 116 | C242(1.22) | LDD0557 | [15] |
| LDCM0241 | AC123 | HCT 116 | C242(0.91) | LDD0558 | [15] |
| LDCM0242 | AC124 | HCT 116 | C242(1.15) | LDD0559 | [15] |
| LDCM0243 | AC125 | HCT 116 | C242(0.97) | LDD0560 | [15] |
| LDCM0244 | AC126 | HCT 116 | C242(0.96) | LDD0561 | [15] |
| LDCM0245 | AC127 | HCT 116 | C242(1.10) | LDD0562 | [15] |
| LDCM0246 | AC128 | HCT 116 | C242(1.20) | LDD0563 | [15] |
| LDCM0247 | AC129 | HCT 116 | C242(0.88) | LDD0564 | [15] |
| LDCM0249 | AC130 | HCT 116 | C242(0.99) | LDD0566 | [15] |
| LDCM0250 | AC131 | HCT 116 | C242(0.91) | LDD0567 | [15] |
| LDCM0251 | AC132 | HCT 116 | C242(0.78) | LDD0568 | [15] |
| LDCM0252 | AC133 | HCT 116 | C242(0.94) | LDD0569 | [15] |
| LDCM0253 | AC134 | HCT 116 | C242(1.02) | LDD0570 | [15] |
| LDCM0254 | AC135 | HCT 116 | C242(0.86) | LDD0571 | [15] |
| LDCM0255 | AC136 | HCT 116 | C242(1.10) | LDD0572 | [15] |
| LDCM0256 | AC137 | HCT 116 | C242(0.80) | LDD0573 | [15] |
| LDCM0257 | AC138 | HCT 116 | C242(1.12) | LDD0574 | [15] |
| LDCM0258 | AC139 | HCT 116 | C242(0.82) | LDD0575 | [15] |
| LDCM0259 | AC14 | HEK-293T | C445(1.09); C242(0.98); C258(1.06) | LDD1512 | [31] |
| LDCM0260 | AC140 | HCT 116 | C242(1.06) | LDD0577 | [15] |
| LDCM0261 | AC141 | HCT 116 | C242(1.12) | LDD0578 | [15] |
| LDCM0262 | AC142 | HCT 116 | C242(0.87) | LDD0579 | [15] |
| LDCM0263 | AC143 | HEK-293T | C242(1.16) | LDD0861 | [15] |
| LDCM0264 | AC144 | HEK-293T | C242(1.01) | LDD0862 | [15] |
| LDCM0265 | AC145 | HEK-293T | C242(0.84) | LDD0863 | [15] |
| LDCM0266 | AC146 | HEK-293T | C242(0.92) | LDD0864 | [15] |
| LDCM0267 | AC147 | HEK-293T | C242(0.93) | LDD0865 | [15] |
| LDCM0268 | AC148 | HEK-293T | C242(0.99) | LDD0866 | [15] |
| LDCM0269 | AC149 | HEK-293T | C242(1.03) | LDD0867 | [15] |
| LDCM0270 | AC15 | HEK-293T | C445(1.14); C242(1.09) | LDD1513 | [31] |
| LDCM0271 | AC150 | HEK-293T | C242(0.99) | LDD0869 | [15] |
| LDCM0272 | AC151 | HEK-293T | C242(0.92) | LDD0870 | [15] |
| LDCM0273 | AC152 | HEK-293T | C242(1.05) | LDD0871 | [15] |
| LDCM0274 | AC153 | HEK-293T | C242(0.93) | LDD0872 | [15] |
| LDCM0621 | AC154 | HEK-293T | C242(1.15) | LDD2162 | [15] |
| LDCM0622 | AC155 | HEK-293T | C242(0.96) | LDD2163 | [15] |
| LDCM0623 | AC156 | HEK-293T | C242(1.11) | LDD2164 | [15] |
| LDCM0624 | AC157 | HEK-293T | C242(0.90) | LDD2165 | [15] |
| LDCM0276 | AC17 | HEK-293T | C445(0.93); C242(0.98); C258(1.04) | LDD1515 | [31] |
| LDCM0277 | AC18 | HEK-293T | C445(0.91); C242(0.98) | LDD1516 | [31] |
| LDCM0278 | AC19 | HEK-293T | C445(0.86); C242(0.76); C258(0.97) | LDD1517 | [31] |
| LDCM0279 | AC2 | HEK-293T | C445(0.93); C242(1.04) | LDD1518 | [31] |
| LDCM0280 | AC20 | HEK-293T | C445(1.03); C242(1.09); C258(0.99) | LDD1519 | [31] |
| LDCM0281 | AC21 | HEK-293T | C445(1.11); C242(0.98); C258(1.19) | LDD1520 | [31] |
| LDCM0282 | AC22 | HEK-293T | C445(1.06); C242(0.97); C258(1.04) | LDD1521 | [31] |
| LDCM0283 | AC23 | HEK-293T | C445(1.09); C242(1.03) | LDD1522 | [31] |
| LDCM0284 | AC24 | HEK-293T | C445(1.05); C242(1.02); C258(1.02) | LDD1523 | [31] |
| LDCM0285 | AC25 | HCT 116 | C242(1.50) | LDD0602 | [15] |
| LDCM0286 | AC26 | HCT 116 | C242(1.37) | LDD0603 | [15] |
| LDCM0287 | AC27 | HCT 116 | C242(1.78) | LDD0604 | [15] |
| LDCM0288 | AC28 | HCT 116 | C242(1.50) | LDD0605 | [15] |
| LDCM0289 | AC29 | HCT 116 | C242(1.71) | LDD0606 | [15] |
| LDCM0290 | AC3 | HEK-293T | C445(0.88); C242(1.06); C258(1.11) | LDD1529 | [31] |
| LDCM0291 | AC30 | HCT 116 | C242(1.51) | LDD0608 | [15] |
| LDCM0292 | AC31 | HCT 116 | C242(1.14) | LDD0609 | [15] |
| LDCM0293 | AC32 | HCT 116 | C242(2.05) | LDD0610 | [15] |
| LDCM0294 | AC33 | HCT 116 | C242(1.96) | LDD0611 | [15] |
| LDCM0295 | AC34 | HCT 116 | C242(2.26) | LDD0612 | [15] |
| LDCM0296 | AC35 | HEK-293T | C445(0.92); C242(0.96); C258(1.08) | LDD1535 | [31] |
| LDCM0297 | AC36 | HEK-293T | C445(0.92); C242(1.06); C258(1.01) | LDD1536 | [31] |
| LDCM0298 | AC37 | HEK-293T | C445(0.92); C242(1.10); C258(1.09) | LDD1537 | [31] |
| LDCM0299 | AC38 | HEK-293T | C445(1.15); C242(1.13); C258(1.12) | LDD1538 | [31] |
| LDCM0300 | AC39 | HEK-293T | C445(0.89); C242(0.98) | LDD1539 | [31] |
| LDCM0301 | AC4 | HEK-293T | C445(1.01); C242(0.90); C258(0.84) | LDD1540 | [31] |
| LDCM0302 | AC40 | HEK-293T | C445(1.07); C242(1.00); C258(1.02) | LDD1541 | [31] |
| LDCM0303 | AC41 | HEK-293T | C445(0.96); C242(0.90); C258(0.91) | LDD1542 | [31] |
| LDCM0304 | AC42 | HEK-293T | C445(0.99); C242(0.89) | LDD1543 | [31] |
| LDCM0305 | AC43 | HEK-293T | C445(0.98); C242(1.72); C258(1.27) | LDD1544 | [31] |
| LDCM0306 | AC44 | HEK-293T | C445(0.85); C242(1.16); C258(1.08) | LDD1545 | [31] |
| LDCM0307 | AC45 | HEK-293T | C445(0.97); C242(1.07); C258(1.12) | LDD1546 | [31] |
| LDCM0308 | AC46 | HCT 116 | C258(0.94) | LDD0625 | [15] |
| LDCM0309 | AC47 | HCT 116 | C258(1.00) | LDD0626 | [15] |
| LDCM0310 | AC48 | HCT 116 | C258(1.10) | LDD0627 | [15] |
| LDCM0311 | AC49 | HCT 116 | C258(0.56) | LDD0628 | [15] |
| LDCM0312 | AC5 | HEK-293T | C445(1.01); C242(1.22); C258(0.96) | LDD1551 | [31] |
| LDCM0313 | AC50 | HCT 116 | C258(0.67) | LDD0630 | [15] |
| LDCM0314 | AC51 | HCT 116 | C258(0.94) | LDD0631 | [15] |
| LDCM0315 | AC52 | HCT 116 | C258(0.92) | LDD0632 | [15] |
| LDCM0316 | AC53 | HCT 116 | C258(0.78) | LDD0633 | [15] |
| LDCM0317 | AC54 | HCT 116 | C258(0.78) | LDD0634 | [15] |
| LDCM0318 | AC55 | HCT 116 | C258(0.60) | LDD0635 | [15] |
| LDCM0319 | AC56 | HCT 116 | C258(0.36) | LDD0636 | [15] |
| LDCM0320 | AC57 | PaTu 8988t | C242(1.31) | LDD1199 | [15] |
| LDCM0321 | AC58 | PaTu 8988t | C242(1.15) | LDD1200 | [15] |
| LDCM0322 | AC59 | PaTu 8988t | C242(1.14) | LDD1201 | [15] |
| LDCM0323 | AC6 | HEK-293T | C445(1.11); C242(0.95); C258(0.98) | LDD1562 | [31] |
| LDCM0324 | AC60 | PaTu 8988t | C242(1.18) | LDD1203 | [15] |
| LDCM0325 | AC61 | PaTu 8988t | C242(1.02) | LDD1204 | [15] |
| LDCM0326 | AC62 | PaTu 8988t | C242(1.32) | LDD1205 | [15] |
| LDCM0327 | AC63 | PaTu 8988t | C242(1.41) | LDD1206 | [15] |
| LDCM0328 | AC64 | PaTu 8988t | C242(1.24) | LDD1207 | [15] |
| LDCM0329 | AC65 | PaTu 8988t | C242(1.15) | LDD1208 | [15] |
| LDCM0330 | AC66 | PaTu 8988t | C242(1.19) | LDD1209 | [15] |
| LDCM0331 | AC67 | PaTu 8988t | C242(1.03) | LDD1210 | [15] |
| LDCM0332 | AC68 | HCT 116 | C258(0.93); C242(1.89) | LDD0649 | [15] |
| LDCM0333 | AC69 | HCT 116 | C258(1.12); C242(1.47) | LDD0650 | [15] |
| LDCM0334 | AC7 | HEK-293T | C445(1.03); C242(1.00) | LDD1568 | [31] |
| LDCM0335 | AC70 | HCT 116 | C258(0.95); C242(1.44) | LDD0652 | [15] |
| LDCM0336 | AC71 | HCT 116 | C242(1.04); C258(1.25) | LDD0653 | [15] |
| LDCM0337 | AC72 | HCT 116 | C258(1.09); C242(1.63) | LDD0654 | [15] |
| LDCM0338 | AC73 | HCT 116 | C258(0.73); C242(1.85) | LDD0655 | [15] |
| LDCM0339 | AC74 | HCT 116 | C258(0.77); C242(1.65) | LDD0656 | [15] |
| LDCM0340 | AC75 | HCT 116 | C258(0.48); C242(2.00) | LDD0657 | [15] |
| LDCM0341 | AC76 | HCT 116 | C258(1.02); C242(1.76) | LDD0658 | [15] |
| LDCM0342 | AC77 | HCT 116 | C258(0.95); C242(1.26) | LDD0659 | [15] |
| LDCM0343 | AC78 | HCT 116 | C258(1.21); C242(1.59) | LDD0660 | [15] |
| LDCM0344 | AC79 | HCT 116 | C258(1.19); C242(1.23) | LDD0661 | [15] |
| LDCM0345 | AC8 | HEK-293T | C445(1.04); C242(0.94); C258(1.10) | LDD1569 | [31] |
| LDCM0346 | AC80 | HCT 116 | C258(1.23); C242(2.05) | LDD0663 | [15] |
| LDCM0347 | AC81 | HCT 116 | C242(0.76); C258(1.08) | LDD0664 | [15] |
| LDCM0348 | AC82 | HCT 116 | C258(0.33); C242(2.89) | LDD0665 | [15] |
| LDCM0349 | AC83 | HCT 116 | C258(0.66) | LDD0666 | [15] |
| LDCM0350 | AC84 | HCT 116 | C258(0.79) | LDD0667 | [15] |
| LDCM0351 | AC85 | HCT 116 | C258(1.10) | LDD0668 | [15] |
| LDCM0352 | AC86 | HCT 116 | C258(1.11) | LDD0669 | [15] |
| LDCM0353 | AC87 | HCT 116 | C258(0.99) | LDD0670 | [15] |
| LDCM0354 | AC88 | HCT 116 | C258(0.86) | LDD0671 | [15] |
| LDCM0355 | AC89 | HCT 116 | C258(0.78) | LDD0672 | [15] |
| LDCM0357 | AC90 | HCT 116 | C258(0.79) | LDD0674 | [15] |
| LDCM0358 | AC91 | HCT 116 | C258(0.66) | LDD0675 | [15] |
| LDCM0359 | AC92 | HCT 116 | C258(0.72) | LDD0676 | [15] |
| LDCM0360 | AC93 | HCT 116 | C258(0.80) | LDD0677 | [15] |
| LDCM0361 | AC94 | HCT 116 | C258(0.98) | LDD0678 | [15] |
| LDCM0362 | AC95 | HCT 116 | C258(0.98) | LDD0679 | [15] |
| LDCM0363 | AC96 | HCT 116 | C258(0.87) | LDD0680 | [15] |
| LDCM0364 | AC97 | HCT 116 | C258(0.83) | LDD0681 | [15] |
| LDCM0365 | AC98 | HEK-293T | C242(0.97) | LDD0963 | [15] |
| LDCM0366 | AC99 | HEK-293T | C242(1.12) | LDD0964 | [15] |
| LDCM0520 | AKOS000195272 | MDA-MB-231 | C258(0.81) | LDD2113 | [11] |
| LDCM0248 | AKOS034007472 | HEK-293T | C445(1.03); C242(1.01); C258(1.05) | LDD1511 | [31] |
| LDCM0356 | AKOS034007680 | HEK-293T | C445(0.95); C242(0.98); C258(0.99) | LDD1570 | [31] |
| LDCM0275 | AKOS034007705 | HEK-293T | C445(1.03); C242(0.99); C258(0.99) | LDD1514 | [31] |
| LDCM0147 | AZ0108 | MDA-MB-468 | 2.03 | LDD0376 | [24] |
| LDCM0108 | Chloroacetamide | HeLa | C258(0.00); H153(0.00) | LDD0222 | [20] |
| LDCM0367 | CL1 | HCT 116 | C258(1.05) | LDD0684 | [15] |
| LDCM0368 | CL10 | HCT 116 | C258(0.89) | LDD0685 | [15] |
| LDCM0369 | CL100 | HEK-293T | C445(0.98); C242(0.89); C258(0.94) | LDD1573 | [31] |
| LDCM0370 | CL101 | HEK-293T | C445(0.71); C242(1.00); C258(1.00) | LDD1574 | [31] |
| LDCM0371 | CL102 | HEK-293T | C445(0.93); C242(0.97) | LDD1575 | [31] |
| LDCM0372 | CL103 | HEK-293T | C445(0.90); C242(1.03) | LDD1576 | [31] |
| LDCM0373 | CL104 | HEK-293T | C445(0.93); C242(1.00); C258(1.00) | LDD1577 | [31] |
| LDCM0374 | CL105 | HEK-293T | C445(0.80); C242(0.92); C258(1.05) | LDD1578 | [31] |
| LDCM0375 | CL106 | HEK-293T | C445(0.97); C242(0.97) | LDD1579 | [31] |
| LDCM0376 | CL107 | HEK-293T | C445(1.00); C242(1.02) | LDD1580 | [31] |
| LDCM0377 | CL108 | HEK-293T | C445(0.95); C242(0.99); C258(0.92) | LDD1581 | [31] |
| LDCM0378 | CL109 | HEK-293T | C445(1.00); C242(0.96); C258(1.01) | LDD1582 | [31] |
| LDCM0379 | CL11 | HCT 116 | C258(0.69) | LDD0696 | [15] |
| LDCM0380 | CL110 | HEK-293T | C445(1.06); C242(1.88) | LDD1584 | [31] |
| LDCM0381 | CL111 | HEK-293T | C445(1.01); C242(1.16) | LDD1585 | [31] |
| LDCM0382 | CL112 | HCT 116 | C242(1.54) | LDD0699 | [15] |
| LDCM0383 | CL113 | HCT 116 | C242(1.61) | LDD0700 | [15] |
| LDCM0384 | CL114 | HCT 116 | C242(1.72) | LDD0701 | [15] |
| LDCM0385 | CL115 | HCT 116 | C242(1.61) | LDD0702 | [15] |
| LDCM0386 | CL116 | HCT 116 | C242(1.37) | LDD0703 | [15] |
| LDCM0387 | CL117 | HEK-293T | C445(0.88); C242(0.95); C258(0.97) | LDD1591 | [31] |
| LDCM0388 | CL118 | HEK-293T | C445(1.00); C242(0.94) | LDD1592 | [31] |
| LDCM0389 | CL119 | HEK-293T | C445(1.07); C242(0.95) | LDD1593 | [31] |
| LDCM0390 | CL12 | HCT 116 | C258(0.69) | LDD0707 | [15] |
| LDCM0391 | CL120 | HEK-293T | C445(1.03); C242(1.00); C258(0.91) | LDD1595 | [31] |
| LDCM0392 | CL121 | HCT 116 | C258(0.80) | LDD0709 | [15] |
| LDCM0393 | CL122 | HCT 116 | C258(1.14) | LDD0710 | [15] |
| LDCM0394 | CL123 | HCT 116 | C258(0.78) | LDD0711 | [15] |
| LDCM0395 | CL124 | HCT 116 | C258(0.76) | LDD0712 | [15] |
| LDCM0396 | CL125 | PaTu 8988t | C242(1.08) | LDD1275 | [15] |
| LDCM0397 | CL126 | PaTu 8988t | C242(1.33) | LDD1276 | [15] |
| LDCM0398 | CL127 | PaTu 8988t | C242(1.06) | LDD1277 | [15] |
| LDCM0399 | CL128 | PaTu 8988t | C242(1.04) | LDD1278 | [15] |
| LDCM0400 | CL13 | HCT 116 | C258(0.94) | LDD0717 | [15] |
| LDCM0401 | CL14 | HCT 116 | C258(1.04) | LDD0718 | [15] |
| LDCM0402 | CL15 | HCT 116 | C258(0.49) | LDD0719 | [15] |
| LDCM0403 | CL16 | HEK-293T | C445(0.90); C242(0.94); C258(0.95) | LDD1607 | [31] |
| LDCM0404 | CL17 | HEK-293T | C445(0.91); C242(1.08); C258(0.95) | LDD1608 | [31] |
| LDCM0405 | CL18 | HEK-293T | C445(0.86); C242(0.97) | LDD1609 | [31] |
| LDCM0406 | CL19 | HEK-293T | C445(0.95); C242(1.14); C258(1.12) | LDD1610 | [31] |
| LDCM0407 | CL2 | HCT 116 | C258(1.00) | LDD0724 | [15] |
| LDCM0408 | CL20 | HEK-293T | C445(0.91); C242(0.99); C258(1.13) | LDD1612 | [31] |
| LDCM0409 | CL21 | HEK-293T | C445(1.14); C242(1.05); C258(1.03) | LDD1613 | [31] |
| LDCM0410 | CL22 | HEK-293T | C445(1.28); C242(1.15); C258(1.13) | LDD1614 | [31] |
| LDCM0411 | CL23 | HEK-293T | C445(1.17); C242(1.15) | LDD1615 | [31] |
| LDCM0412 | CL24 | HEK-293T | C445(1.19); C242(0.94); C258(1.05) | LDD1616 | [31] |
| LDCM0413 | CL25 | HEK-293T | C445(0.70); C242(1.13); C258(1.05) | LDD1617 | [31] |
| LDCM0414 | CL26 | HEK-293T | C445(0.98); C242(0.96) | LDD1618 | [31] |
| LDCM0415 | CL27 | HEK-293T | C445(1.11); C242(0.97) | LDD1619 | [31] |
| LDCM0416 | CL28 | HEK-293T | C445(1.01); C242(1.02); C258(1.12) | LDD1620 | [31] |
| LDCM0417 | CL29 | HEK-293T | C445(1.00); C242(0.94); C258(1.04) | LDD1621 | [31] |
| LDCM0418 | CL3 | HCT 116 | C258(1.04) | LDD0735 | [15] |
| LDCM0419 | CL30 | HEK-293T | C445(1.00); C242(1.03) | LDD1623 | [31] |
| LDCM0420 | CL31 | HEK-293T | C242(1.05) | LDD1018 | [15] |
| LDCM0421 | CL32 | HEK-293T | C242(1.02) | LDD1019 | [15] |
| LDCM0422 | CL33 | HEK-293T | C242(1.08) | LDD1020 | [15] |
| LDCM0423 | CL34 | HEK-293T | C242(1.10) | LDD1021 | [15] |
| LDCM0424 | CL35 | HEK-293T | C242(1.04) | LDD1022 | [15] |
| LDCM0425 | CL36 | HEK-293T | C242(0.86) | LDD1023 | [15] |
| LDCM0426 | CL37 | HEK-293T | C242(1.18) | LDD1024 | [15] |
| LDCM0428 | CL39 | HEK-293T | C242(0.95) | LDD1026 | [15] |
| LDCM0429 | CL4 | HCT 116 | C258(1.12) | LDD0746 | [15] |
| LDCM0430 | CL40 | HEK-293T | C242(1.00) | LDD1028 | [15] |
| LDCM0431 | CL41 | HEK-293T | C242(0.89) | LDD1029 | [15] |
| LDCM0432 | CL42 | HEK-293T | C242(0.88) | LDD1030 | [15] |
| LDCM0433 | CL43 | HEK-293T | C242(0.97) | LDD1031 | [15] |
| LDCM0434 | CL44 | HEK-293T | C242(1.07) | LDD1032 | [15] |
| LDCM0435 | CL45 | HEK-293T | C242(0.96) | LDD1033 | [15] |
| LDCM0436 | CL46 | HCT 116 | C242(0.97) | LDD0753 | [15] |
| LDCM0437 | CL47 | HCT 116 | C242(0.77) | LDD0754 | [15] |
| LDCM0438 | CL48 | HCT 116 | C242(0.96) | LDD0755 | [15] |
| LDCM0439 | CL49 | HCT 116 | C242(1.11) | LDD0756 | [15] |
| LDCM0440 | CL5 | HCT 116 | C258(0.99) | LDD0757 | [15] |
| LDCM0441 | CL50 | HCT 116 | C242(1.02) | LDD0758 | [15] |
| LDCM0442 | CL51 | HCT 116 | C242(1.09) | LDD0759 | [15] |
| LDCM0443 | CL52 | HCT 116 | C242(0.99) | LDD0760 | [15] |
| LDCM0444 | CL53 | HCT 116 | C242(1.32) | LDD0761 | [15] |
| LDCM0445 | CL54 | HCT 116 | C242(0.98) | LDD0762 | [15] |
| LDCM0446 | CL55 | HCT 116 | C242(0.92) | LDD0763 | [15] |
| LDCM0447 | CL56 | HCT 116 | C242(1.16) | LDD0764 | [15] |
| LDCM0448 | CL57 | HCT 116 | C242(0.86) | LDD0765 | [15] |
| LDCM0449 | CL58 | HCT 116 | C242(0.82) | LDD0766 | [15] |
| LDCM0450 | CL59 | HCT 116 | C242(1.12) | LDD0767 | [15] |
| LDCM0451 | CL6 | HCT 116 | C258(1.08) | LDD0768 | [15] |
| LDCM0452 | CL60 | HCT 116 | C242(0.86) | LDD0769 | [15] |
| LDCM0453 | CL61 | PaTu 8988t | C242(0.72) | LDD1332 | [15] |
| LDCM0454 | CL62 | PaTu 8988t | C242(0.75) | LDD1333 | [15] |
| LDCM0455 | CL63 | PaTu 8988t | C242(0.86) | LDD1334 | [15] |
| LDCM0456 | CL64 | PaTu 8988t | C242(0.79) | LDD1335 | [15] |
| LDCM0457 | CL65 | PaTu 8988t | C242(0.49) | LDD1336 | [15] |
| LDCM0458 | CL66 | PaTu 8988t | C242(1.19) | LDD1337 | [15] |
| LDCM0459 | CL67 | PaTu 8988t | C242(1.03) | LDD1338 | [15] |
| LDCM0460 | CL68 | PaTu 8988t | C242(1.03) | LDD1339 | [15] |
| LDCM0461 | CL69 | PaTu 8988t | C242(1.16) | LDD1340 | [15] |
| LDCM0462 | CL7 | HCT 116 | C258(0.97) | LDD0779 | [15] |
| LDCM0463 | CL70 | PaTu 8988t | C242(1.32) | LDD1342 | [15] |
| LDCM0464 | CL71 | PaTu 8988t | C242(0.64) | LDD1343 | [15] |
| LDCM0465 | CL72 | PaTu 8988t | C242(0.59) | LDD1344 | [15] |
| LDCM0466 | CL73 | PaTu 8988t | C242(1.99) | LDD1345 | [15] |
| LDCM0467 | CL74 | PaTu 8988t | C242(0.56) | LDD1346 | [15] |
| LDCM0469 | CL76 | PaTu 8988t | C242(1.23) | LDD1348 | [15] |
| LDCM0470 | CL77 | PaTu 8988t | C242(1.20) | LDD1349 | [15] |
| LDCM0471 | CL78 | PaTu 8988t | C242(1.15) | LDD1350 | [15] |
| LDCM0472 | CL79 | PaTu 8988t | C242(1.00) | LDD1351 | [15] |
| LDCM0473 | CL8 | HCT 116 | C258(0.66) | LDD0790 | [15] |
| LDCM0474 | CL80 | PaTu 8988t | C242(1.09) | LDD1353 | [15] |
| LDCM0475 | CL81 | PaTu 8988t | C242(1.17) | LDD1354 | [15] |
| LDCM0476 | CL82 | PaTu 8988t | C242(1.18) | LDD1355 | [15] |
| LDCM0477 | CL83 | PaTu 8988t | C242(1.47) | LDD1356 | [15] |
| LDCM0478 | CL84 | PaTu 8988t | C242(1.53) | LDD1357 | [15] |
| LDCM0479 | CL85 | PaTu 8988t | C242(0.99) | LDD1358 | [15] |
| LDCM0480 | CL86 | PaTu 8988t | C242(1.14) | LDD1359 | [15] |
| LDCM0481 | CL87 | PaTu 8988t | C242(1.45) | LDD1360 | [15] |
| LDCM0482 | CL88 | PaTu 8988t | C242(1.34) | LDD1361 | [15] |
| LDCM0483 | CL89 | PaTu 8988t | C242(1.44) | LDD1362 | [15] |
| LDCM0484 | CL9 | HCT 116 | C258(0.90) | LDD0801 | [15] |
| LDCM0485 | CL90 | PaTu 8988t | C242(1.34) | LDD1364 | [15] |
| LDCM0486 | CL91 | HEK-293T | C445(0.94); C242(1.03); C258(1.15) | LDD1689 | [31] |
| LDCM0487 | CL92 | HEK-293T | C445(0.82); C242(0.94); C258(1.27) | LDD1690 | [31] |
| LDCM0488 | CL93 | HEK-293T | C445(1.35); C242(1.18); C258(1.31) | LDD1691 | [31] |
| LDCM0489 | CL94 | HEK-293T | C445(1.22); C242(1.04); C258(1.21) | LDD1692 | [31] |
| LDCM0490 | CL95 | HEK-293T | C445(1.08); C242(1.09) | LDD1693 | [31] |
| LDCM0491 | CL96 | HEK-293T | C445(1.09); C242(0.95); C258(1.06) | LDD1694 | [31] |
| LDCM0492 | CL97 | HEK-293T | C445(0.85); C242(0.96); C258(1.05) | LDD1695 | [31] |
| LDCM0493 | CL98 | HEK-293T | C445(0.97); C242(0.94) | LDD1696 | [31] |
| LDCM0494 | CL99 | HEK-293T | C445(0.96); C242(0.95) | LDD1697 | [31] |
| LDCM0033 | Curcusone1d | MCF-7 | 2.19 | LDD0188 | [12] |
| LDCM0495 | E2913 | HEK-293T | C445(0.97); C242(0.97) | LDD1698 | [31] |
| LDCM0213 | Electrophilic fragment 2 | MDA-MB-231 | C445(8.25); C258(3.96) | LDD1702 | [11] |
| LDCM0082 | FK866 | A-549 | 2.58 | LDD0142 | [28] |
| LDCM0468 | Fragment33 | PaTu 8988t | C242(0.79) | LDD1347 | [15] |
| LDCM0427 | Fragment51 | HEK-293T | C242(1.16) | LDD1025 | [15] |
| LDCM0107 | IAA | HeLa | C258(0.00); H153(0.00) | LDD0221 | [20] |
| LDCM0022 | KB02 | HEK-293T | C445(0.83); C242(1.12); C258(1.09) | LDD1492 | [31] |
| LDCM0023 | KB03 | HEK-293T | C445(0.83); C242(1.04); C258(1.00) | LDD1497 | [31] |
| LDCM0024 | KB05 | G361 | C445(2.01) | LDD3311 | [32] |
| LDCM0109 | NEM | HeLa | H172(0.00); C258(0.00) | LDD0223 | [20] |
| LDCM0500 | Nucleophilic fragment 13a | MDA-MB-231 | C258(1.01) | LDD2093 | [11] |
| LDCM0504 | Nucleophilic fragment 15a | MDA-MB-231 | C258(0.91) | LDD2097 | [11] |
| LDCM0506 | Nucleophilic fragment 16a | MDA-MB-231 | C258(0.99) | LDD2099 | [11] |
| LDCM0518 | Nucleophilic fragment 22a | MDA-MB-231 | C258(1.05) | LDD2111 | [11] |
| LDCM0522 | Nucleophilic fragment 24a | MDA-MB-231 | C258(0.57) | LDD2115 | [11] |
| LDCM0532 | Nucleophilic fragment 29a | MDA-MB-231 | C258(0.69) | LDD2125 | [11] |
| LDCM0533 | Nucleophilic fragment 29b | MDA-MB-231 | C258(0.10) | LDD2126 | [11] |
| LDCM0534 | Nucleophilic fragment 30a | MDA-MB-231 | C258(0.73) | LDD2127 | [11] |
| LDCM0536 | Nucleophilic fragment 31 | MDA-MB-231 | C258(1.12) | LDD2129 | [11] |
| LDCM0540 | Nucleophilic fragment 35 | MDA-MB-231 | C258(0.56) | LDD2133 | [11] |
| LDCM0541 | Nucleophilic fragment 36 | MDA-MB-231 | C258(0.84) | LDD2134 | [11] |
| LDCM0542 | Nucleophilic fragment 37 | MDA-MB-231 | C258(1.31) | LDD2135 | [11] |
| LDCM0544 | Nucleophilic fragment 39 | MDA-MB-231 | C258(0.84) | LDD2137 | [11] |
| LDCM0547 | Nucleophilic fragment 41 | MDA-MB-231 | C258(0.62) | LDD2141 | [11] |
| LDCM0551 | Nucleophilic fragment 5b | MDA-MB-231 | C258(0.18) | LDD2145 | [11] |
| LDCM0552 | Nucleophilic fragment 6a | MDA-MB-231 | C258(0.82) | LDD2146 | [11] |
| LDCM0148 | Olaparib | MDA-MB-468 | 1.88 | LDD0377 | [24] |
| LDCM0014 | Panhematin | K562 | 3.00 | LDD0063 | [13] |
| LDCM0099 | Phenelzine | MDA-MB-231 | 7.69 | LDD0283 | [10] |
| LDCM0131 | RA190 | MM1.R | 3.43 | LDD0299 | [14] |
| LDCM0084 | Ro 48-8071 | A-549 | 2.46 | LDD0144 | [28] |
The Interaction Atlas With This Target
References