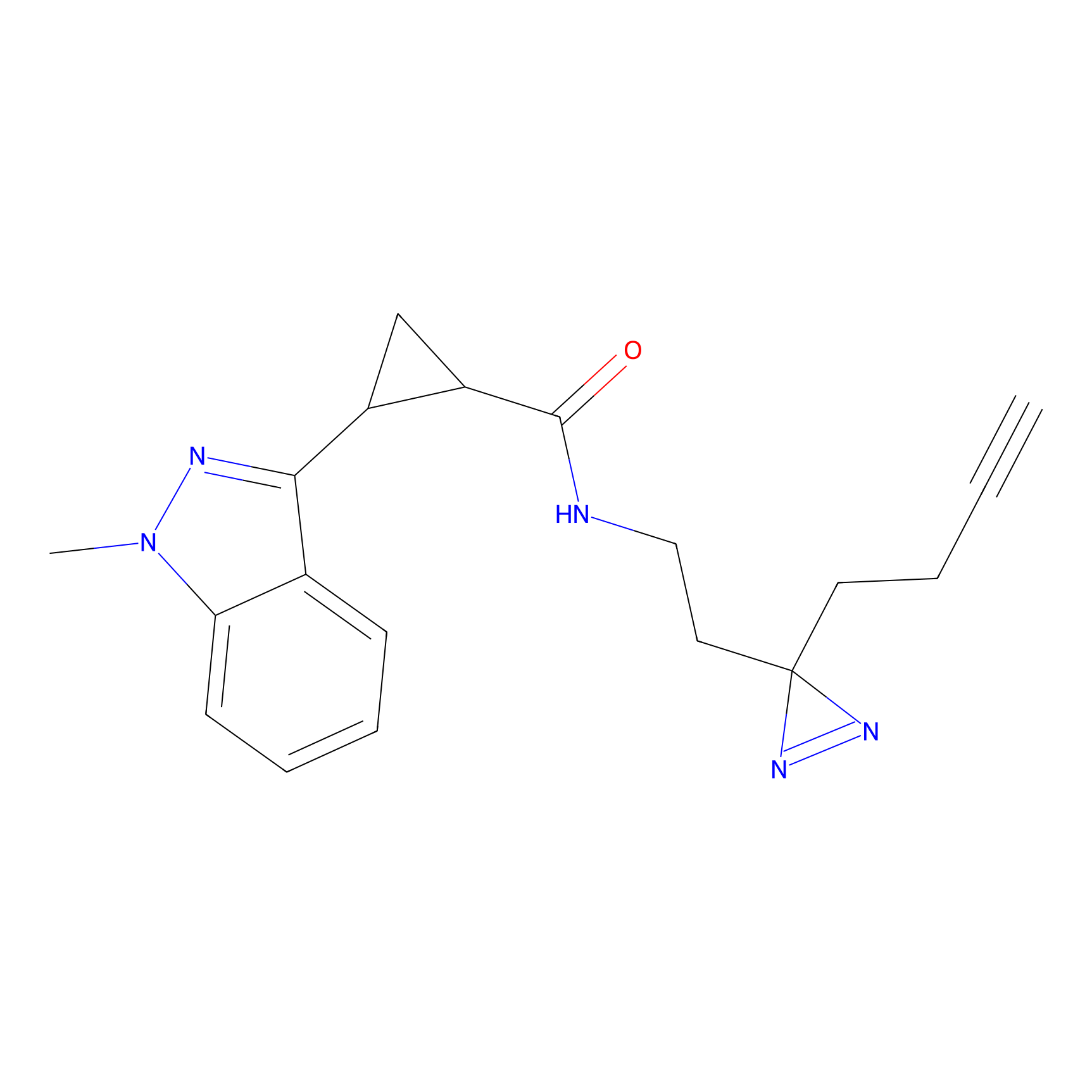
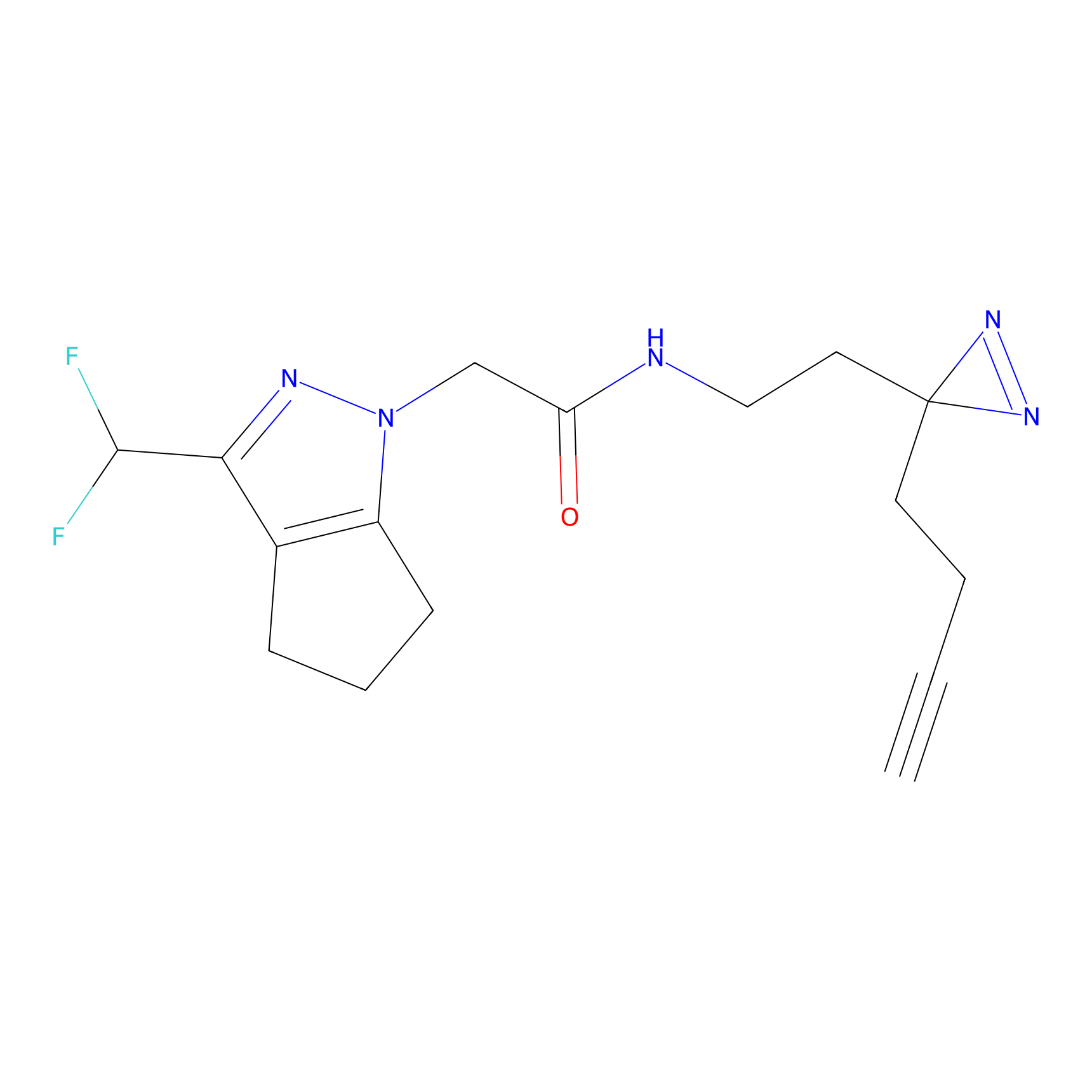
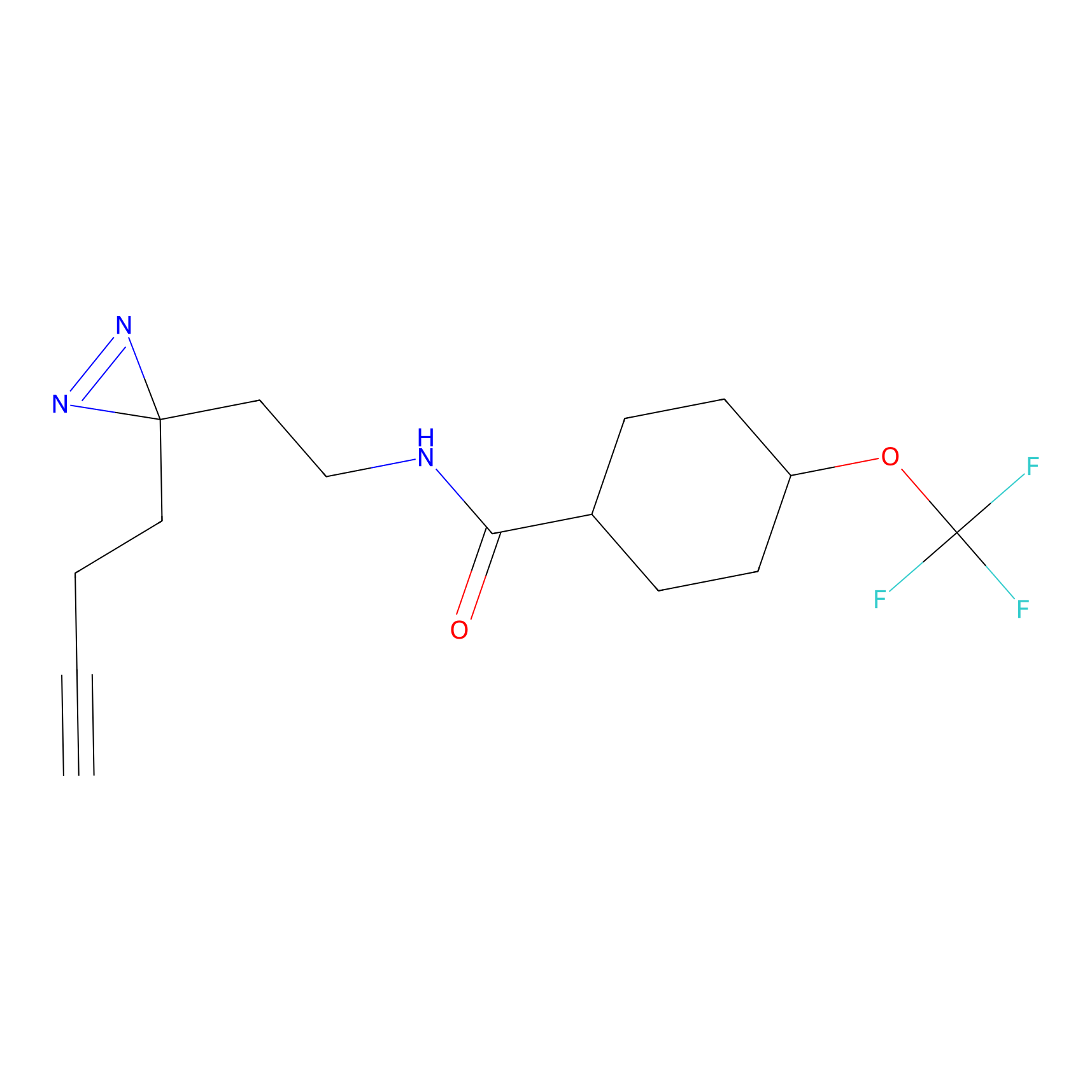
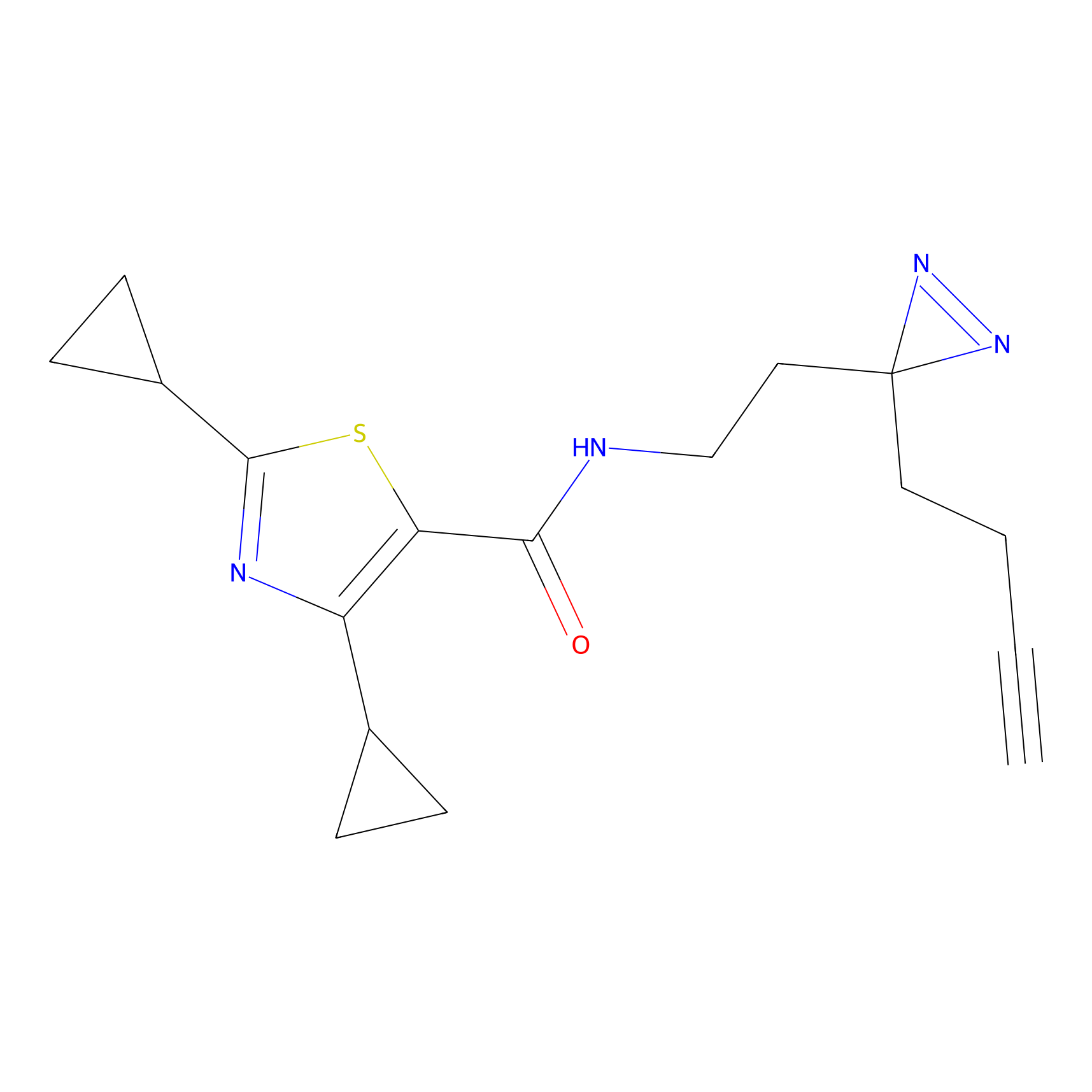
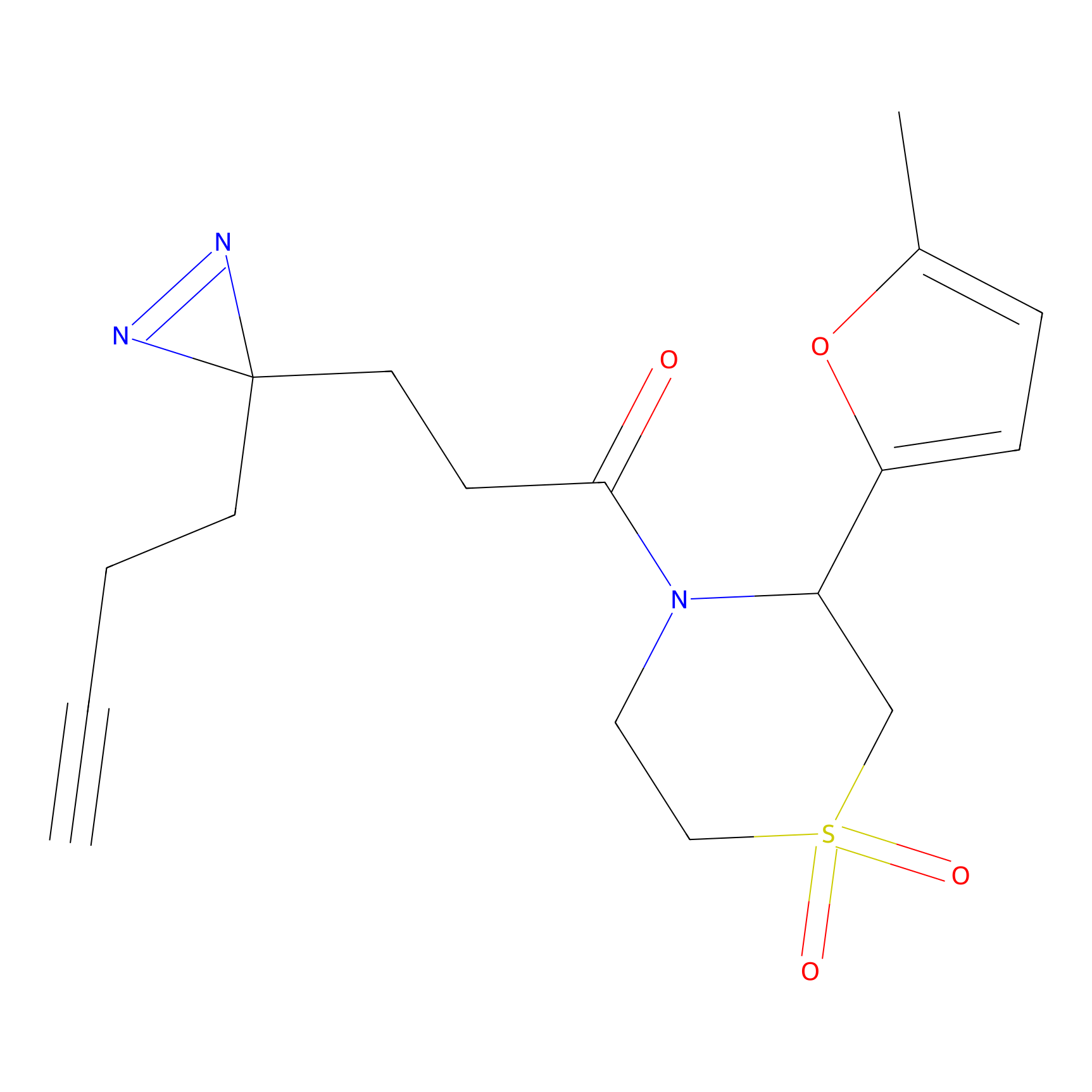
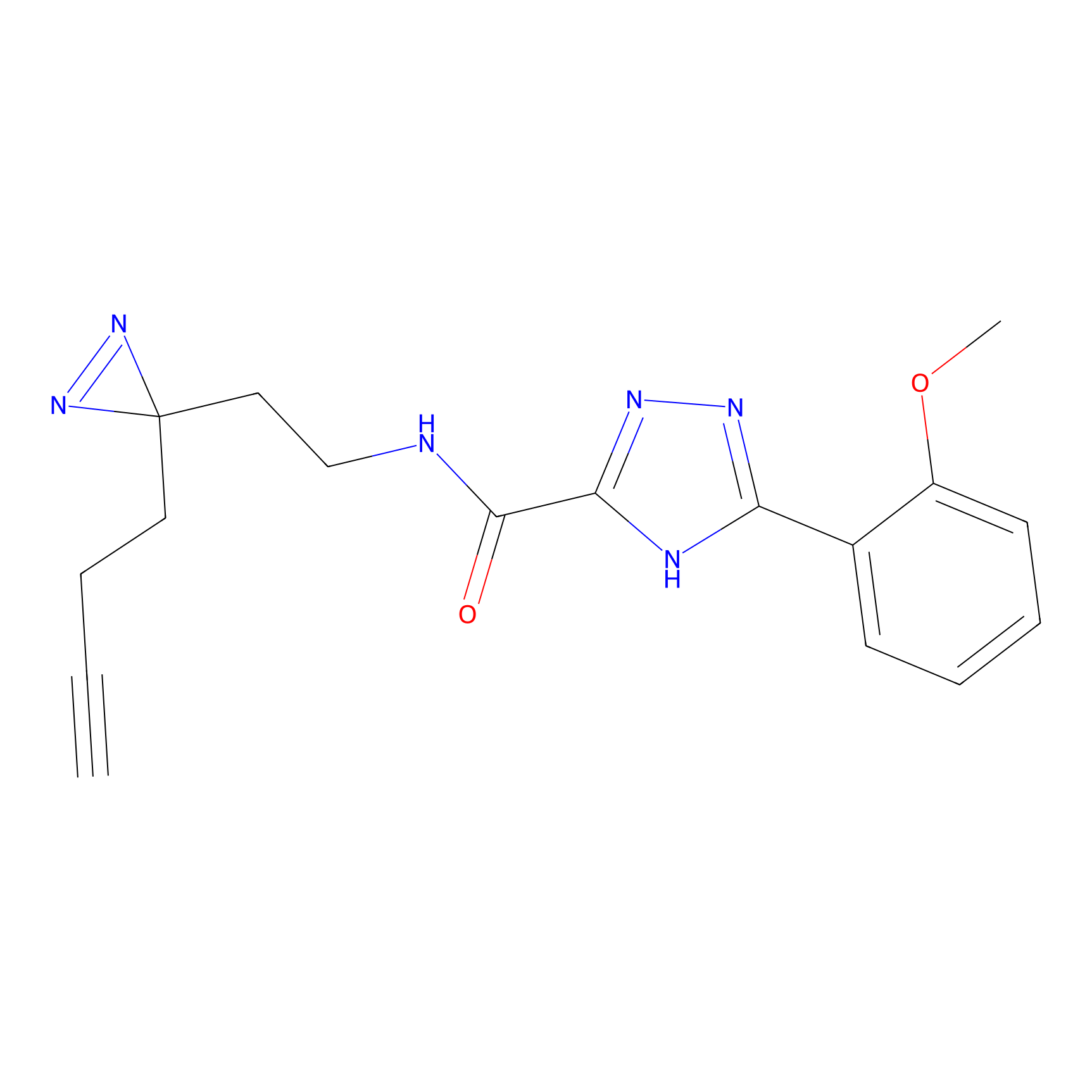
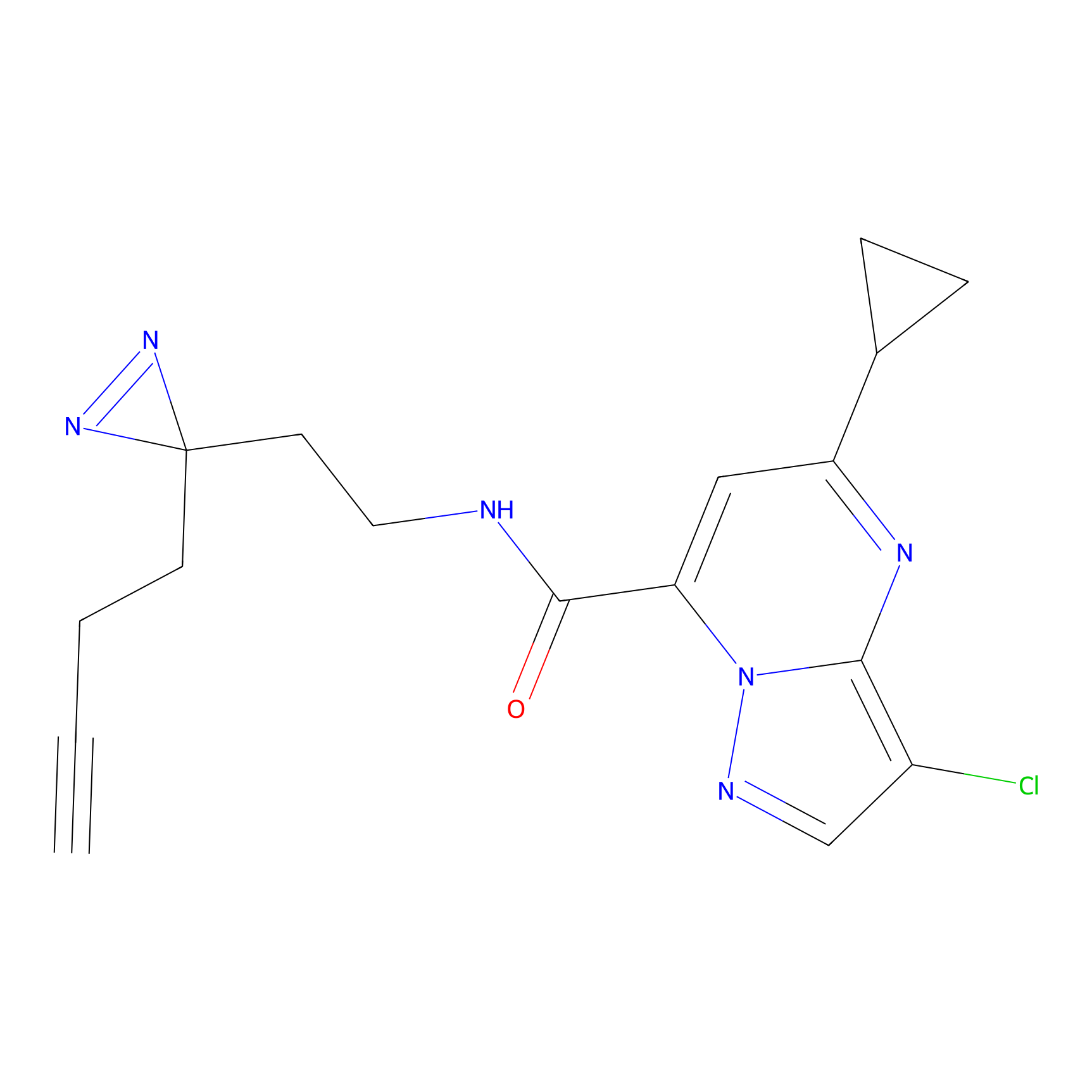
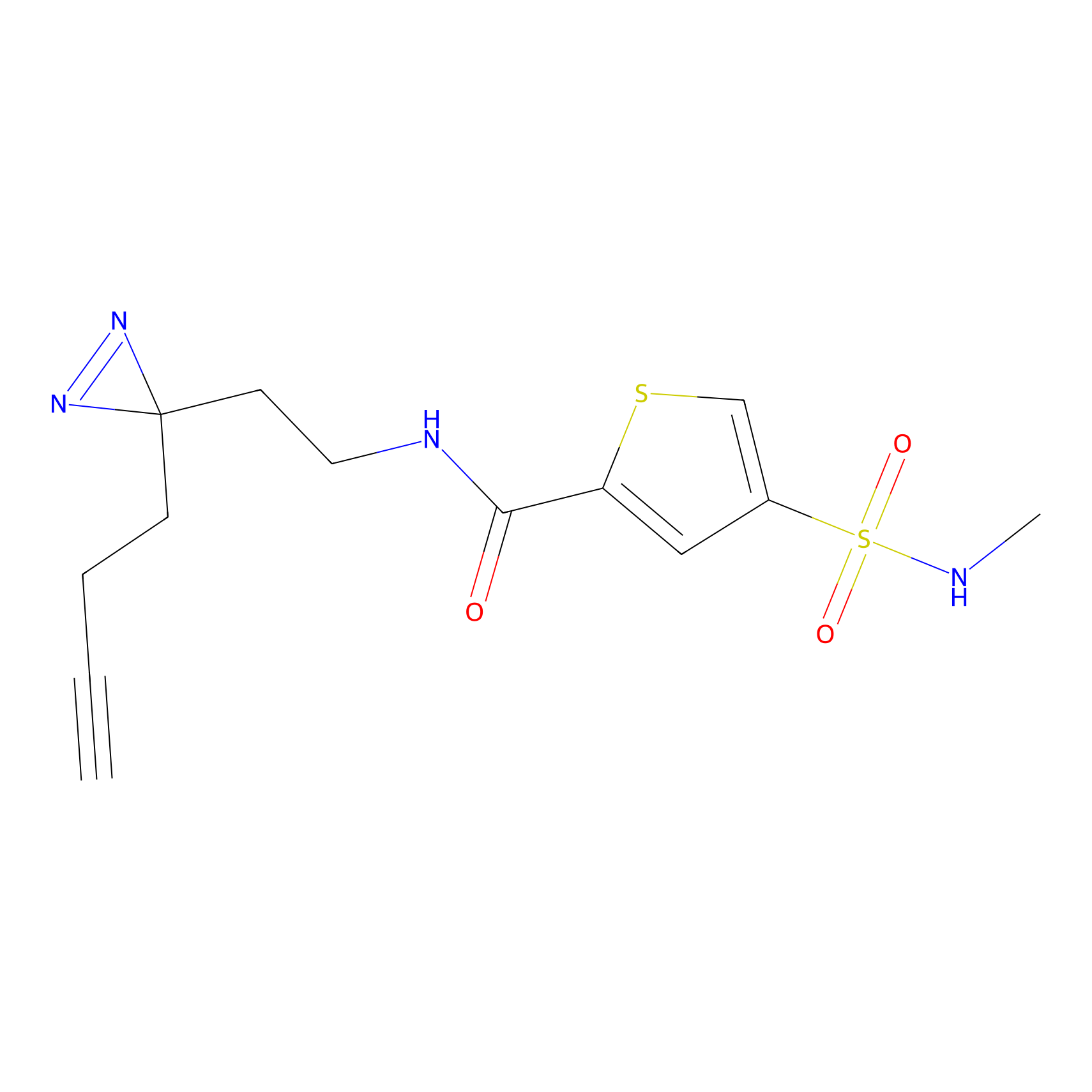
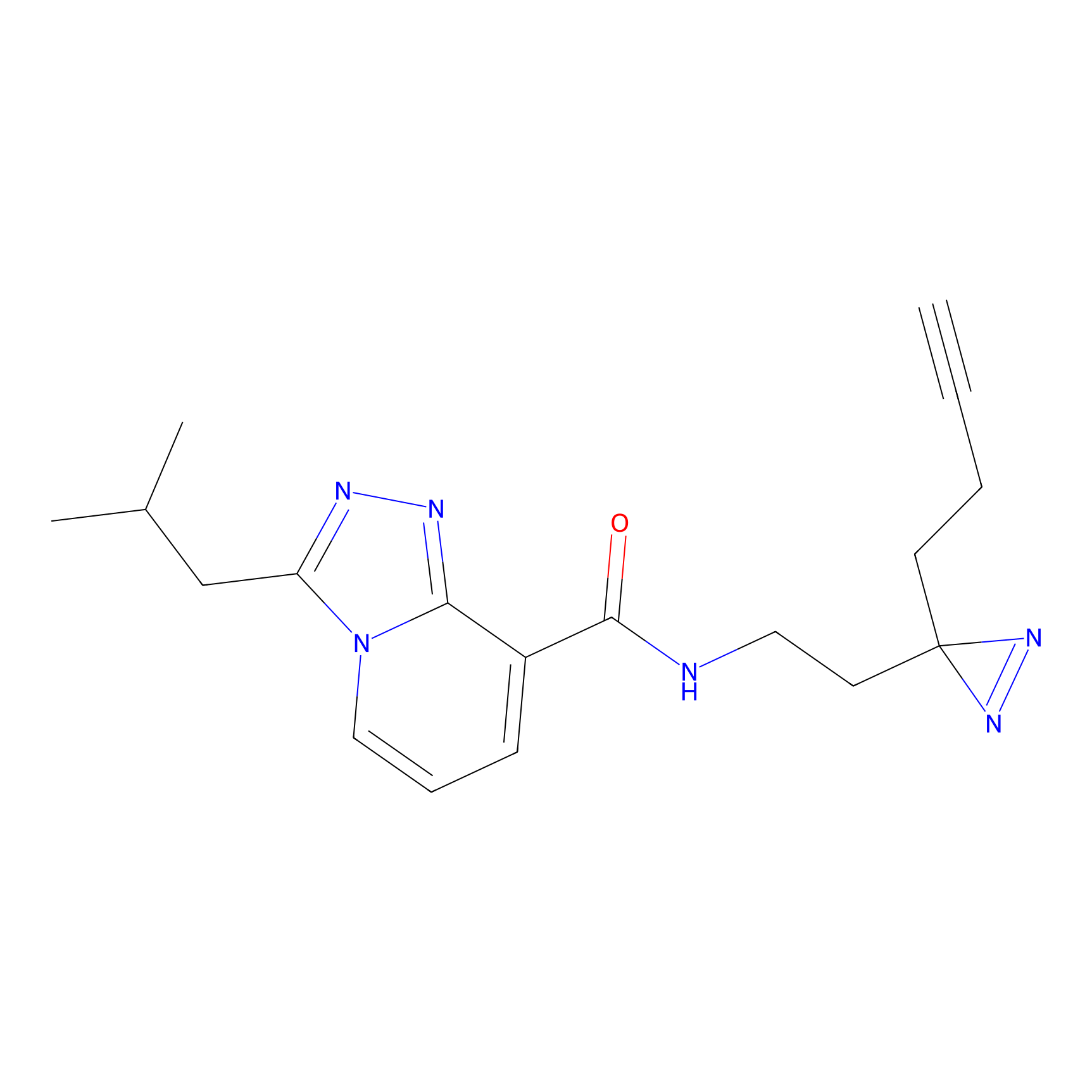
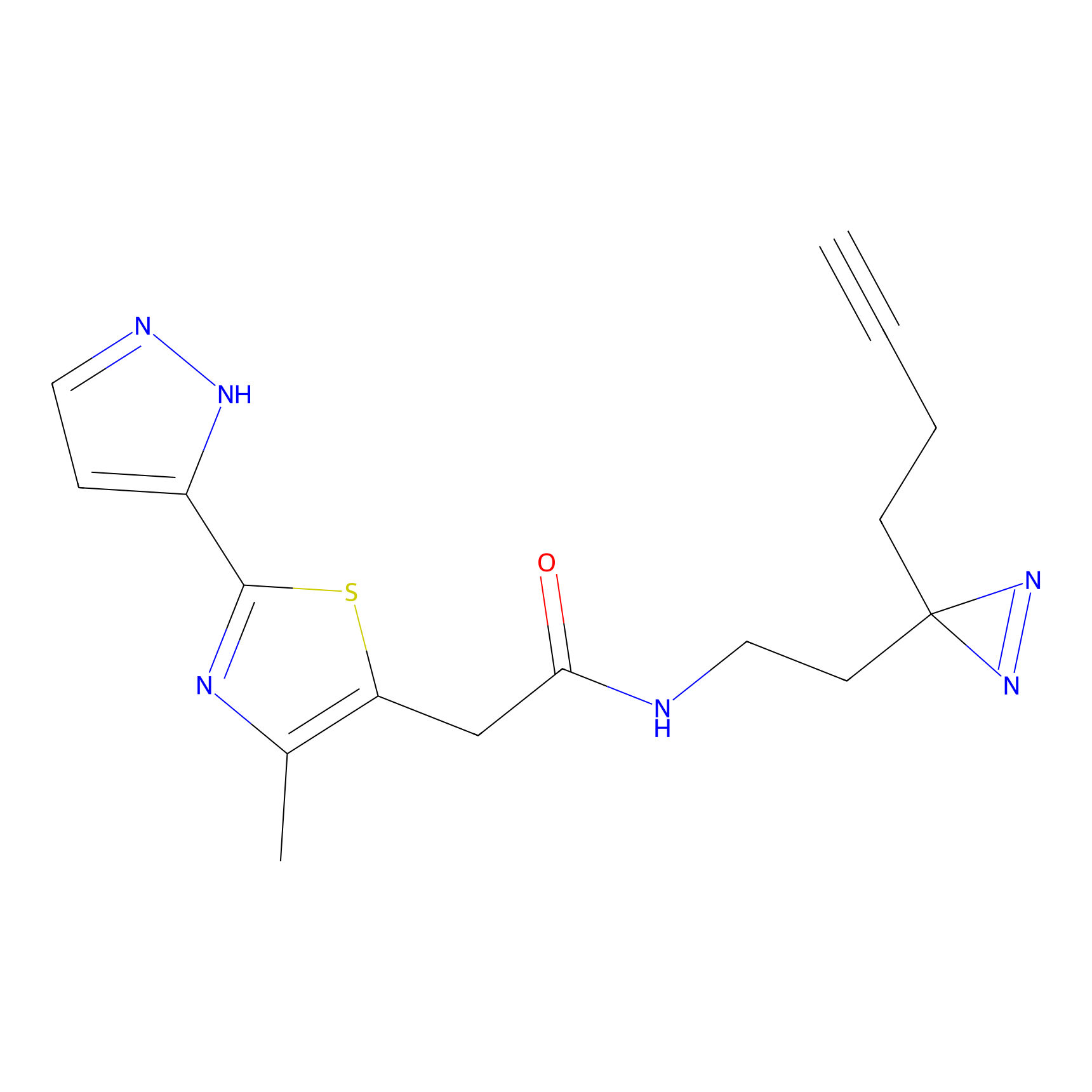
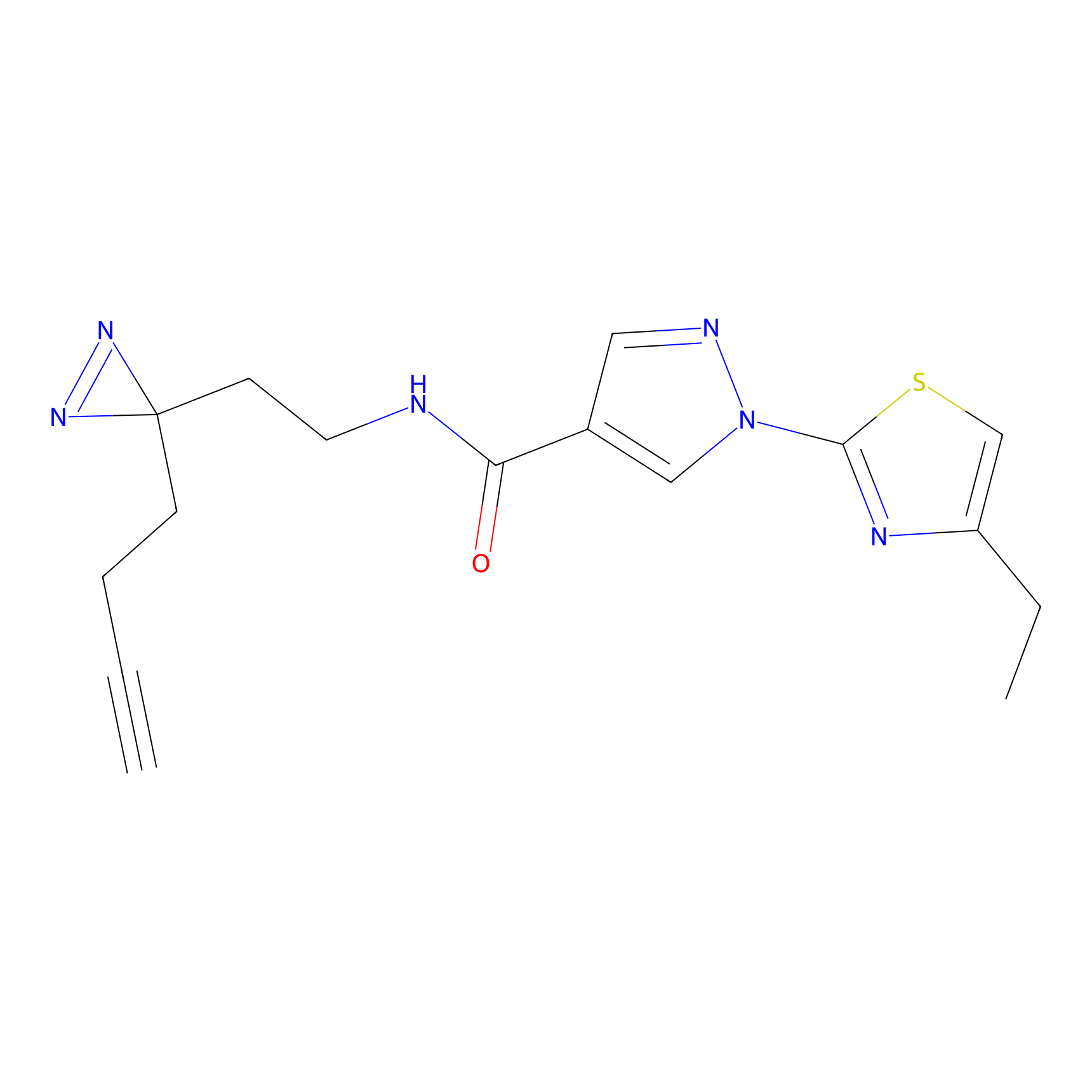
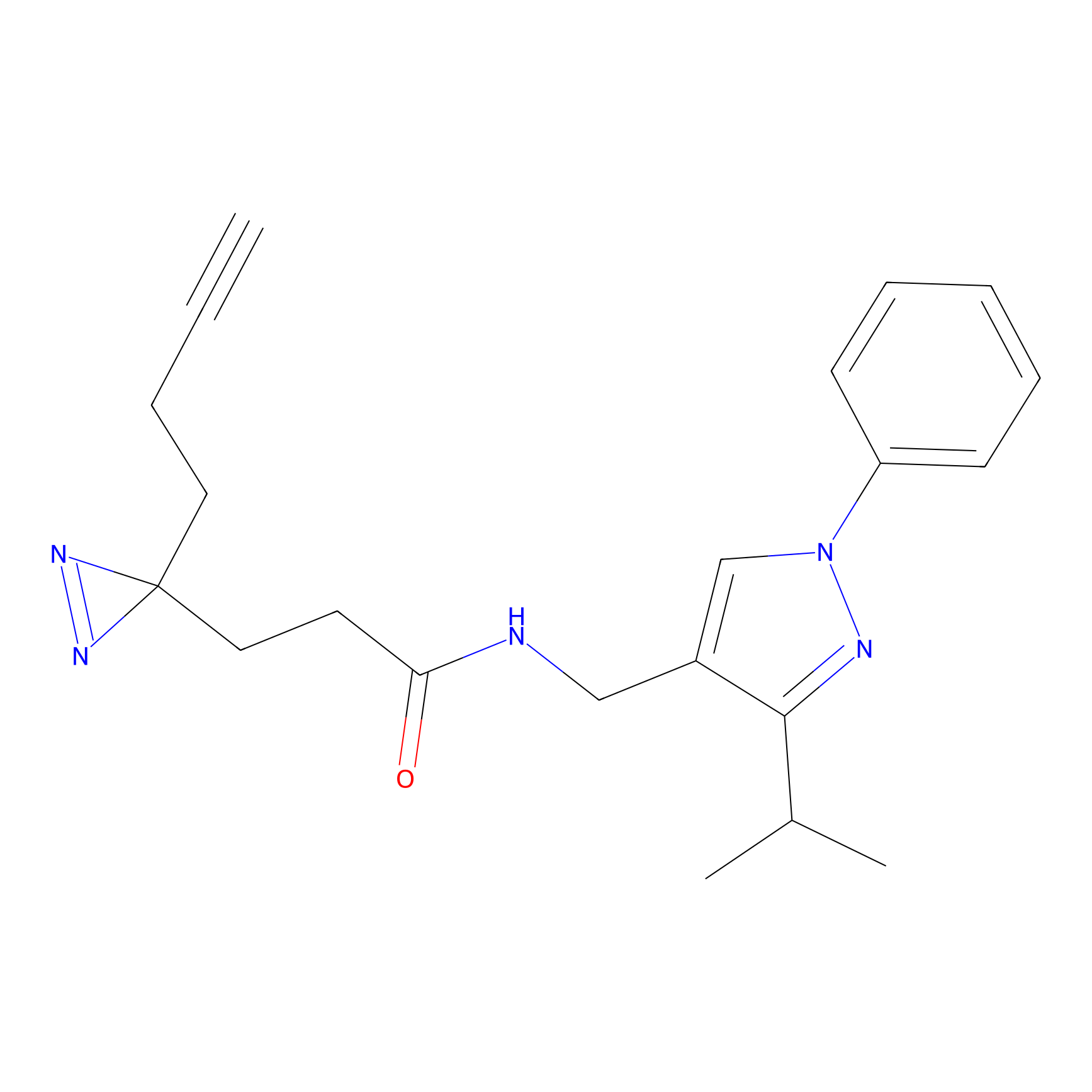
Details of the Target
General Information of Target
Target Site Mutations in Different Cell Lines
Probe(s) Labeling This Target
ABPP Probe
| Probe name | Structure | Binding Site(Ratio) | Interaction ID | Ref | |
|---|---|---|---|---|---|
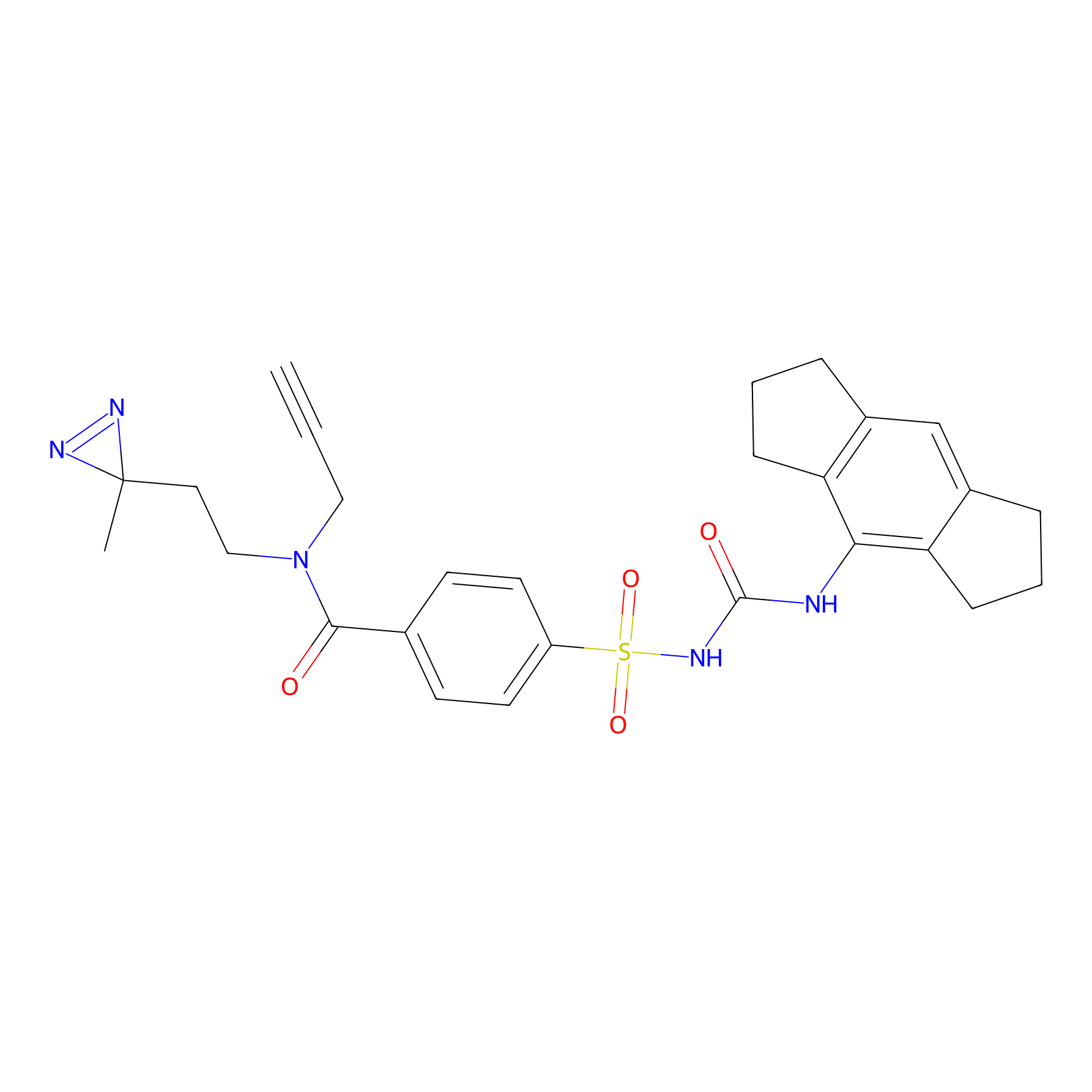
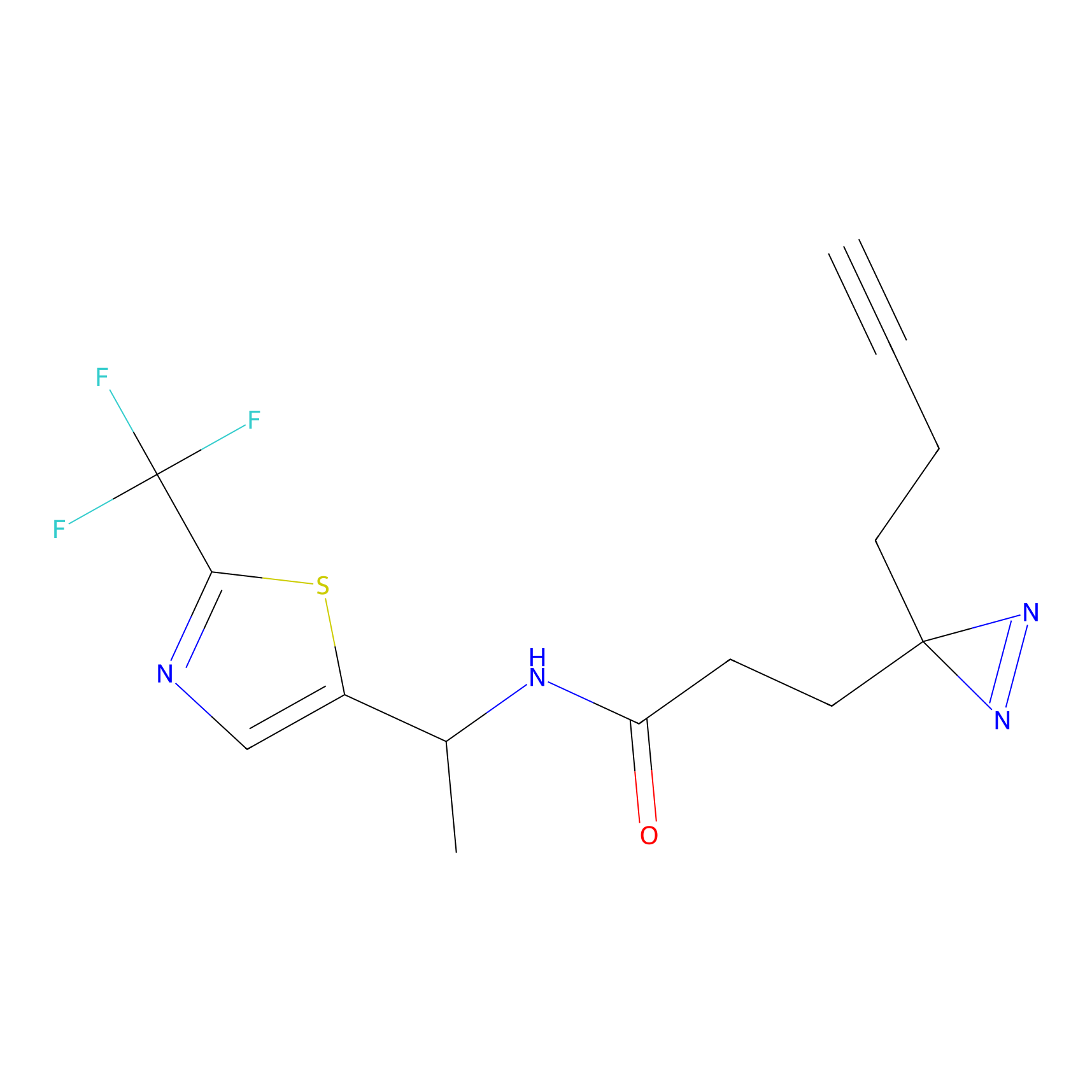
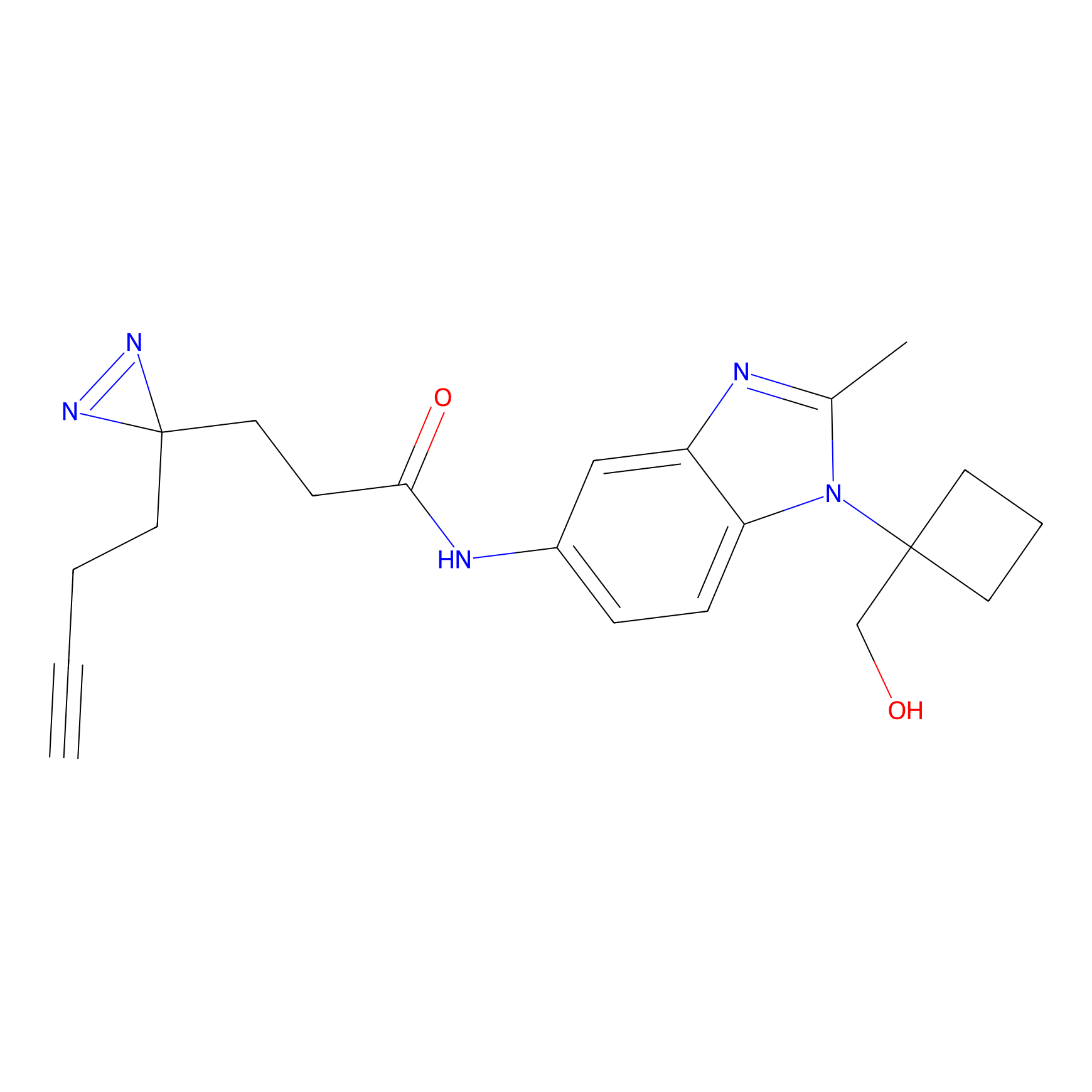
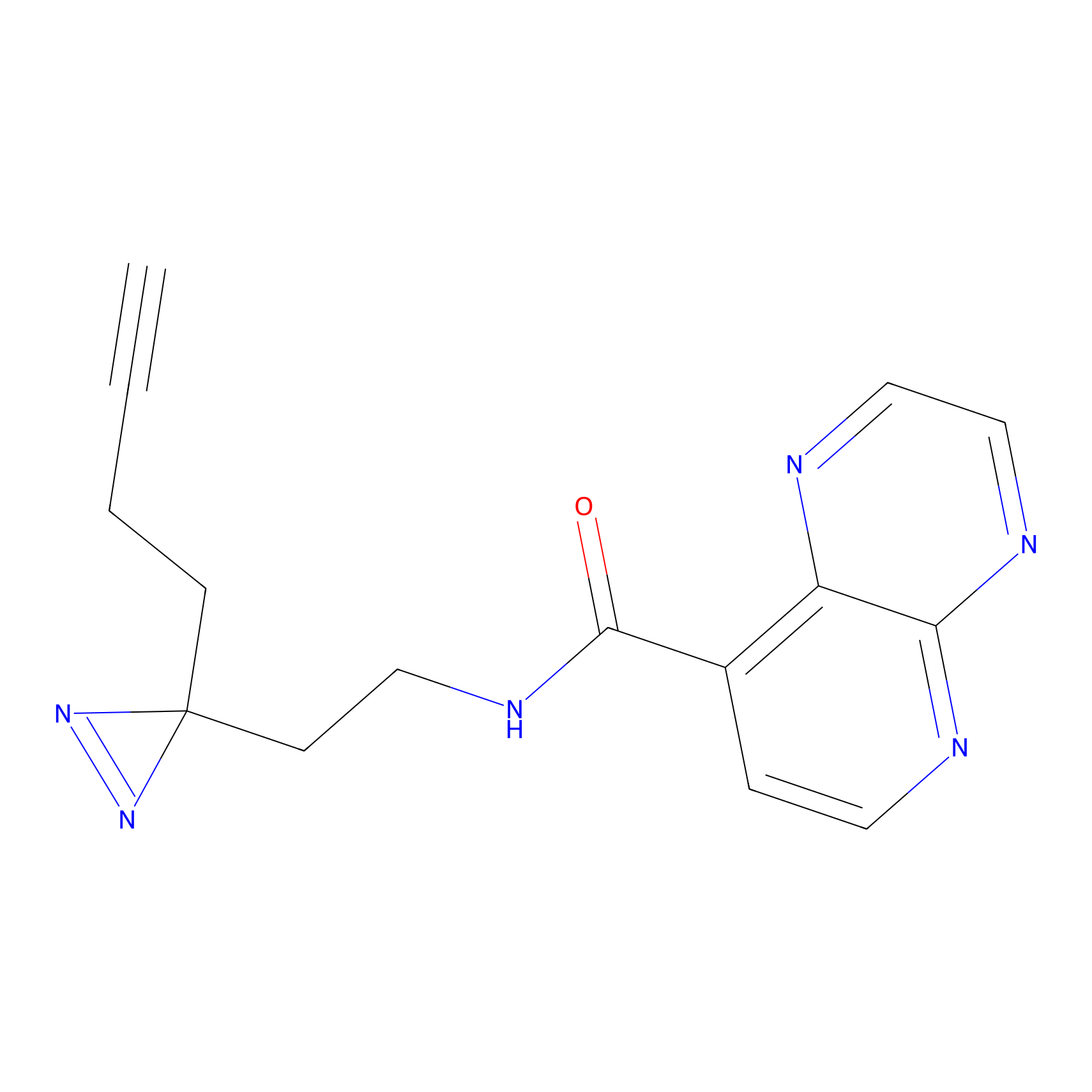
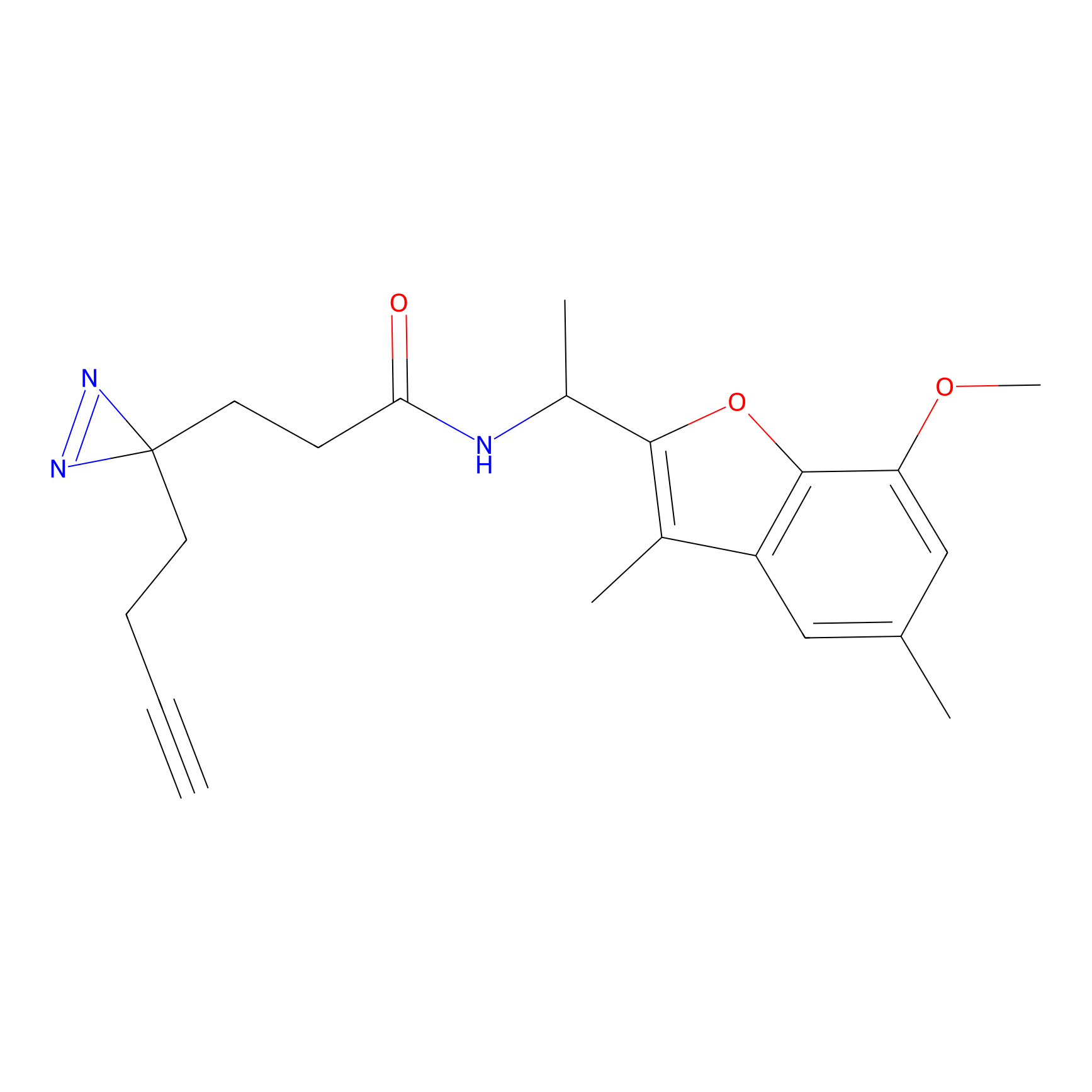
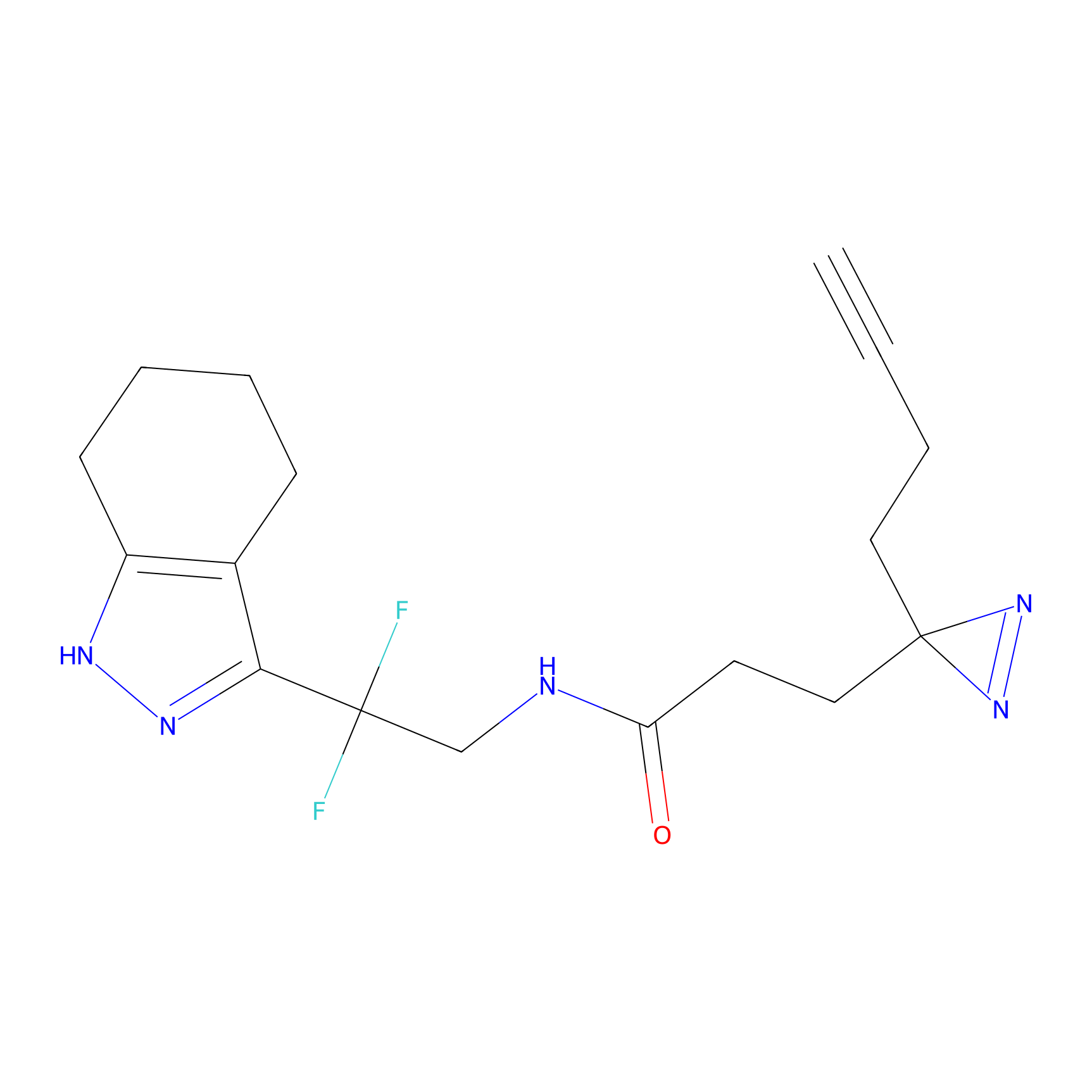
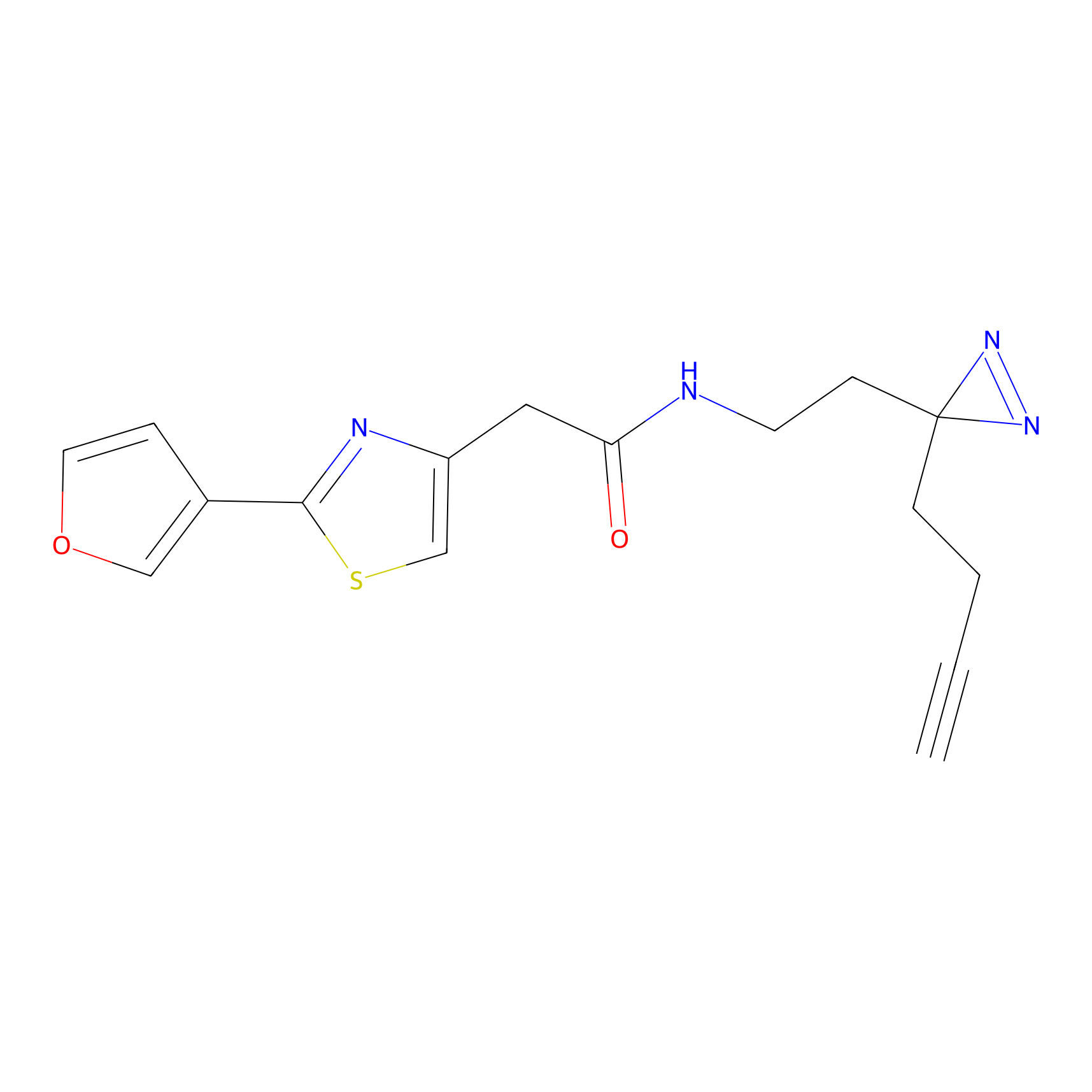
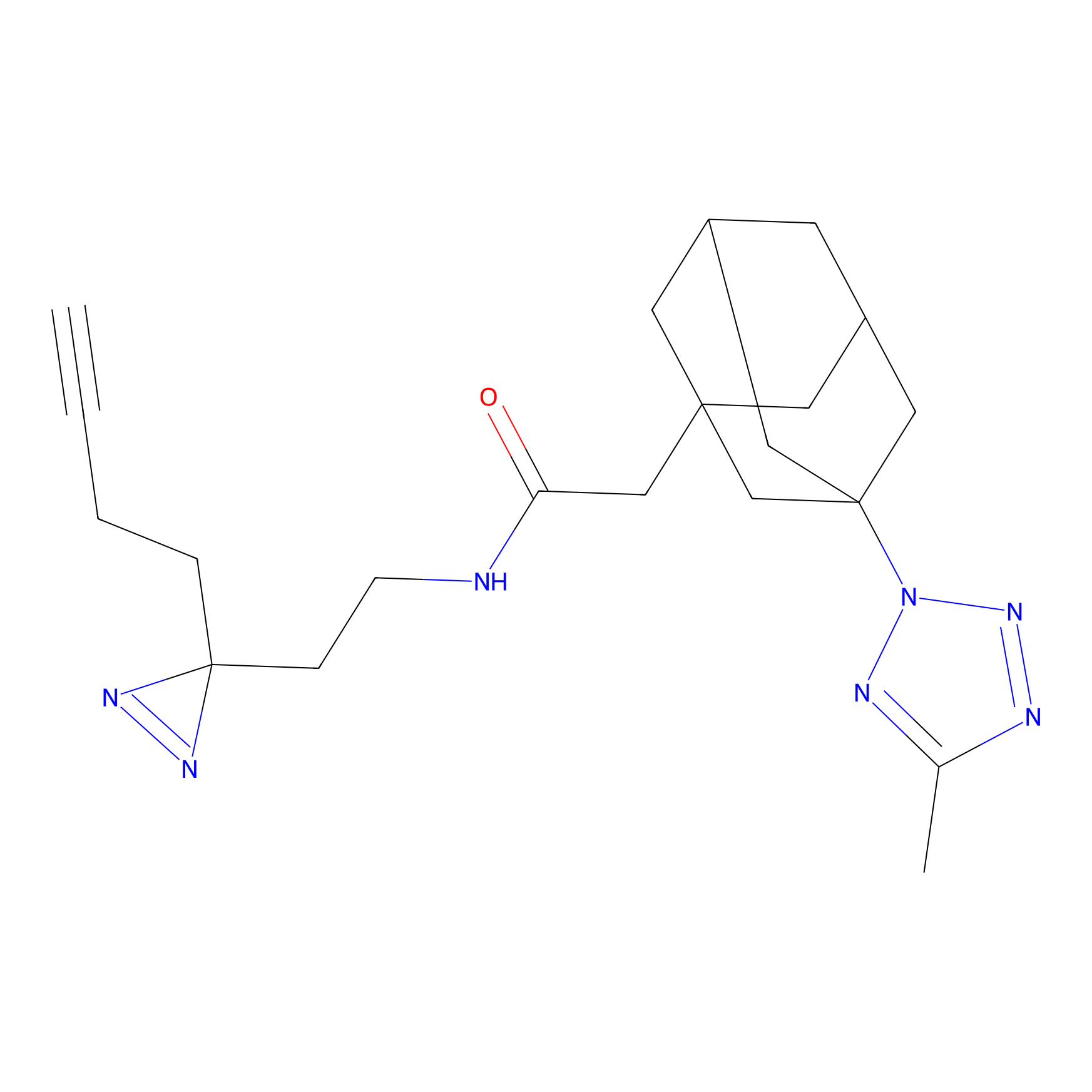
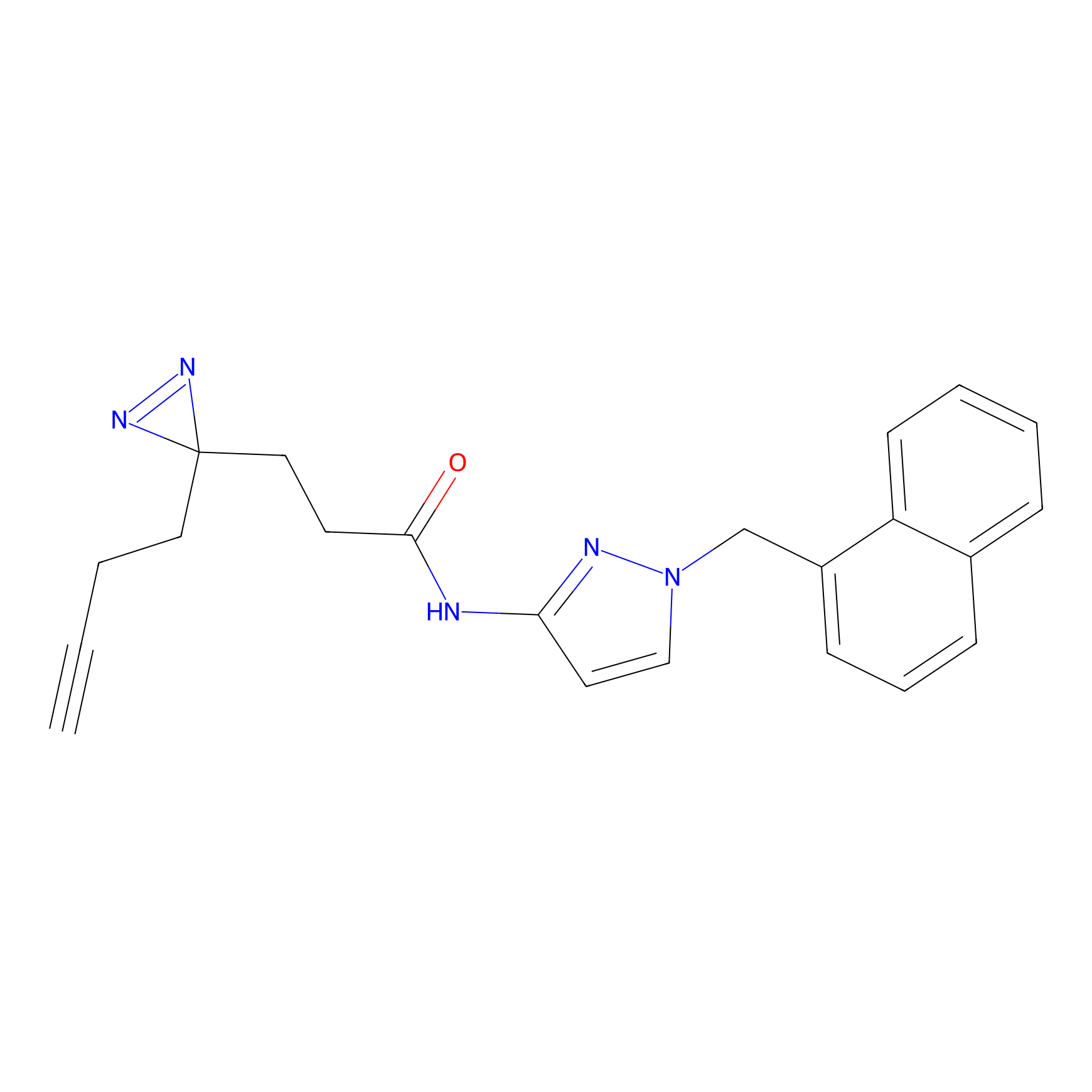
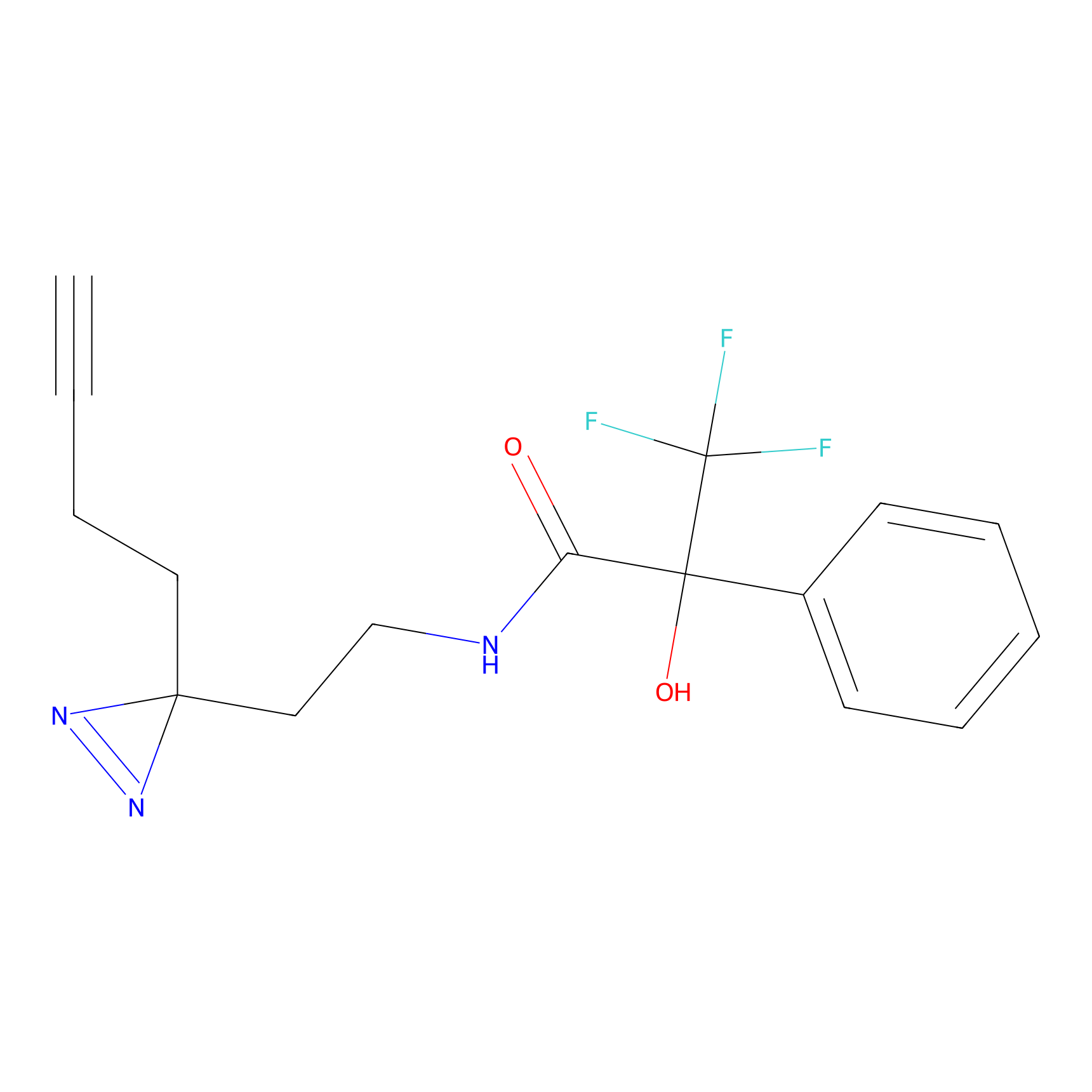
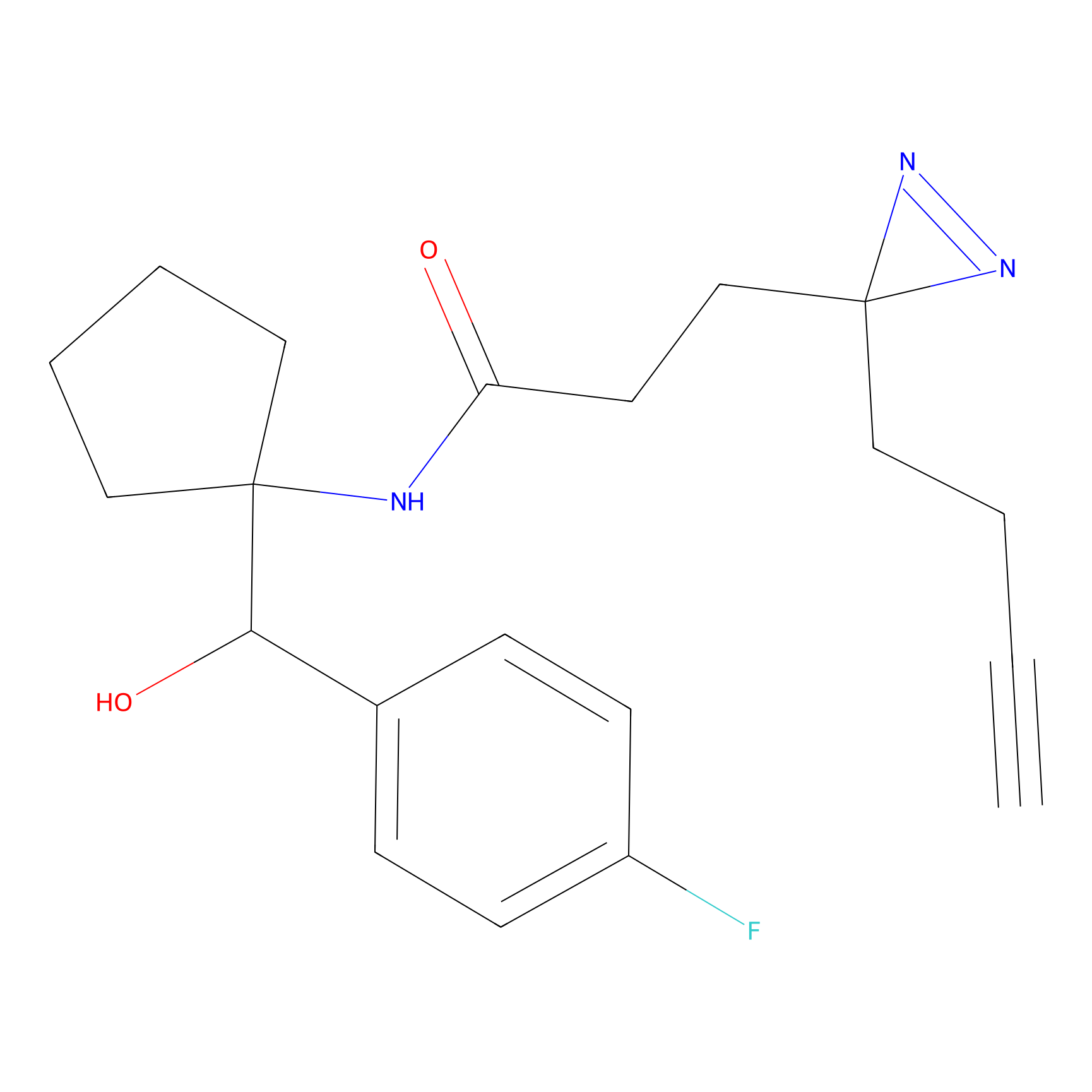
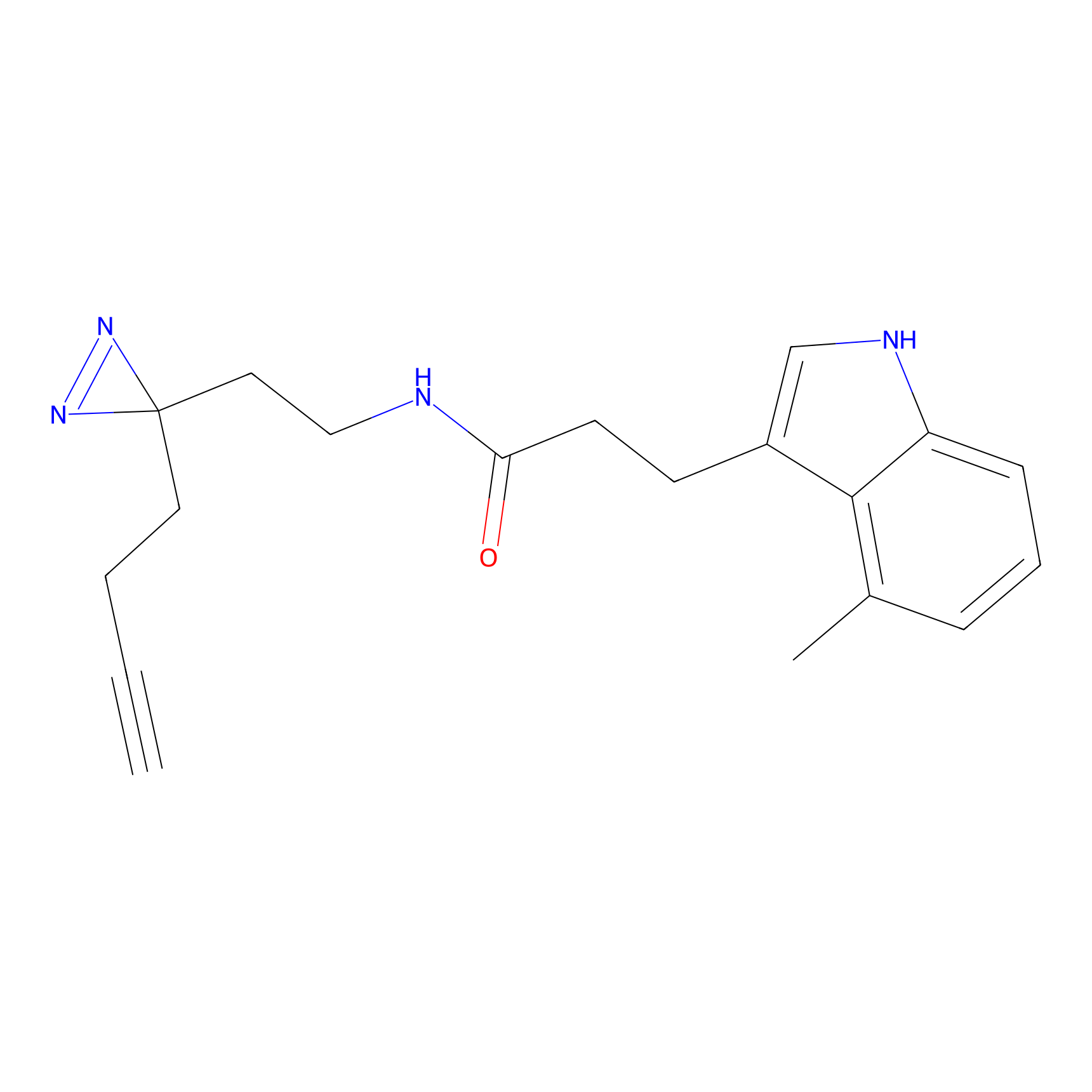
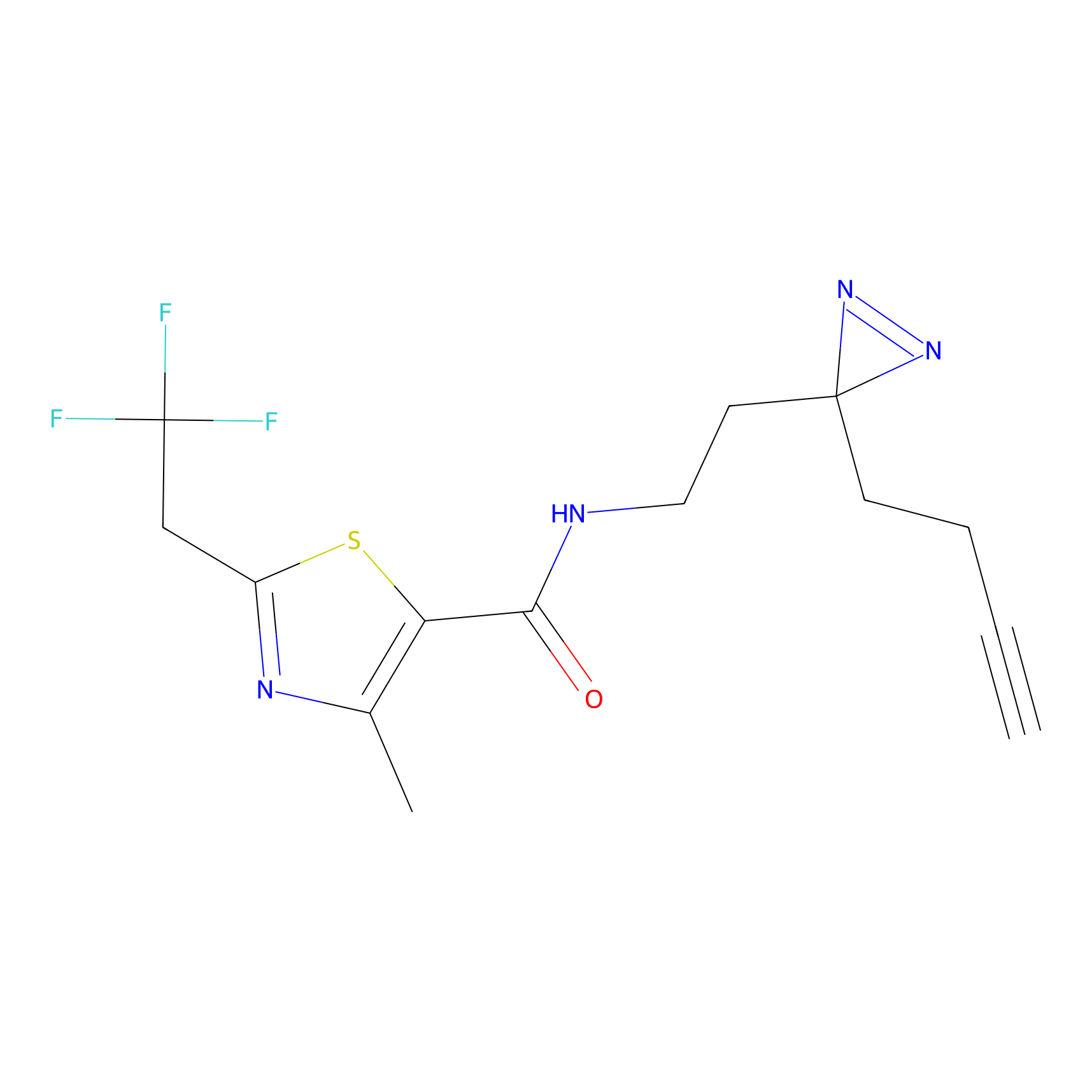
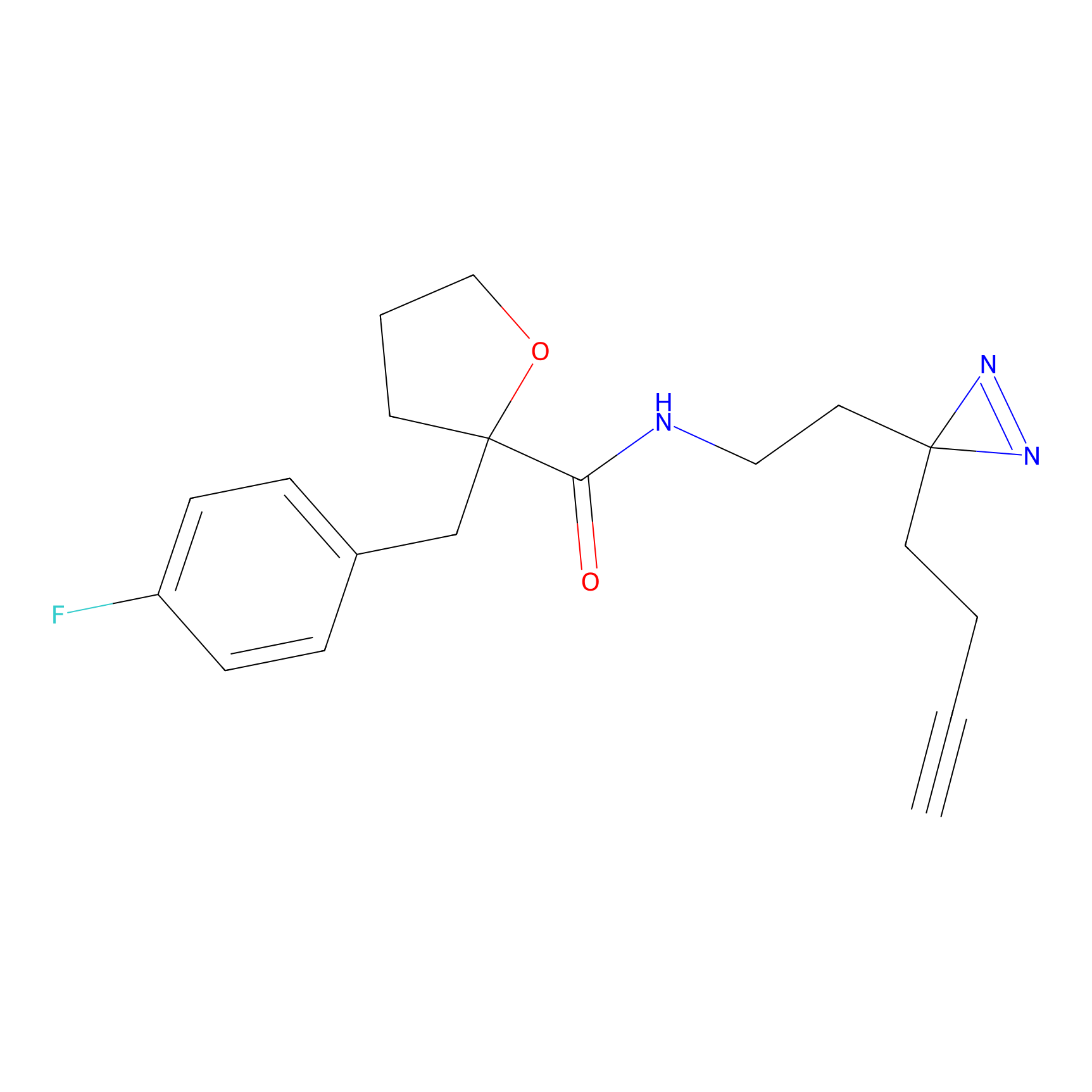
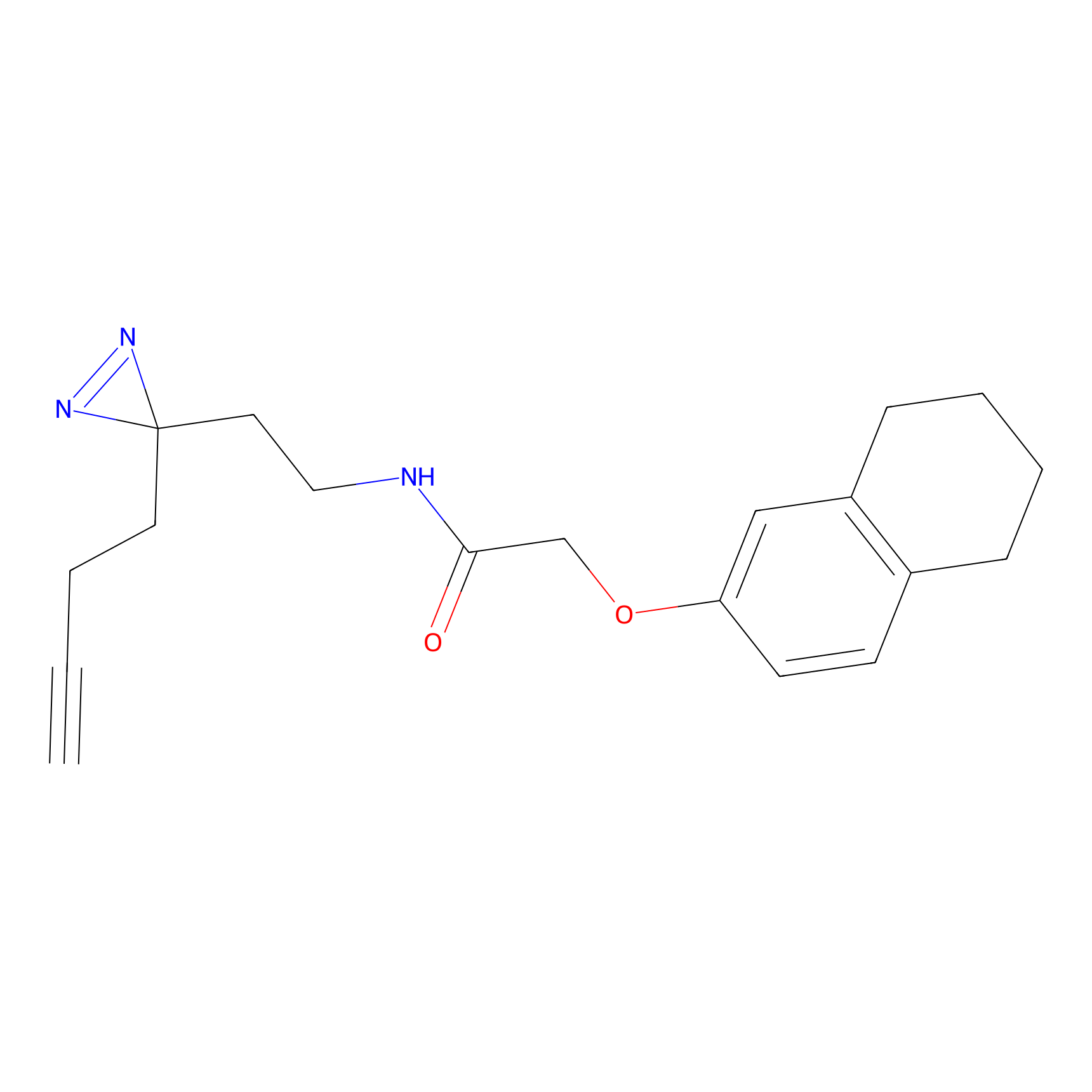
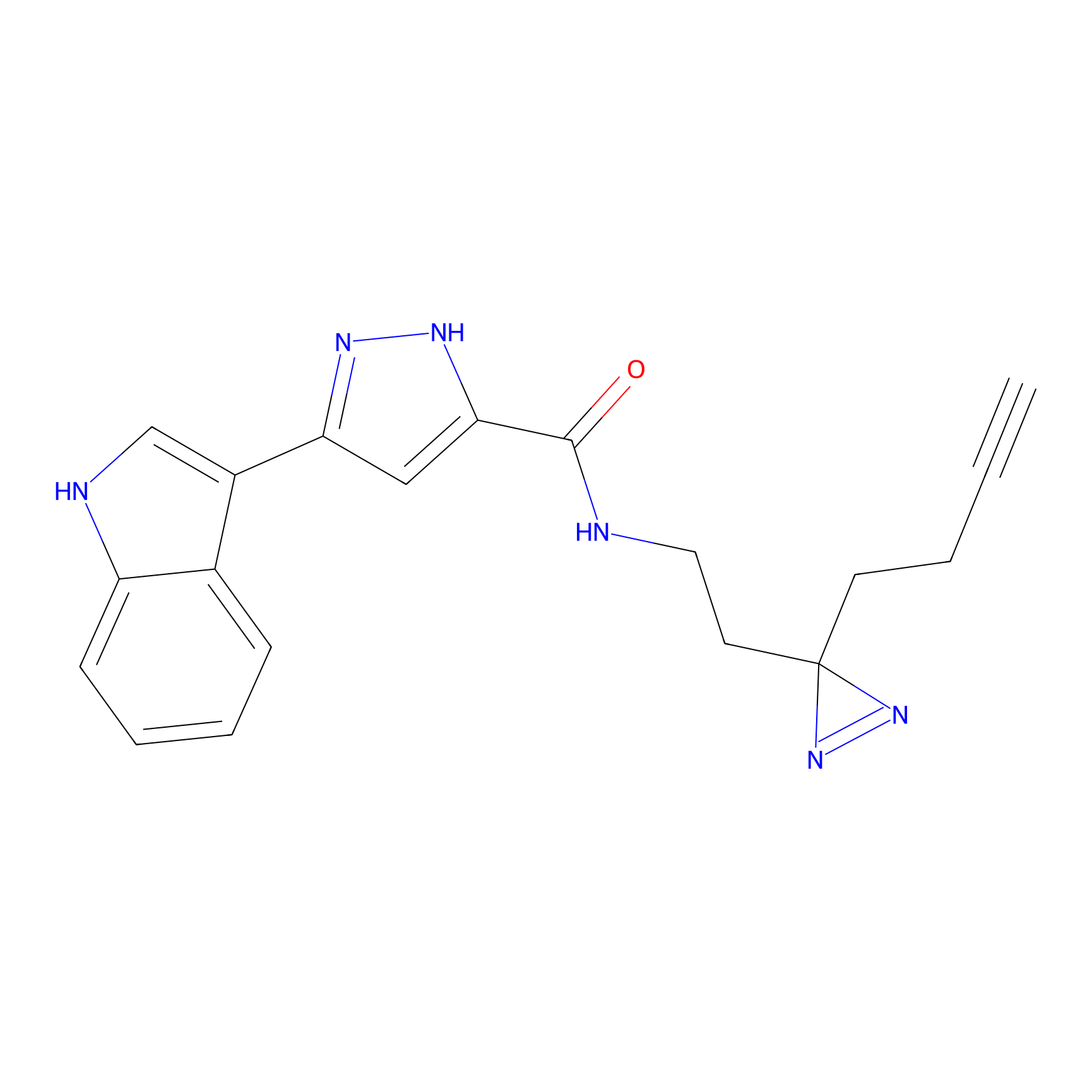
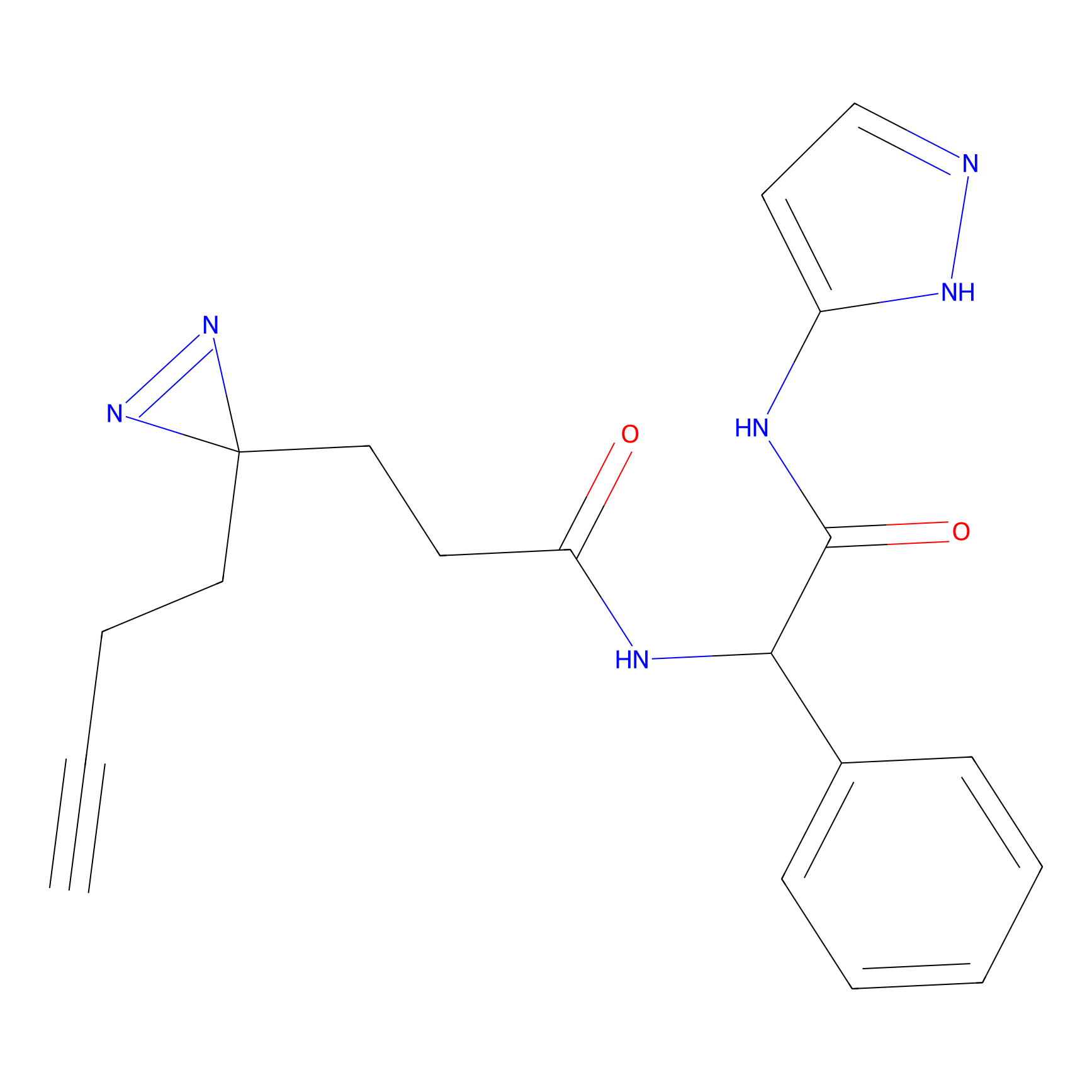
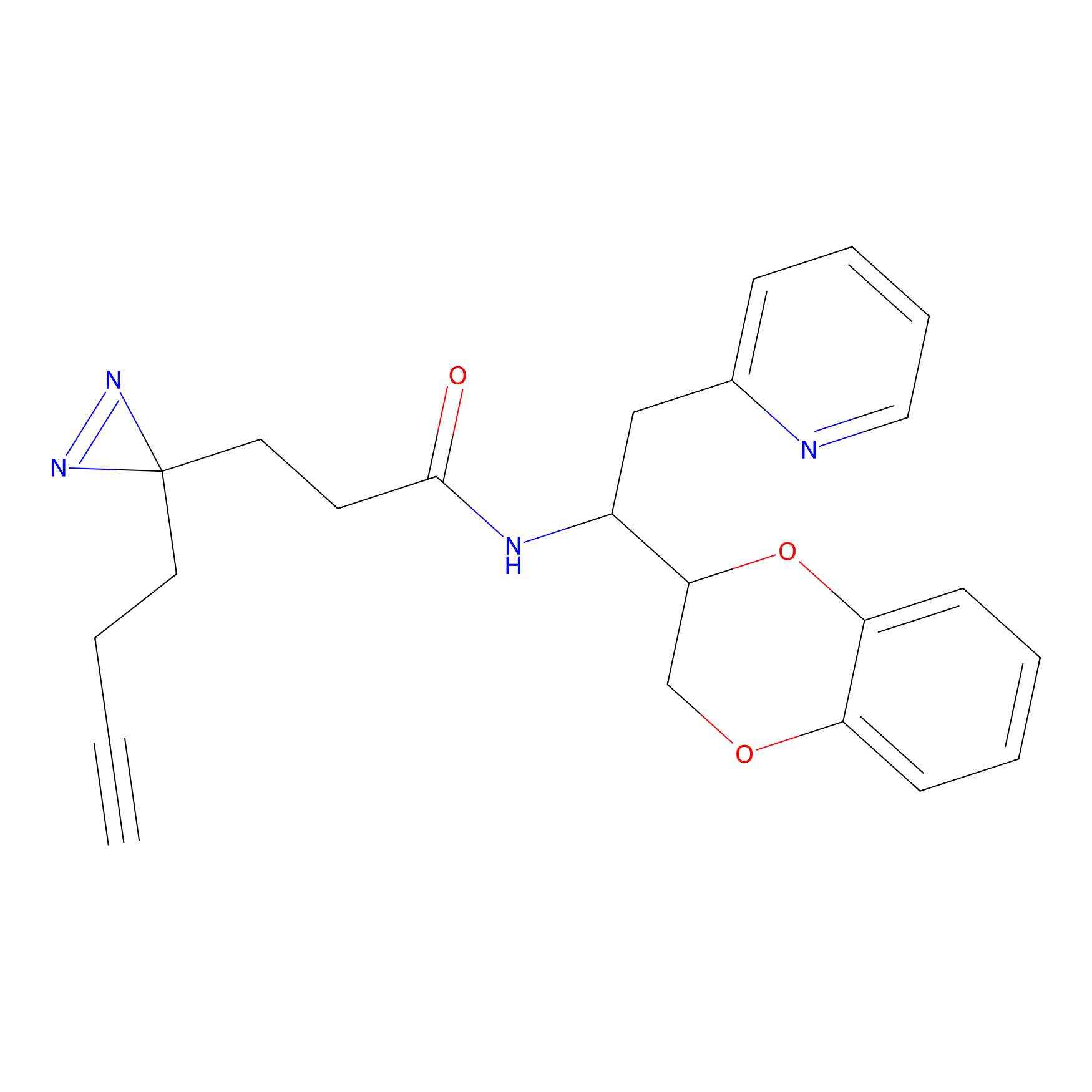
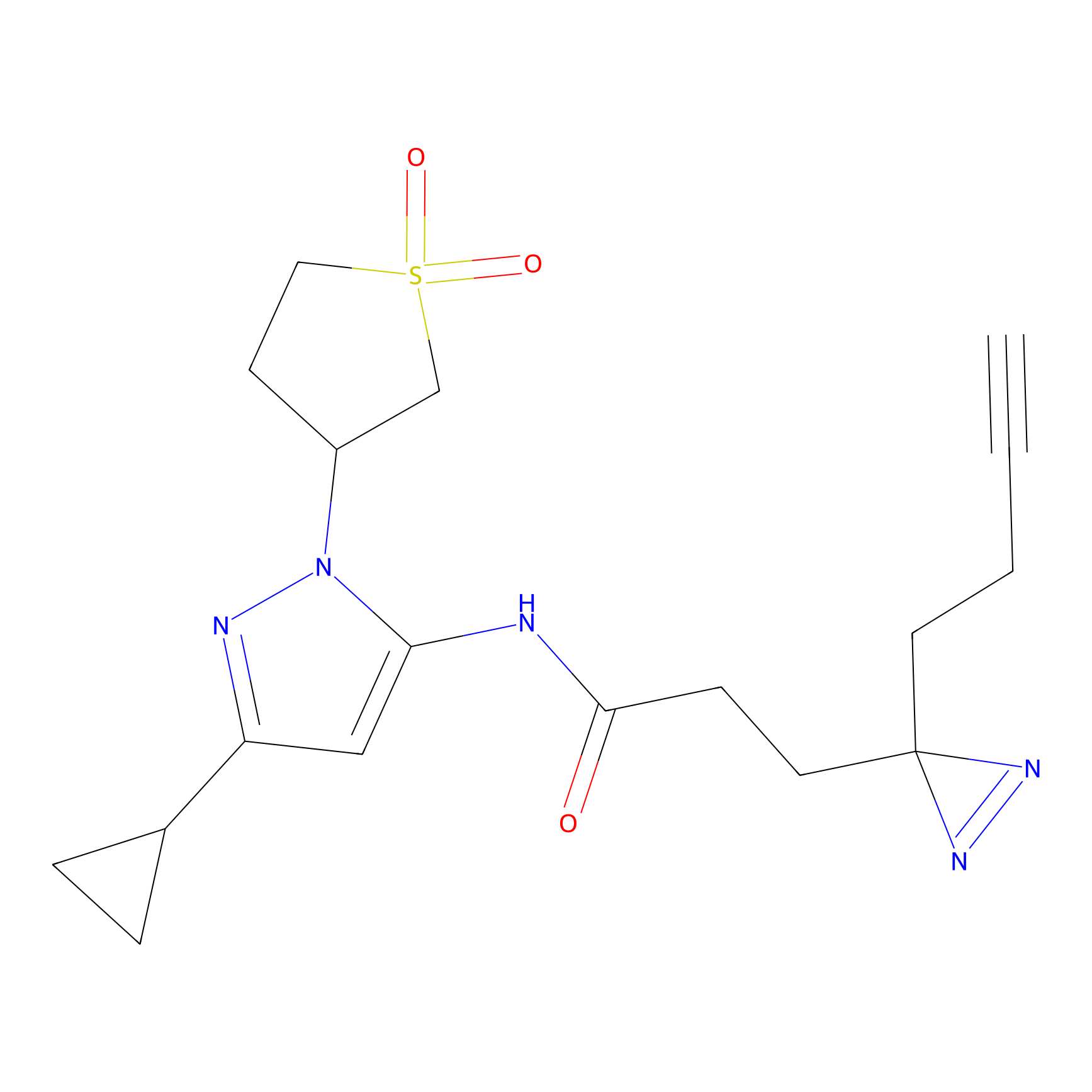
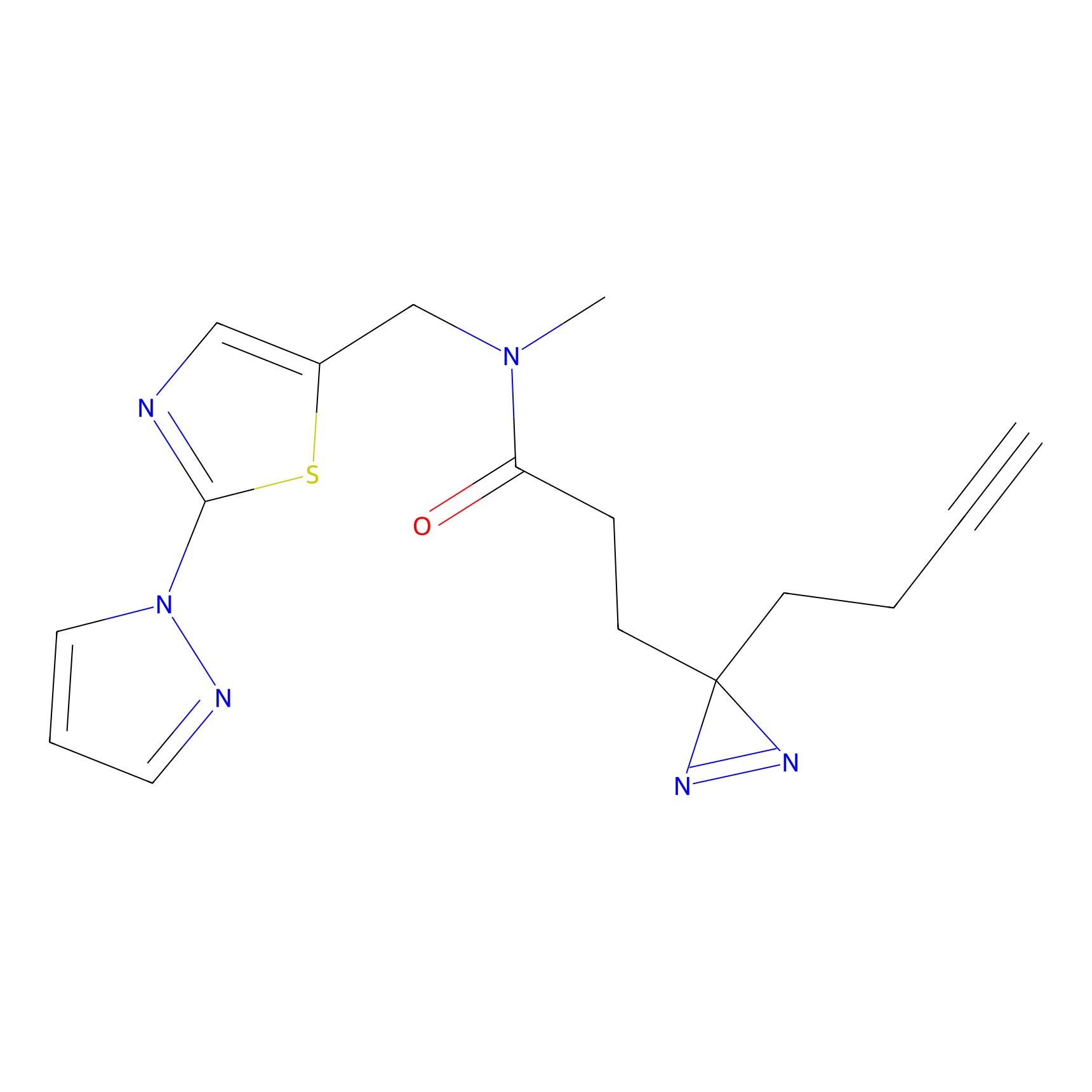
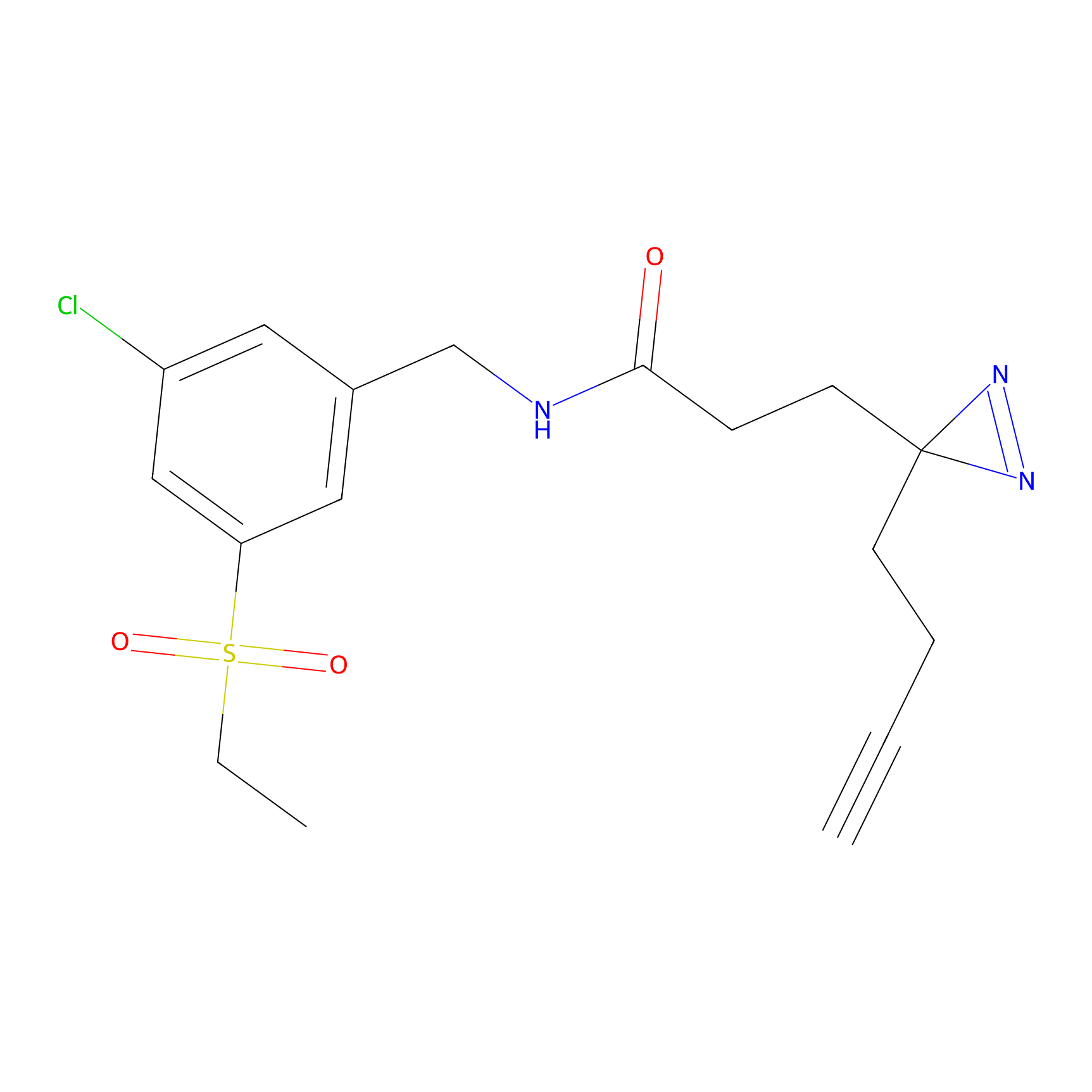
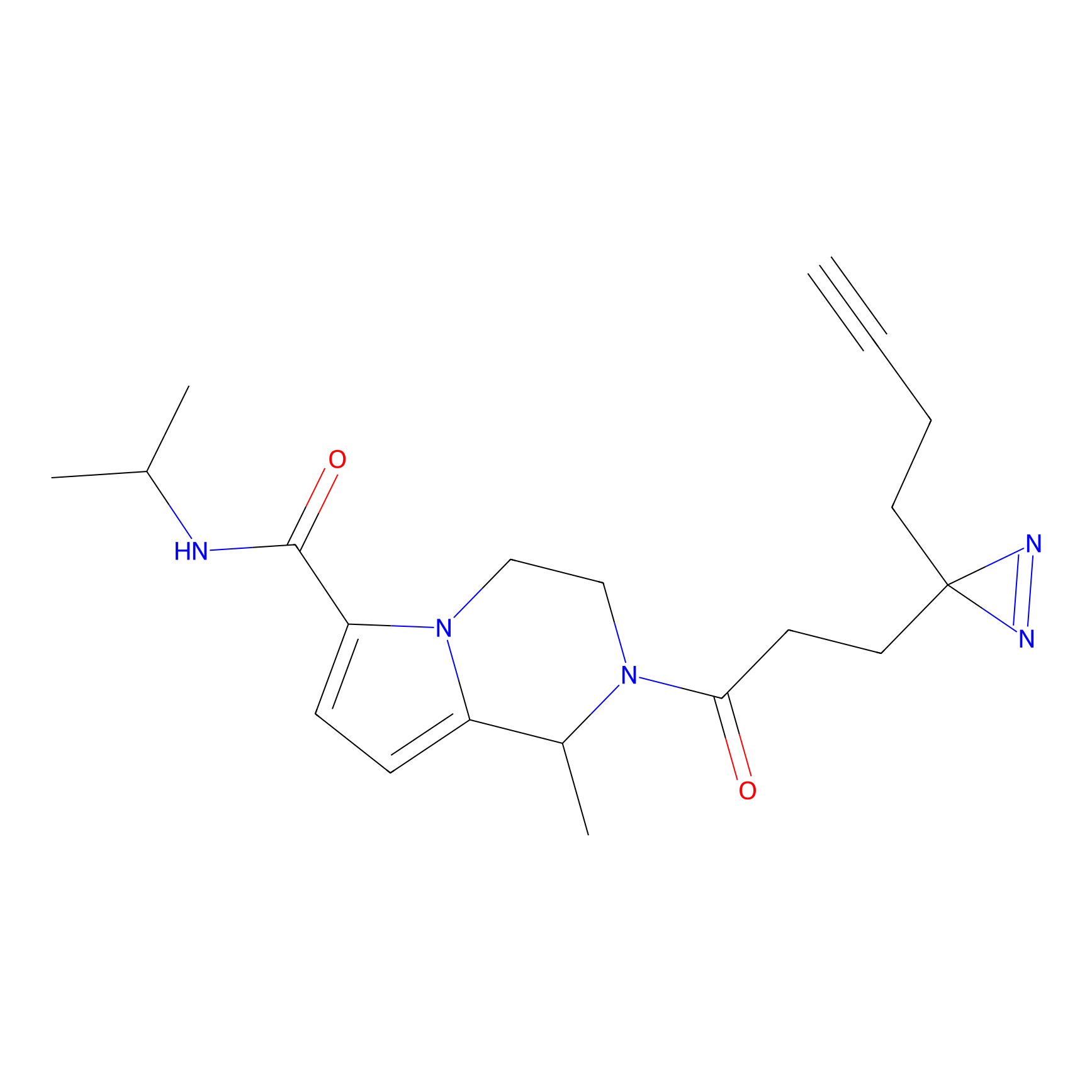
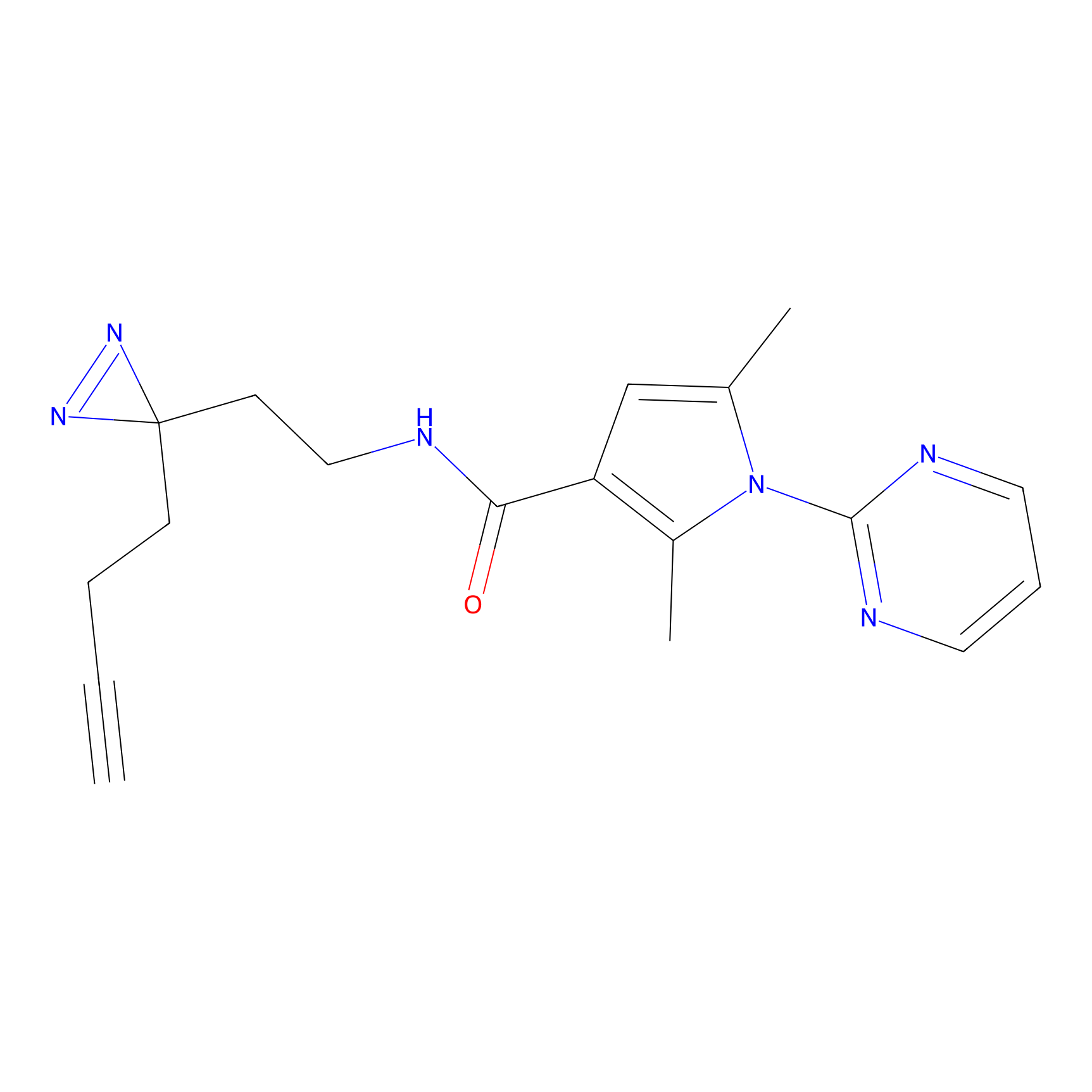
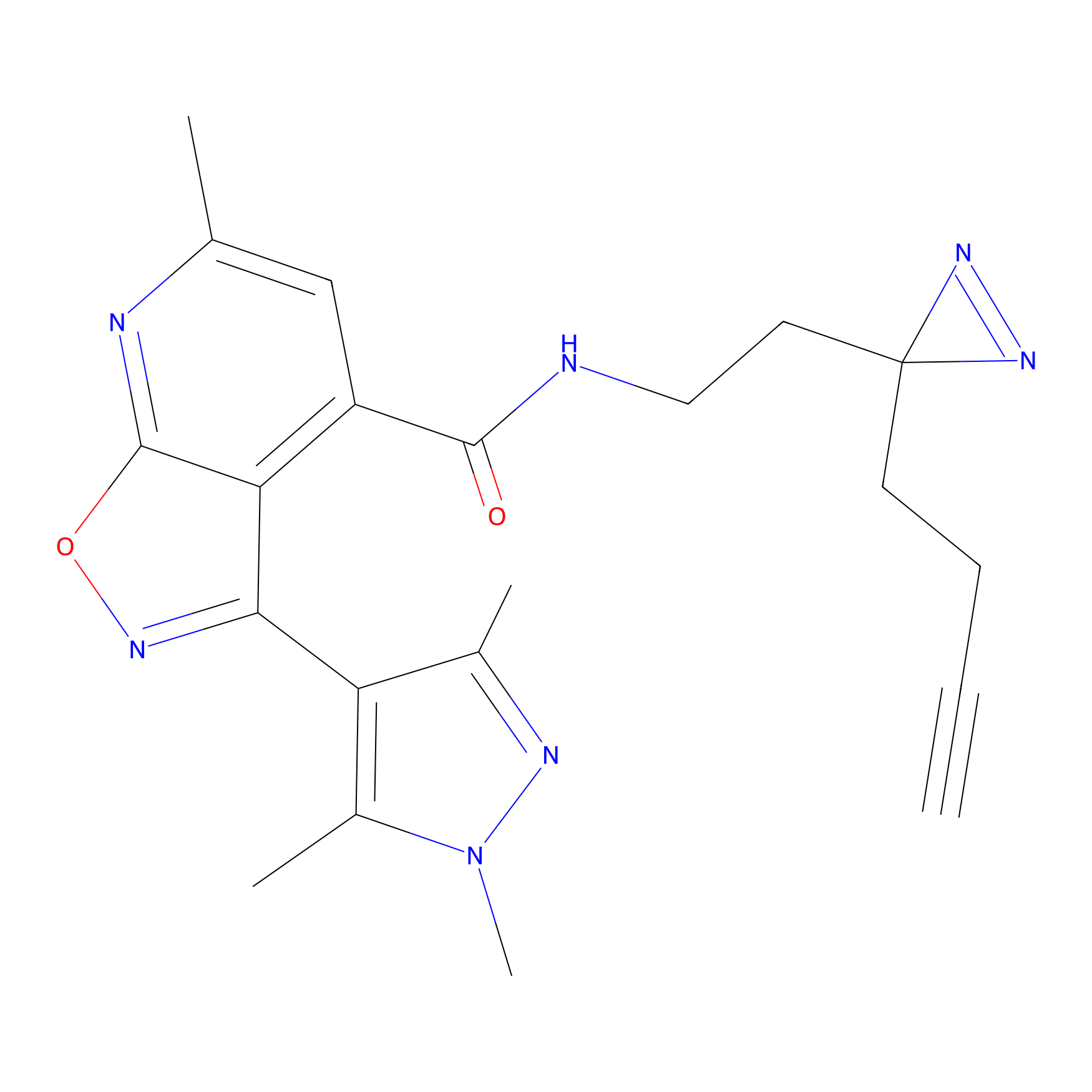
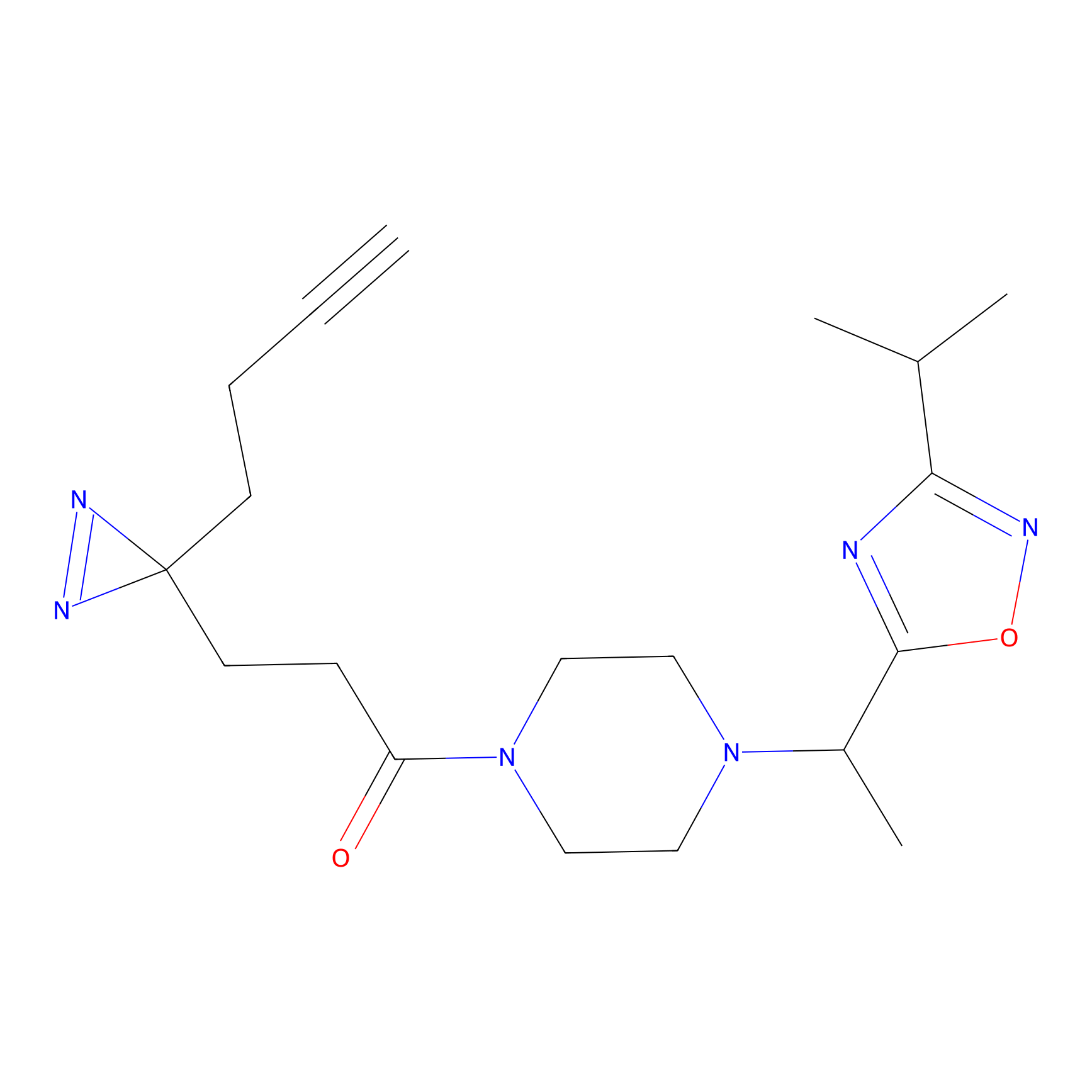
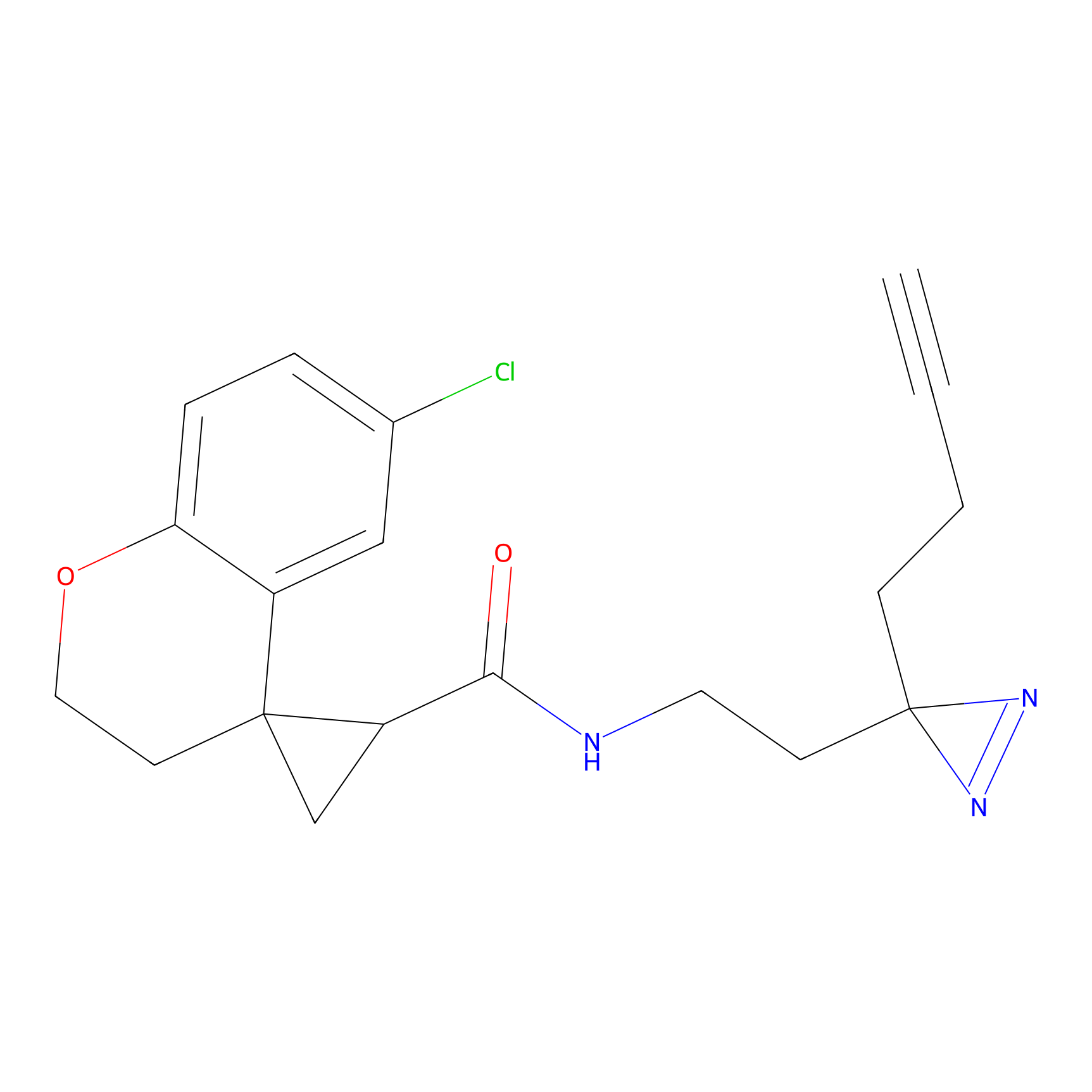
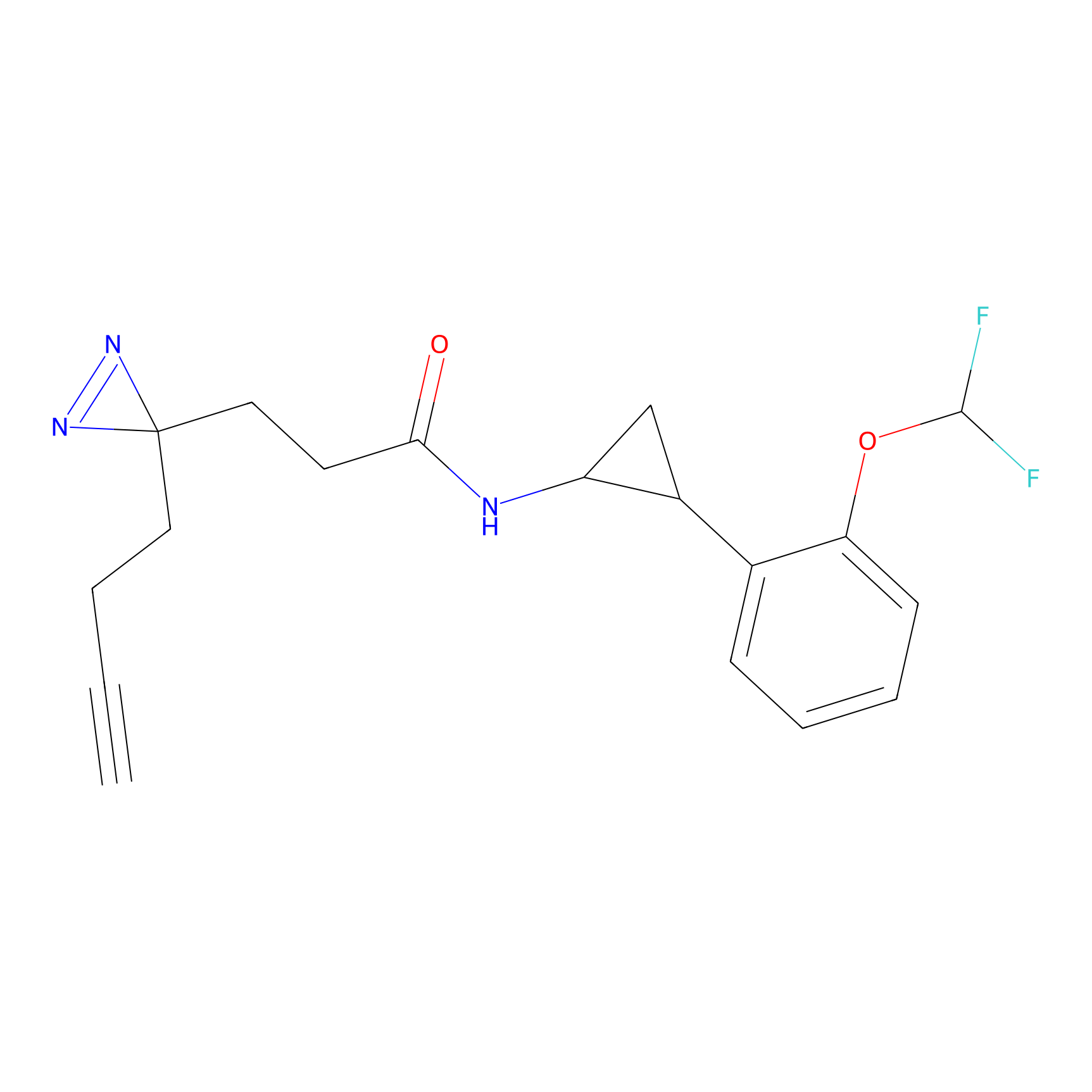
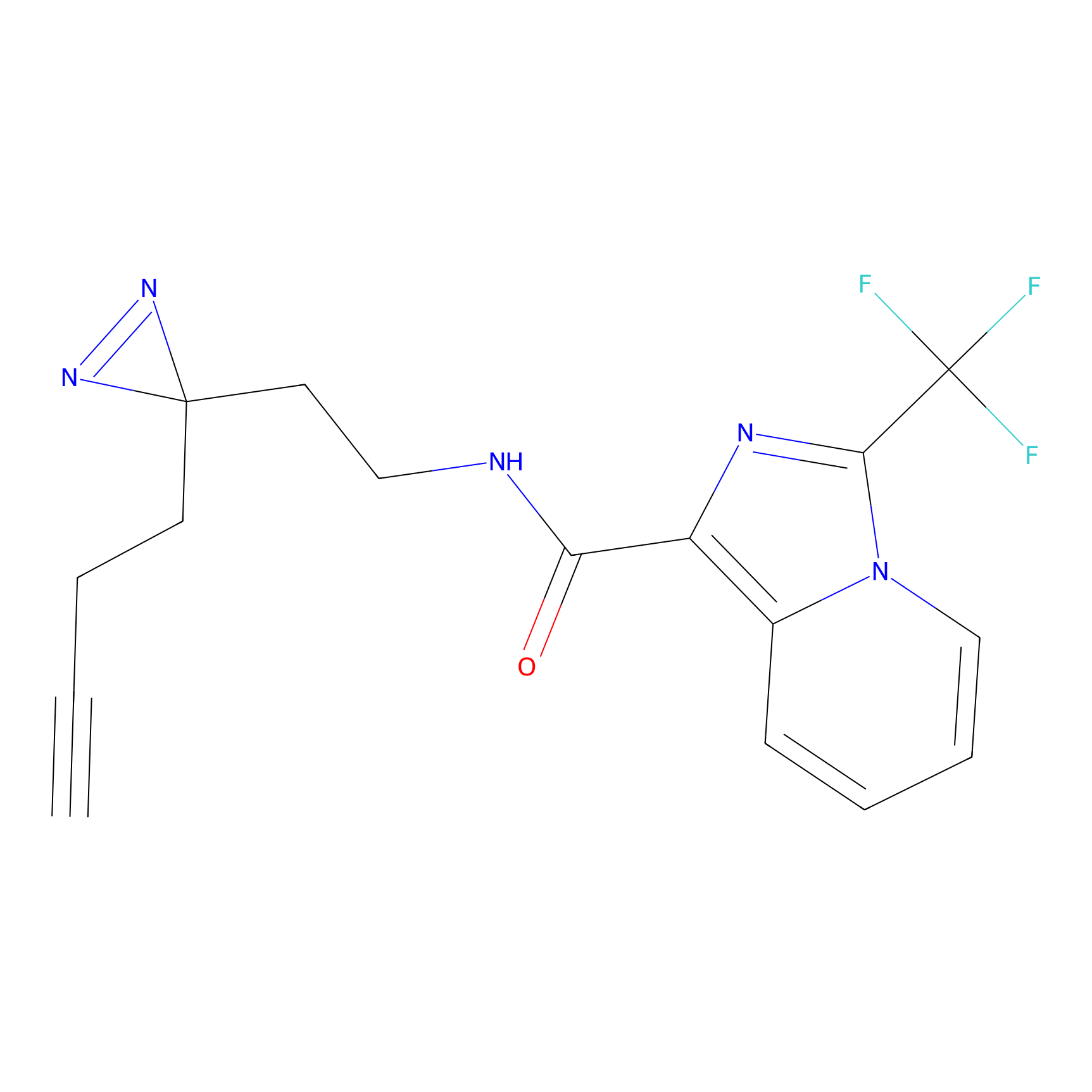
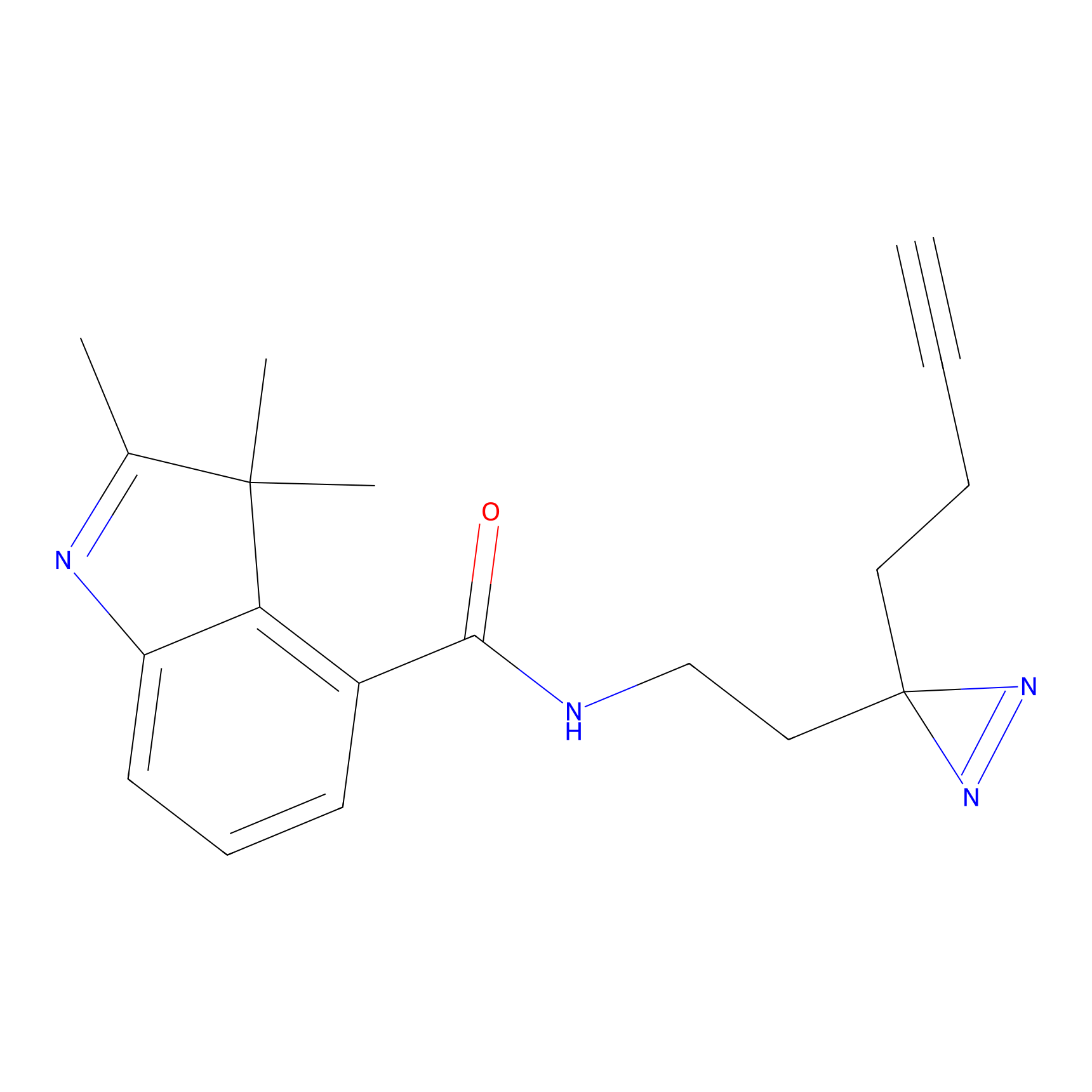
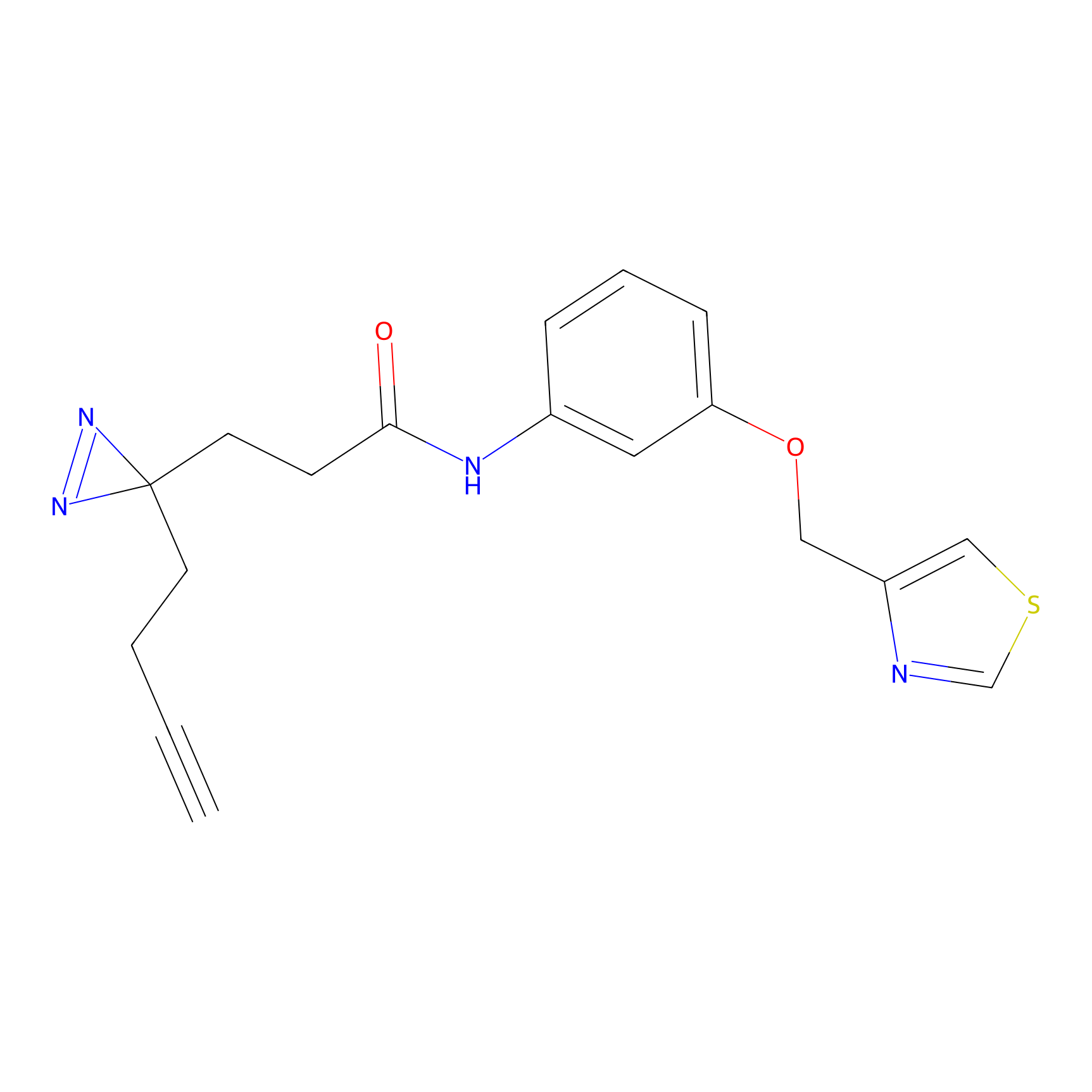
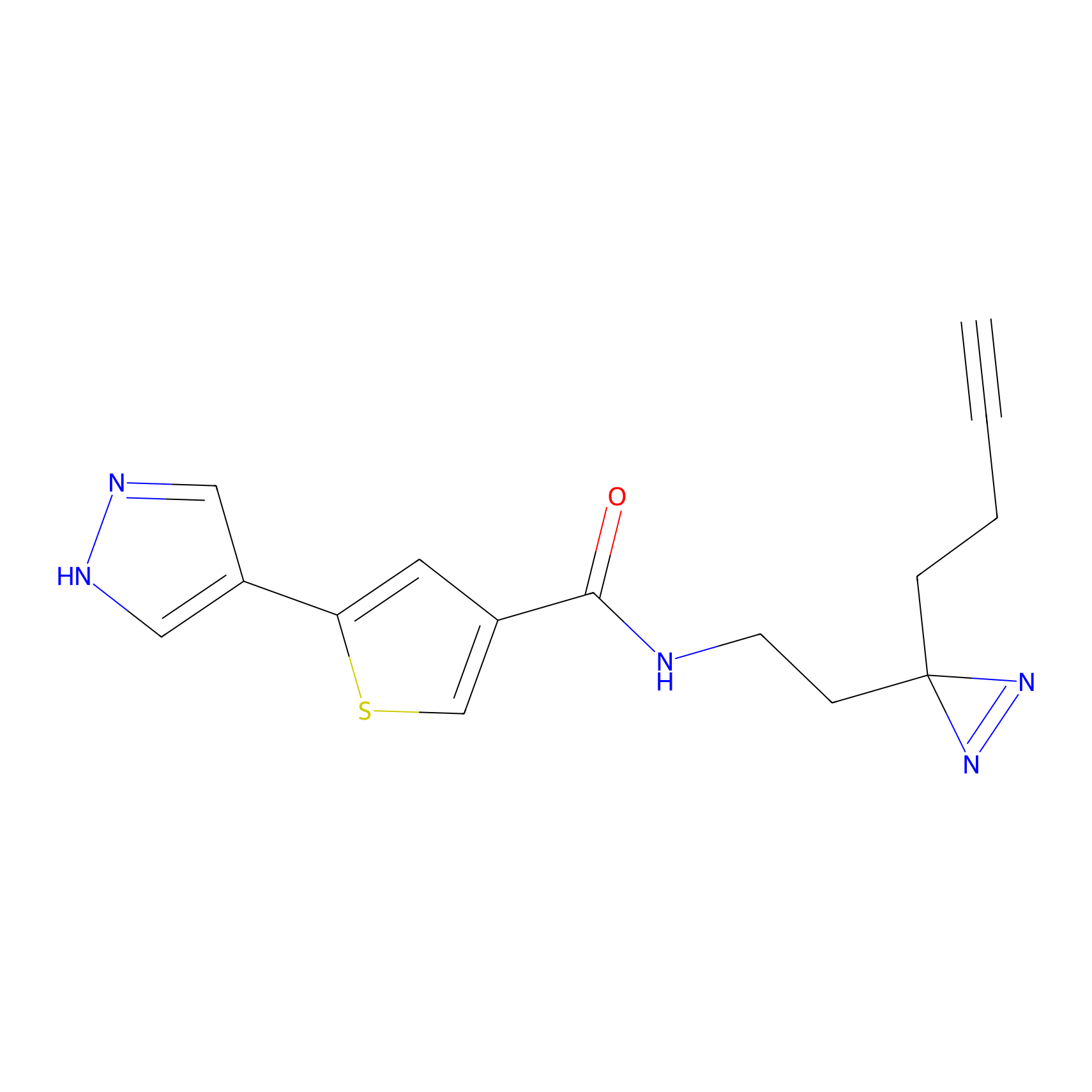
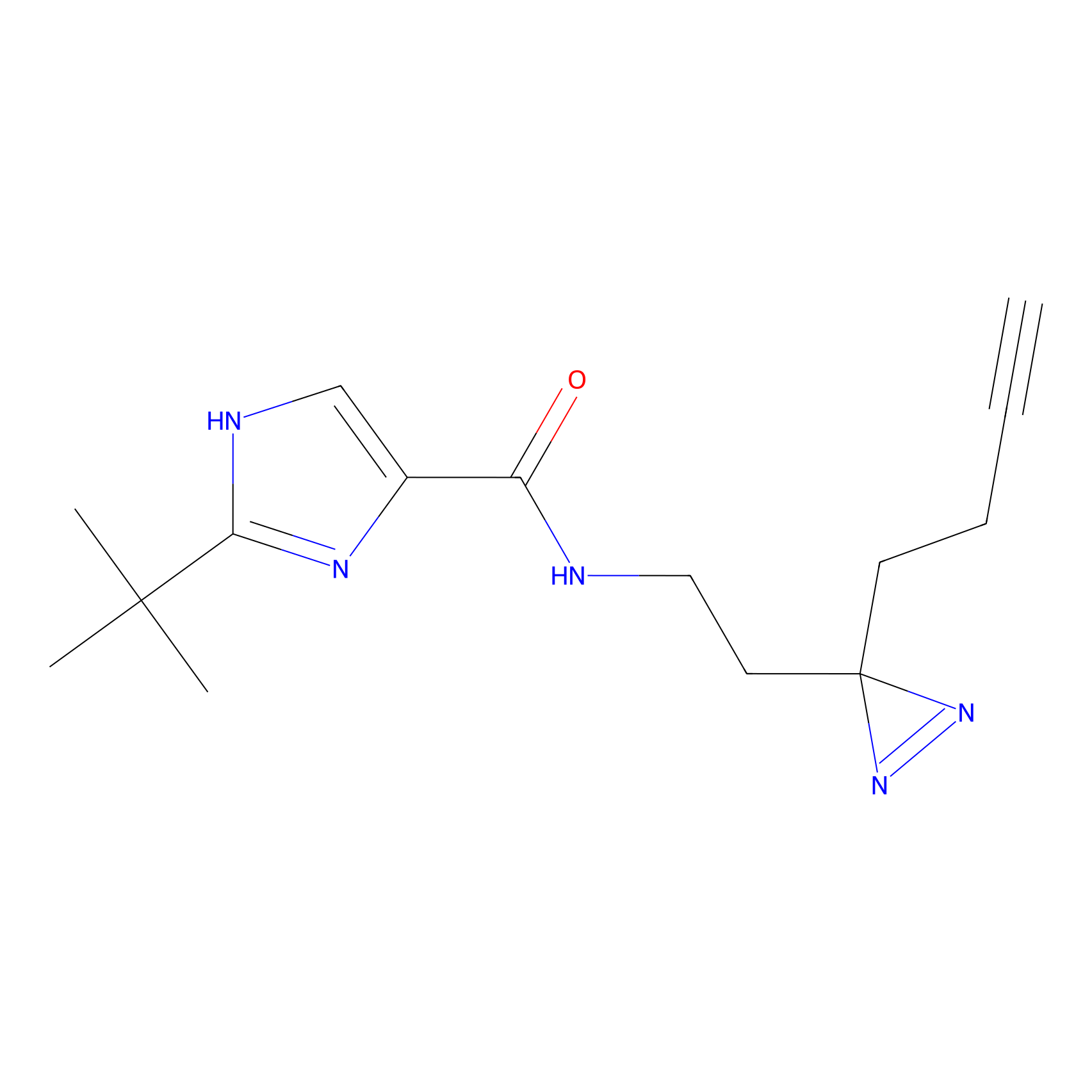
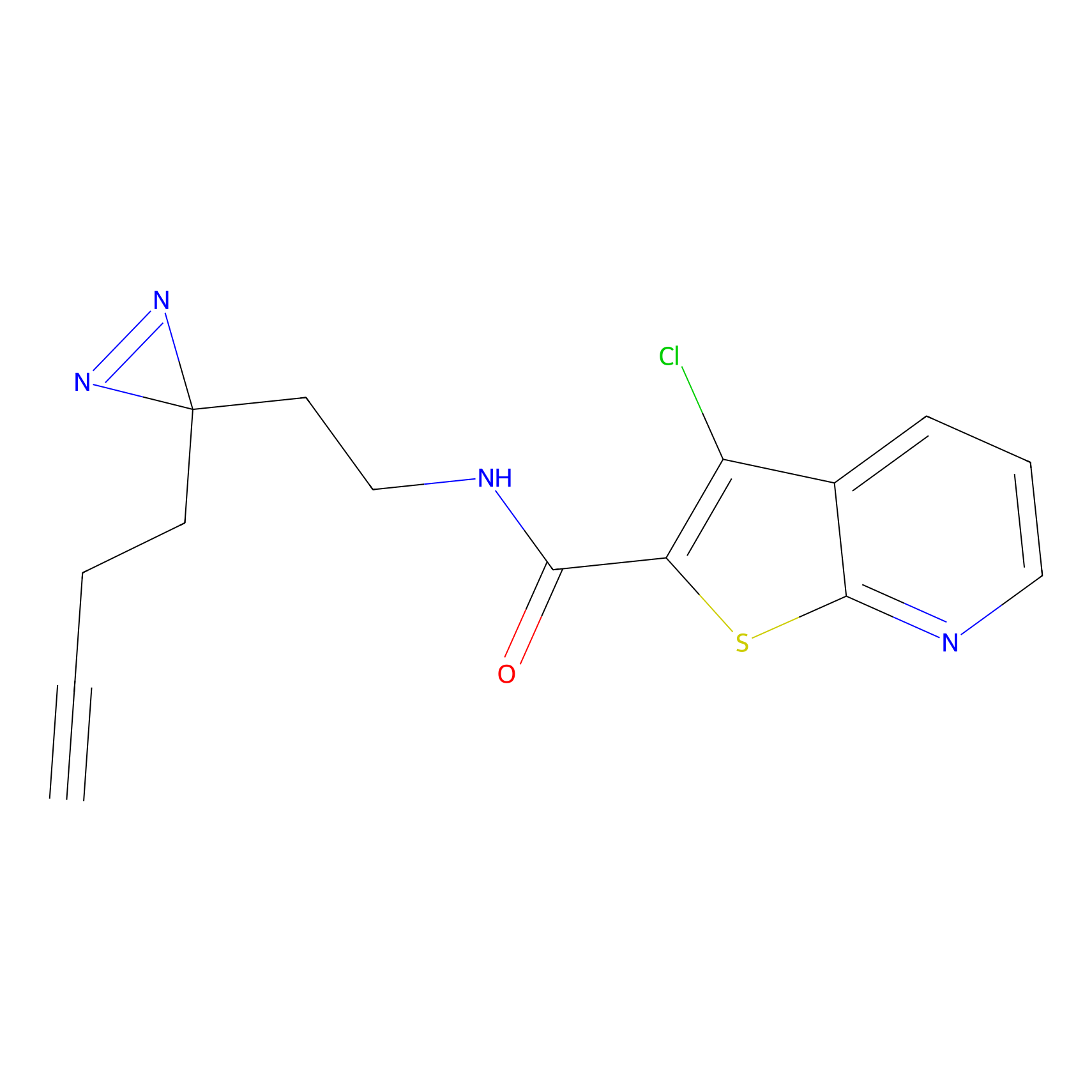
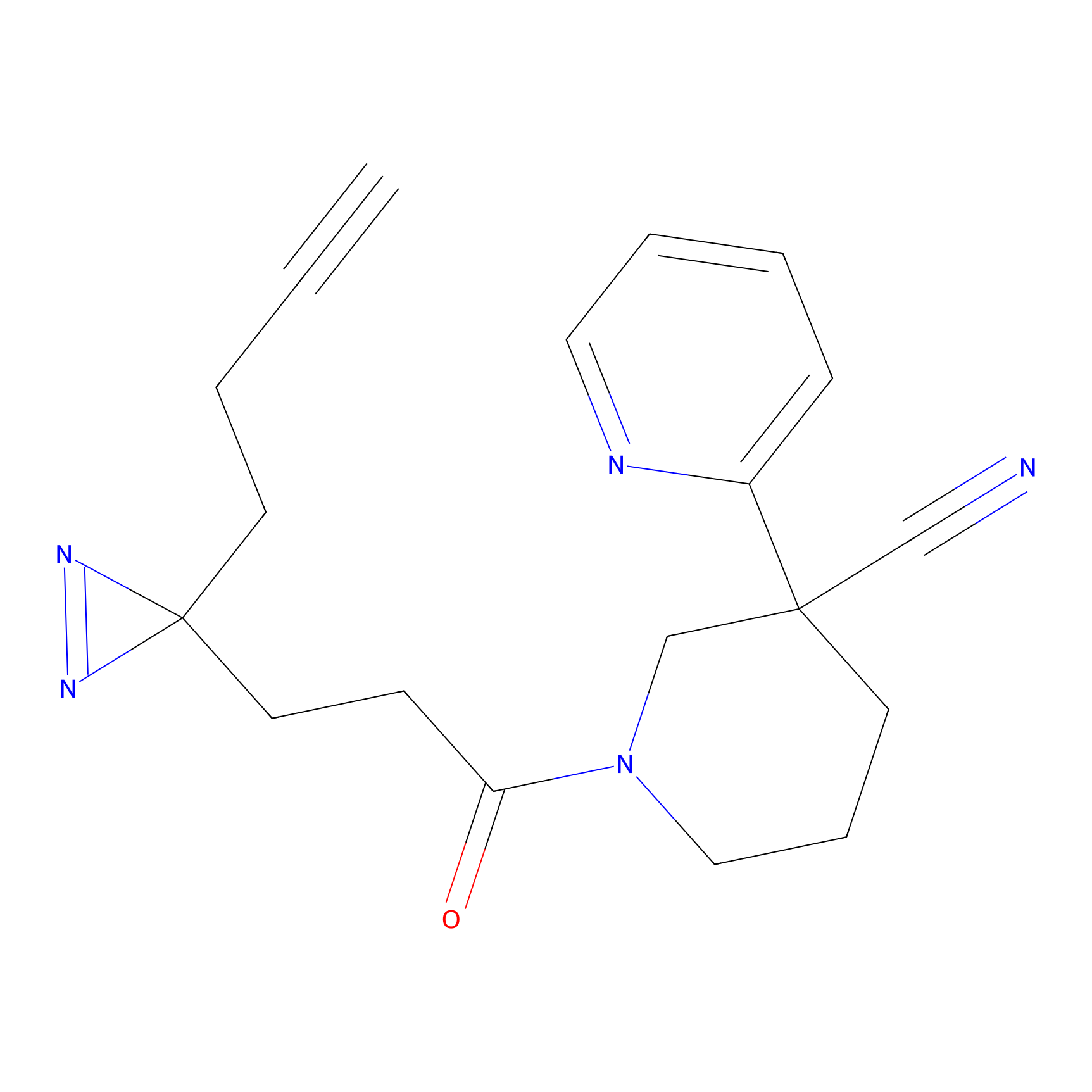
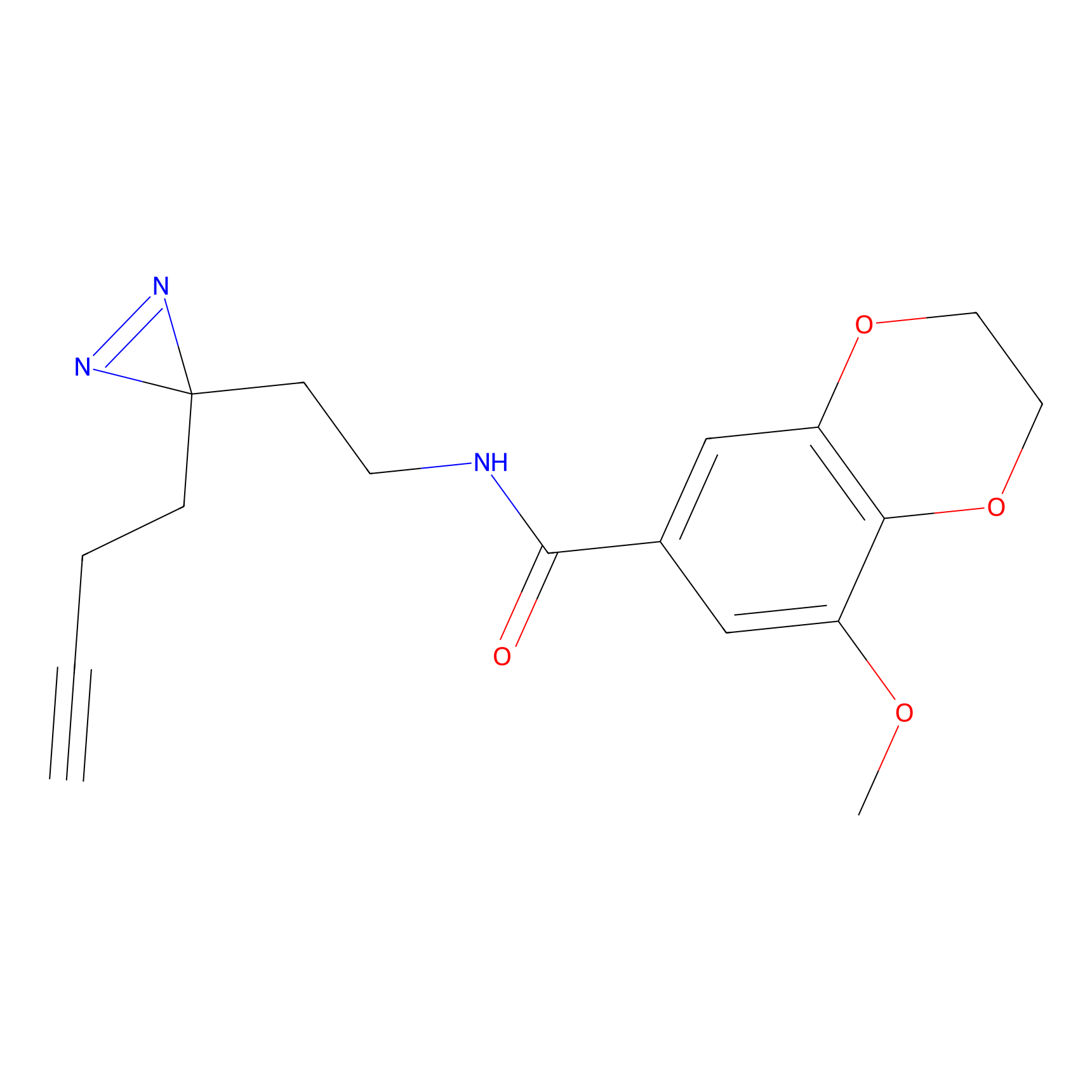
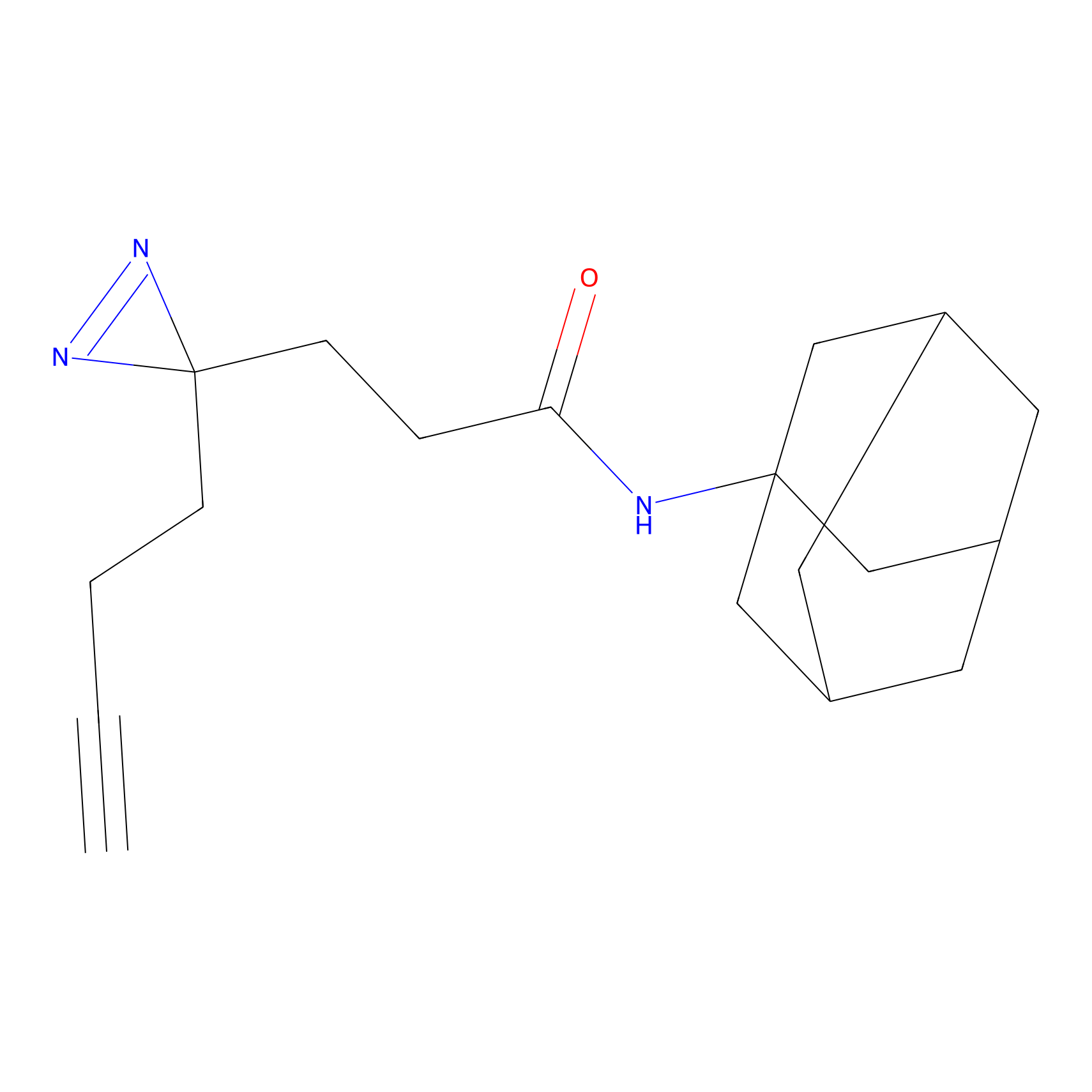
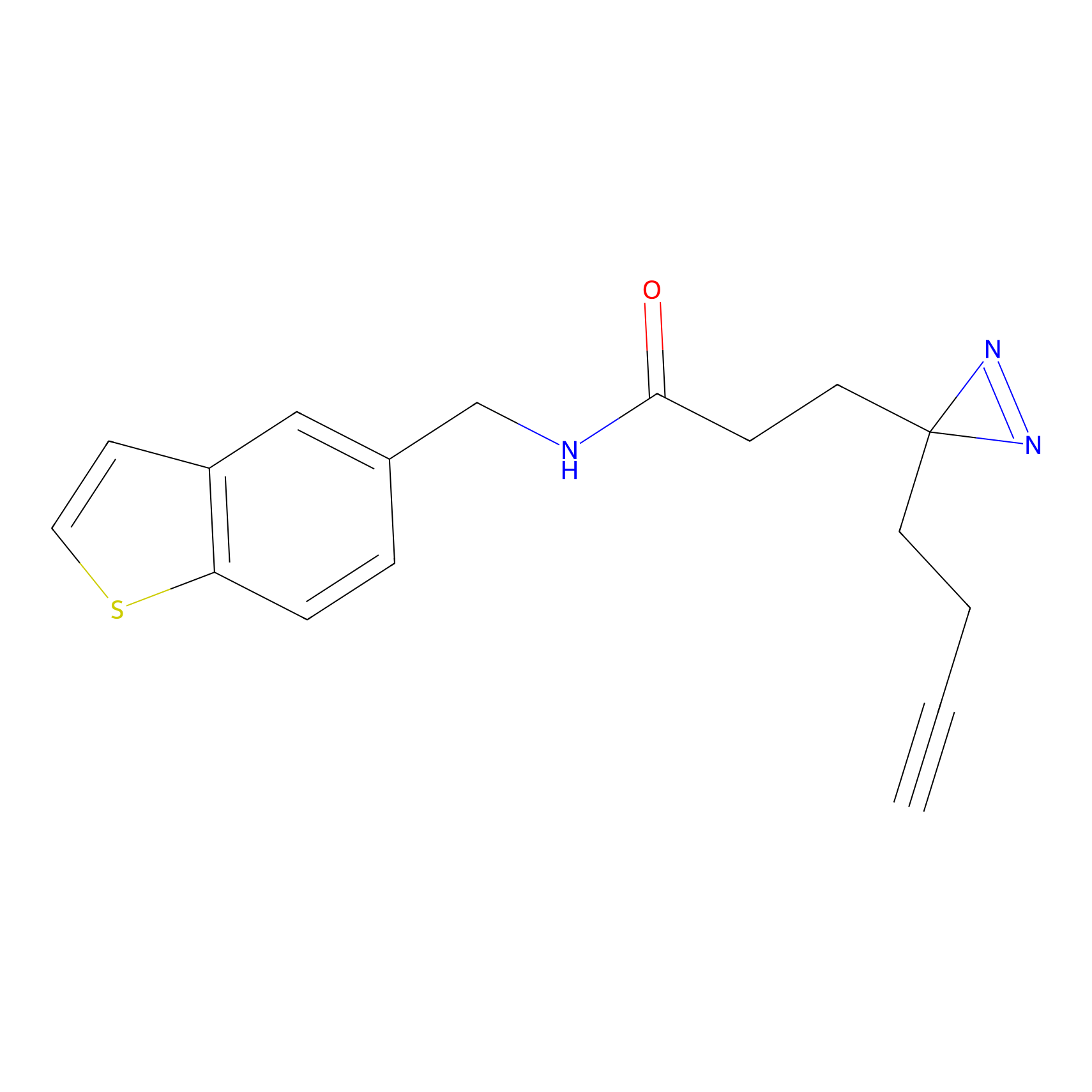
|
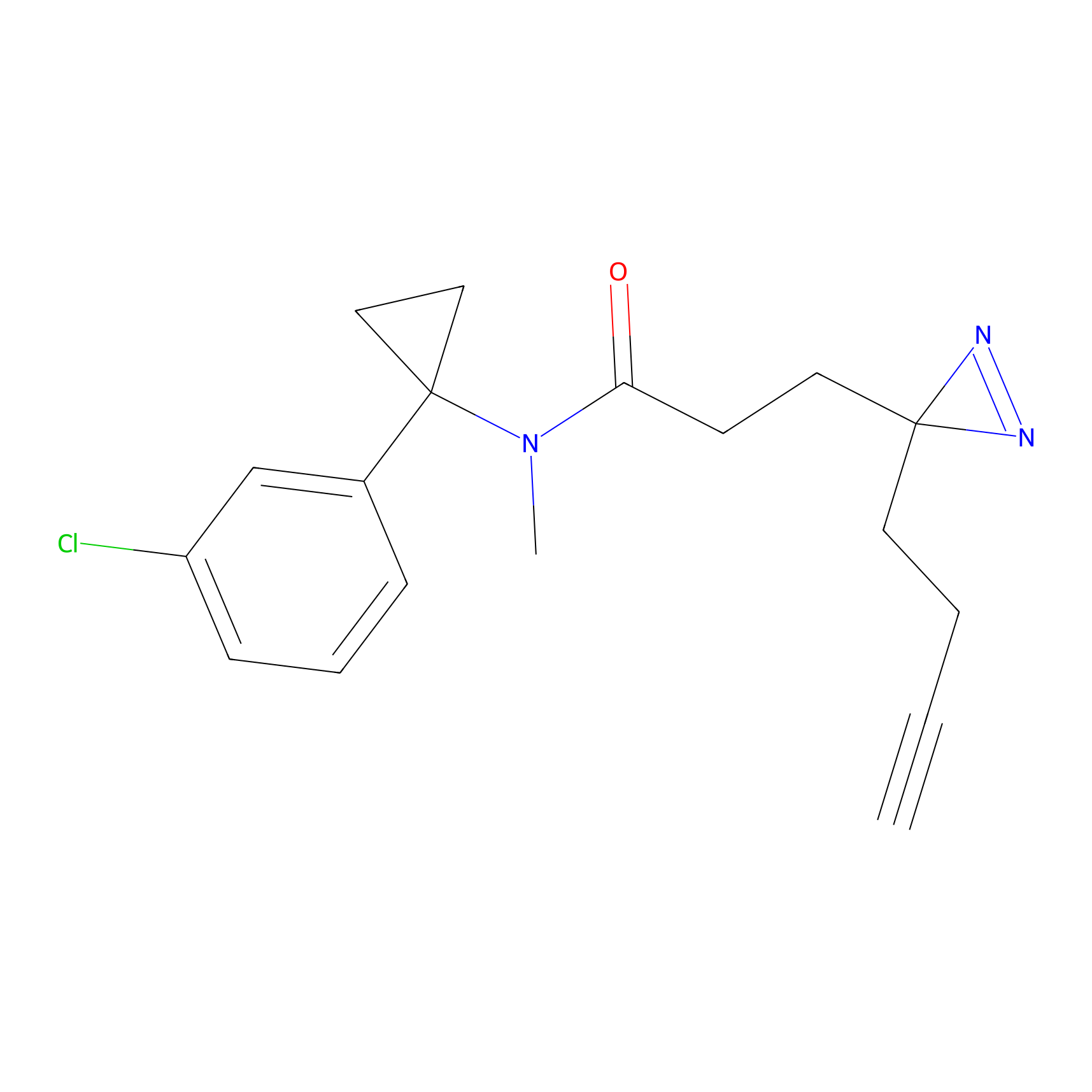
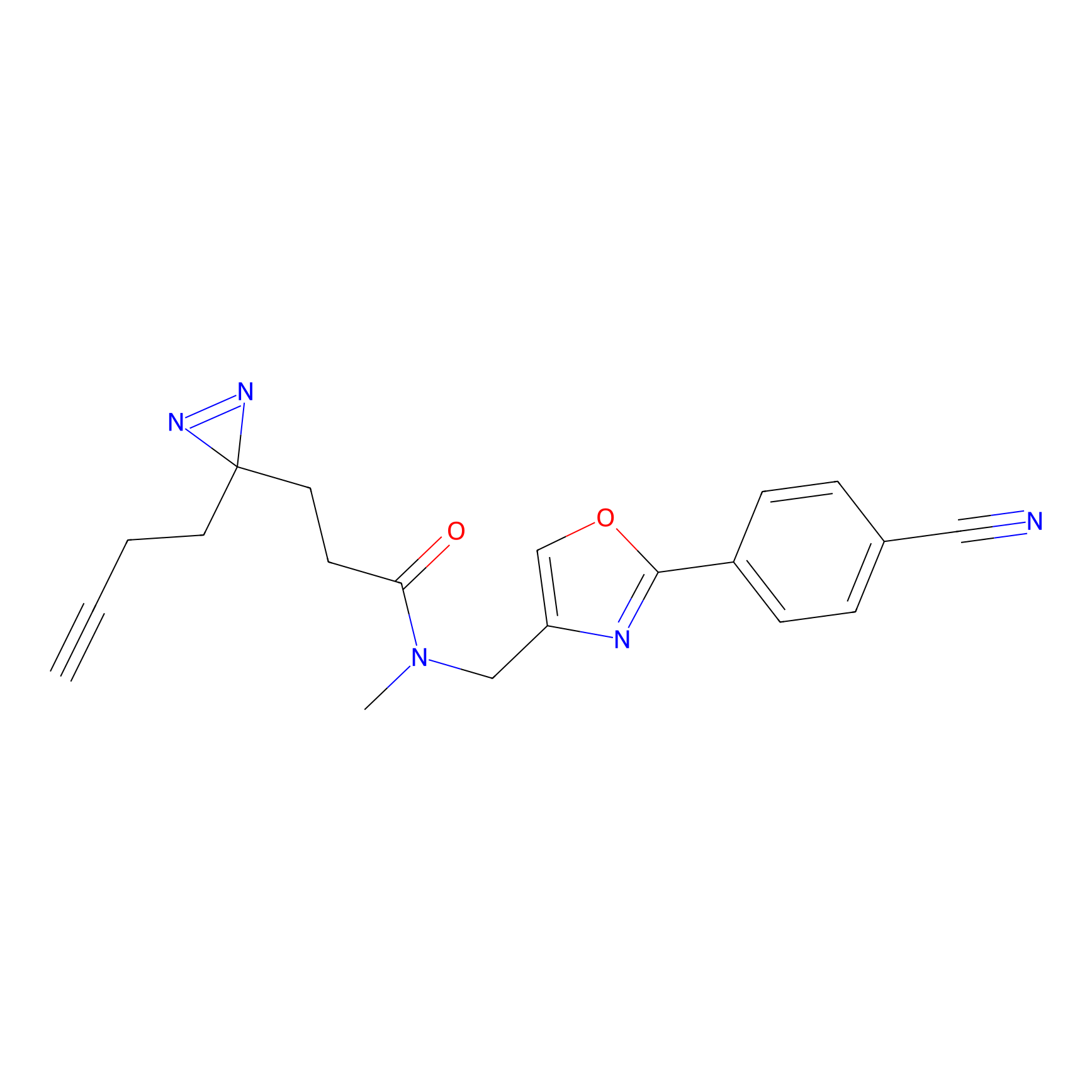
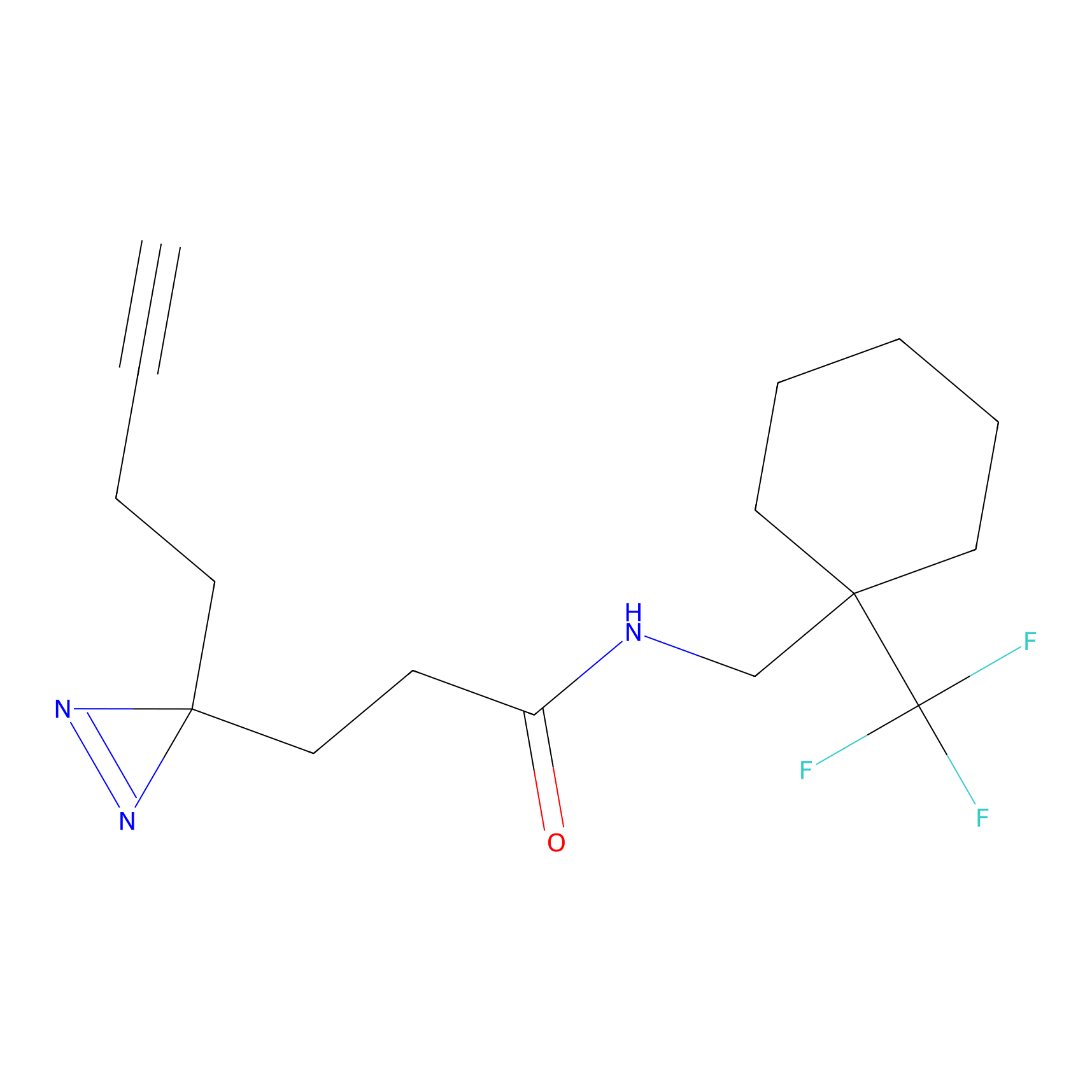
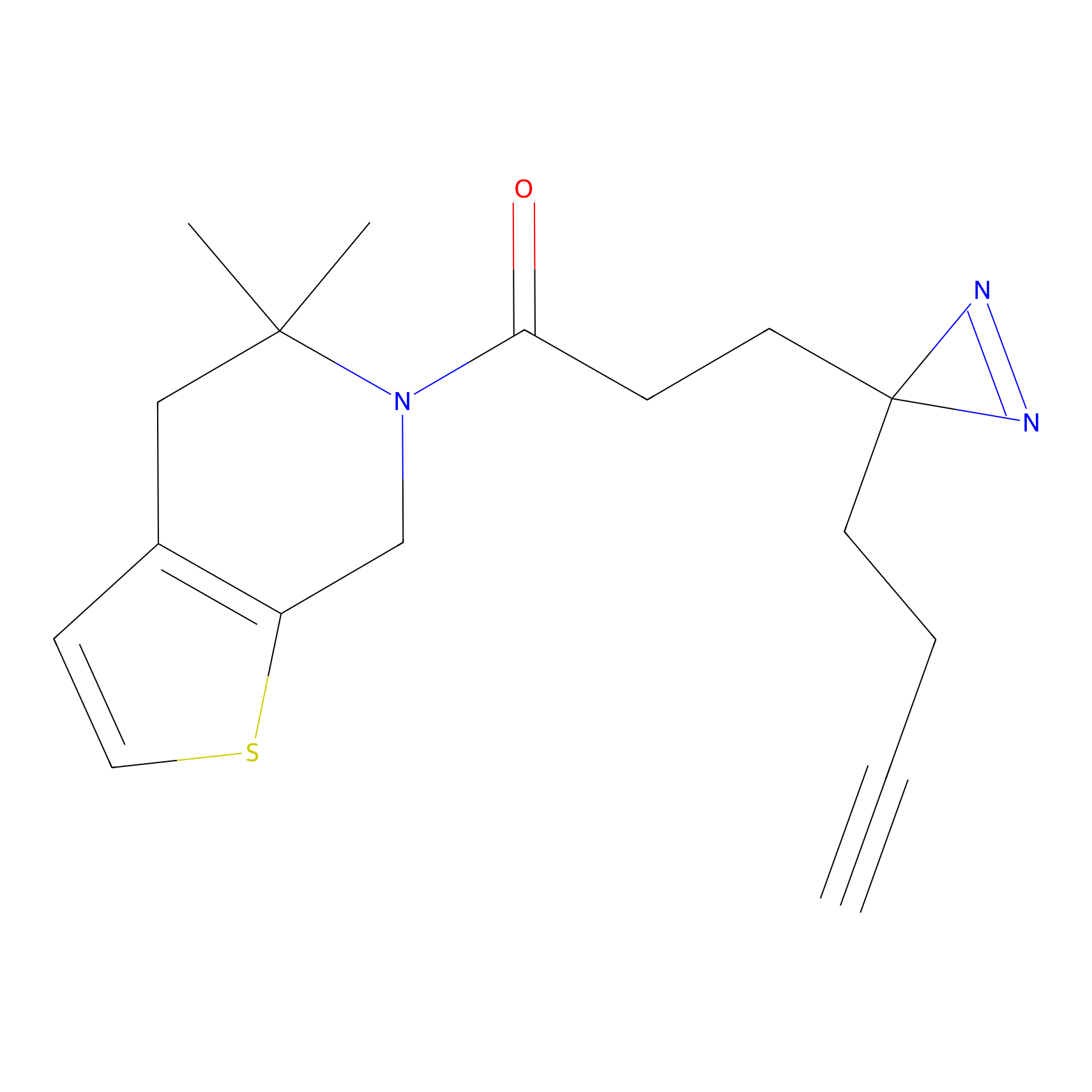
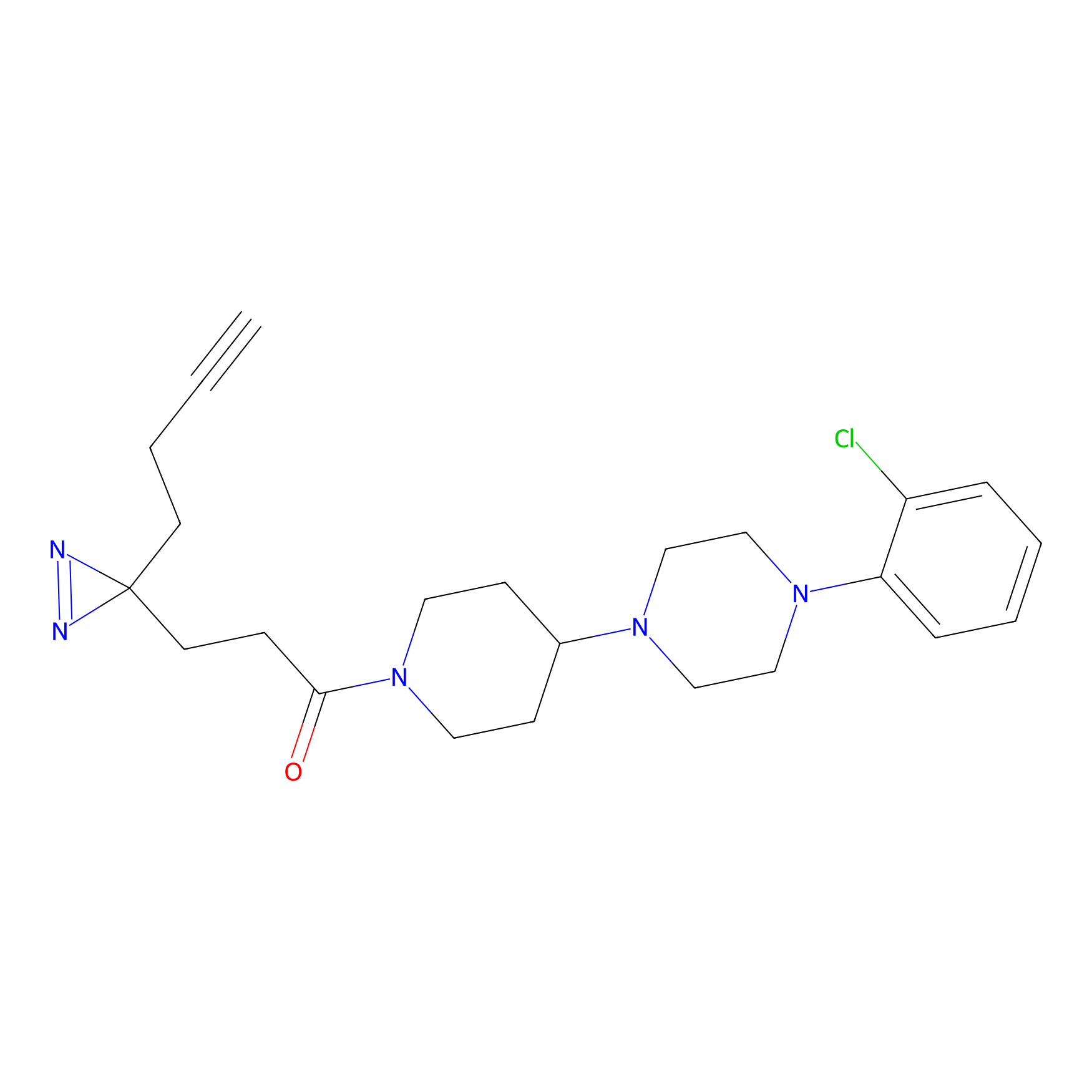
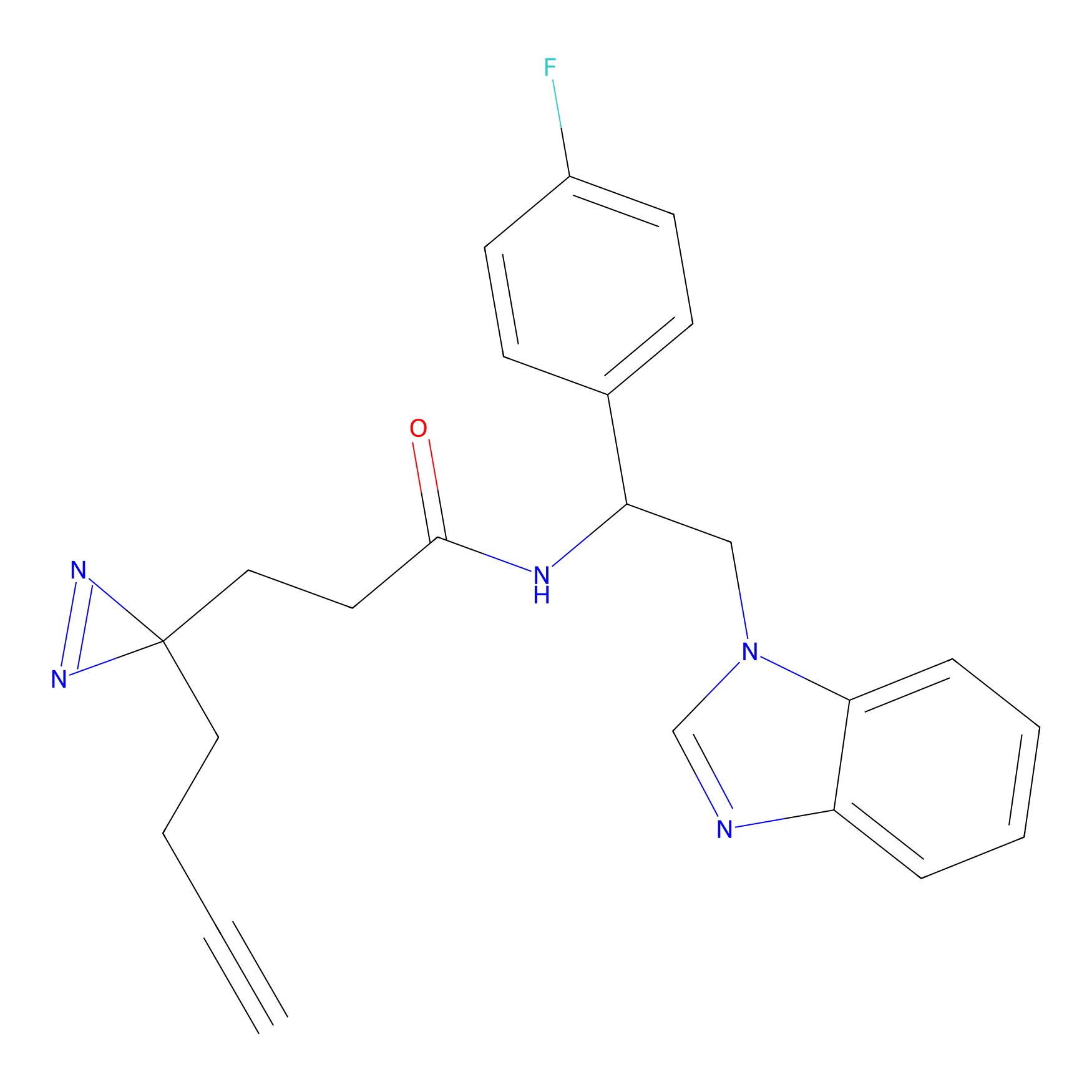
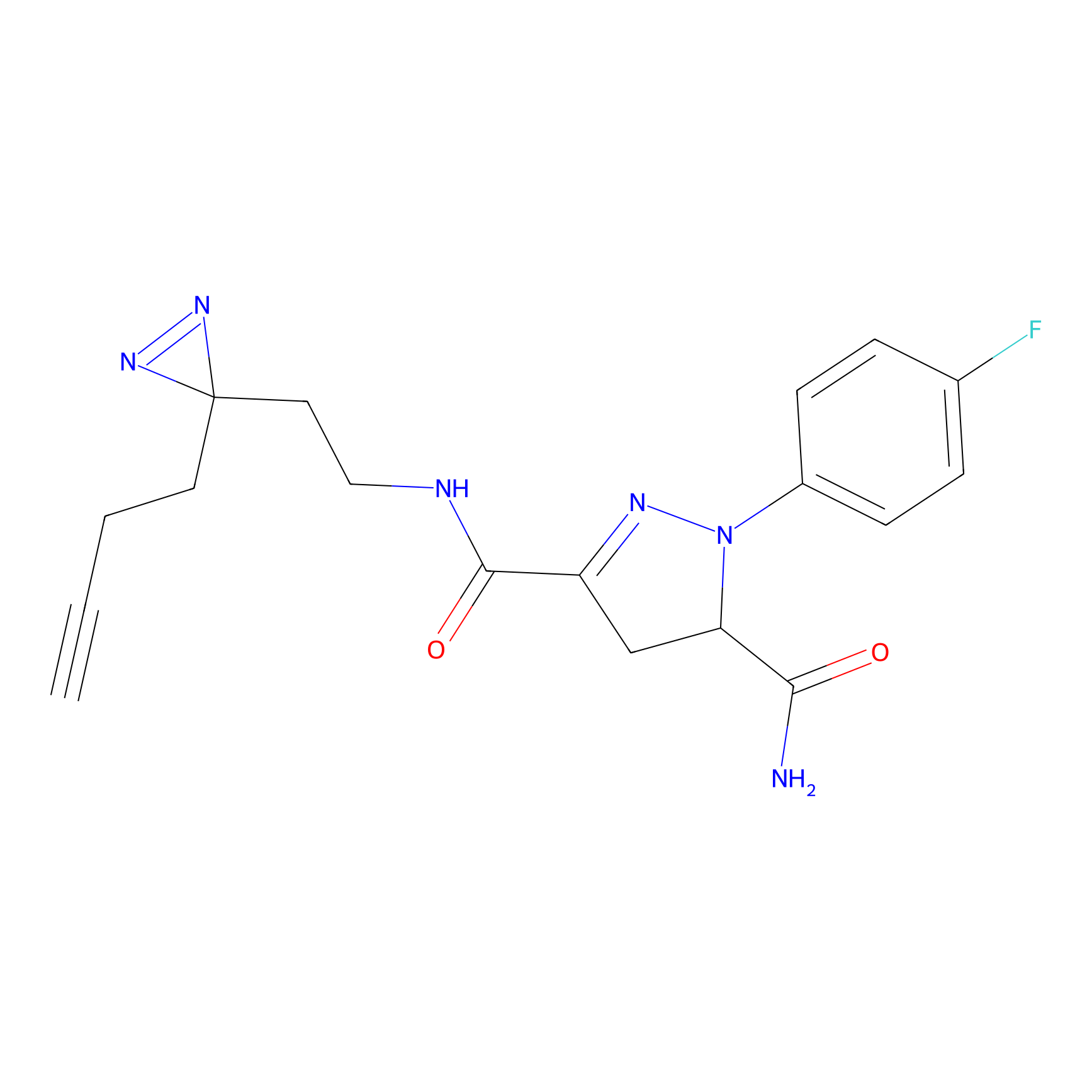
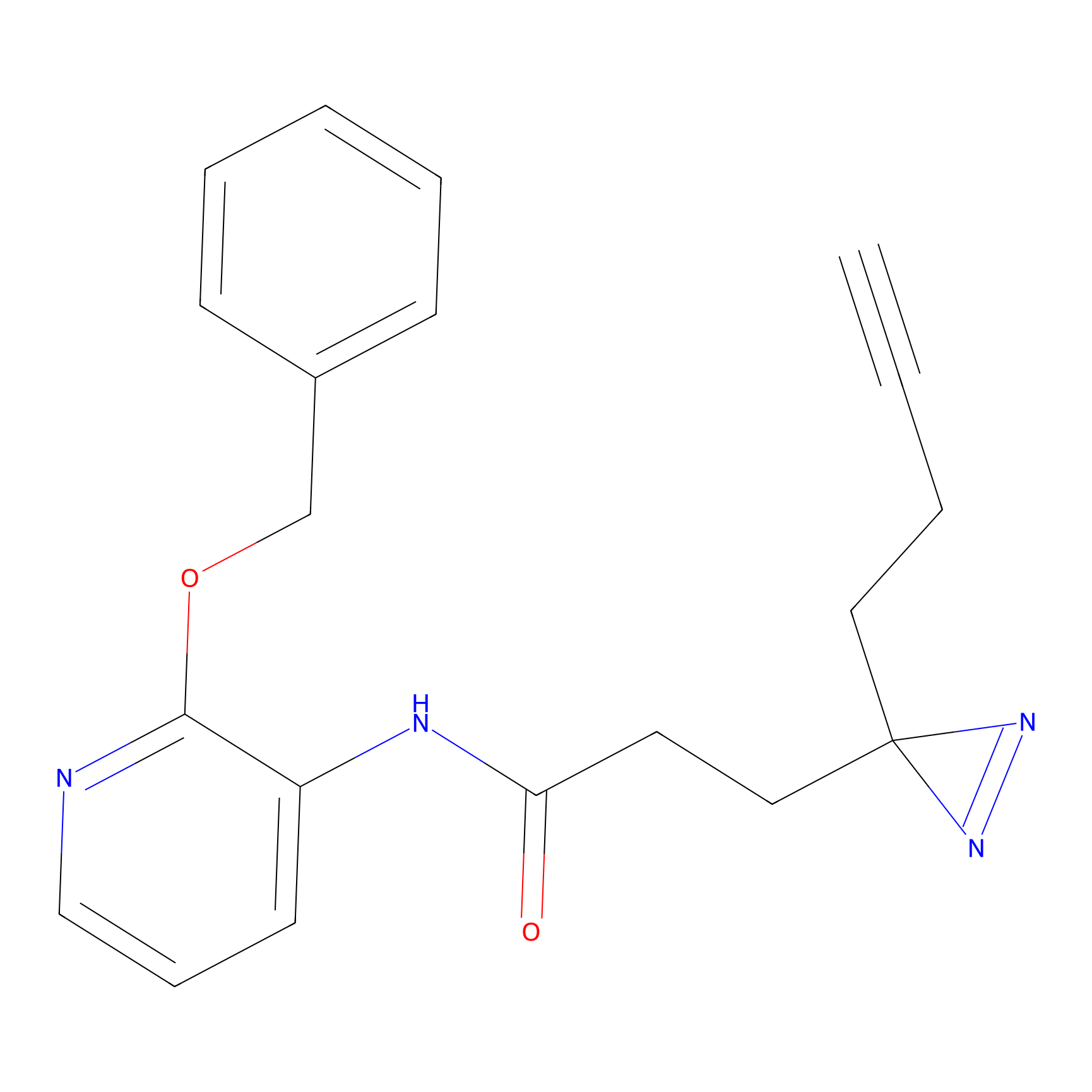
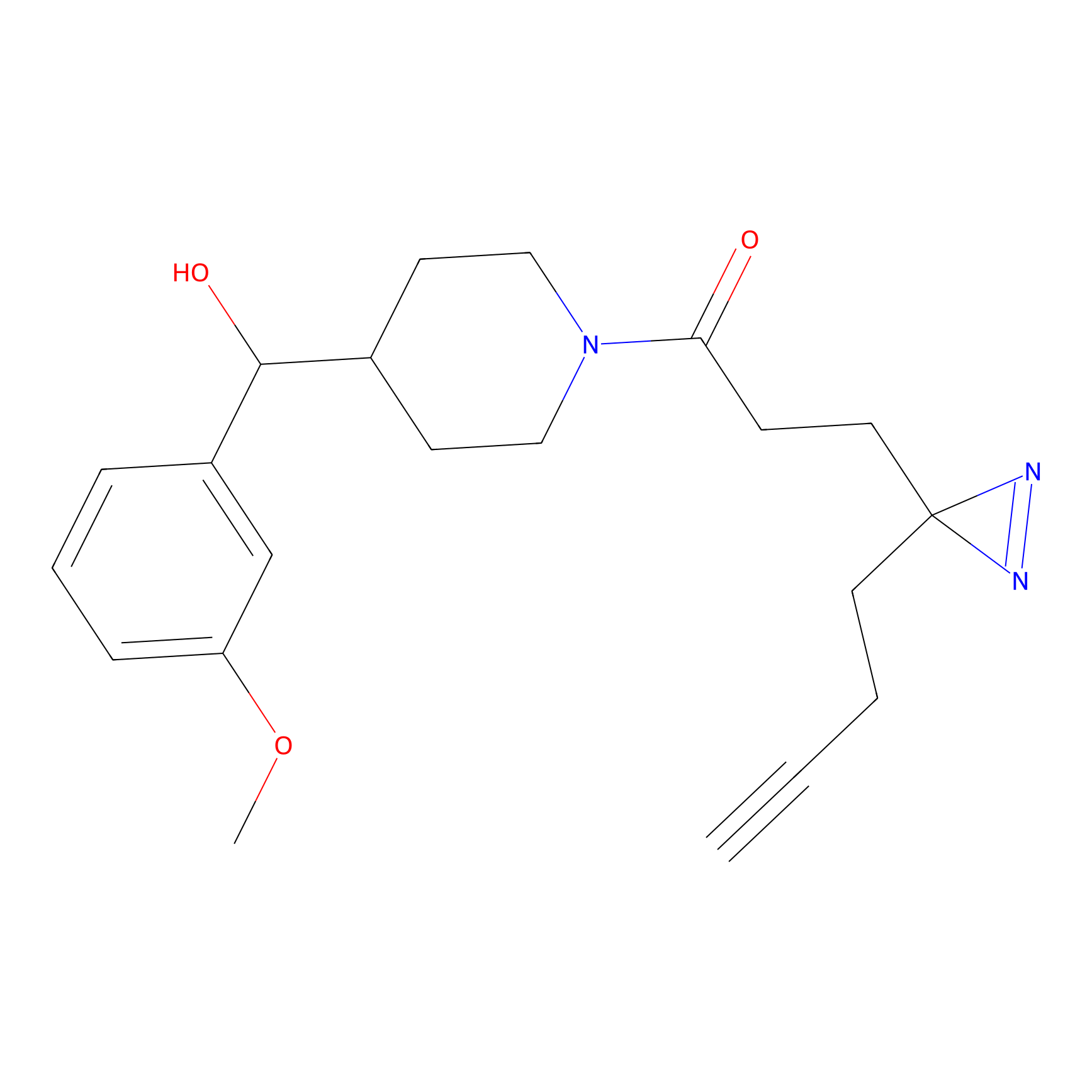
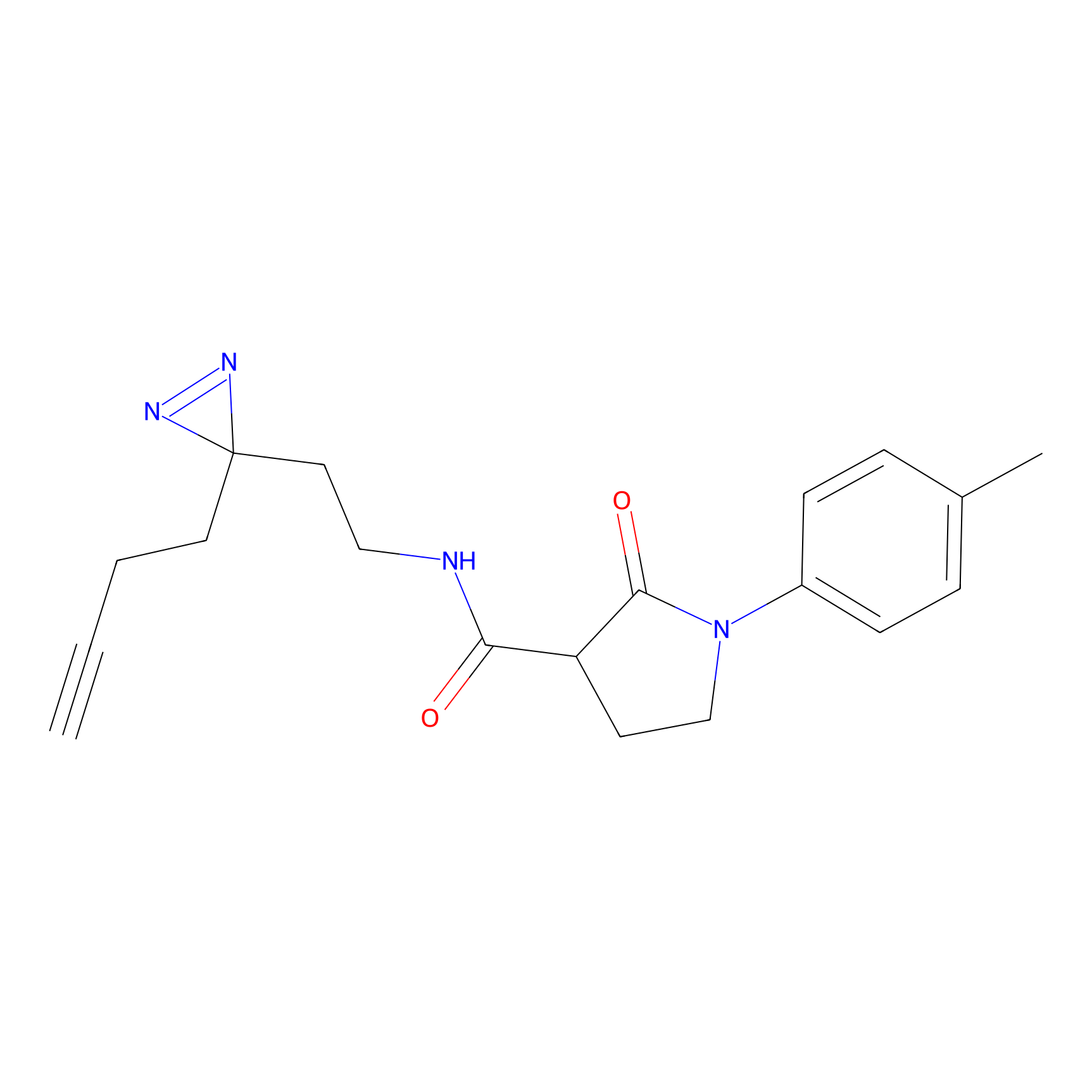
FBPP2 Probe Info |
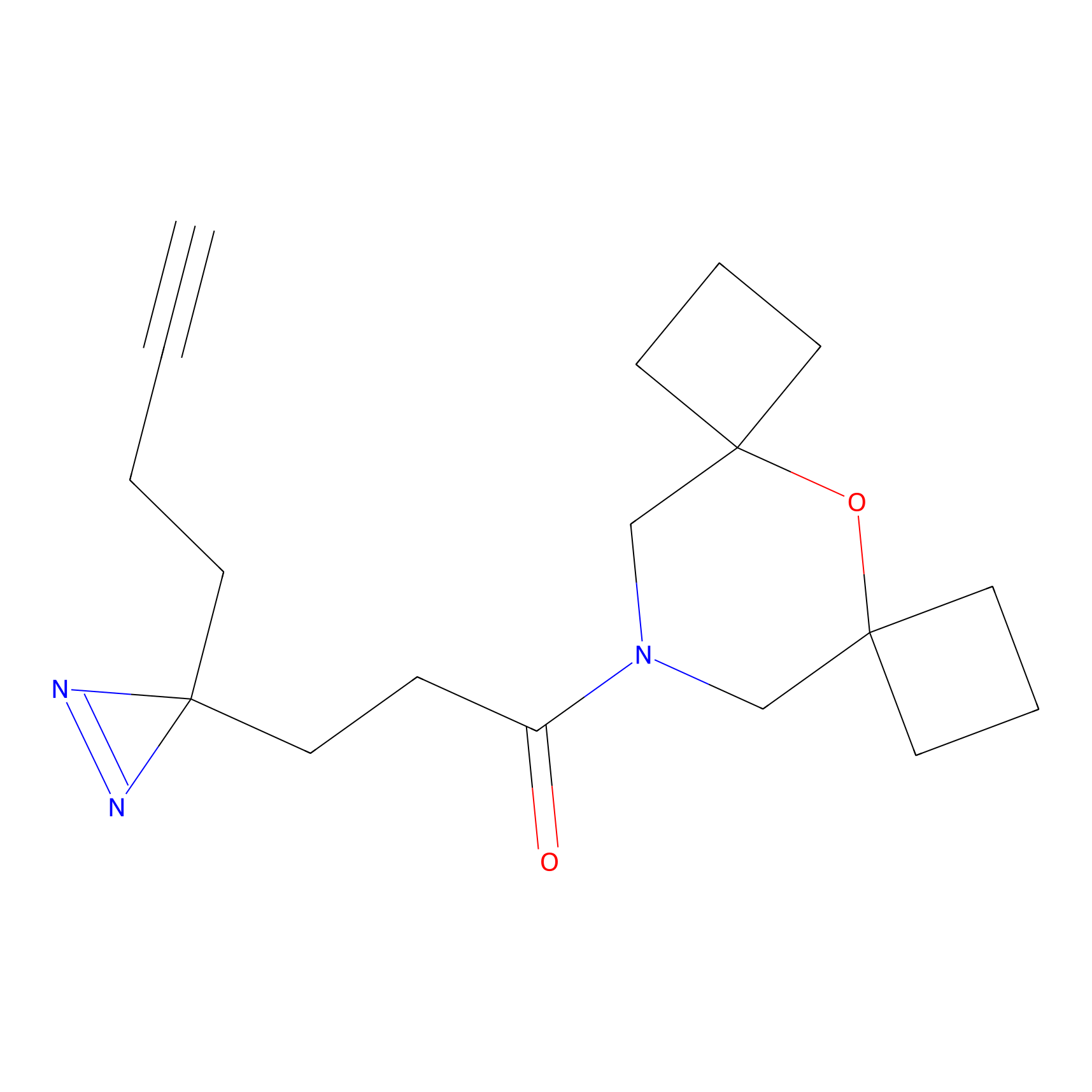
 |
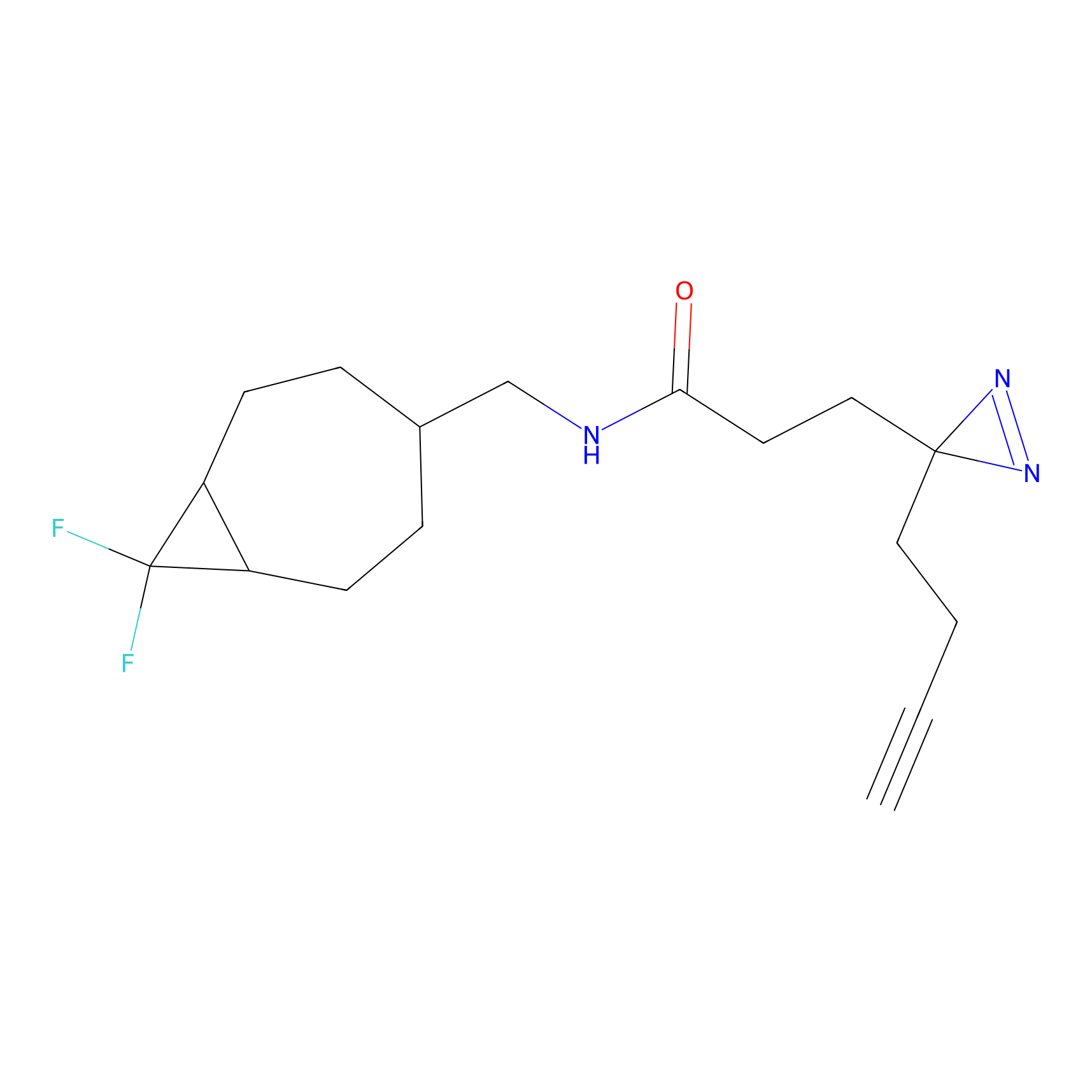
24.62 | LDD0318 | [1] | |
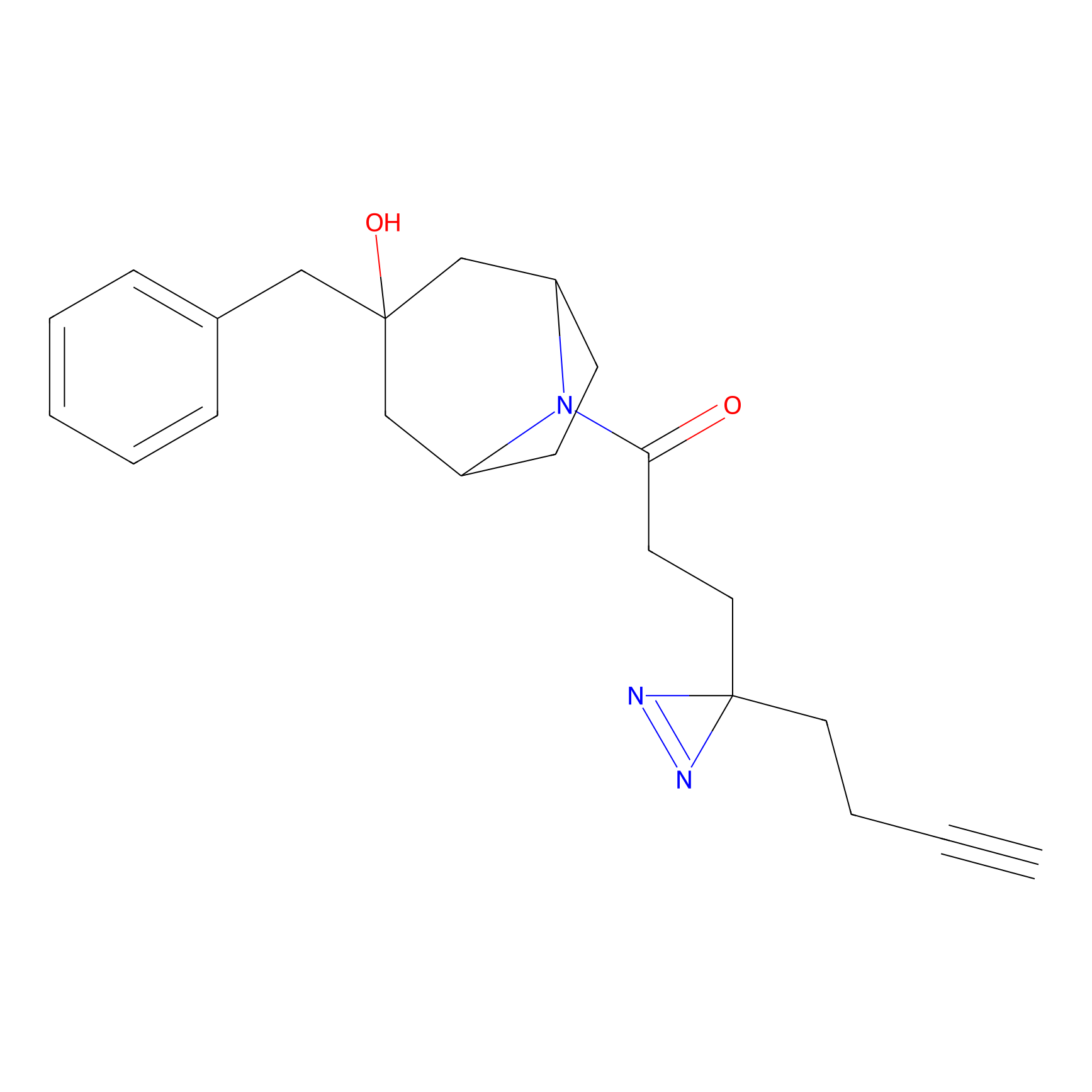
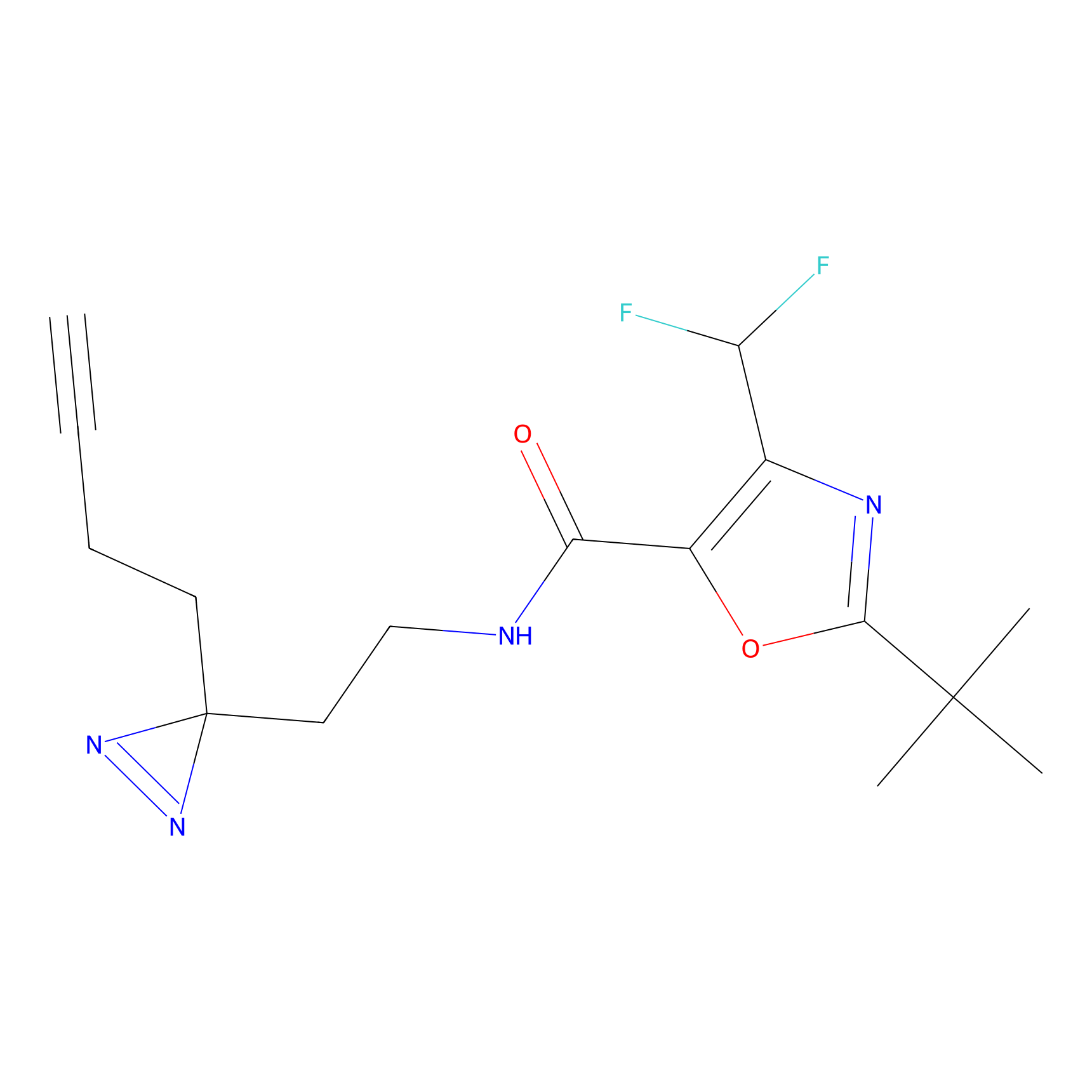
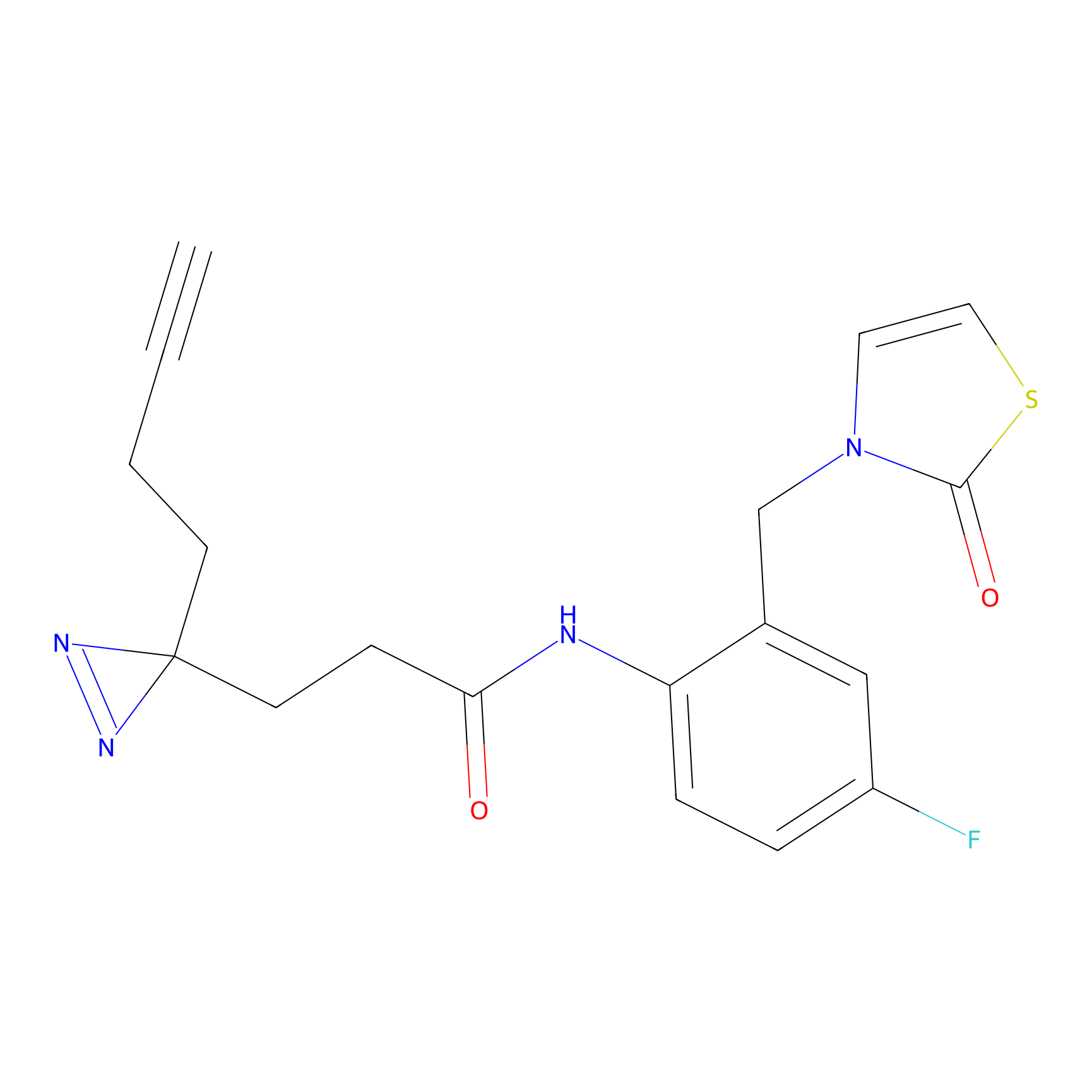
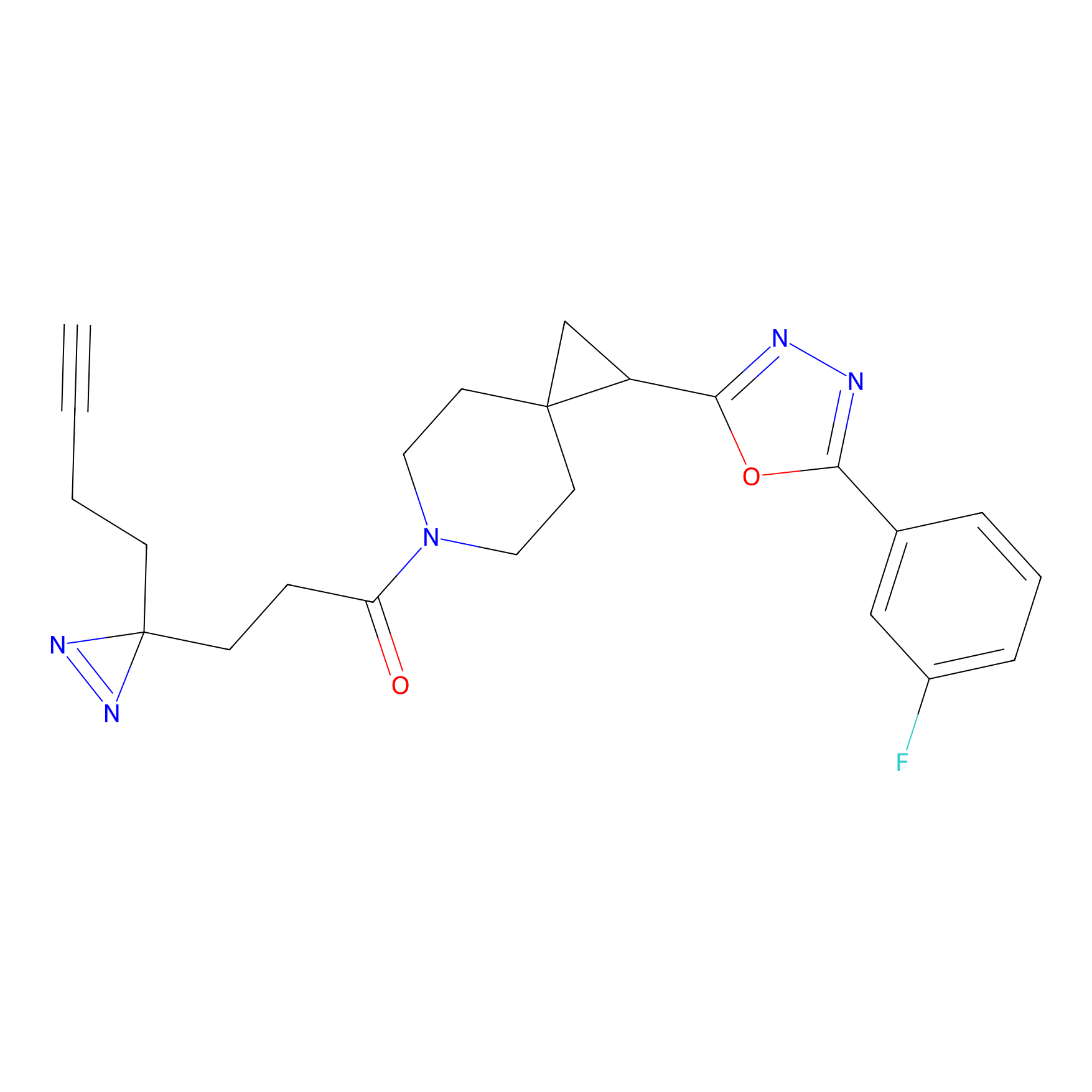
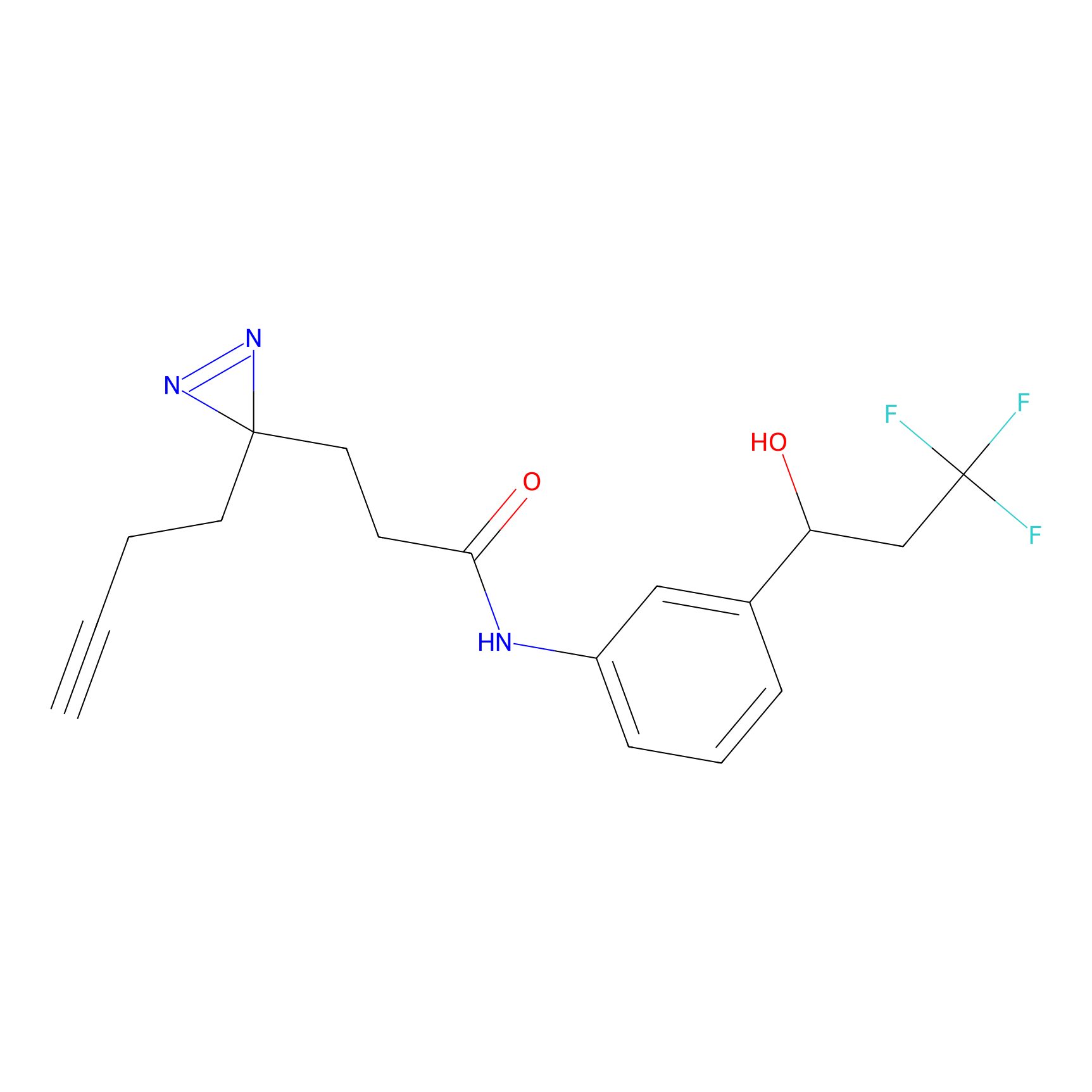
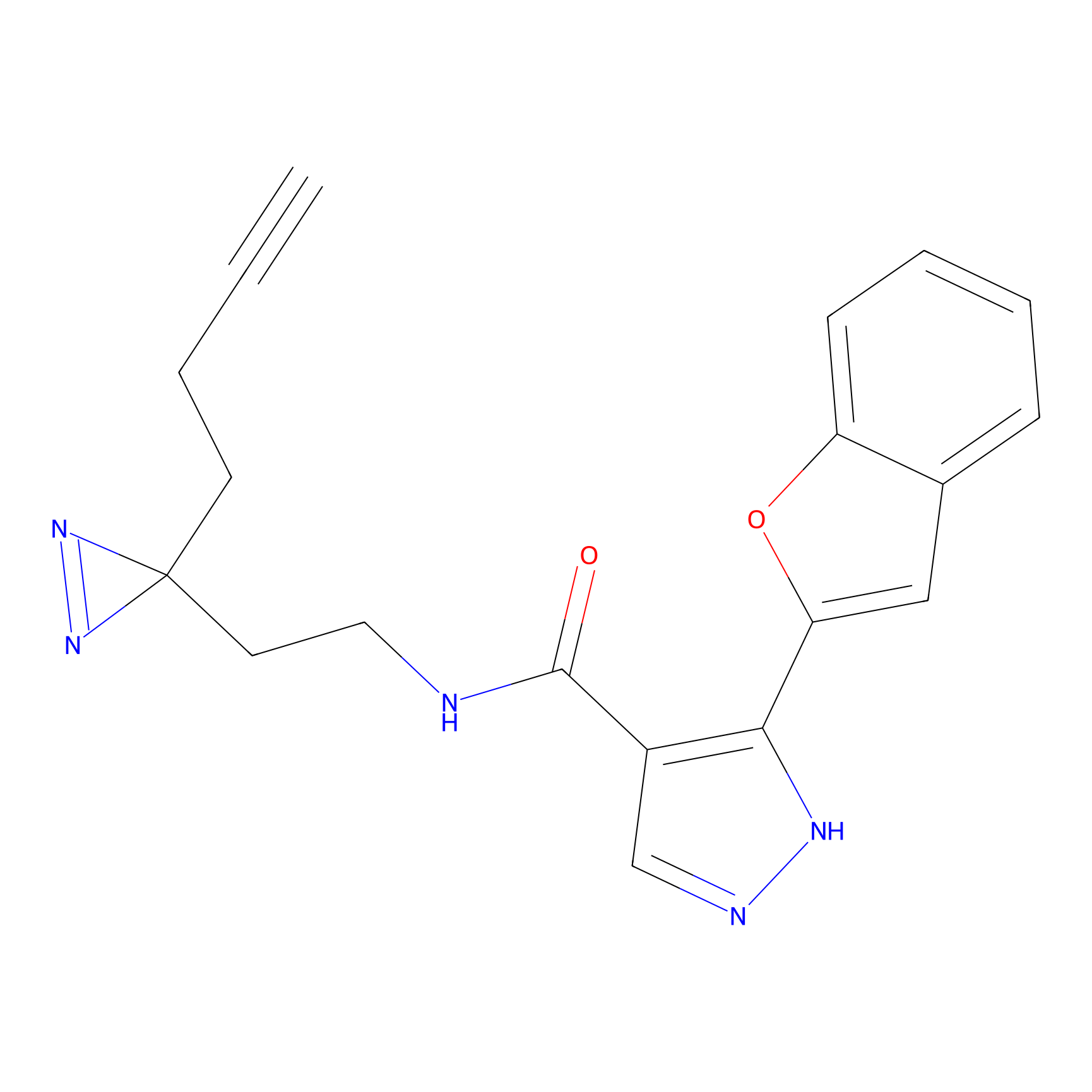
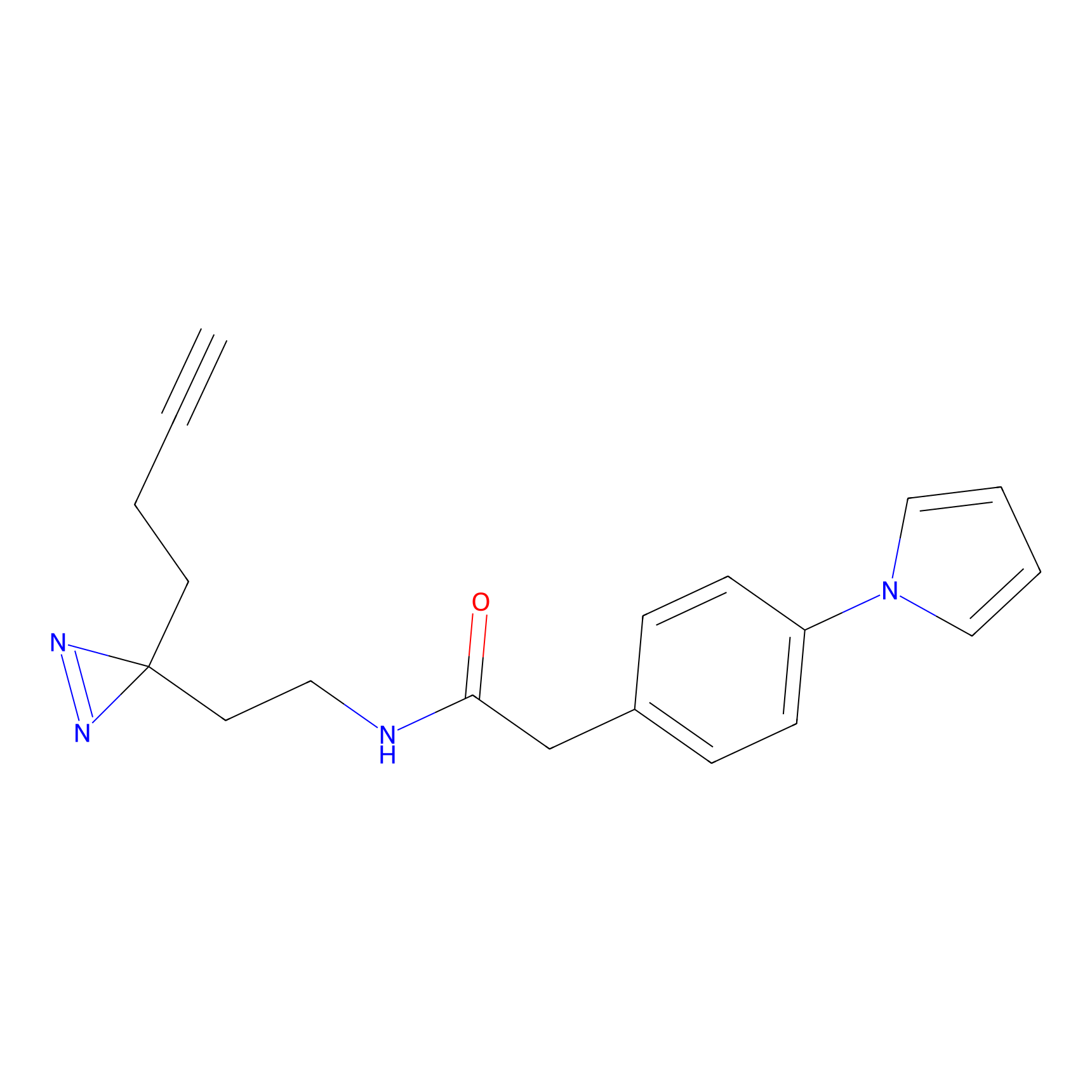
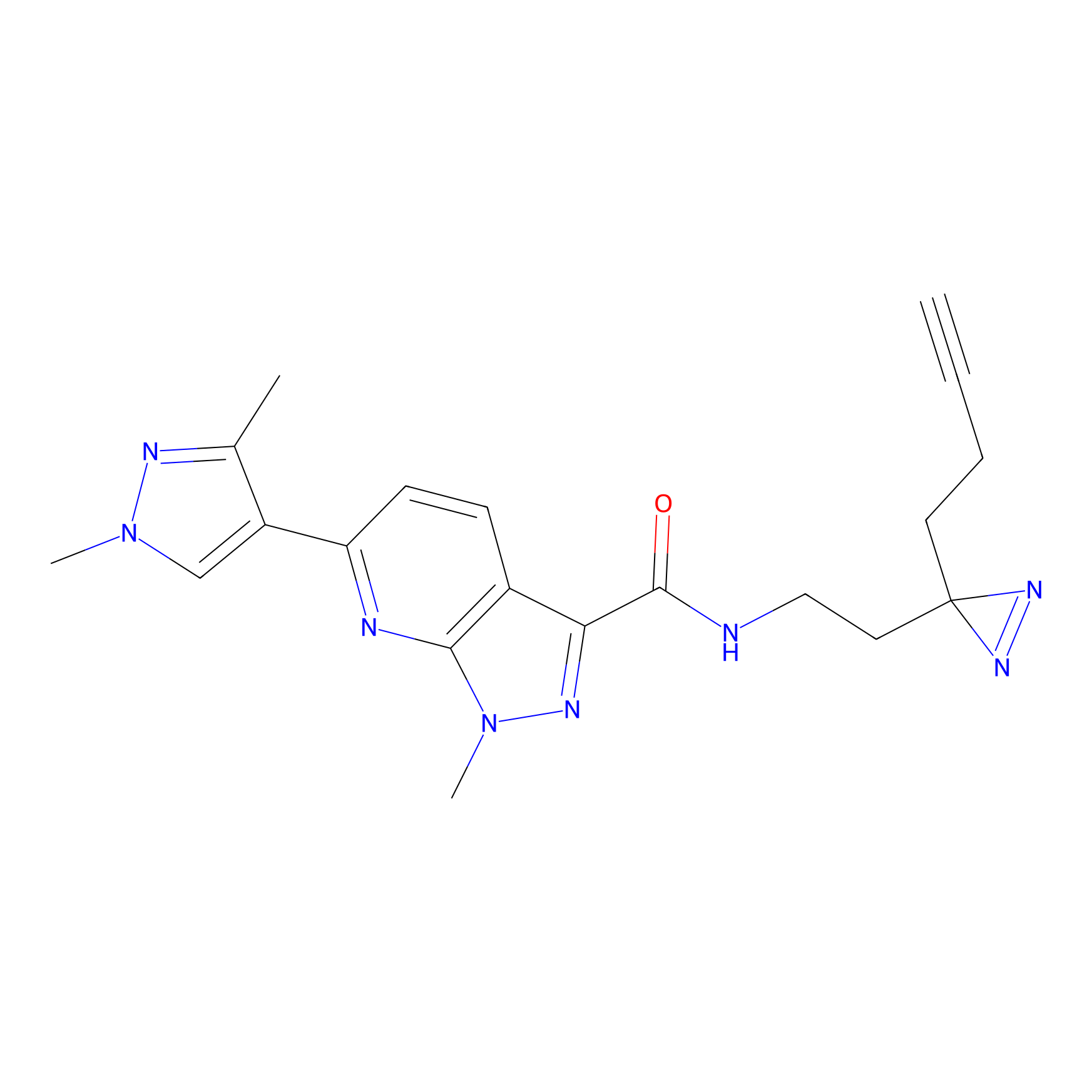
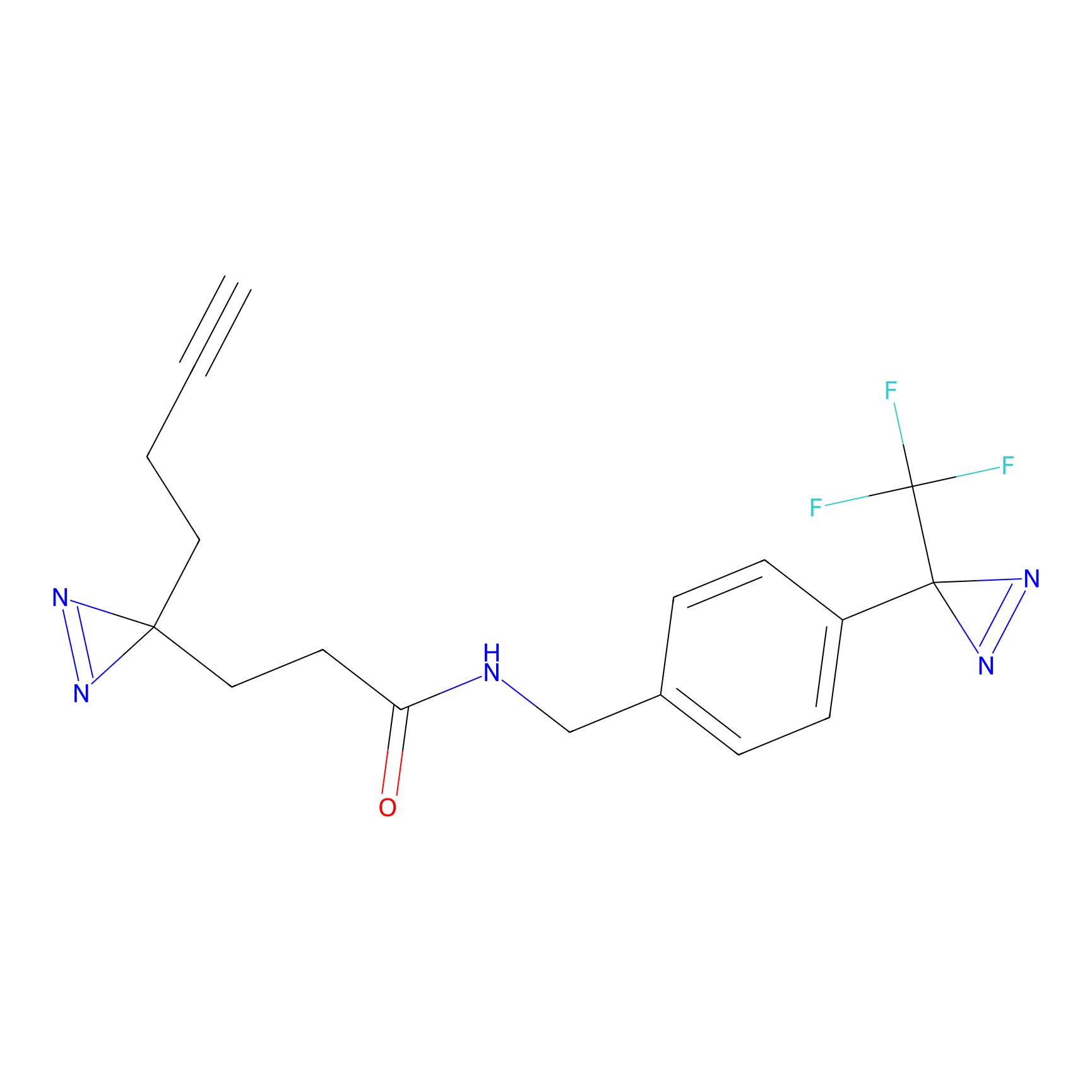
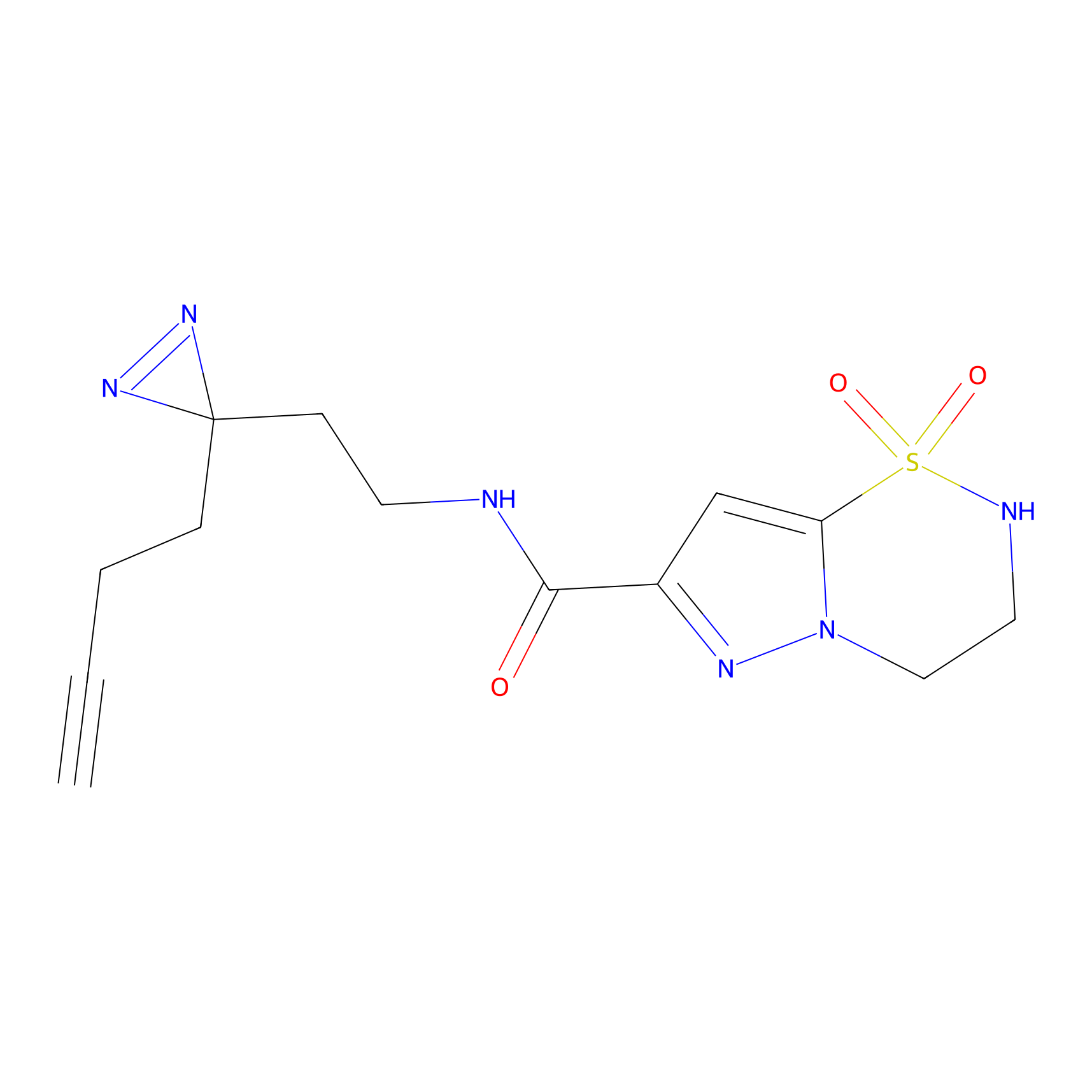
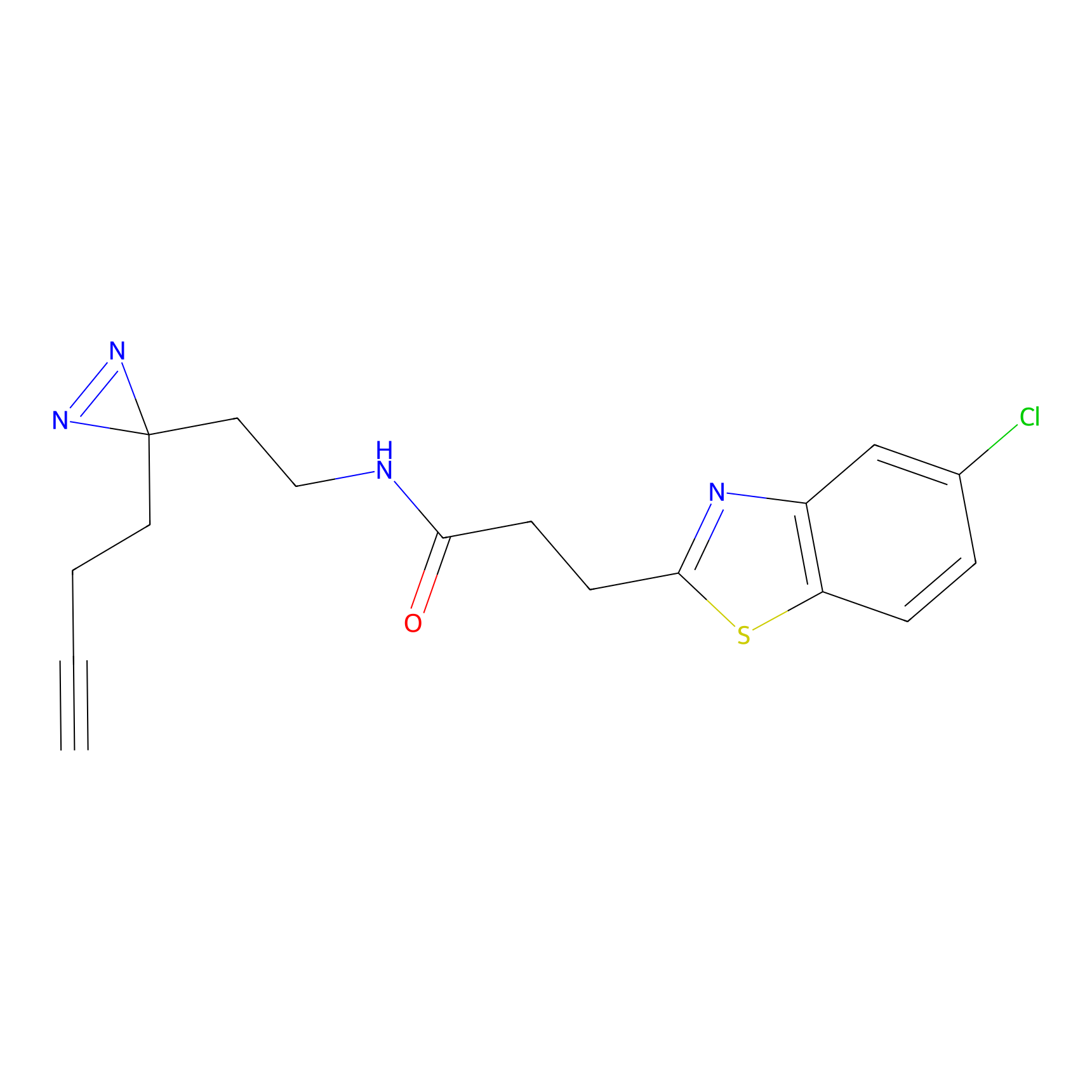
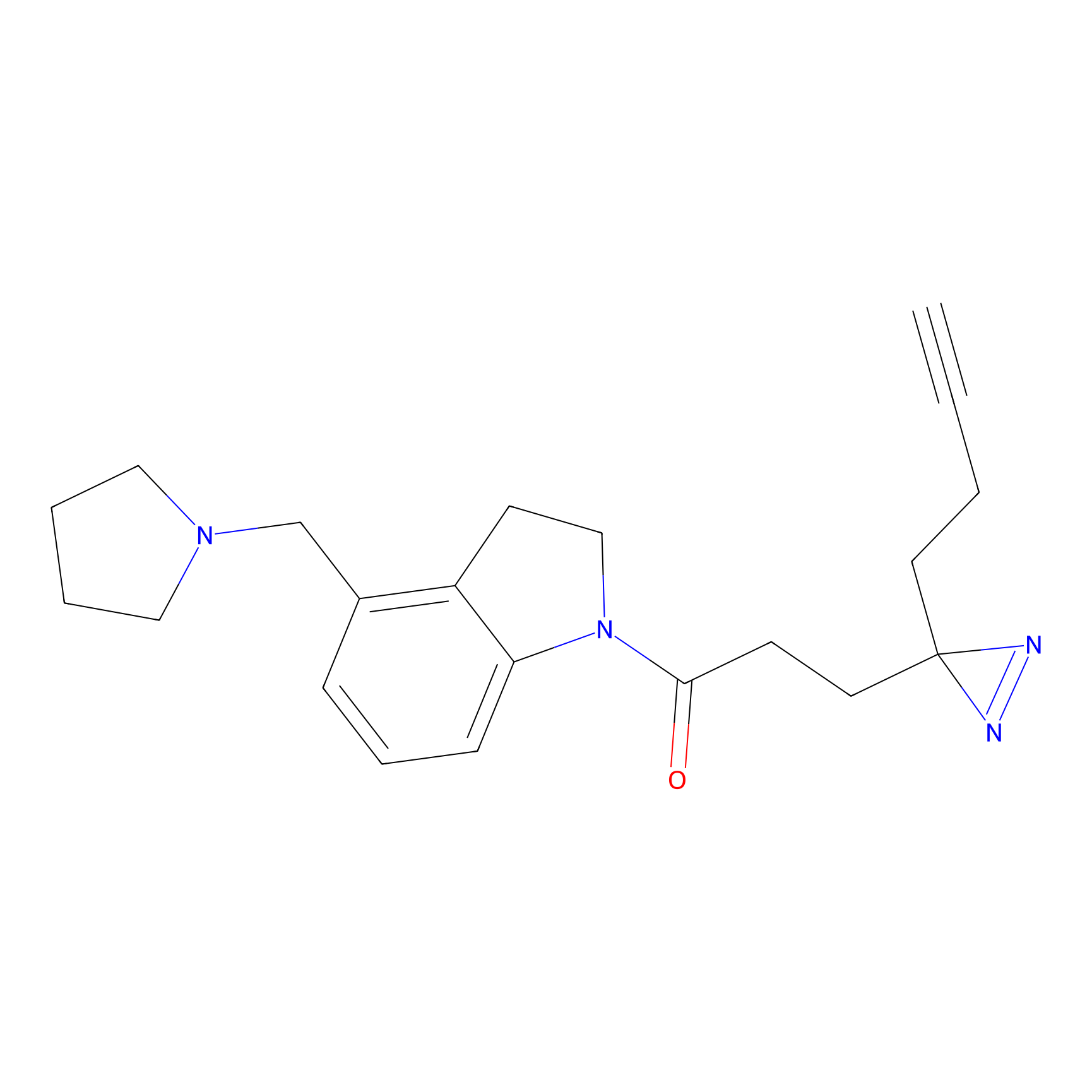
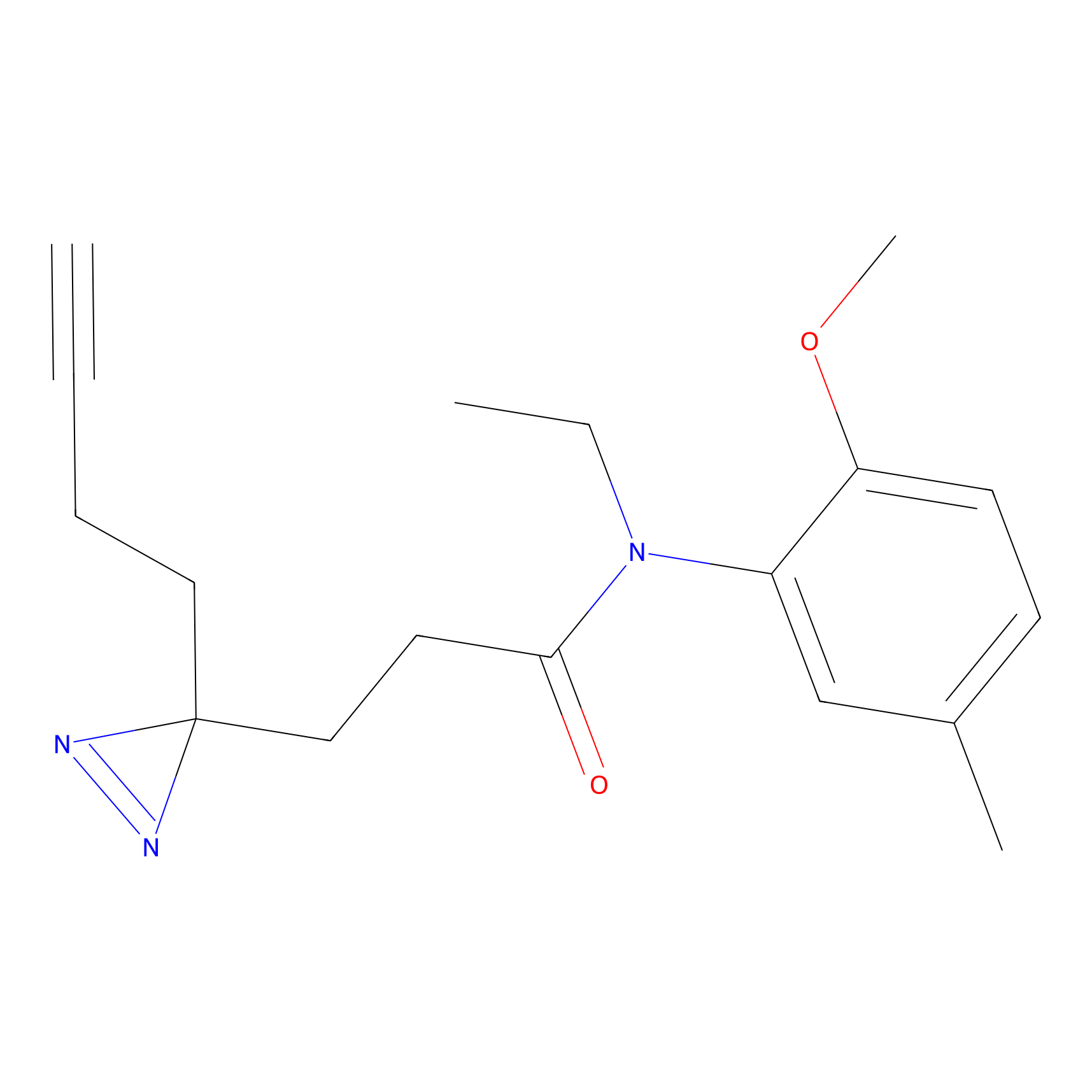
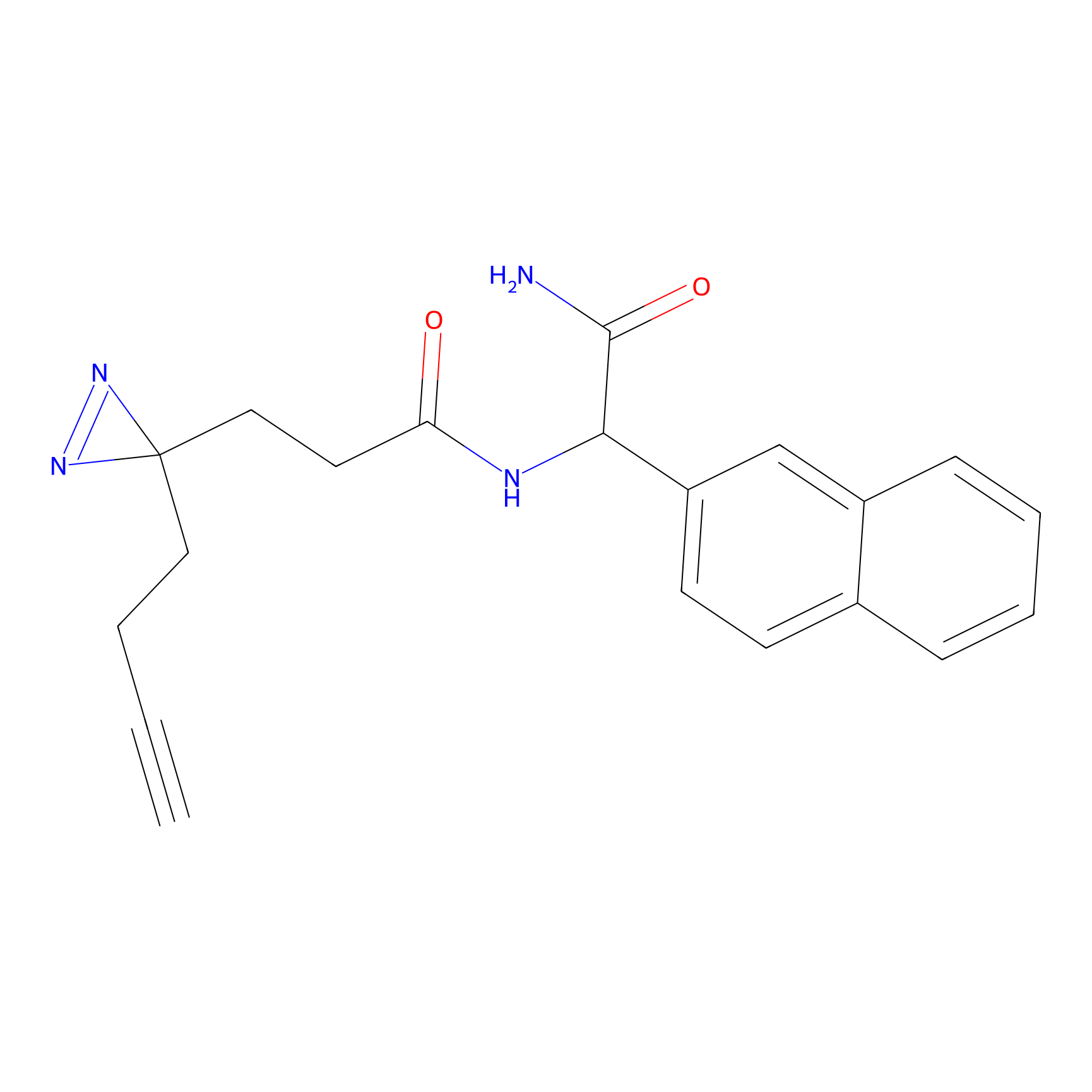
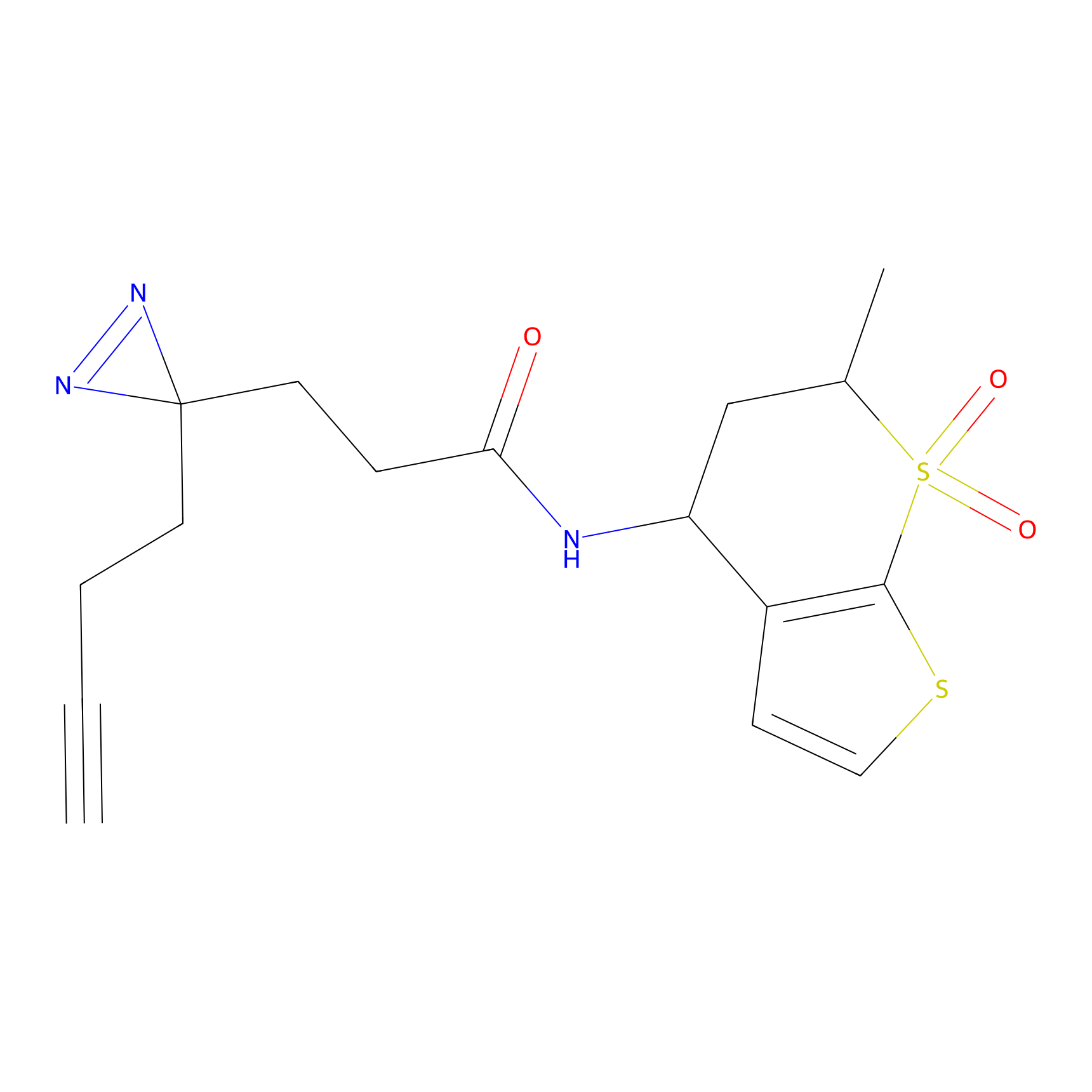
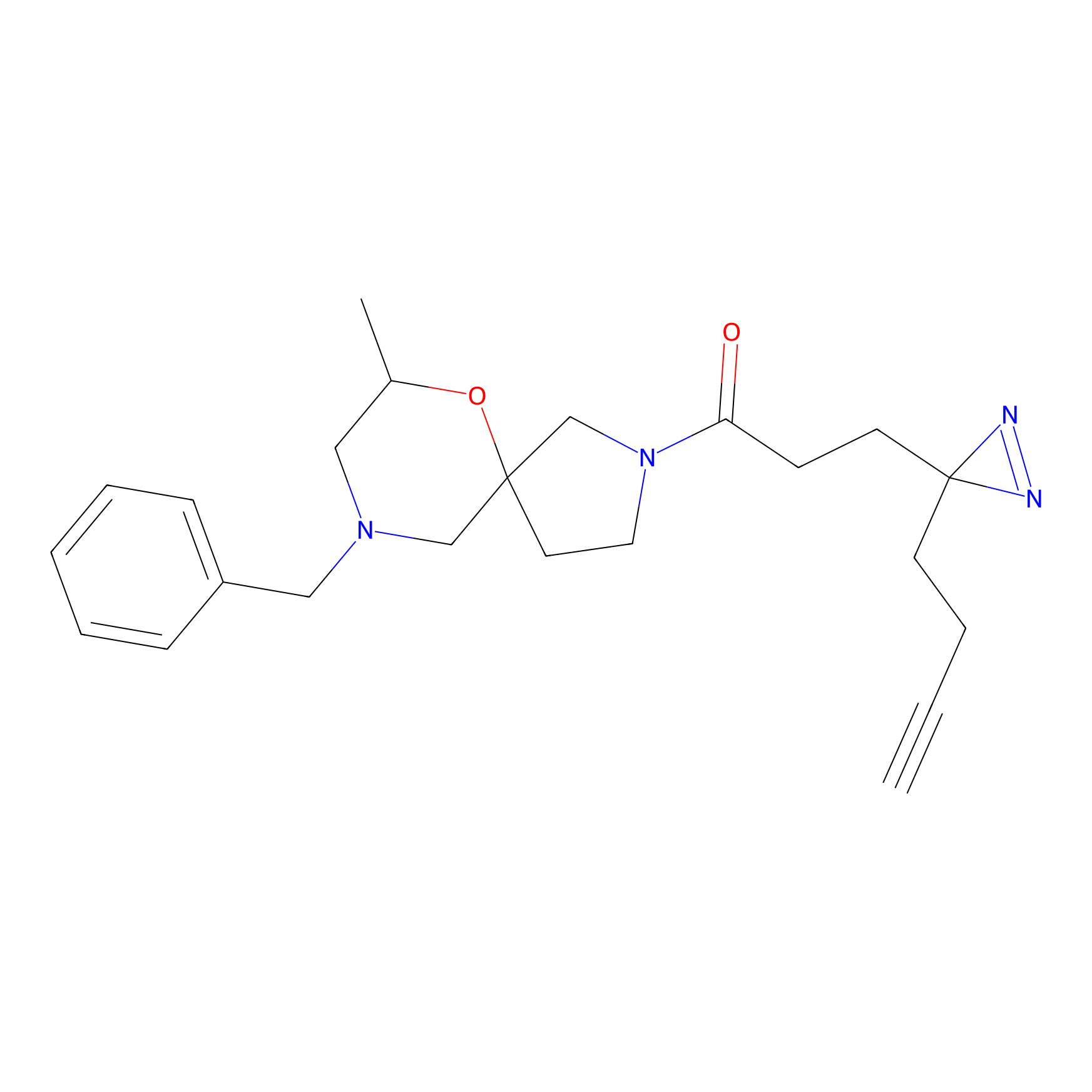
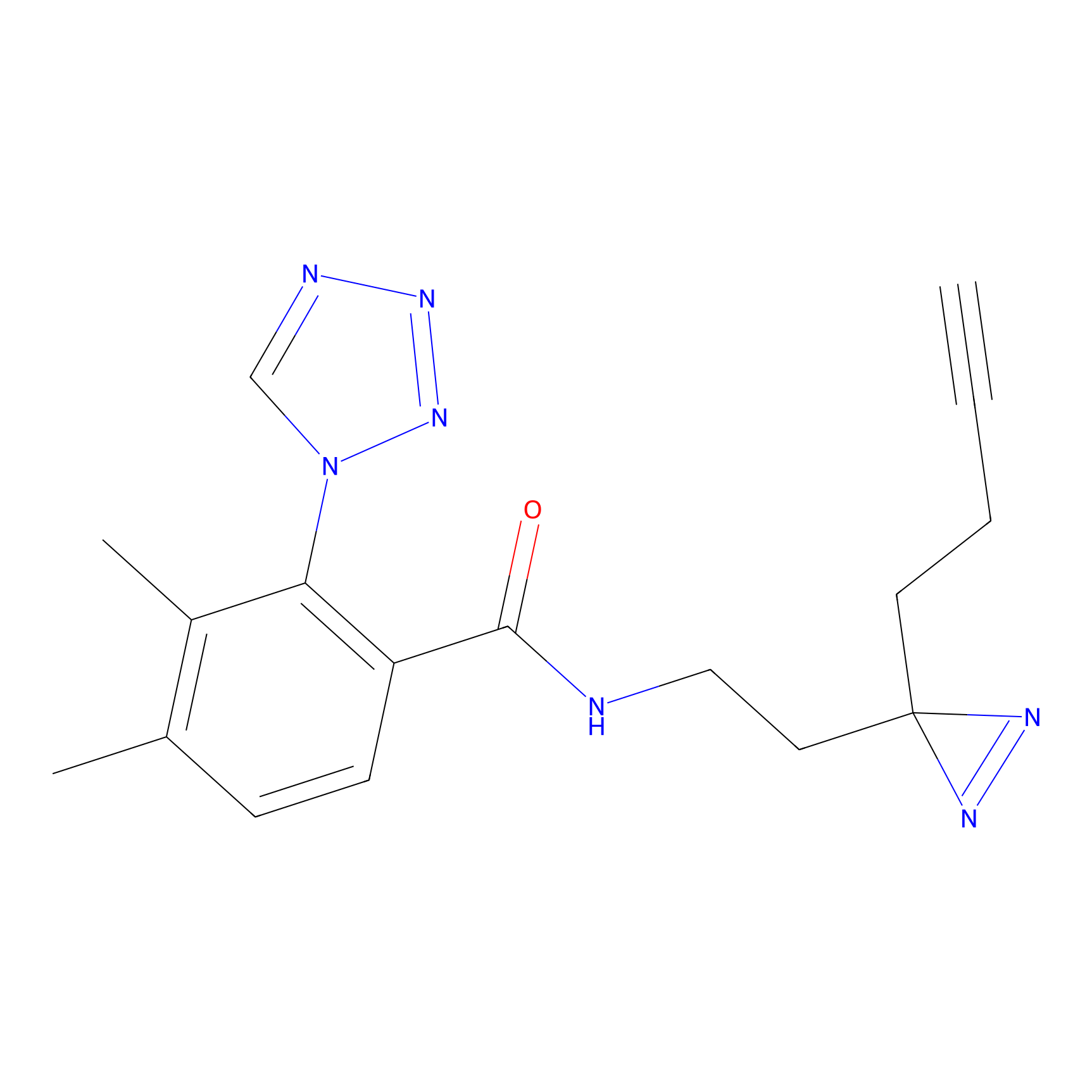
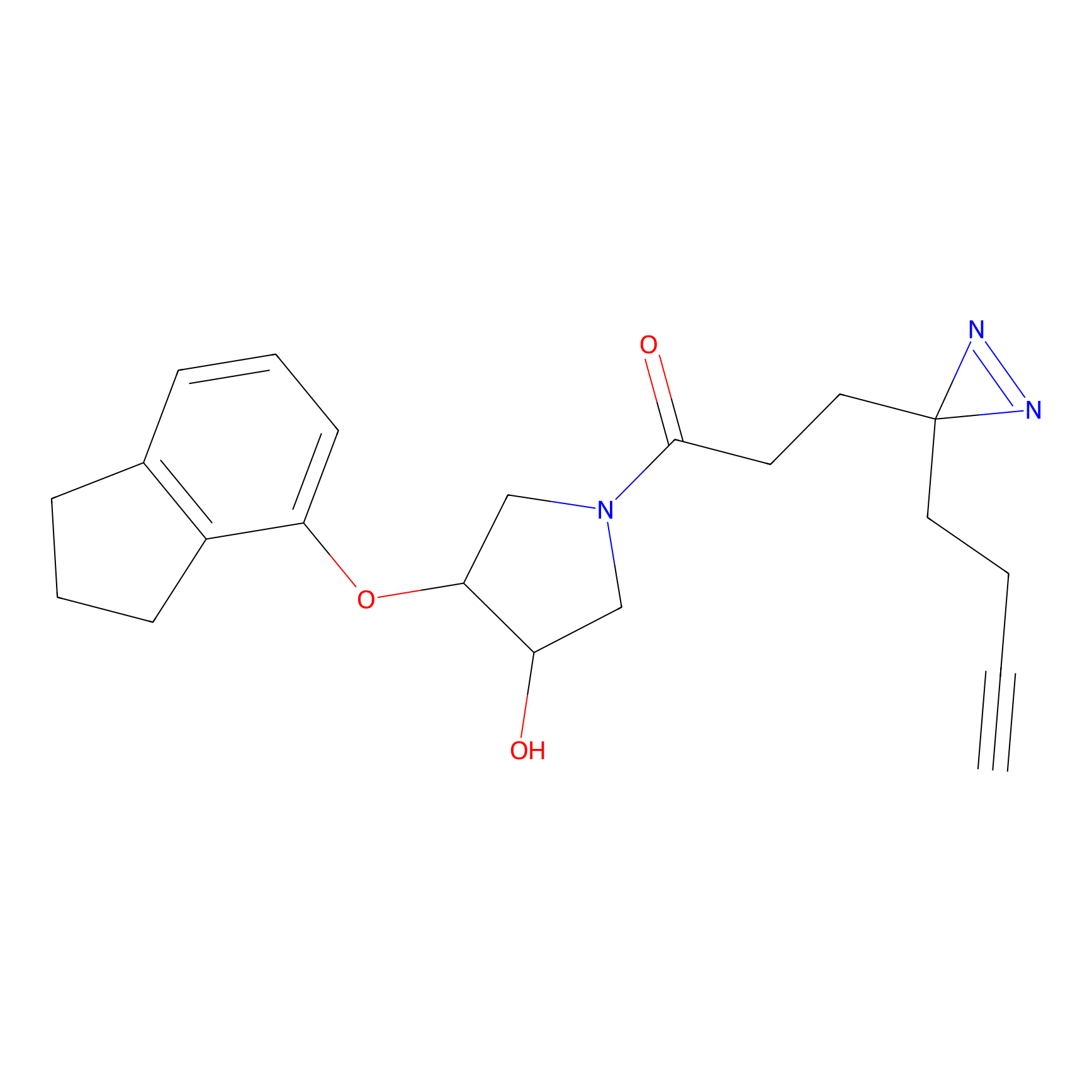
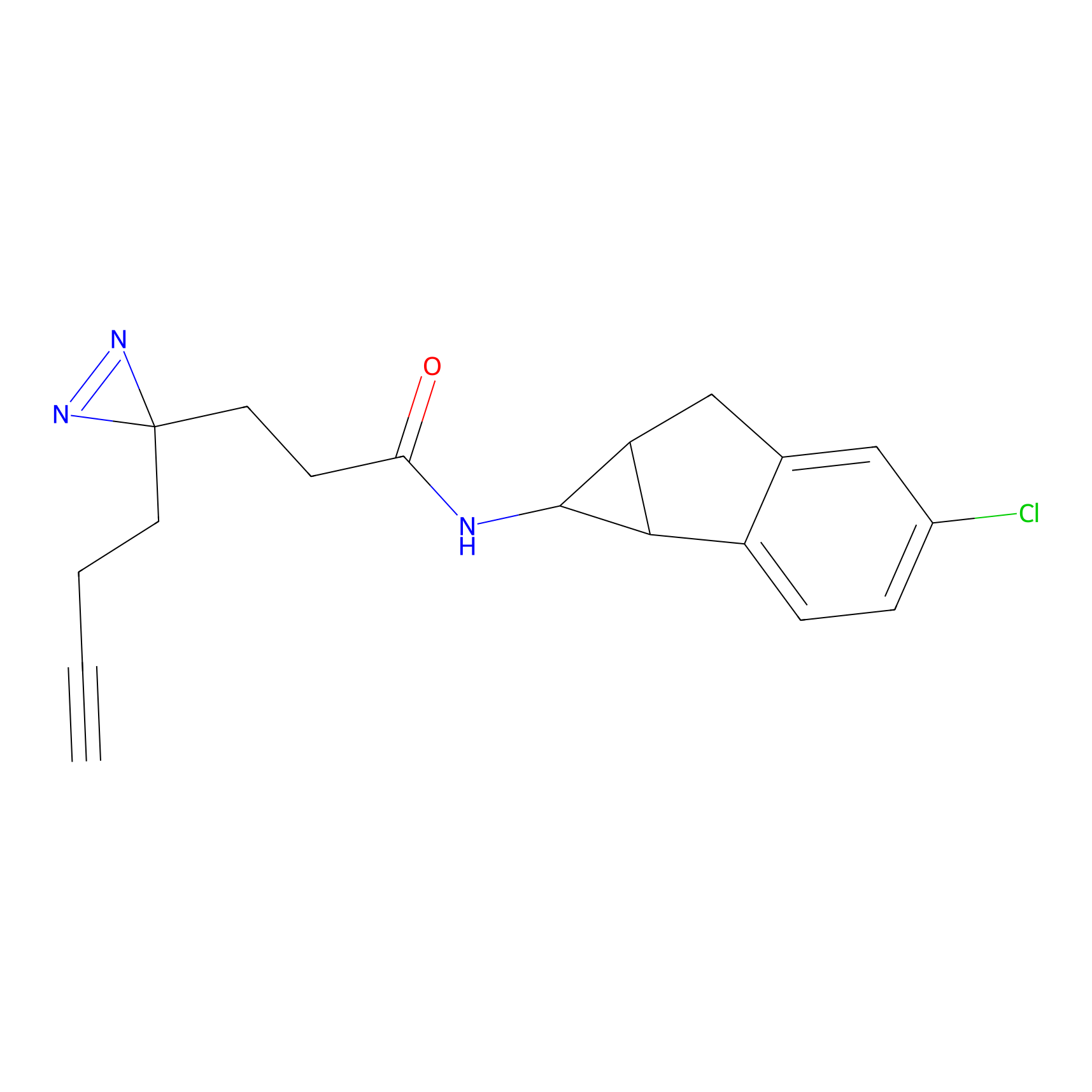
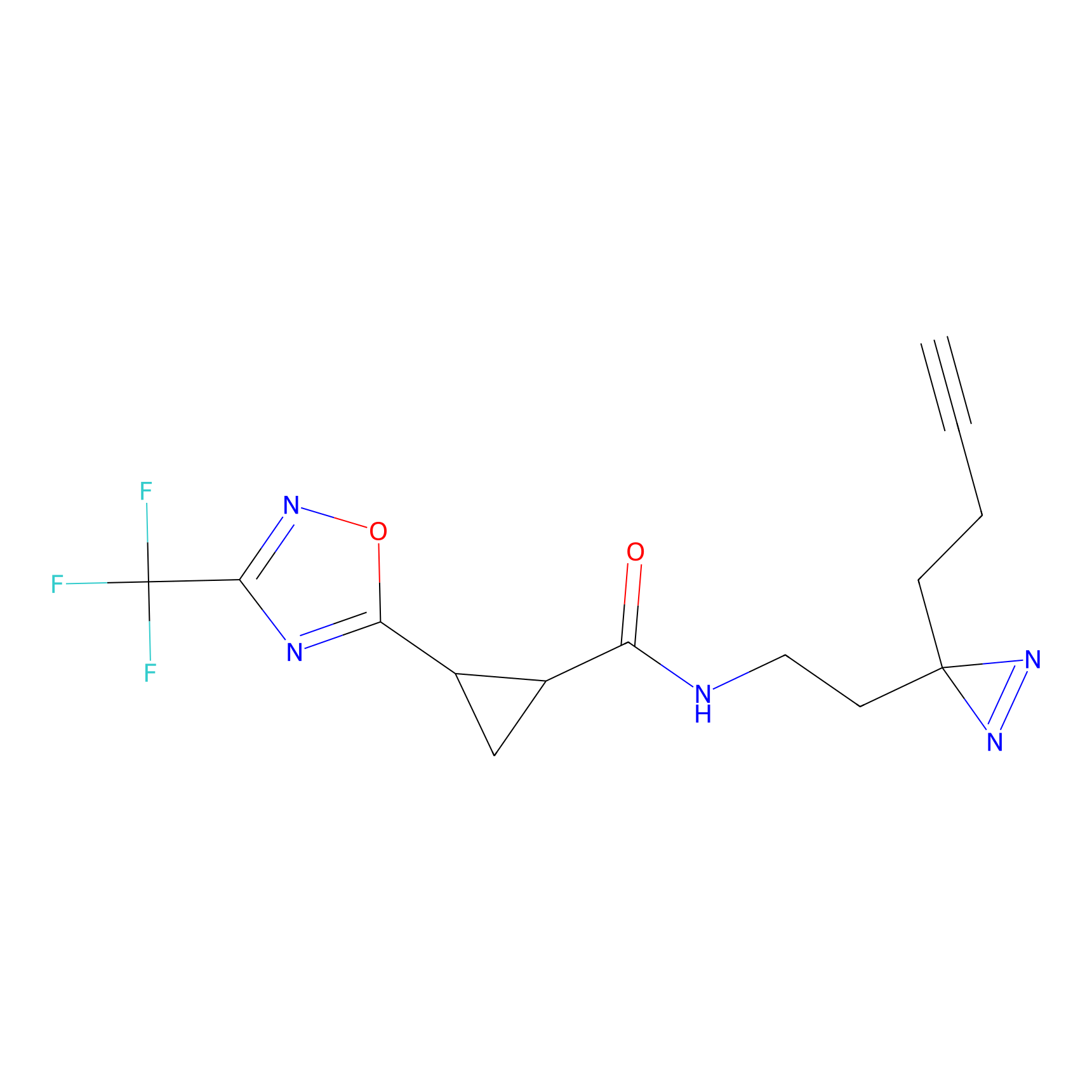
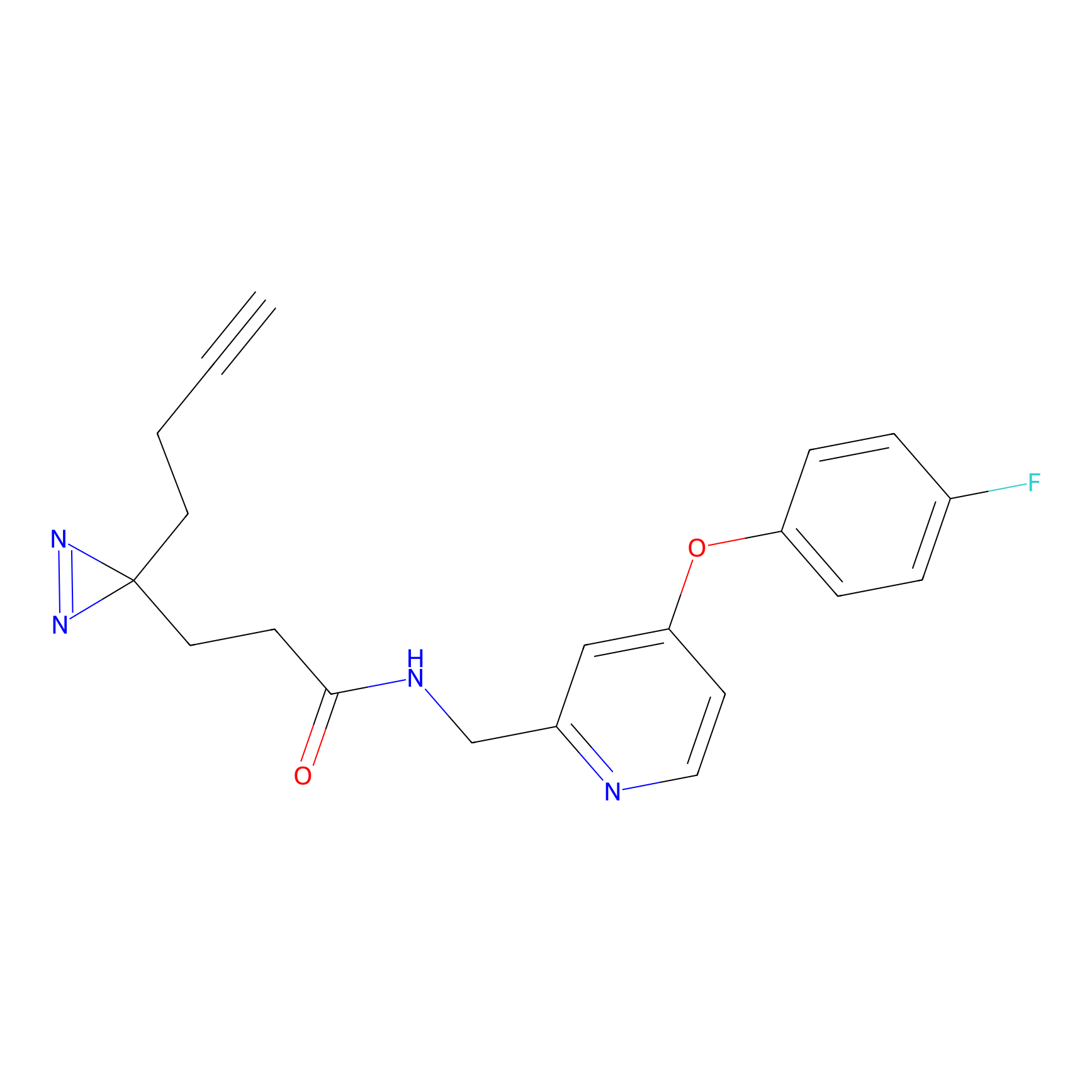
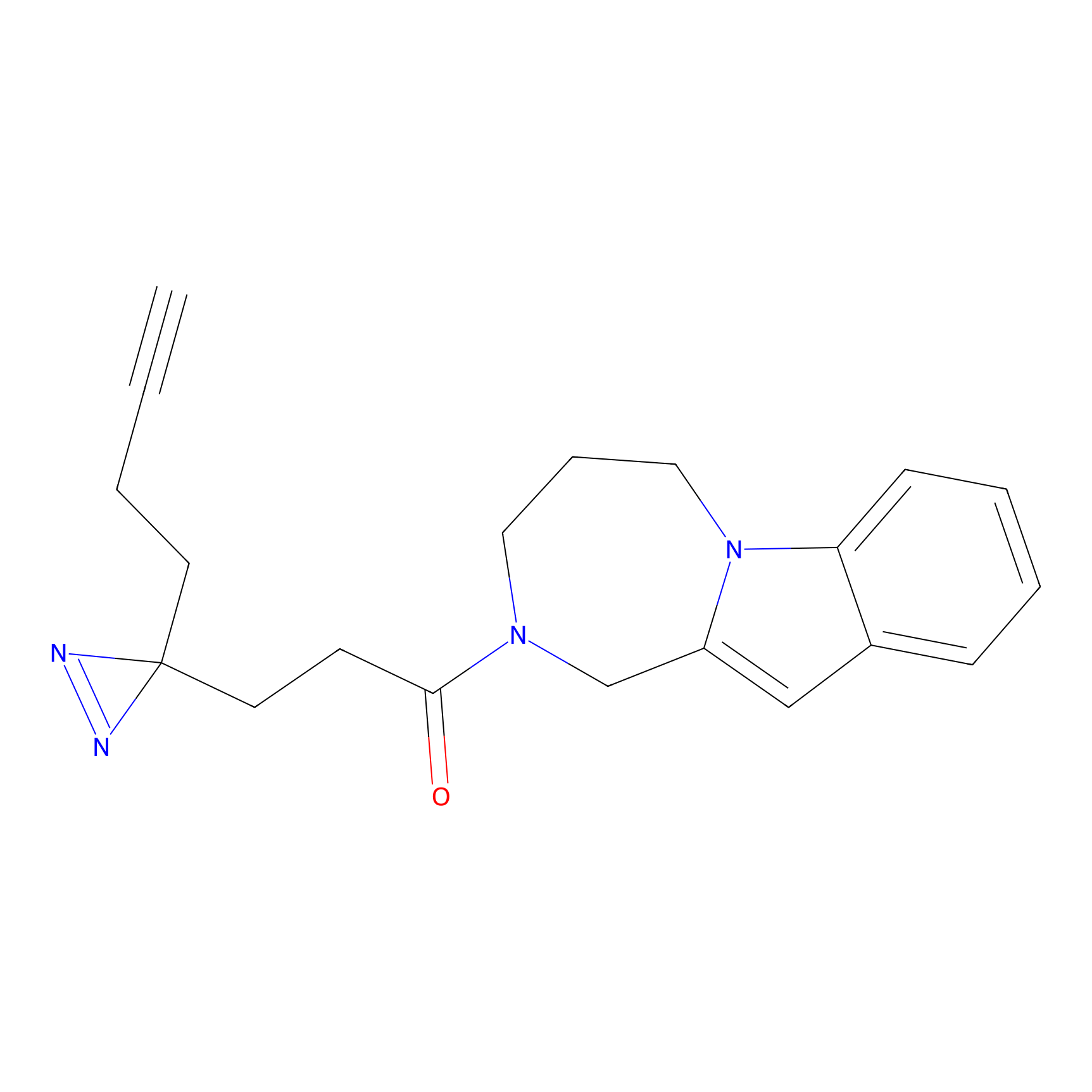
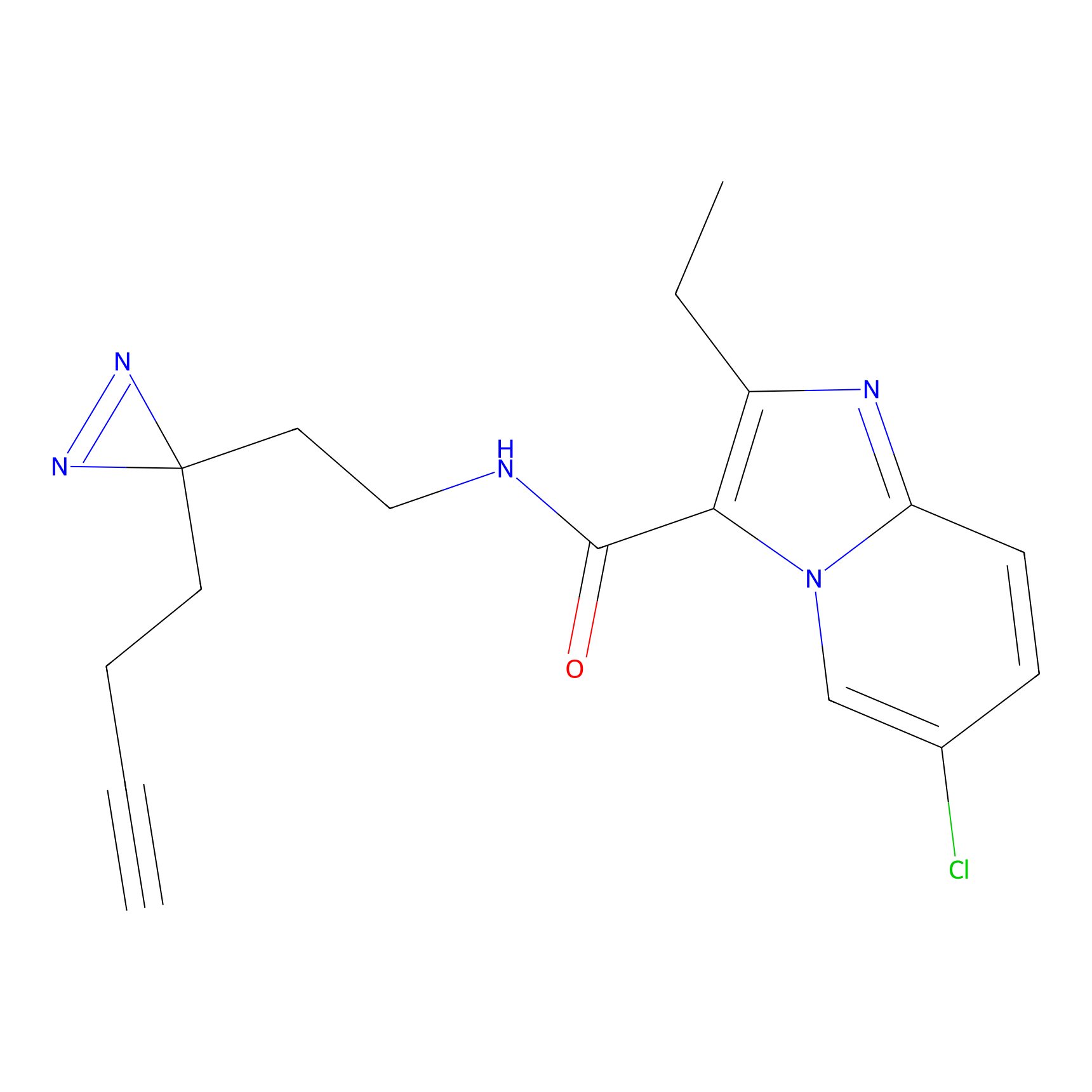
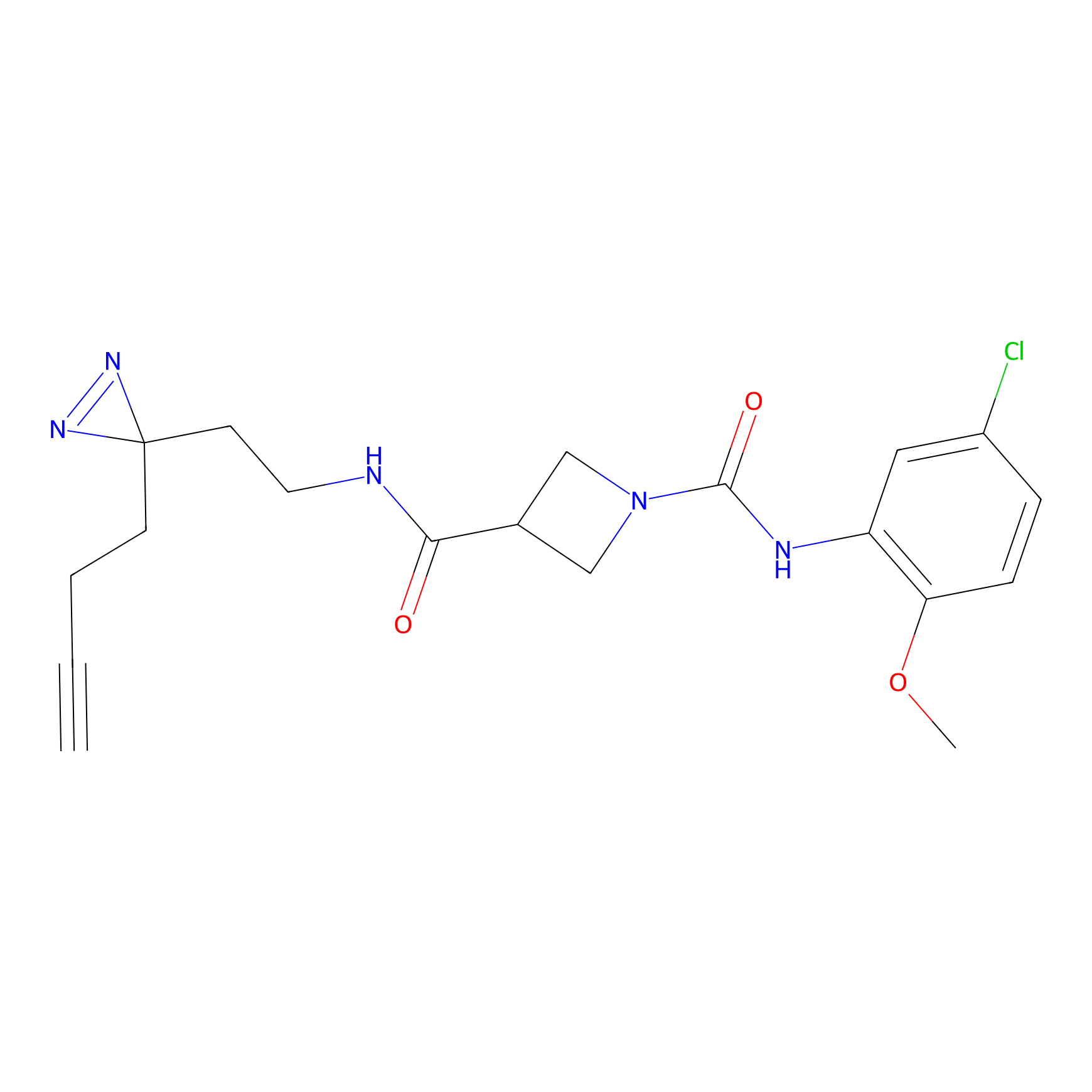
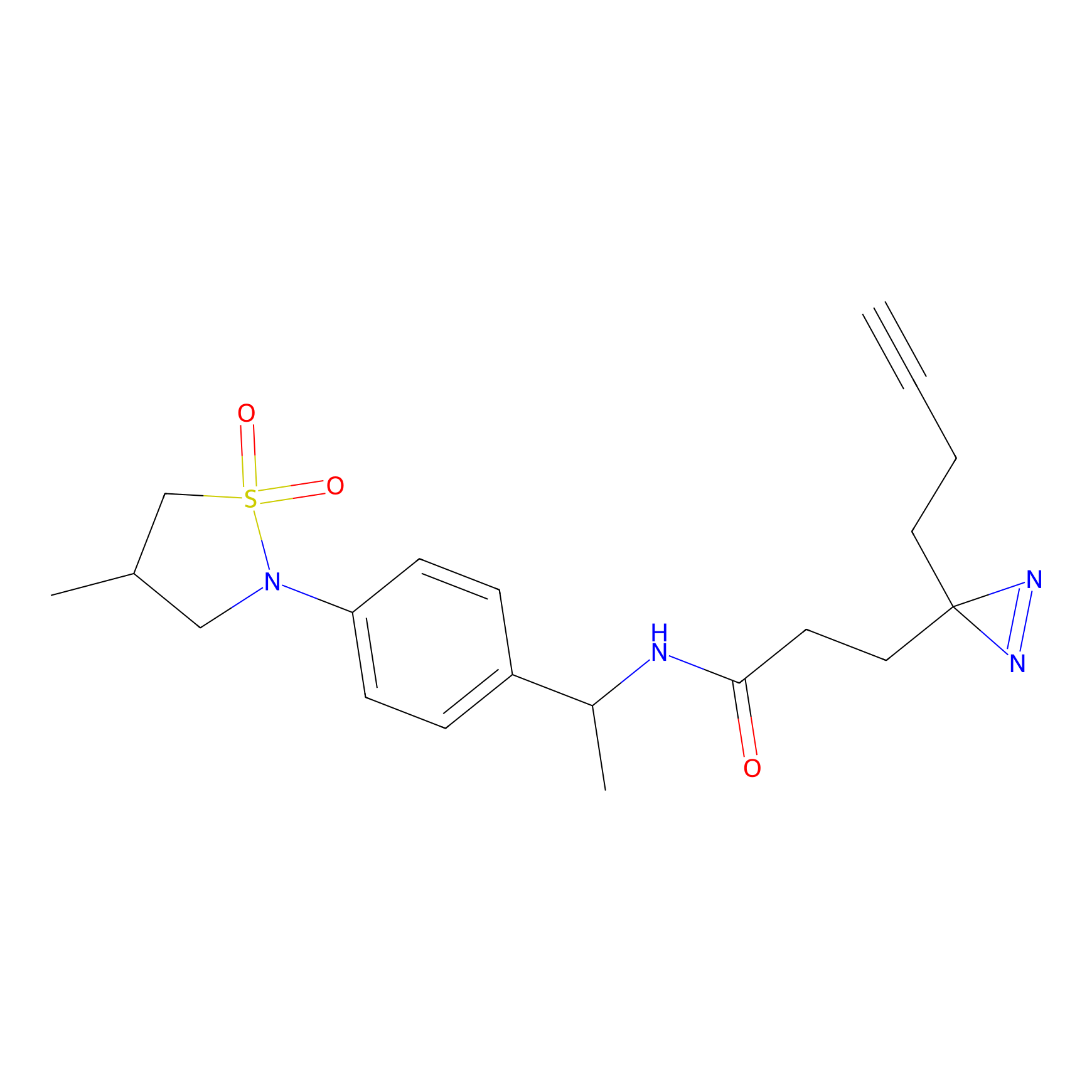
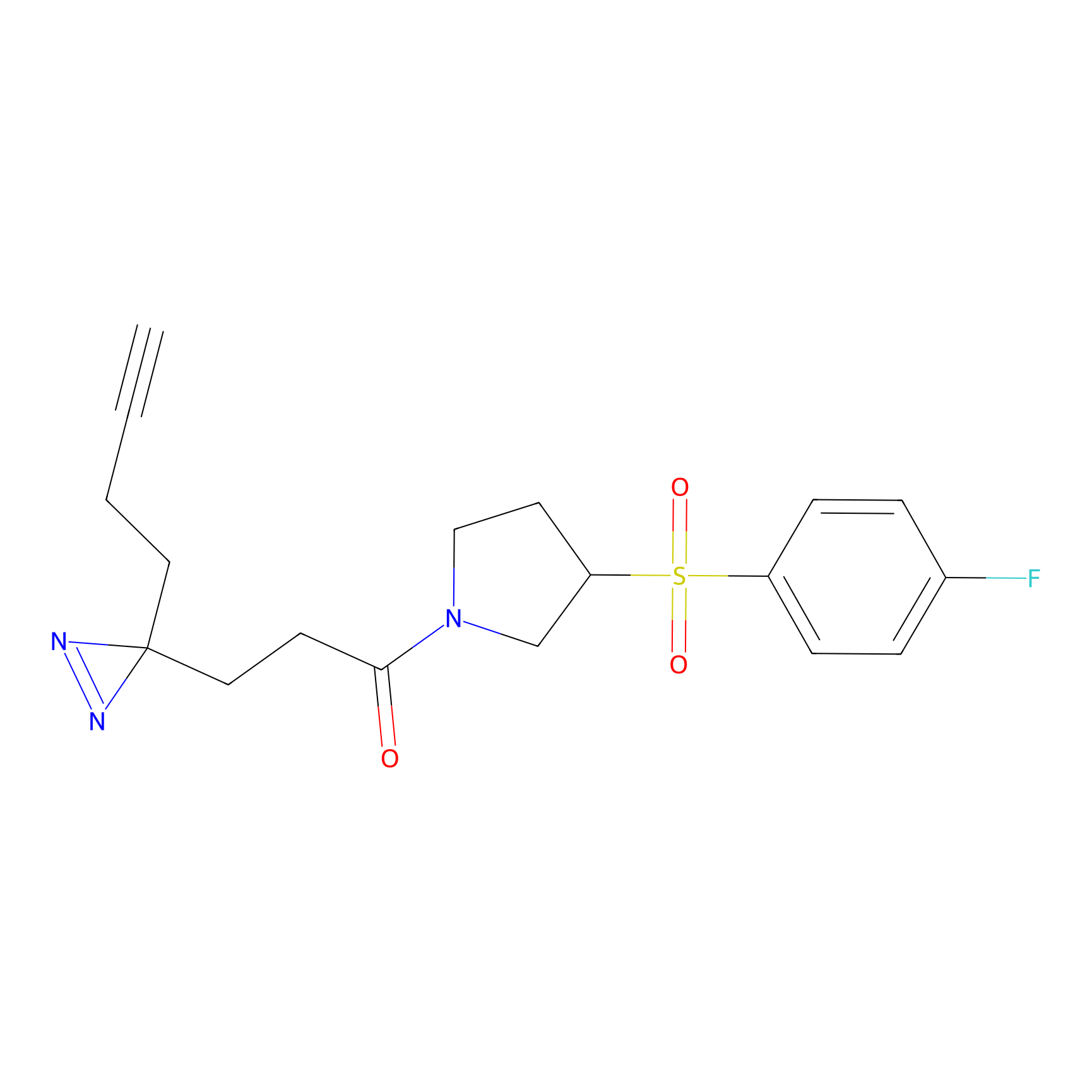
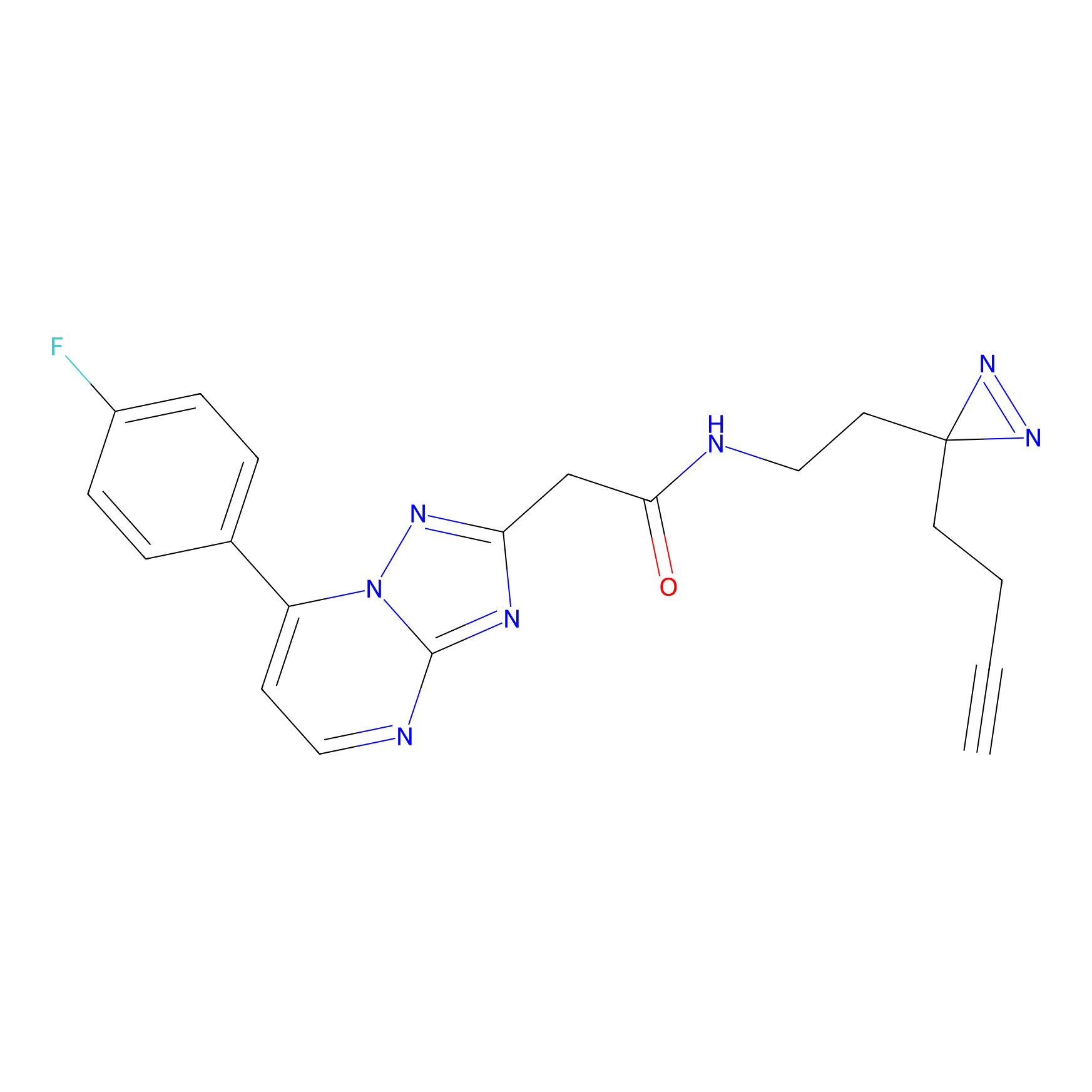
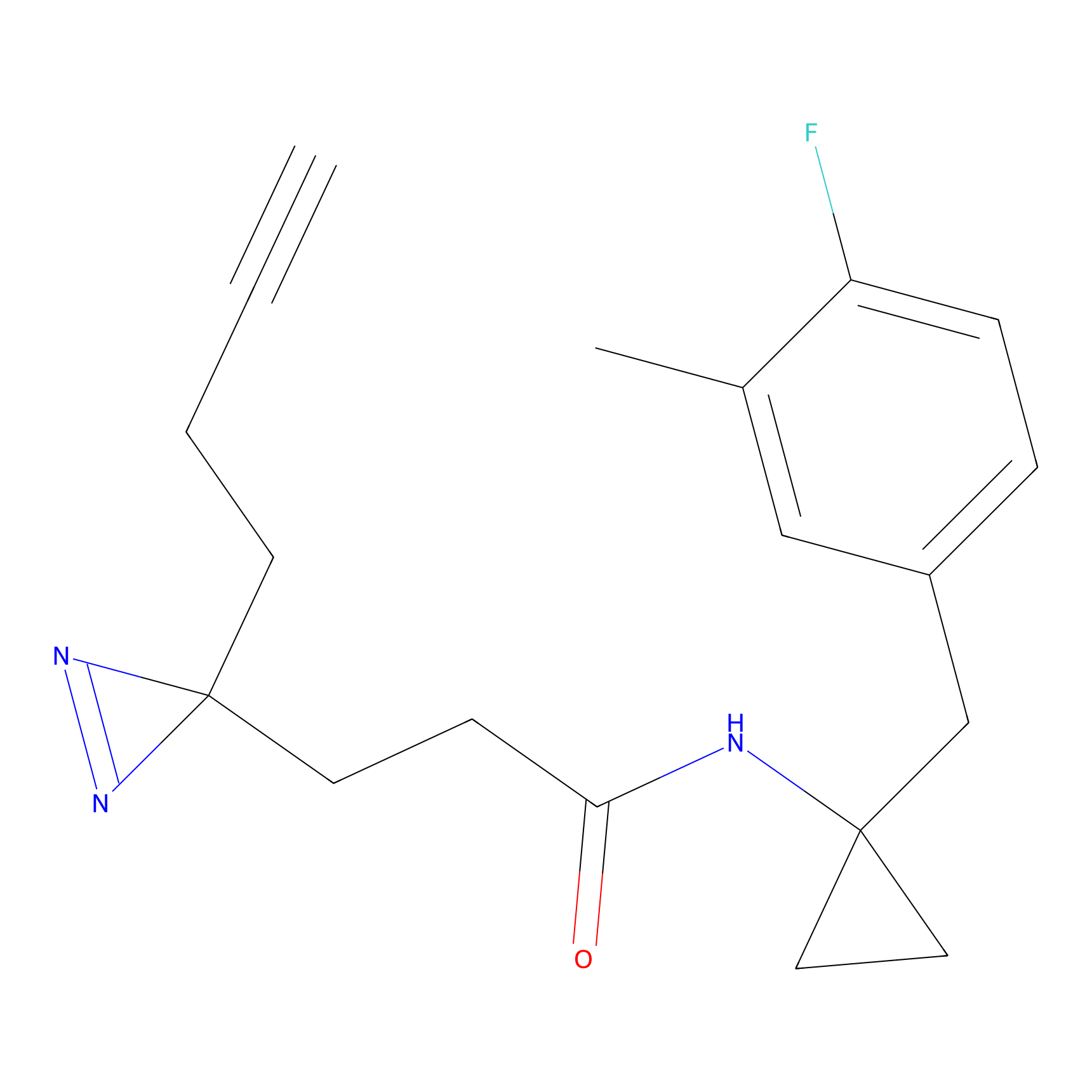
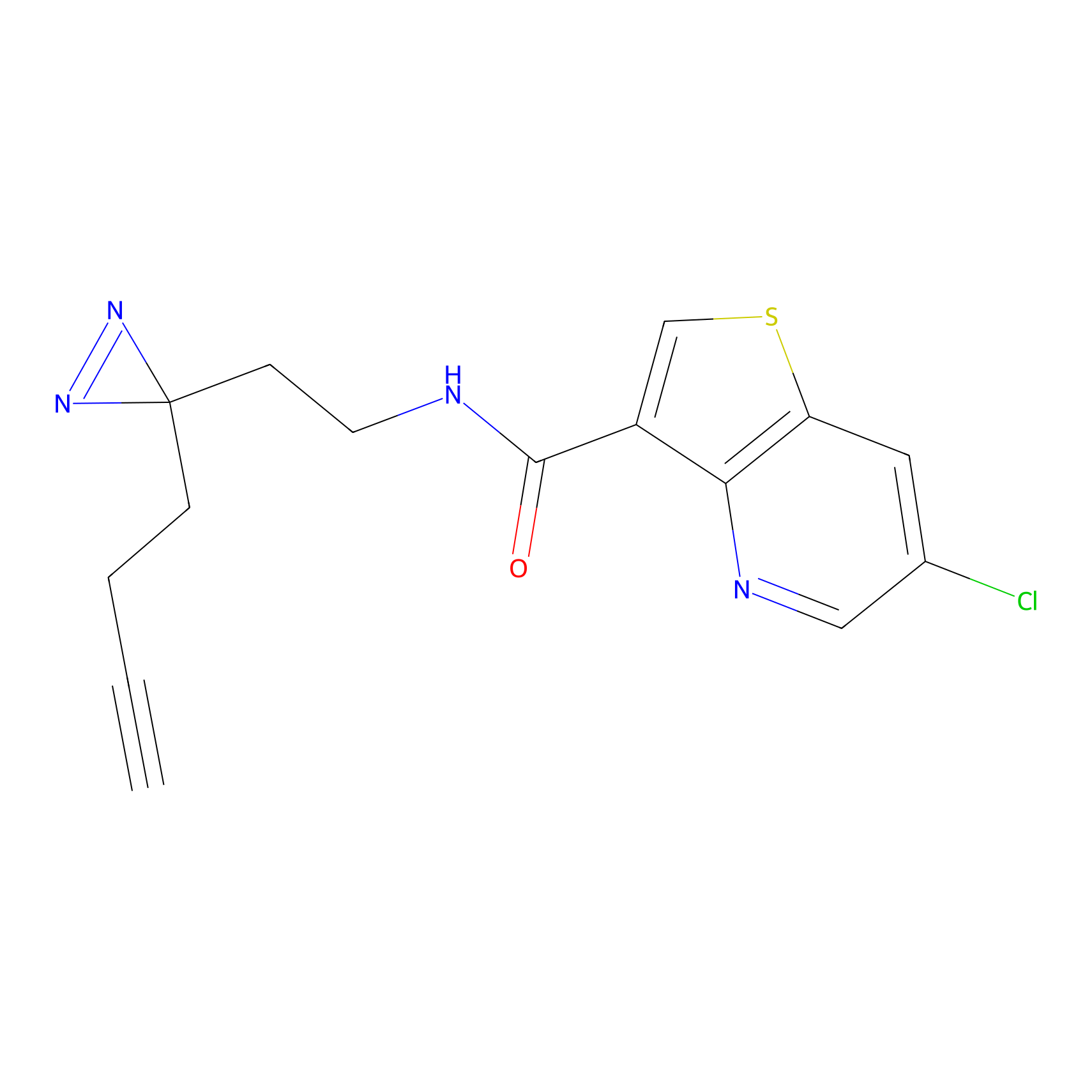
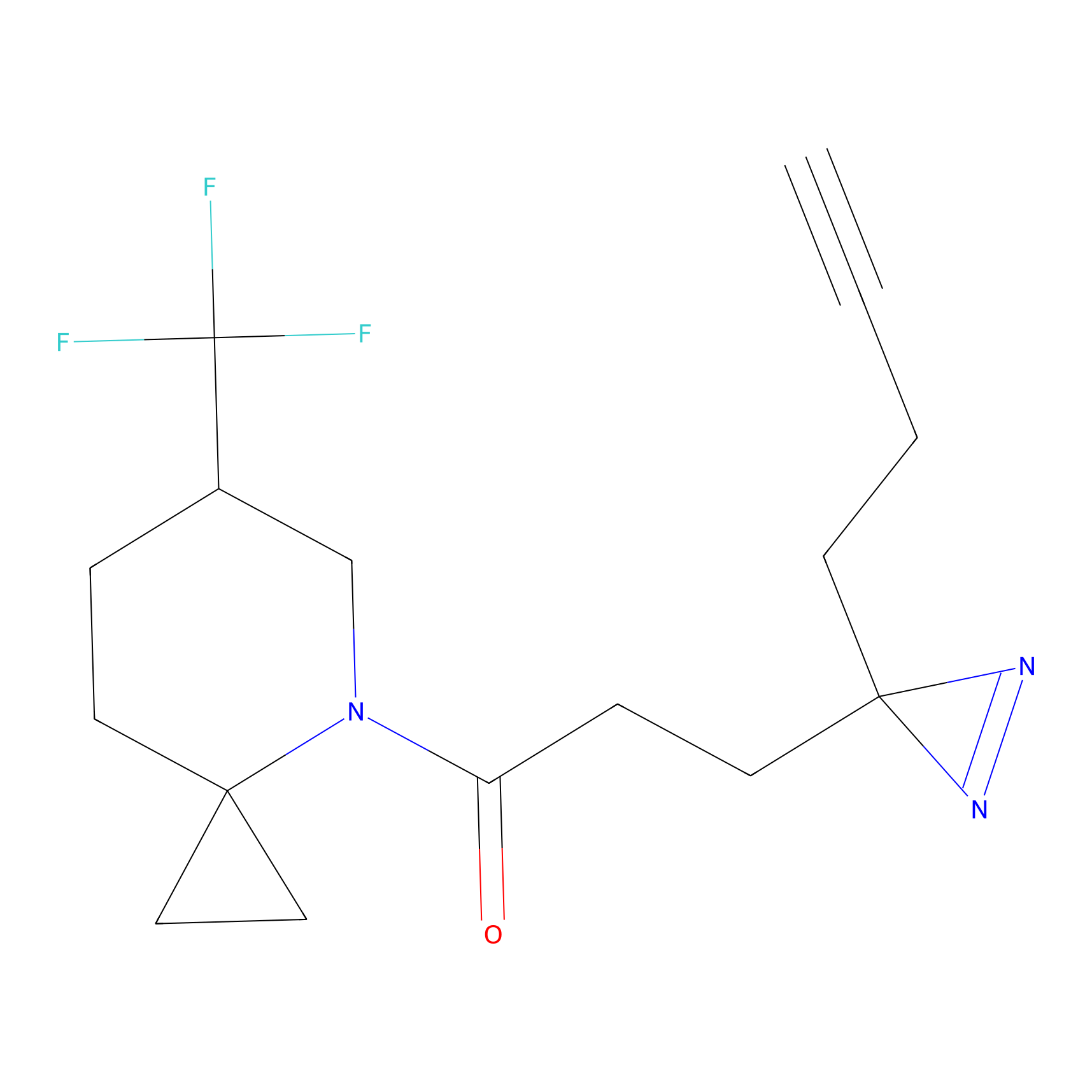
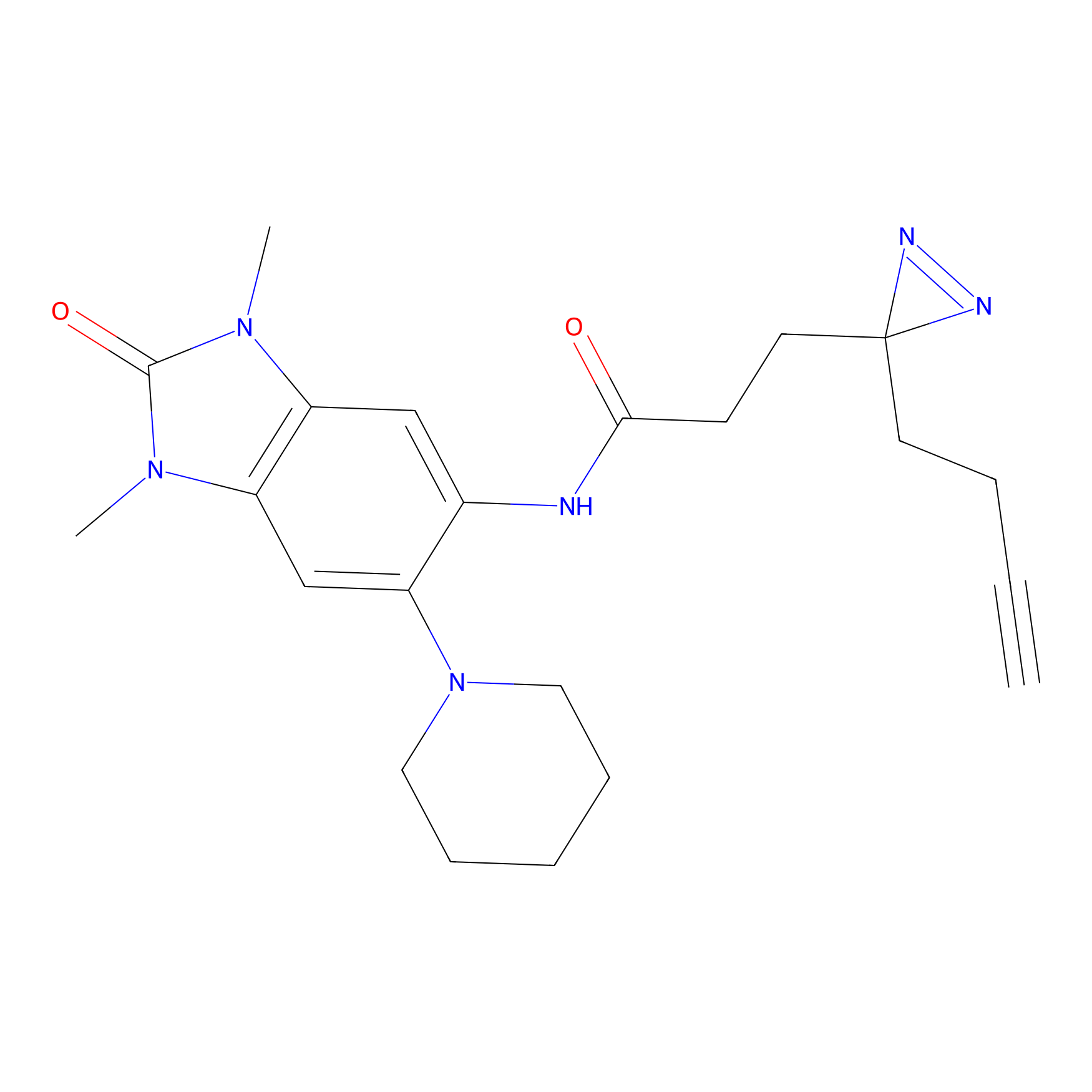
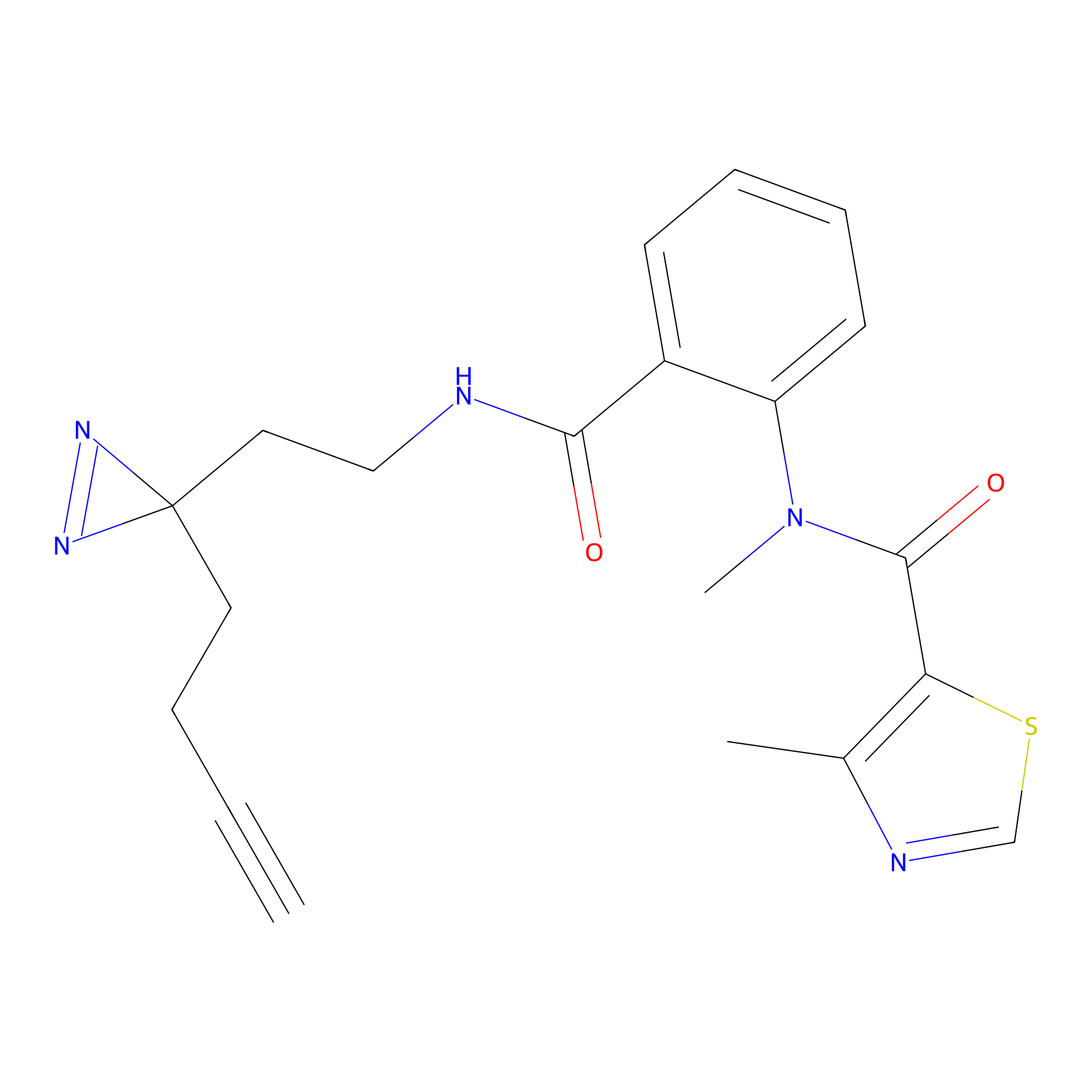
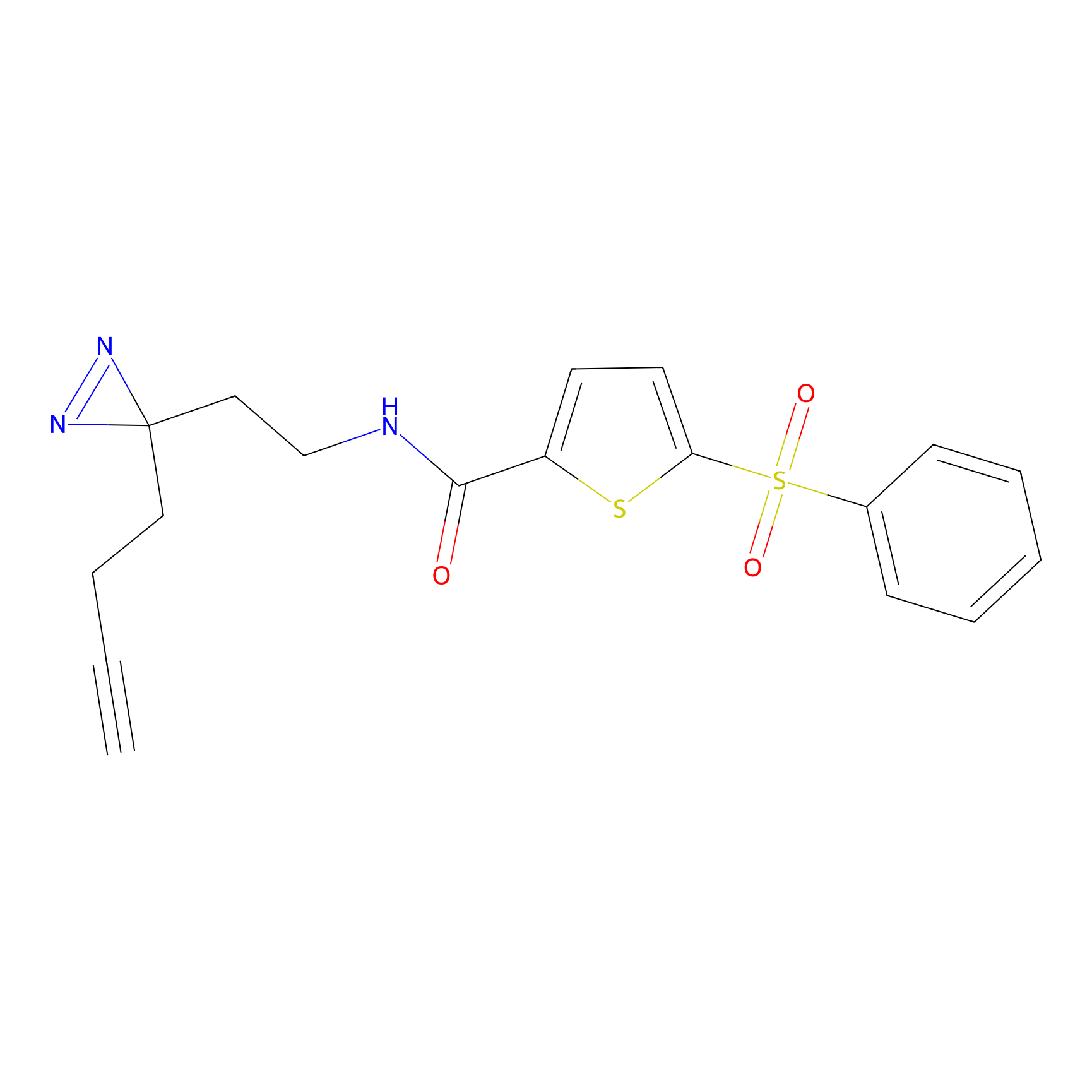
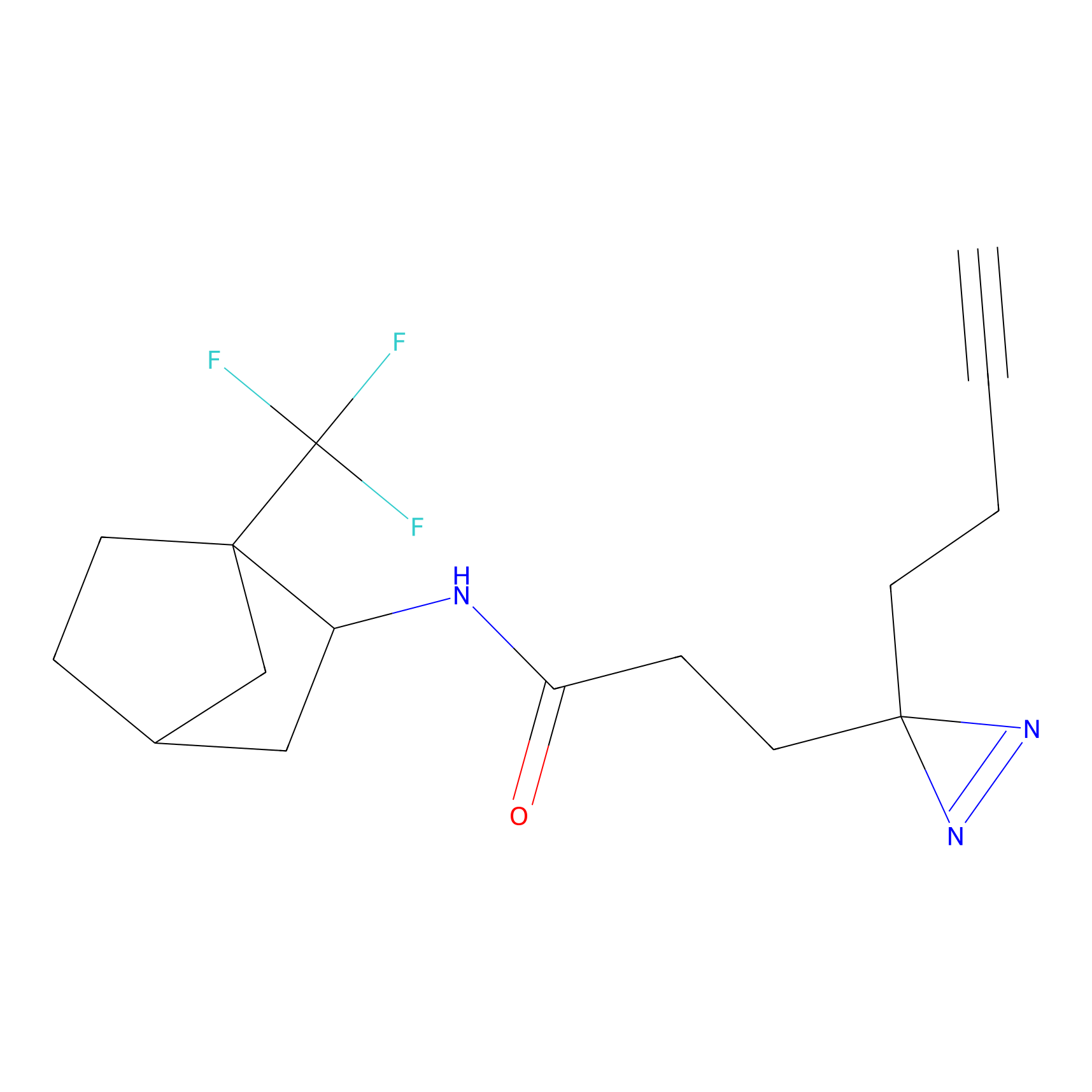
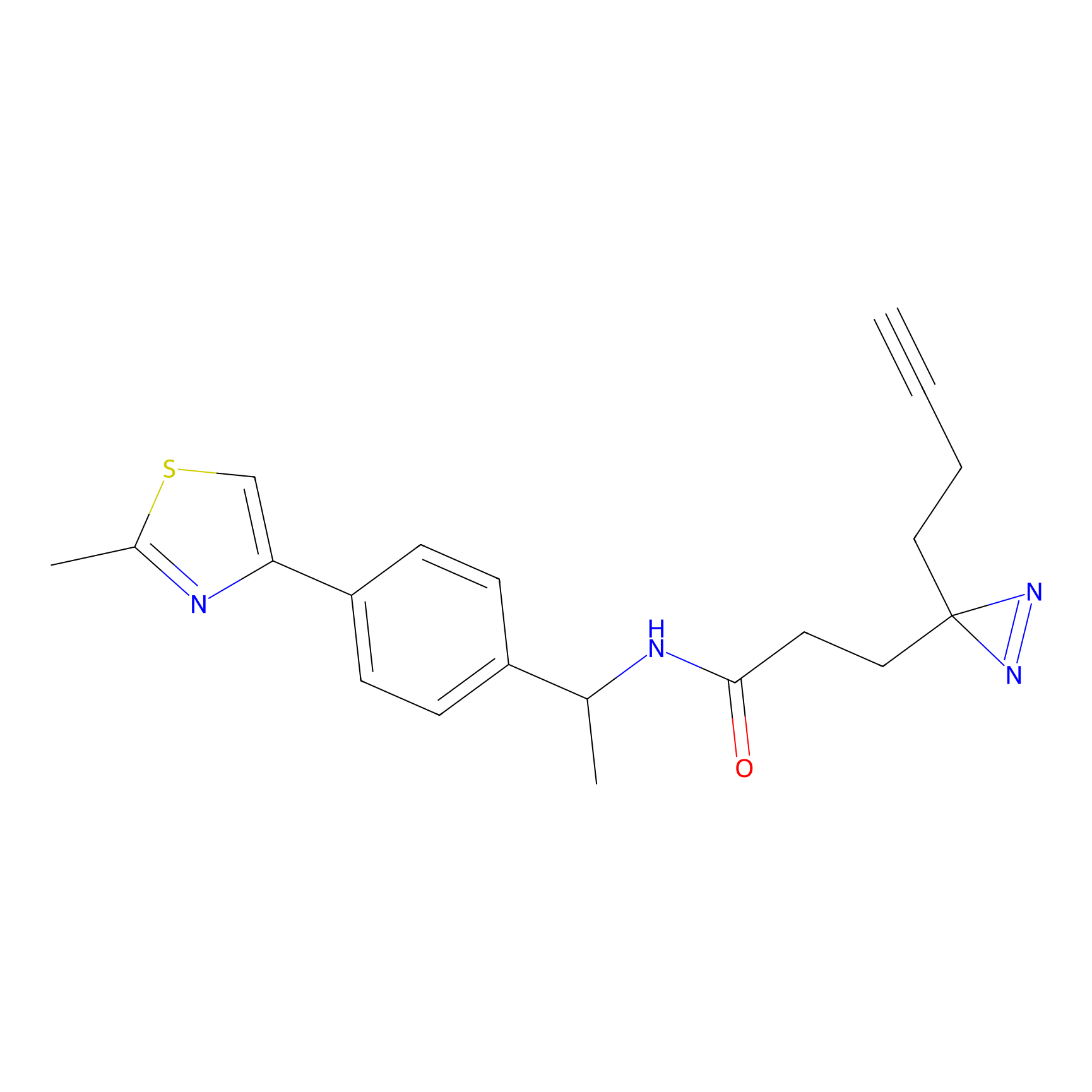
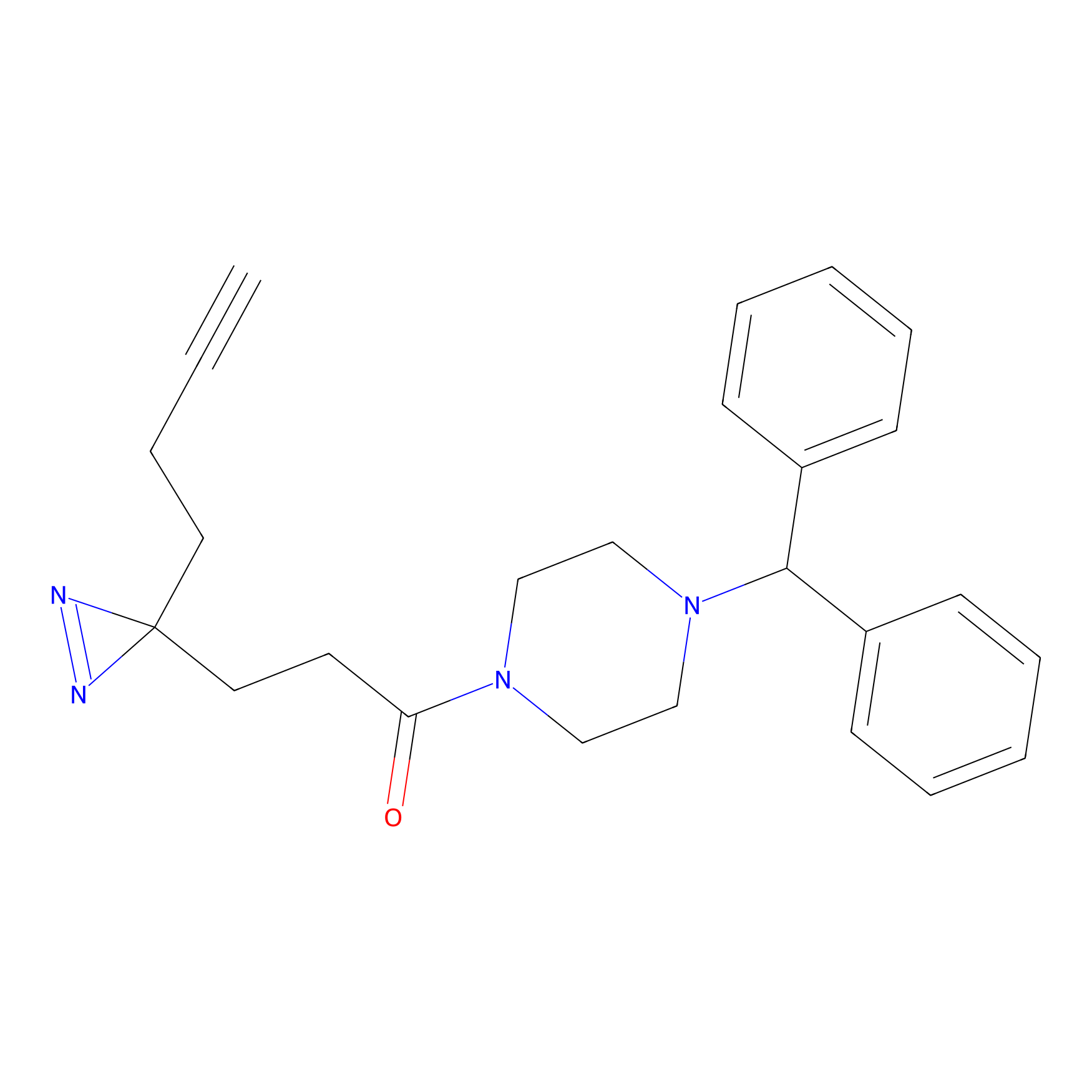
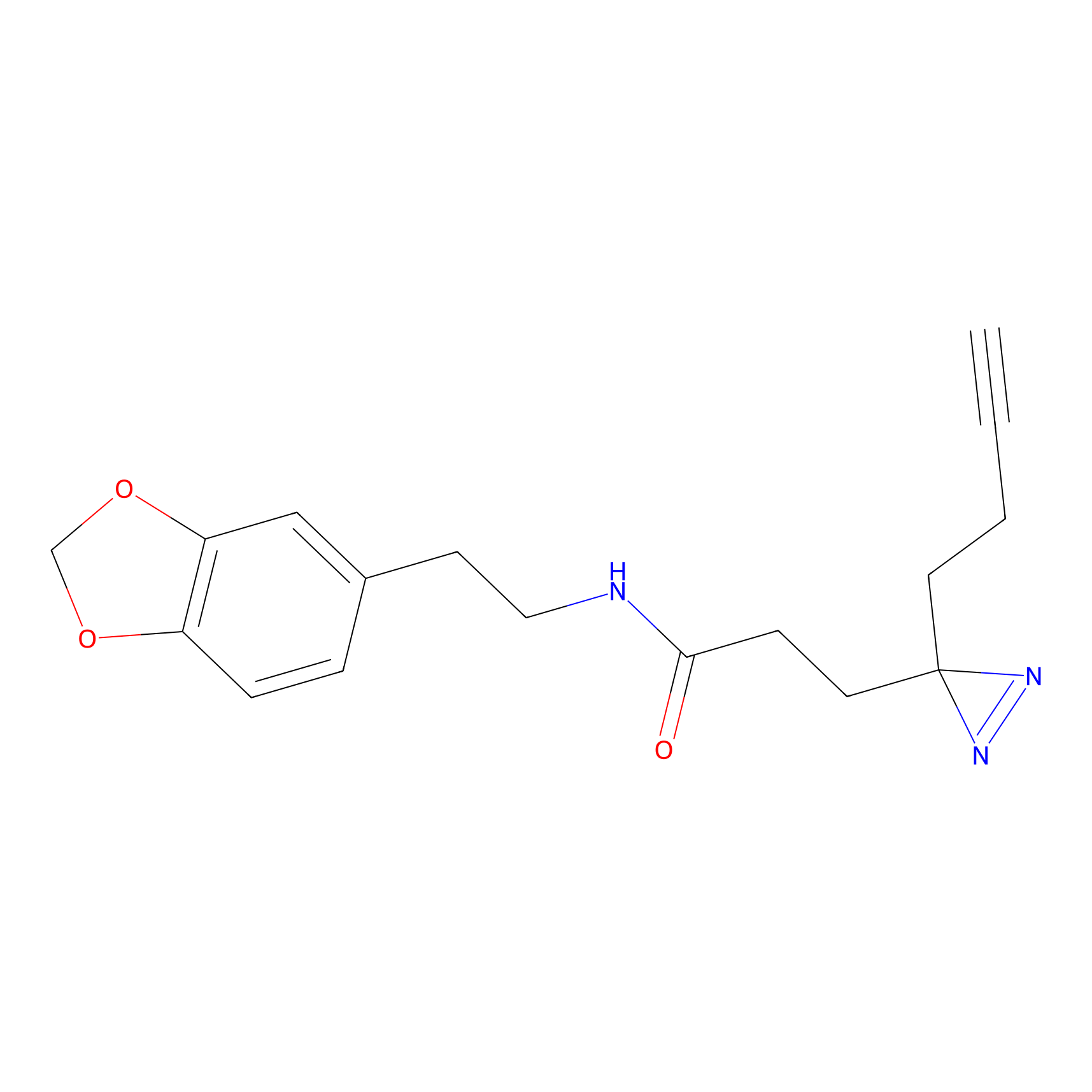
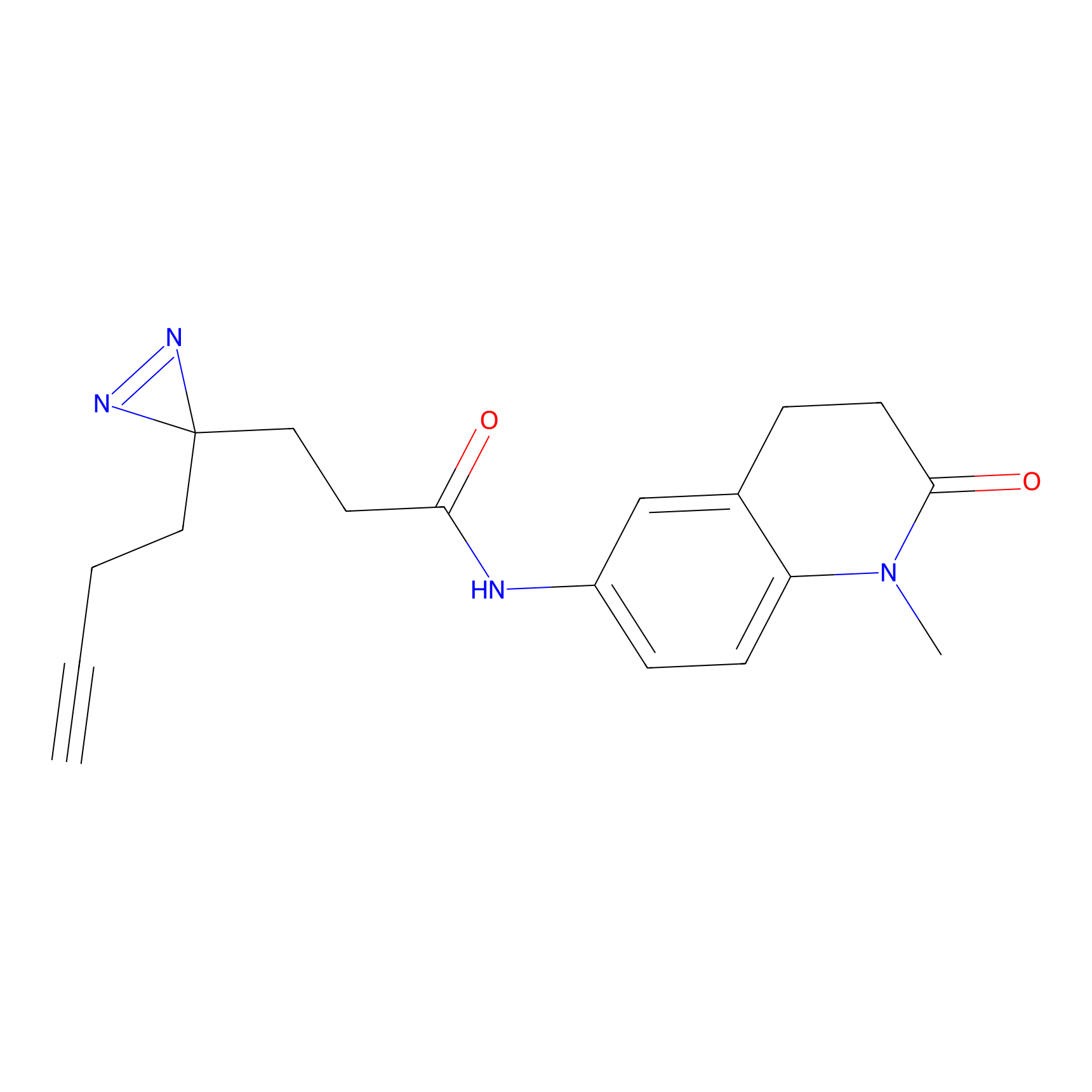
|
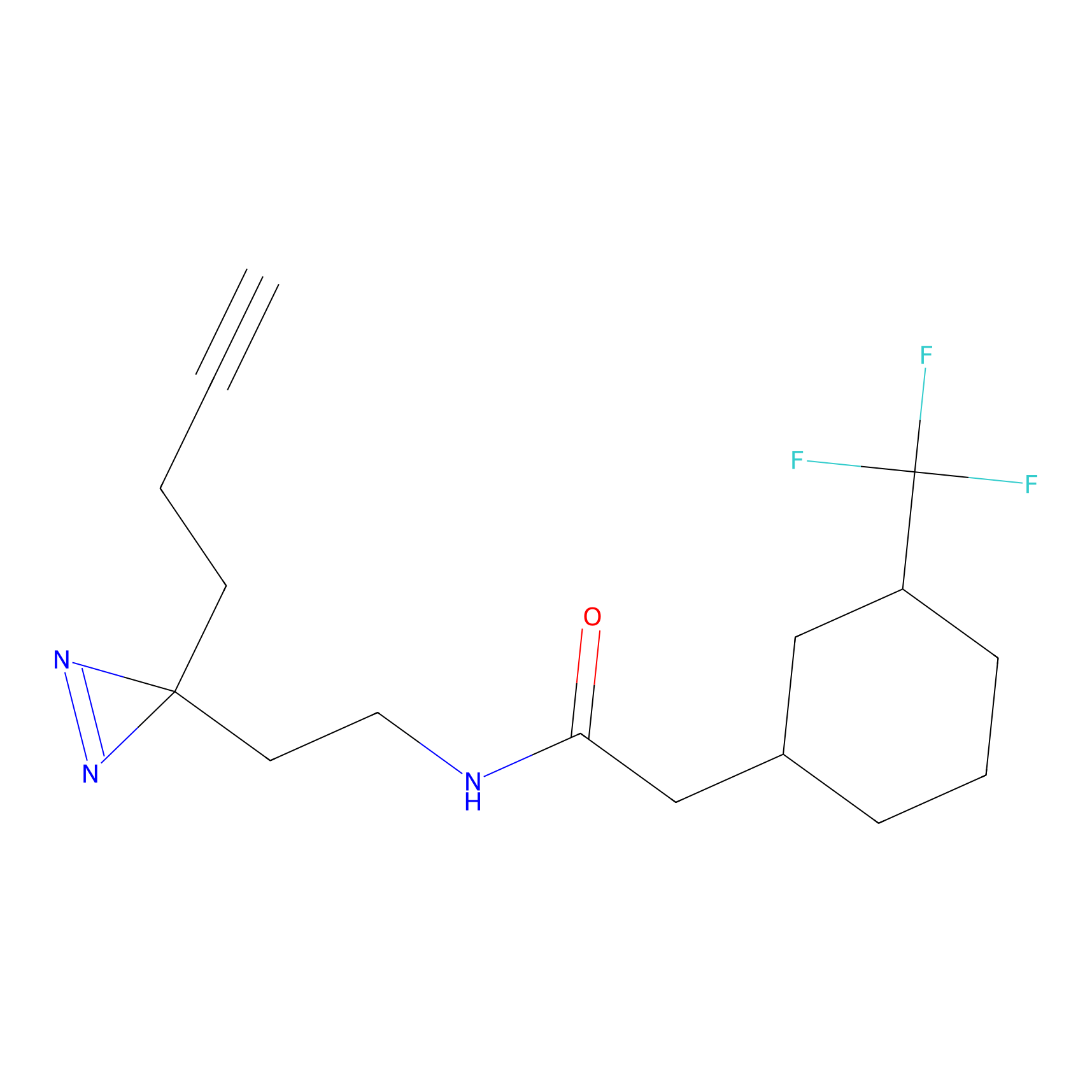
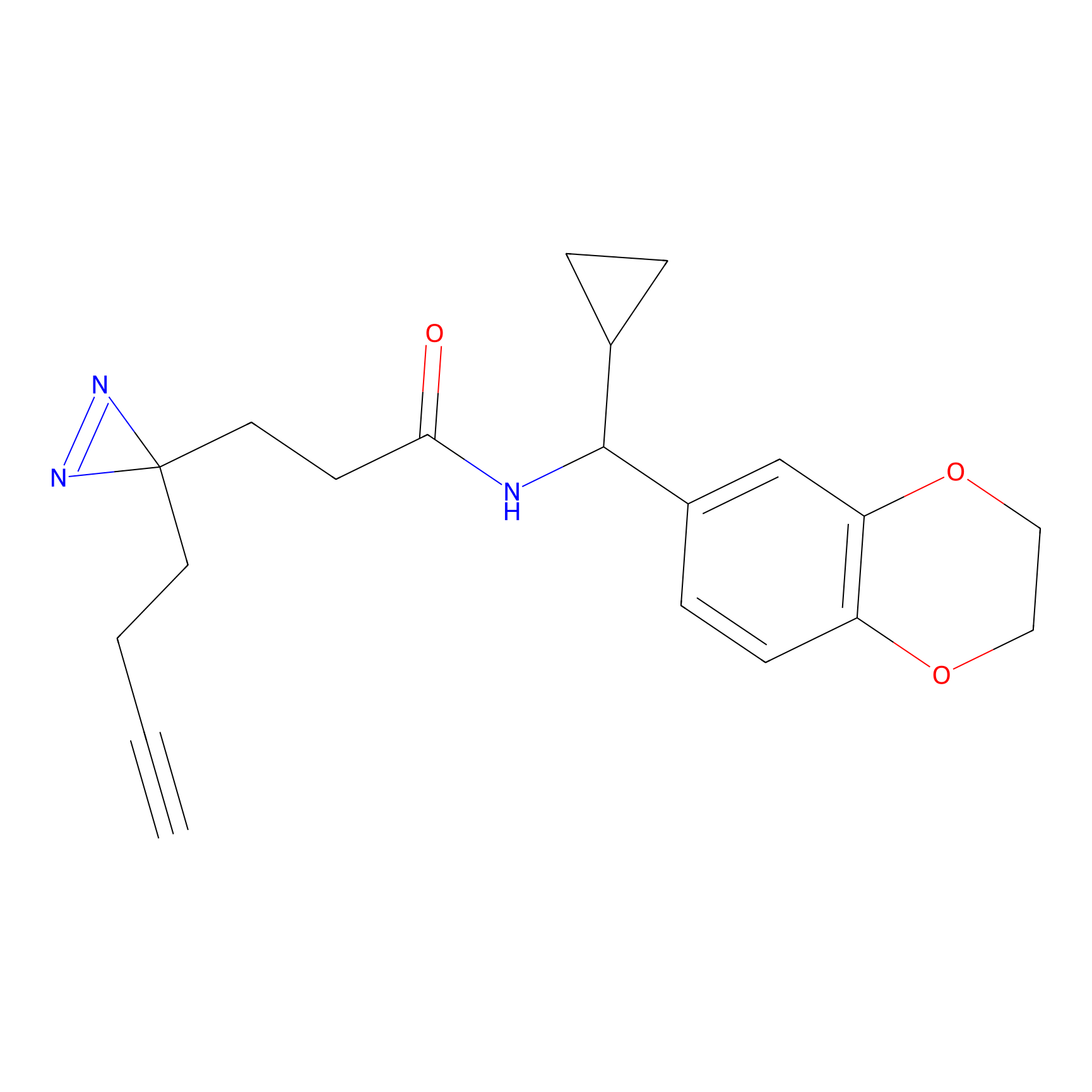
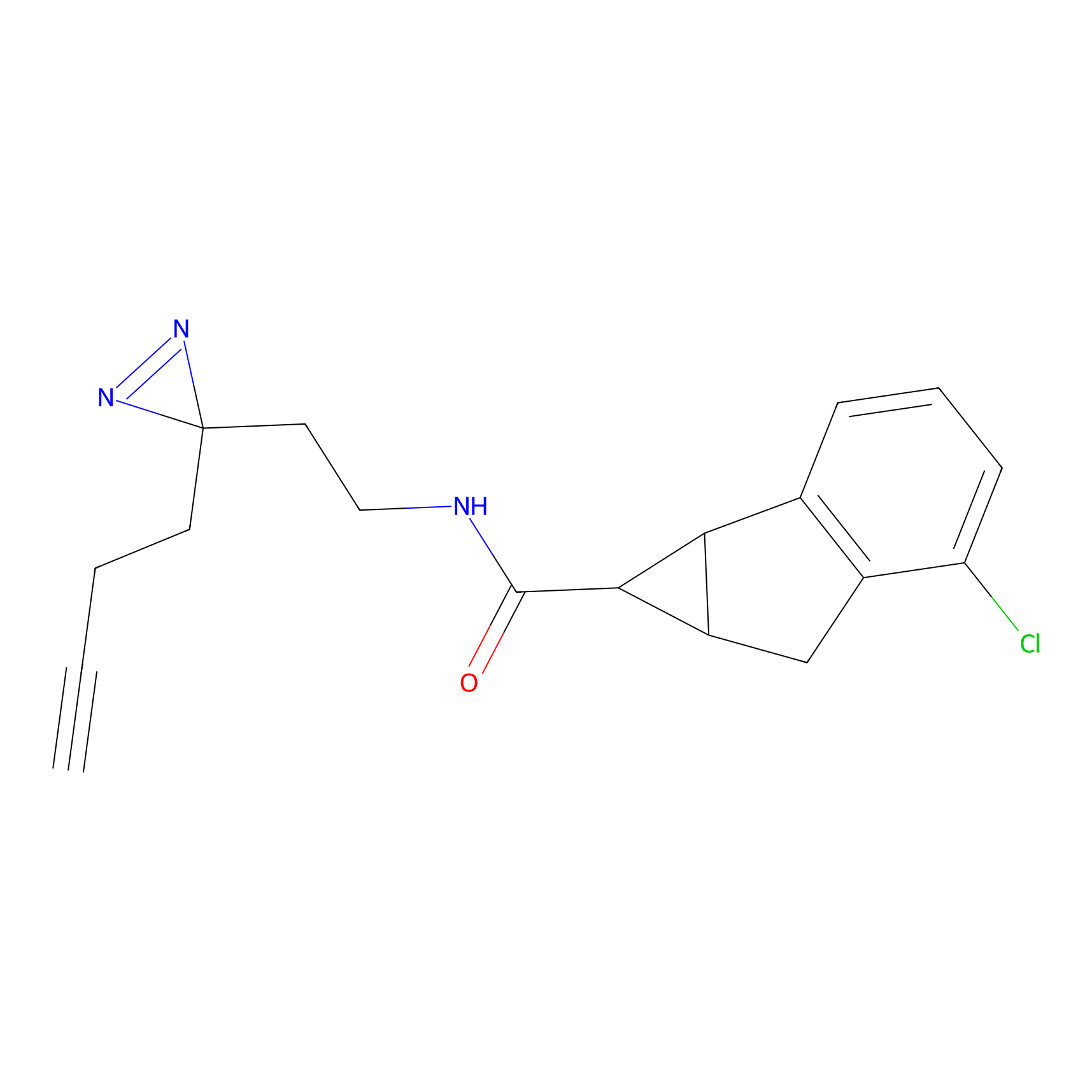
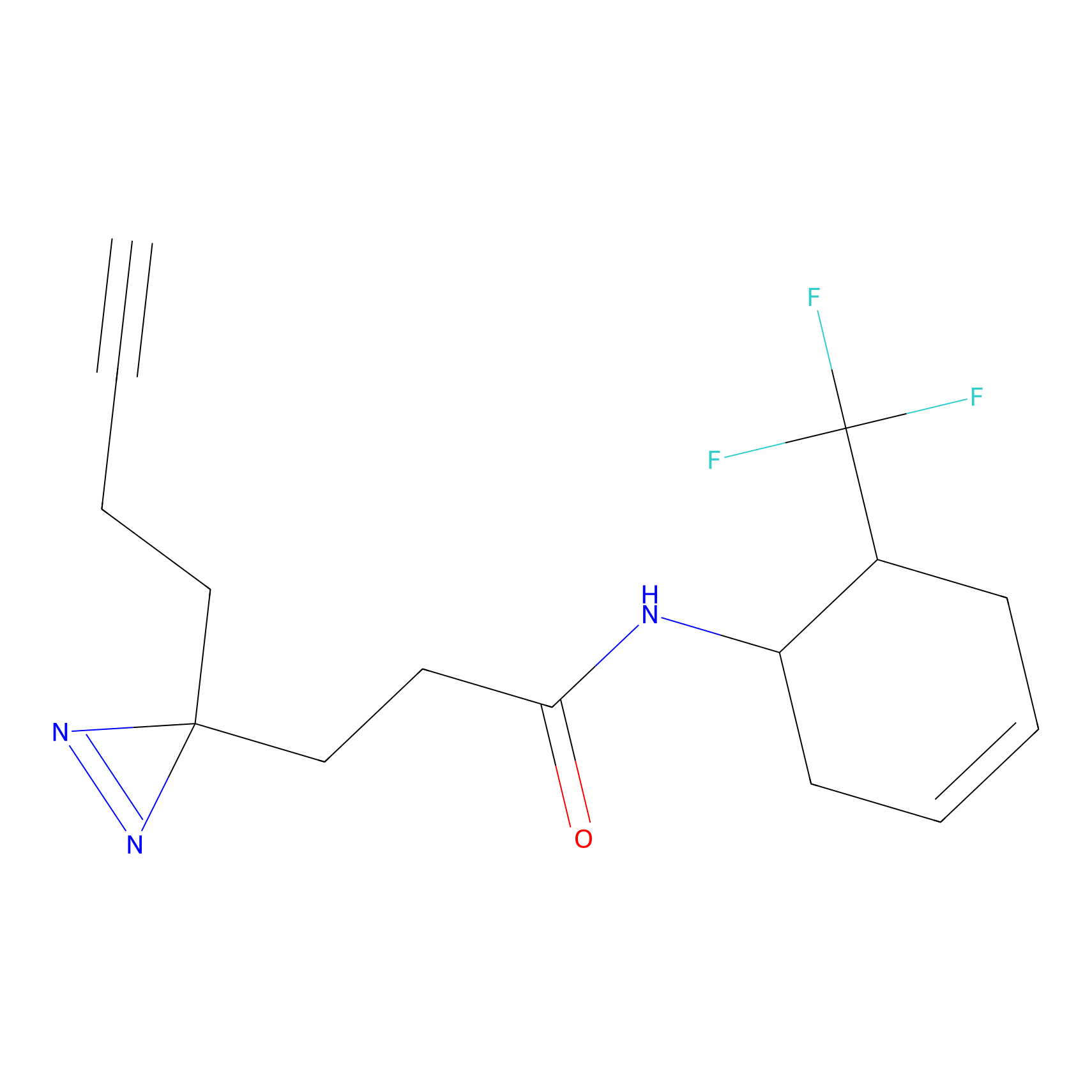
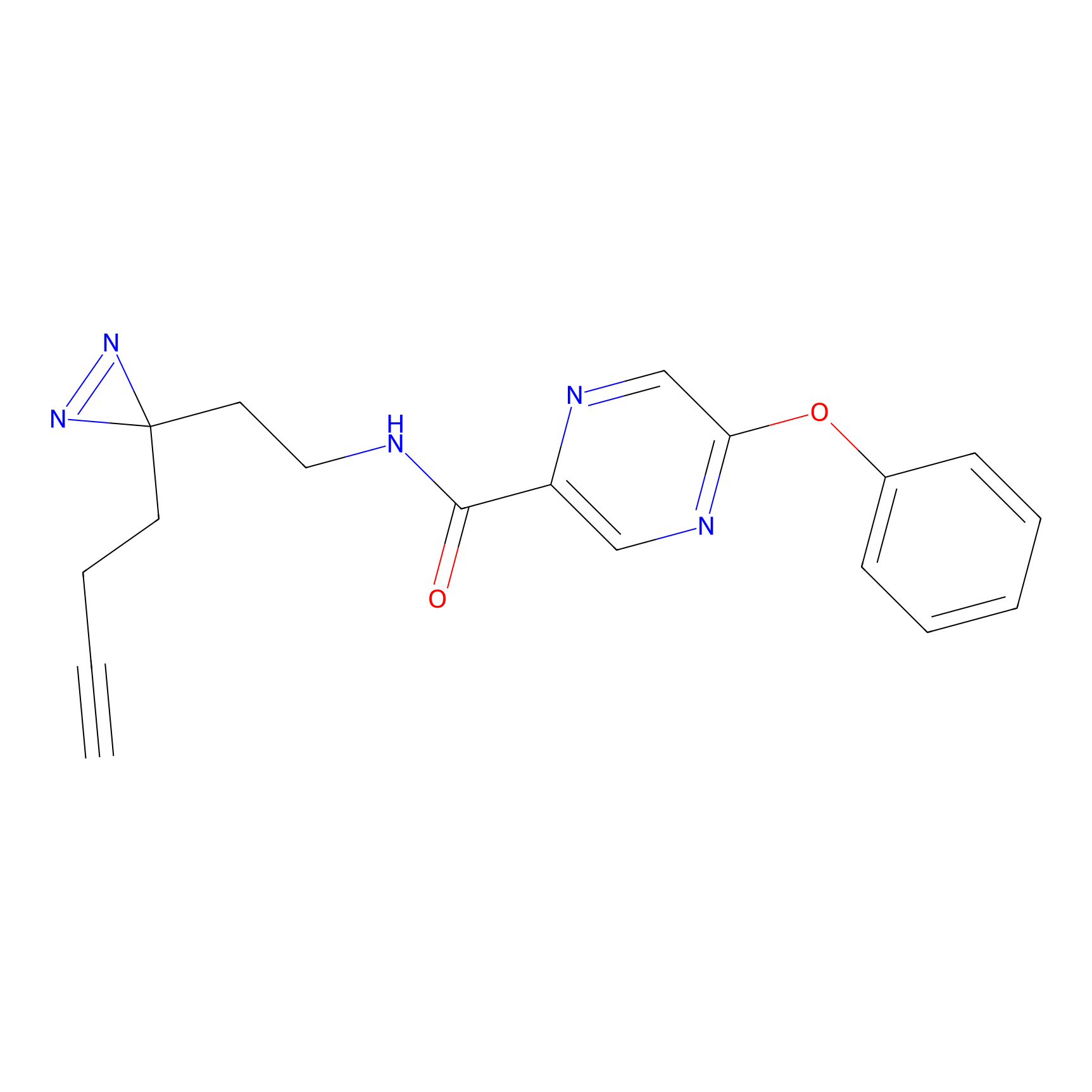
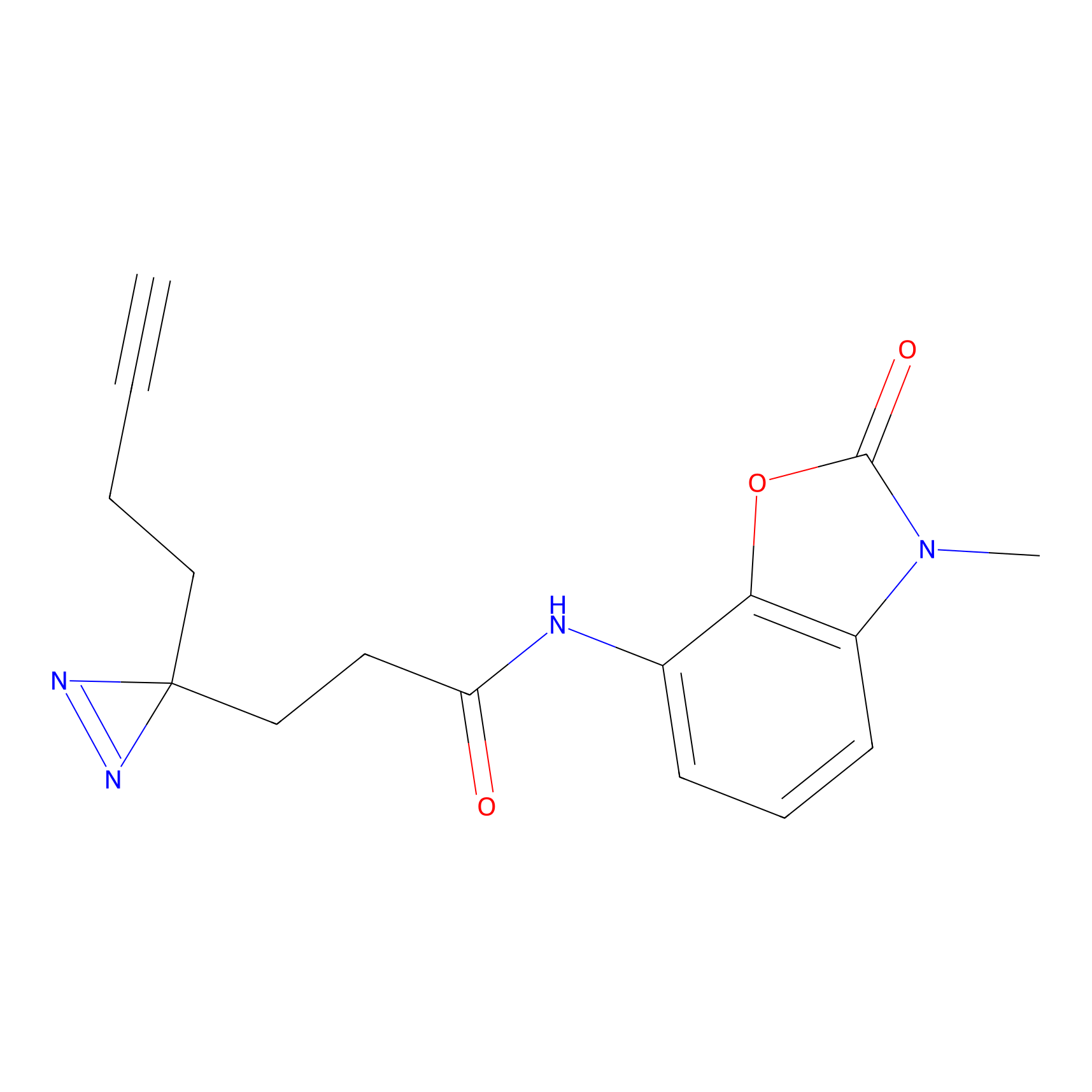
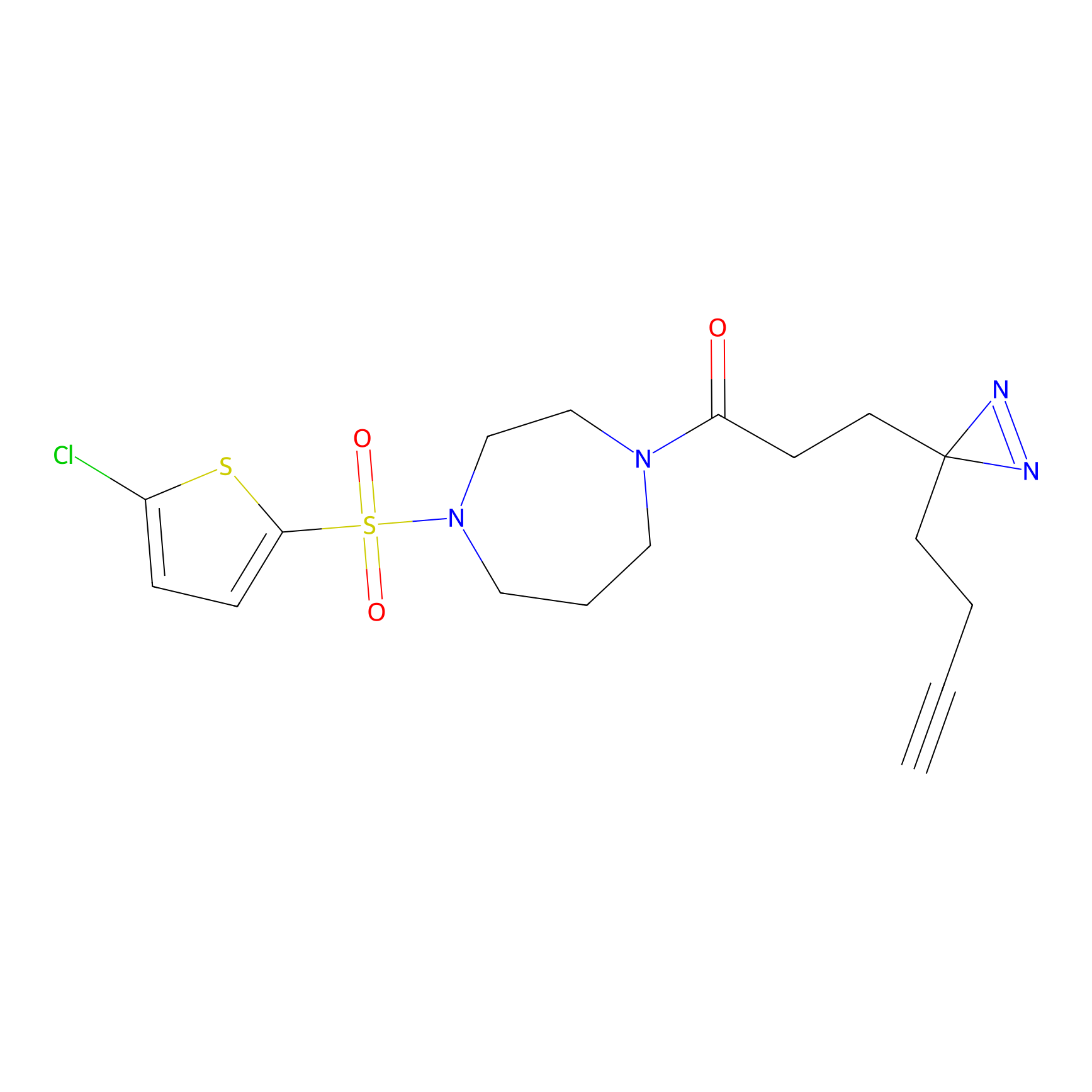
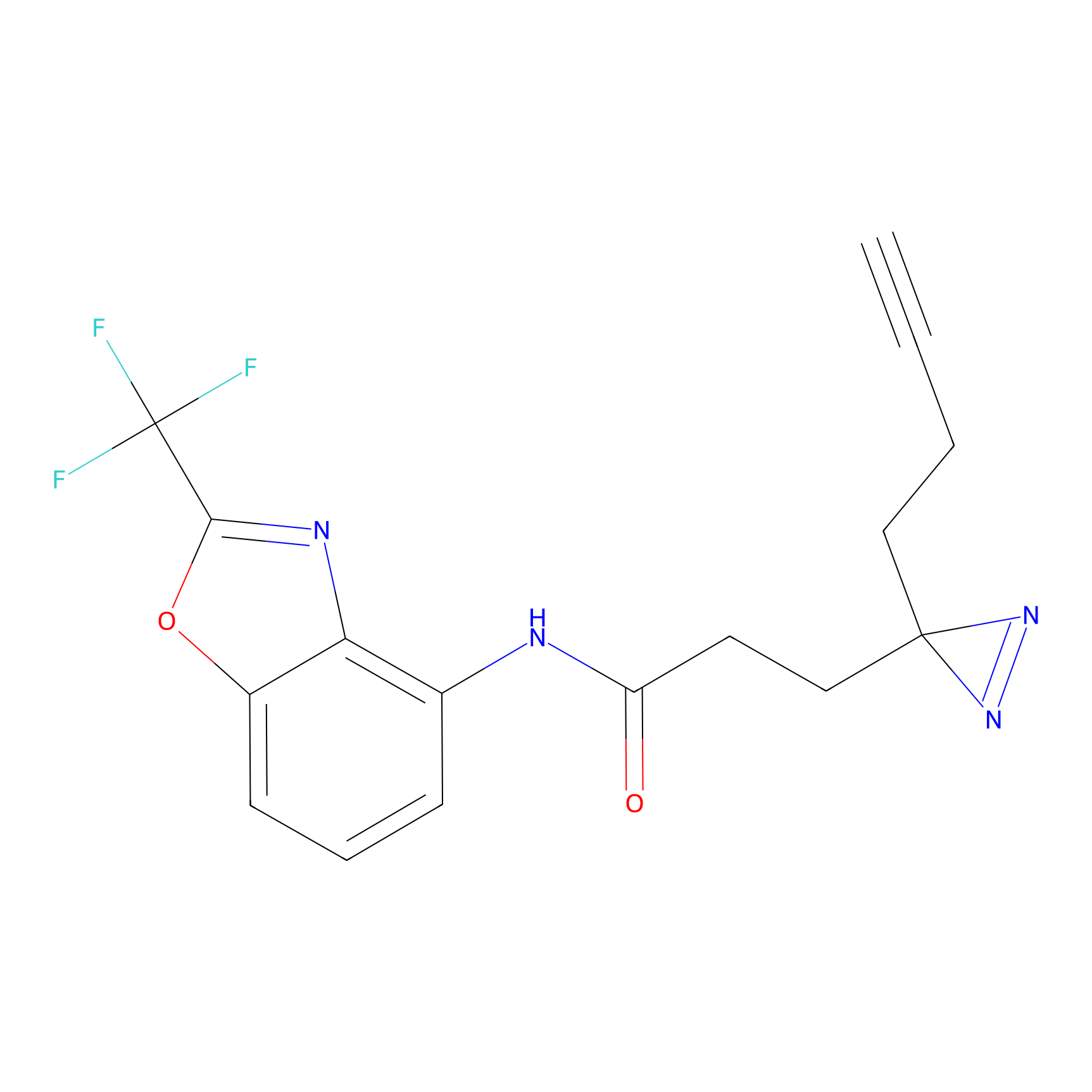
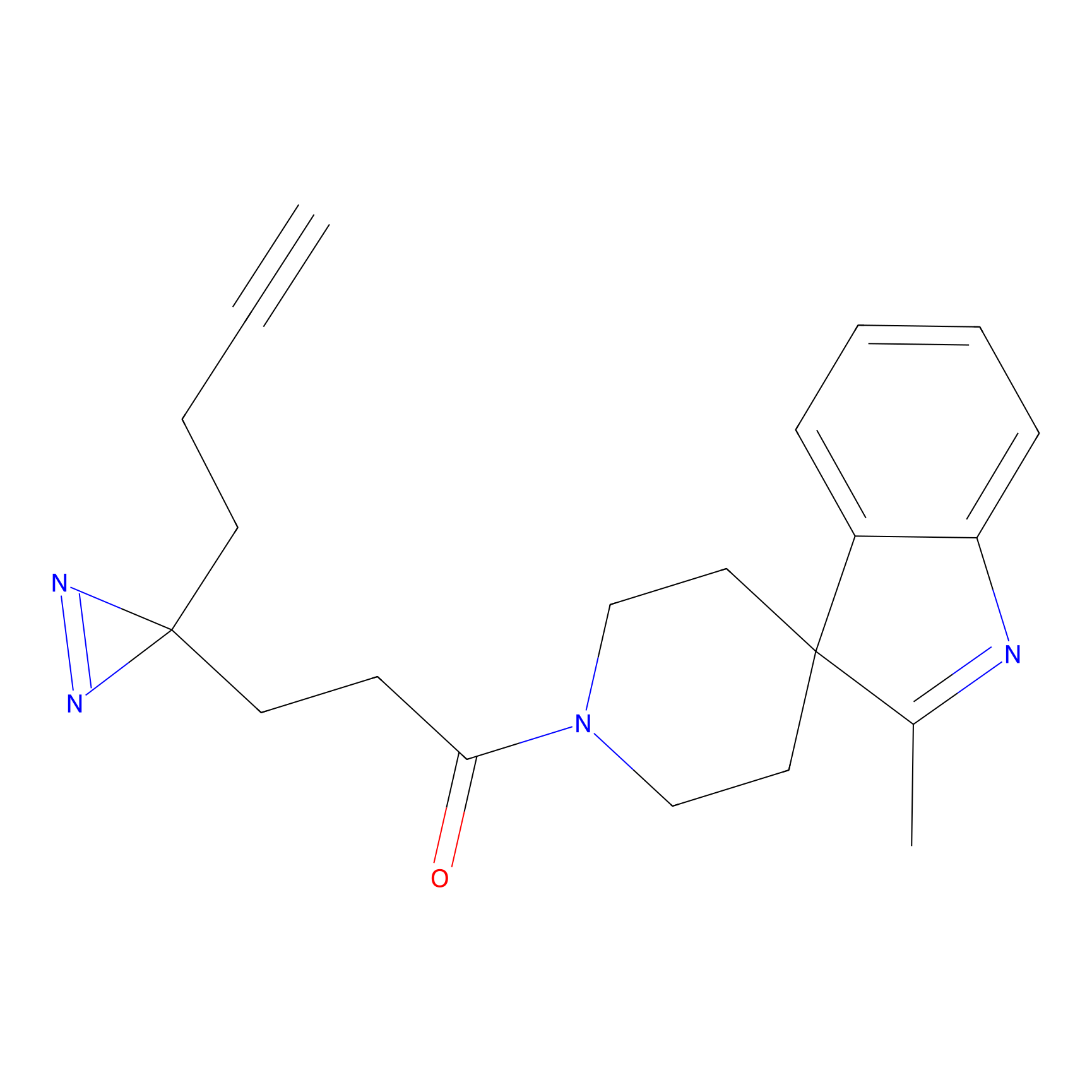
SAA-alkyne Probe Info |
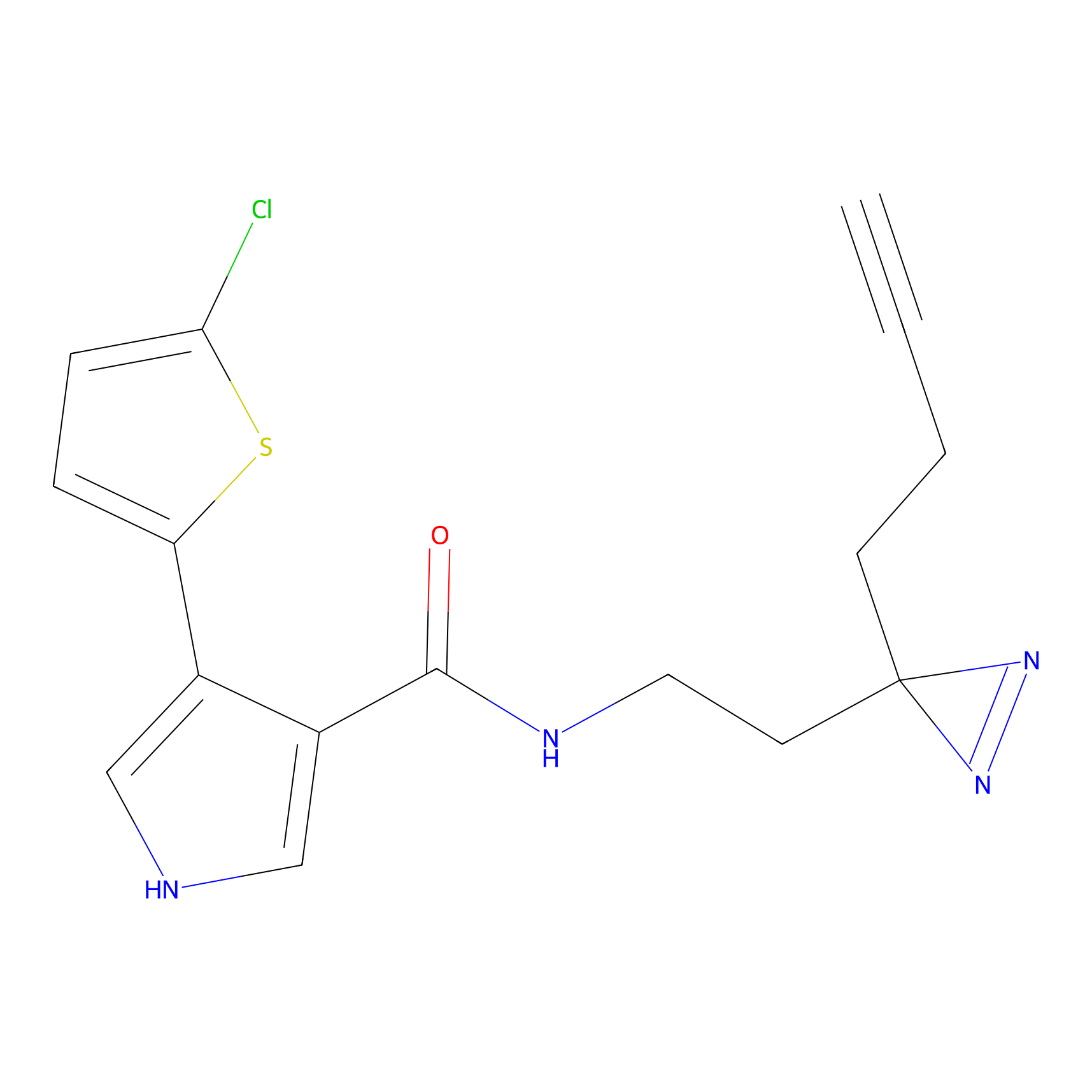
 |
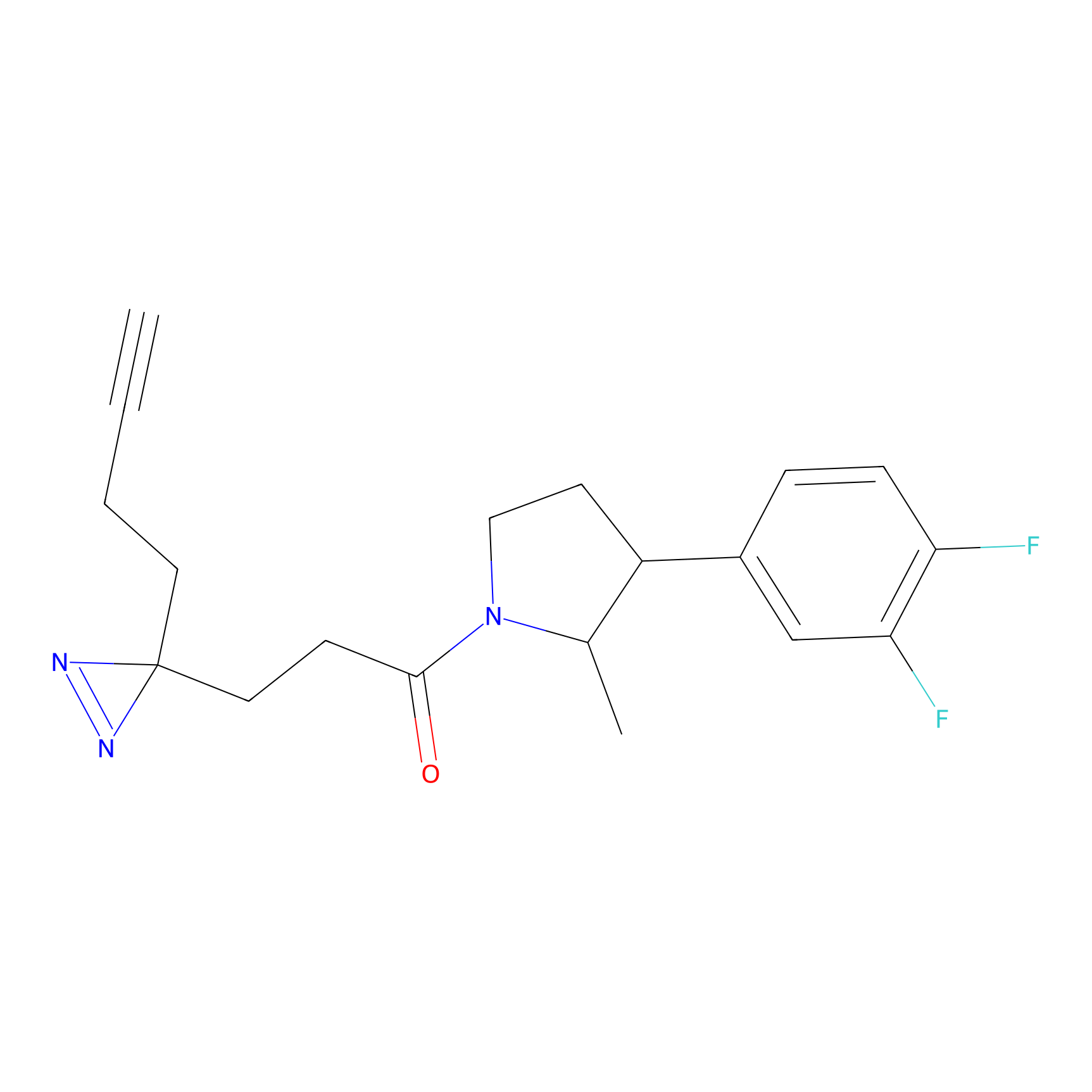
1.12 | LDD0252 | [2] | |
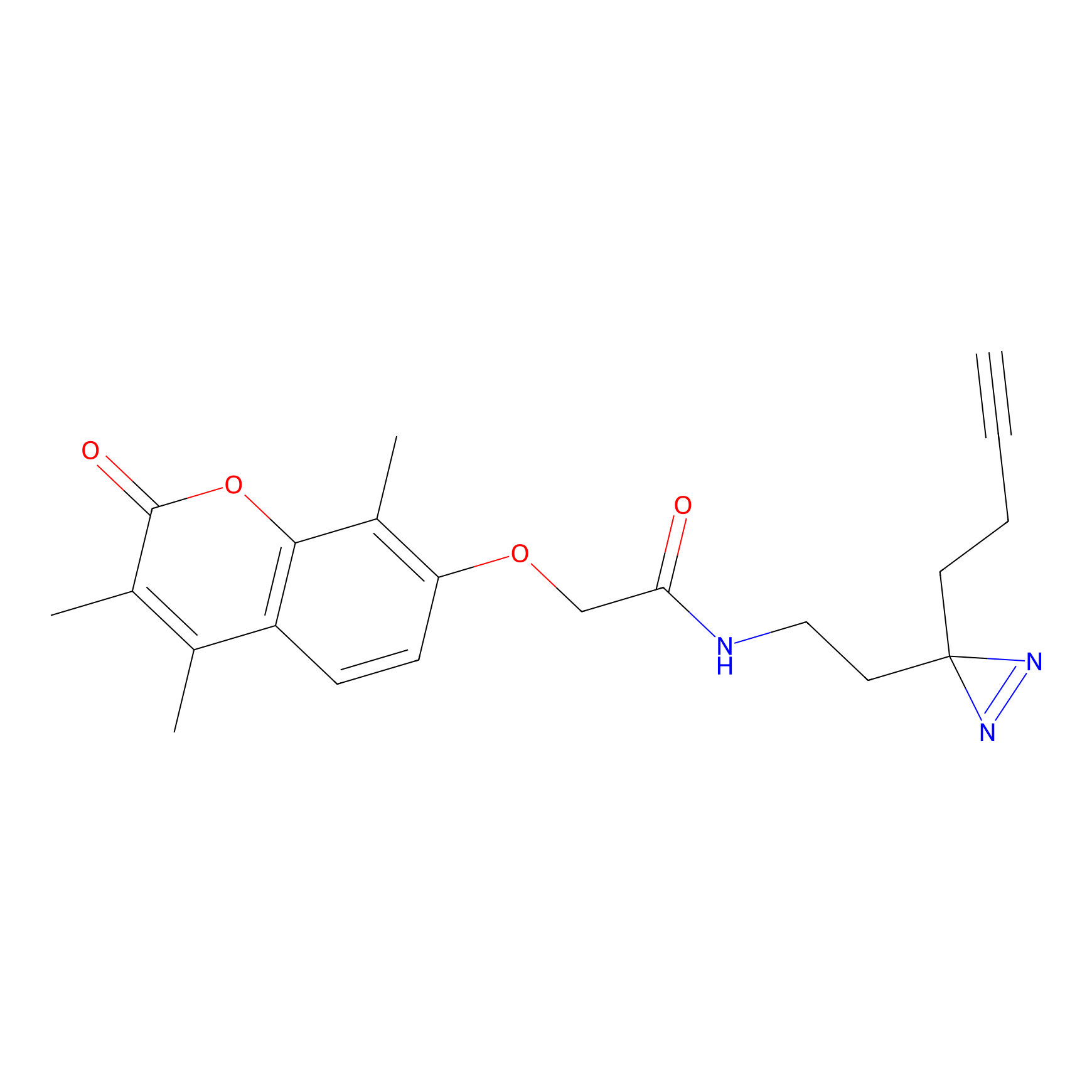
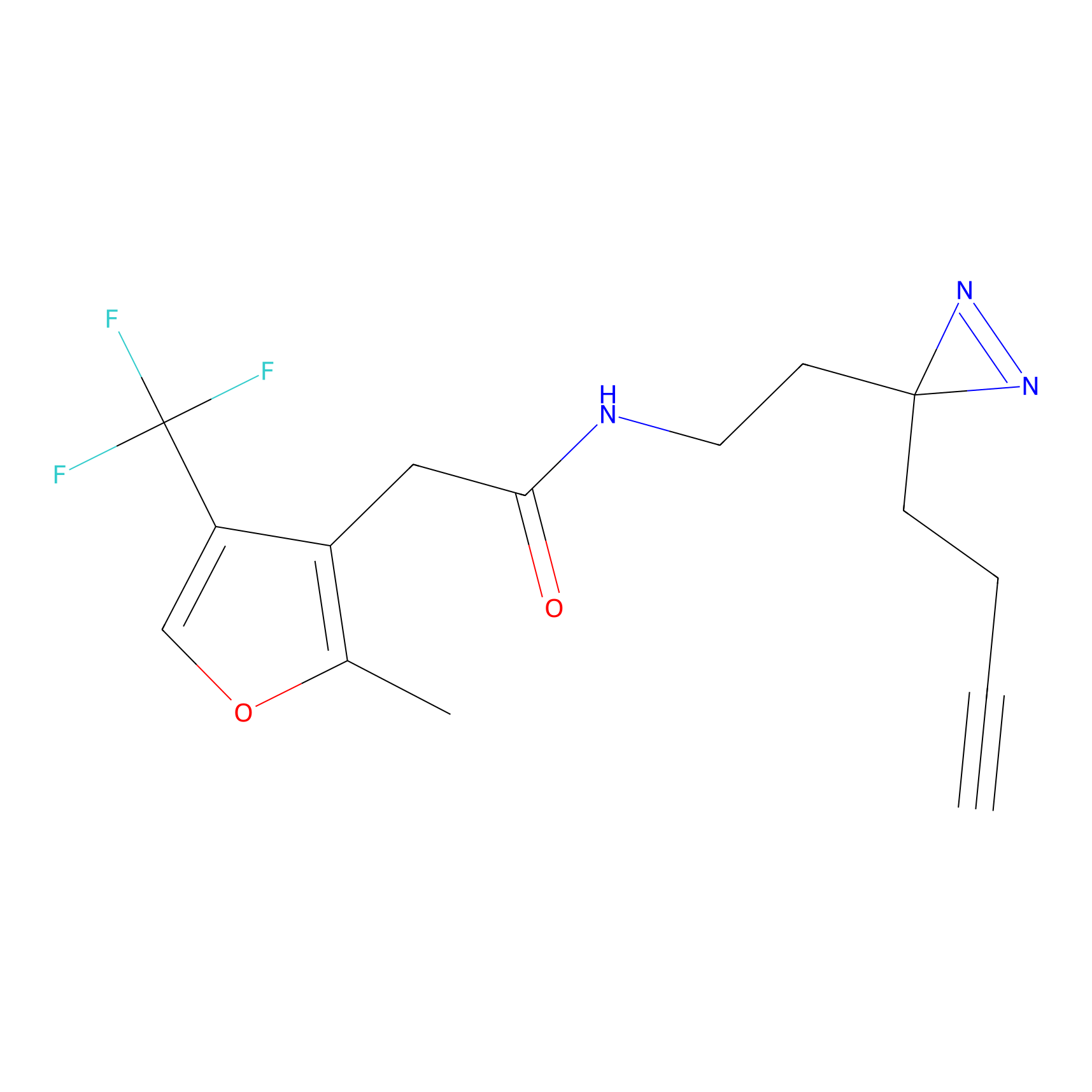
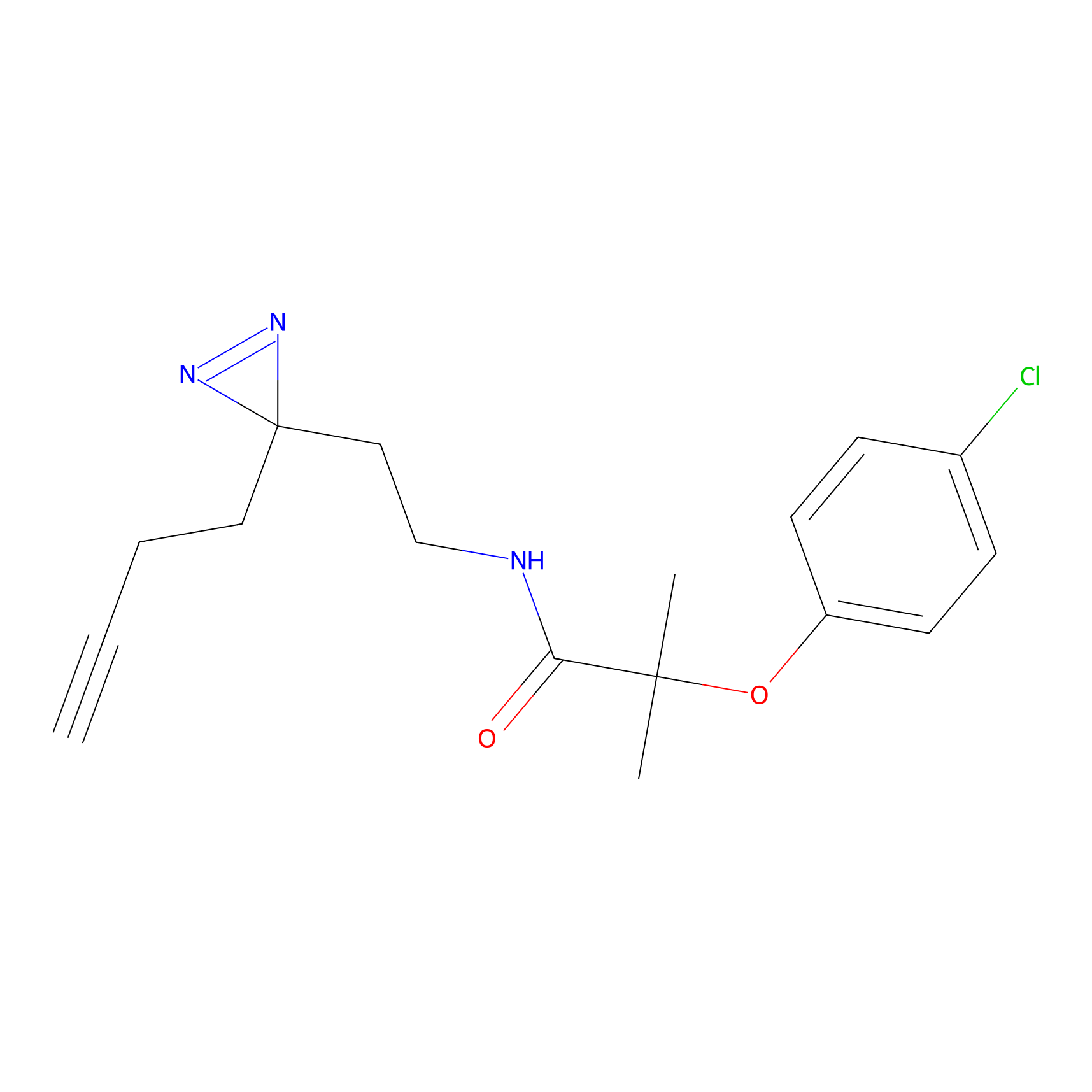
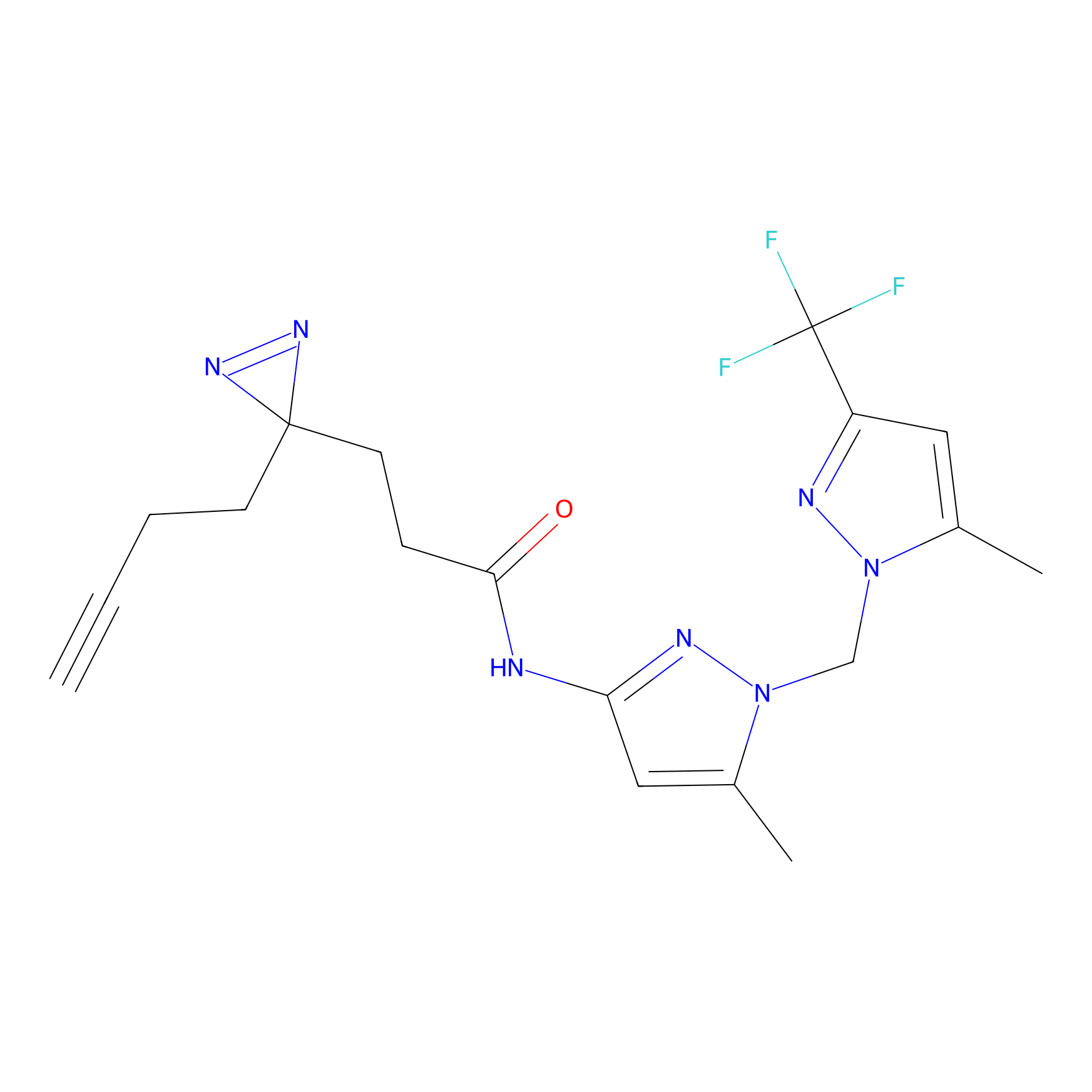
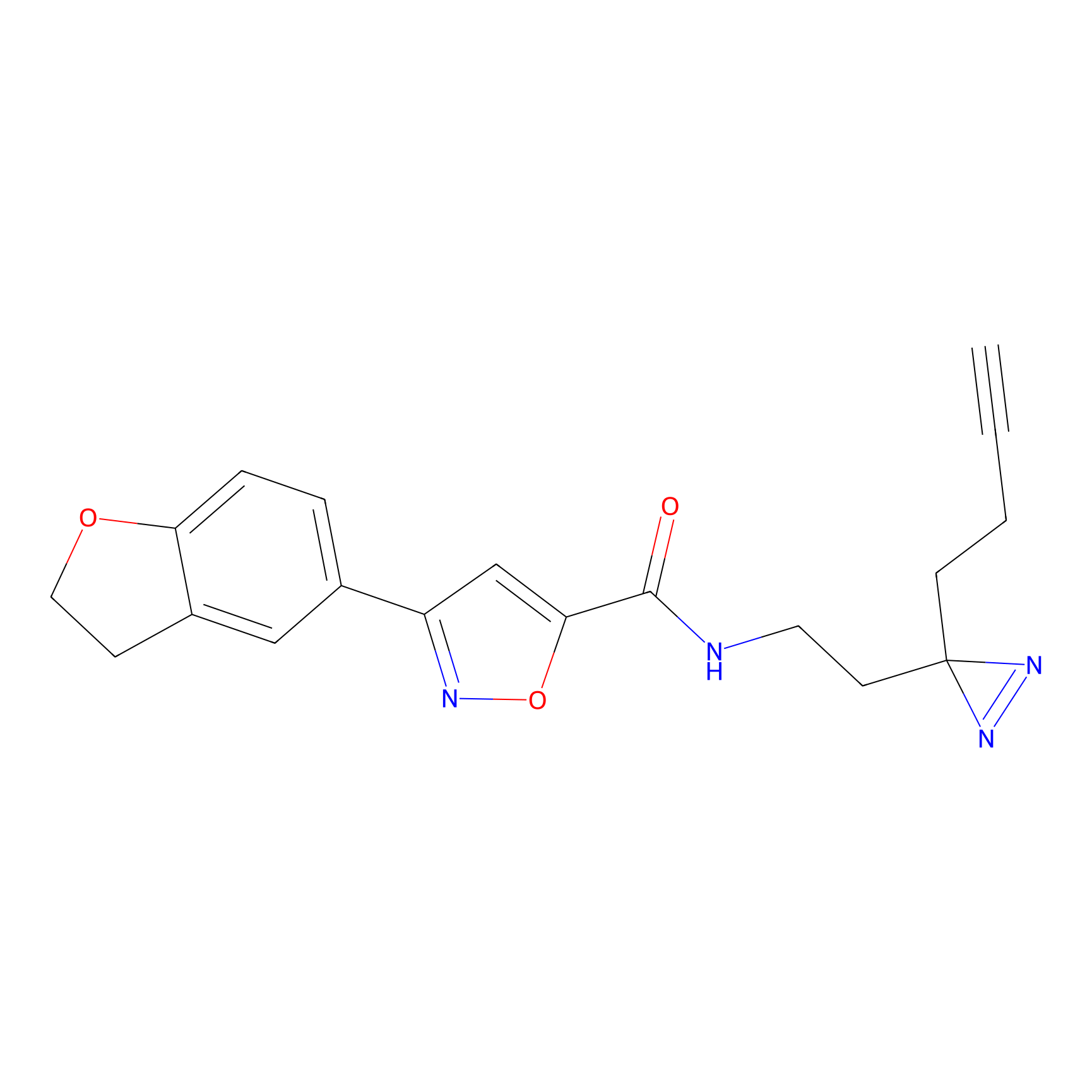
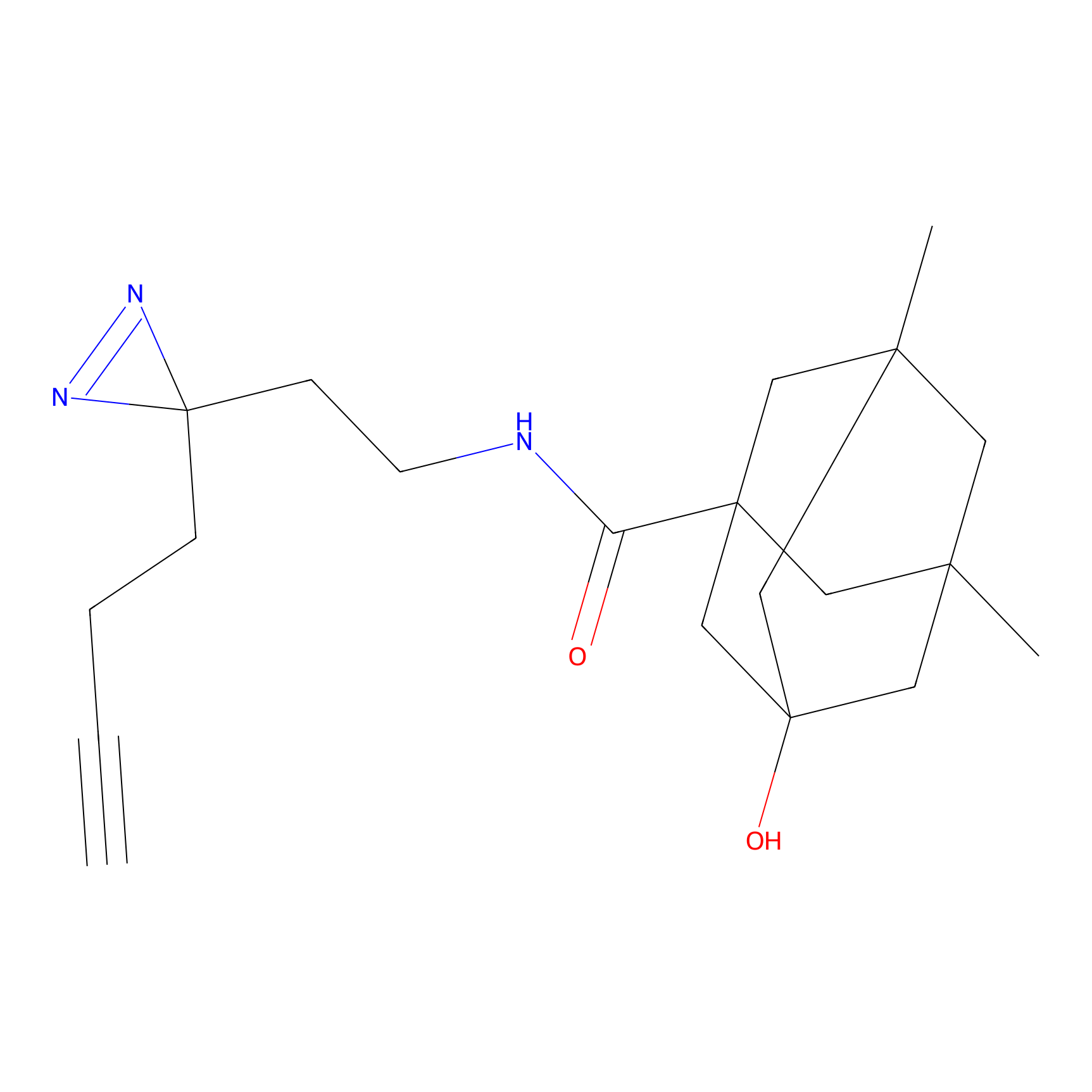
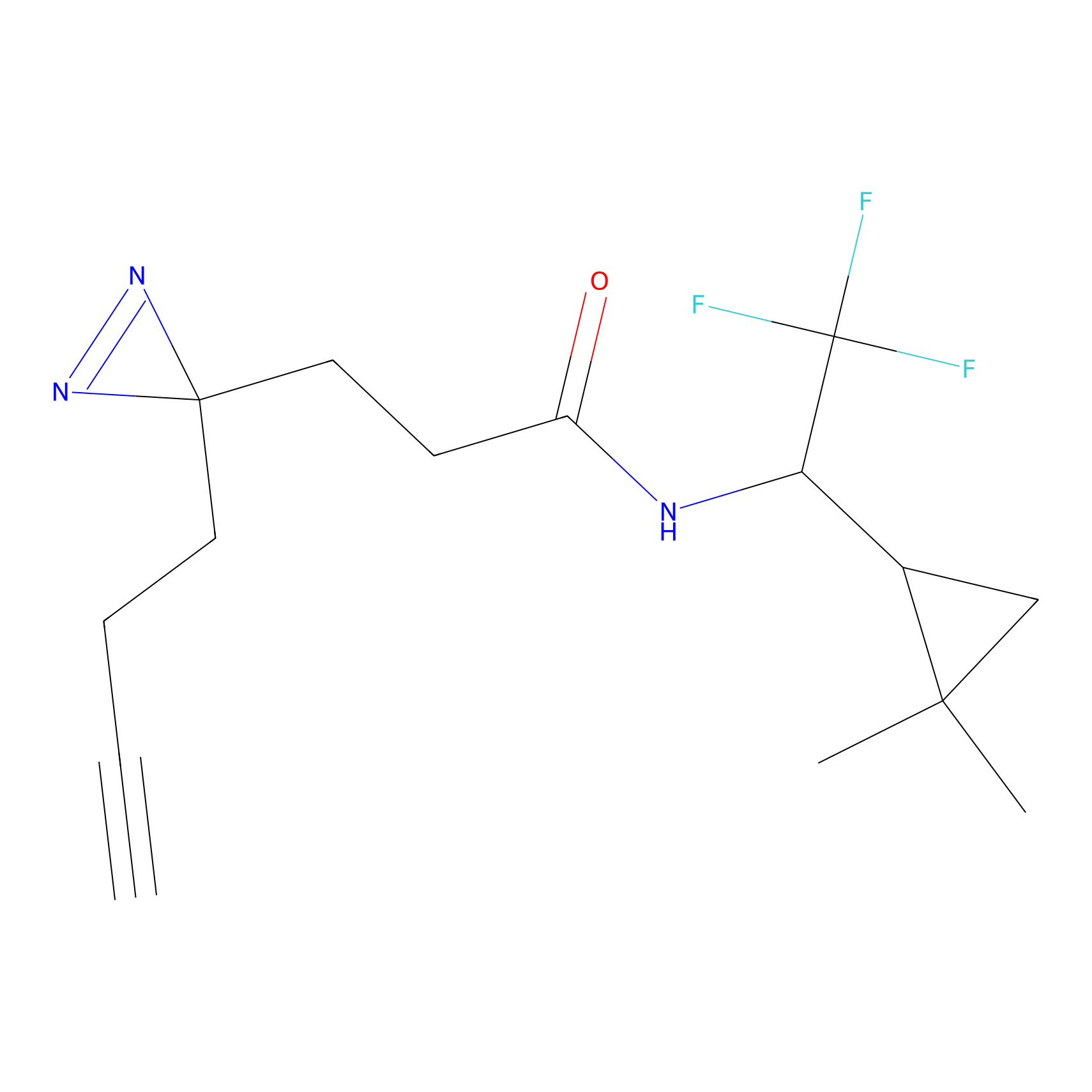
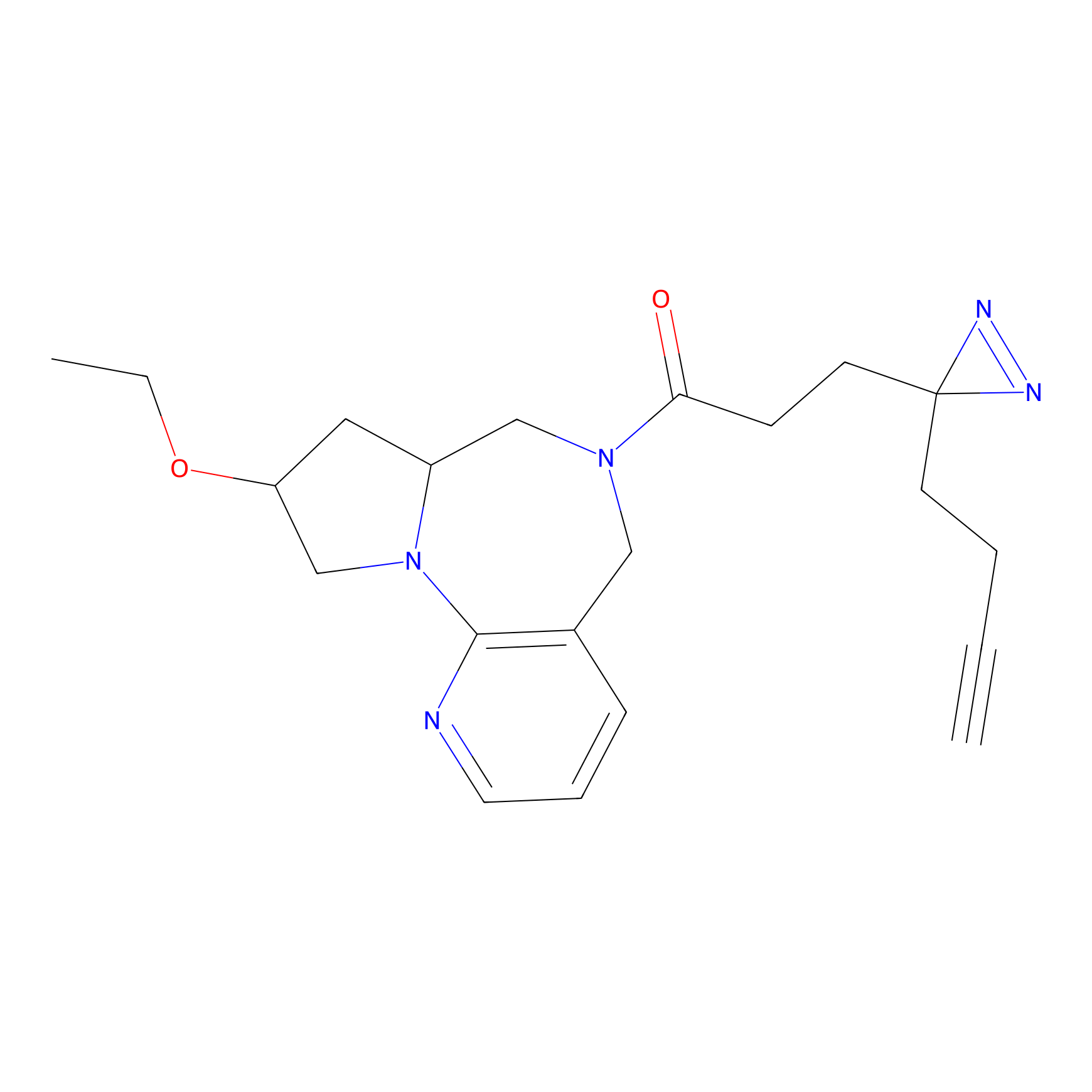
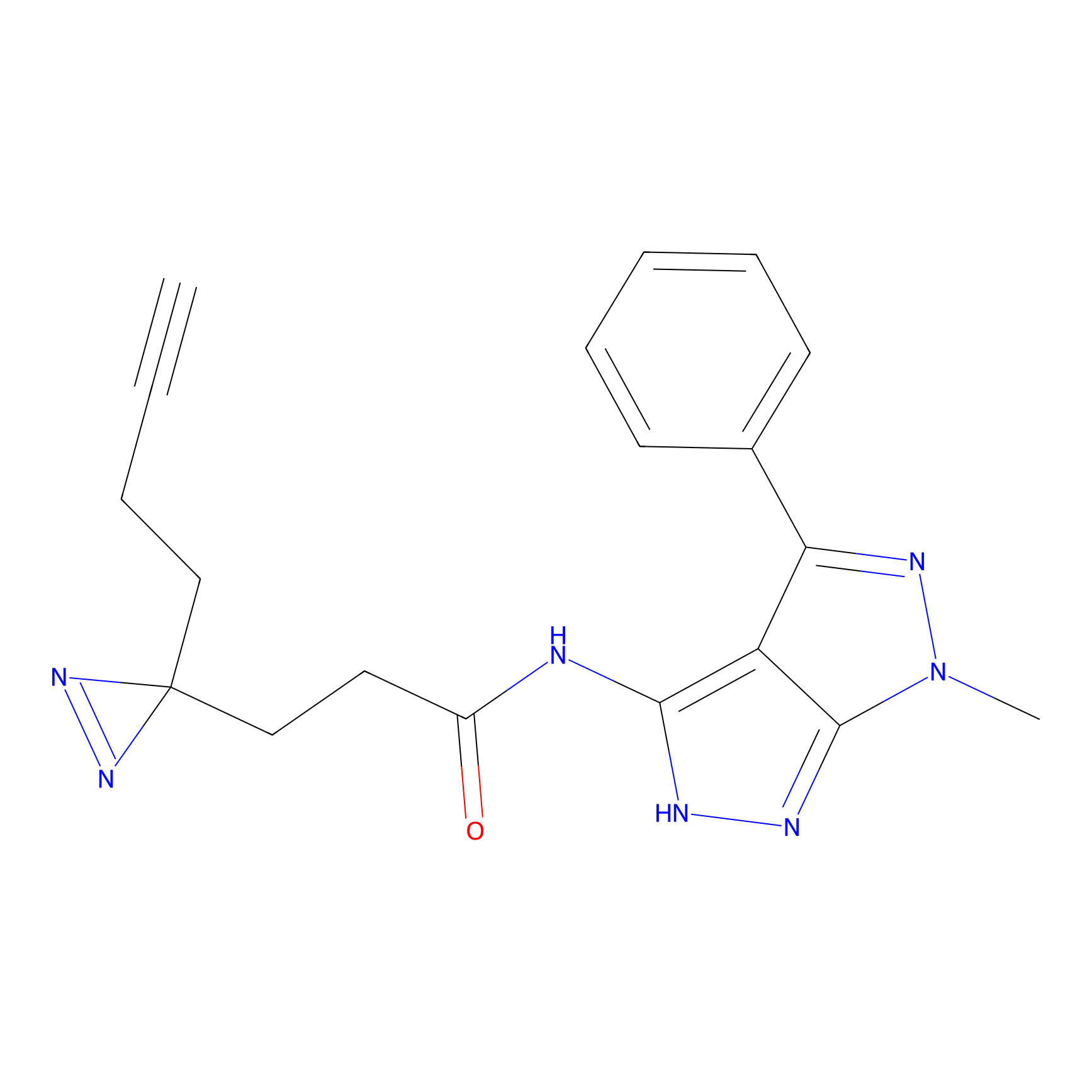
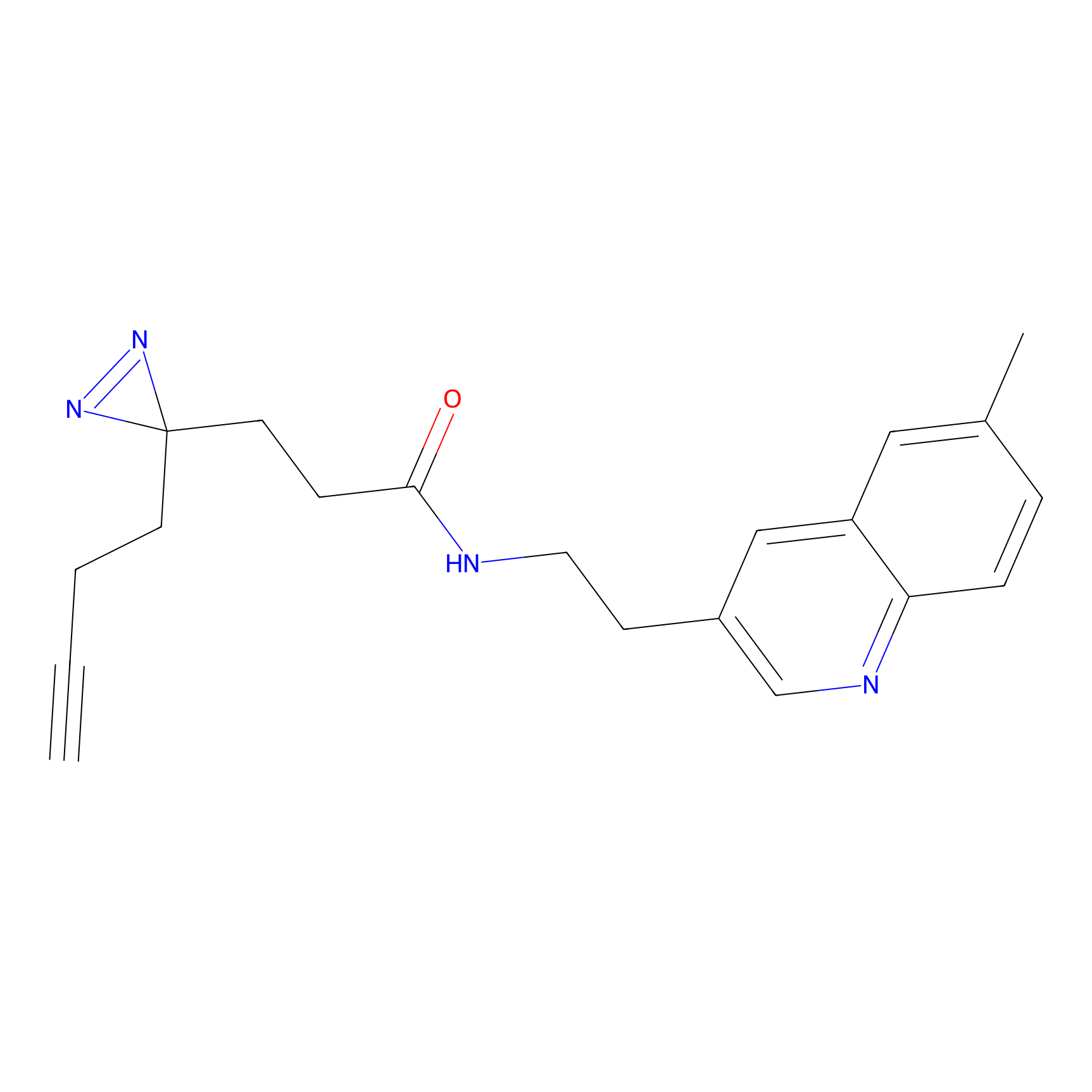
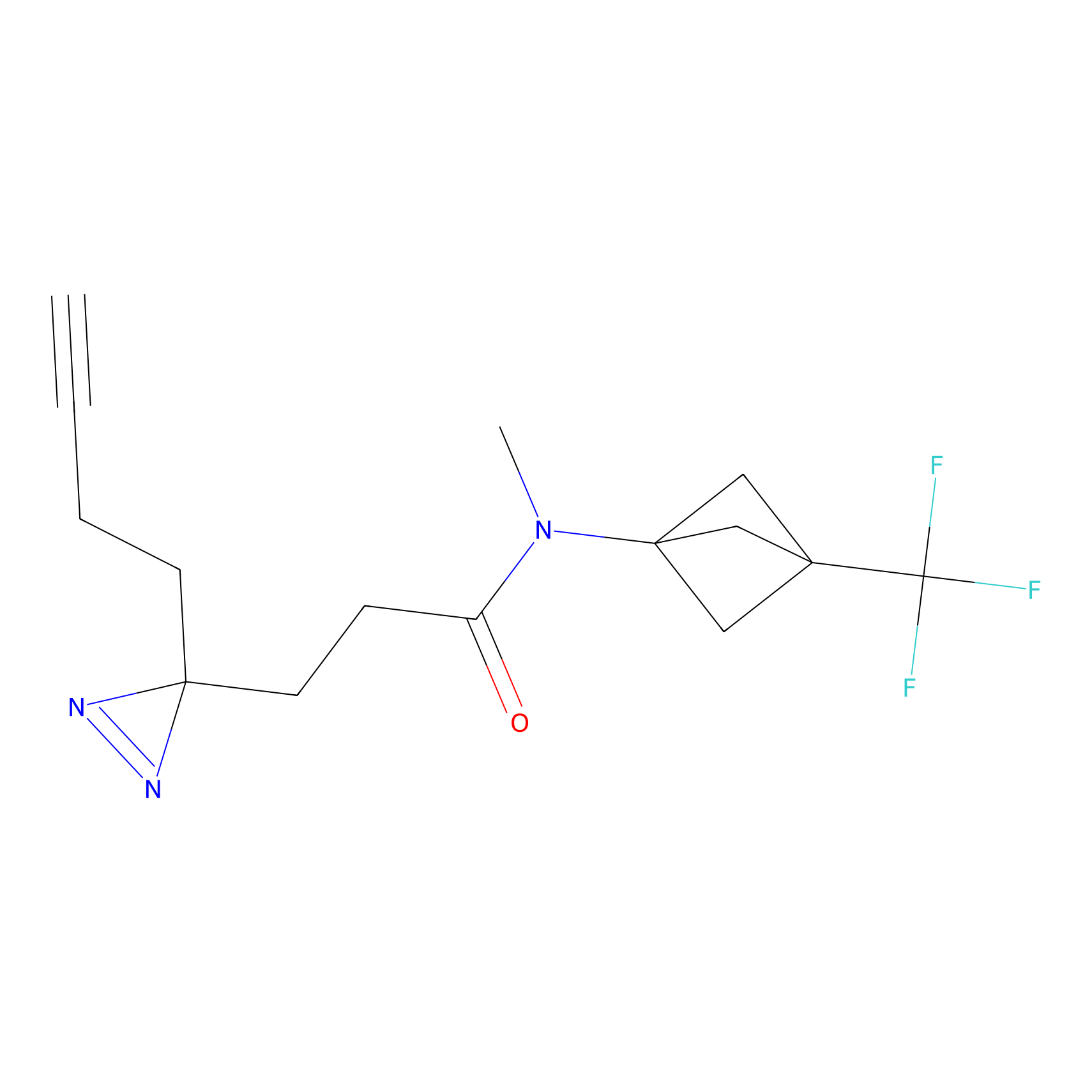
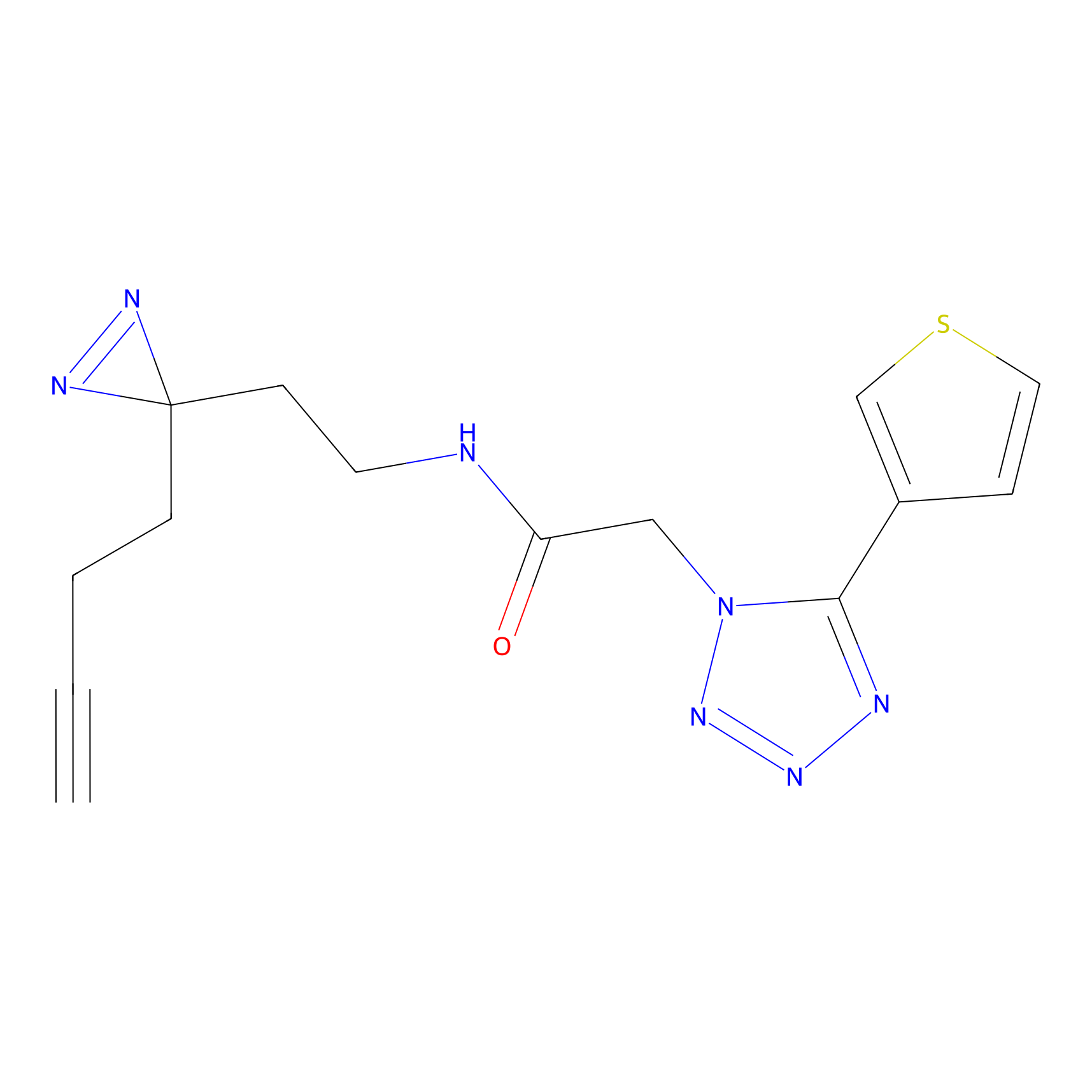
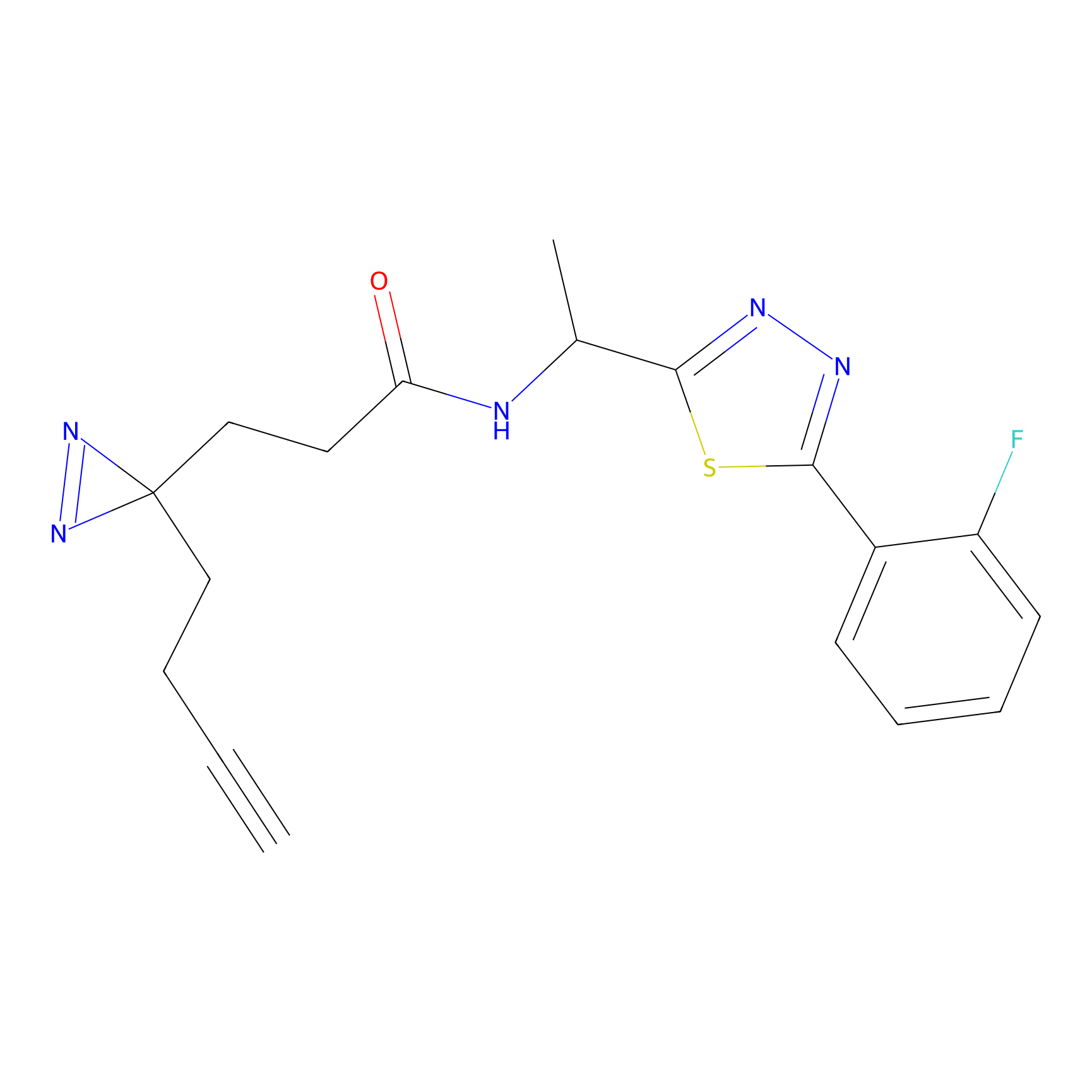
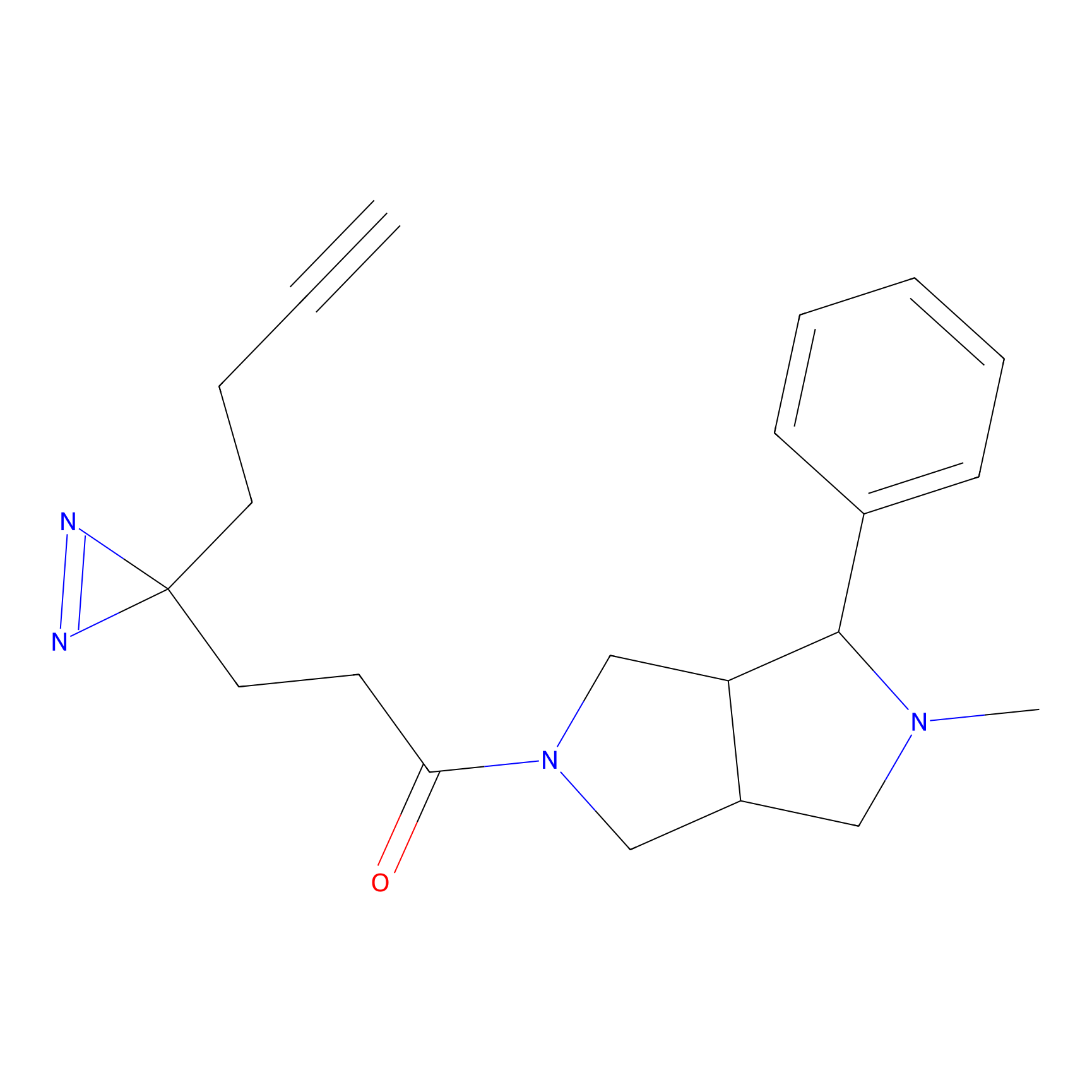
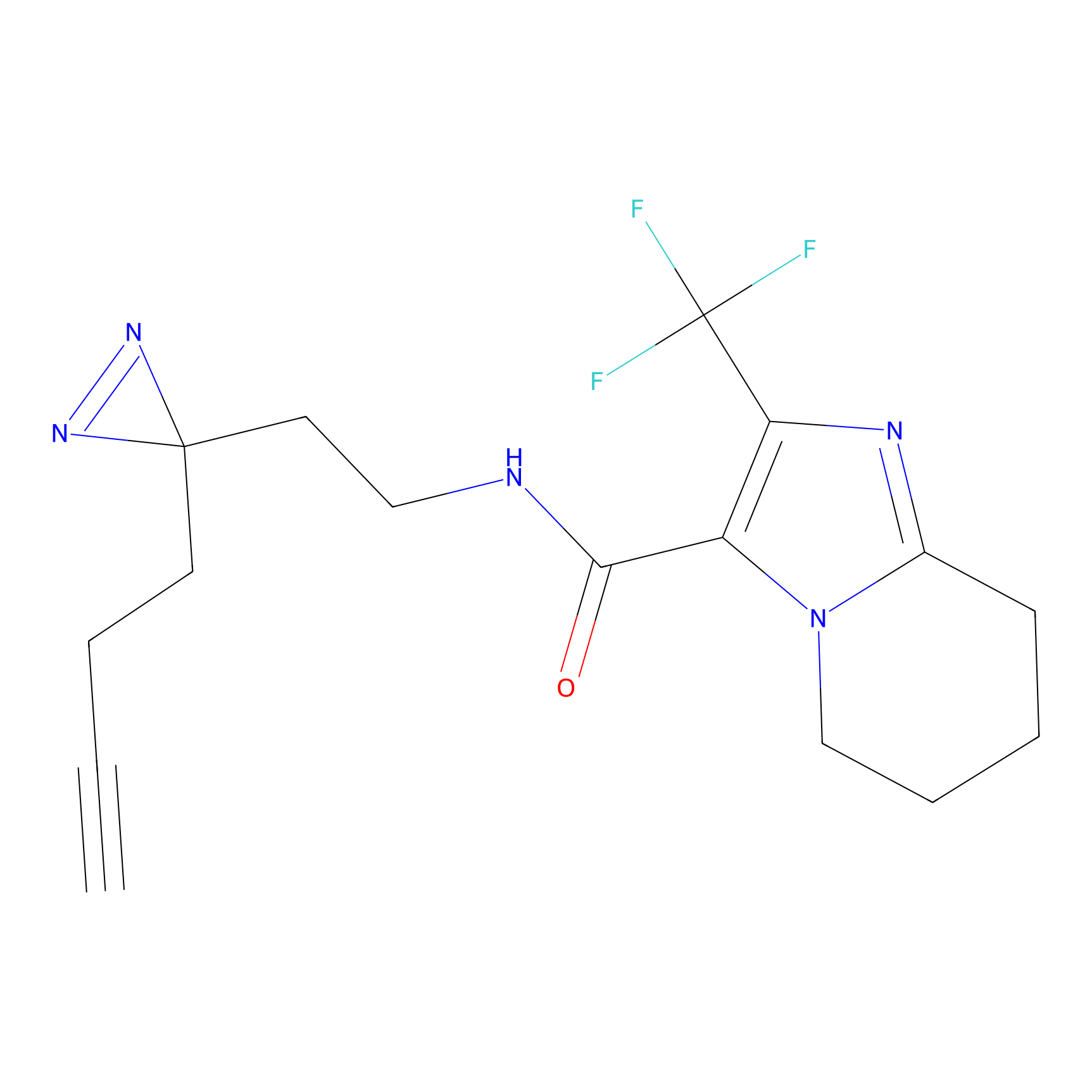
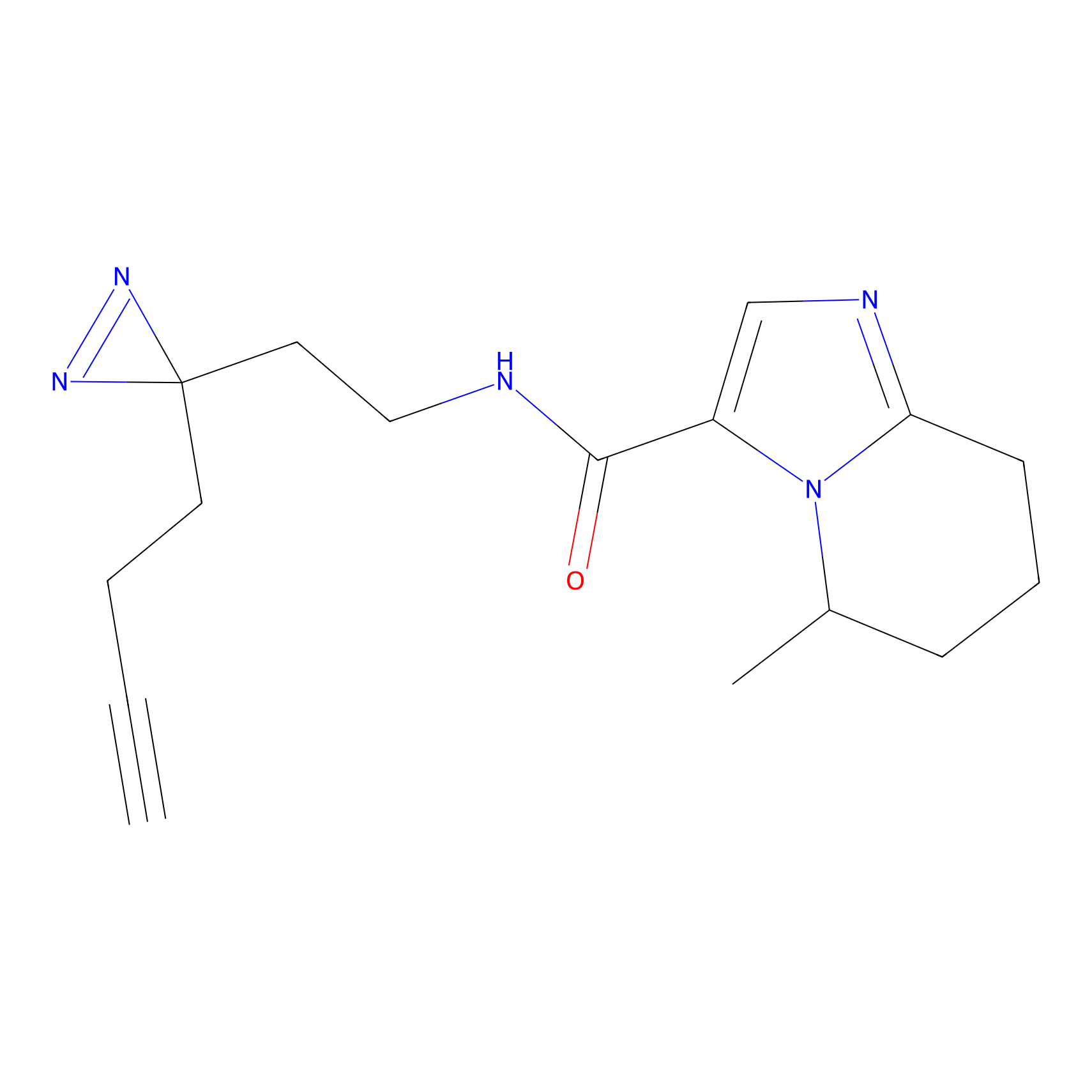
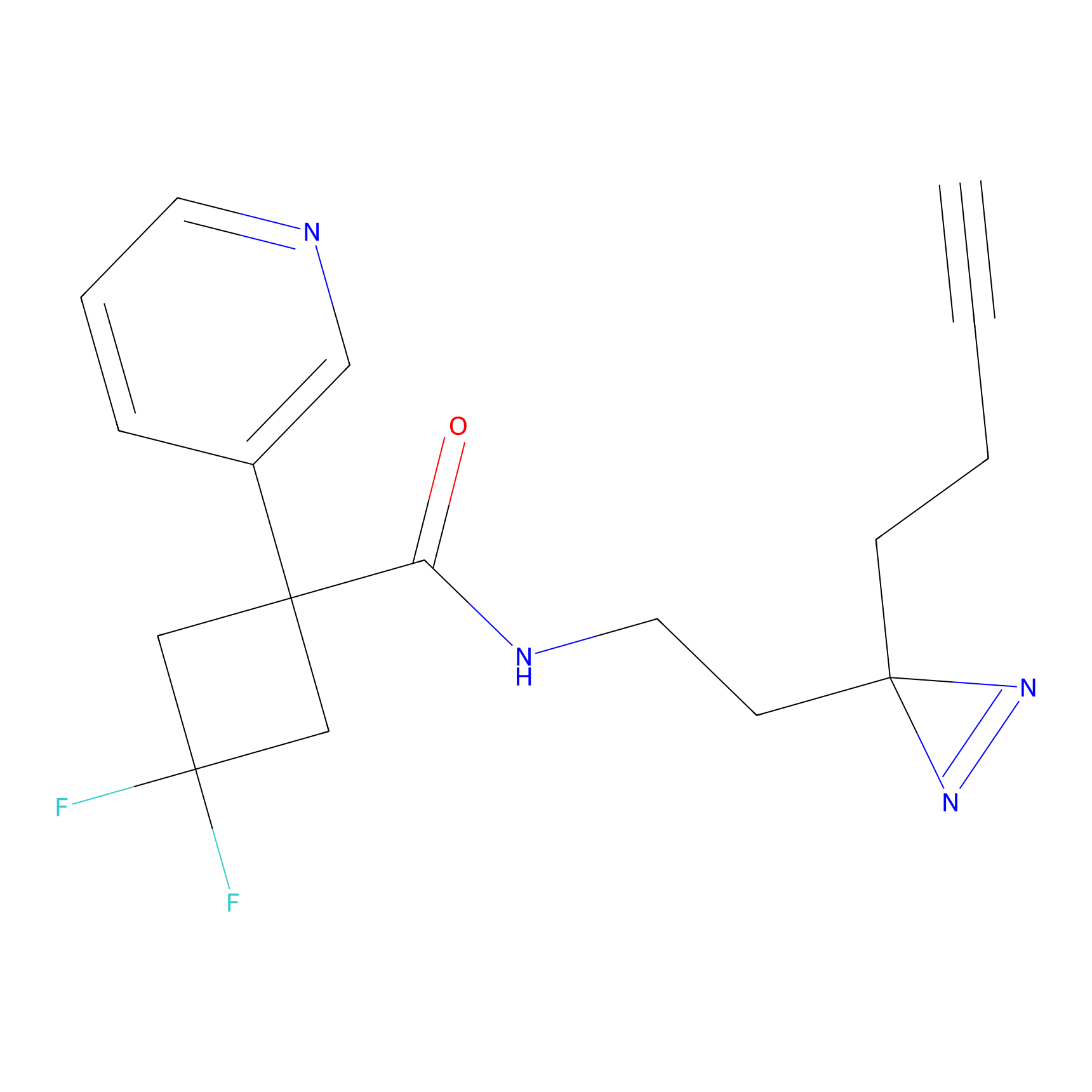
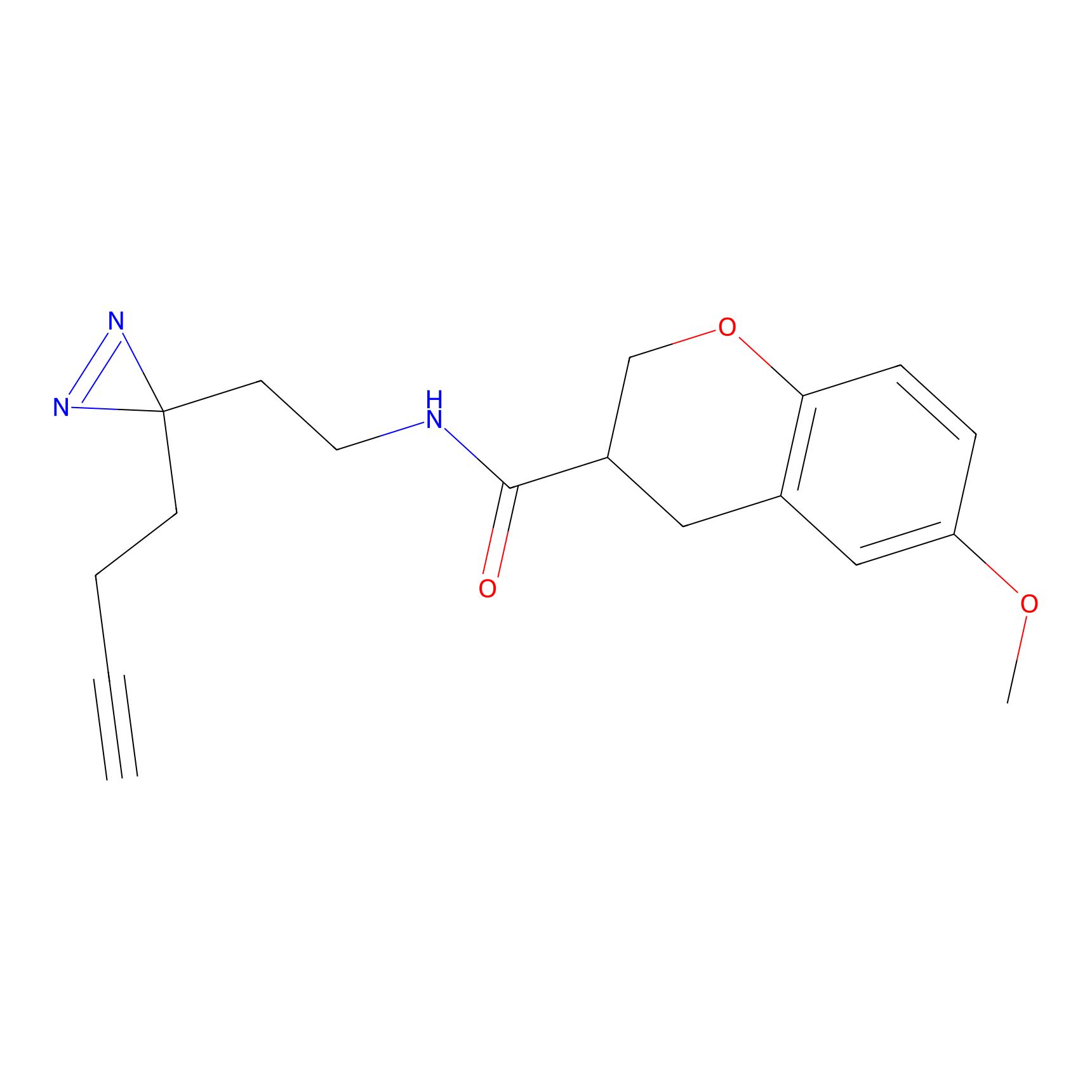
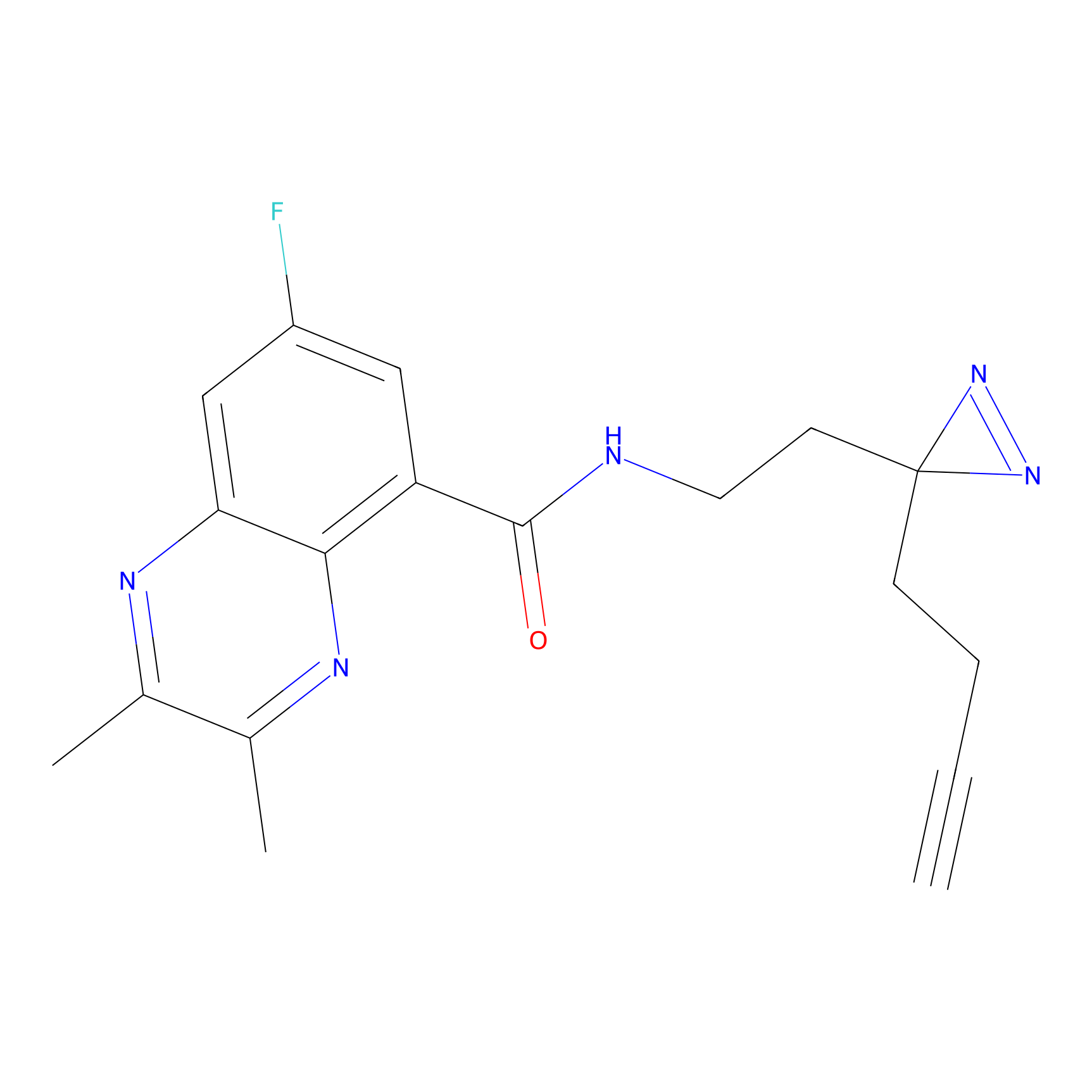
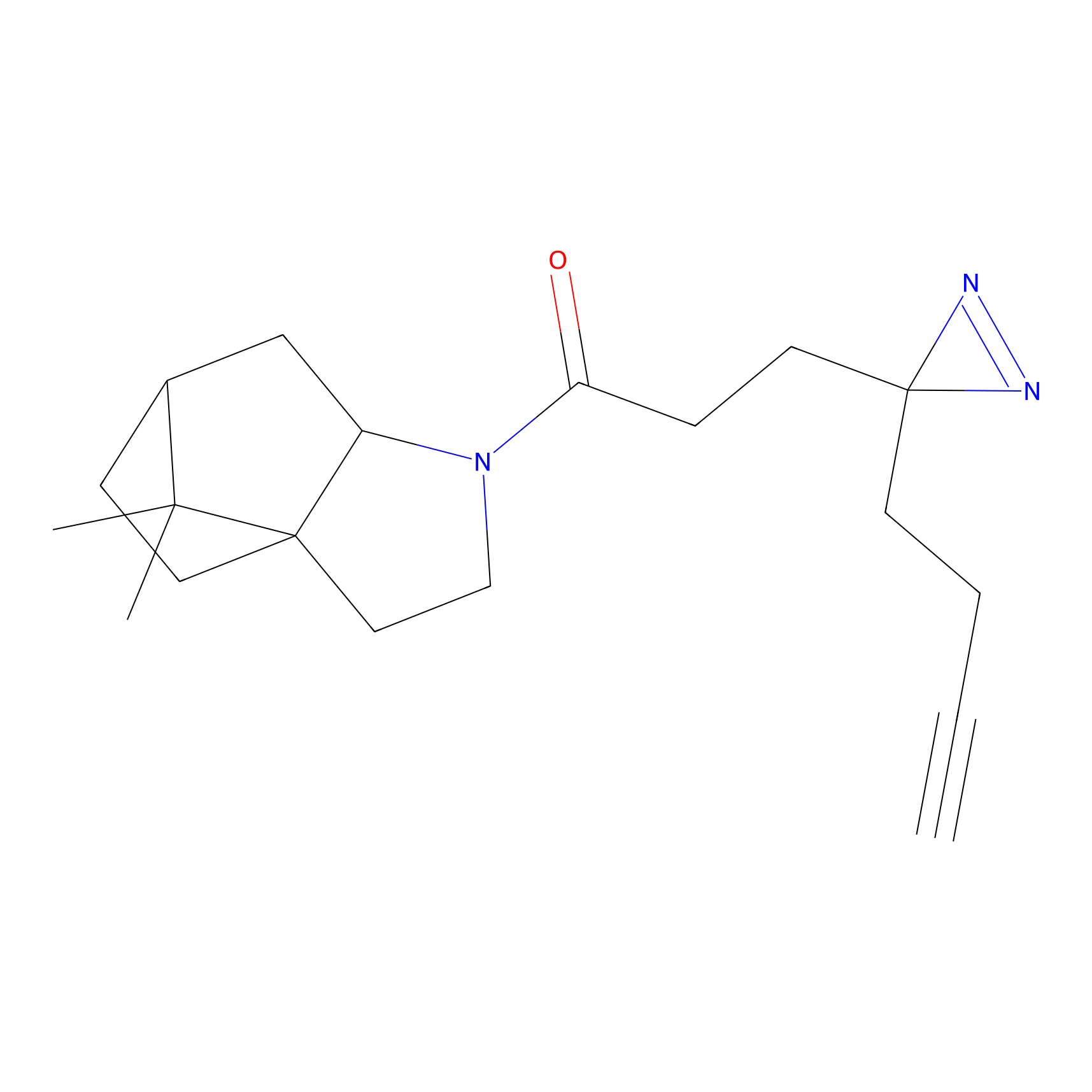
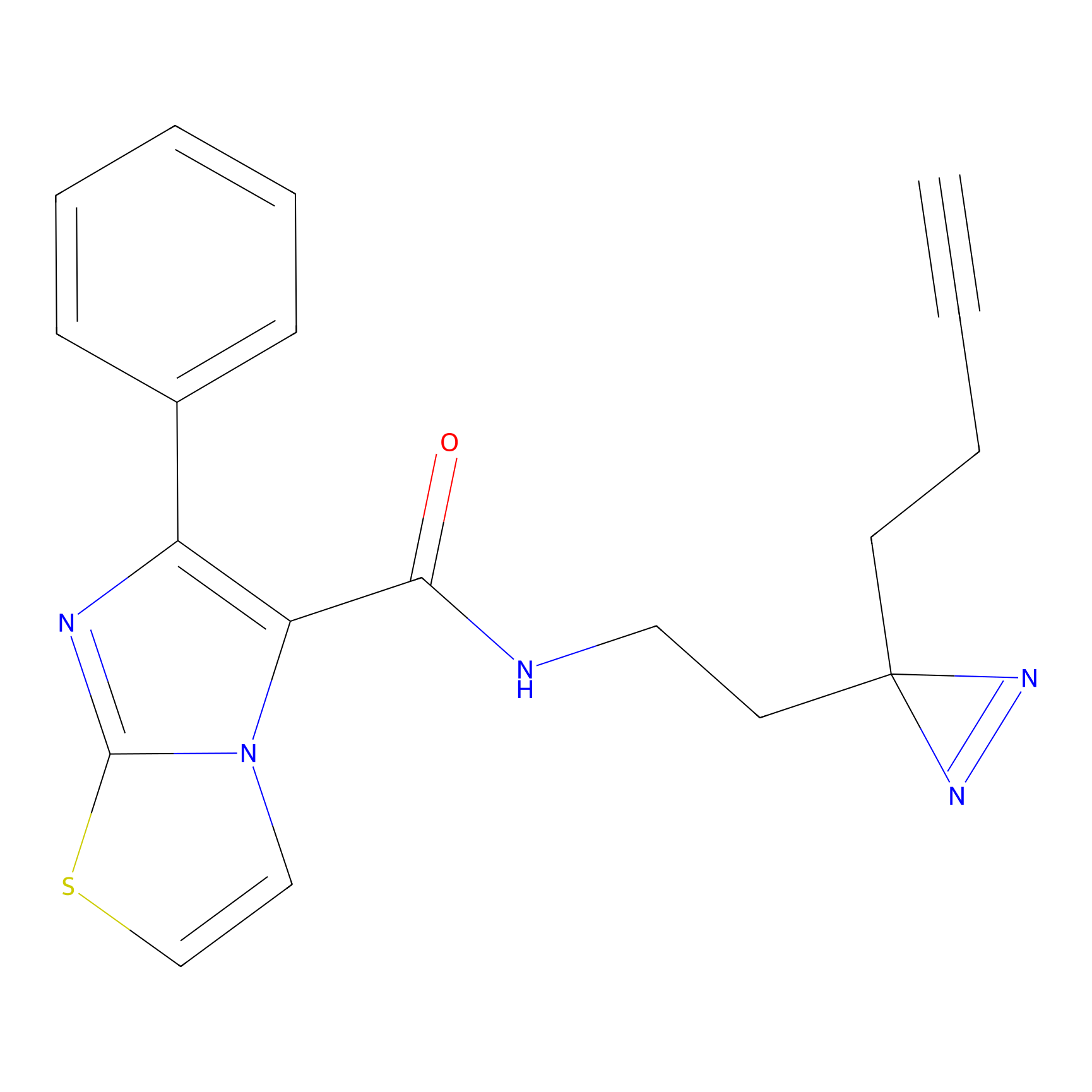
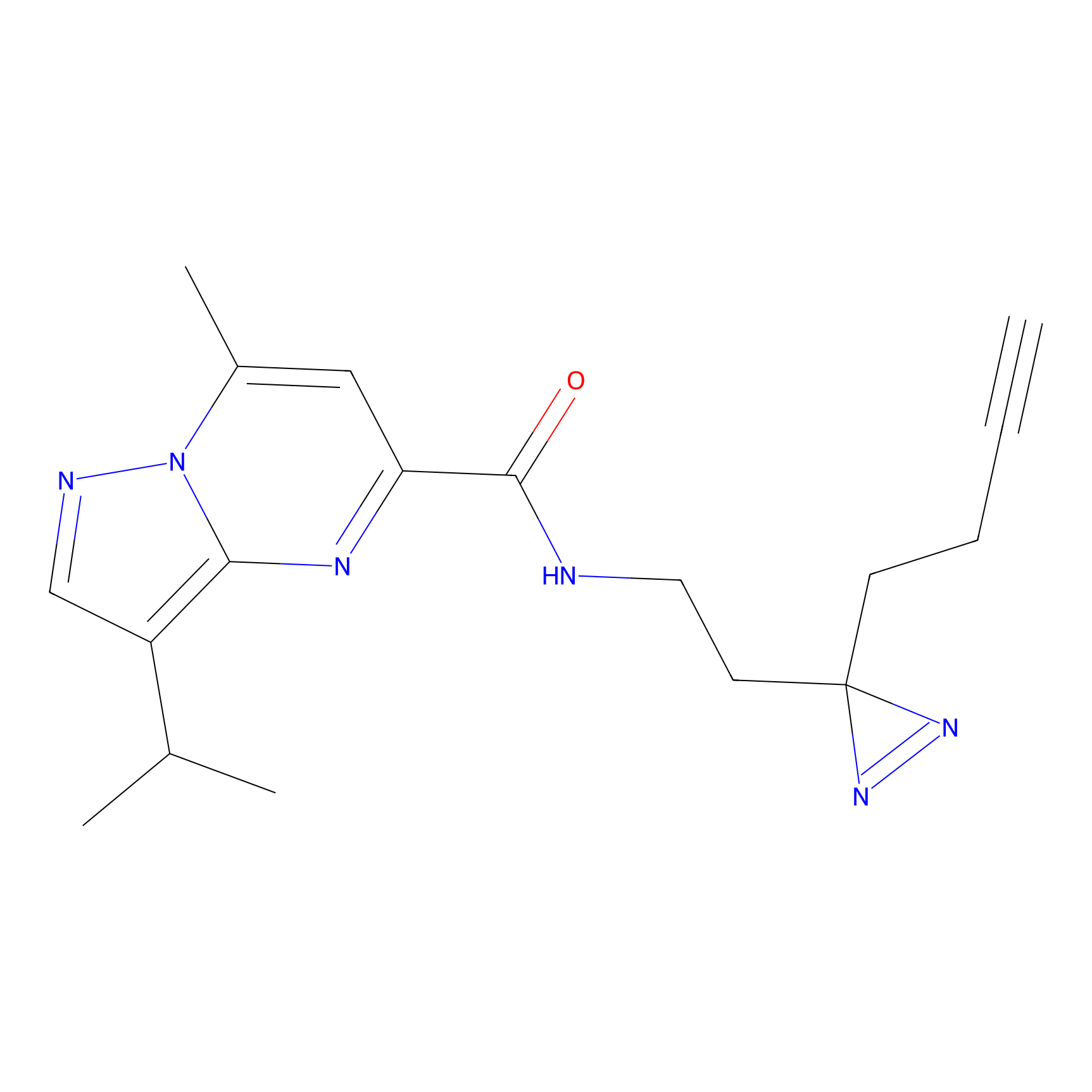
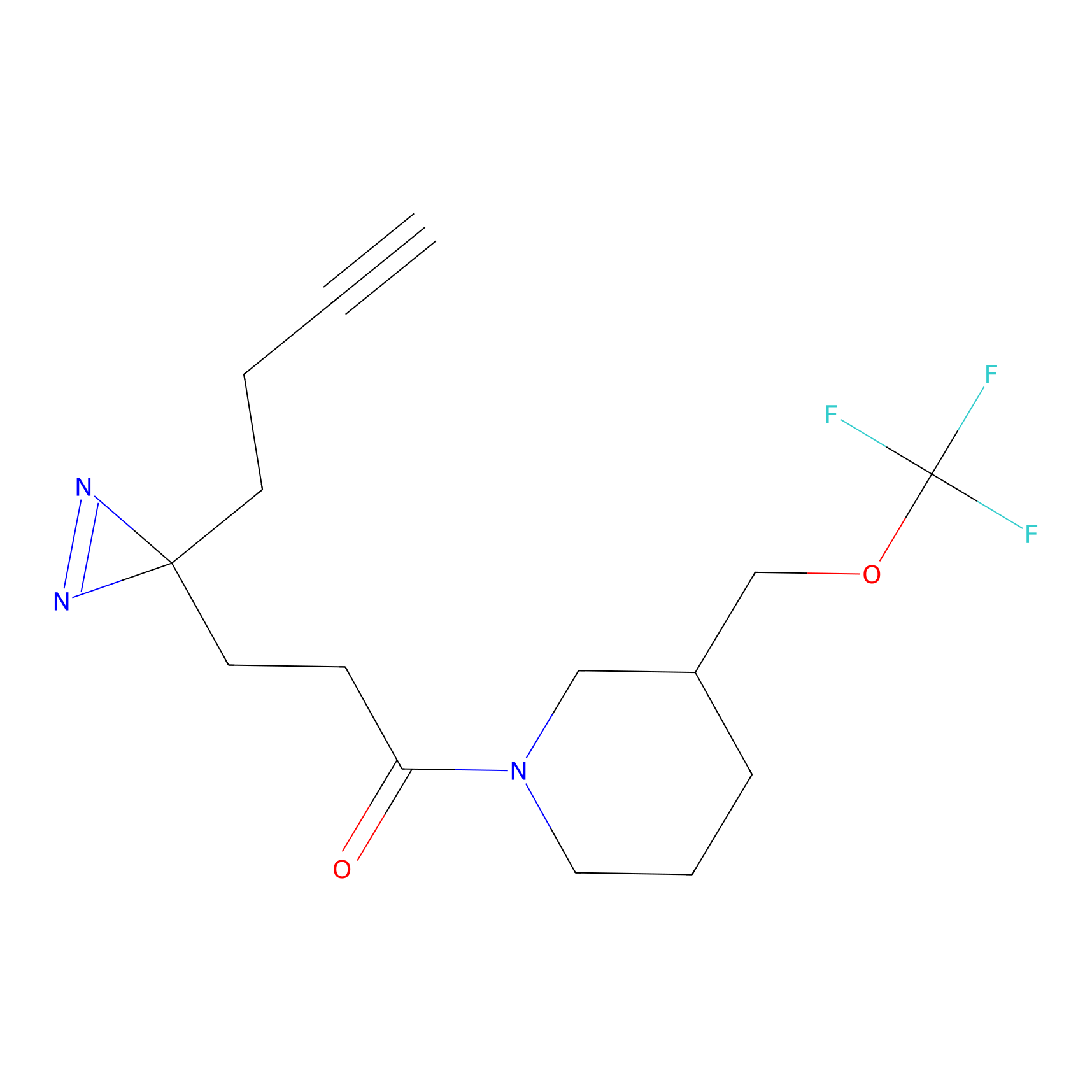
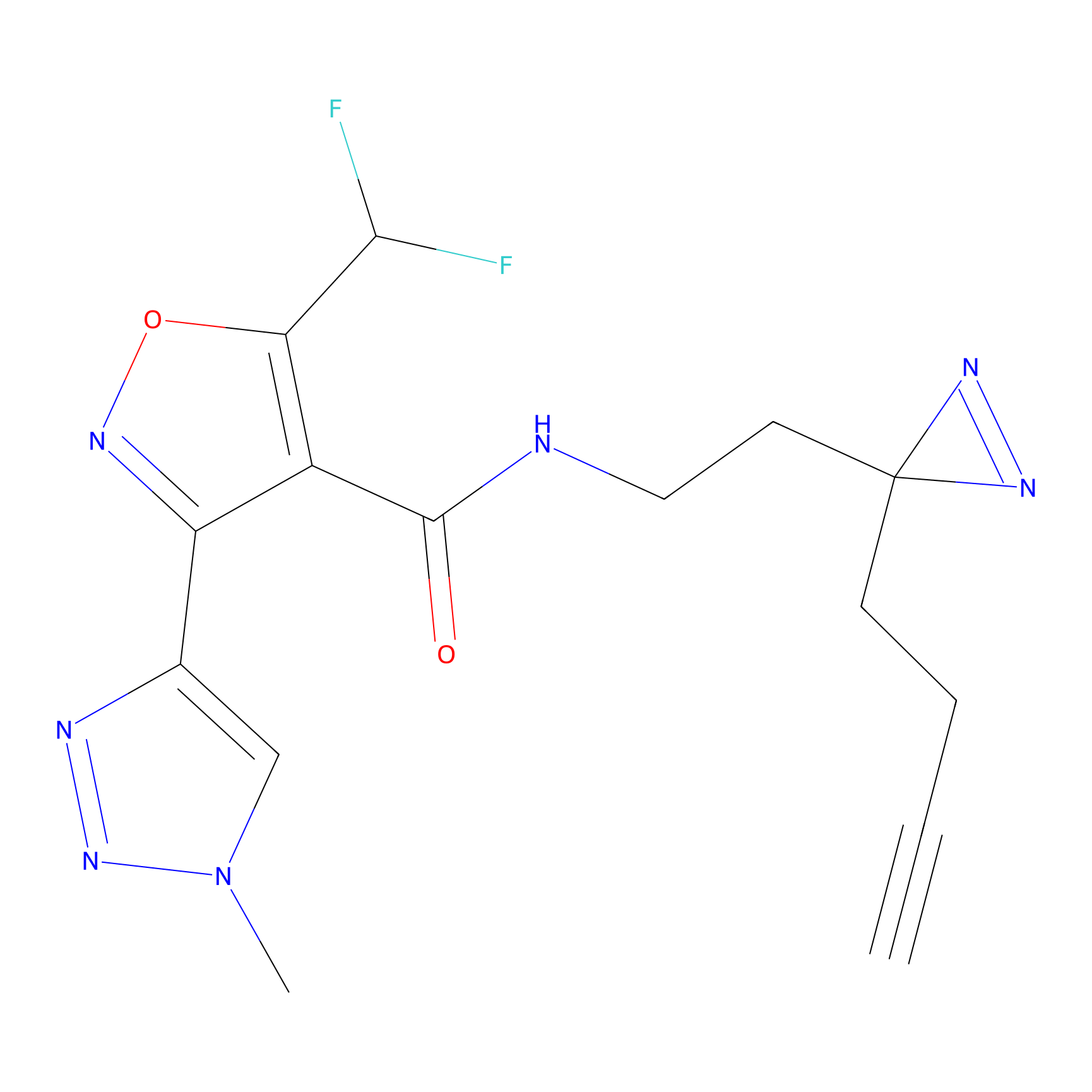
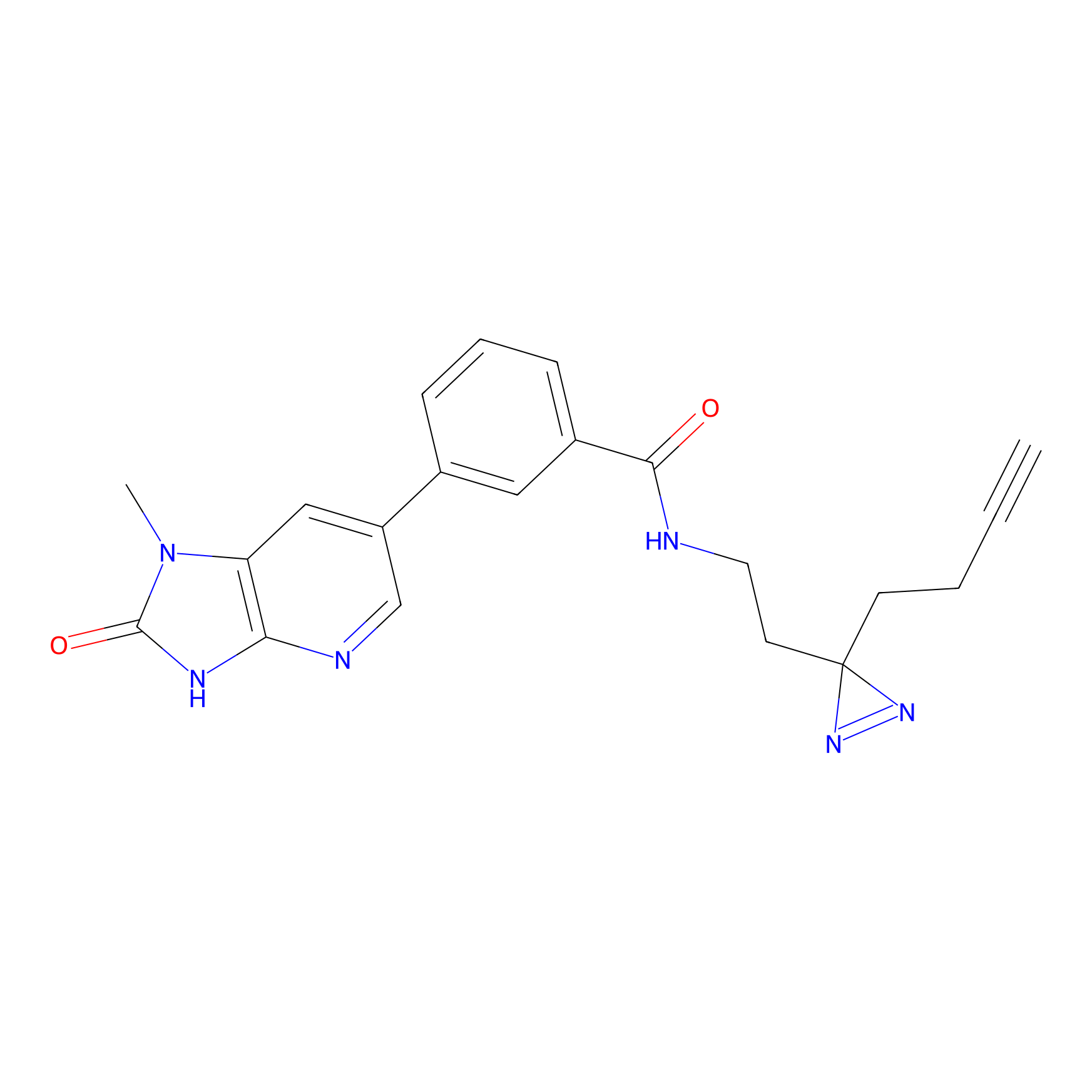
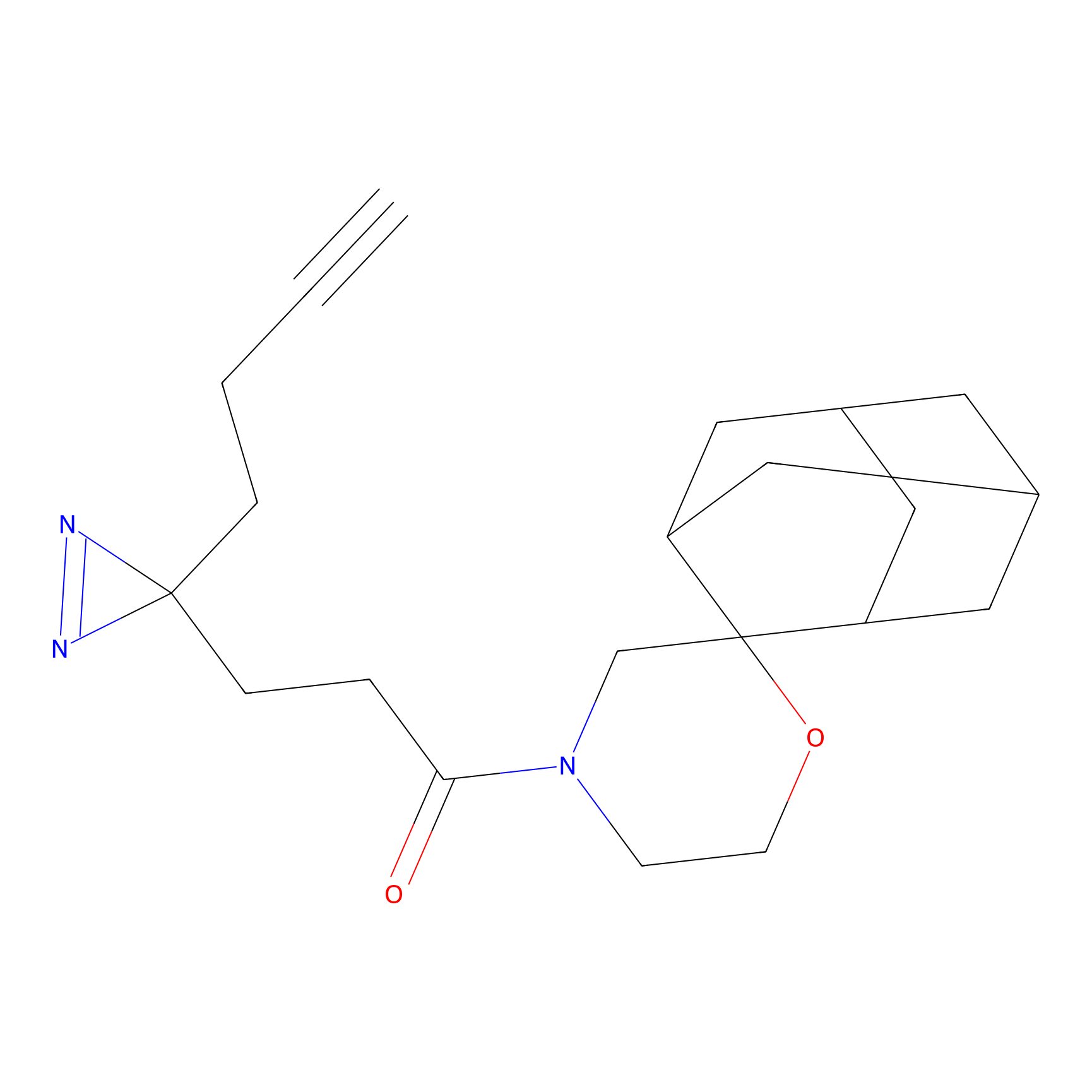
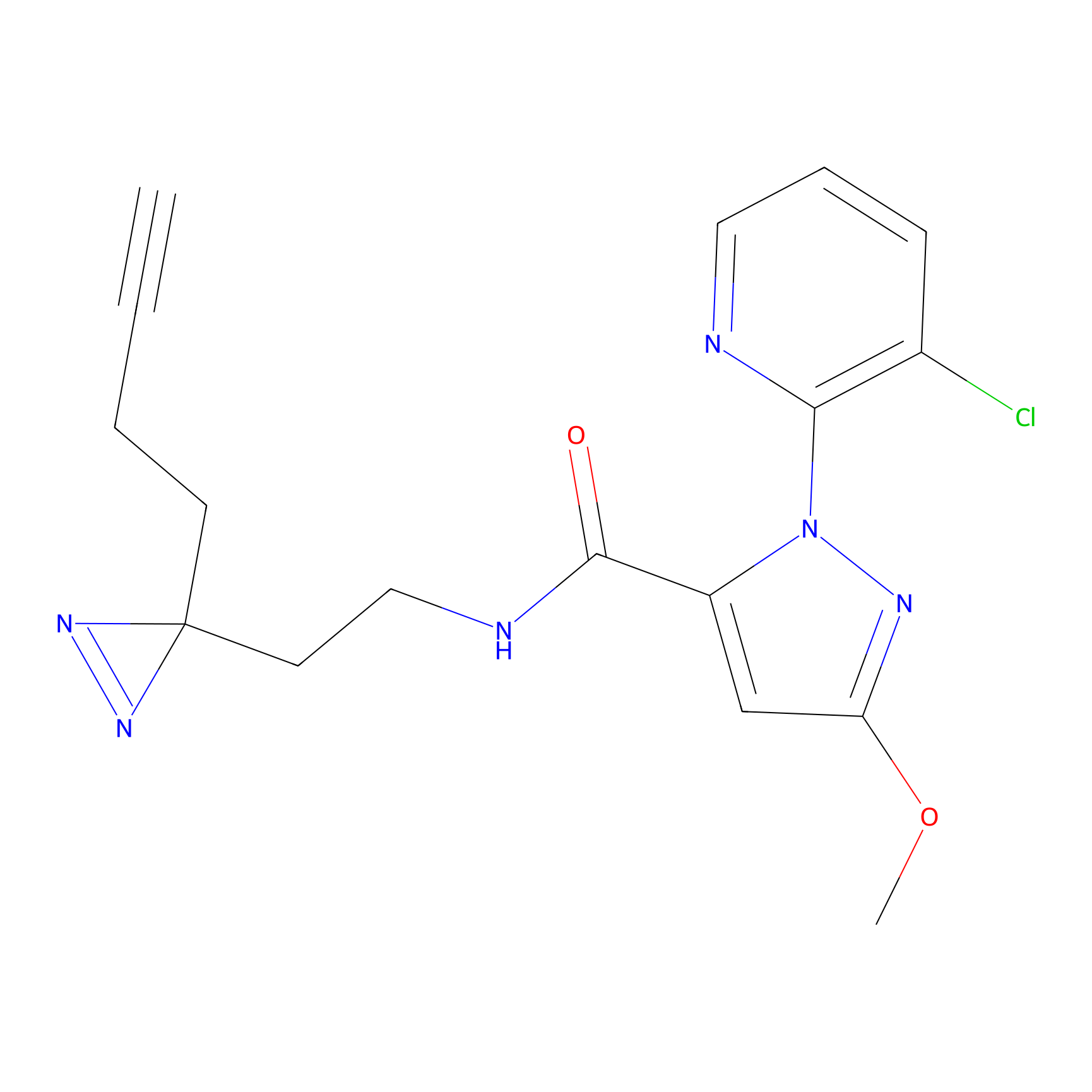
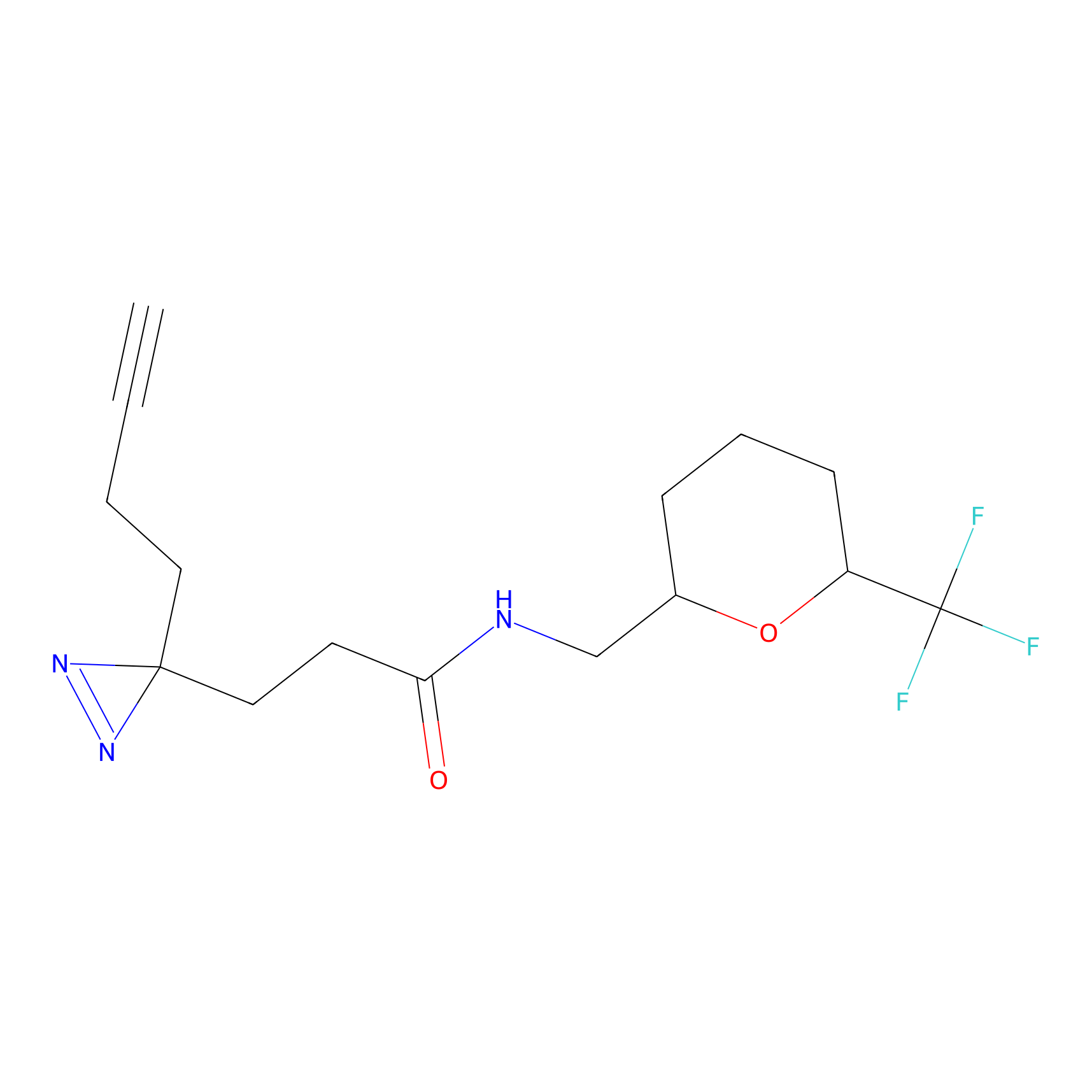
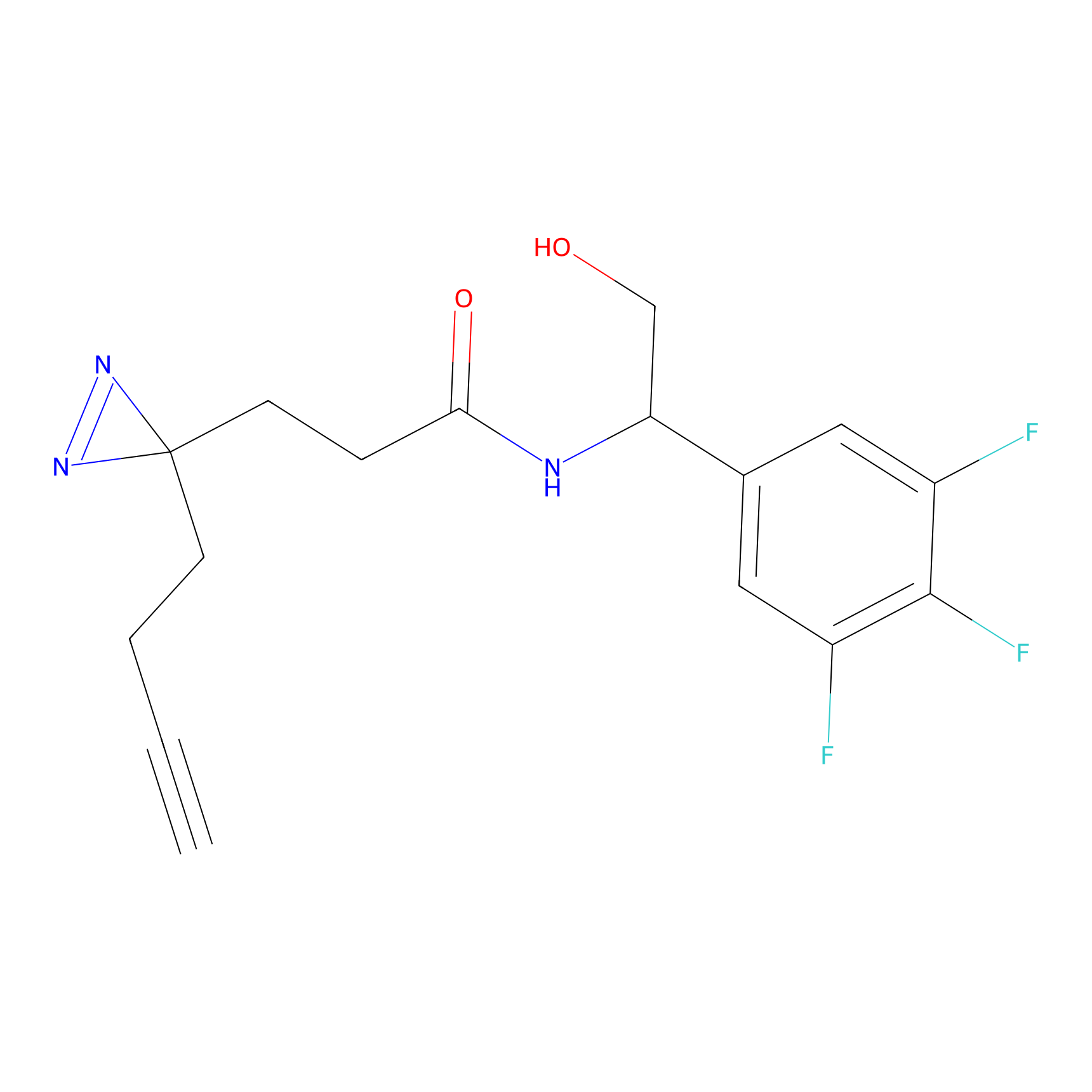
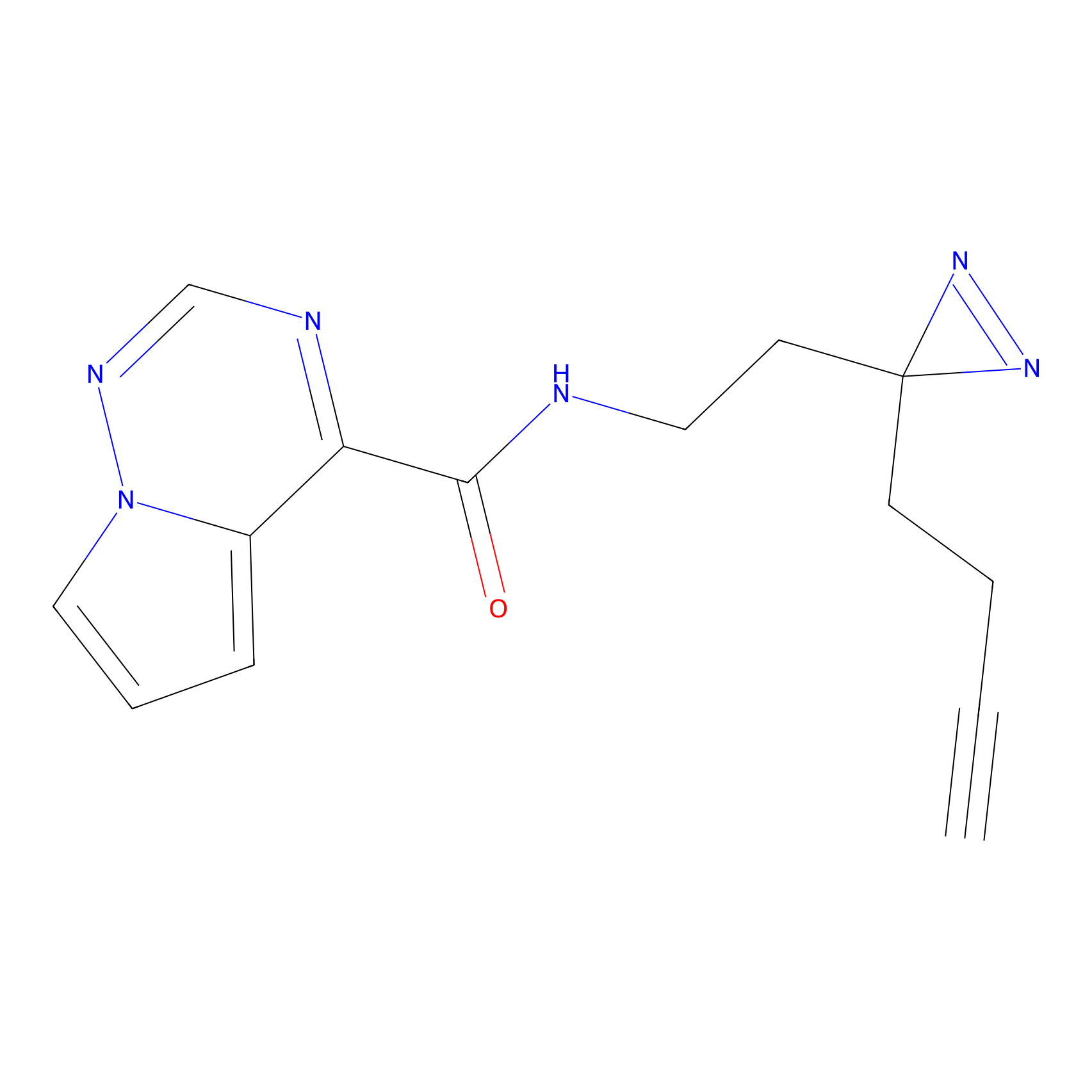
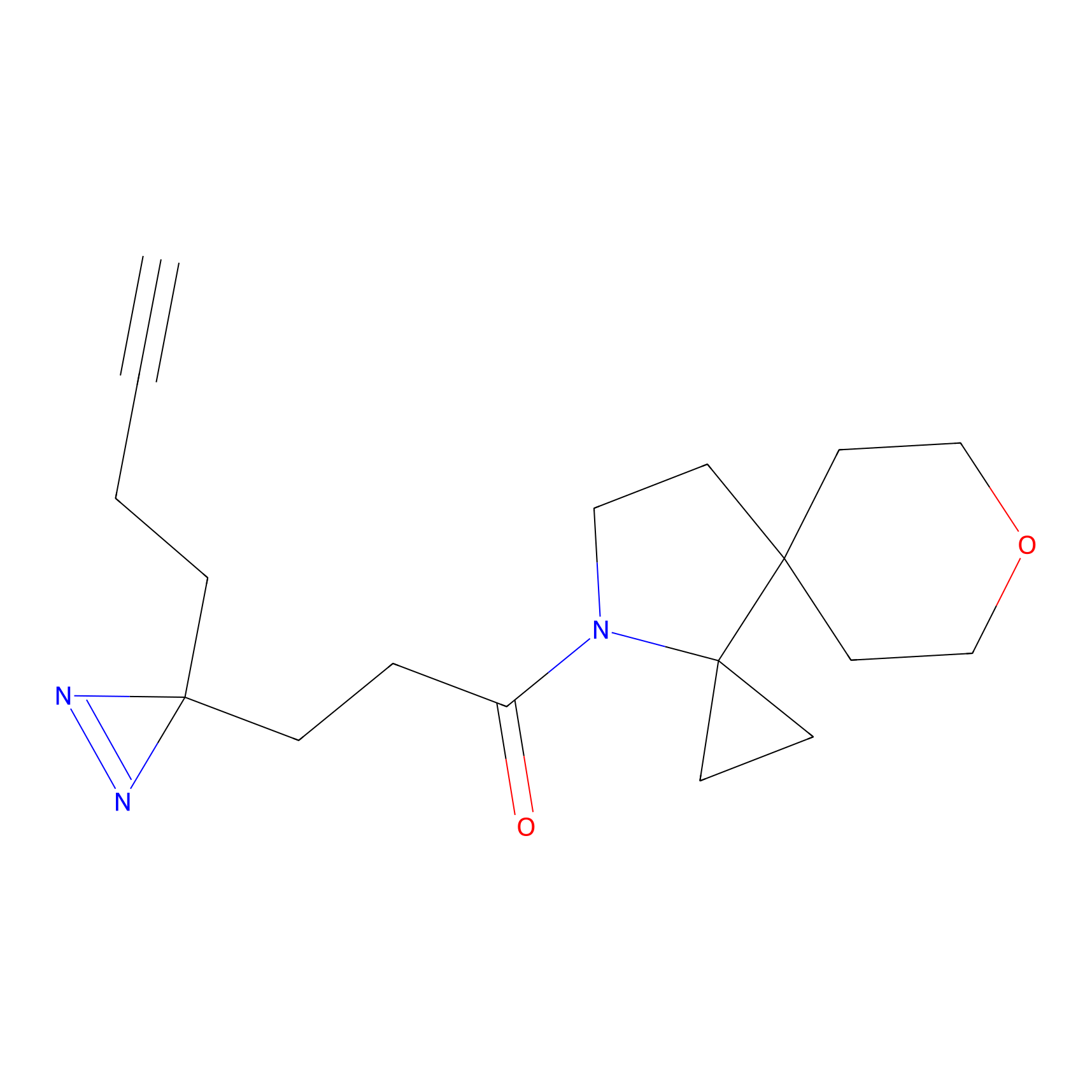
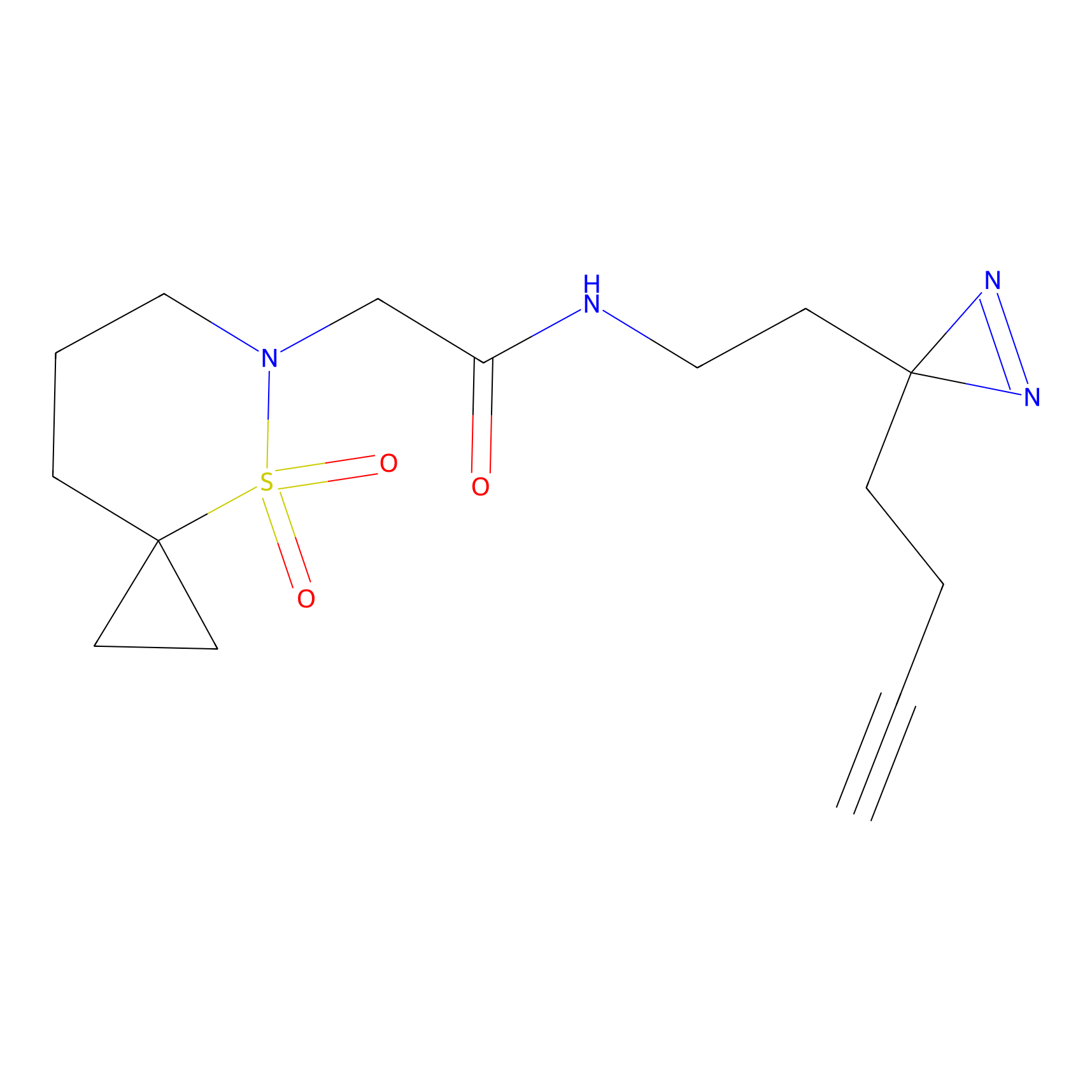
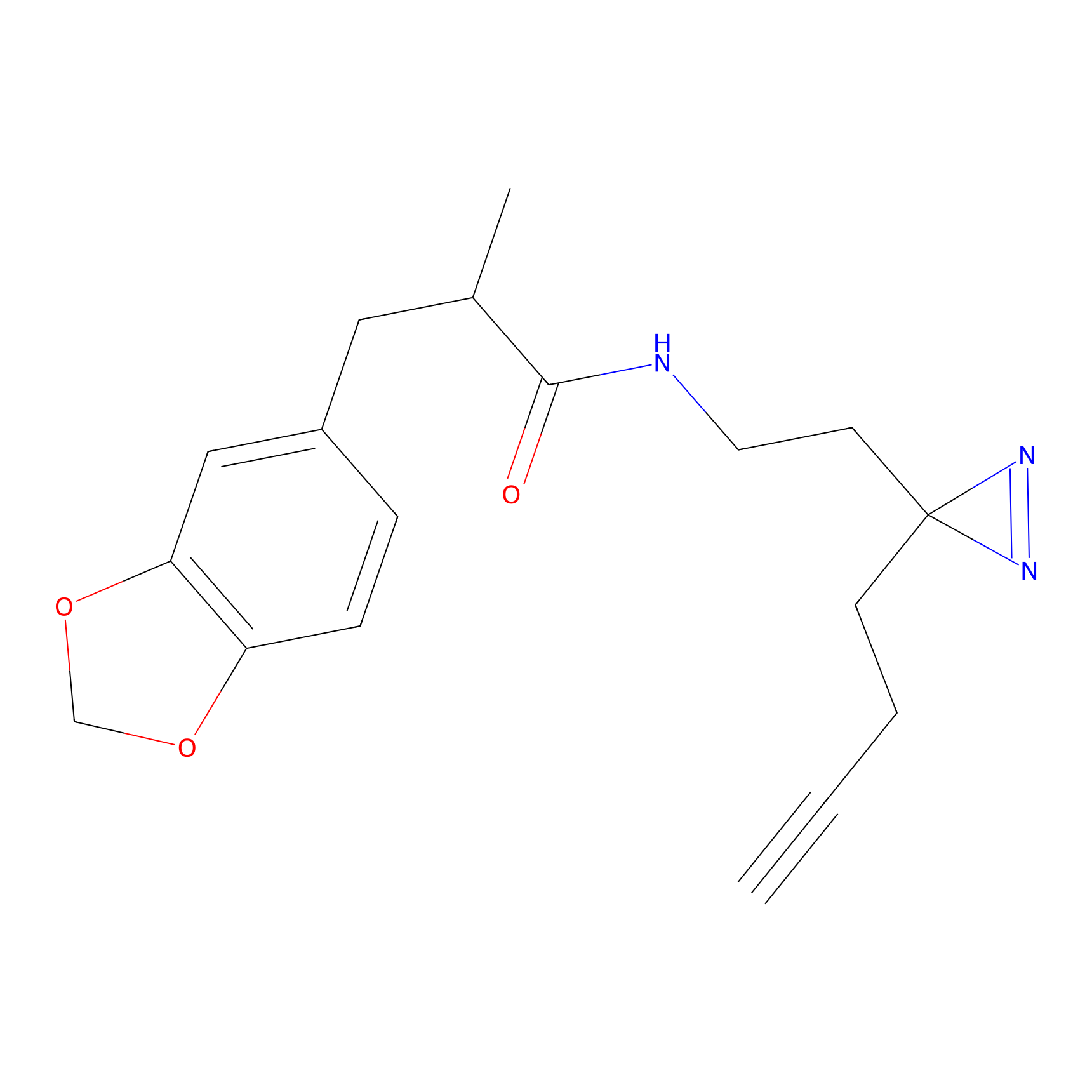
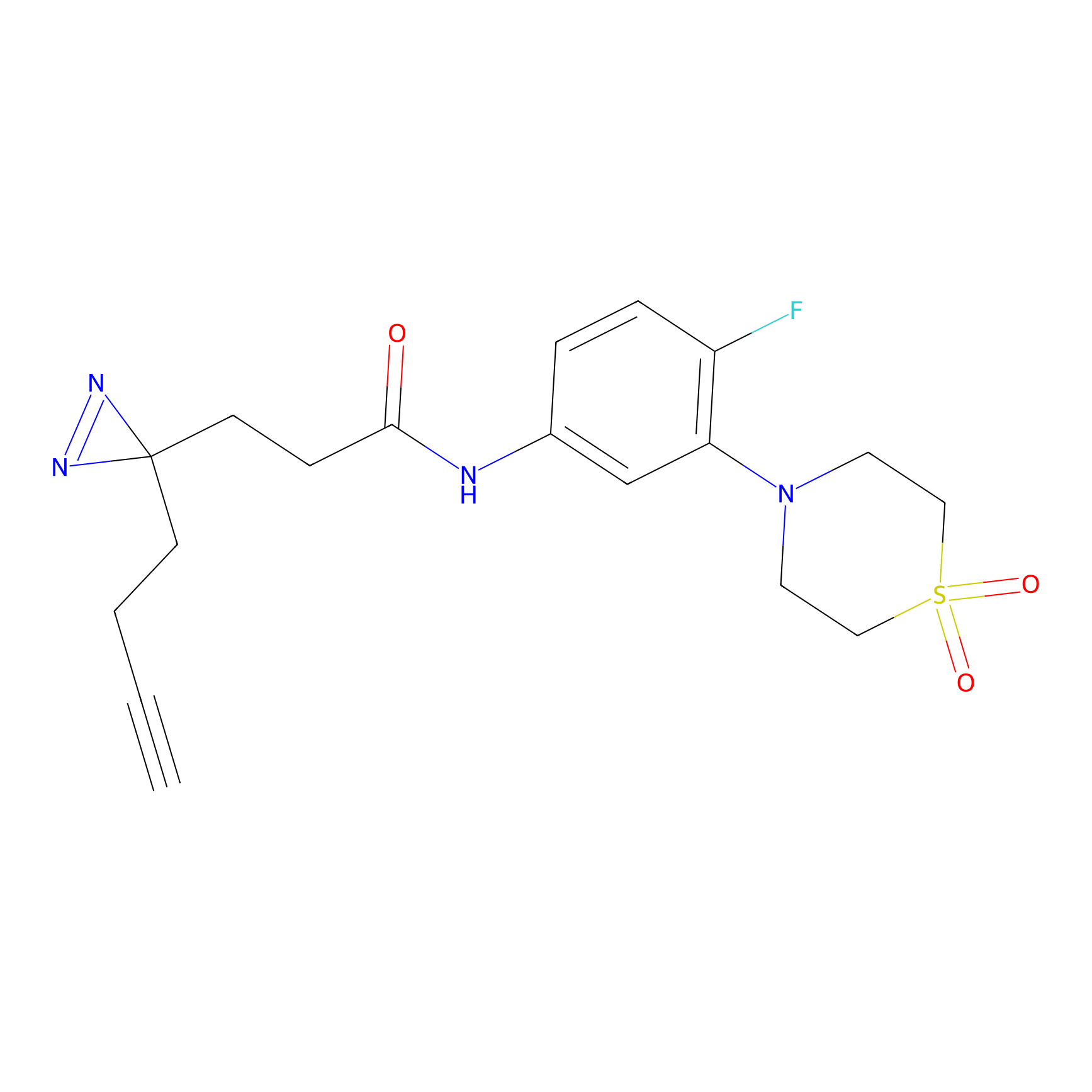
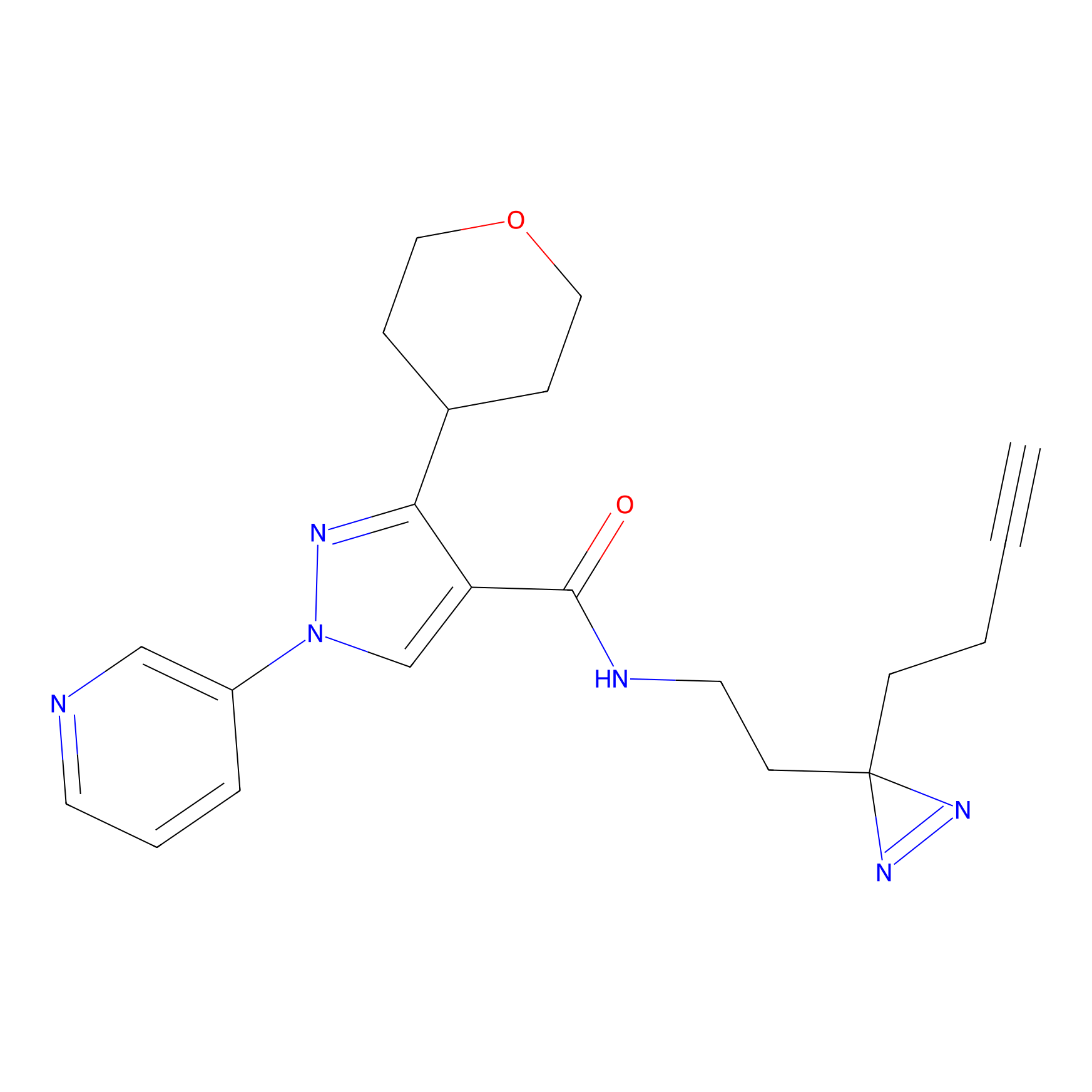
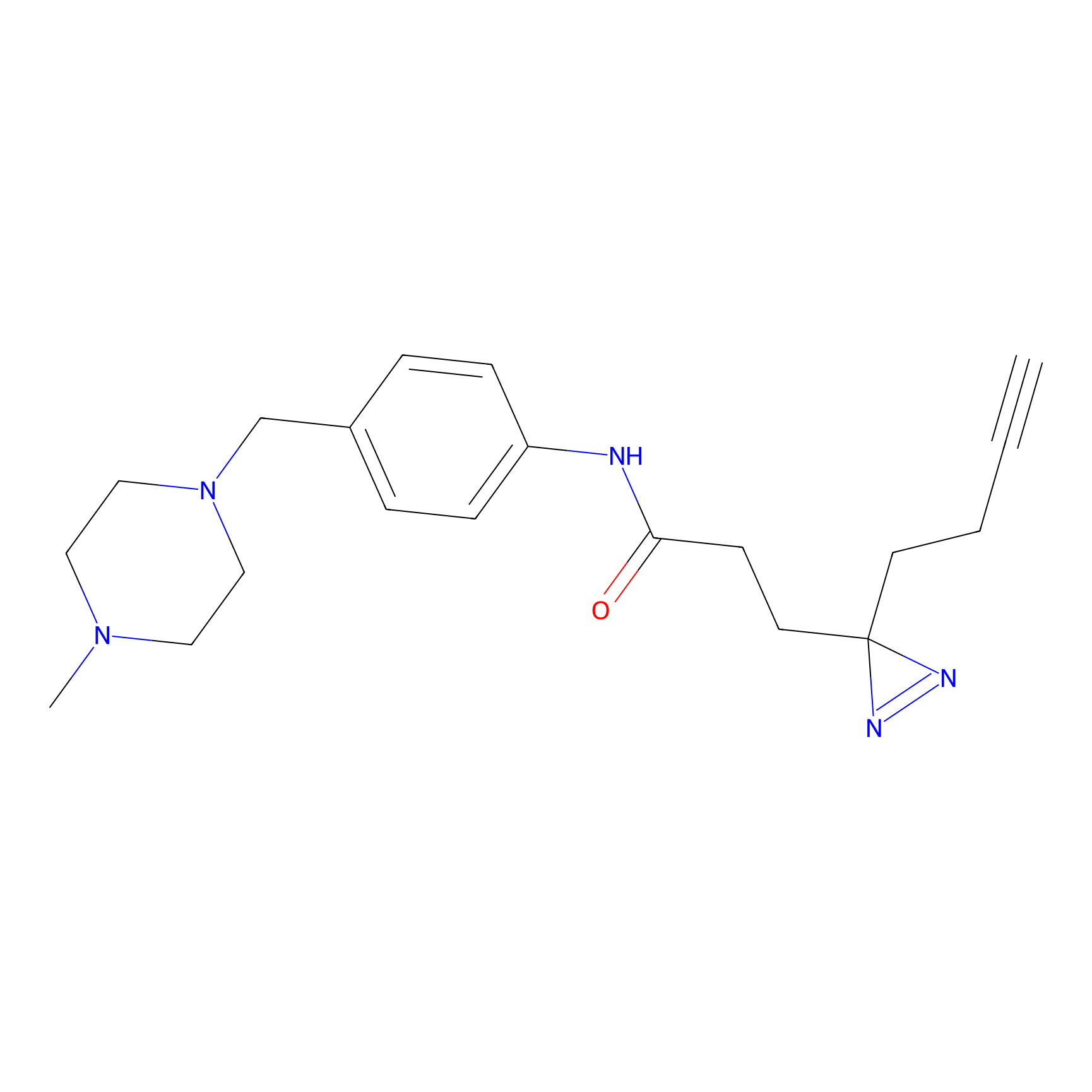
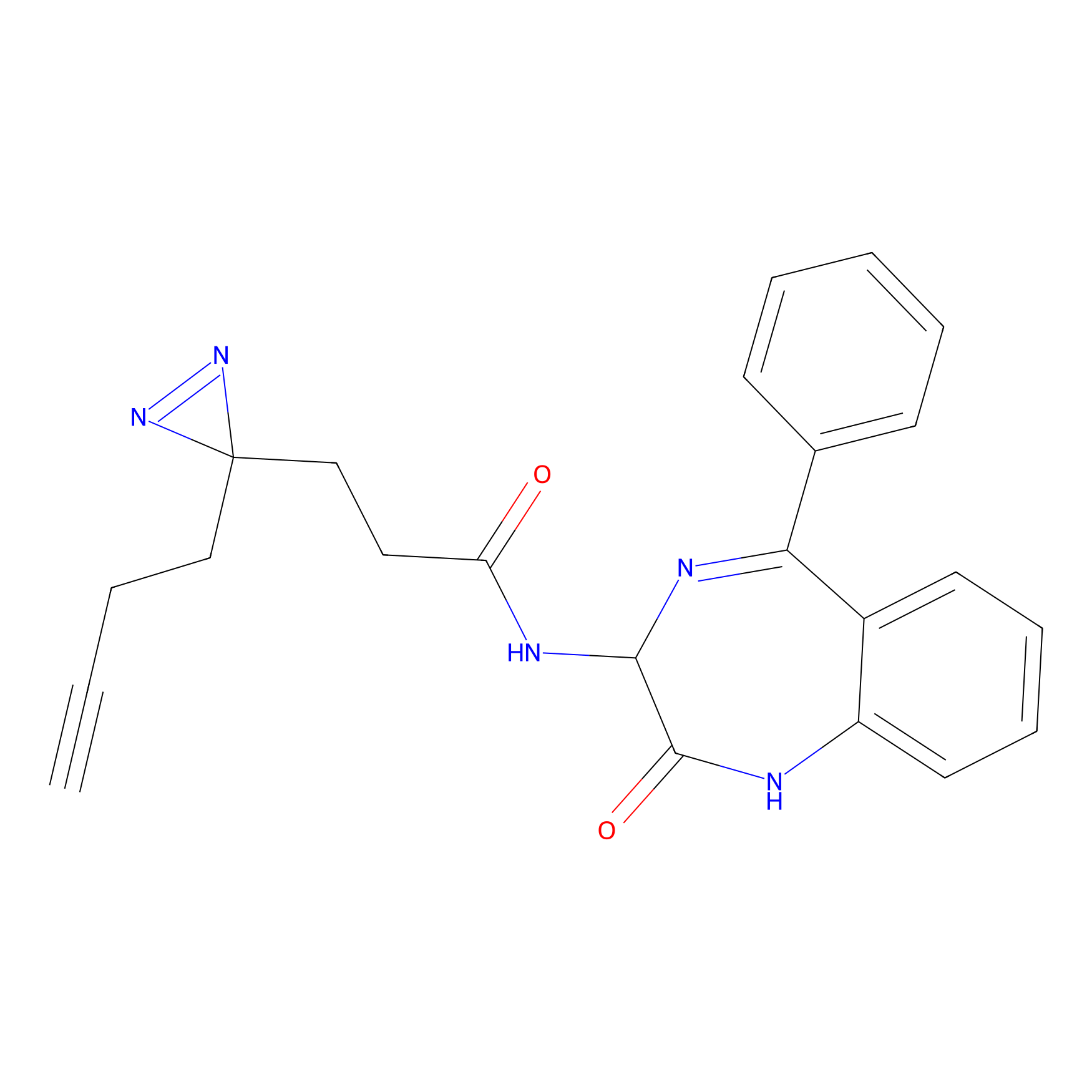
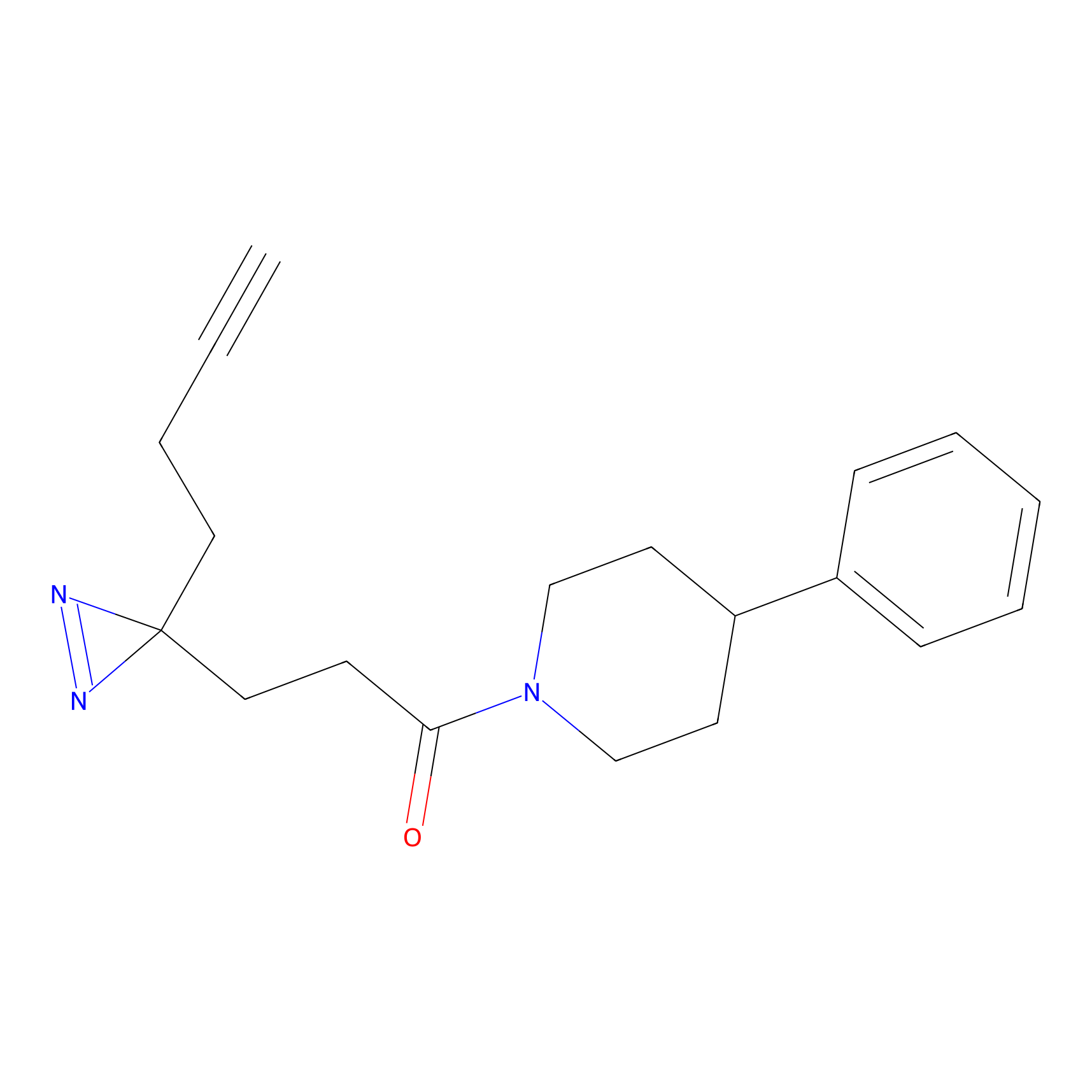
|
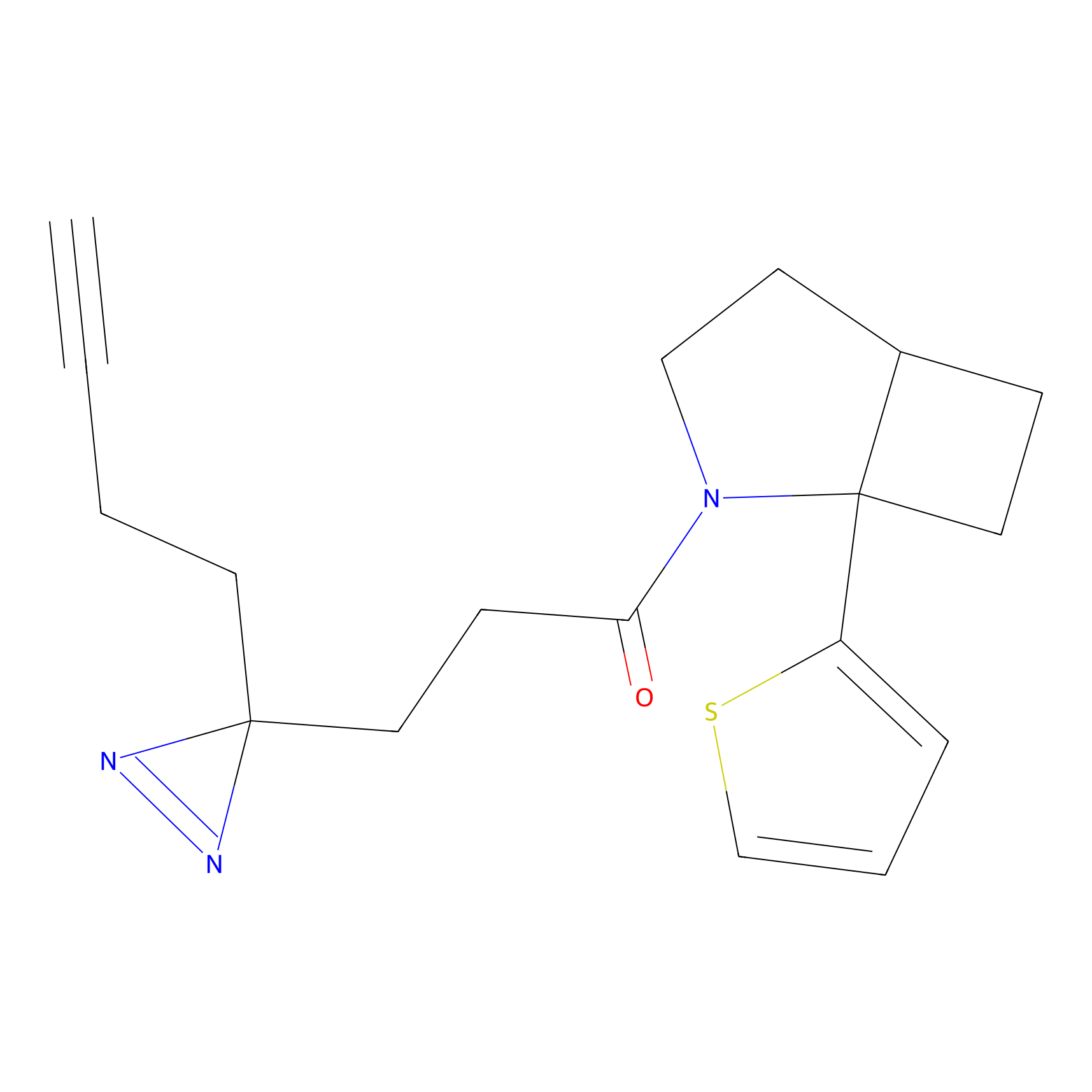
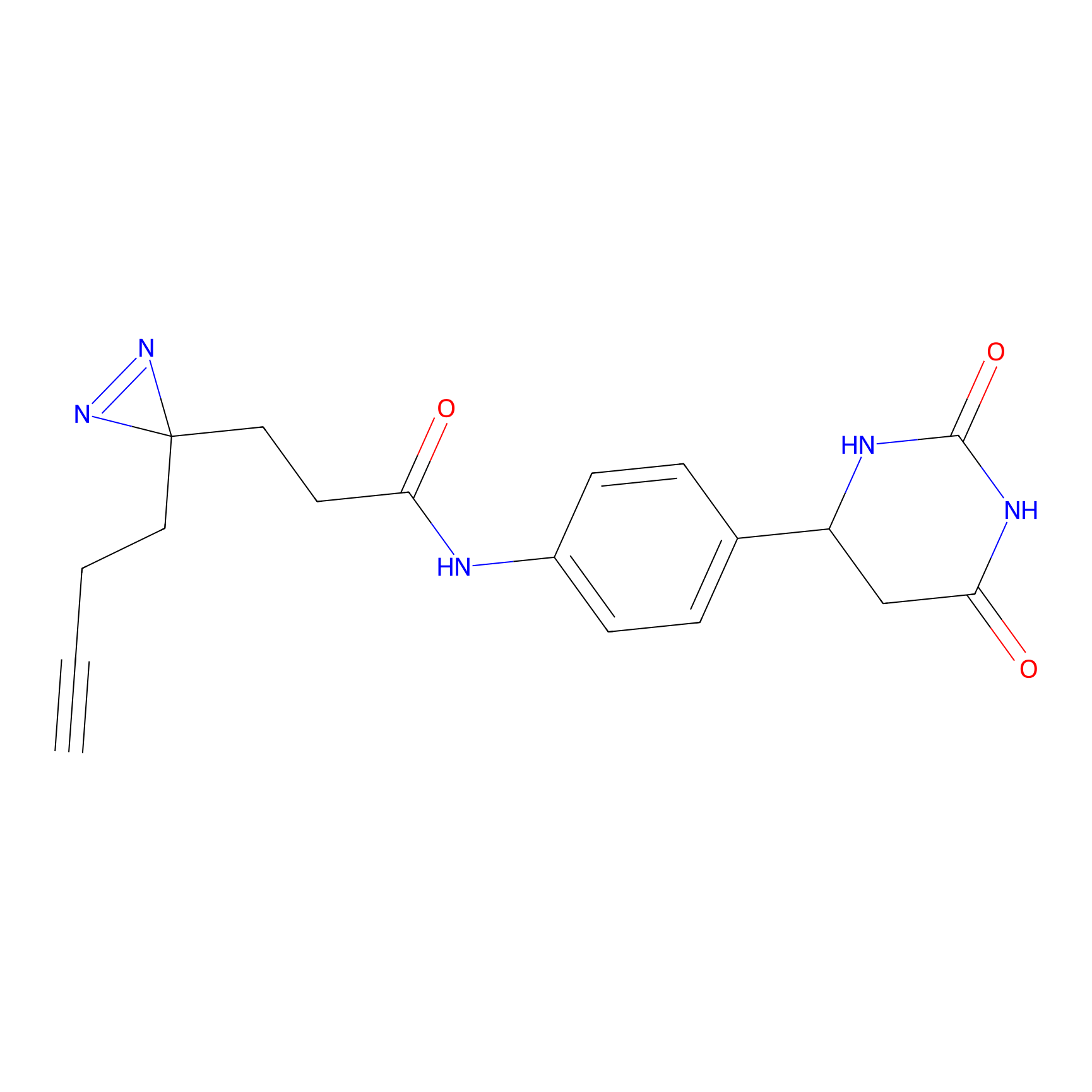
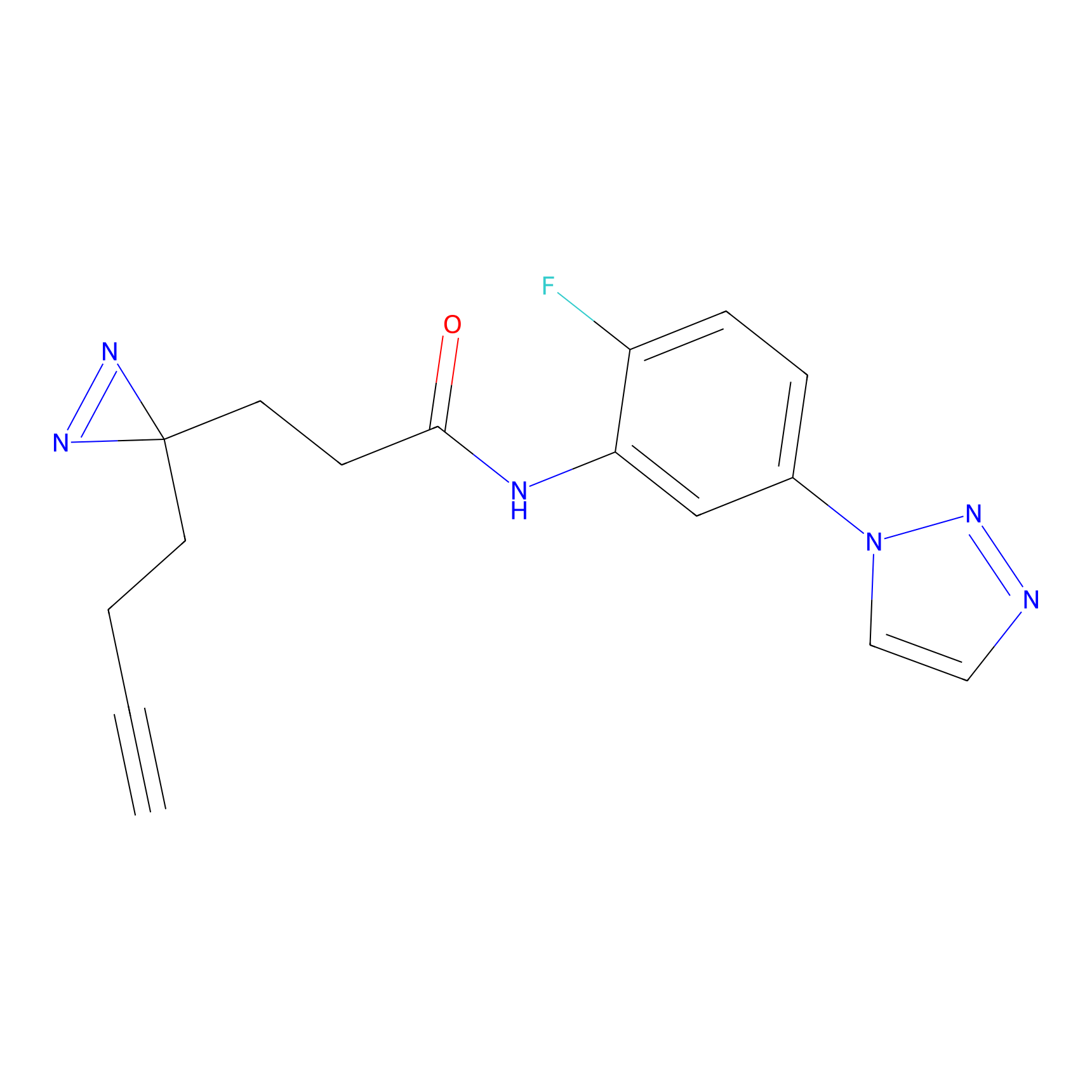
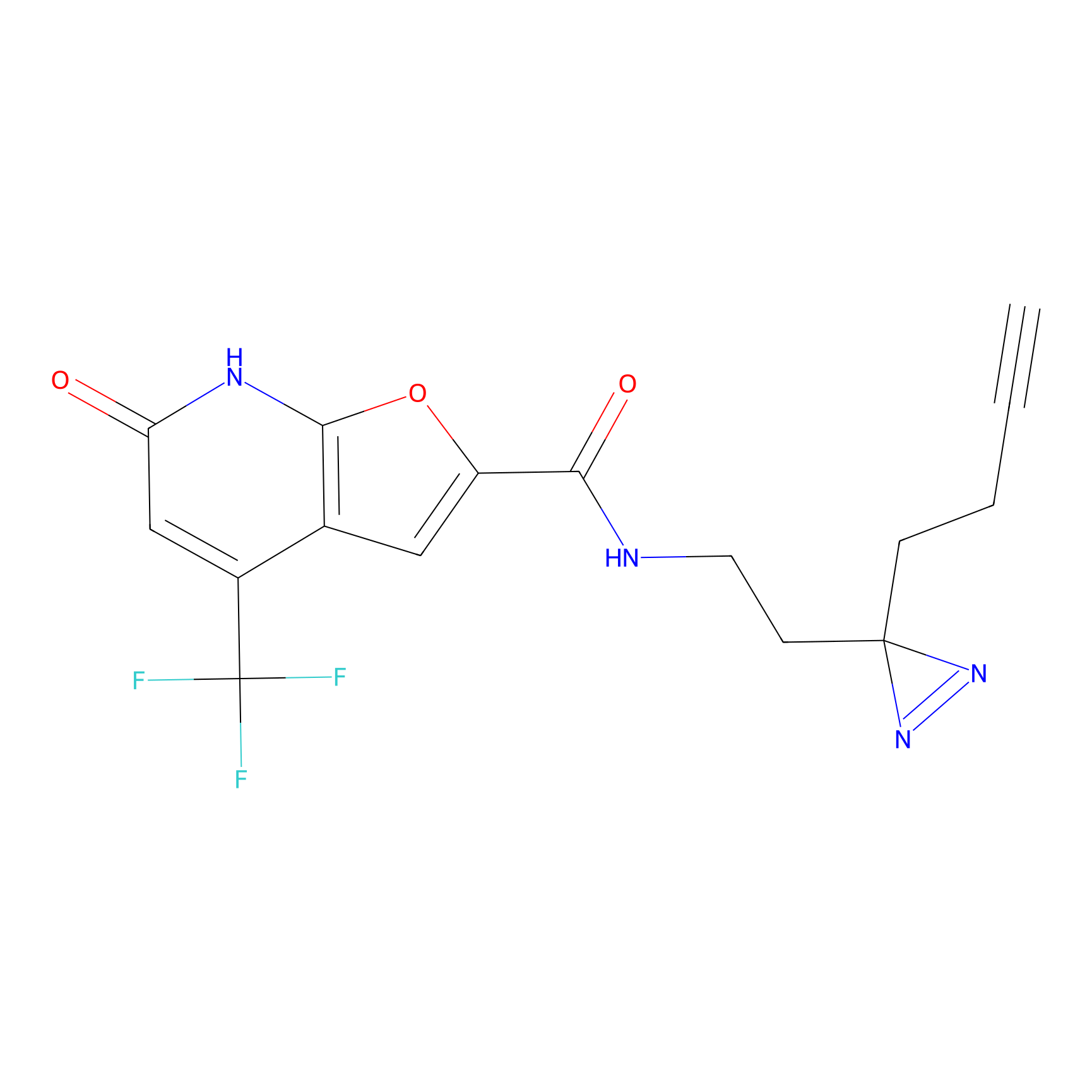
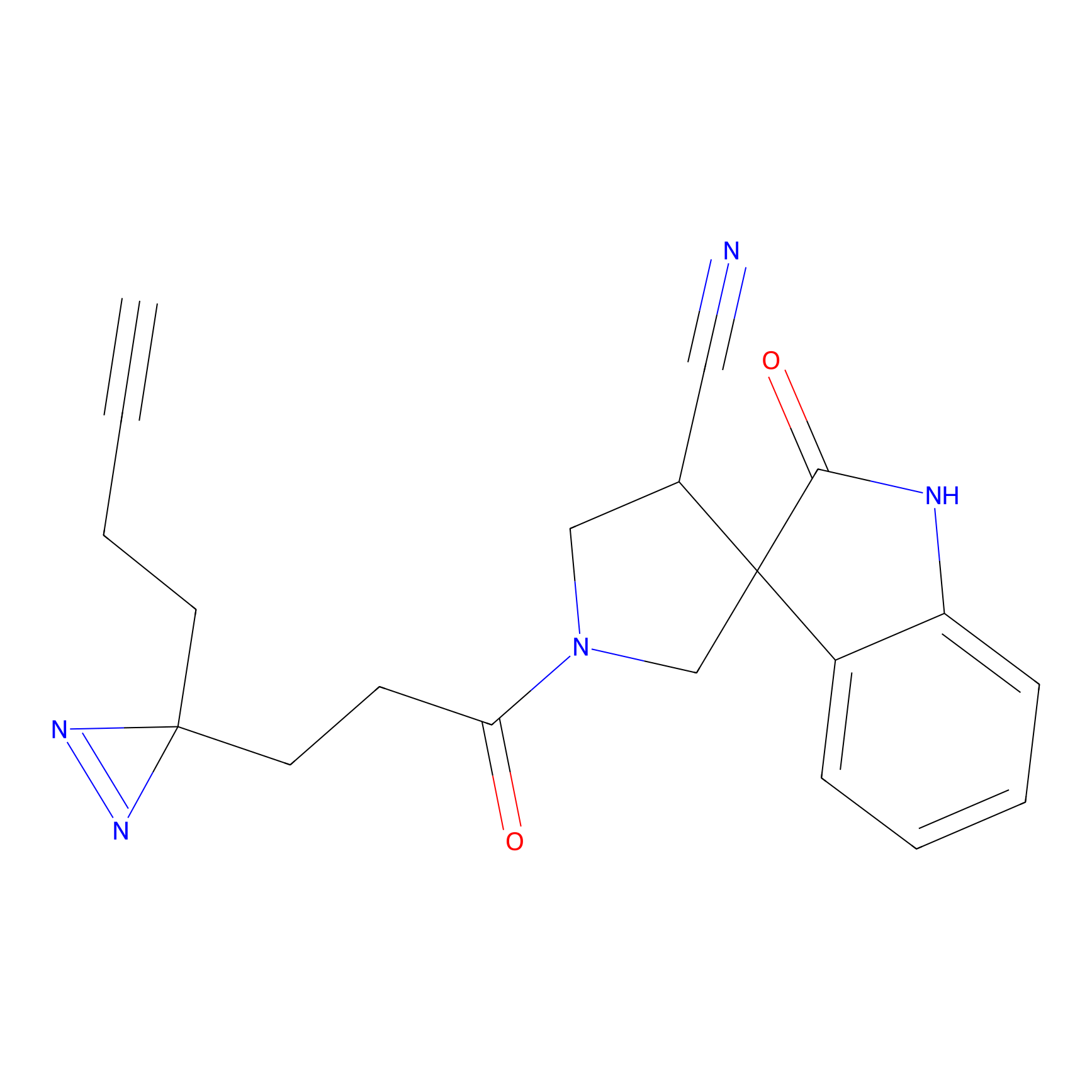
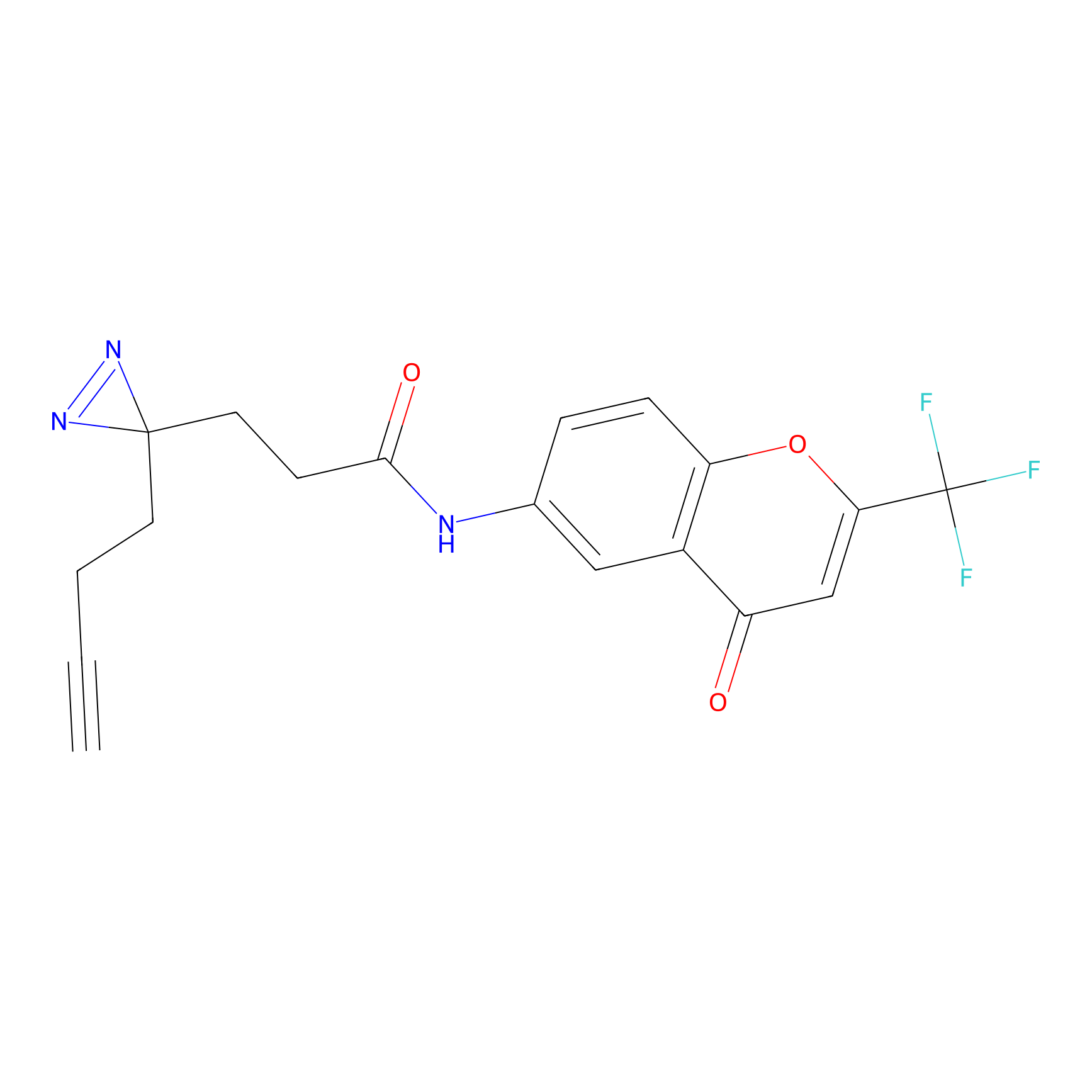
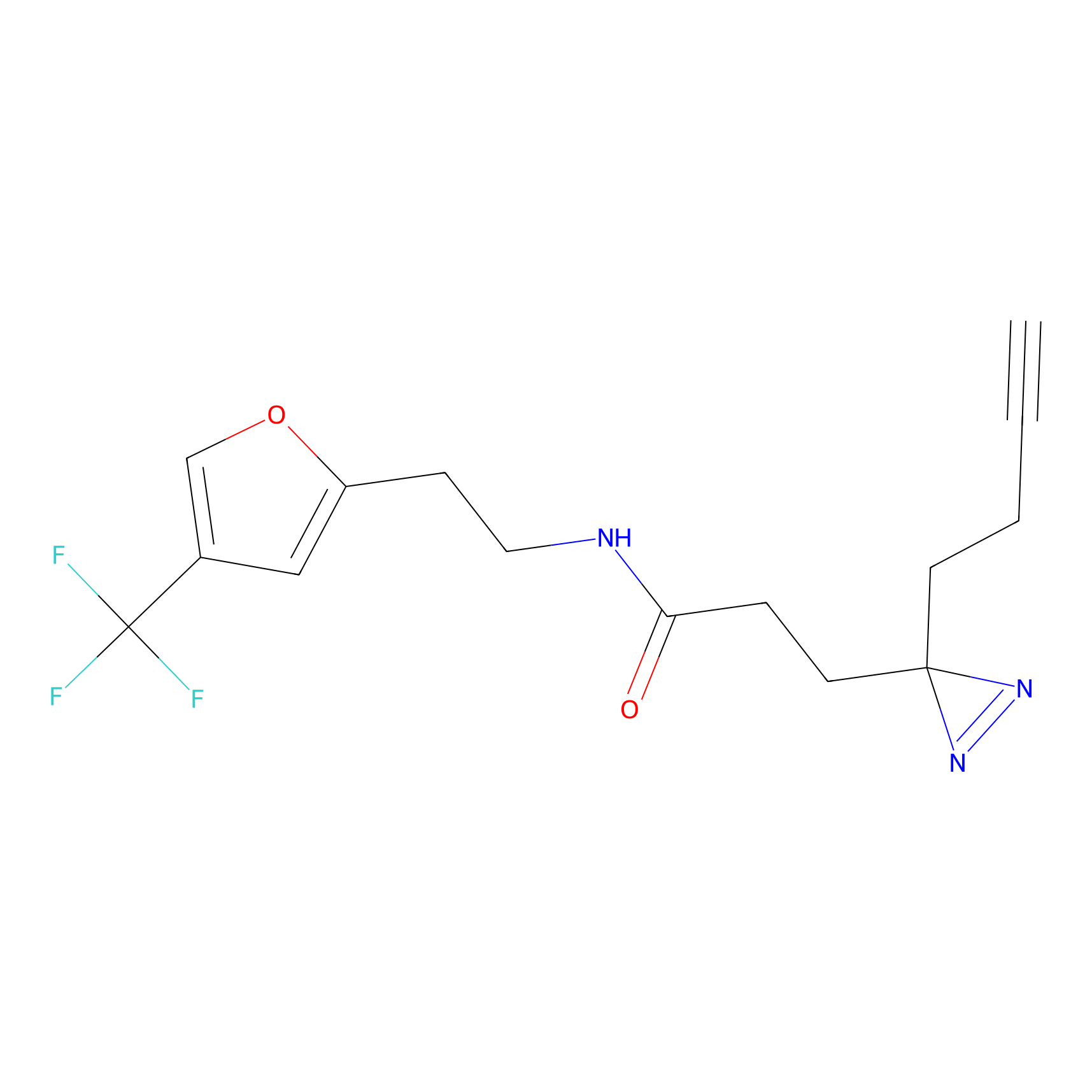
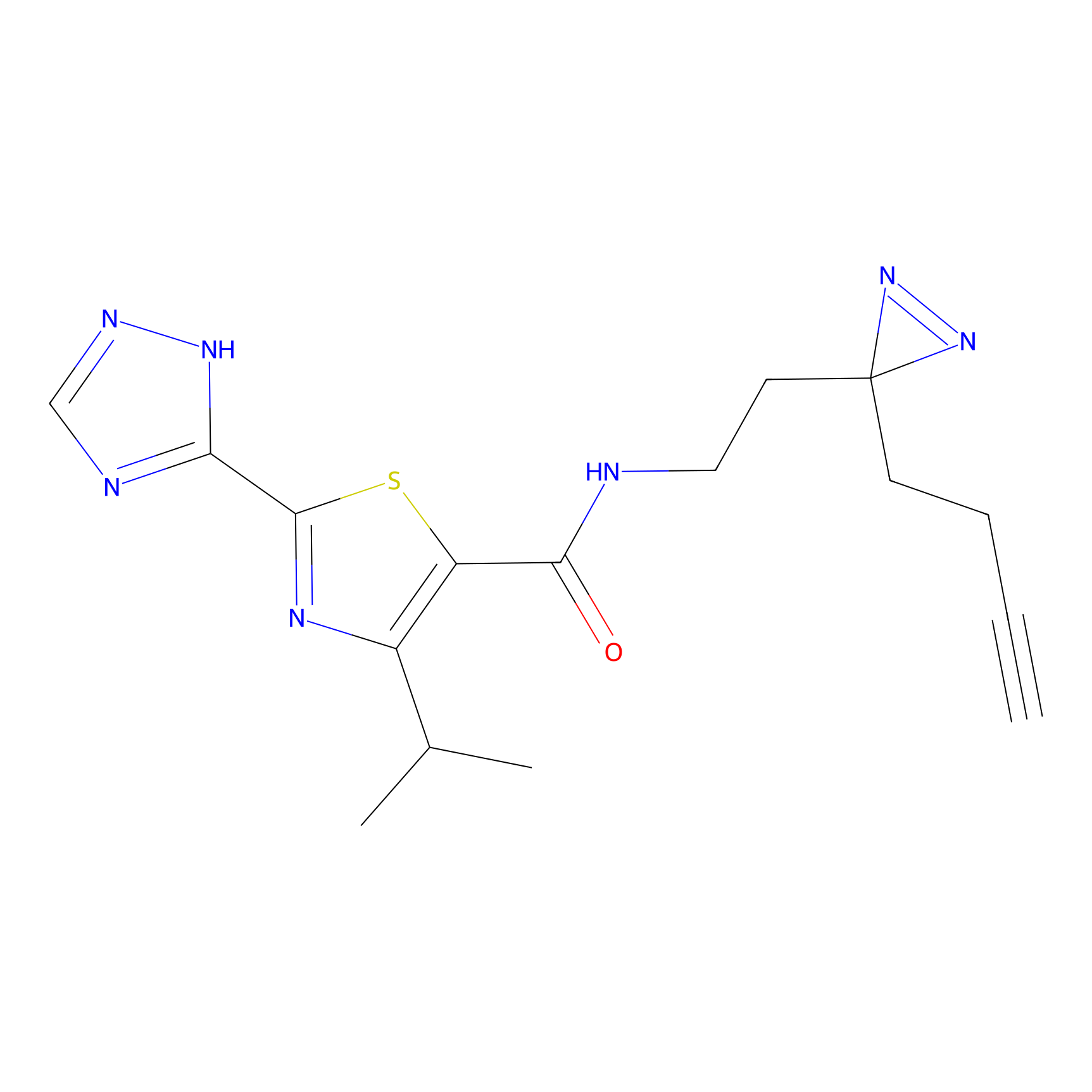
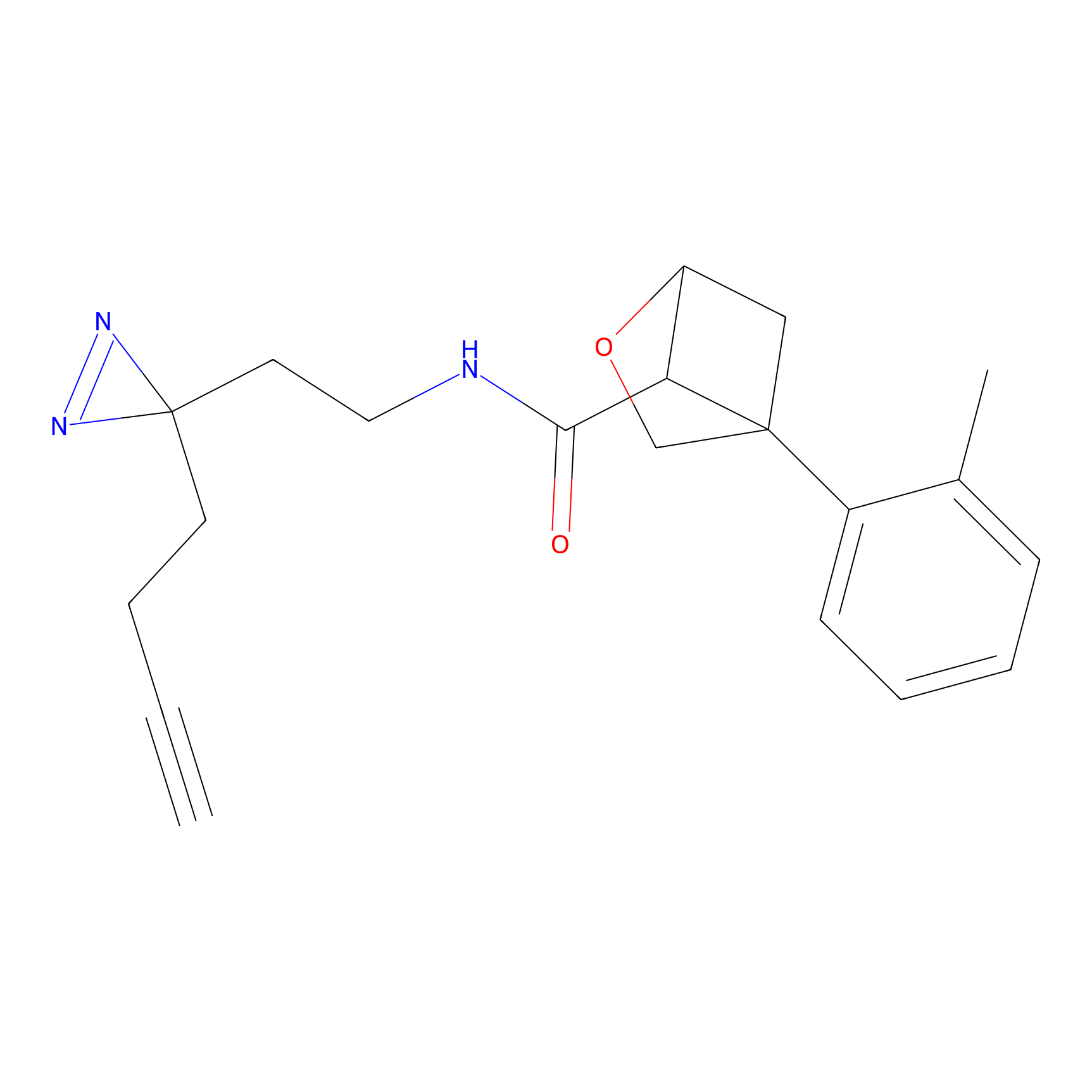
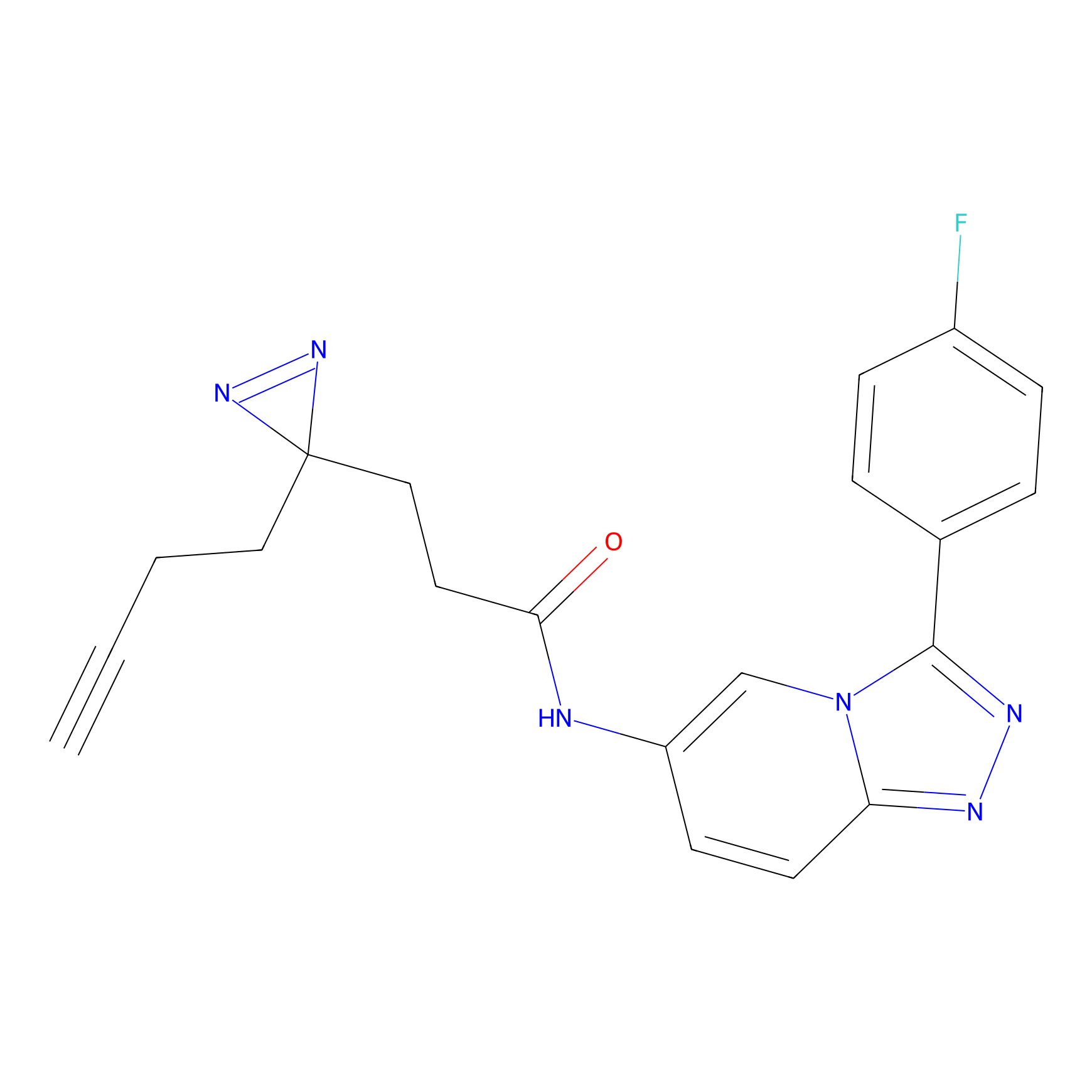
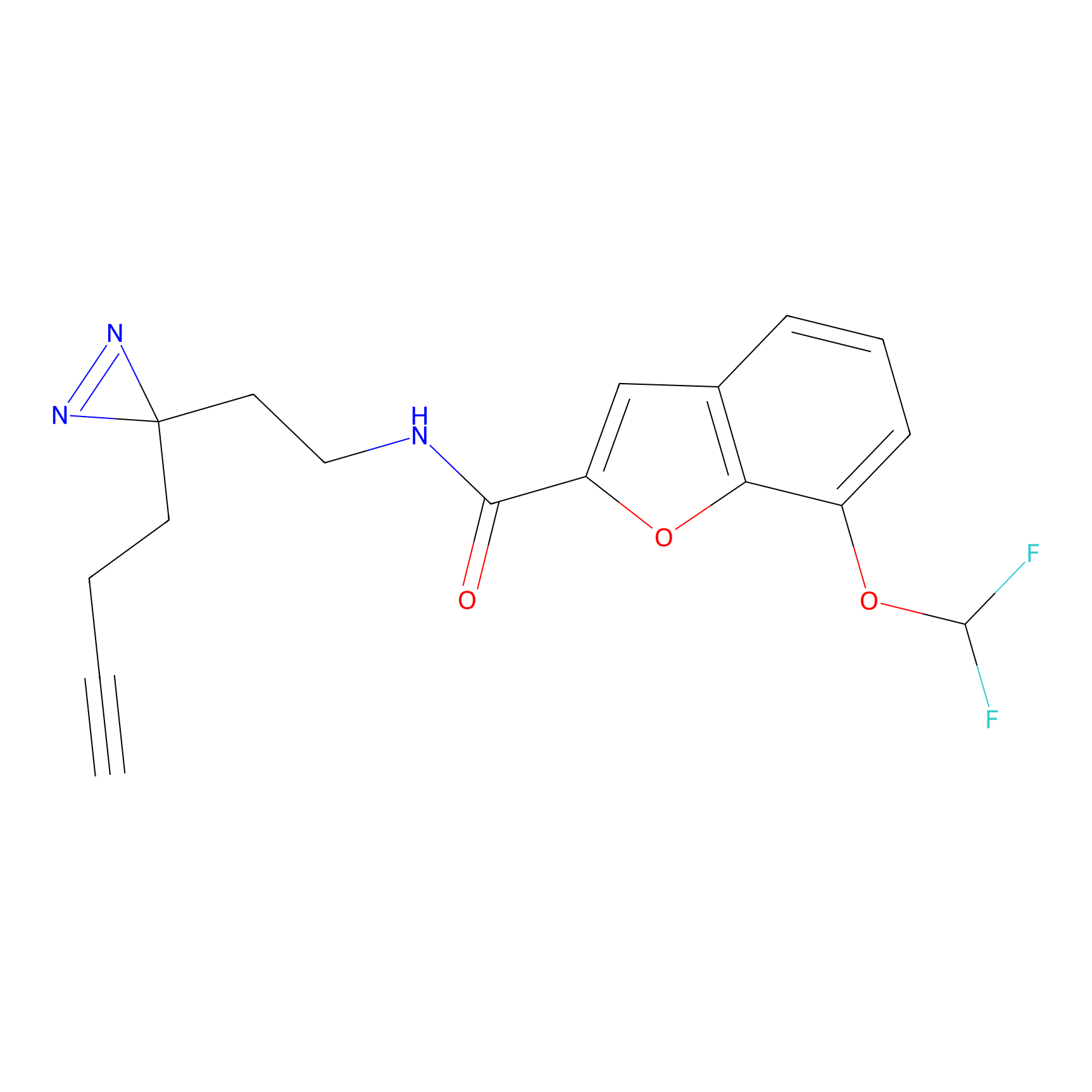
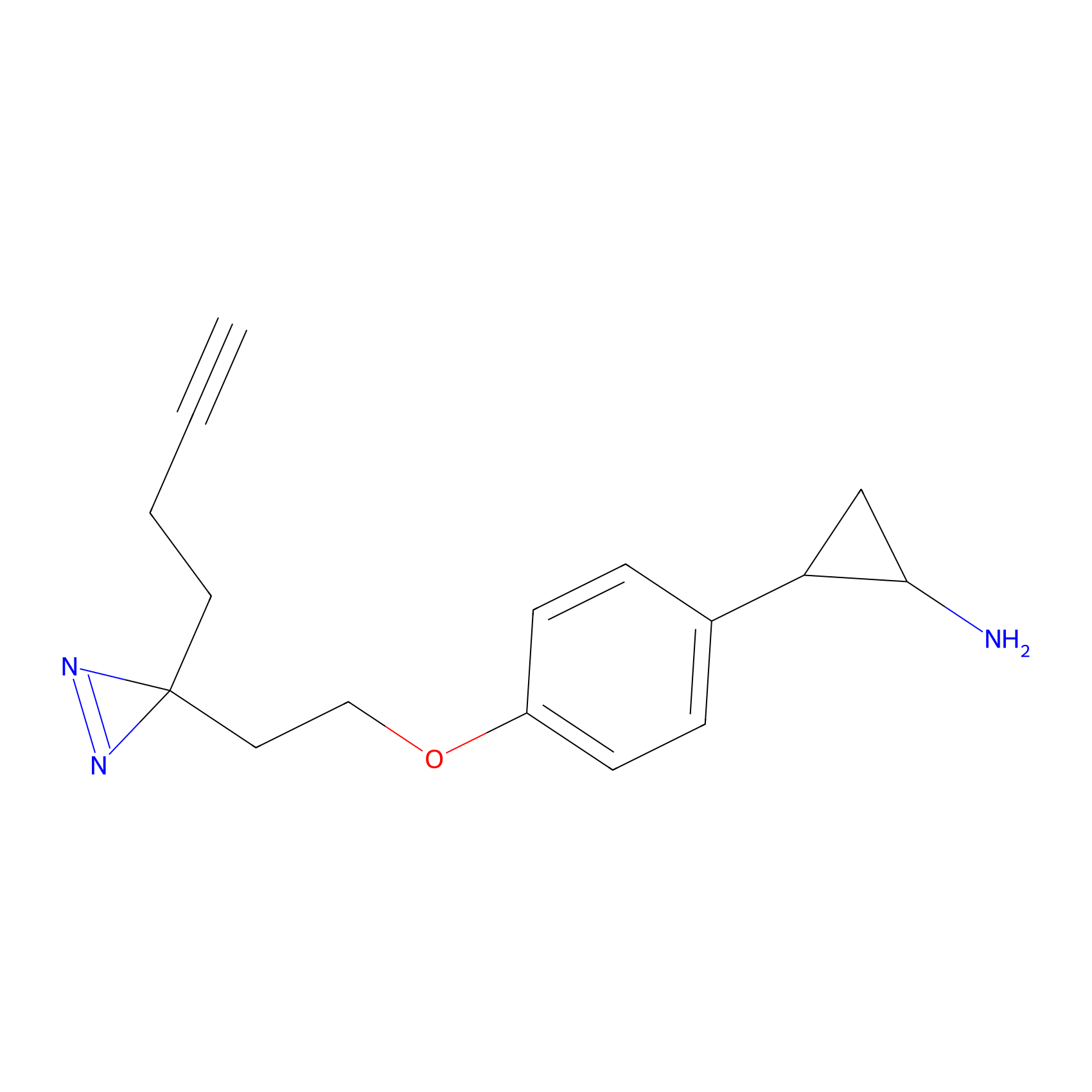
TH214 Probe Info |
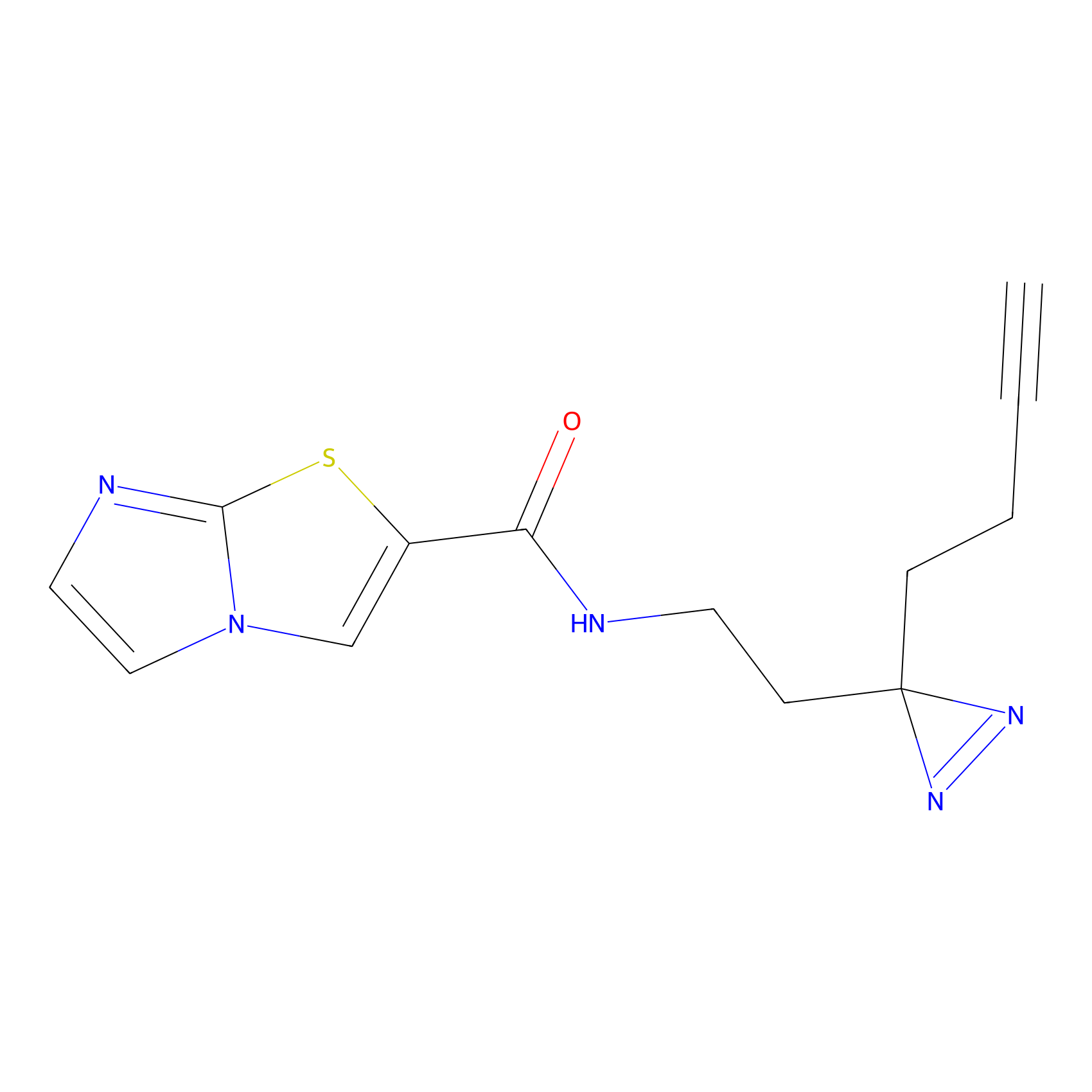
 |
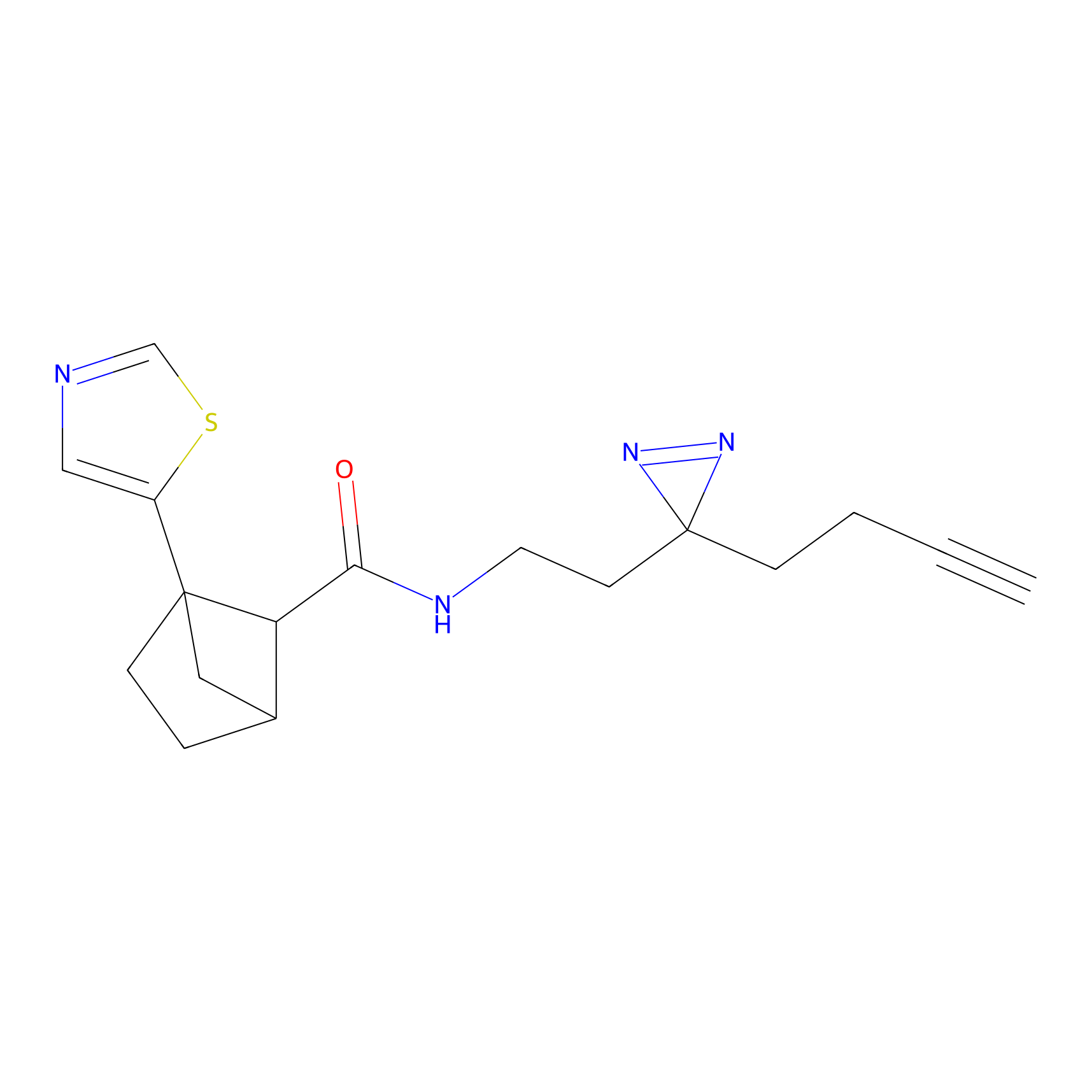
Y169(6.11) | LDD0258 | [3] | |
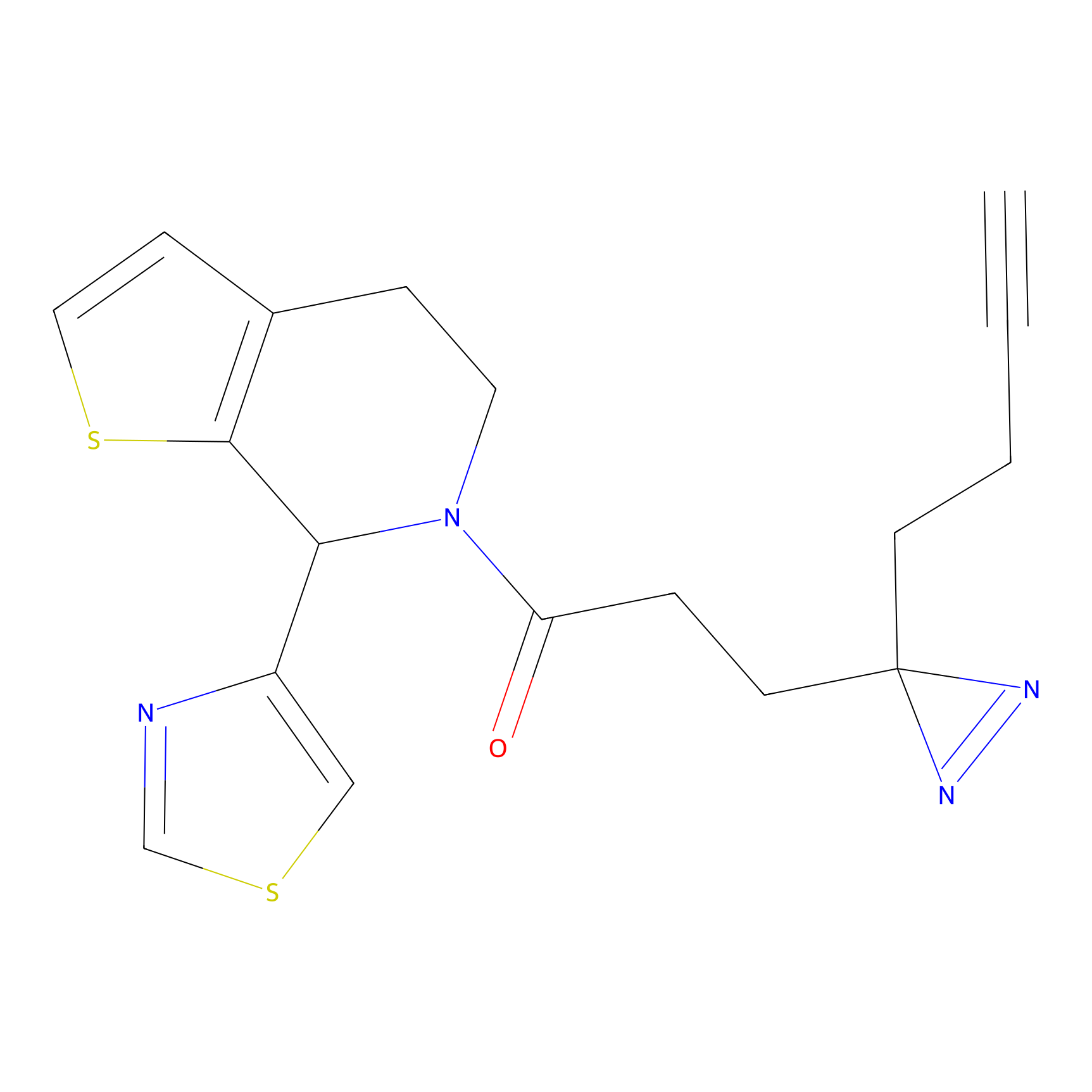
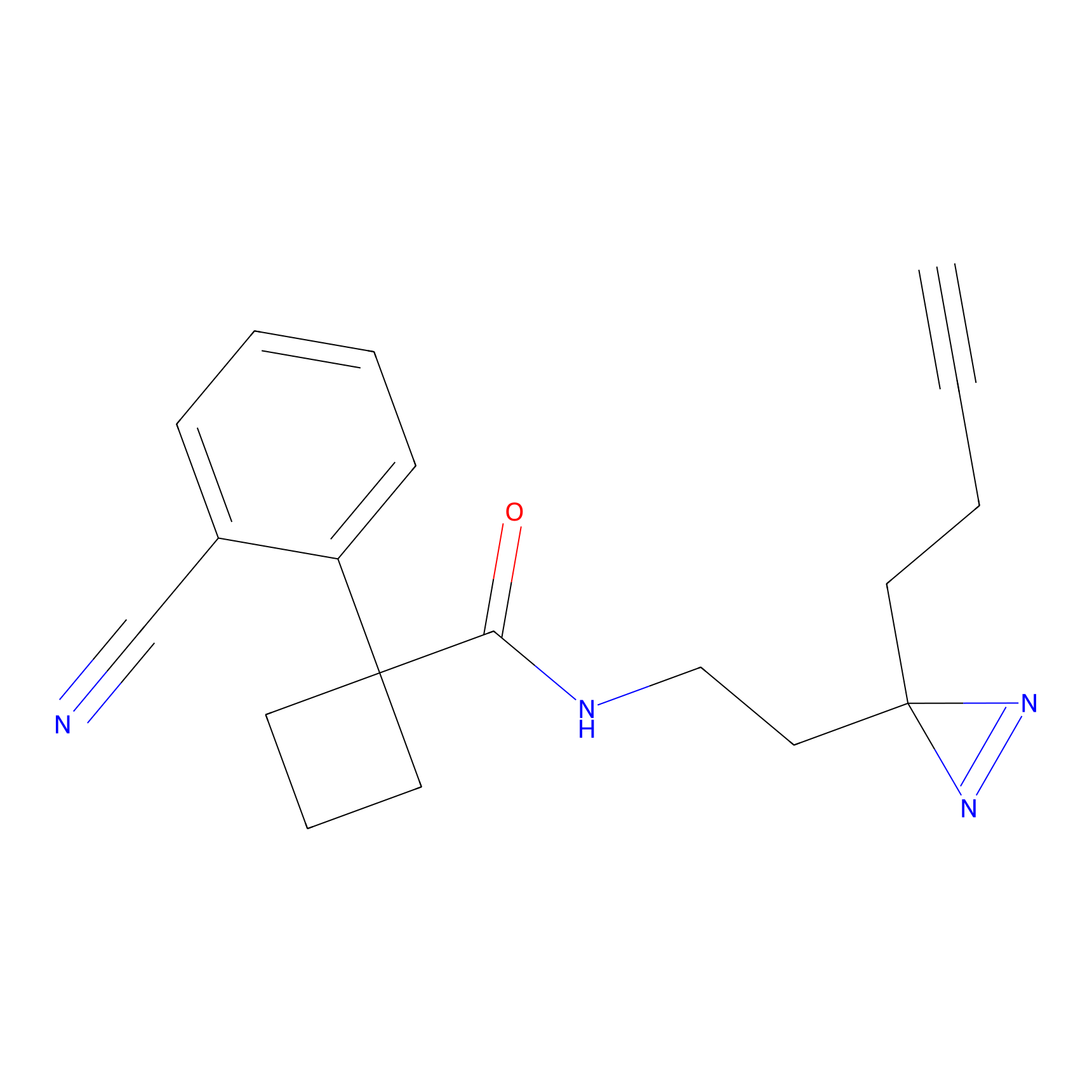
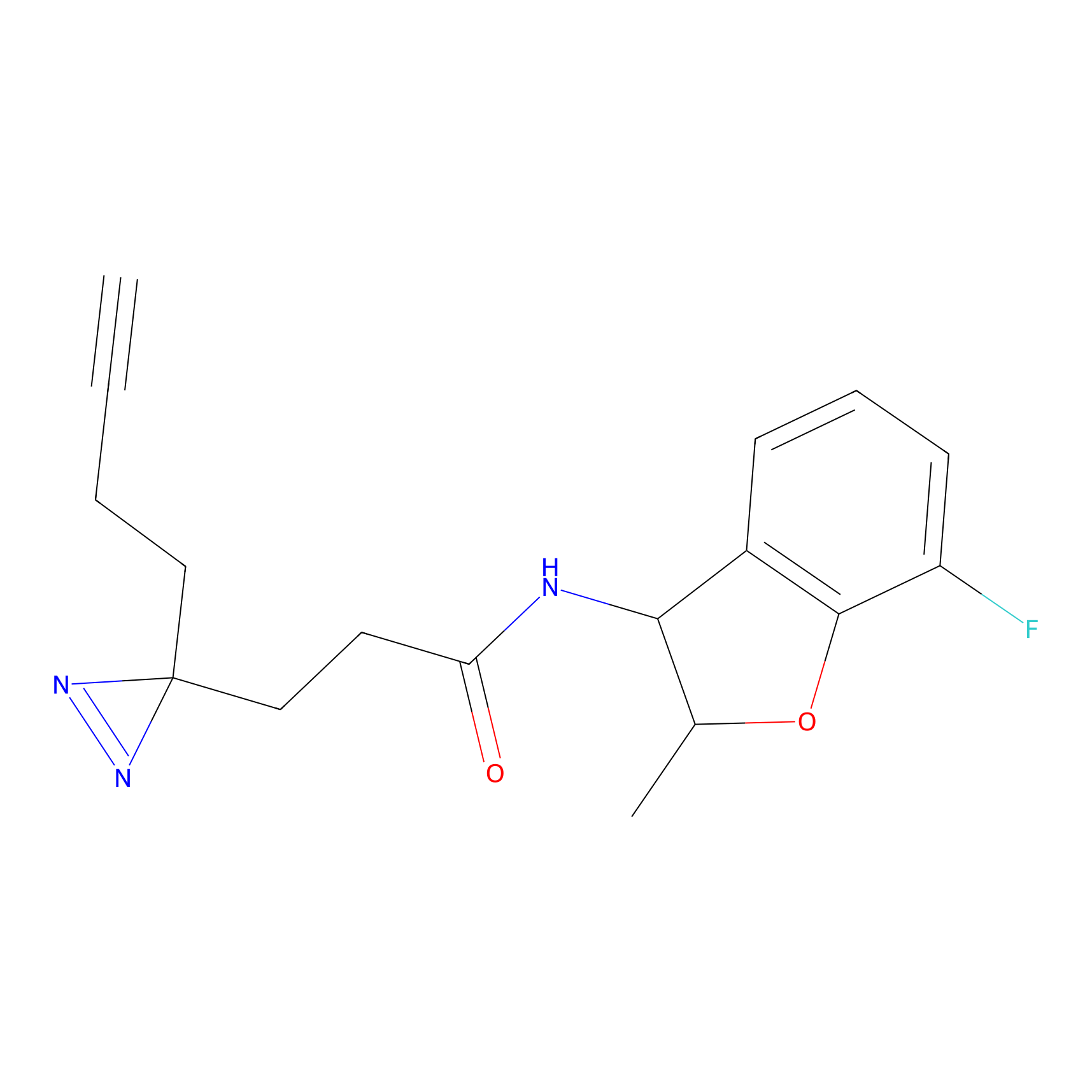
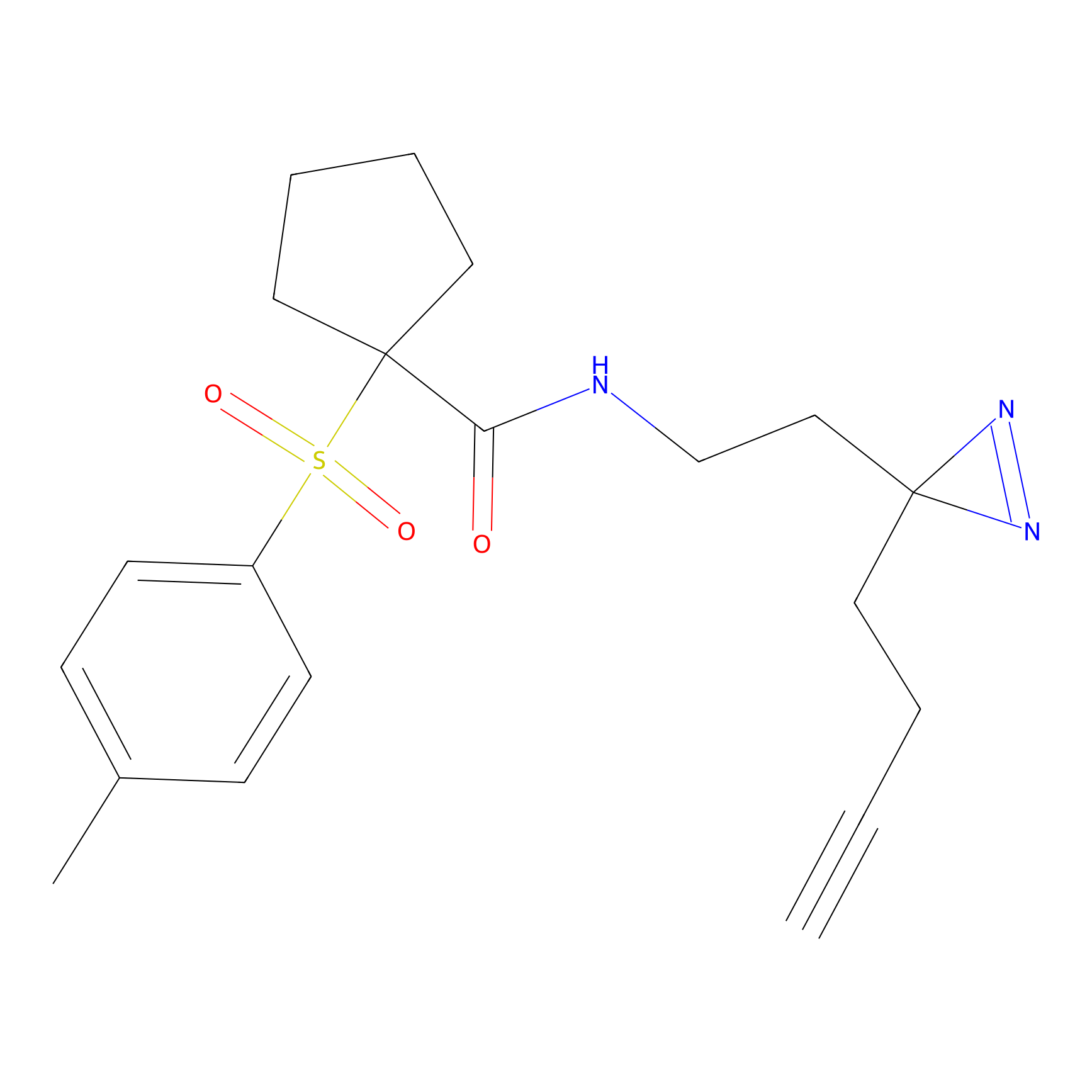
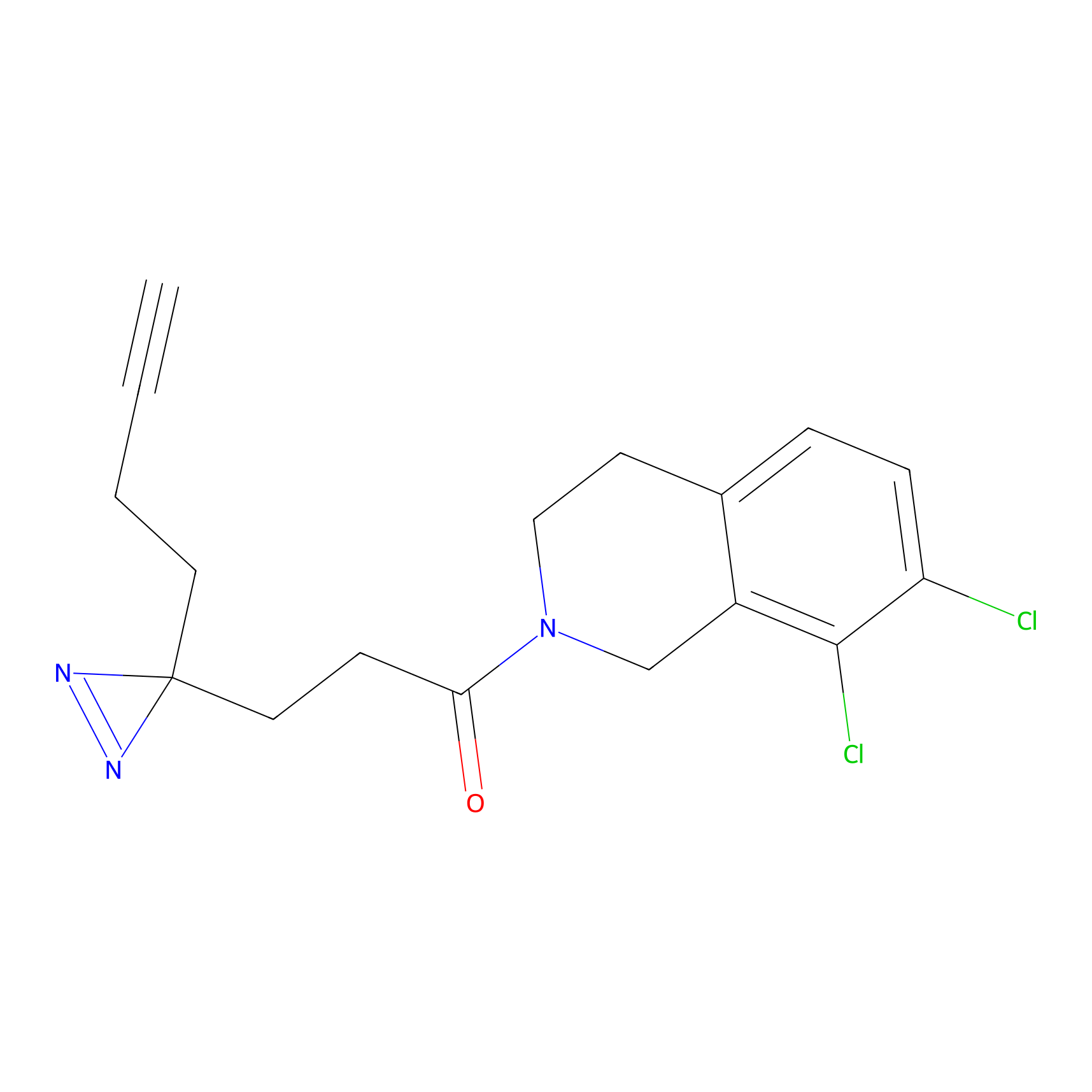
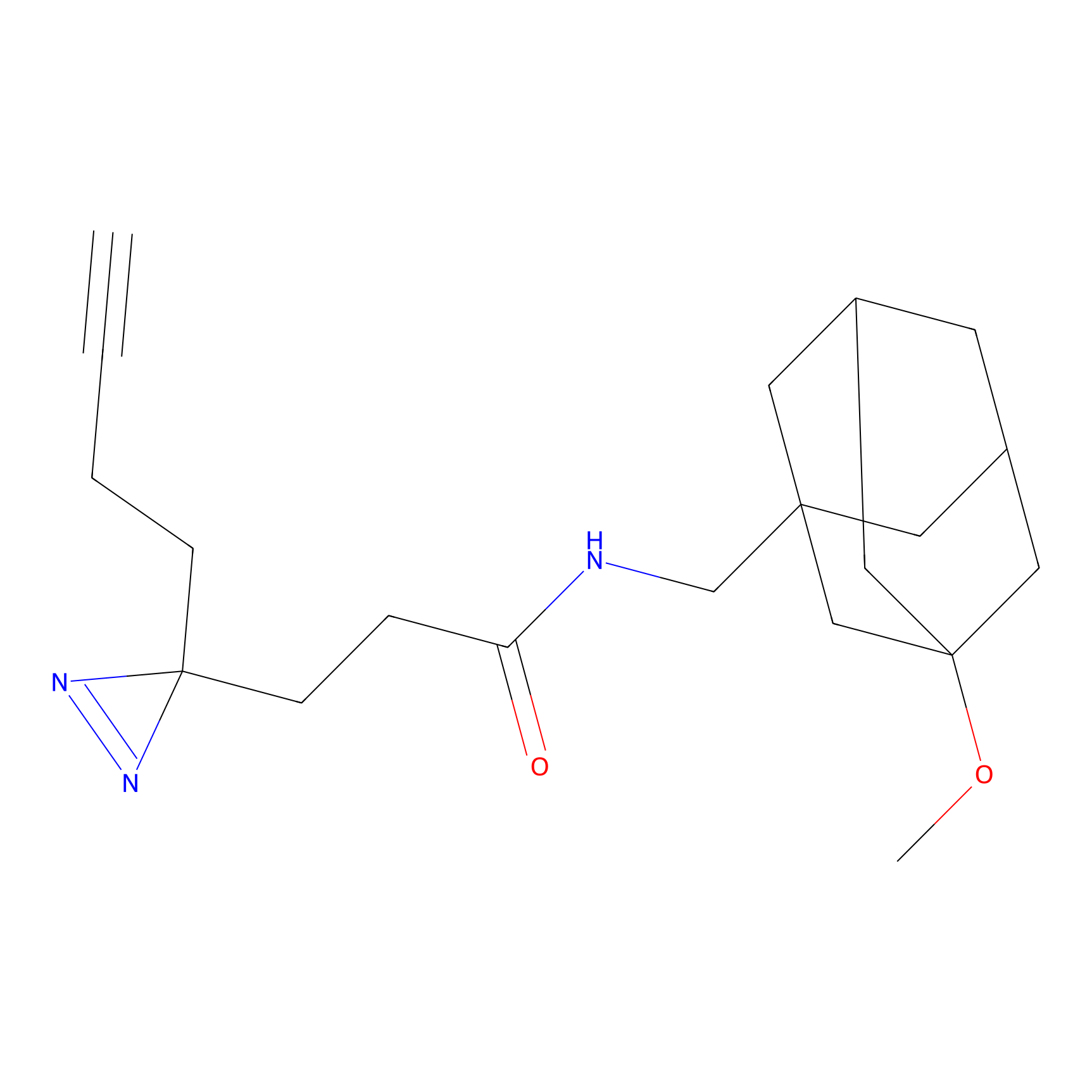
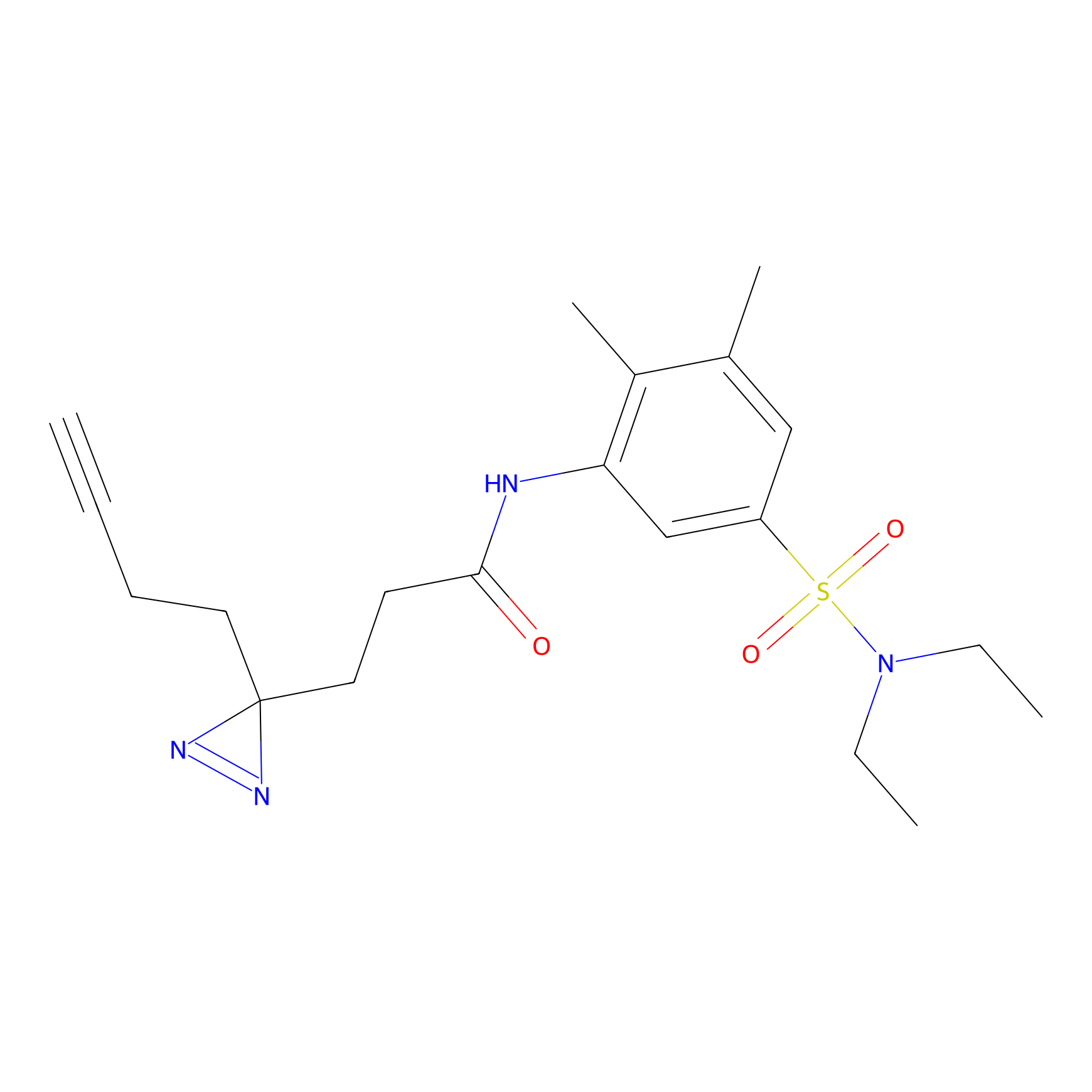
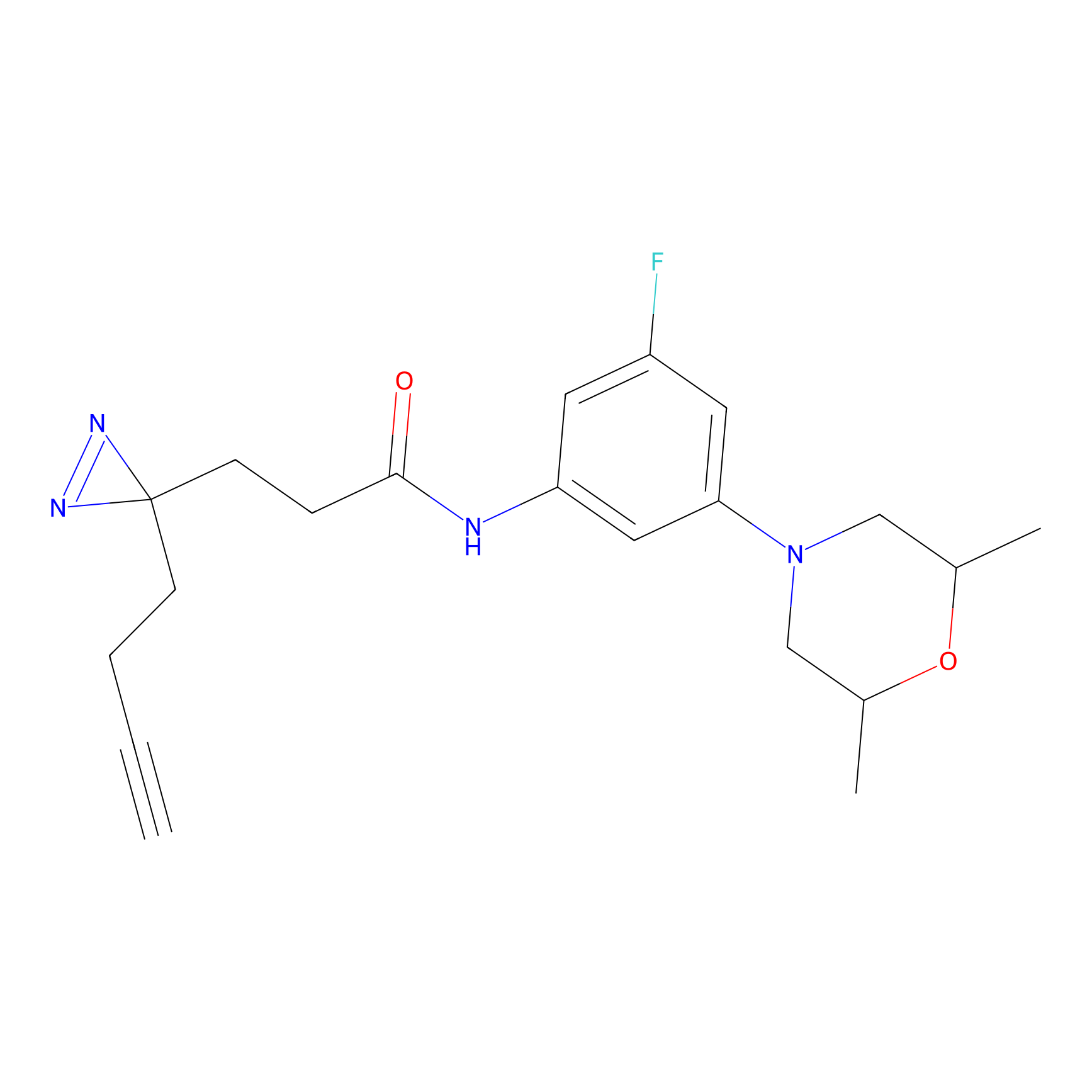
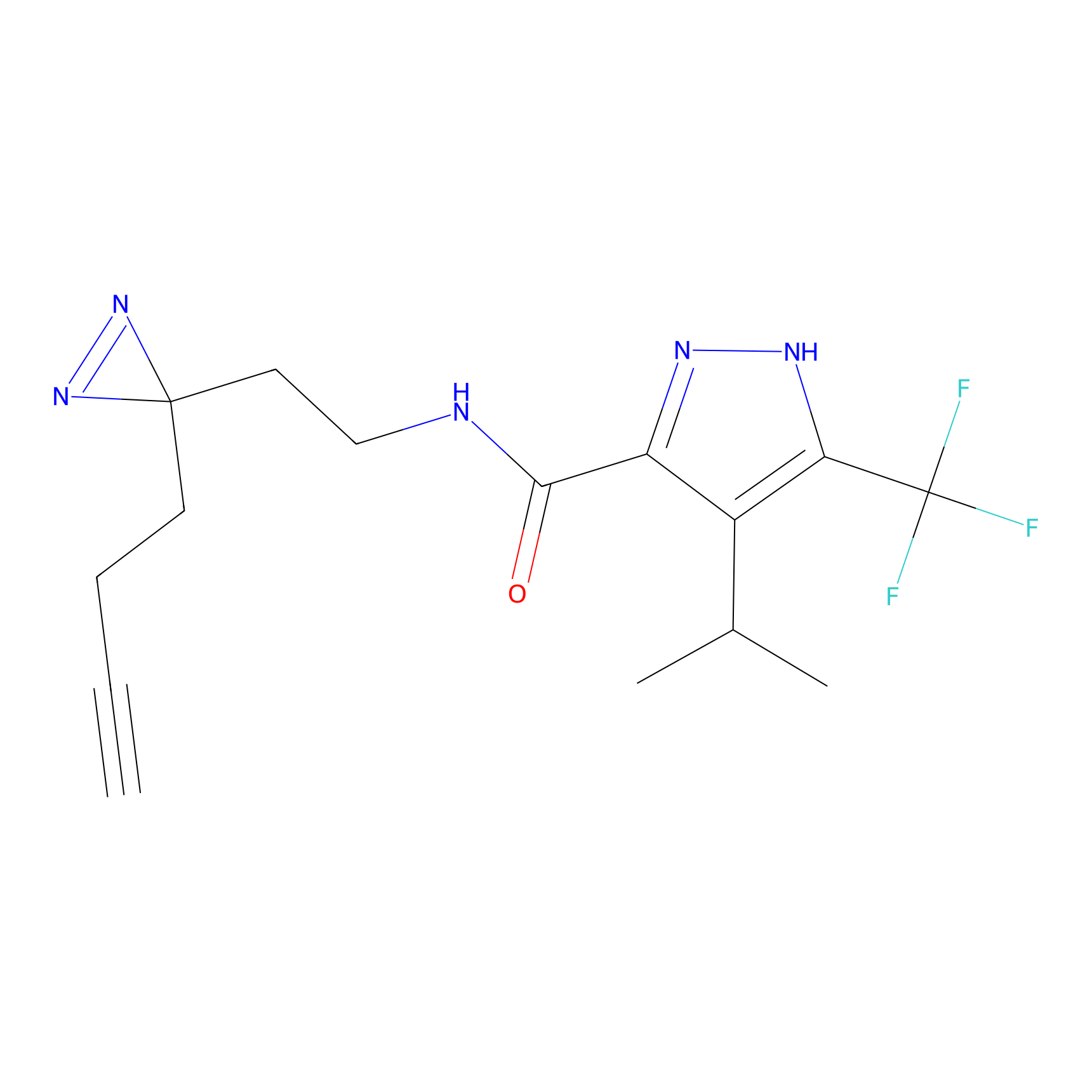
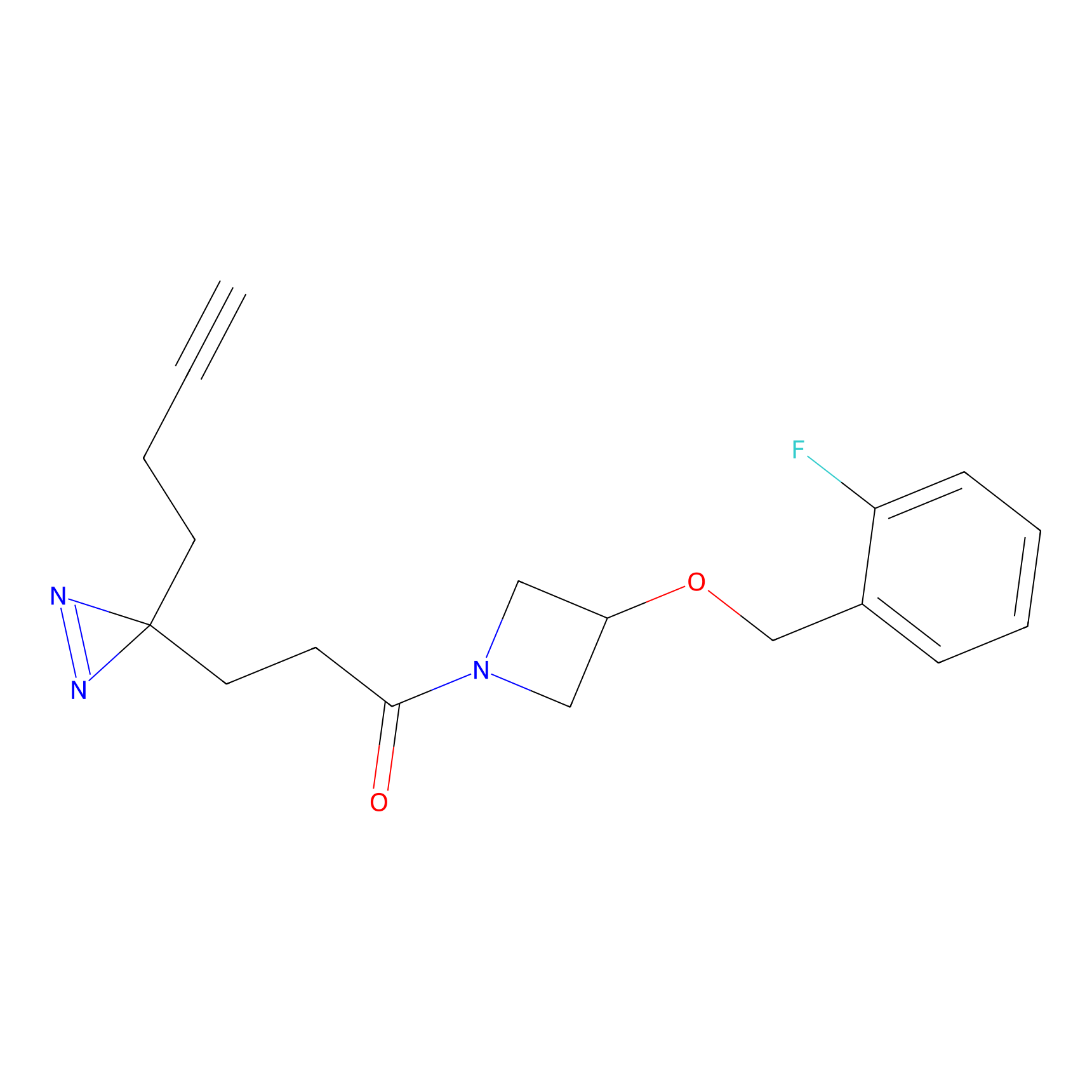
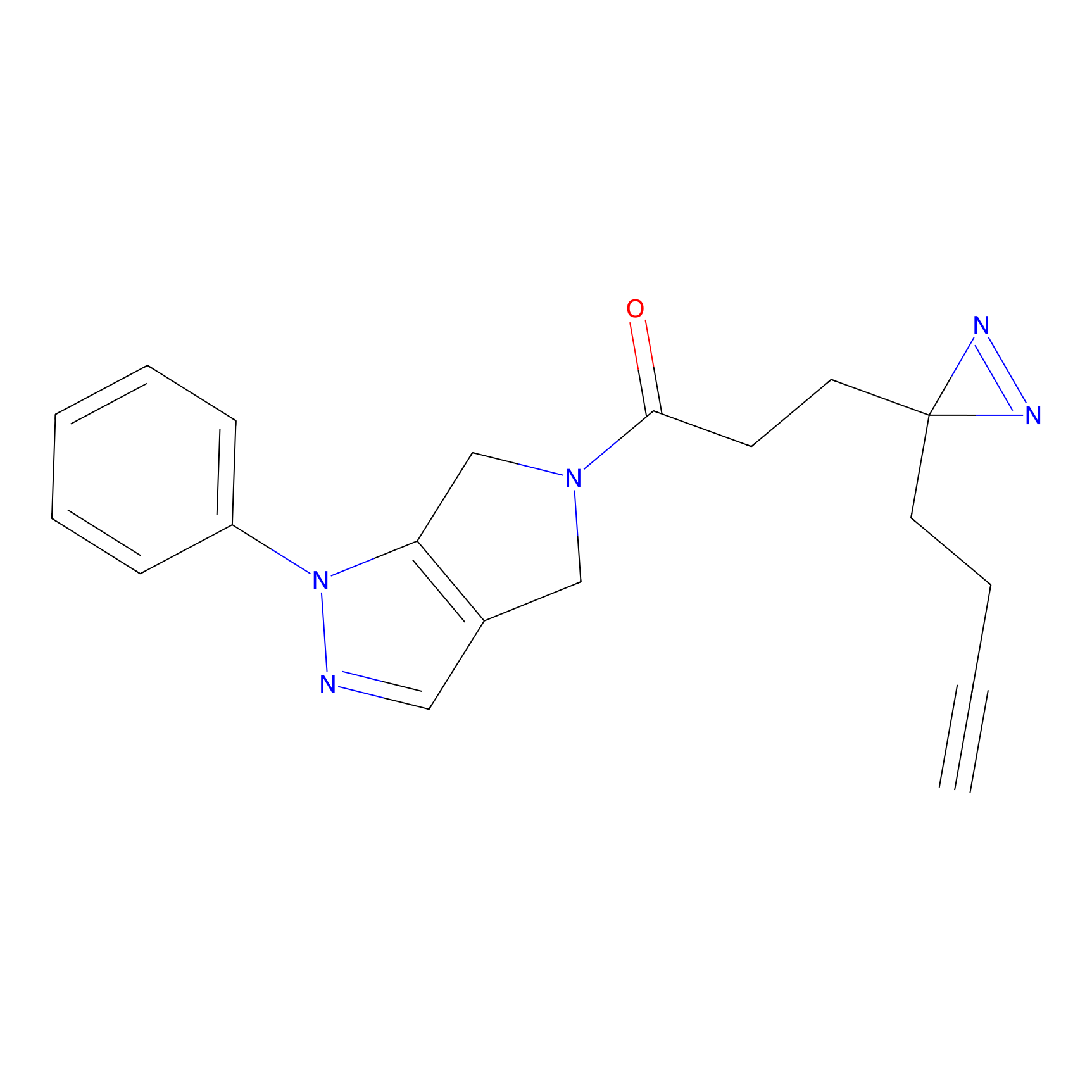
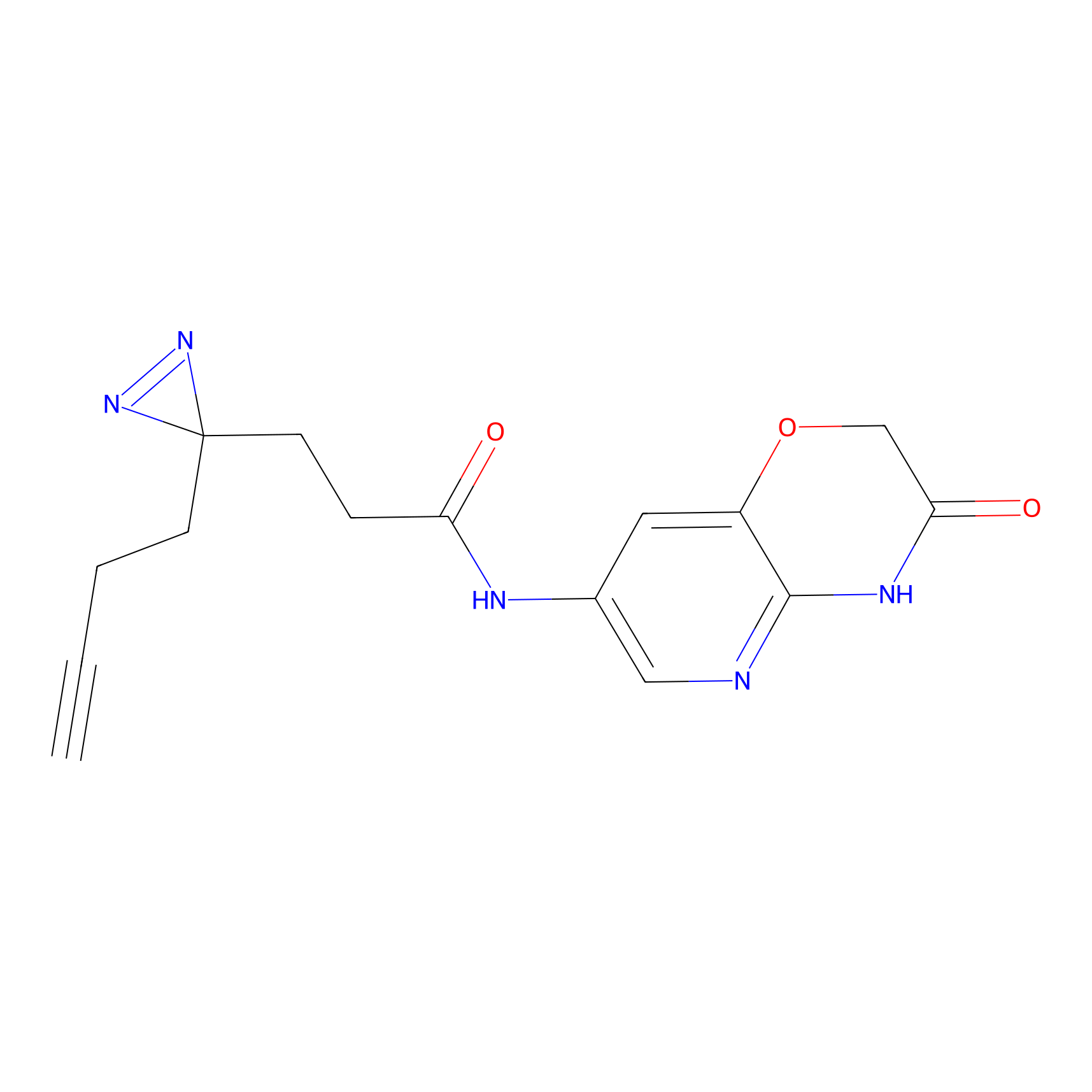
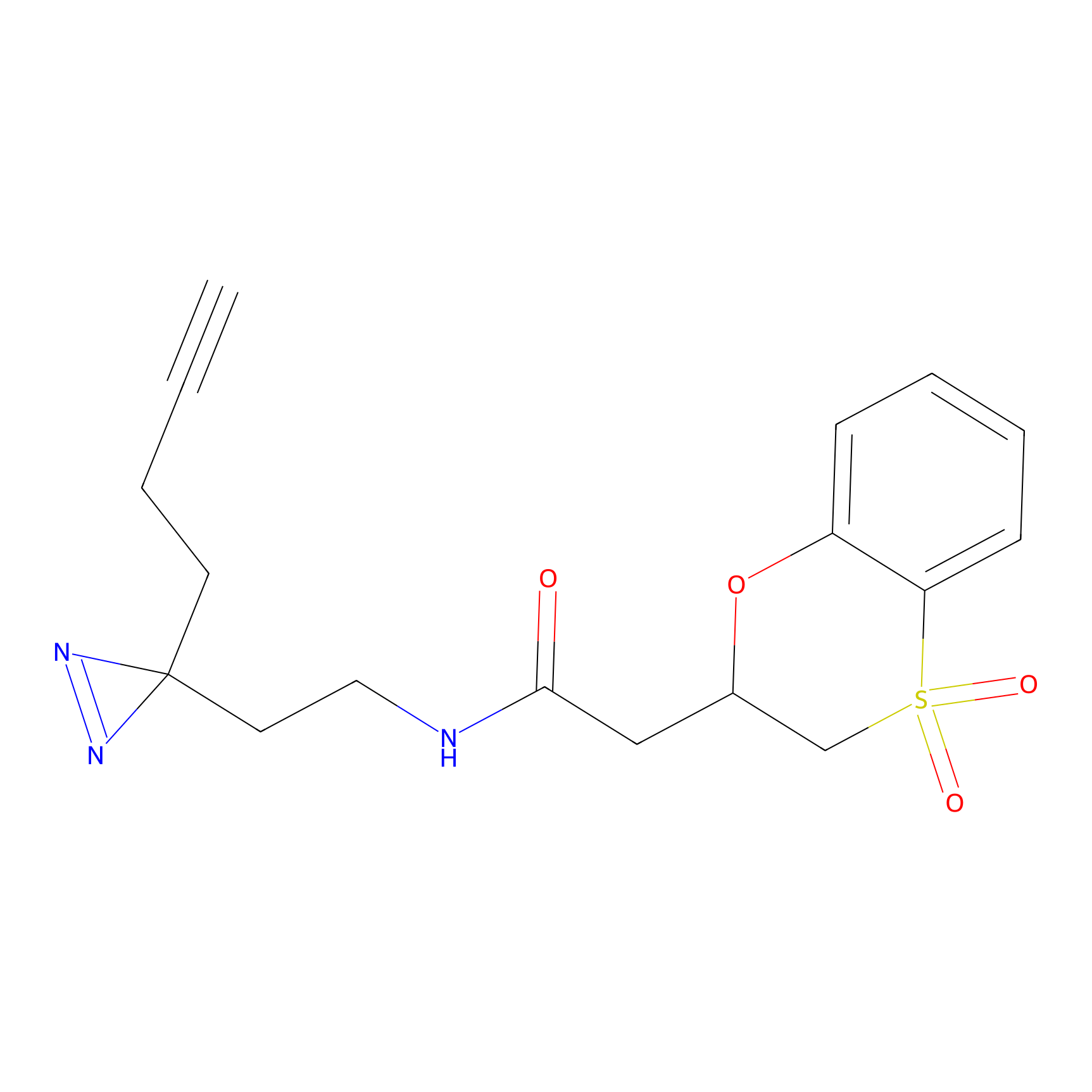
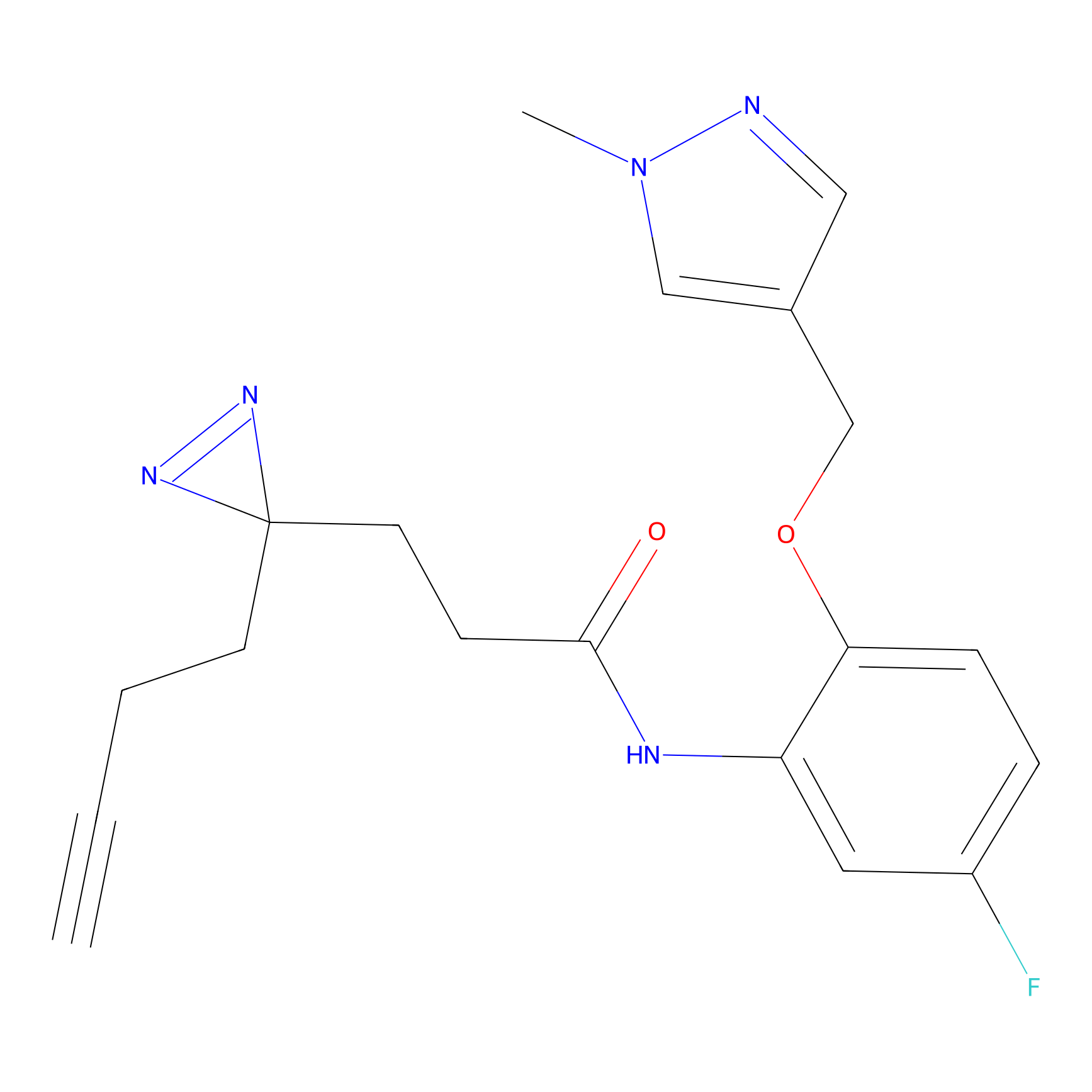
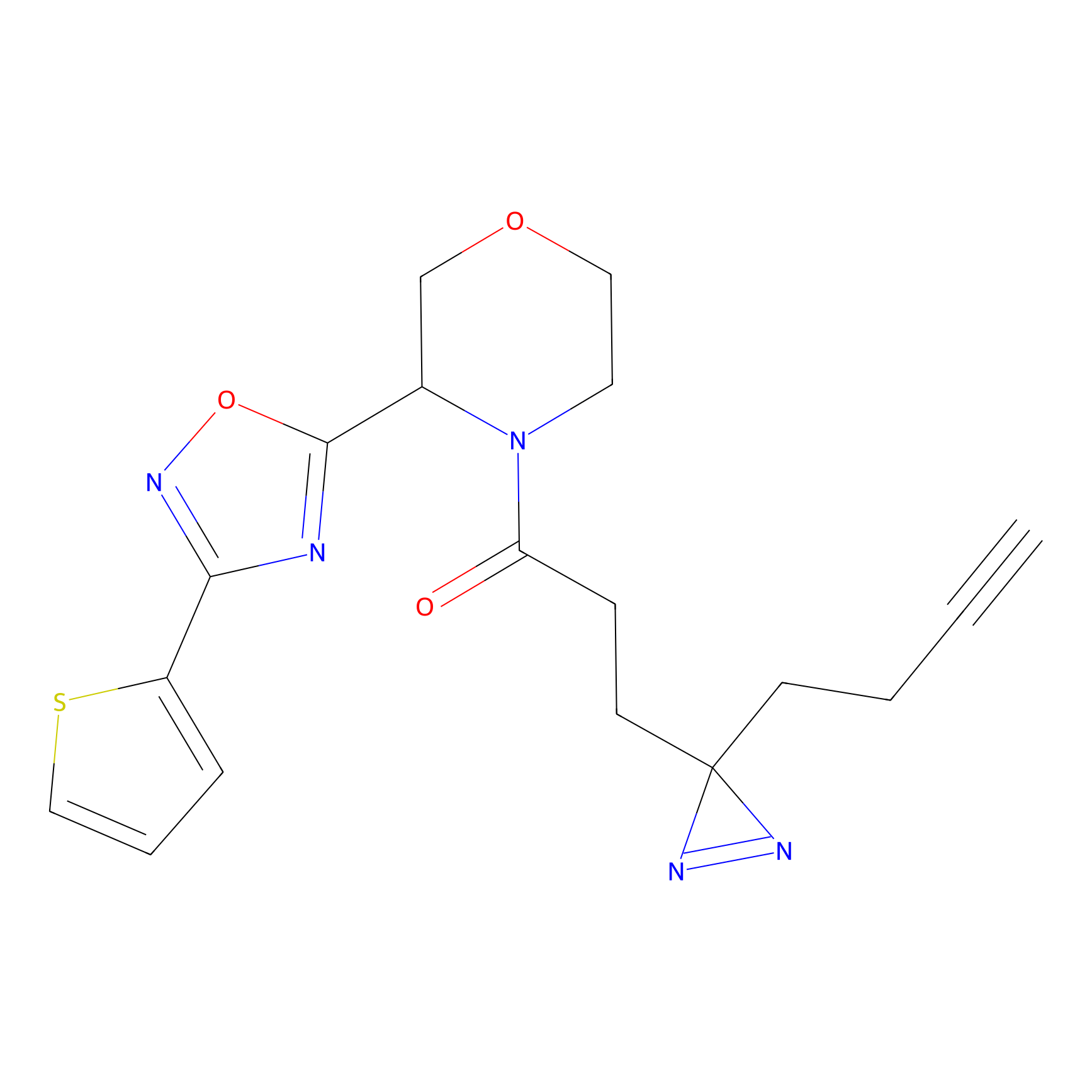
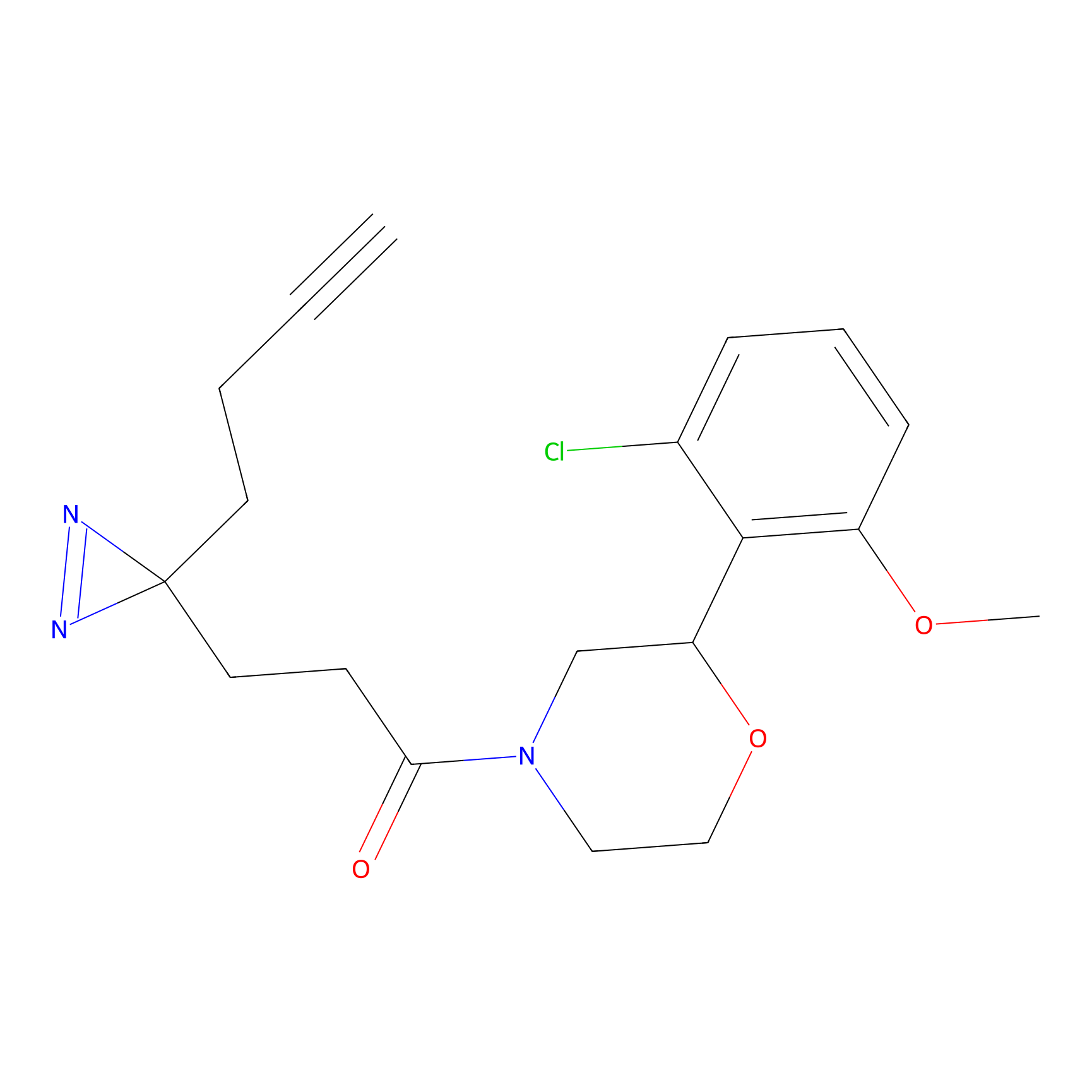
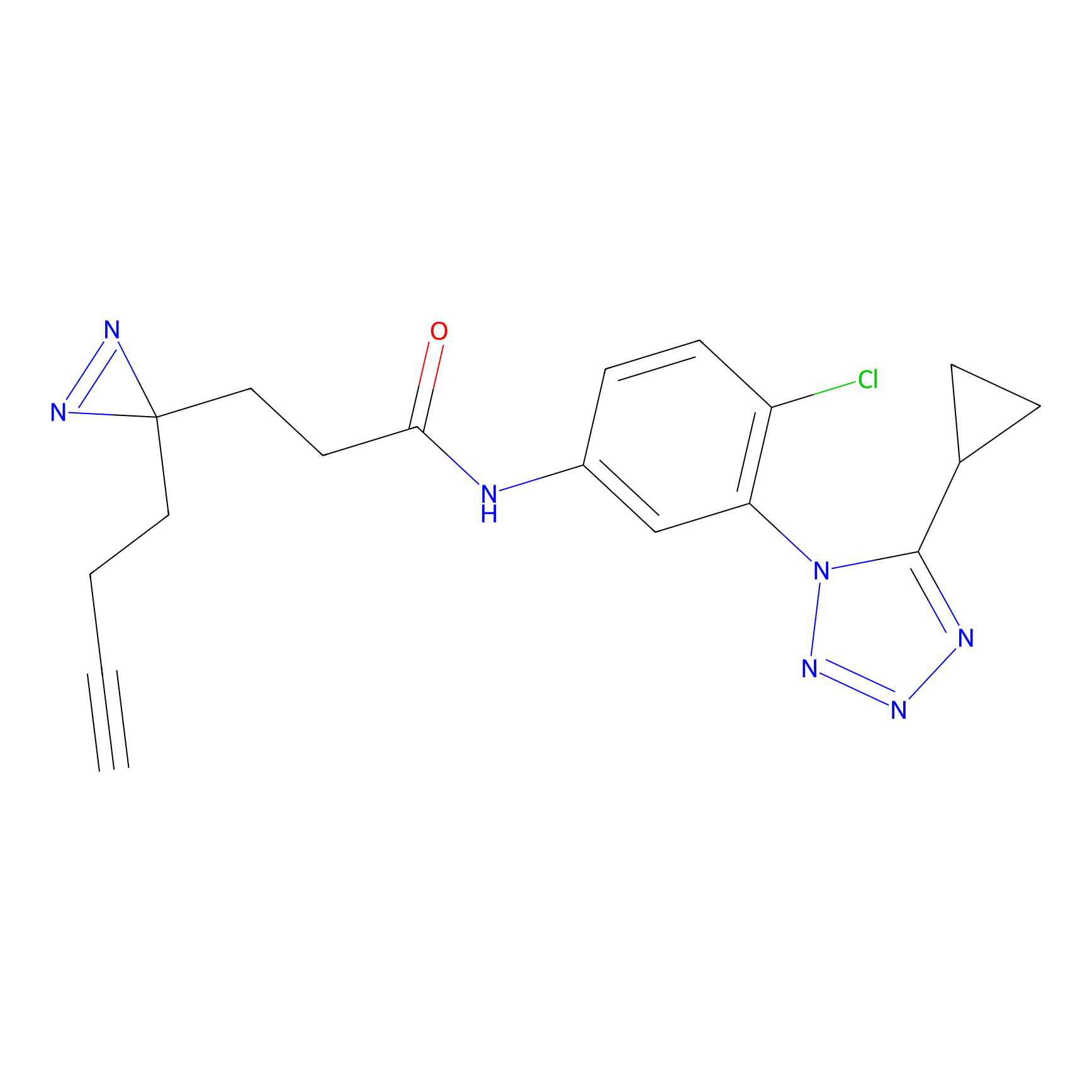
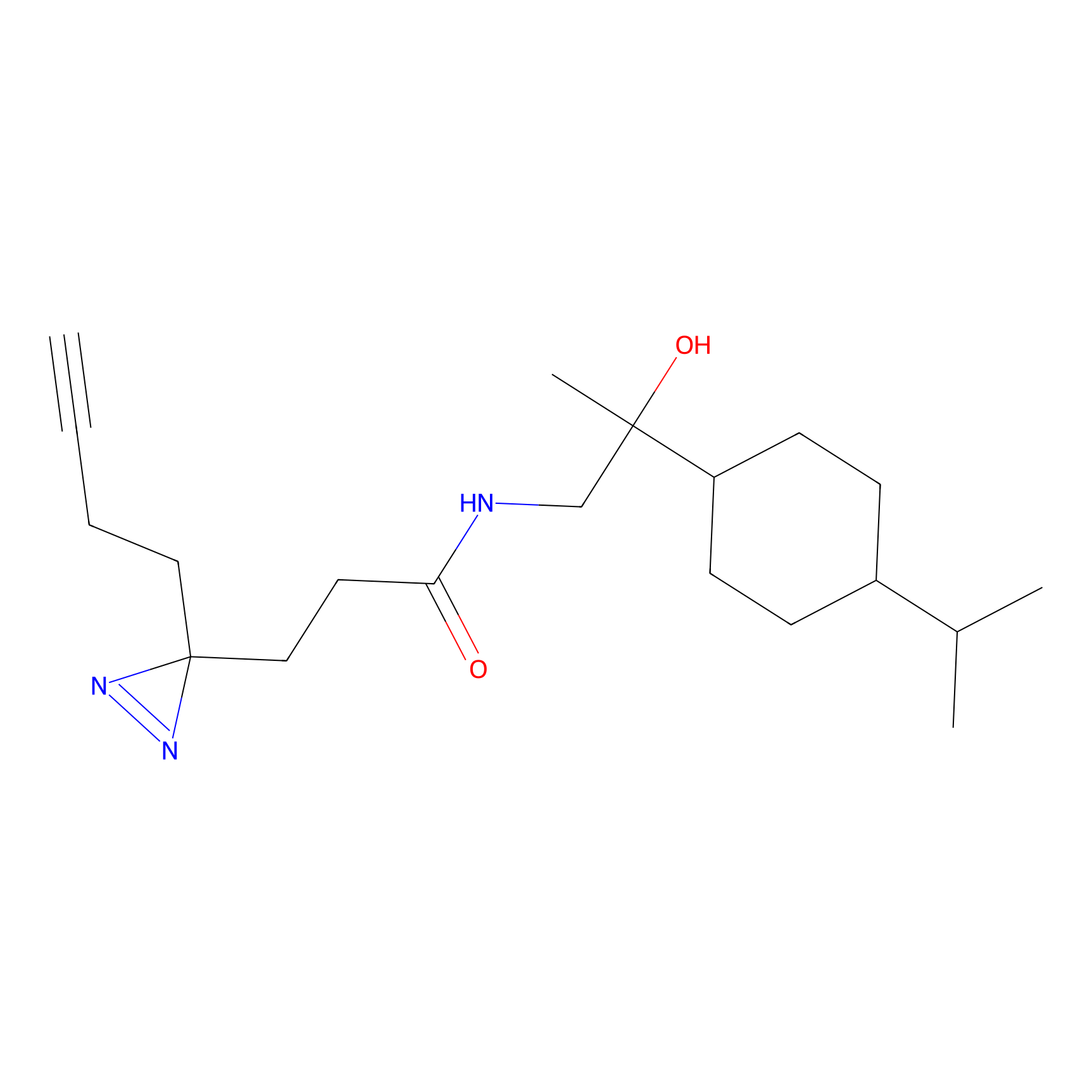
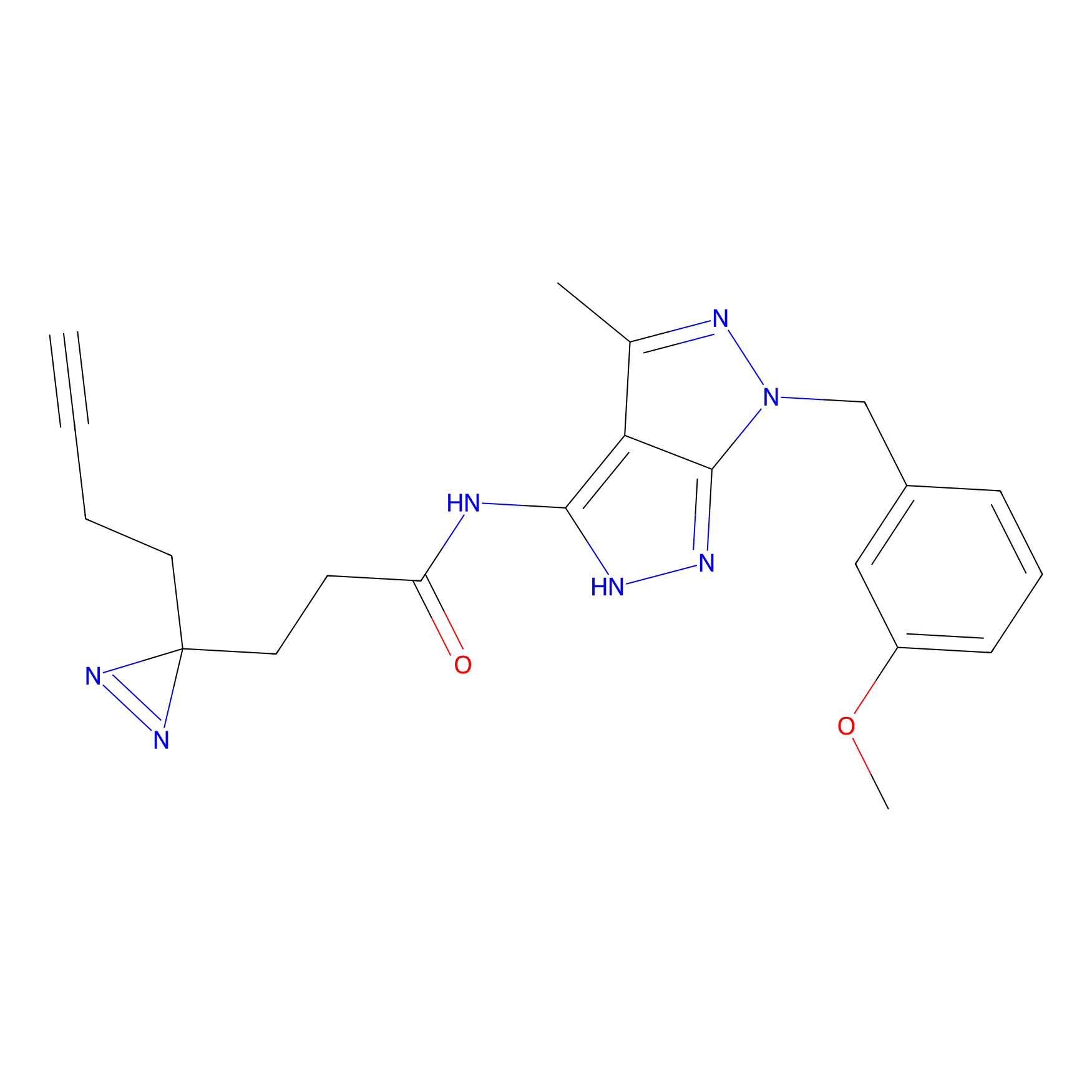
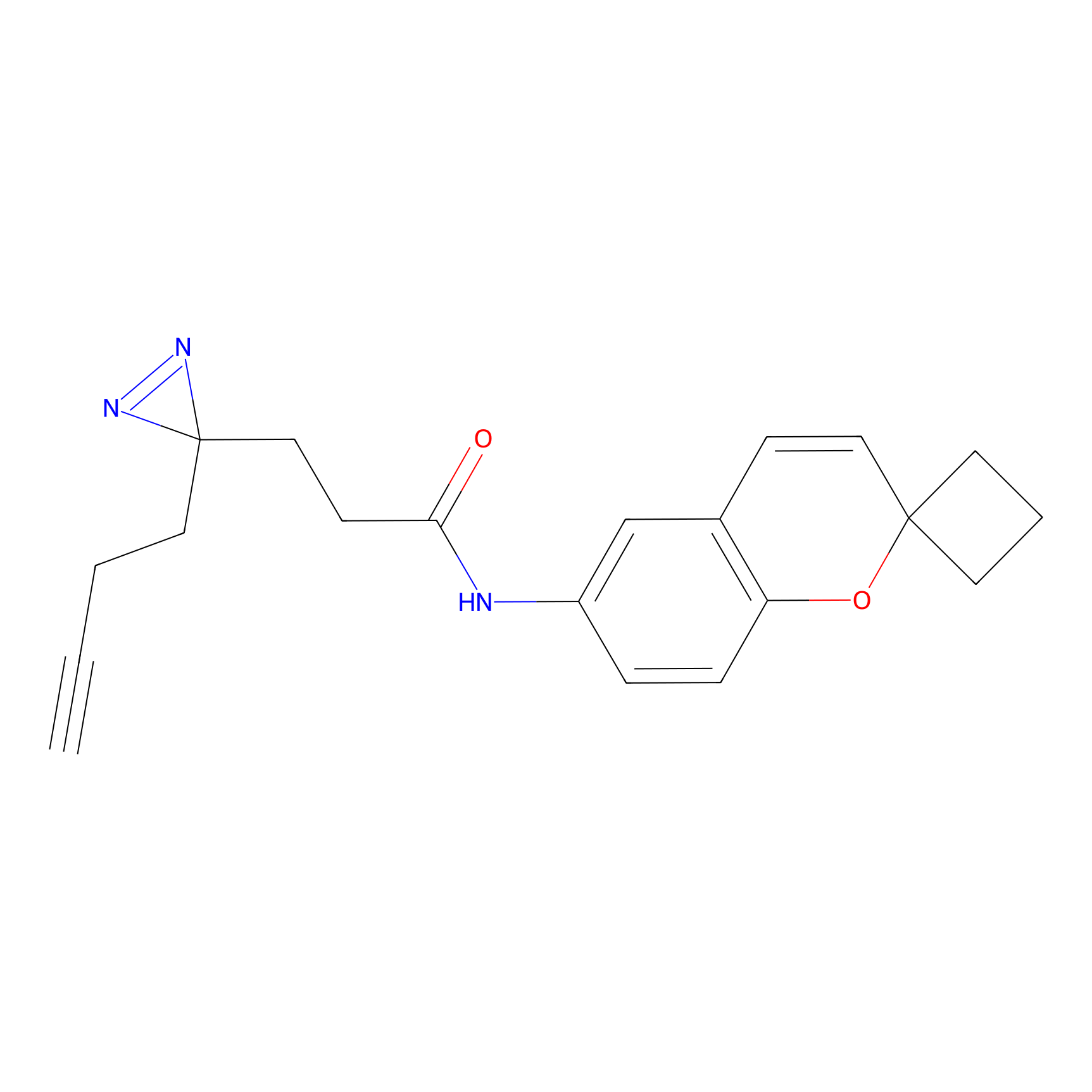
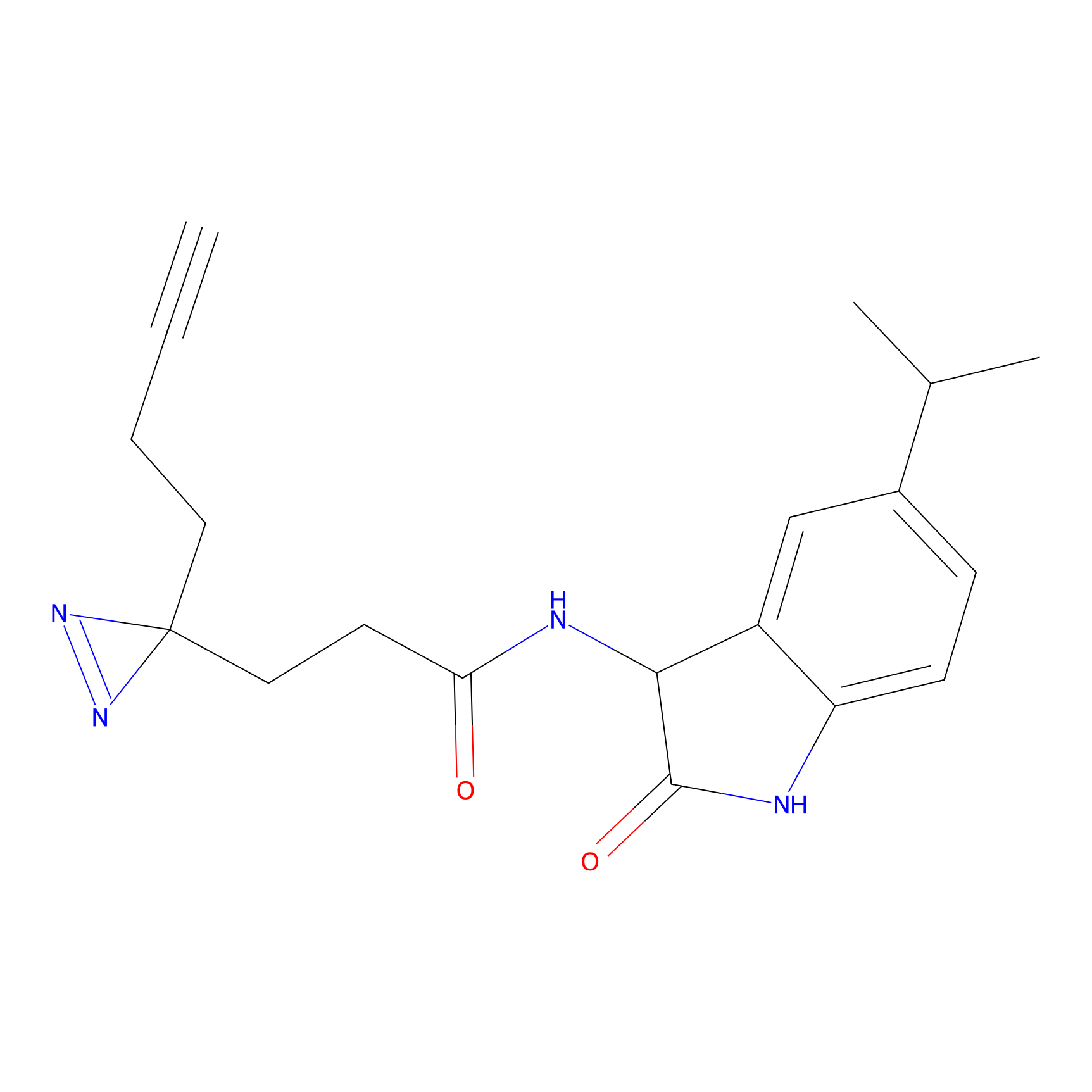
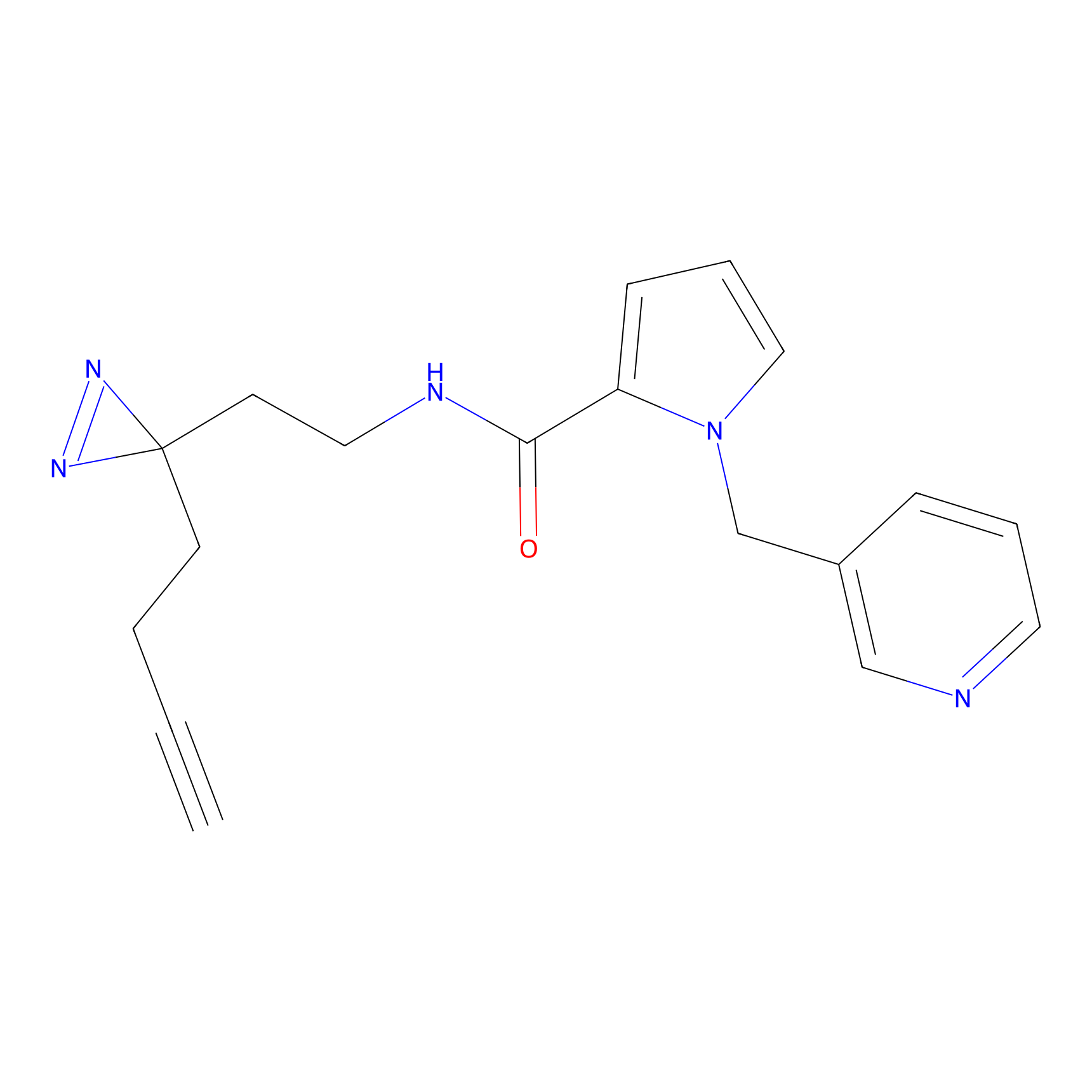
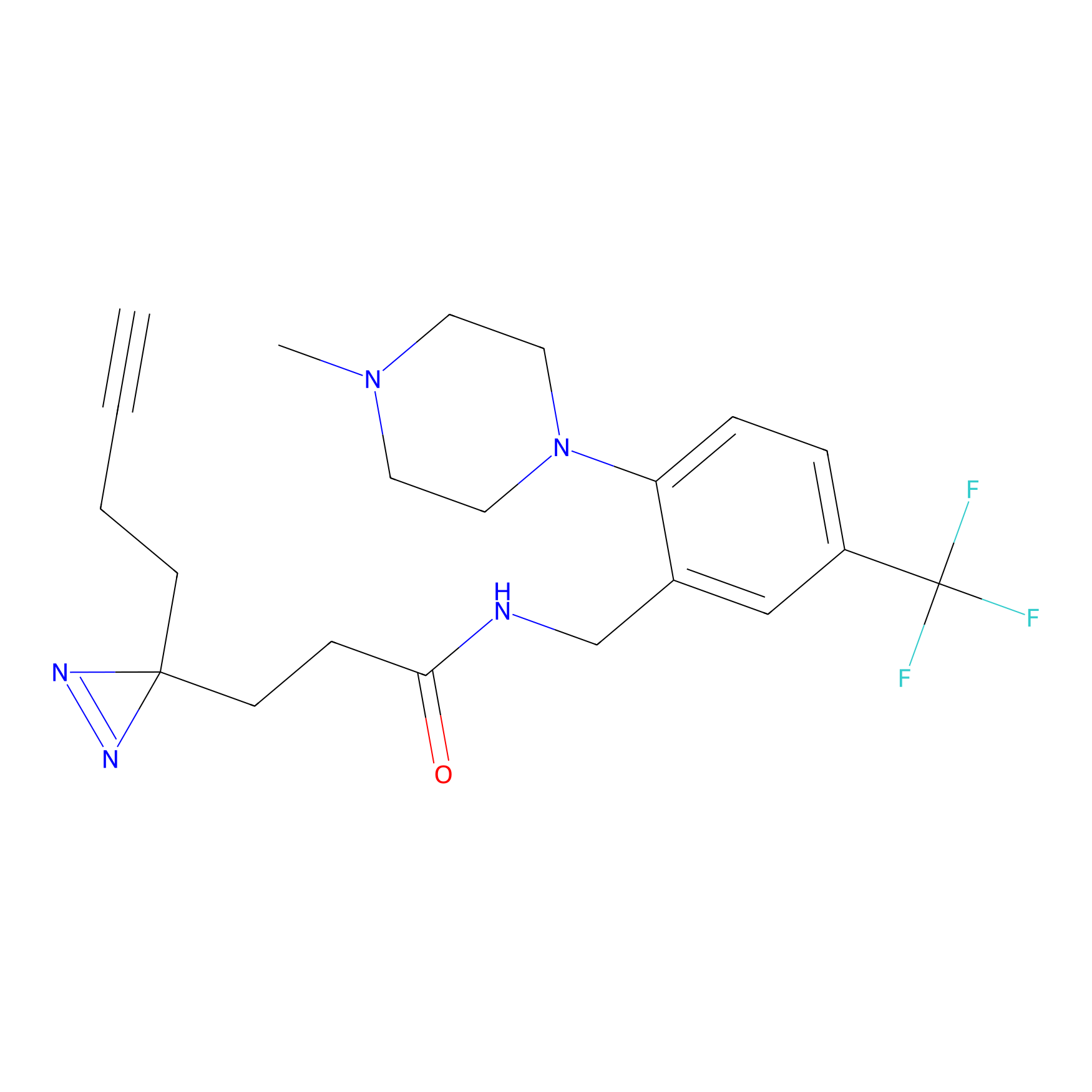
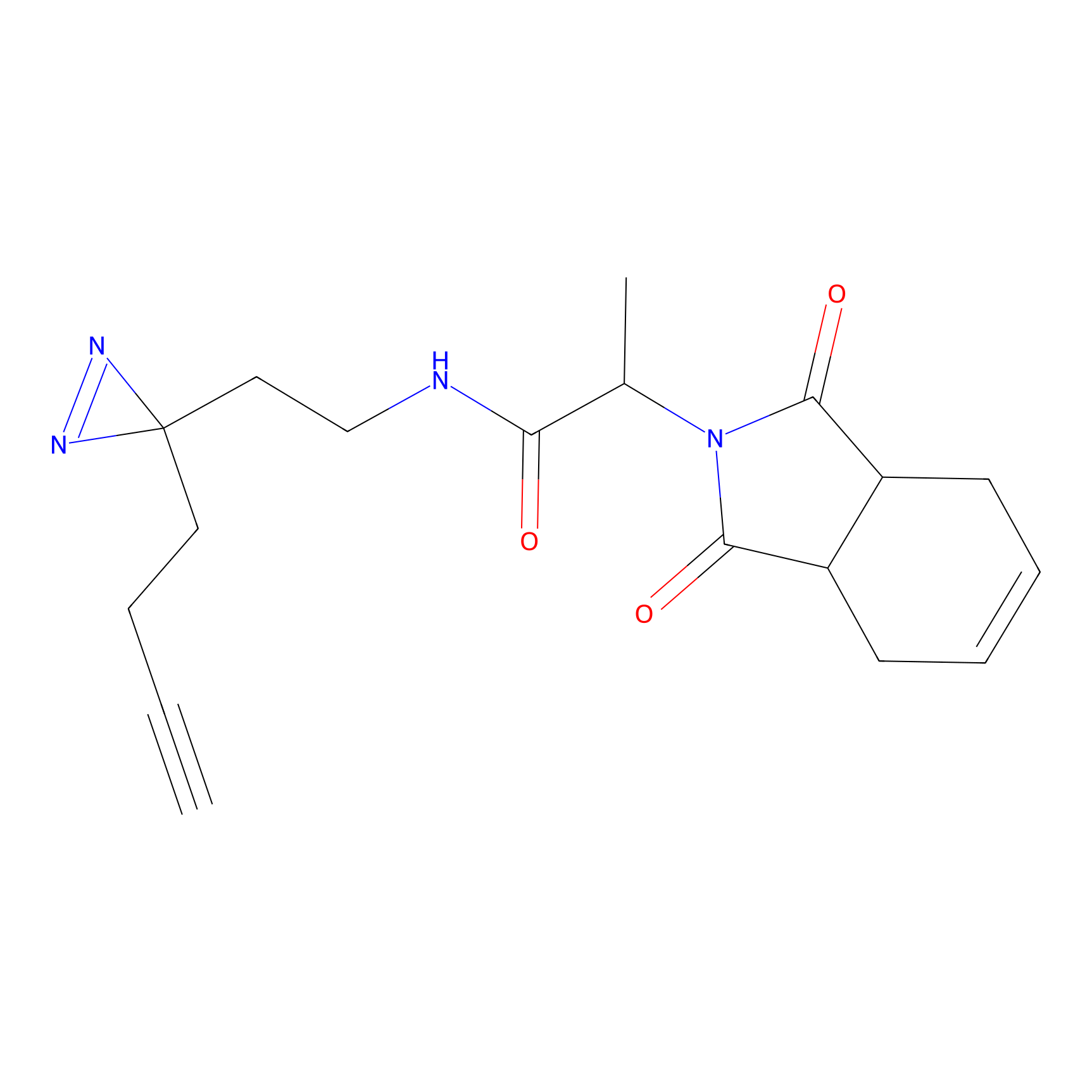
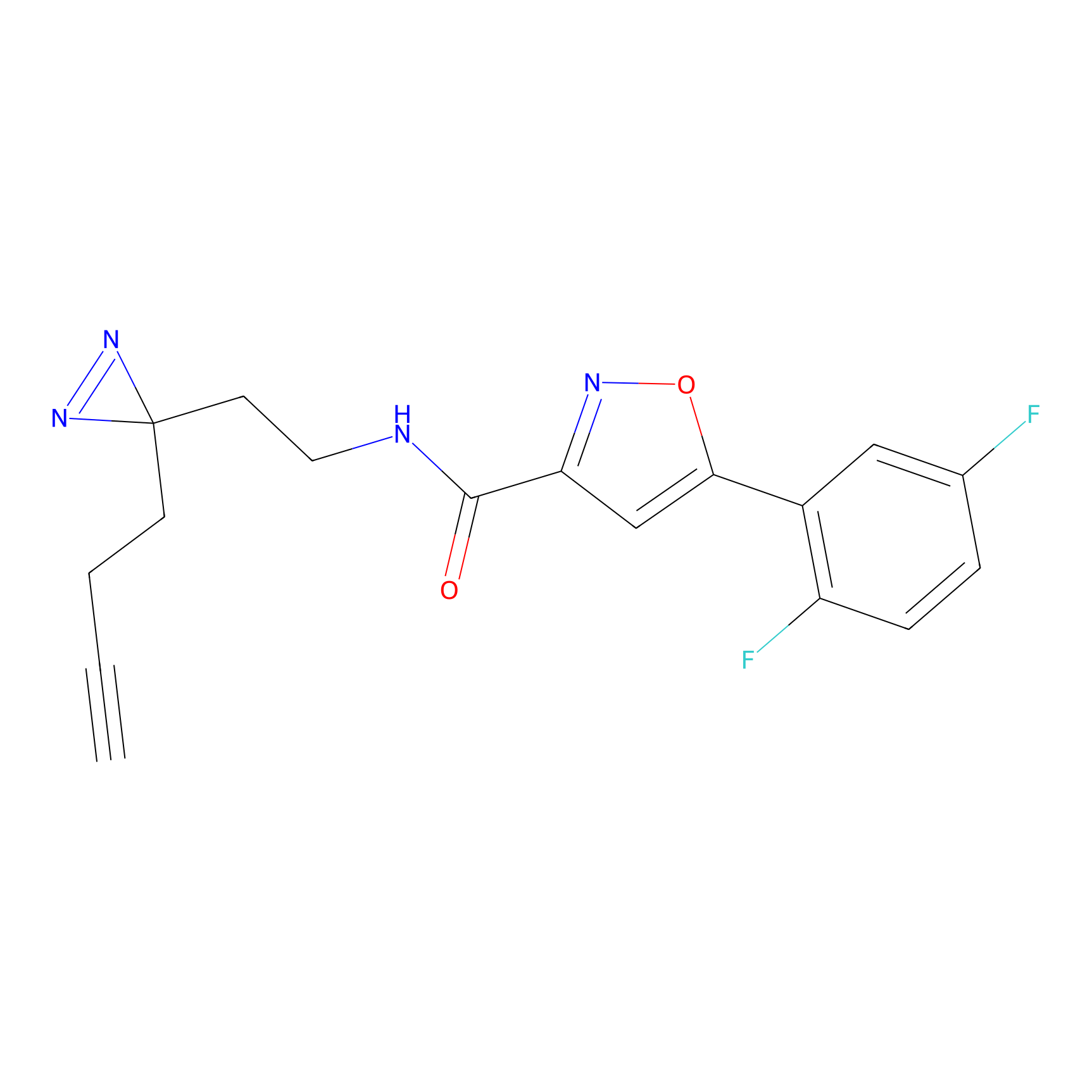
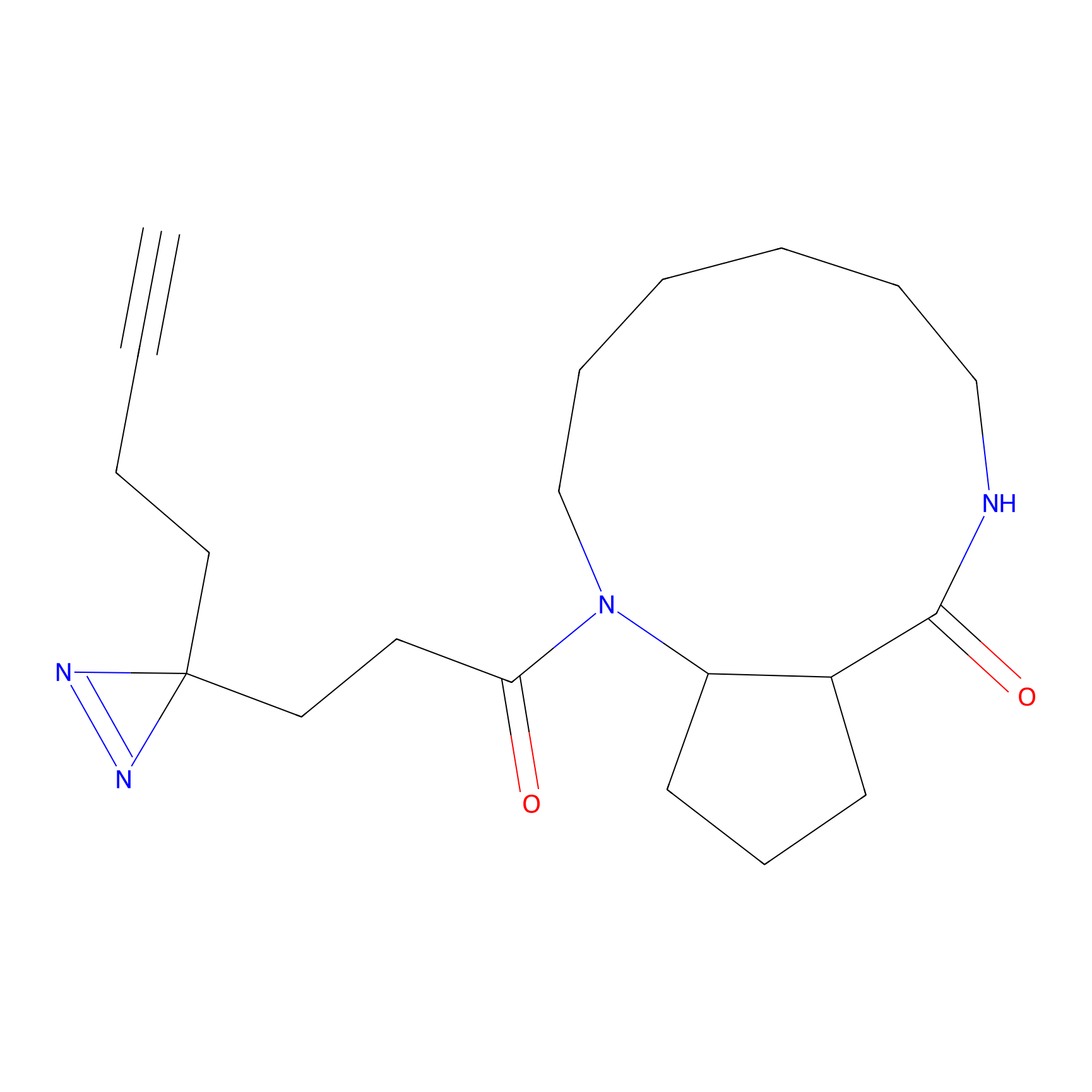
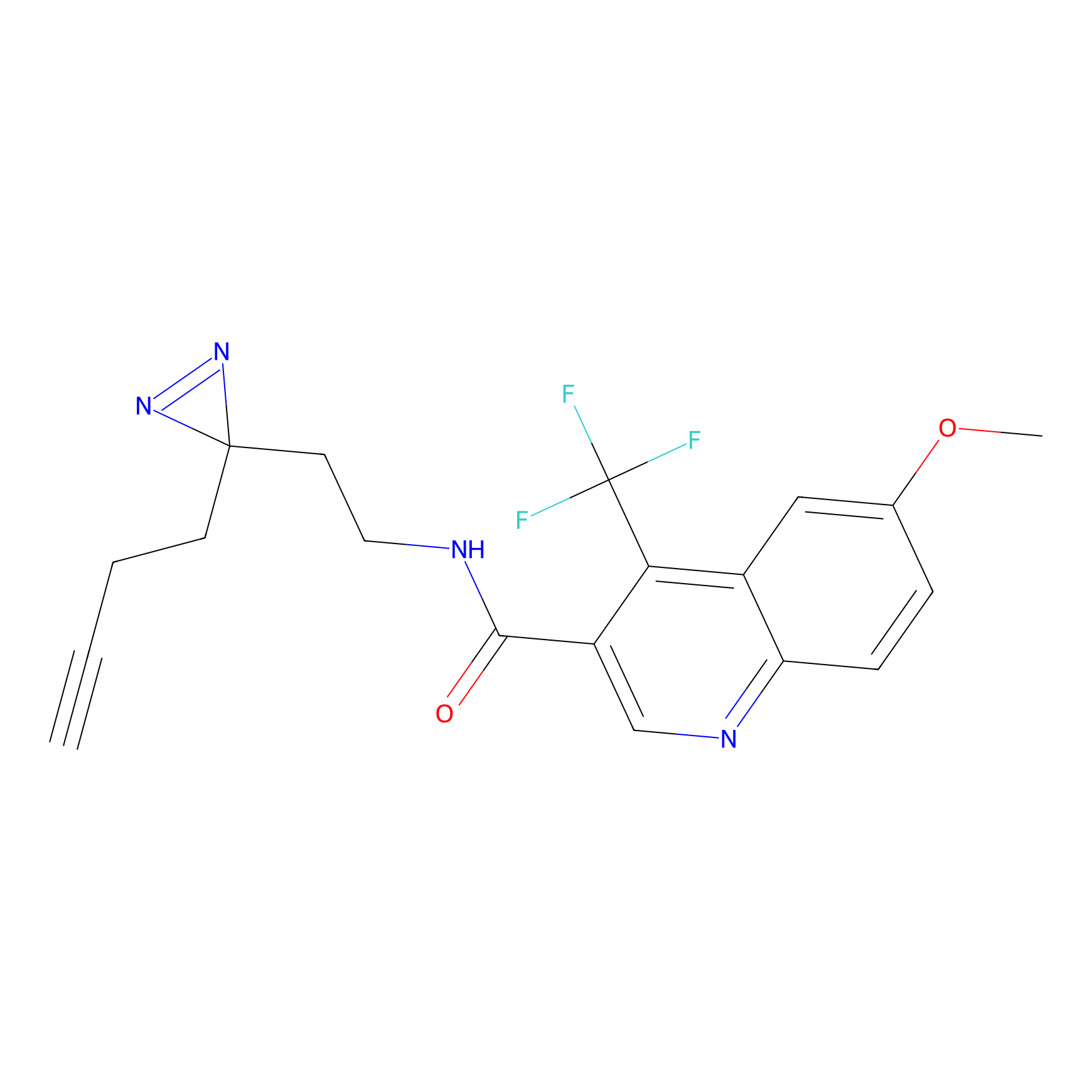
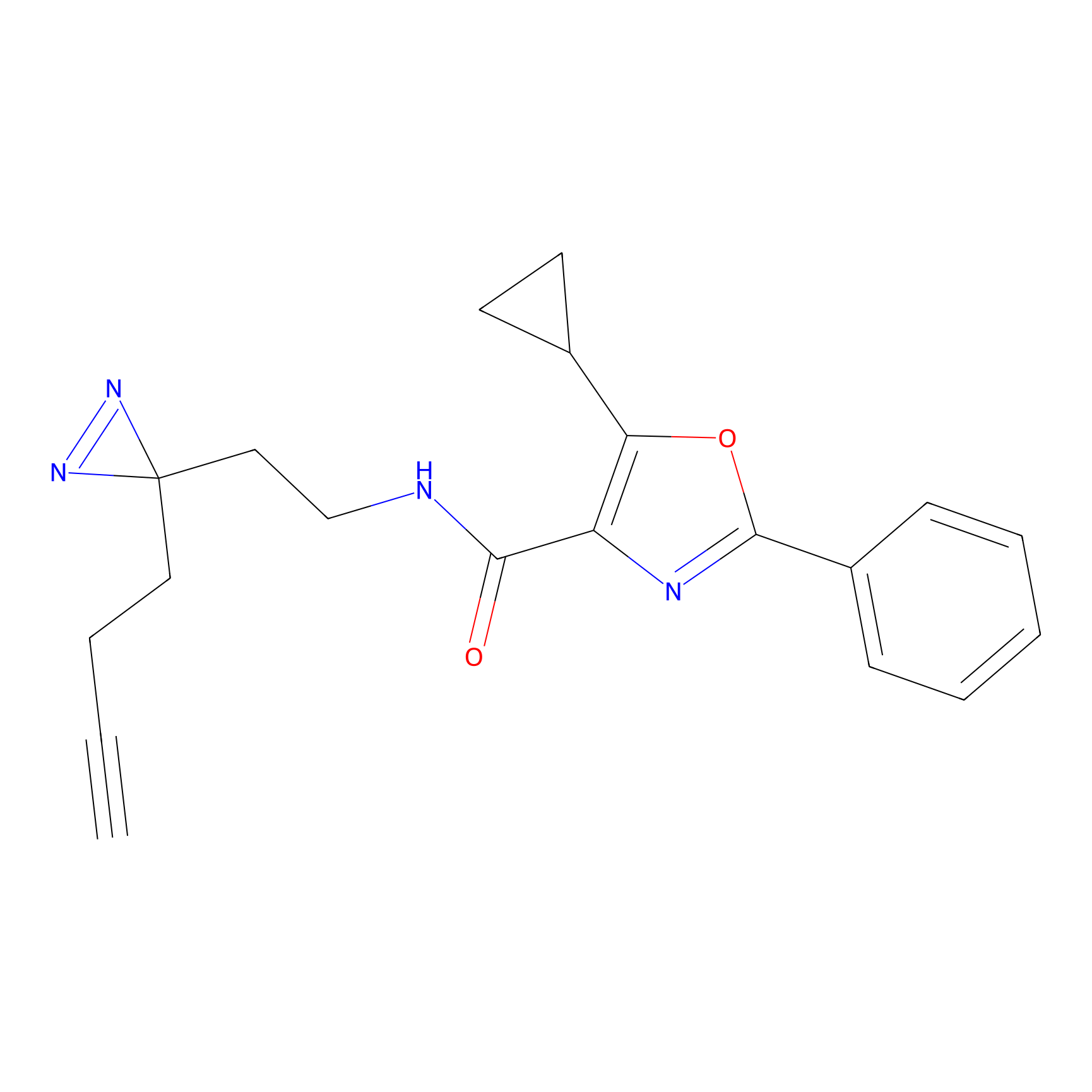
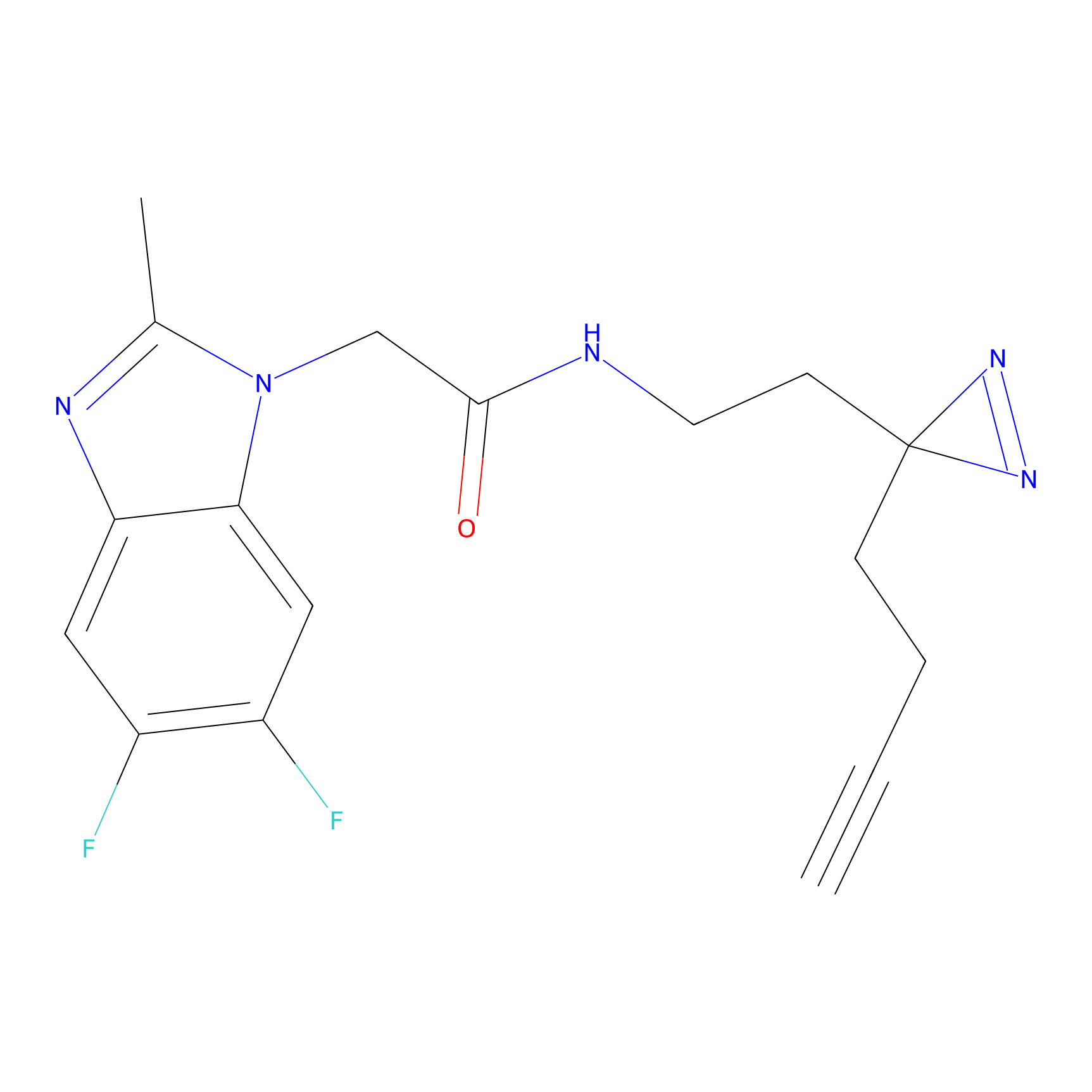
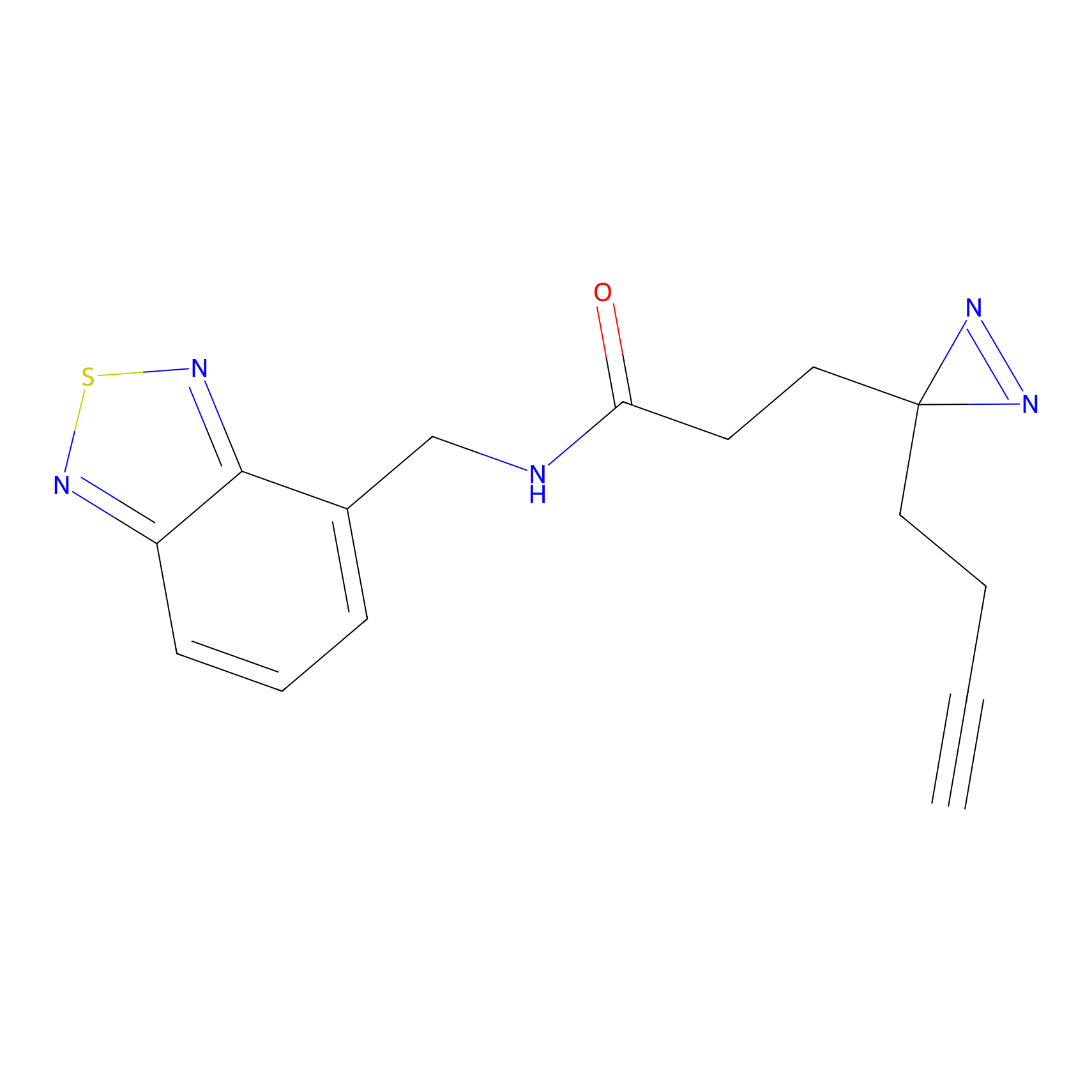
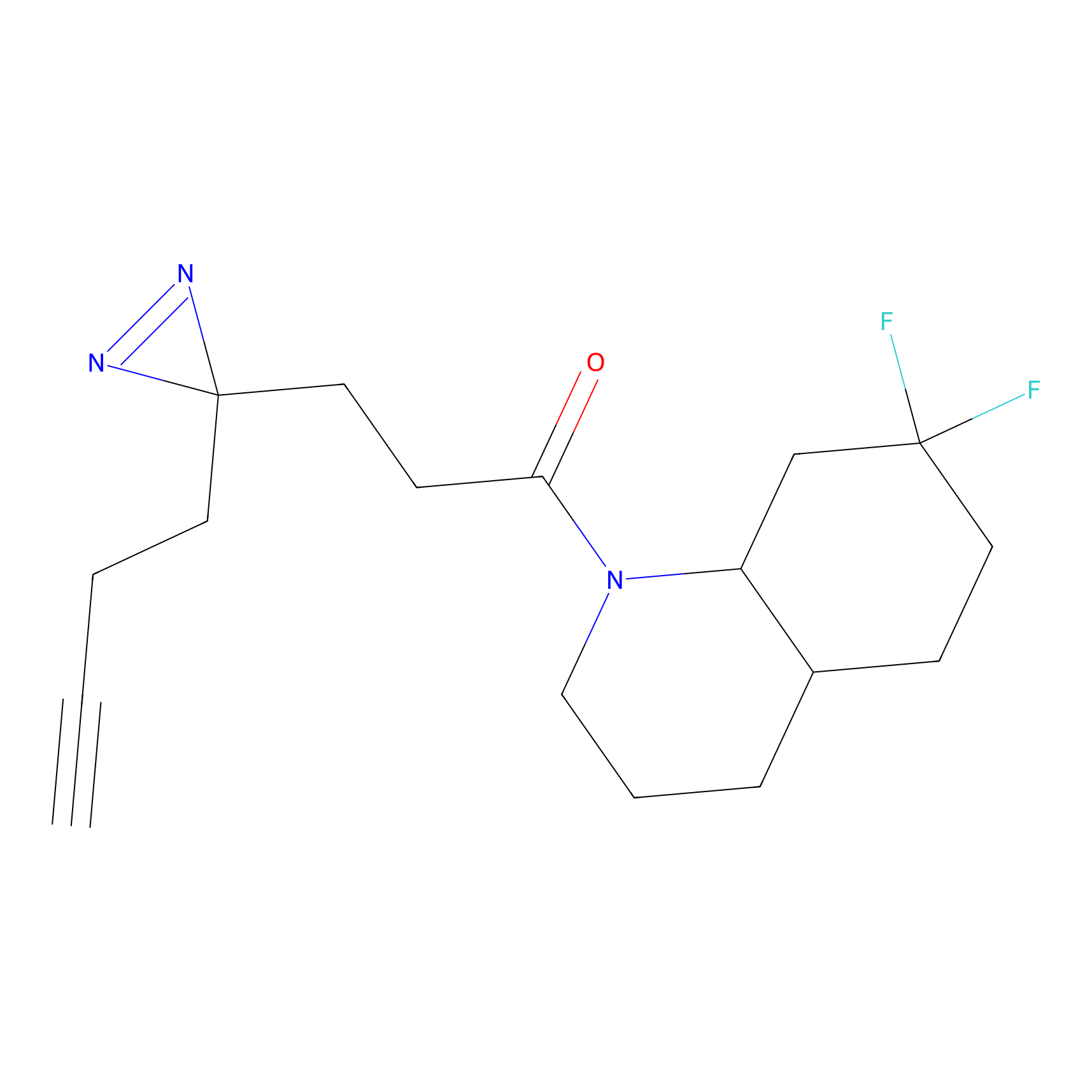
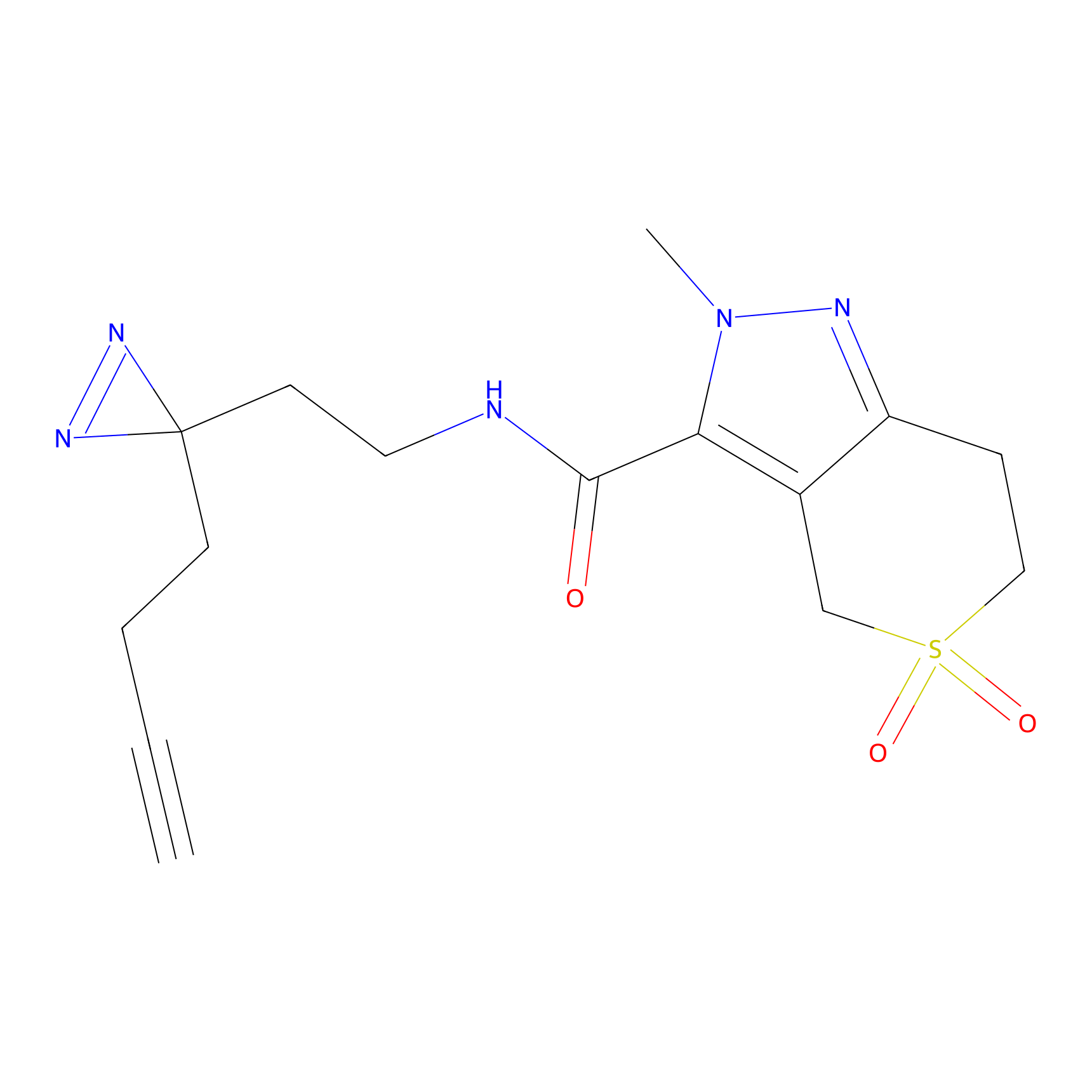
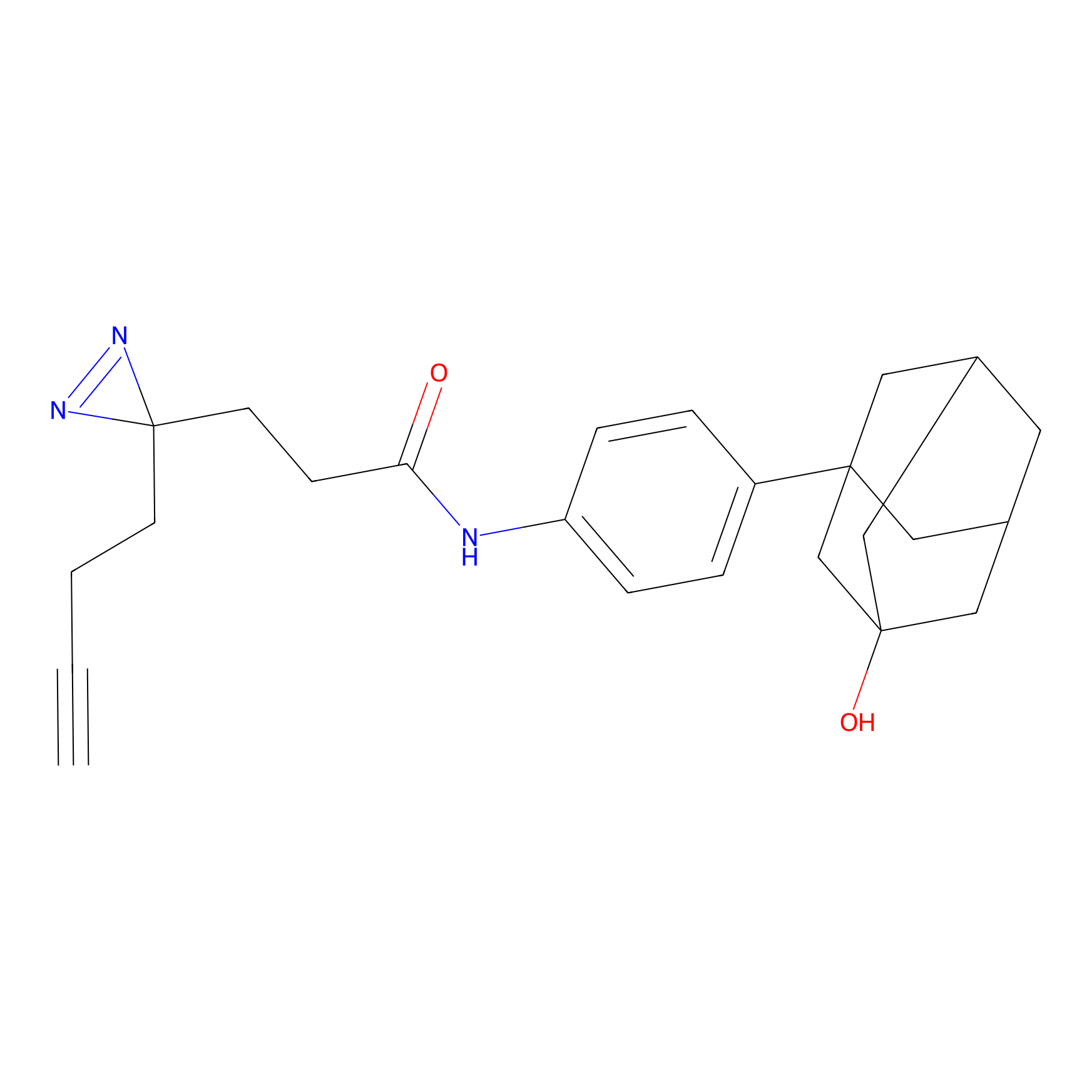
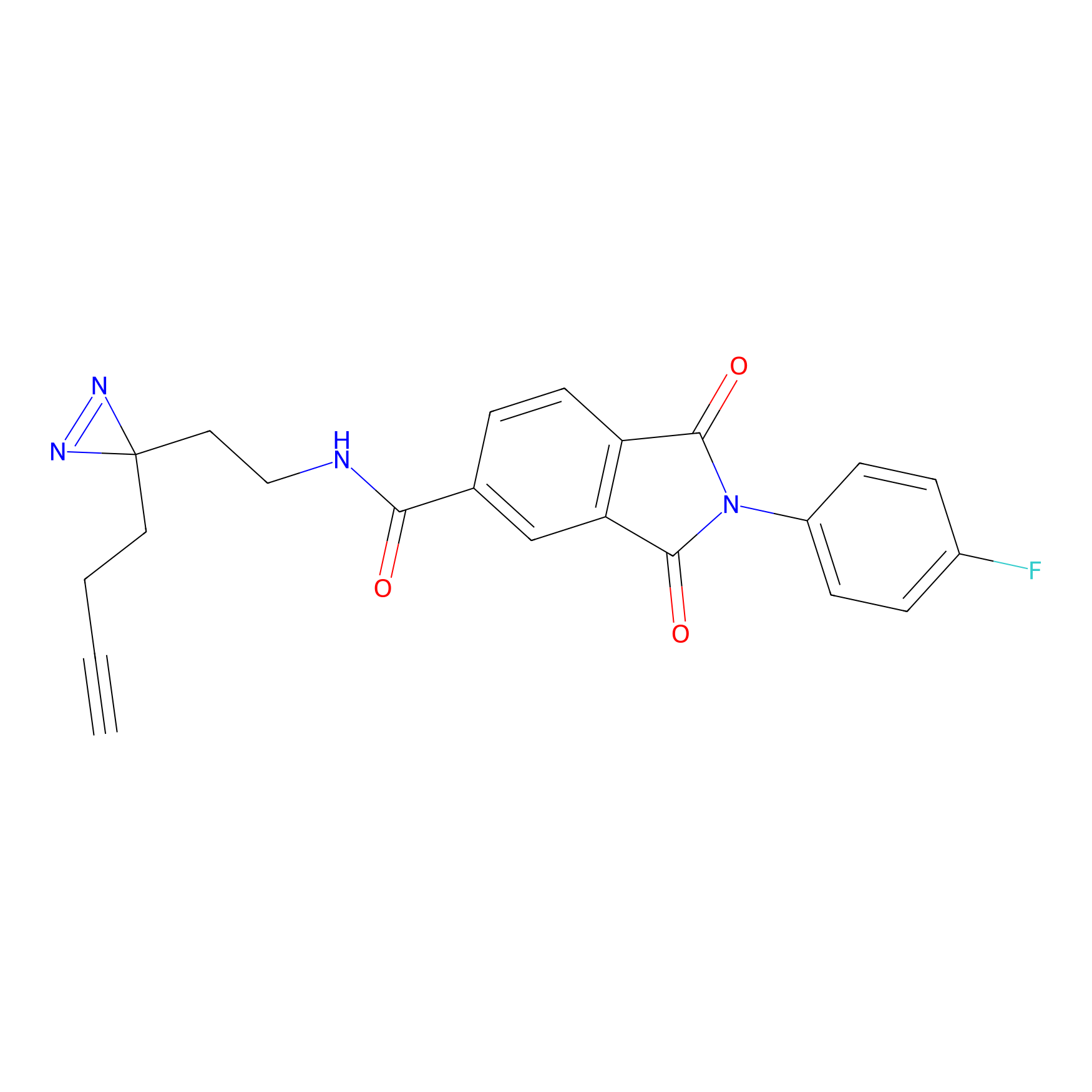
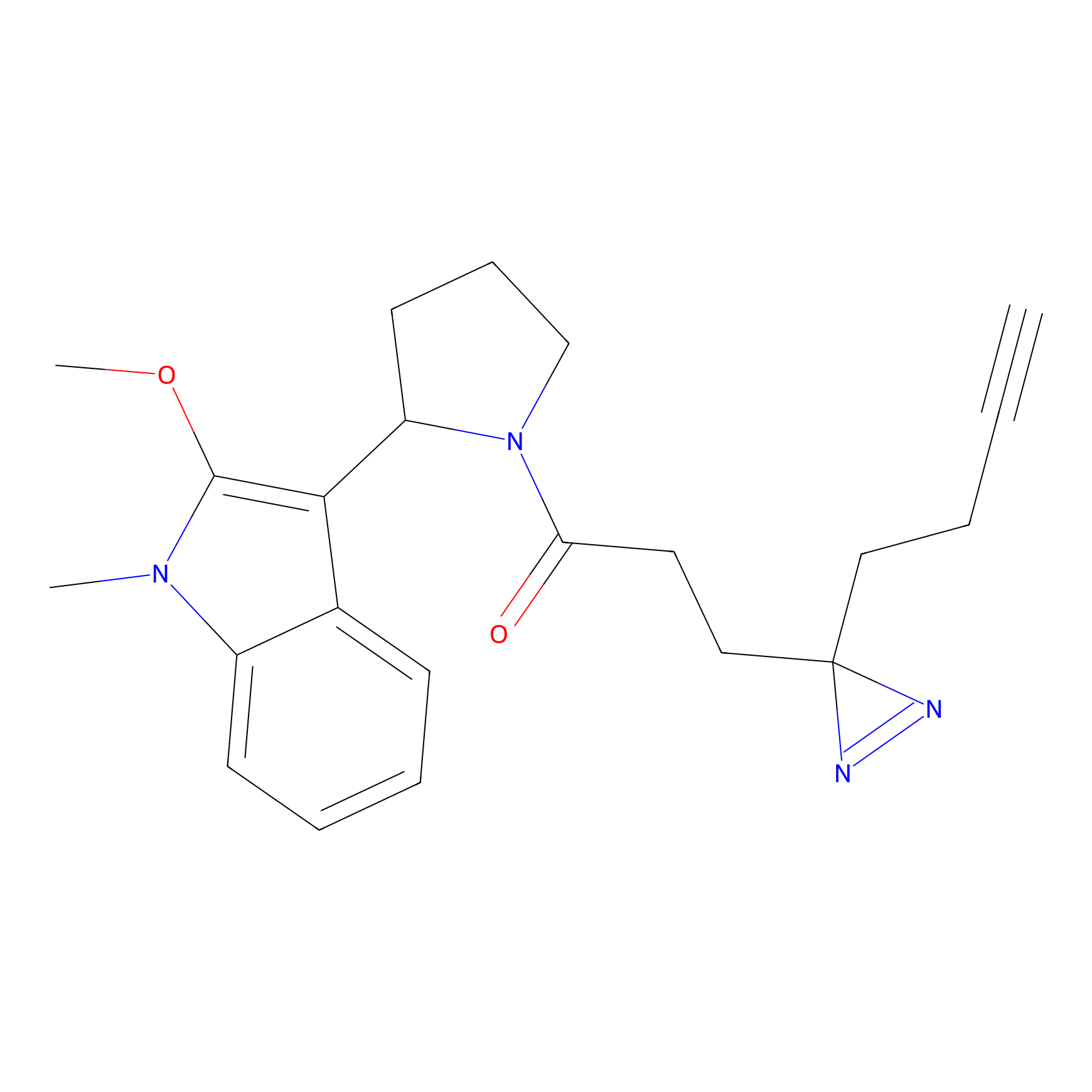
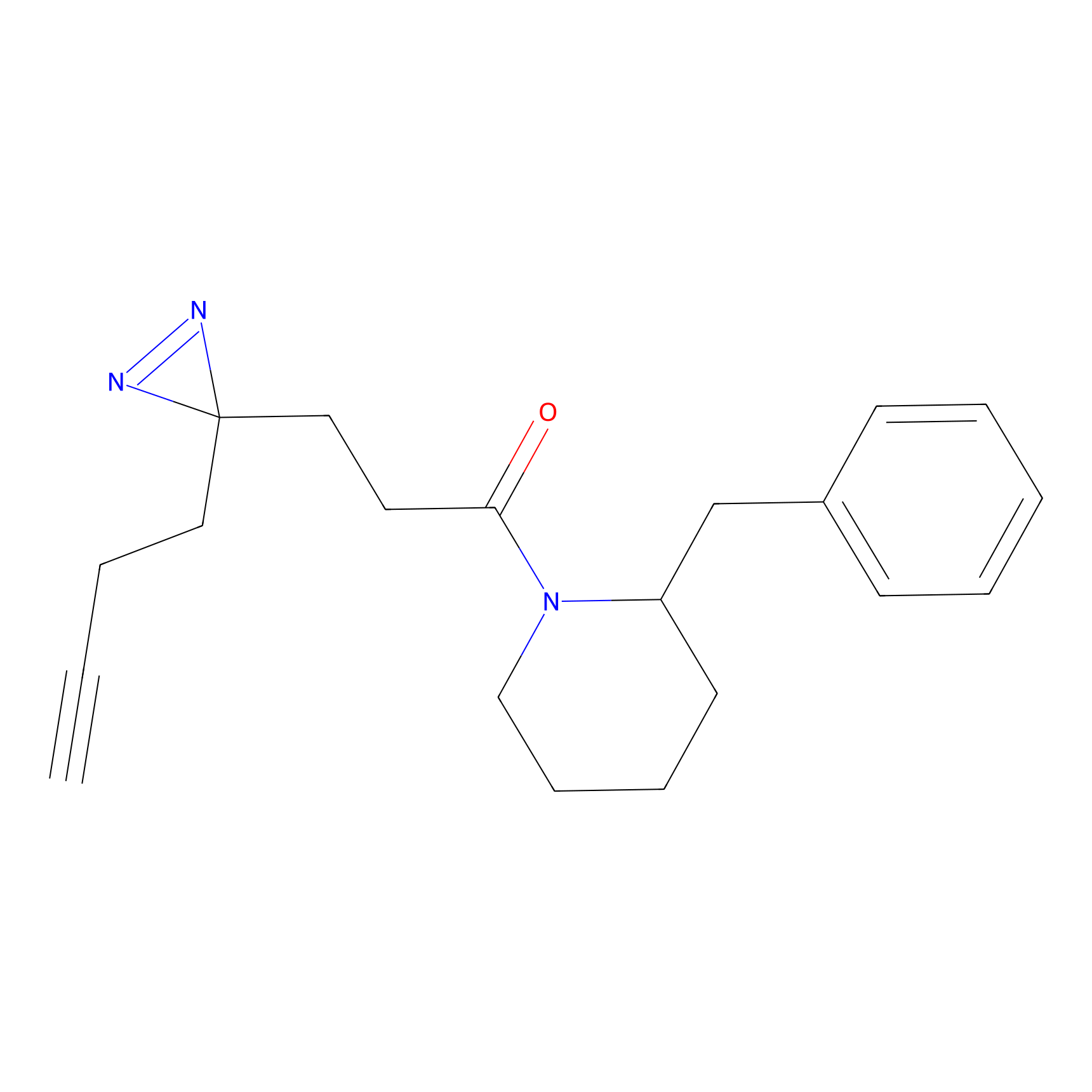
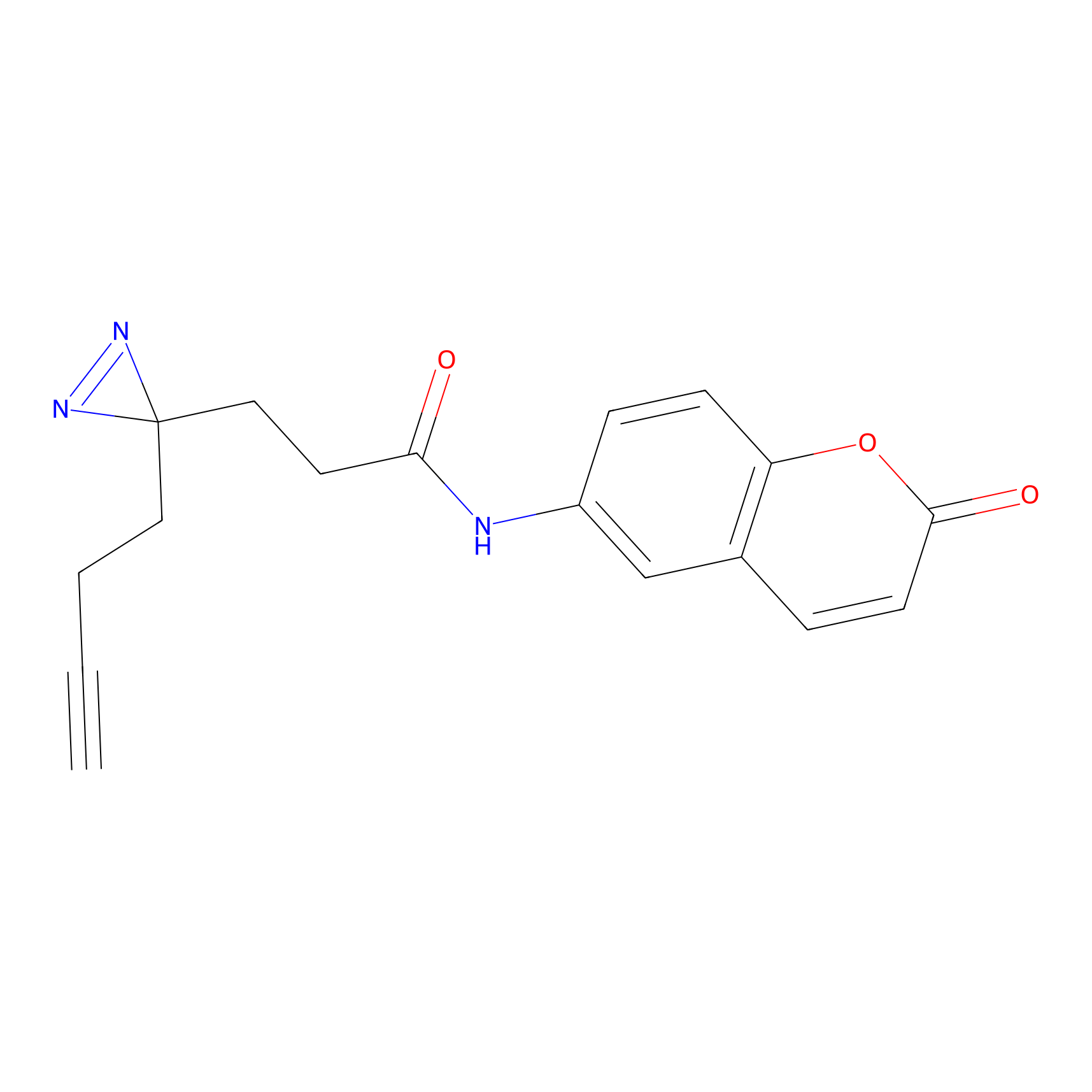
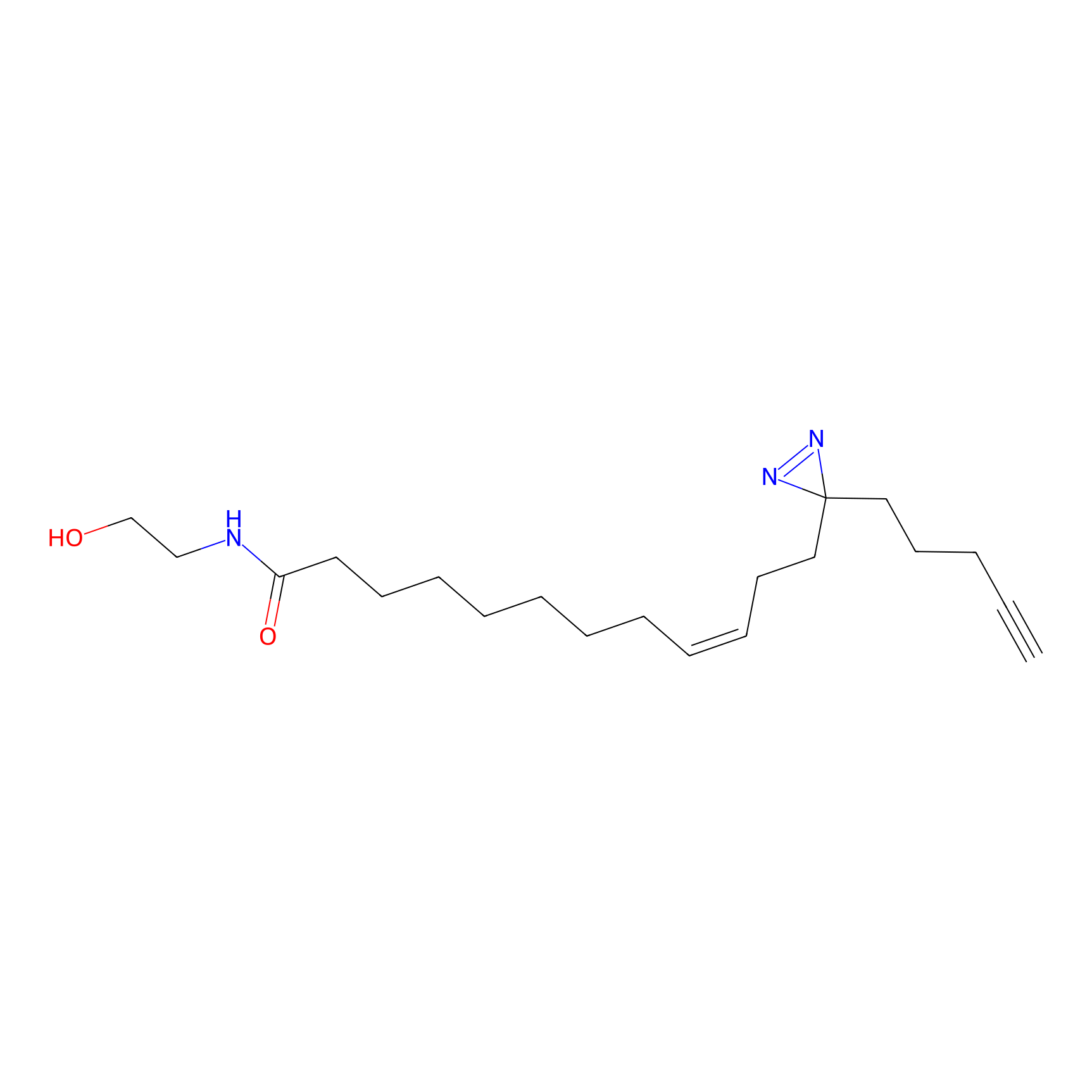
|
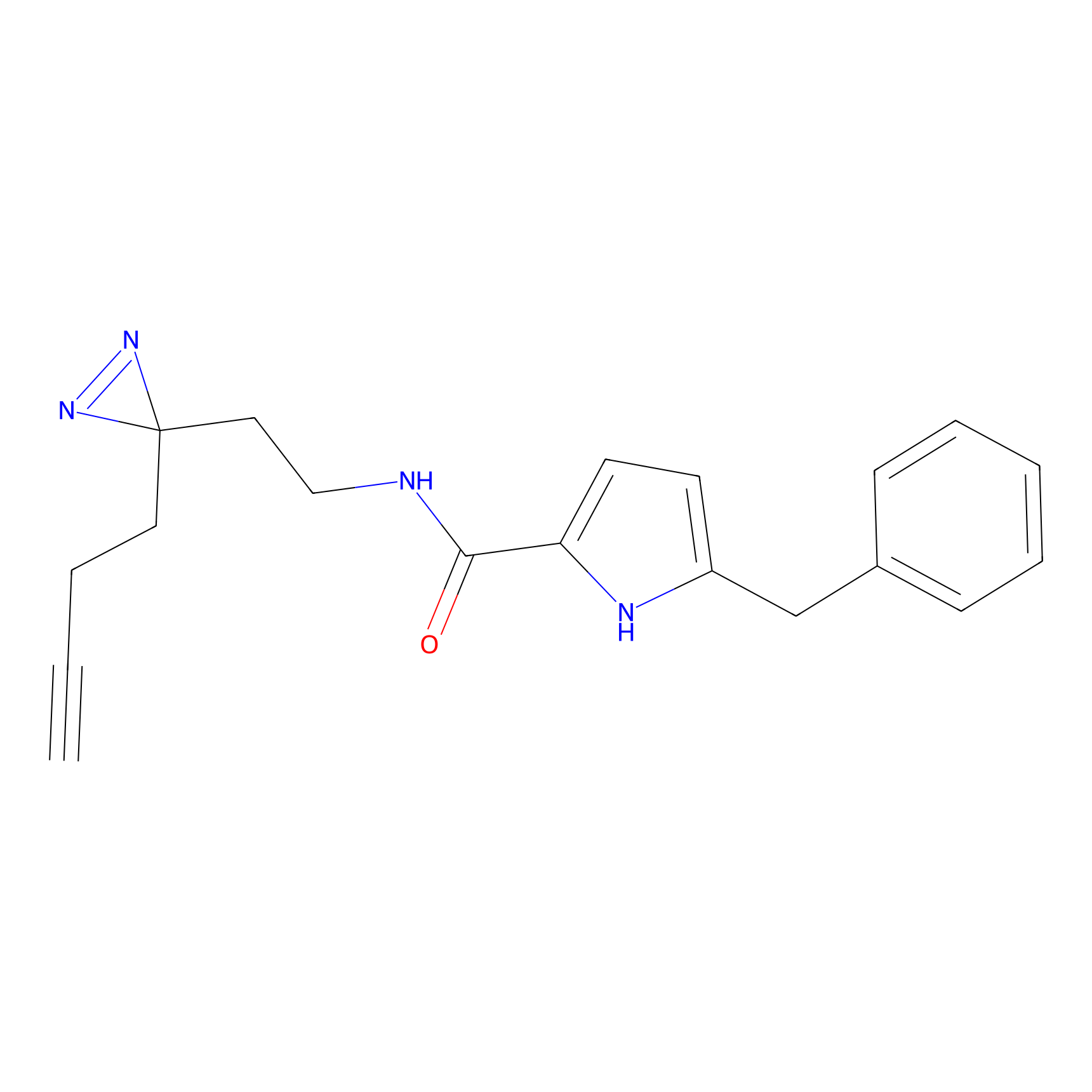
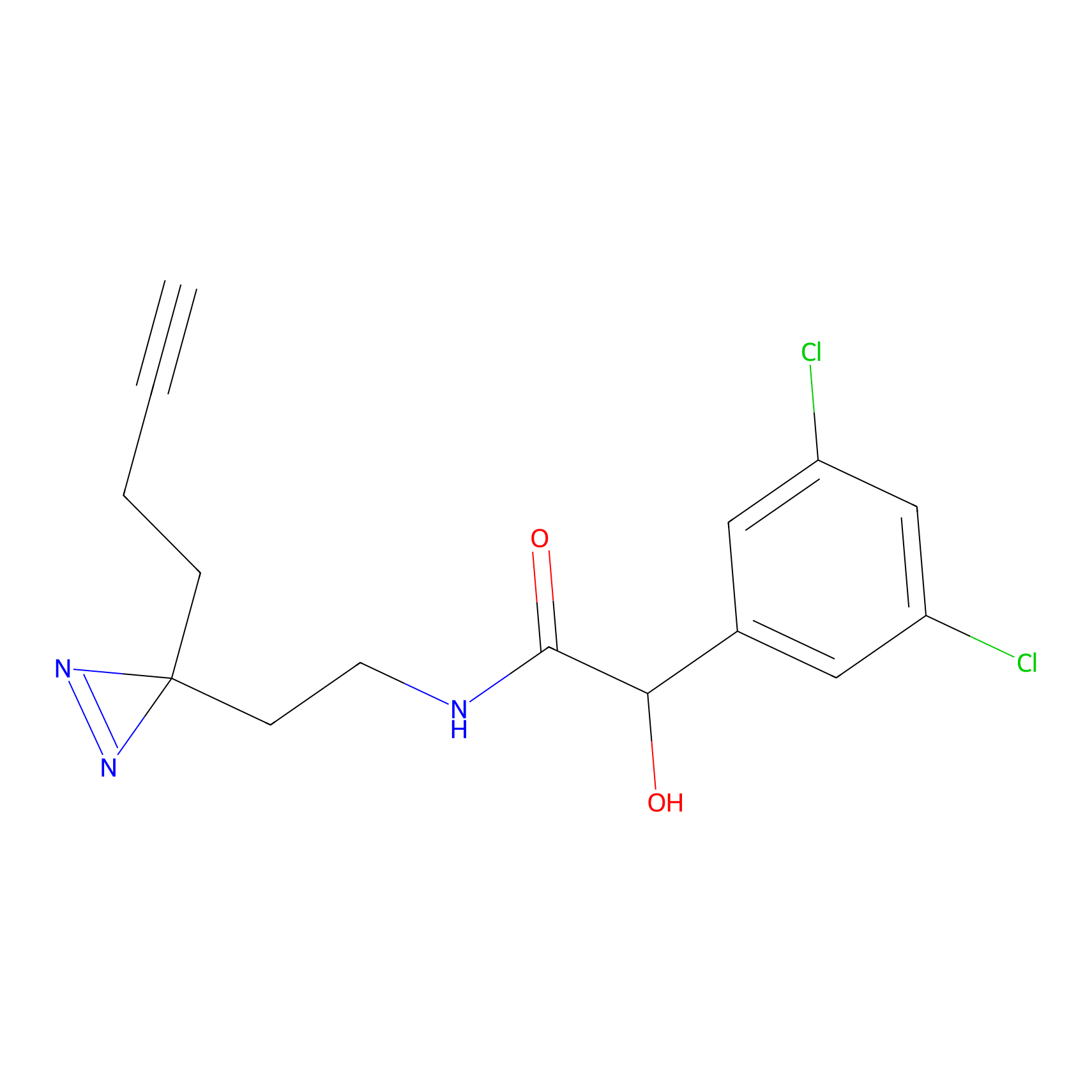
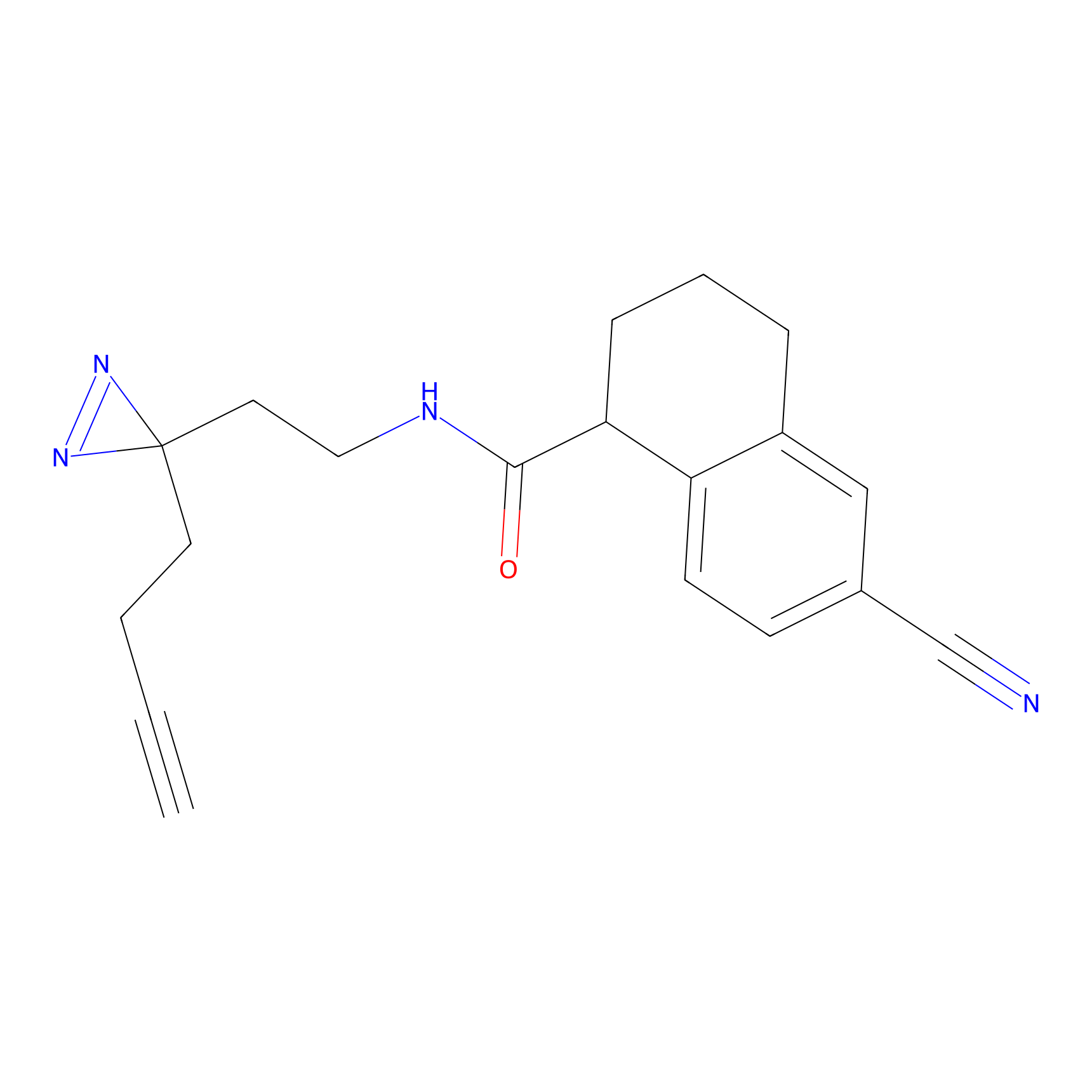
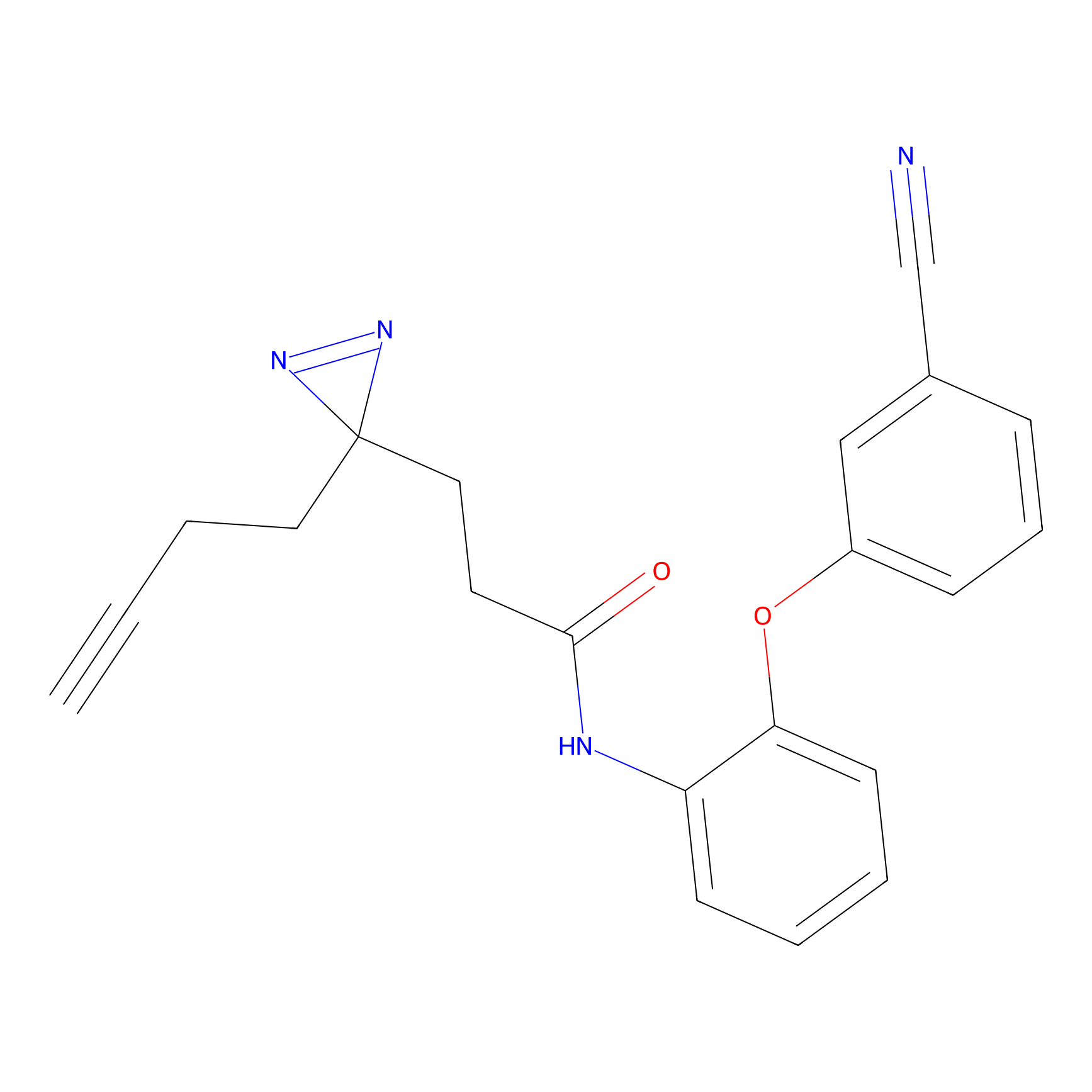
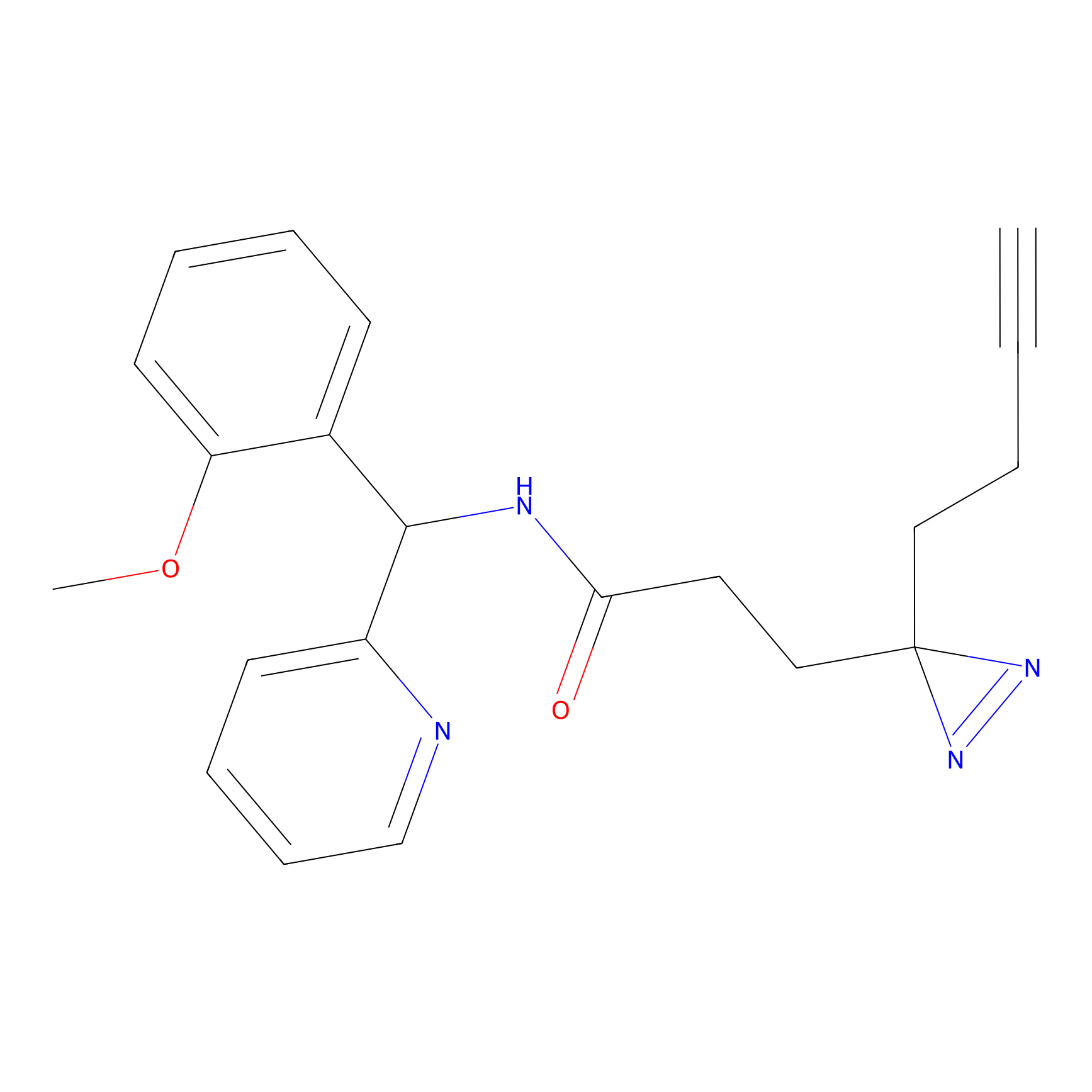
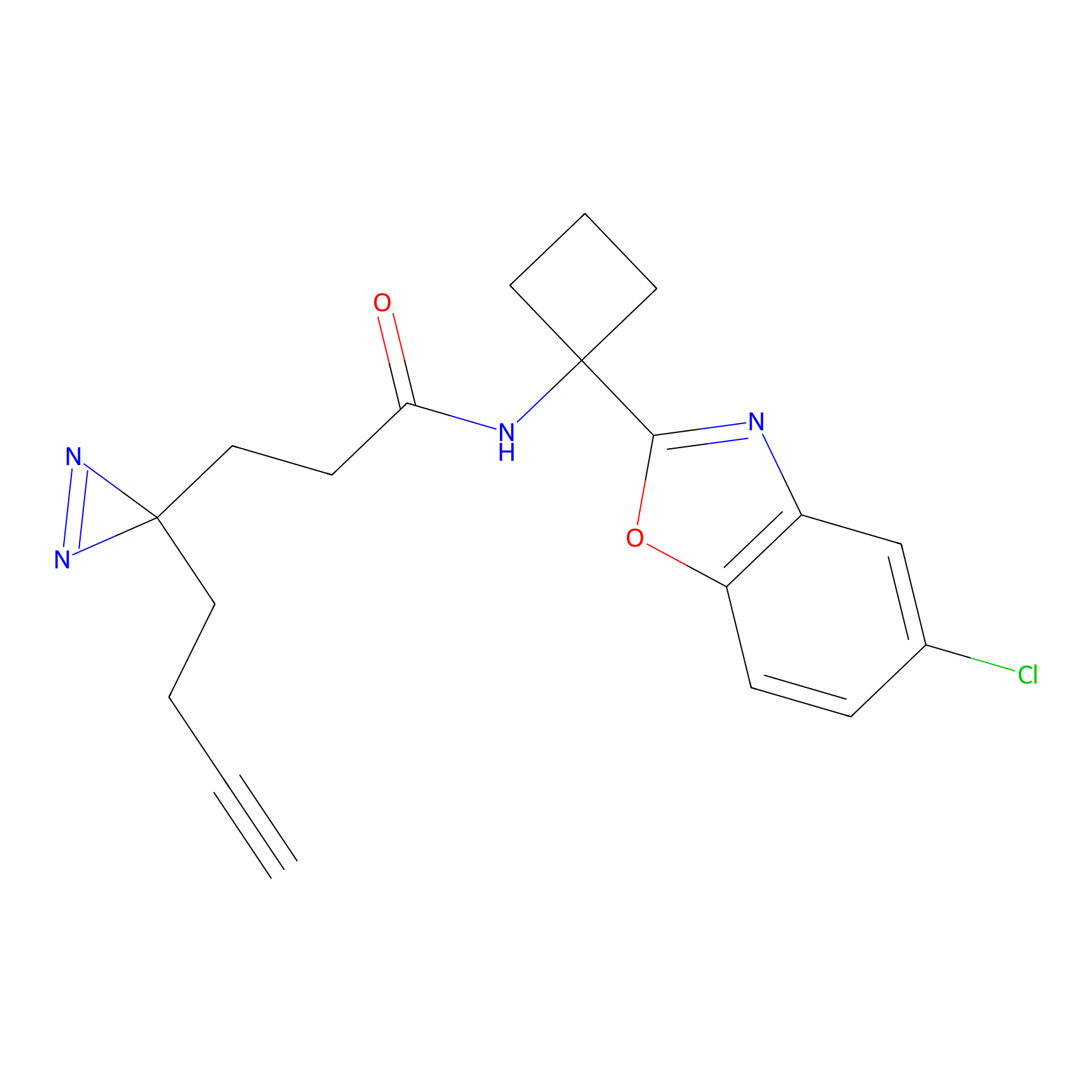
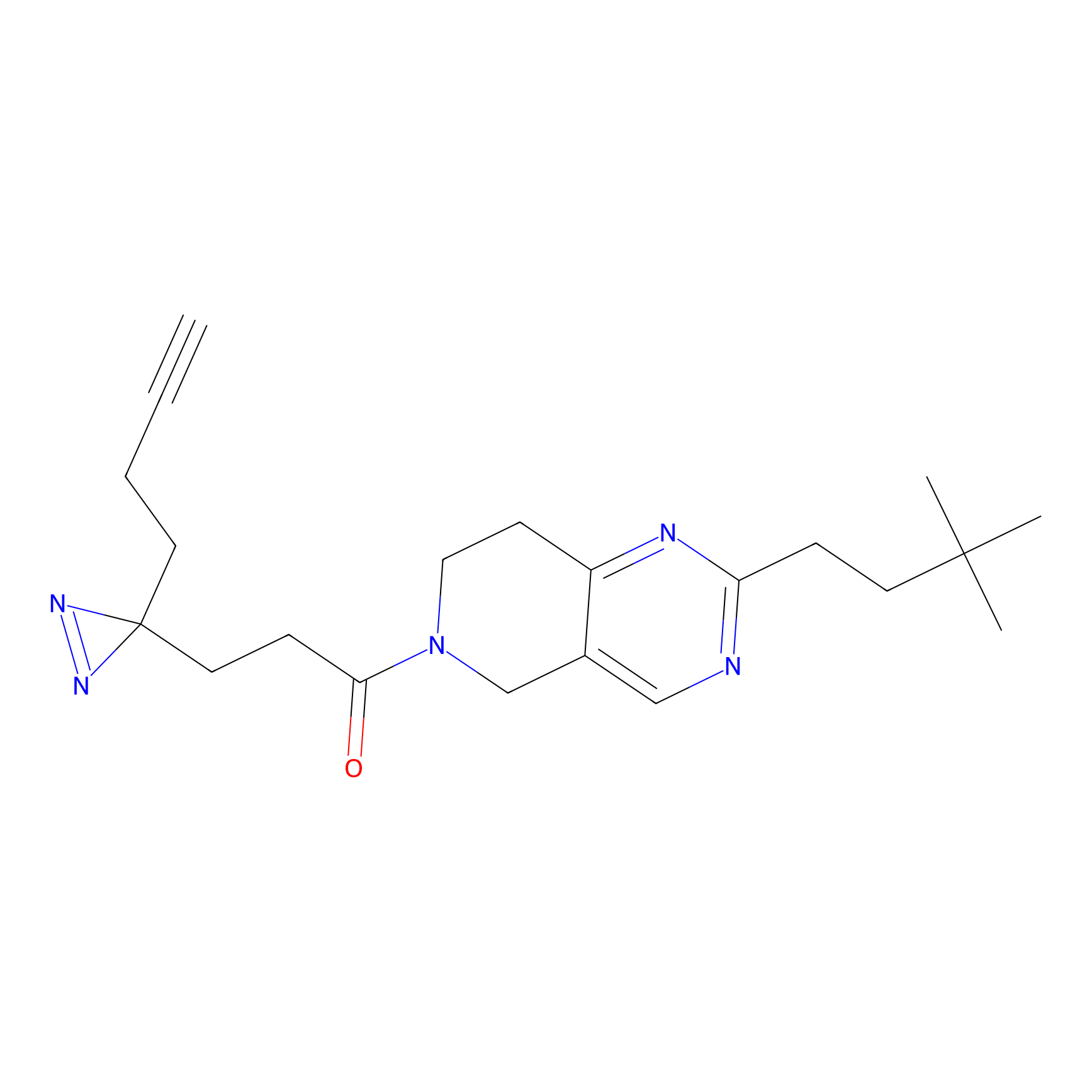
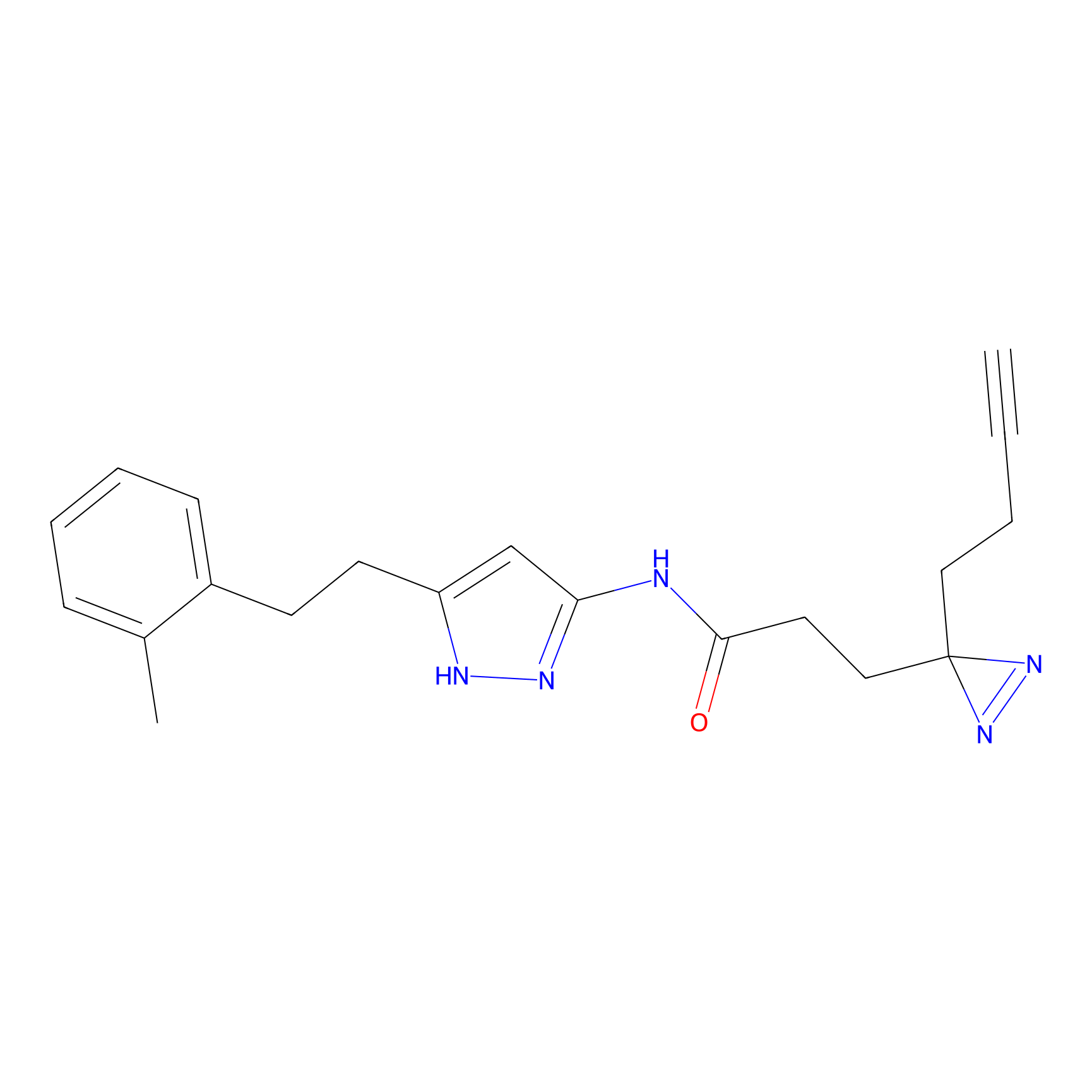
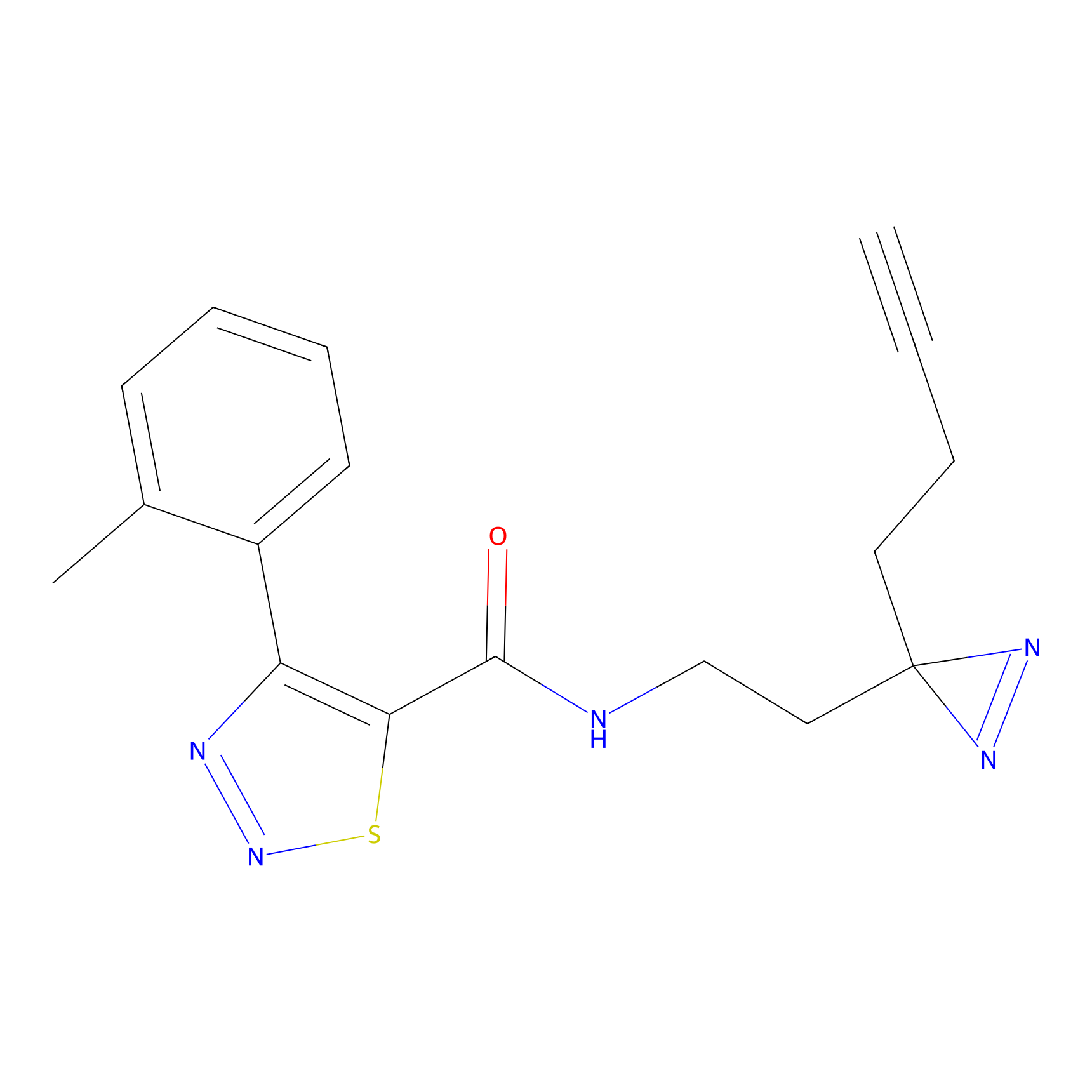
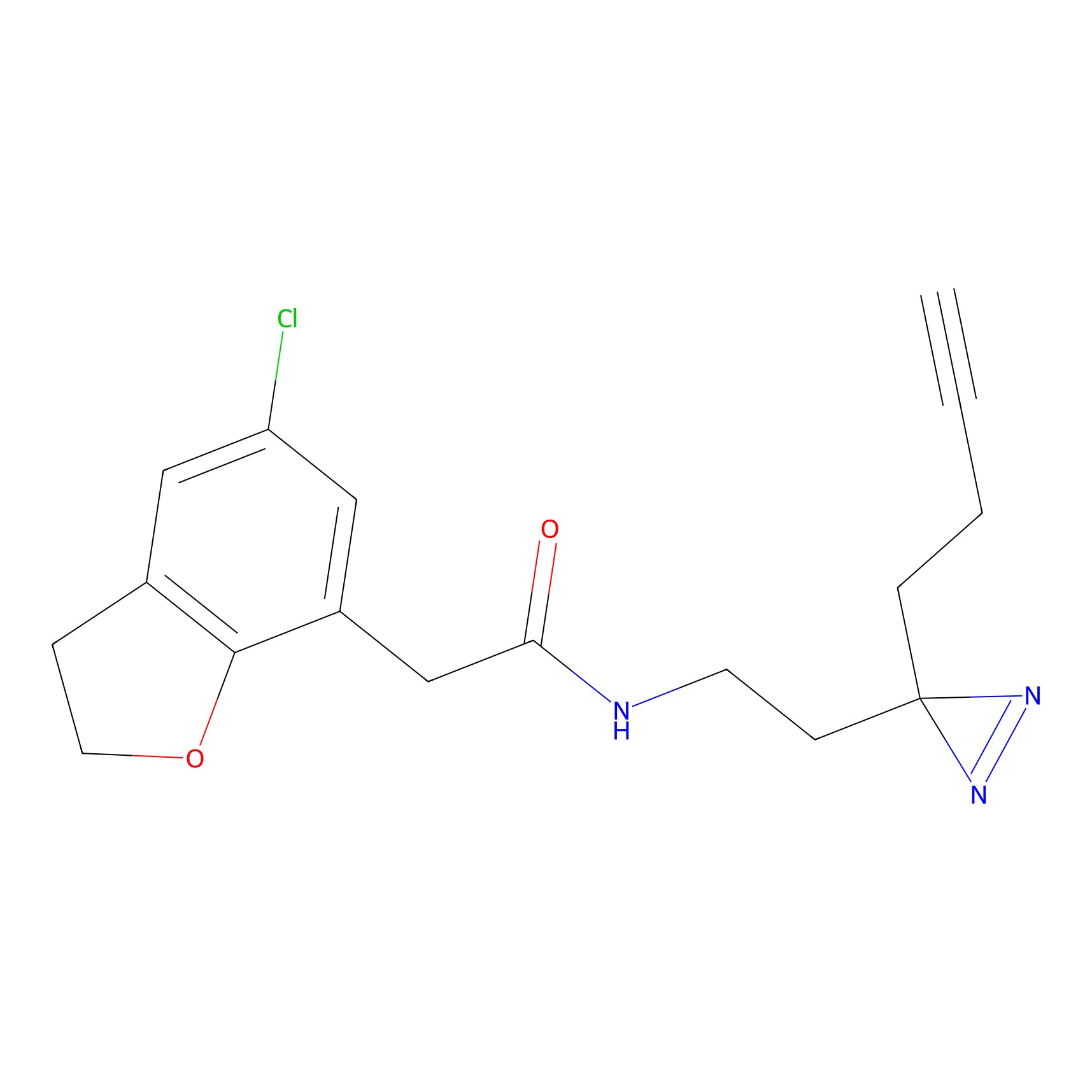
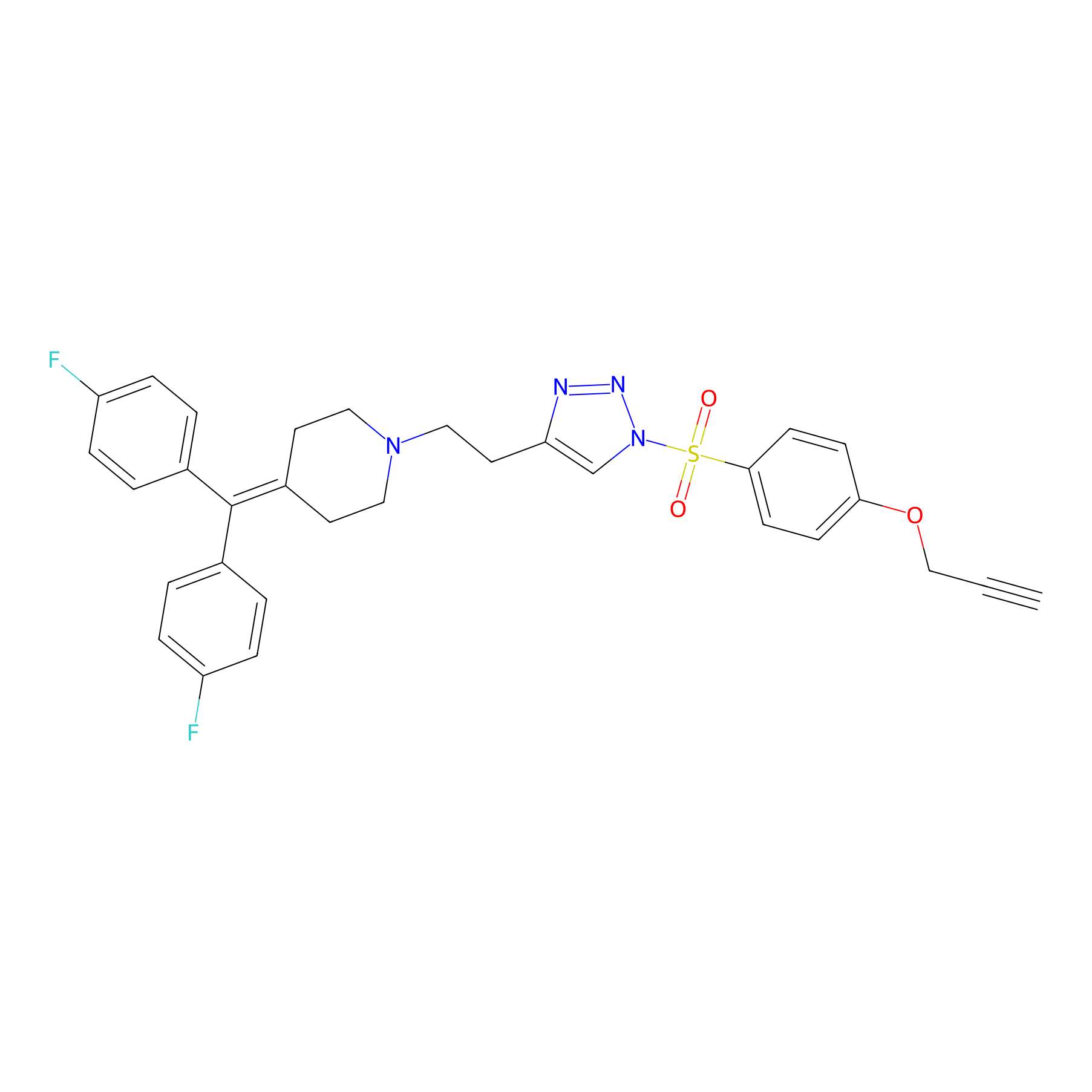
TH216 Probe Info |
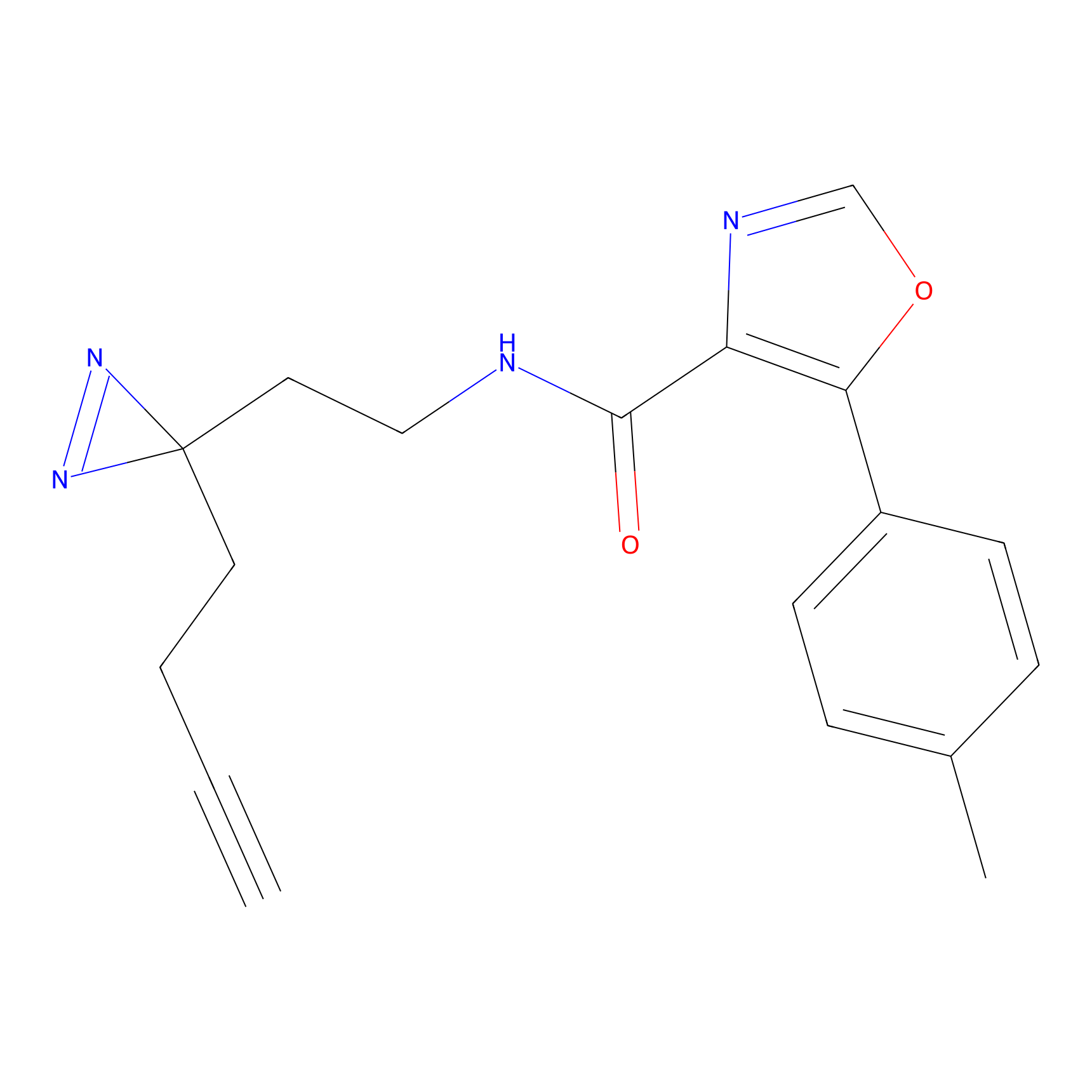
 |
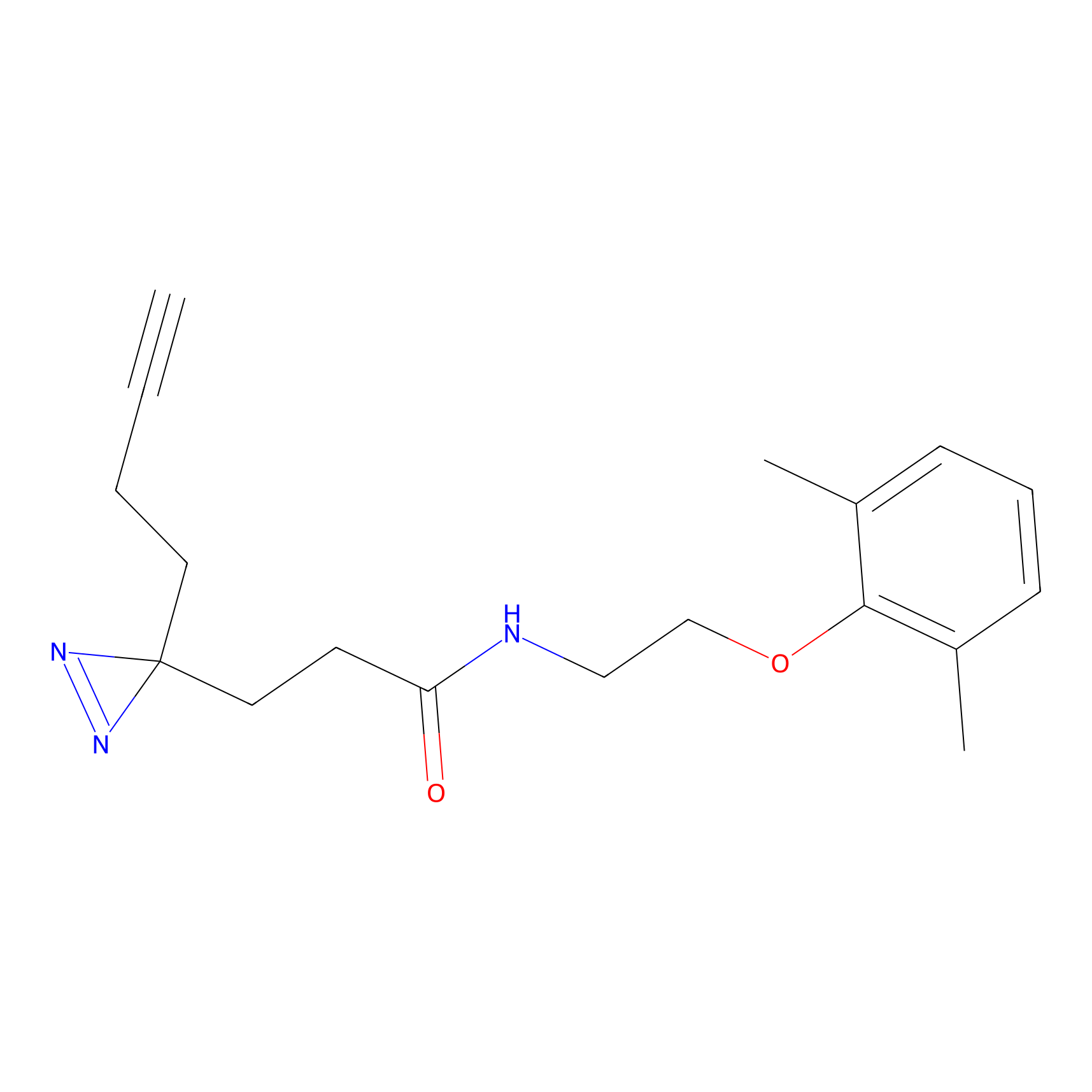
Y169(20.00) | LDD0259 | [3] | |
|
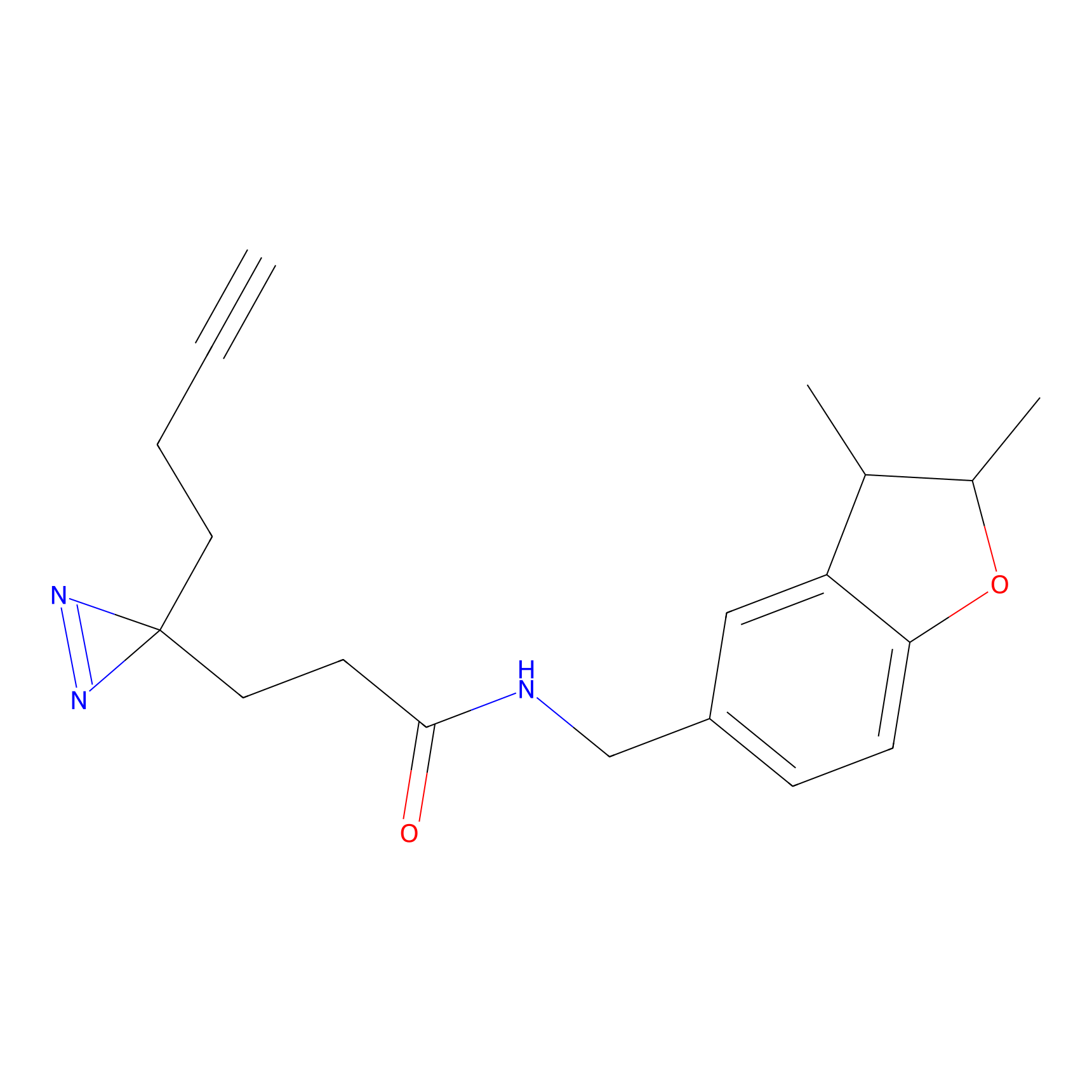
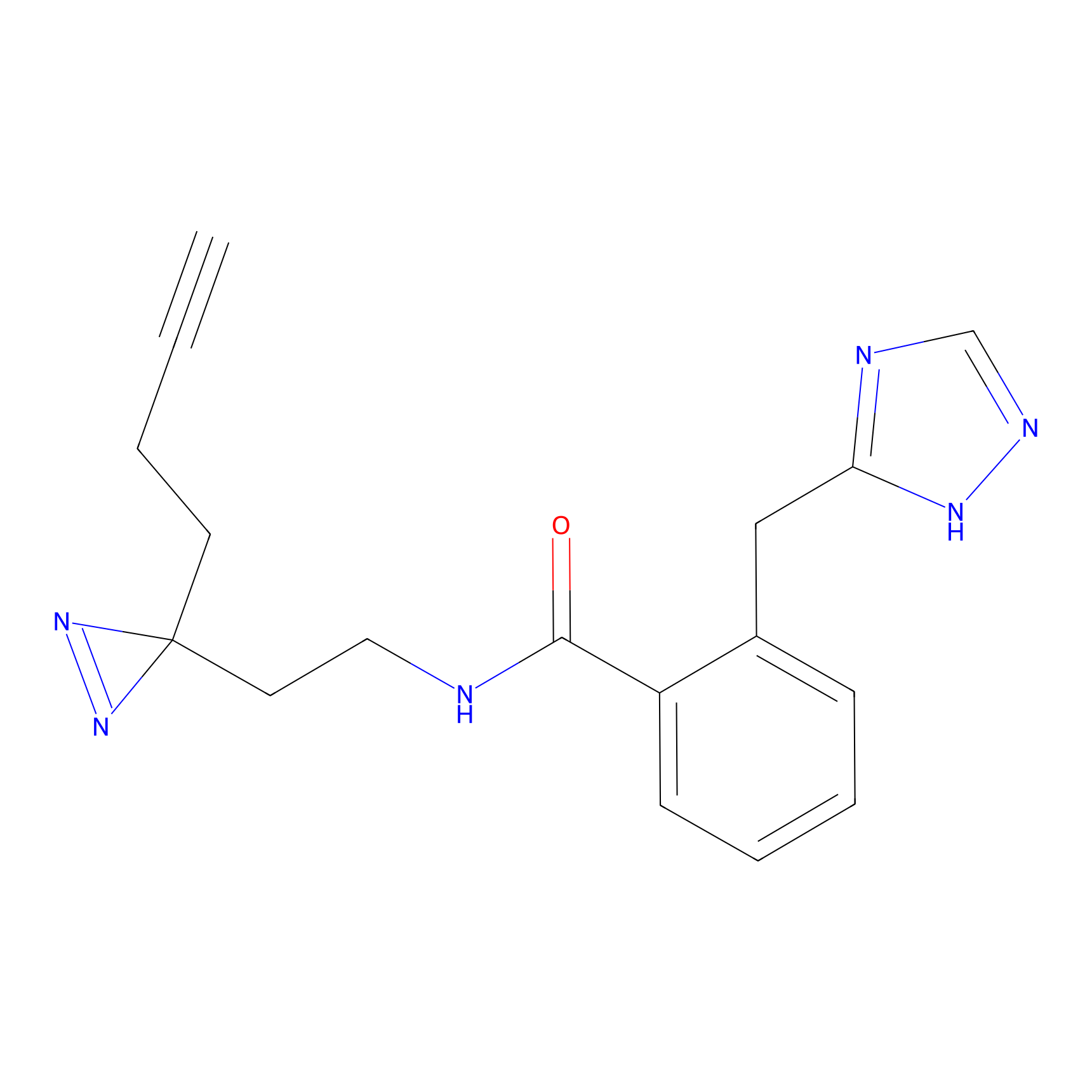
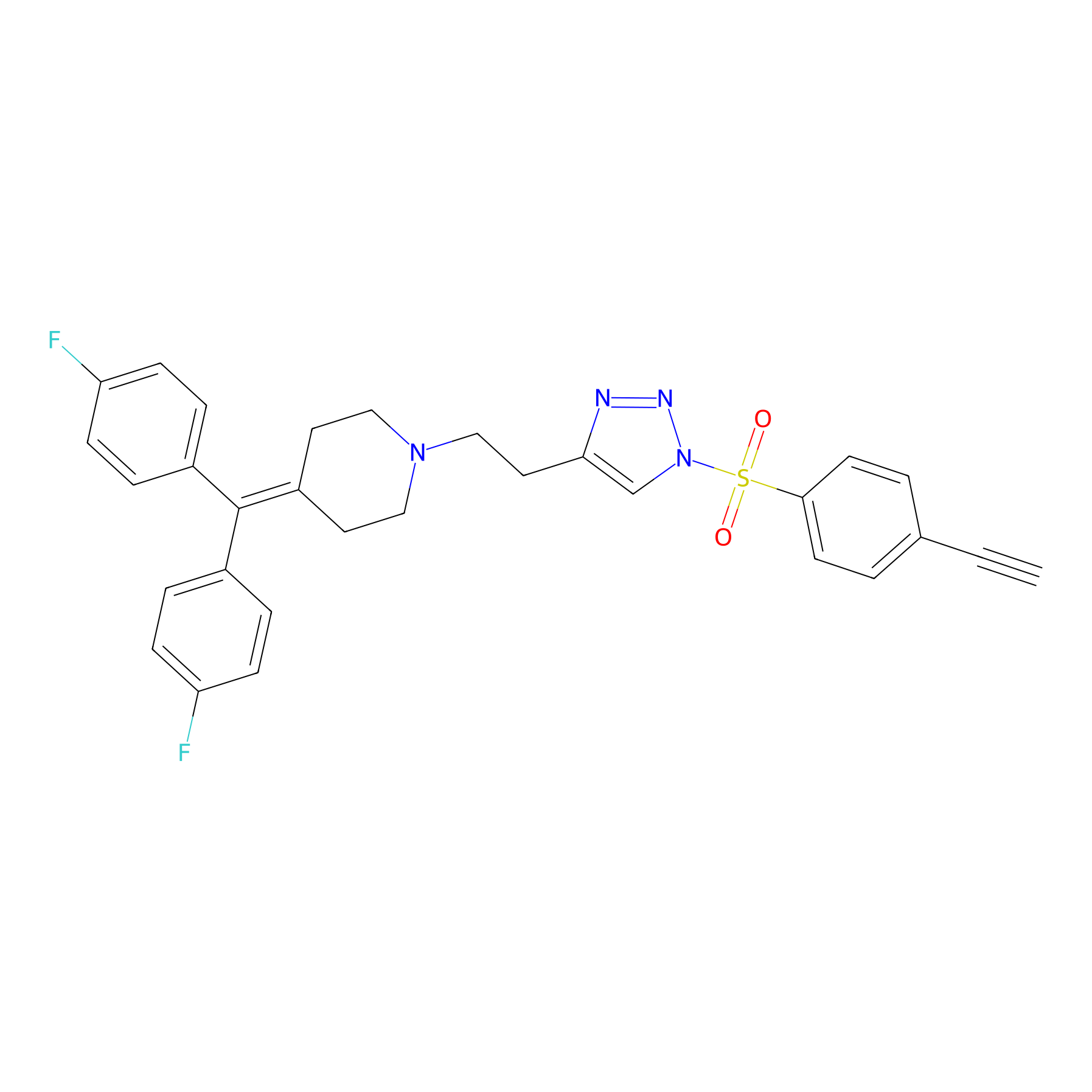
ONAyne Probe Info |
 |
K171(10.00); K69(7.14) | LDD0274 | [4] | |
|
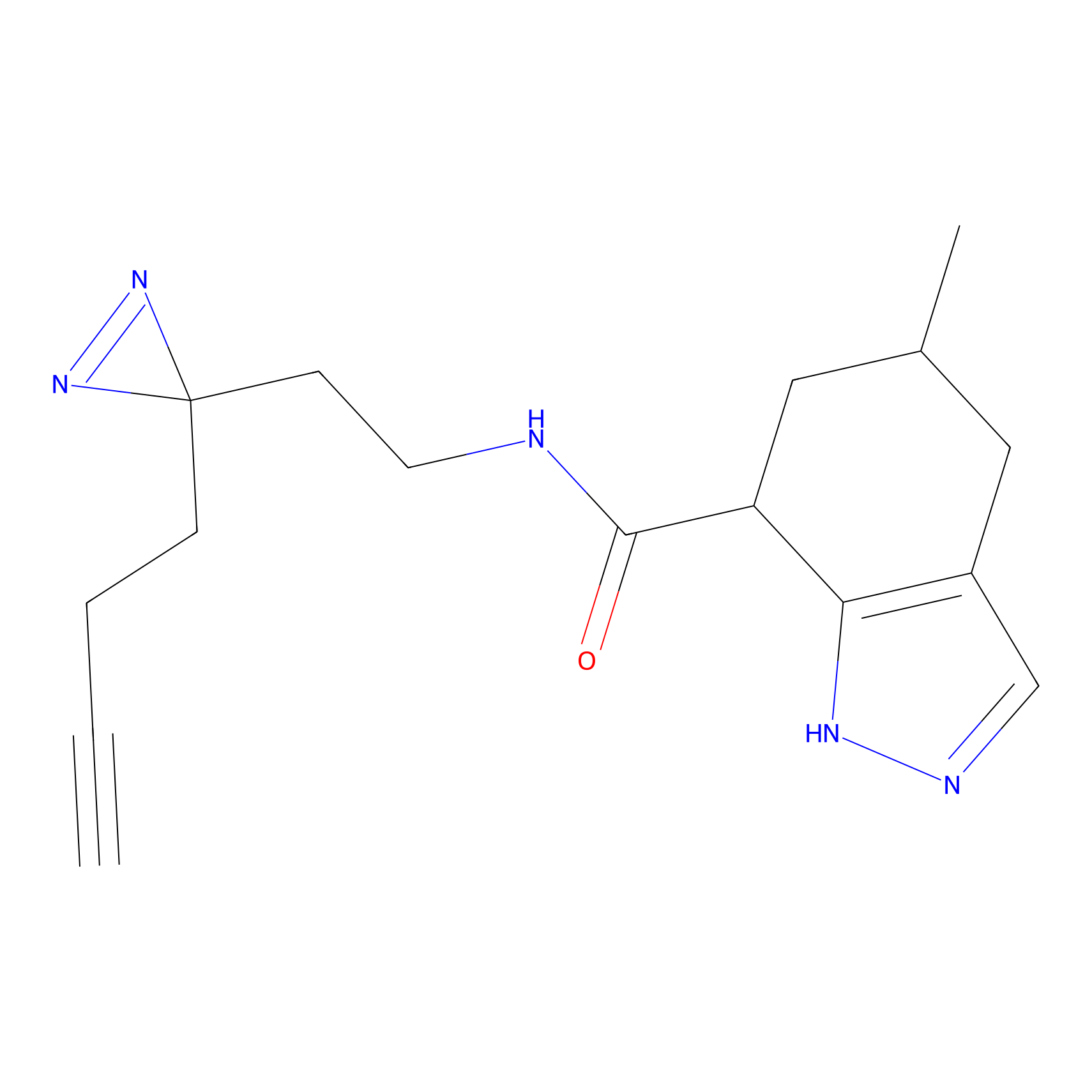
OPA-S-S-alkyne Probe Info |
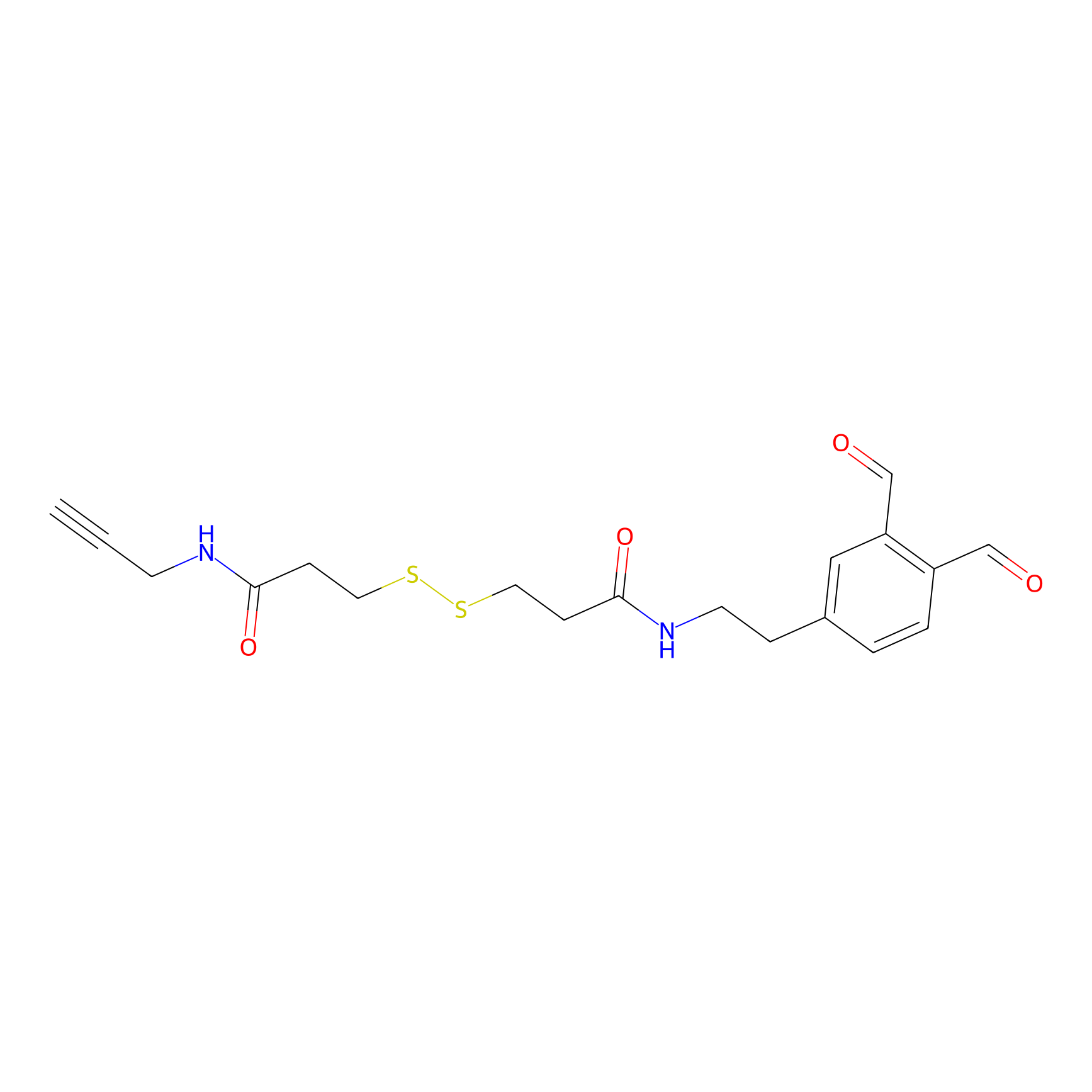 |
K84(1.63); K171(2.34); K184(2.65) | LDD3494 | [5] | |
|
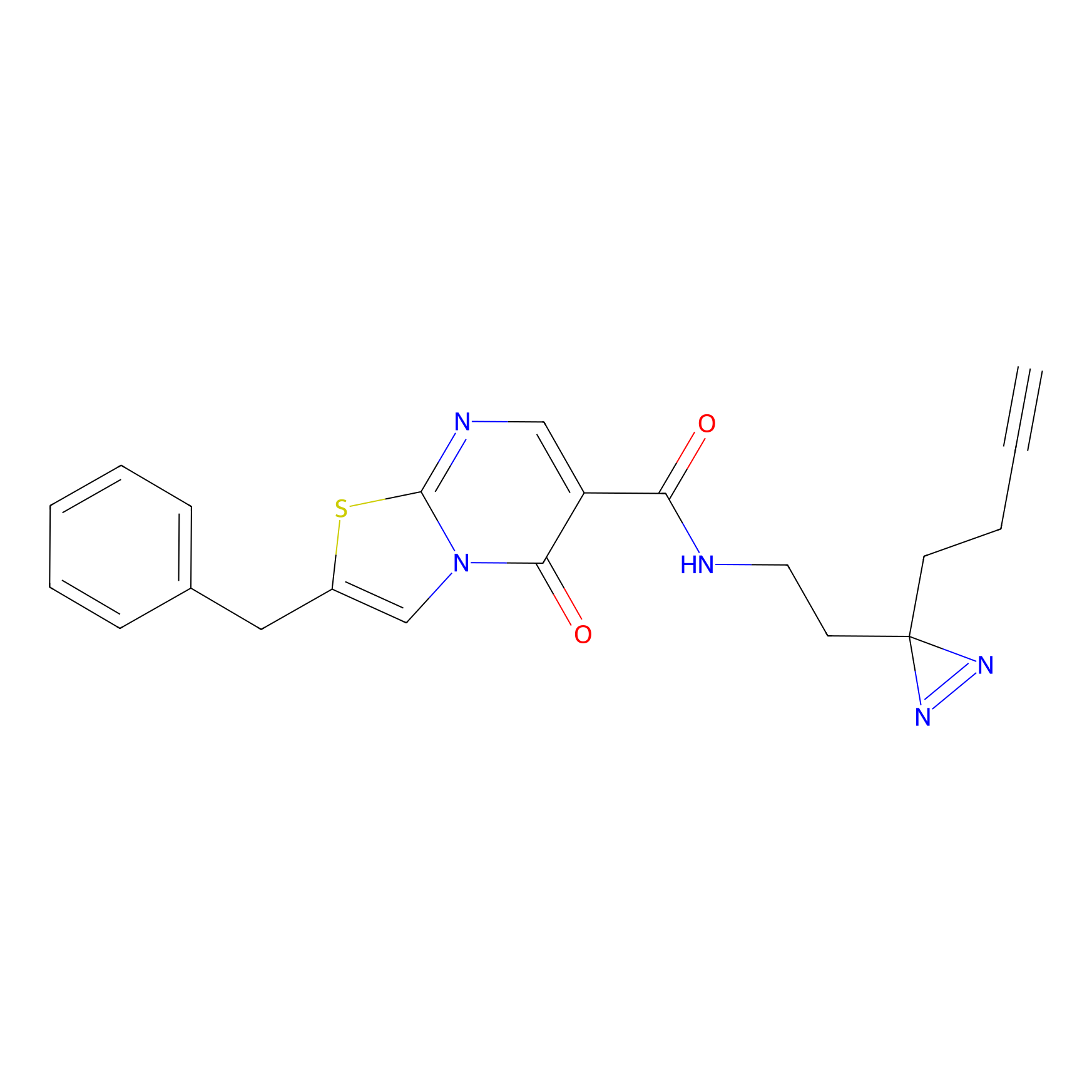
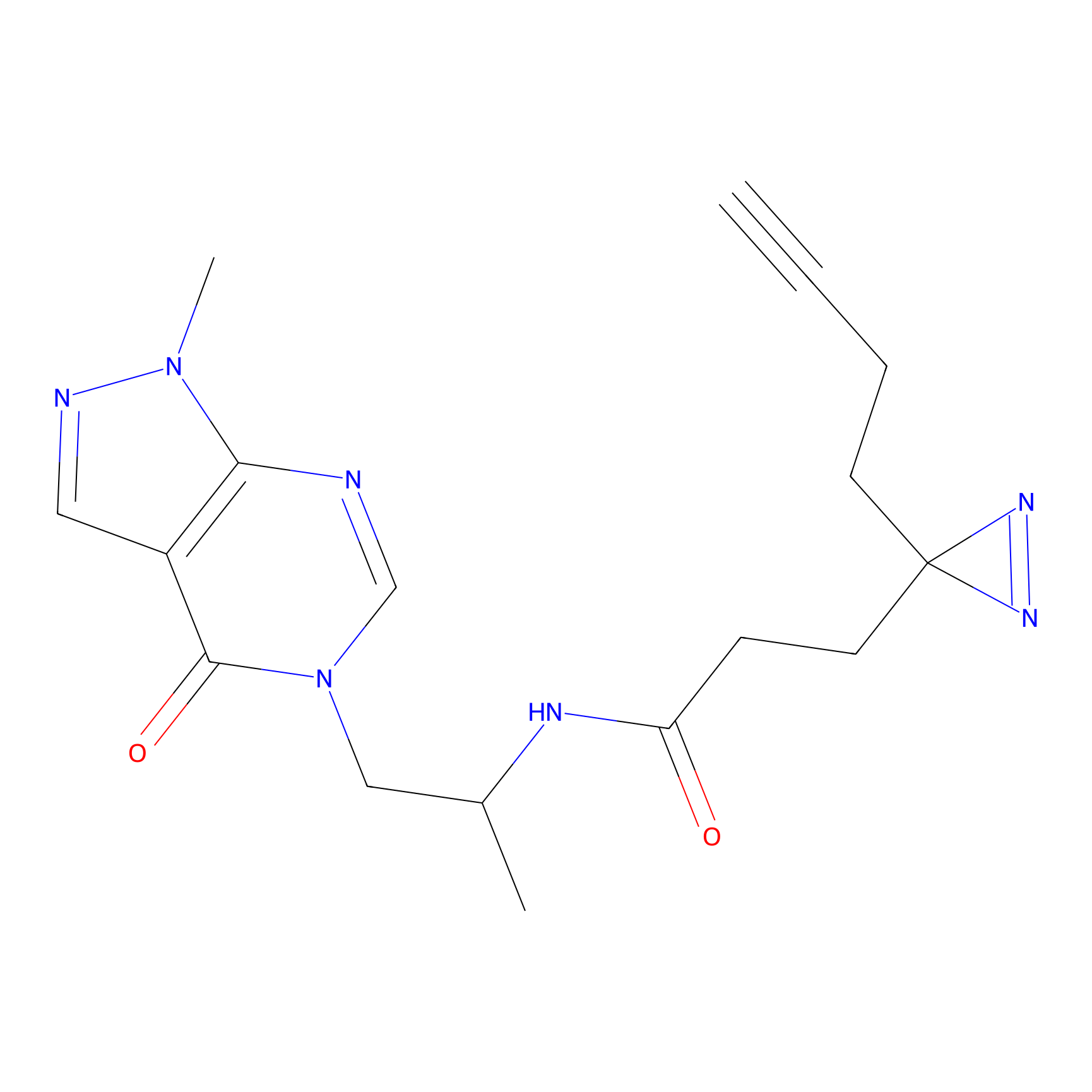
Probe 1 Probe Info |
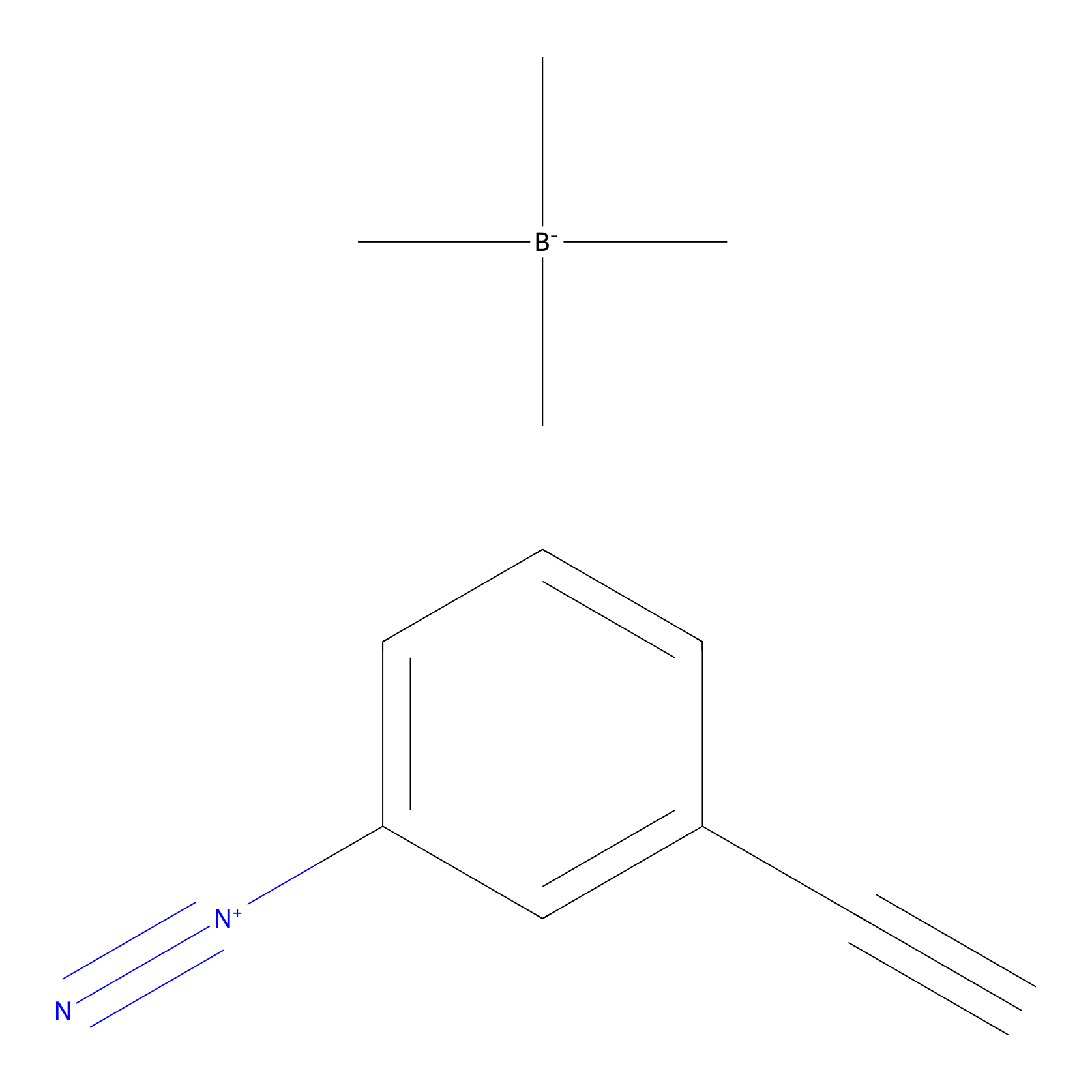 |
Y54(13.68); Y169(20.12); Y389(11.08) | LDD3495 | [6] | |
|
5E-2FA Probe Info |
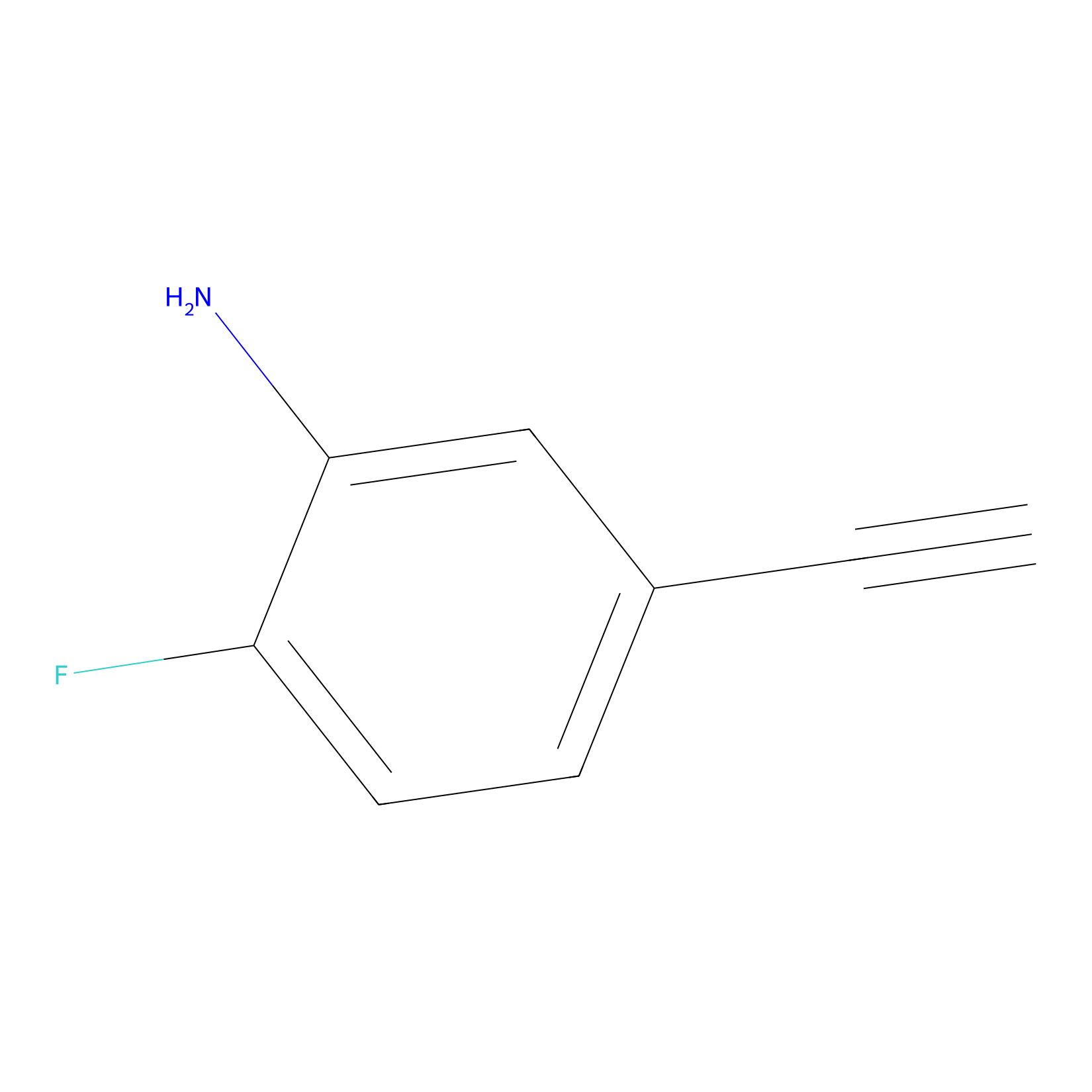 |
H132(0.00); H390(0.00); H300(0.00); H379(0.00) | LDD2235 | [7] | |
|
ATP probe Probe Info |
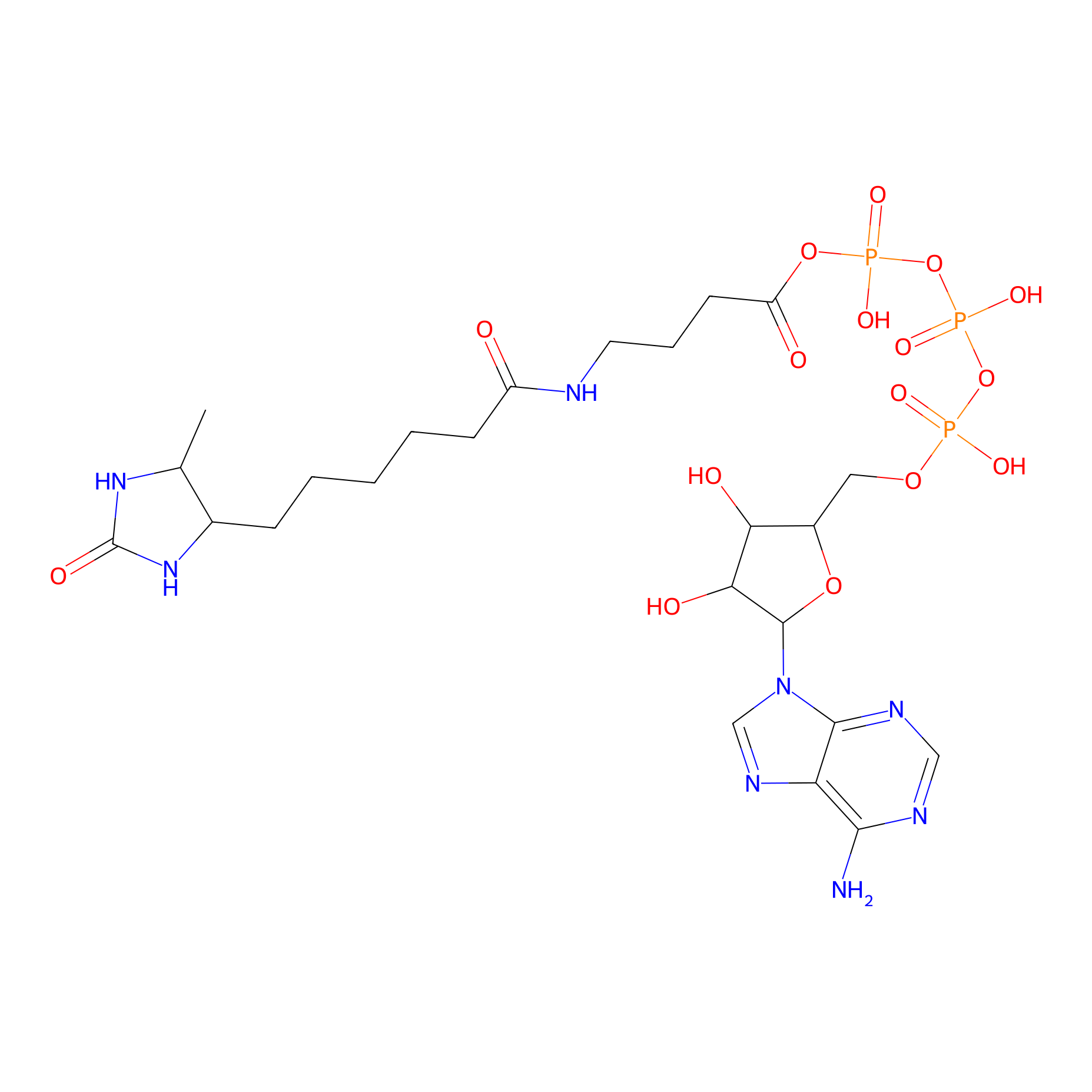 |
K399(0.00); K400(0.00); K226(0.00); K230(0.00) | LDD0199 | [8] | |
|
m-APA Probe Info |
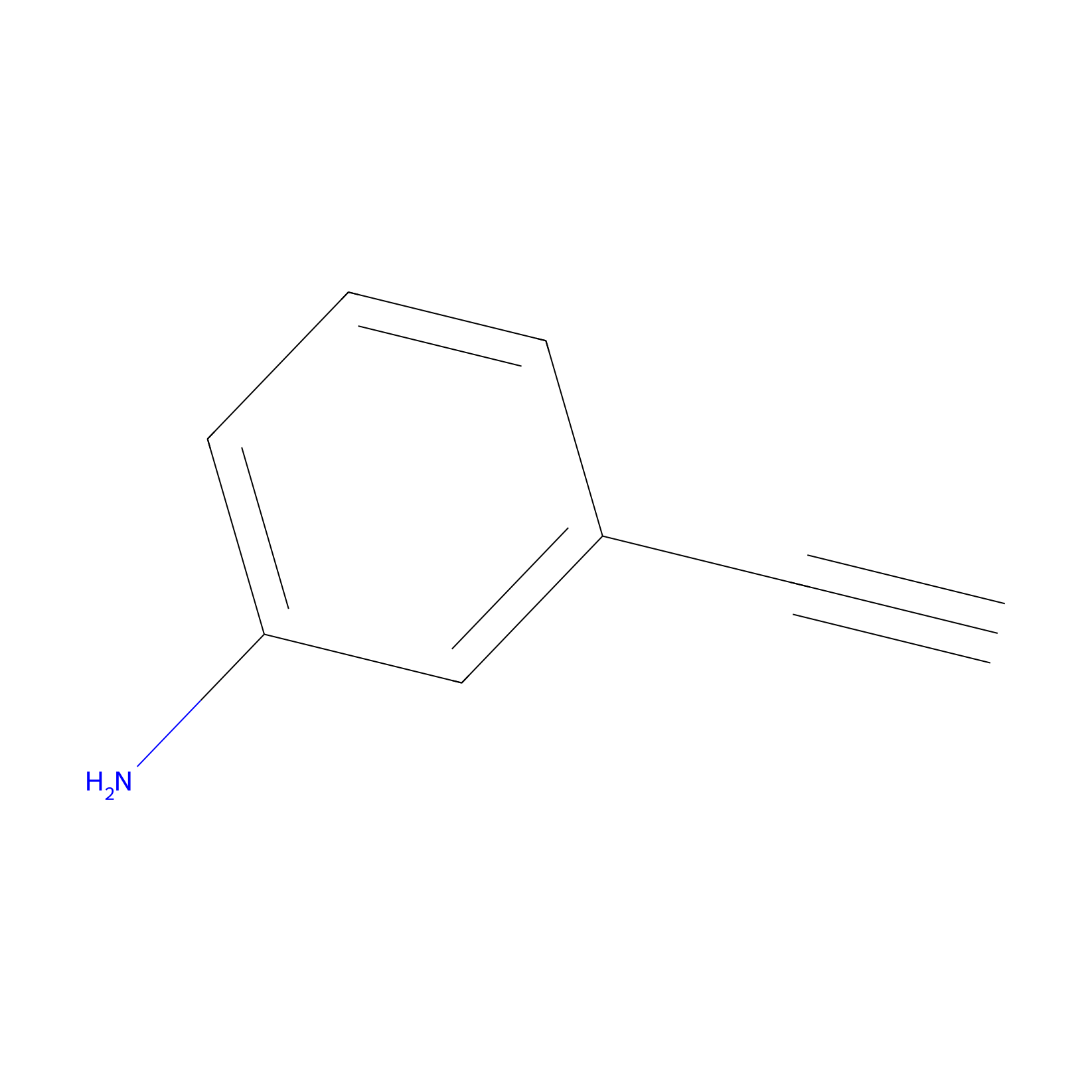 |
H300(0.00); H132(0.00); H390(0.00); H253(0.00) | LDD2231 | [7] | |
|
STPyne Probe Info |
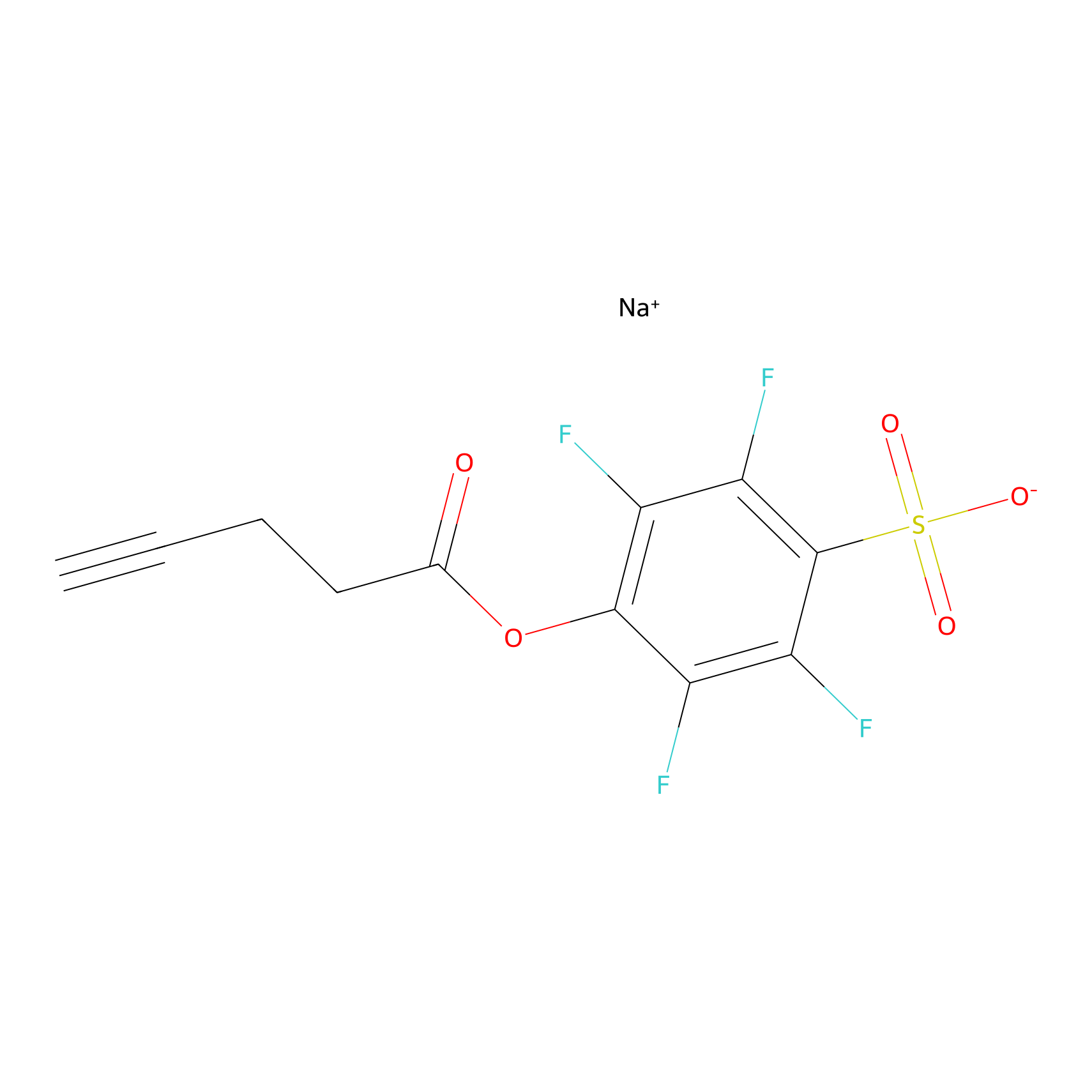 |
N.A. | LDD0009 | [9] | |
|
1c-yne Probe Info |
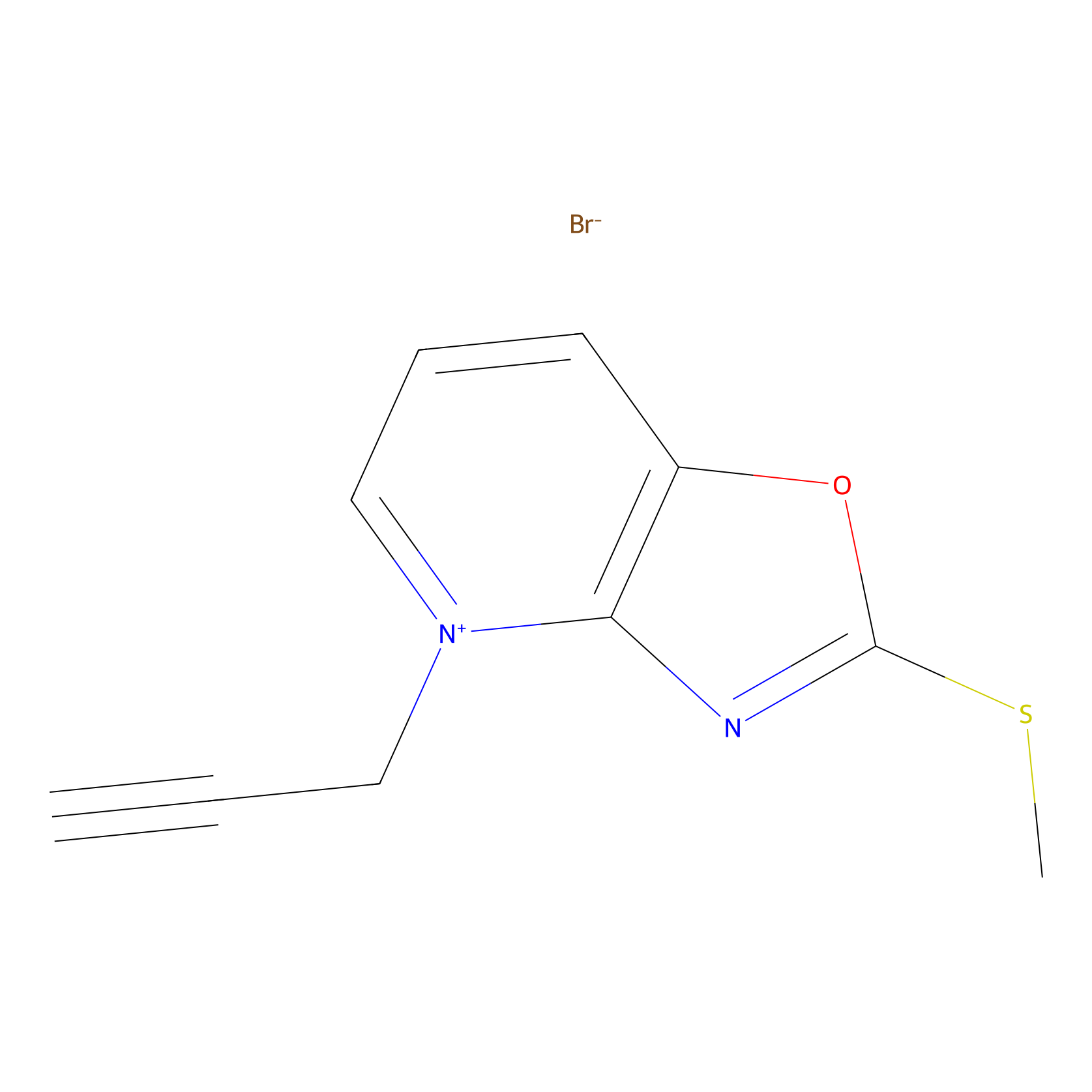 |
N.A. | LDD0228 | [10] | |
|
Acrolein Probe Info |
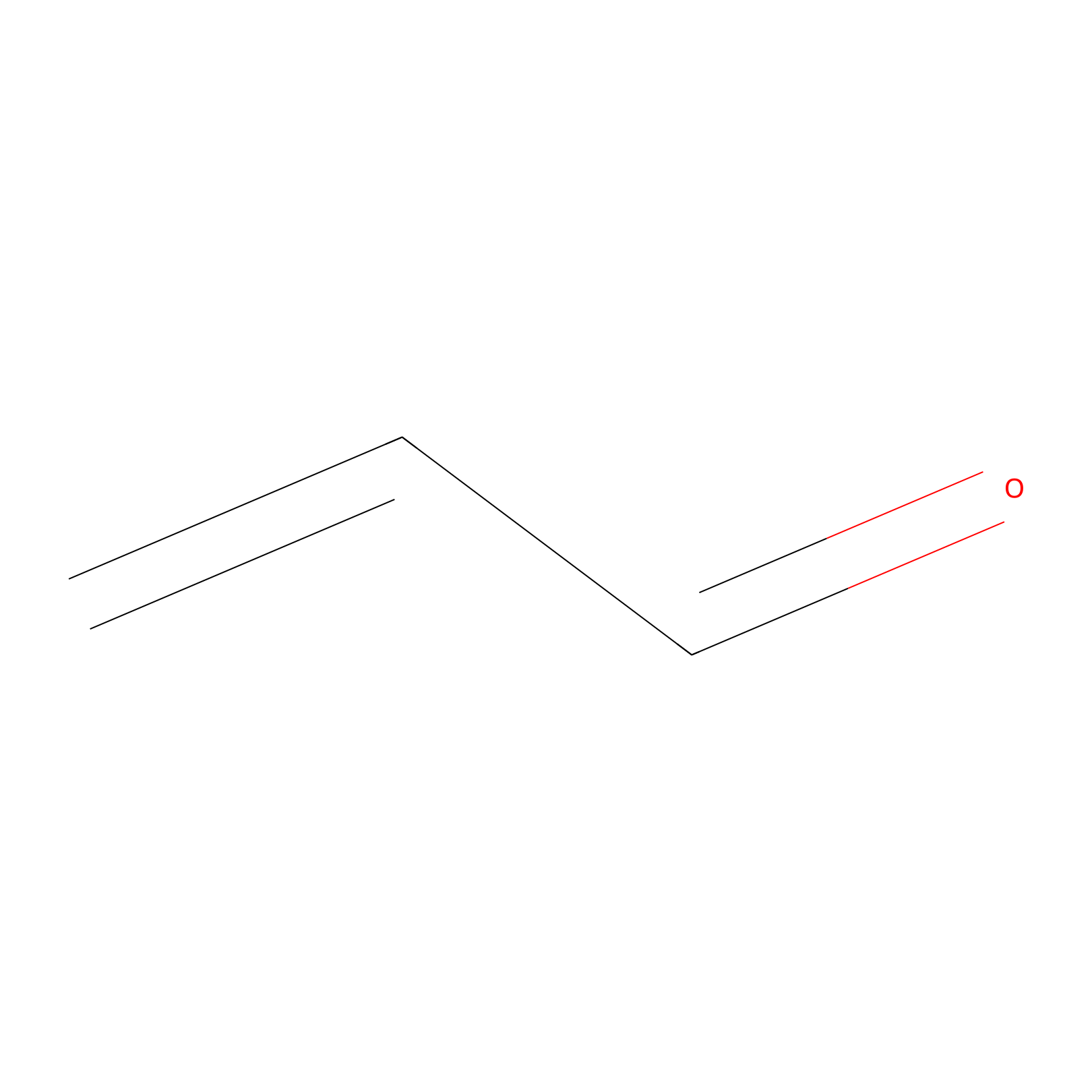 |
N.A. | LDD0217 | [11] | |
|
W1 Probe Info |
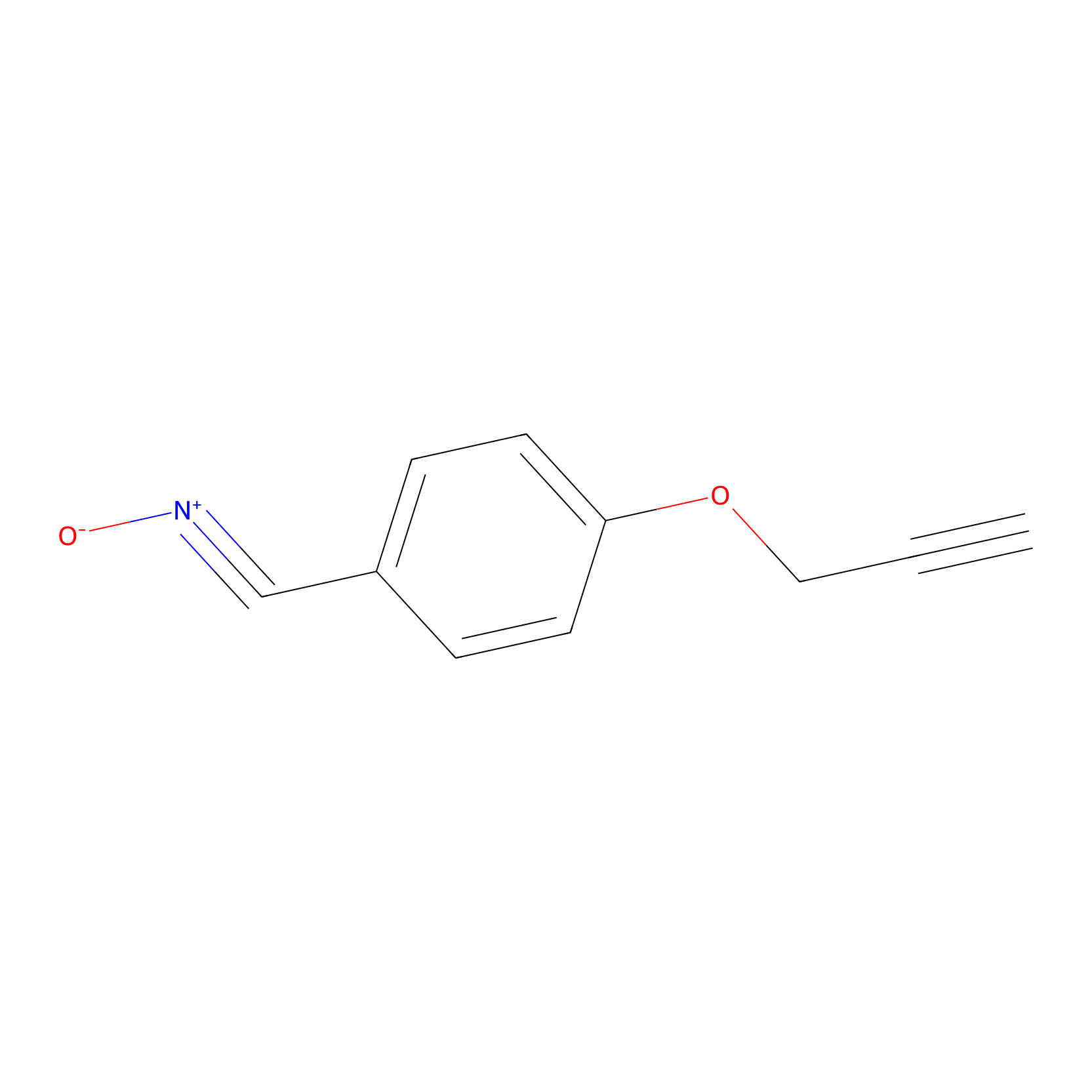 |
E299(0.00); N303(0.00); H300(0.00) | LDD0236 | [12] | |
PAL-AfBPP Probe
Competitor(s) Related to This Target
| Competitor ID | Name | Cell line | Binding Site(Ratio) | Interaction ID | Ref |
|---|---|---|---|---|---|
| LDCM0108 | Chloroacetamide | HeLa | H300(0.00); H38(0.00) | LDD0222 | [11] |
| LDCM0185 | Compound 17 | HEK-293T | 3.75 | LDD0512 | [15] |
| LDCM0191 | Compound 21 | HEK-293T | 10.00 | LDD0508 | [15] |
| LDCM0190 | Compound 34 | HEK-293T | 12.35 | LDD0510 | [15] |
| LDCM0192 | Compound 35 | HEK-293T | 9.52 | LDD0509 | [15] |
| LDCM0193 | Compound 36 | HEK-293T | 12.05 | LDD0511 | [15] |
| LDCM0107 | IAA | HeLa | H300(0.00); H38(0.00) | LDD0221 | [11] |
| LDCM0109 | NEM | HeLa | H38(0.00); H168(0.00); H97(0.00); H300(0.00) | LDD0223 | [11] |
The Interaction Atlas With This Target
References