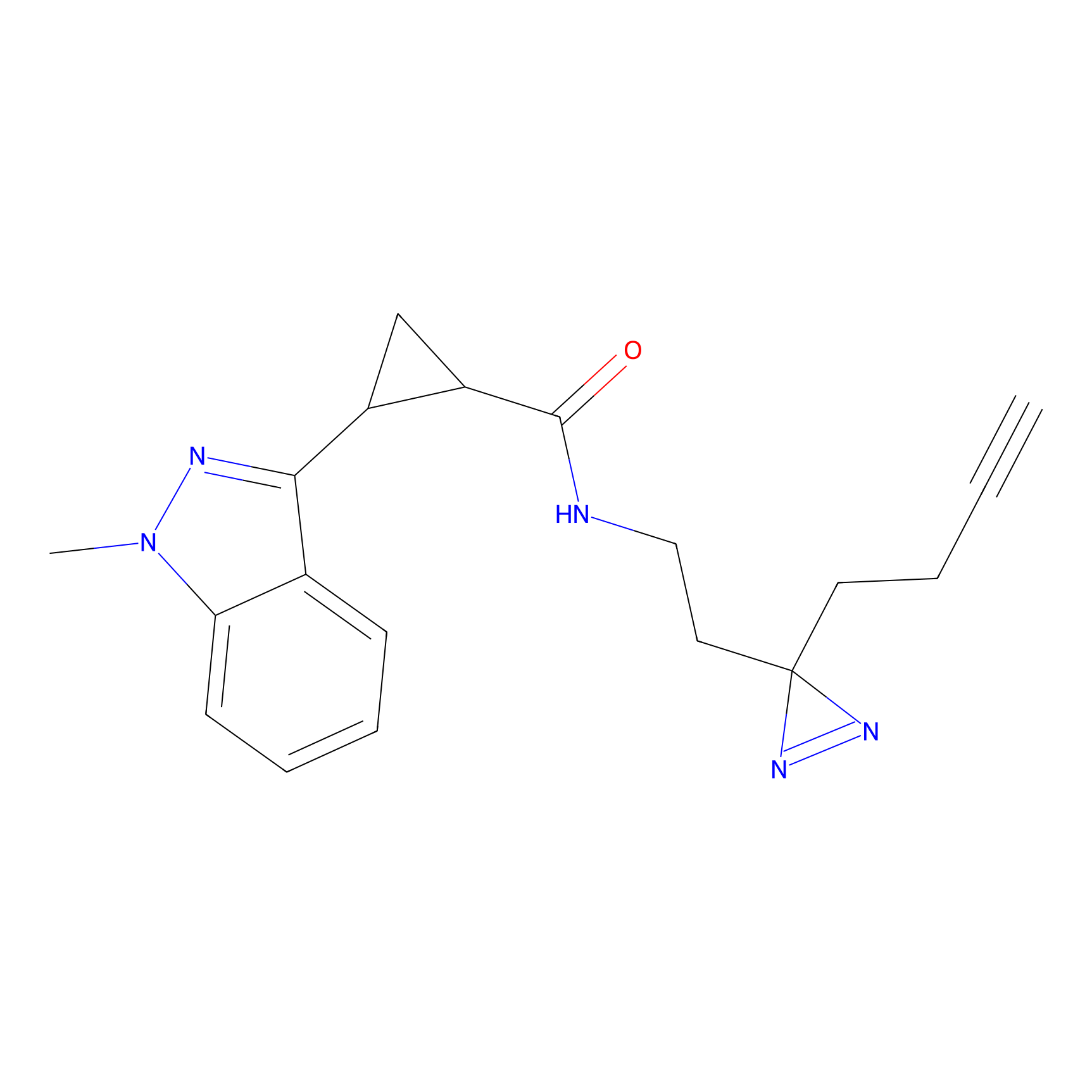
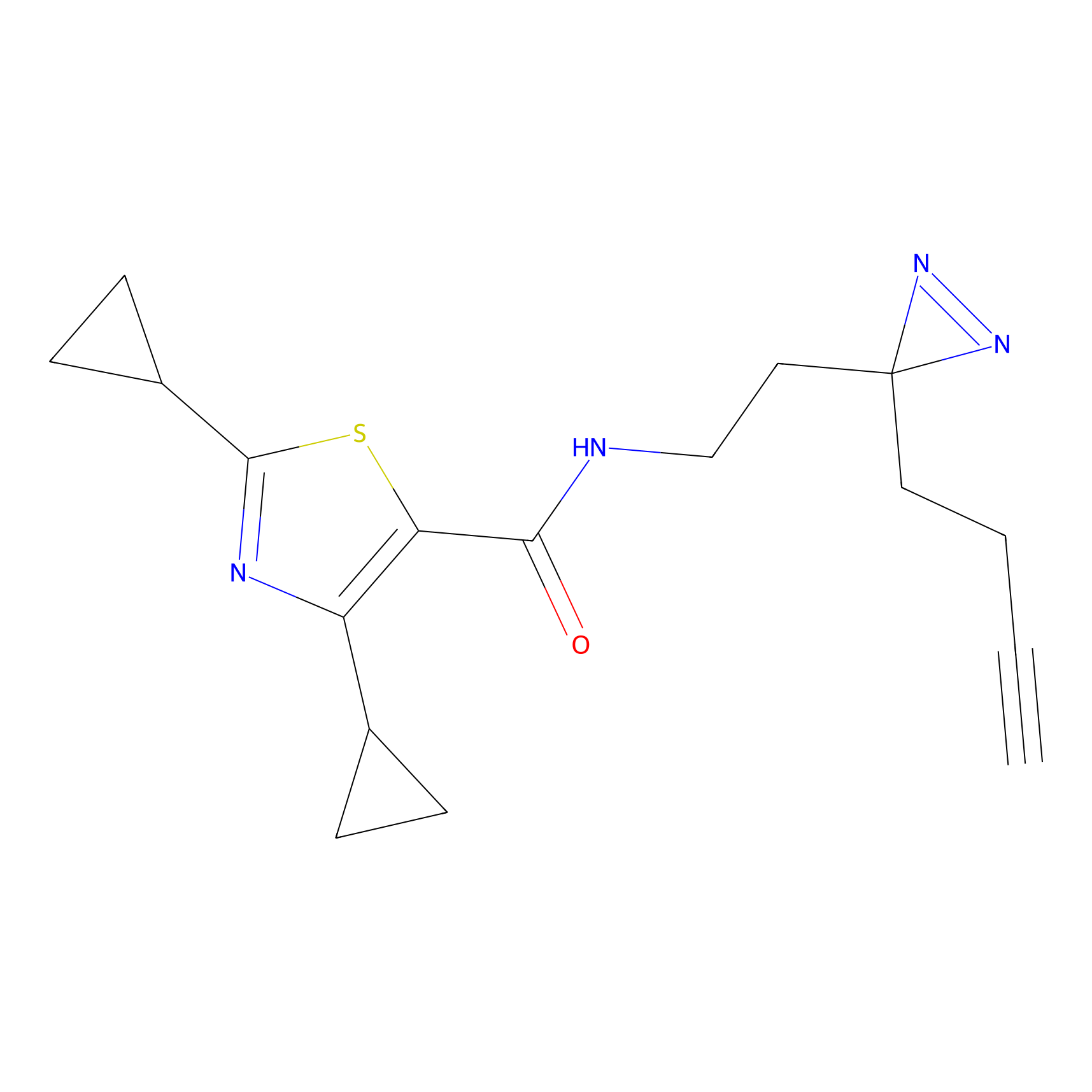
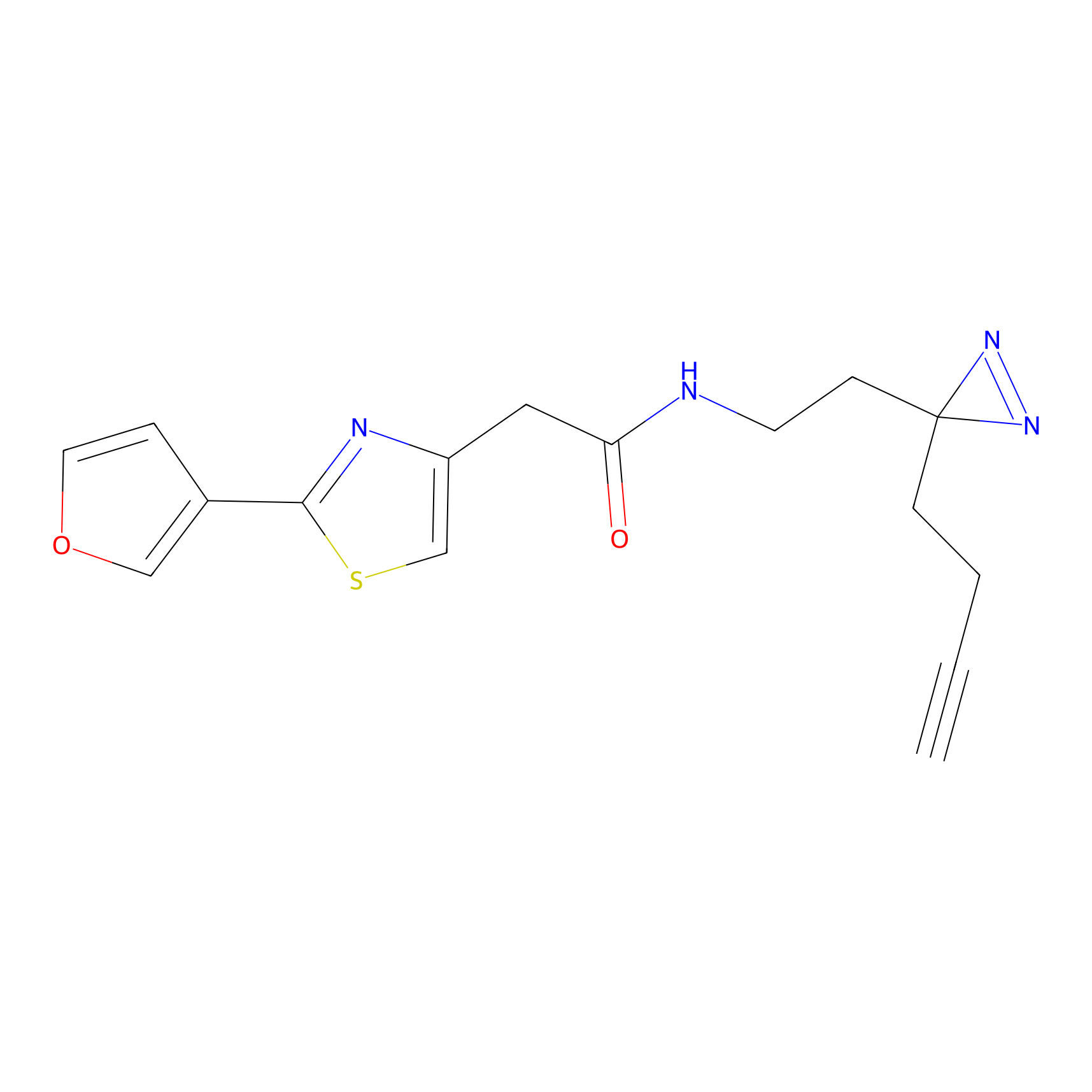
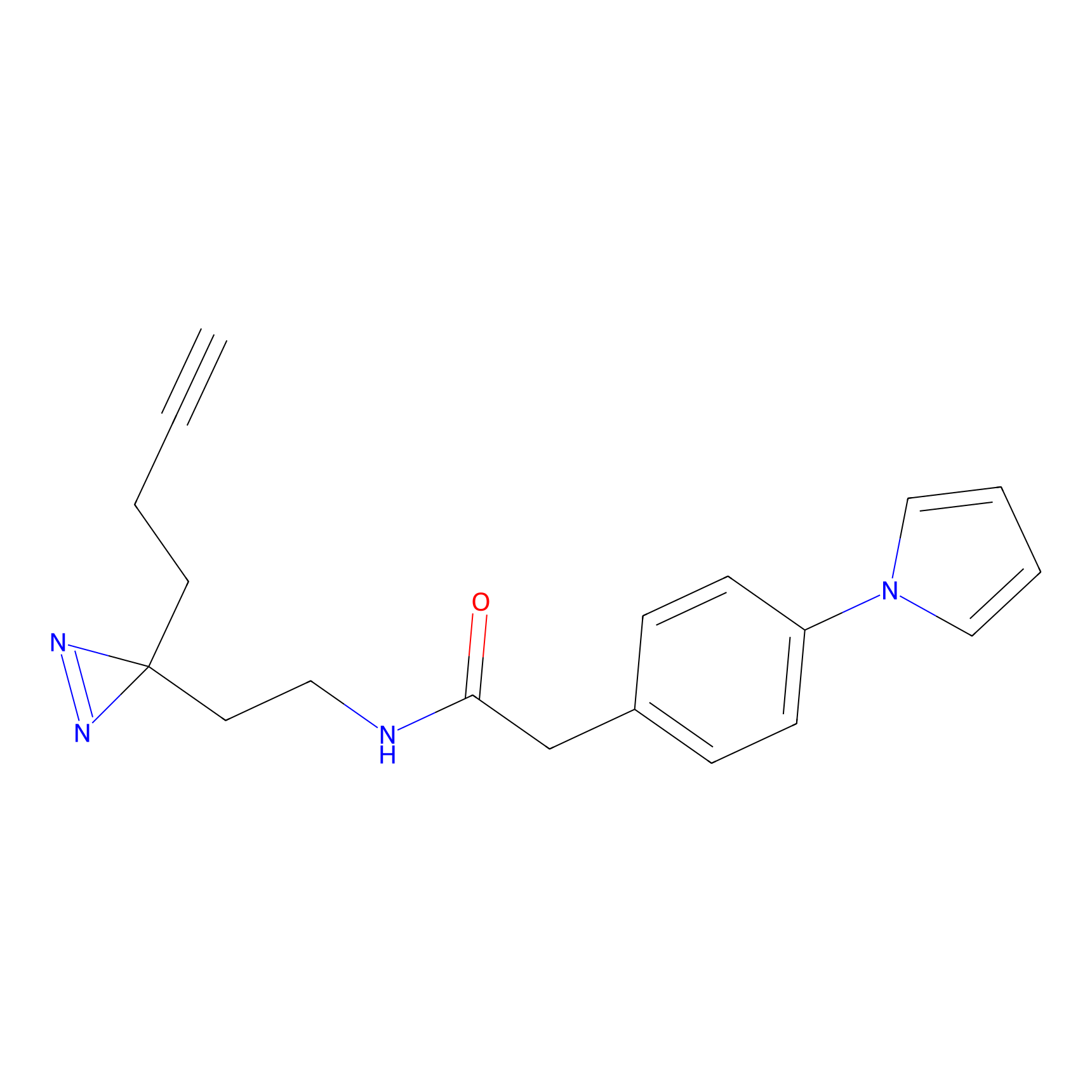
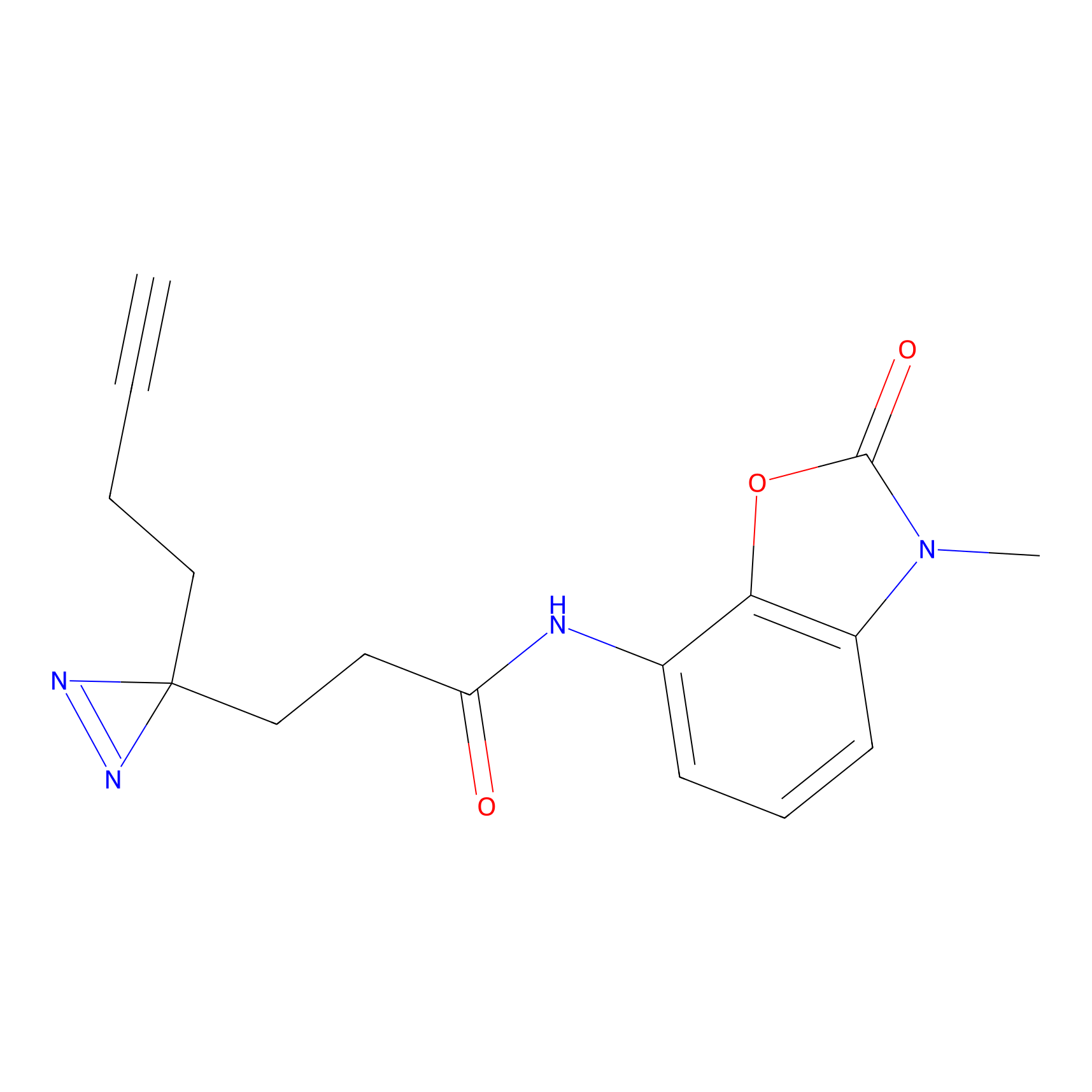
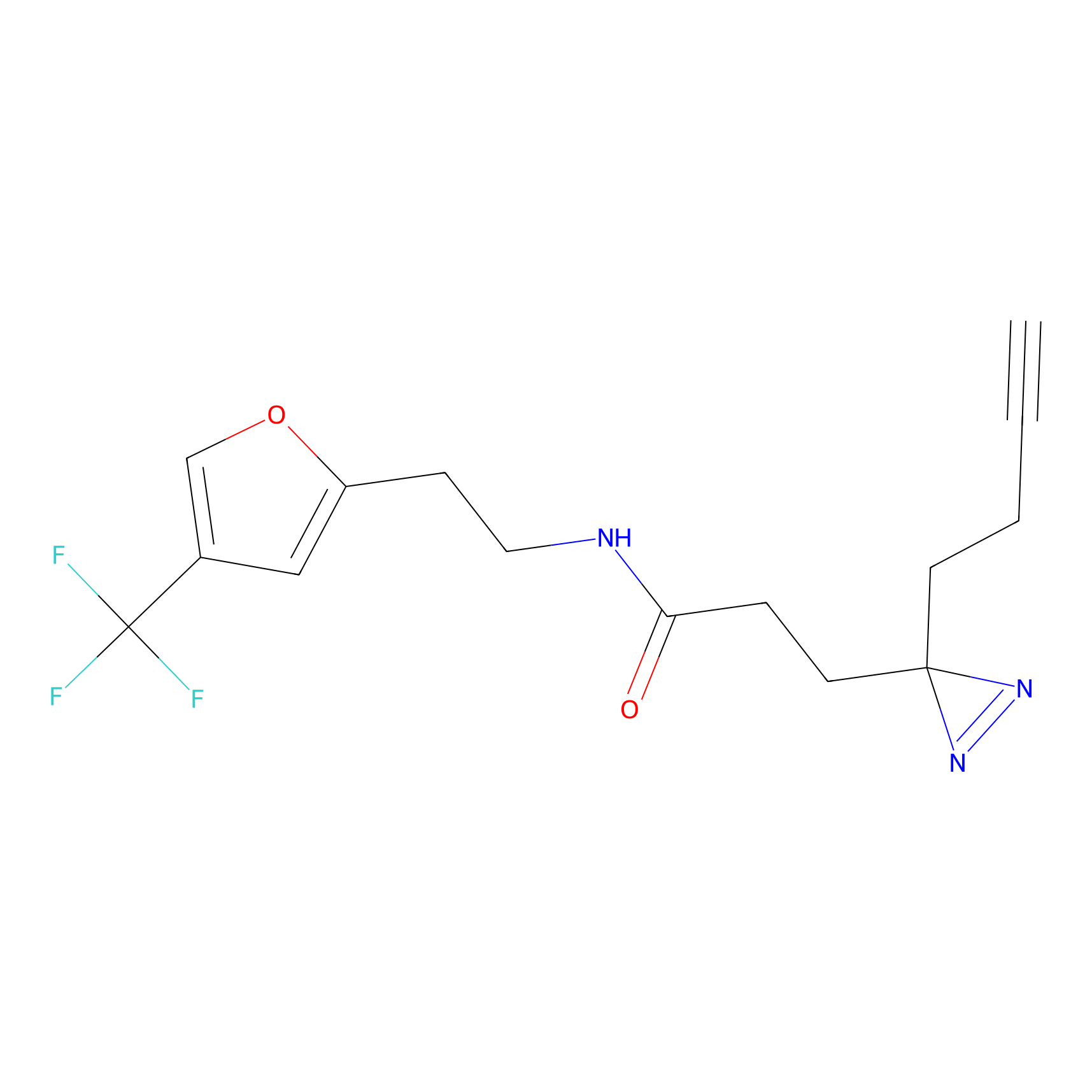
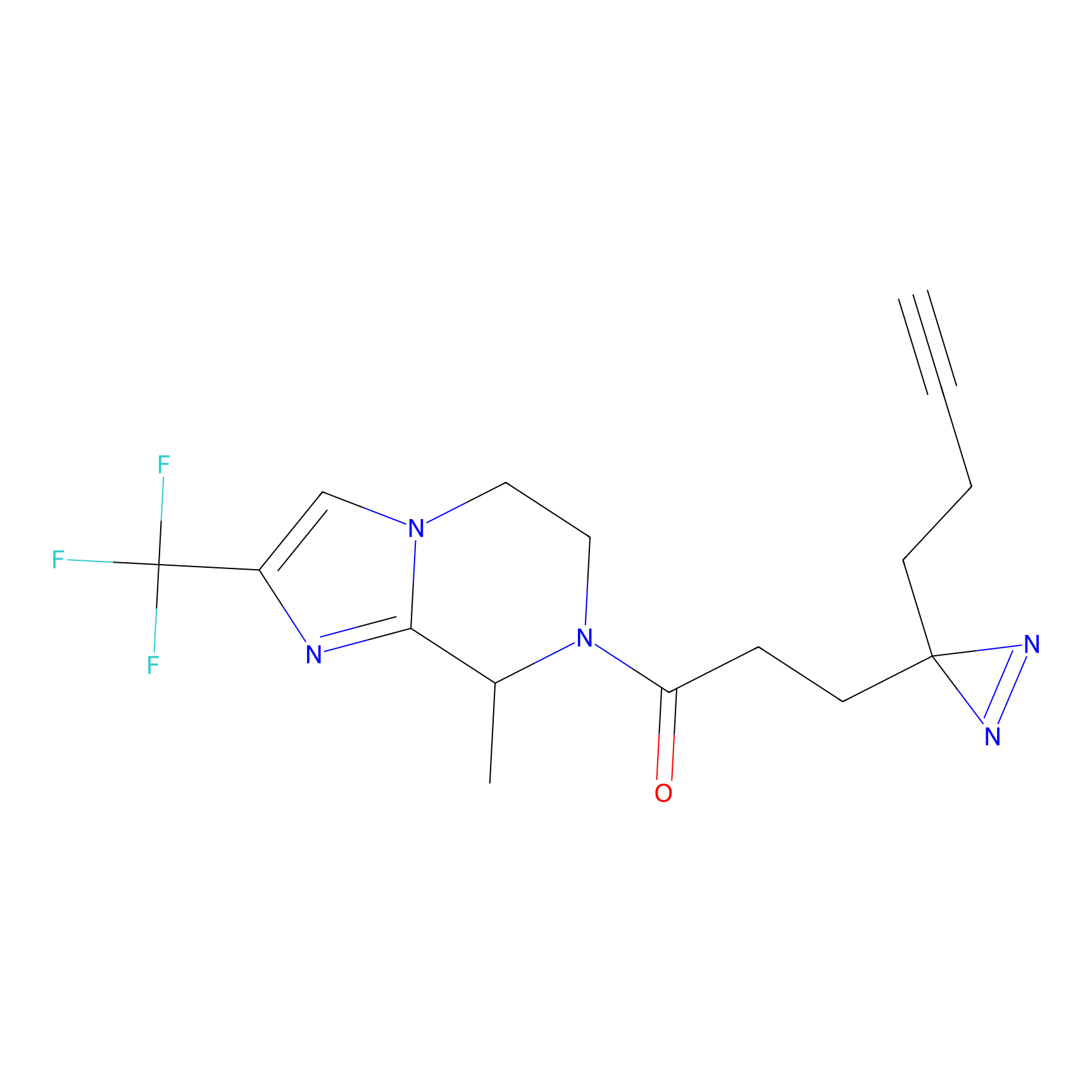
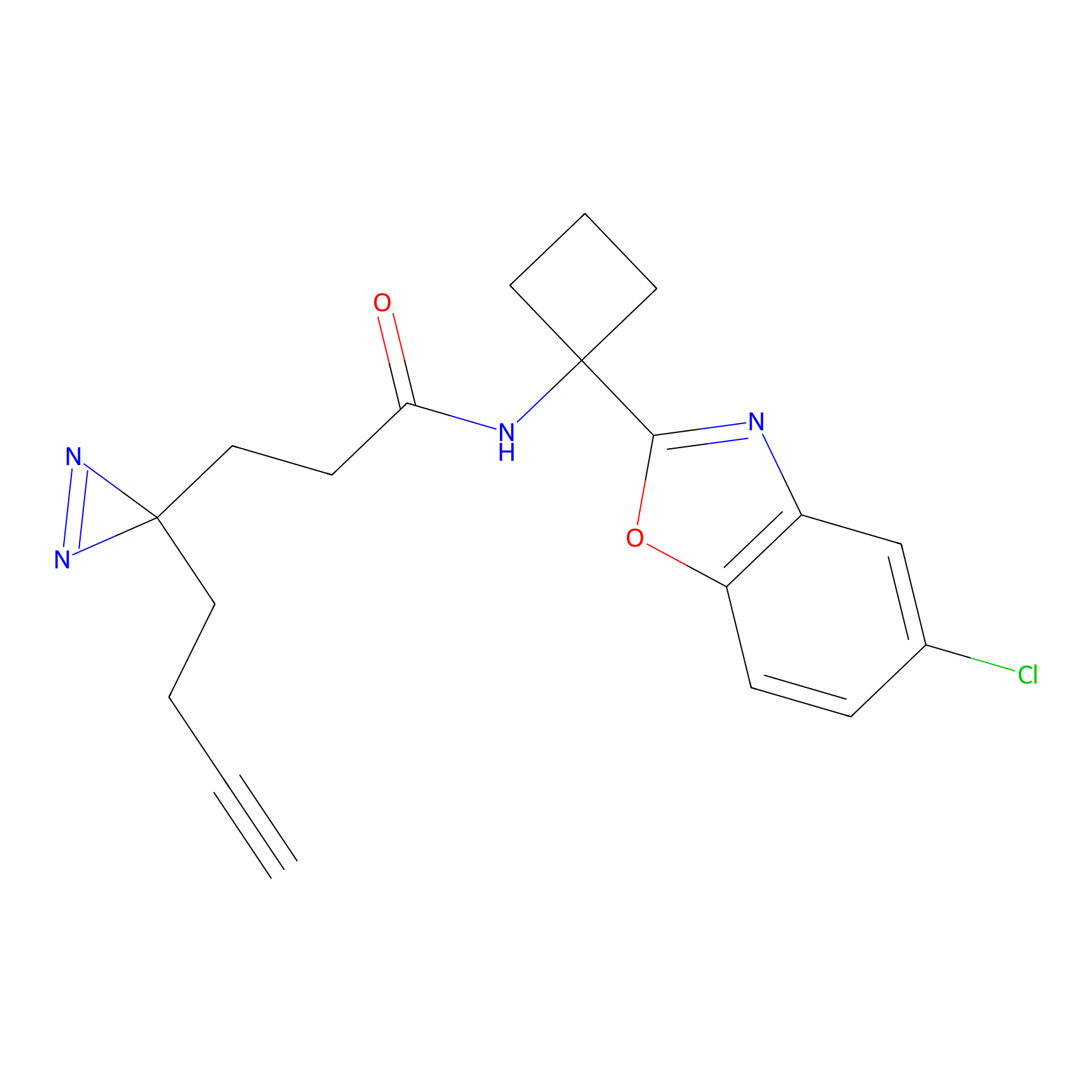
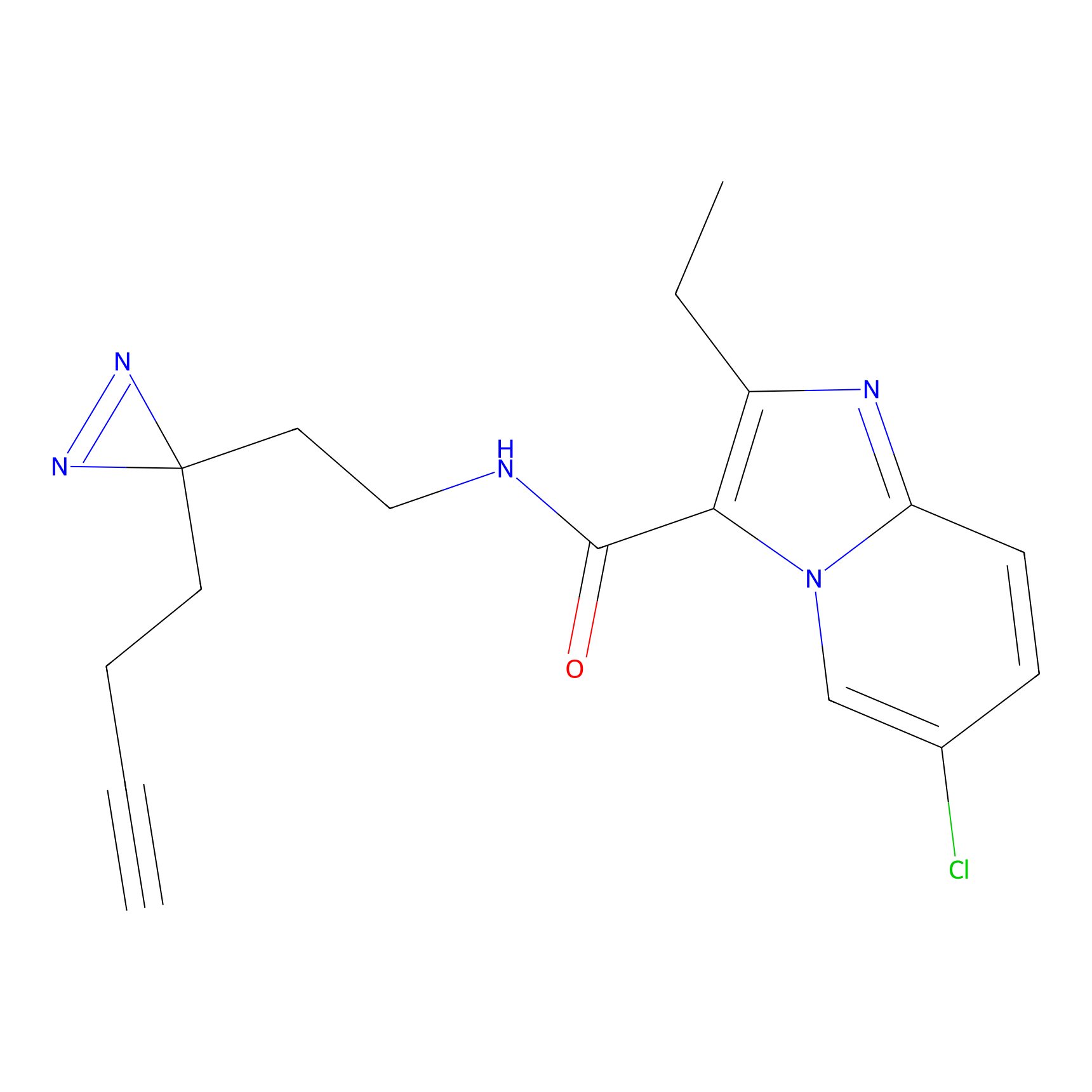
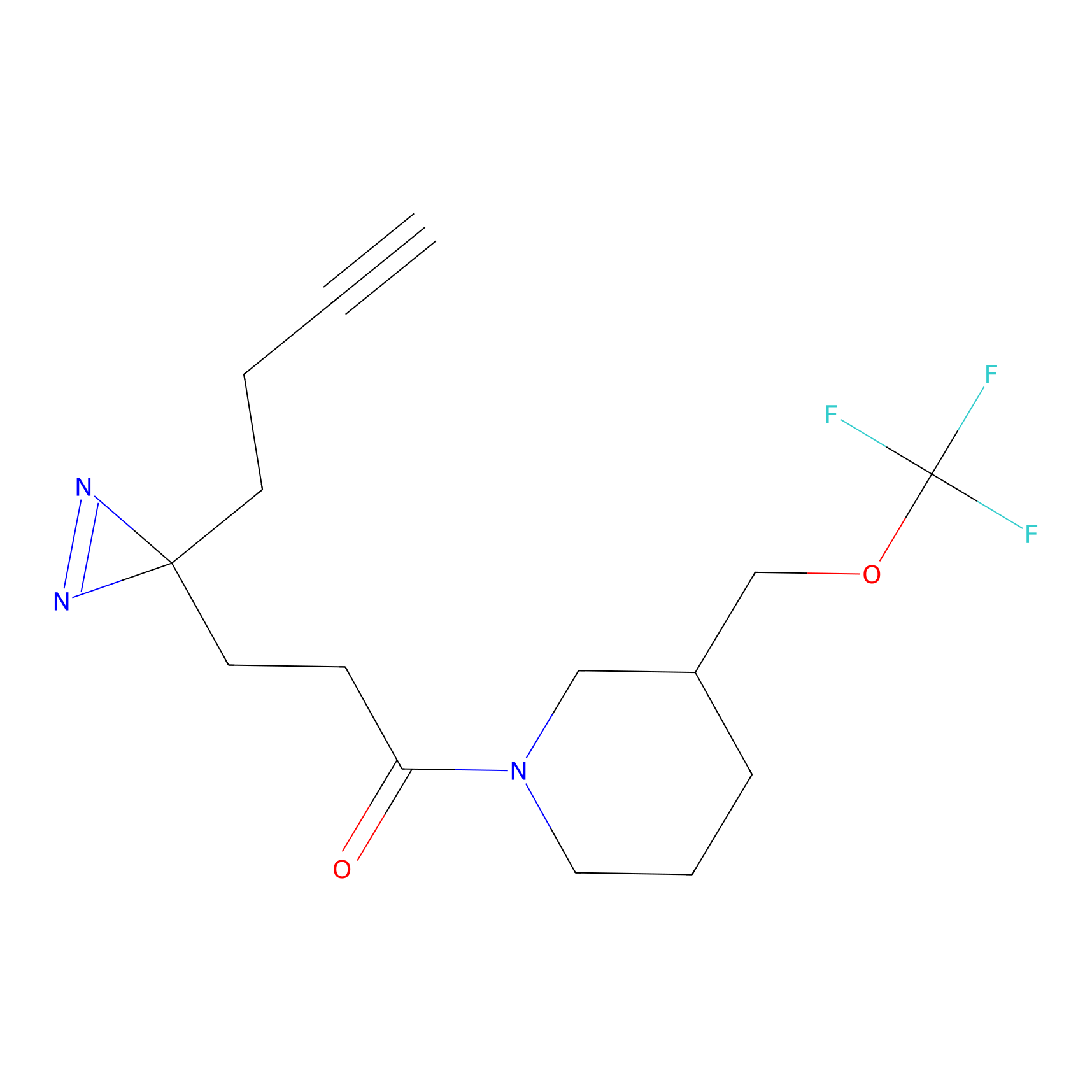
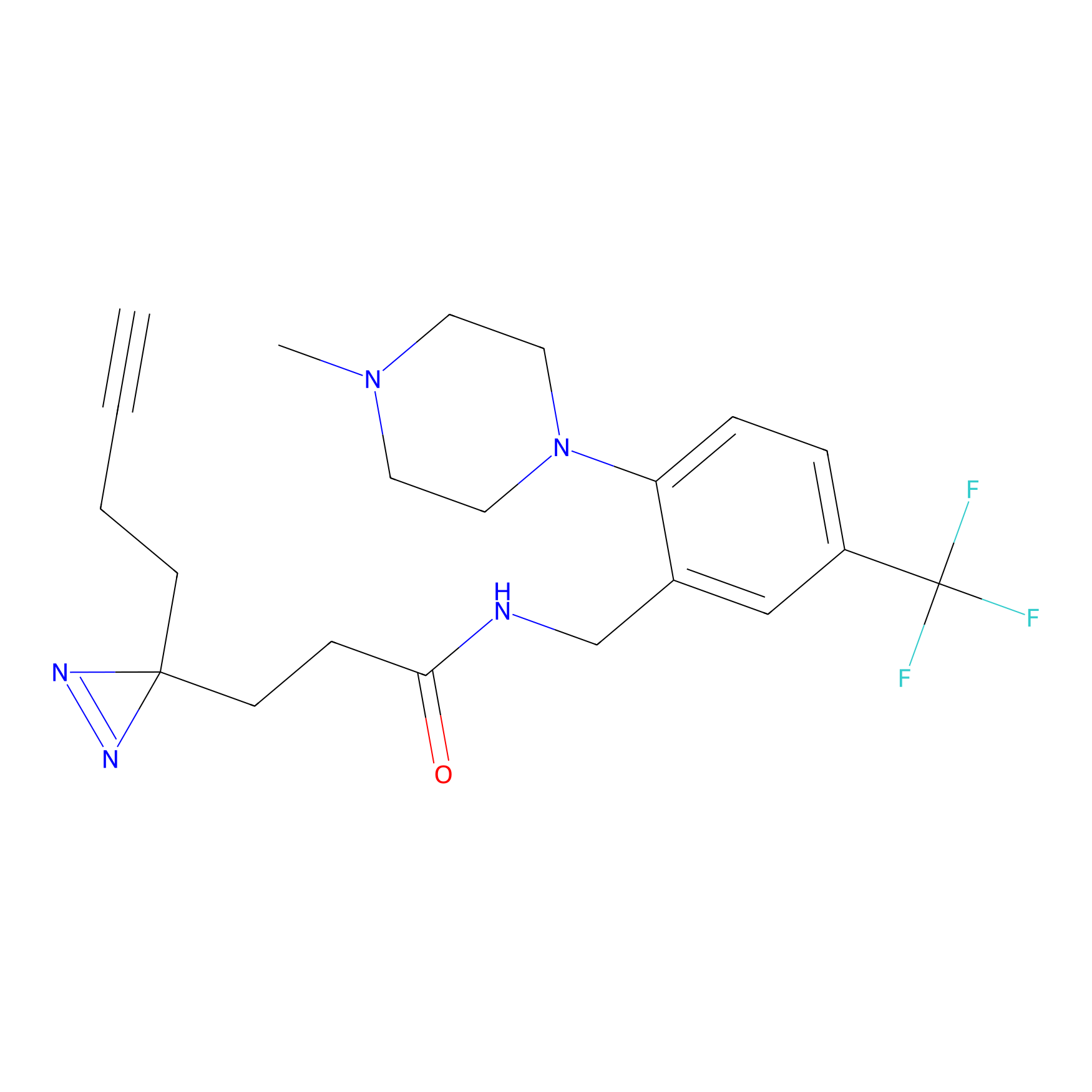
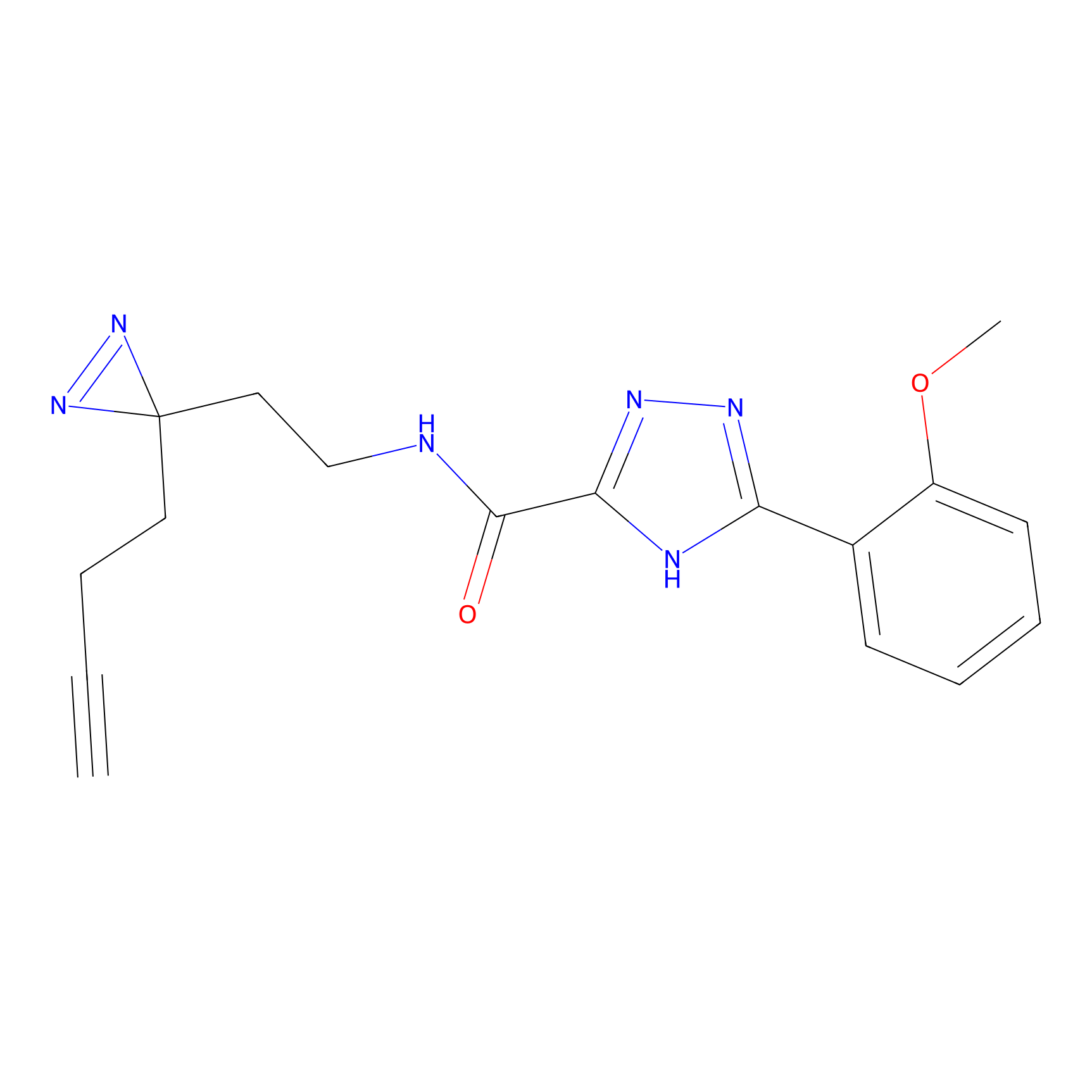
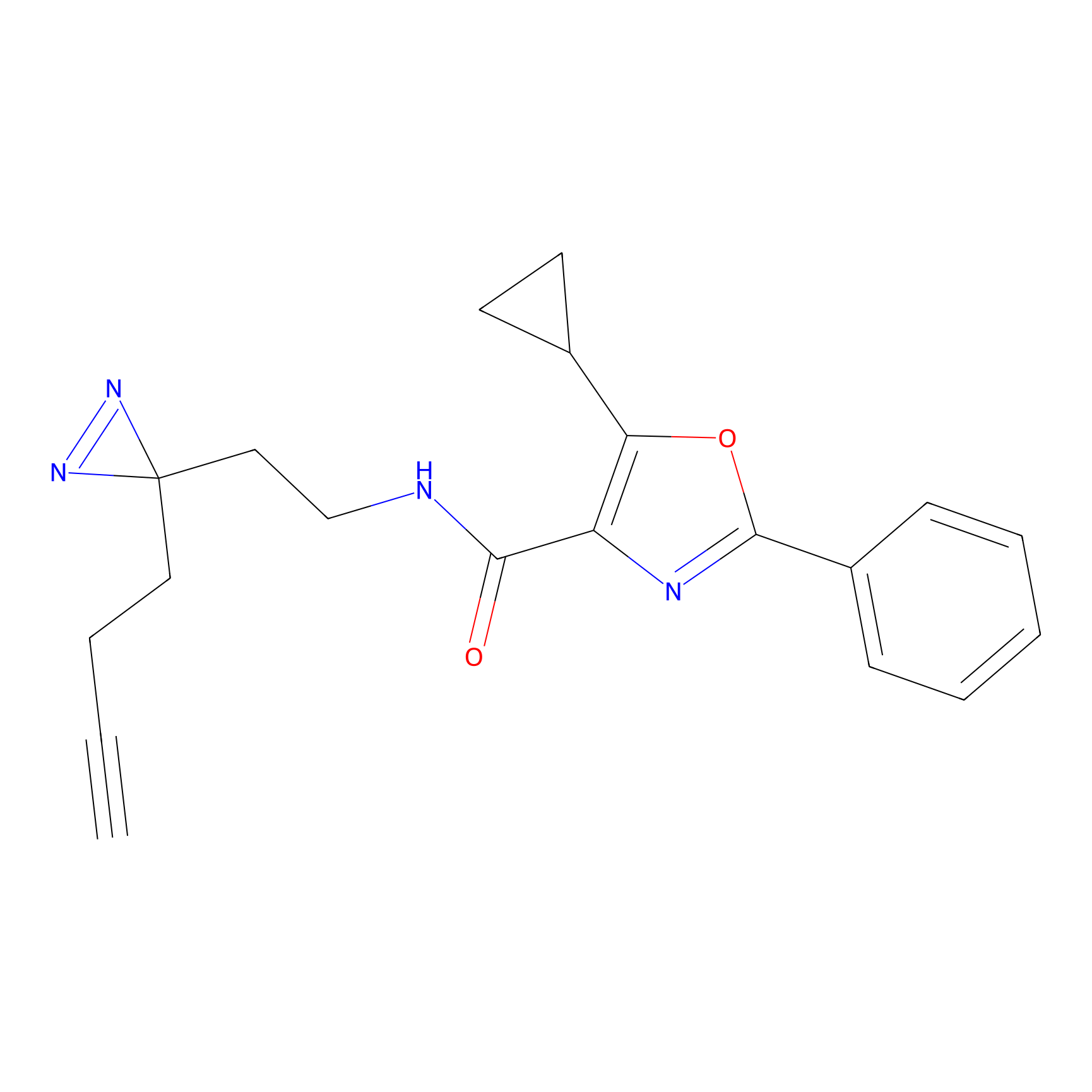
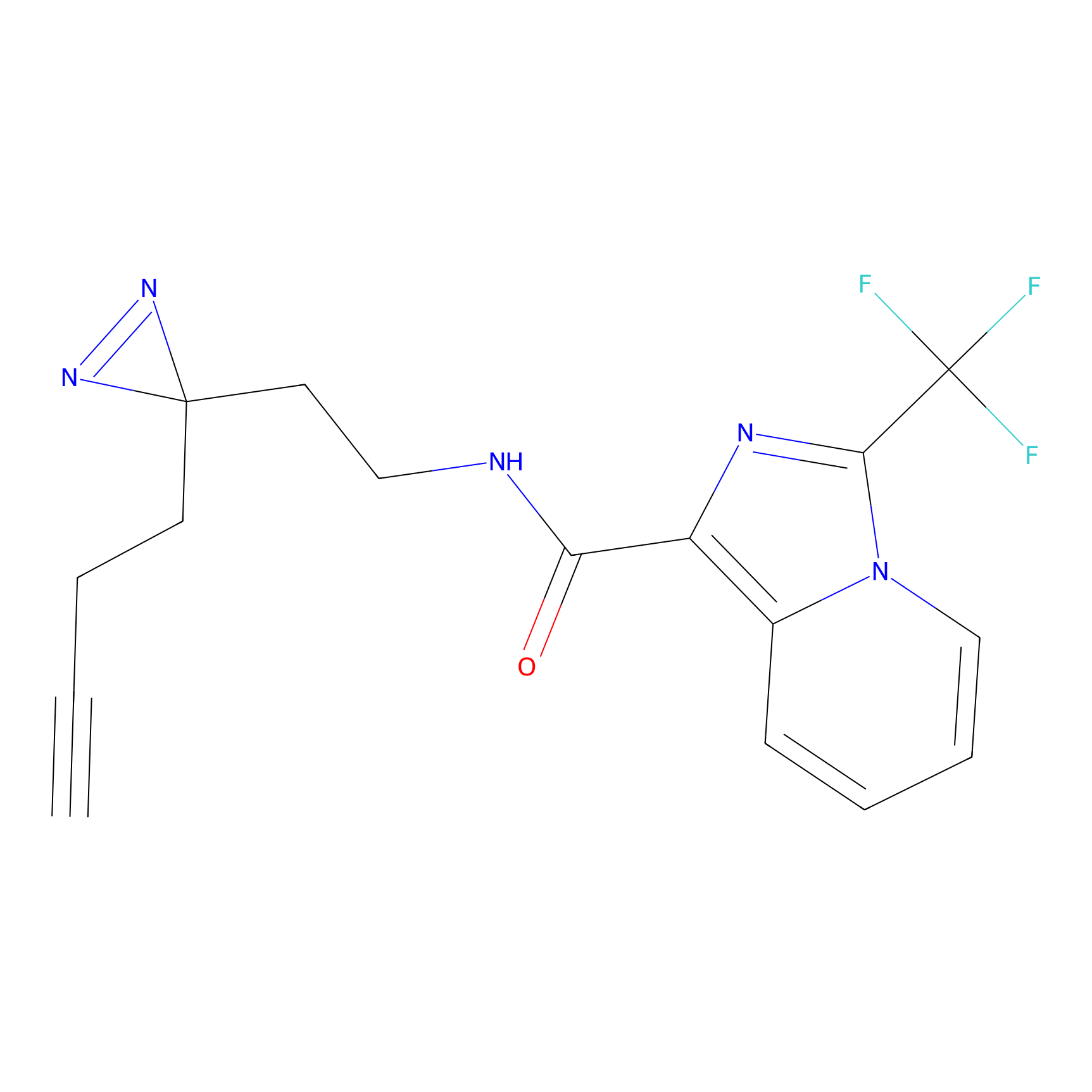
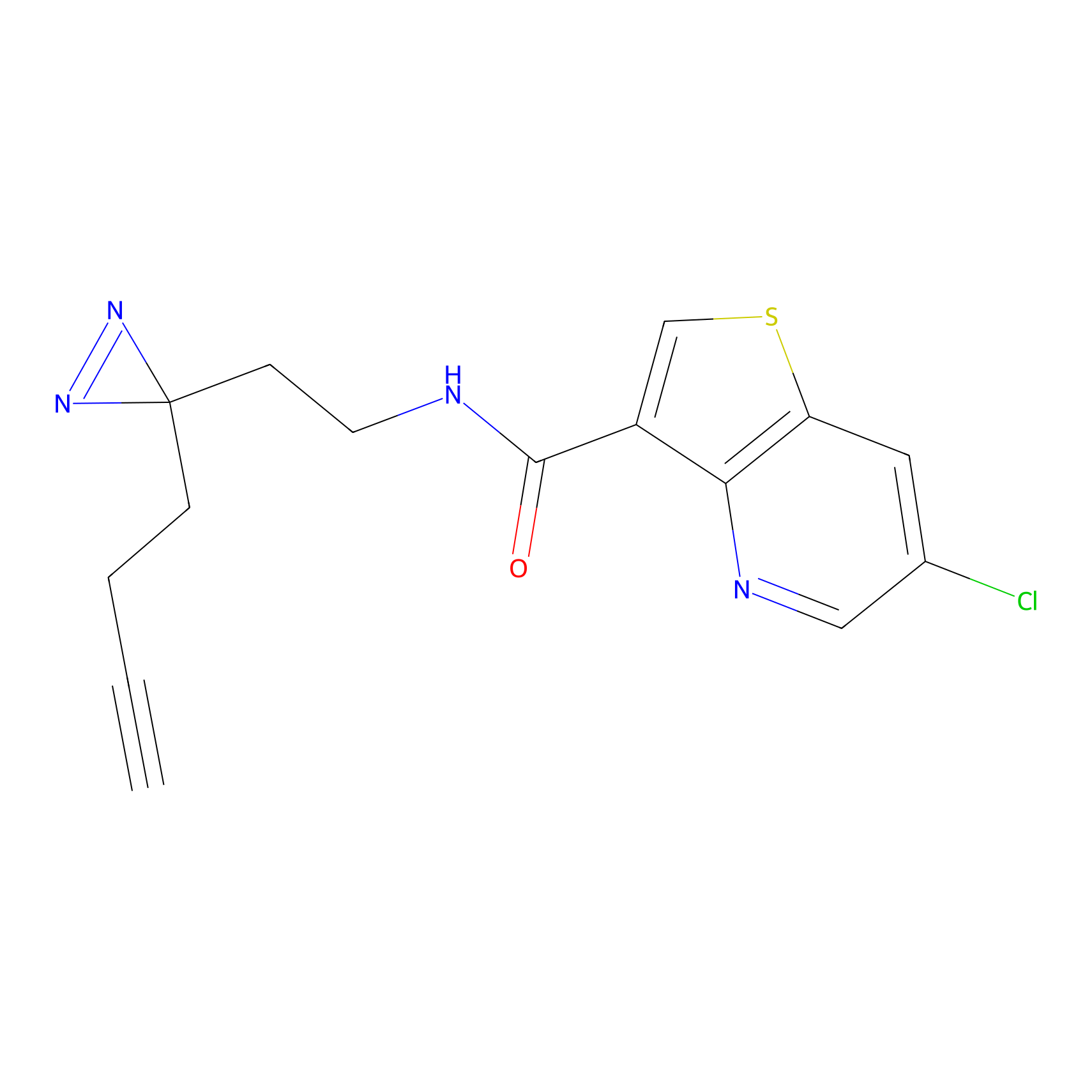
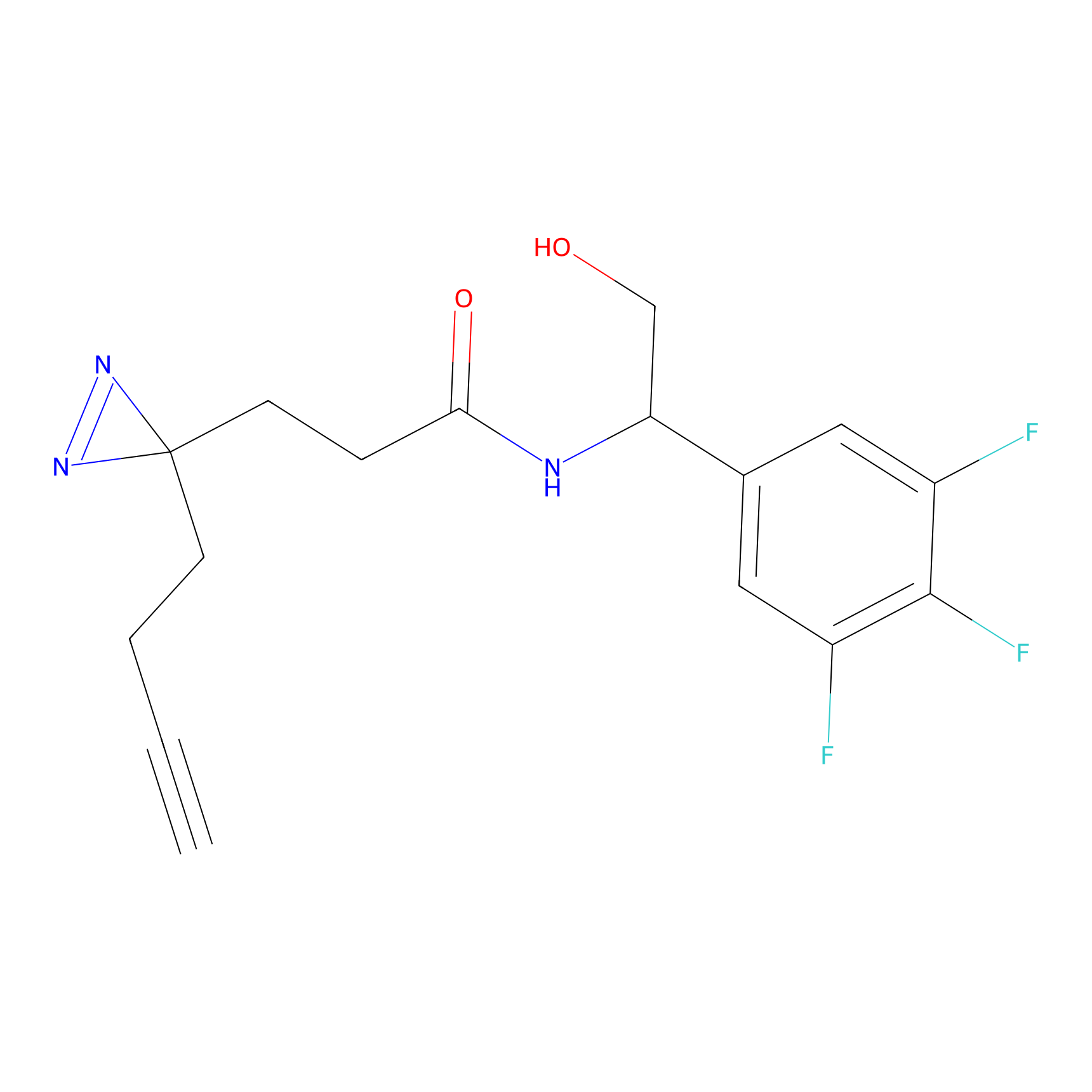
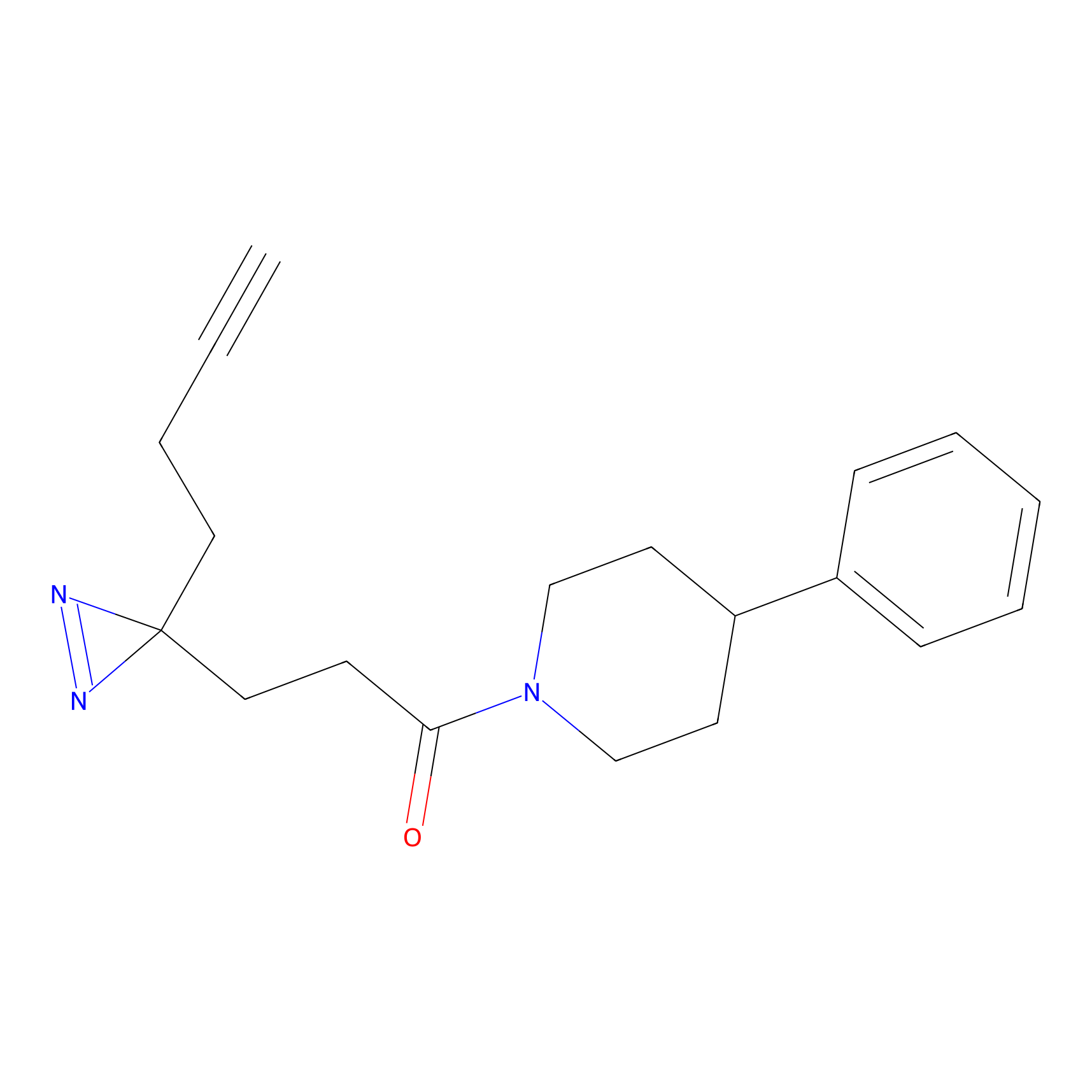
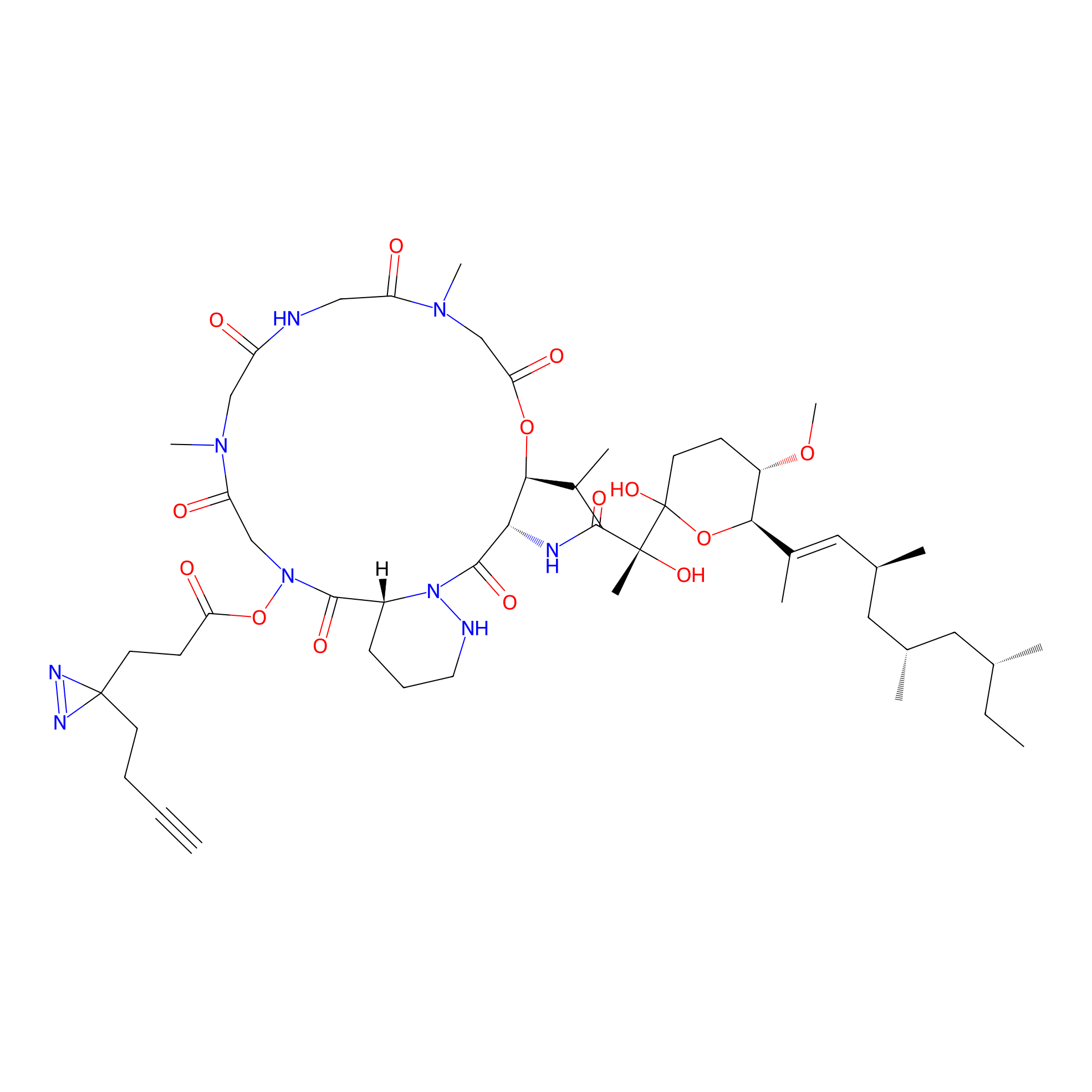
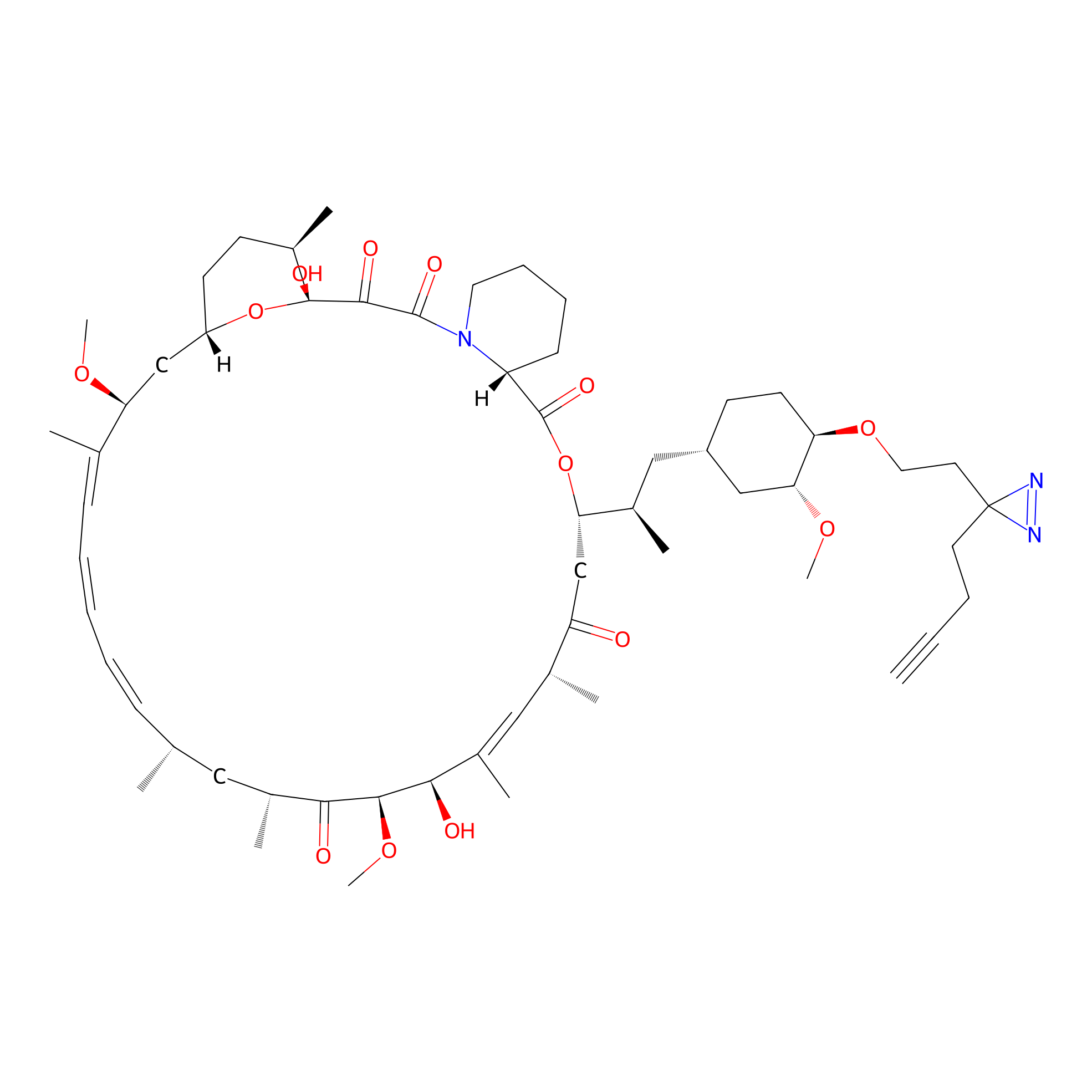
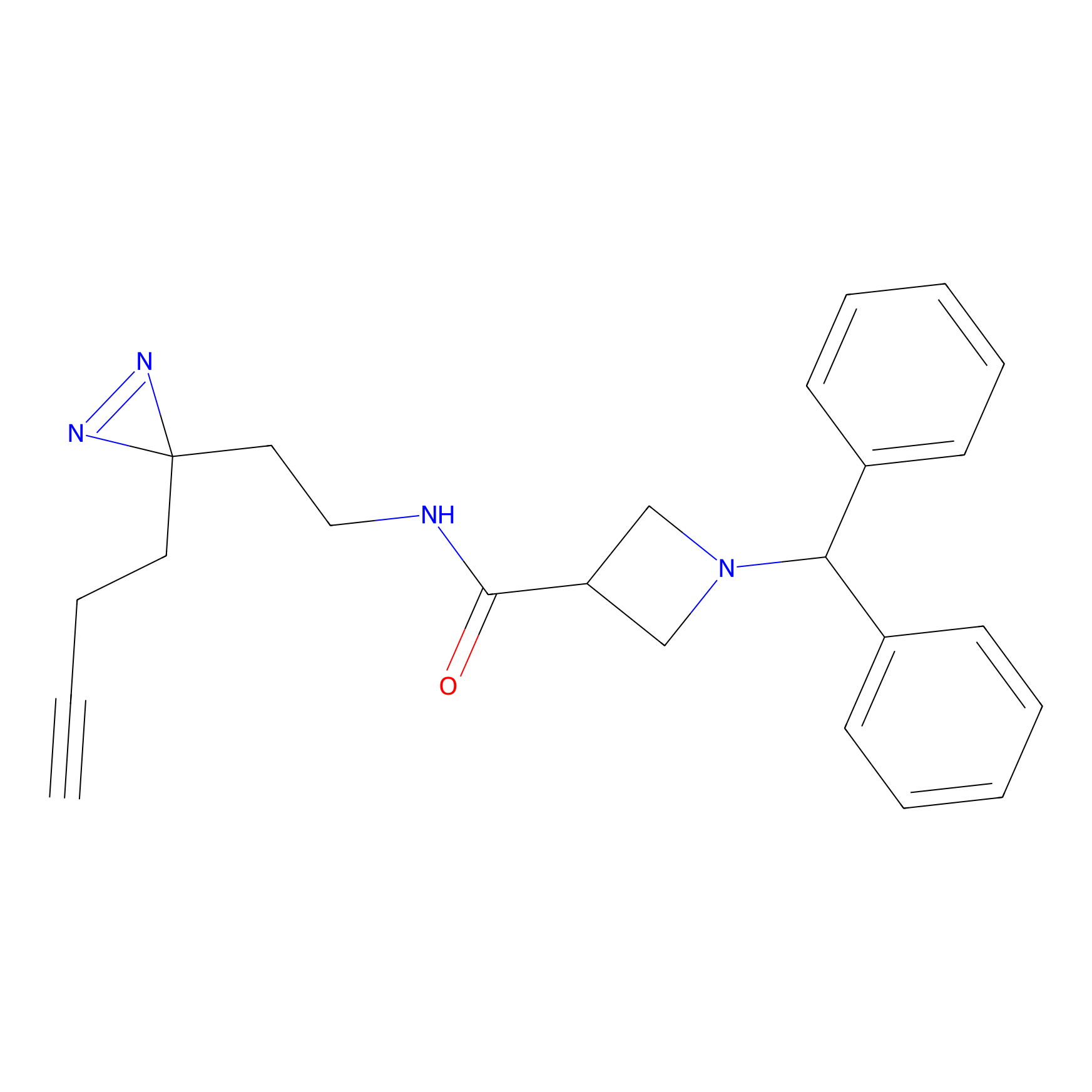
Details of the Target
General Information of Target
| Target ID | LDTP11813 | |||||
|---|---|---|---|---|---|---|
| Target Name | Solute carrier family 25 member 32 (SLC25A32) | |||||
| Gene Name | SLC25A32 | |||||
| Gene ID | 81034 | |||||
| Synonyms |
MFT; MFTC; Solute carrier family 25 member 32; Mitochondrial FAD transporter |
|||||
| 3D Structure | ||||||
| Sequence |
MGNGGRSGLQQGKGNVDGVAATPTAASASCQYRCIECNQEAKELYRDYNHGVLKITICKS
CQKPVDKYIEYDPVIILINAILCKAQAYRHILFNTQINIHGKLCIFCLLCEAYLRWWQLQ DSNQNTAPDDLIRYAKEWDFYRMFAIAALEQTAYFIGIFTFLWVERPMTAKKKPNFILLL KALLLSSYGKLLLIPAVIWEHDYTSVCLKLIKVFVLTSNFQAIRVTLNINRKLSFLAVLS GLLLESIMVYFFQSMEWDVGSDYAIFKSQDF |
|||||
| Target Bioclass |
Transporter and channel
|
|||||
| Family |
Mitochondrial carrier (TC 2.A.29) family
|
|||||
| Subcellular location |
Mitochondrion inner membrane
|
|||||
| Function |
Facilitates flavin adenine dinucleotide (FAD) translocation across the mitochondrial inner membrane into the mitochondrial matrix where it acts as a redox cofactor to assist flavoenzyme activities in fundamental metabolic processes including fatty acid beta-oxidation, amino acid and choline metabolism as well as mitochondrial electron transportation. In particular, provides FAD to DLD dehydrogenase of the glycine cleavage system, part of mitochondrial one-carbon metabolic pathway involved in neural tube closure in early embryogenesis.
|
|||||
| Uniprot ID | ||||||
| Ensemble ID | ||||||
| HGNC ID | ||||||
Probe(s) Labeling This Target
ABPP Probe
| Probe name | Structure | Binding Site(Ratio) | Interaction ID | Ref | |
|---|---|---|---|---|---|
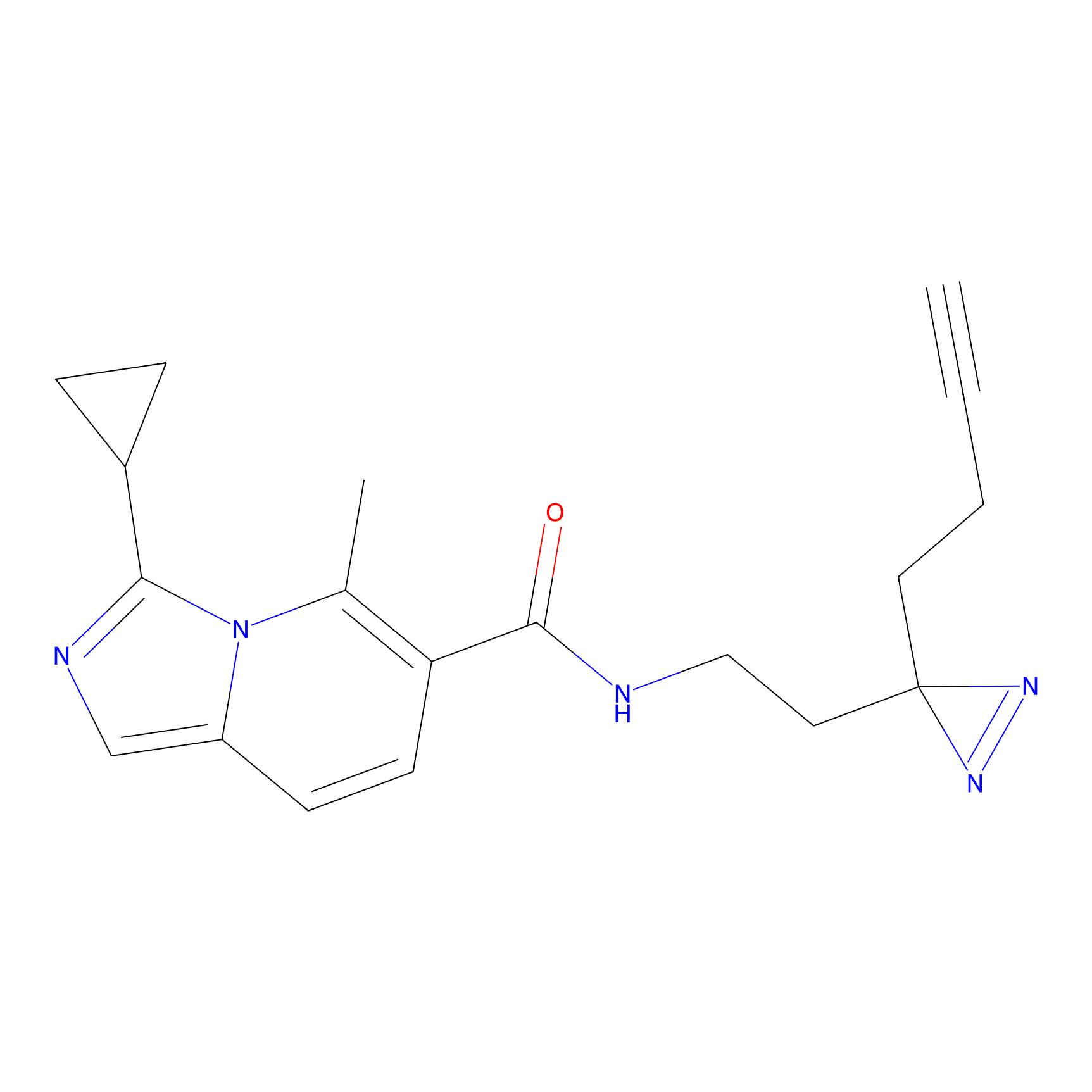
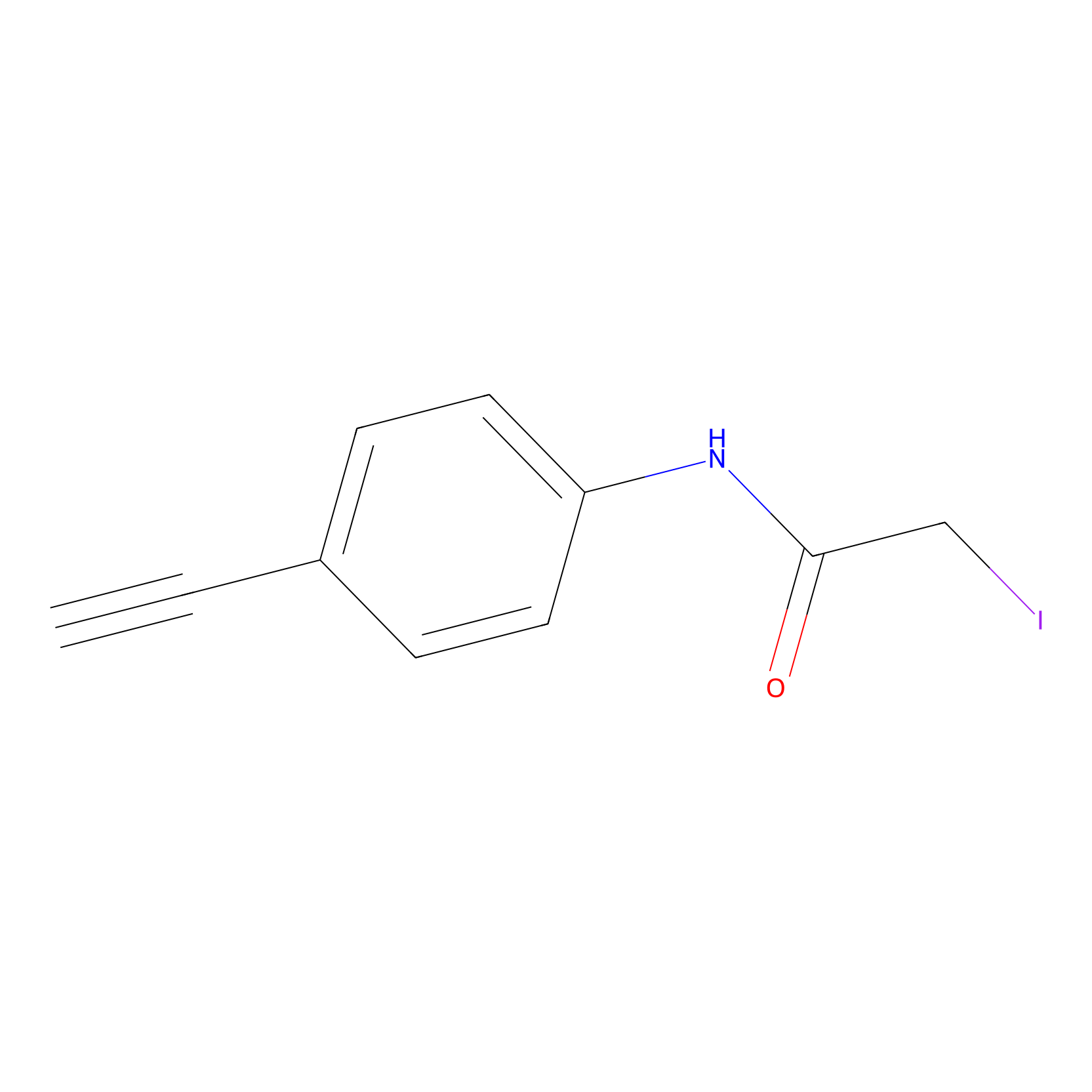
|
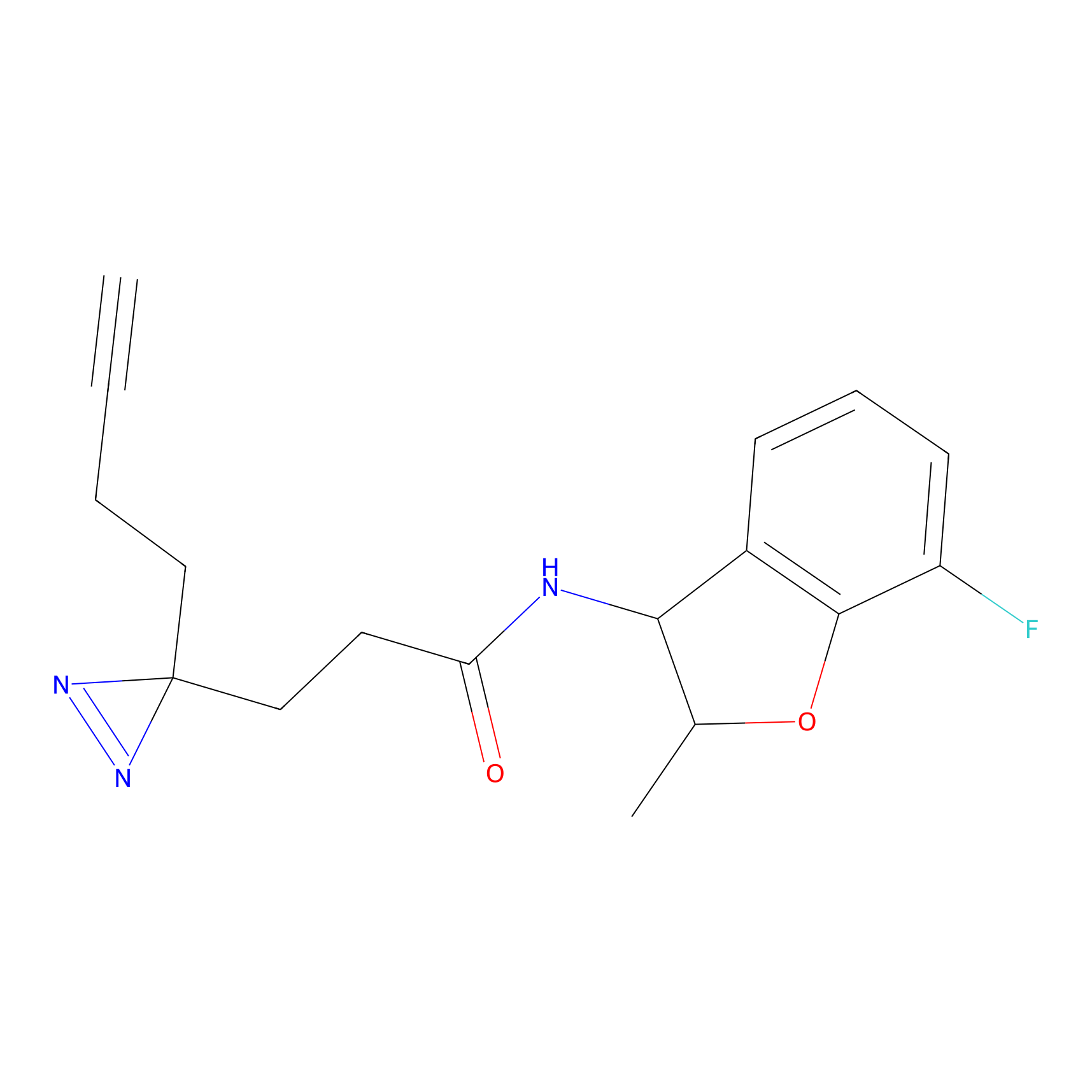
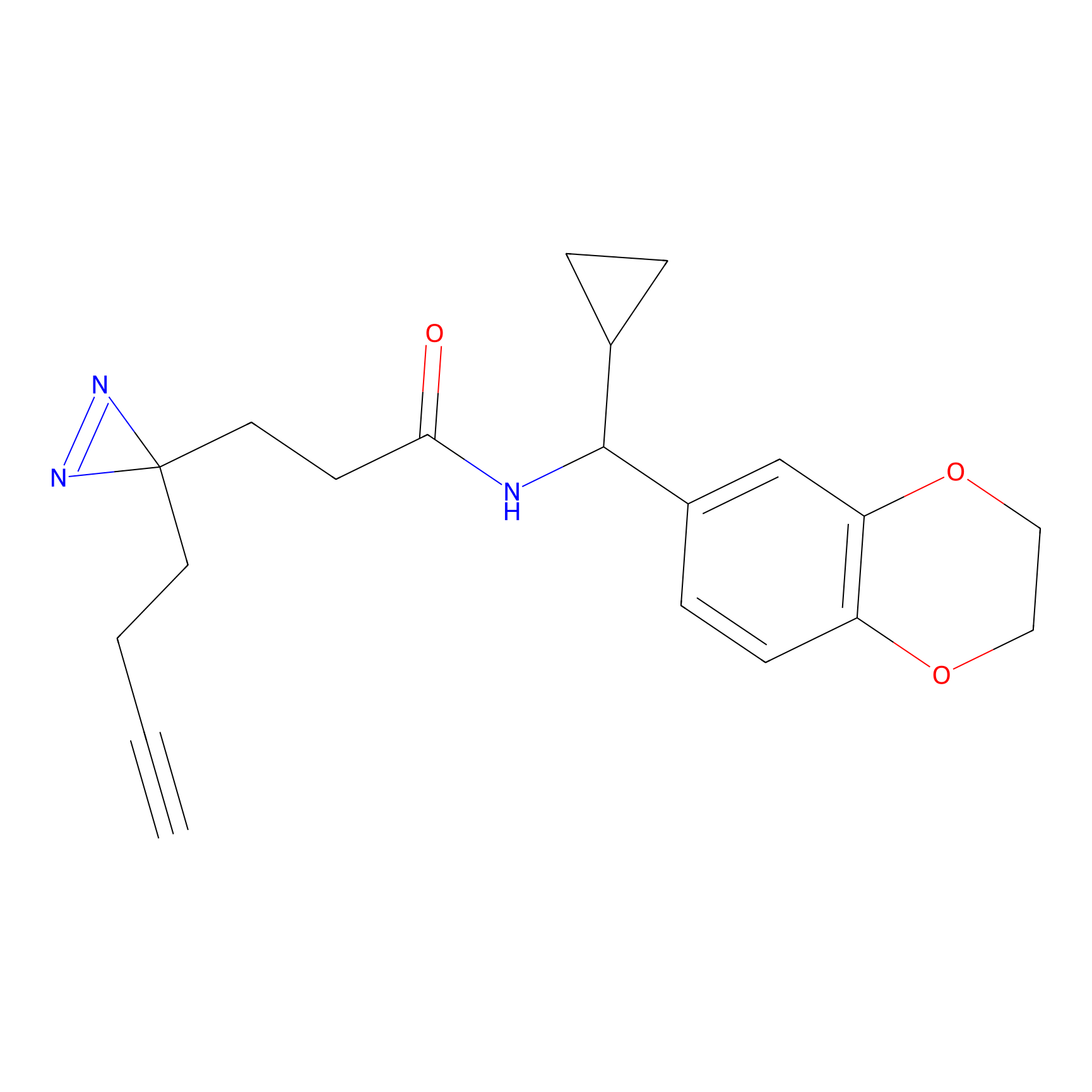
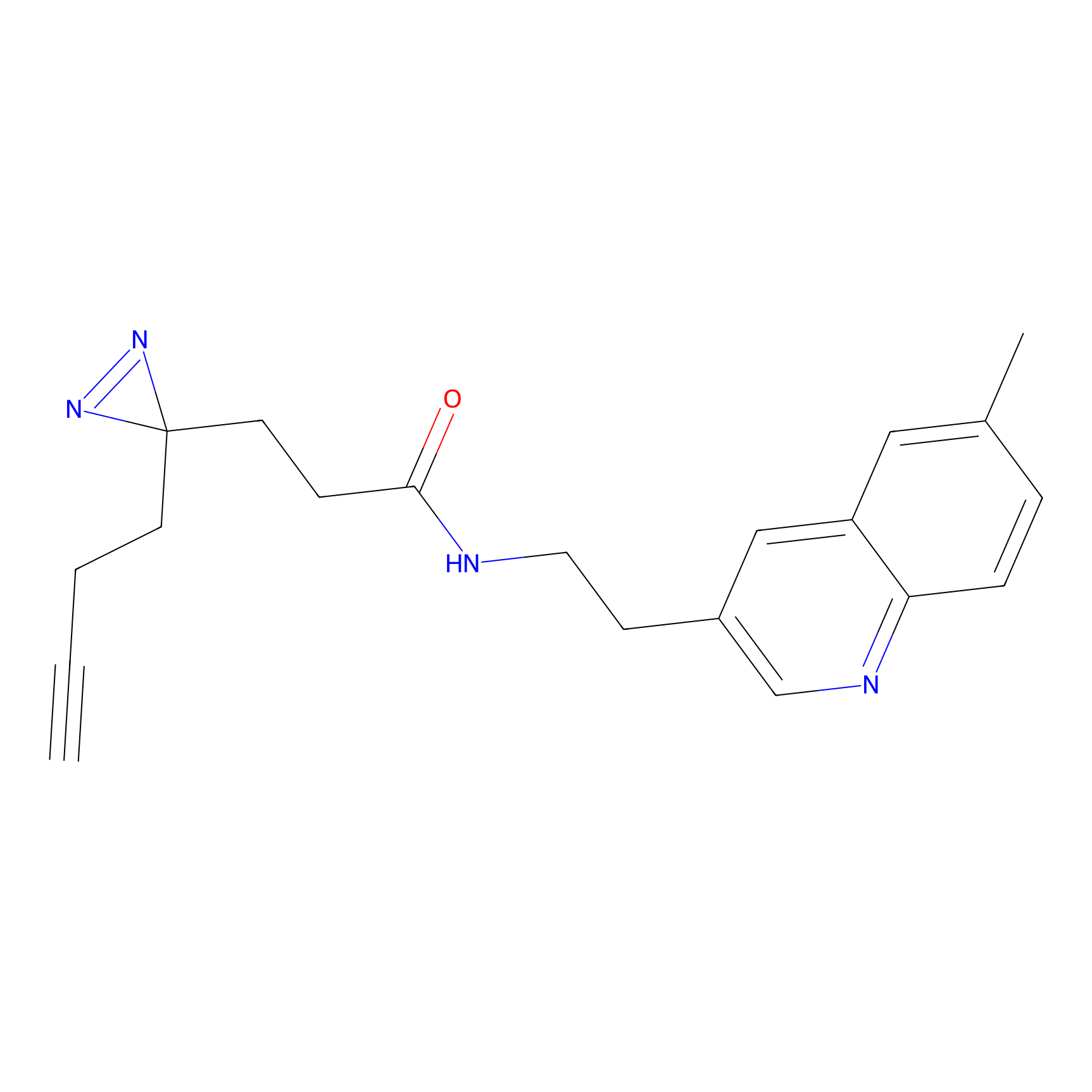
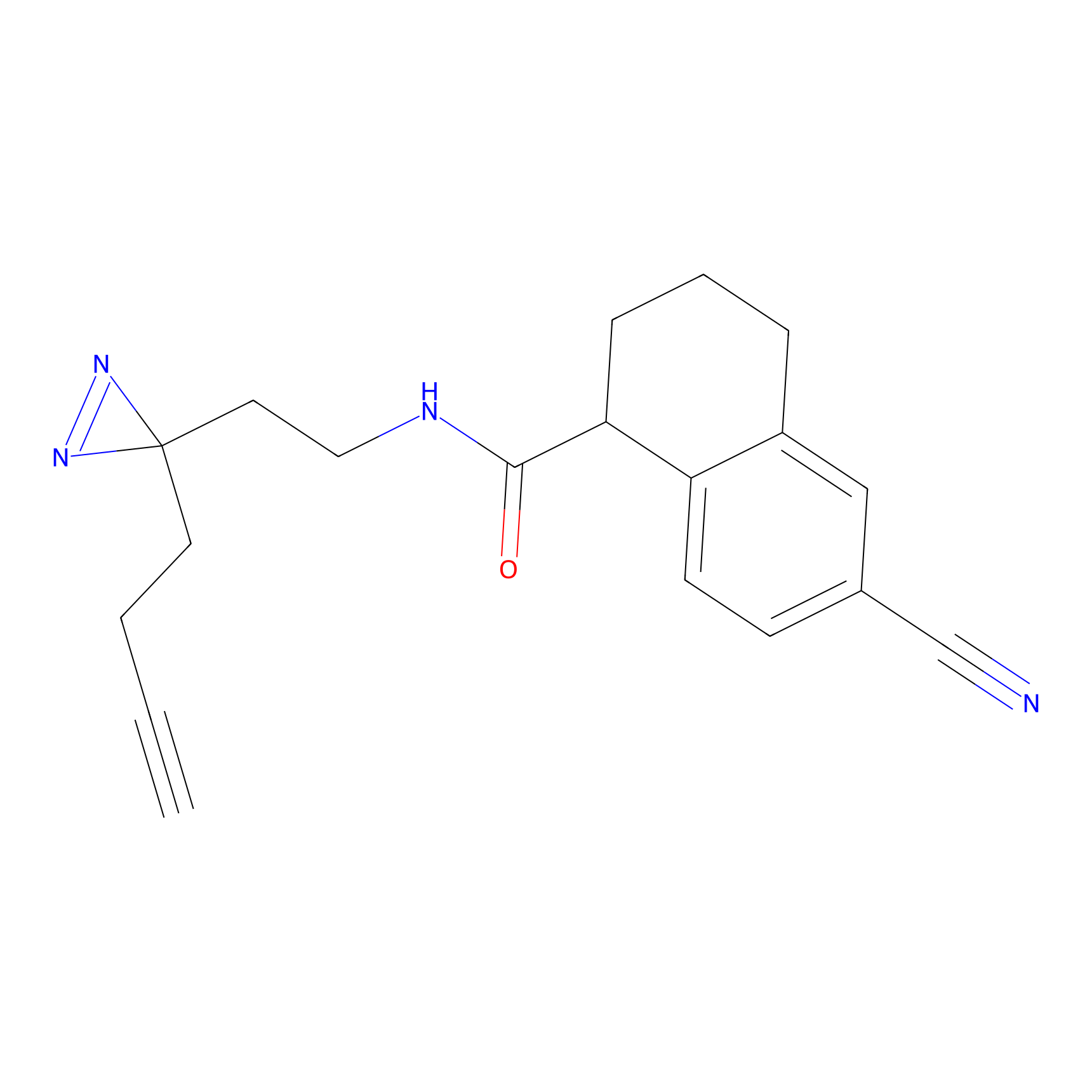
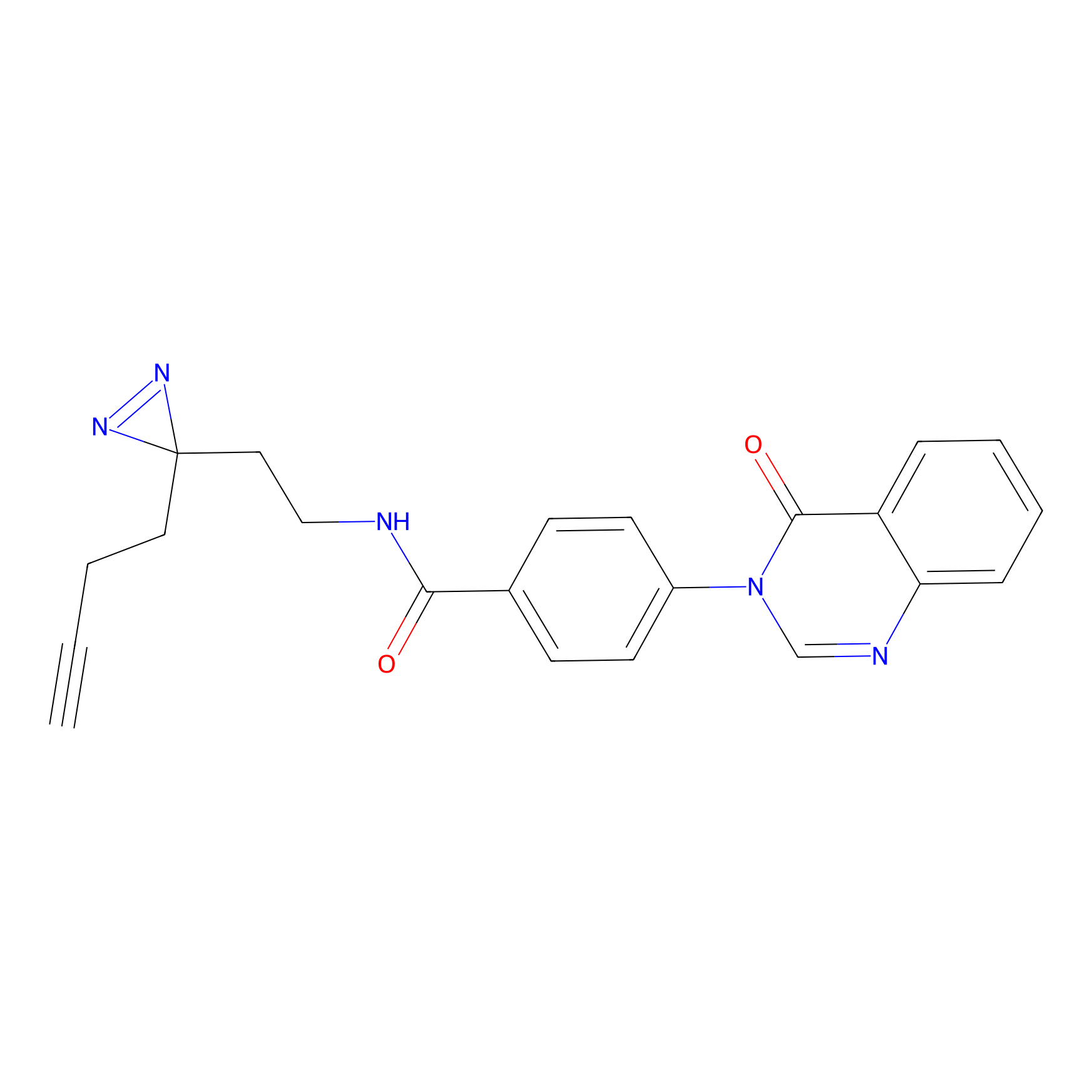
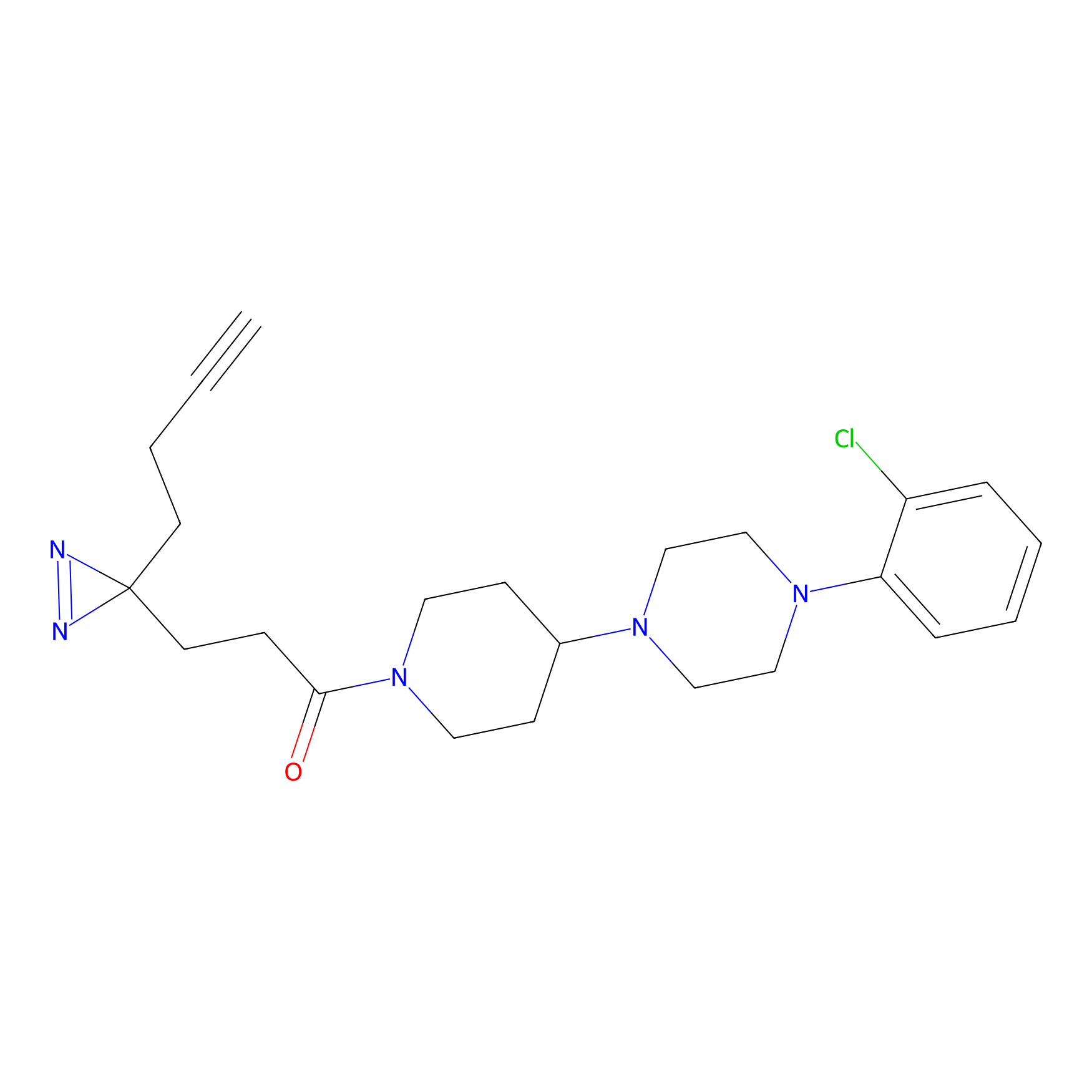
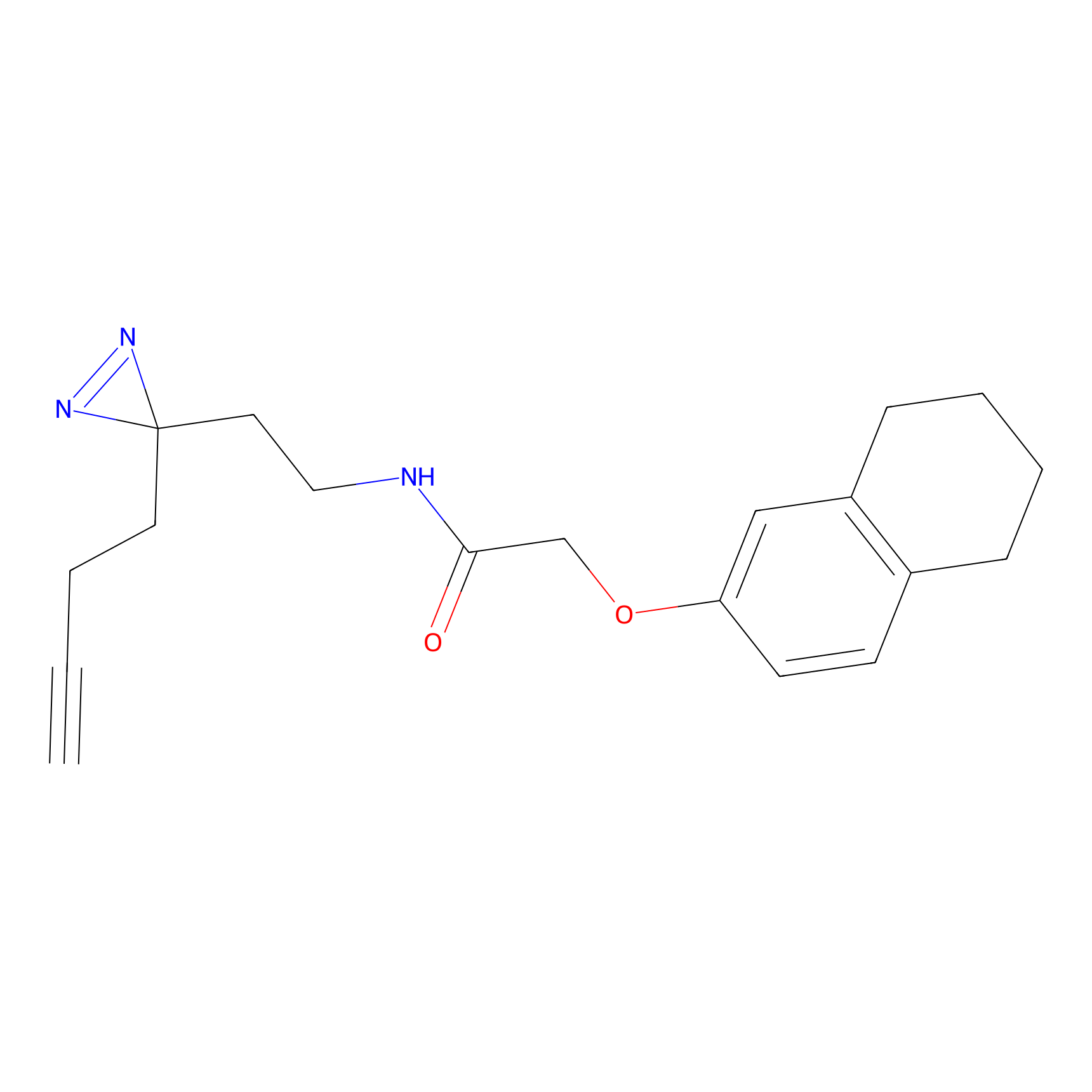
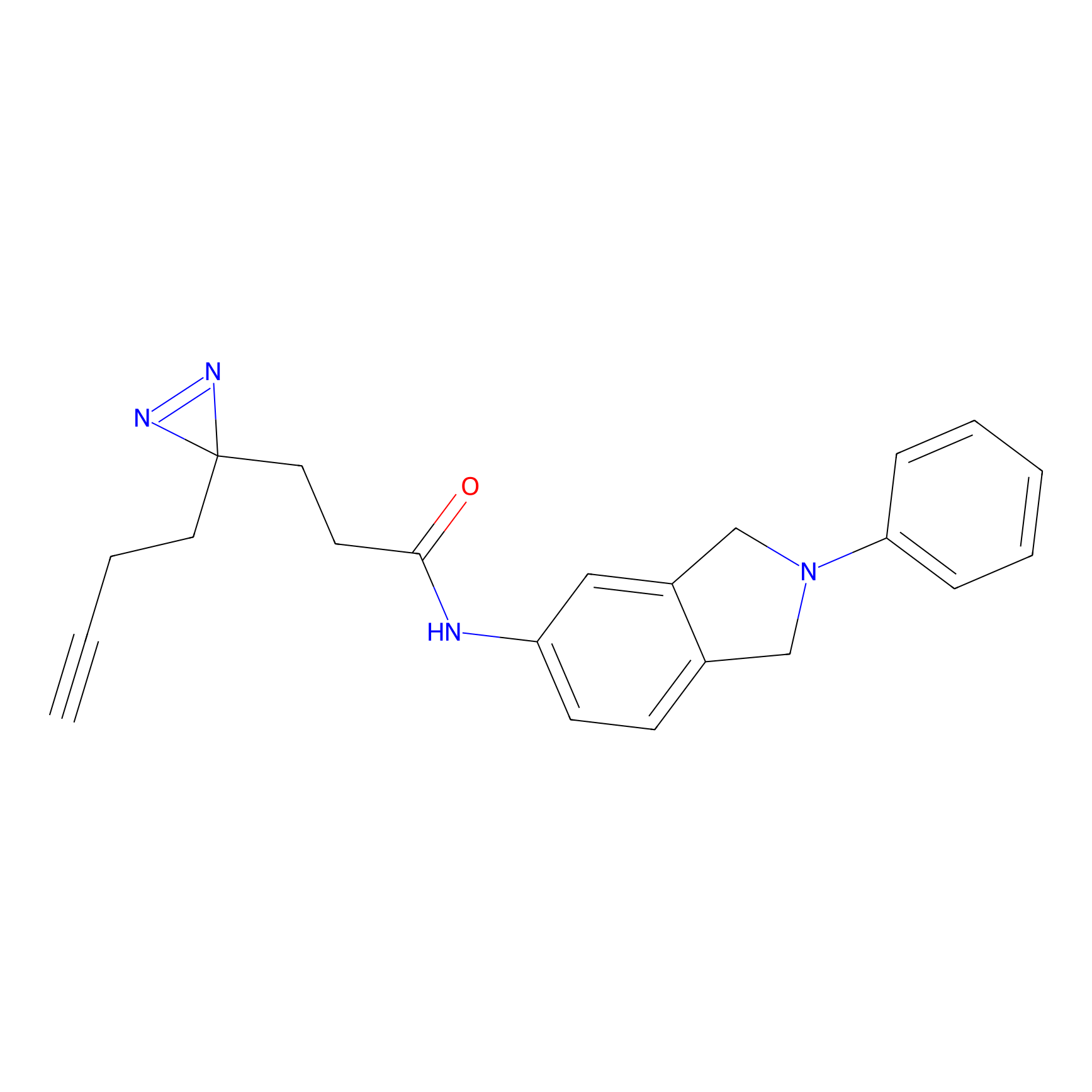
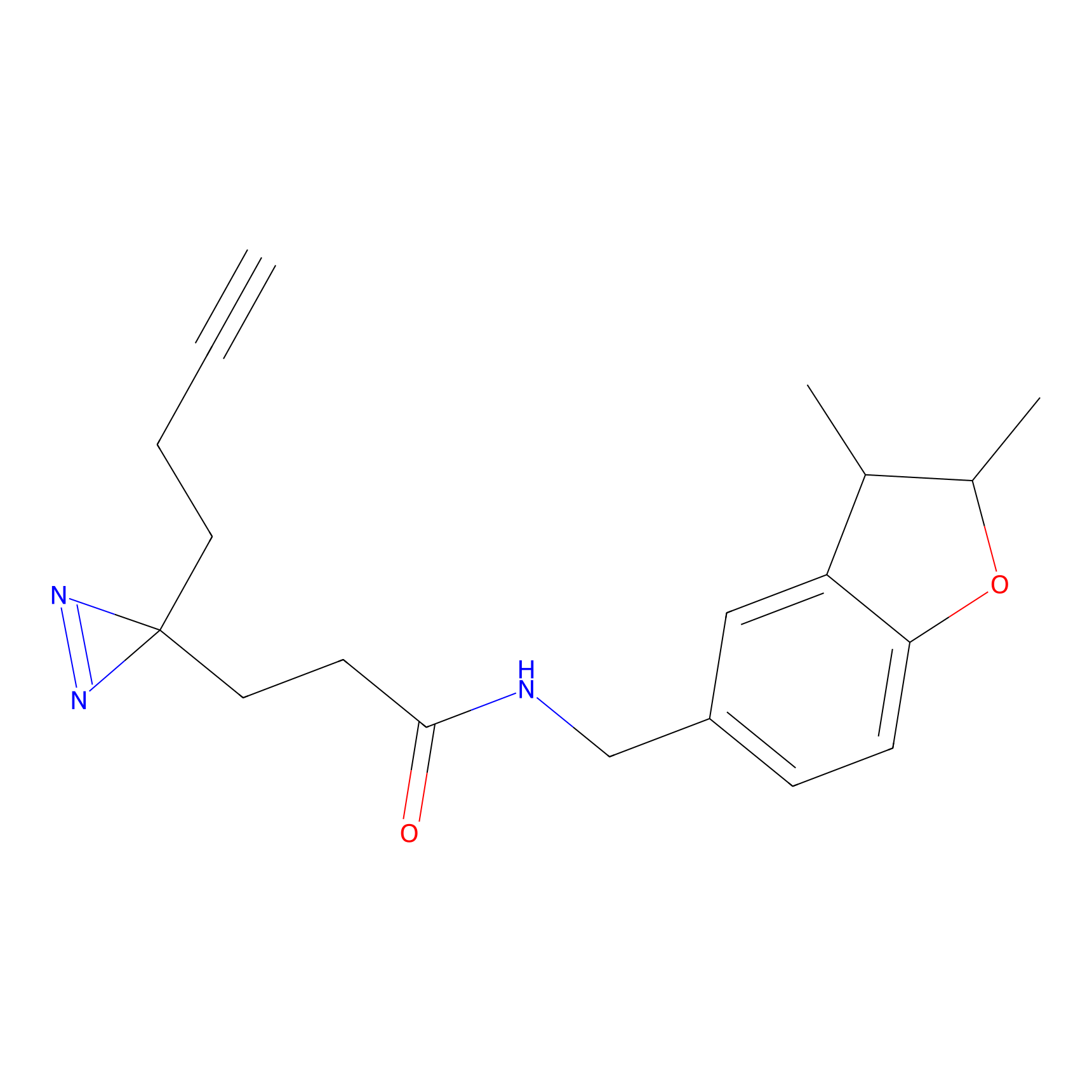
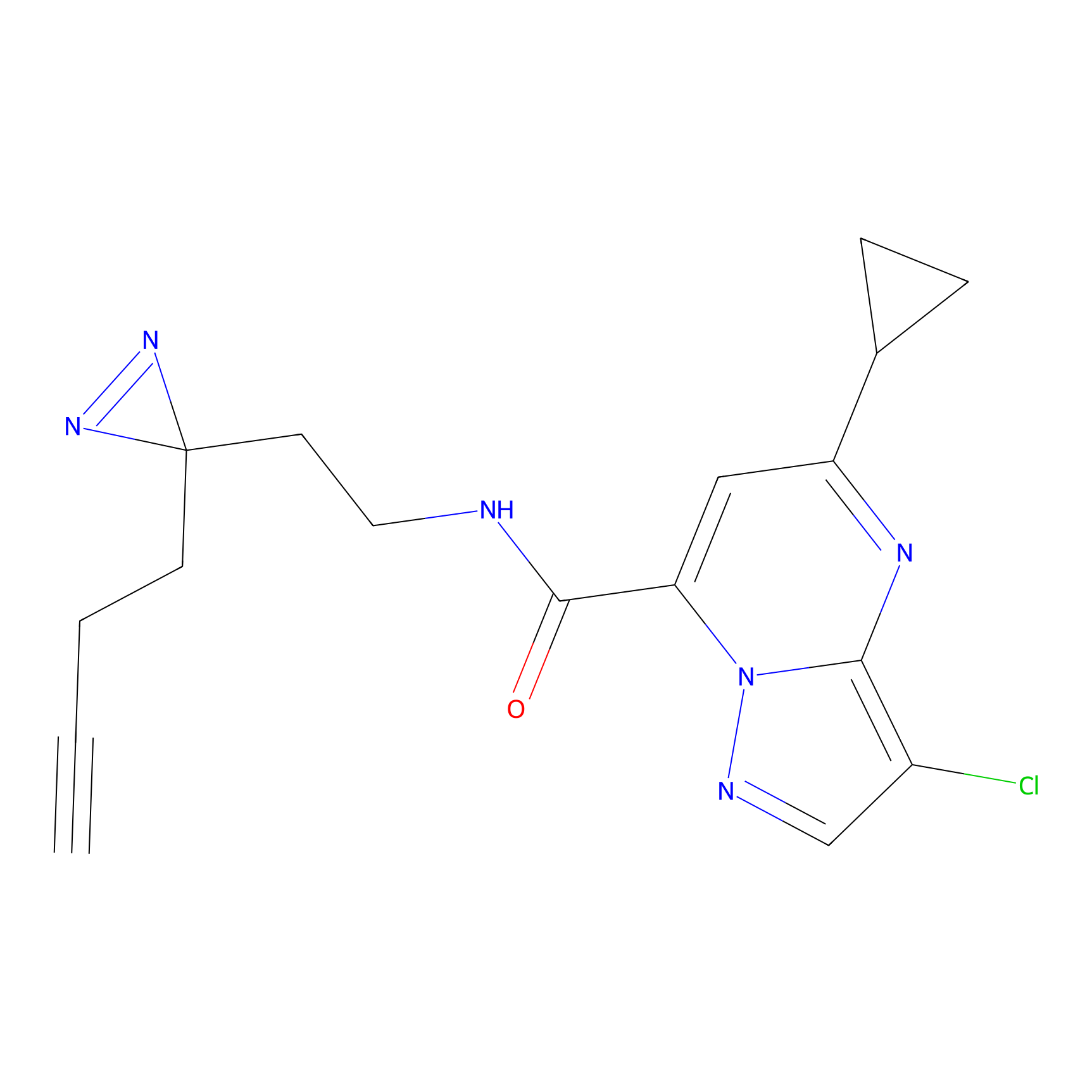
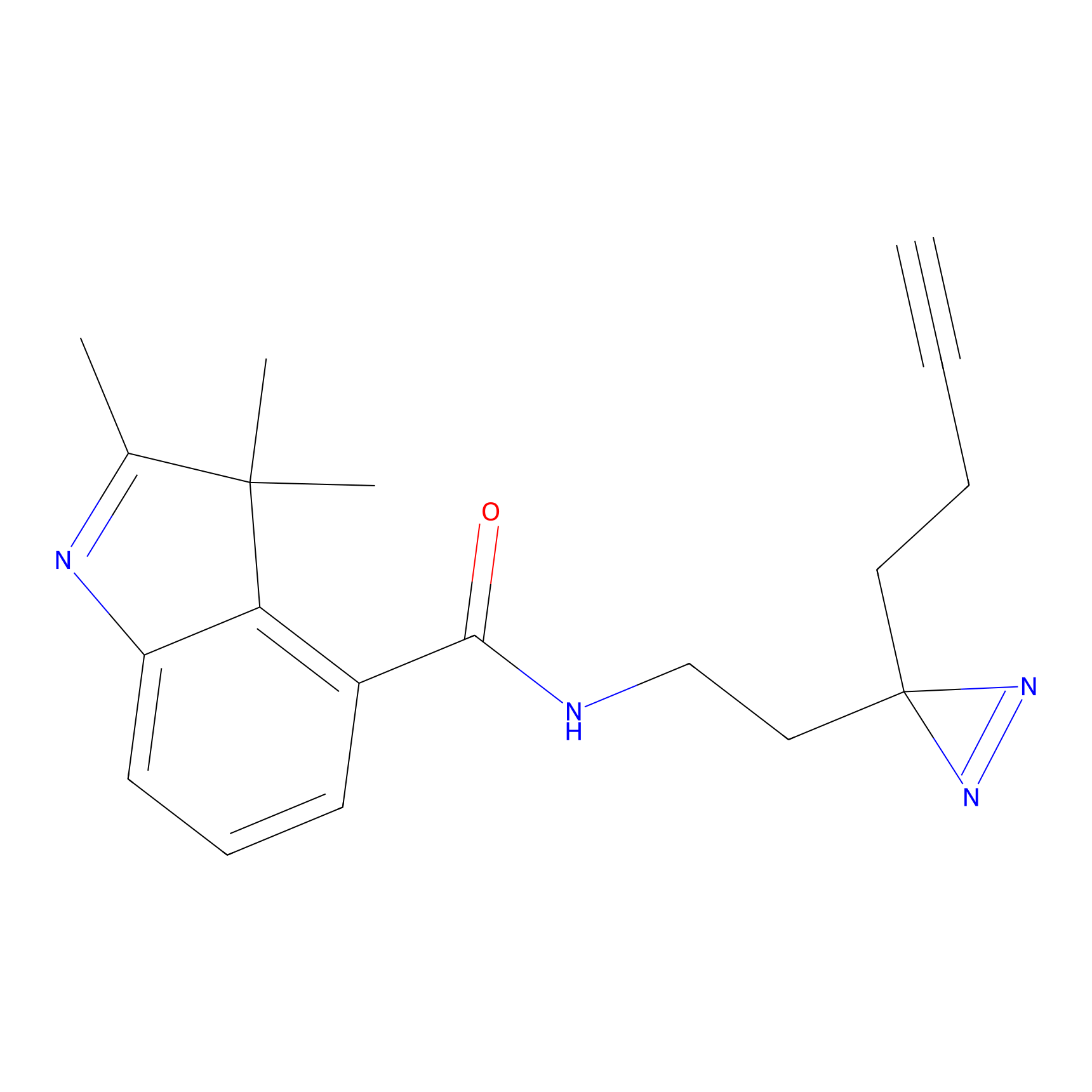
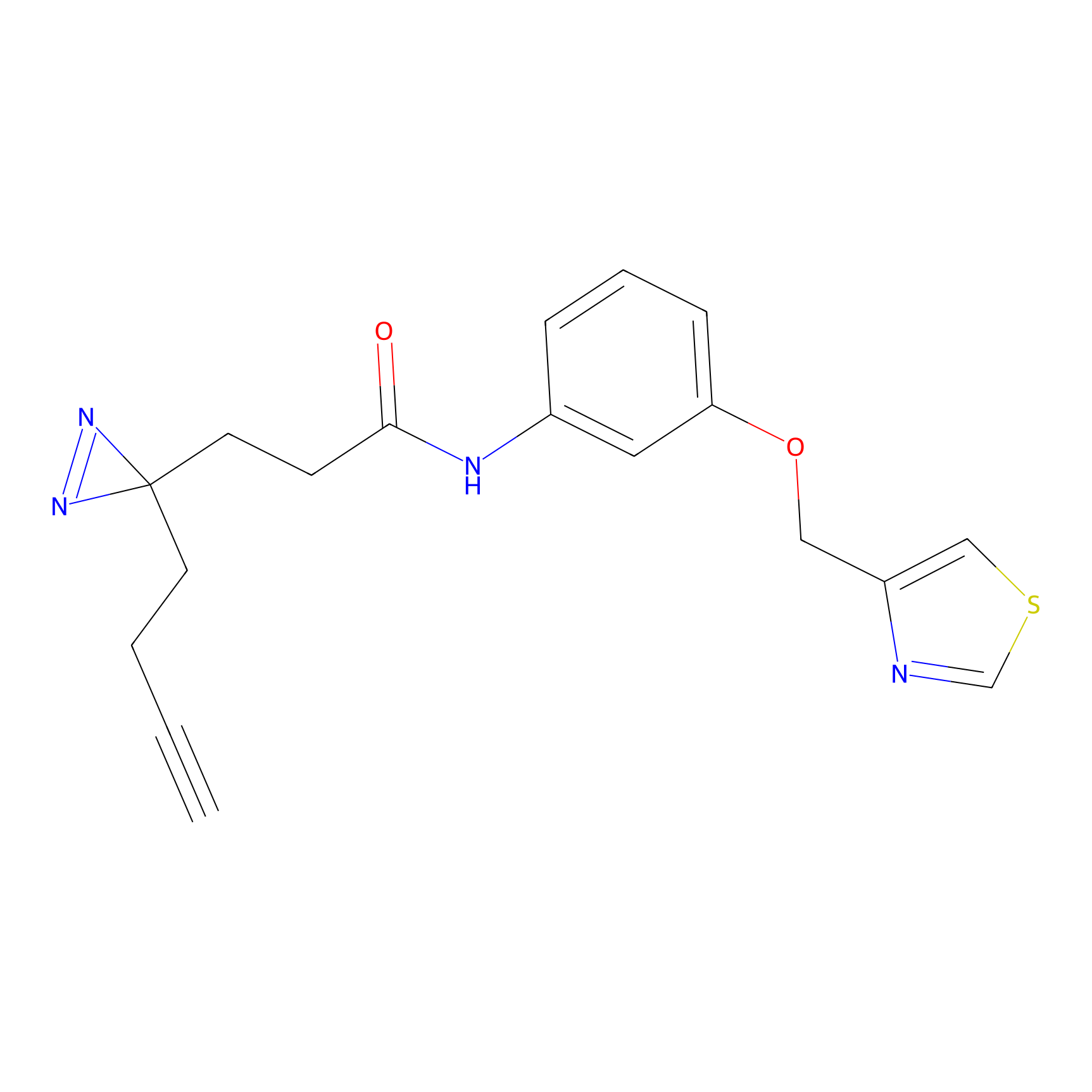
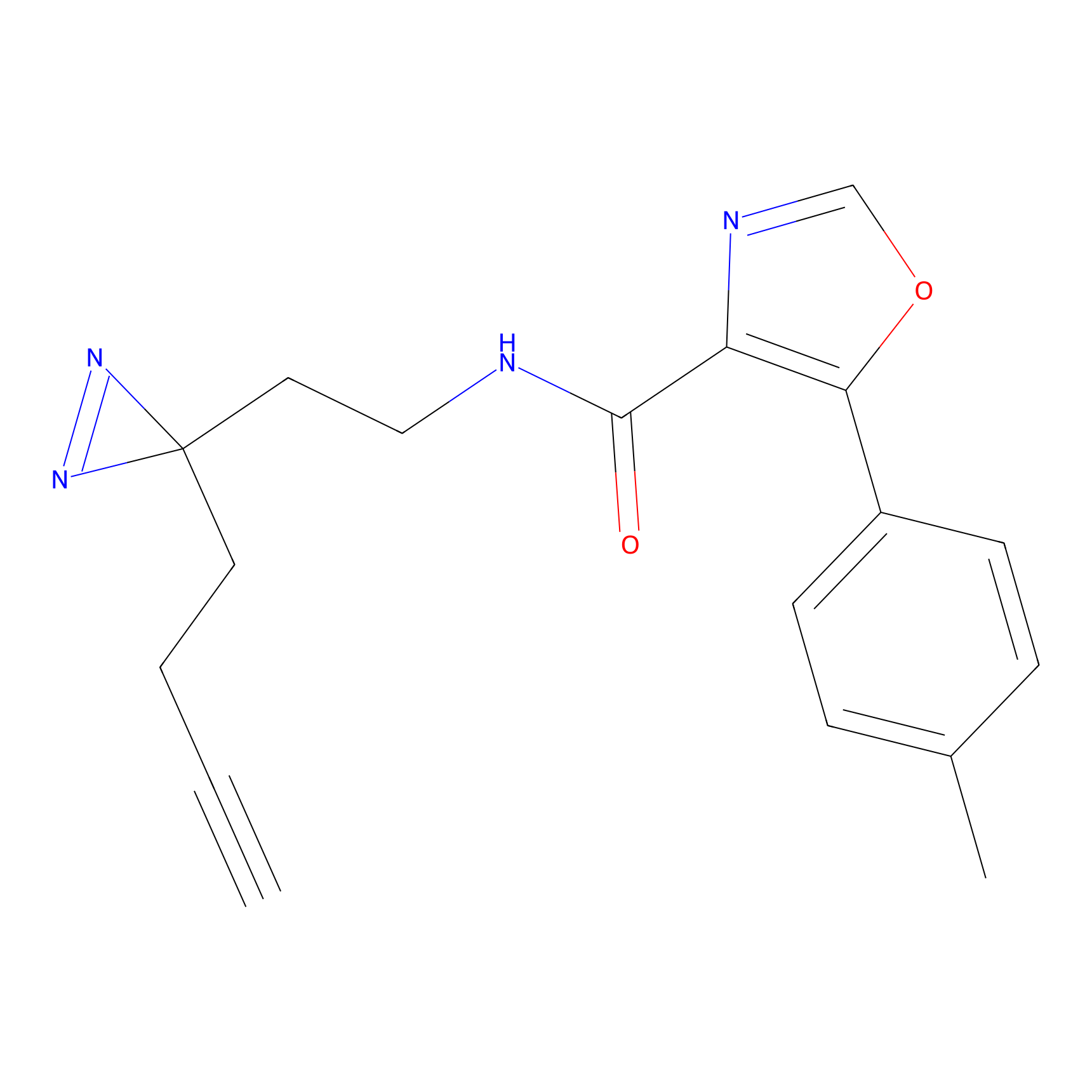
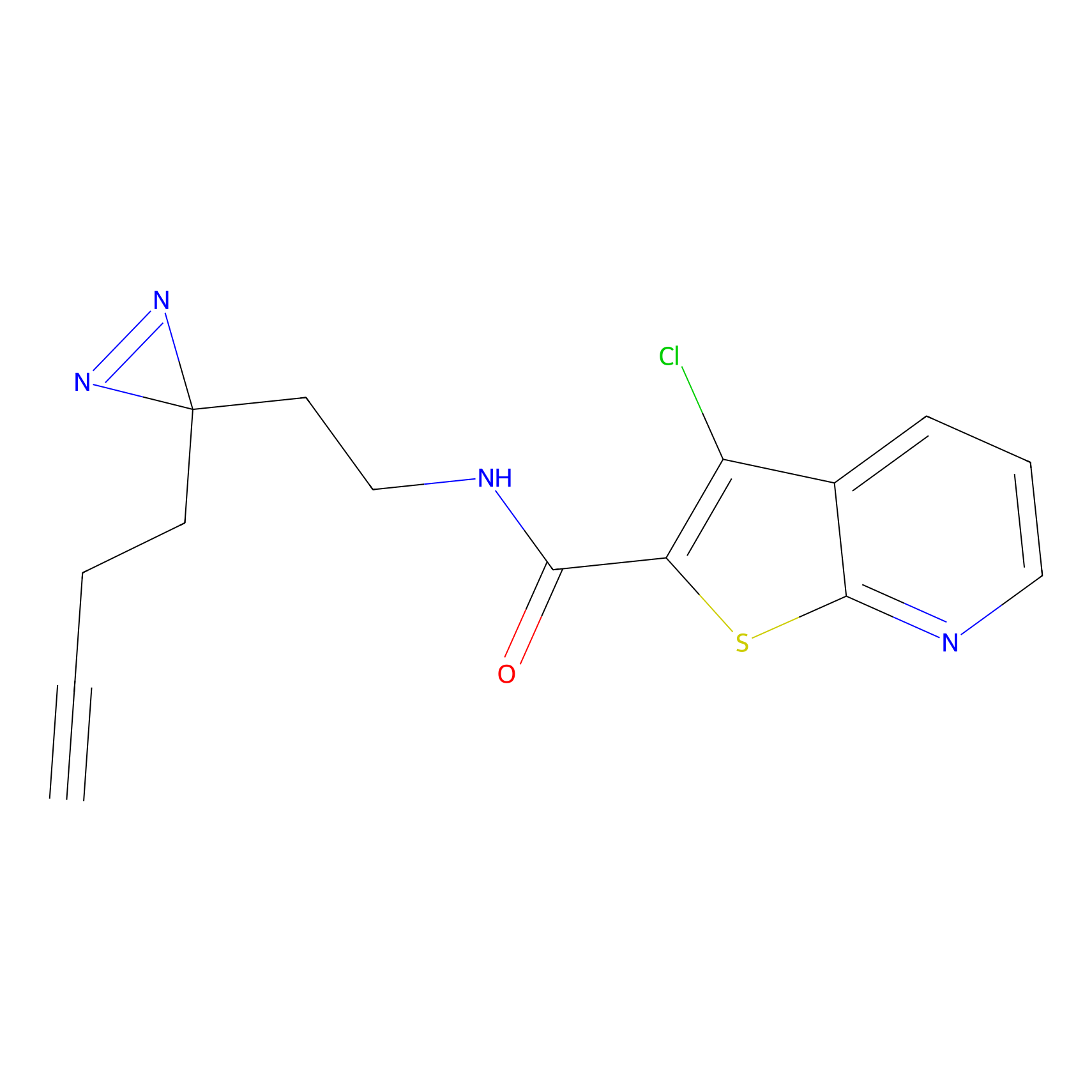
EA-probe Probe Info |
 |
N.A. | LDD0440 | [1] | |
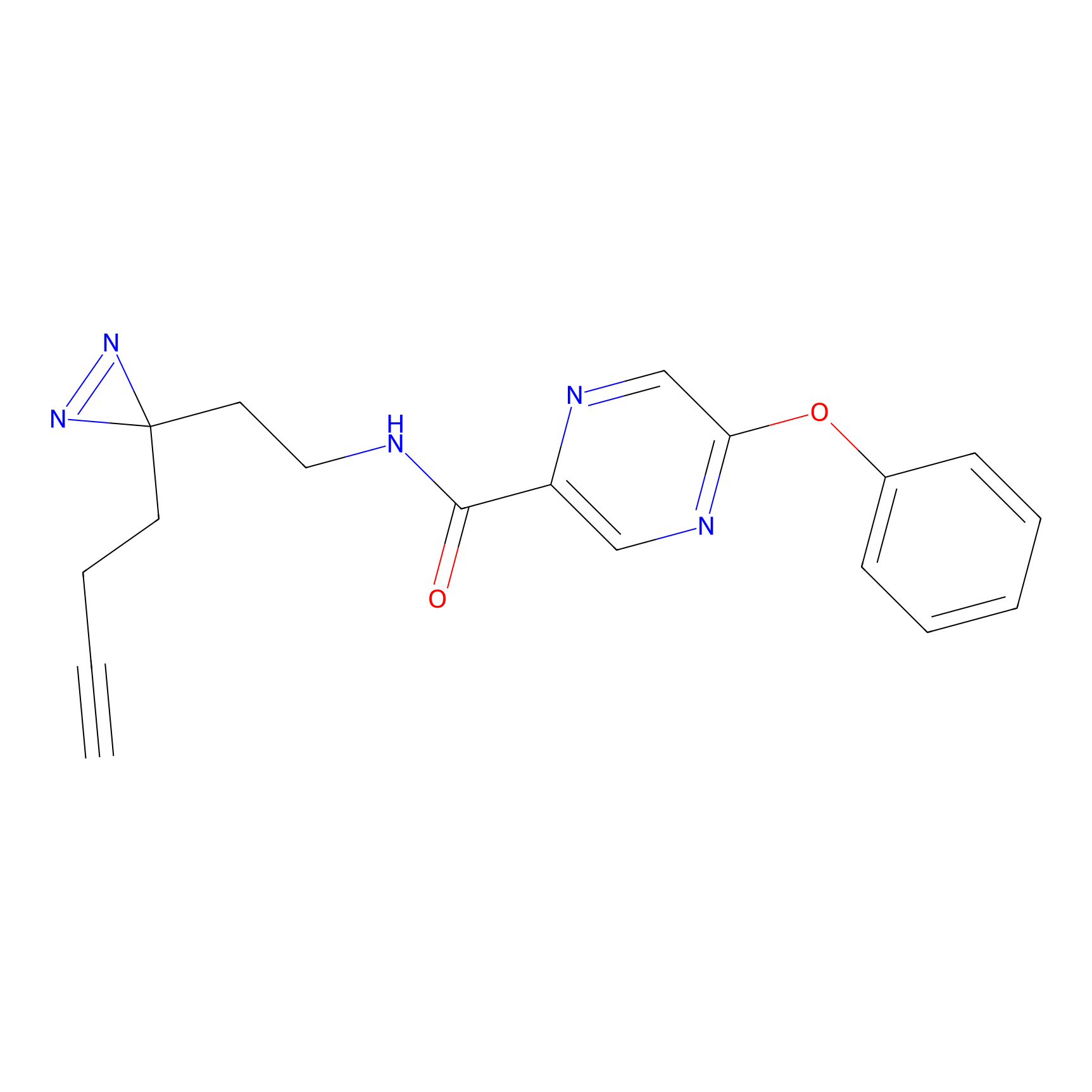
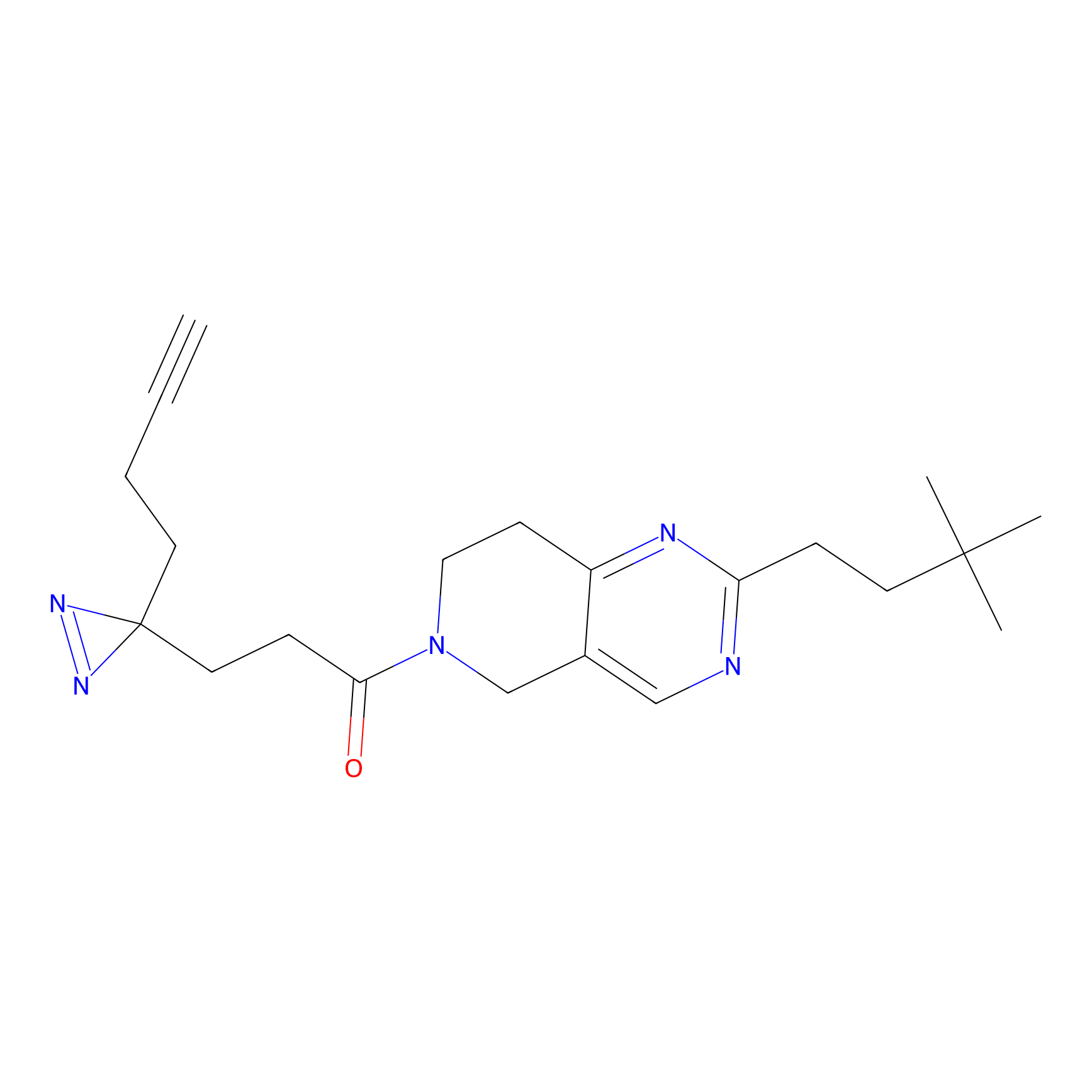
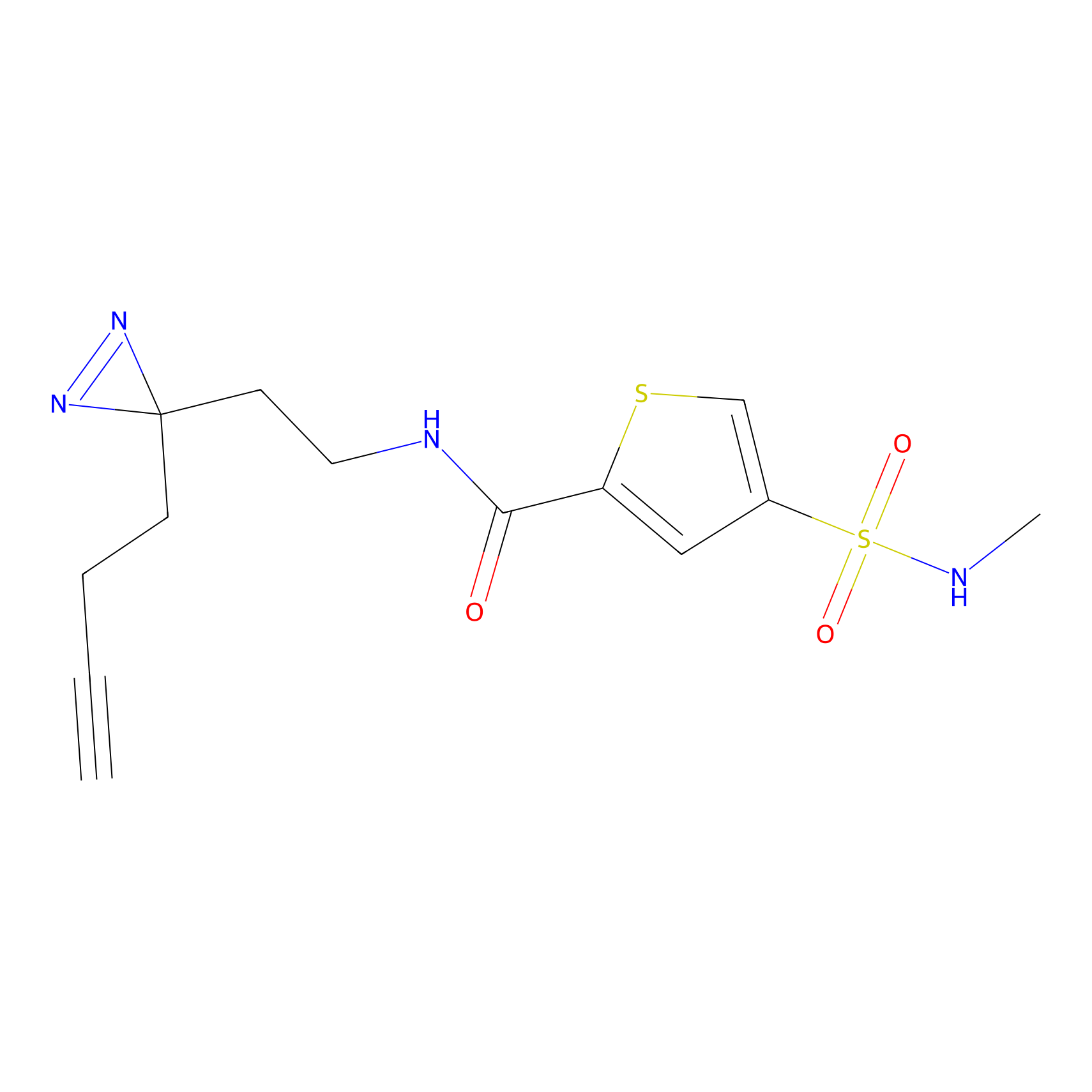
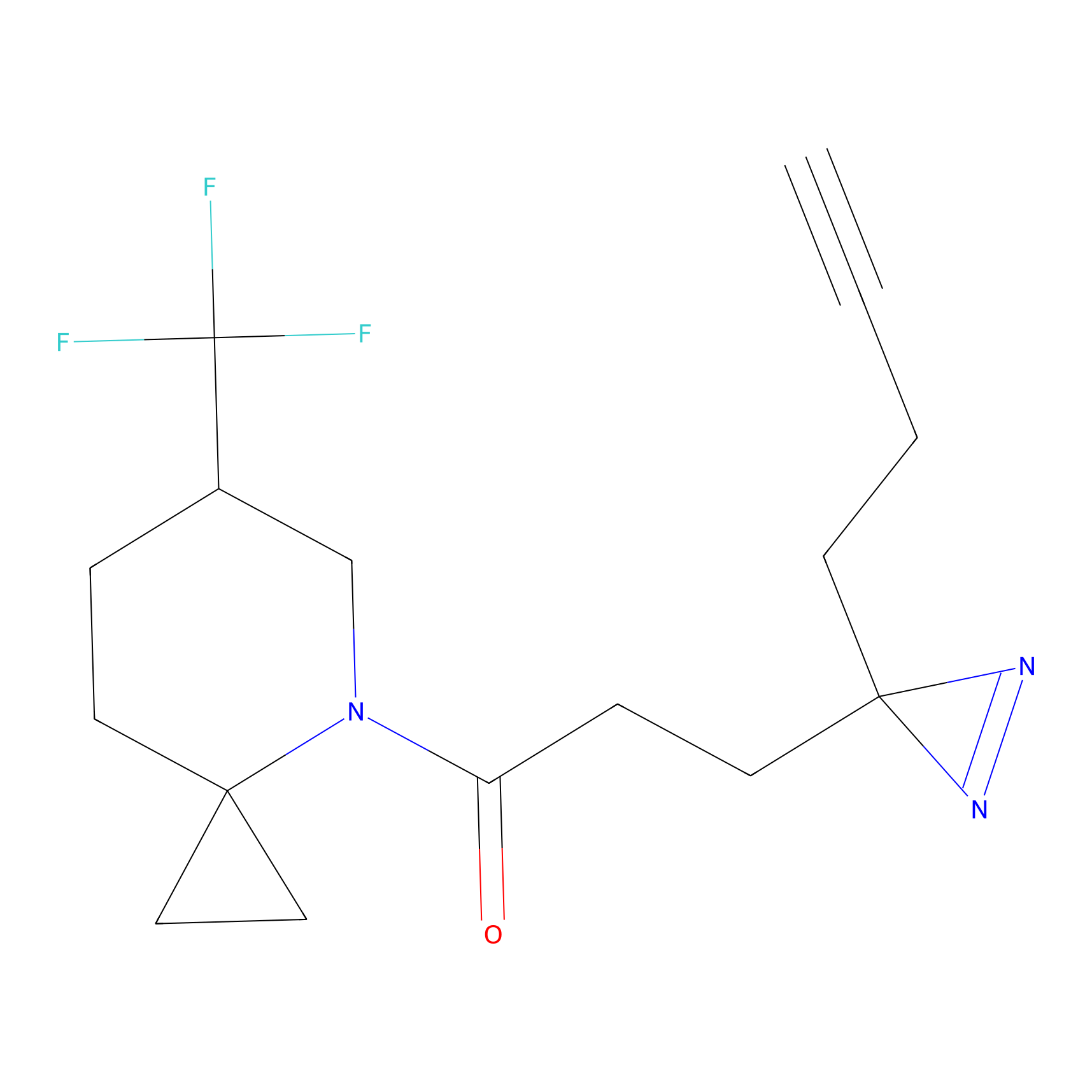
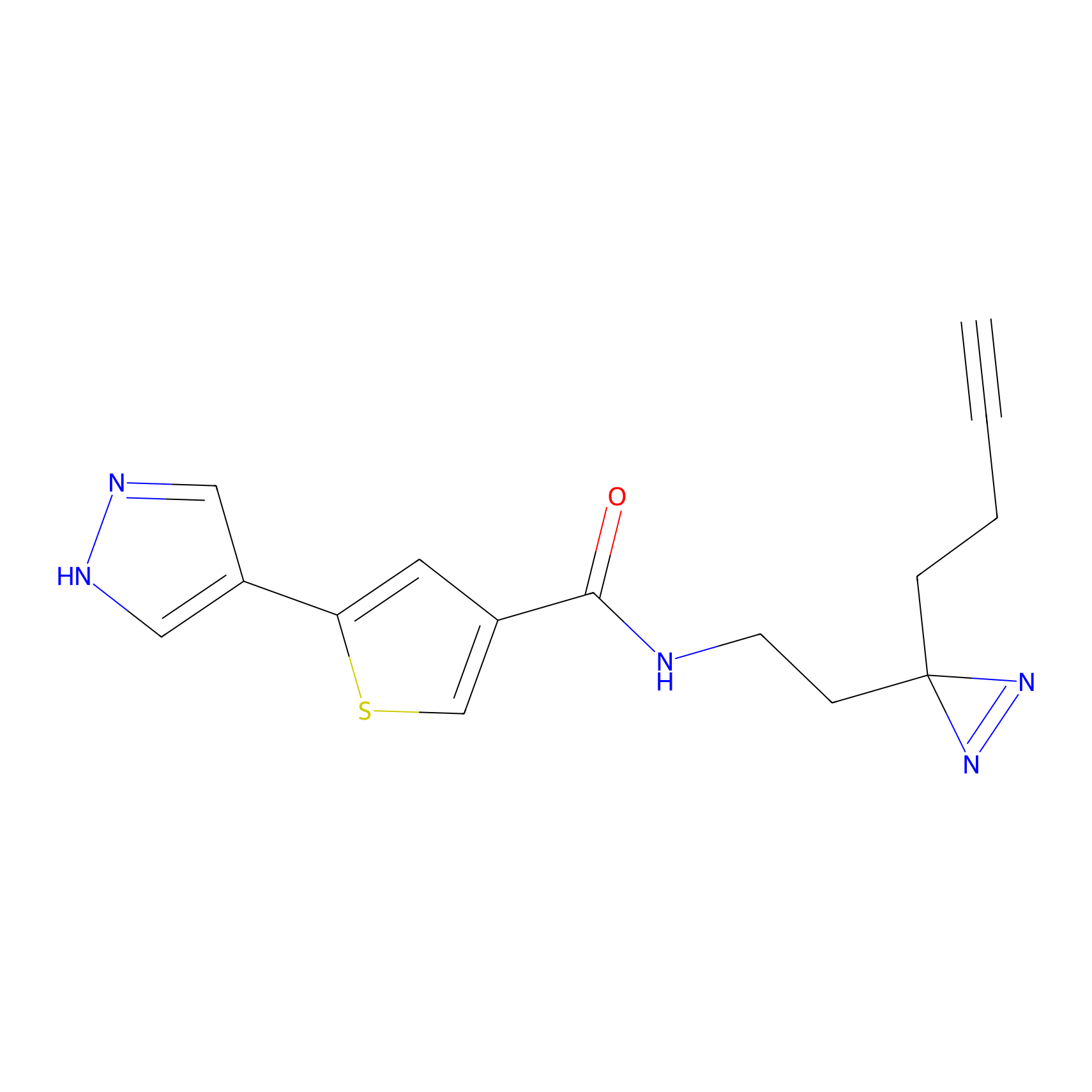
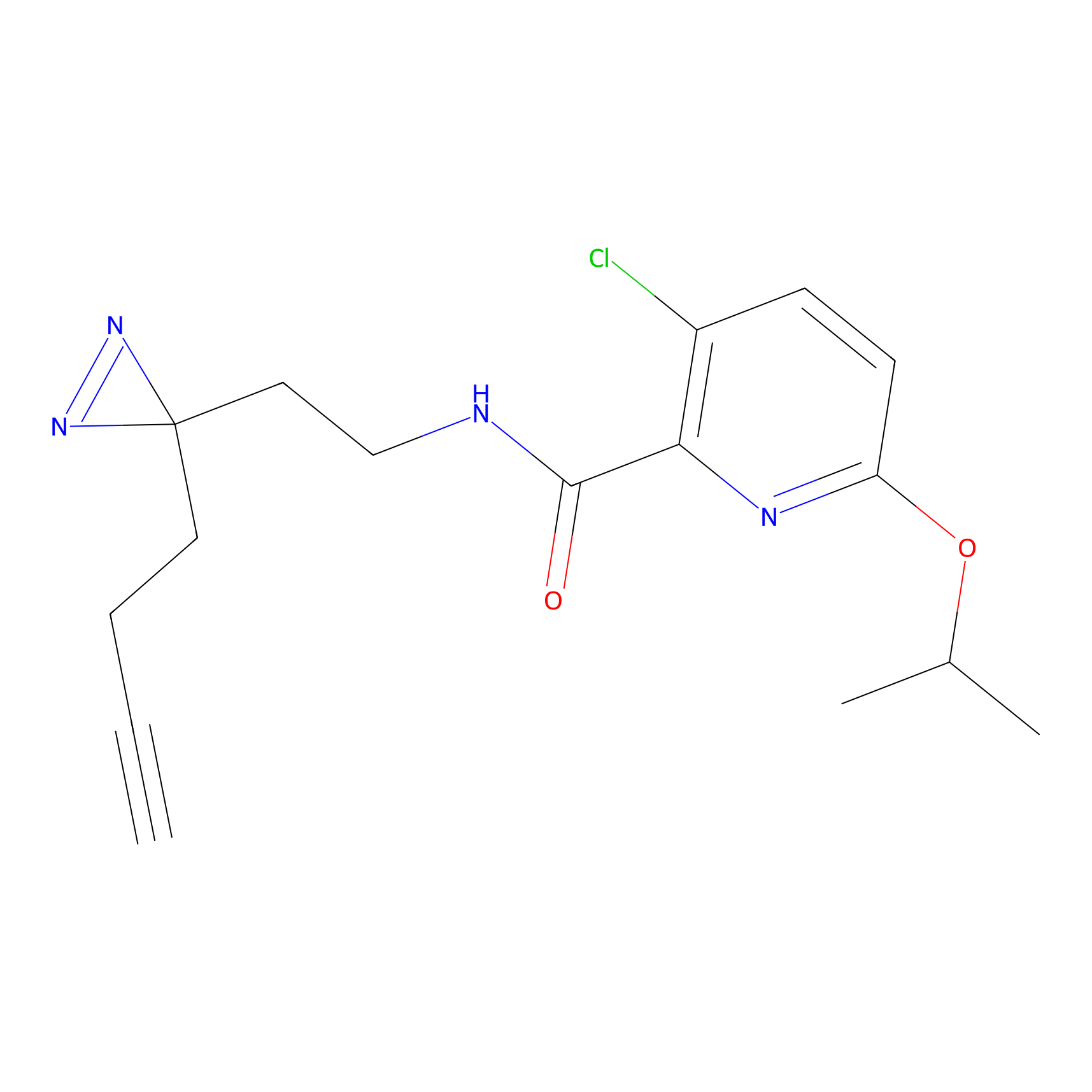
|
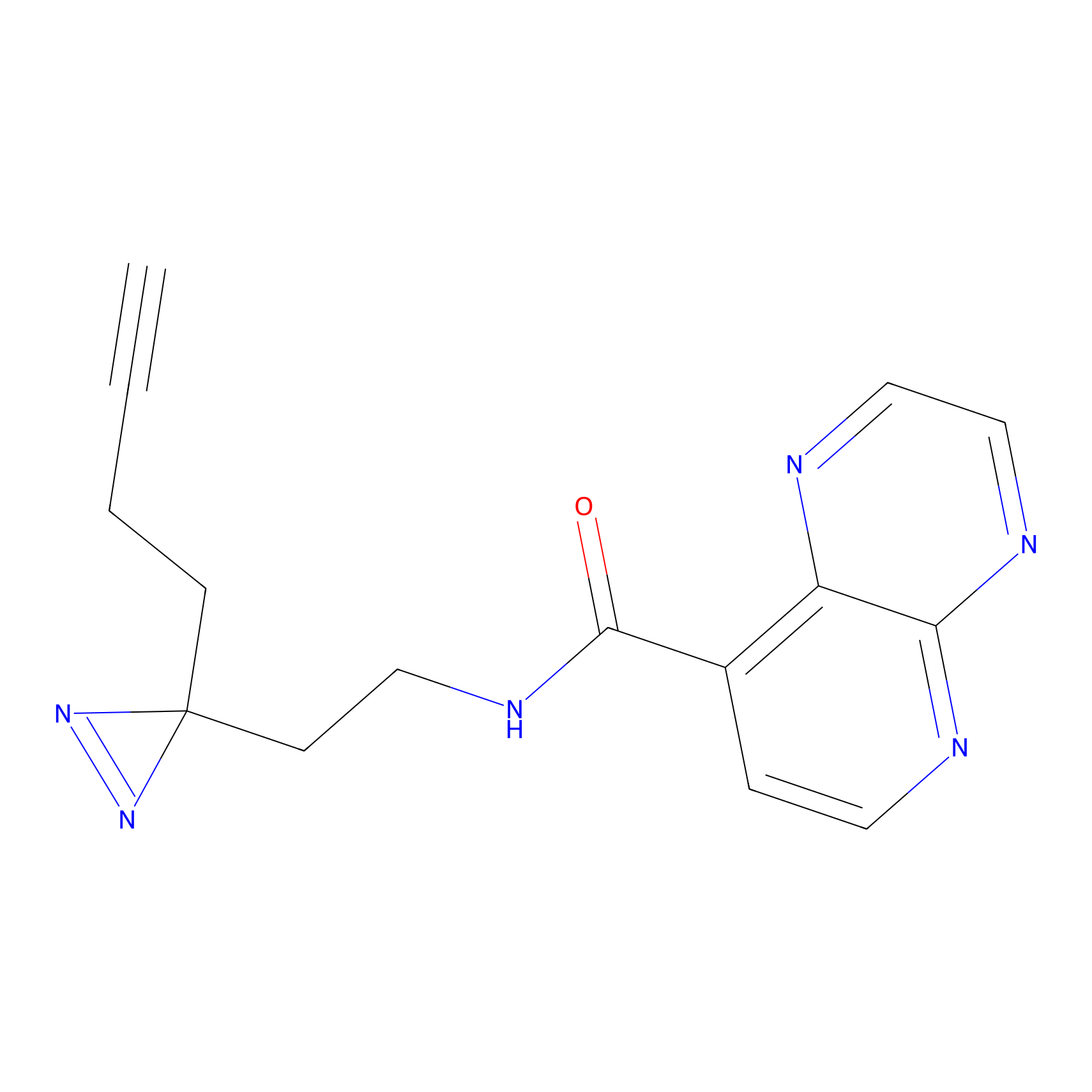
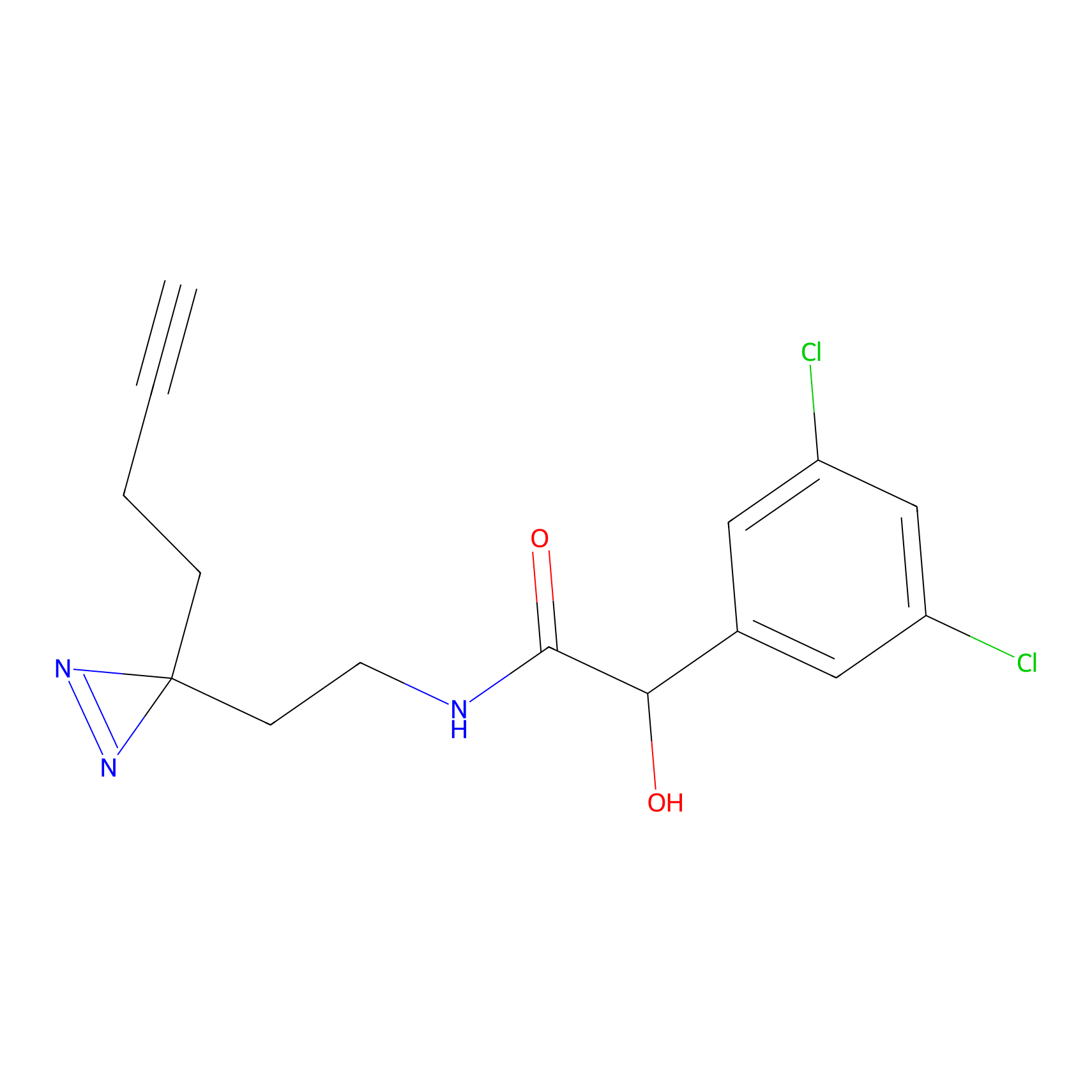
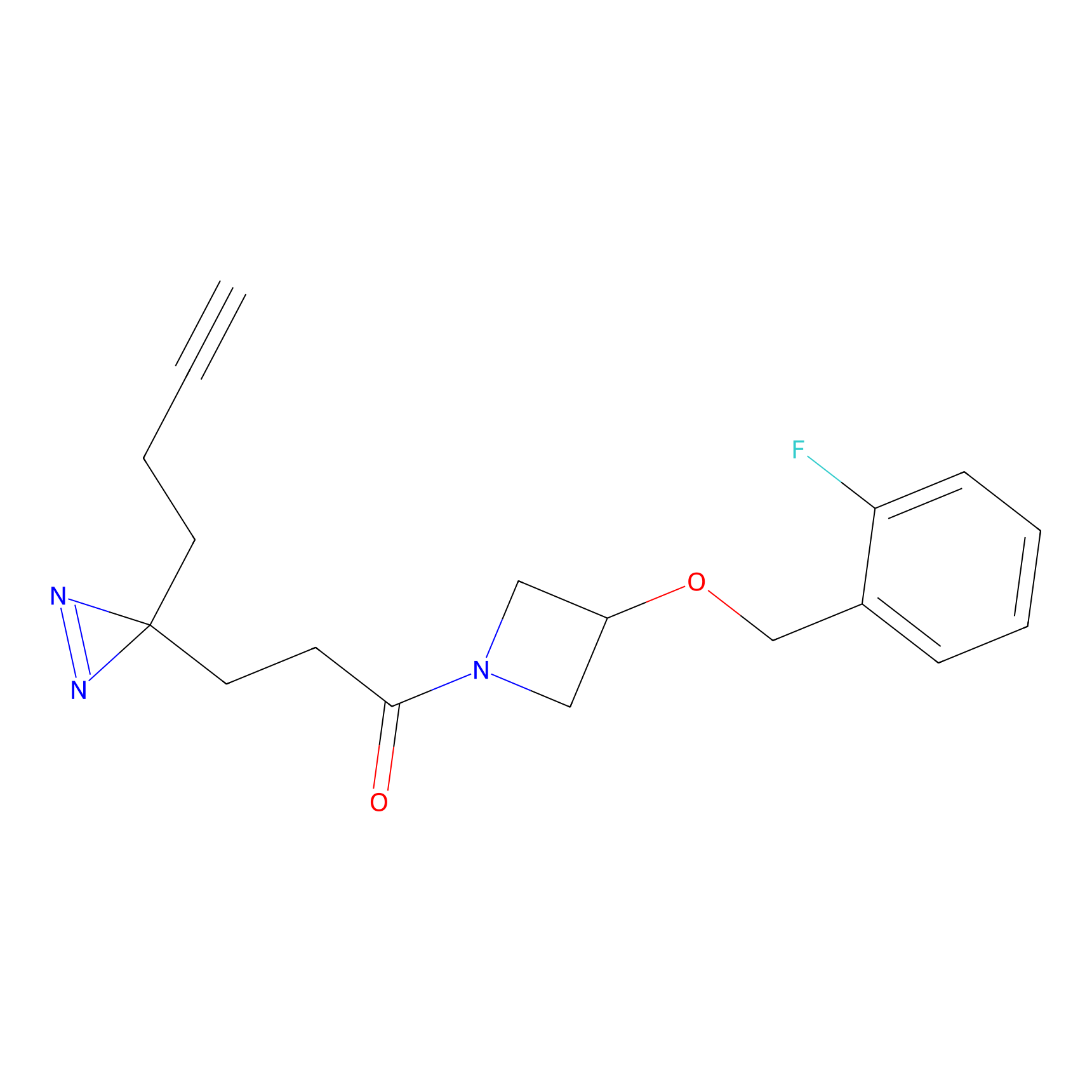
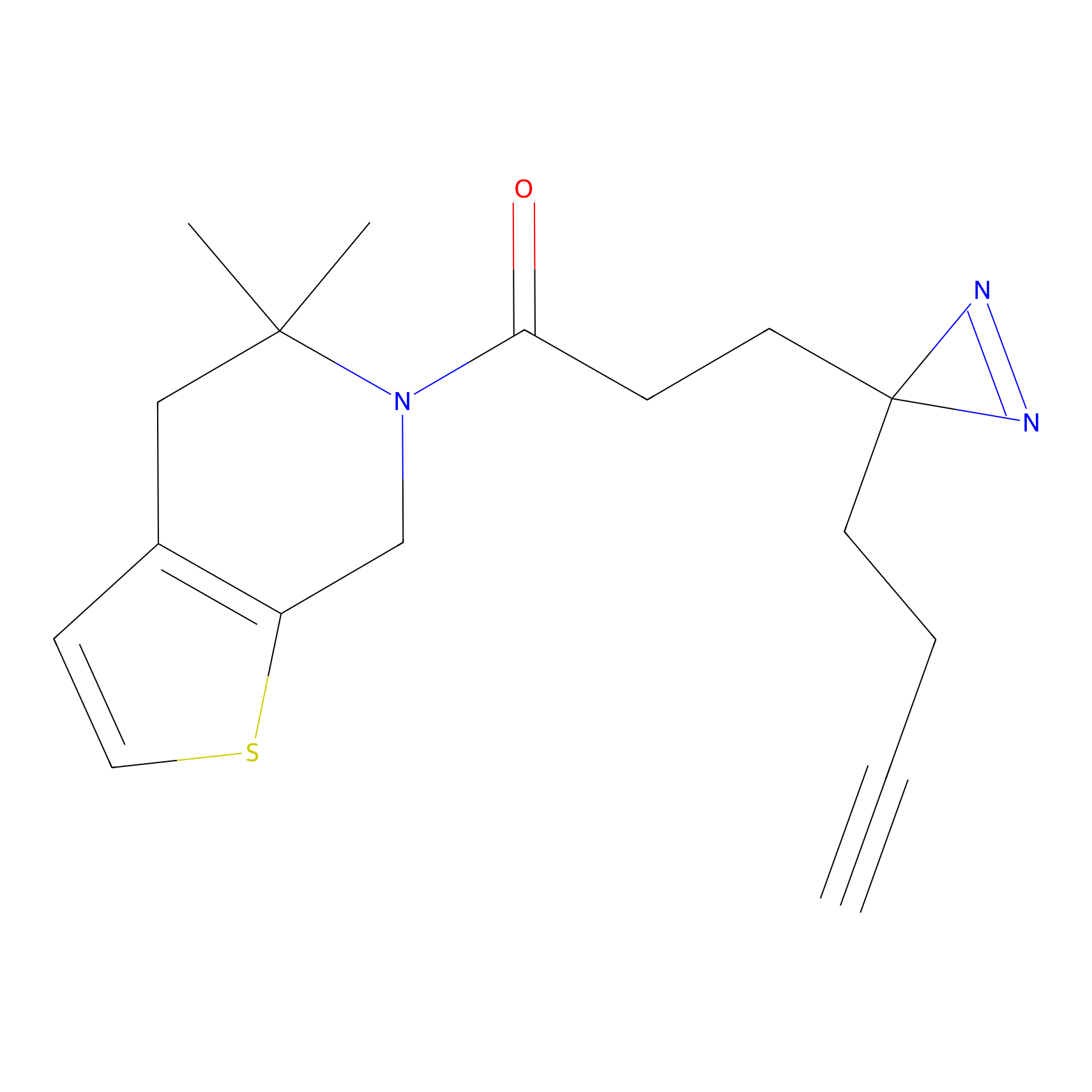
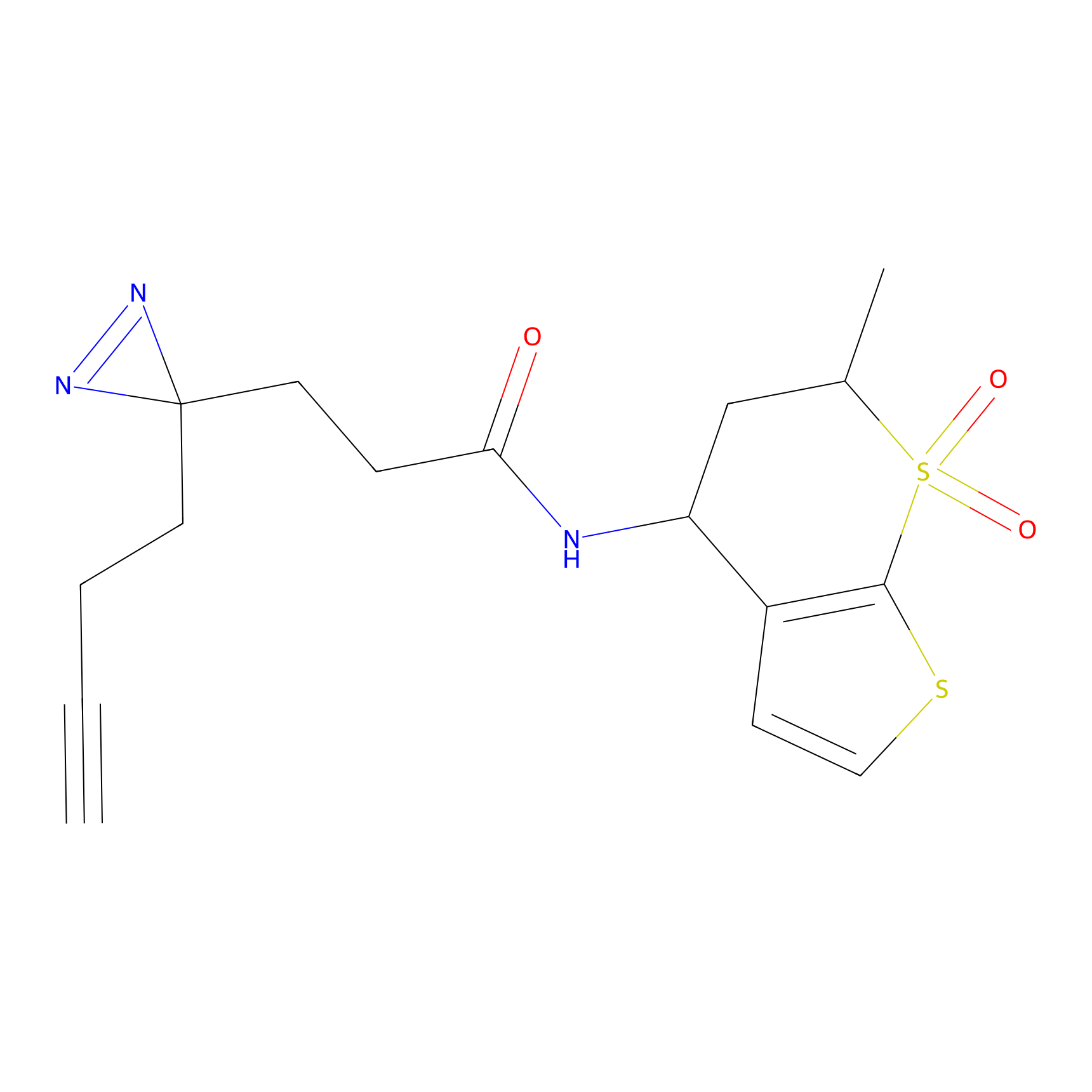
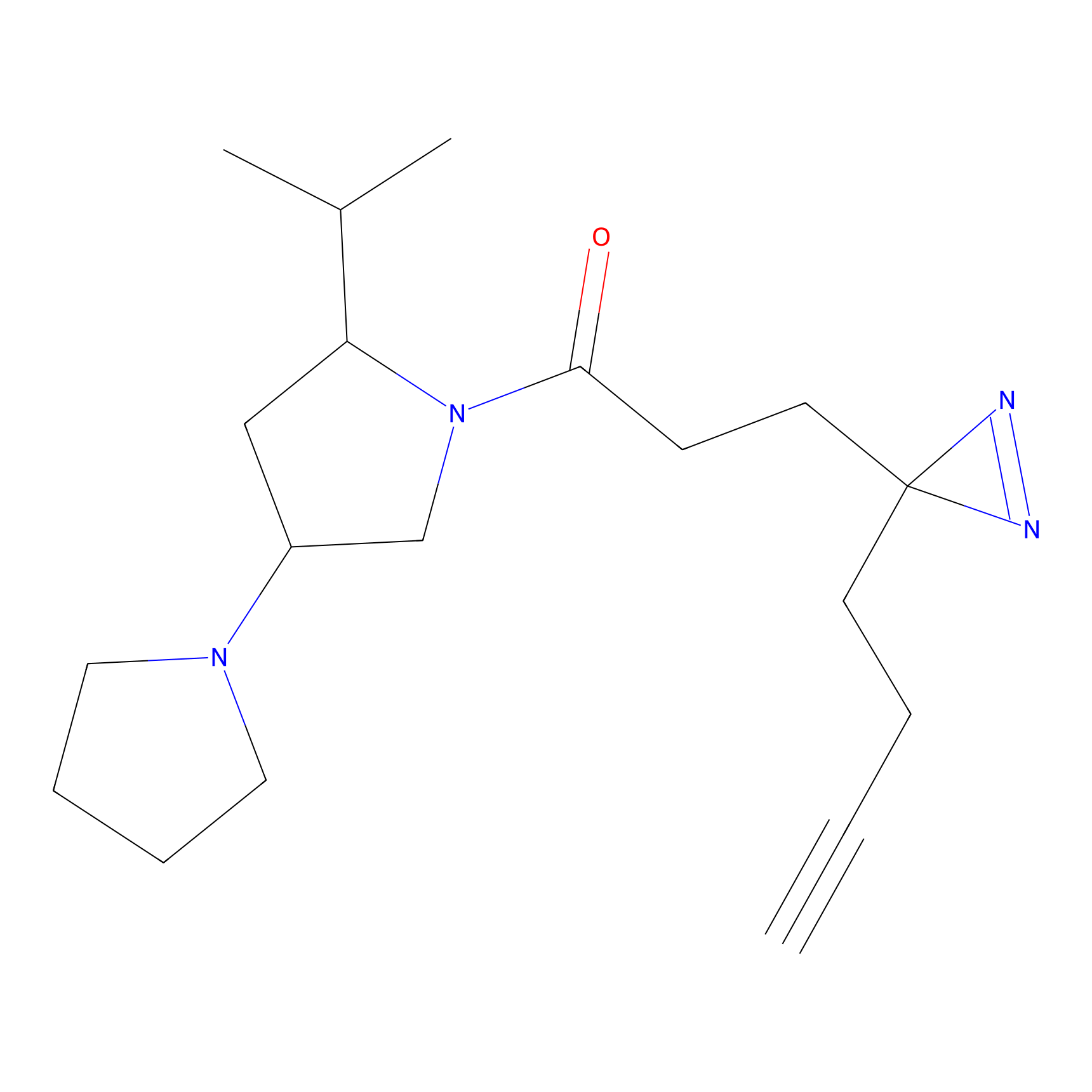
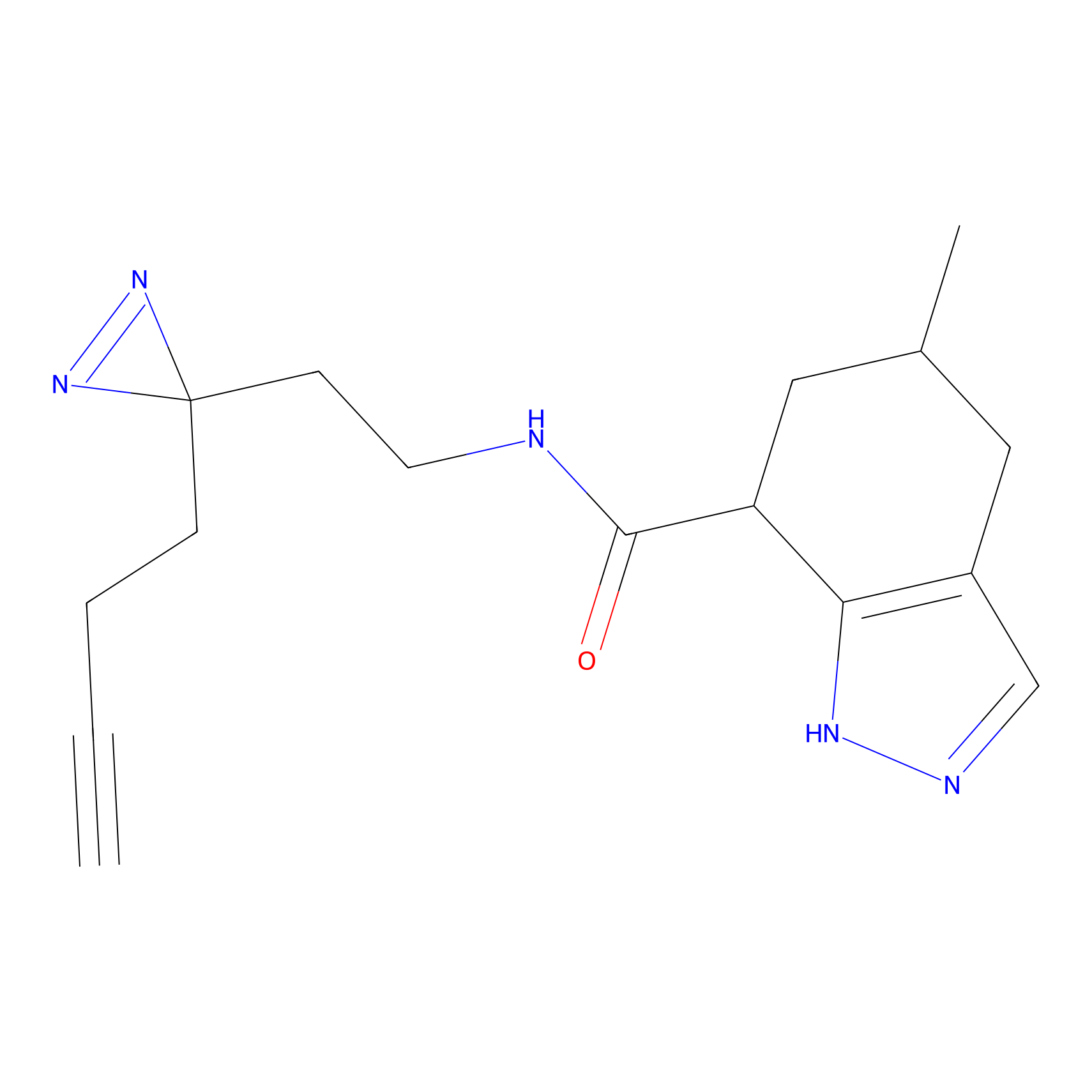
DBIA Probe Info |
 |
C68(0.90) | LDD1492 | [2] | |
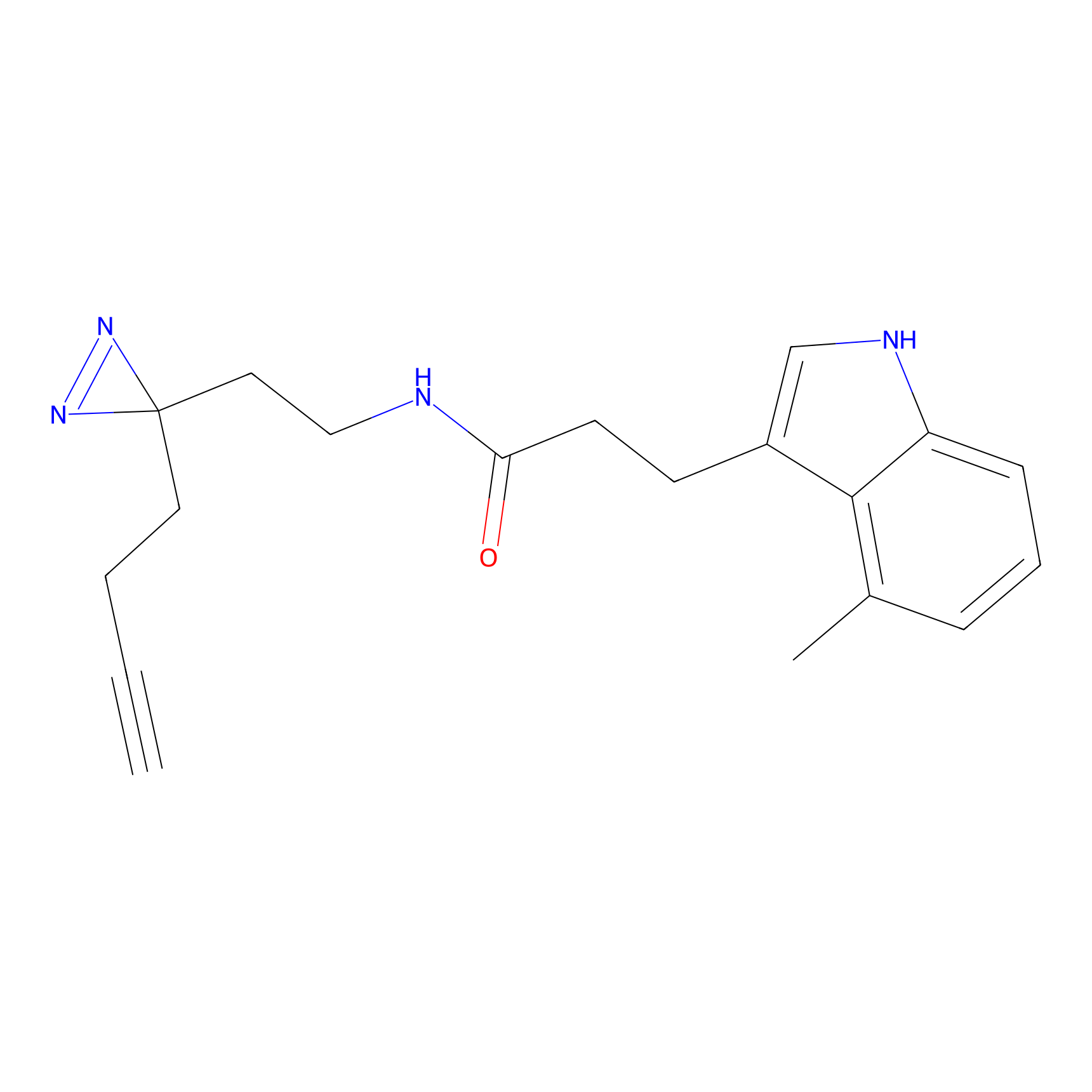
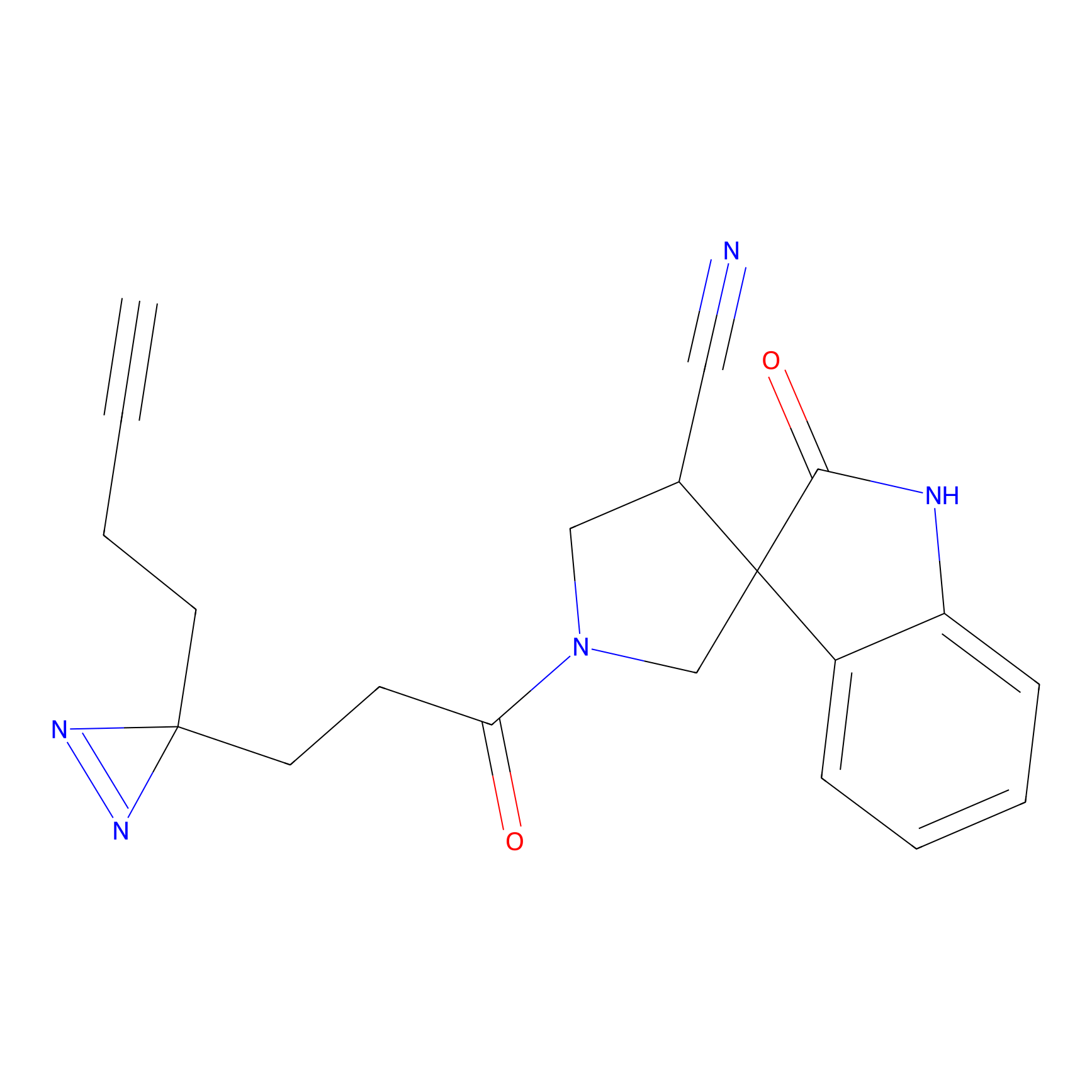
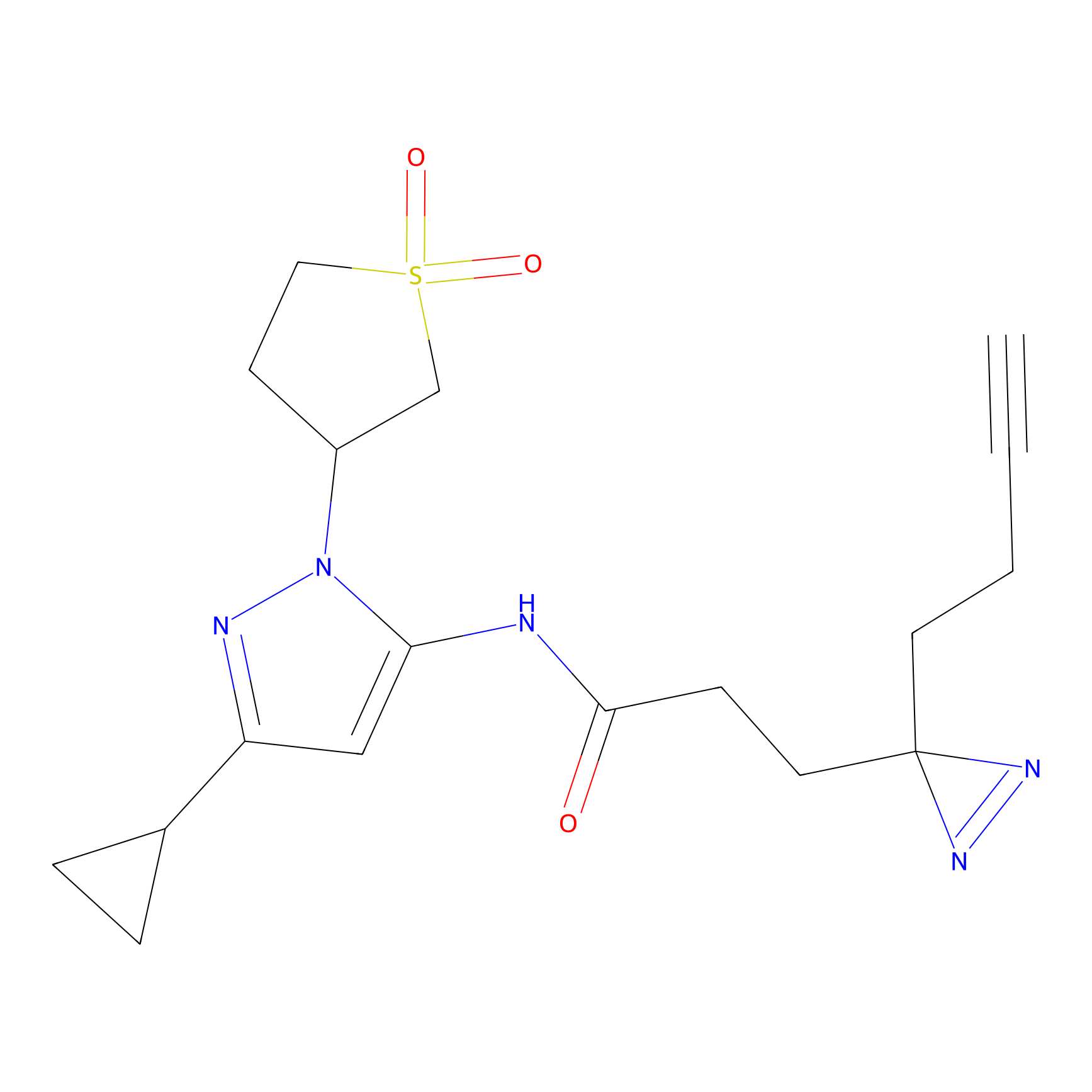
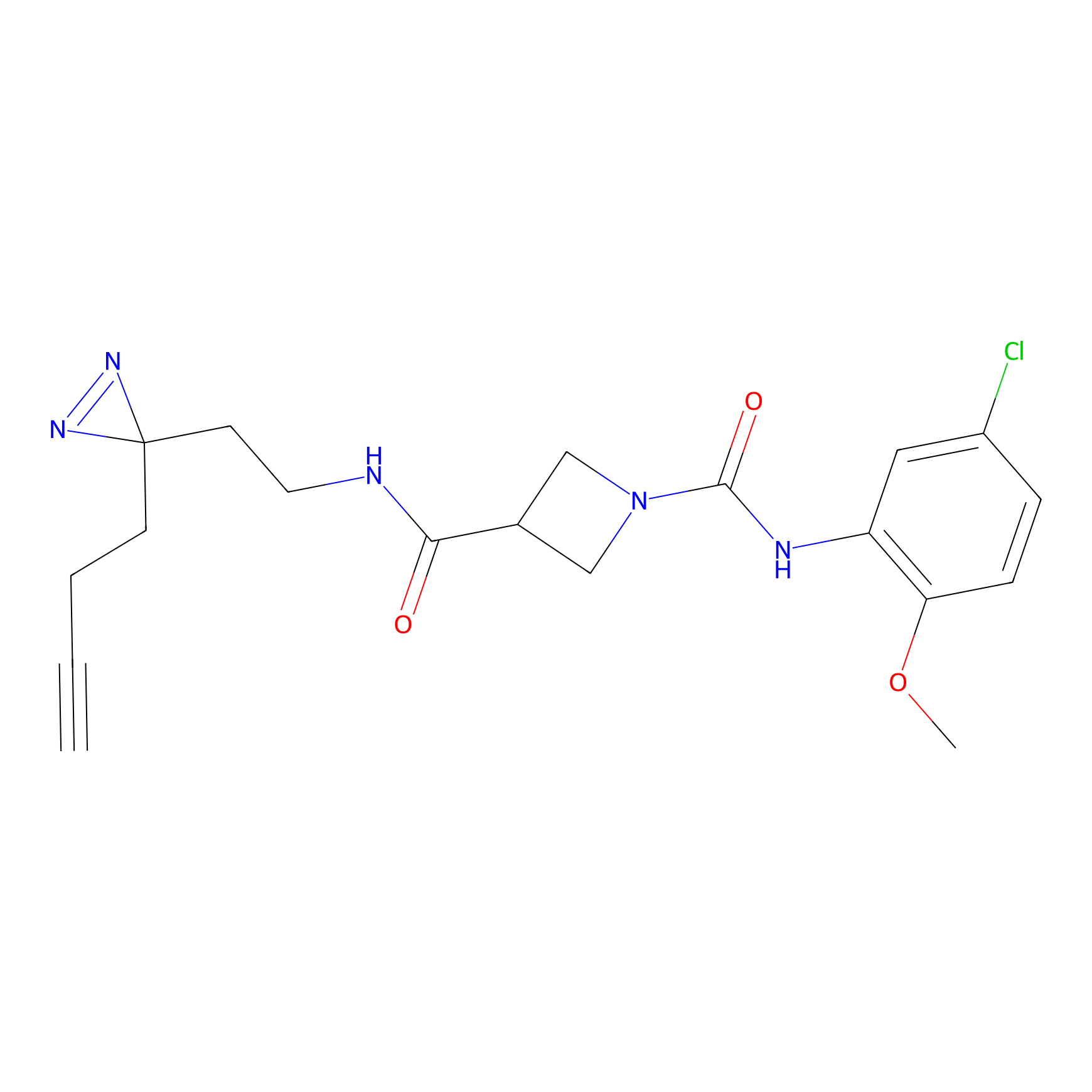
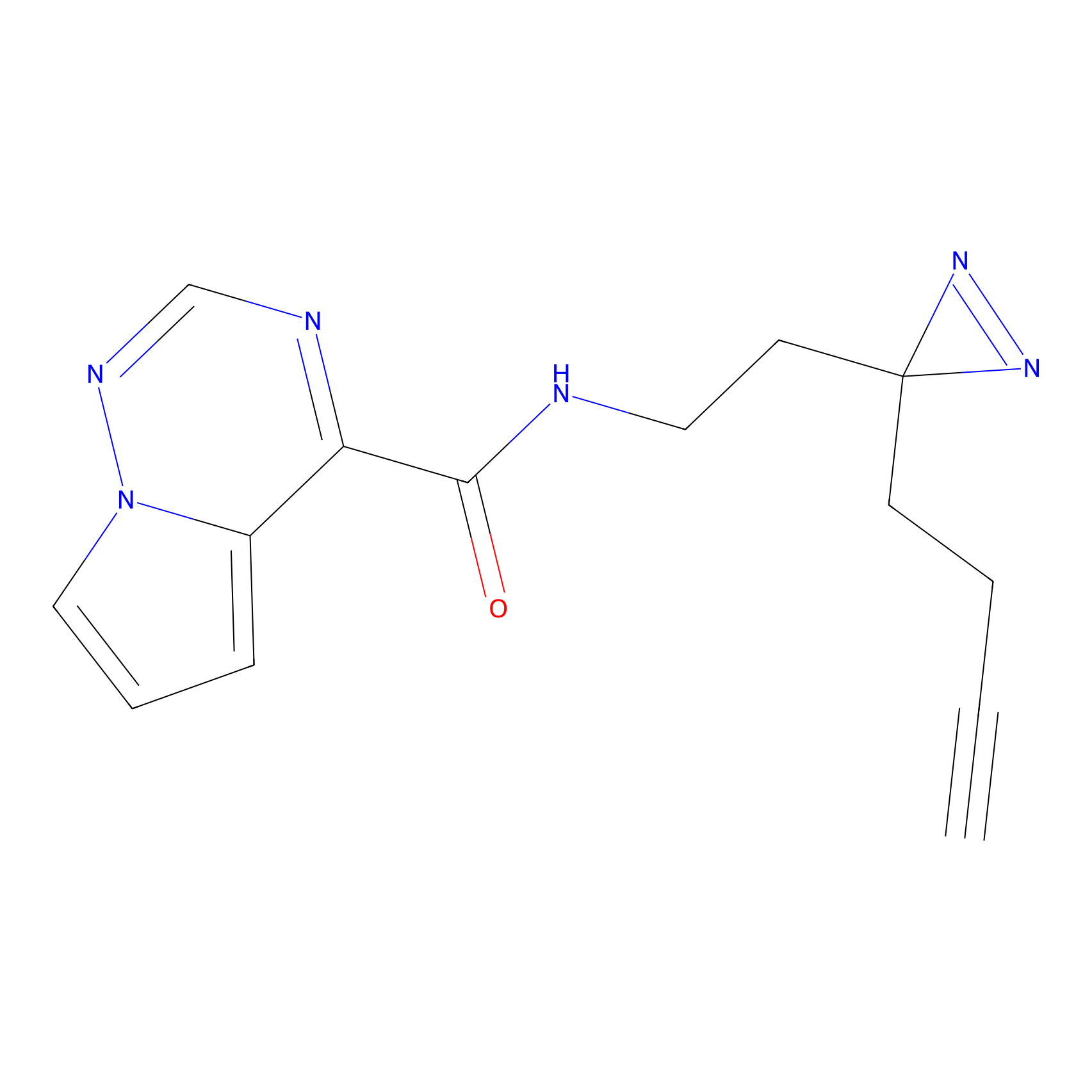
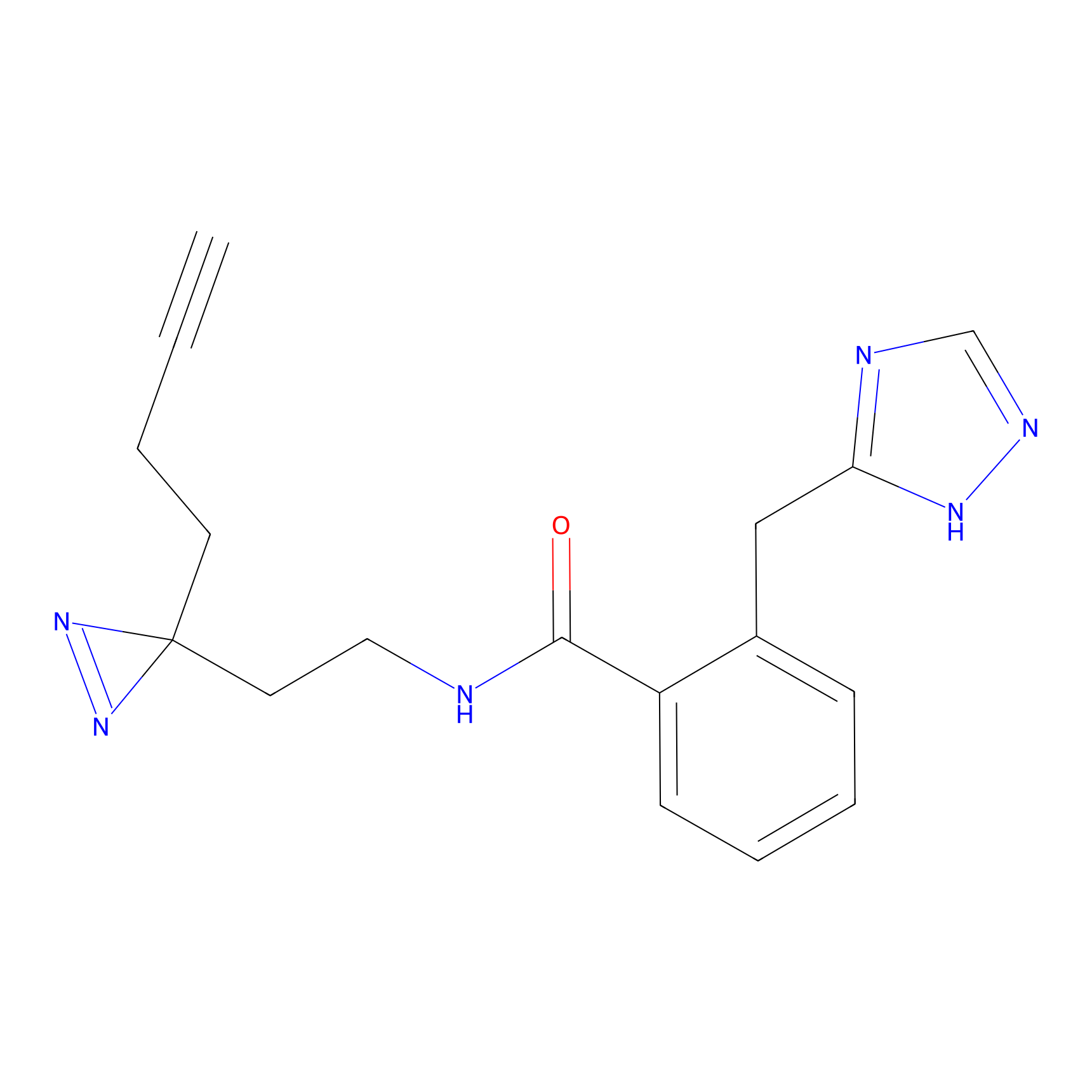
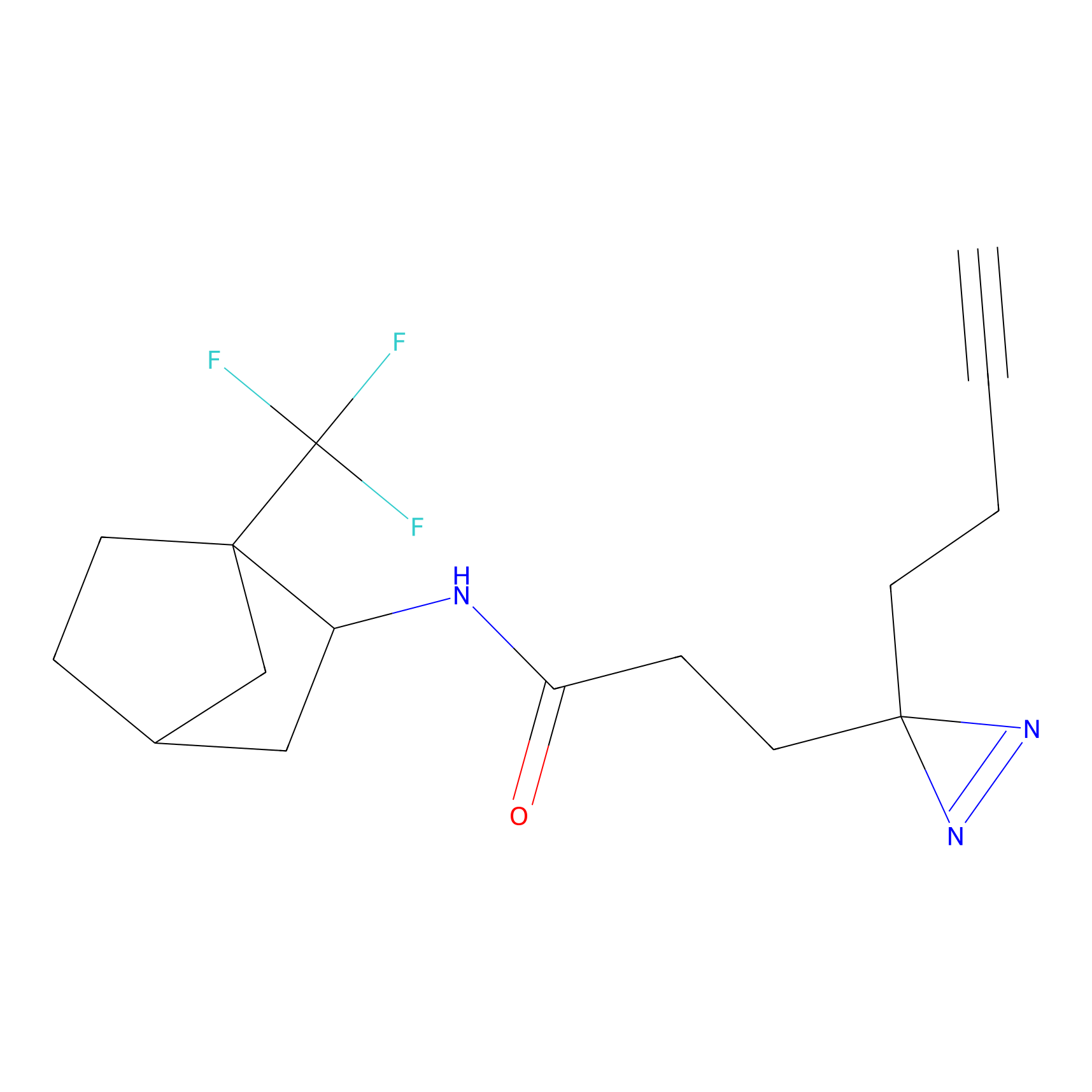
|
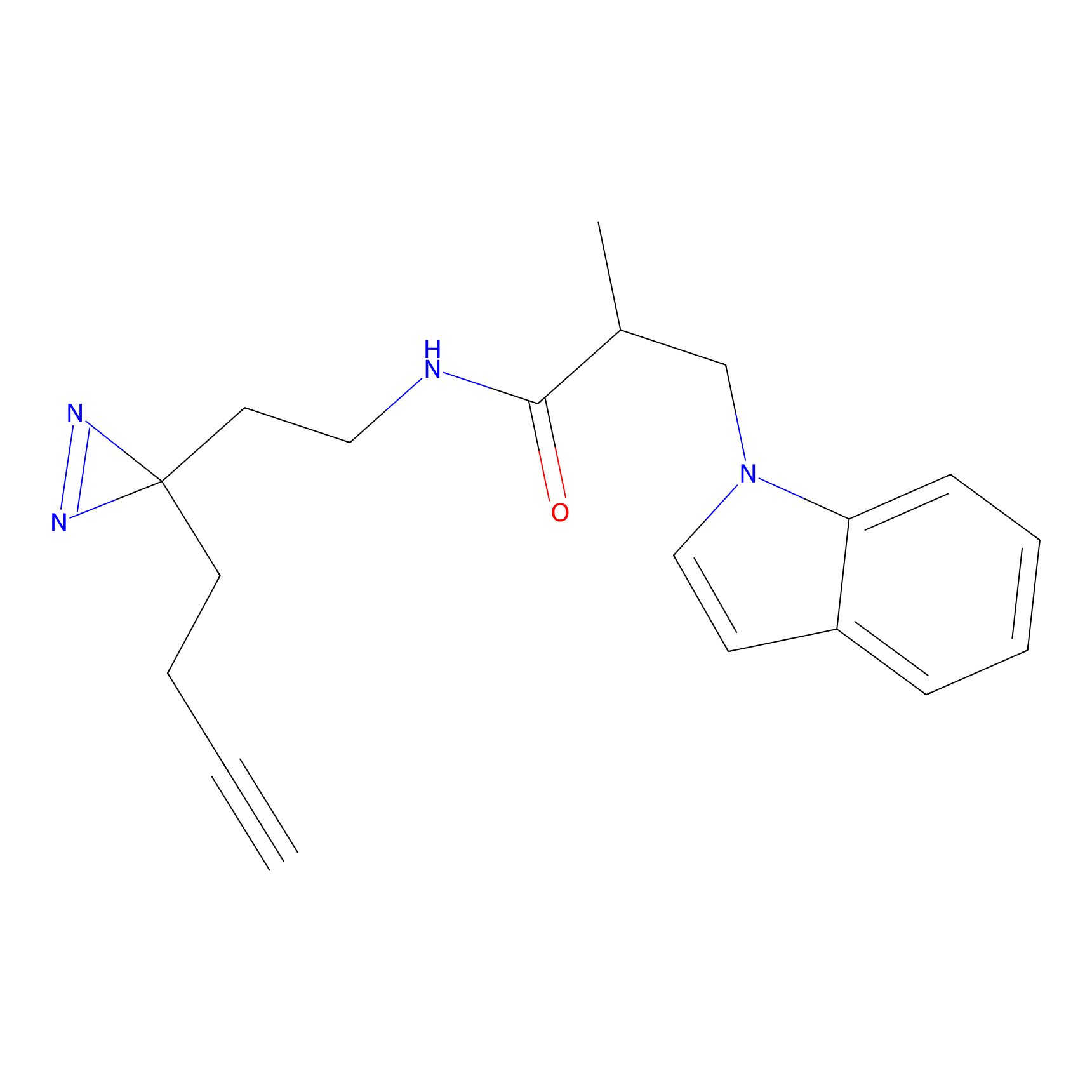
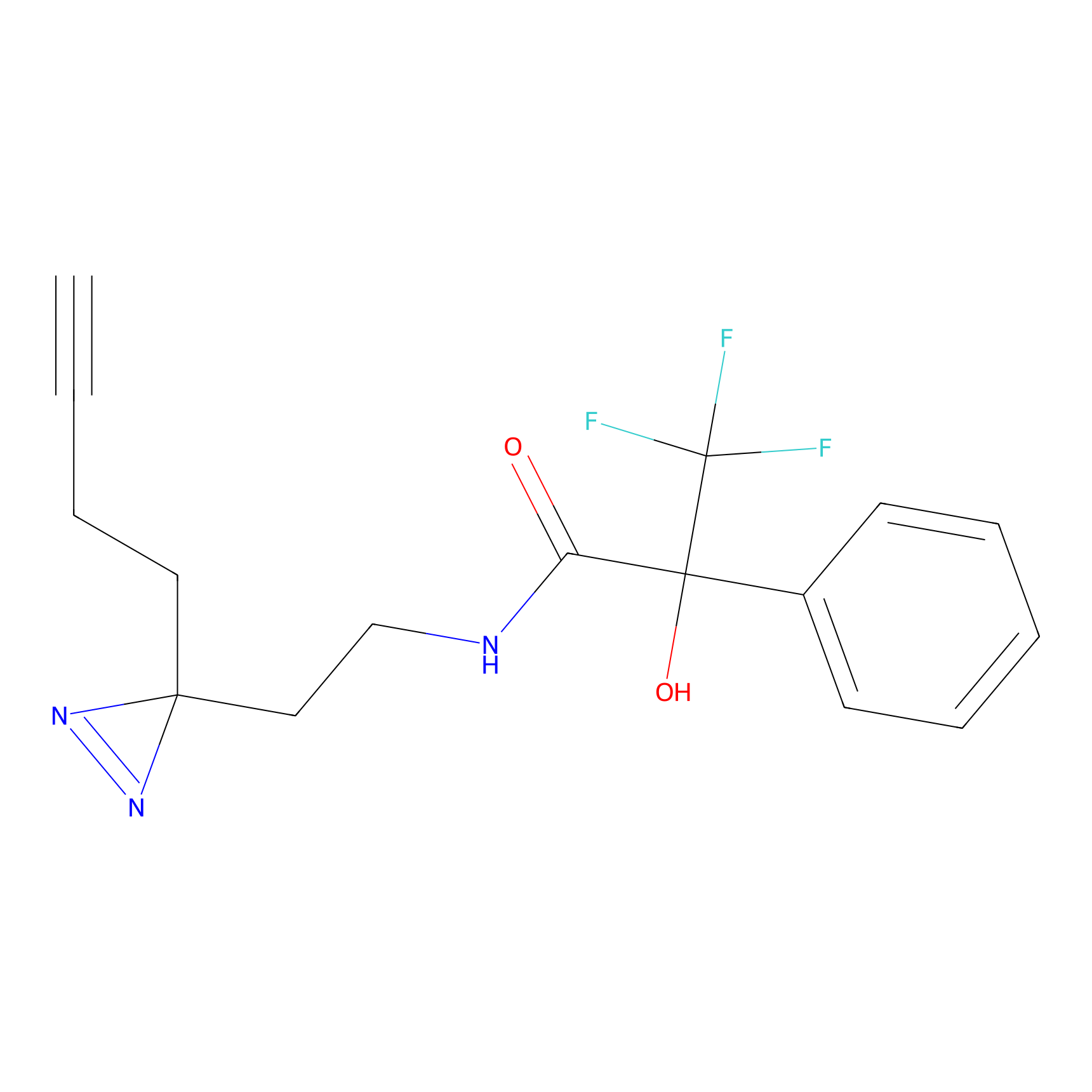
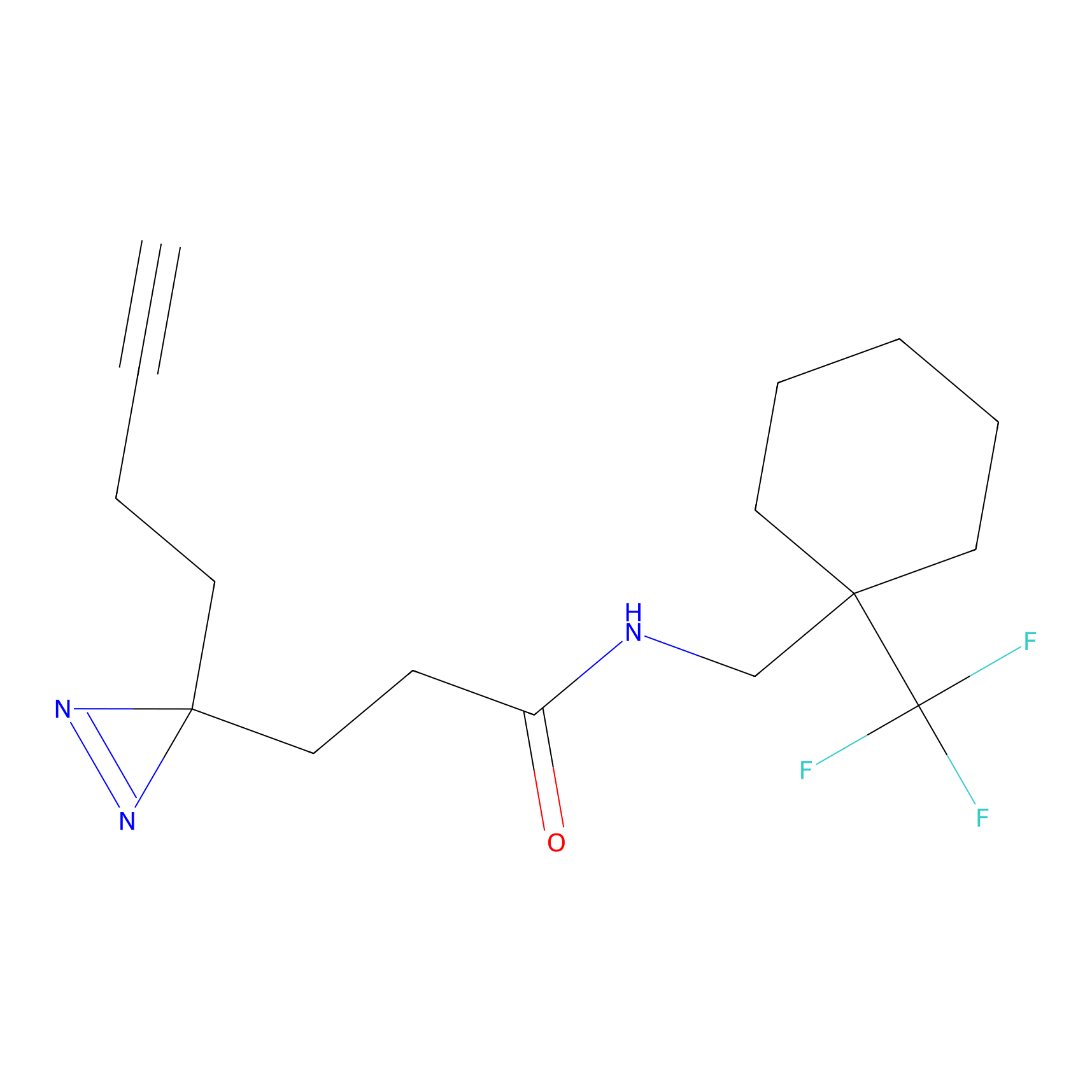
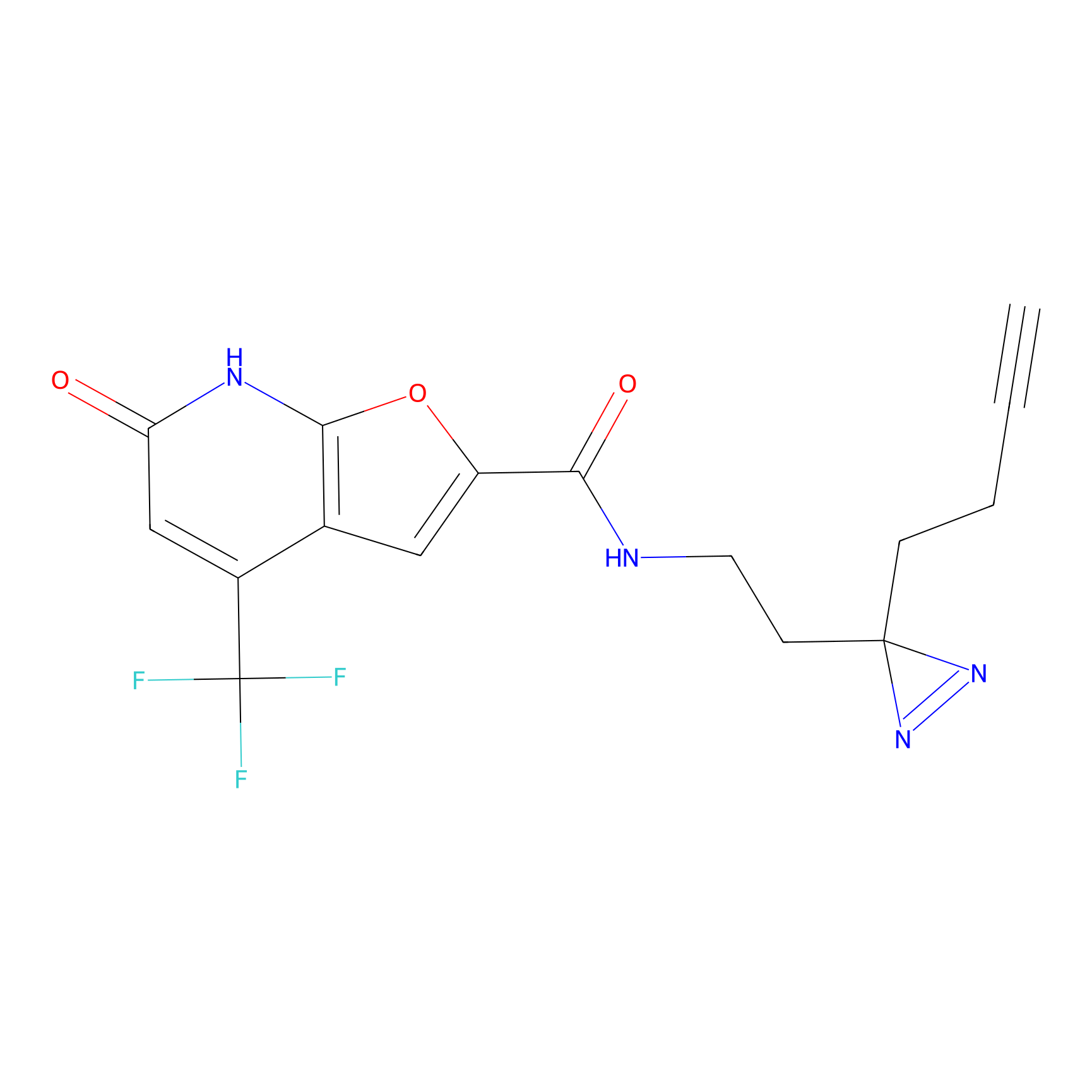
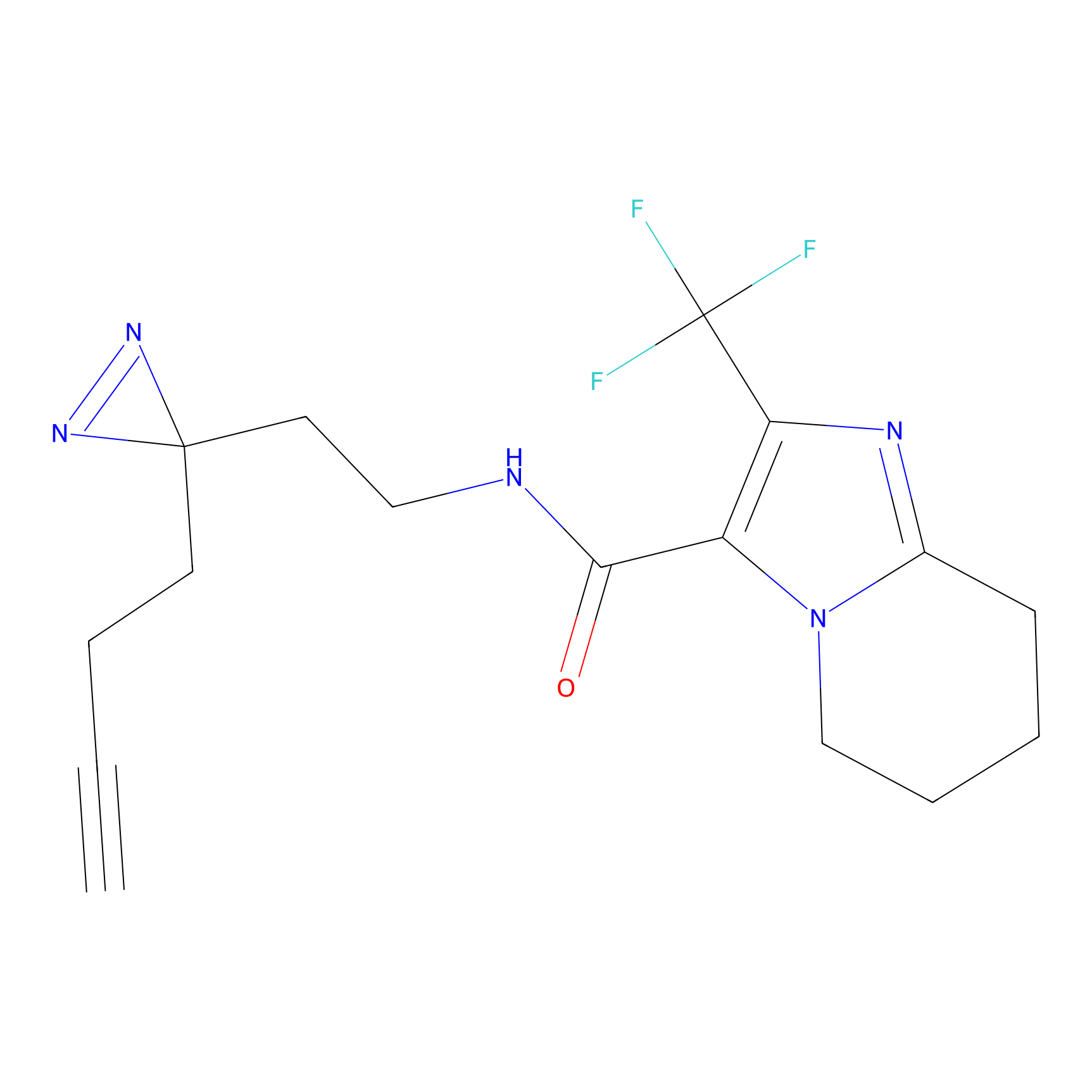
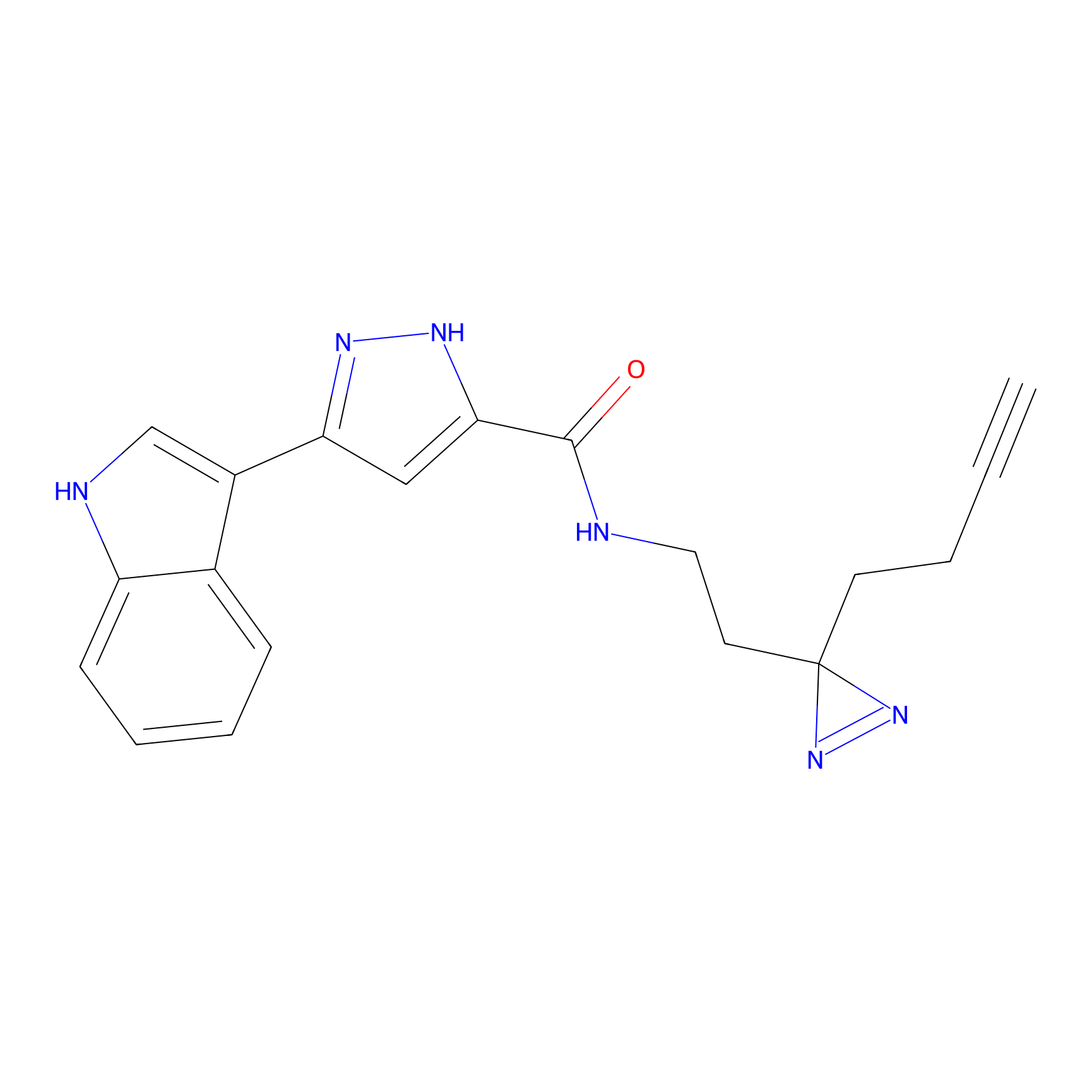
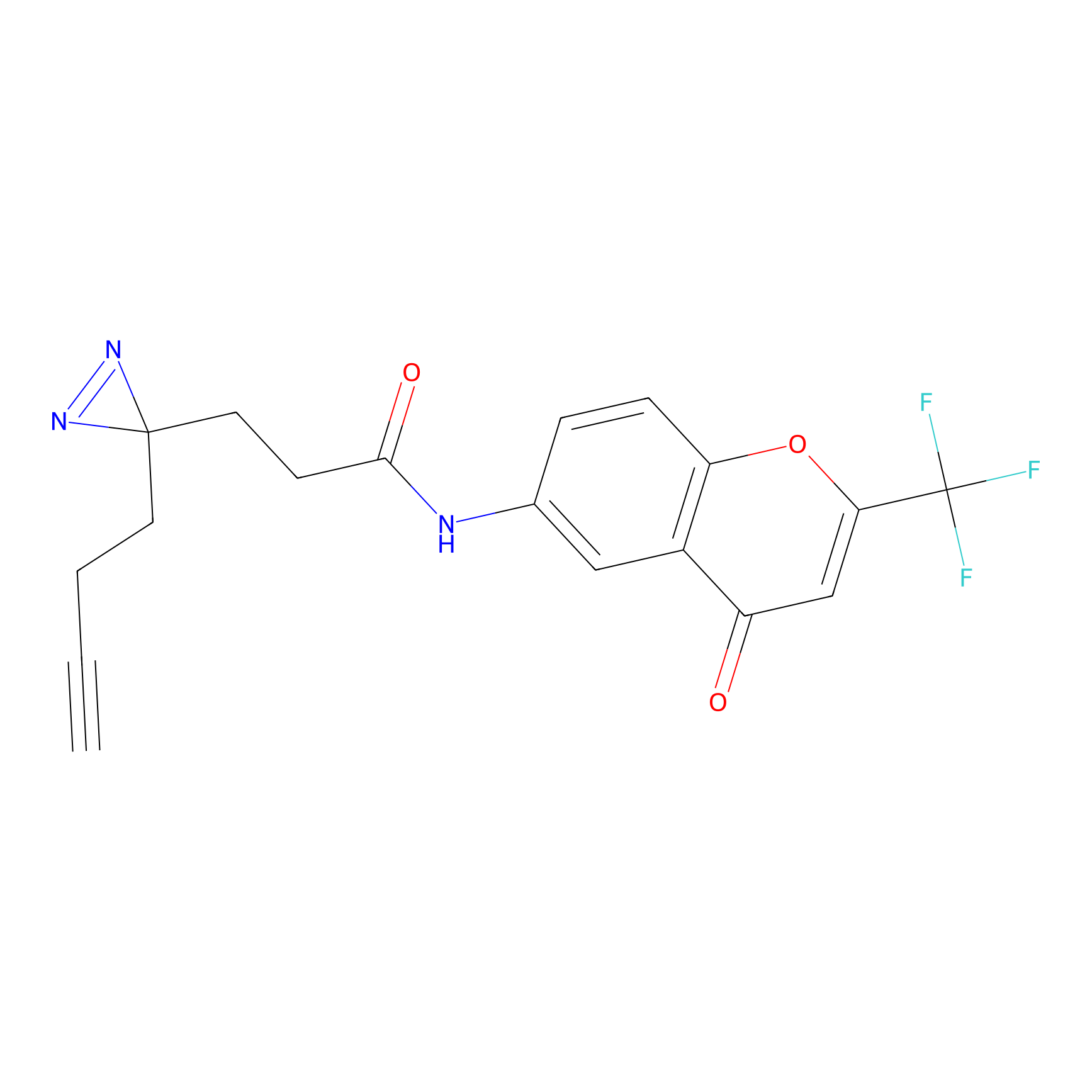
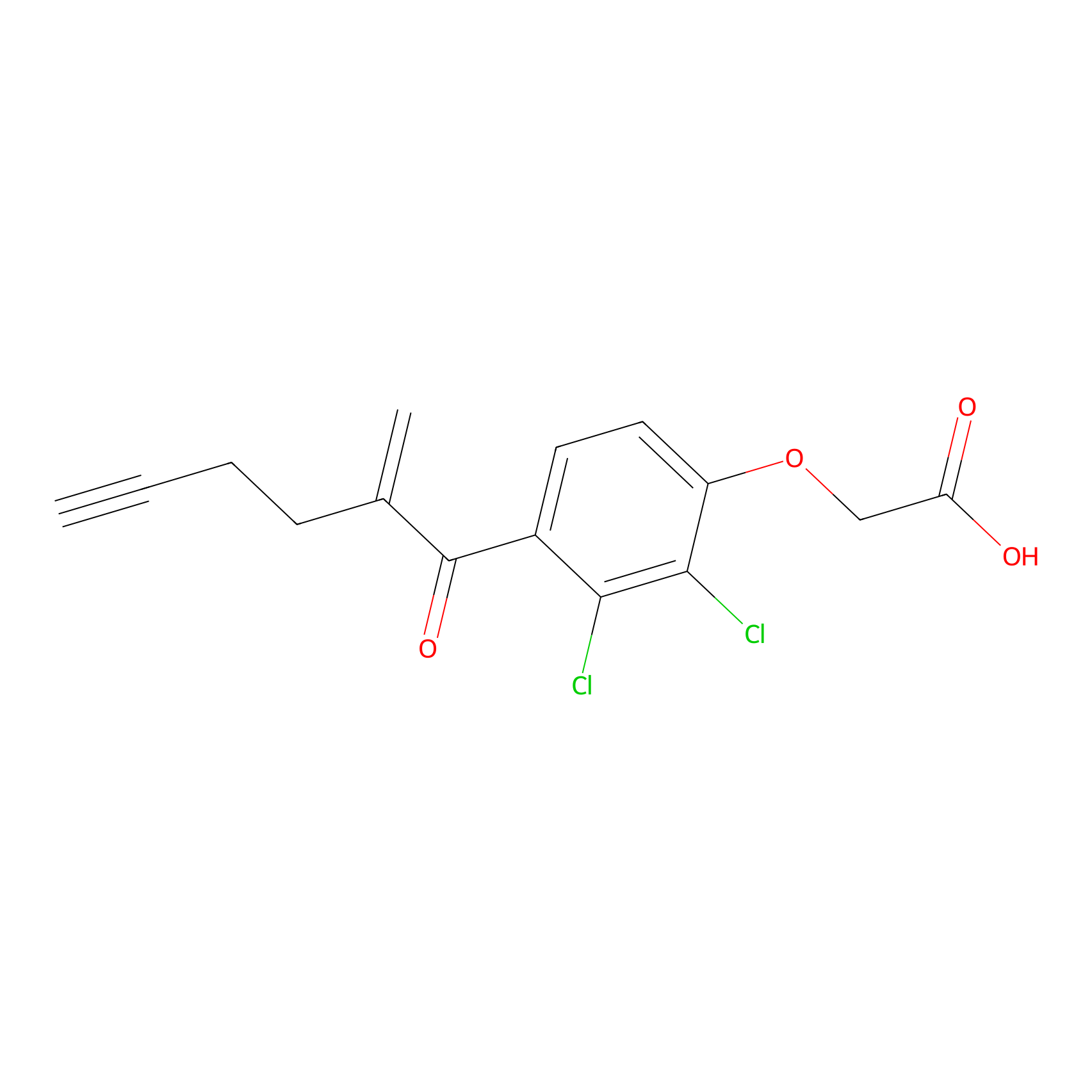
4-Iodoacetamidophenylacetylene Probe Info |
 |
N.A. | LDD0038 | [3] | |
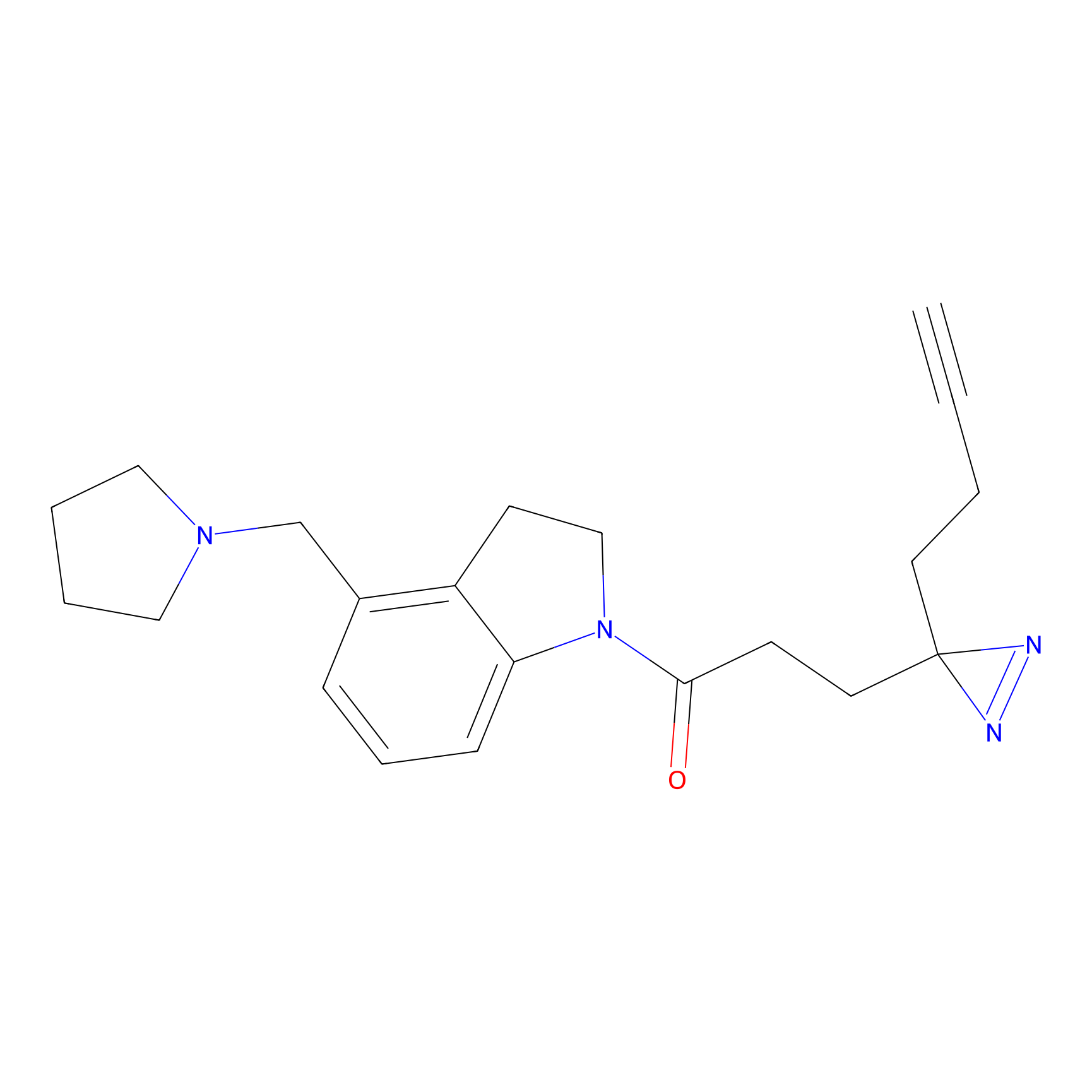
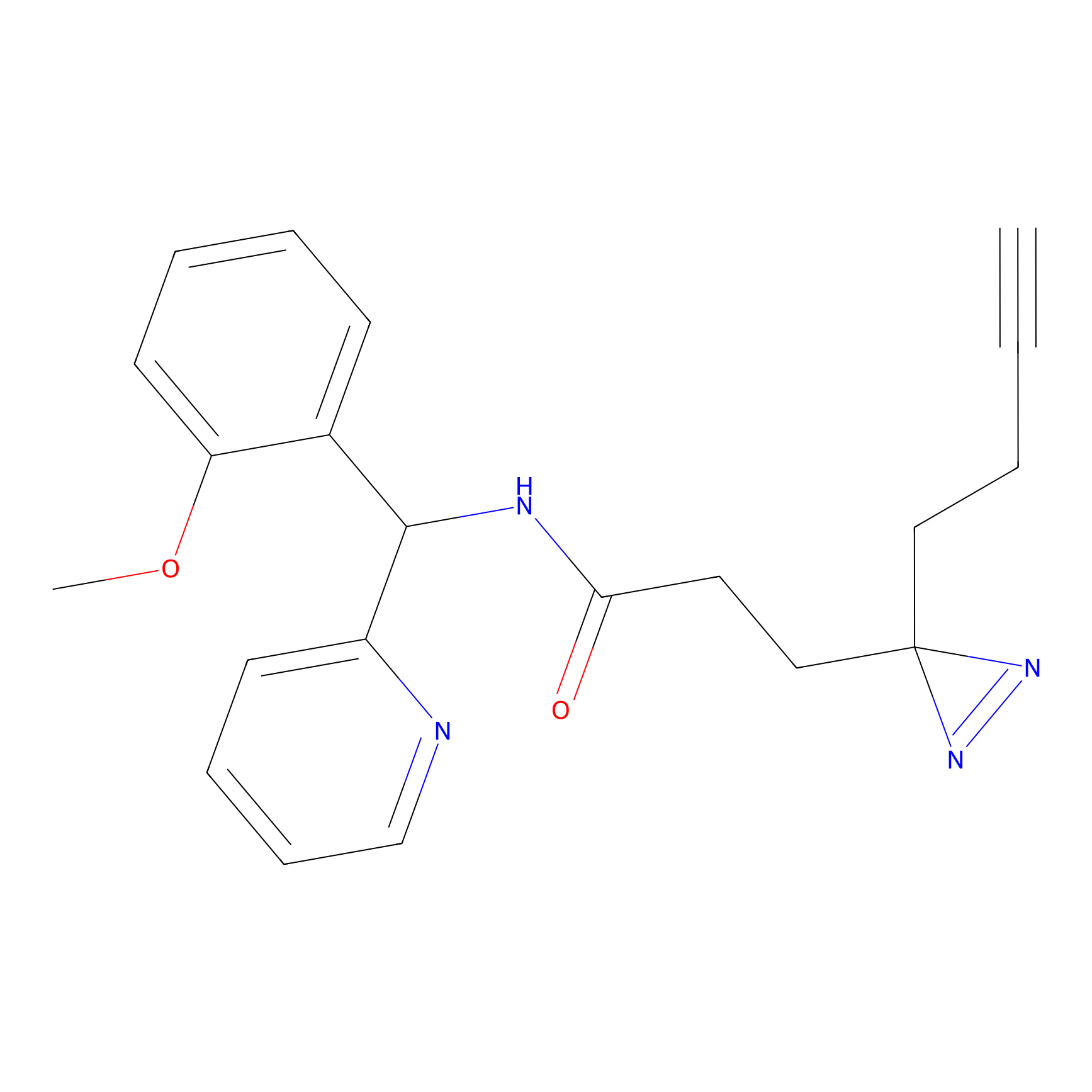
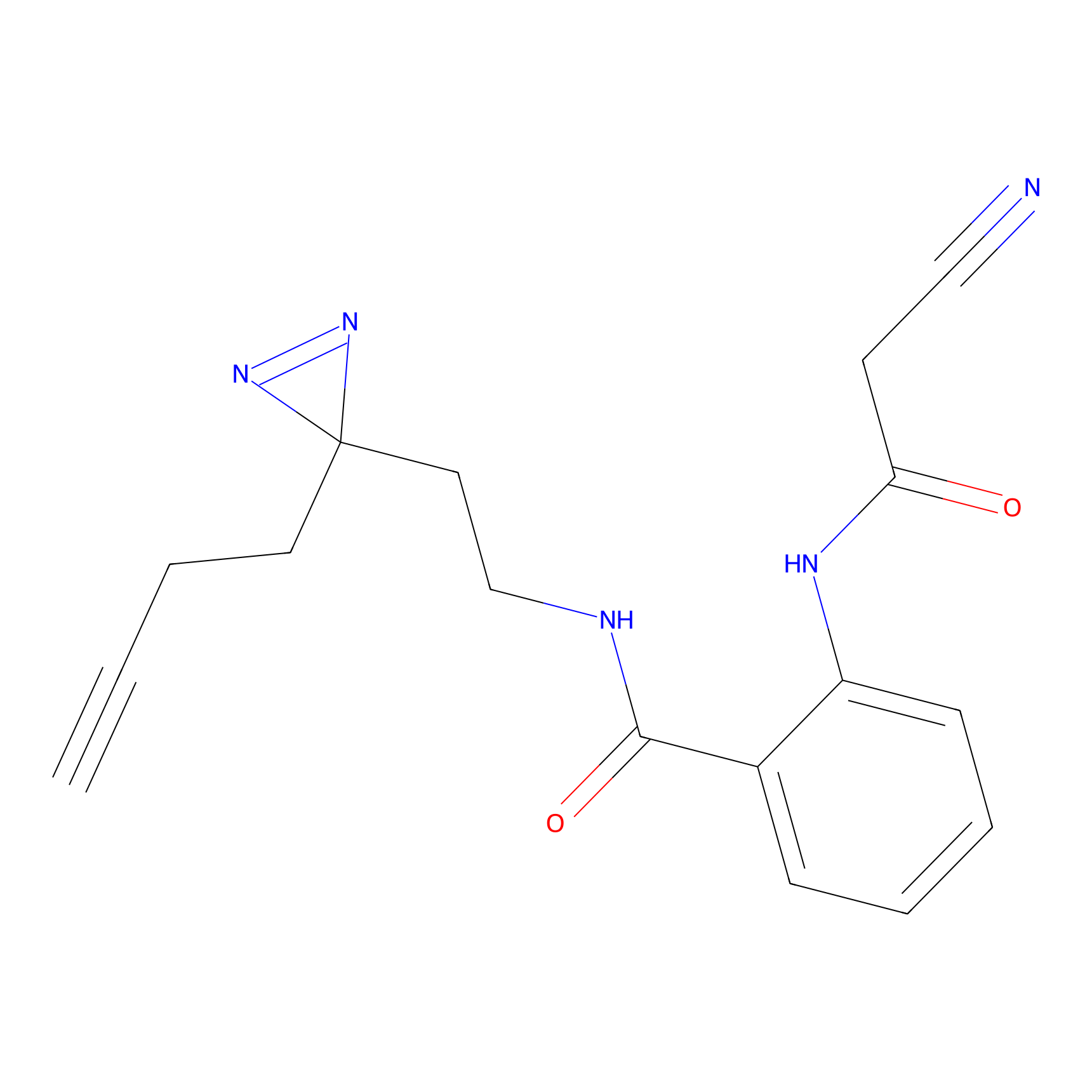
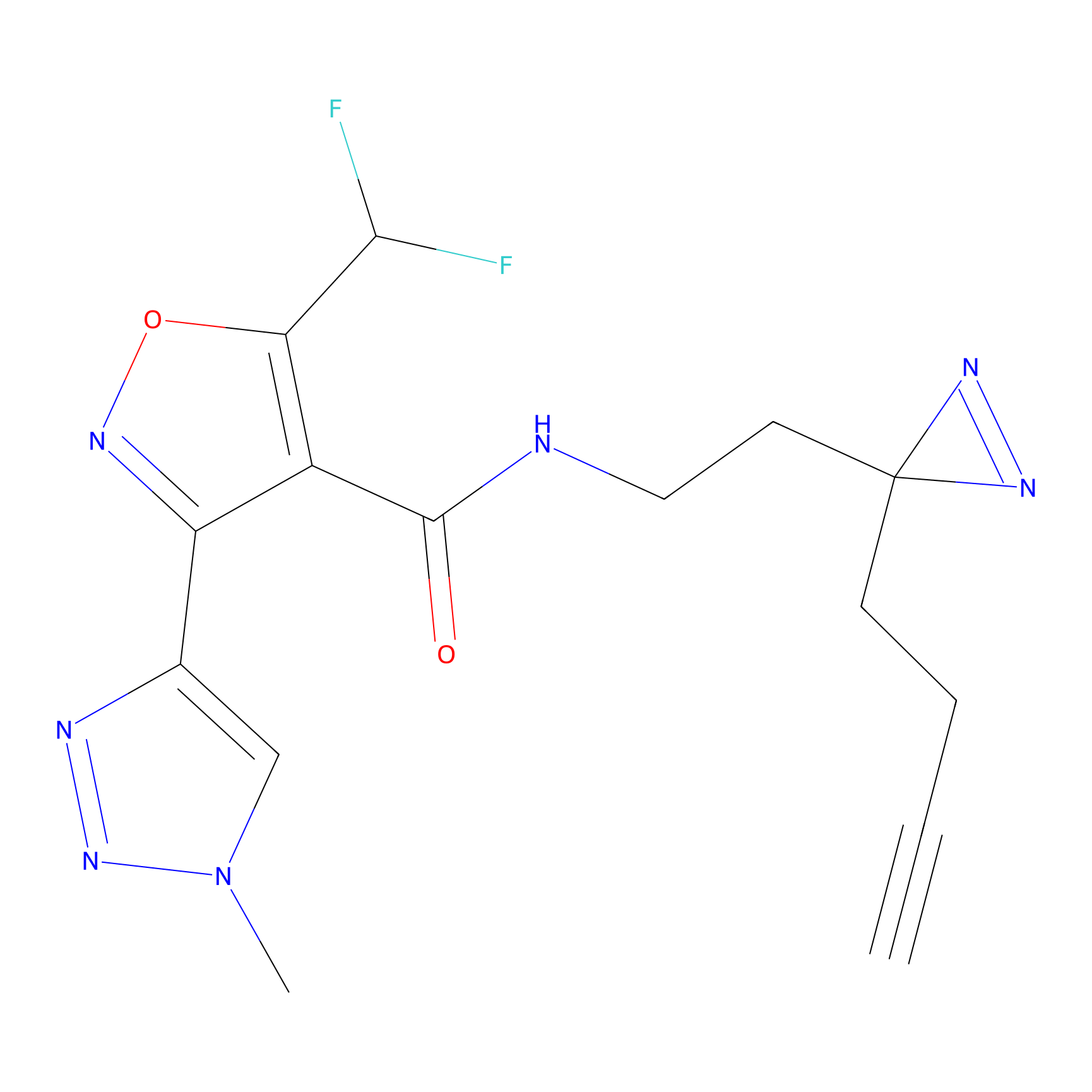
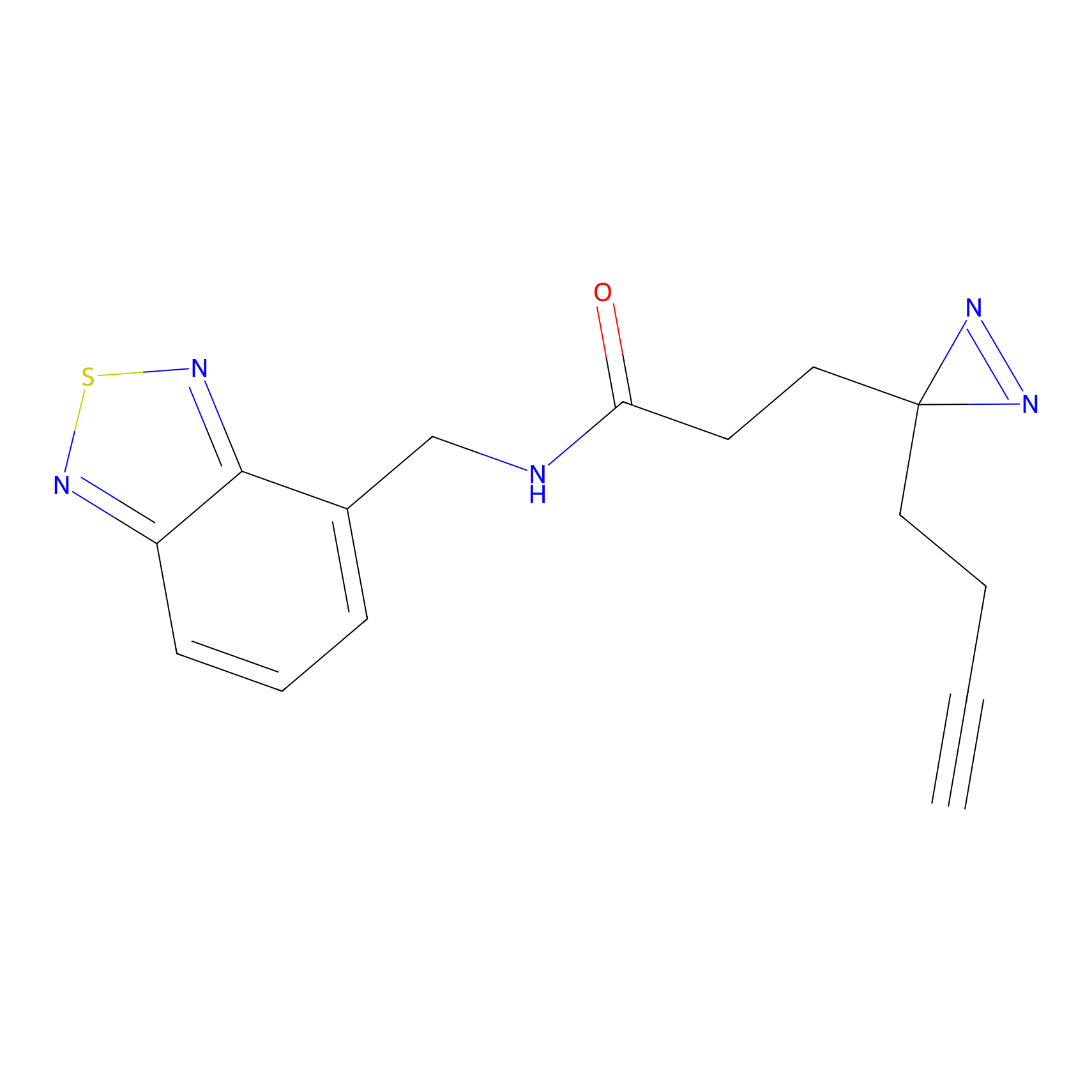
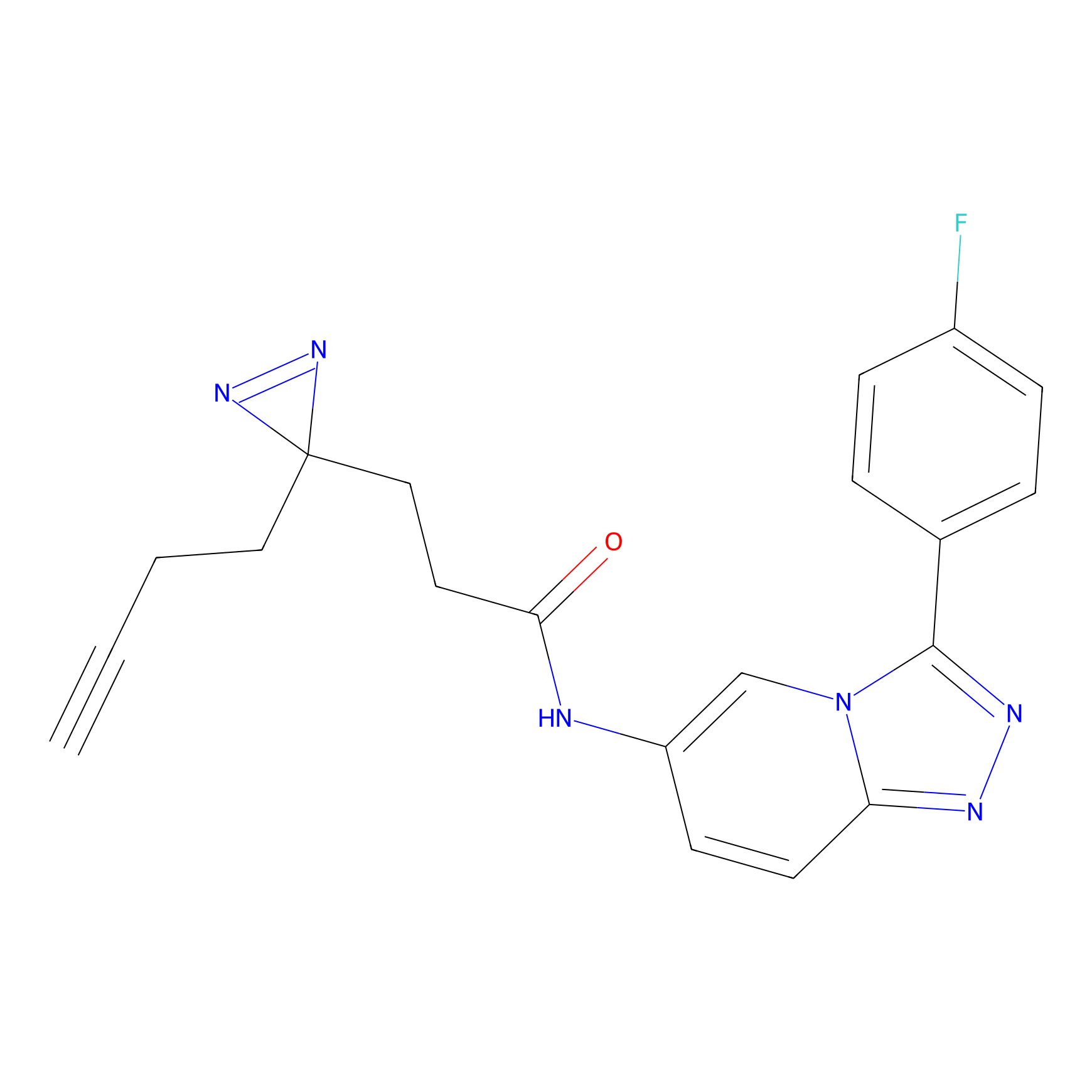
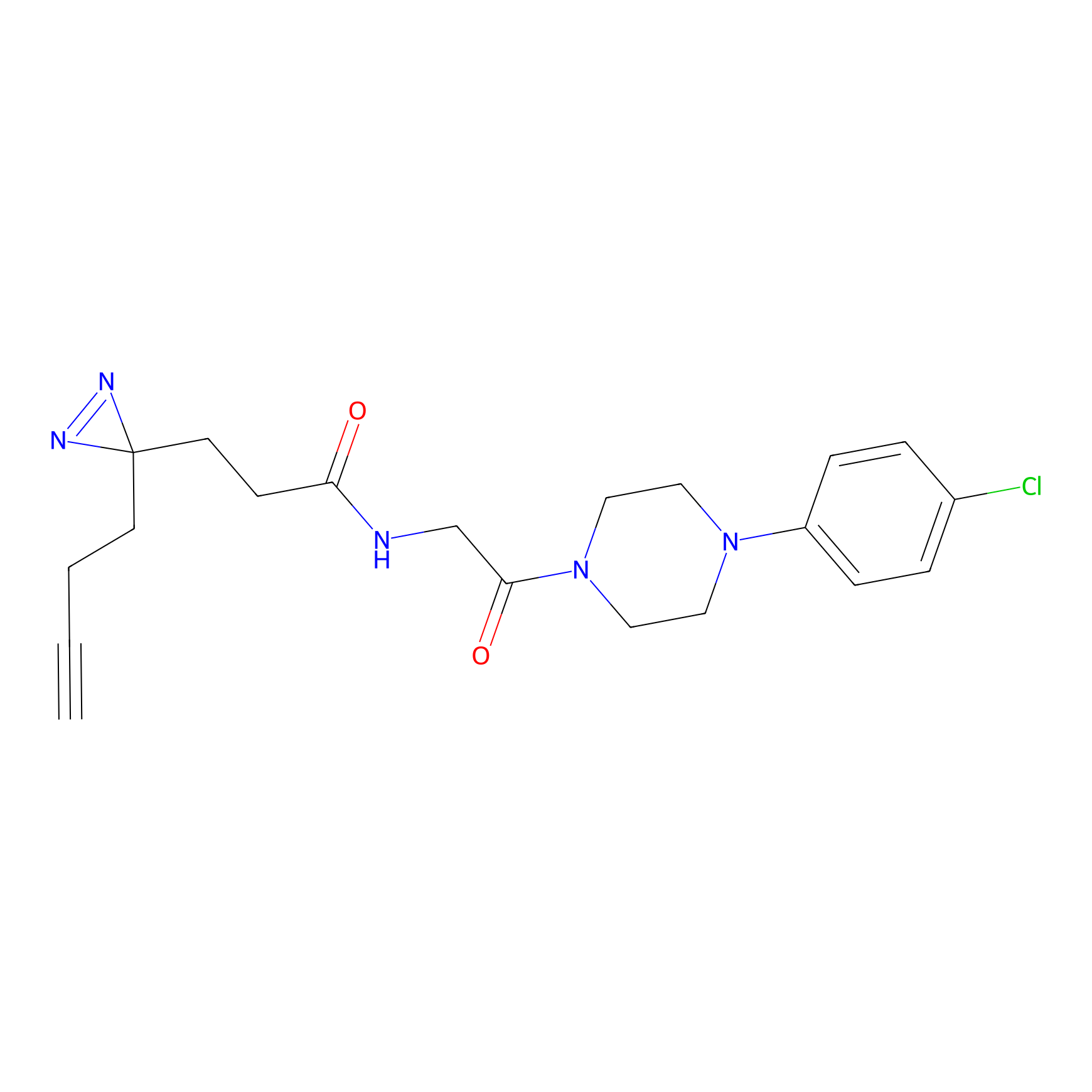
|
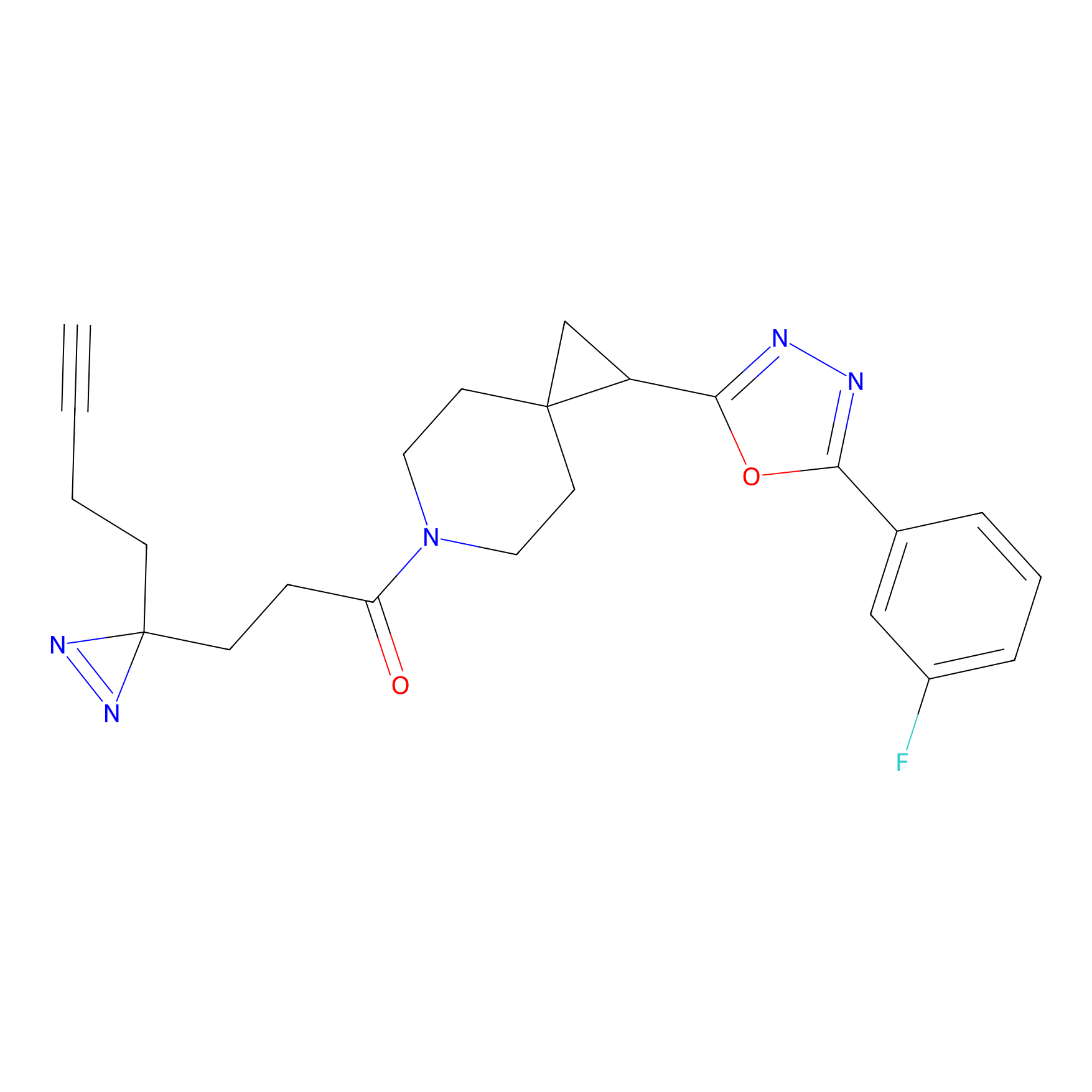
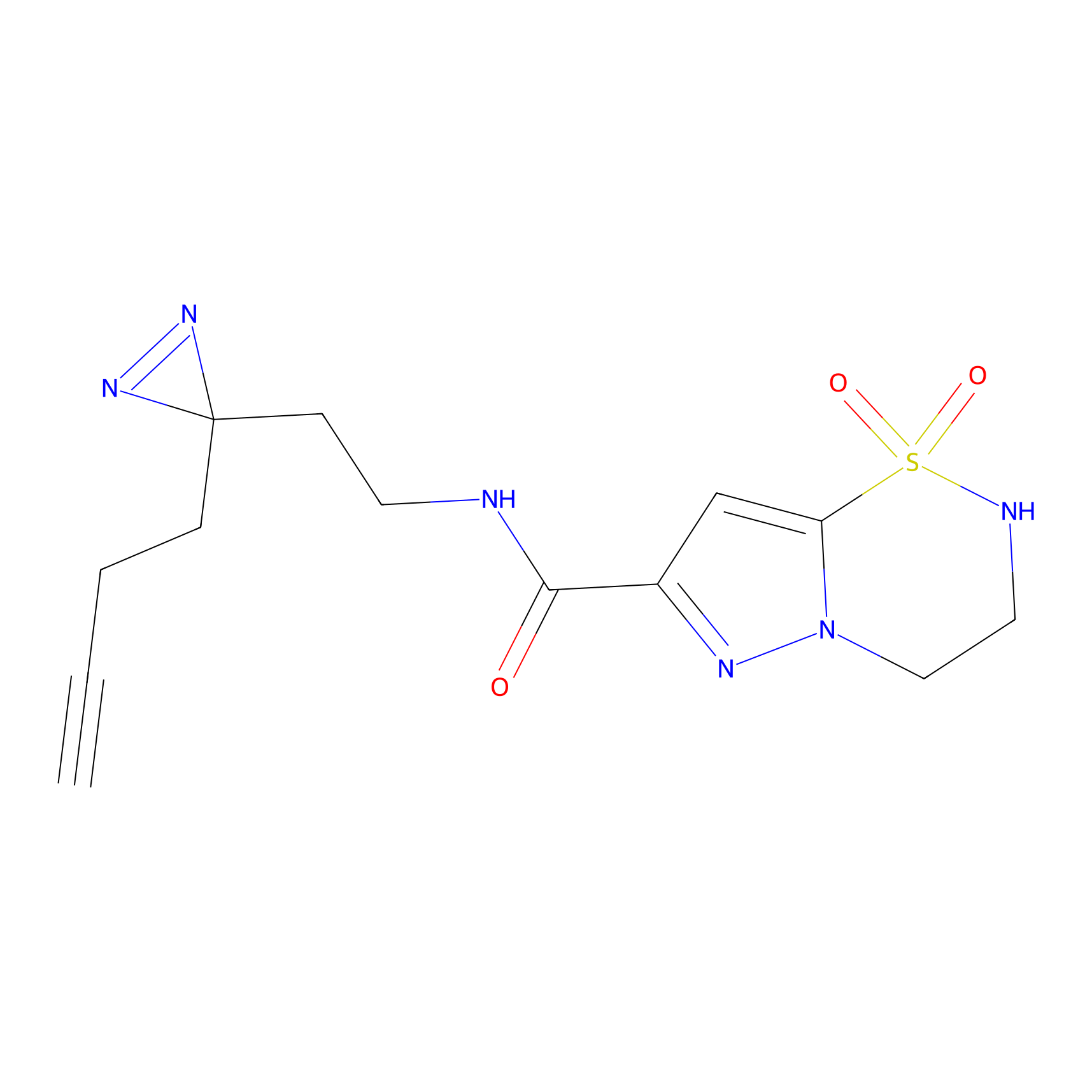
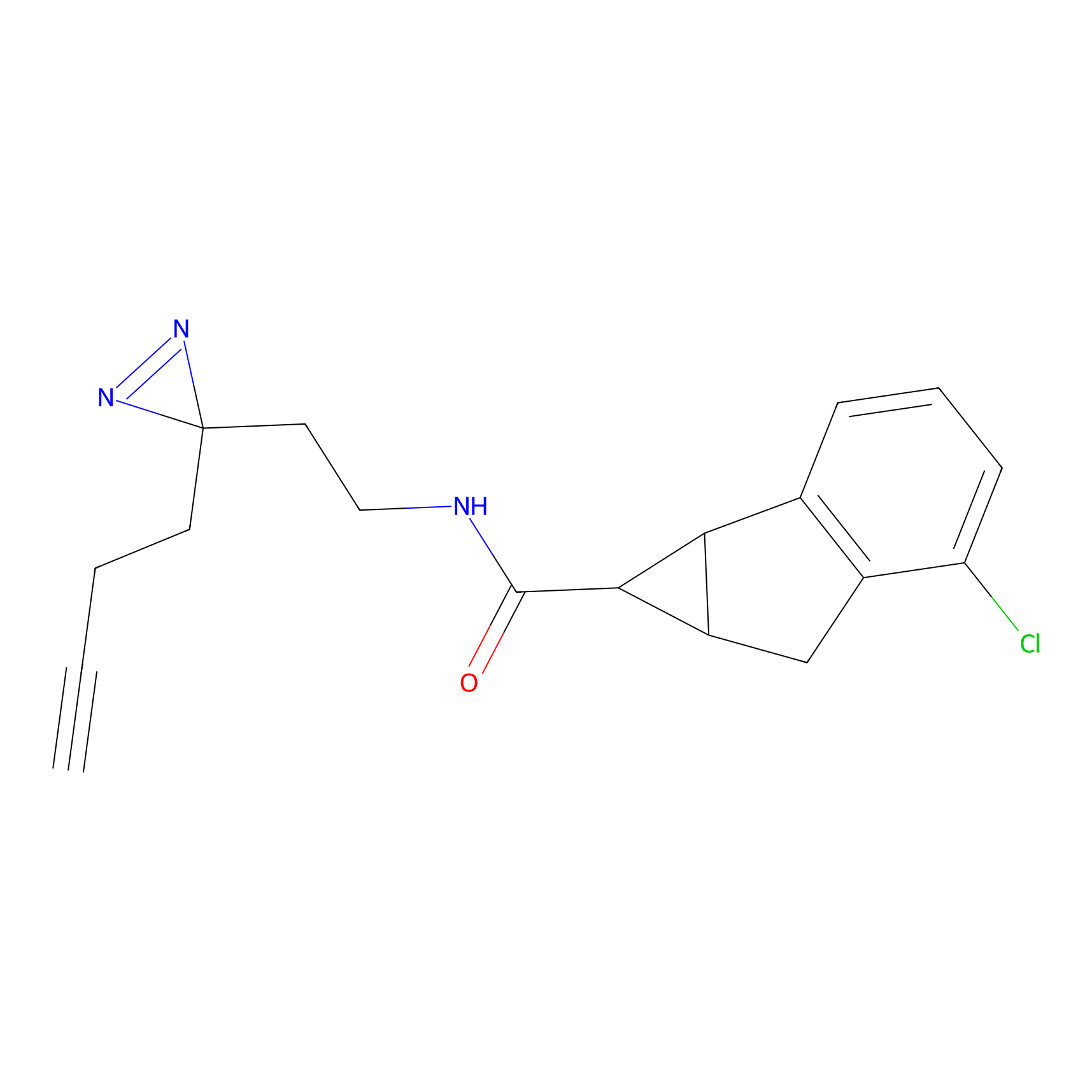
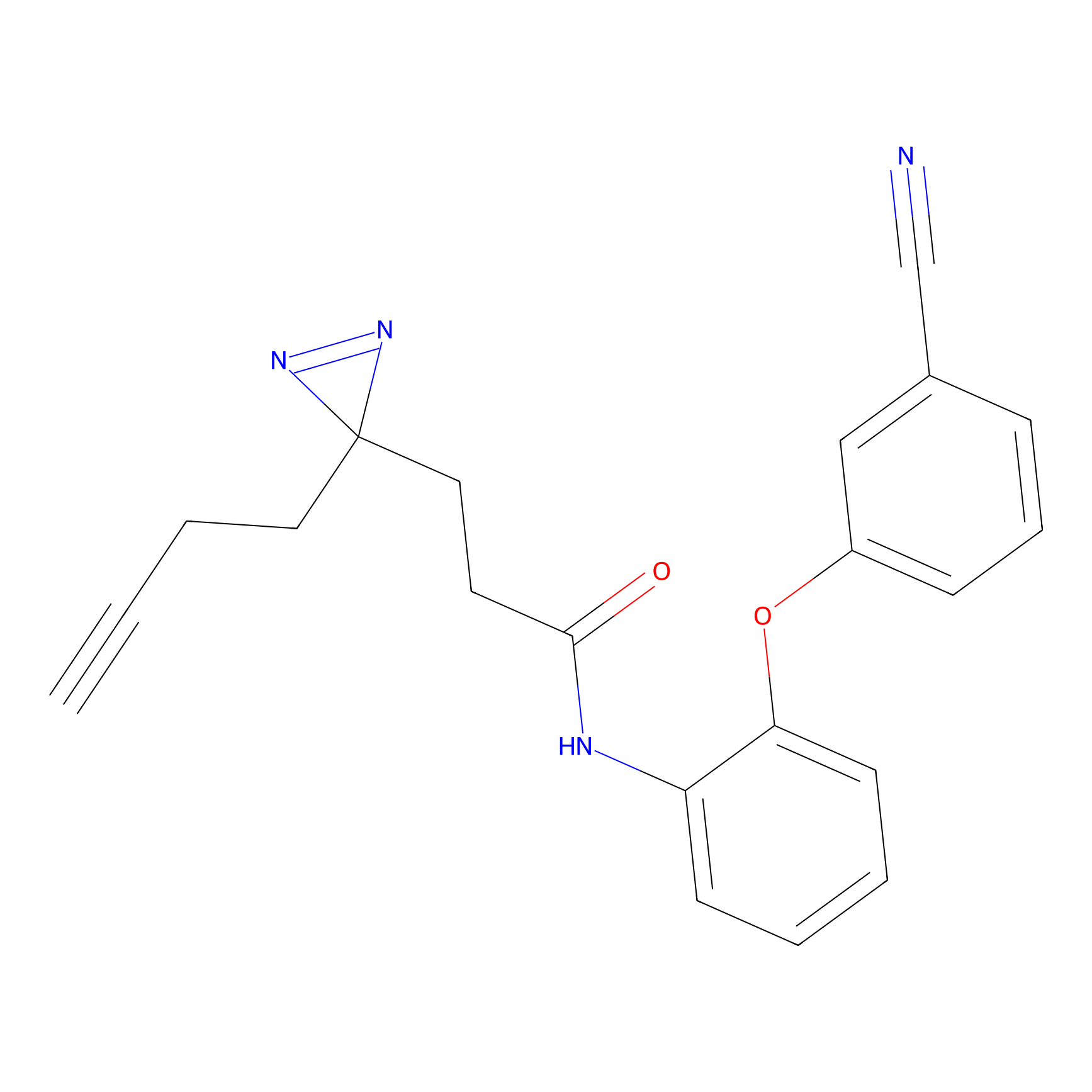
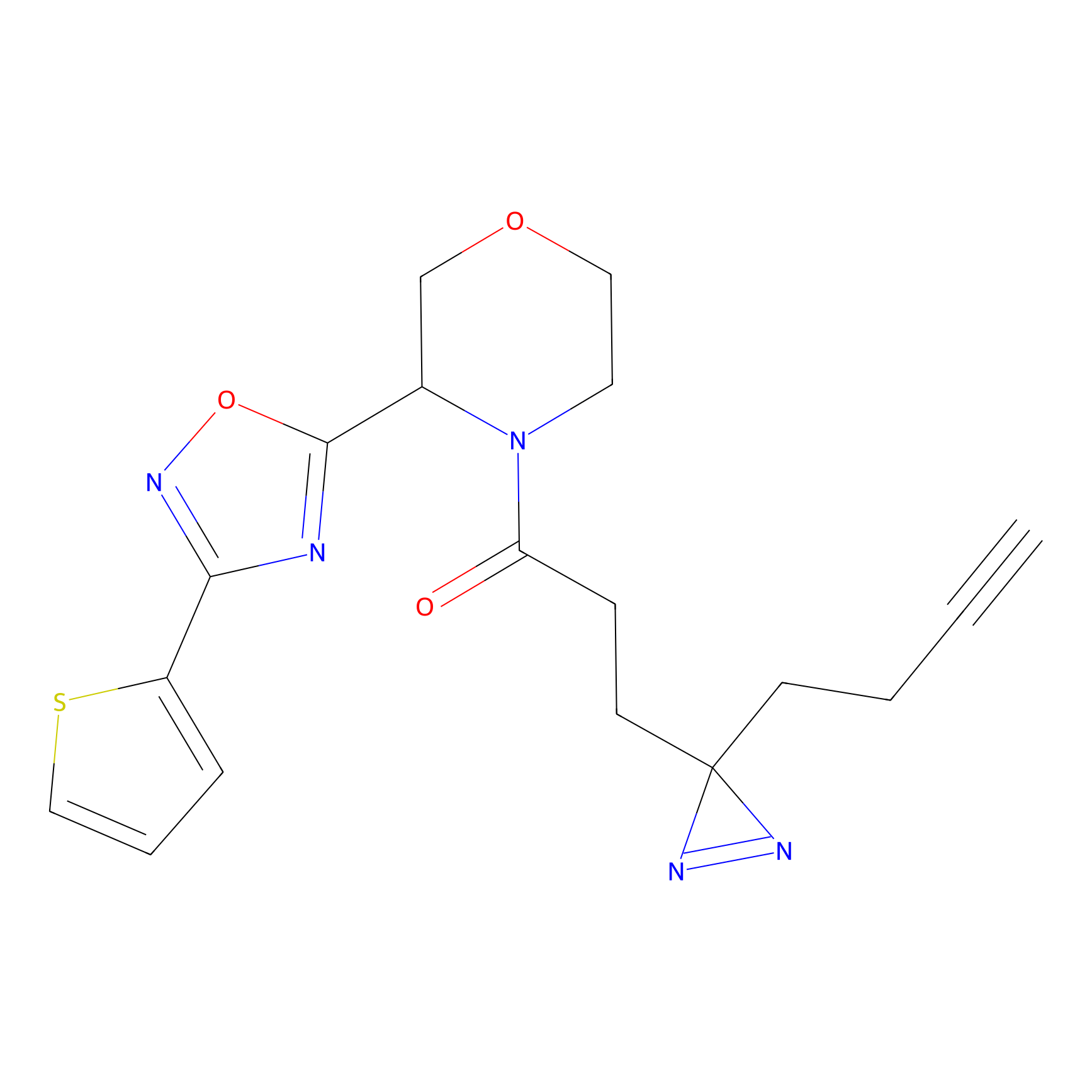
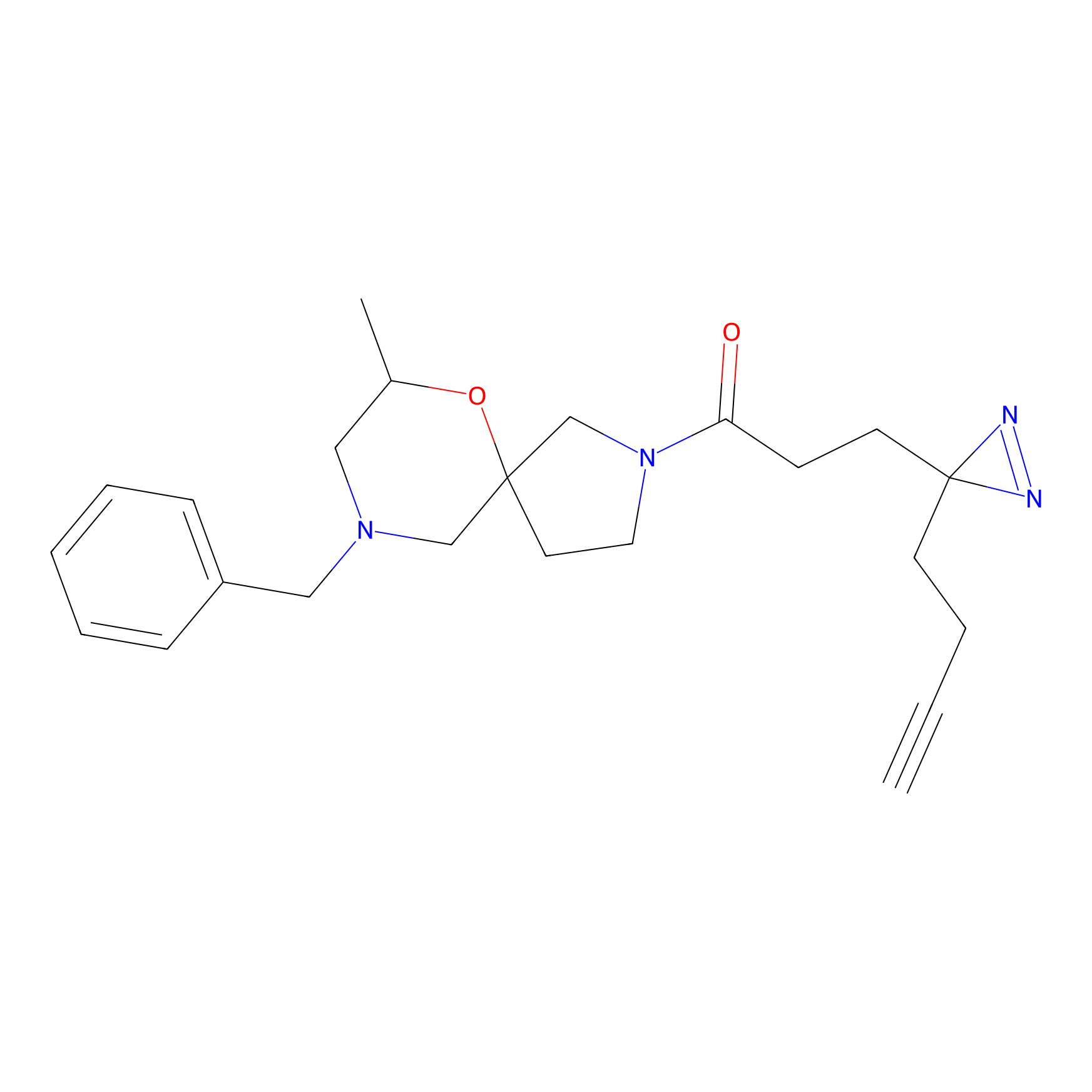
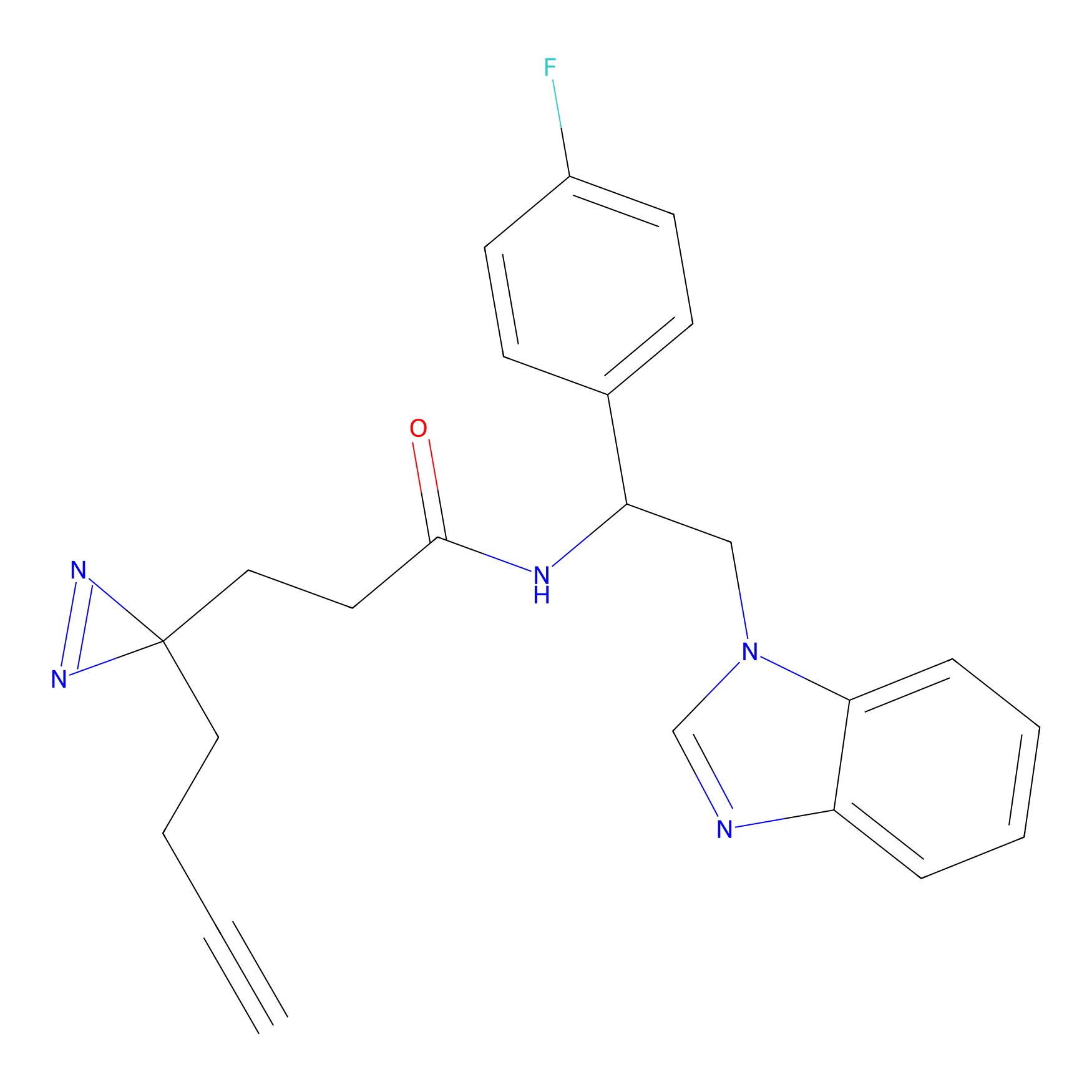
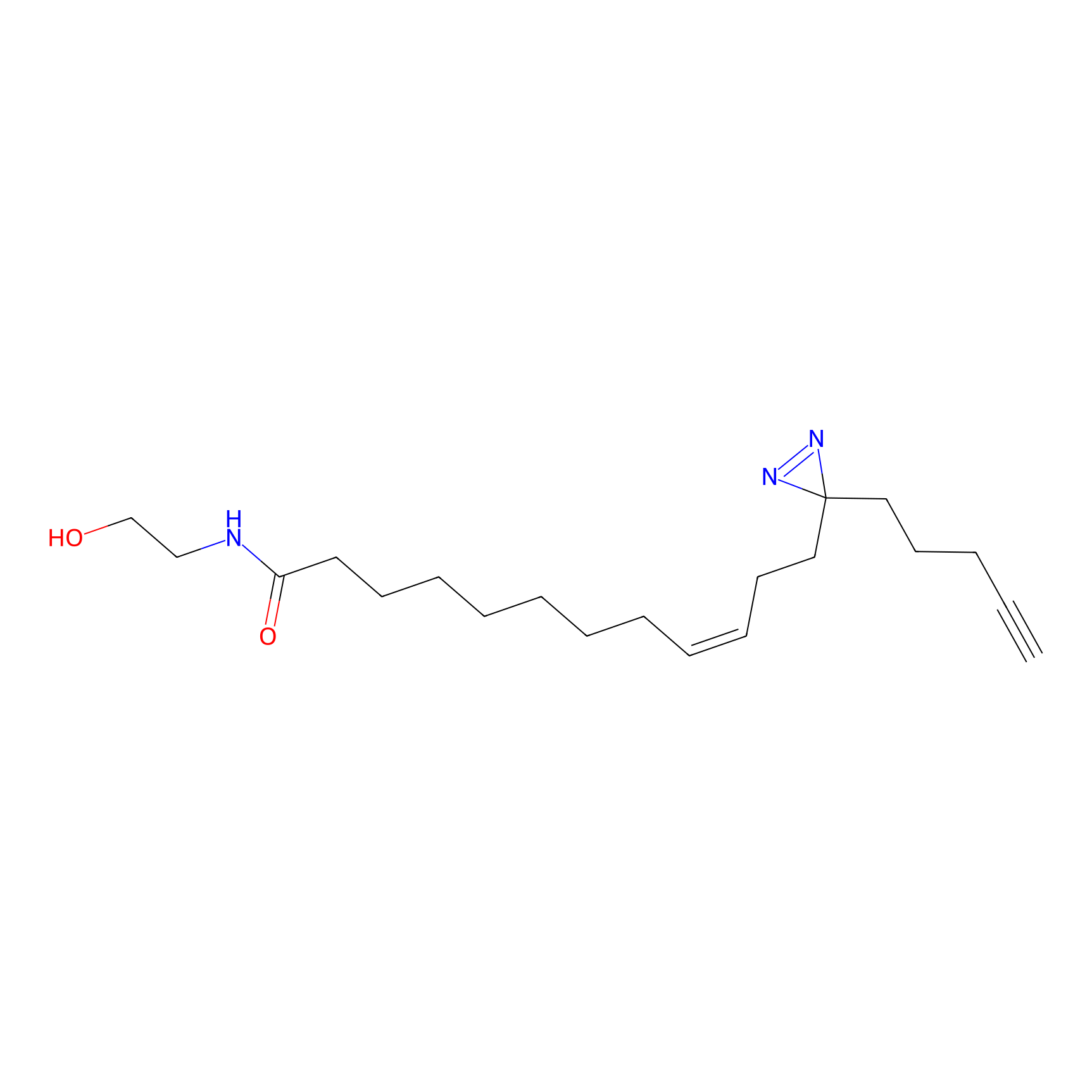
IA-alkyne Probe Info |
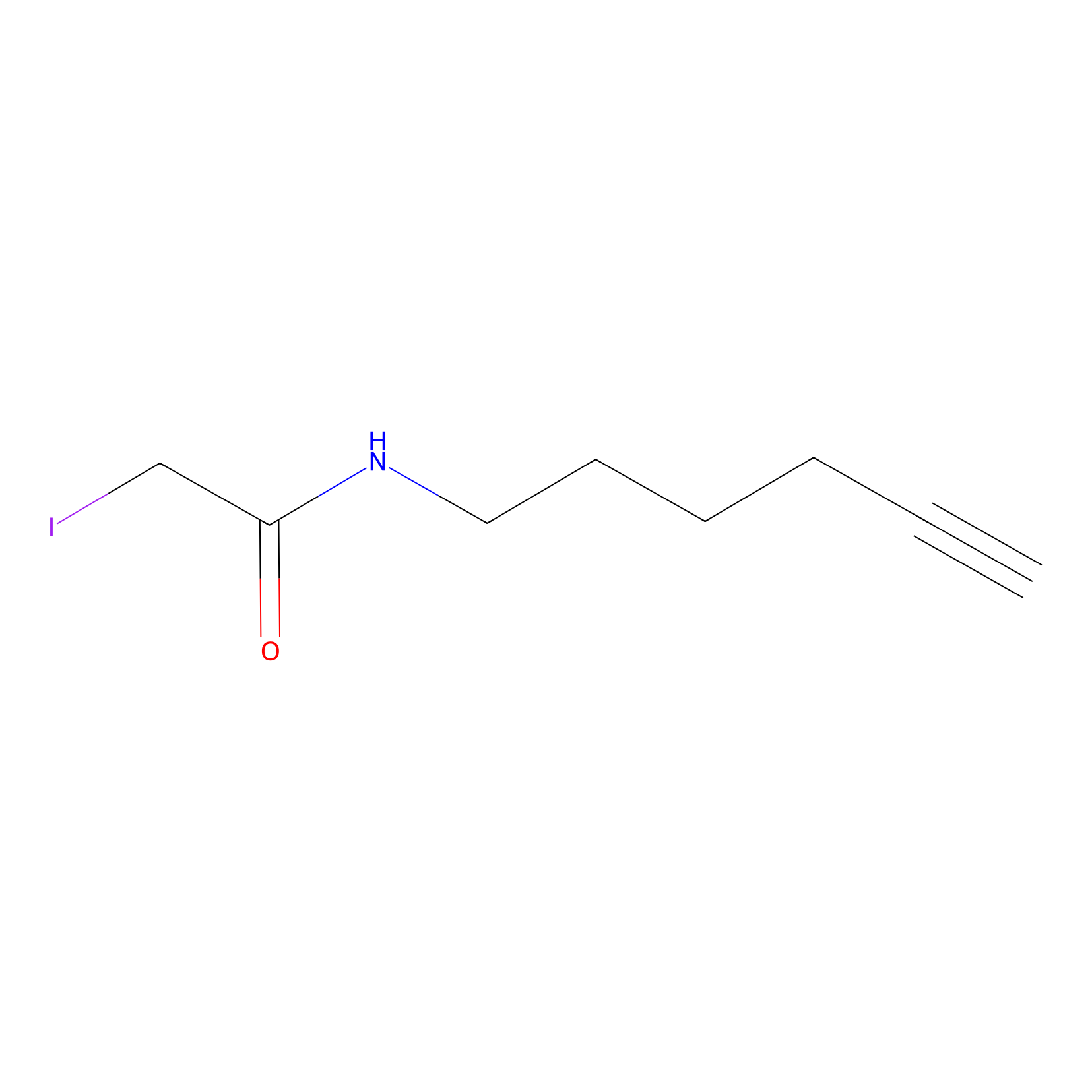 |
N.A. | LDD0036 | [3] | |
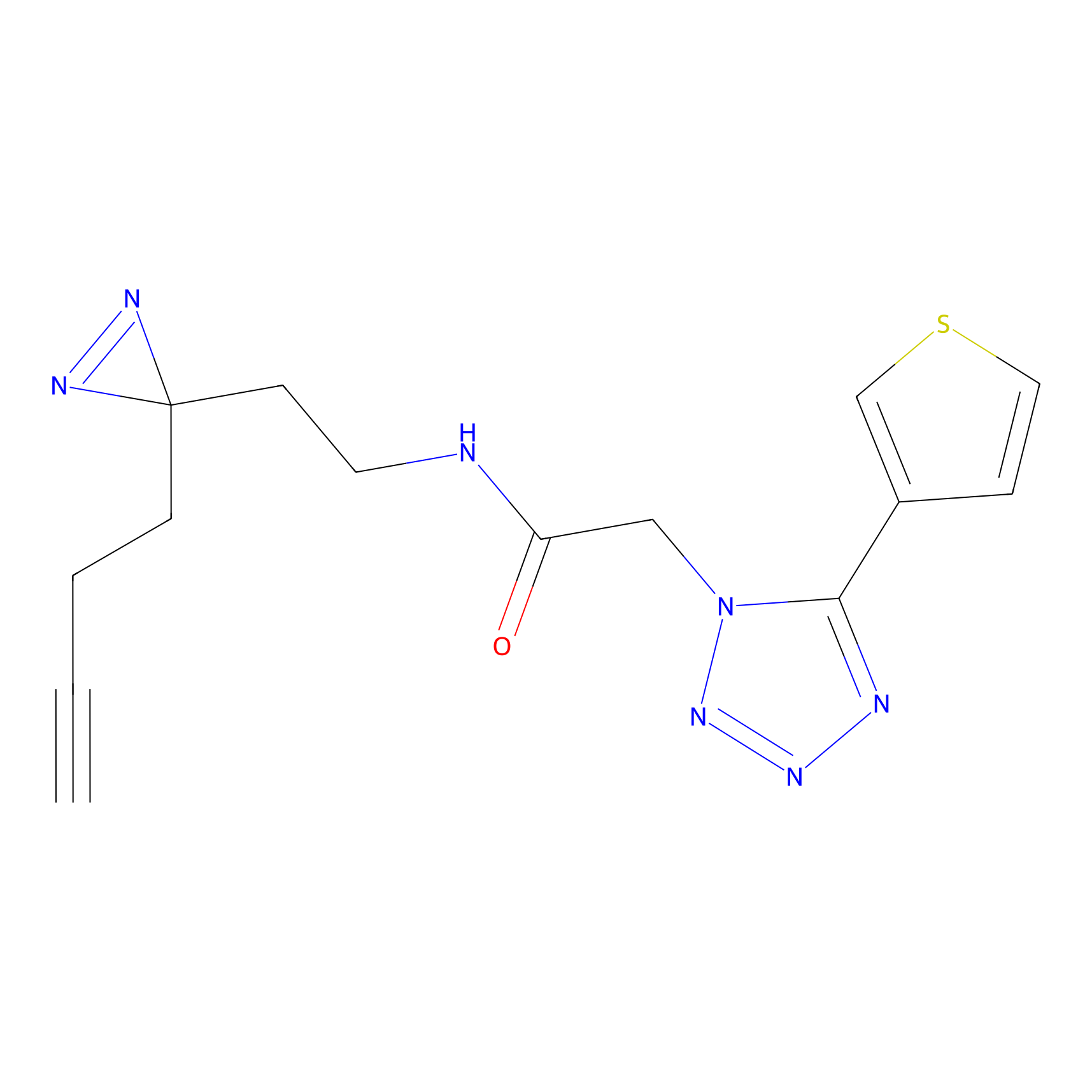
|
Lodoacetamide azide Probe Info |
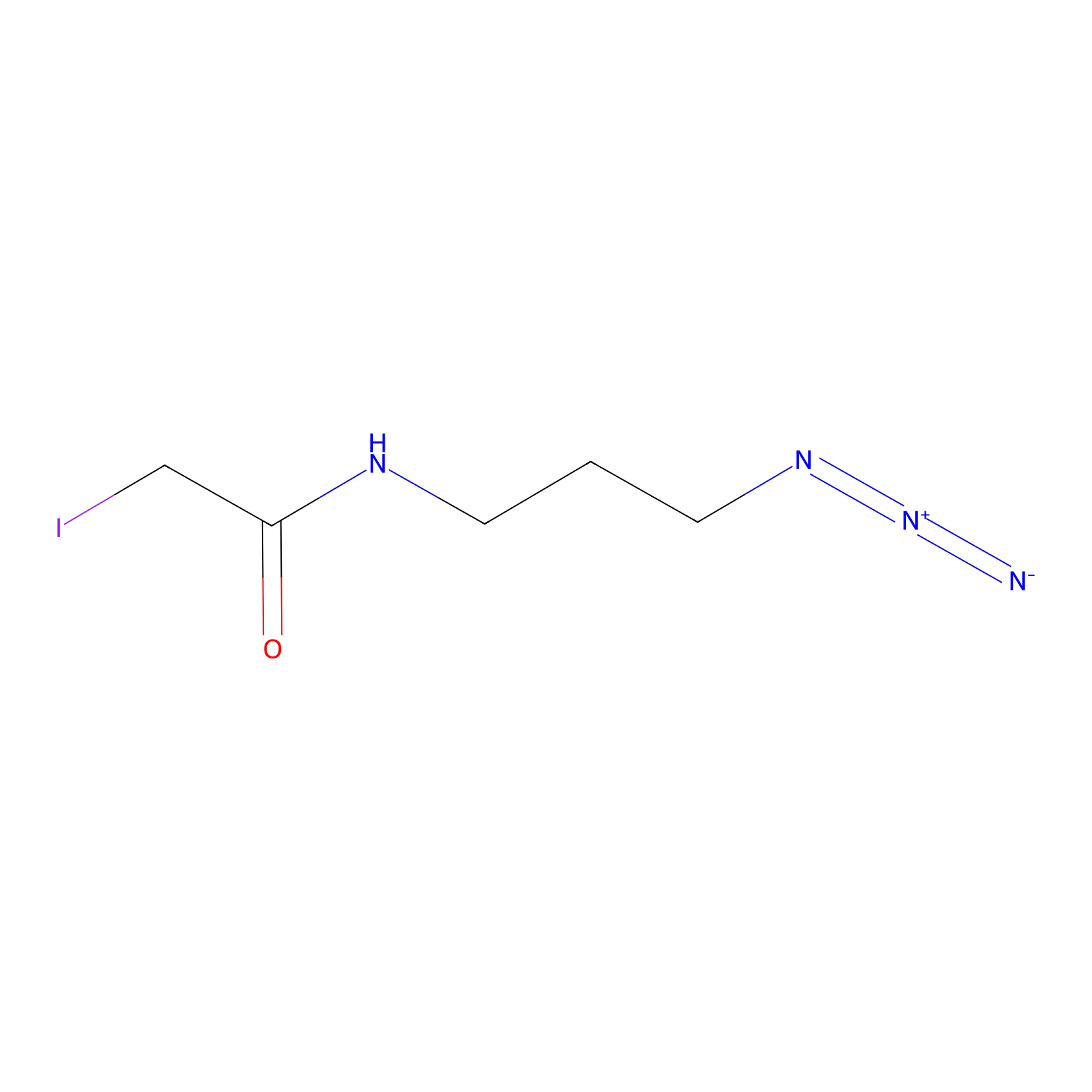 |
N.A. | LDD0037 | [3] | |
|
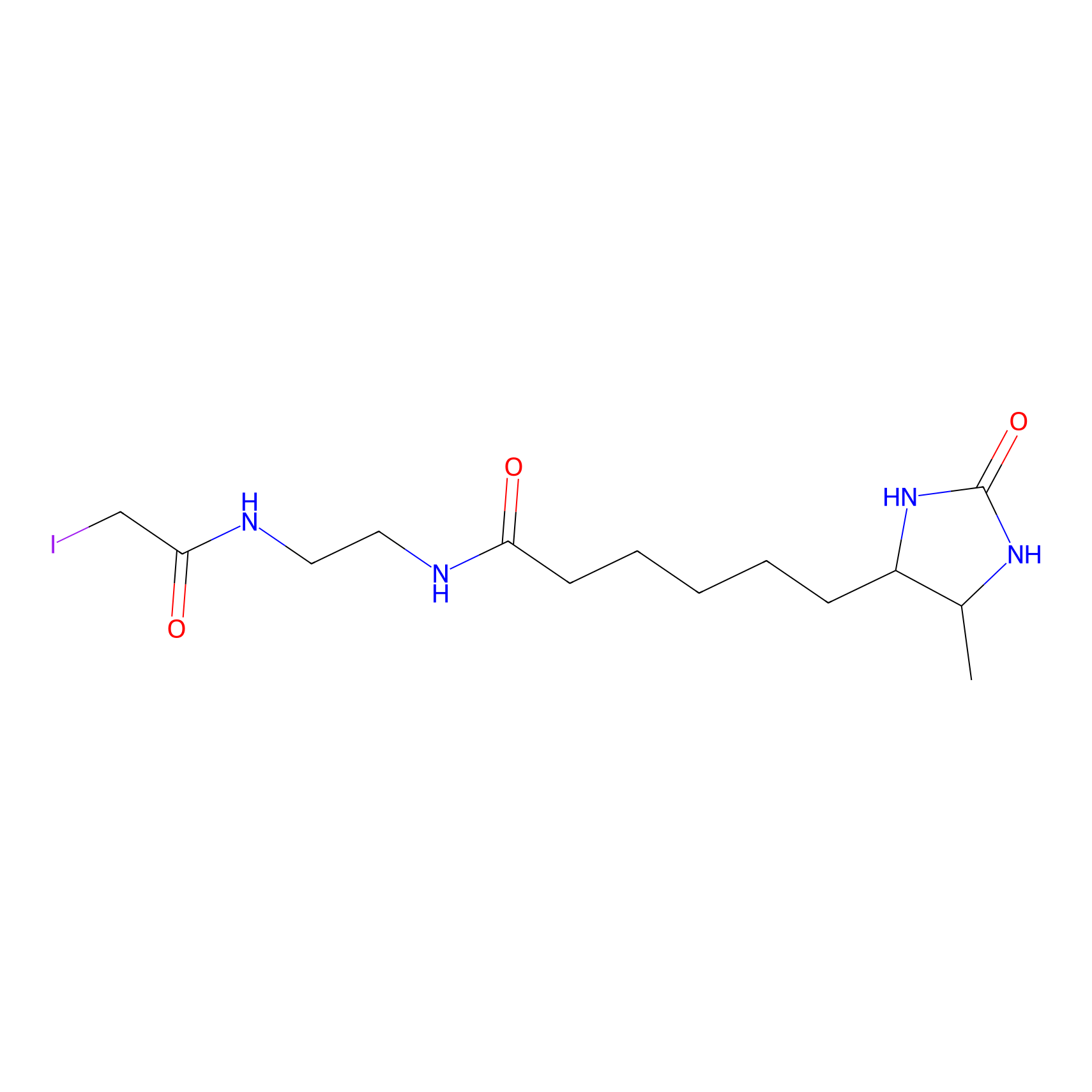
IPM Probe Info |
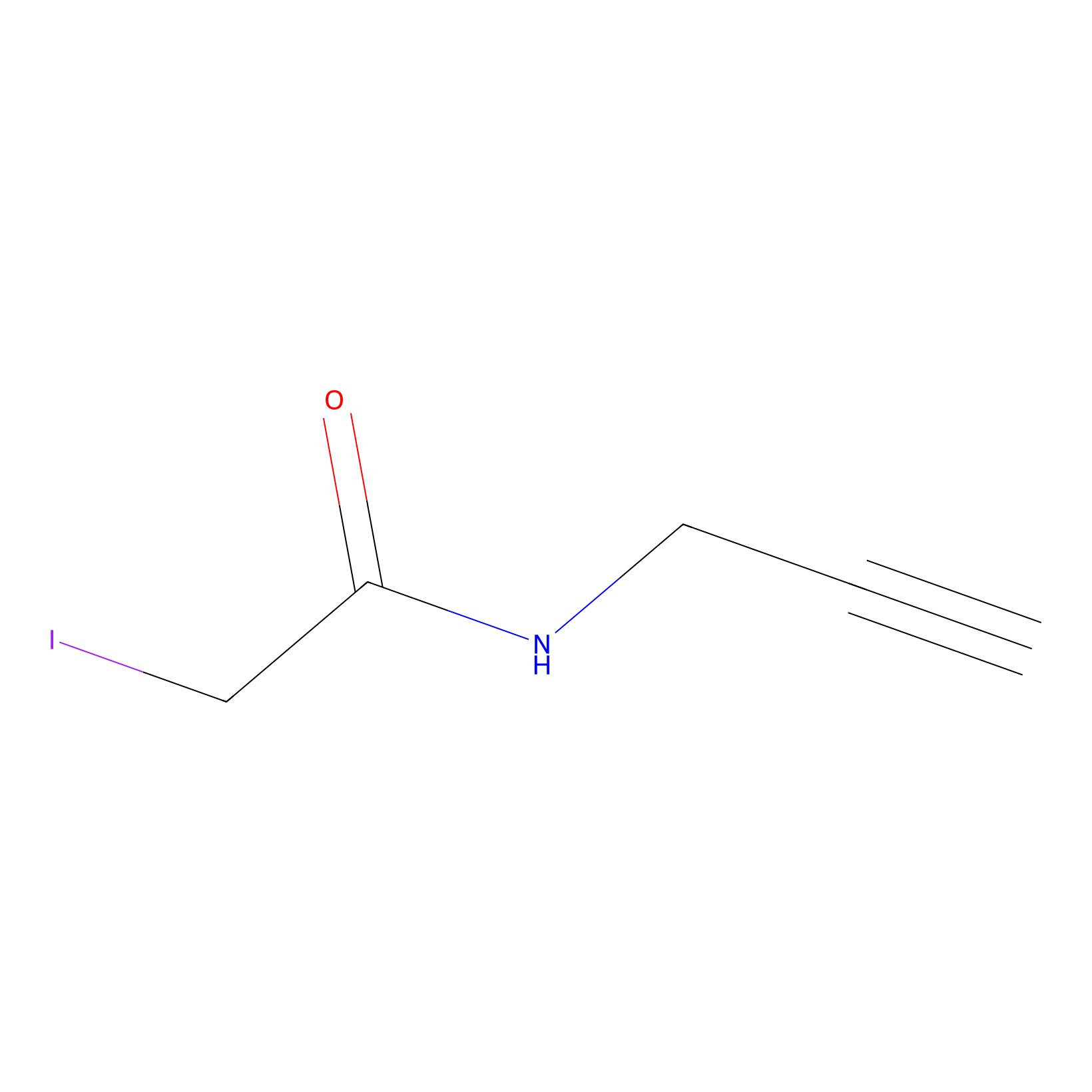 |
N.A. | LDD2156 | [4] | |
|
STPyne Probe Info |
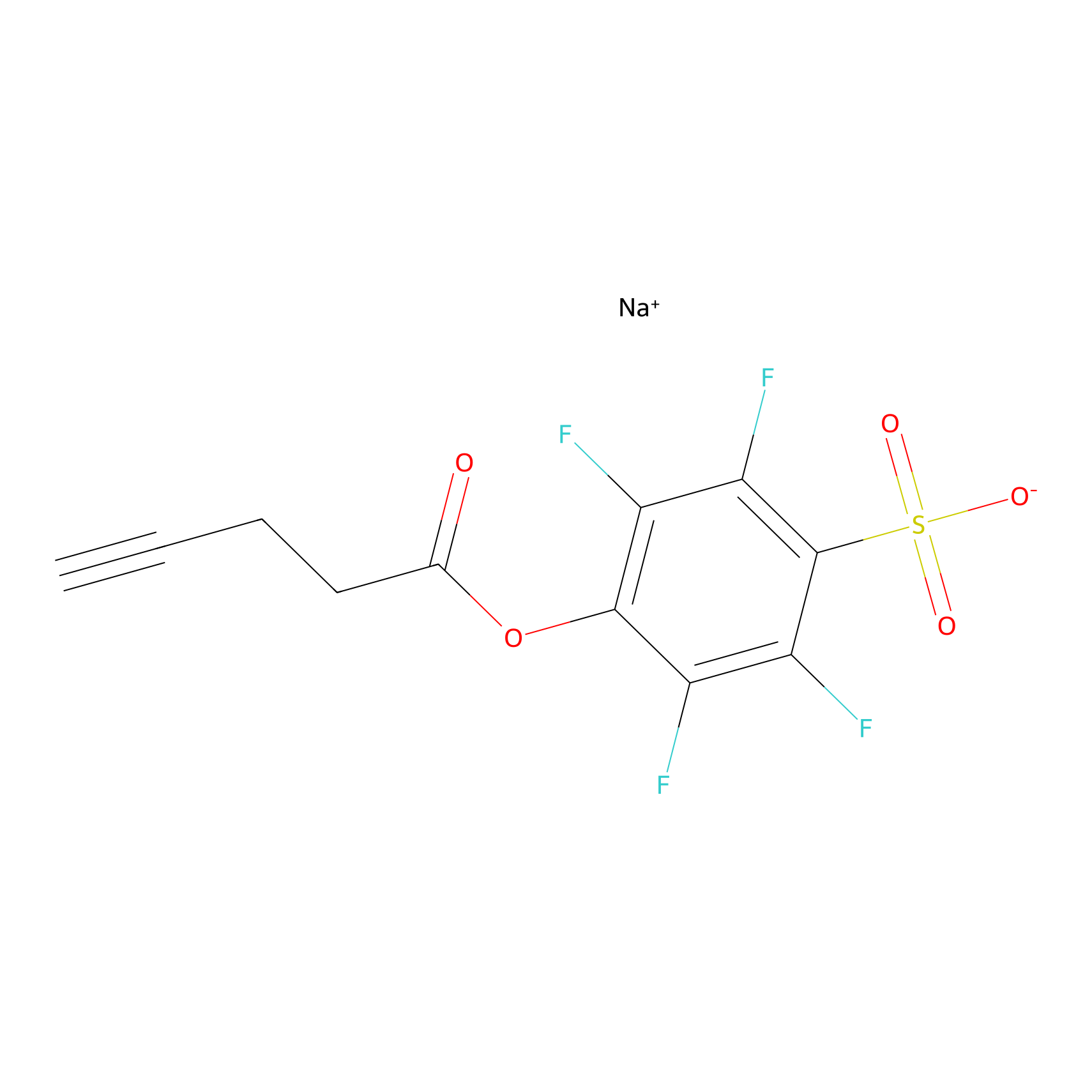 |
N.A. | LDD0009 | [5] | |
|
AOyne Probe Info |
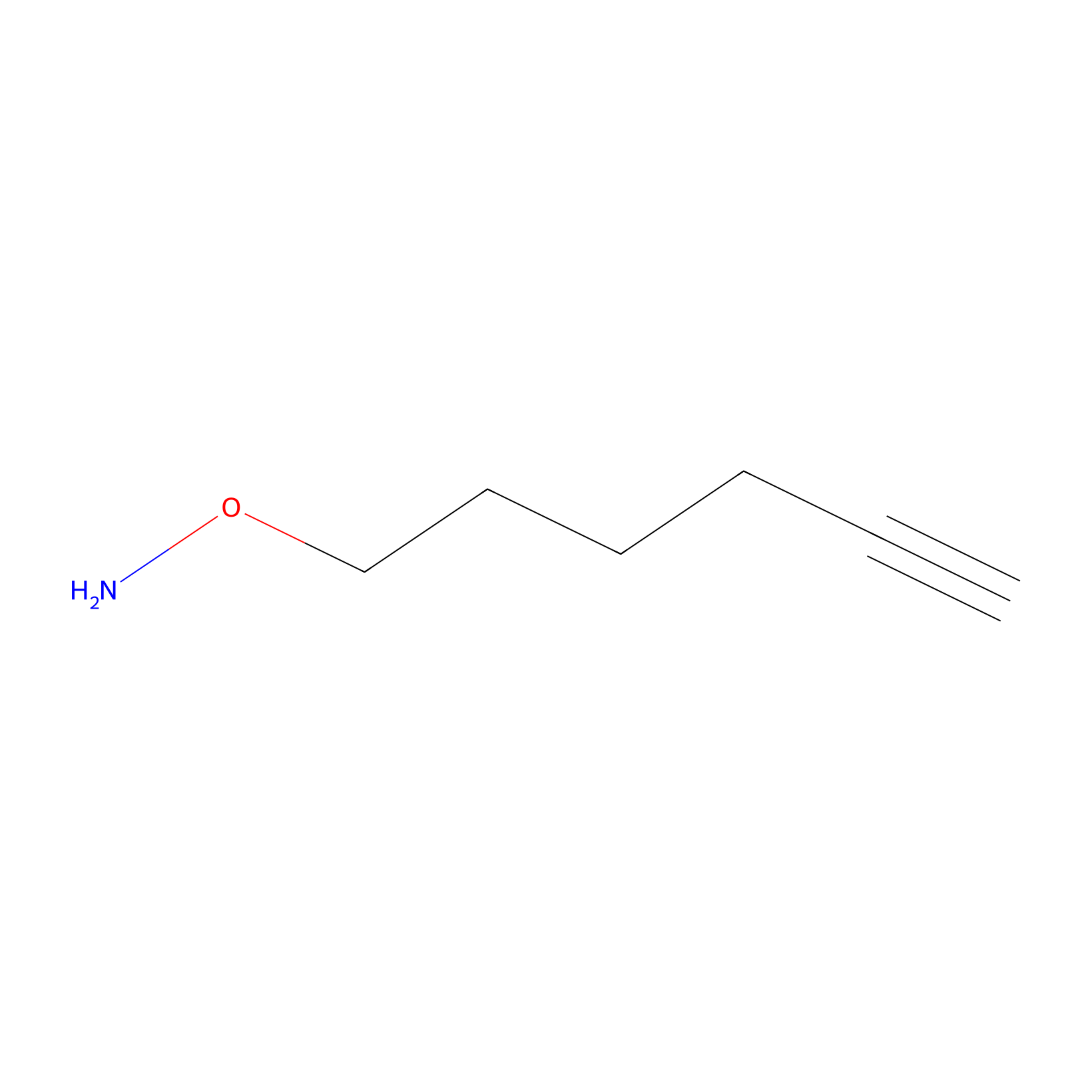 |
15.00 | LDD0443 | [6] | |
PAL-AfBPP Probe
Competitor(s) Related to This Target
| Competitor ID | Name | Cell line | Binding Site(Ratio) | Interaction ID | Ref |
|---|---|---|---|---|---|
| LDCM0214 | AC1 | HEK-293T | C68(1.11) | LDD1507 | [2] |
| LDCM0226 | AC11 | HEK-293T | C68(0.97) | LDD1509 | [2] |
| LDCM0276 | AC17 | HEK-293T | C68(1.11) | LDD1515 | [2] |
| LDCM0278 | AC19 | HEK-293T | C68(0.94) | LDD1517 | [2] |
| LDCM0285 | AC25 | HEK-293T | C68(0.96) | LDD1524 | [2] |
| LDCM0287 | AC27 | HEK-293T | C68(1.07) | LDD1526 | [2] |
| LDCM0290 | AC3 | HEK-293T | C68(0.97) | LDD1529 | [2] |
| LDCM0294 | AC33 | HEK-293T | C68(1.09) | LDD1533 | [2] |
| LDCM0296 | AC35 | HEK-293T | C68(0.88) | LDD1535 | [2] |
| LDCM0303 | AC41 | HEK-293T | C68(1.12) | LDD1542 | [2] |
| LDCM0305 | AC43 | HEK-293T | C68(0.97) | LDD1544 | [2] |
| LDCM0311 | AC49 | HEK-293T | C68(1.03) | LDD1550 | [2] |
| LDCM0314 | AC51 | HEK-293T | C68(0.94) | LDD1553 | [2] |
| LDCM0320 | AC57 | HEK-293T | C68(1.23) | LDD1559 | [2] |
| LDCM0322 | AC59 | HEK-293T | C68(1.07) | LDD1561 | [2] |
| LDCM0356 | AKOS034007680 | HEK-293T | C68(1.15) | LDD1570 | [2] |
| LDCM0367 | CL1 | HEK-293T | C68(0.96) | LDD1571 | [2] |
| LDCM0370 | CL101 | HEK-293T | C68(0.97) | LDD1574 | [2] |
| LDCM0374 | CL105 | HEK-293T | C68(0.96) | LDD1578 | [2] |
| LDCM0378 | CL109 | HEK-293T | C68(0.96) | LDD1582 | [2] |
| LDCM0383 | CL113 | HEK-293T | C68(0.90) | LDD1587 | [2] |
| LDCM0387 | CL117 | HEK-293T | C68(0.95) | LDD1591 | [2] |
| LDCM0392 | CL121 | HEK-293T | C68(0.96) | LDD1596 | [2] |
| LDCM0396 | CL125 | HEK-293T | C68(0.98) | LDD1600 | [2] |
| LDCM0400 | CL13 | HEK-293T | C68(0.98) | LDD1604 | [2] |
| LDCM0404 | CL17 | HEK-293T | C68(1.06) | LDD1608 | [2] |
| LDCM0406 | CL19 | HEK-293T | C68(0.93) | LDD1610 | [2] |
| LDCM0413 | CL25 | HEK-293T | C68(0.97) | LDD1617 | [2] |
| LDCM0417 | CL29 | HEK-293T | C68(1.05) | LDD1621 | [2] |
| LDCM0420 | CL31 | HEK-293T | C68(0.93) | LDD1624 | [2] |
| LDCM0426 | CL37 | HEK-293T | C68(1.01) | LDD1630 | [2] |
| LDCM0431 | CL41 | HEK-293T | C68(1.03) | LDD1635 | [2] |
| LDCM0433 | CL43 | HEK-293T | C68(0.97) | LDD1637 | [2] |
| LDCM0439 | CL49 | HEK-293T | C68(1.05) | LDD1643 | [2] |
| LDCM0440 | CL5 | HEK-293T | C68(1.04) | LDD1644 | [2] |
| LDCM0444 | CL53 | HEK-293T | C68(1.03) | LDD1647 | [2] |
| LDCM0446 | CL55 | HEK-293T | C68(0.99) | LDD1649 | [2] |
| LDCM0453 | CL61 | HEK-293T | C68(1.07) | LDD1656 | [2] |
| LDCM0457 | CL65 | HEK-293T | C68(1.22) | LDD1660 | [2] |
| LDCM0459 | CL67 | HEK-293T | C68(0.86) | LDD1662 | [2] |
| LDCM0462 | CL7 | HEK-293T | C68(0.96) | LDD1665 | [2] |
| LDCM0466 | CL73 | HEK-293T | C68(1.03) | LDD1669 | [2] |
| LDCM0470 | CL77 | HEK-293T | C68(1.10) | LDD1673 | [2] |
| LDCM0472 | CL79 | HEK-293T | C68(0.90) | LDD1675 | [2] |
| LDCM0479 | CL85 | HEK-293T | C68(1.03) | LDD1682 | [2] |
| LDCM0483 | CL89 | HEK-293T | C68(1.12) | LDD1686 | [2] |
| LDCM0486 | CL91 | HEK-293T | C68(1.04) | LDD1689 | [2] |
| LDCM0492 | CL97 | HEK-293T | C68(0.99) | LDD1695 | [2] |
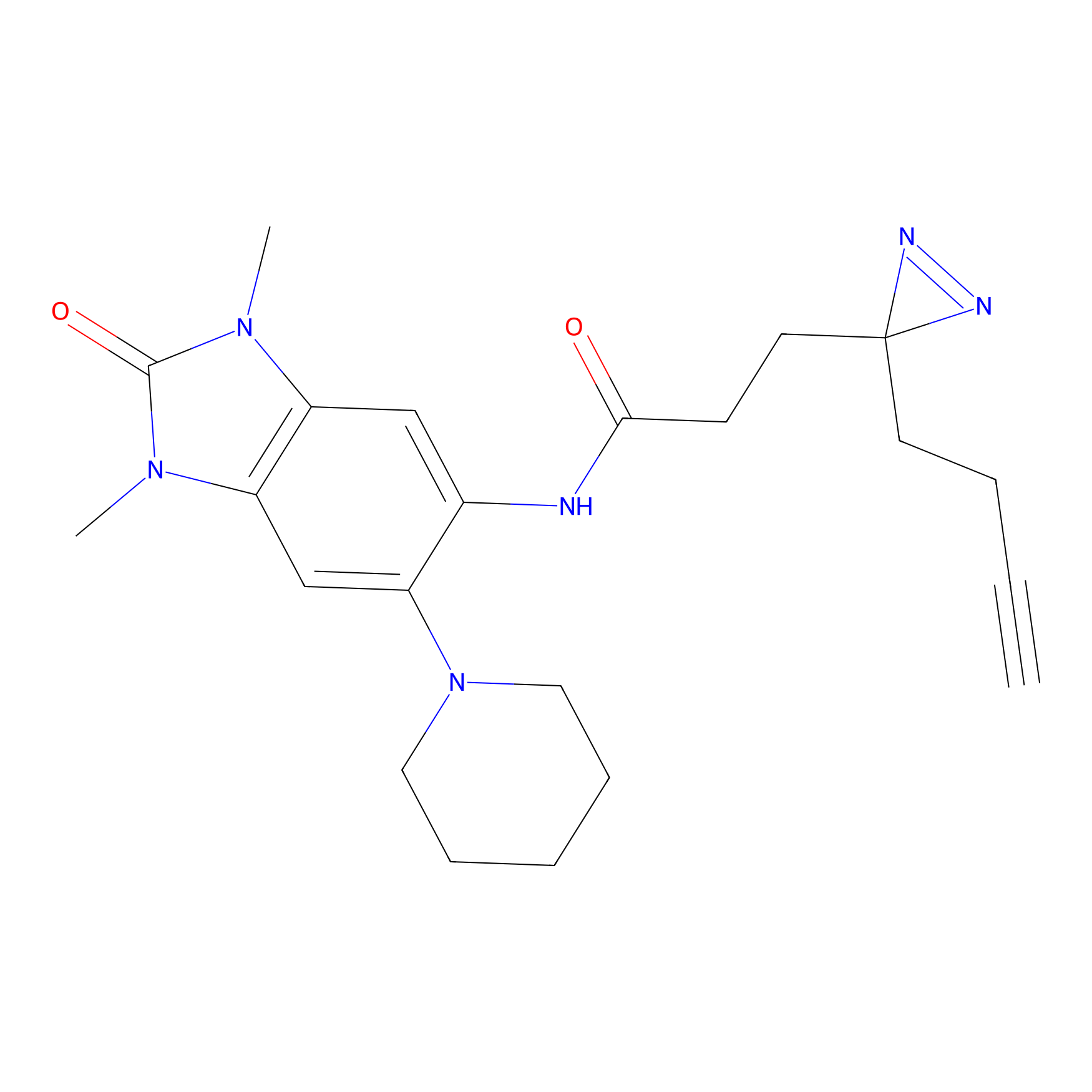
| LDCM0175 | Ethacrynic acid | HeLa | N.A. | LDD0440 | [1] |
| LDCM0625 | F8 | Ramos | C68(3.12) | LDD2187 | [13] |
| LDCM0576 | Fragment14 | Ramos | C68(3.81) | LDD2193 | [13] |
| LDCM0586 | Fragment28 | Ramos | C68(2.48) | LDD2198 | [13] |
| LDCM0569 | Fragment7 | Ramos | C68(5.32) | LDD2186 | [13] |
| LDCM0022 | KB02 | HEK-293T | C68(0.90) | LDD1492 | [2] |
| LDCM0023 | KB03 | HEK-293T | C68(0.98) | LDD1497 | [2] |
| LDCM0024 | KB05 | HEK-293T | C68(0.98) | LDD1502 | [2] |
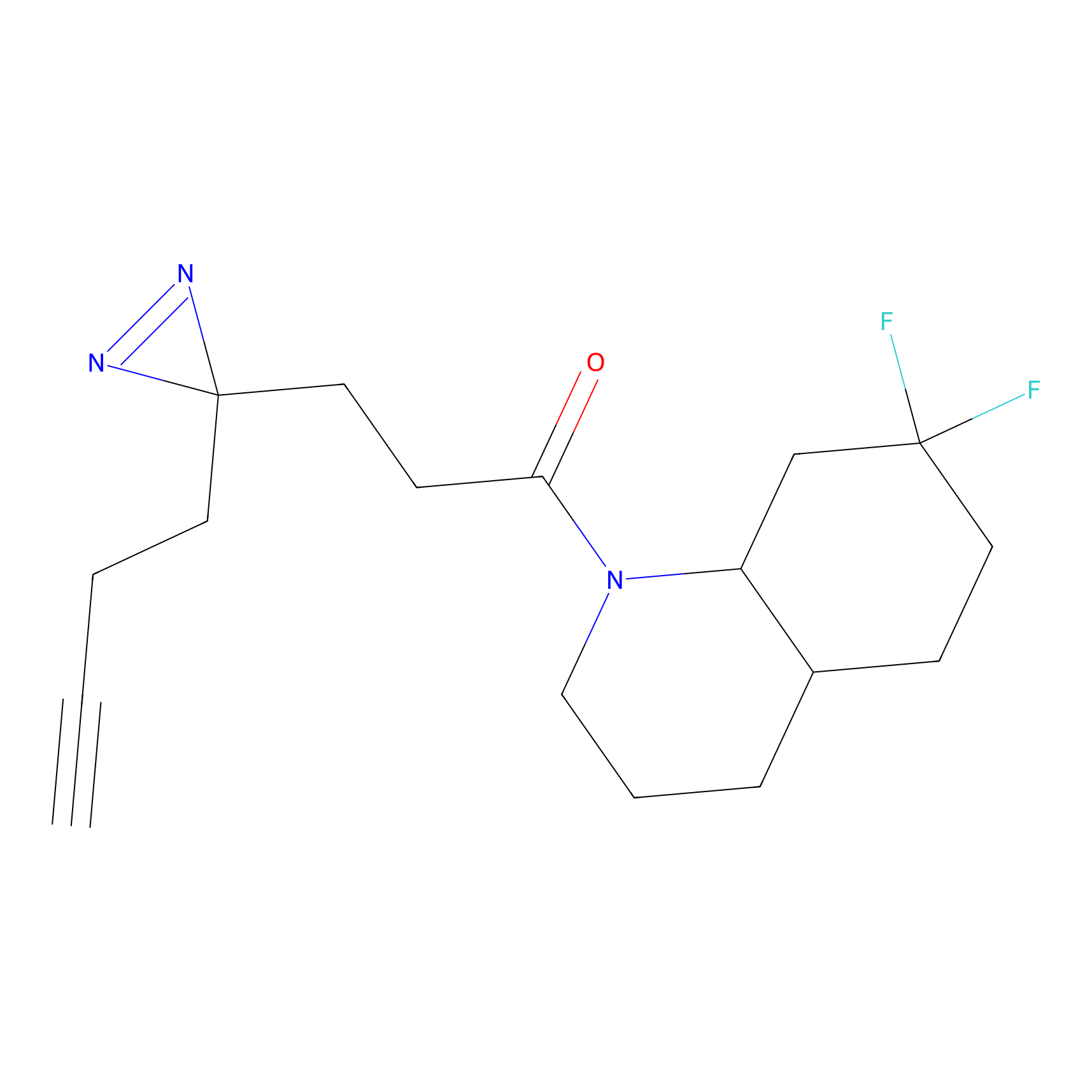
| LDCM0090 | Rapamycin | JHH-7 | 2.99 | LDD0213 | [10] |
The Interaction Atlas With This Target
References