Details of the Target
General Information of Target
| Target ID | LDTP16813 | |||||
|---|---|---|---|---|---|---|
| Target Name | Tetraspanin-3 (TSPAN3) | |||||
| Gene Name | TSPAN3 | |||||
| Gene ID | 10099 | |||||
| Synonyms |
TM4SF8; Tetraspanin-3; Tspan-3; Tetraspanin TM4-A; Transmembrane 4 superfamily member 8 |
|||||
| 3D Structure | ||||||
| Sequence |
MAVRQWVIALALAALLVVDREVPVAAGKLPFSRMPICEHMVESPTCSQMSNLVCGTDGLT
YTNECQLCLARIKTKQDIQIMKDGKC |
|||||
| Target Bioclass |
Transporter and channel
|
|||||
| Family |
Tetraspanin (TM4SF) family
|
|||||
| Subcellular location |
Membrane
|
|||||
| Function | Regulates the proliferation and migration of oligodendrocytes, a process essential for normal myelination and repair. | |||||
| Uniprot ID | ||||||
| Ensemble ID | ||||||
| HGNC ID | ||||||
Target Site Mutations in Different Cell Lines
Probe(s) Labeling This Target
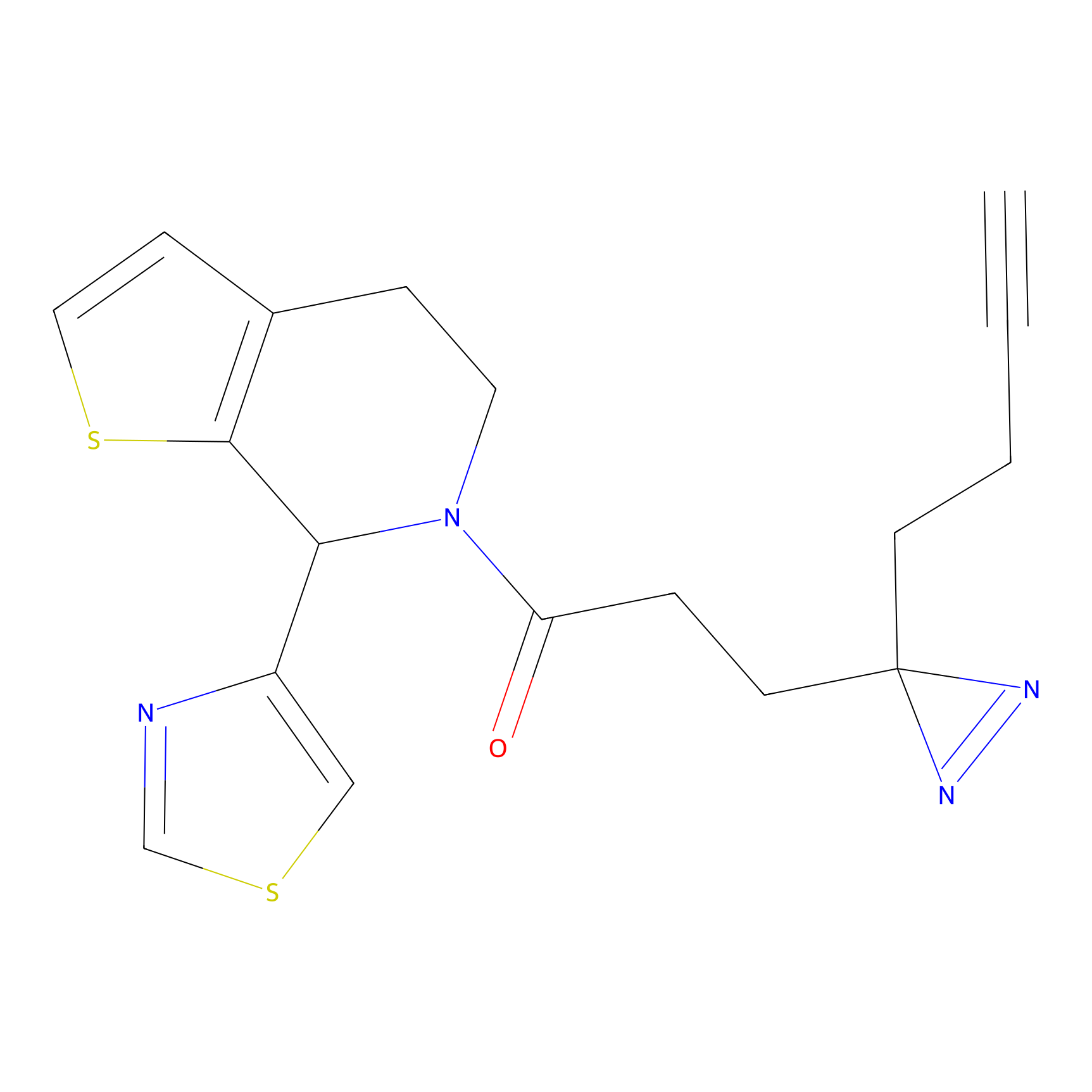
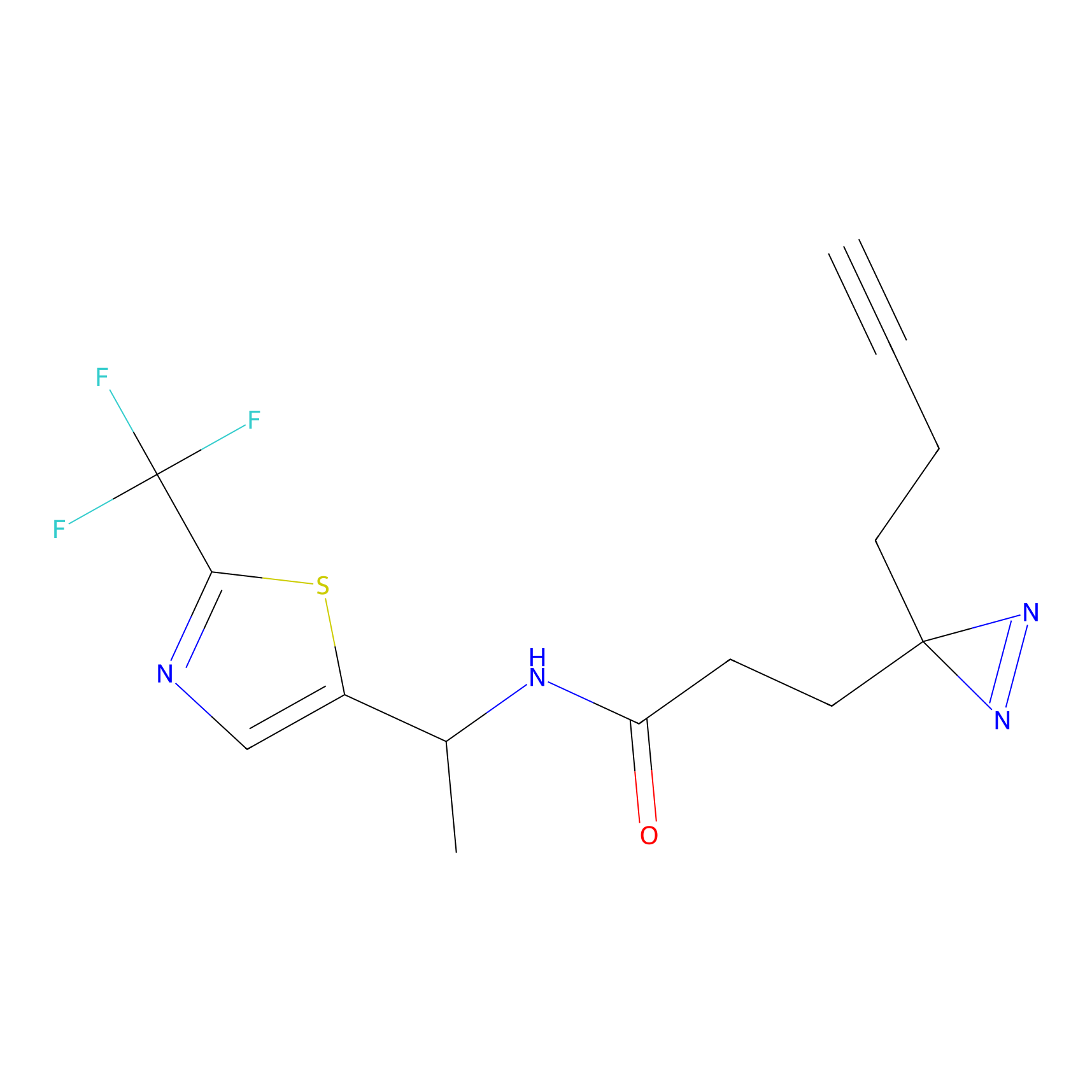
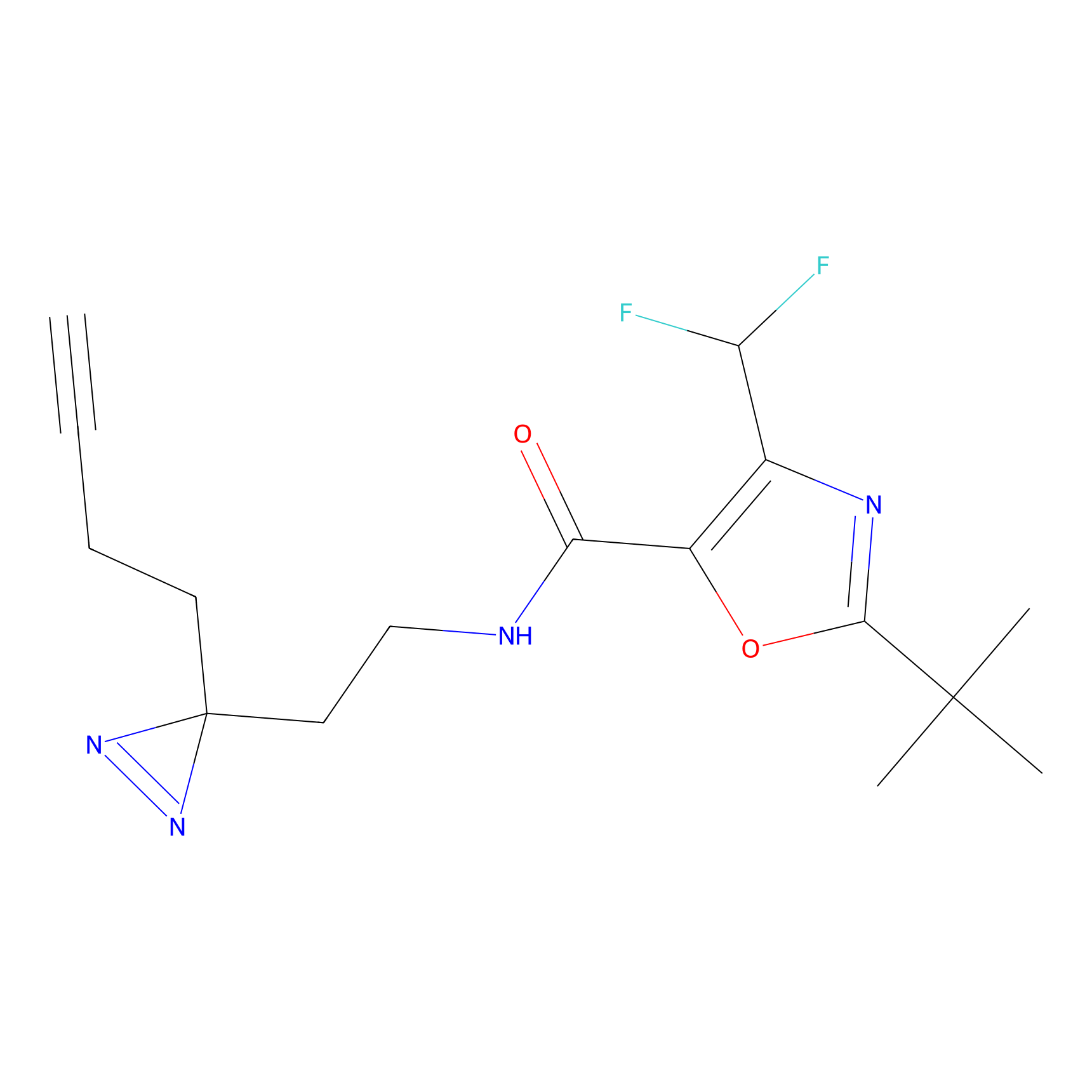
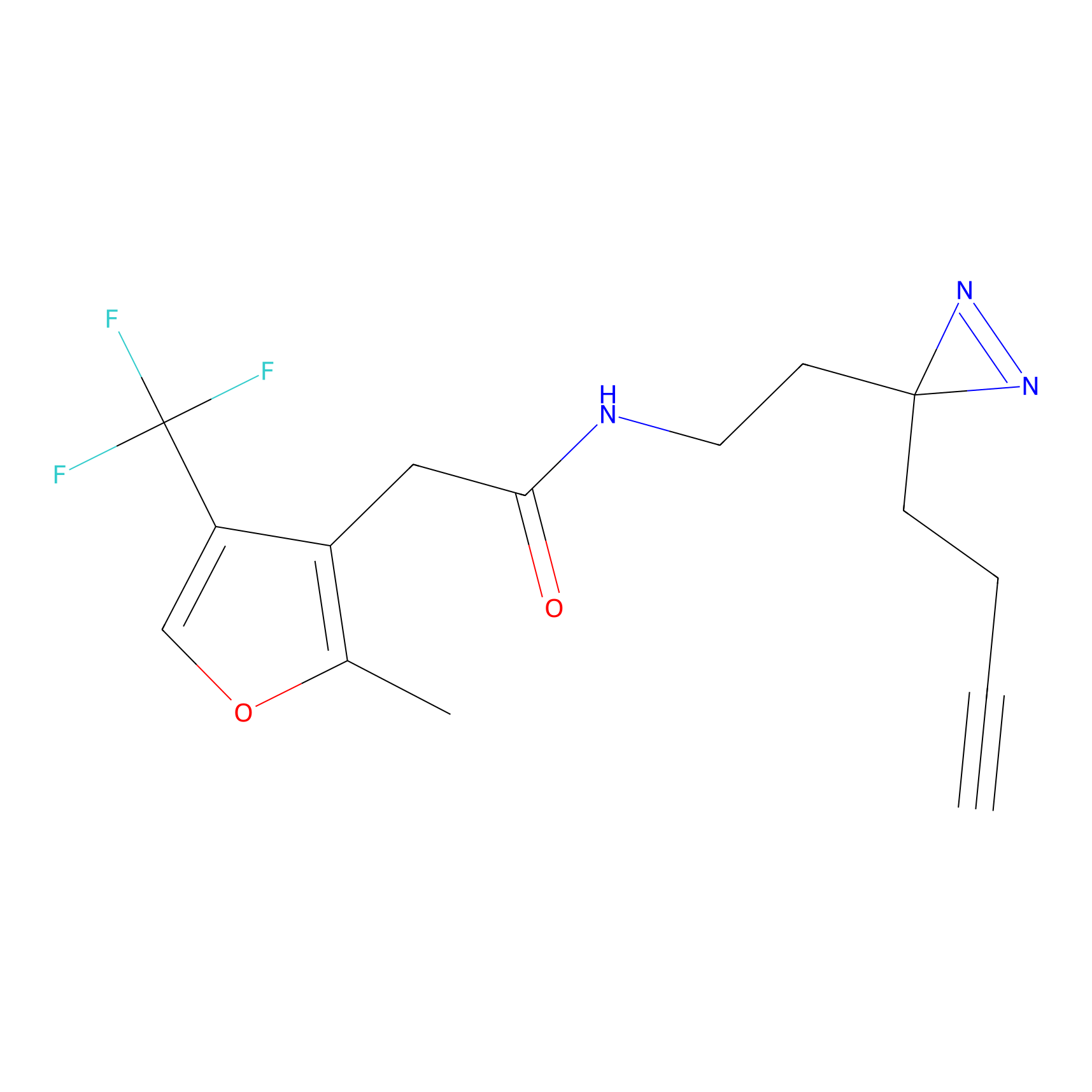
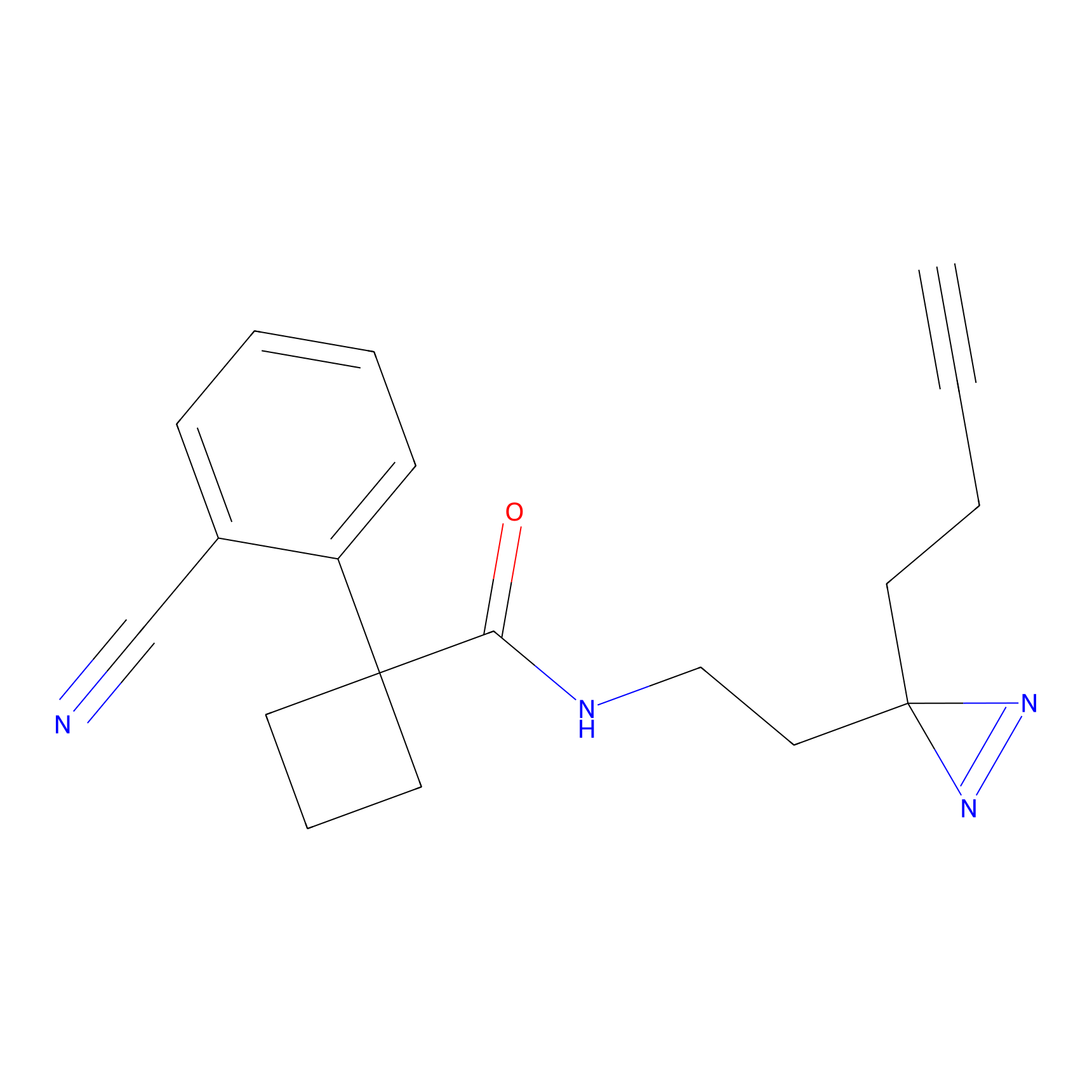
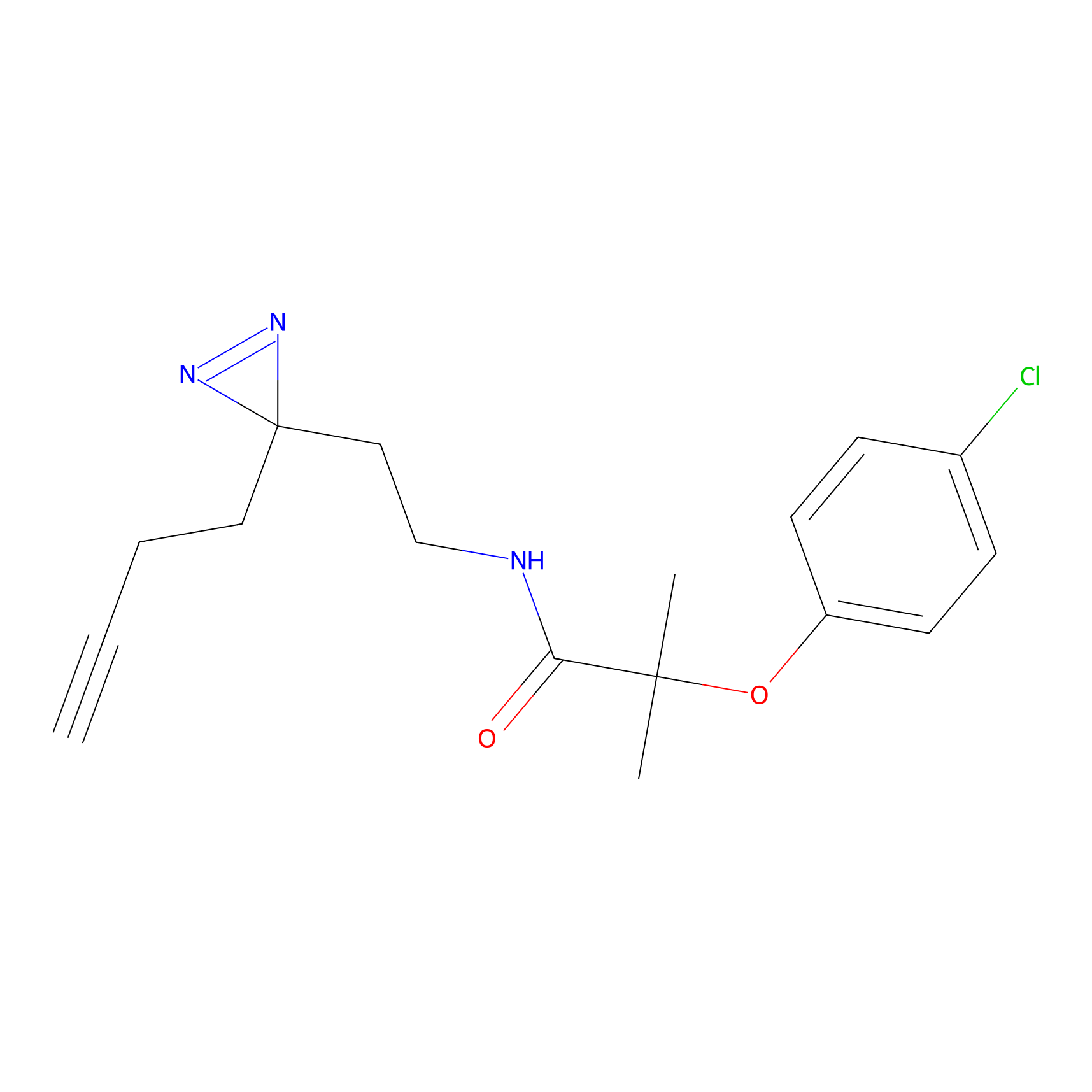
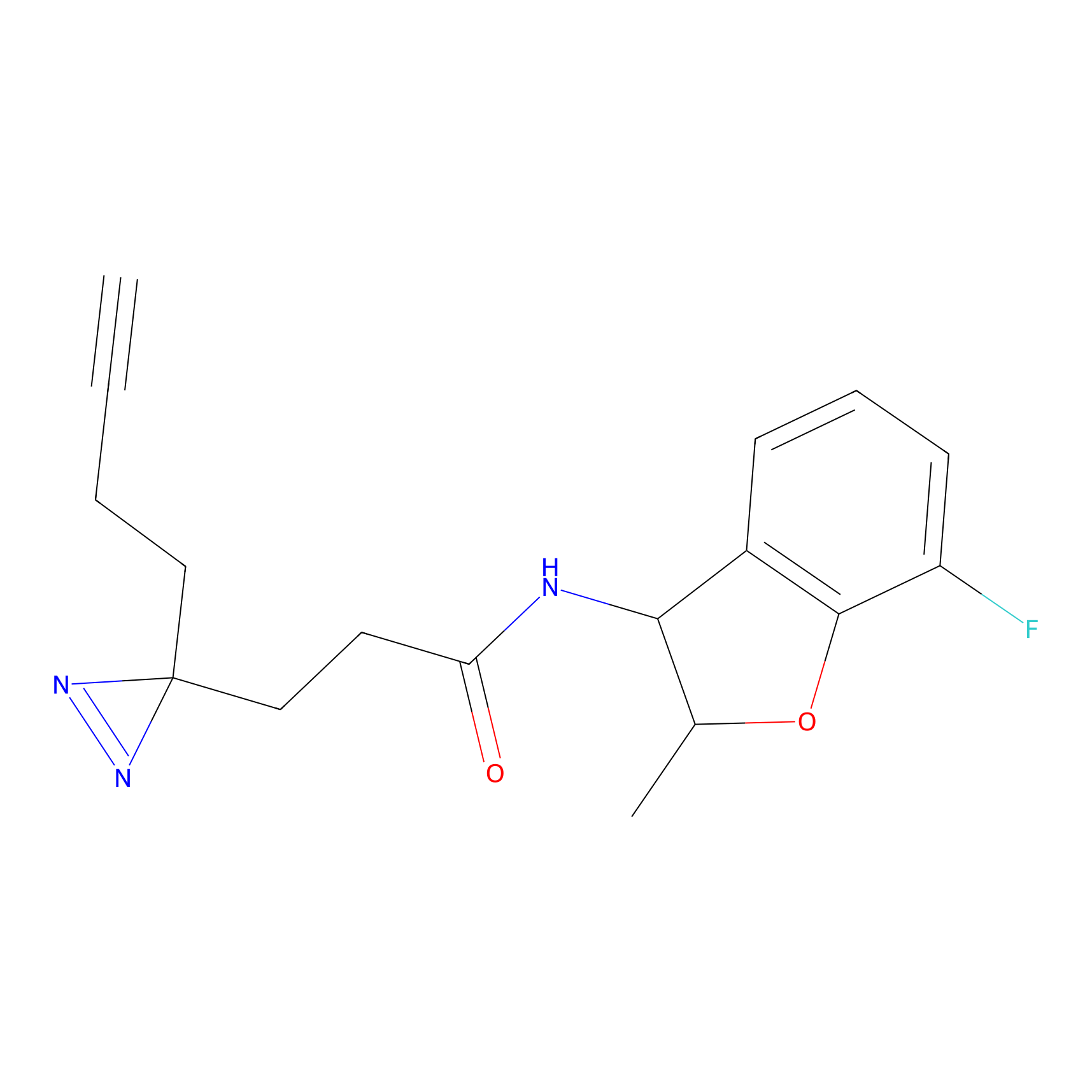
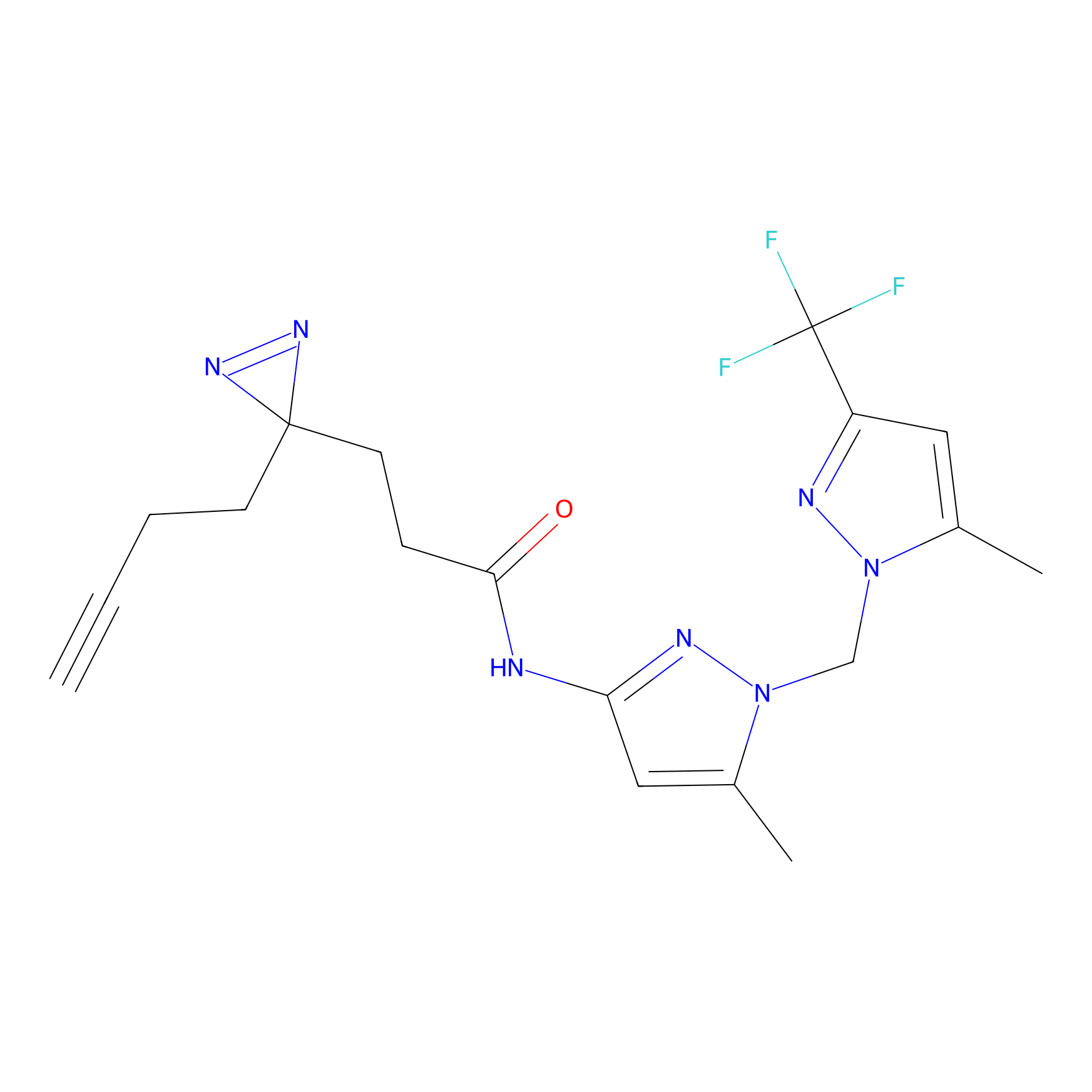
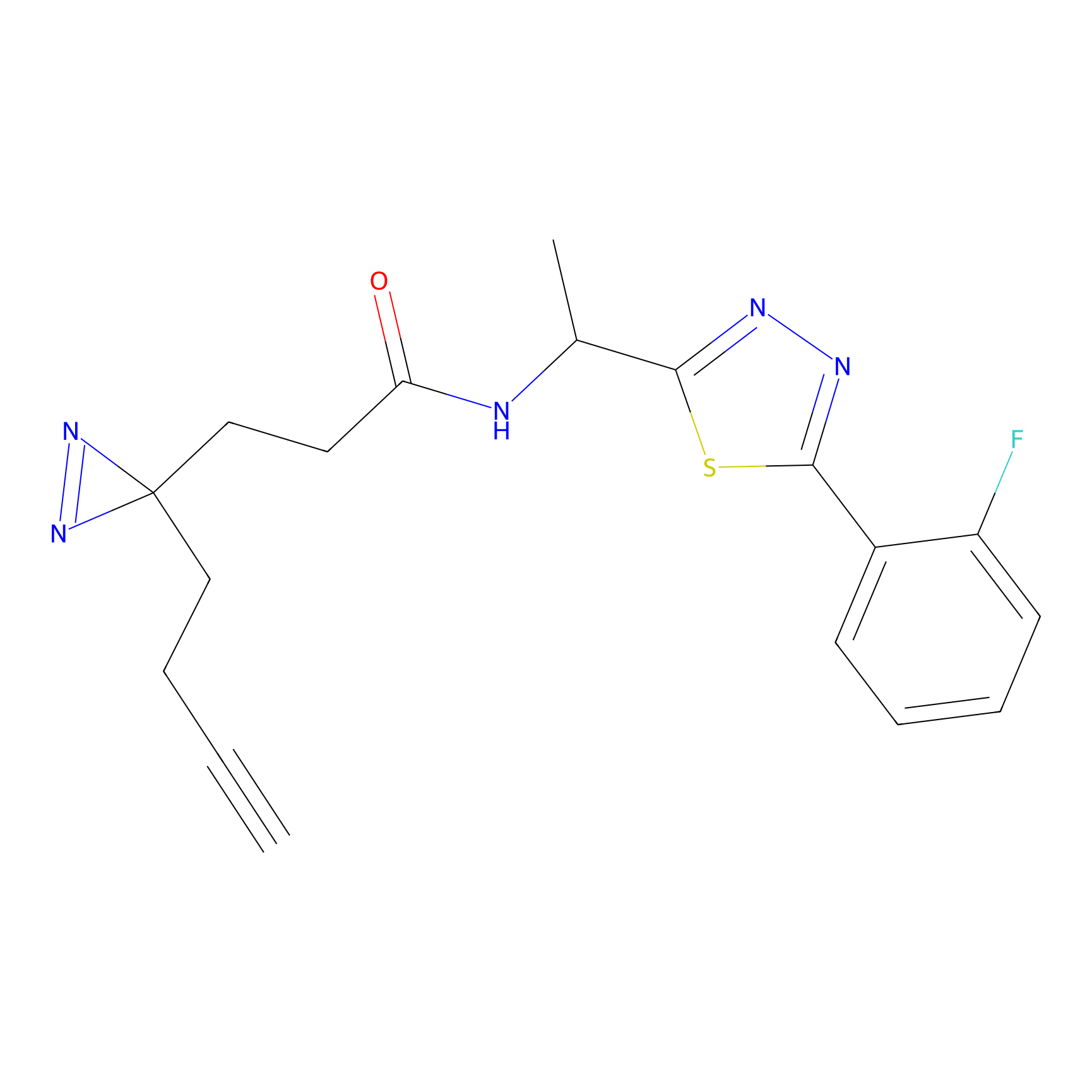
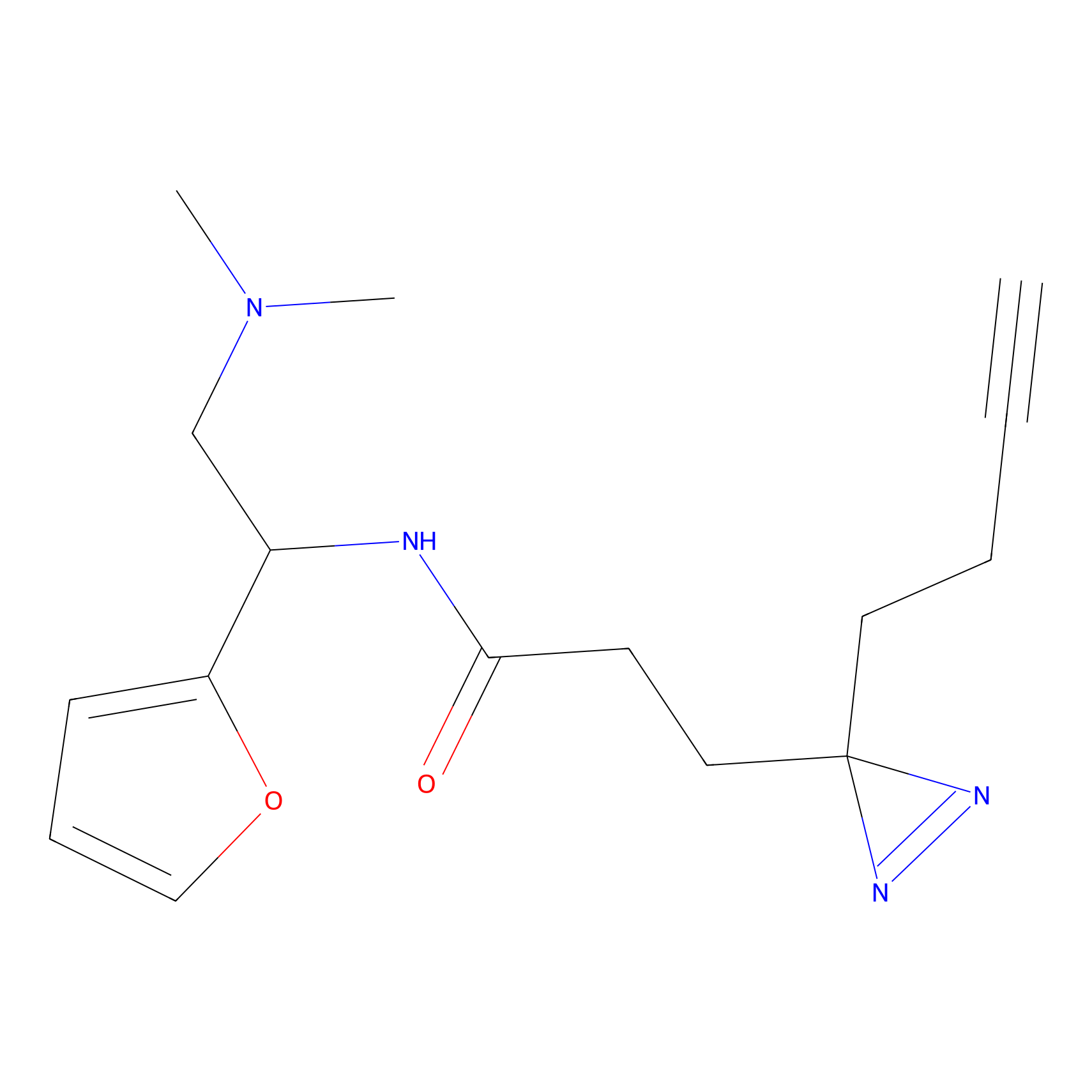
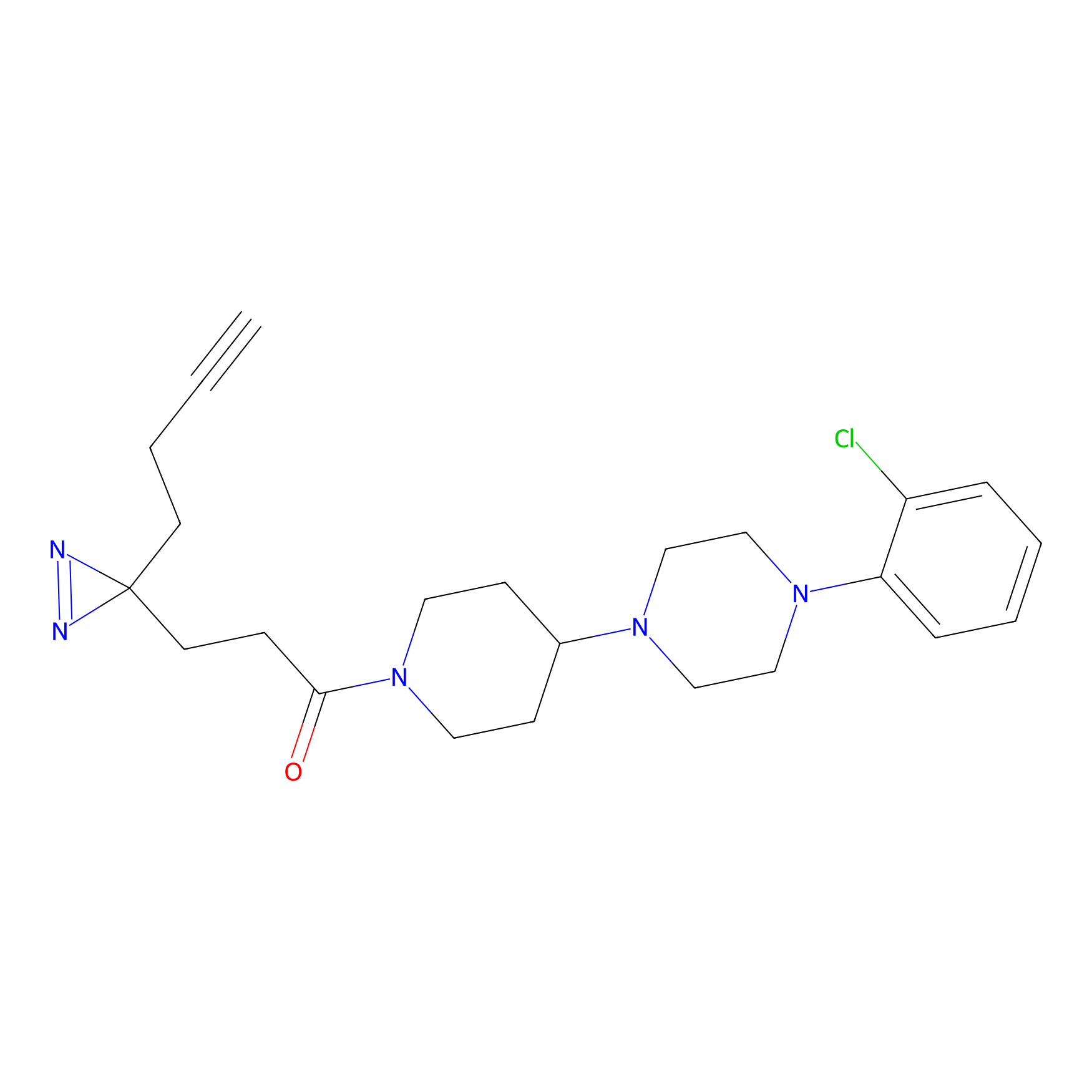
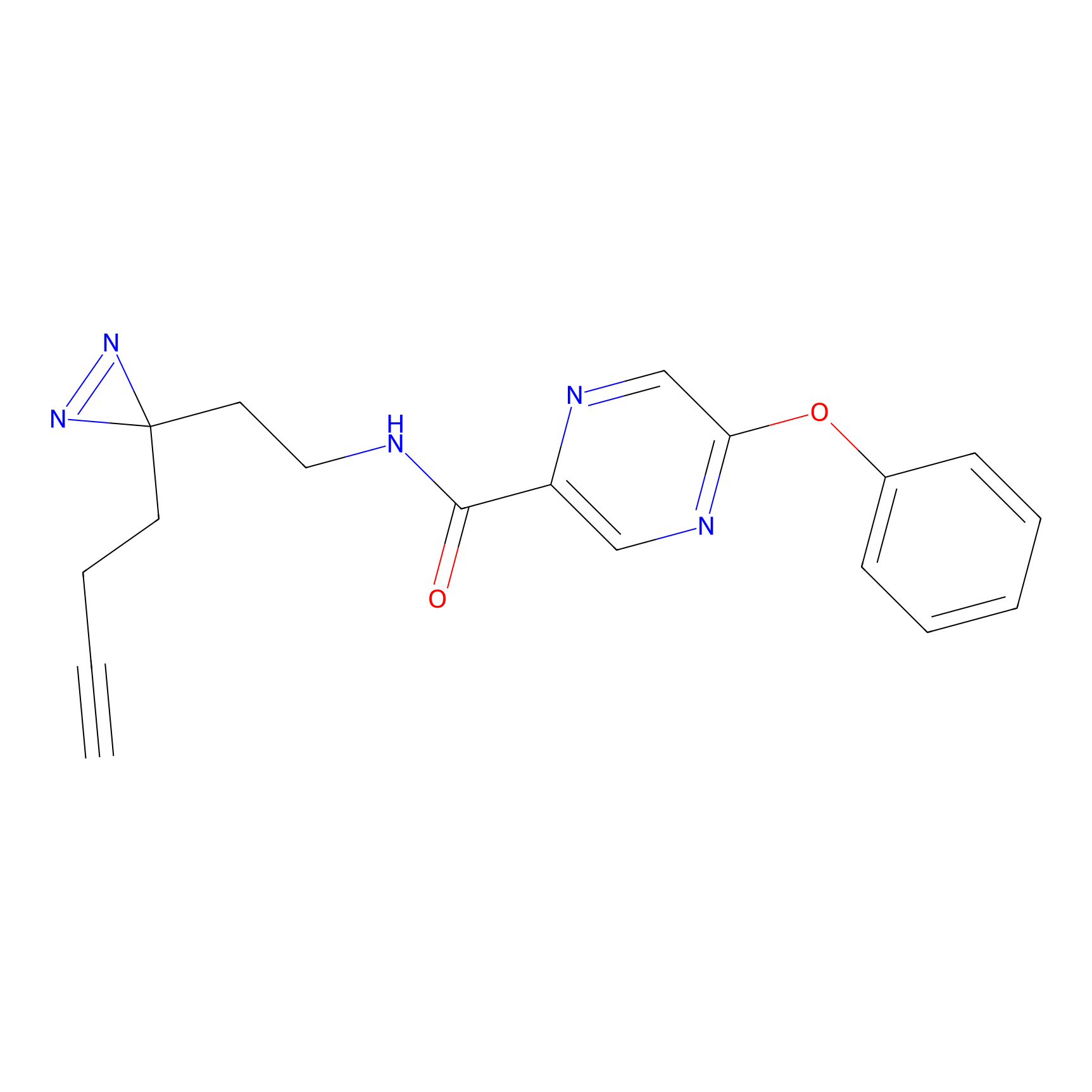
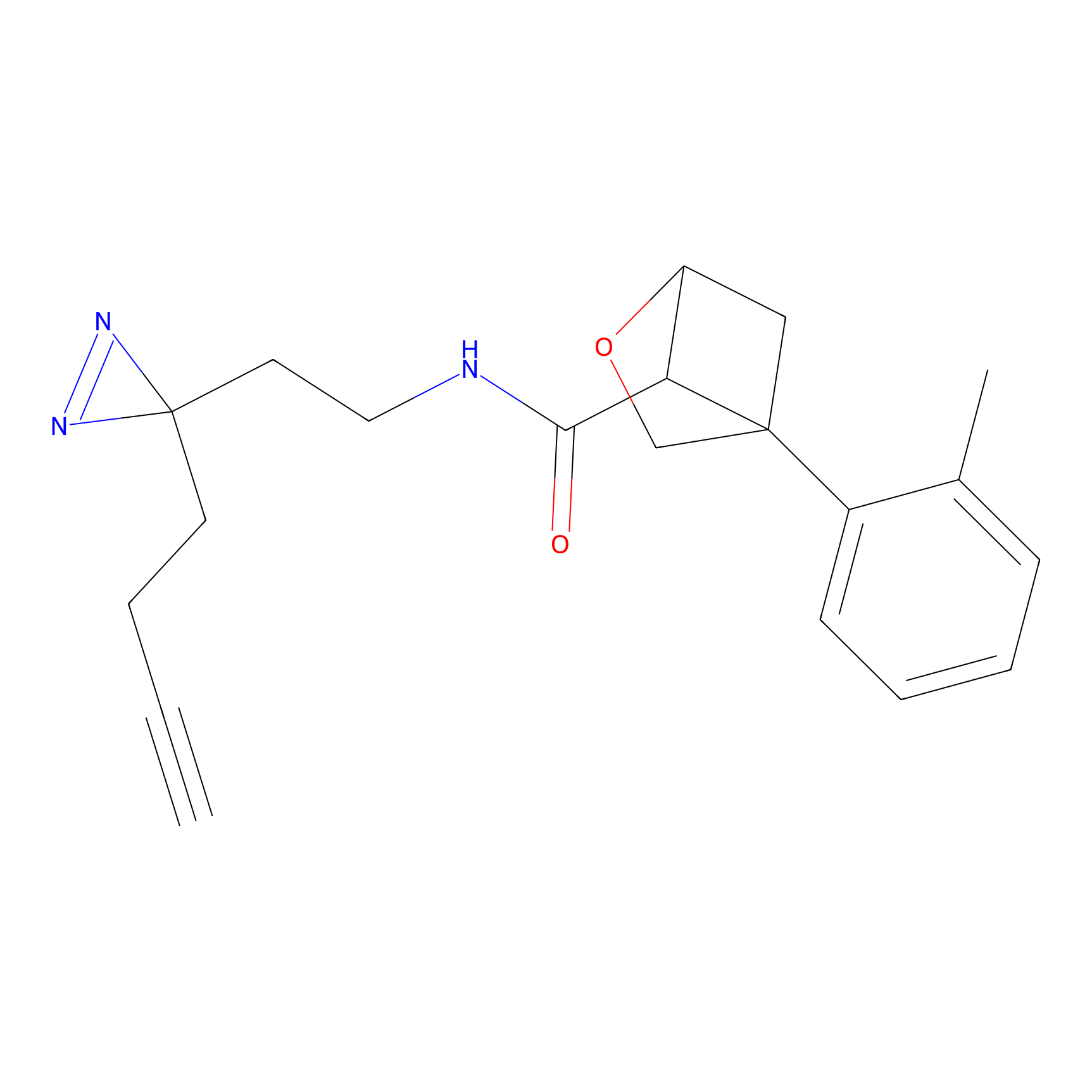
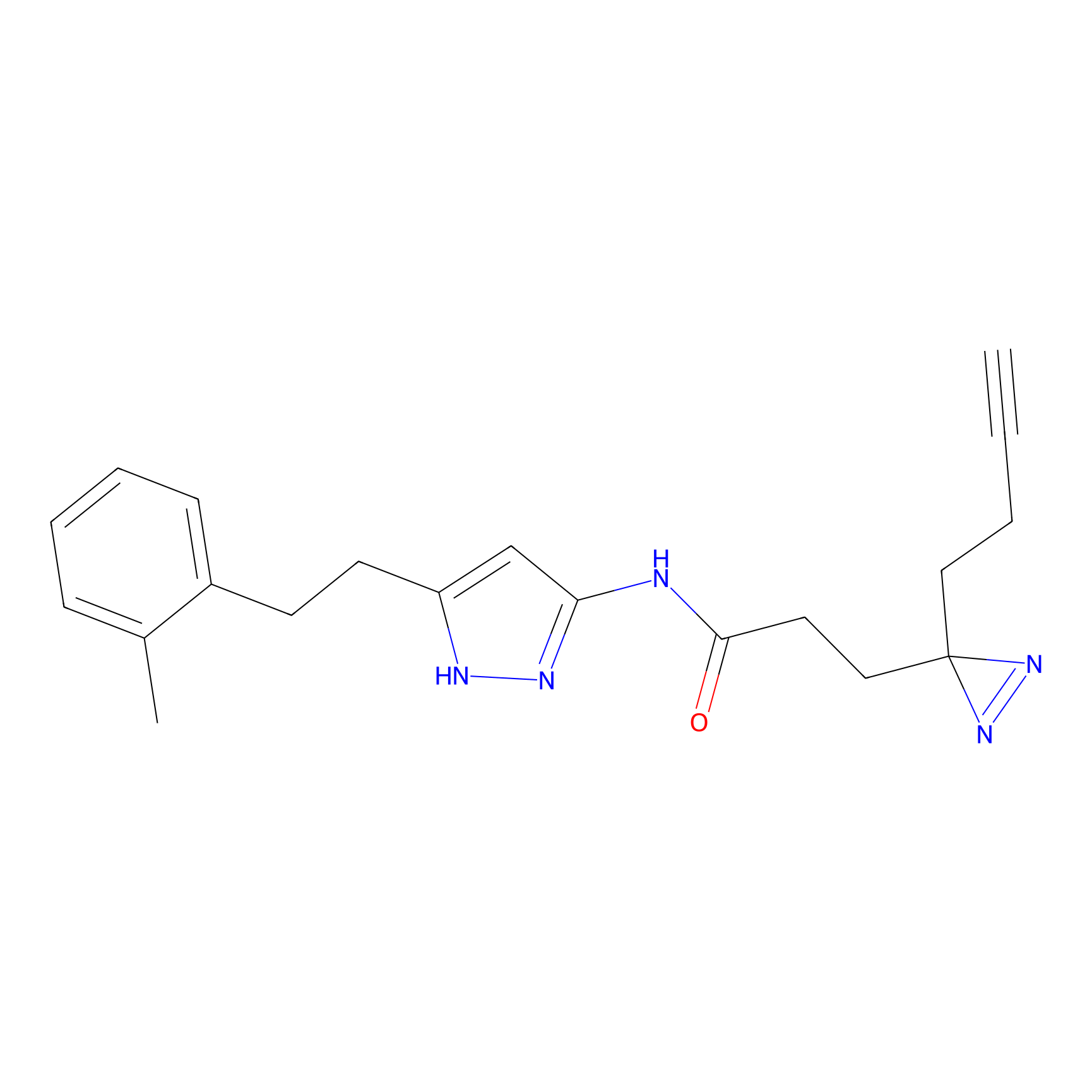
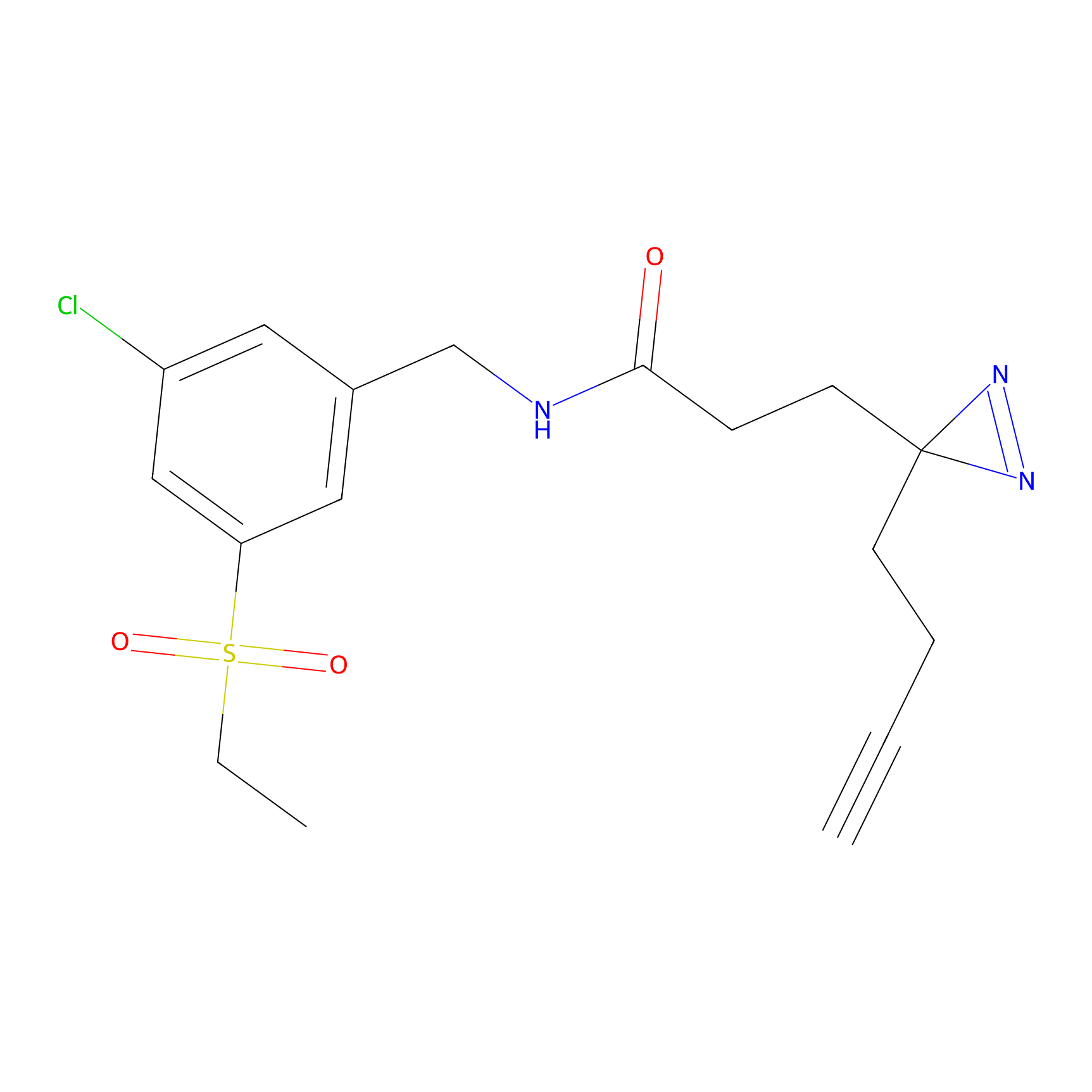
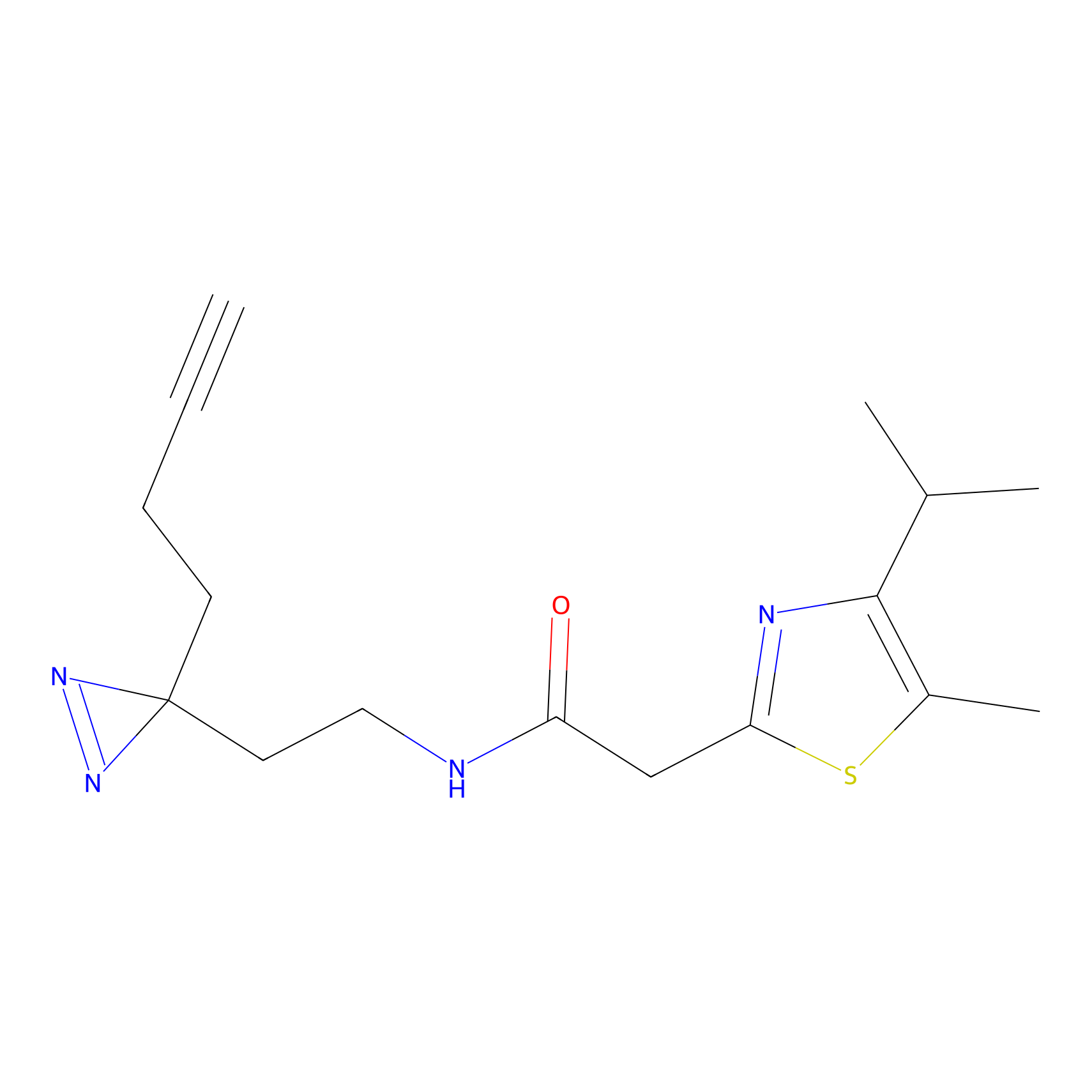
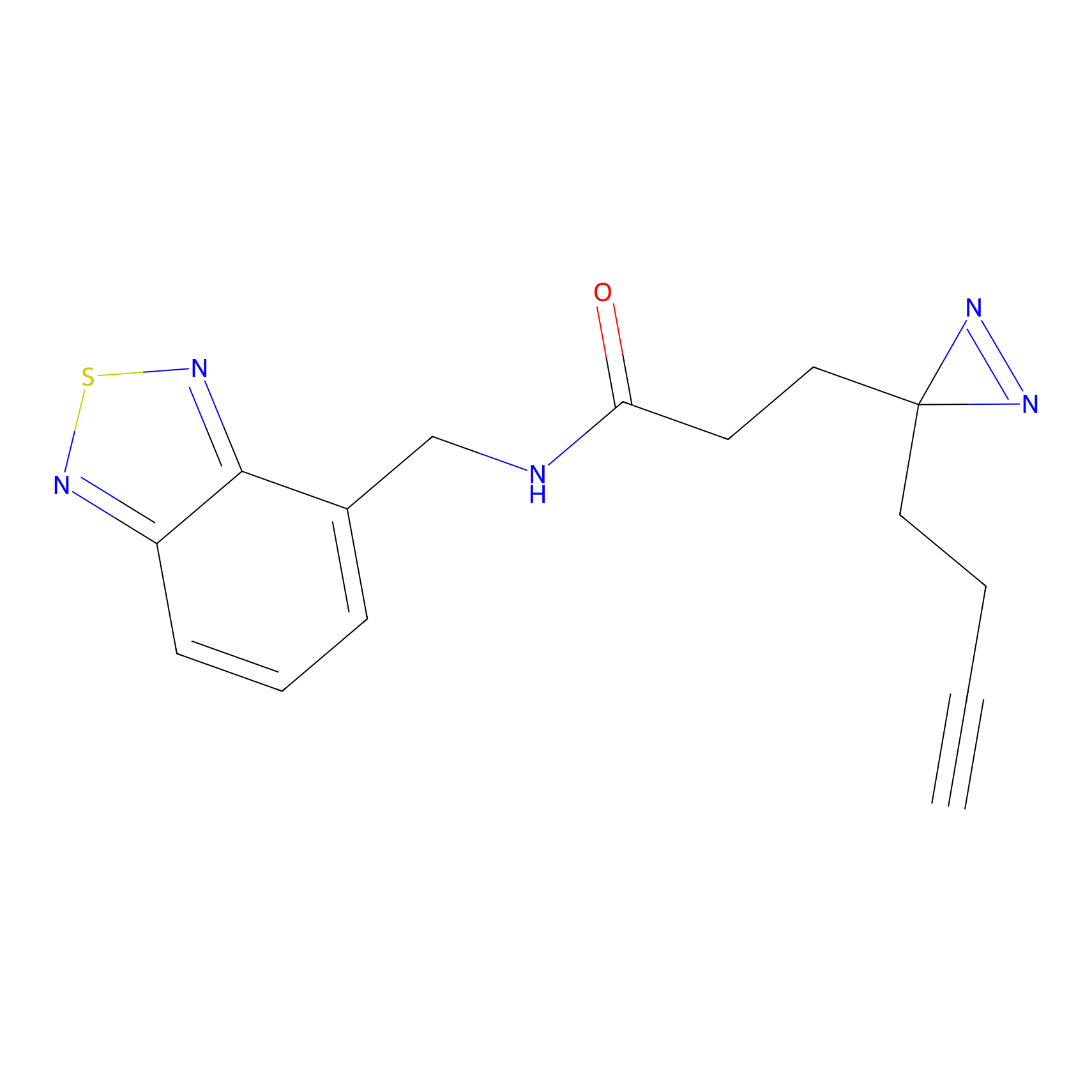
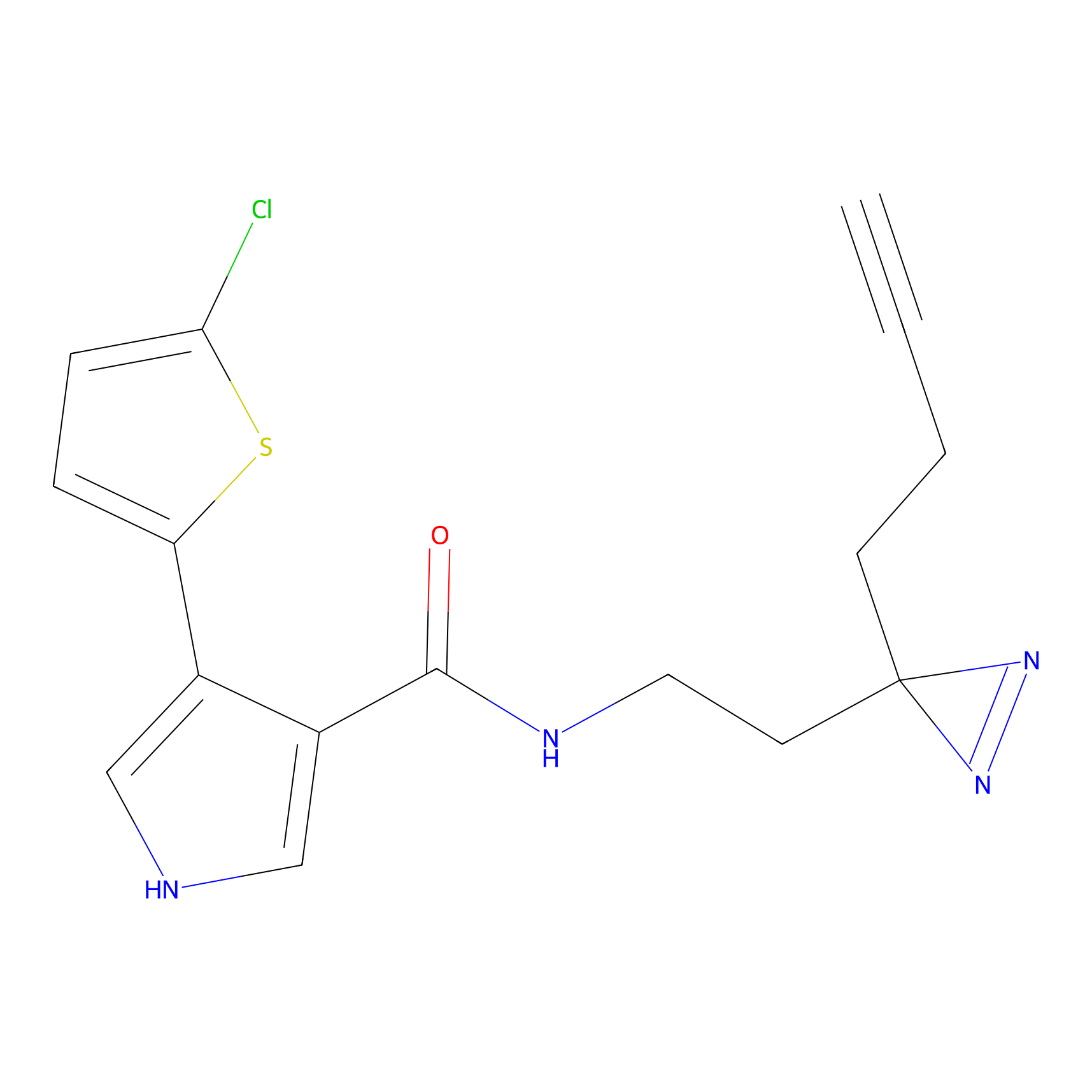
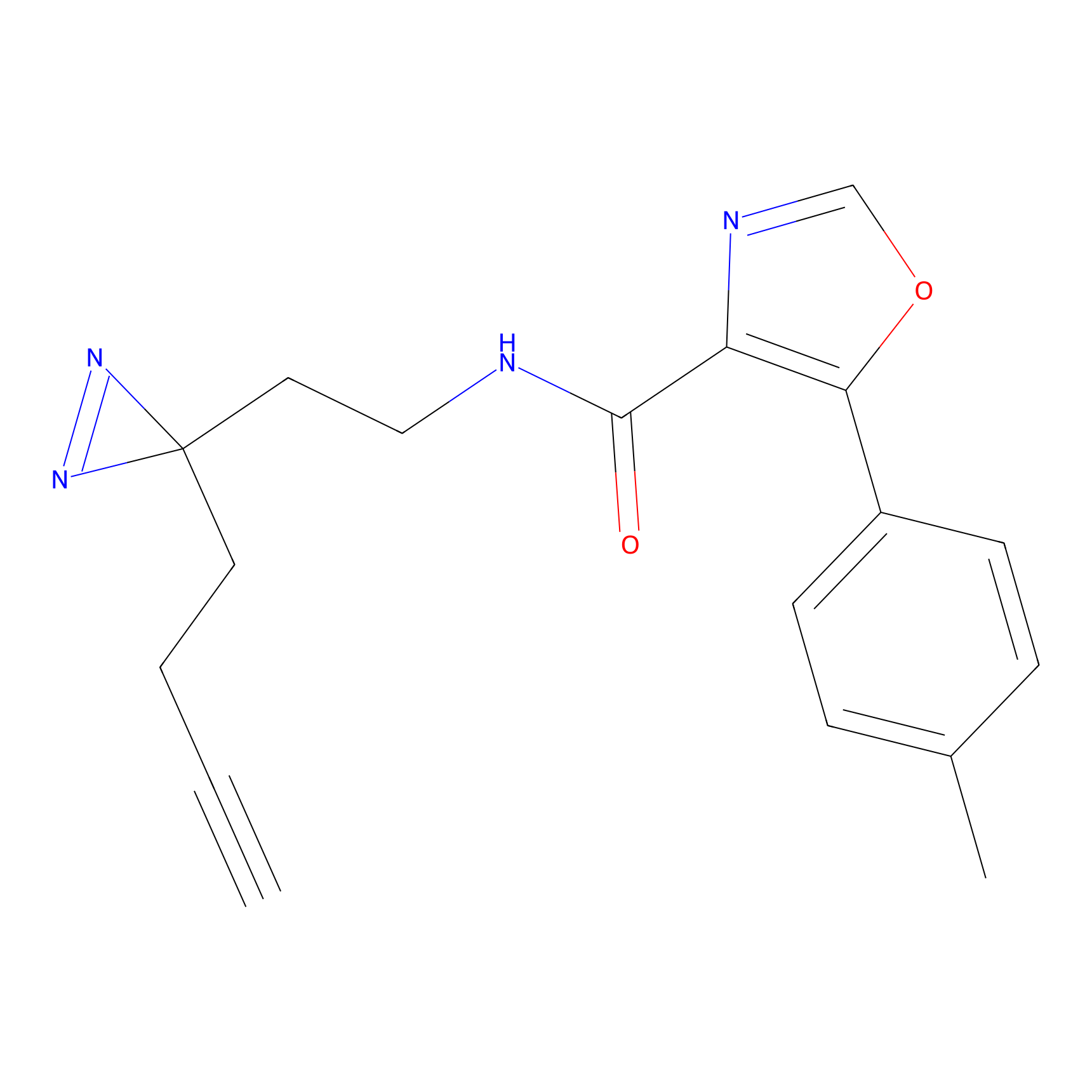
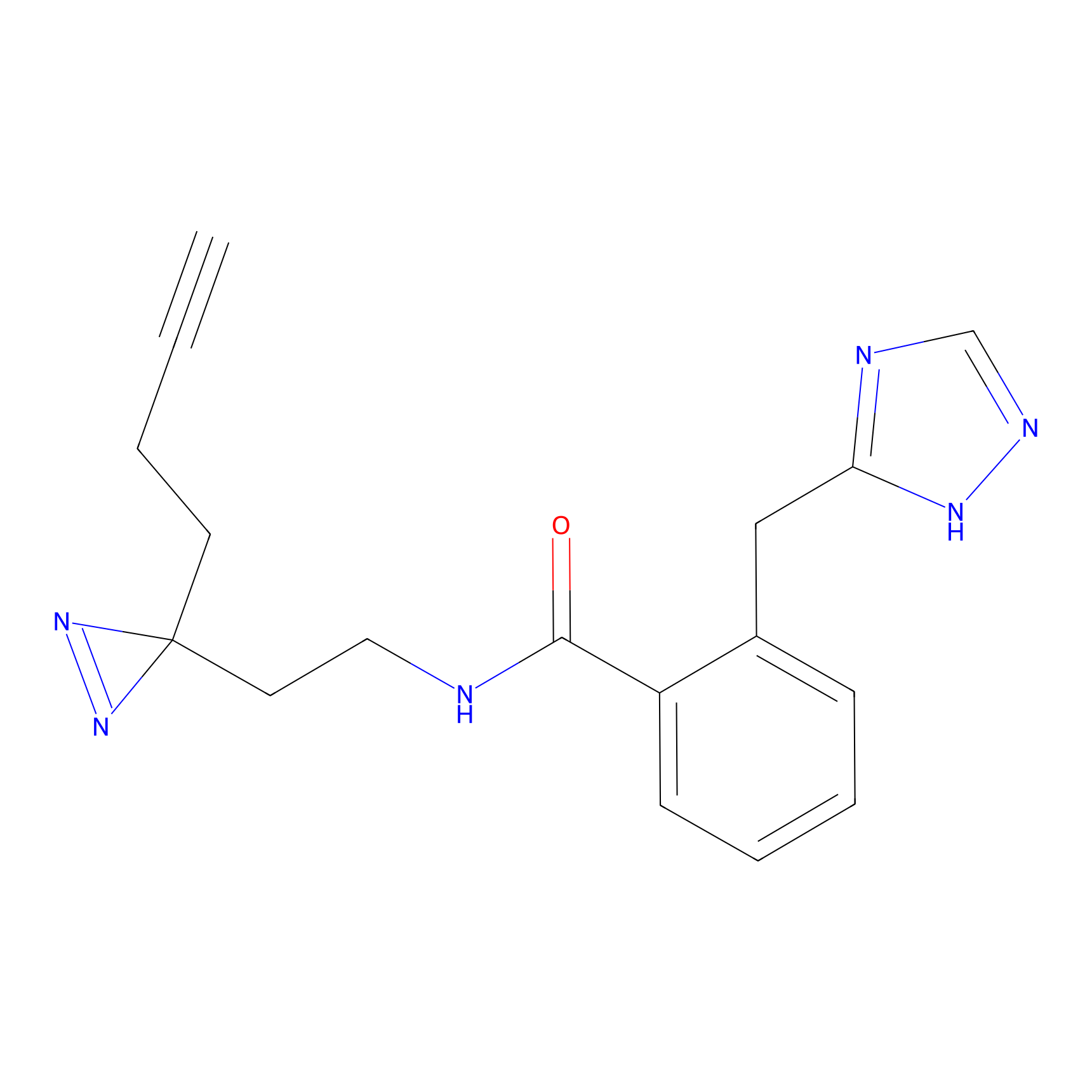
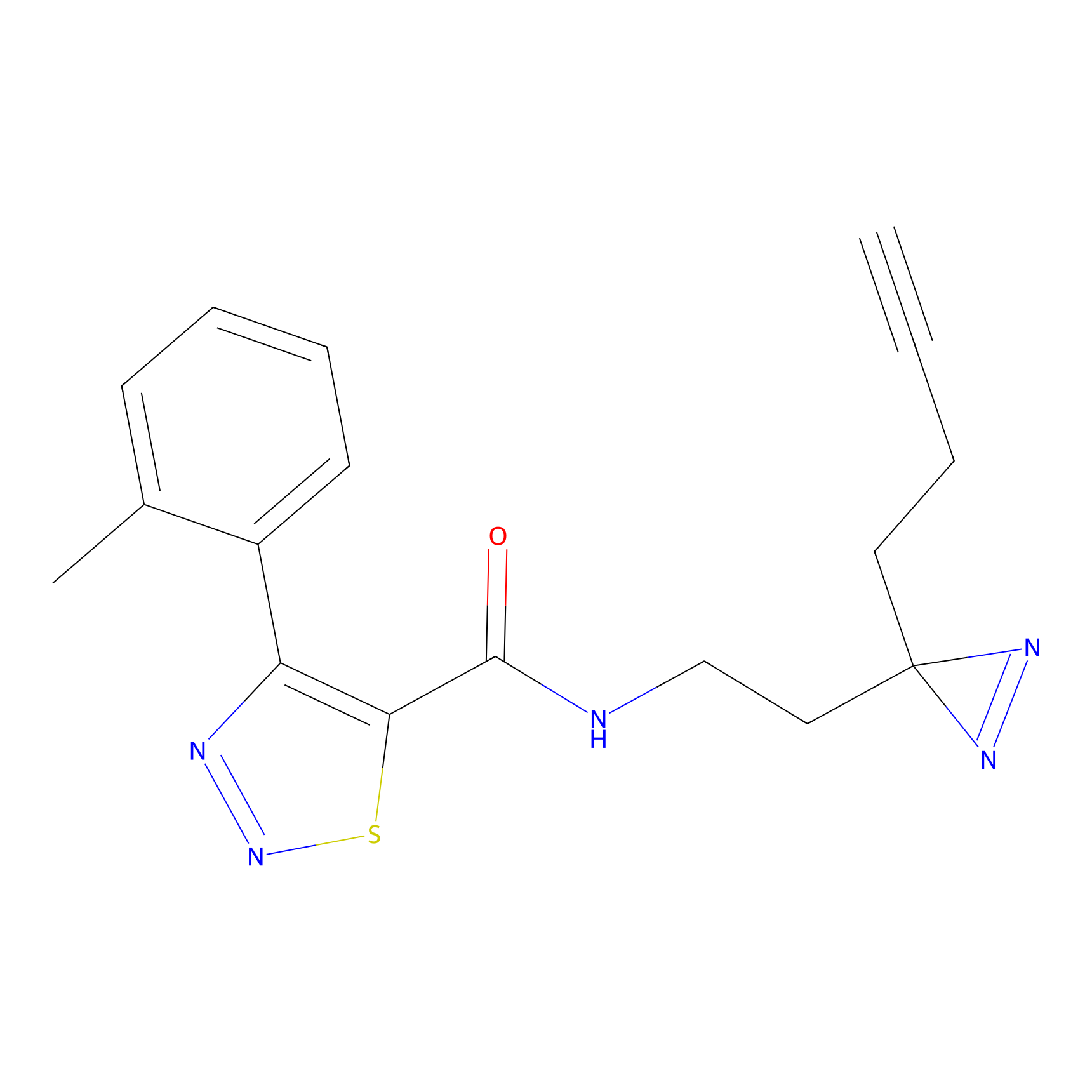
ABPP Probe
| Probe name | Structure | Binding Site(Ratio) | Interaction ID | Ref | |
|---|---|---|---|---|---|
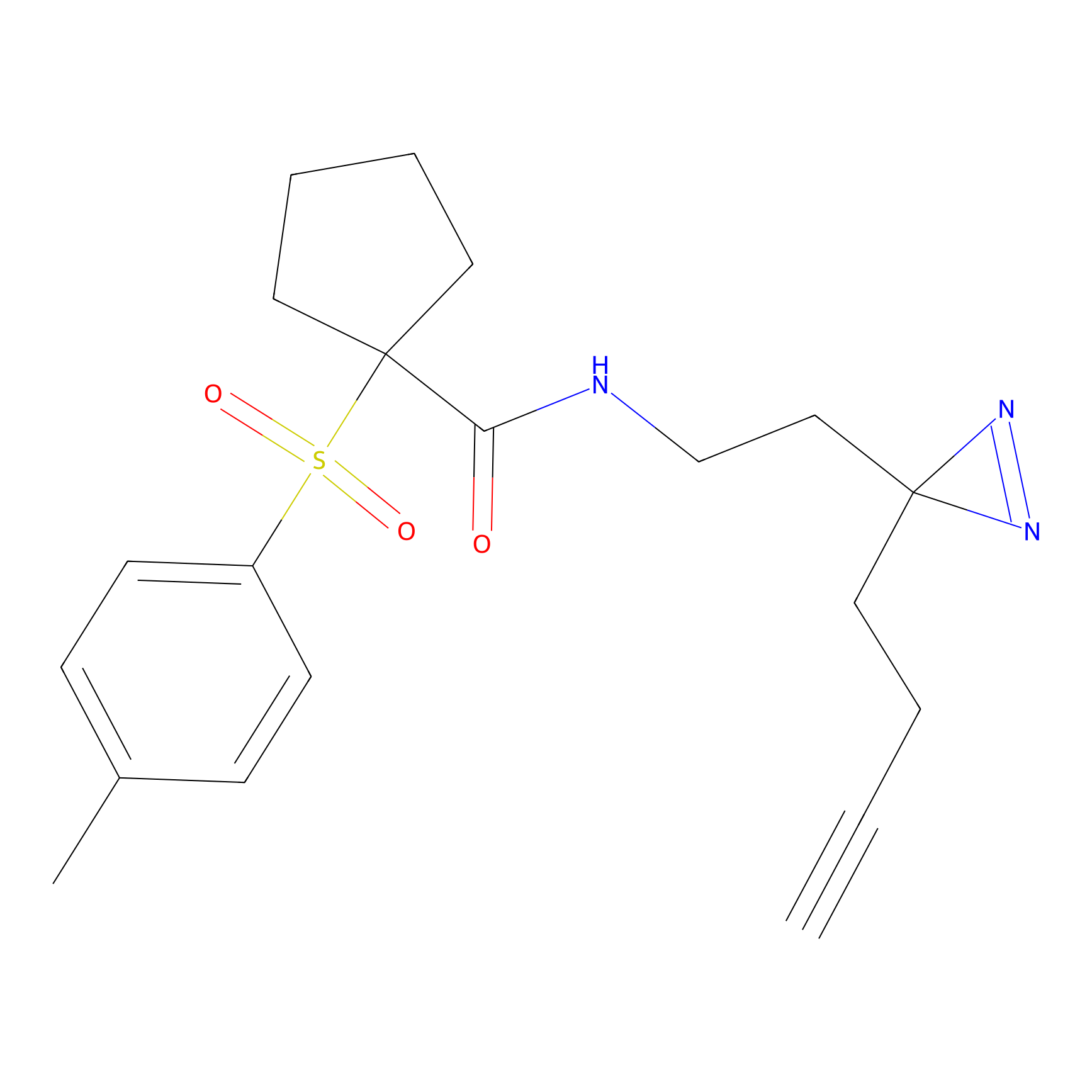
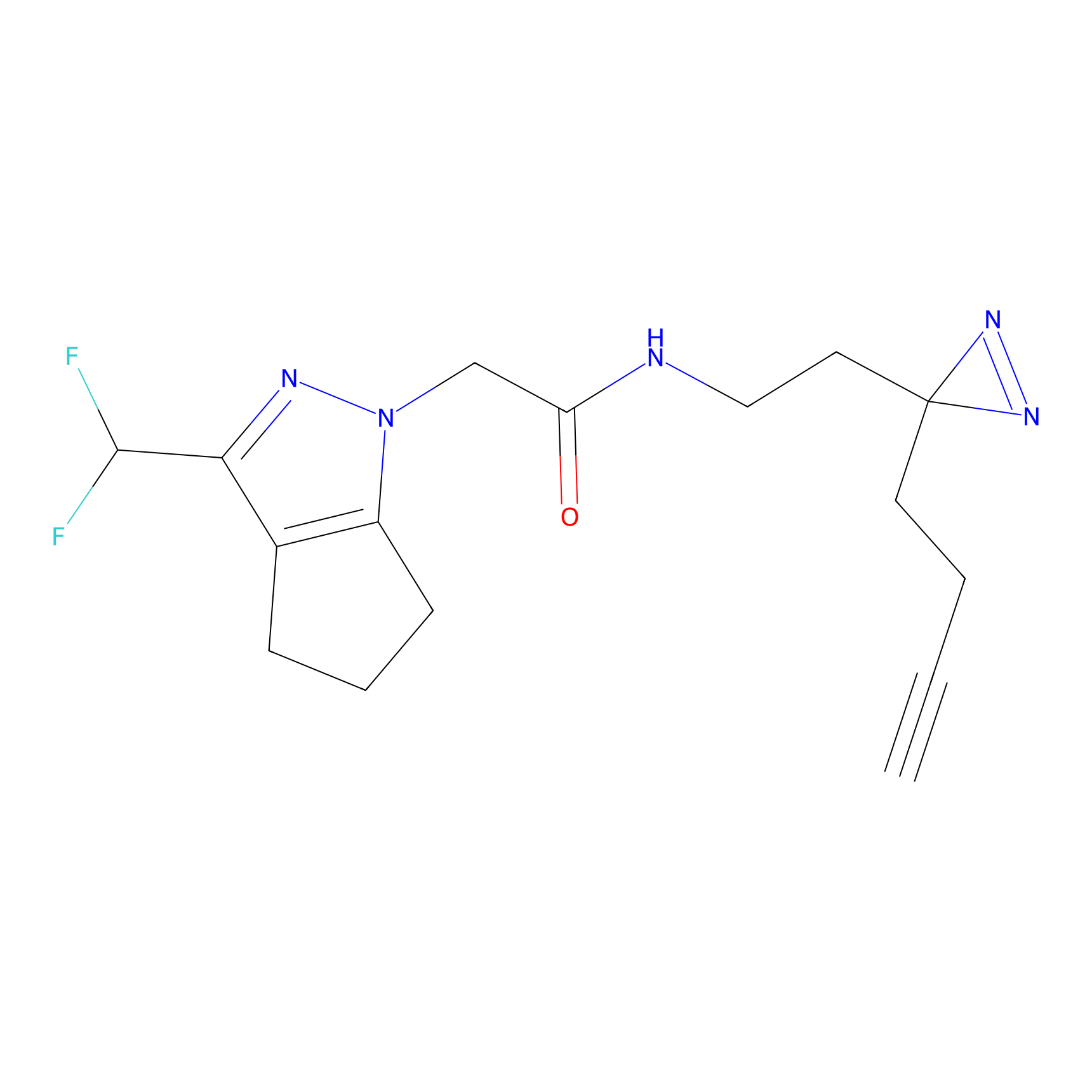
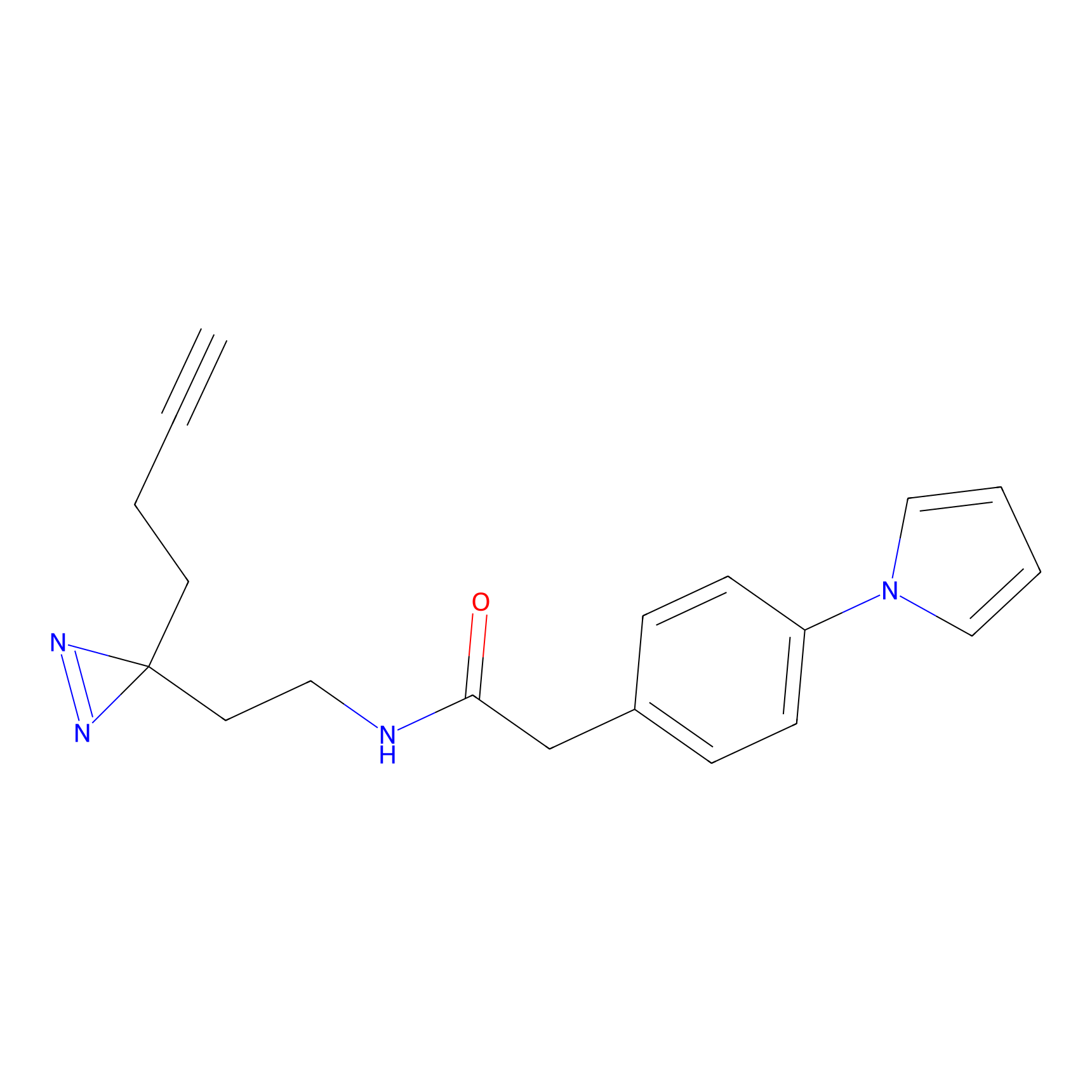
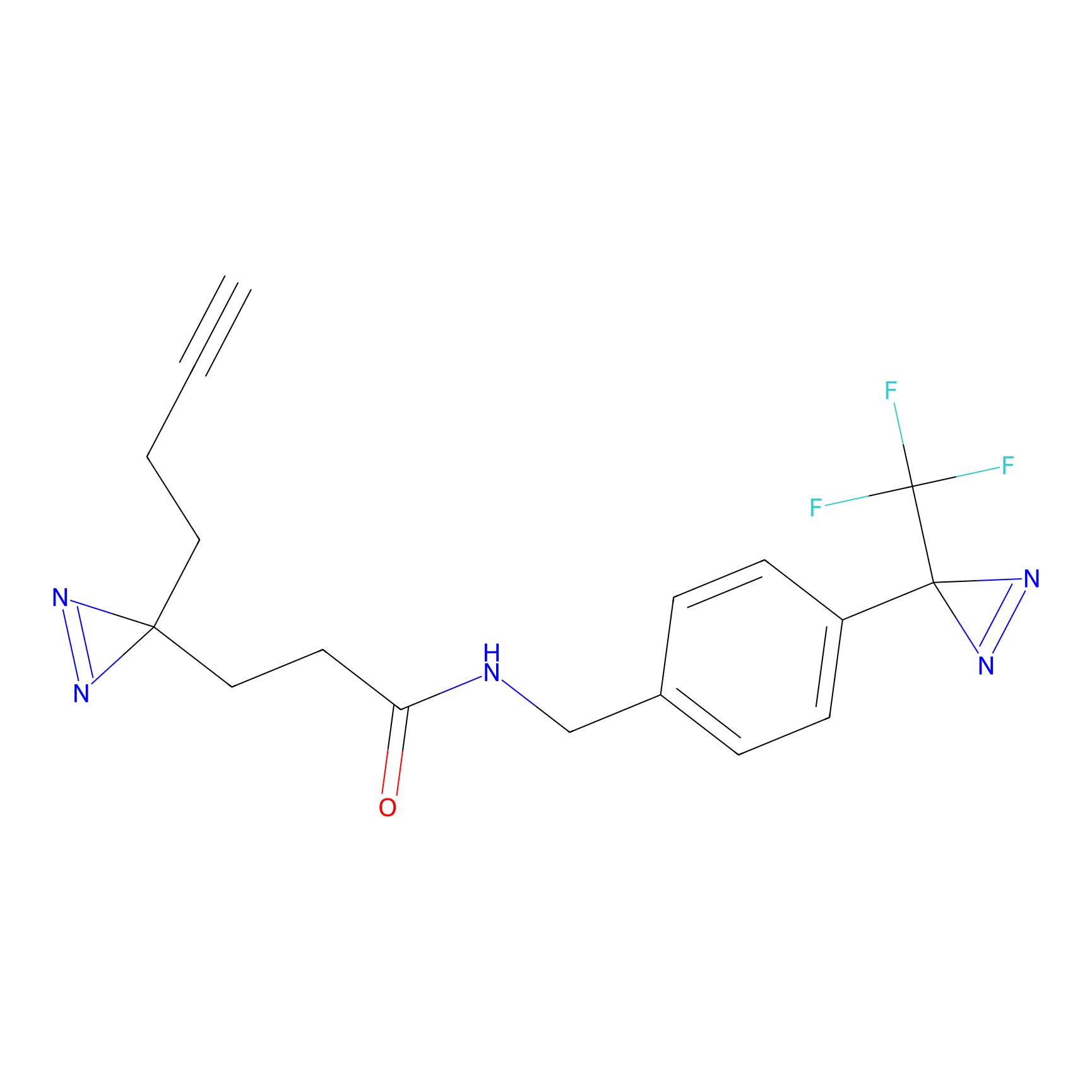
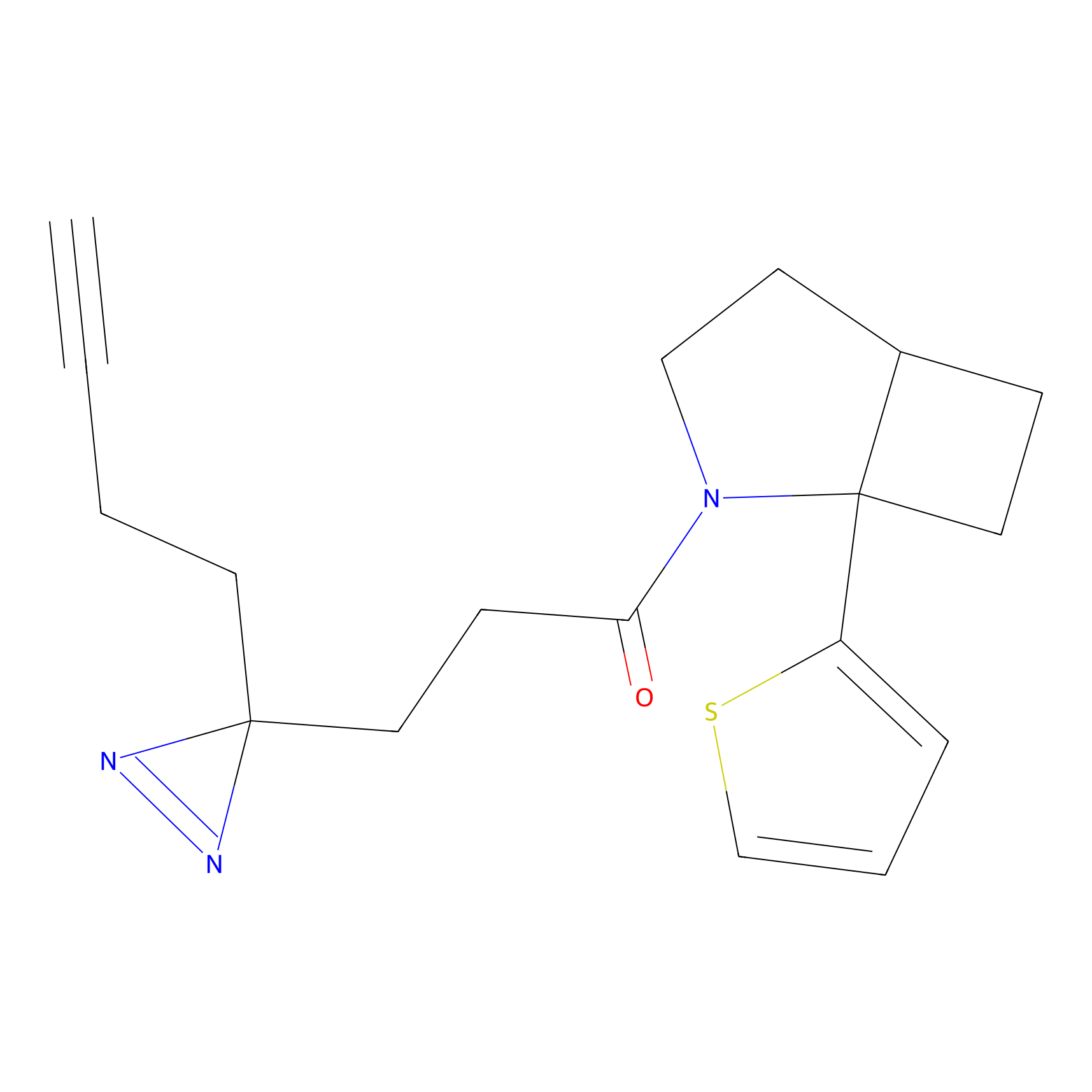
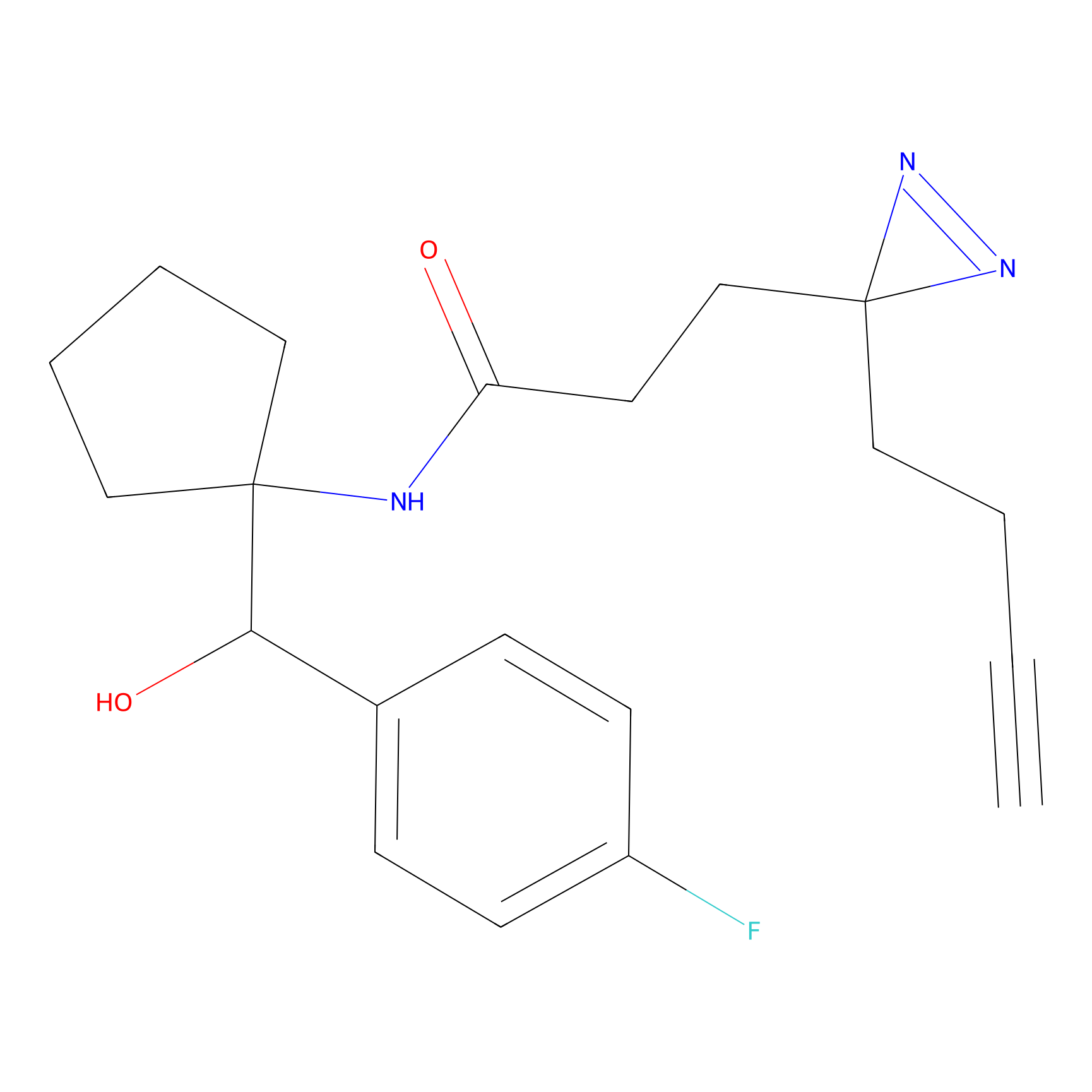
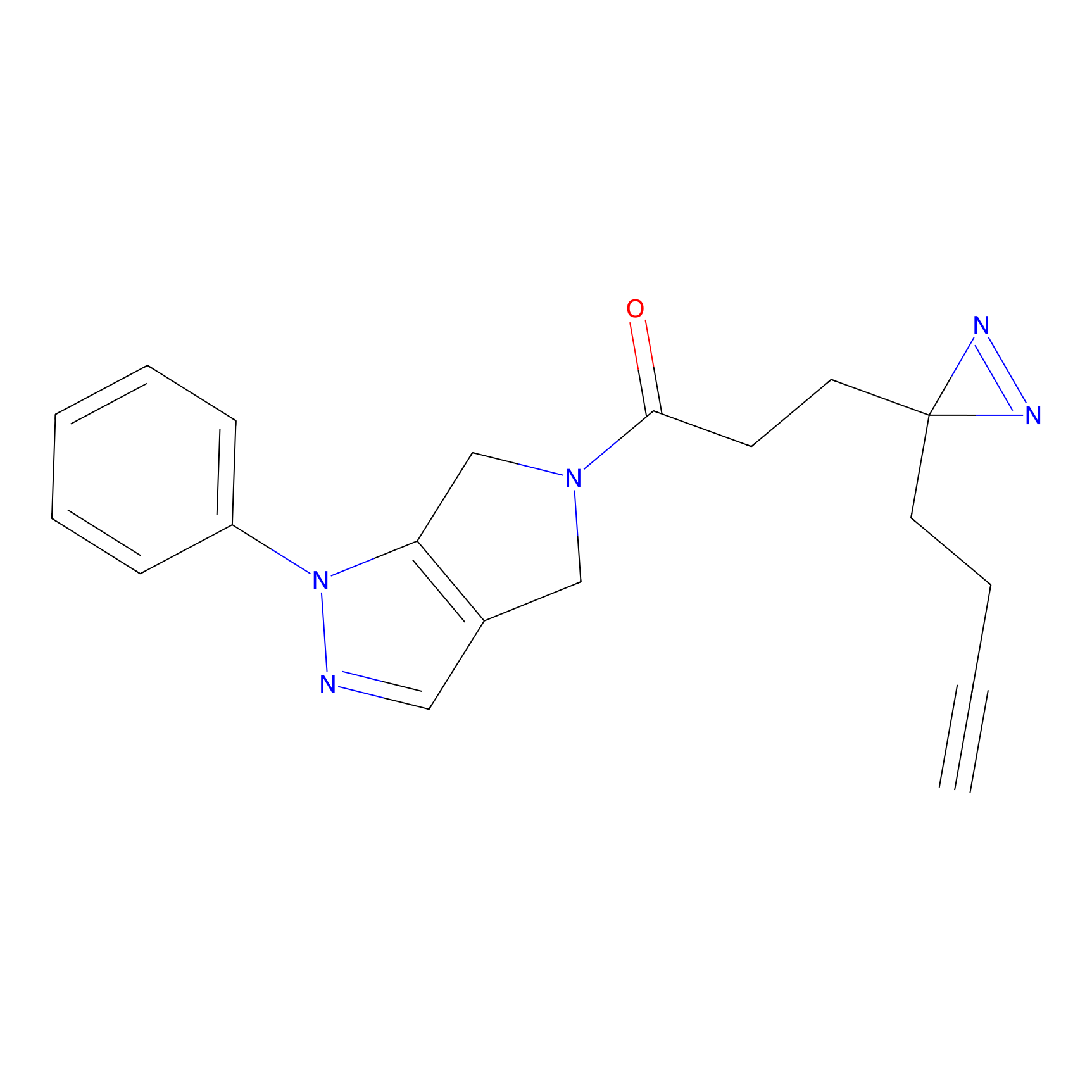
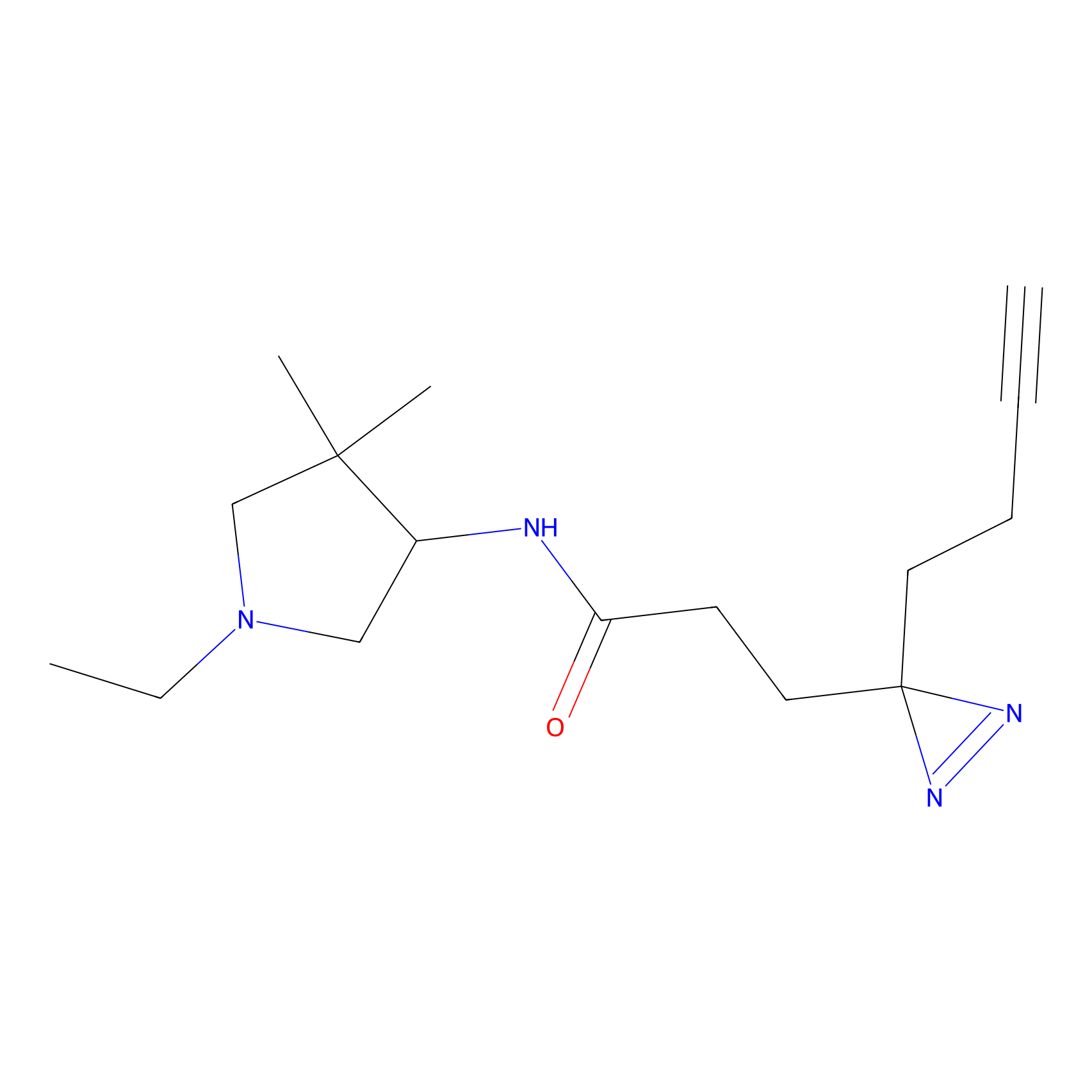
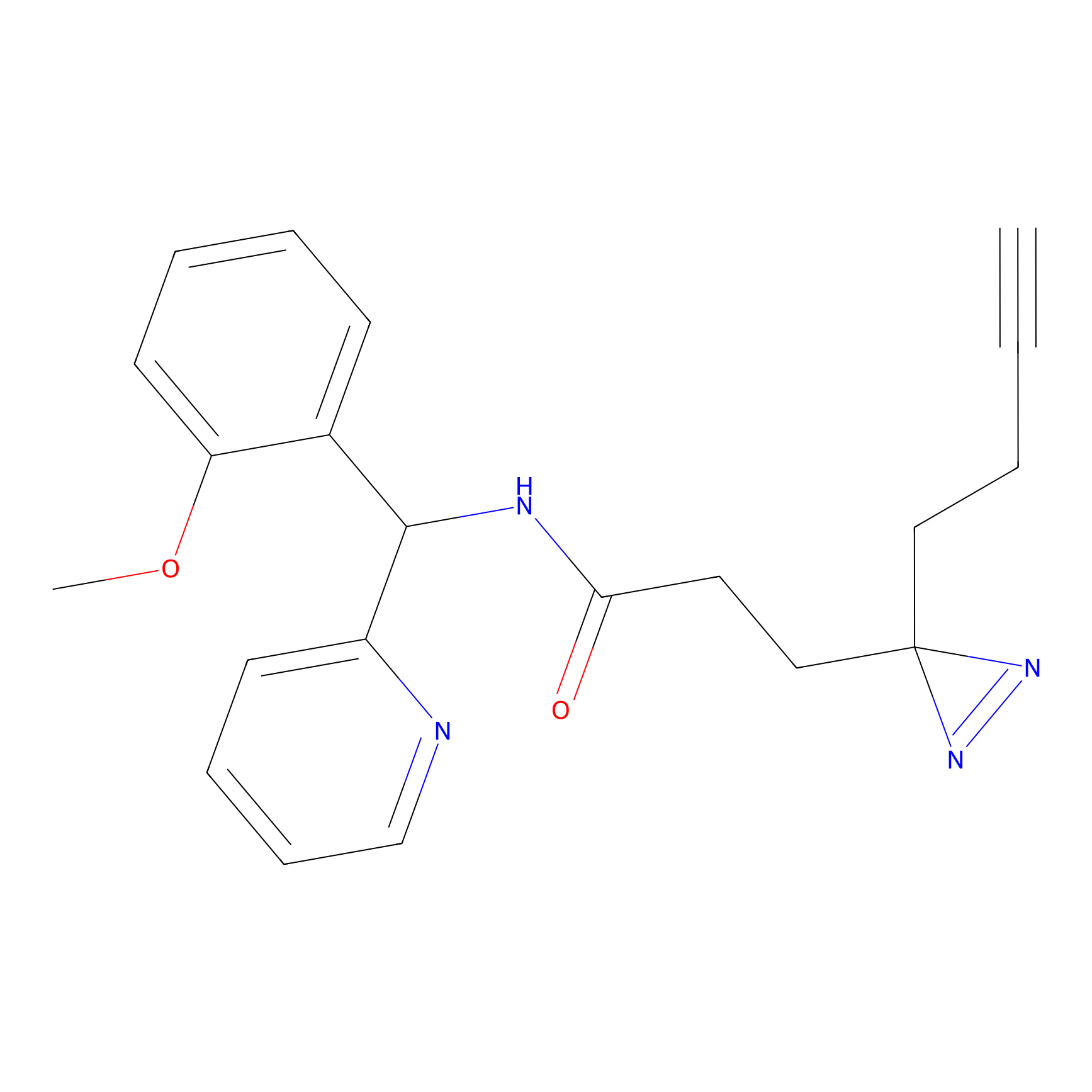
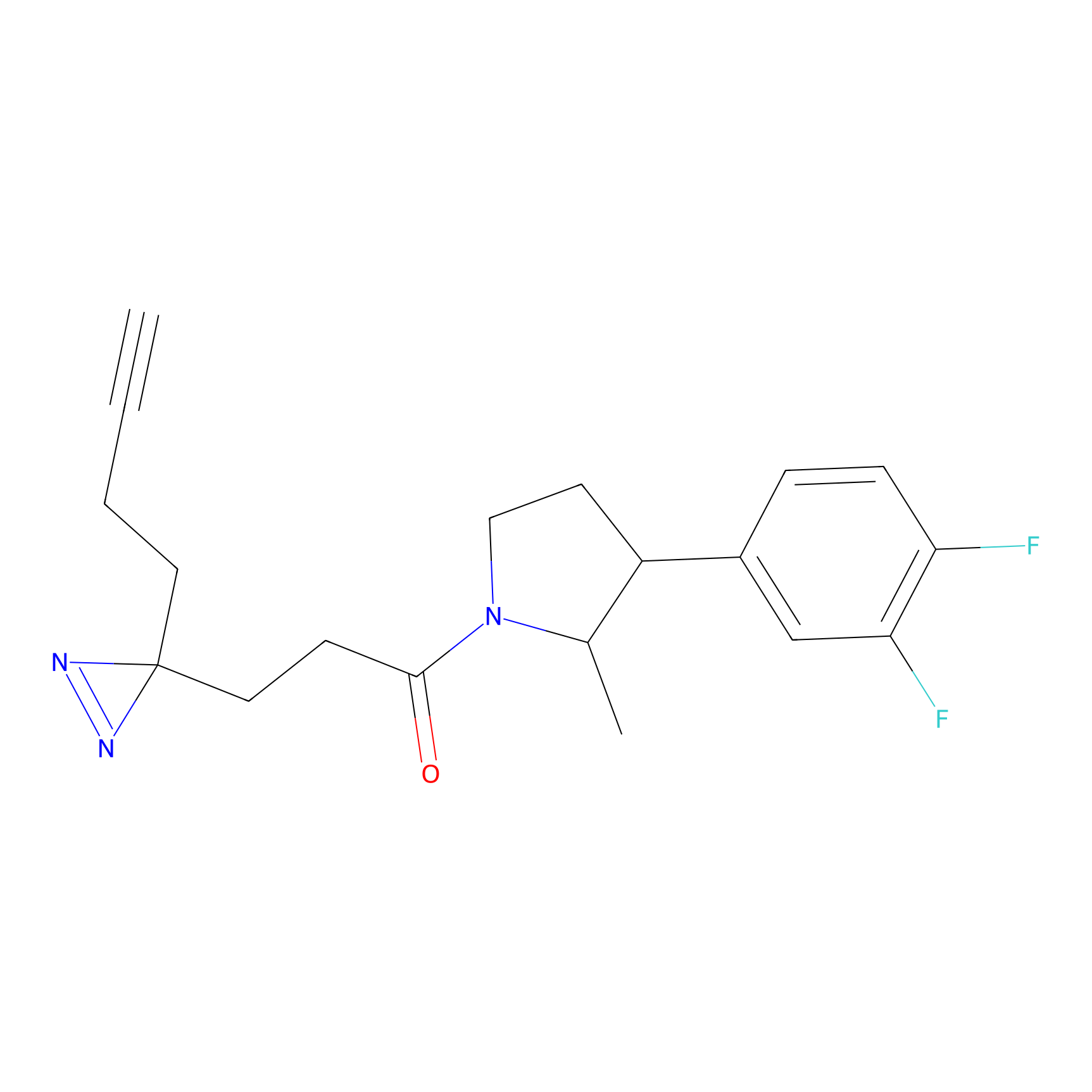
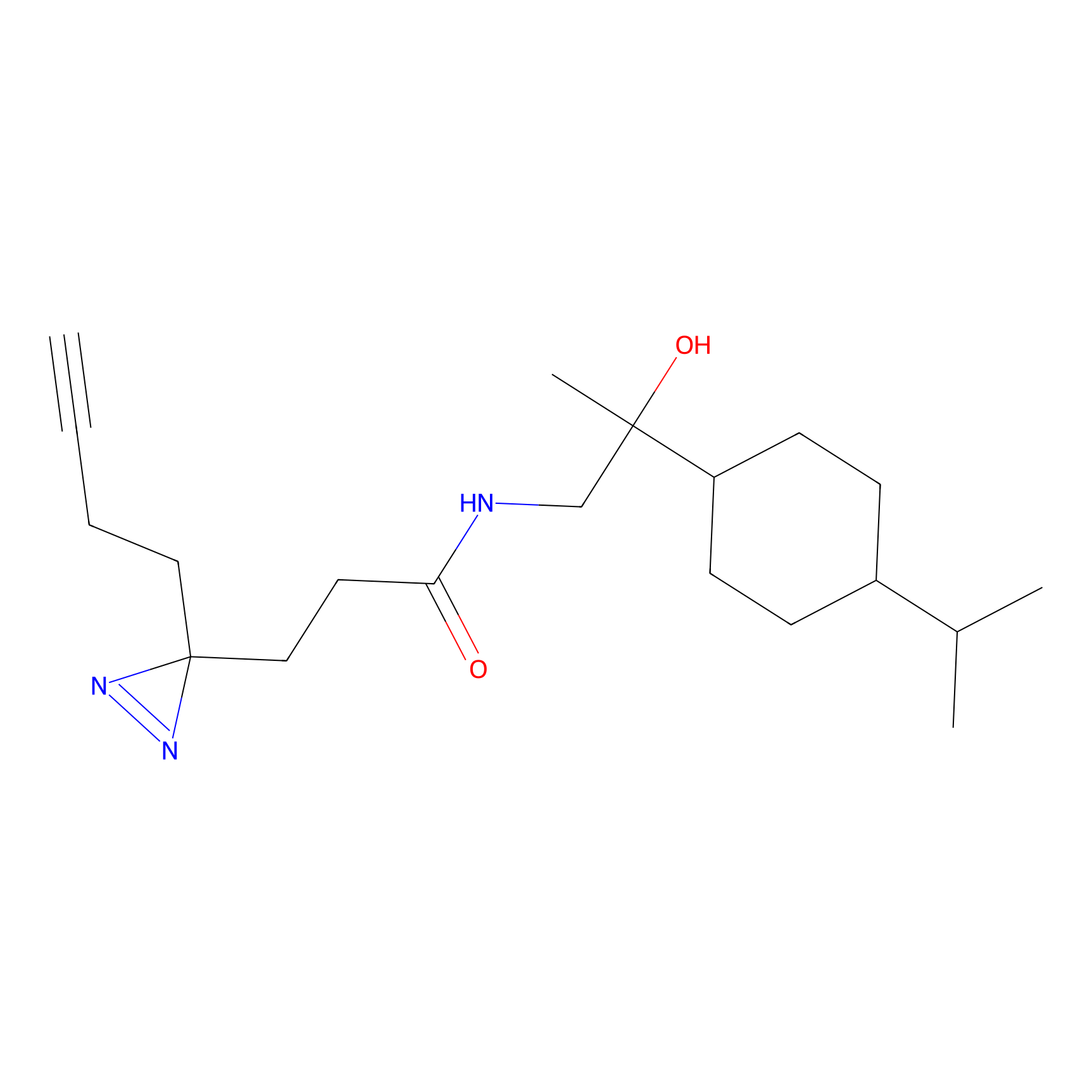
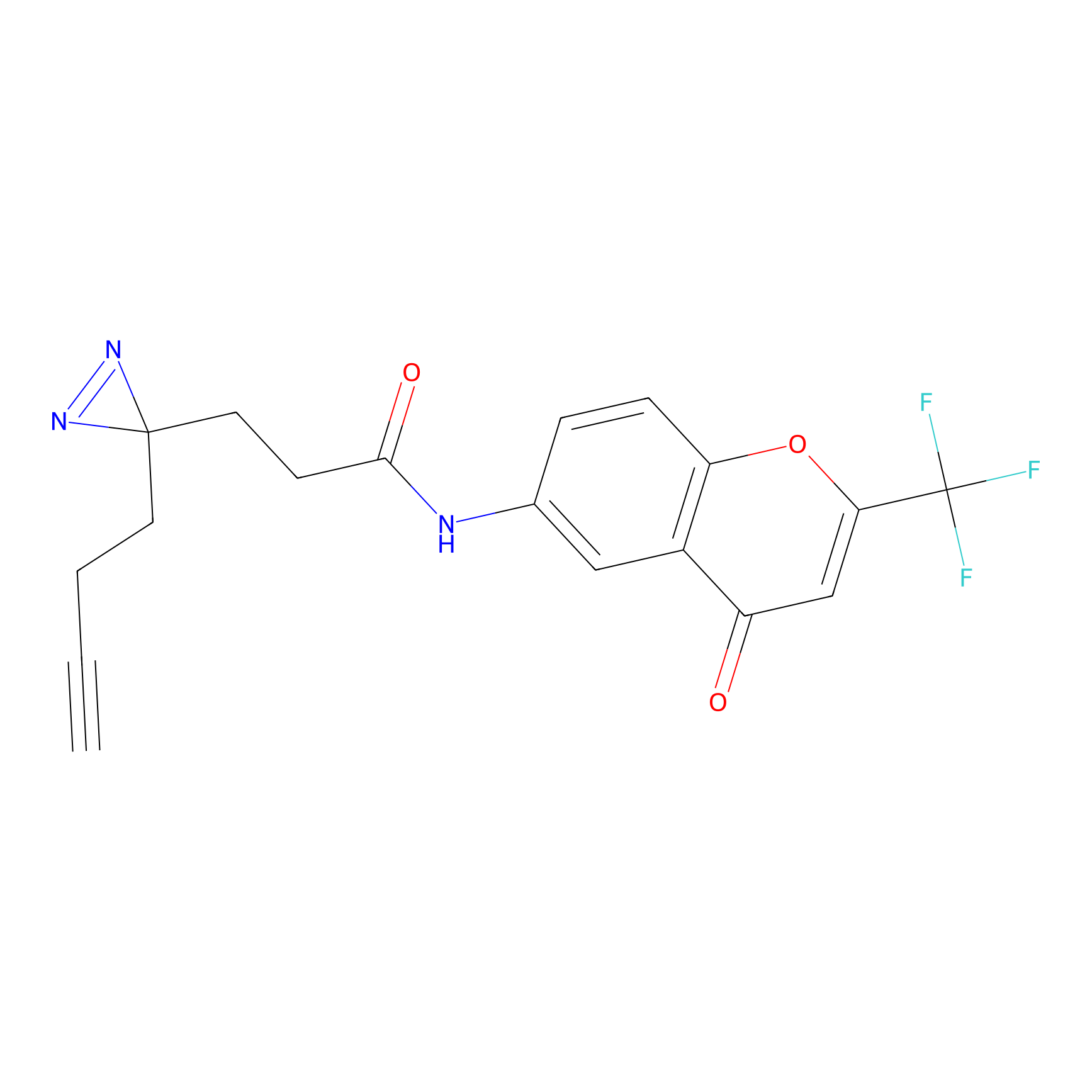
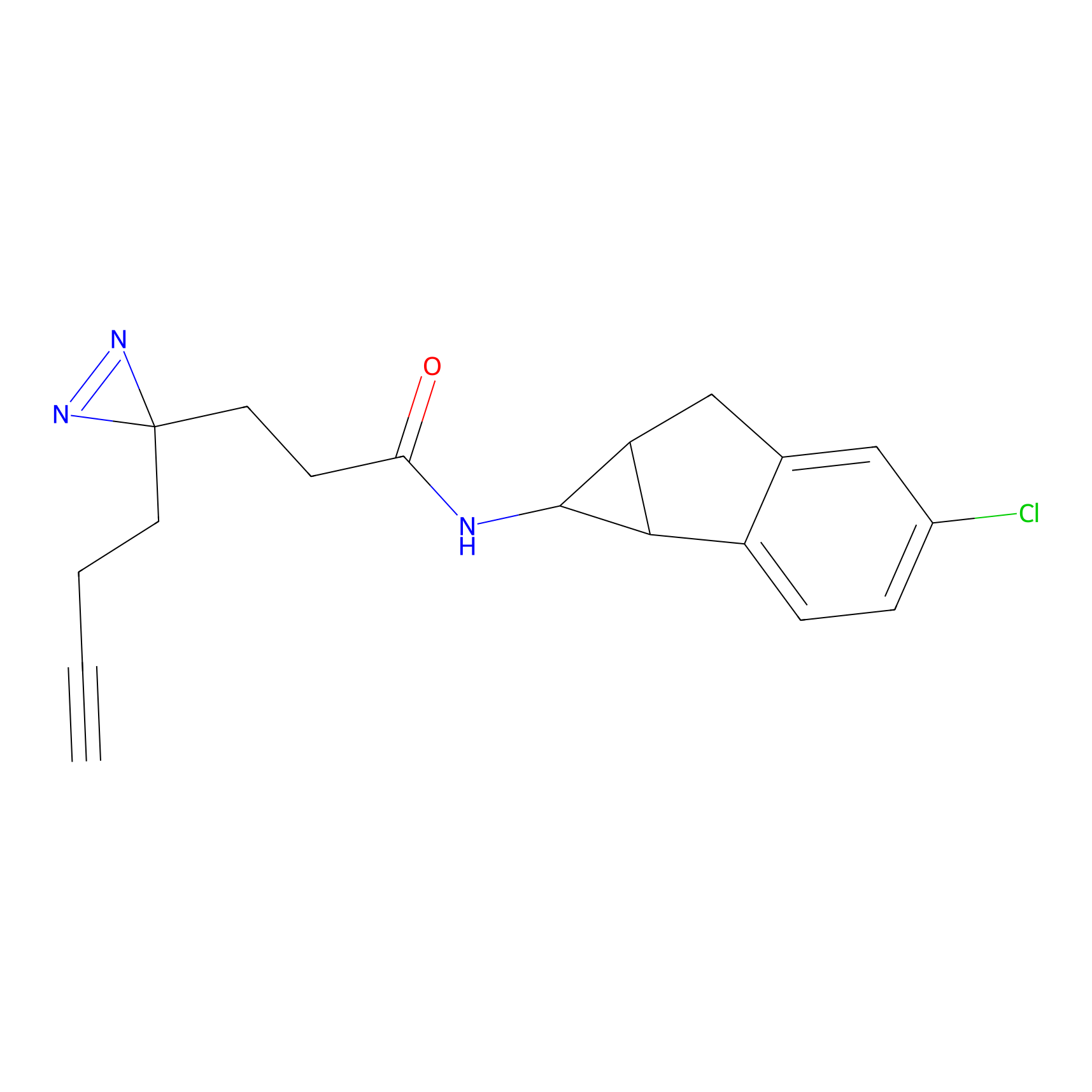
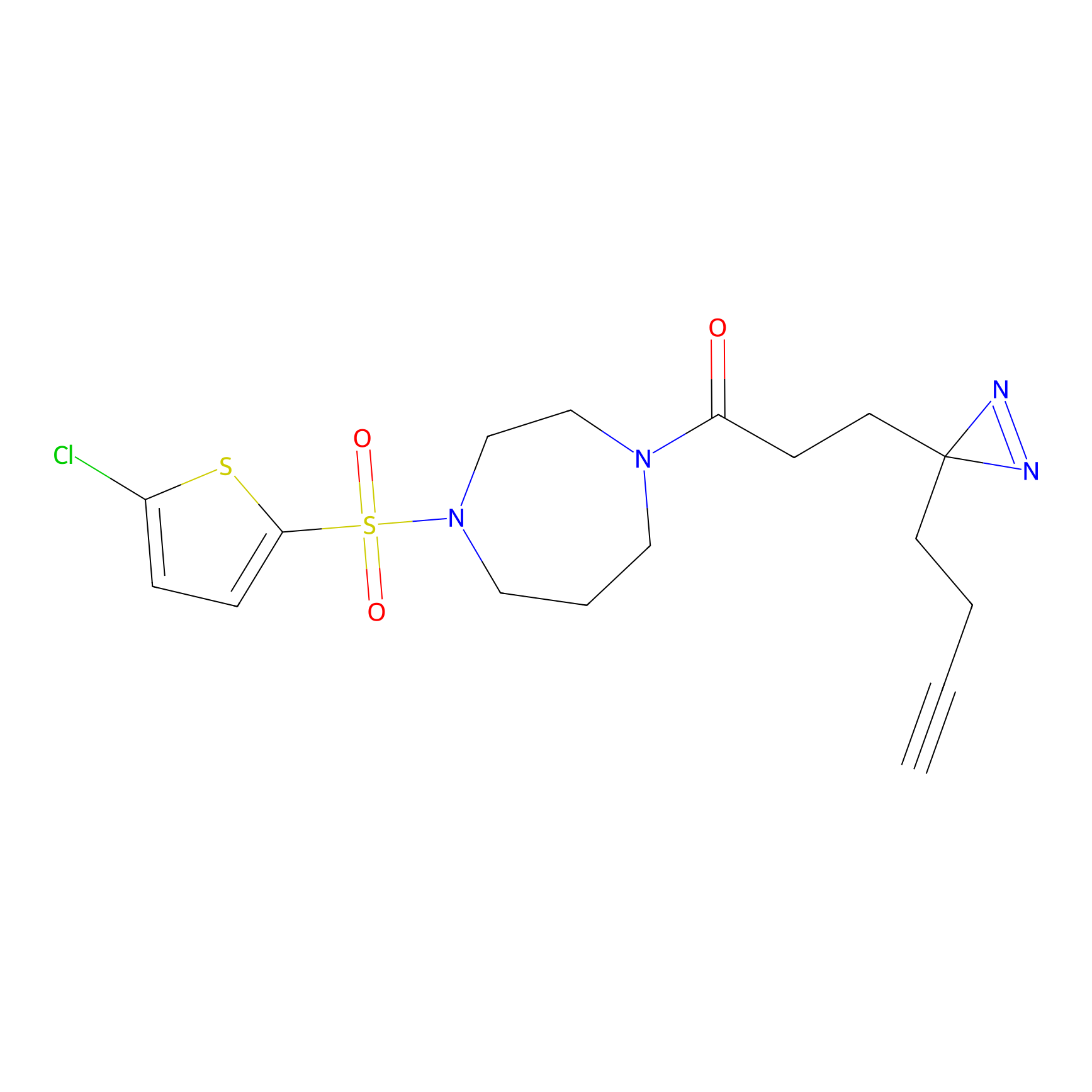
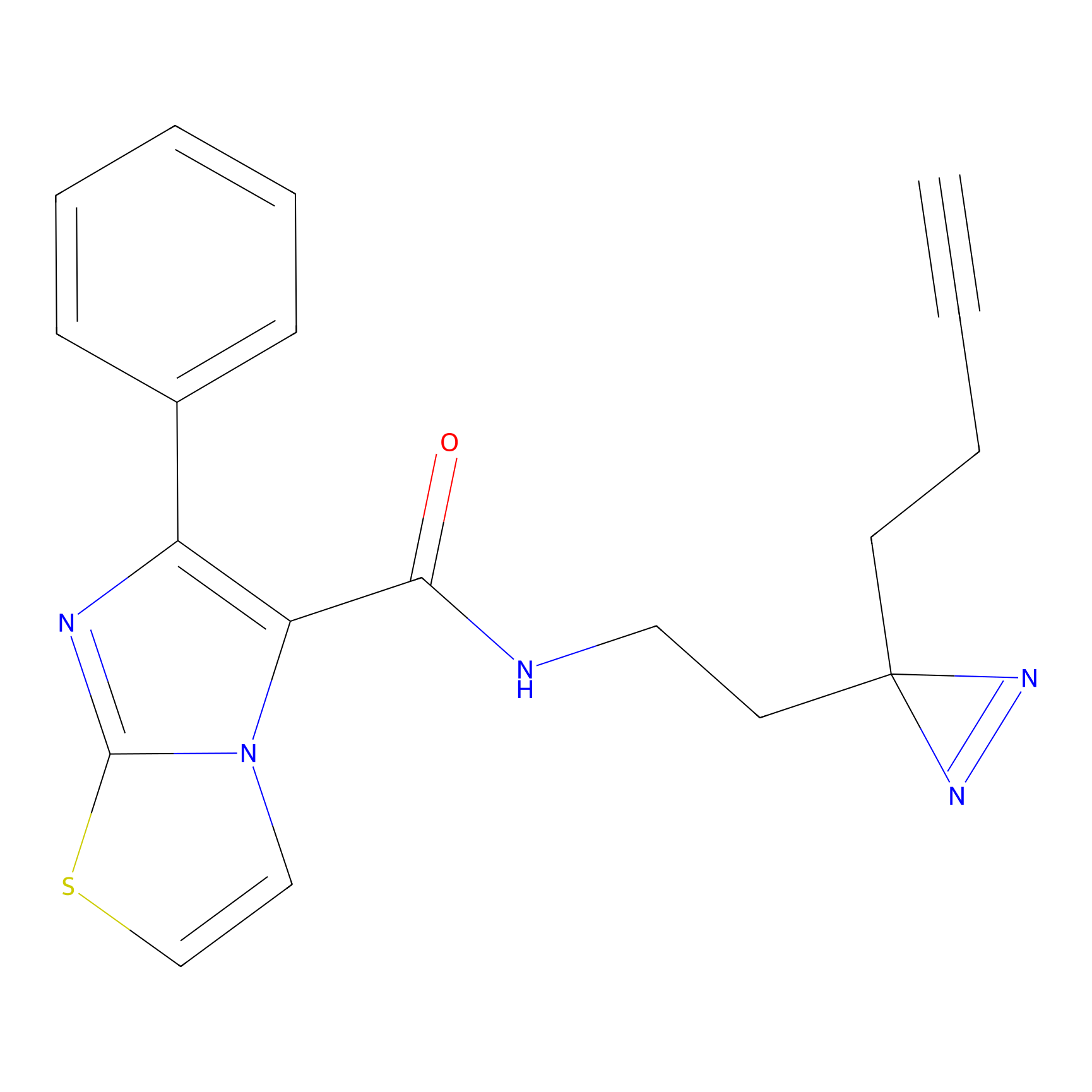
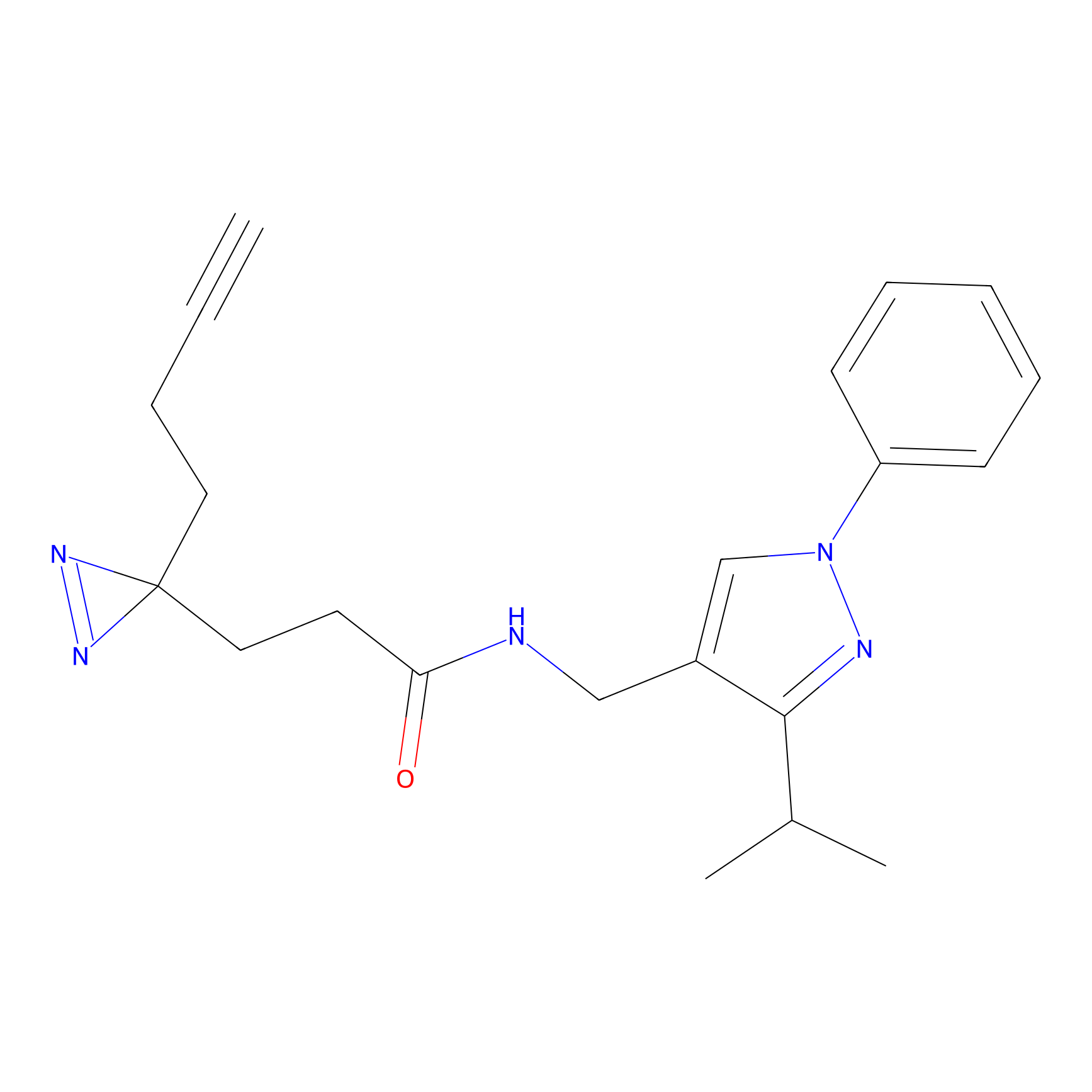
|
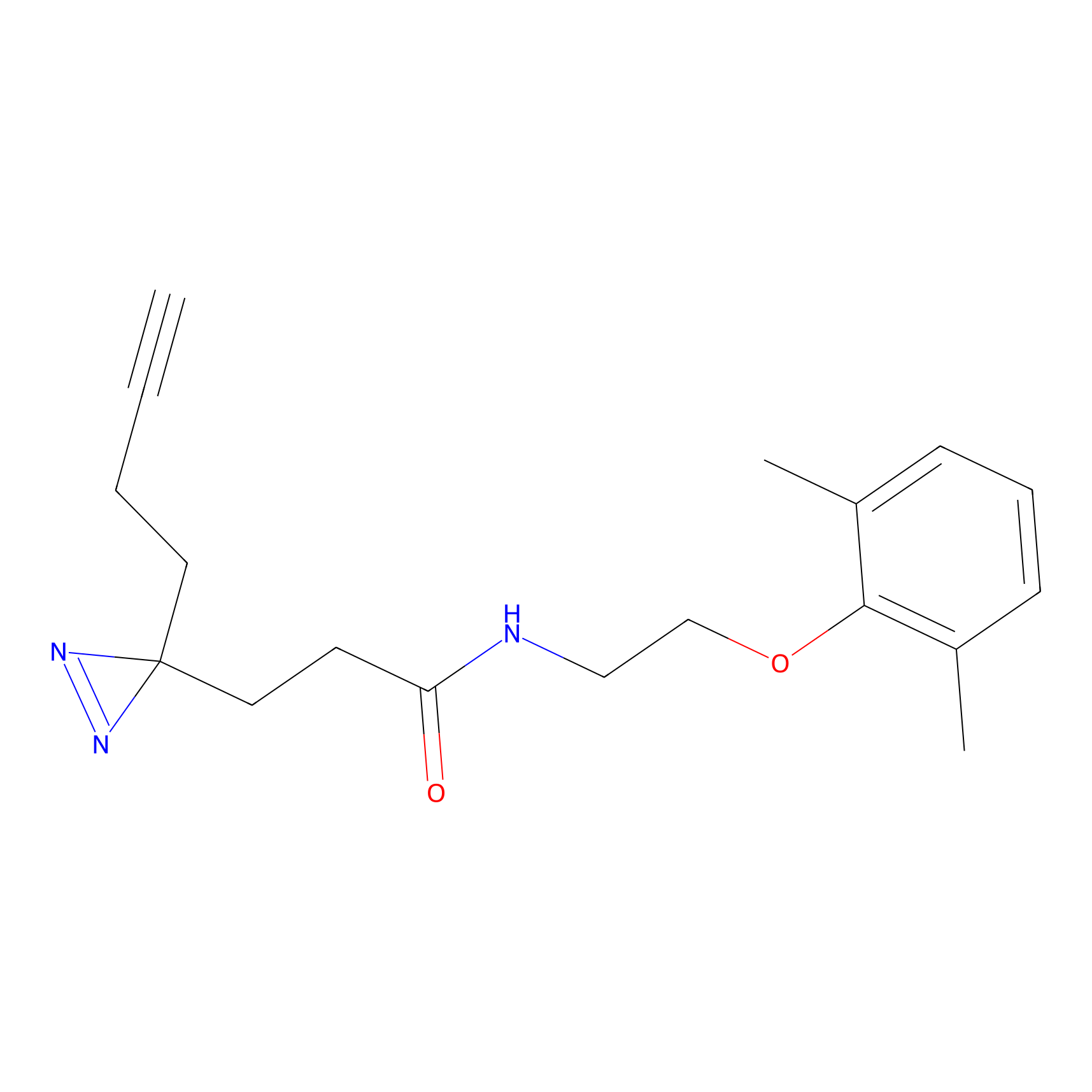
FBPP2 Probe Info |
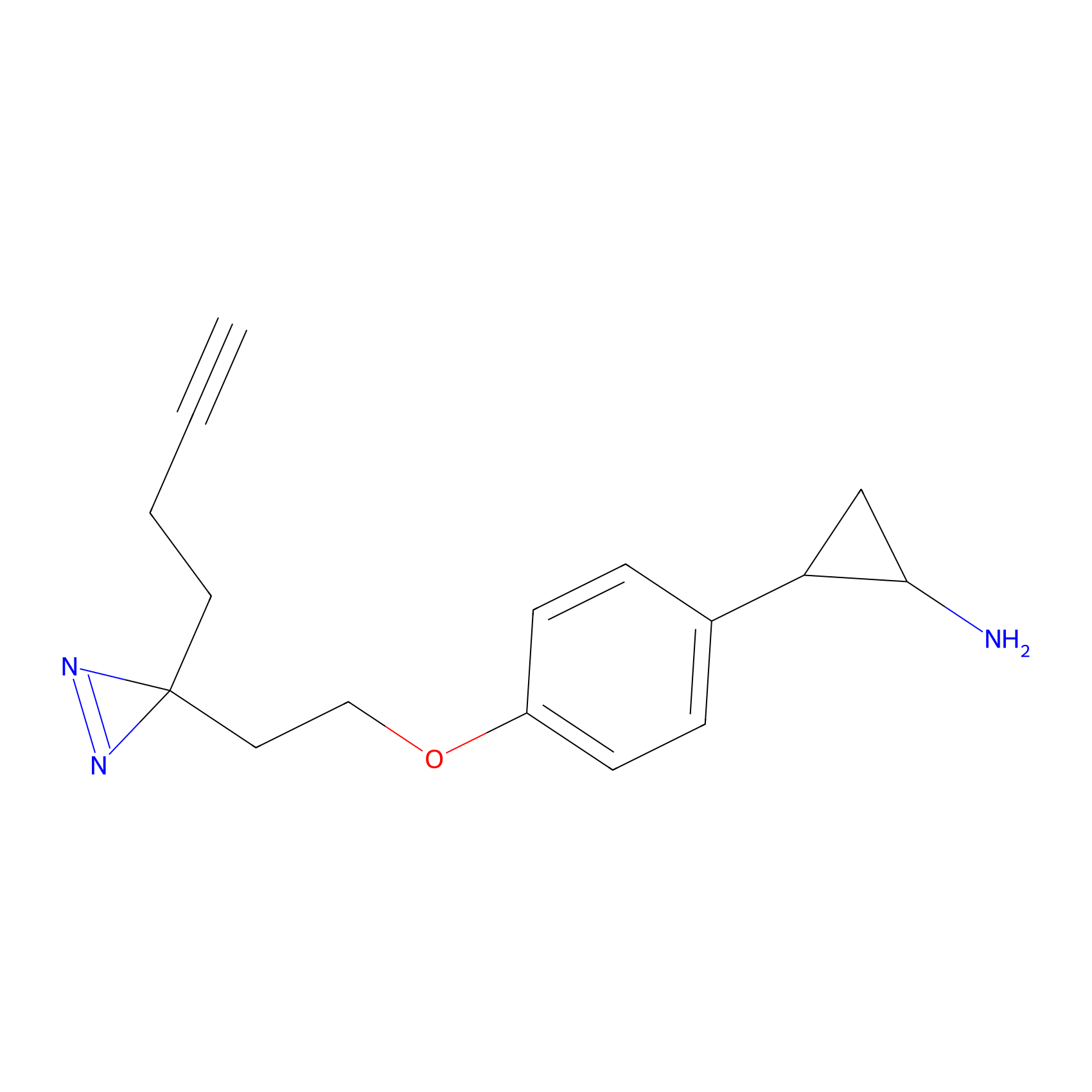 |
9.86 | LDD0318 | [1] | |
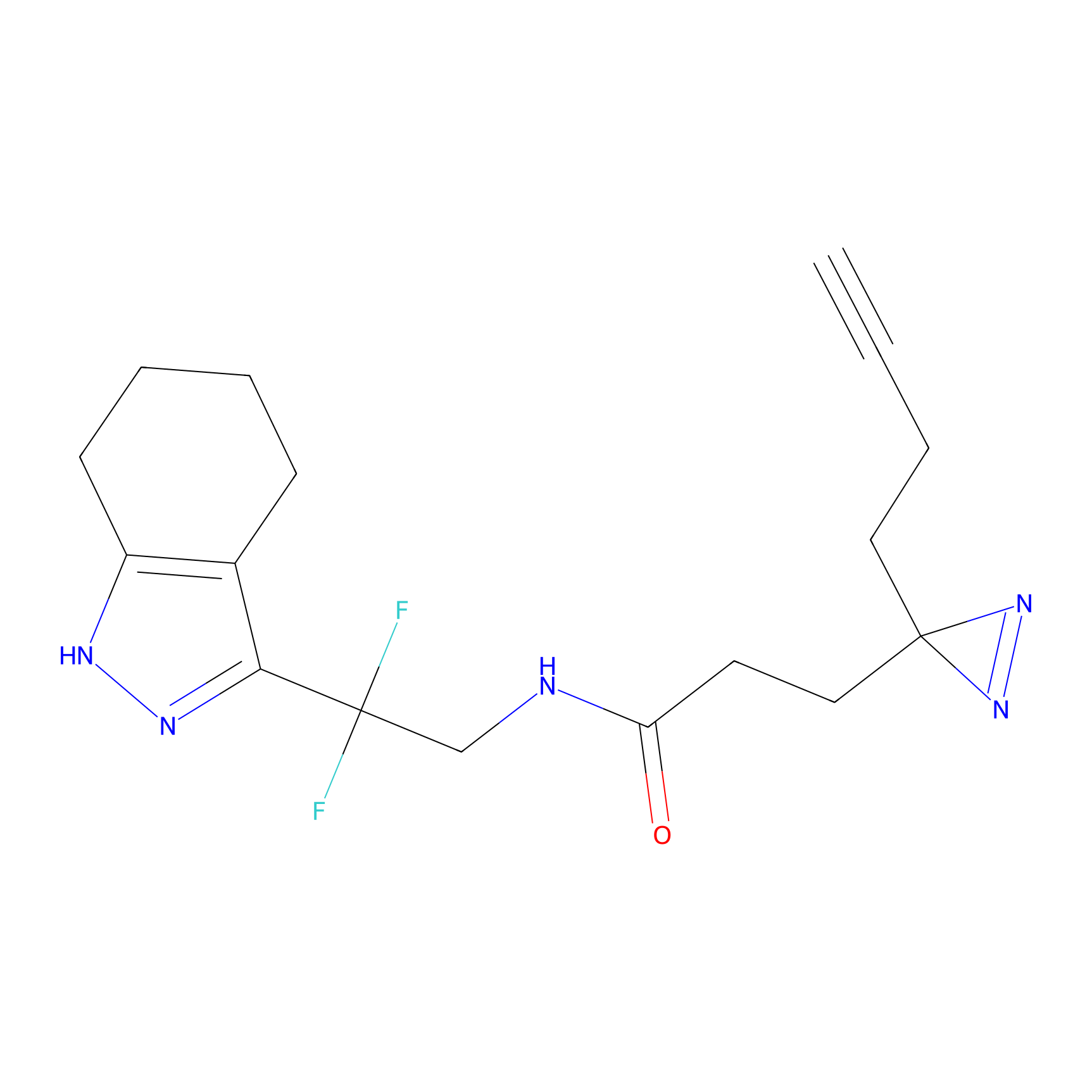
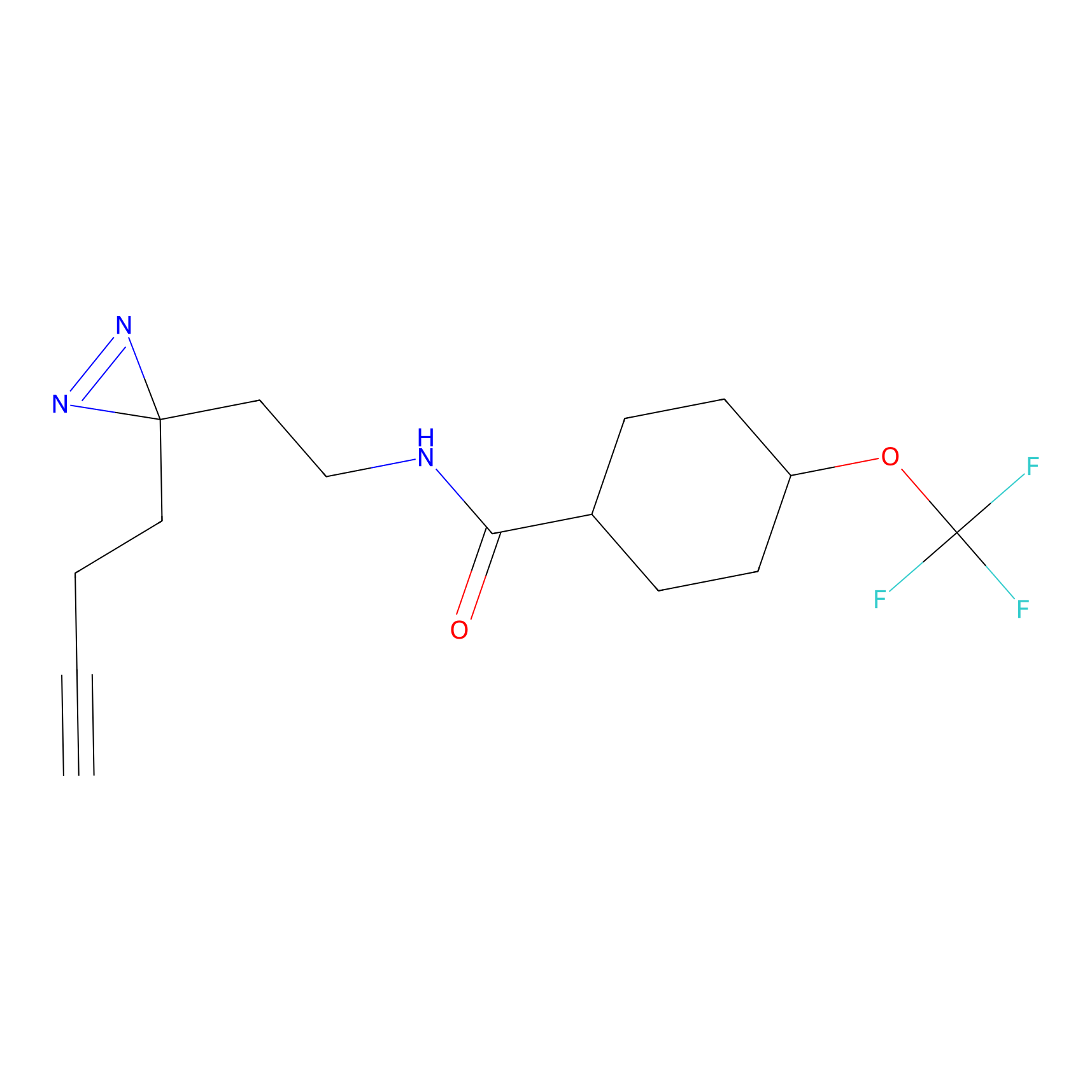
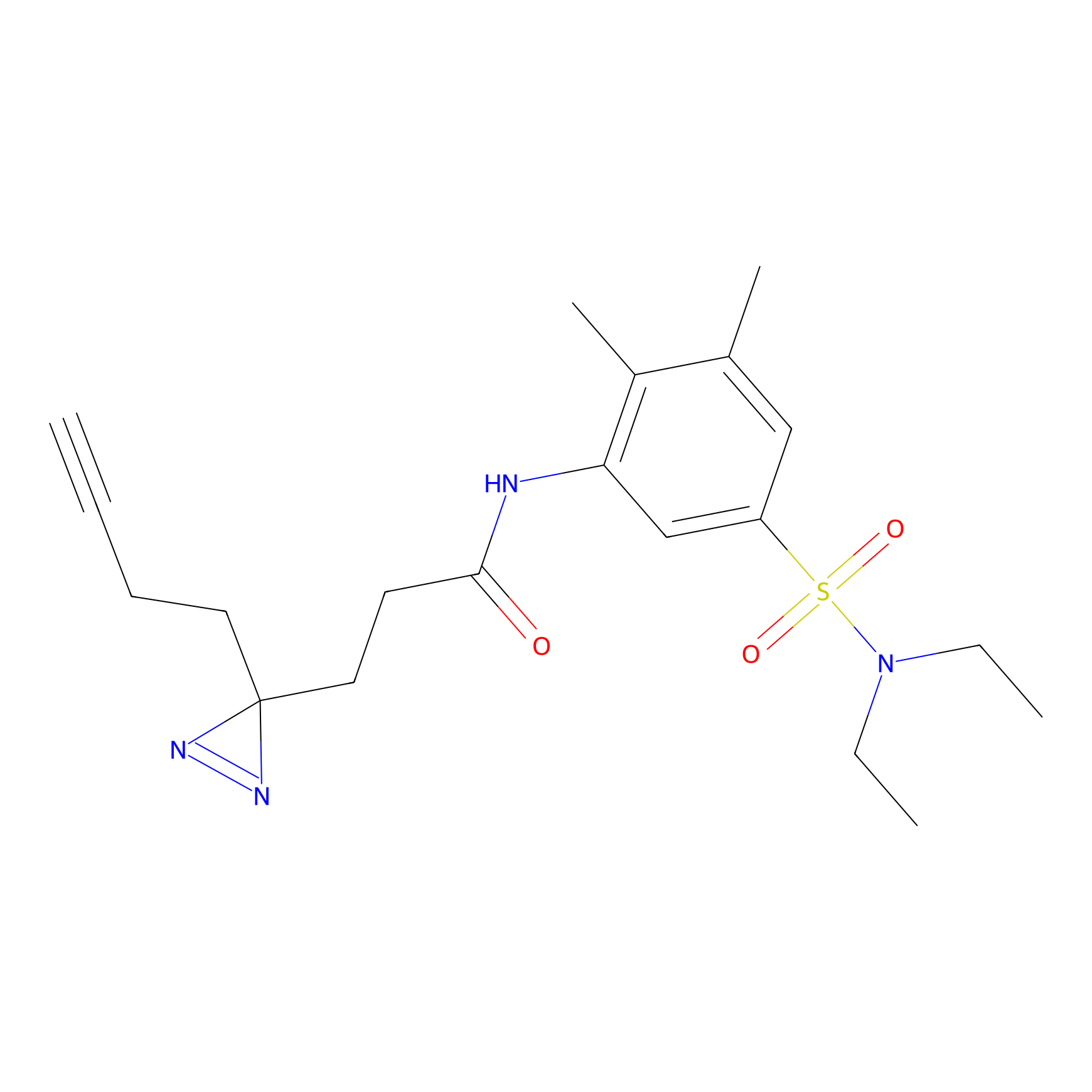
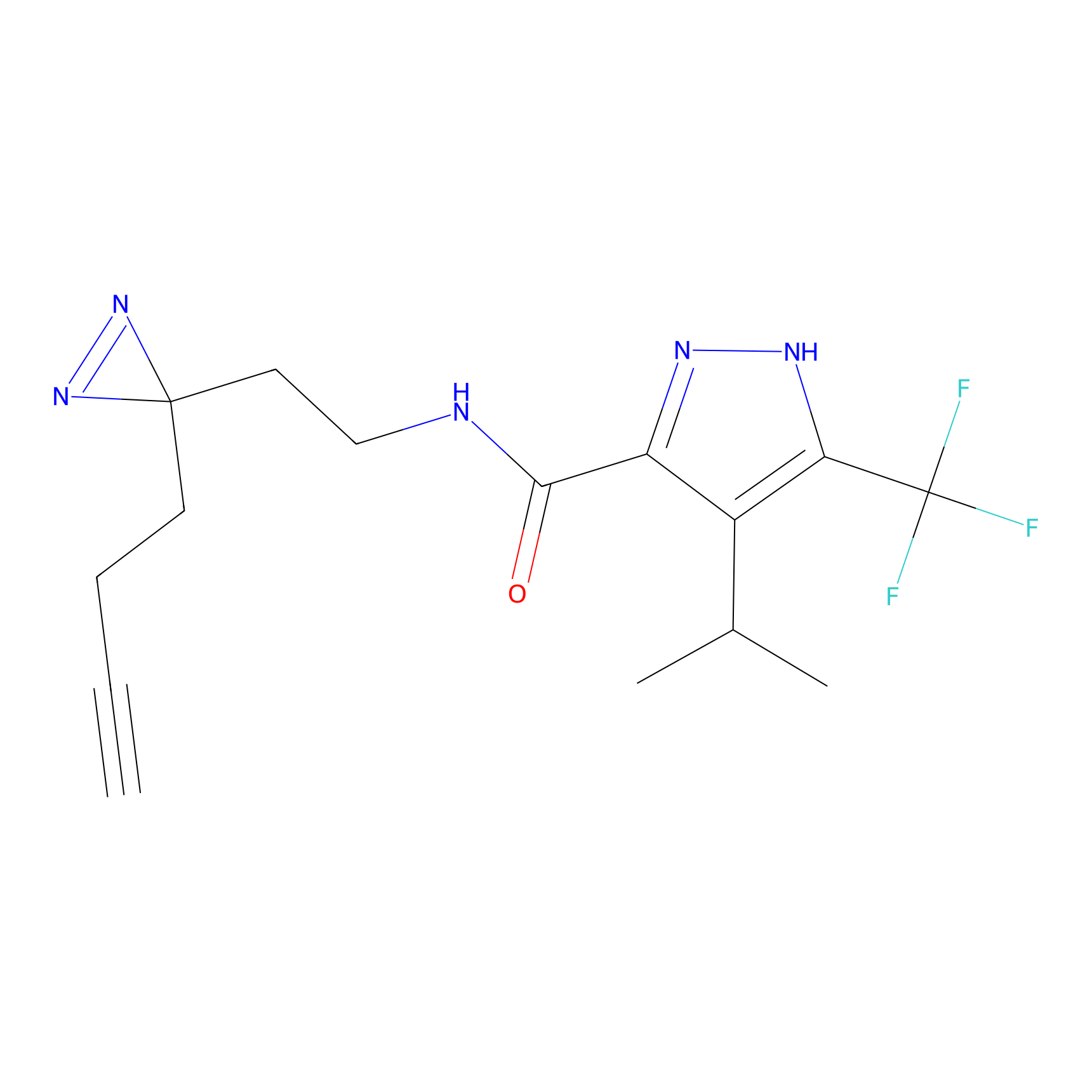
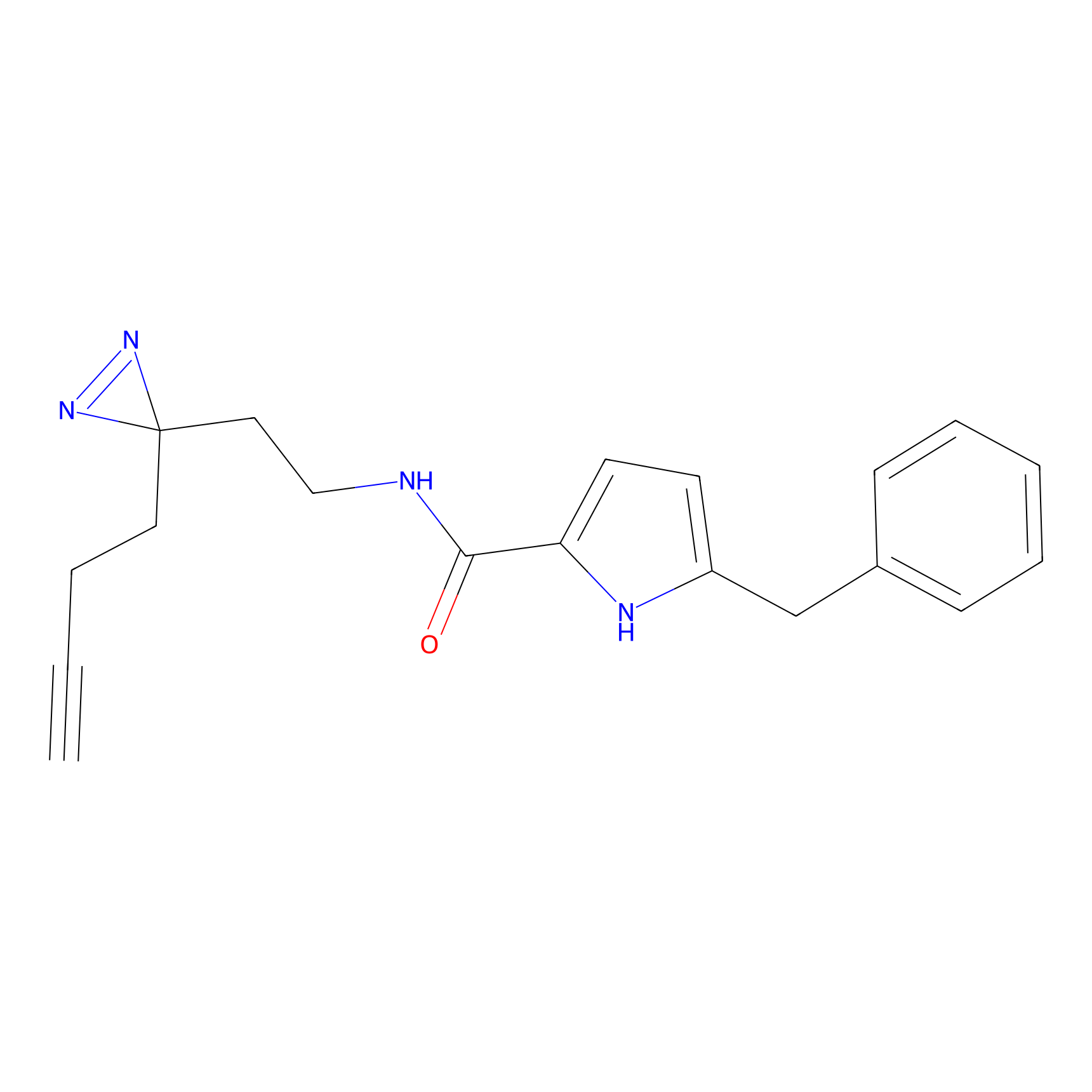
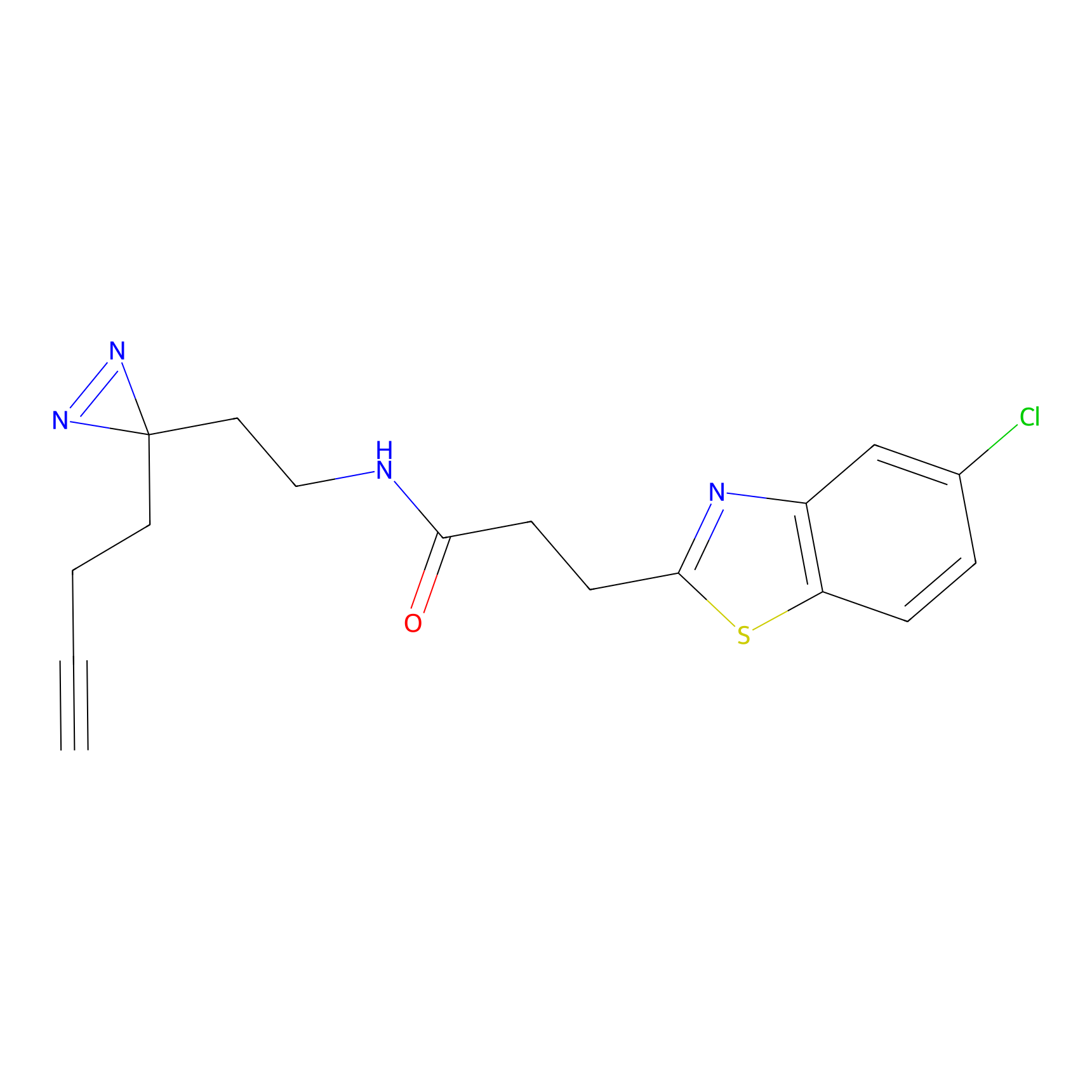
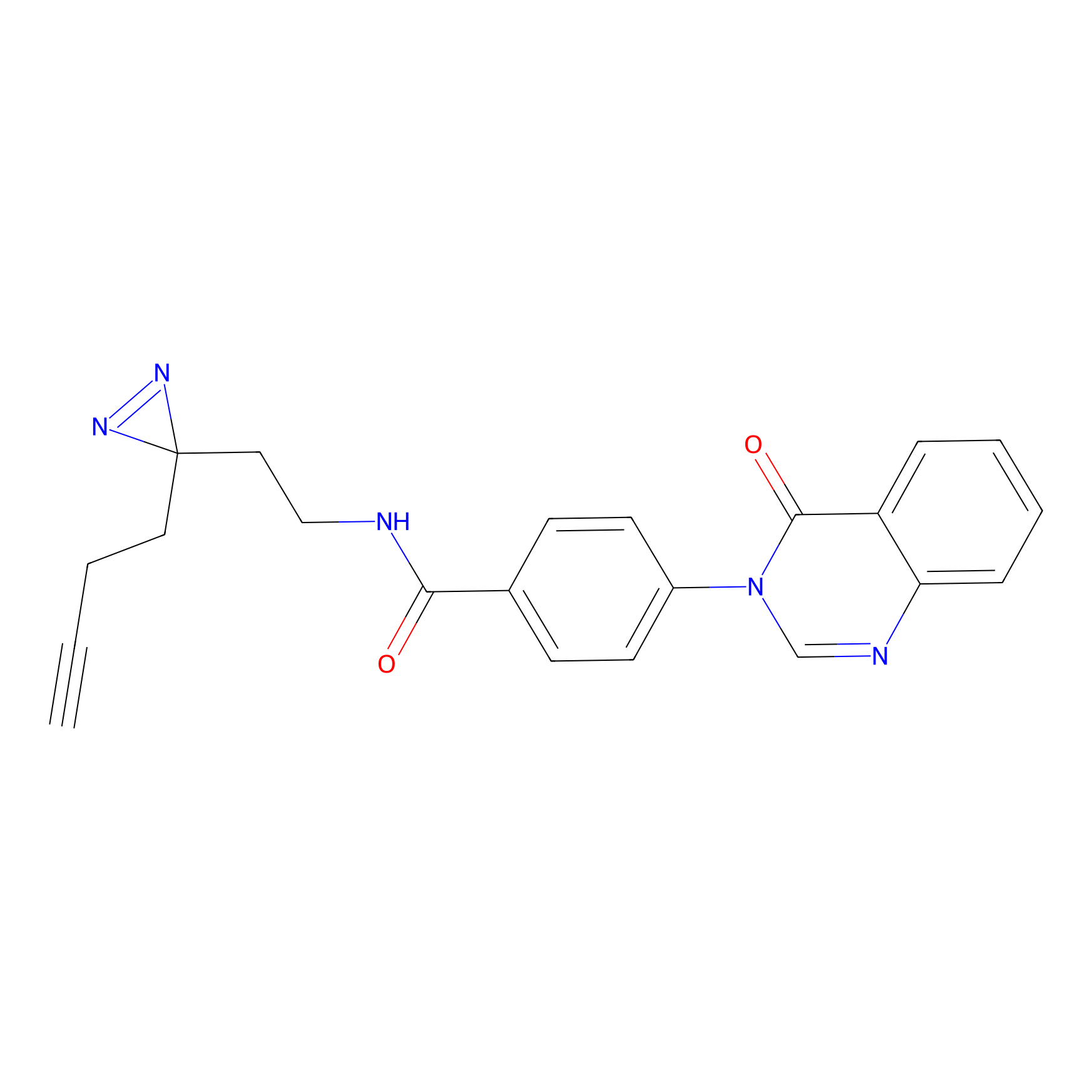
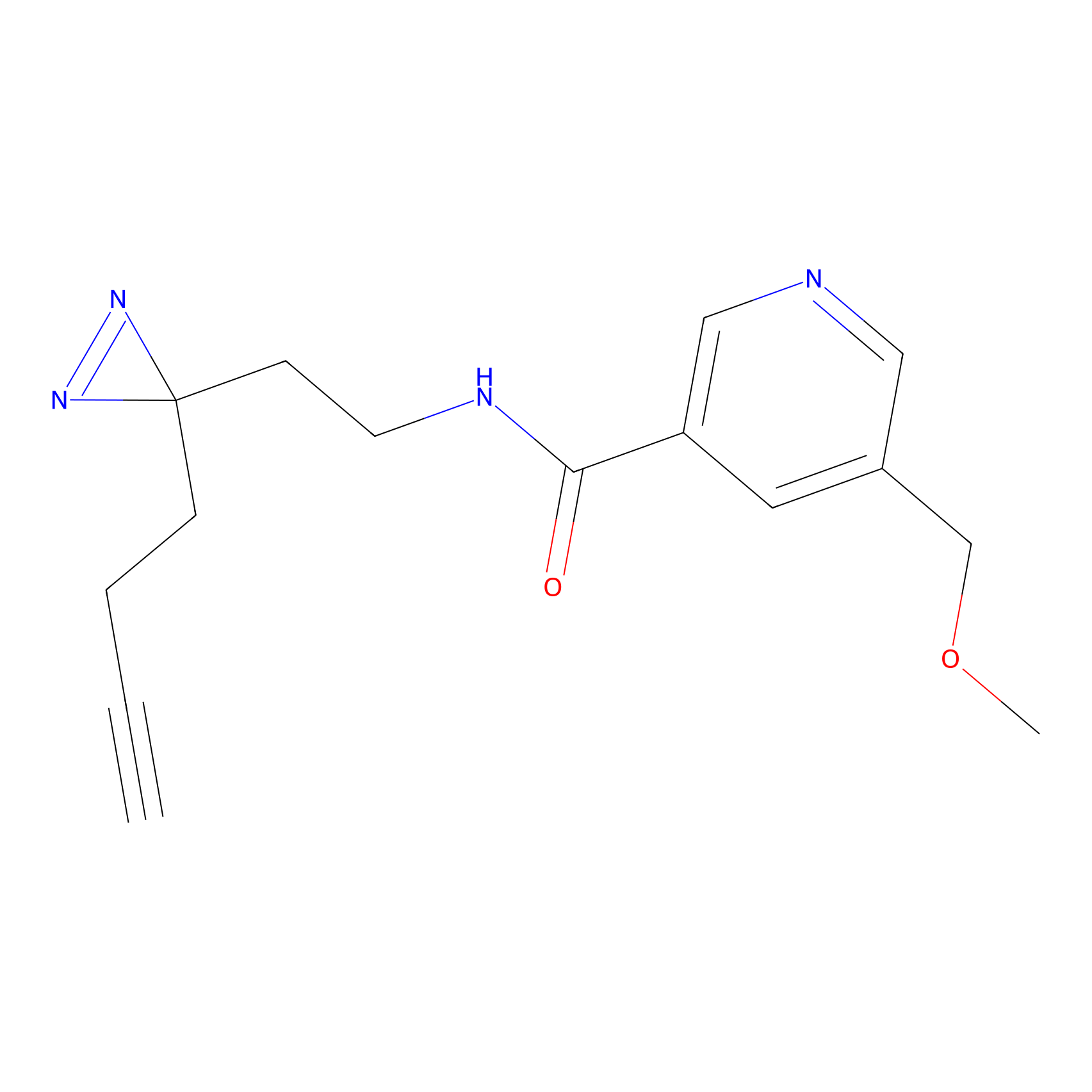
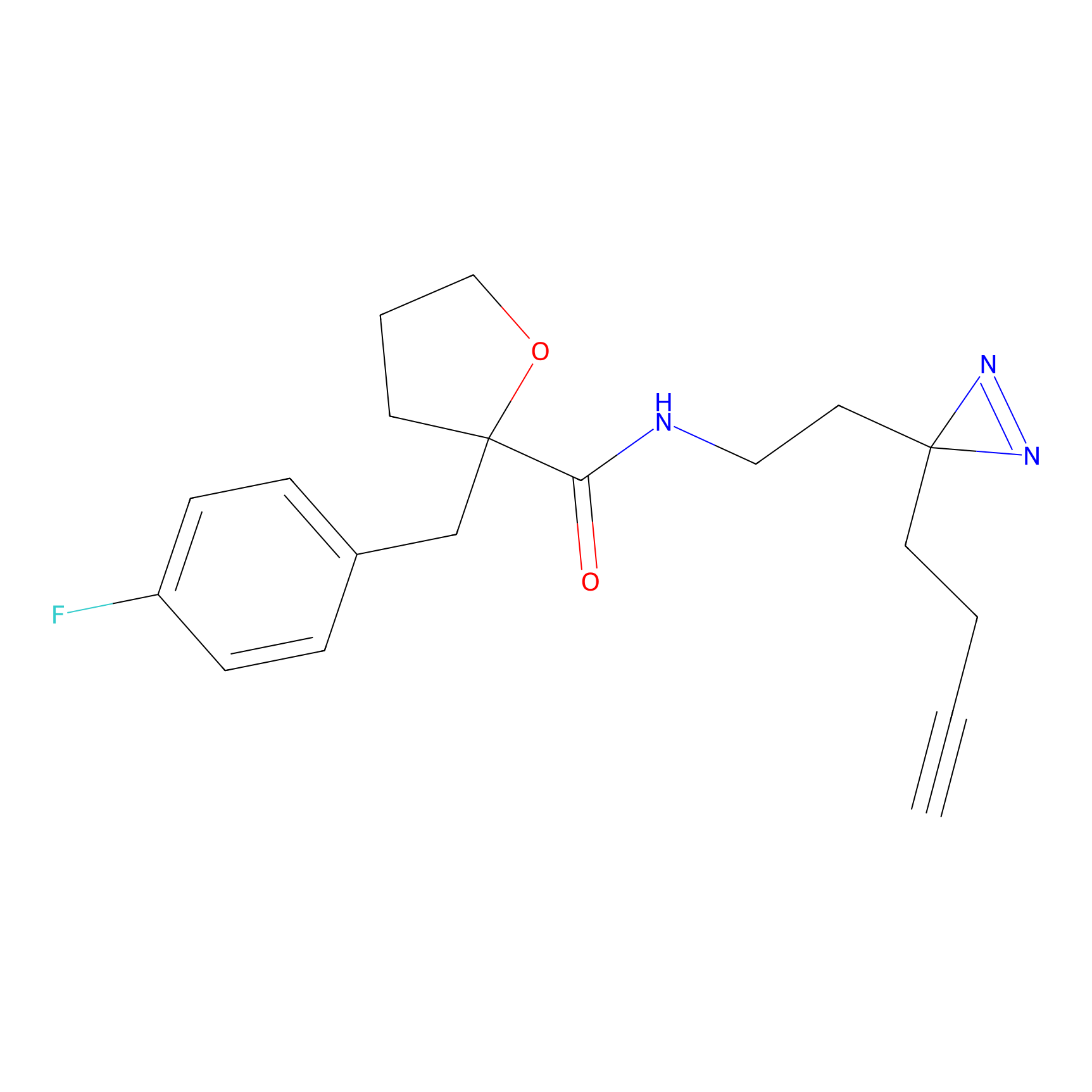
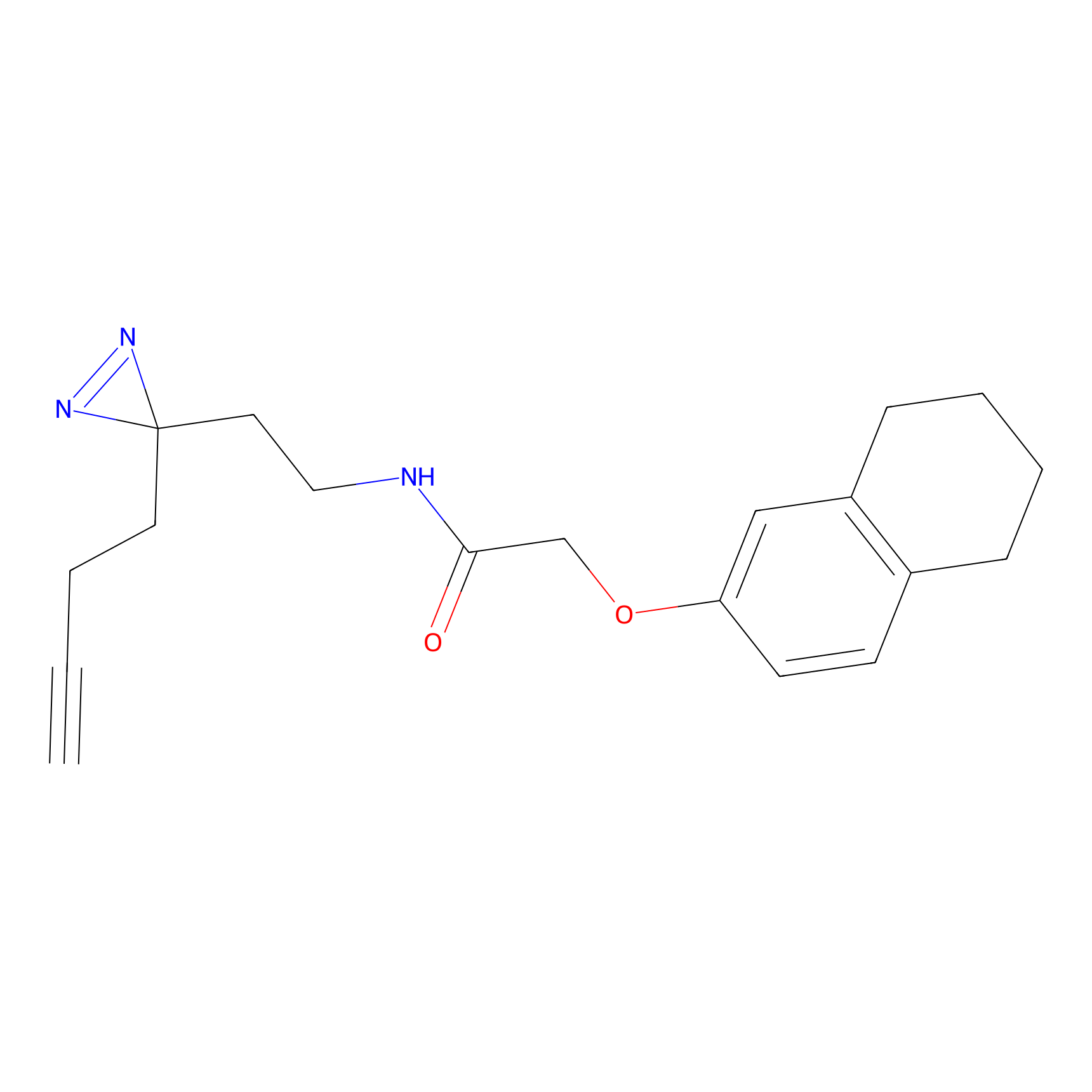
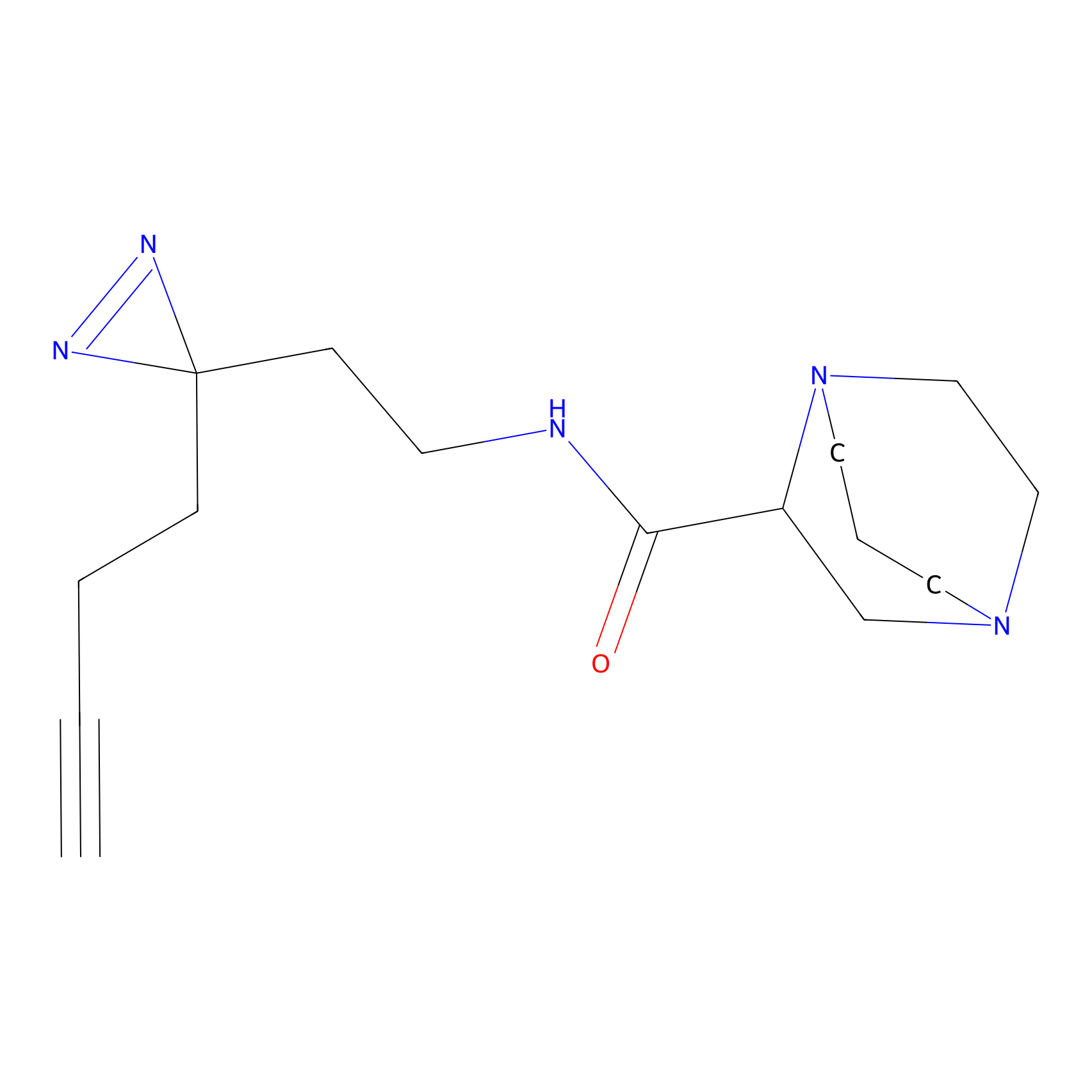
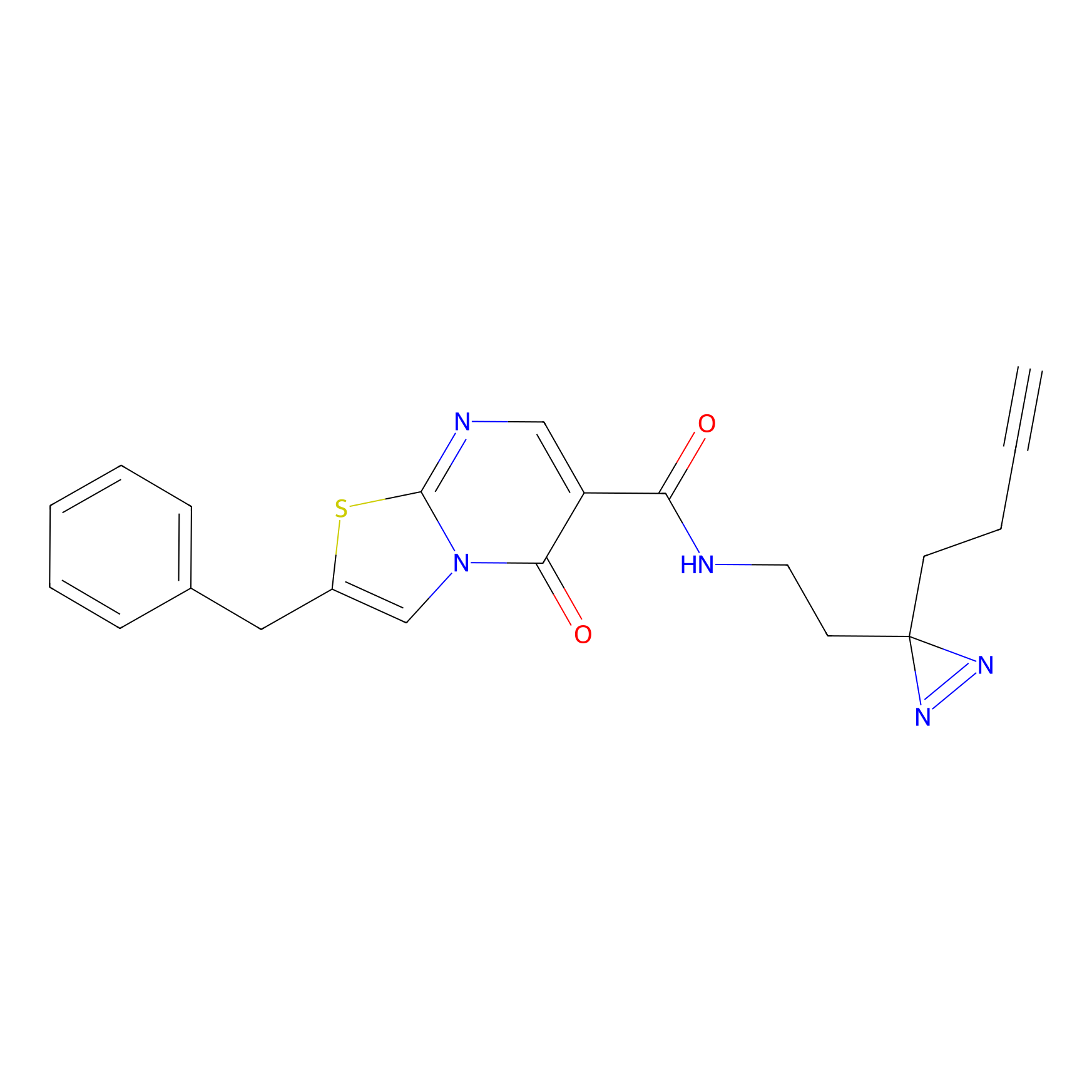
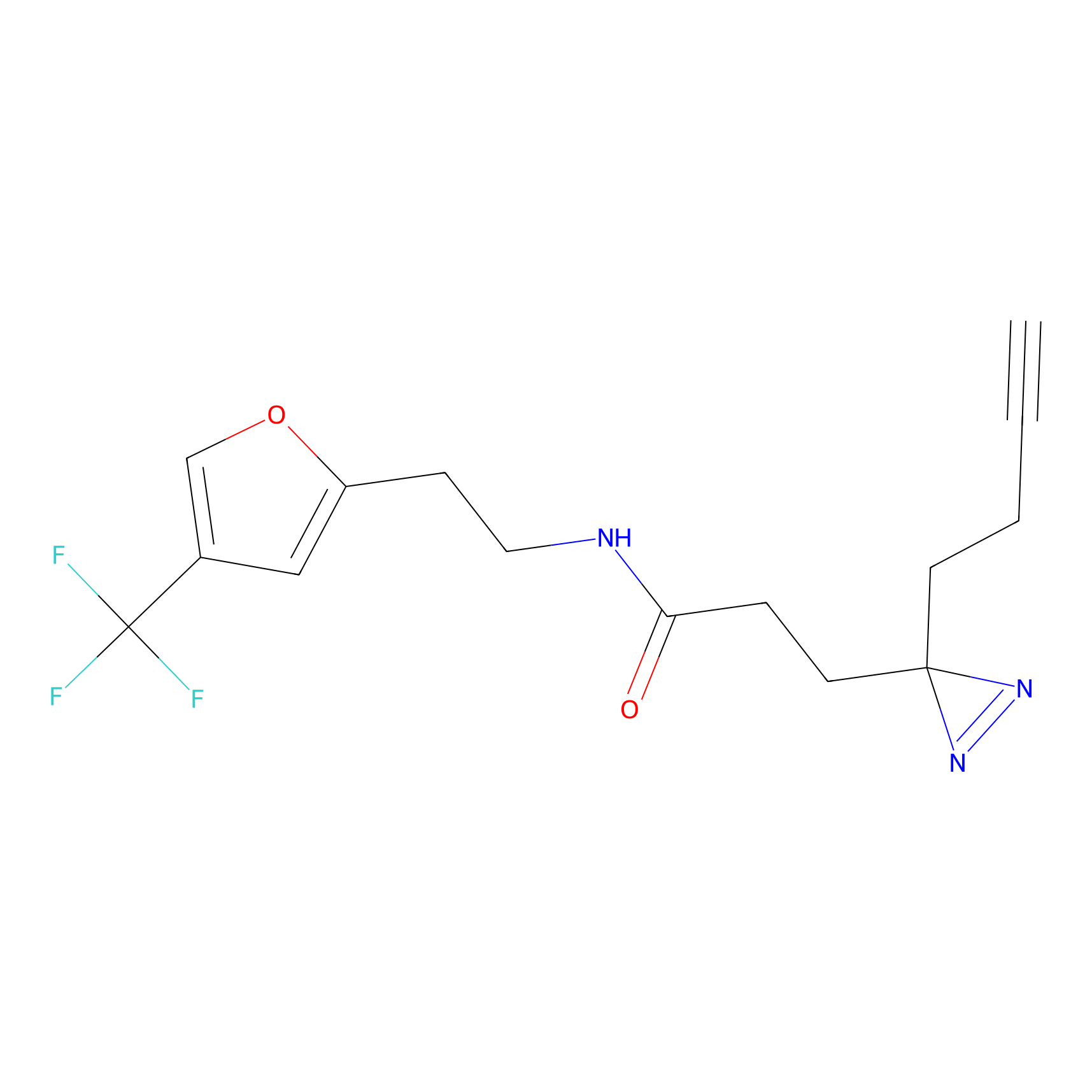
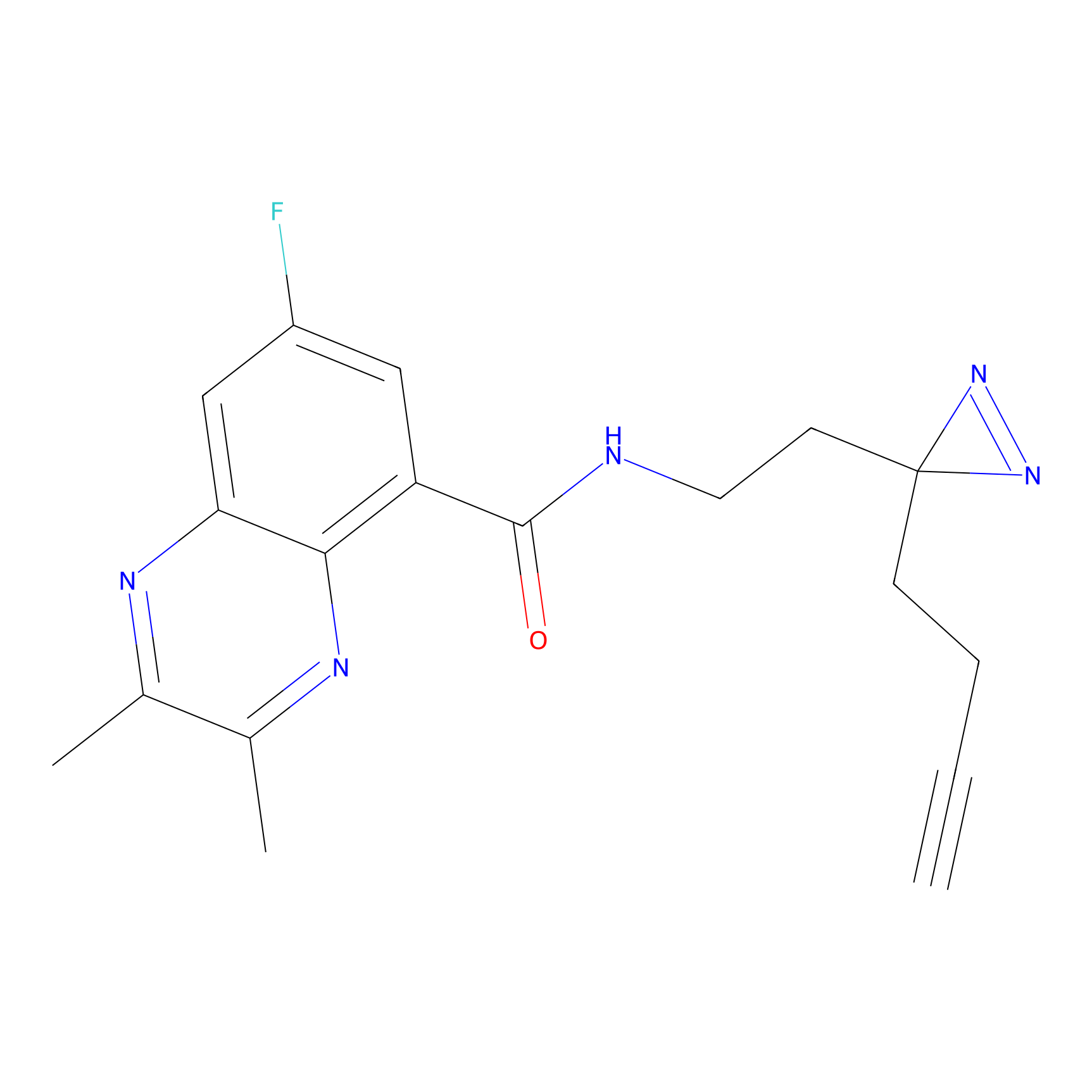
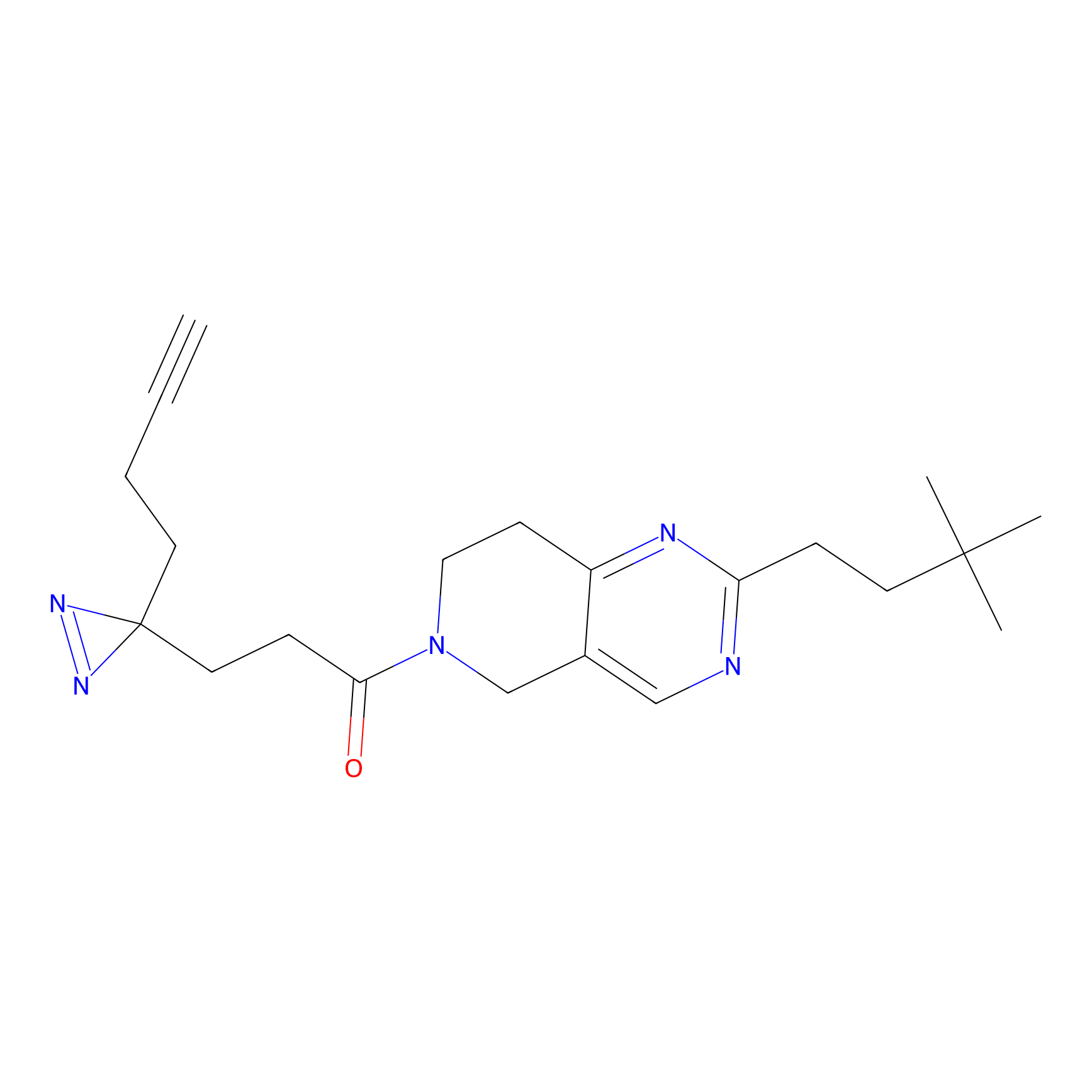
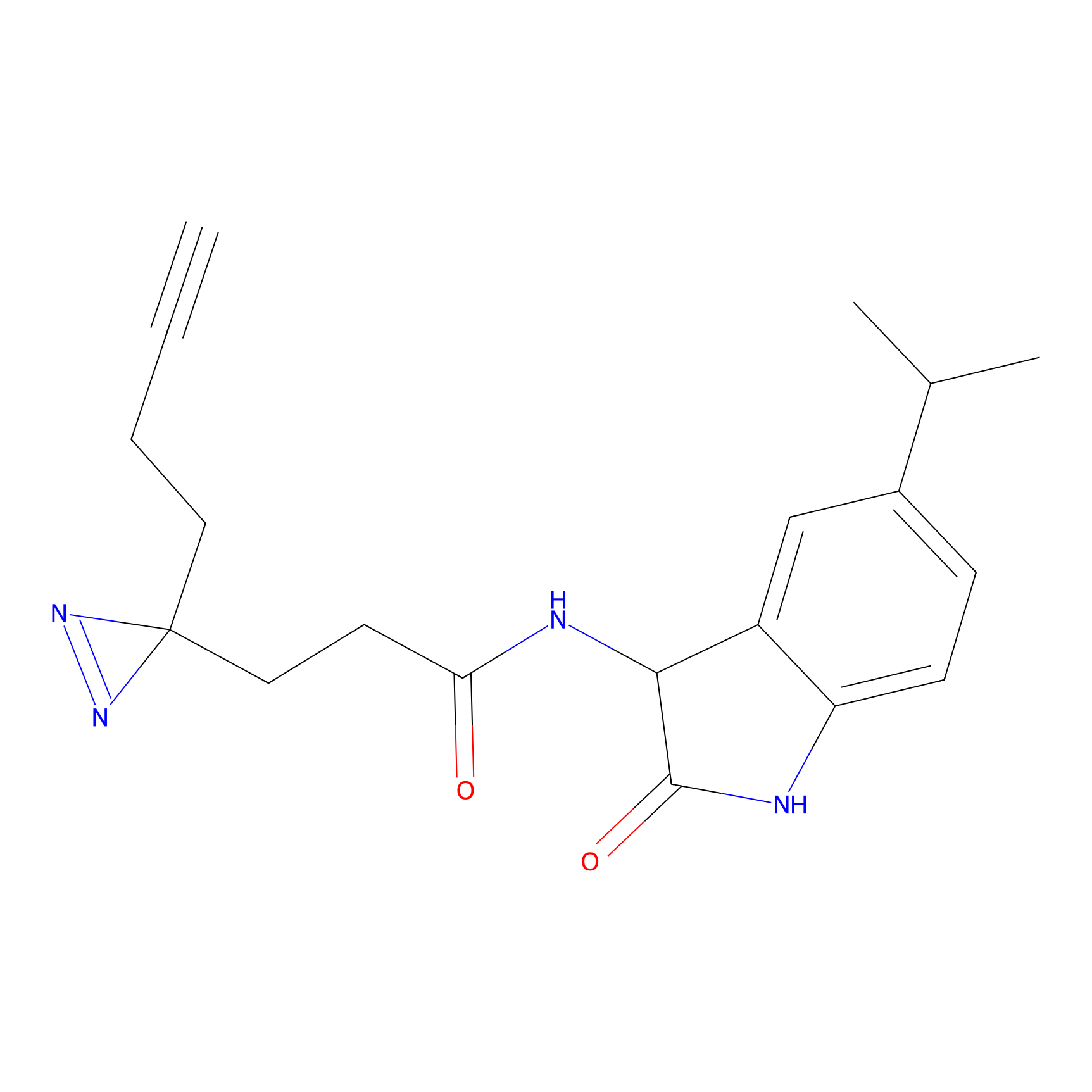
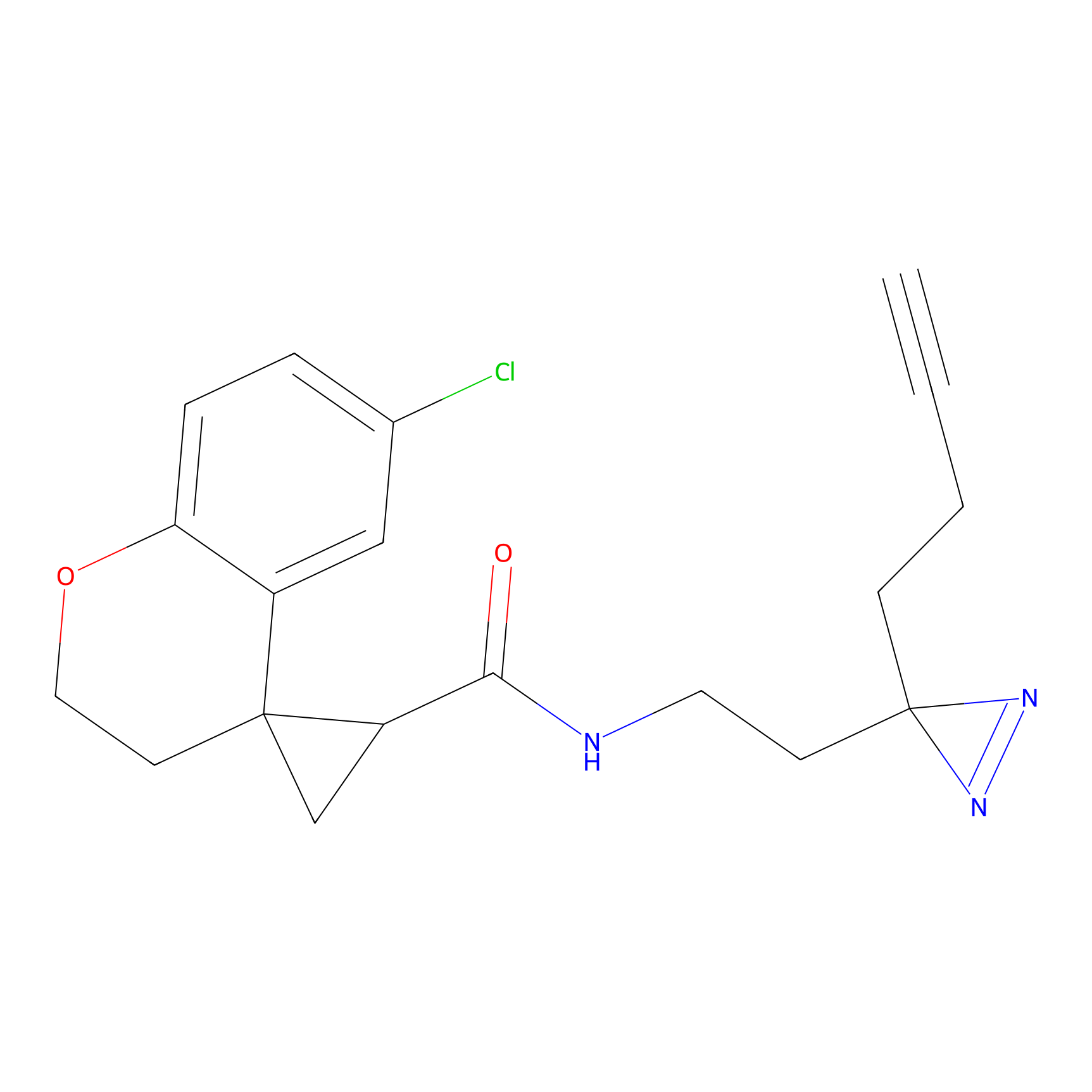
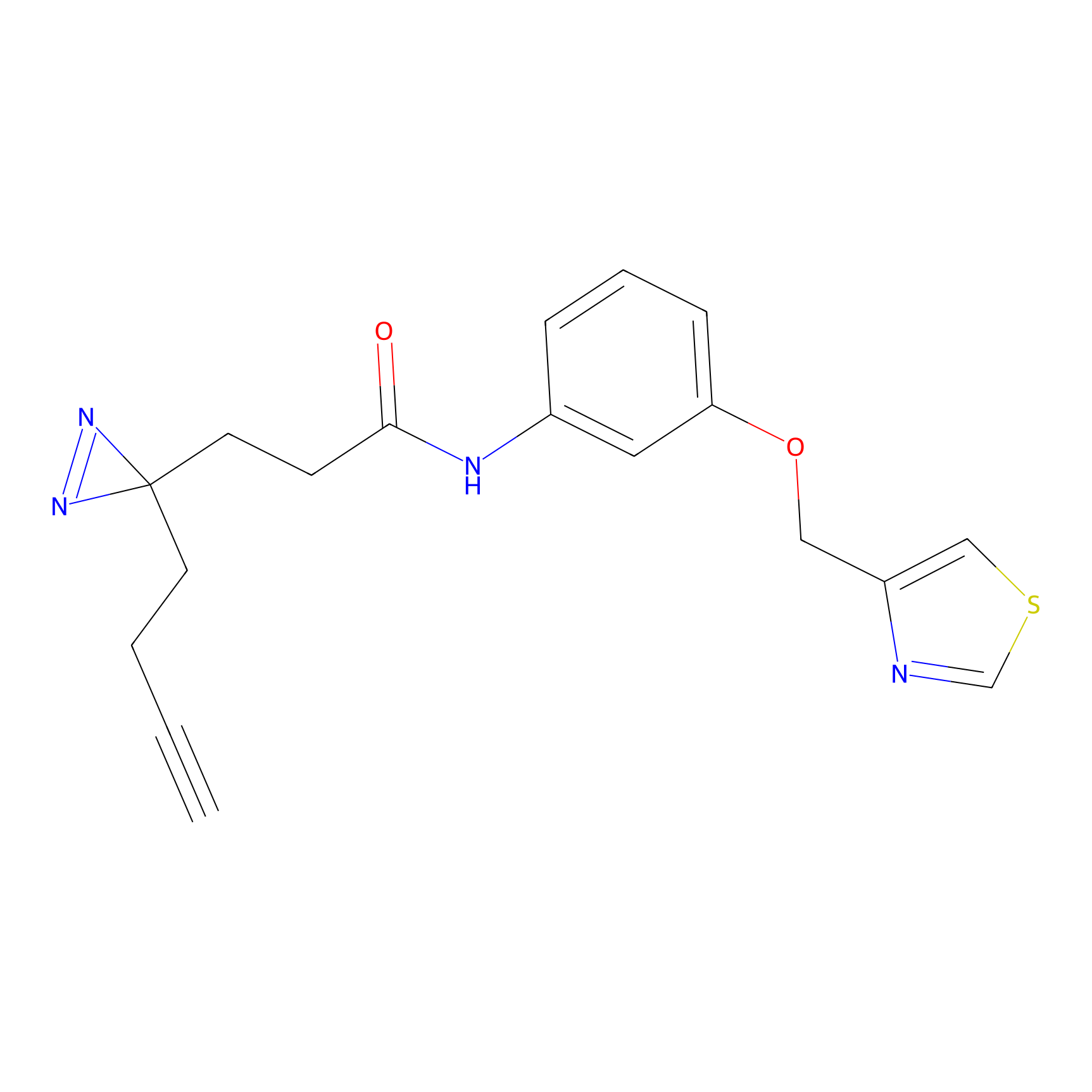
|
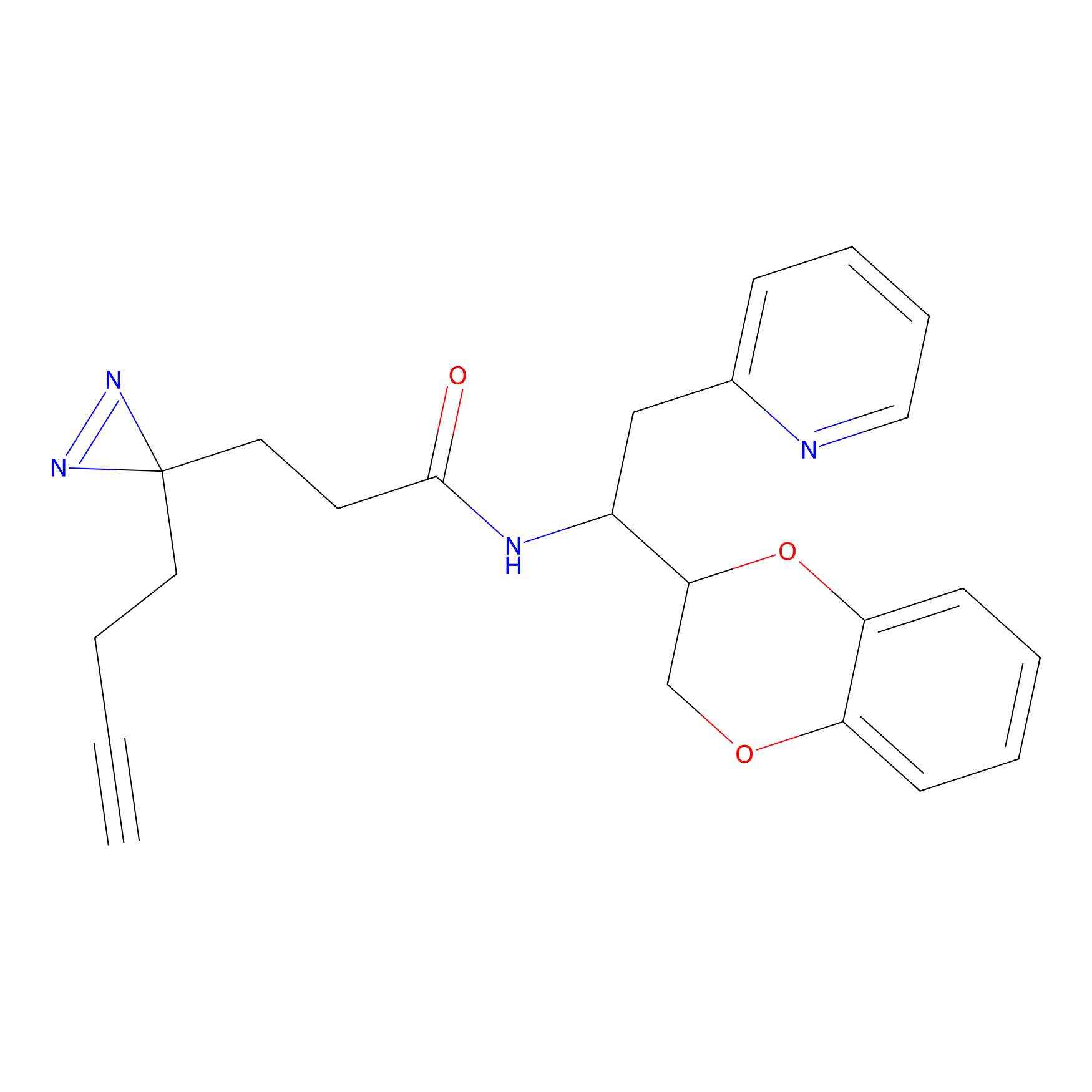
TH211 Probe Info |
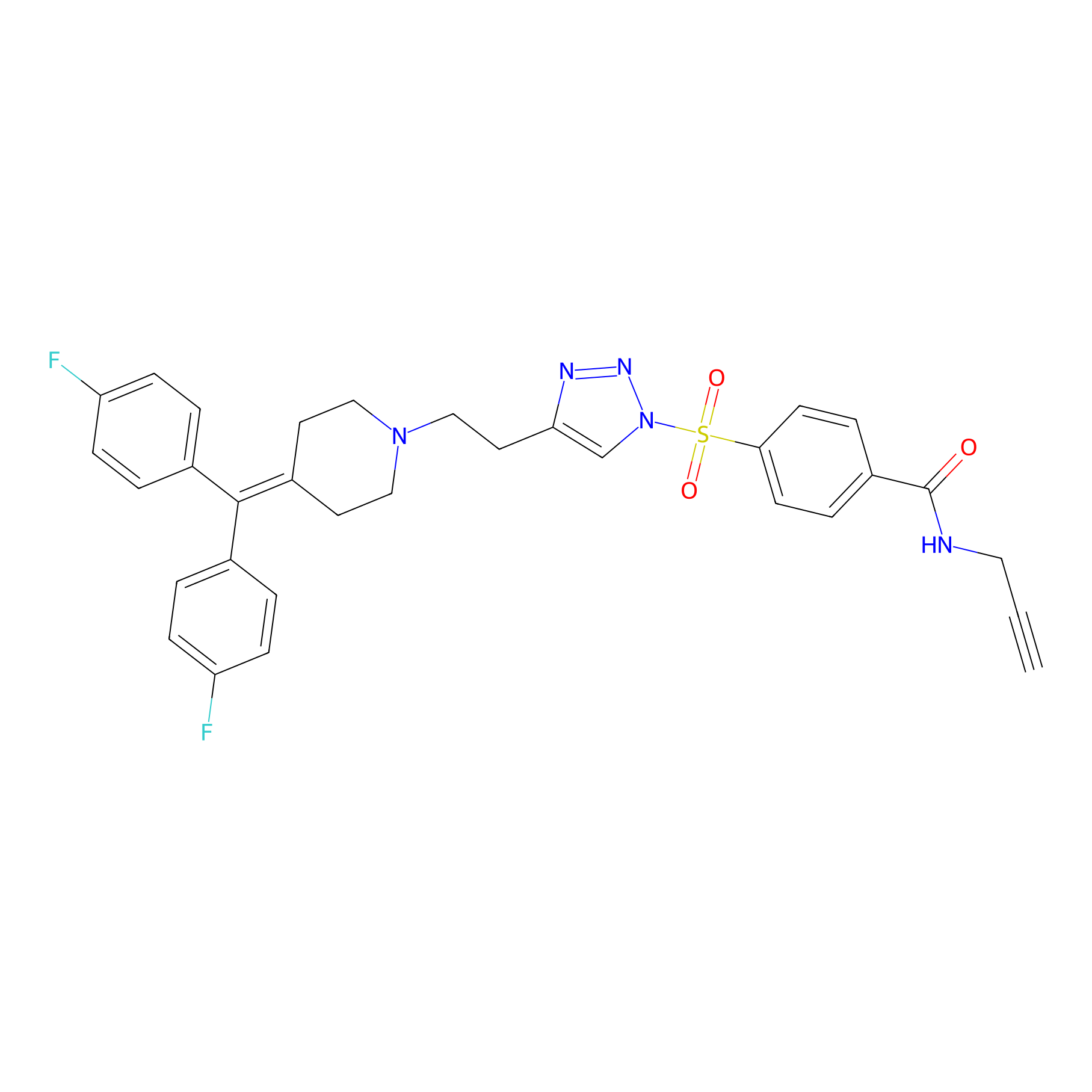 |
Y140(6.92) | LDD0257 | [2] | |
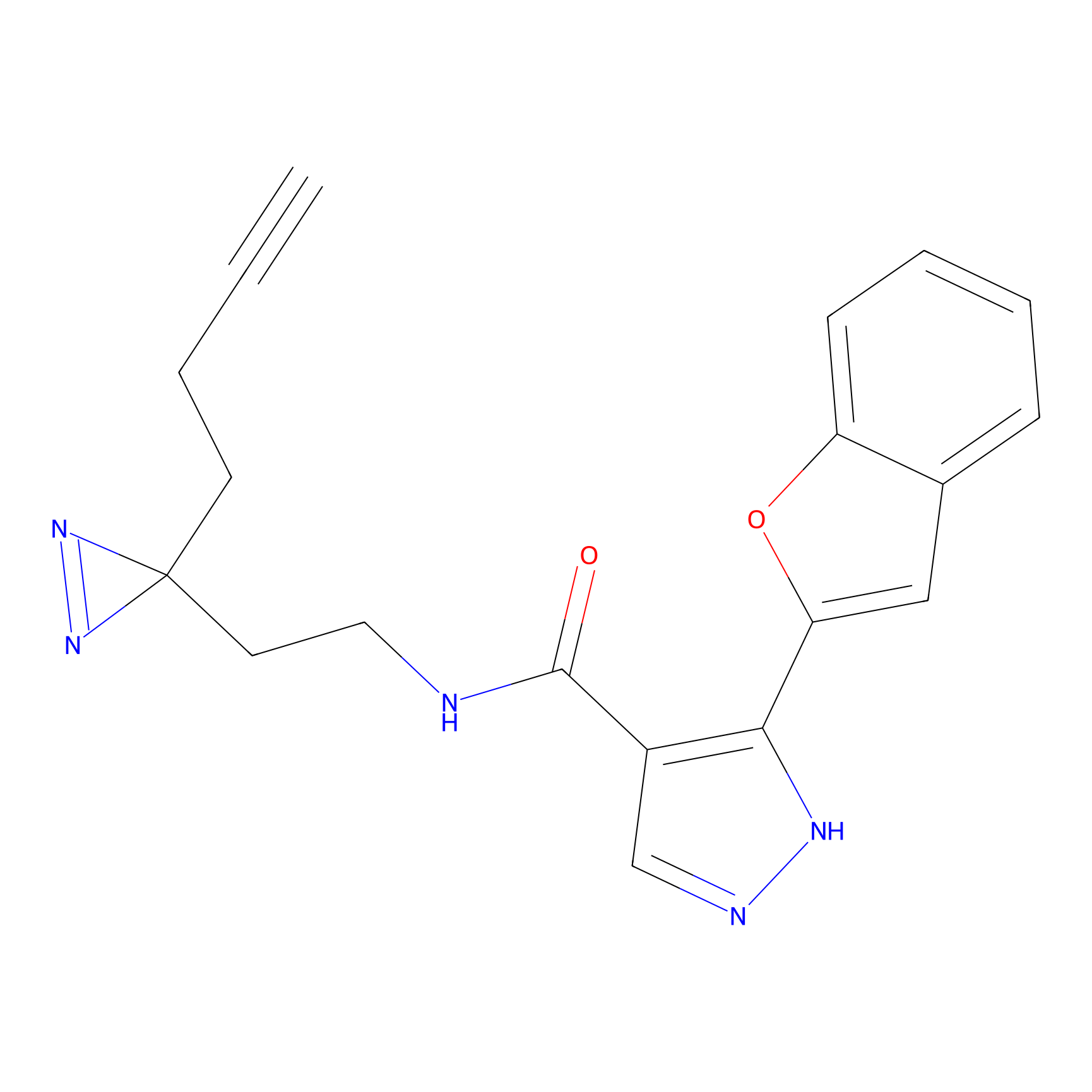
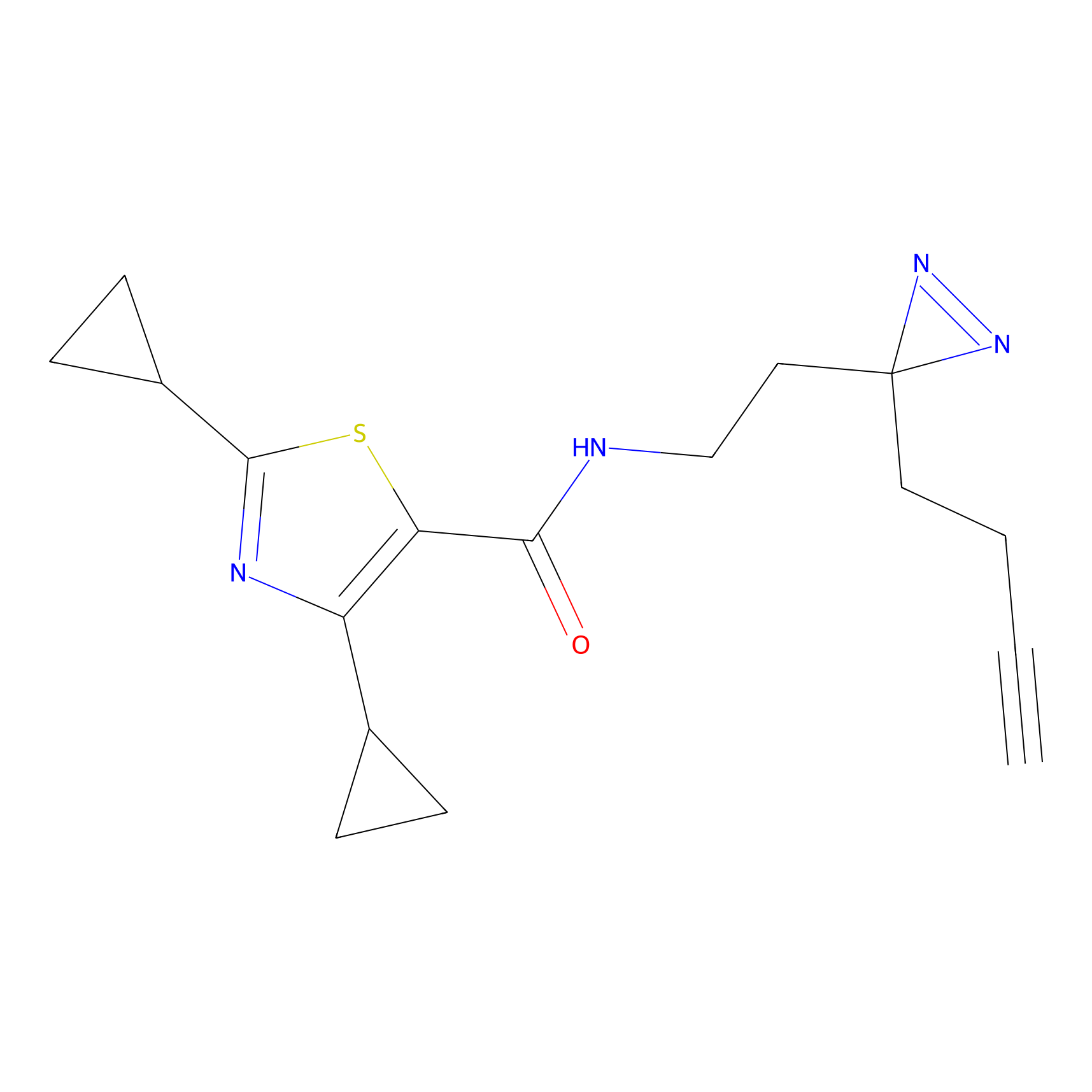
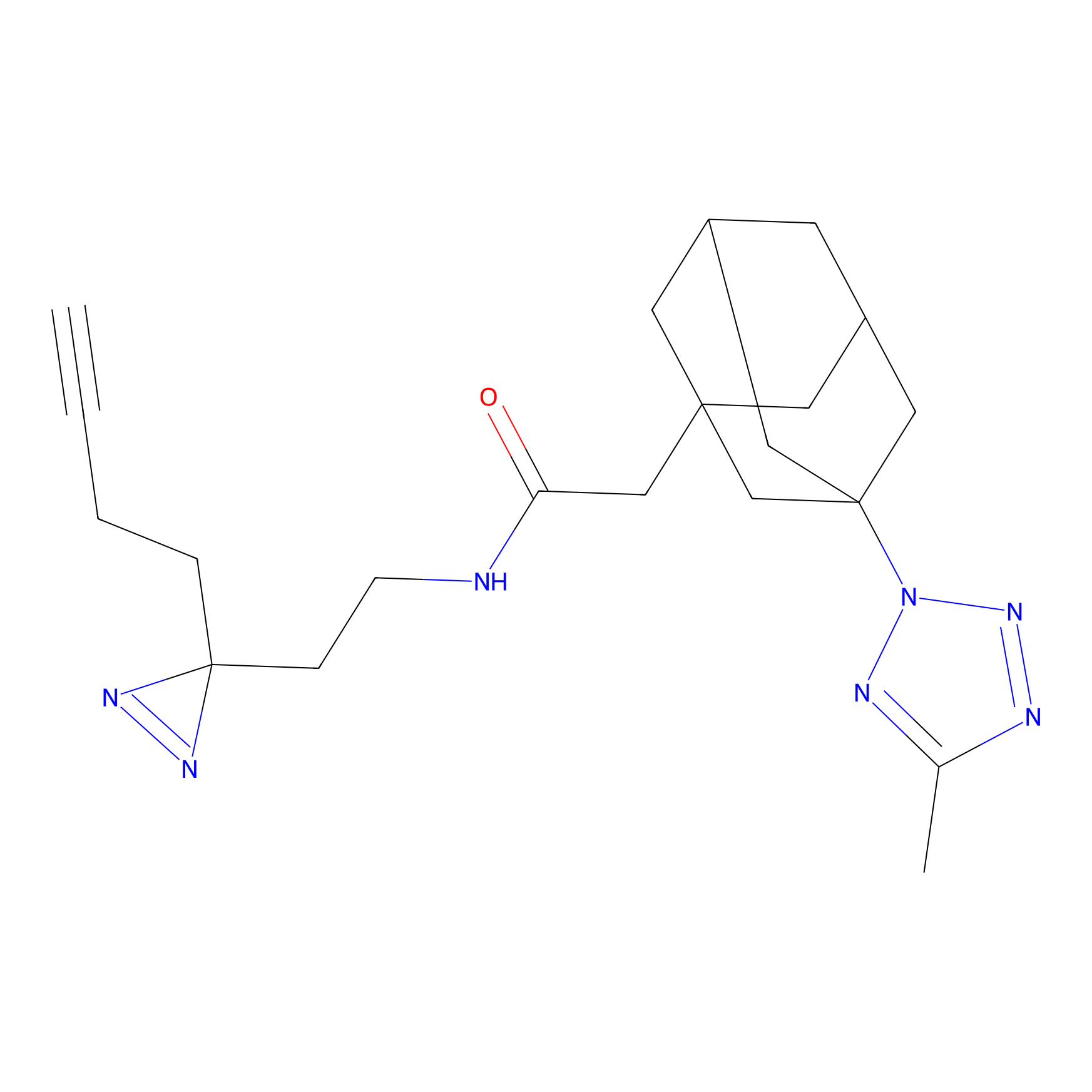
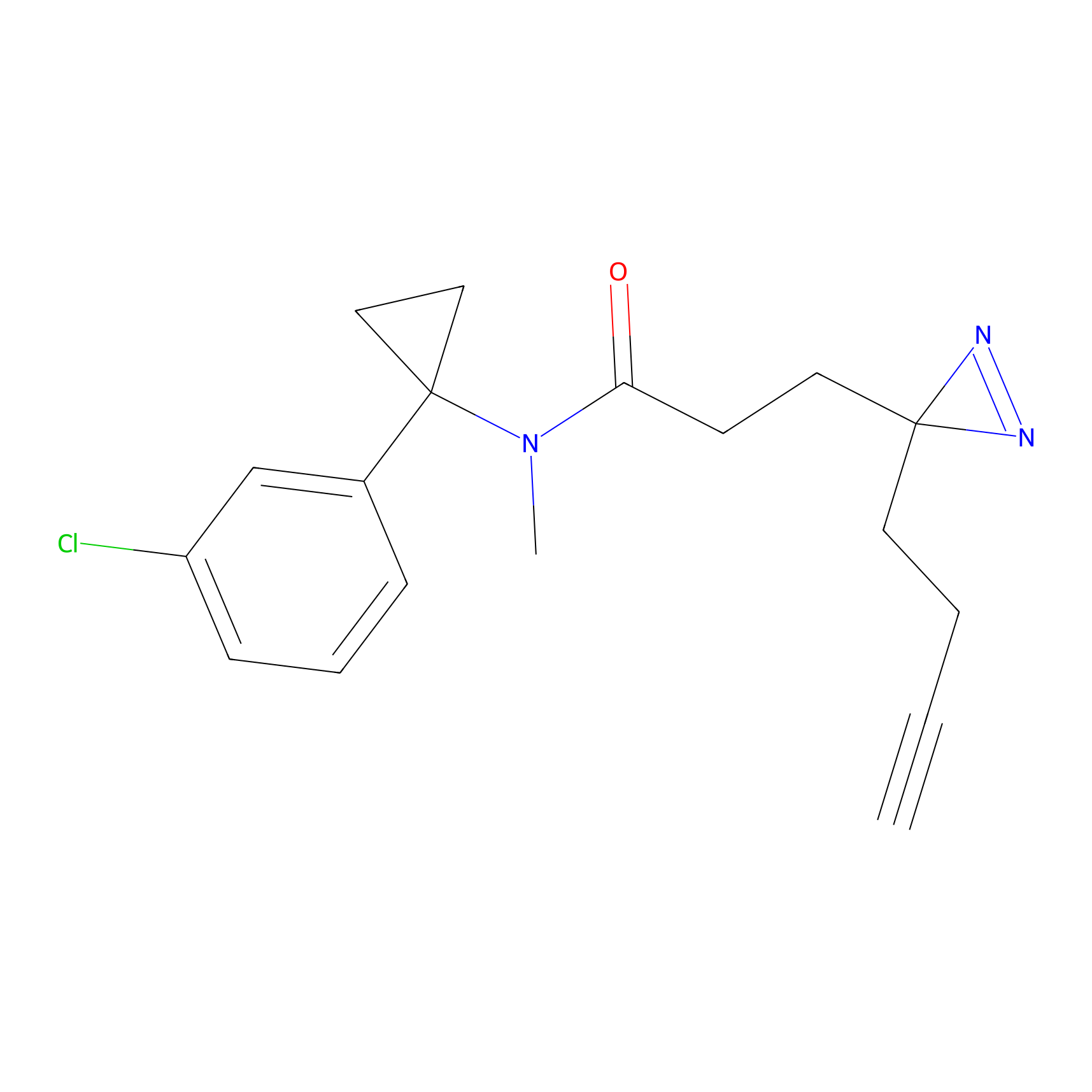
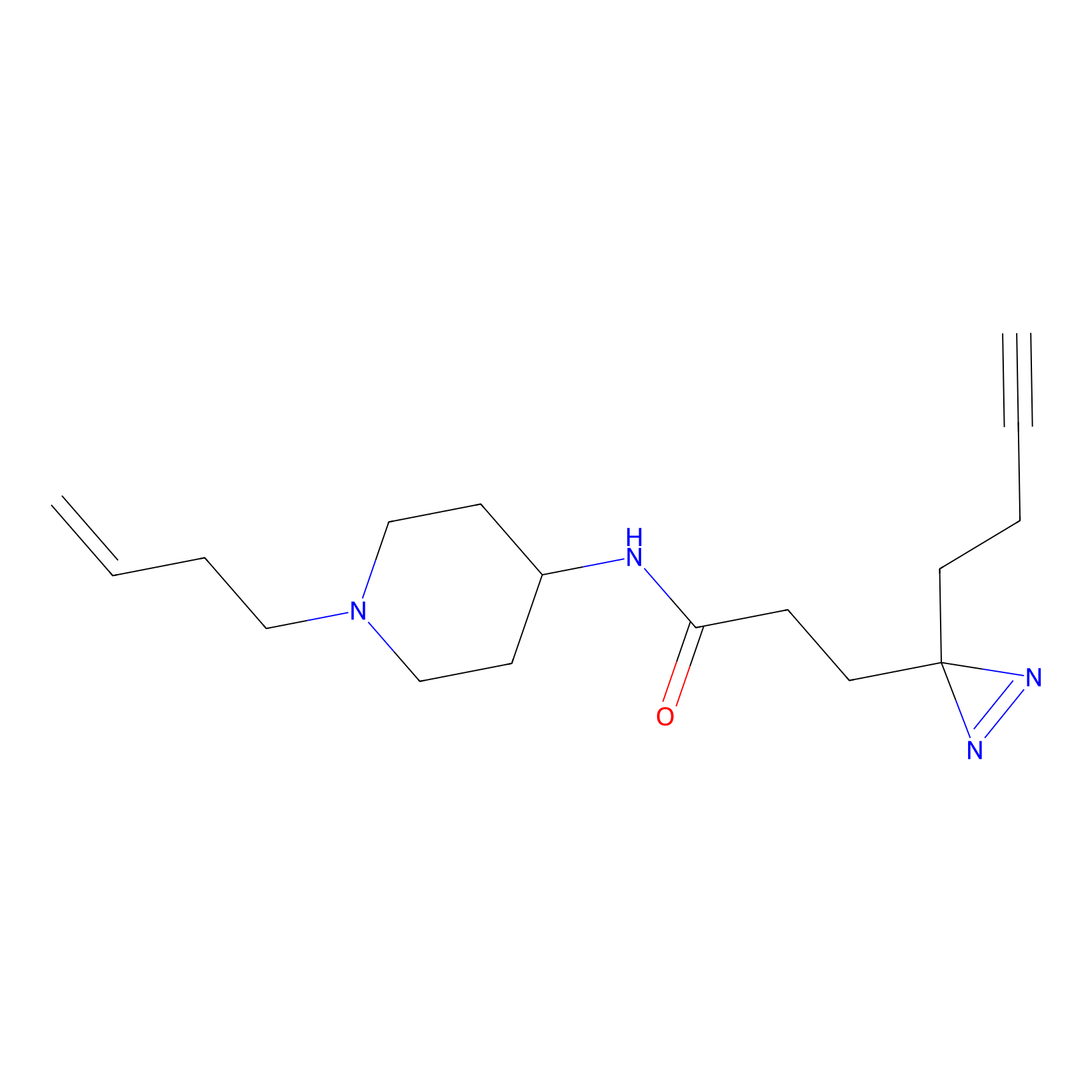
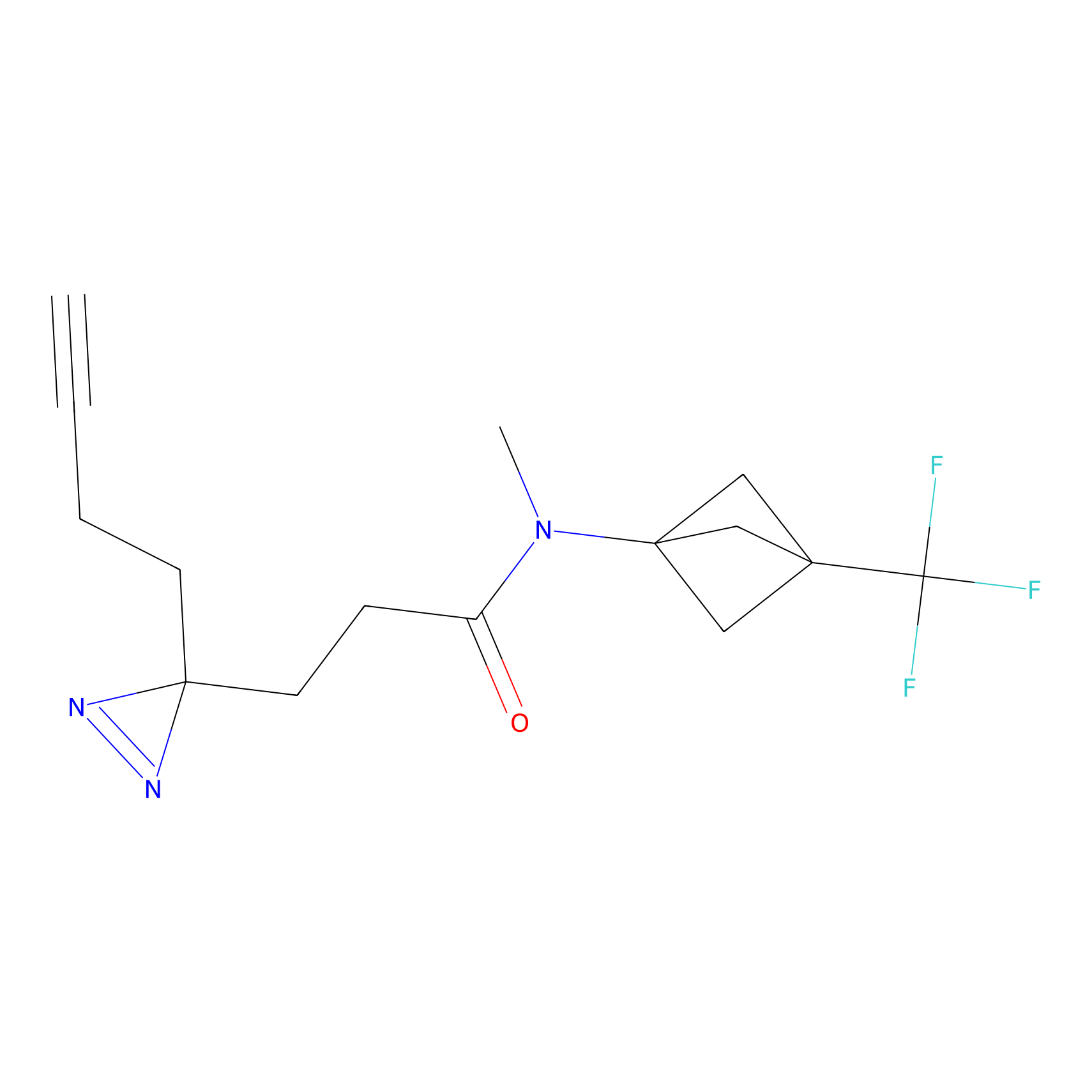
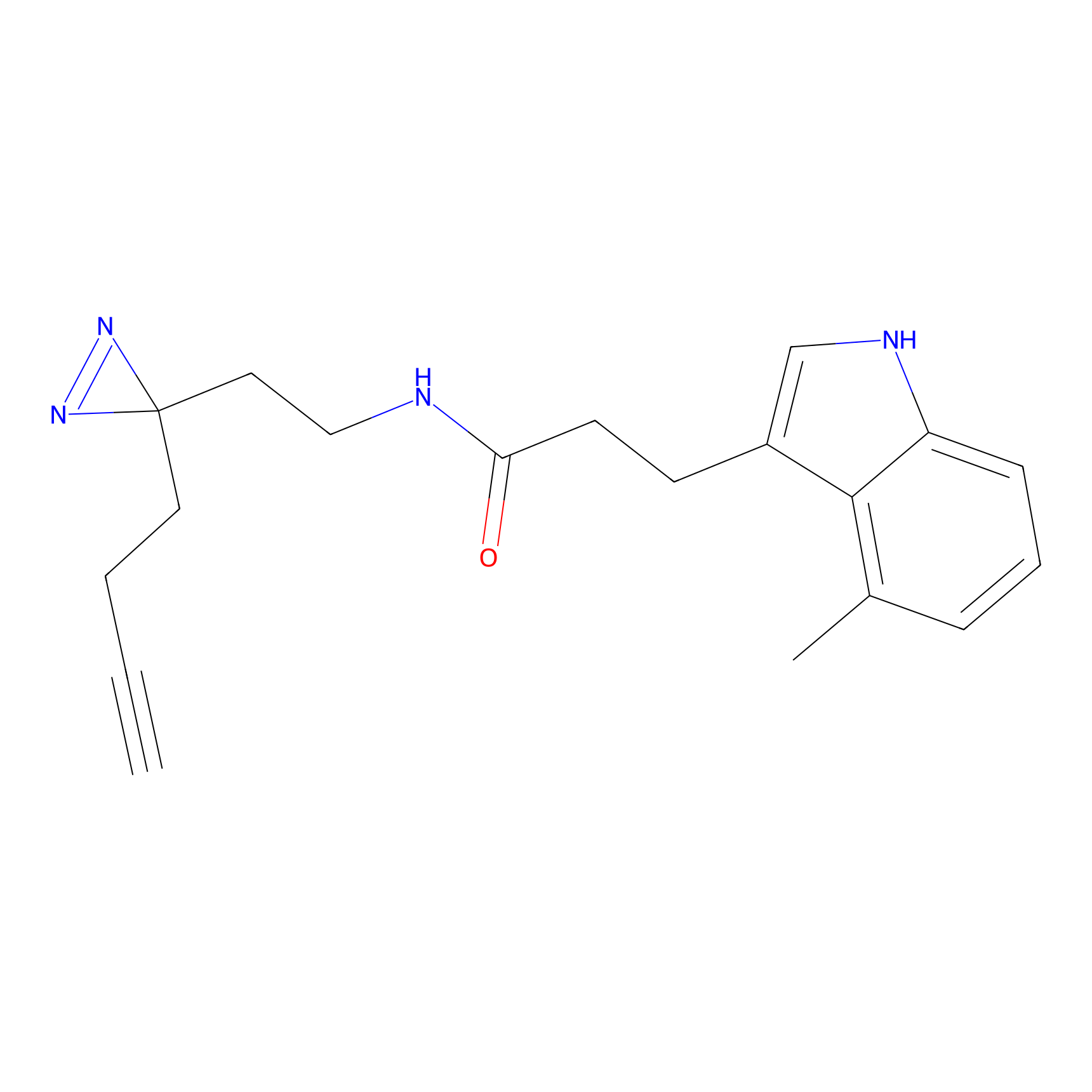
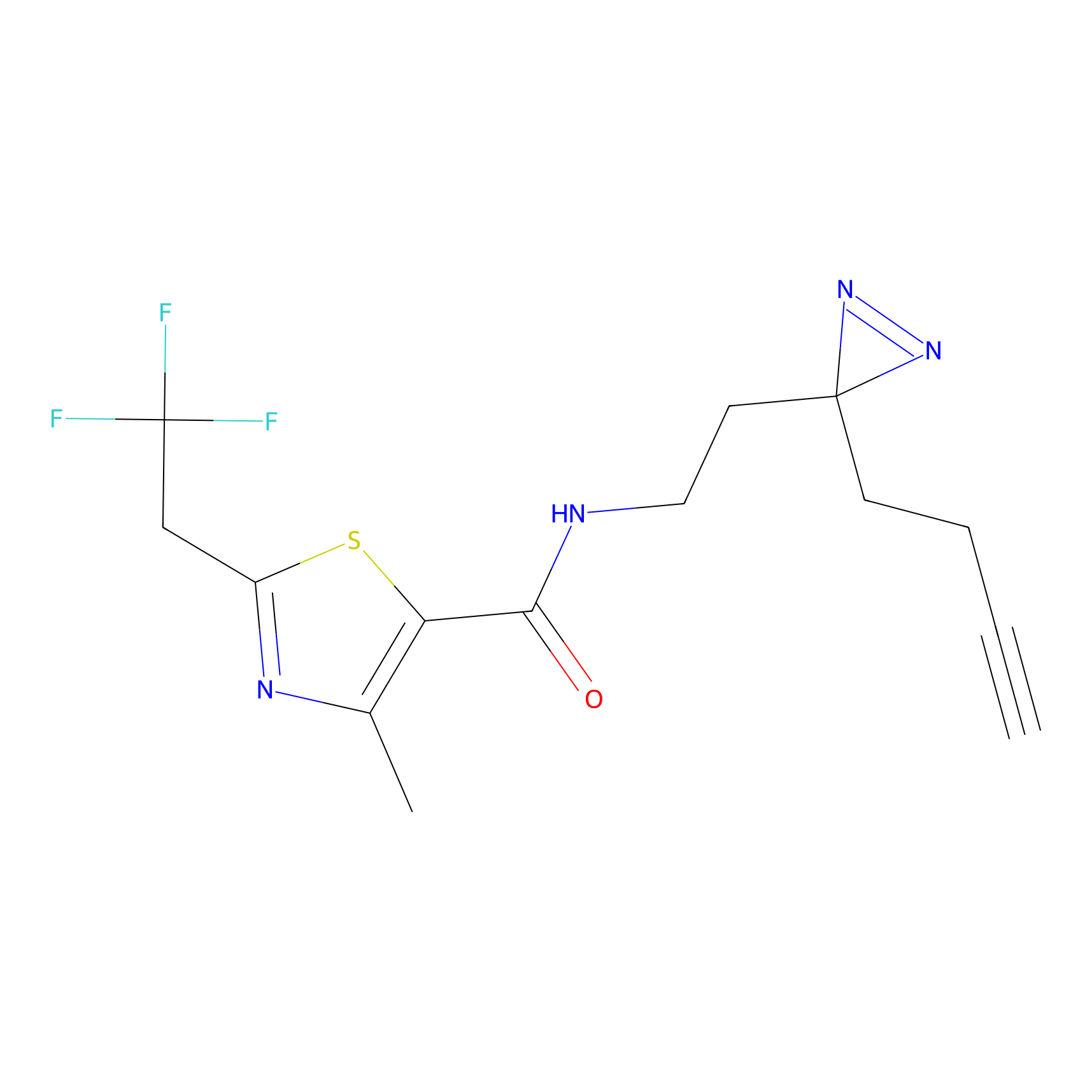
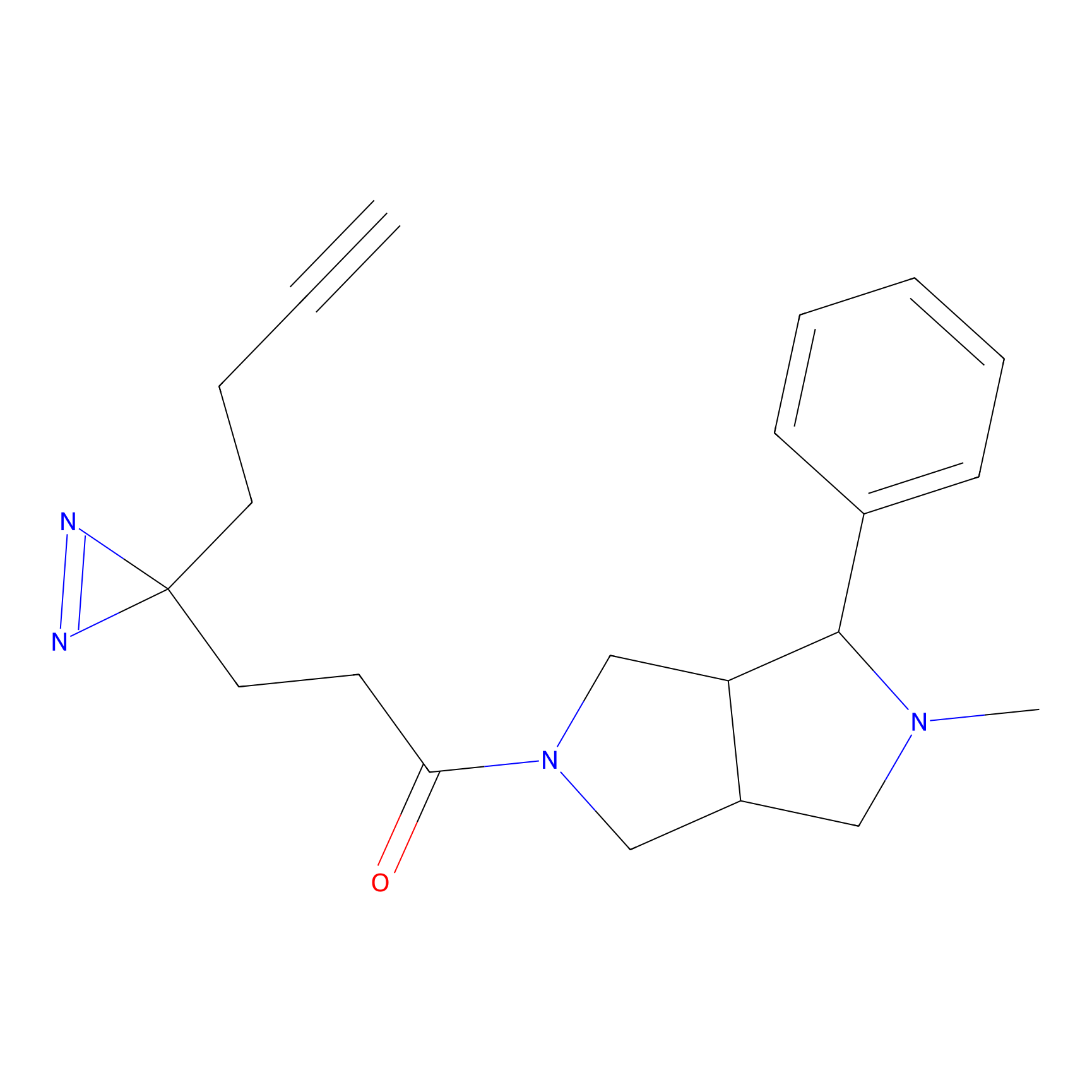
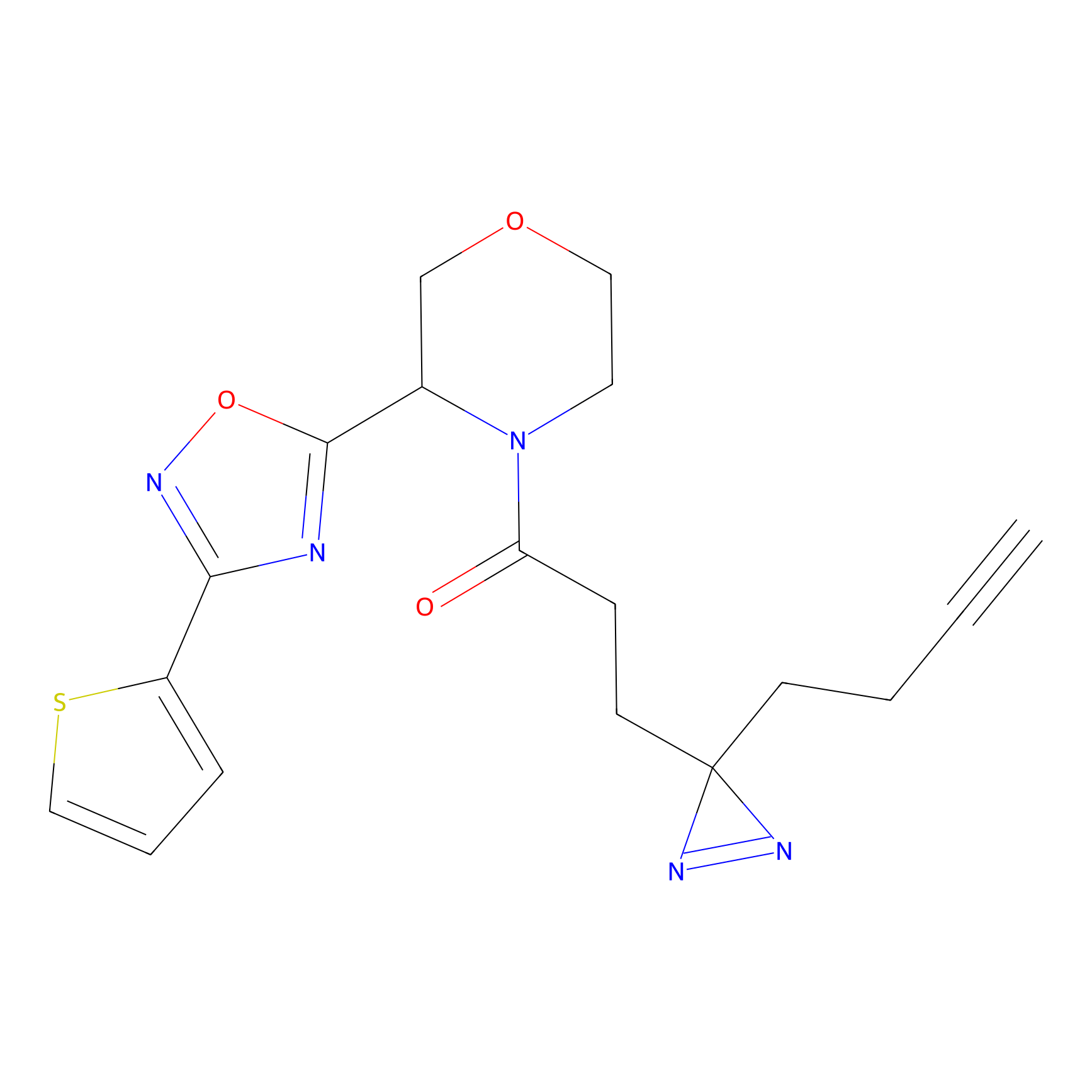
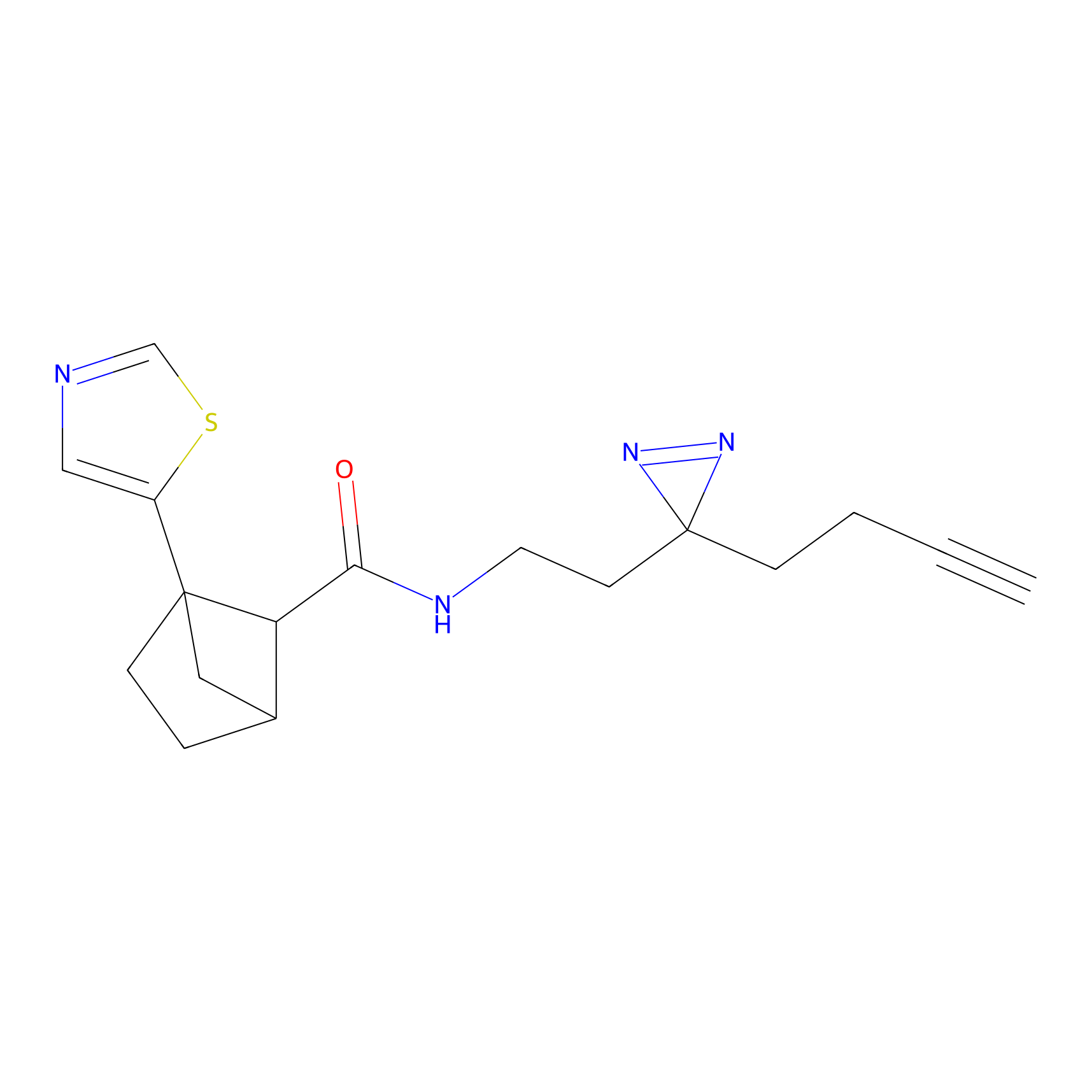
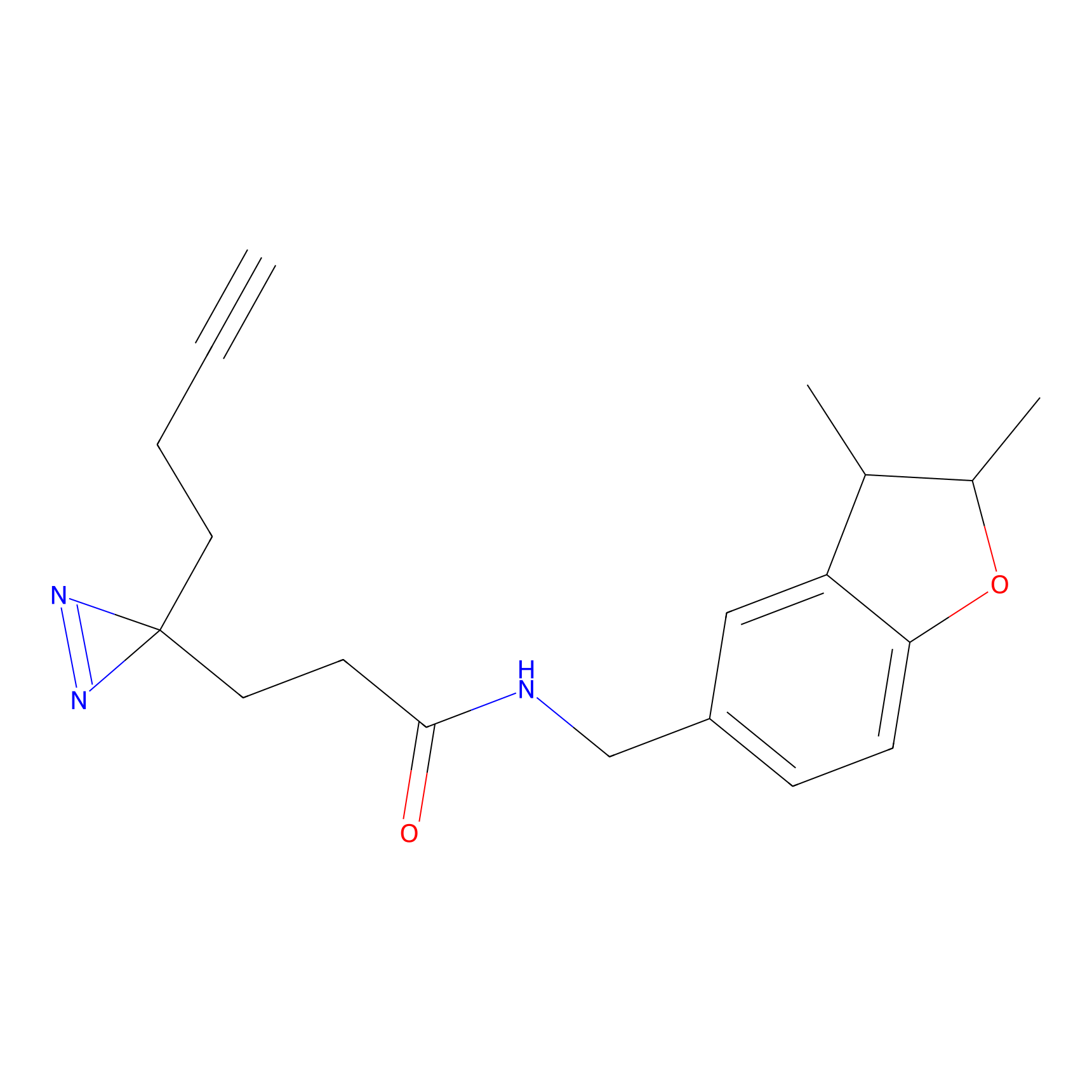
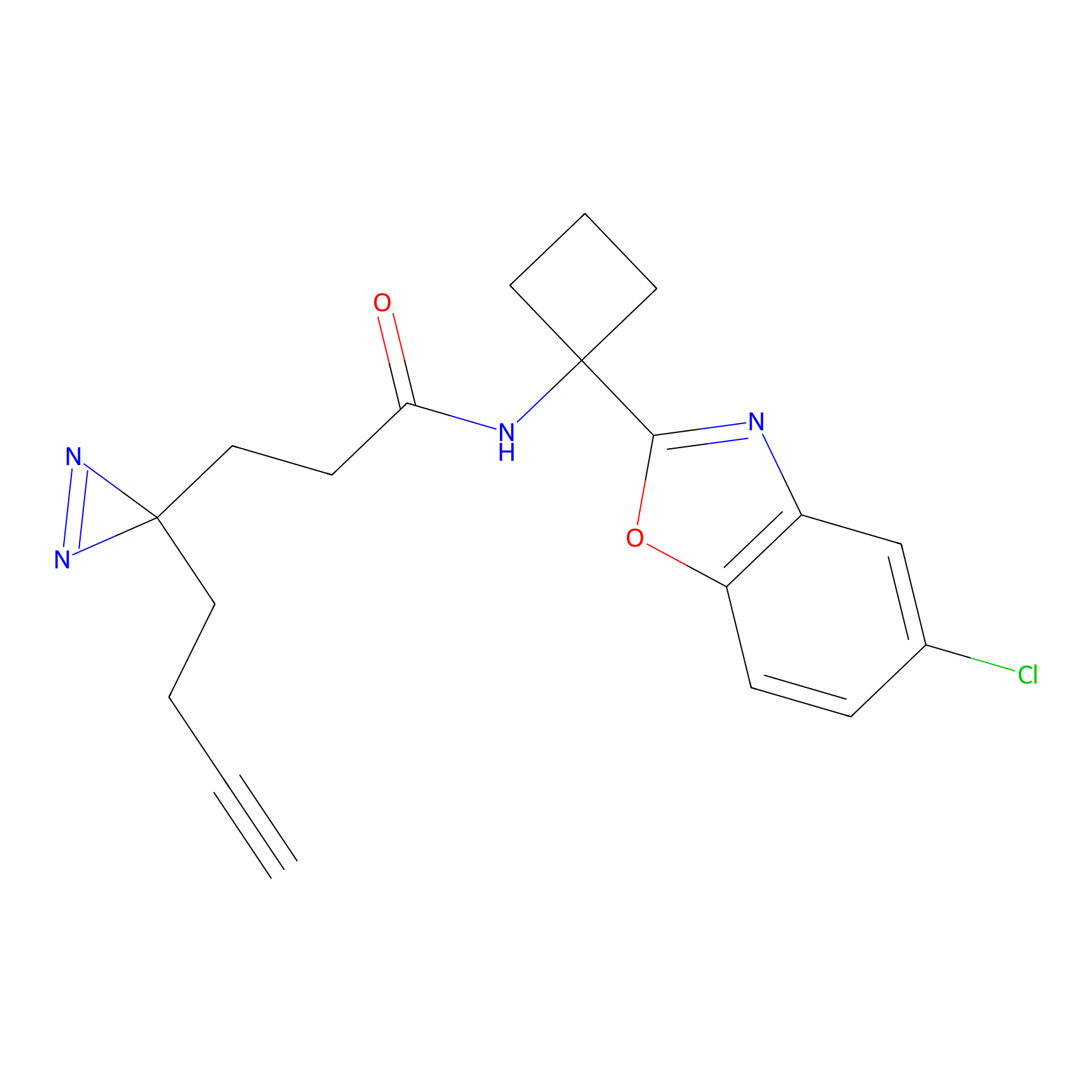
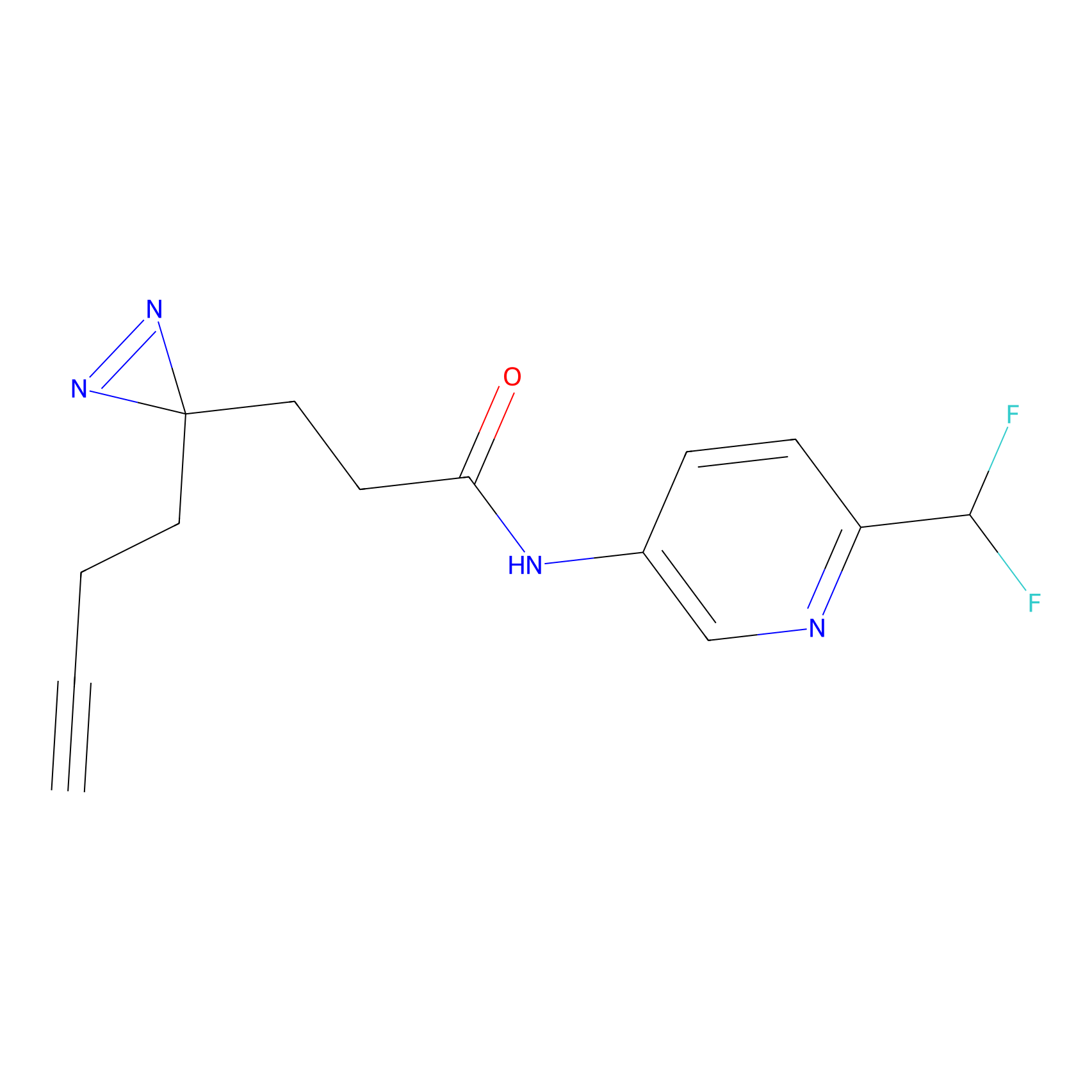
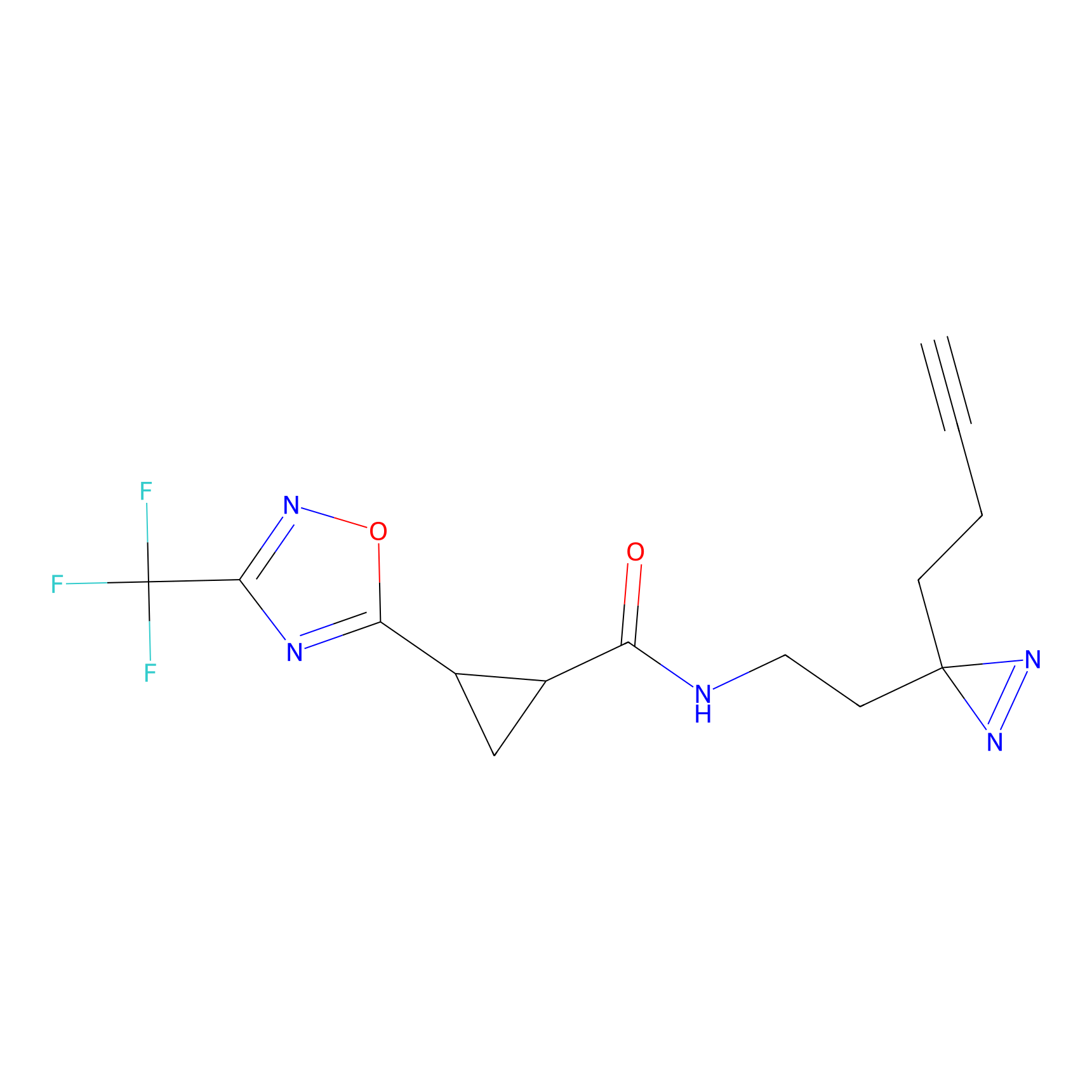
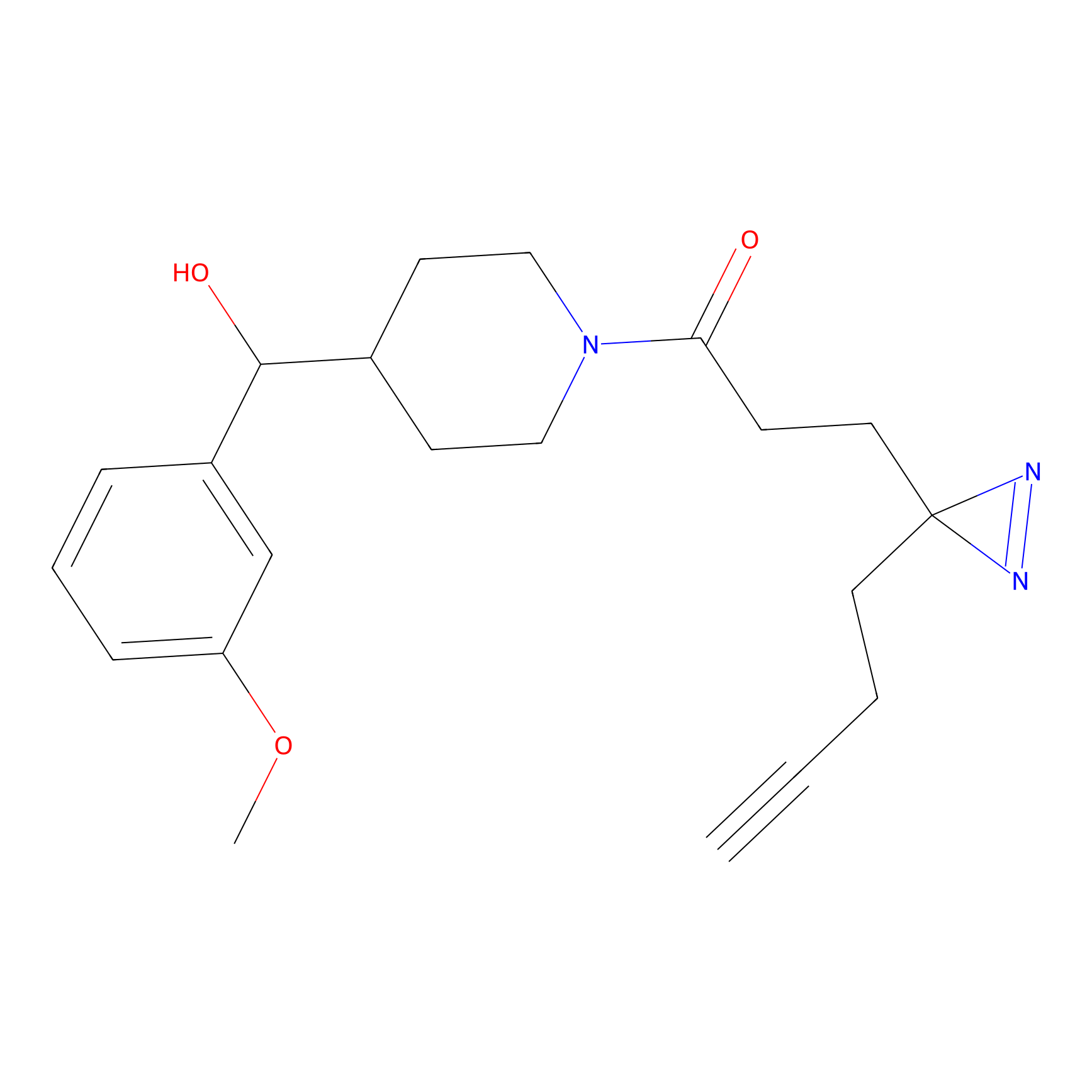
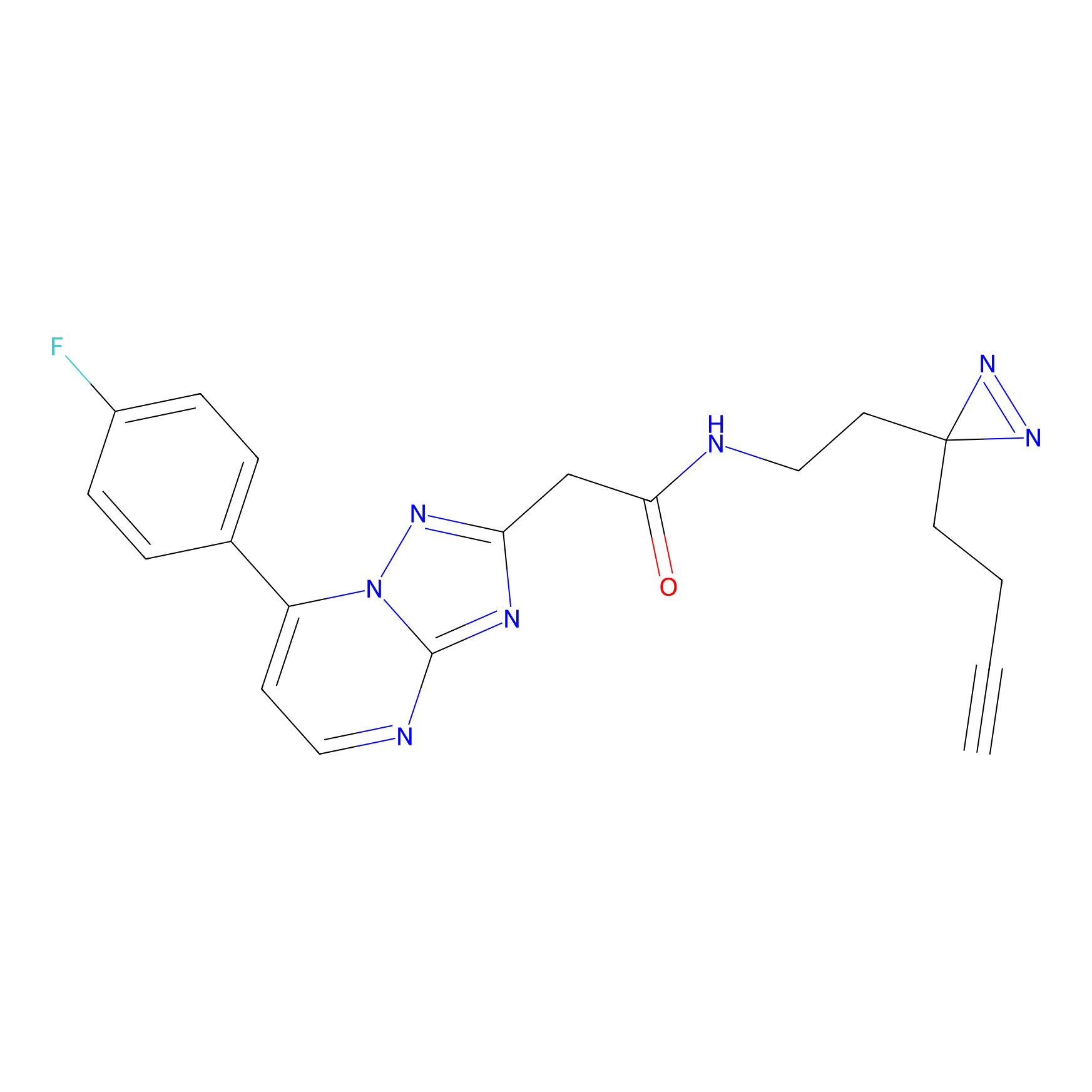
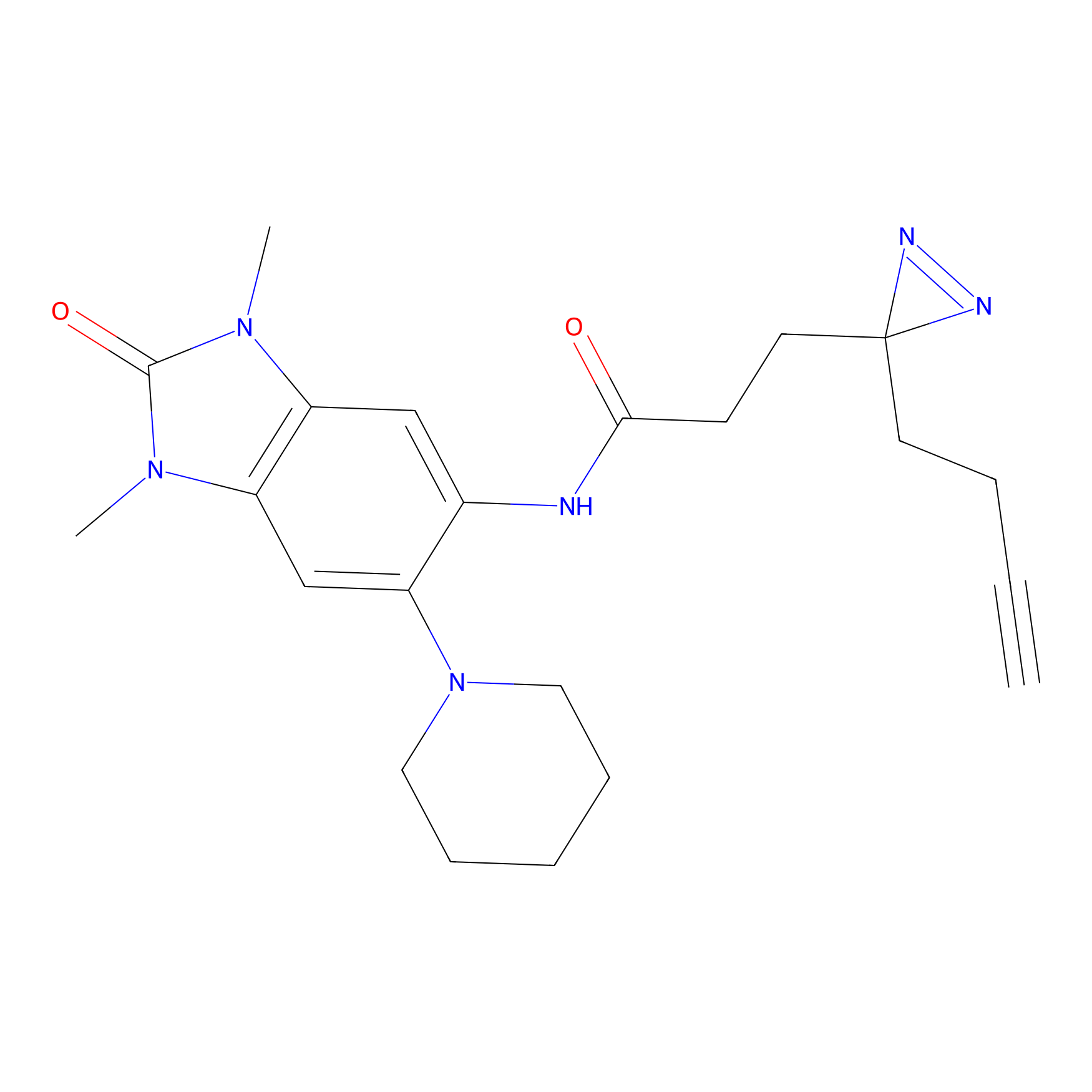
|
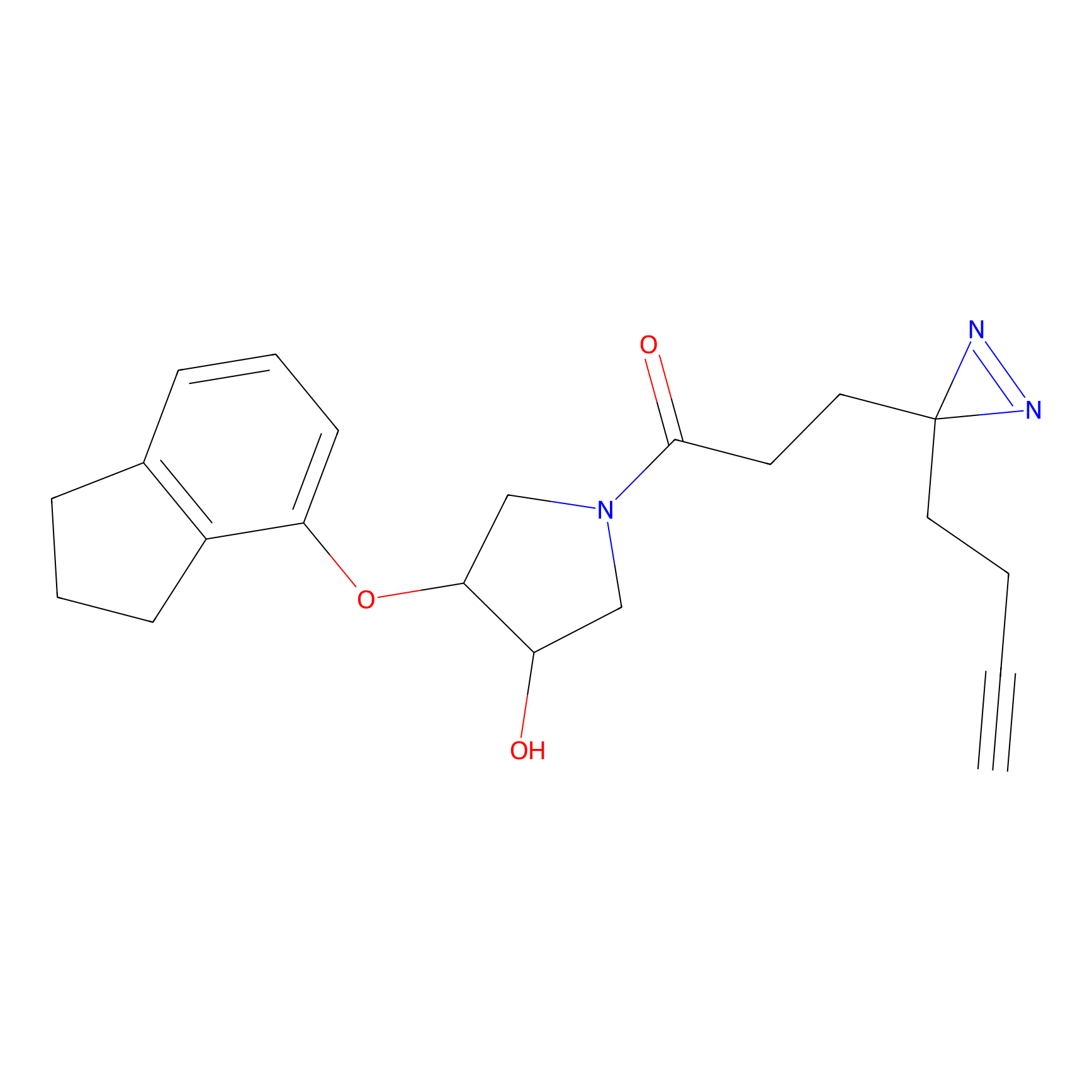
STPyne Probe Info |
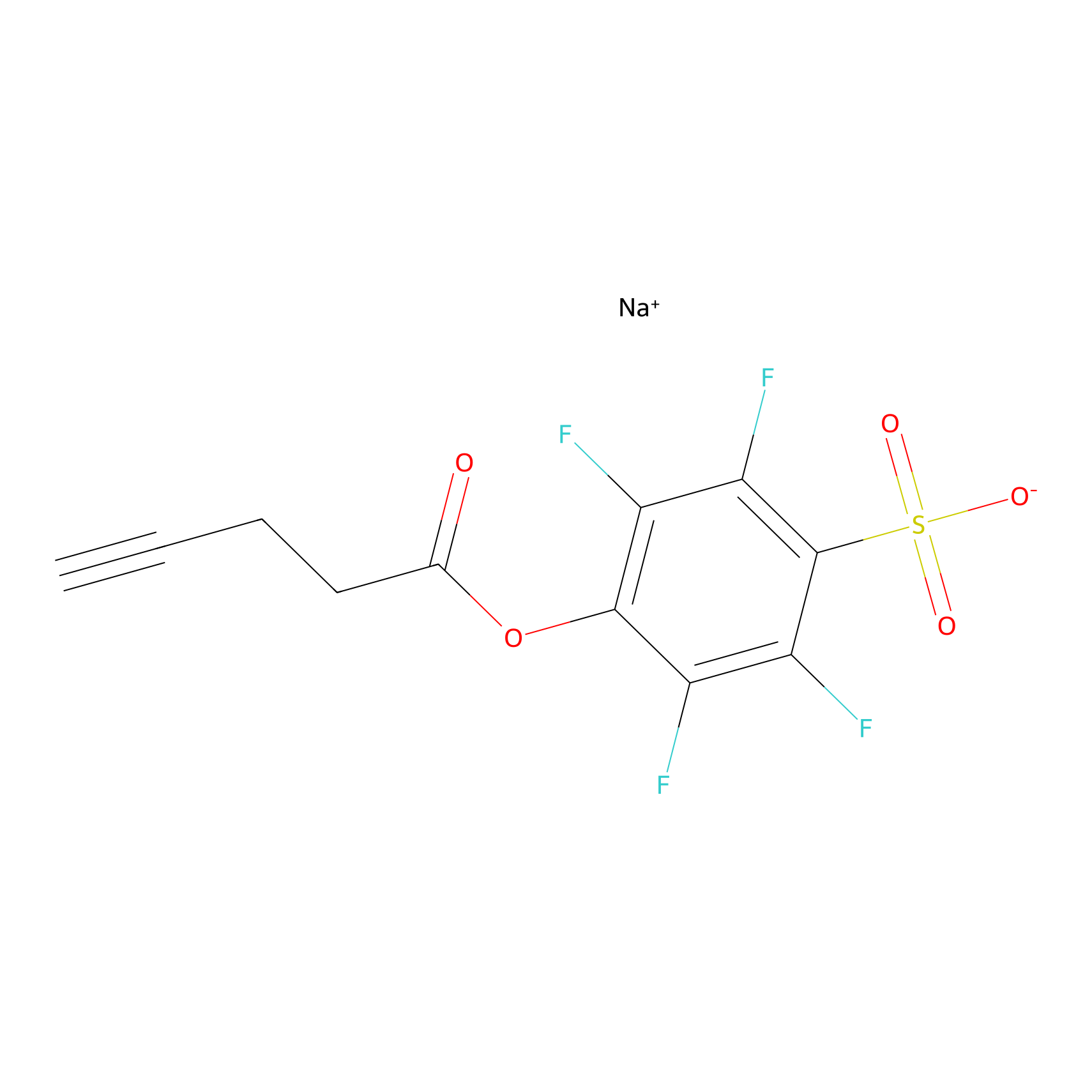 |
K121(1.93) | LDD0277 | [3] | |
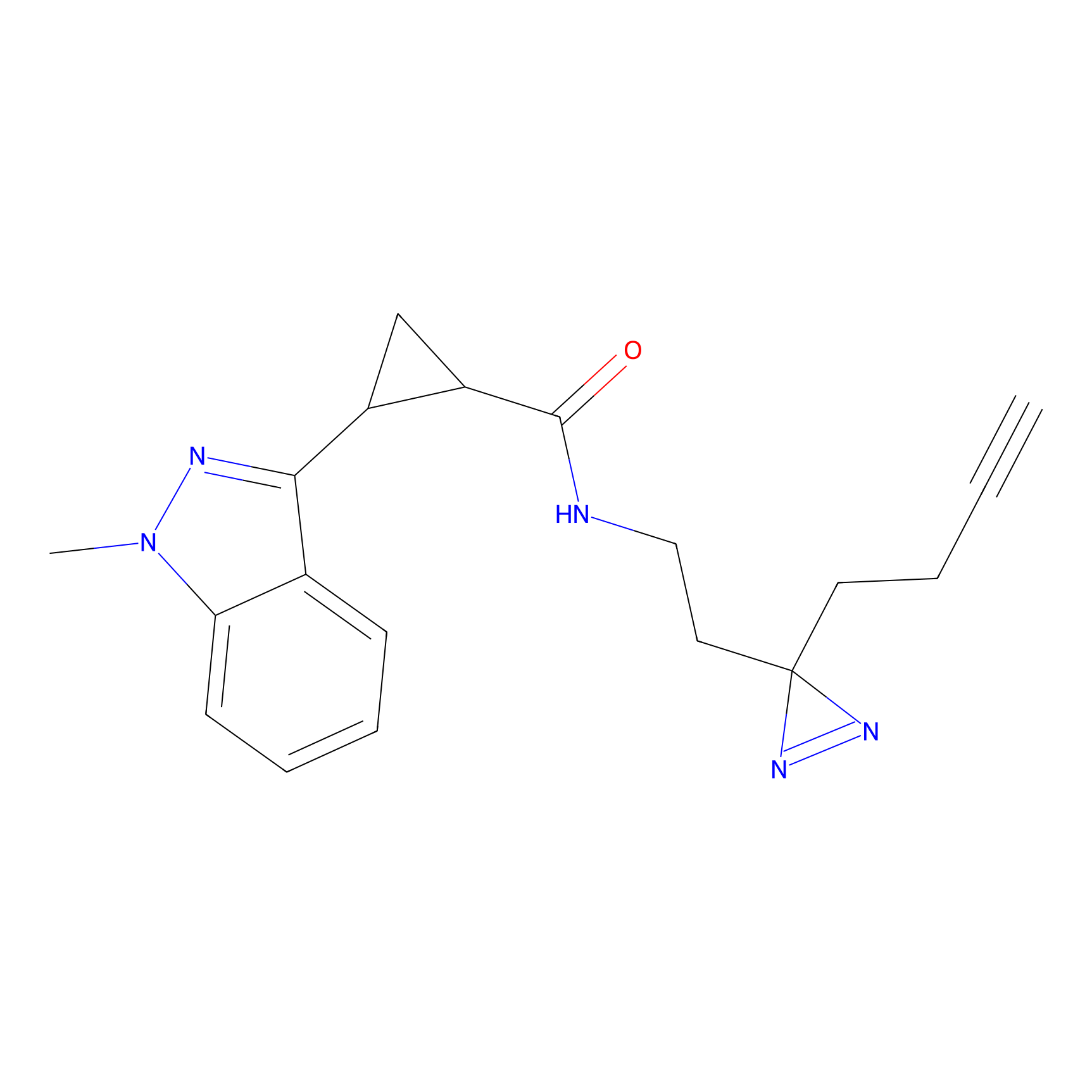
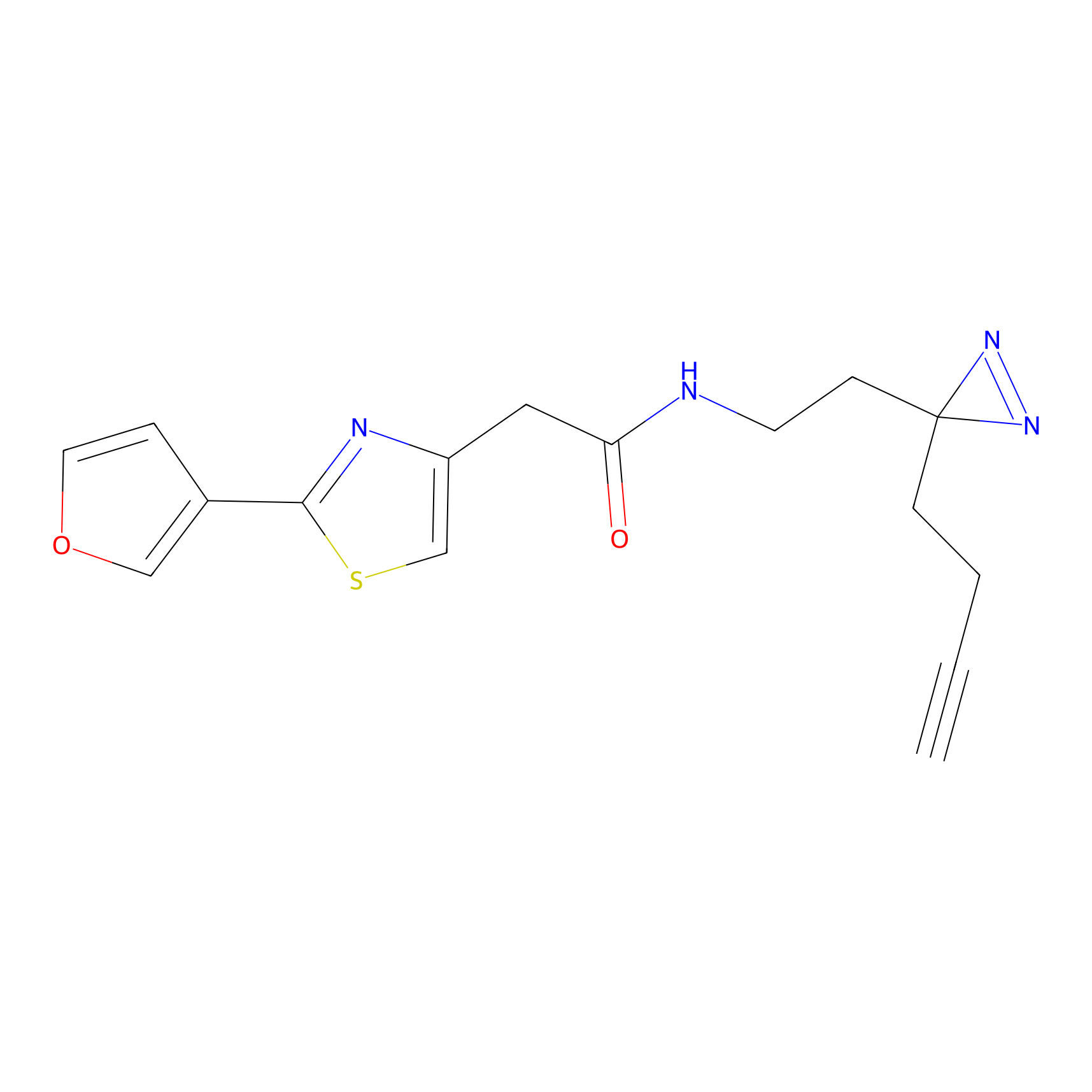
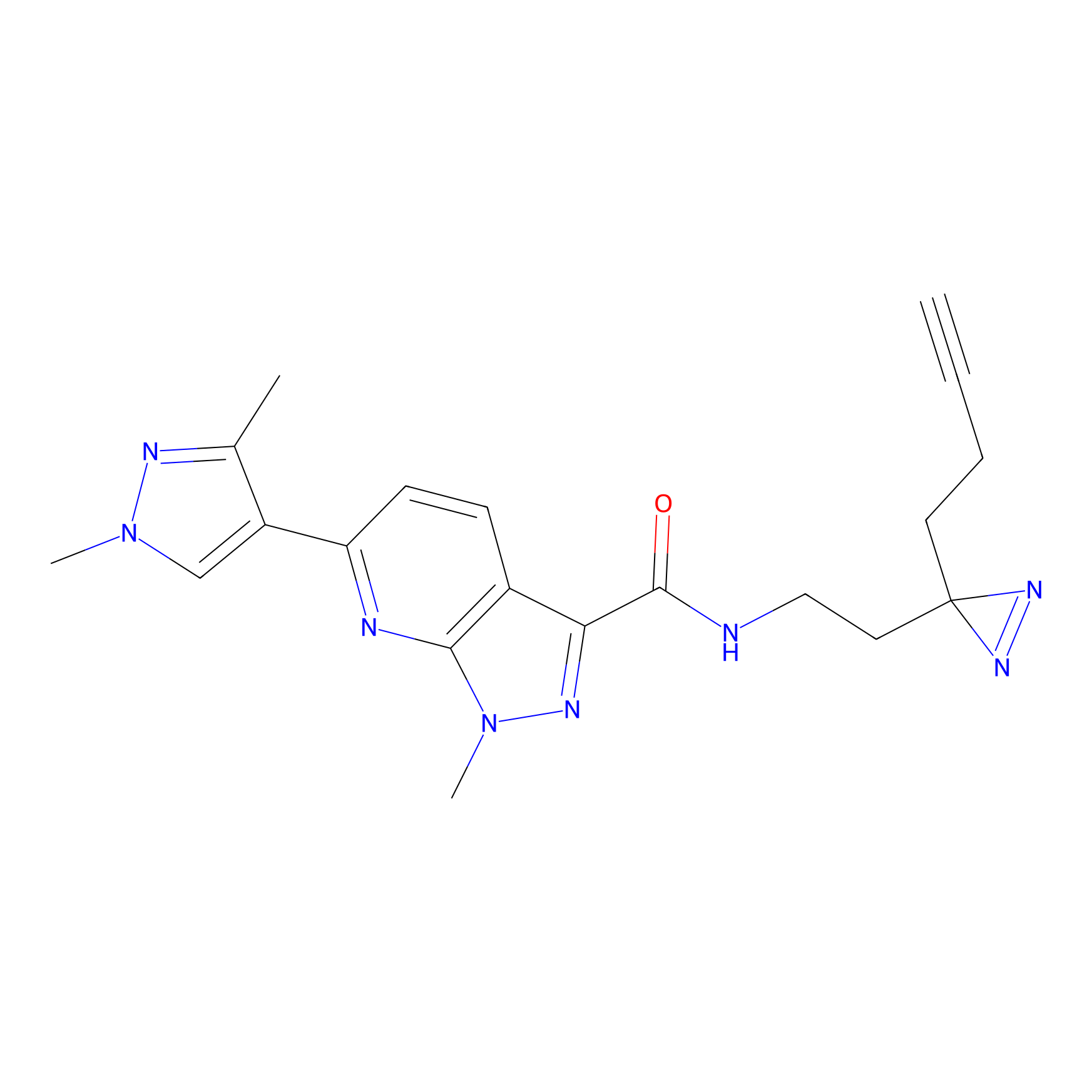
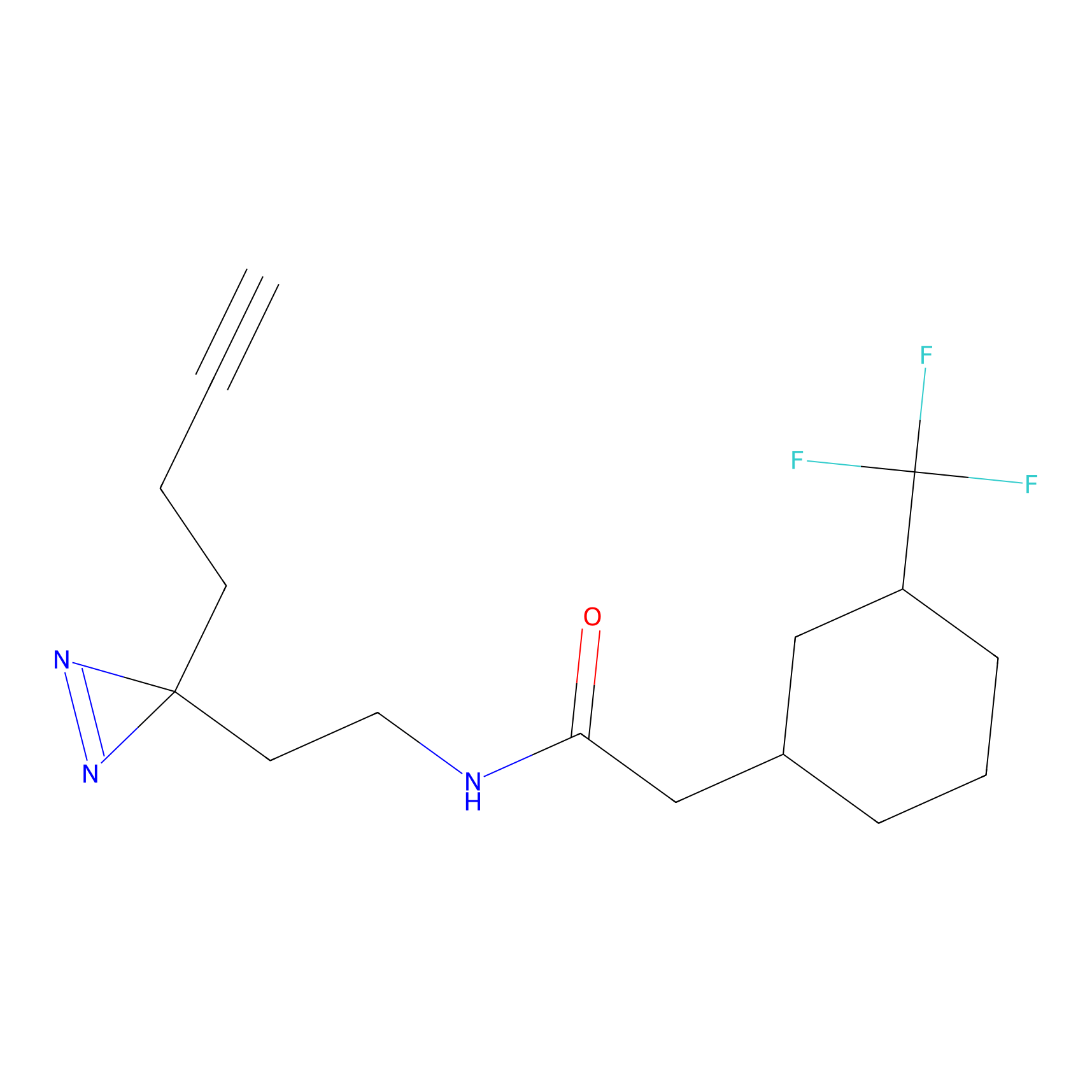
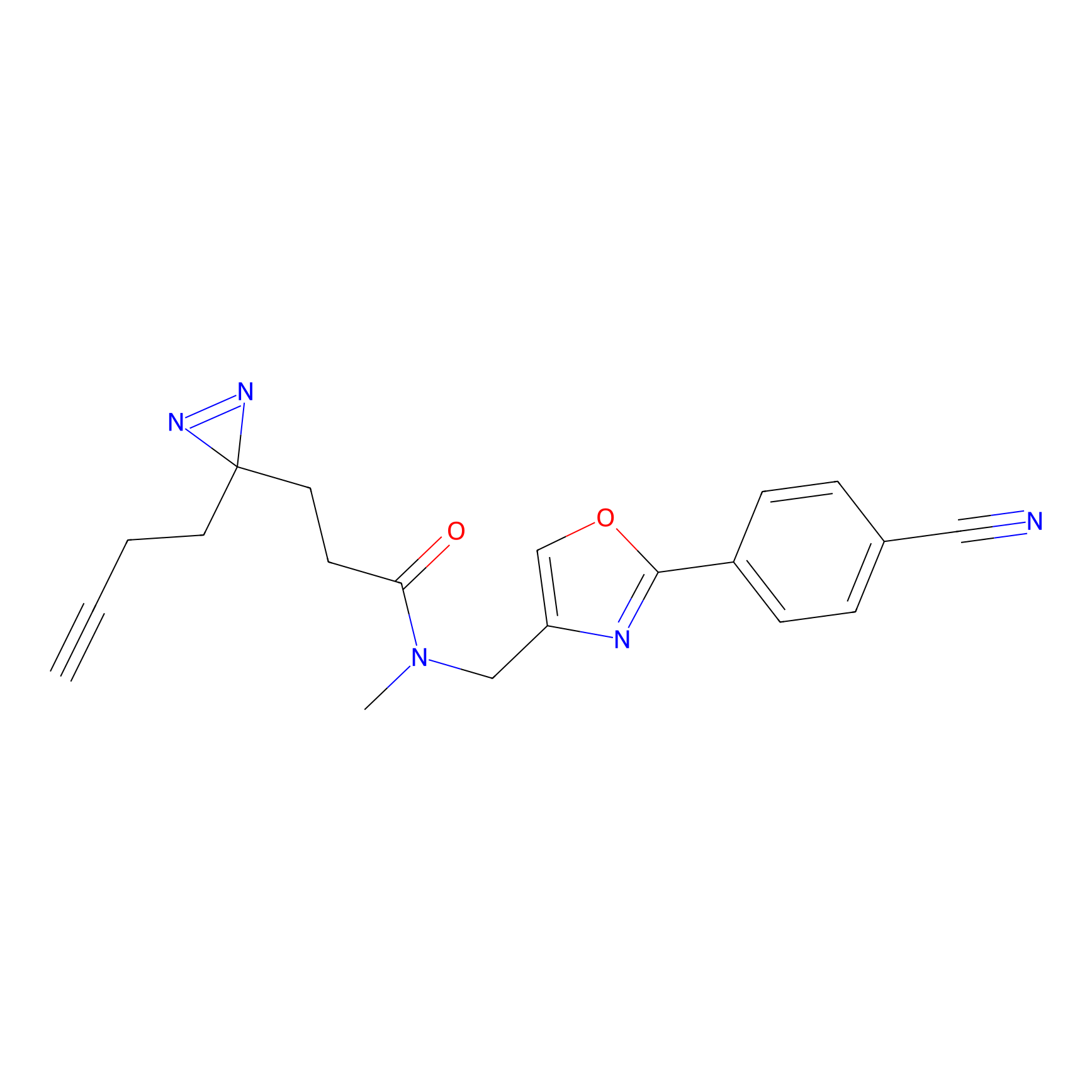
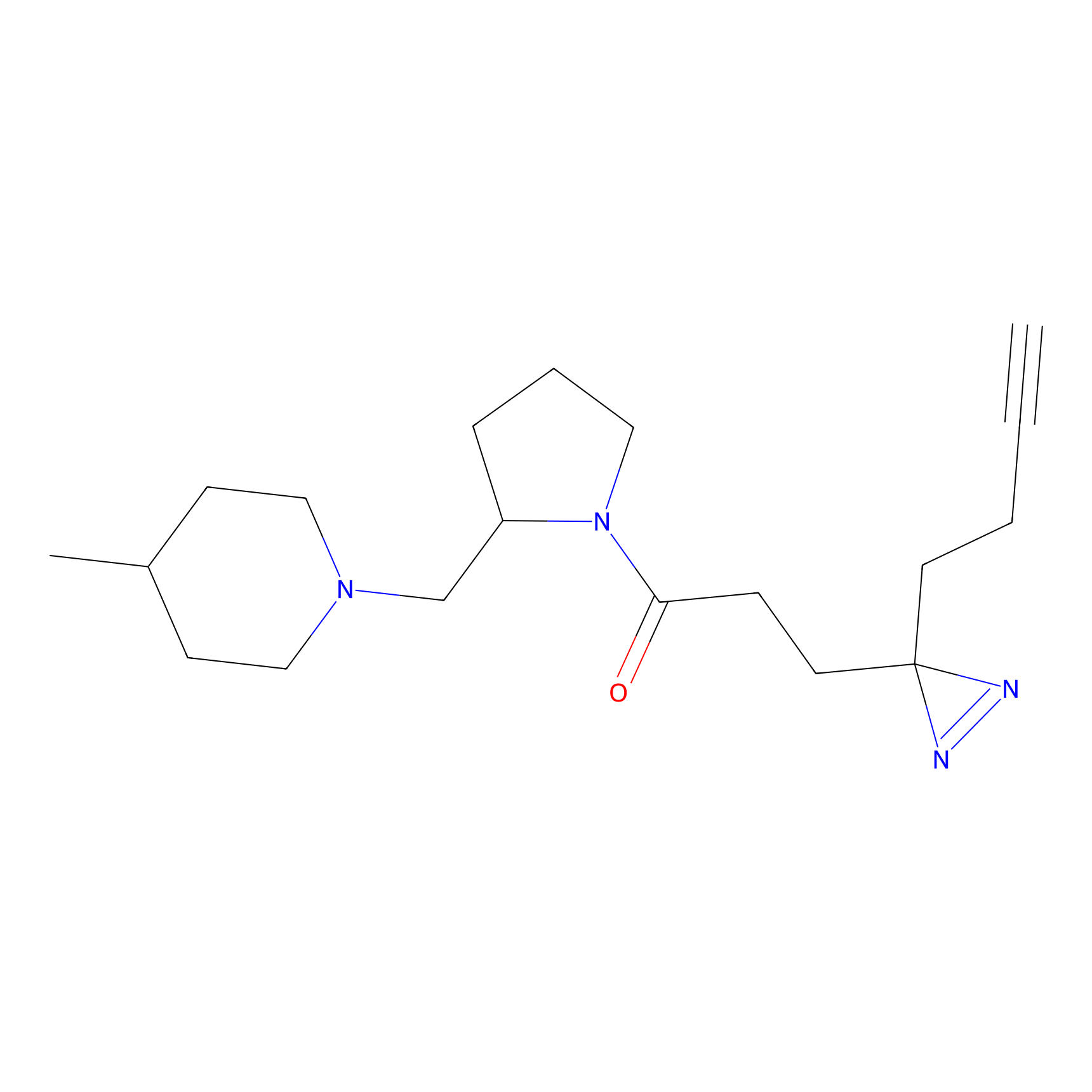
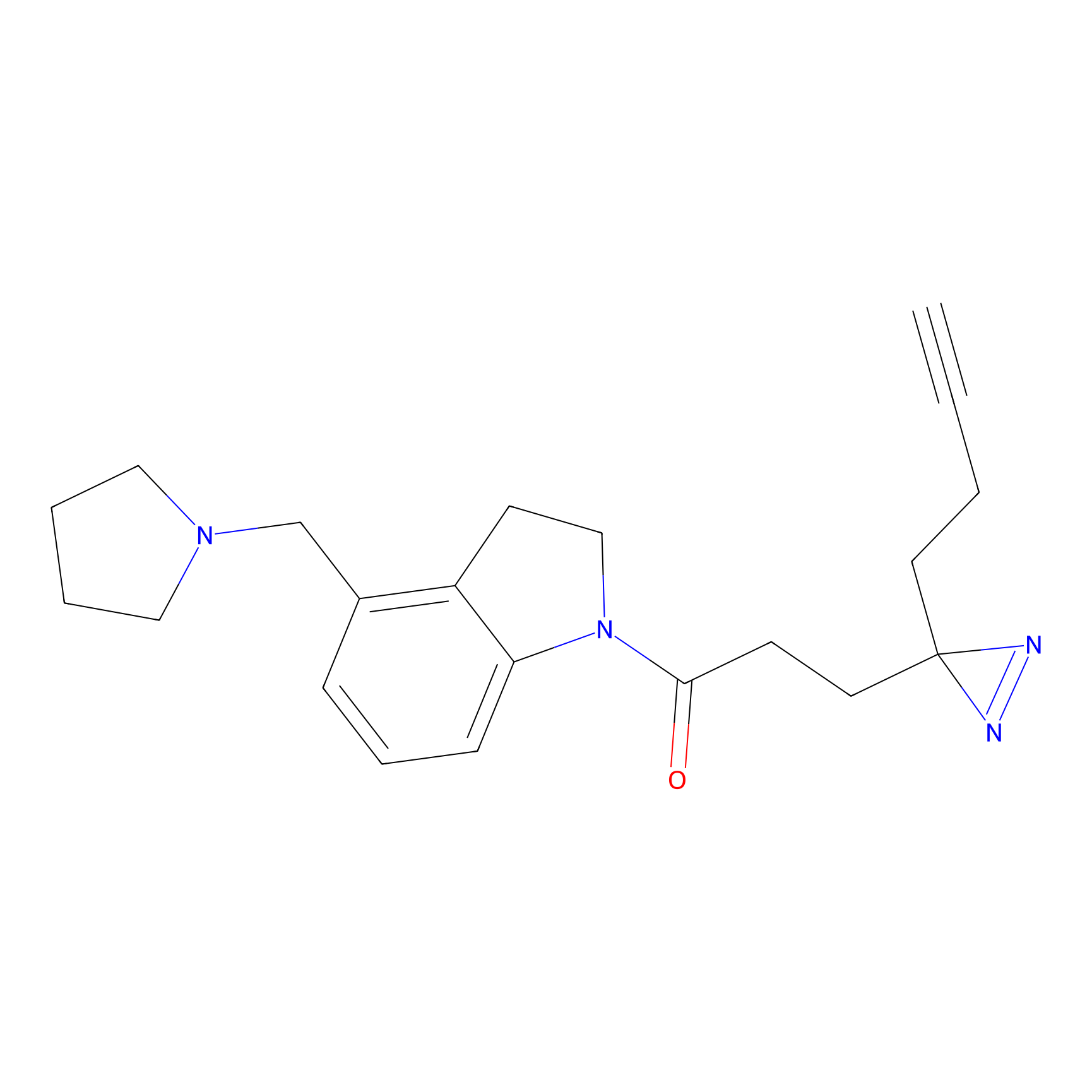
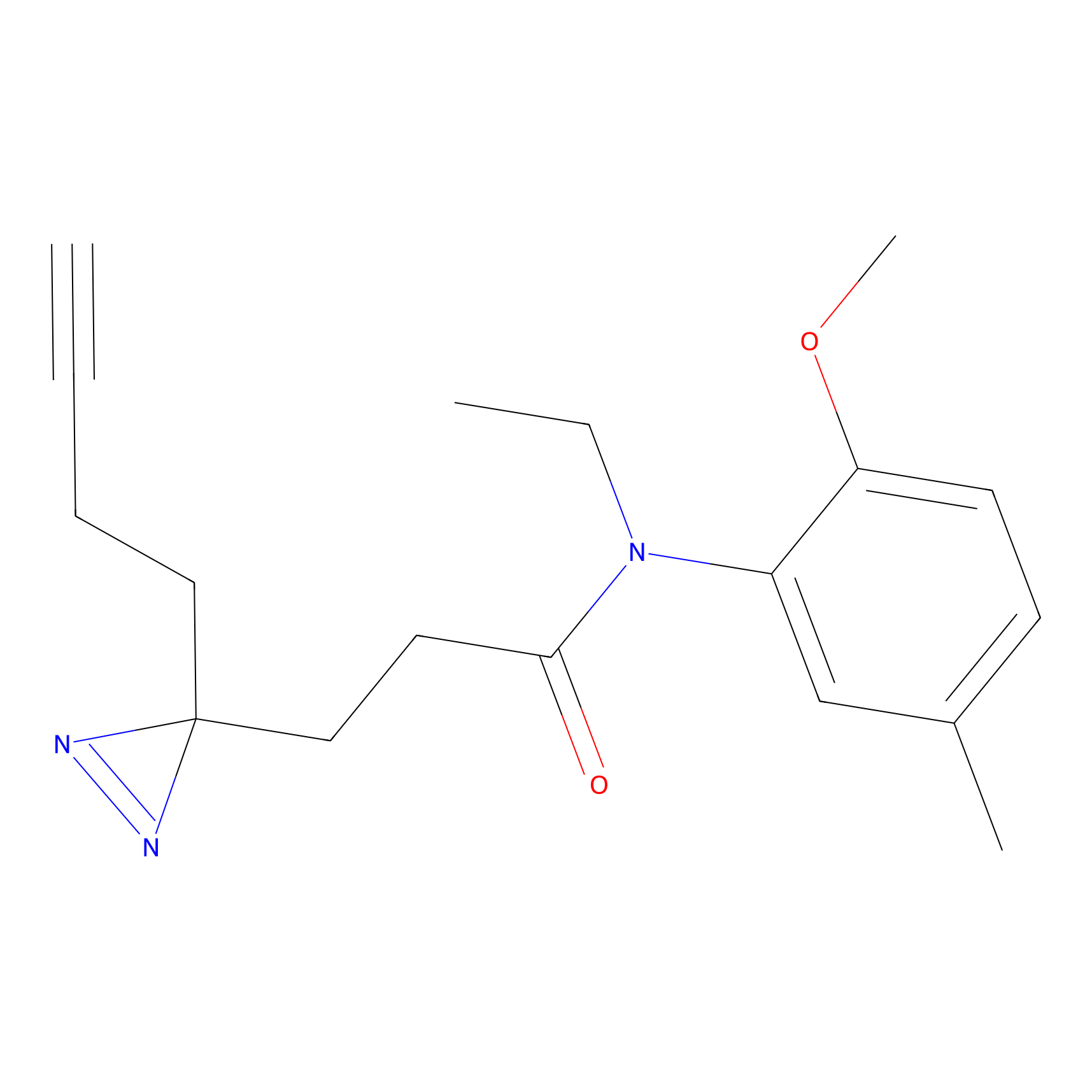
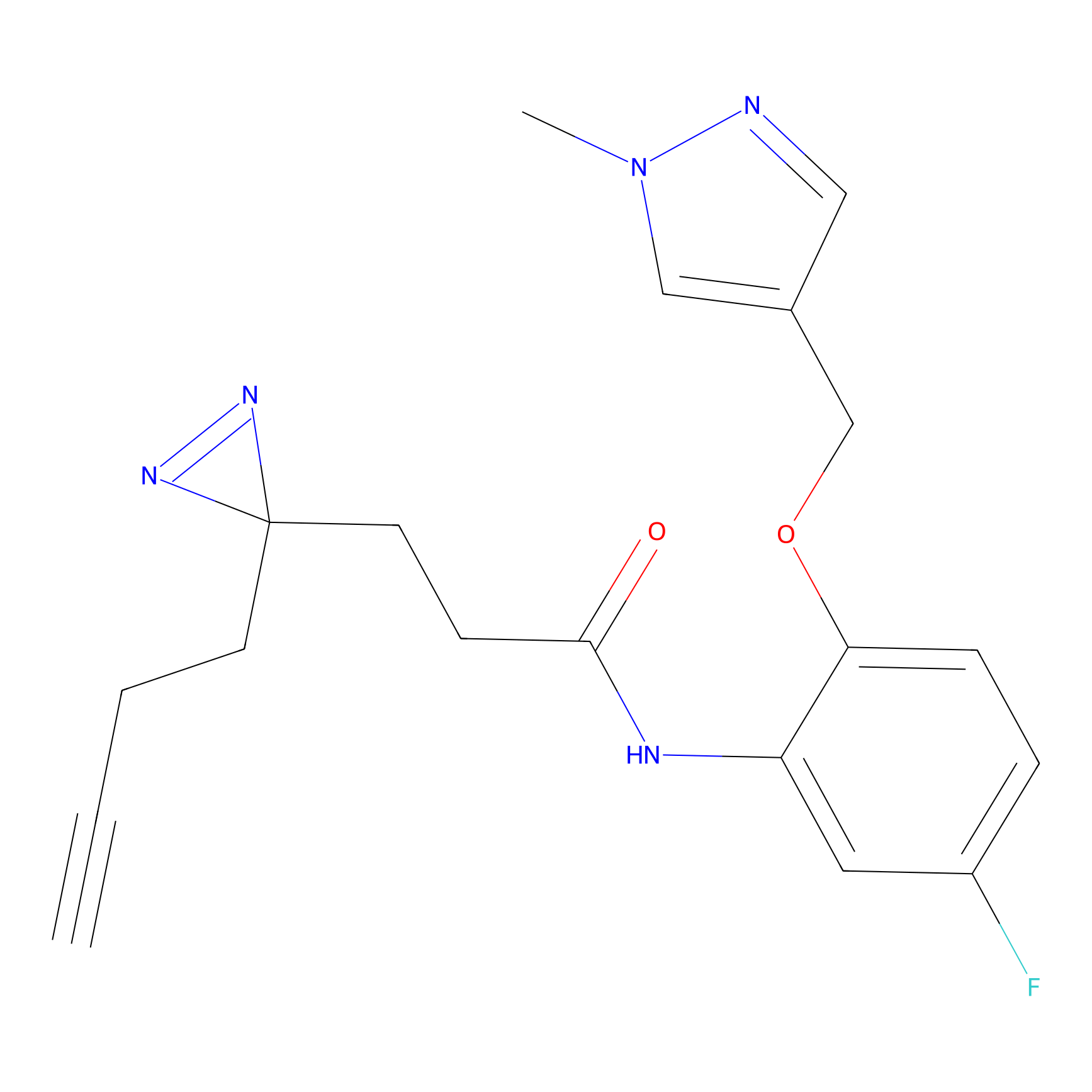
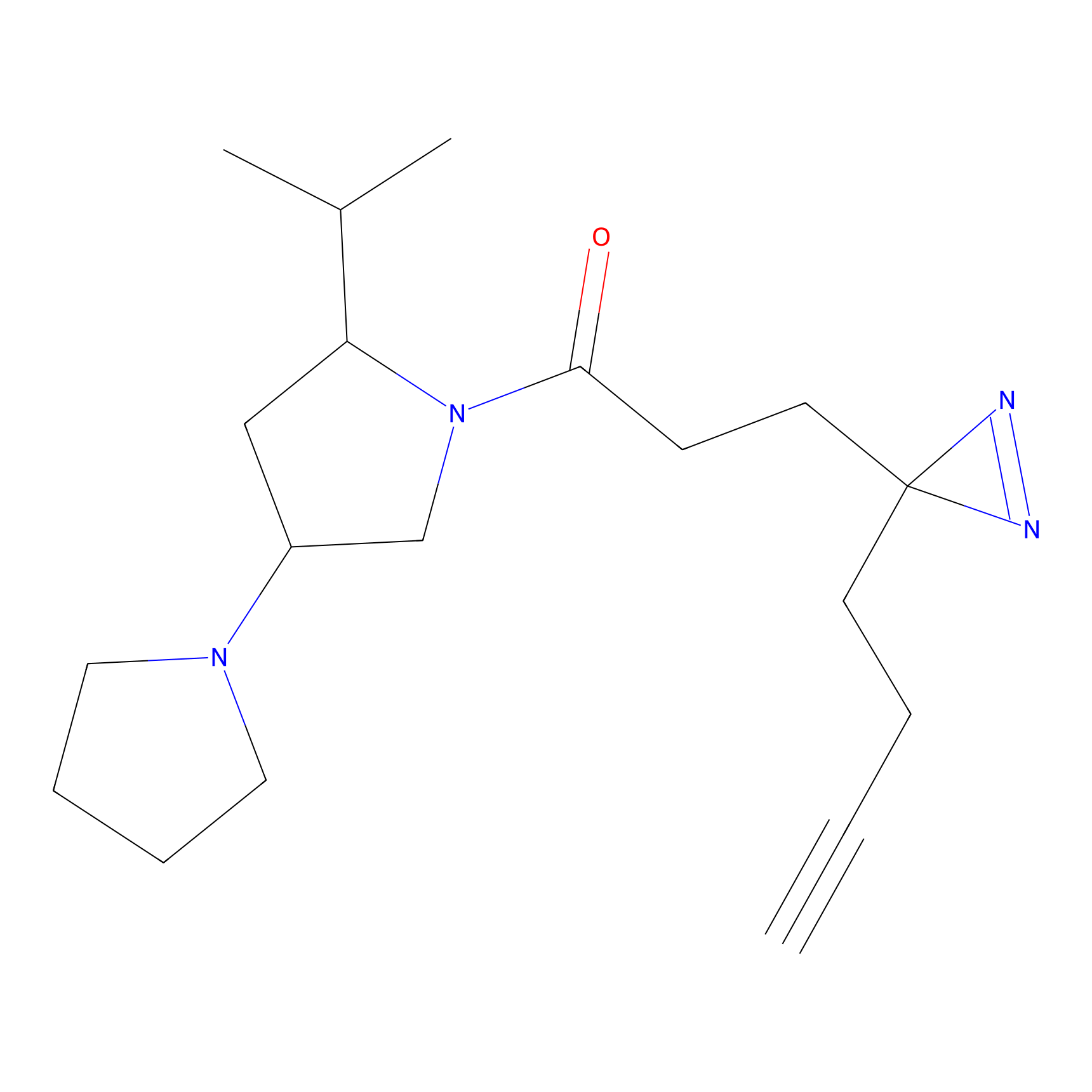
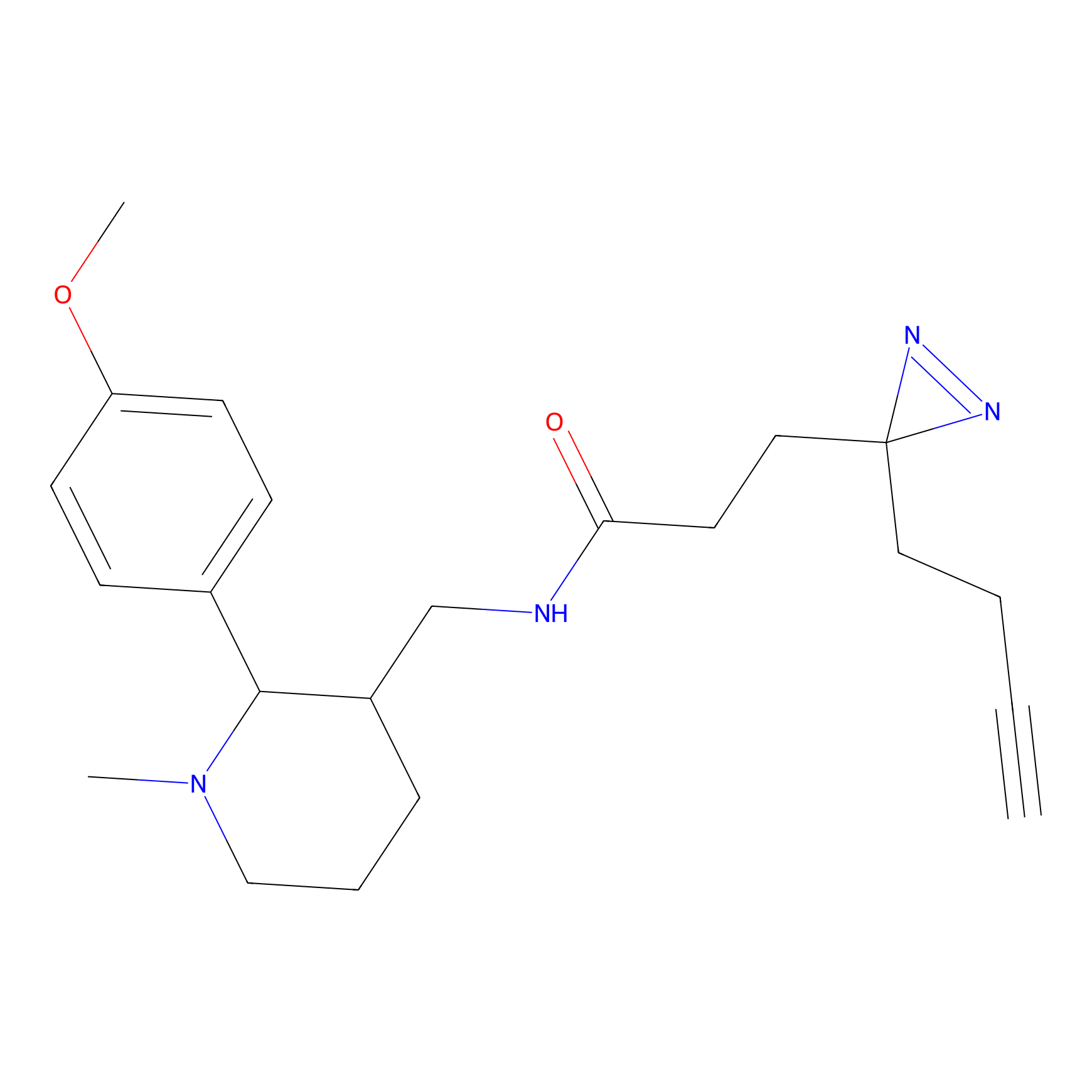
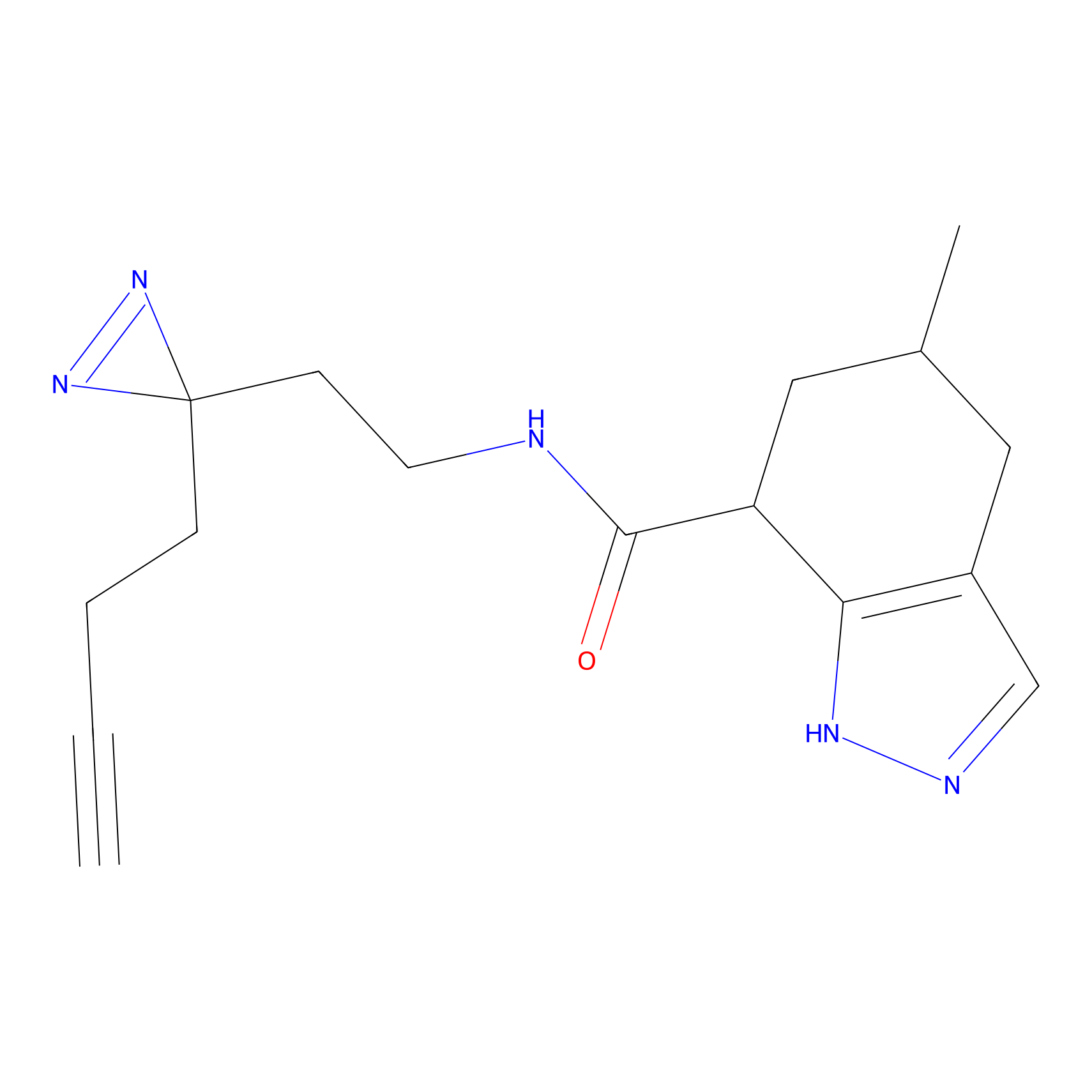
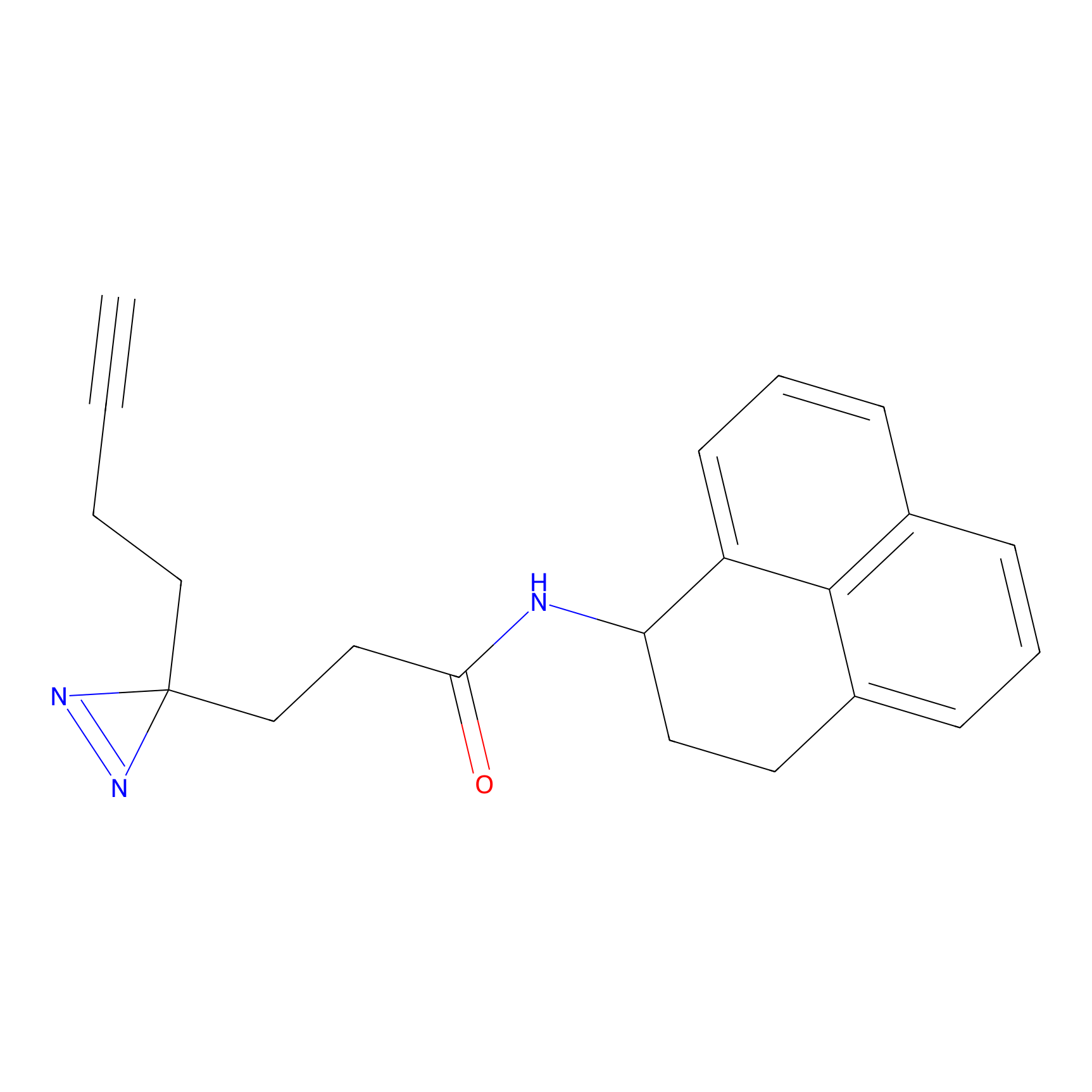
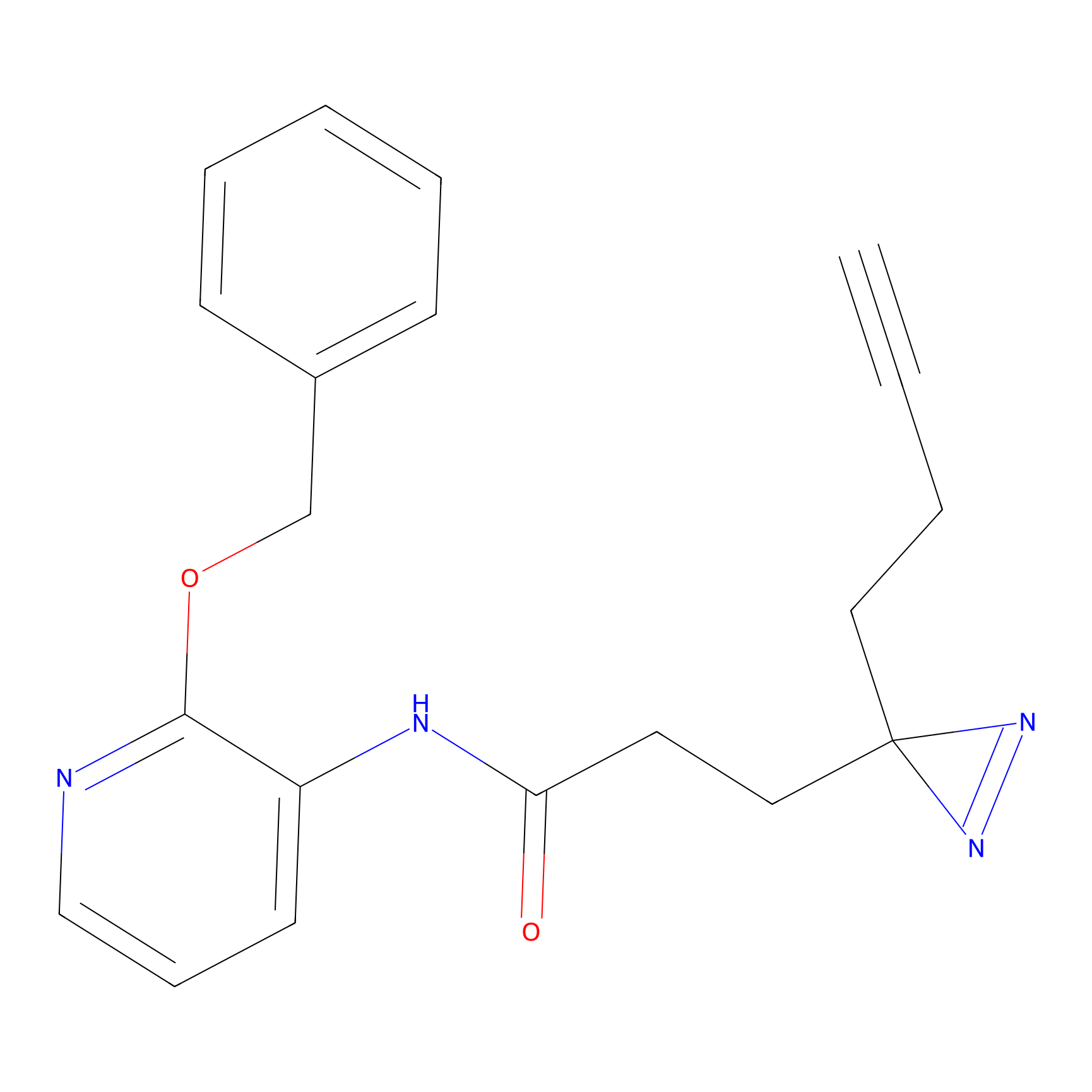
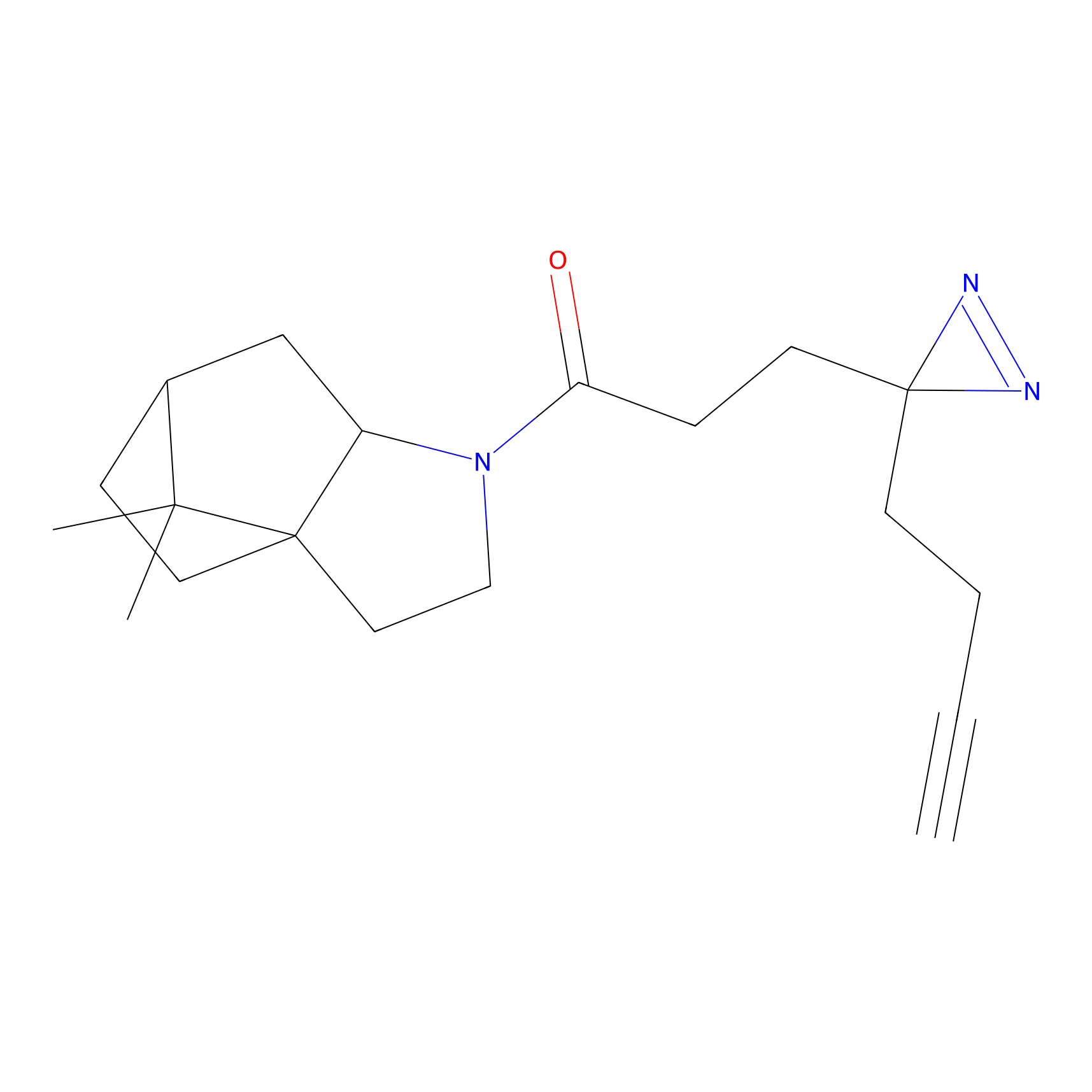
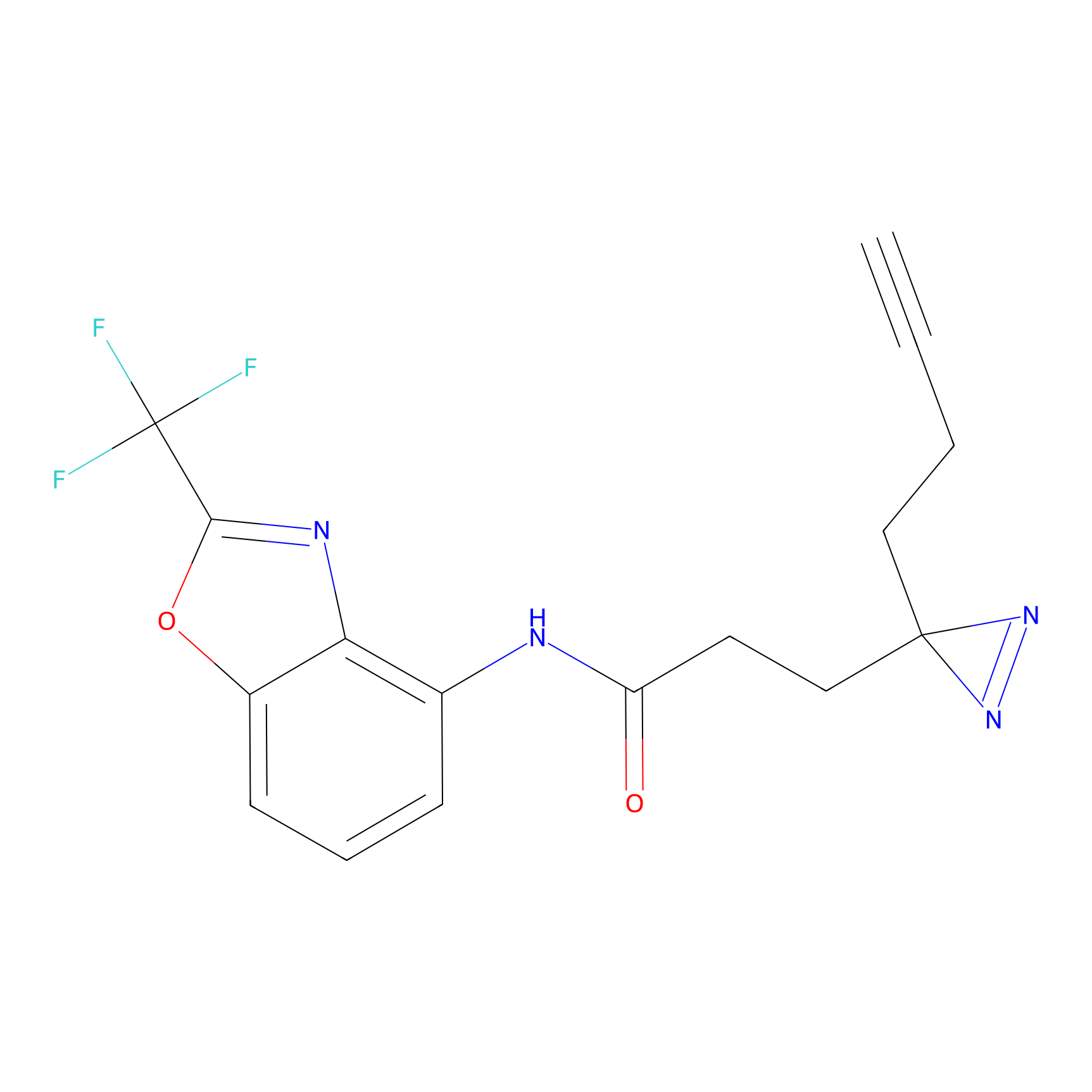
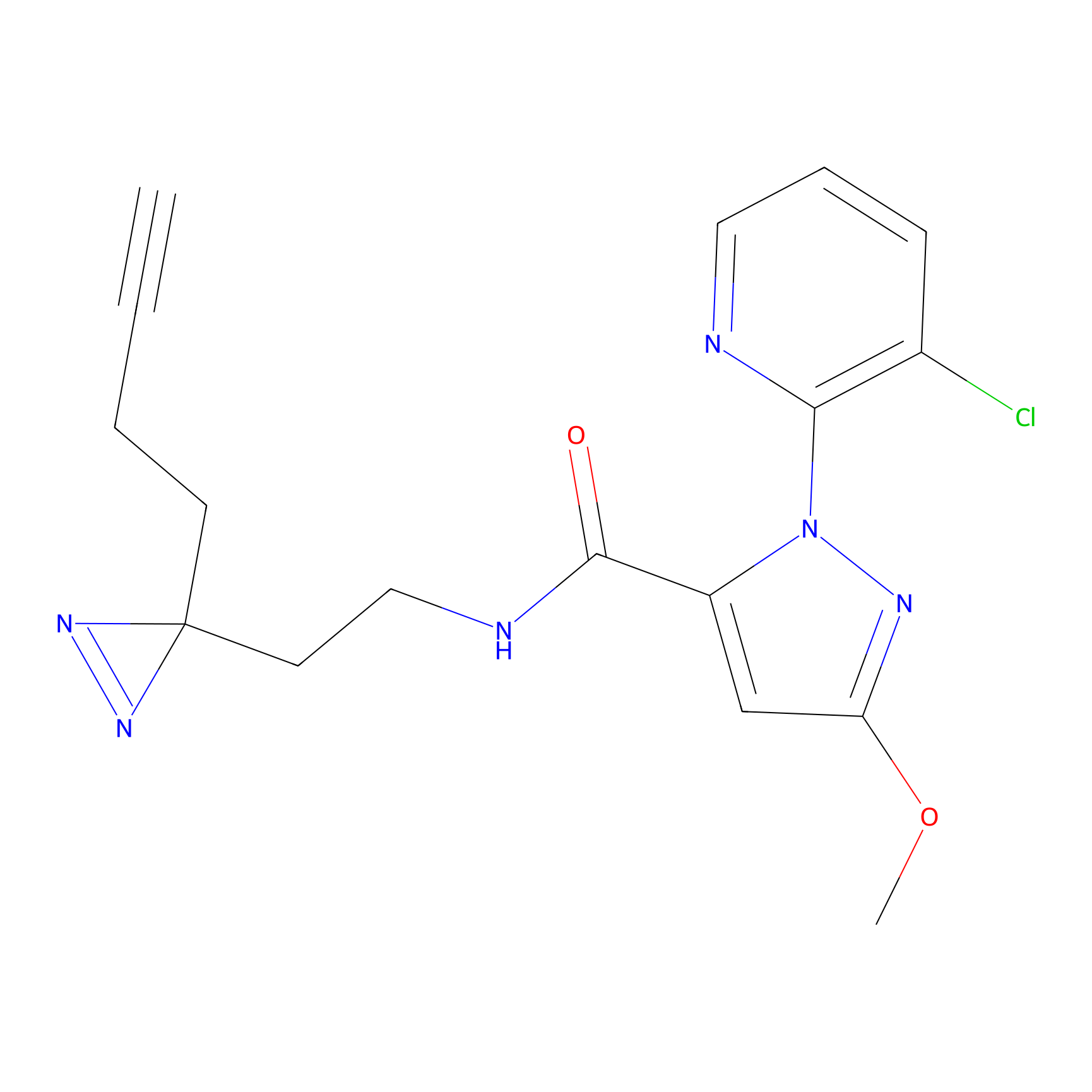
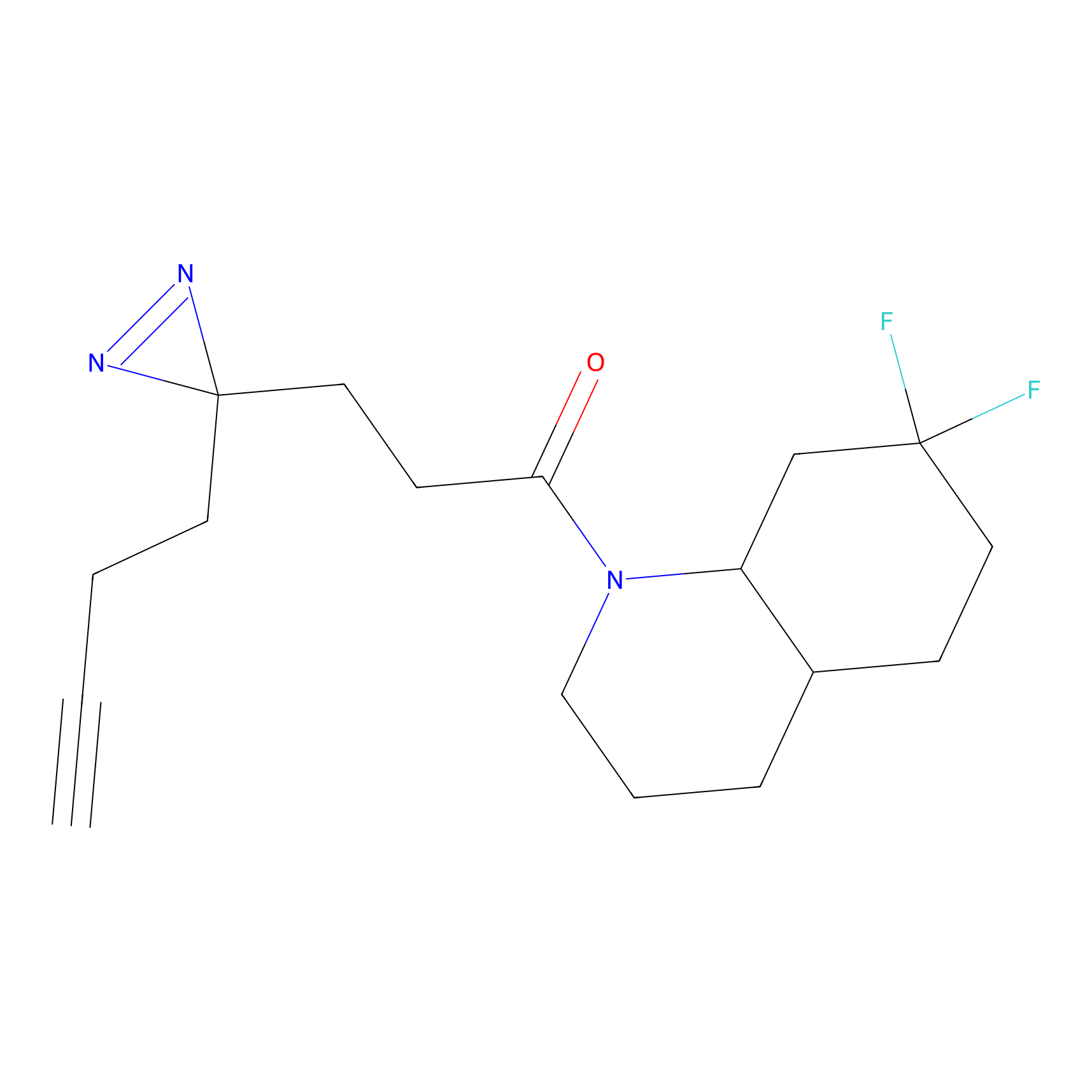
|
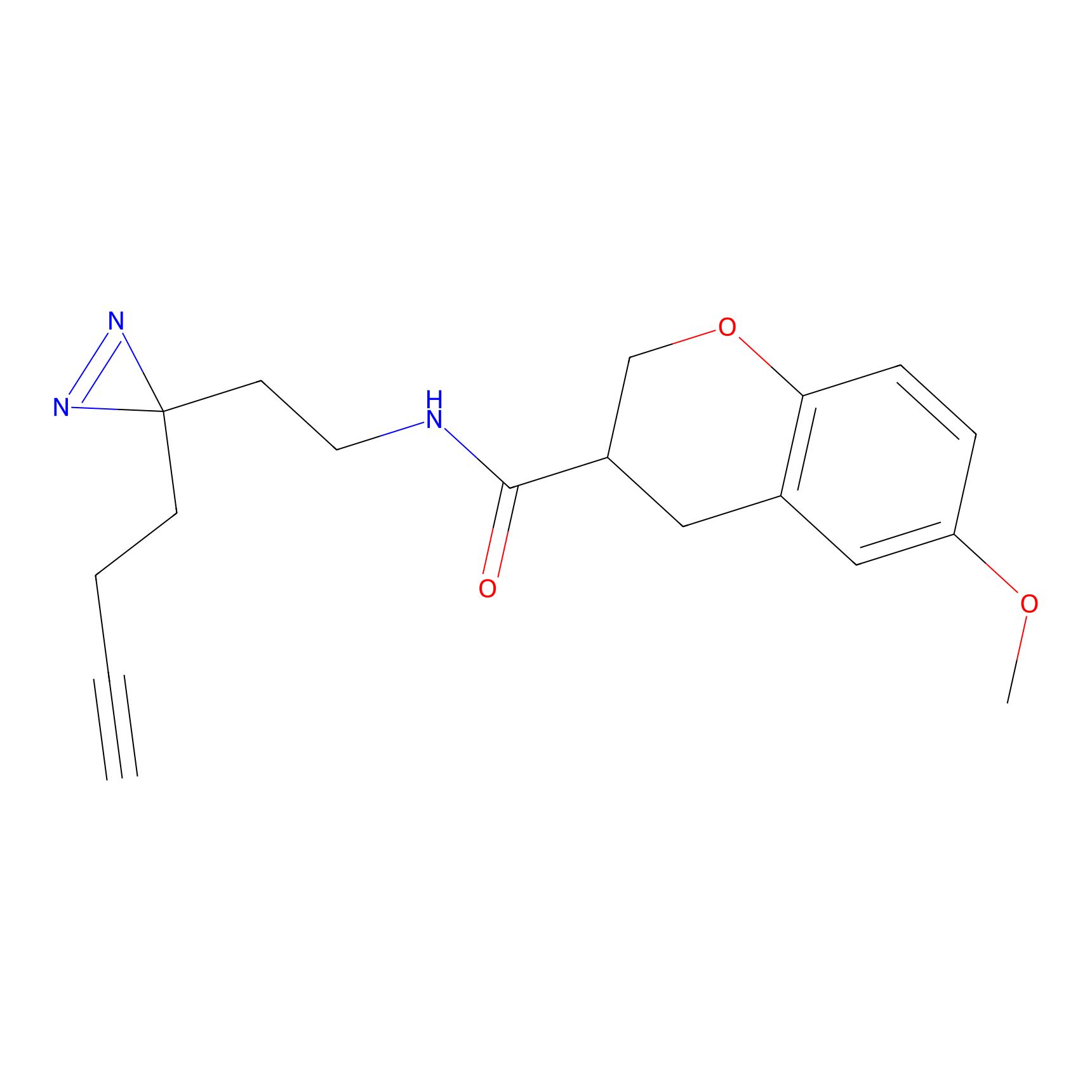
OPA-S-S-alkyne Probe Info |
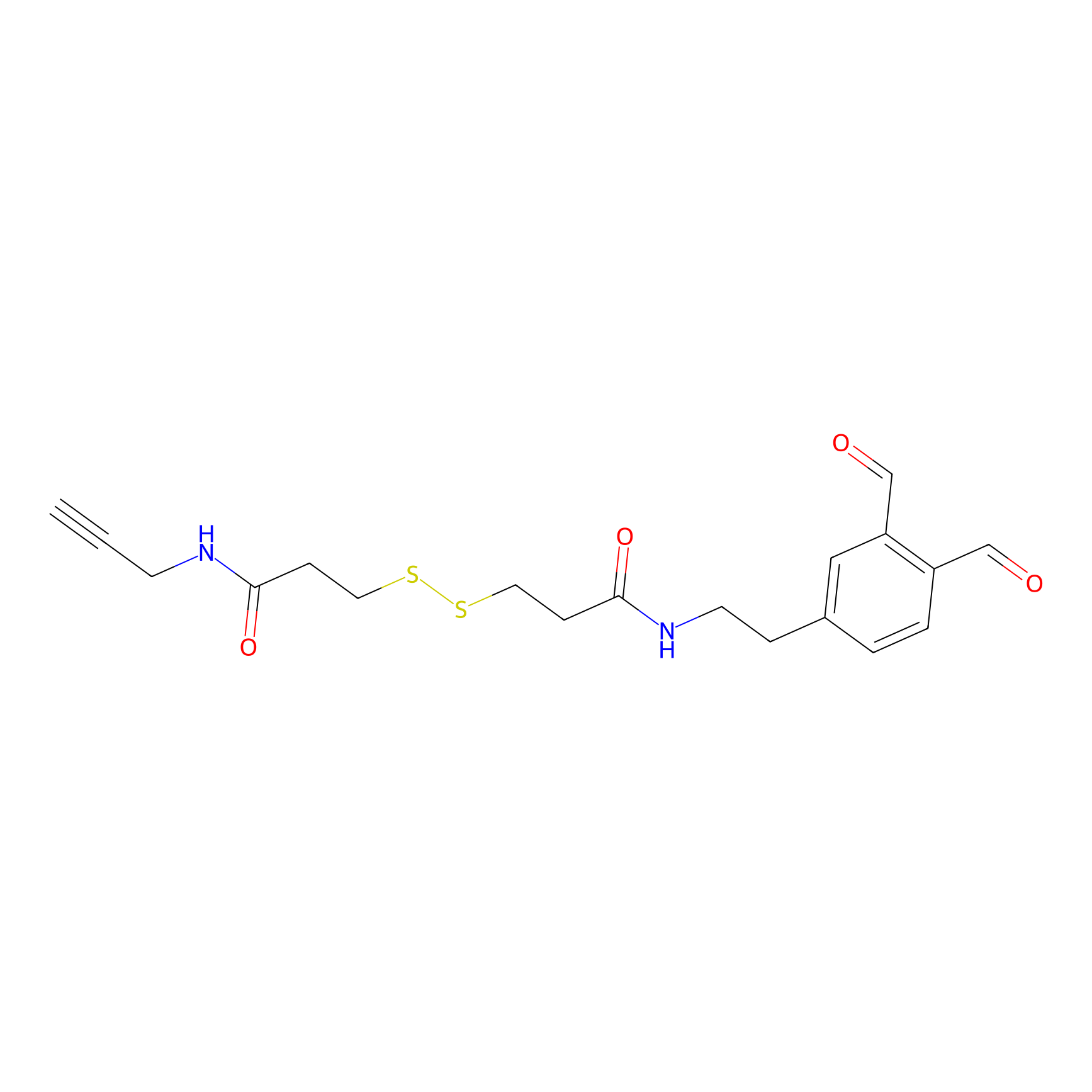 |
K110(4.07) | LDD3494 | [4] | |
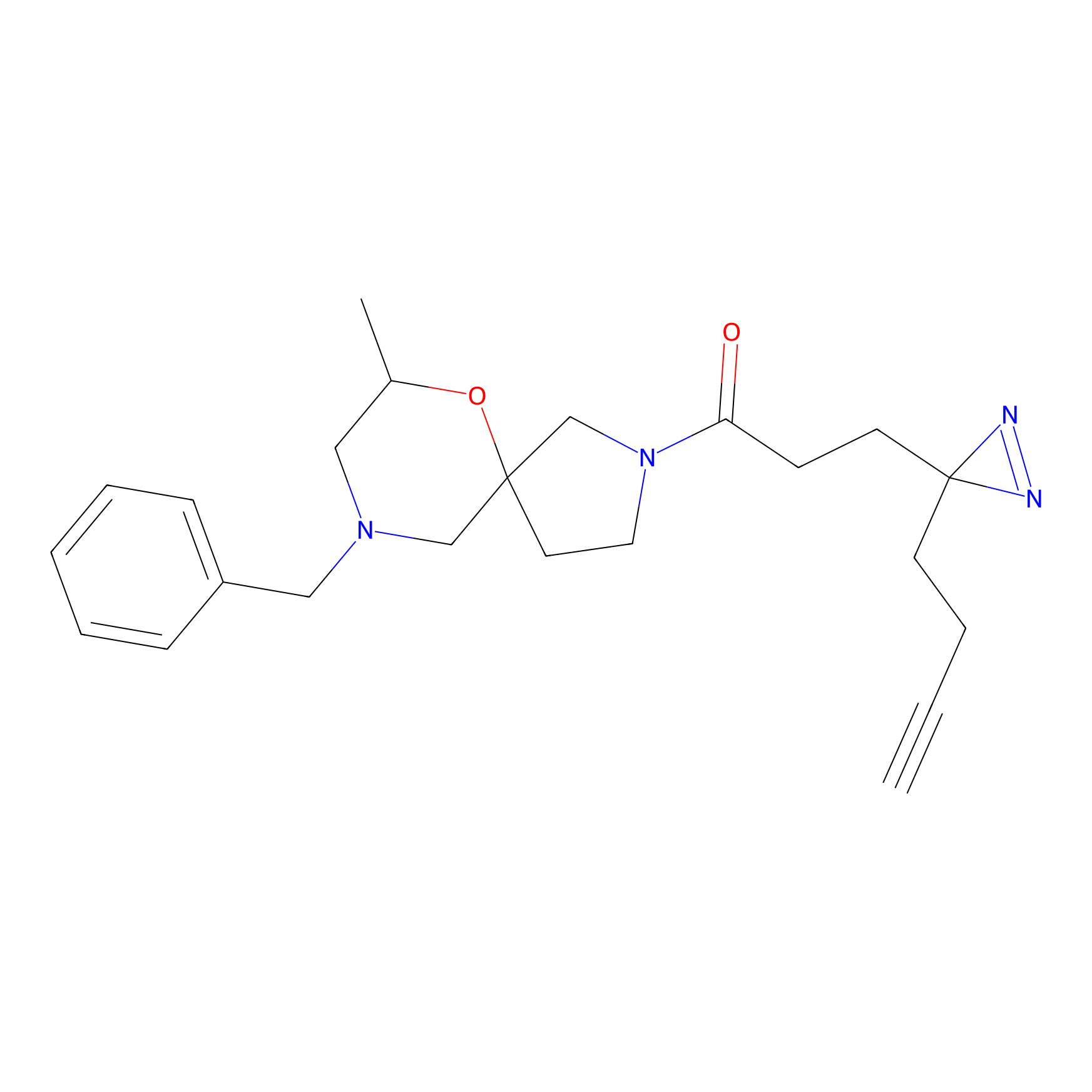
|
BTD Probe Info |
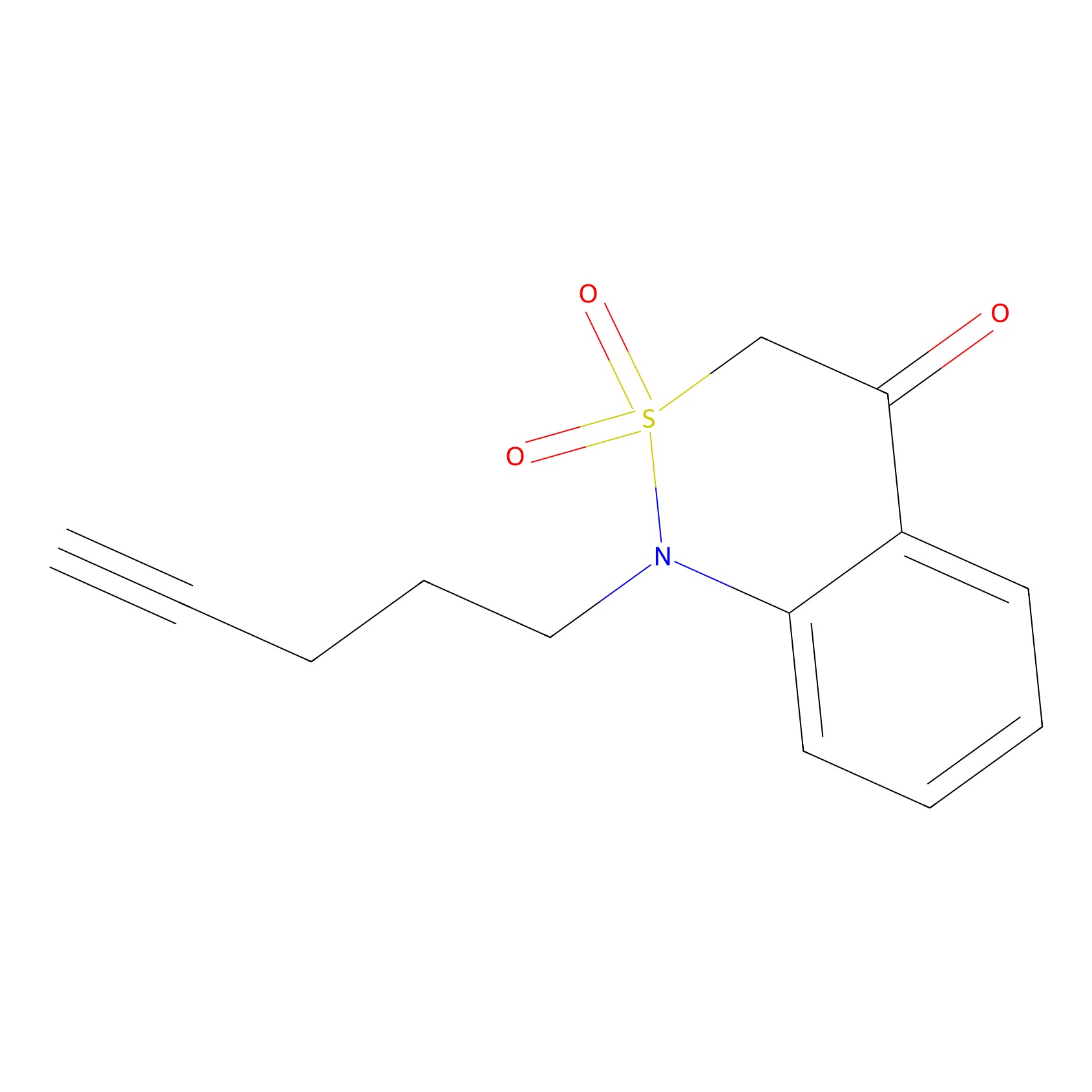 |
C4(1.71) | LDD2108 | [5] | |
|
IA-alkyne Probe Info |
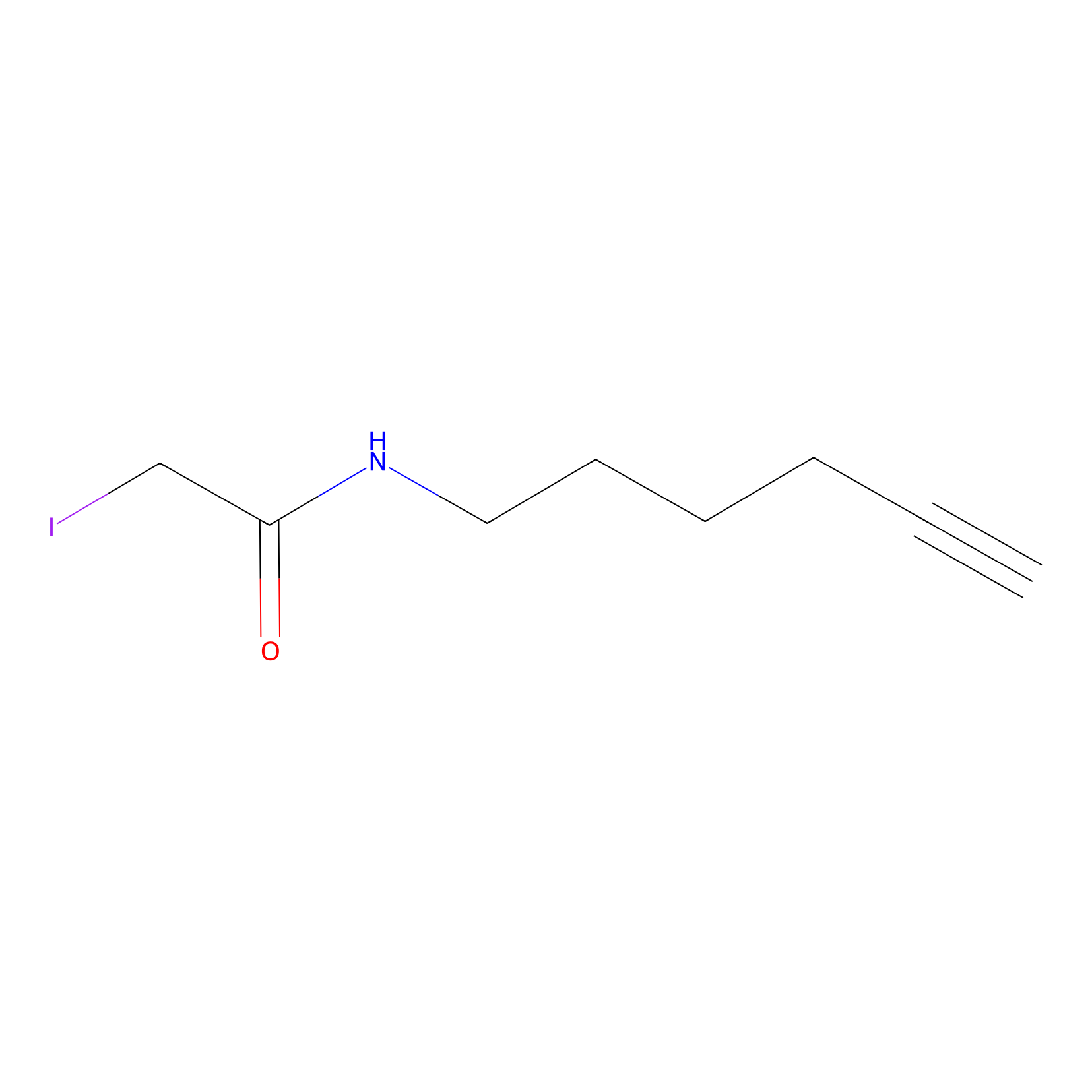 |
C618(0.00); C533(0.00) | LDD0161 | [6] | |
|
WYneN Probe Info |
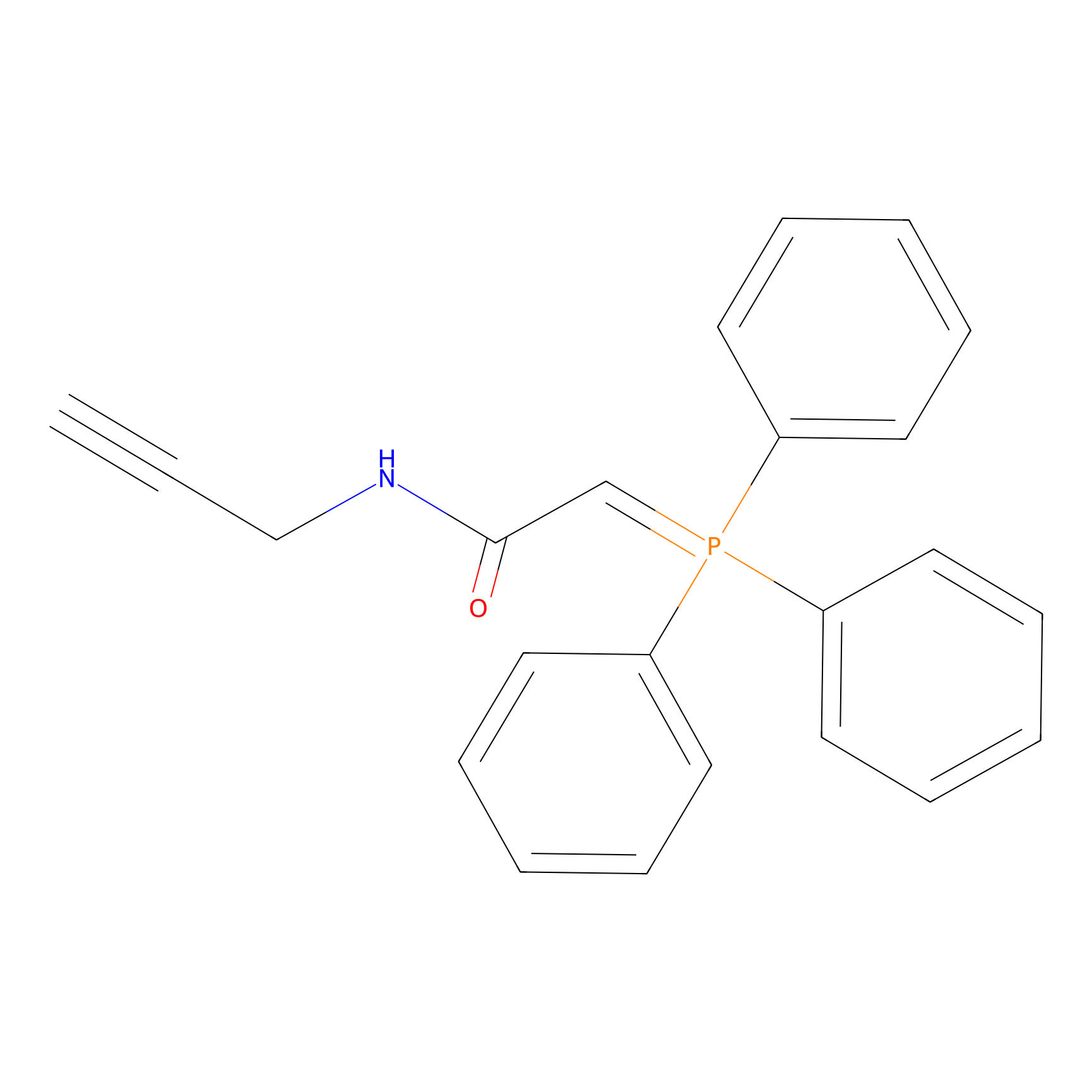 |
N.A. | LDD0021 | [7] | |
|
IPM Probe Info |
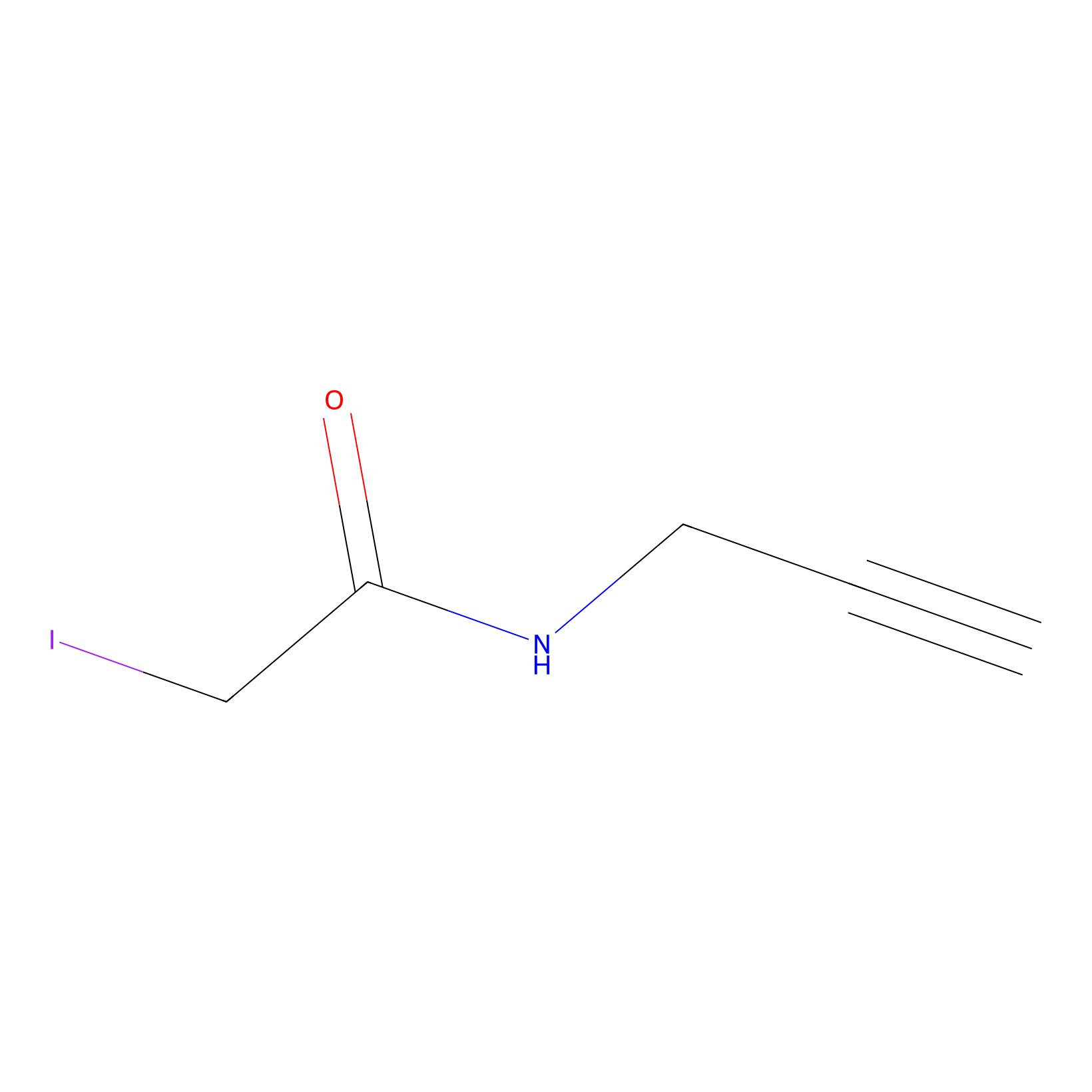 |
N.A. | LDD0005 | [7] | |
PAL-AfBPP Probe
Competitor(s) Related to This Target
References