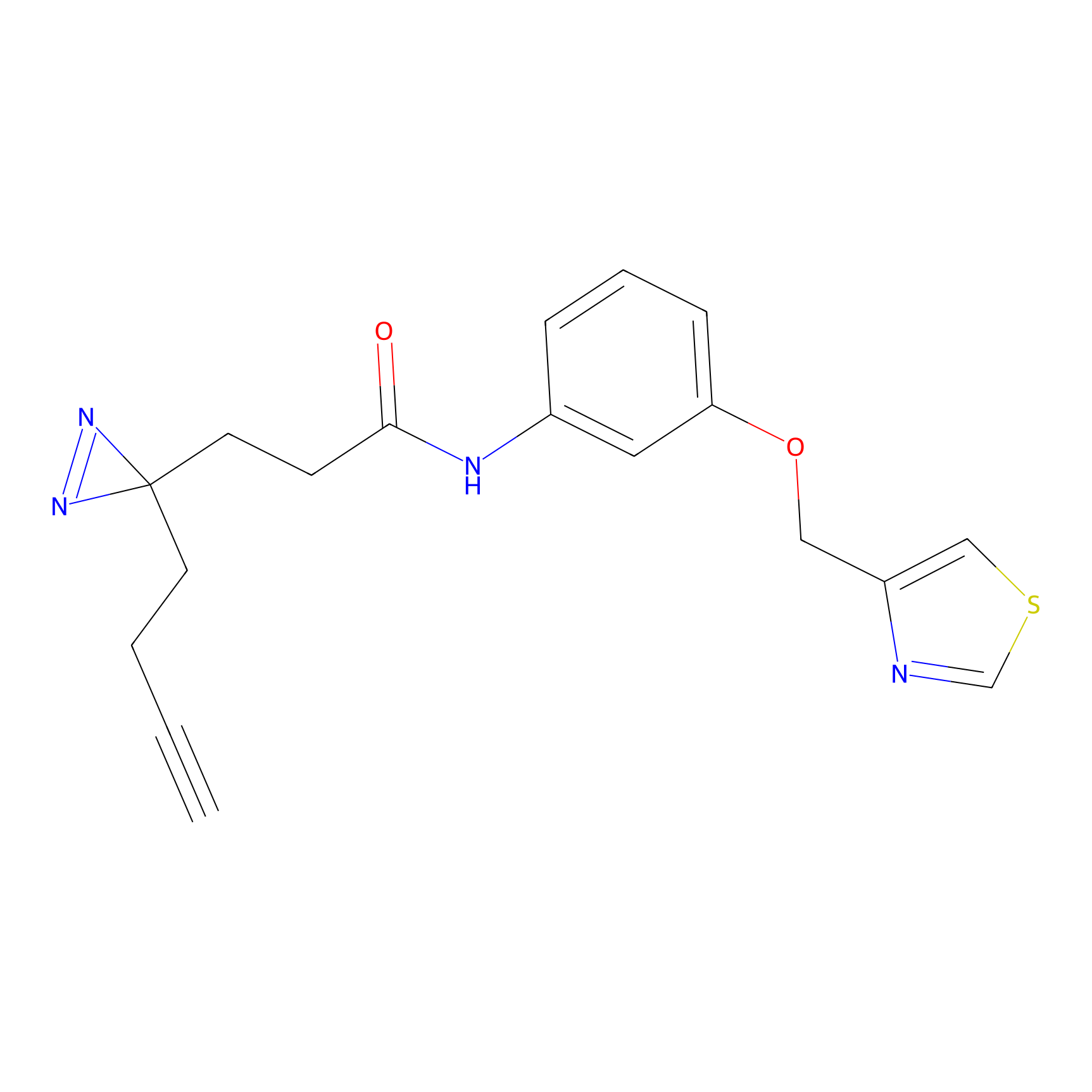
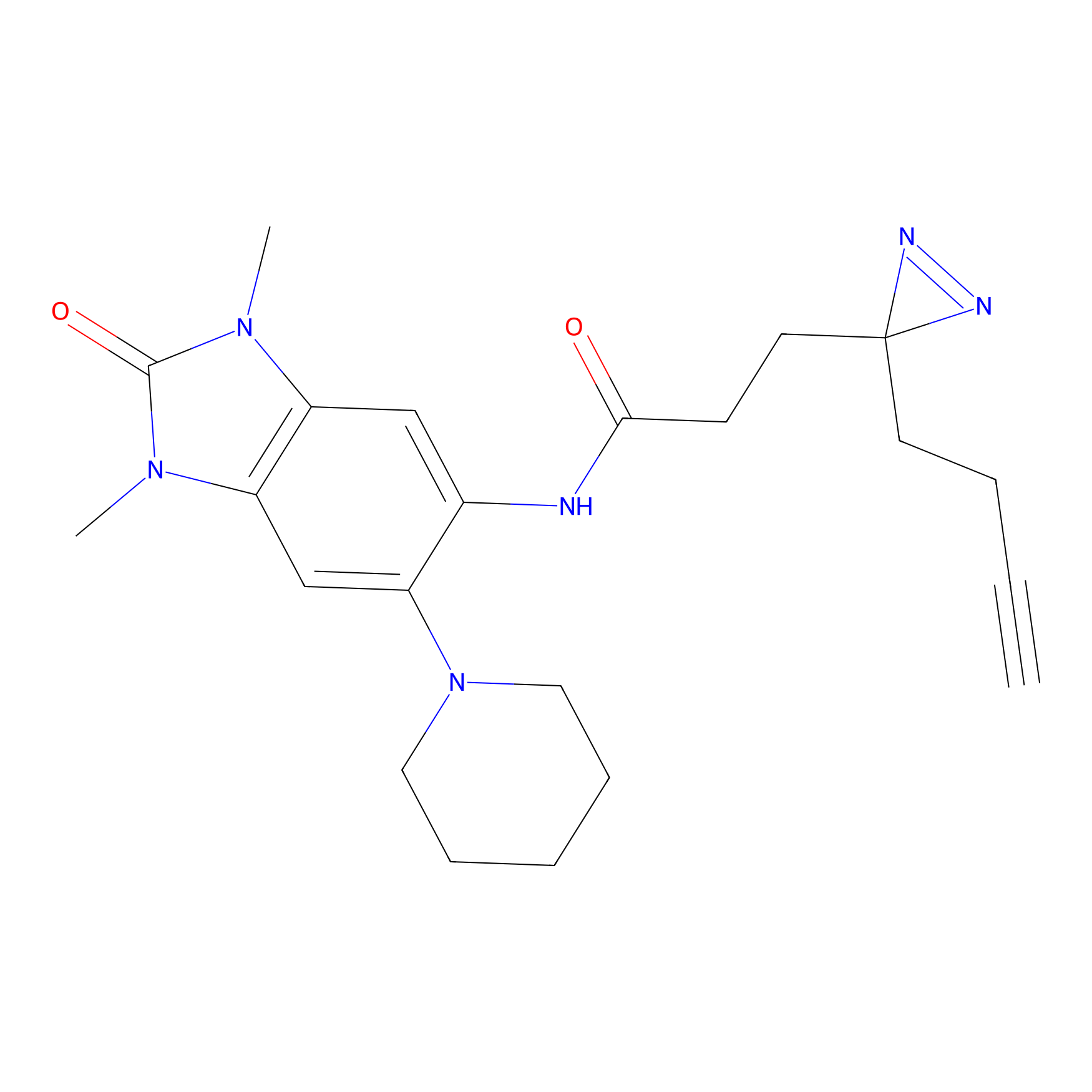
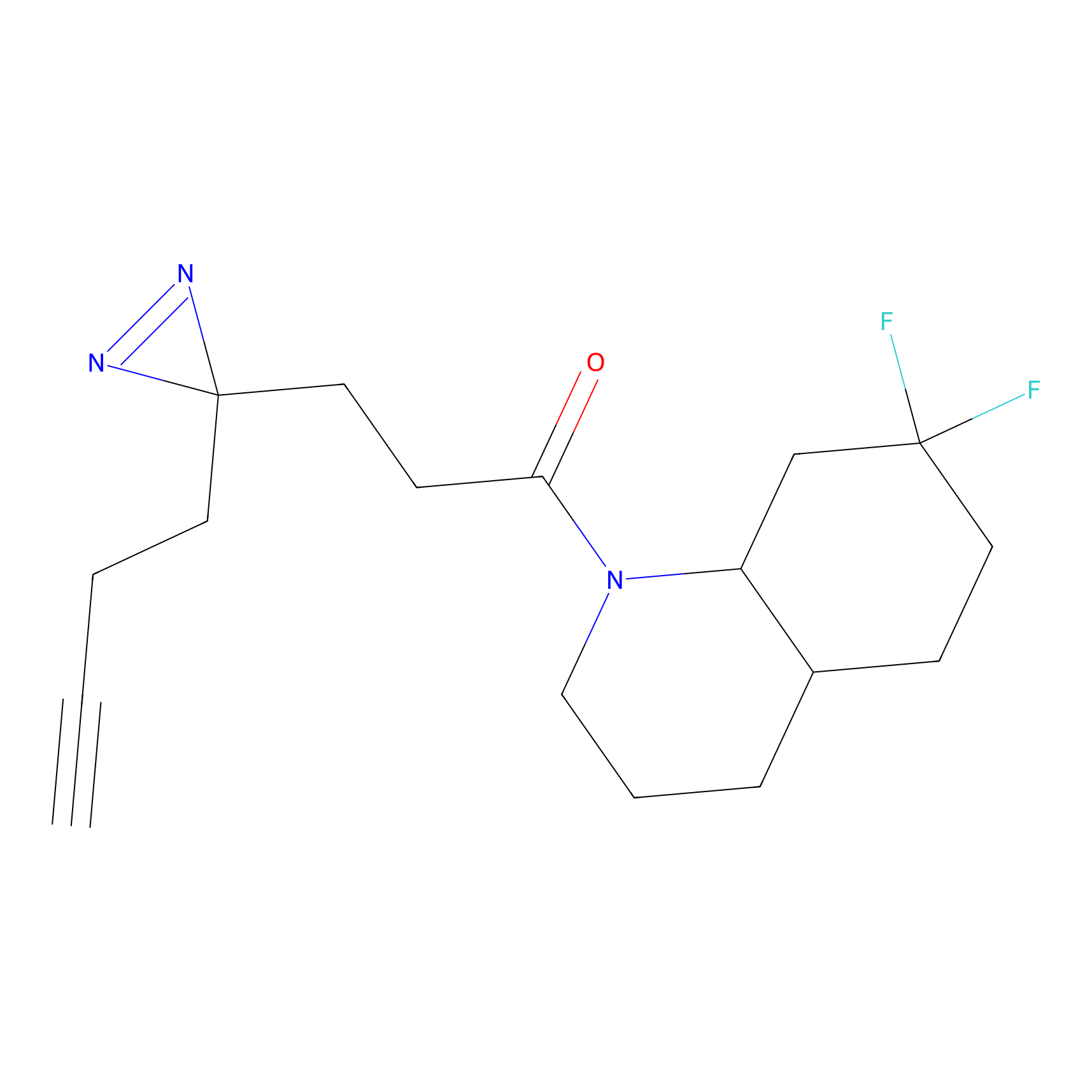
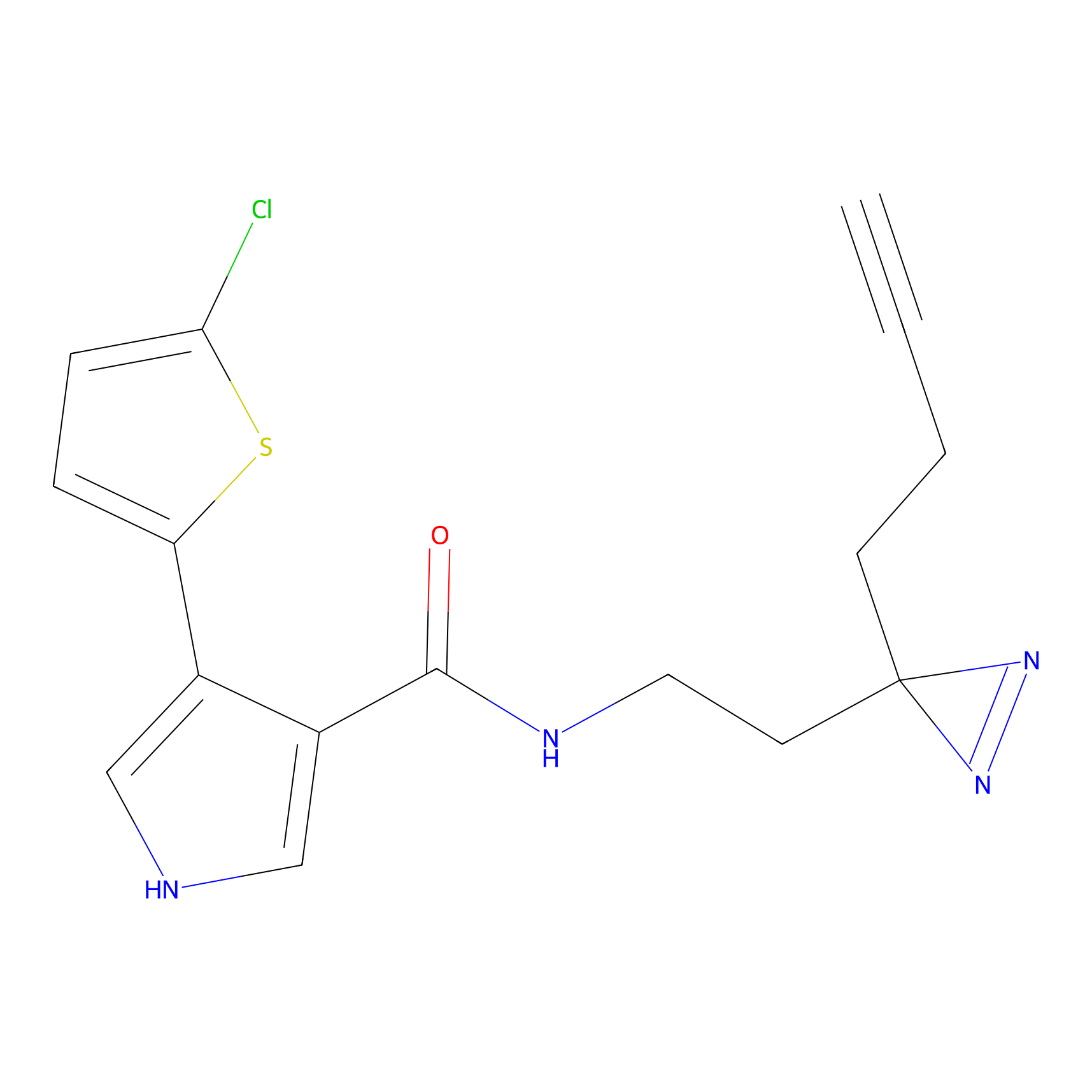
Details of the Target
General Information of Target
| Target ID | LDTP18883 | |||||
|---|---|---|---|---|---|---|
| Target Name | Protein CEBPZOS (CEBPZOS) | |||||
| Gene Name | CEBPZOS | |||||
| Gene ID | 100505876 | |||||
| Synonyms |
CEBPZ-AS1; Protein CEBPZOS; CEBPZ antisense RNA 1; CEBPZ opposite strand |
|||||
| 3D Structure | ||||||
| Sequence |
MDWFHCNQCFRKDGAHFFVTSCGHIFCKKCVTLEKCAVCGTACKHLALSDNLKPQEKMFF
KSPVETALQYFSHISQVWSFQKKQTDLLIAFYKHRITKLETAMQEAQQALVSQDKELSVL RKENGELKKFLAILKESPSRYQGSRSITPRPVGITSPSQSVTPRPSFQHSSQVVSRSSSA ESIPYREAGFGSLGQGGRGLQGRRTPRDSYNETPSPASTHSLSYRTSSASSGQGIFSFRP SPNGHSGHTRVLTPNNFAQRESTTTLESLPSFQLPVLQTLYQQRRHMGLPSGREAWTTSR |
|||||
| Target Bioclass |
Other
|
|||||
| Subcellular location |
Mitochondrion membrane
|
|||||
| Uniprot ID | ||||||
| Ensemble ID | ||||||
| HGNC ID | ||||||
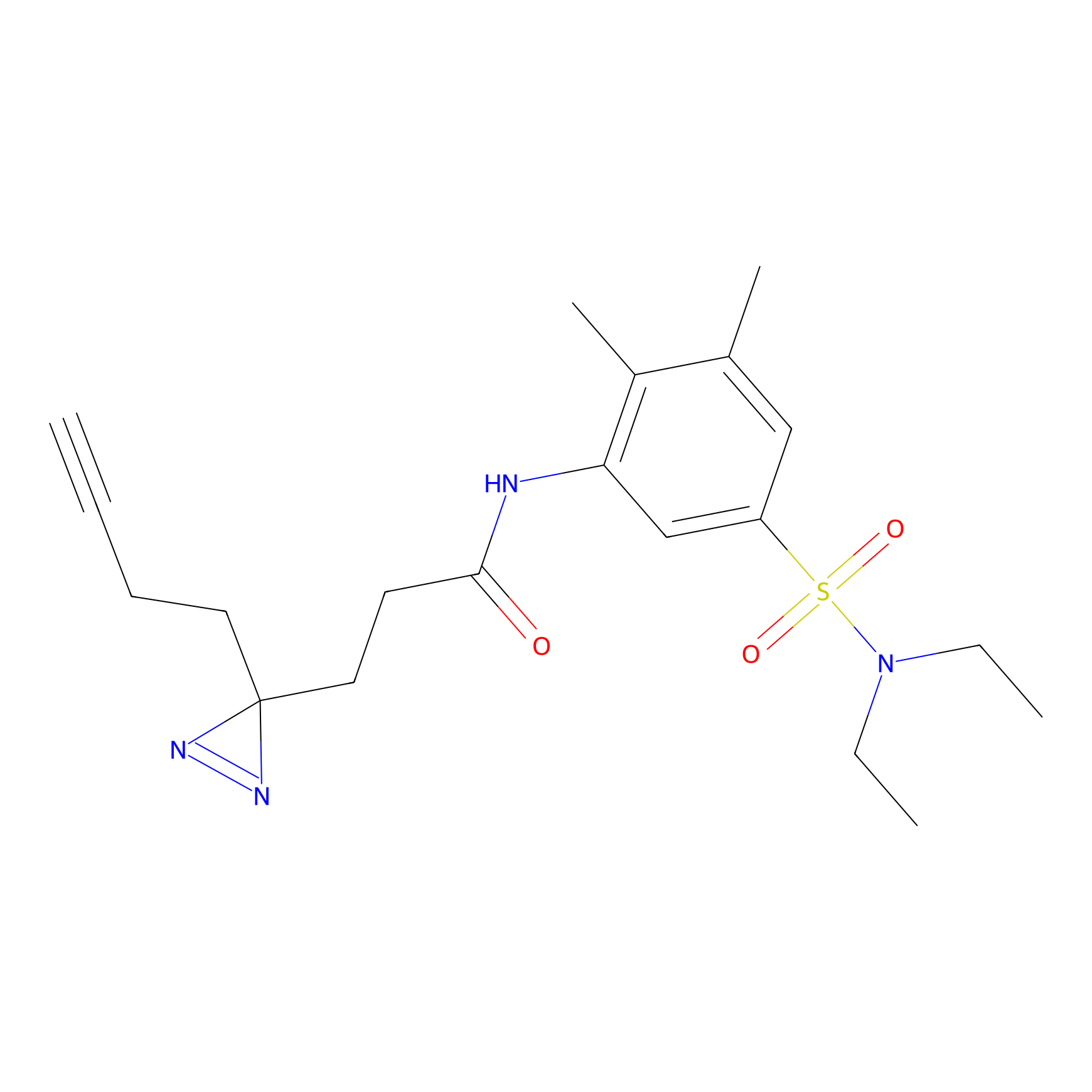
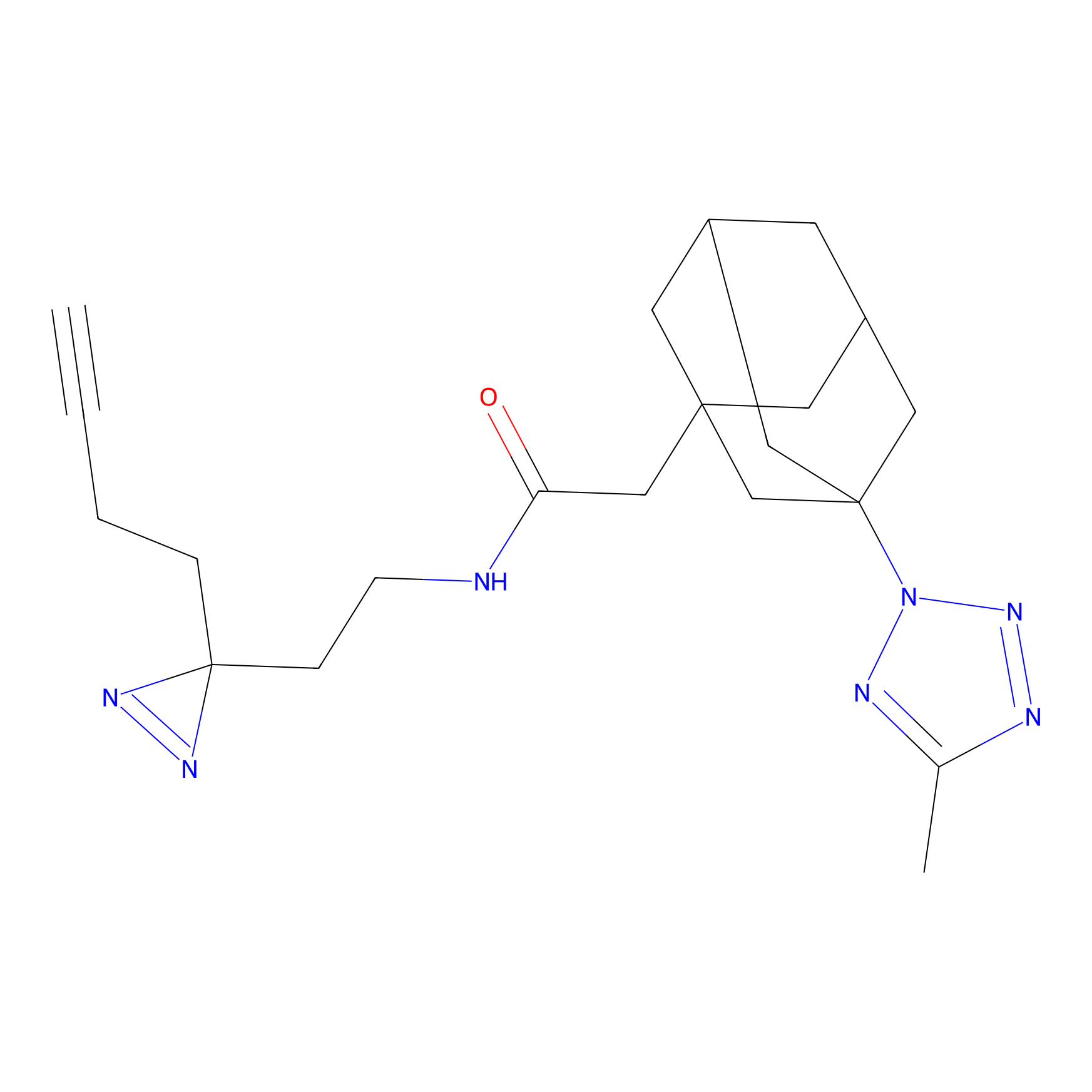
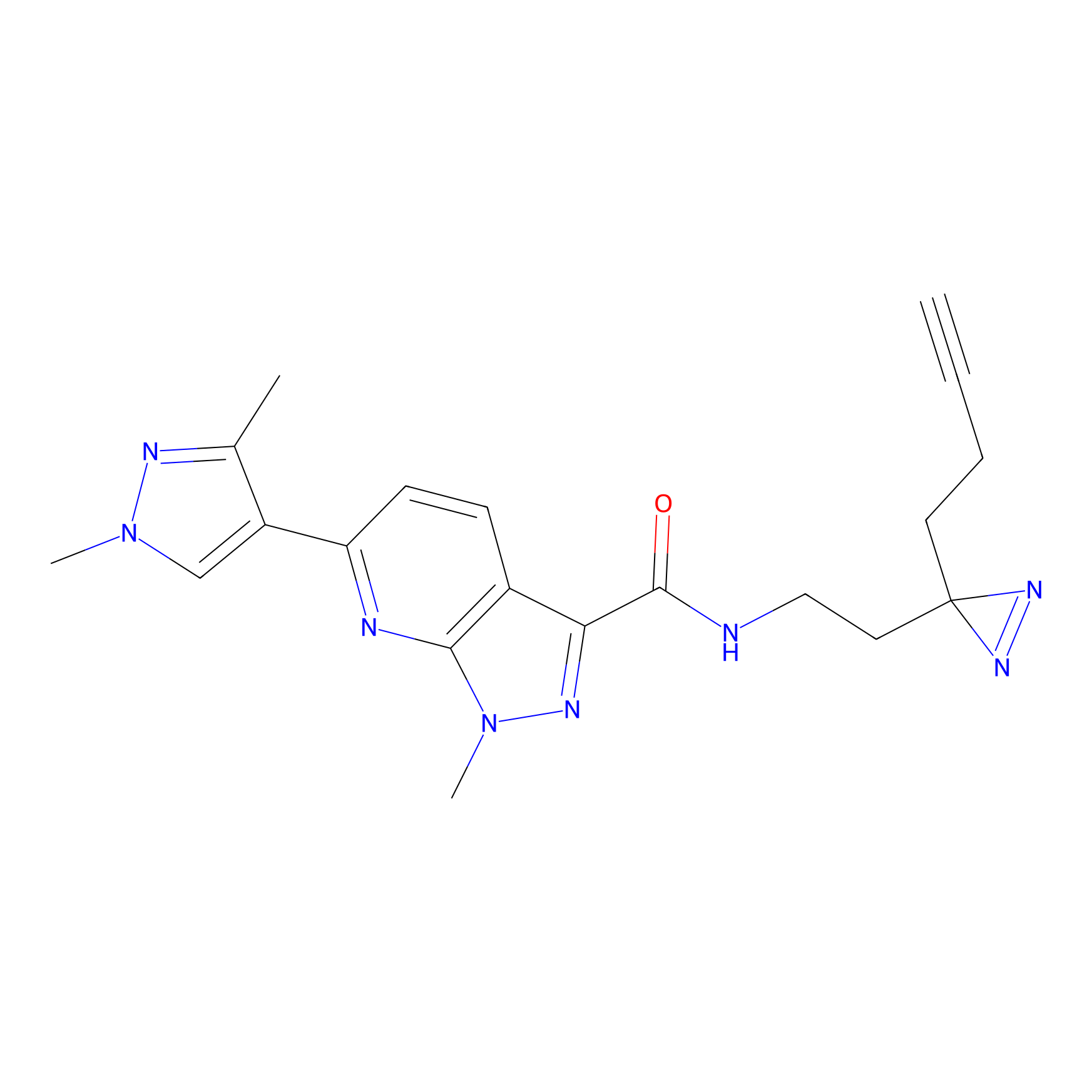
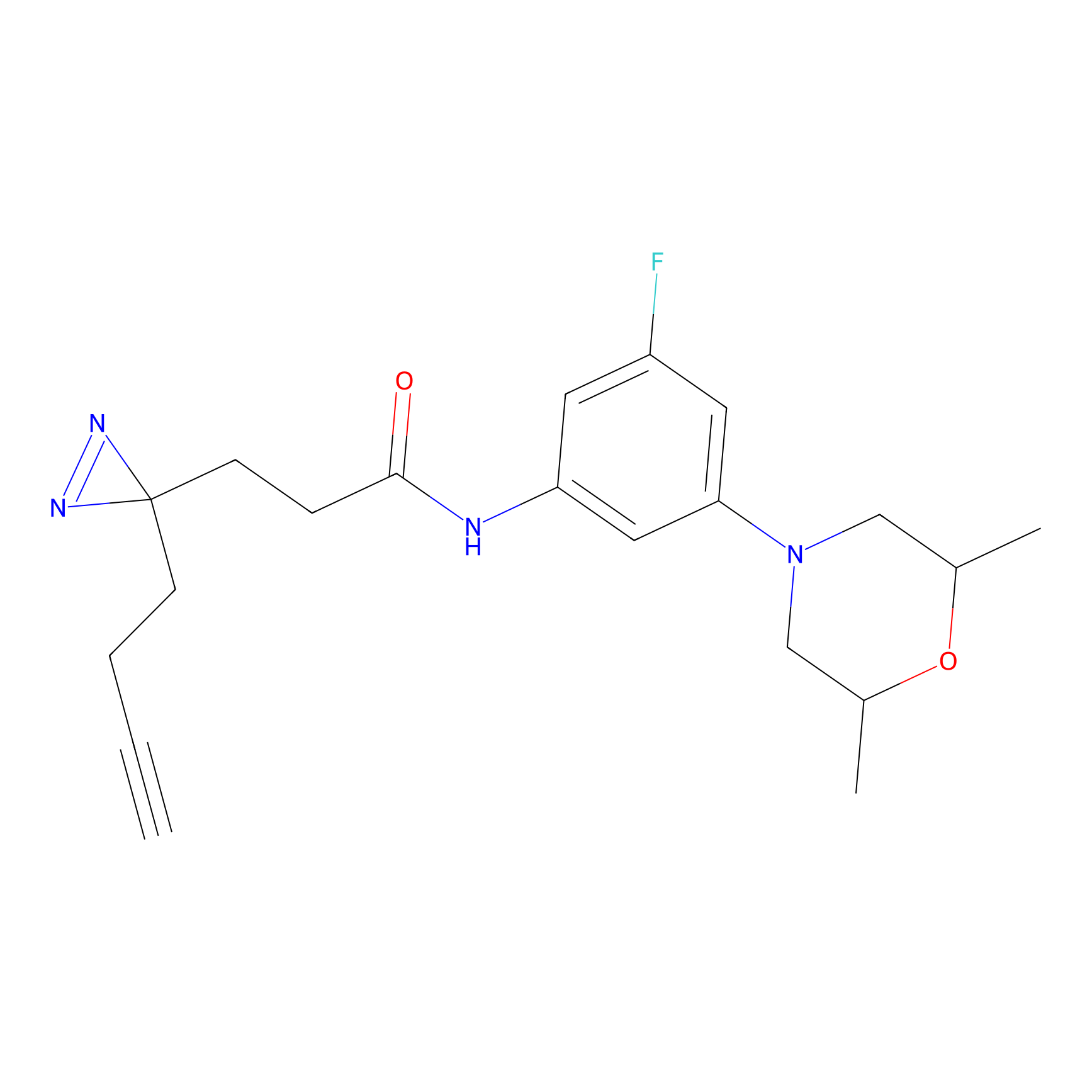
Probe(s) Labeling This Target
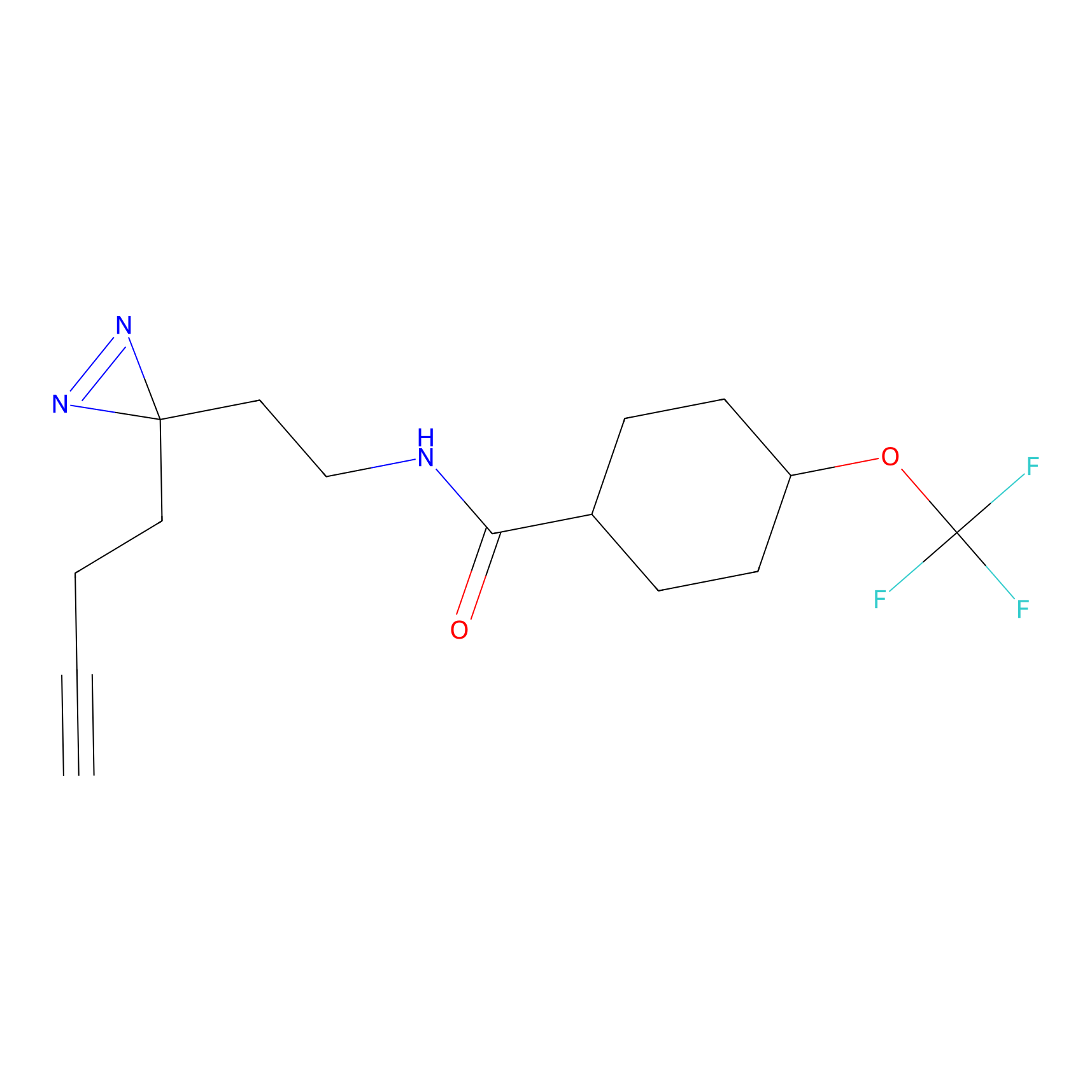
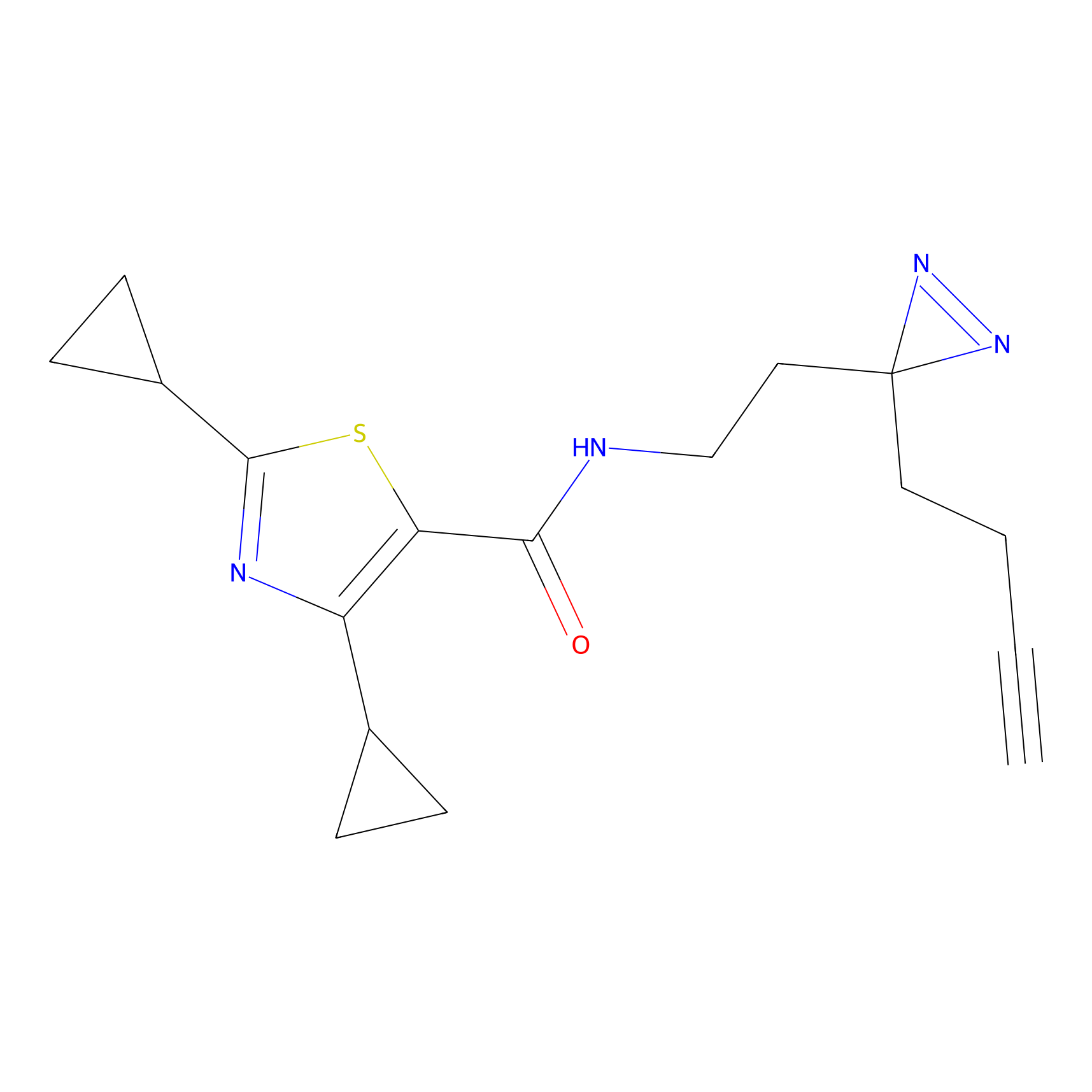
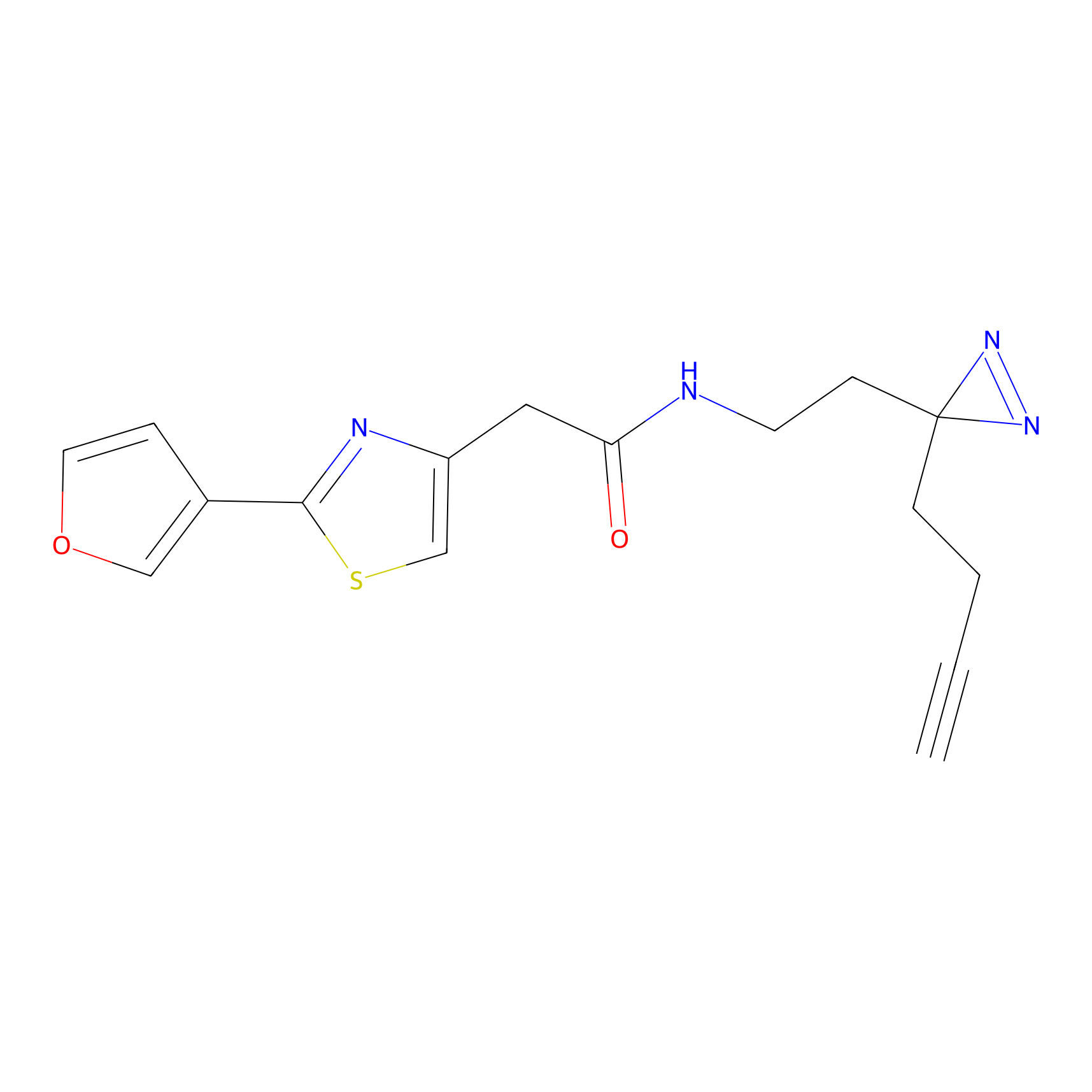
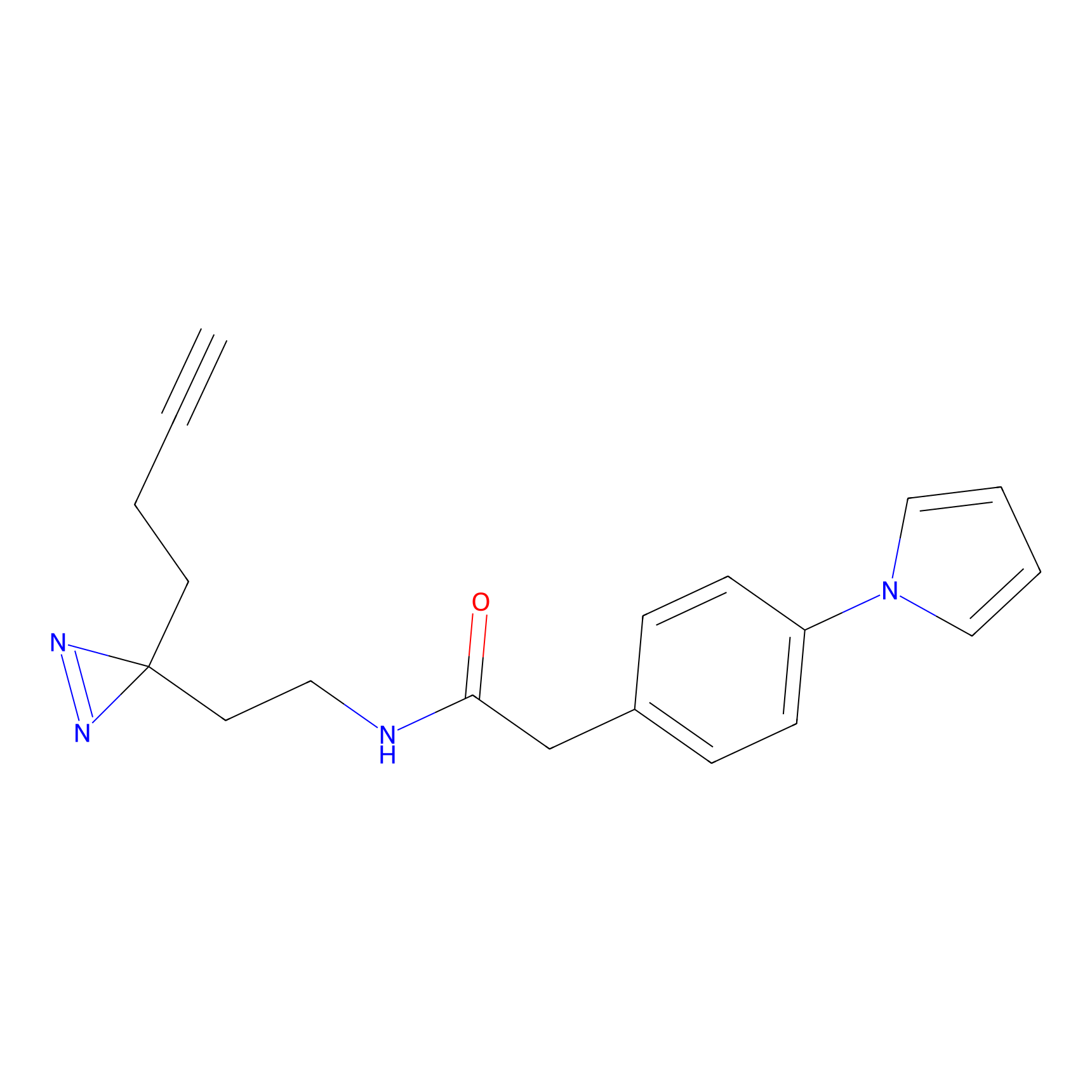
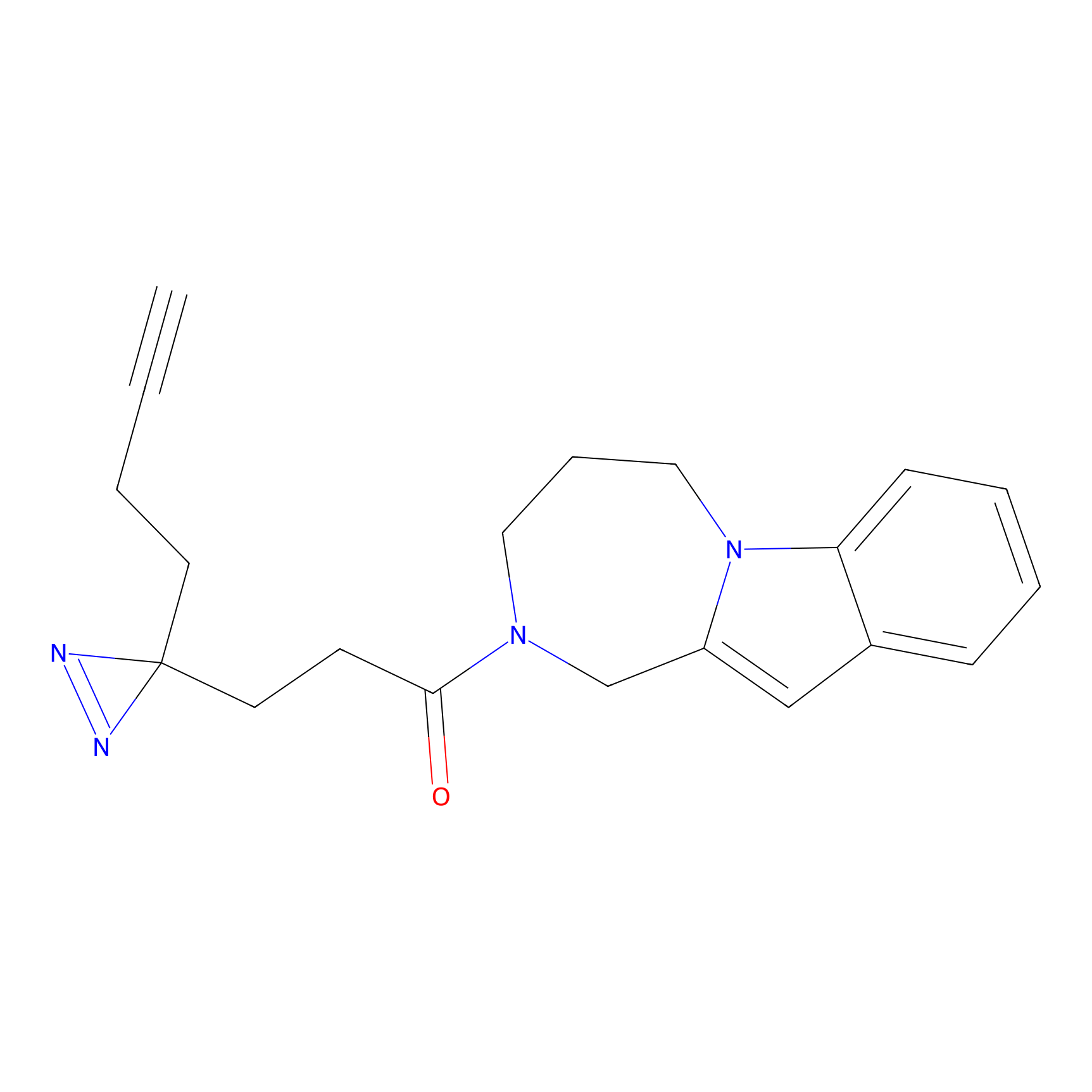
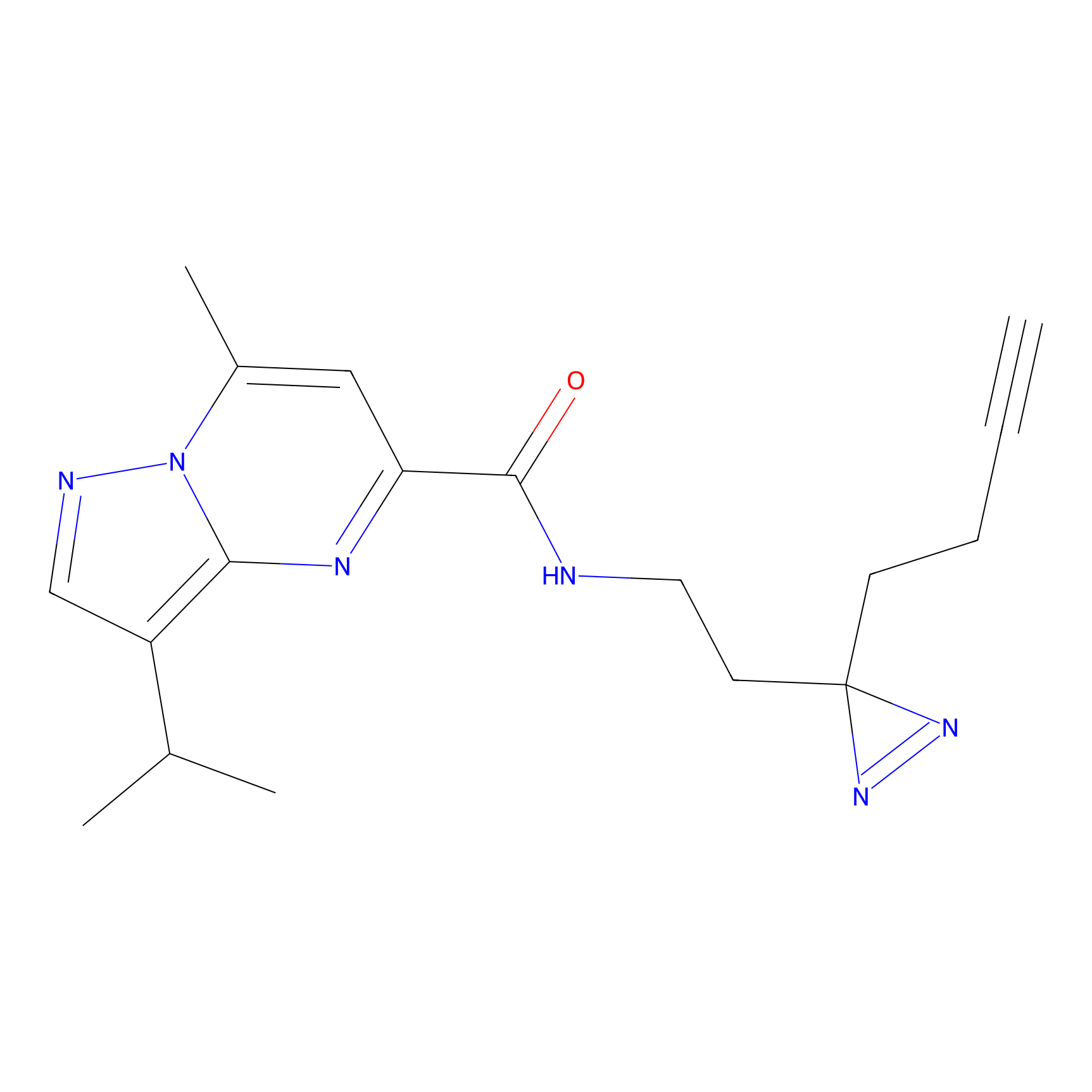
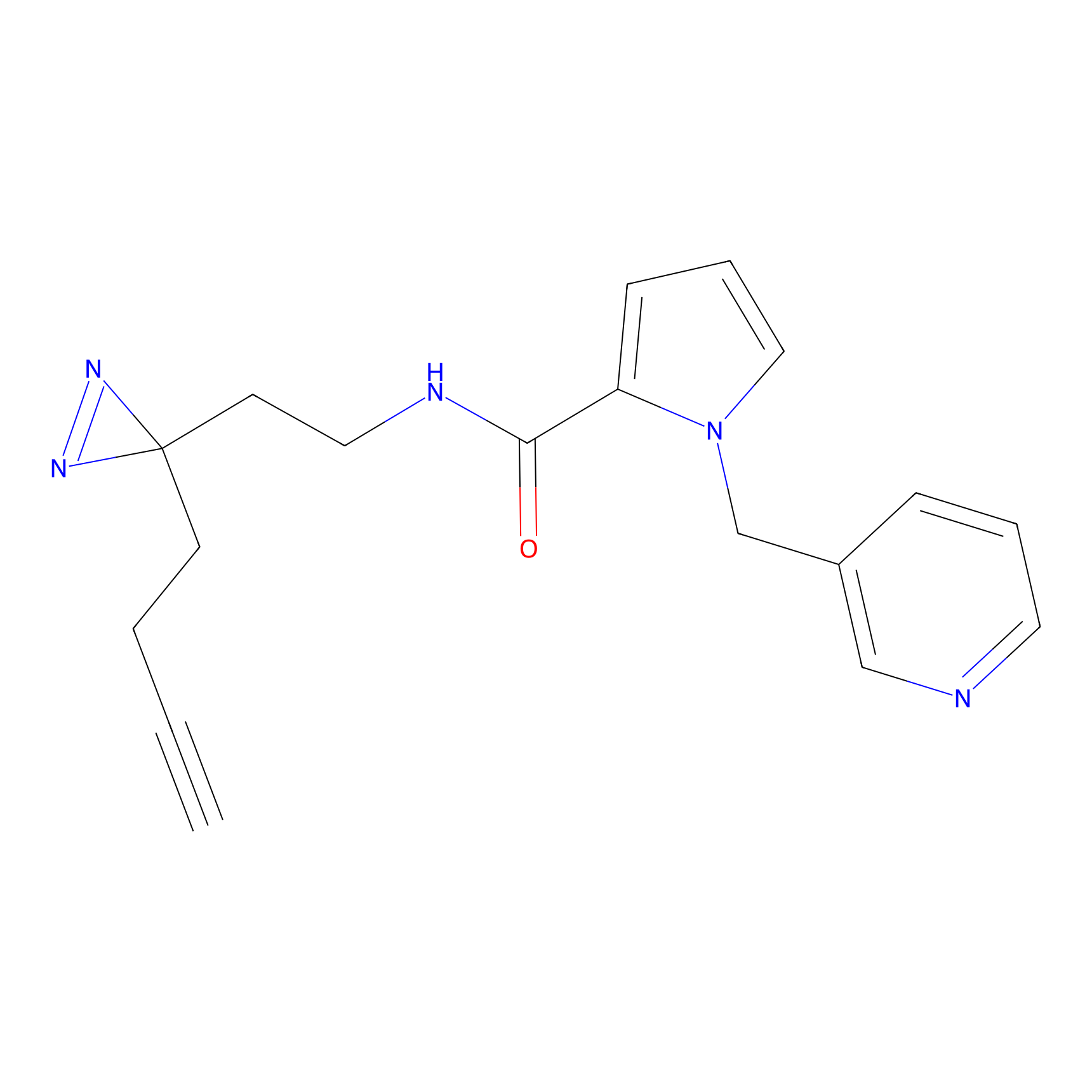
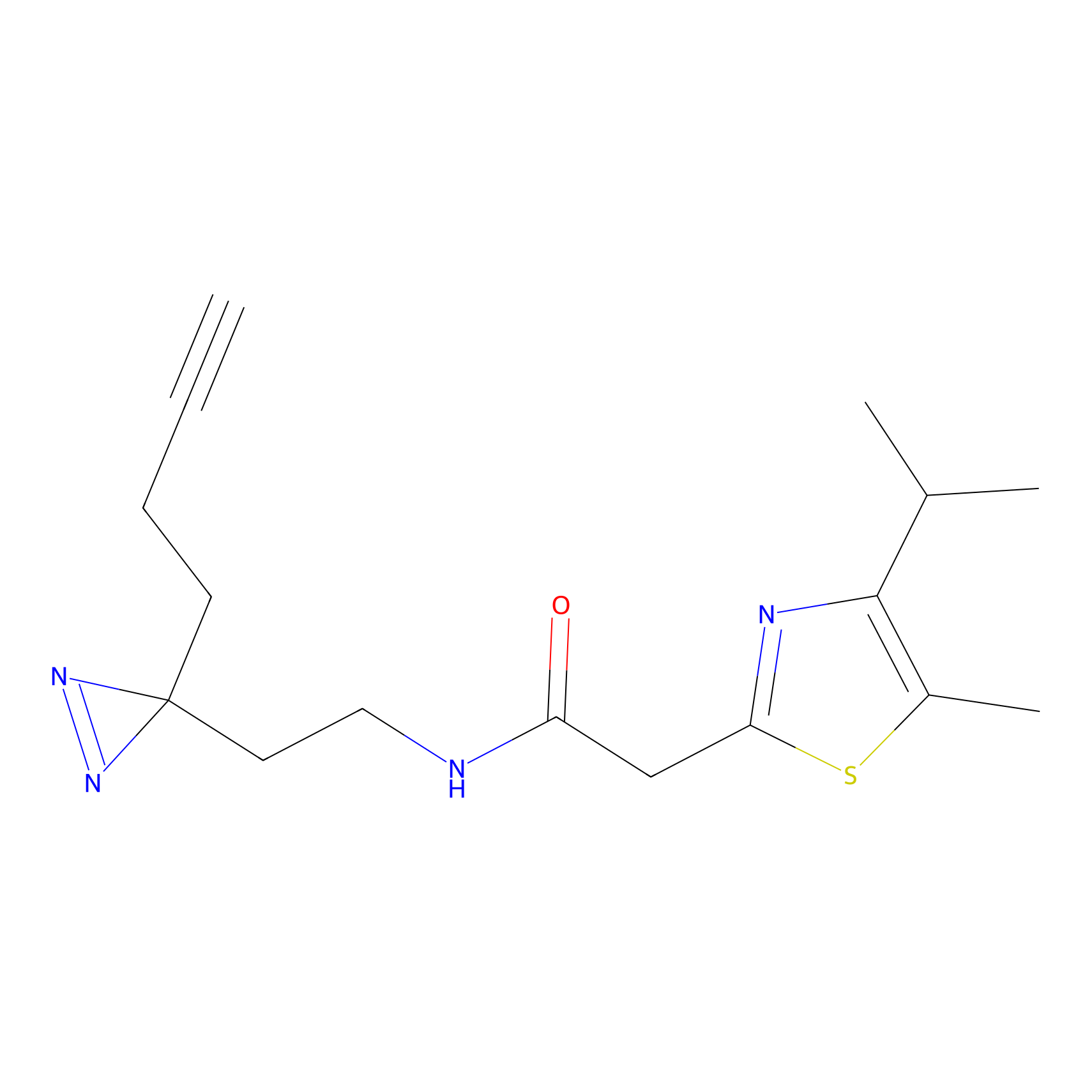
ABPP Probe
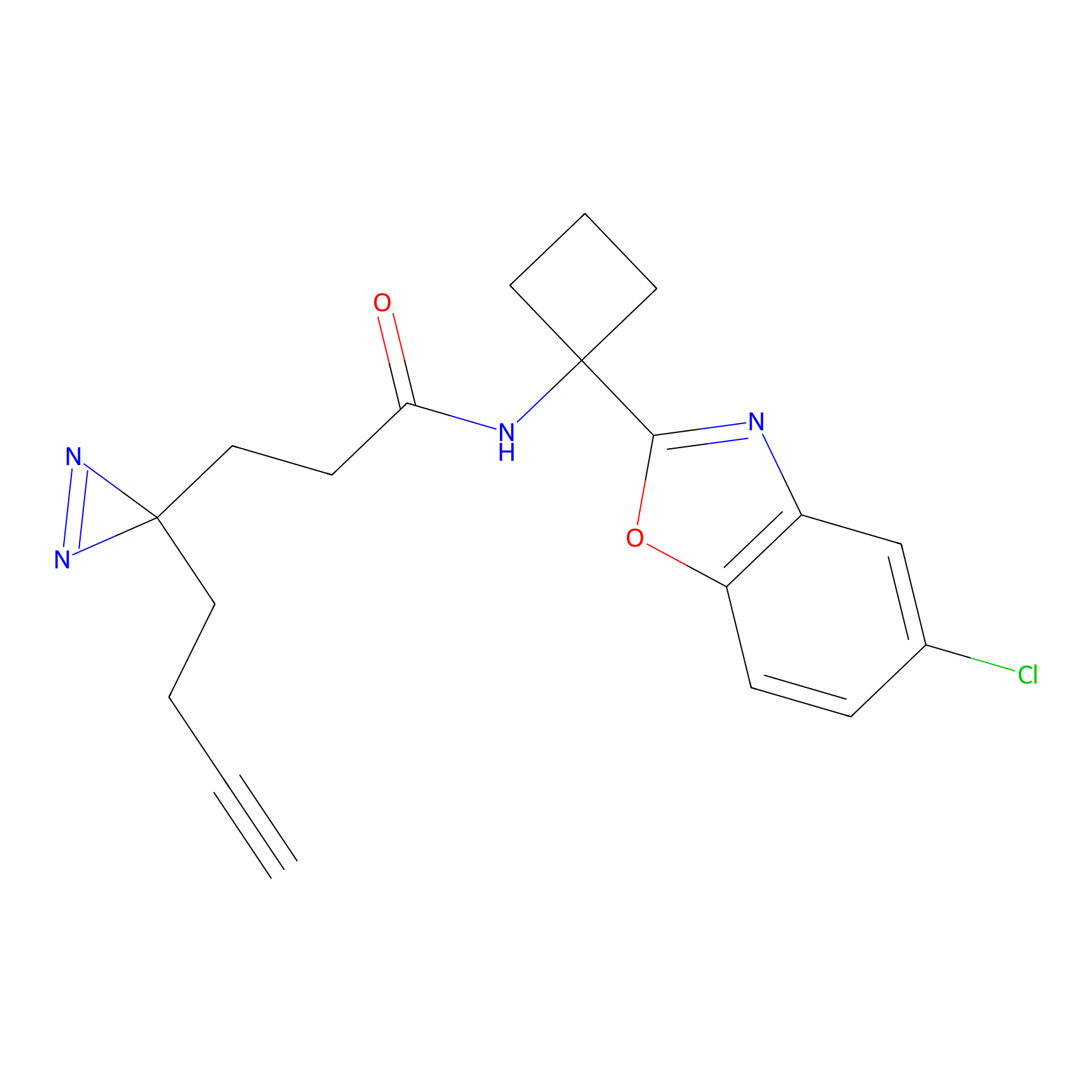
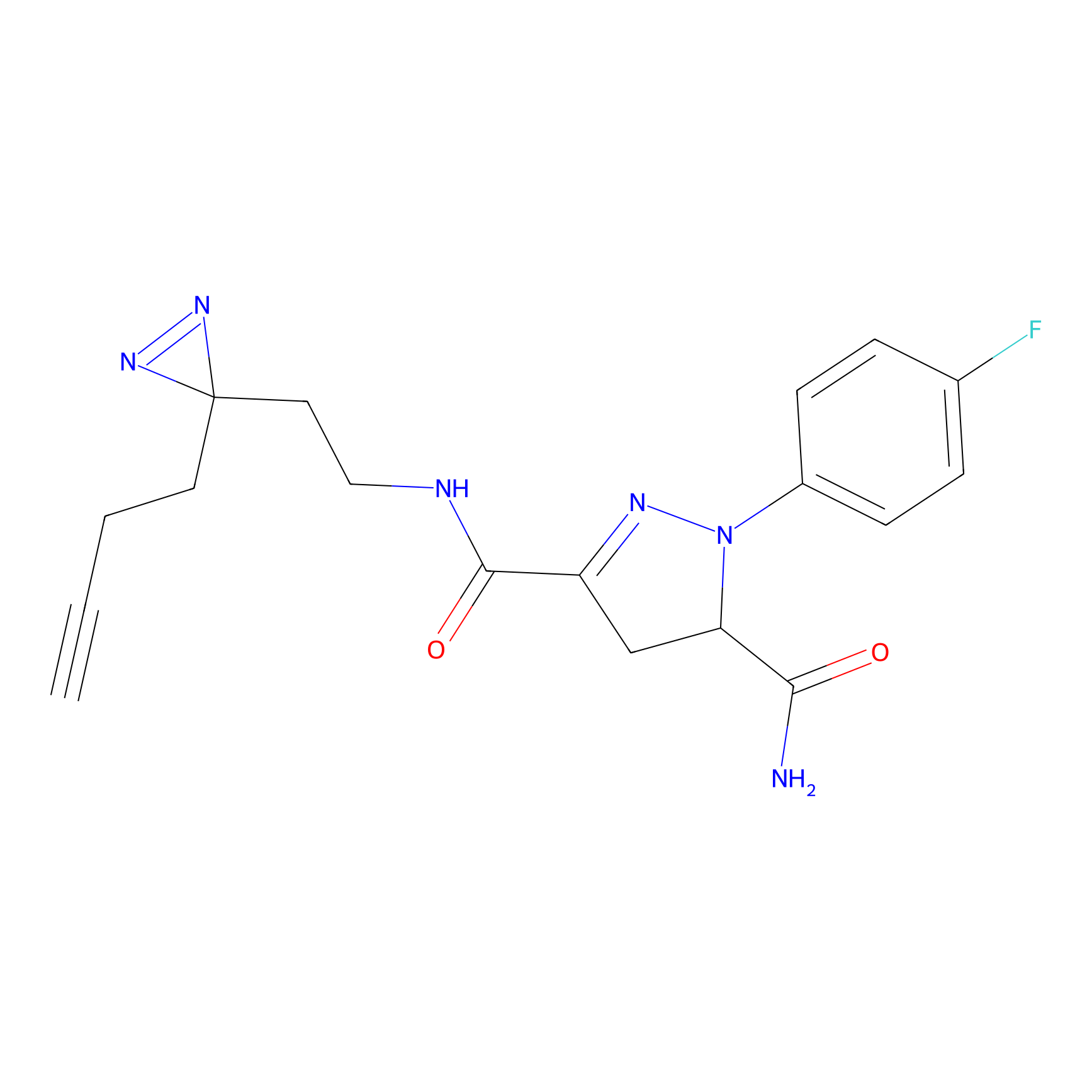
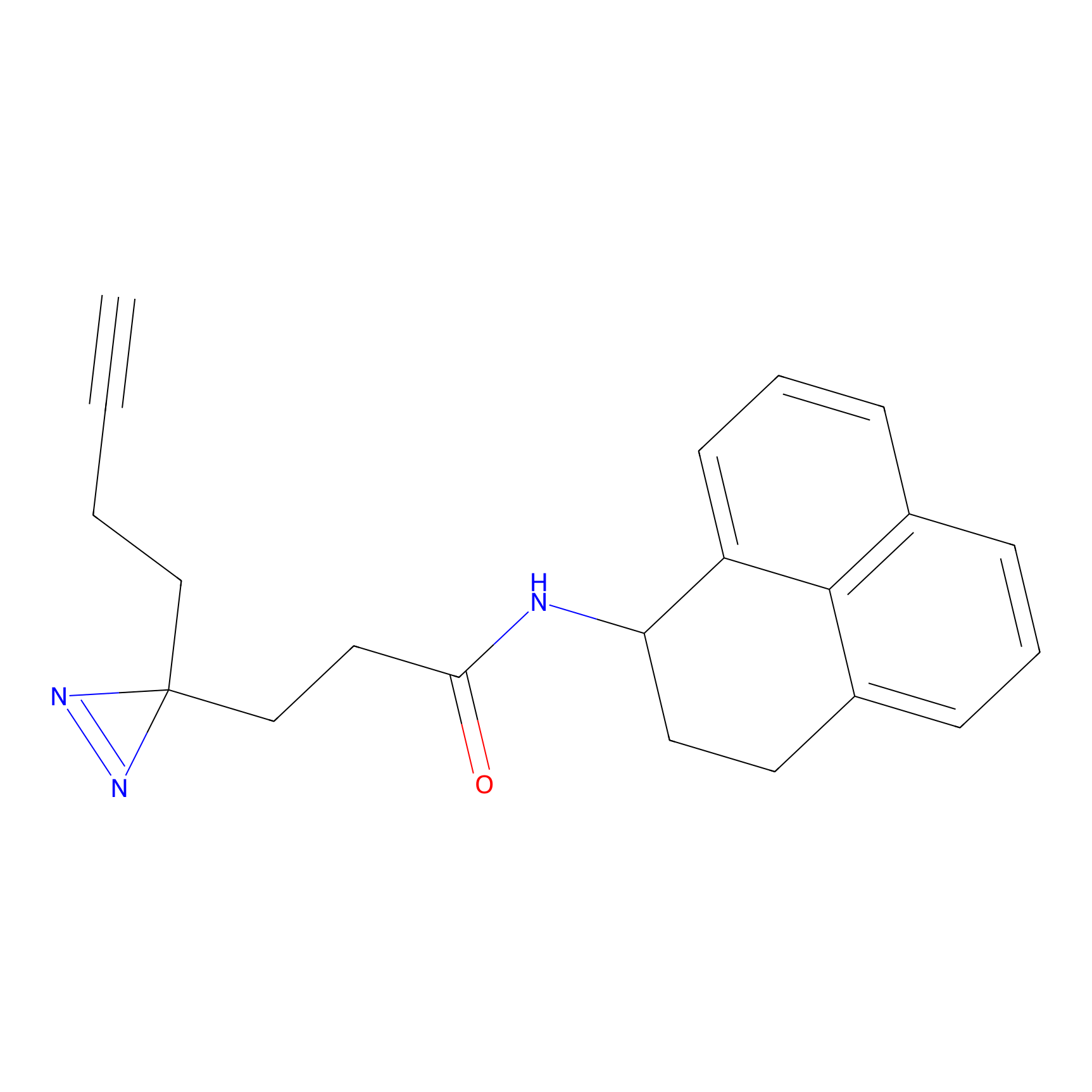
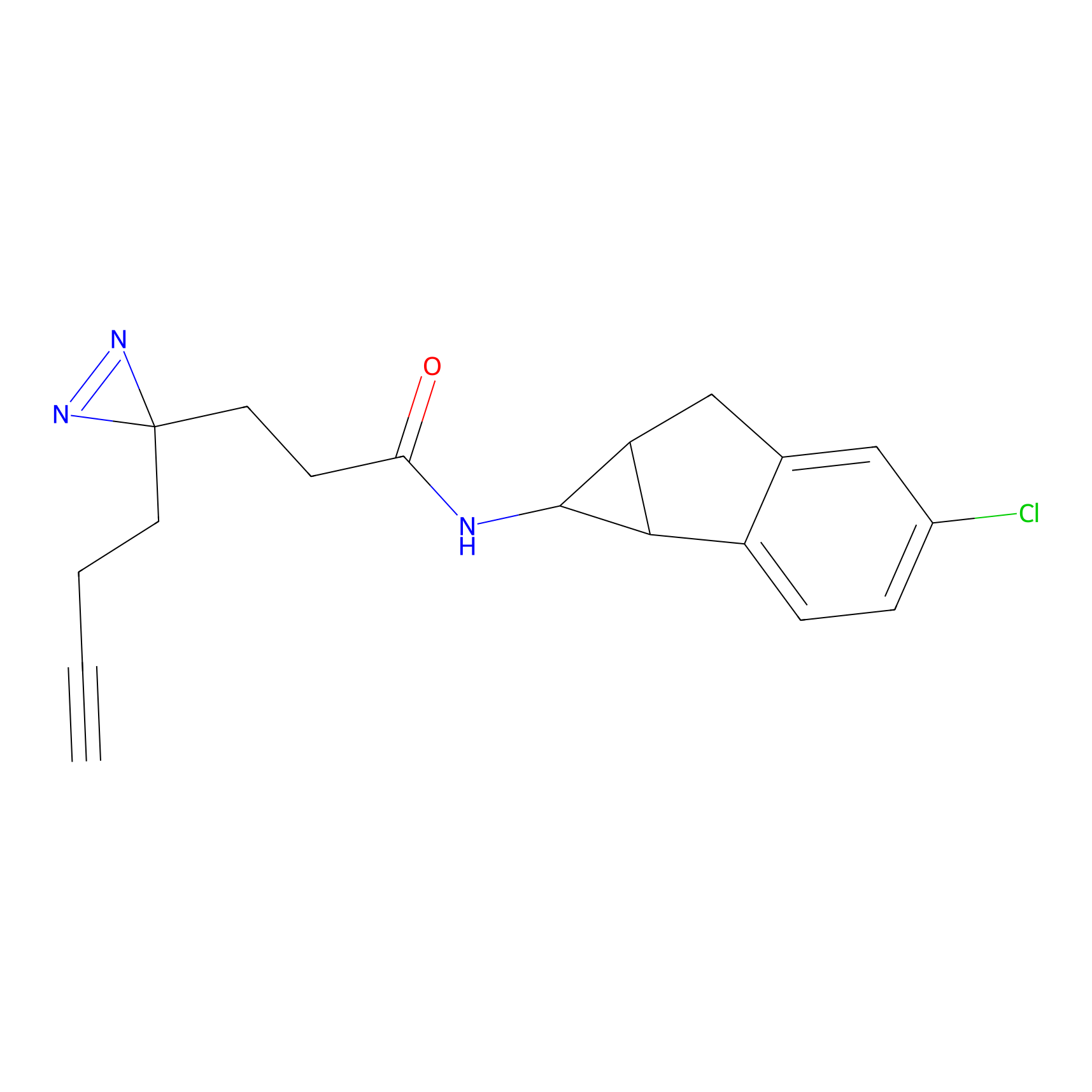
| Probe name | Structure | Binding Site(Ratio) | Interaction ID | Ref | |
|---|---|---|---|---|---|
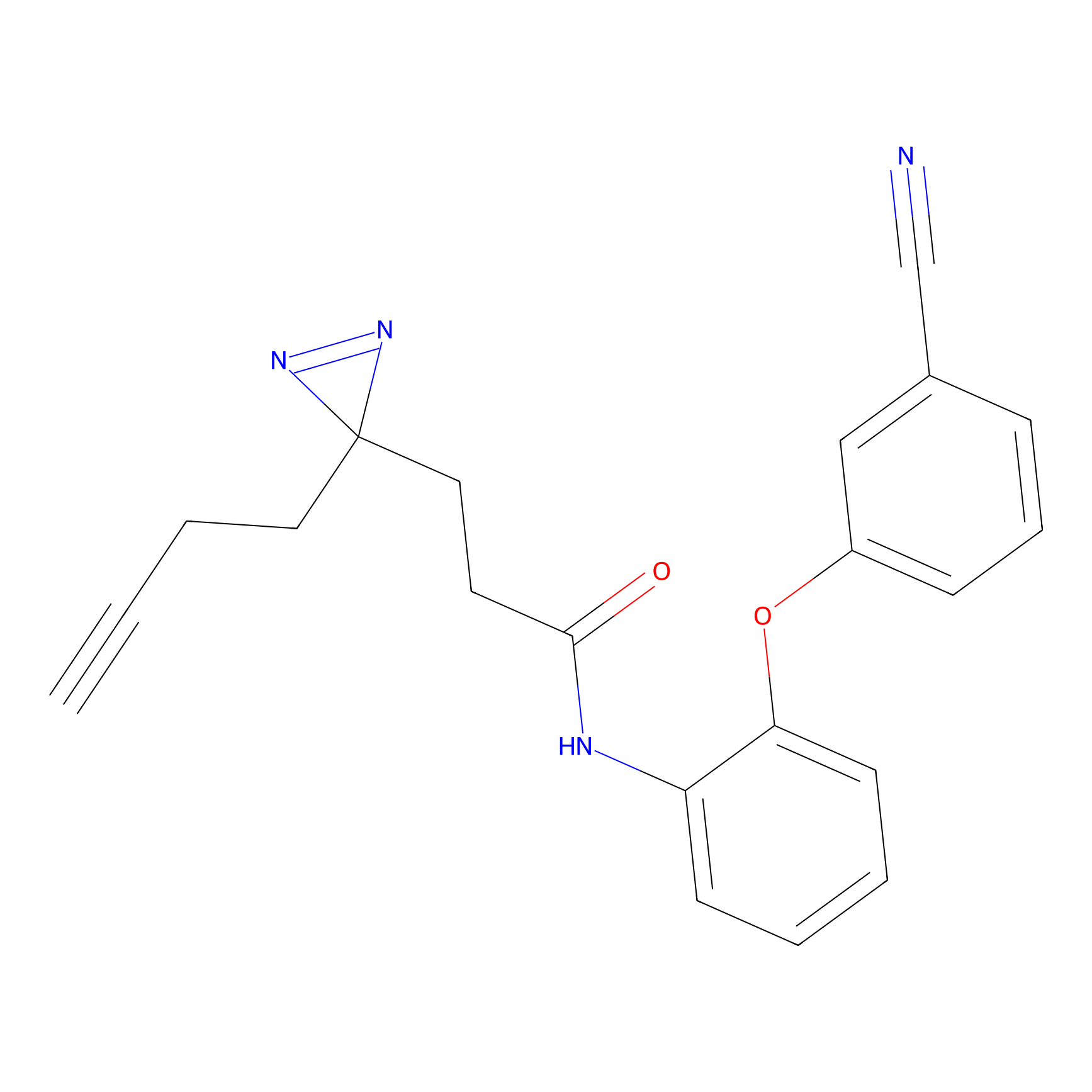
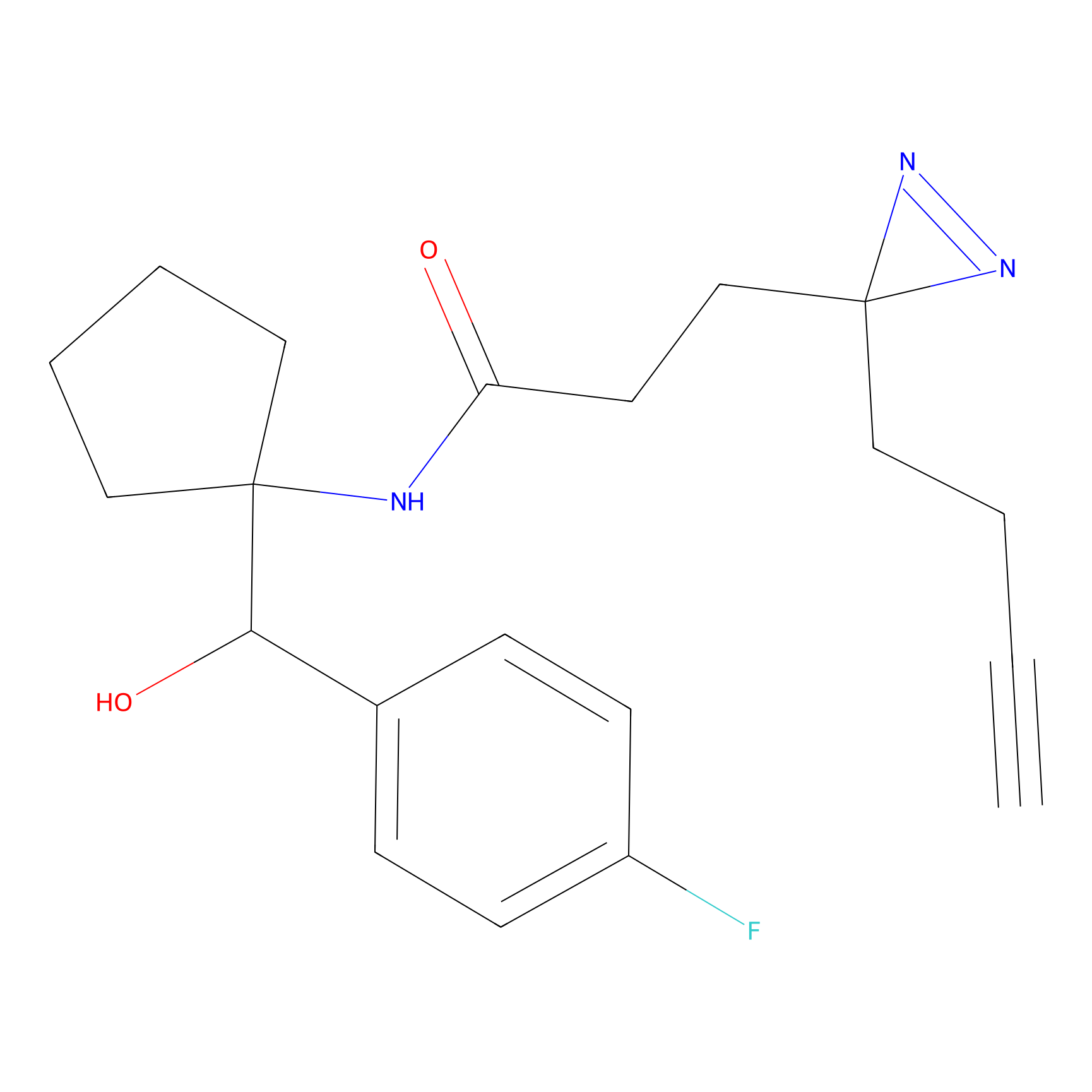
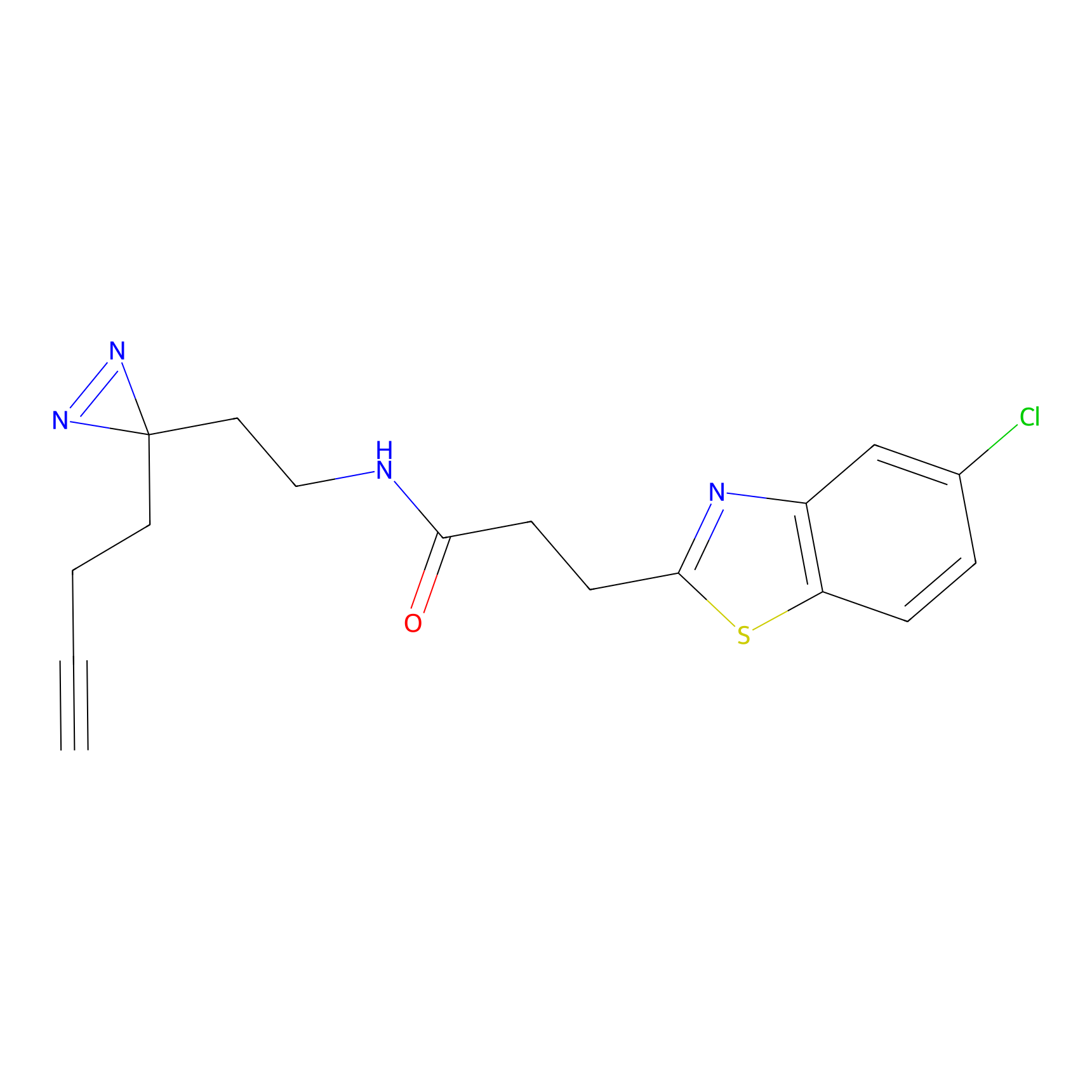
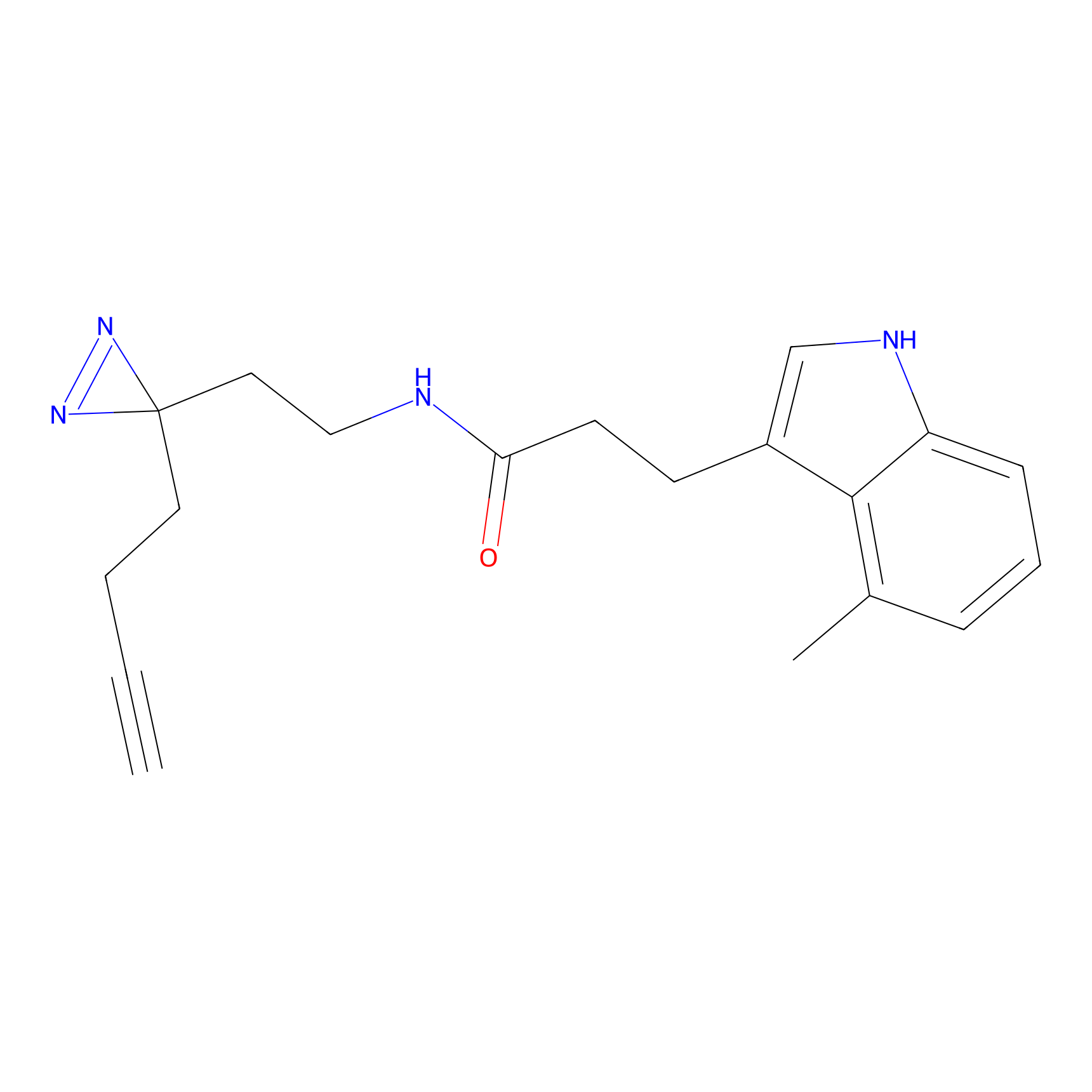
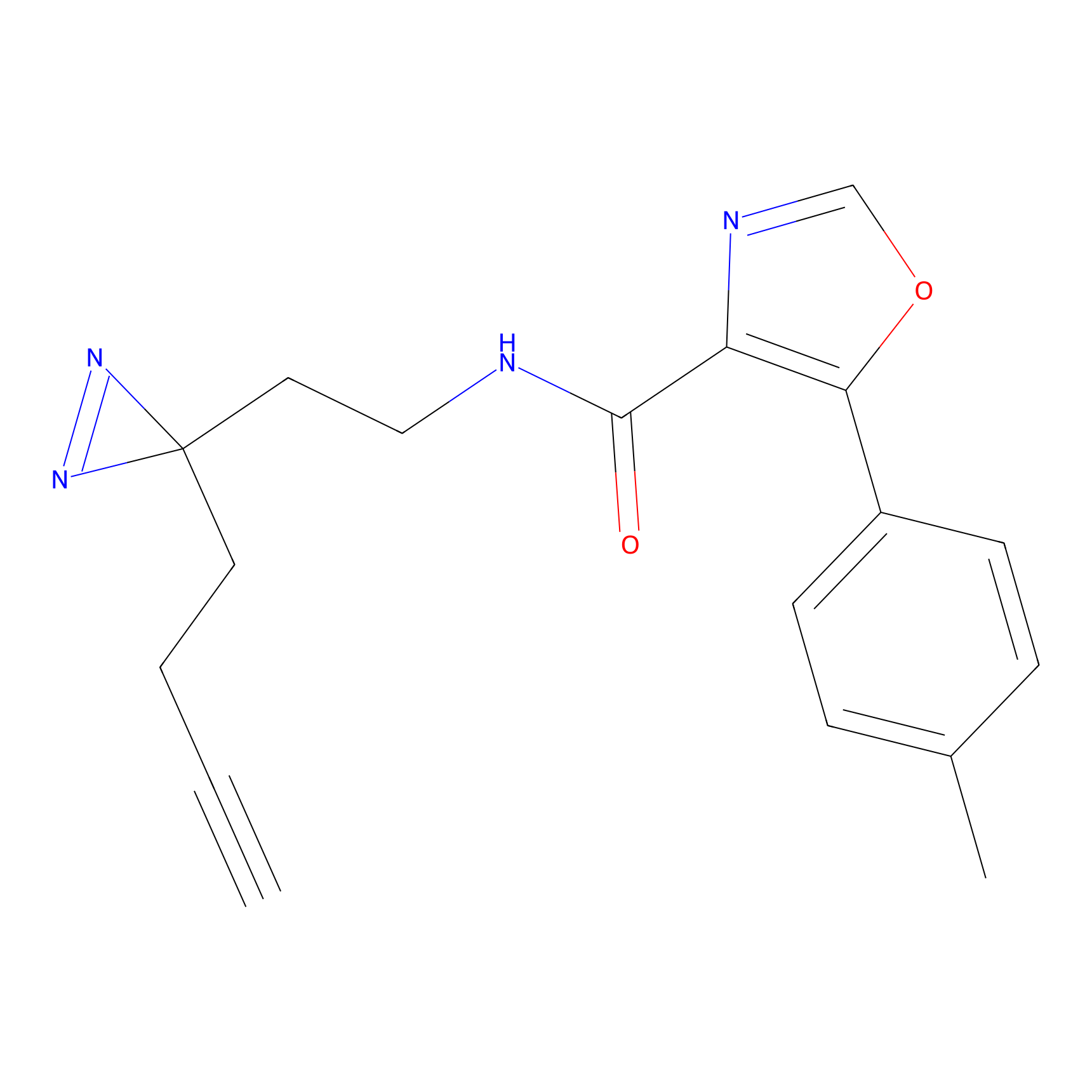
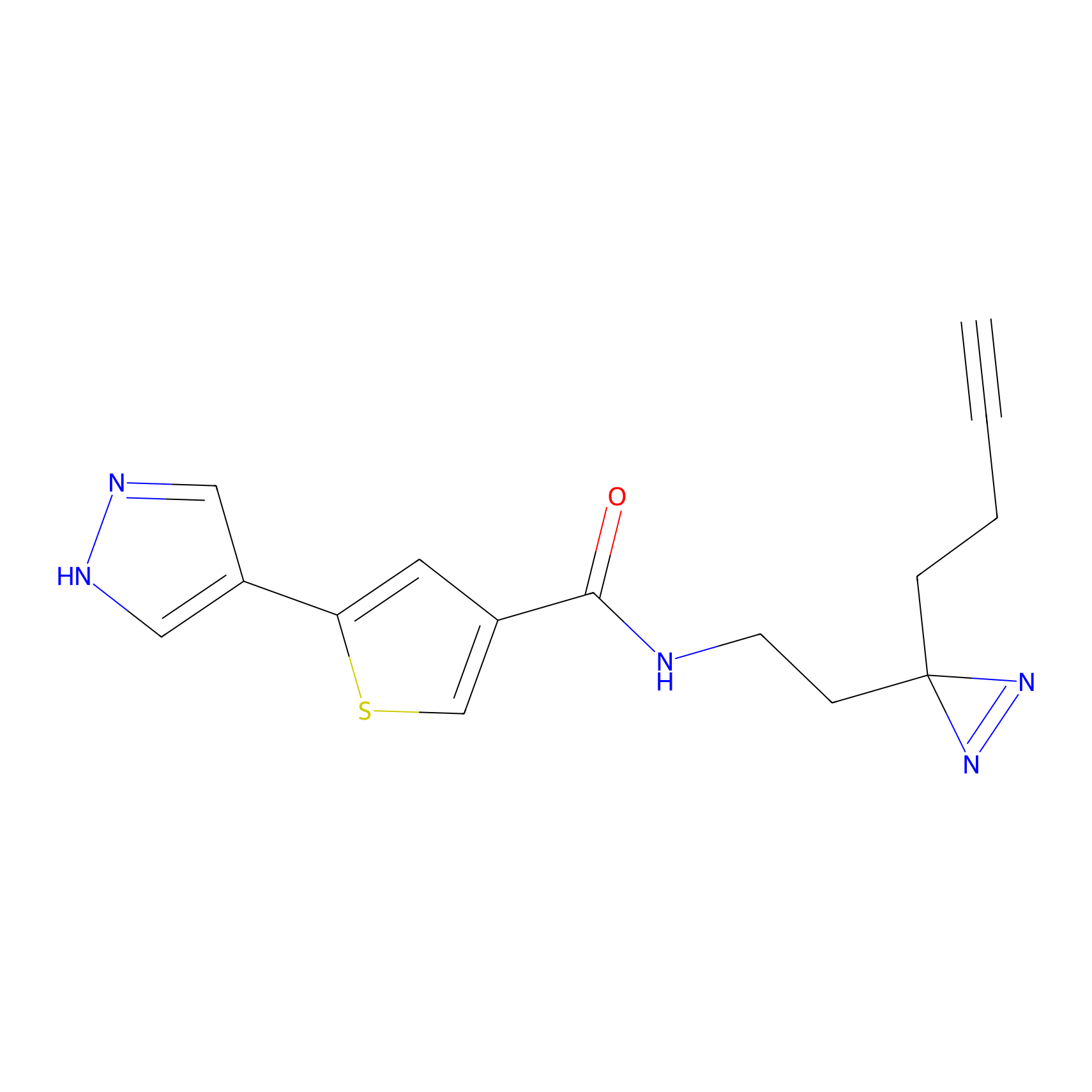
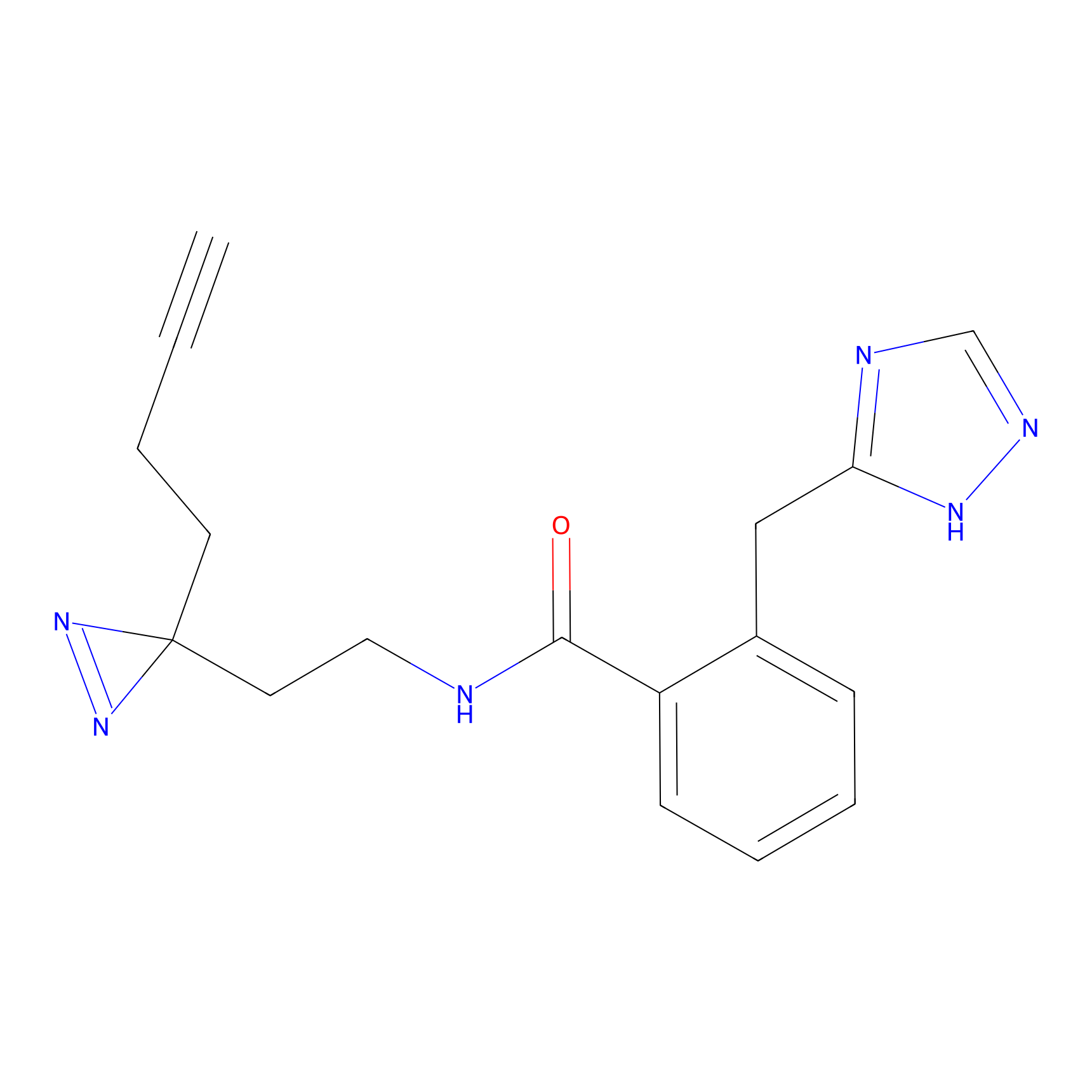
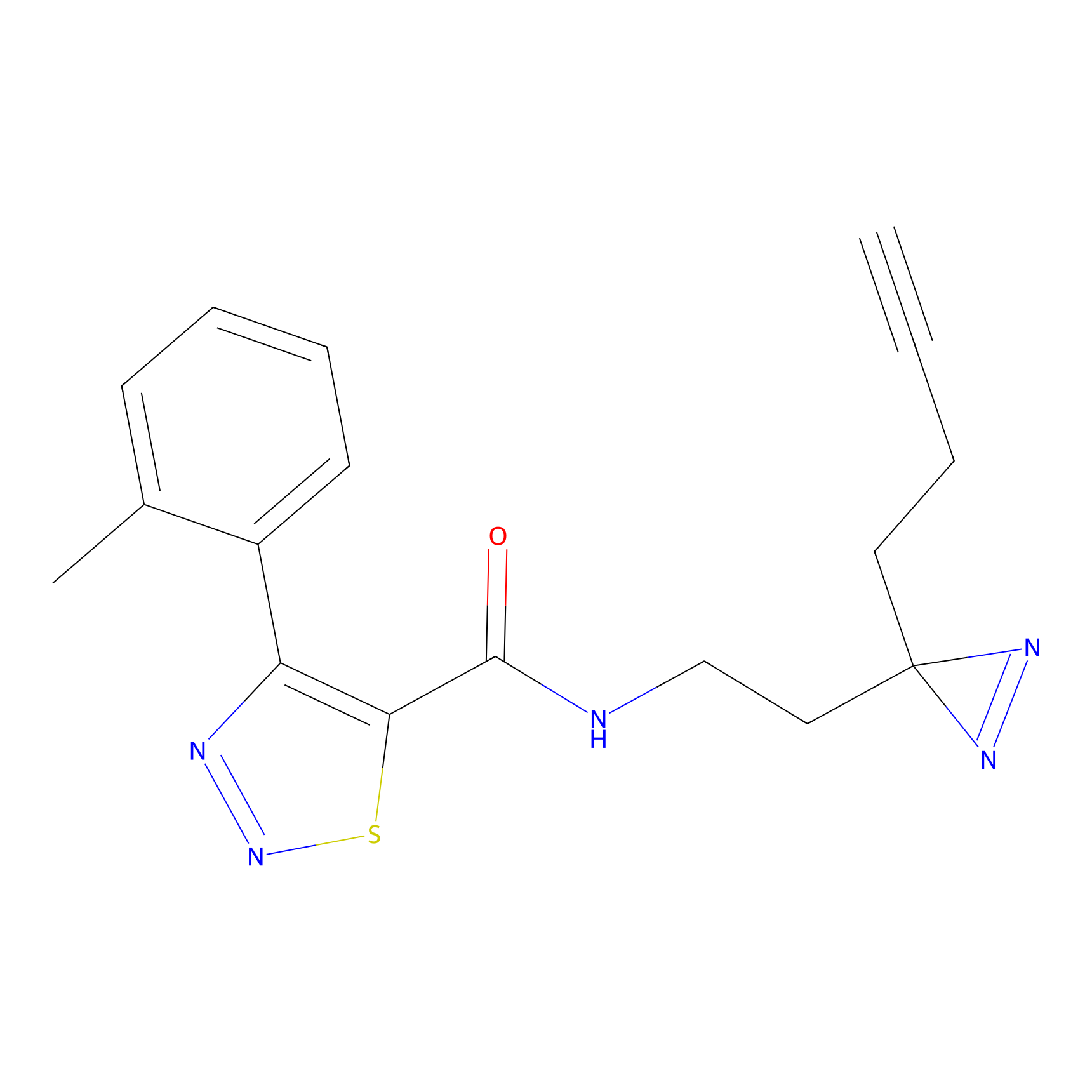
|
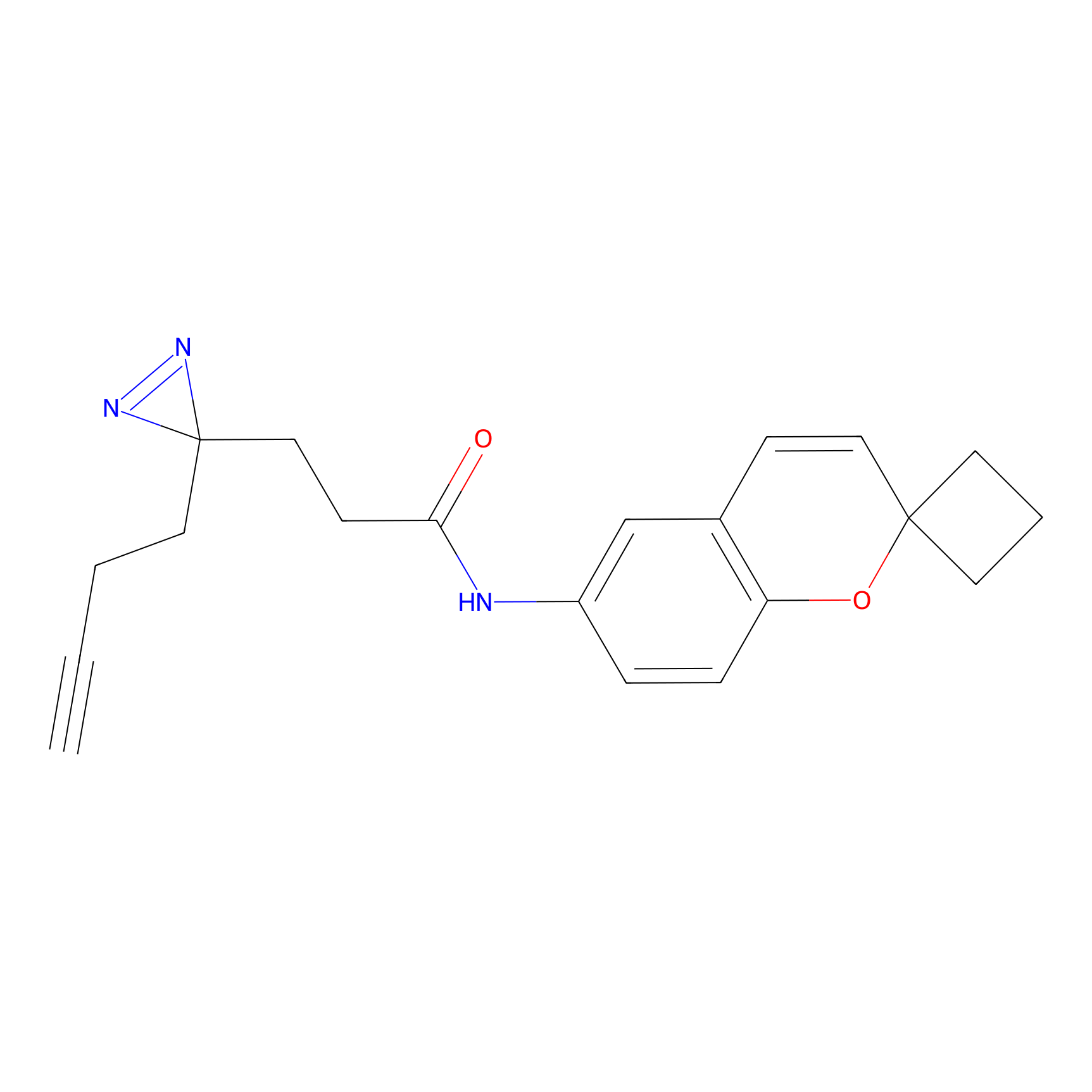
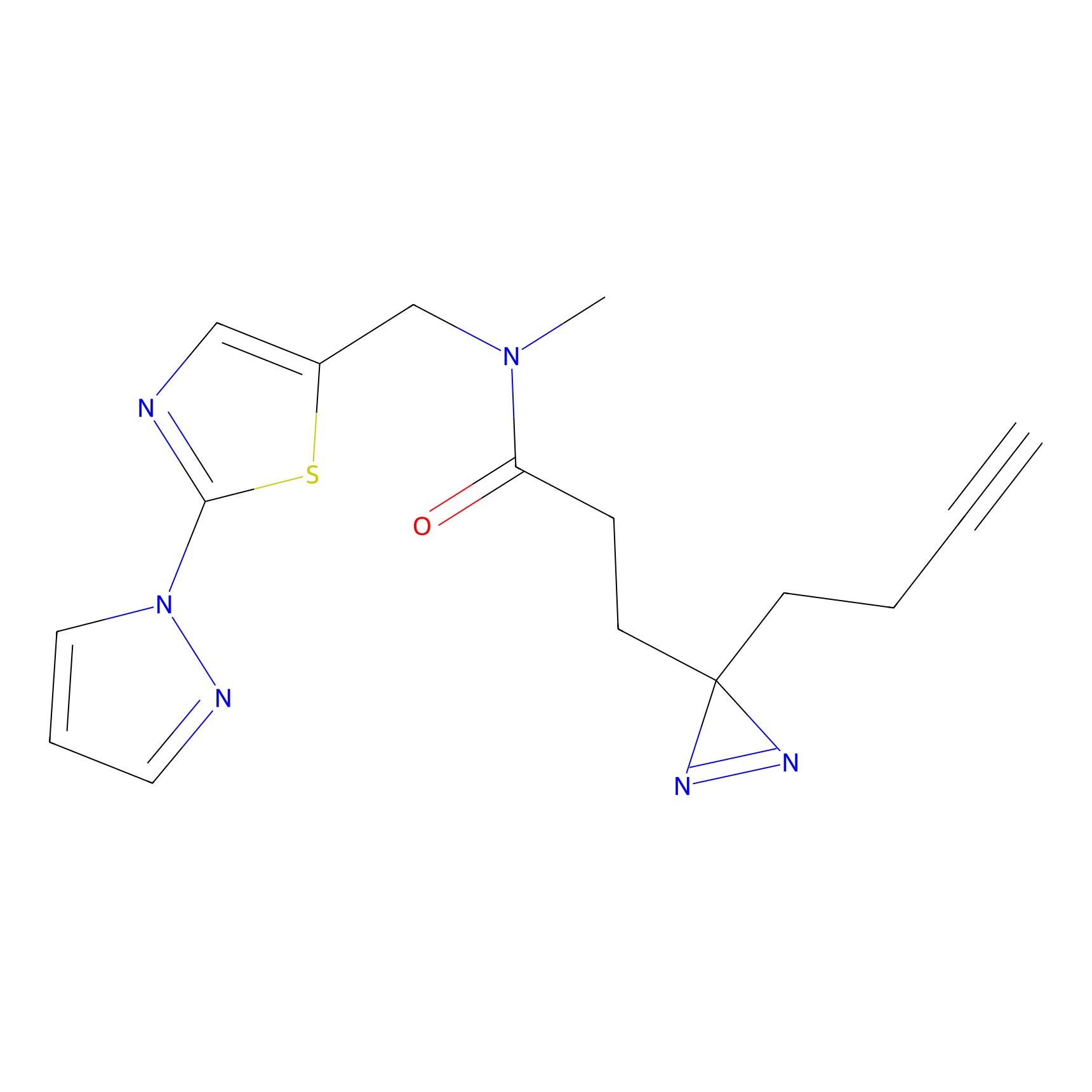
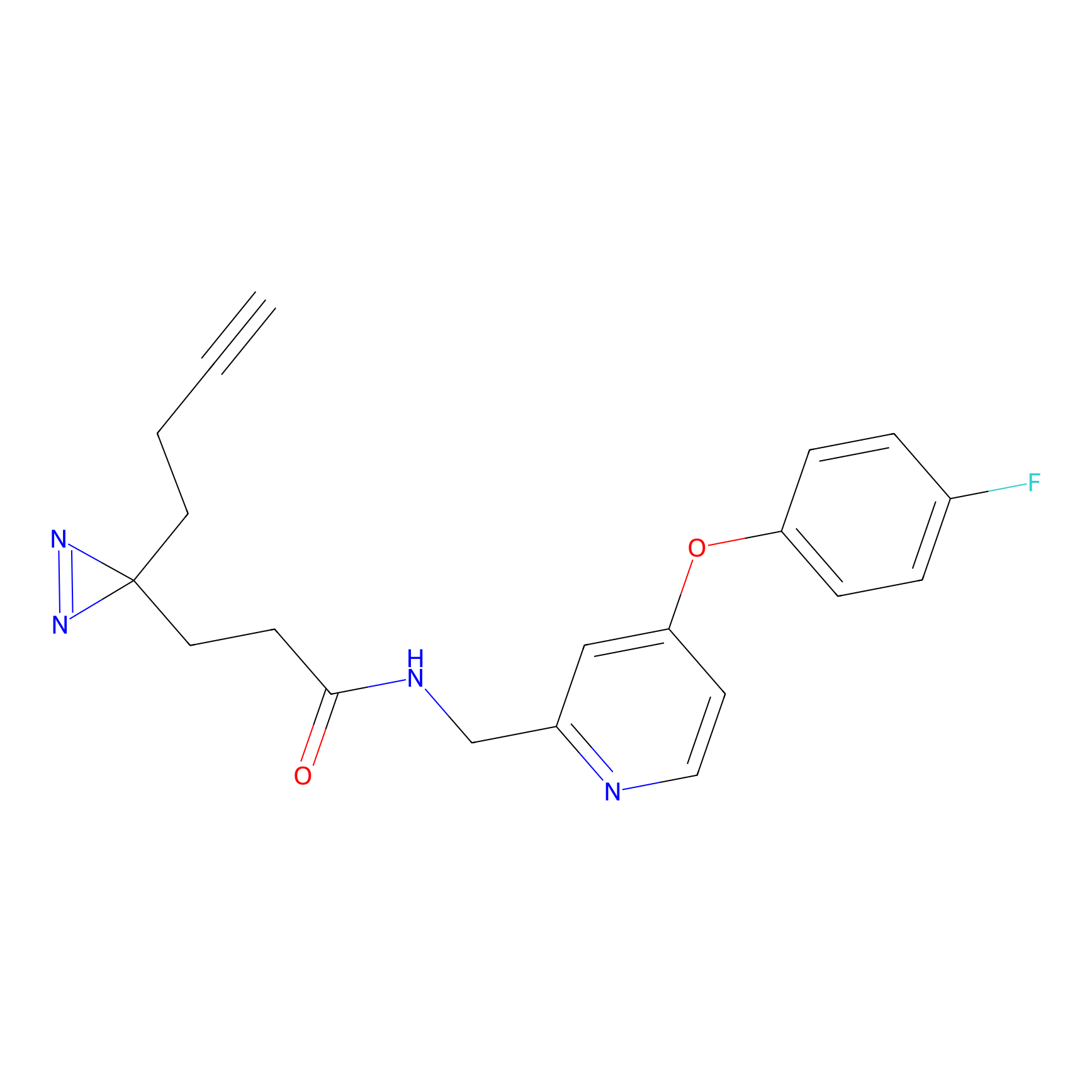
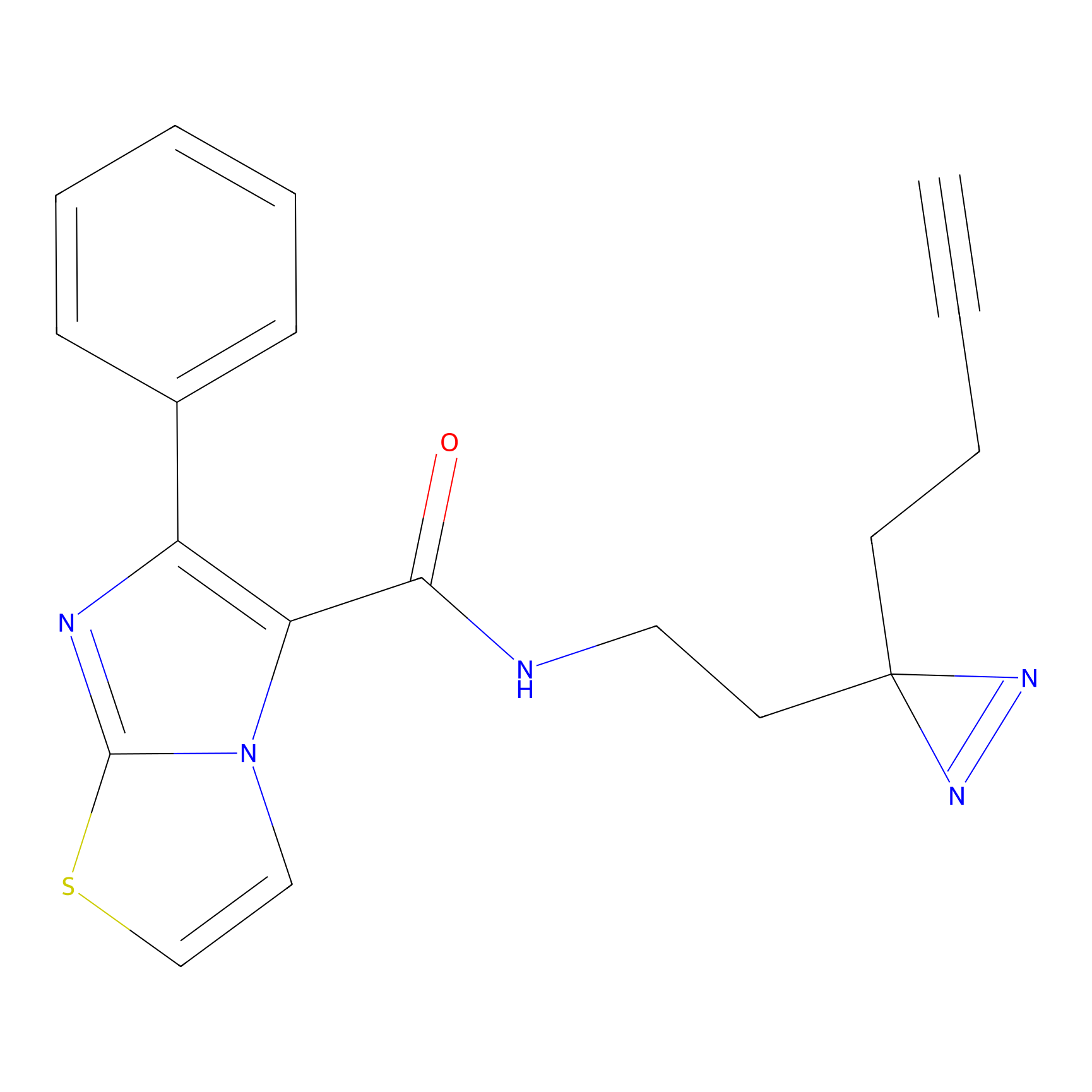
STPyne Probe Info |
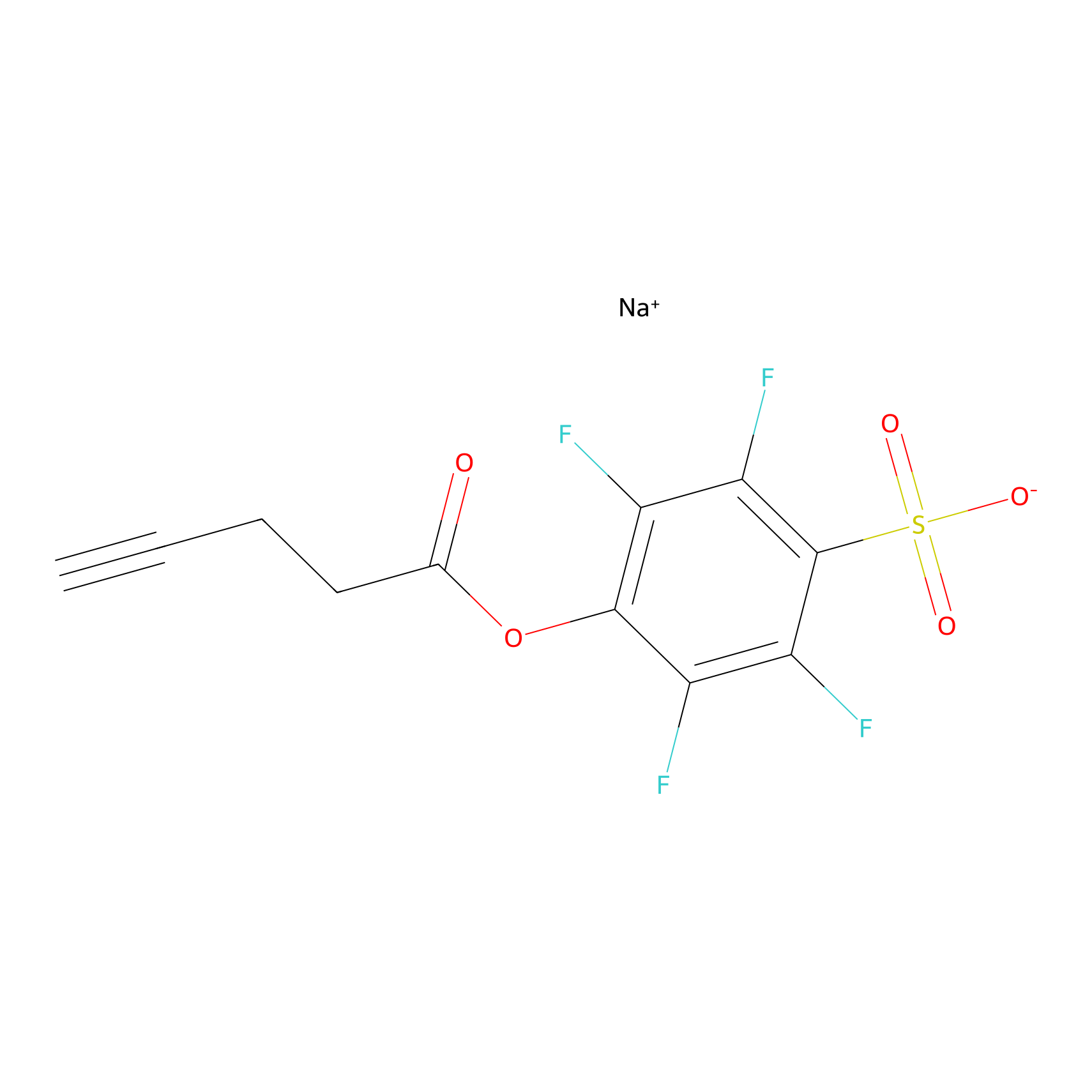 |
N.A. | LDD0009 | [1] | |
PAL-AfBPP Probe
References