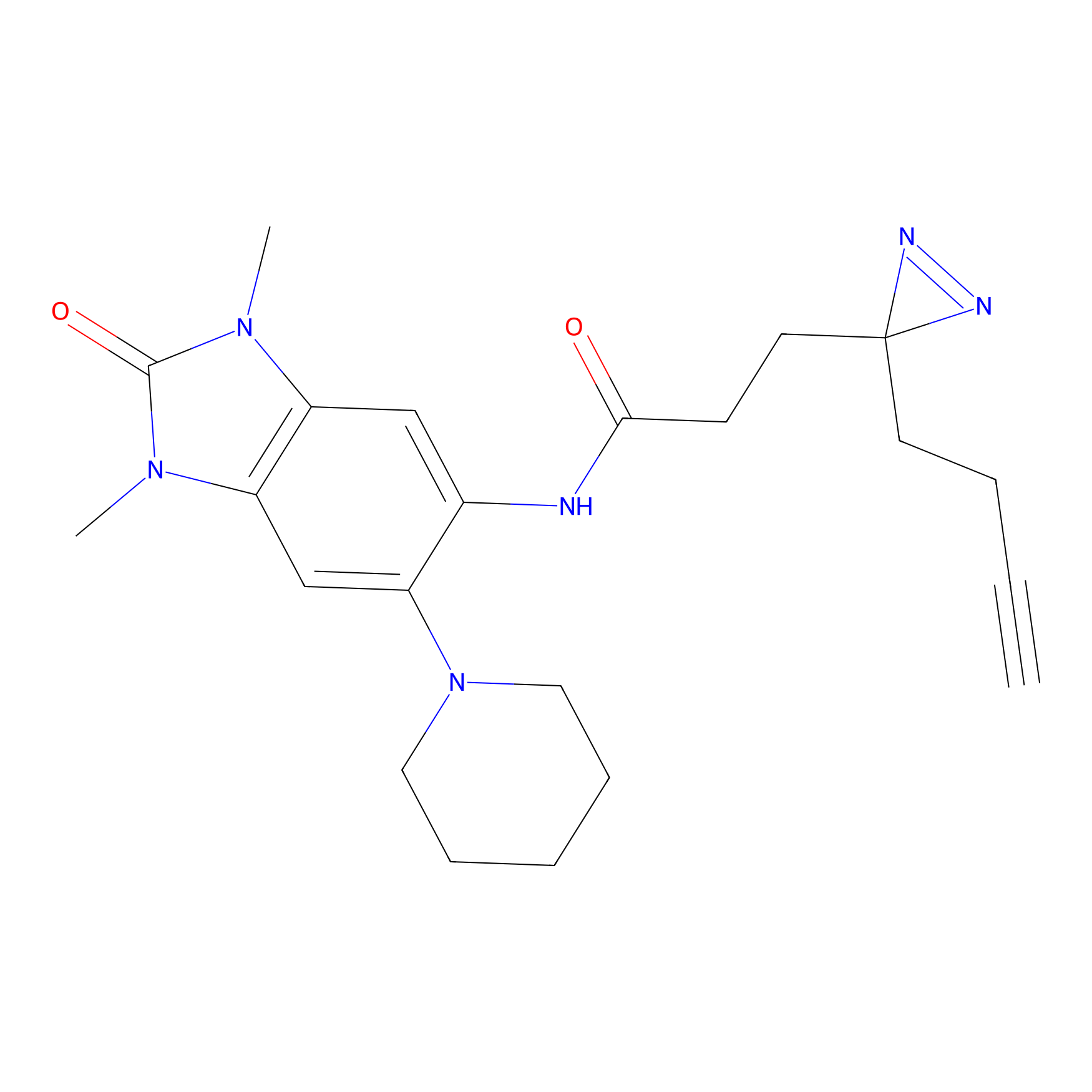
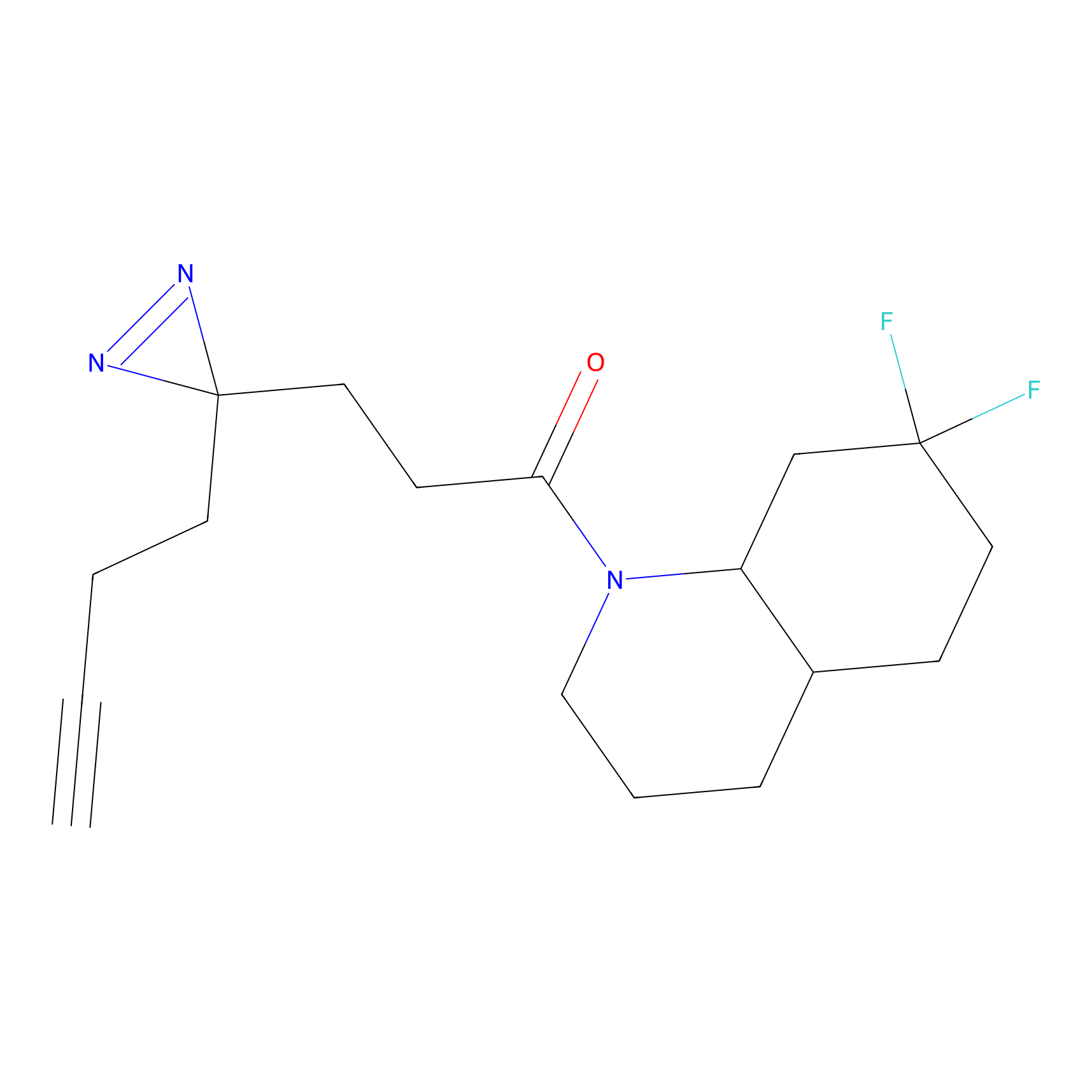
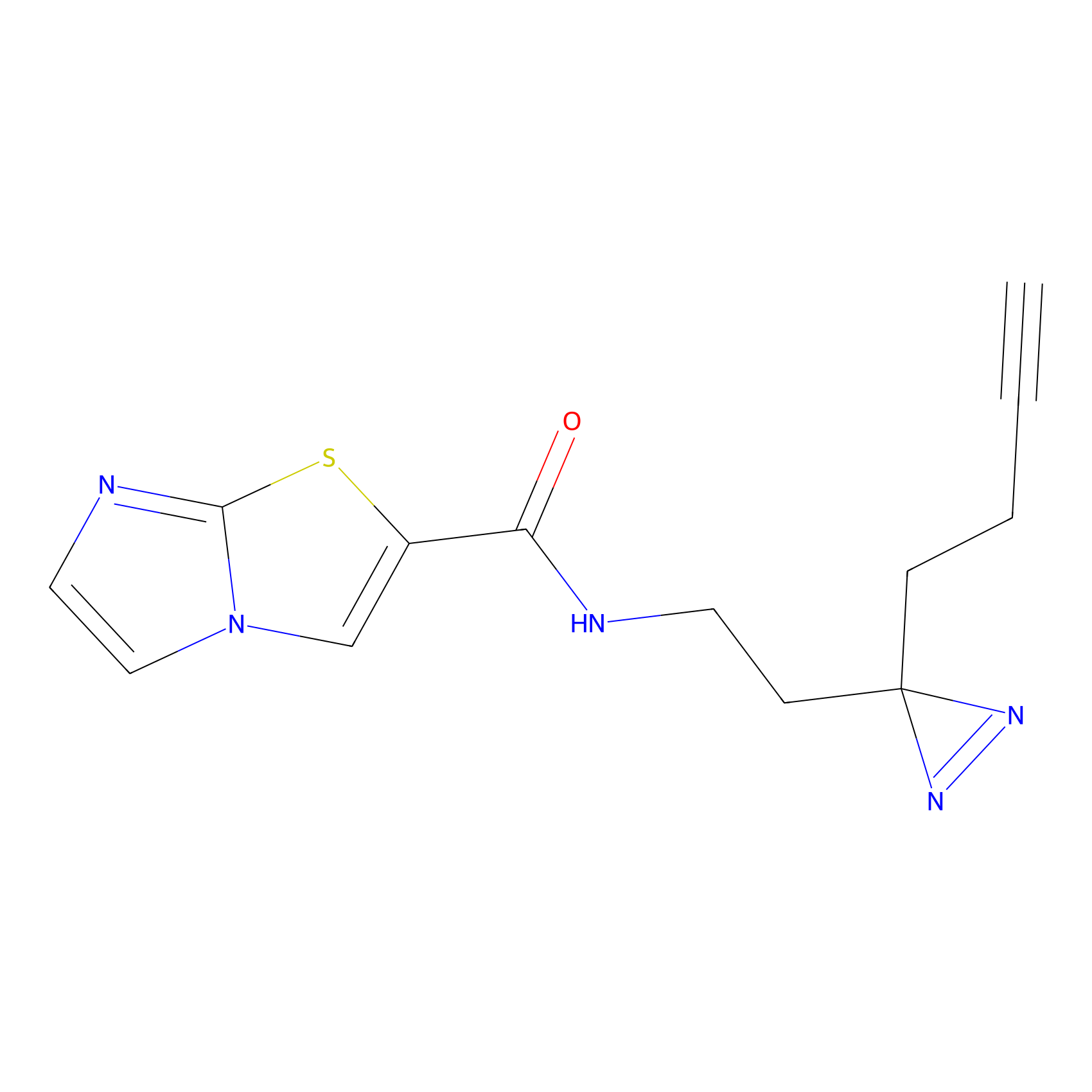
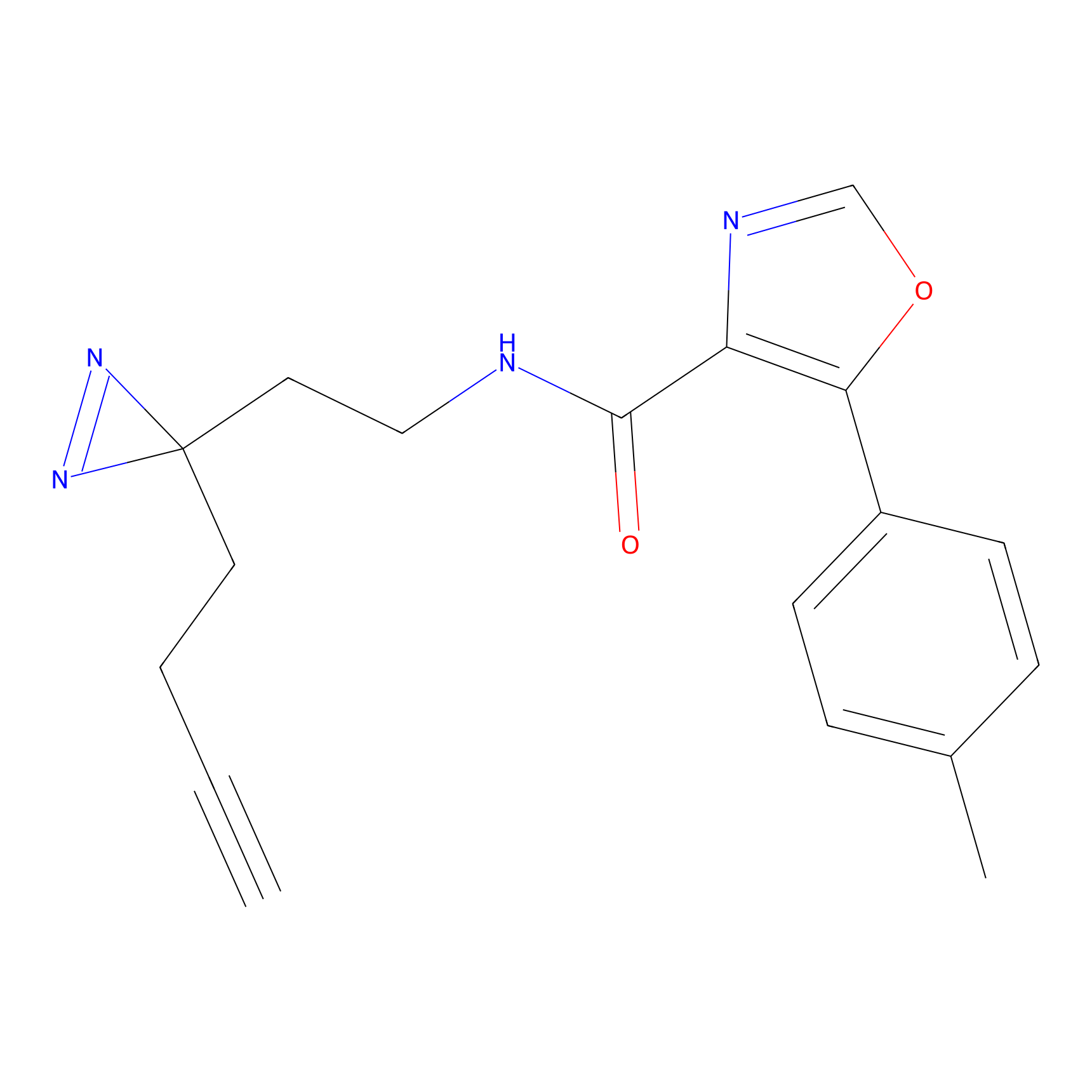
Details of the Target
General Information of Target
Target Site Mutations in Different Cell Lines
Probe(s) Labeling This Target
ABPP Probe
| Probe name | Structure | Binding Site(Ratio) | Interaction ID | Ref | |
|---|---|---|---|---|---|
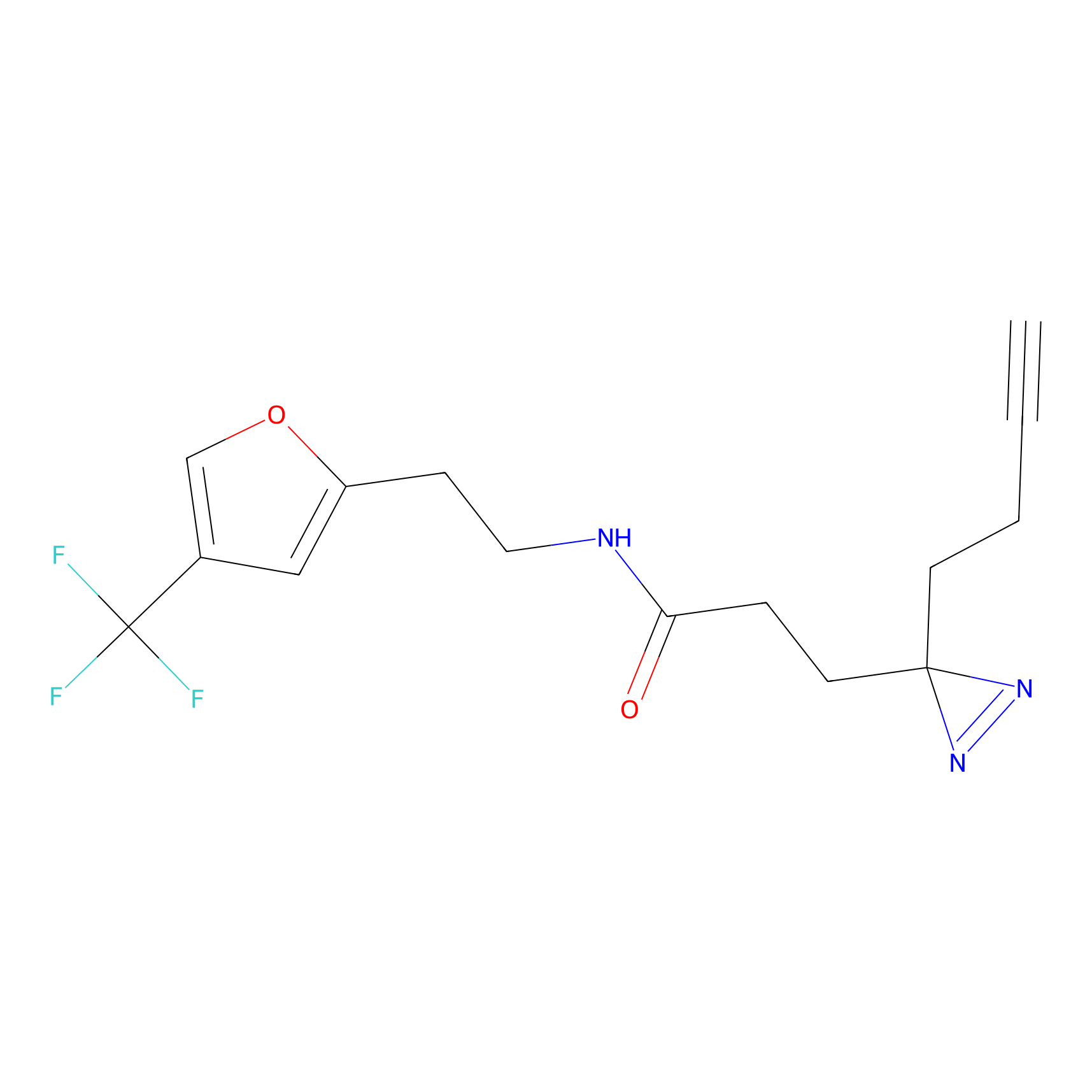
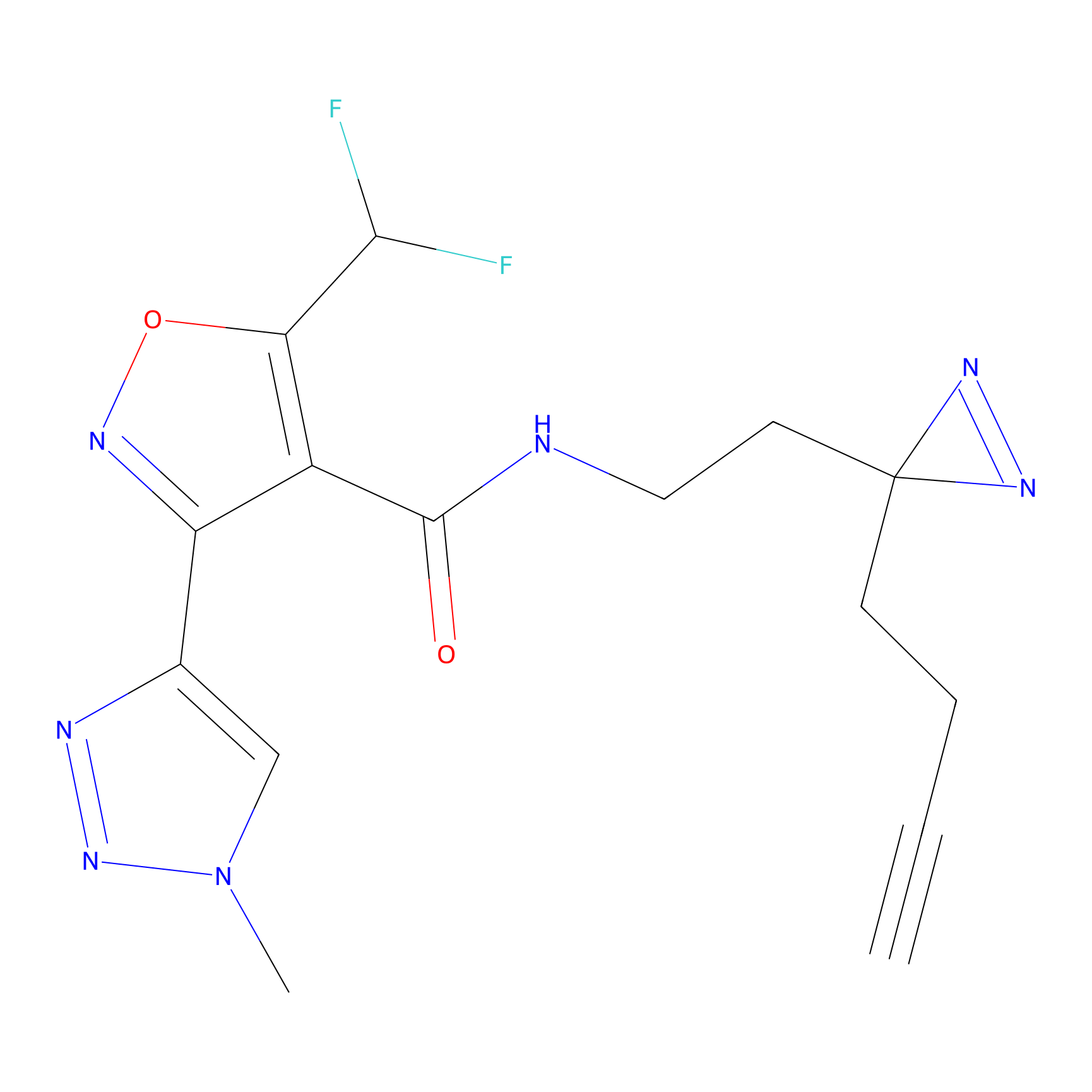
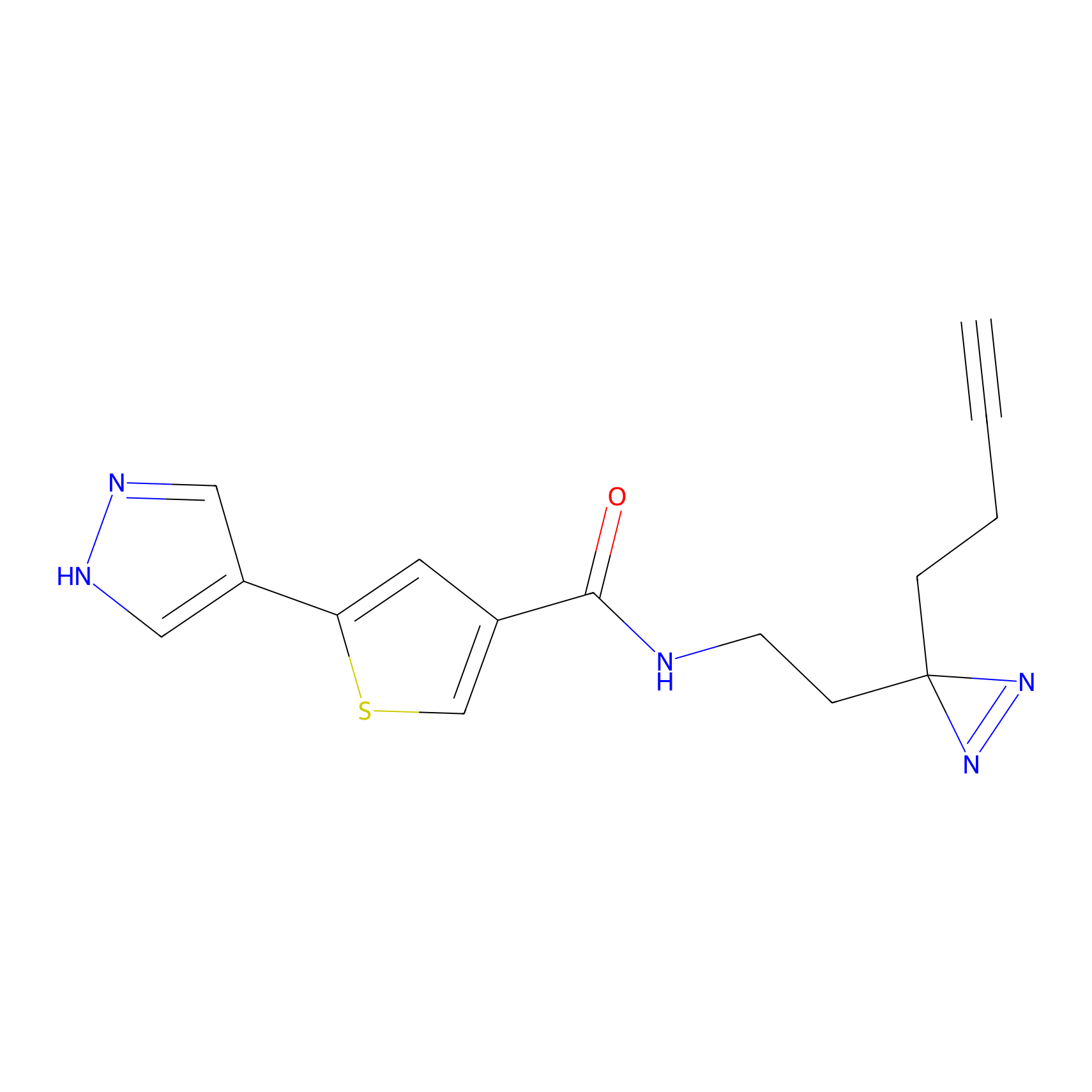
|
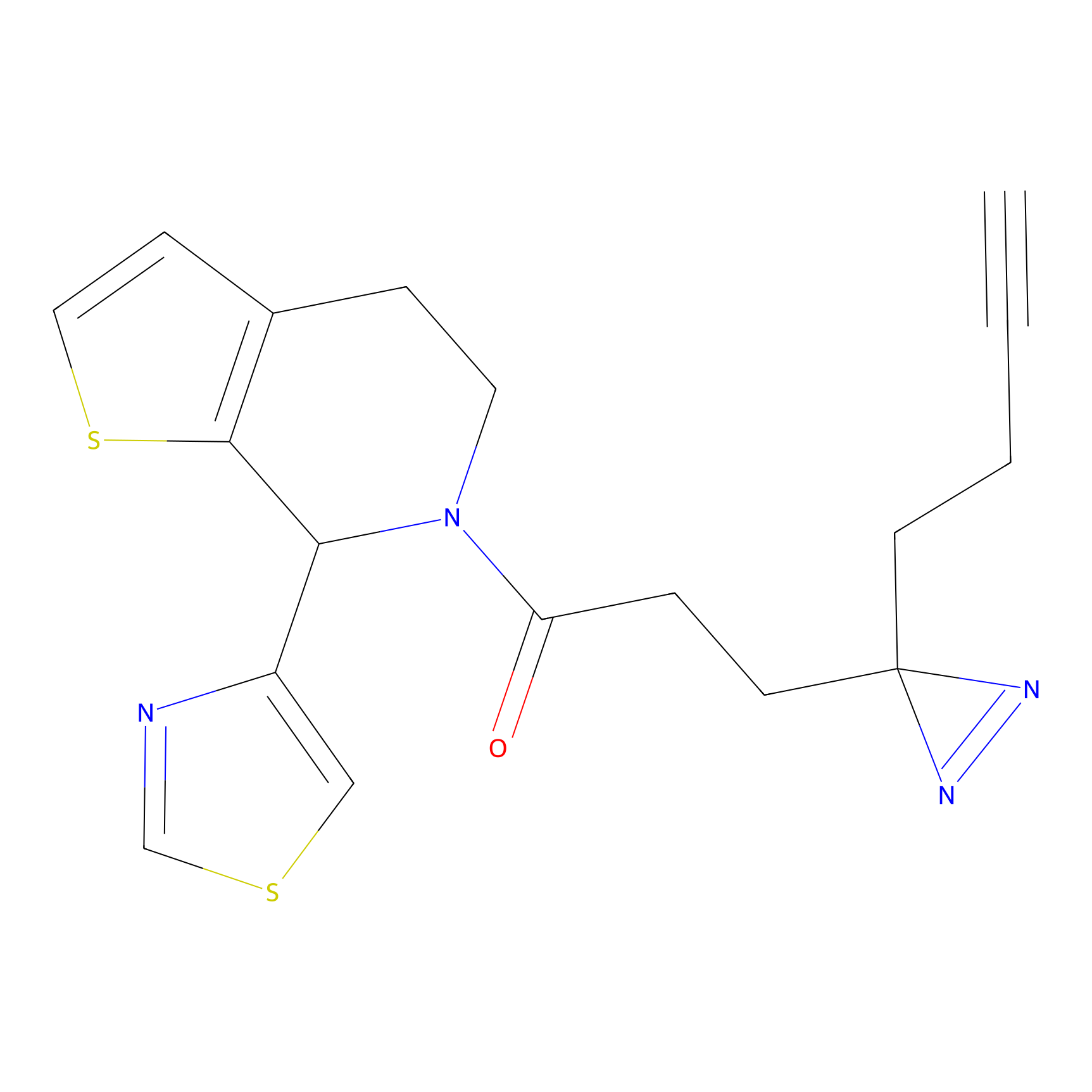
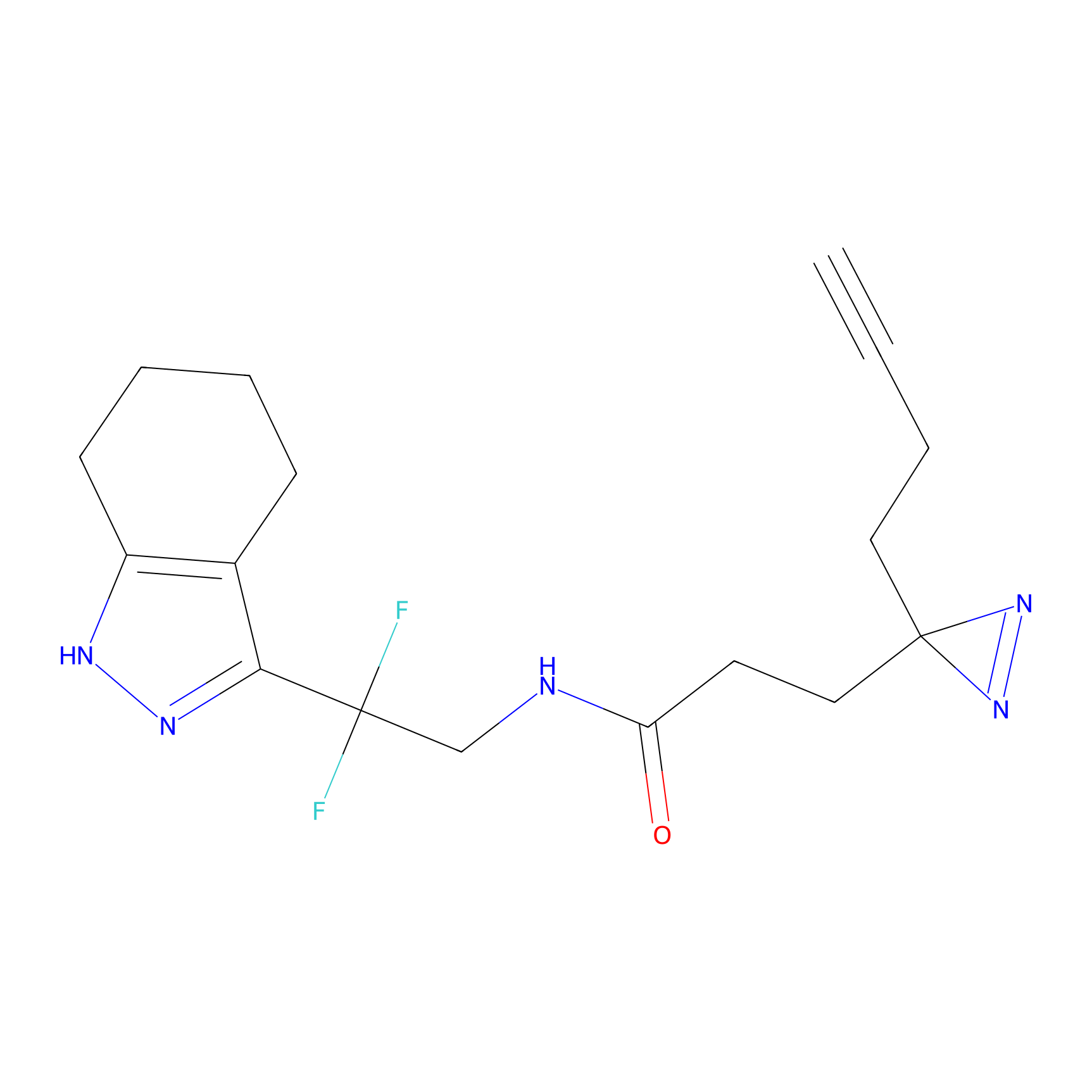
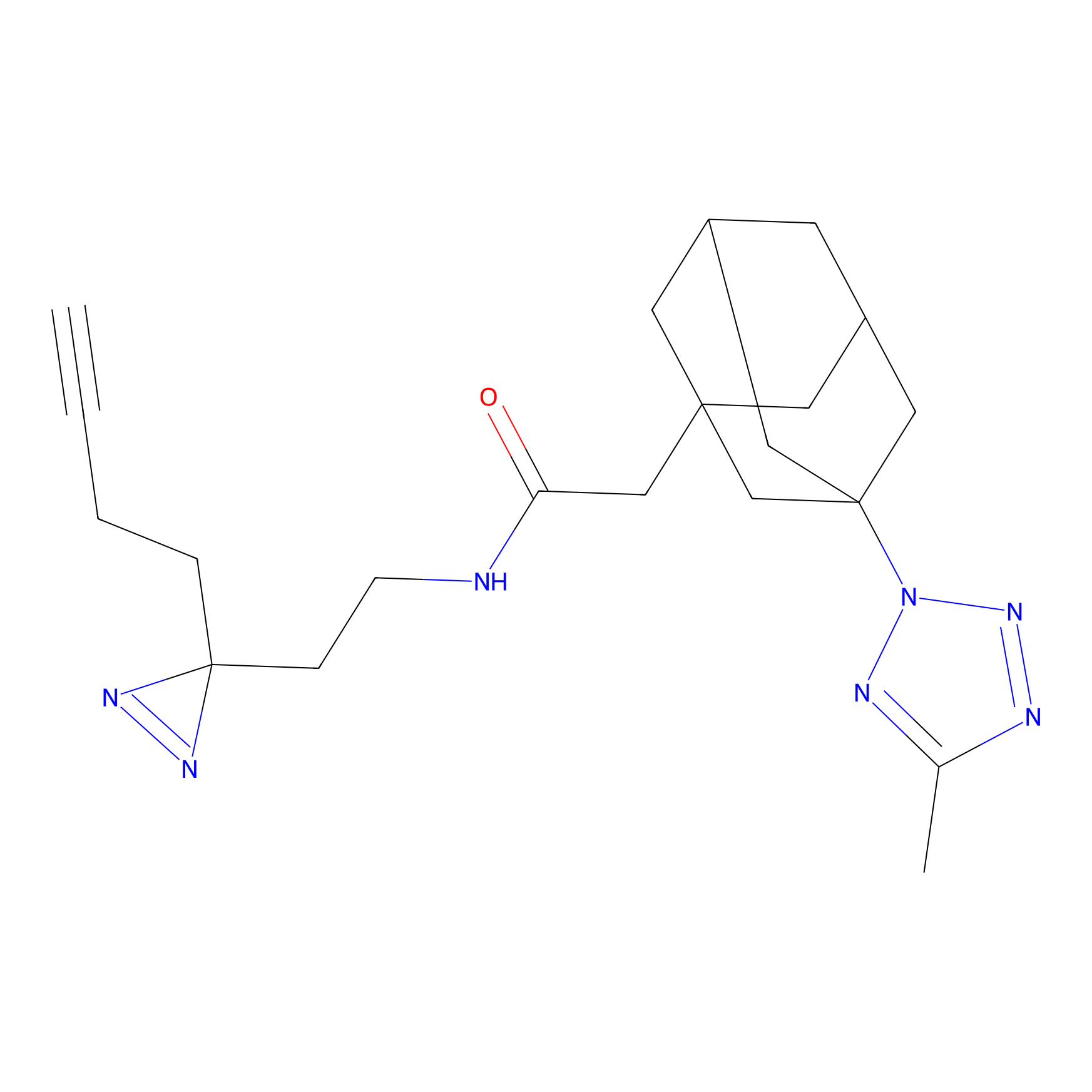
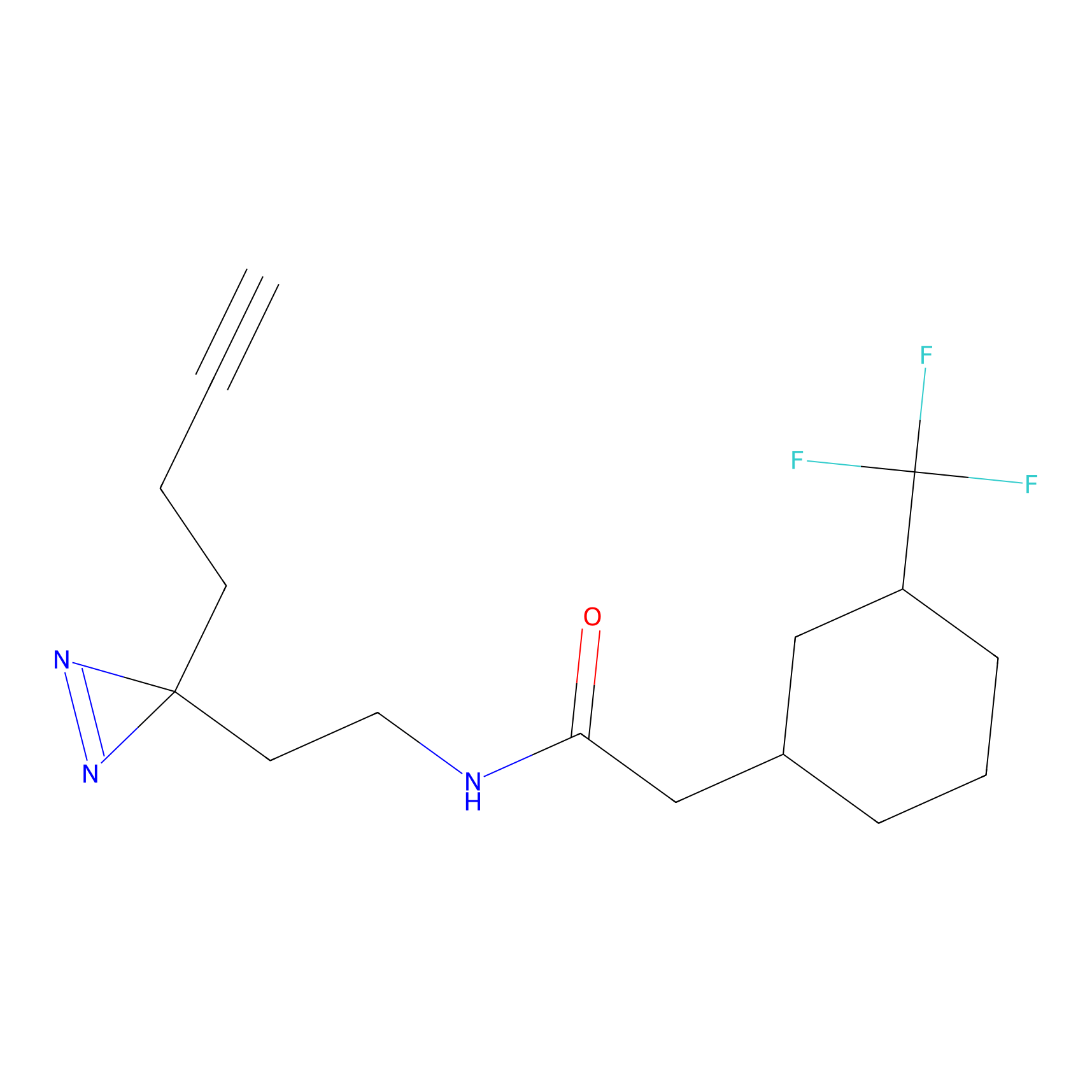
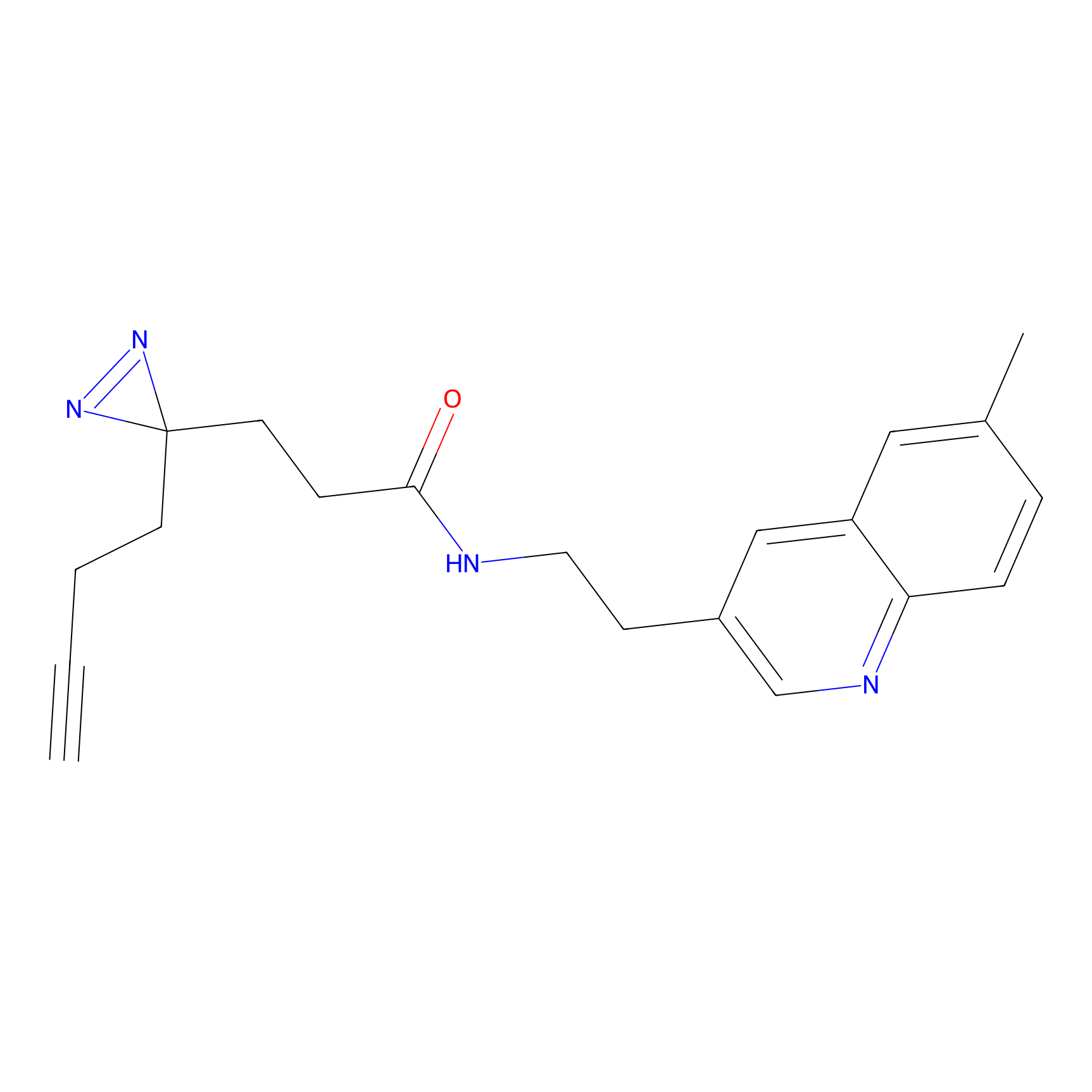
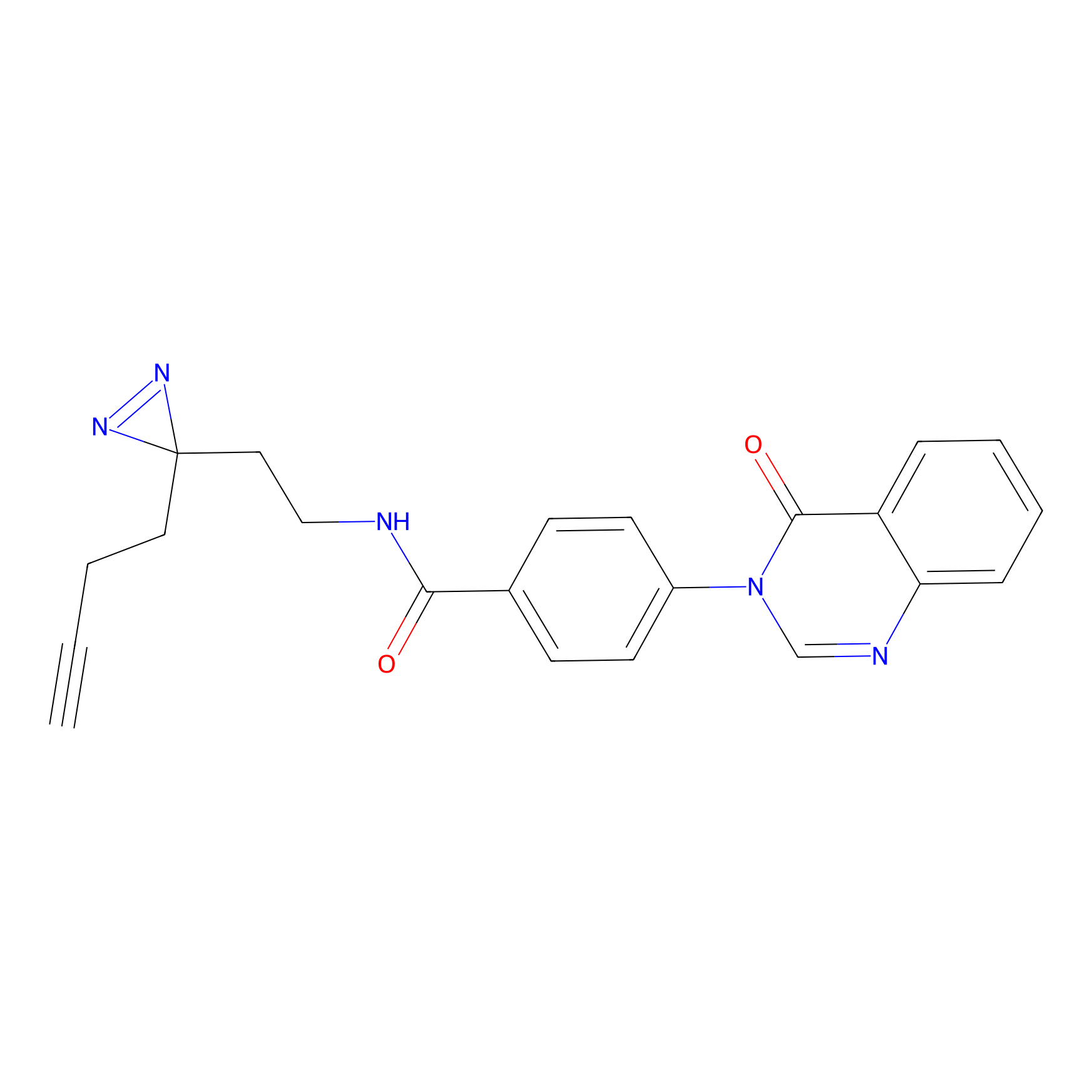
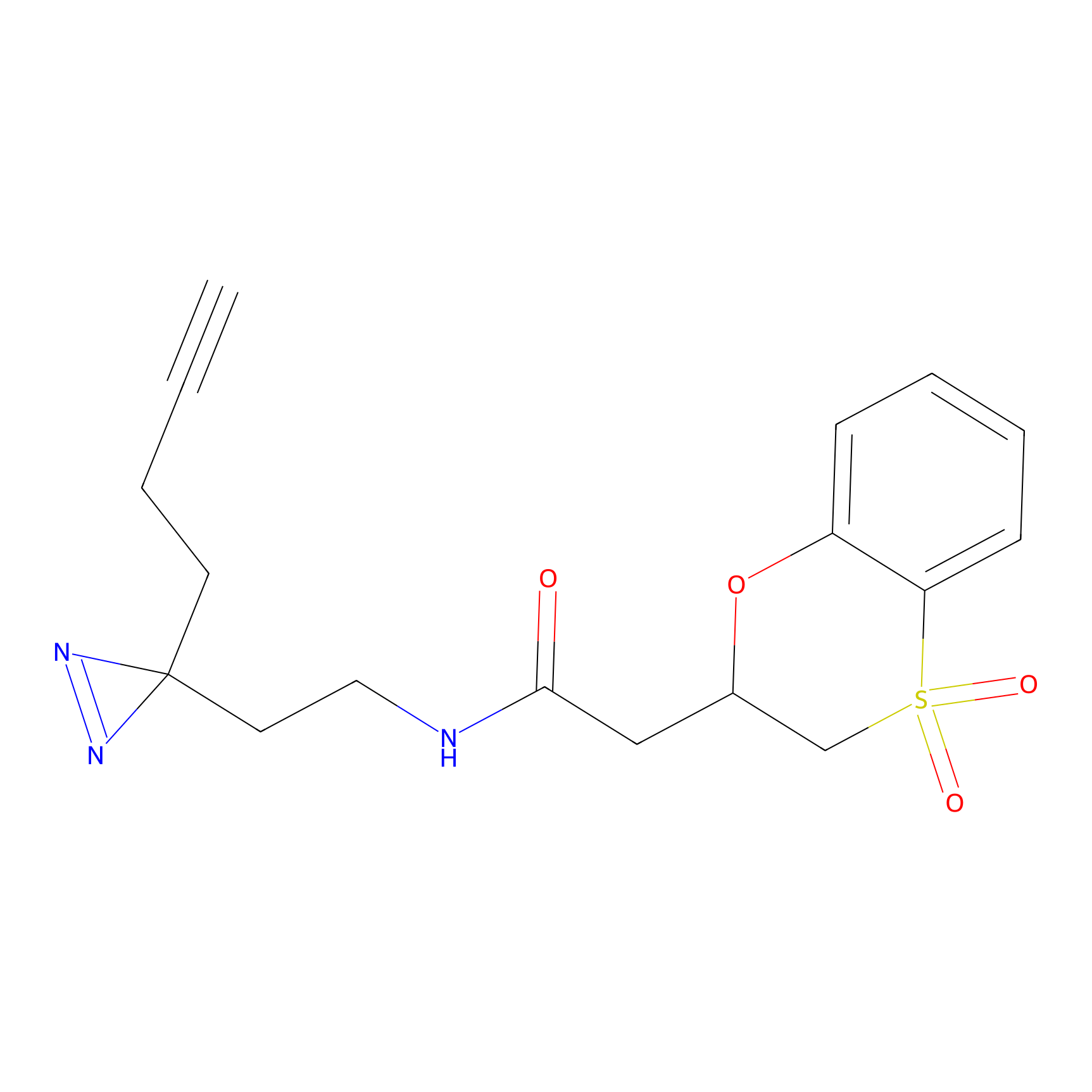
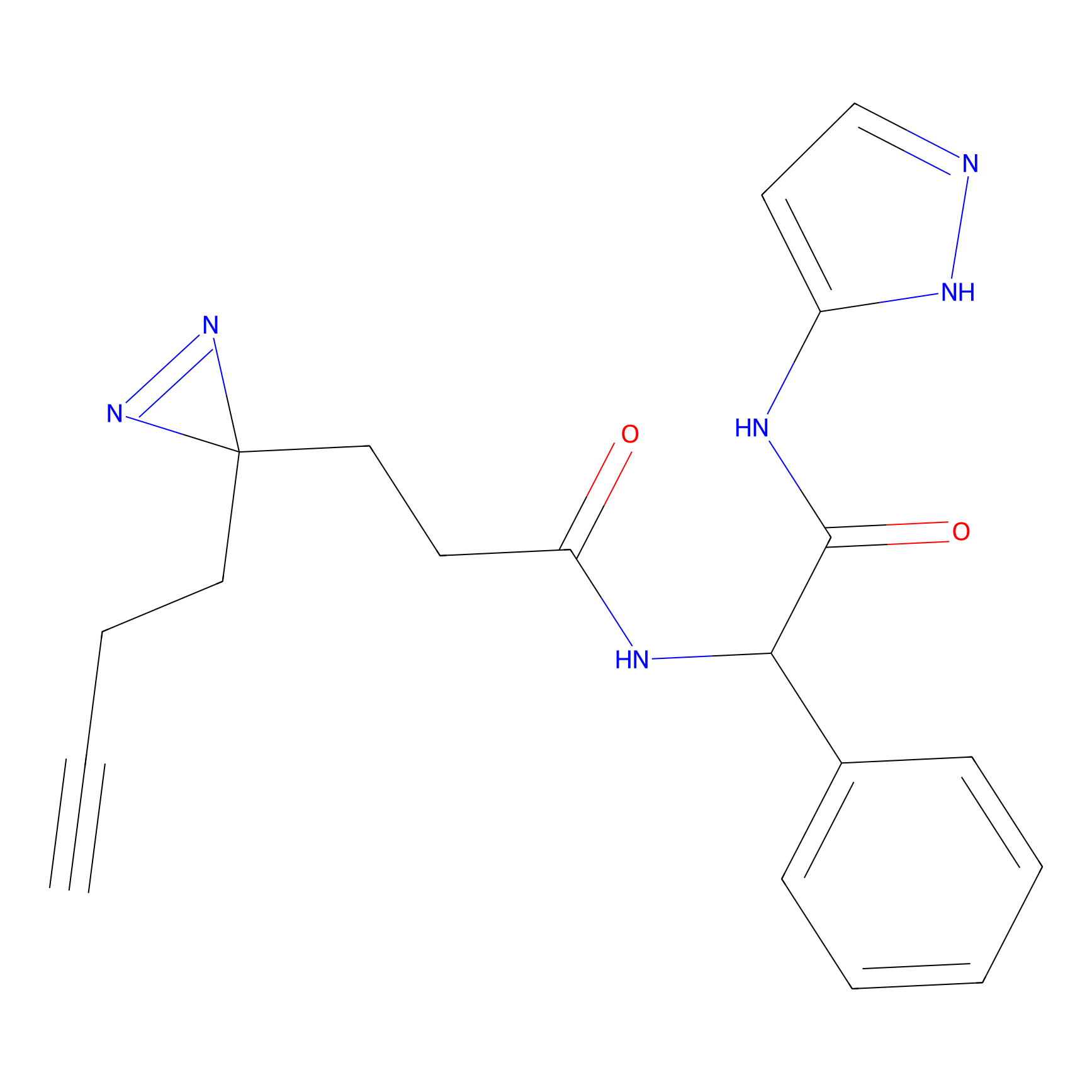
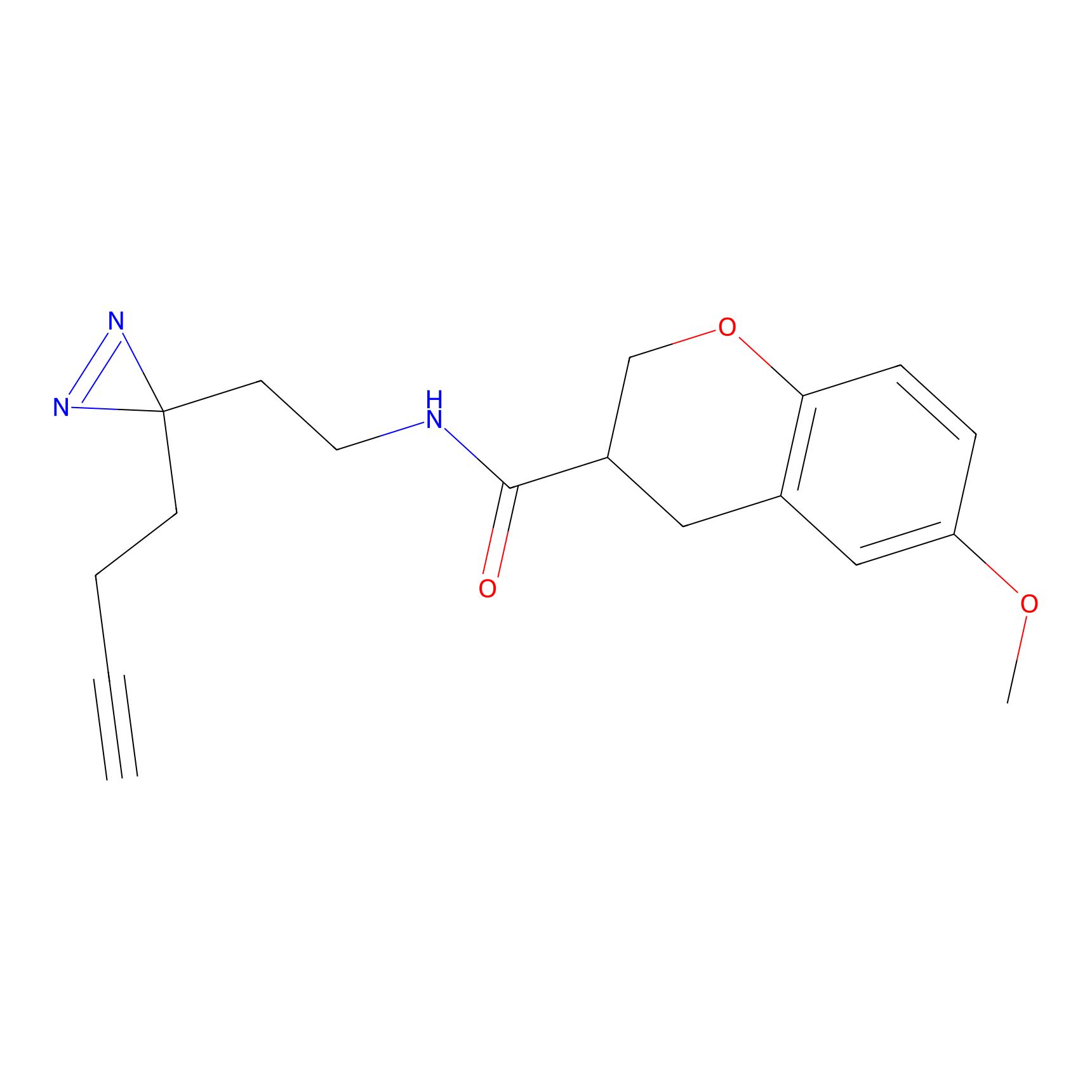
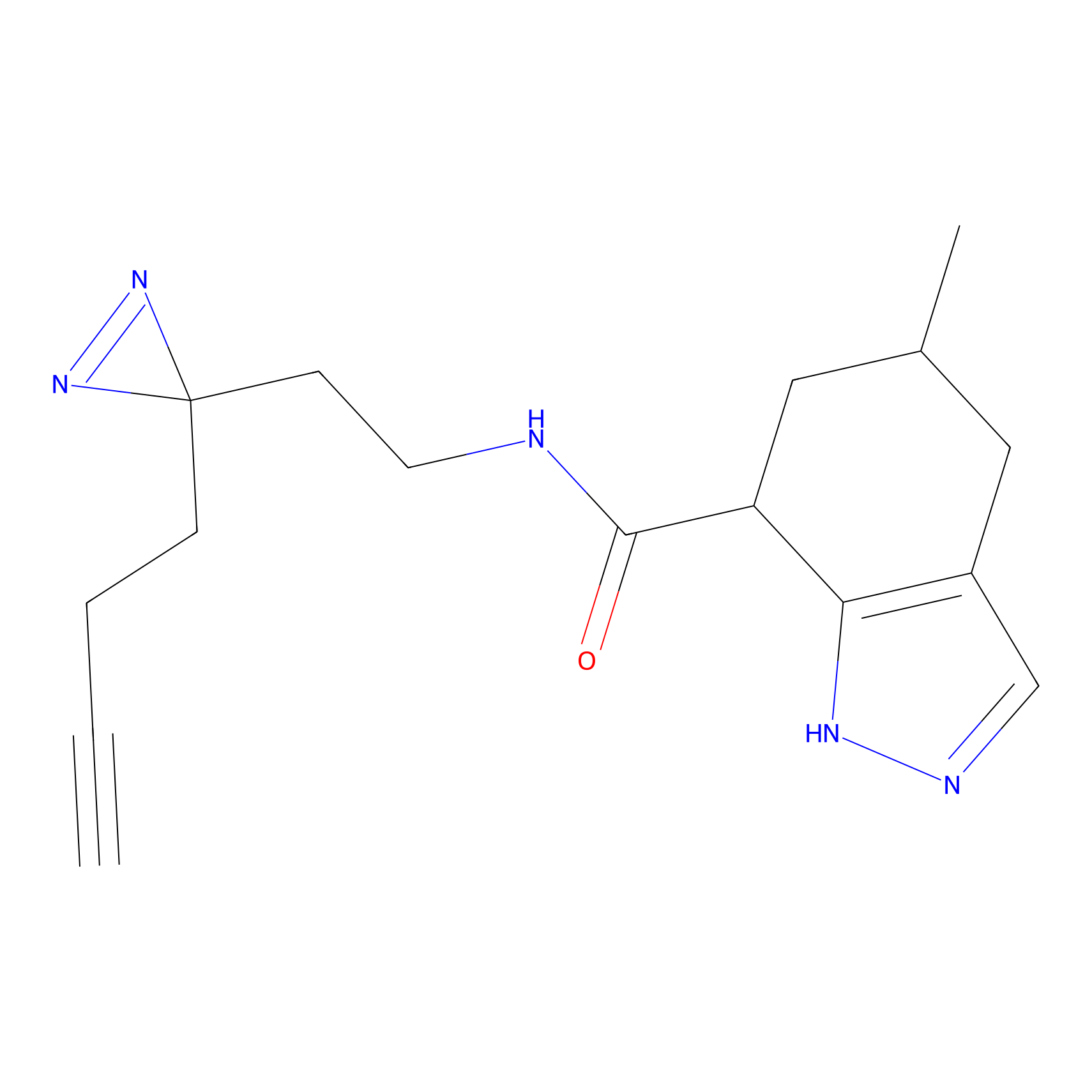
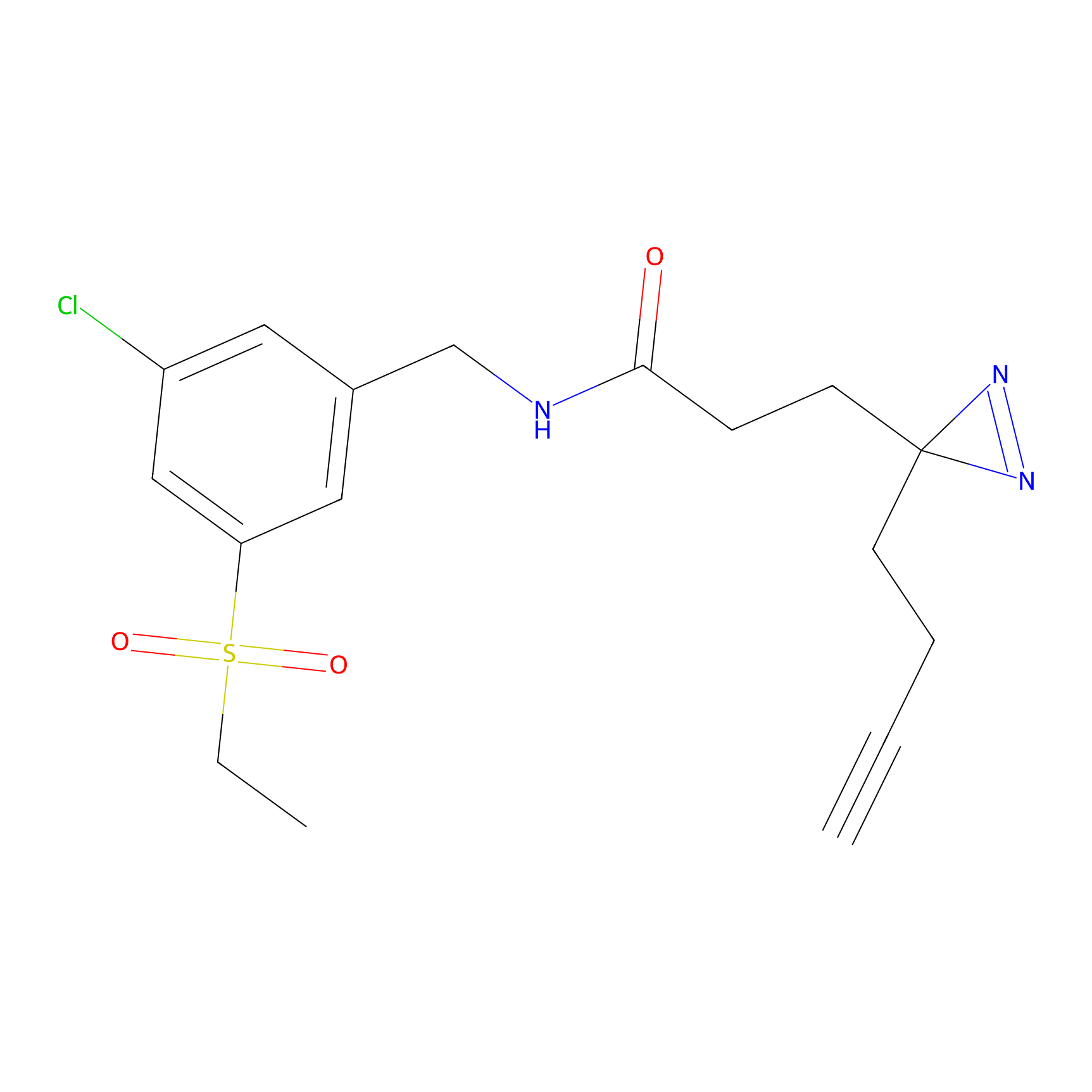
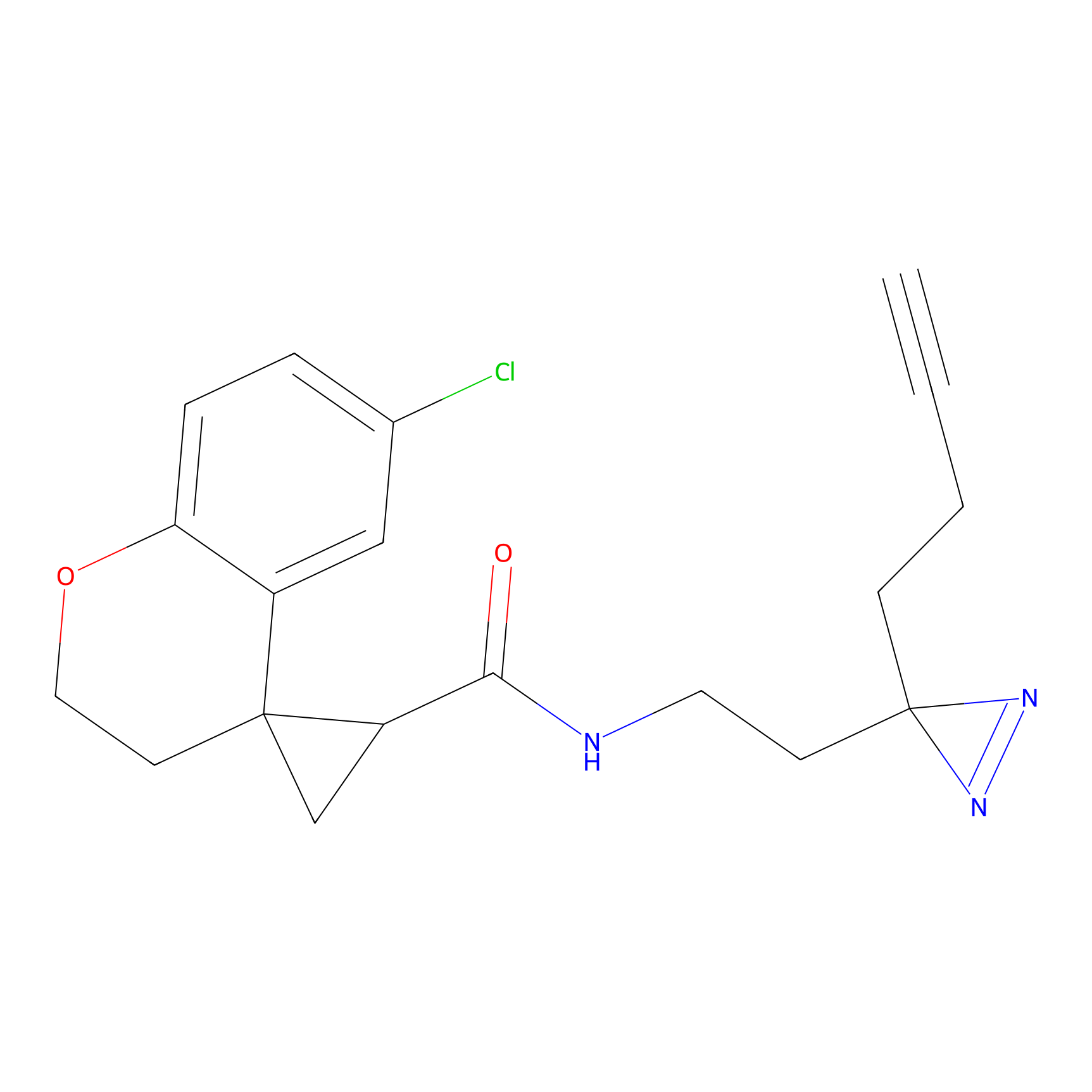
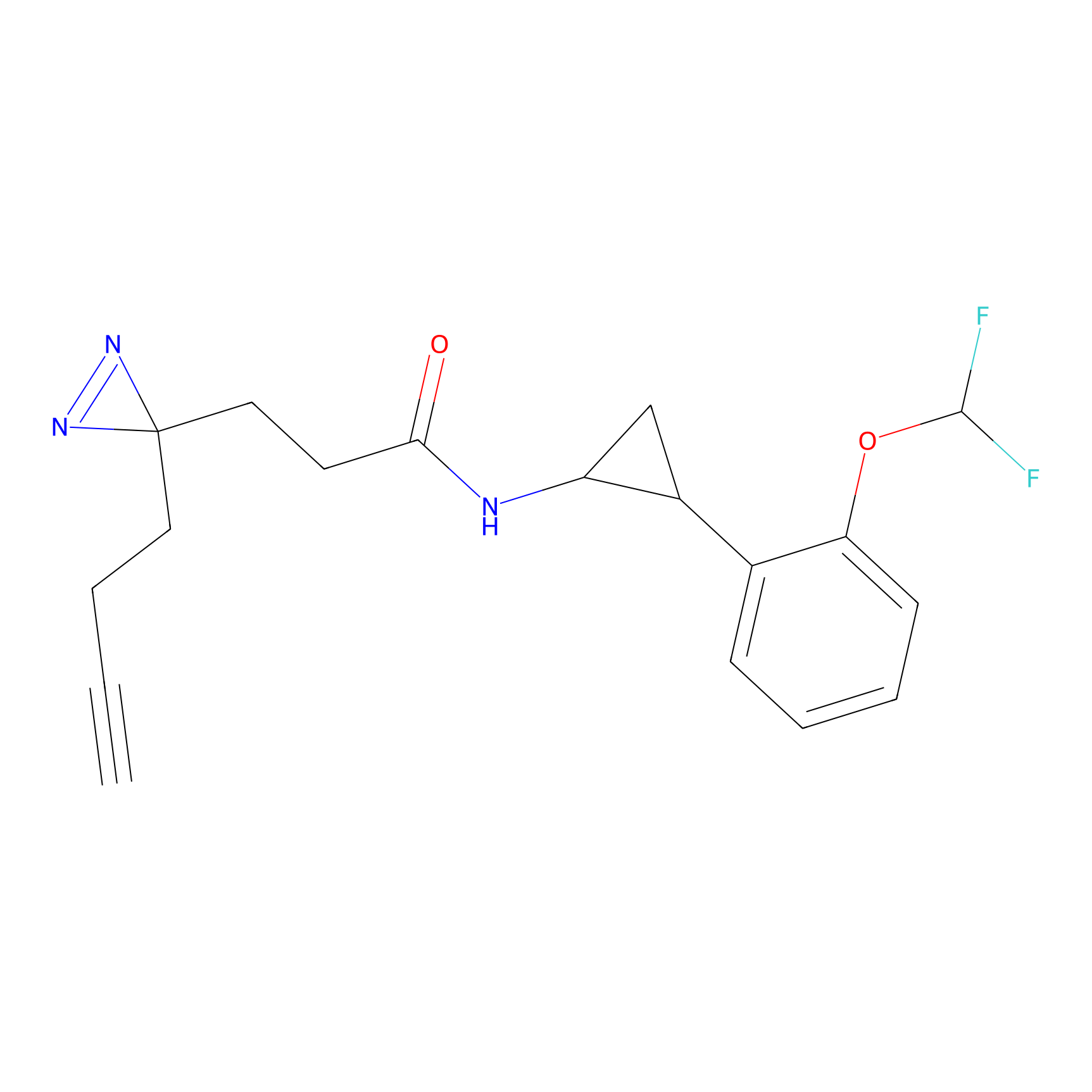
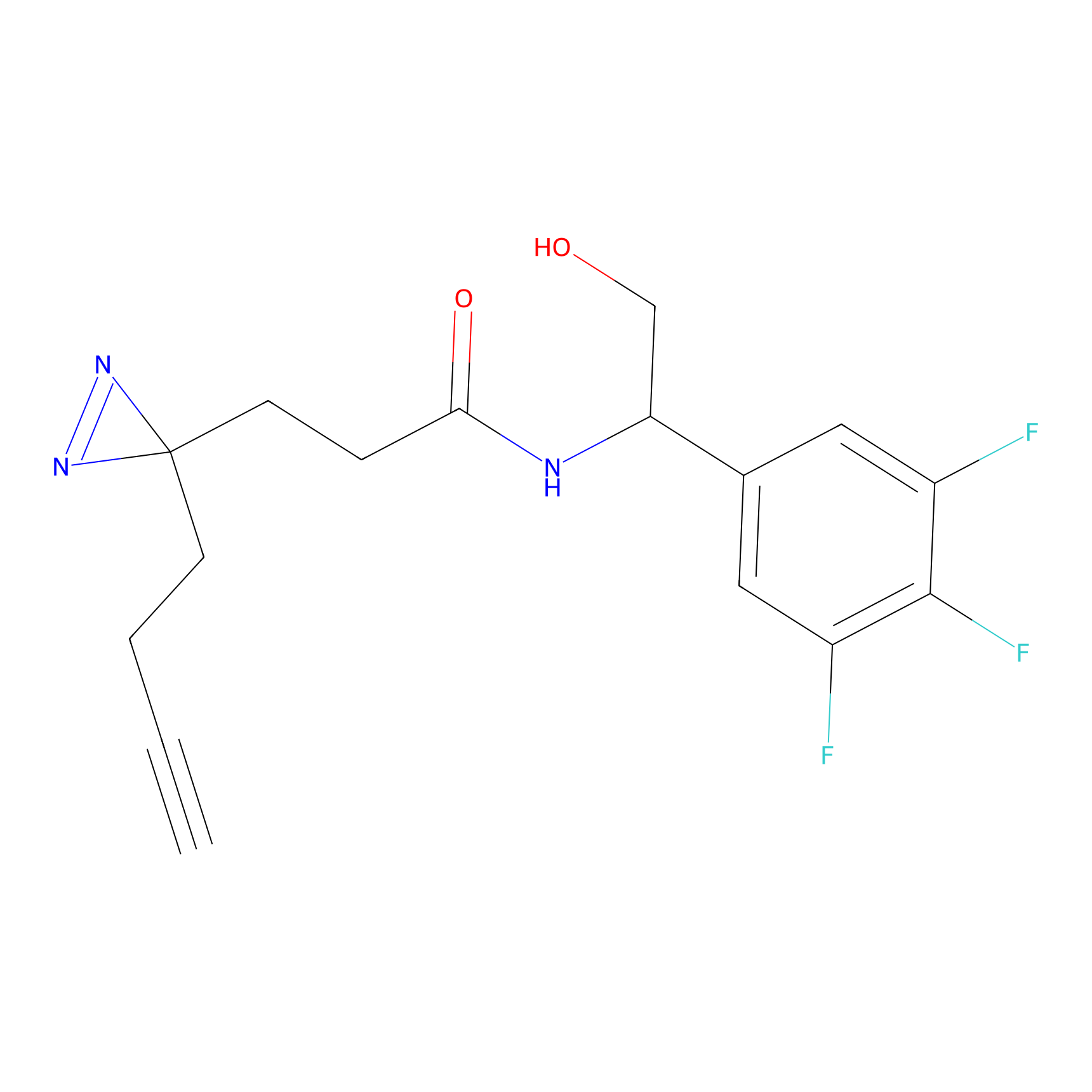
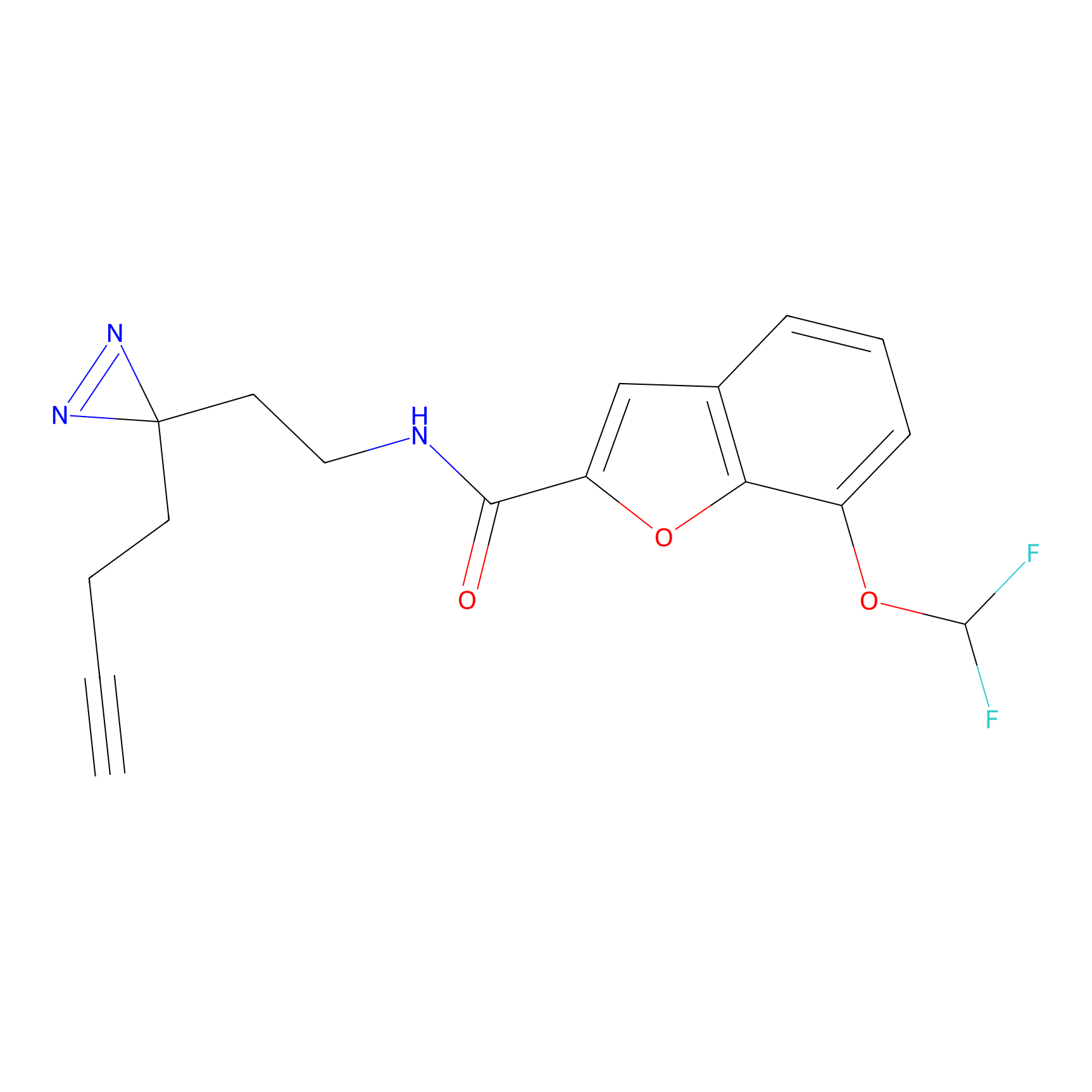
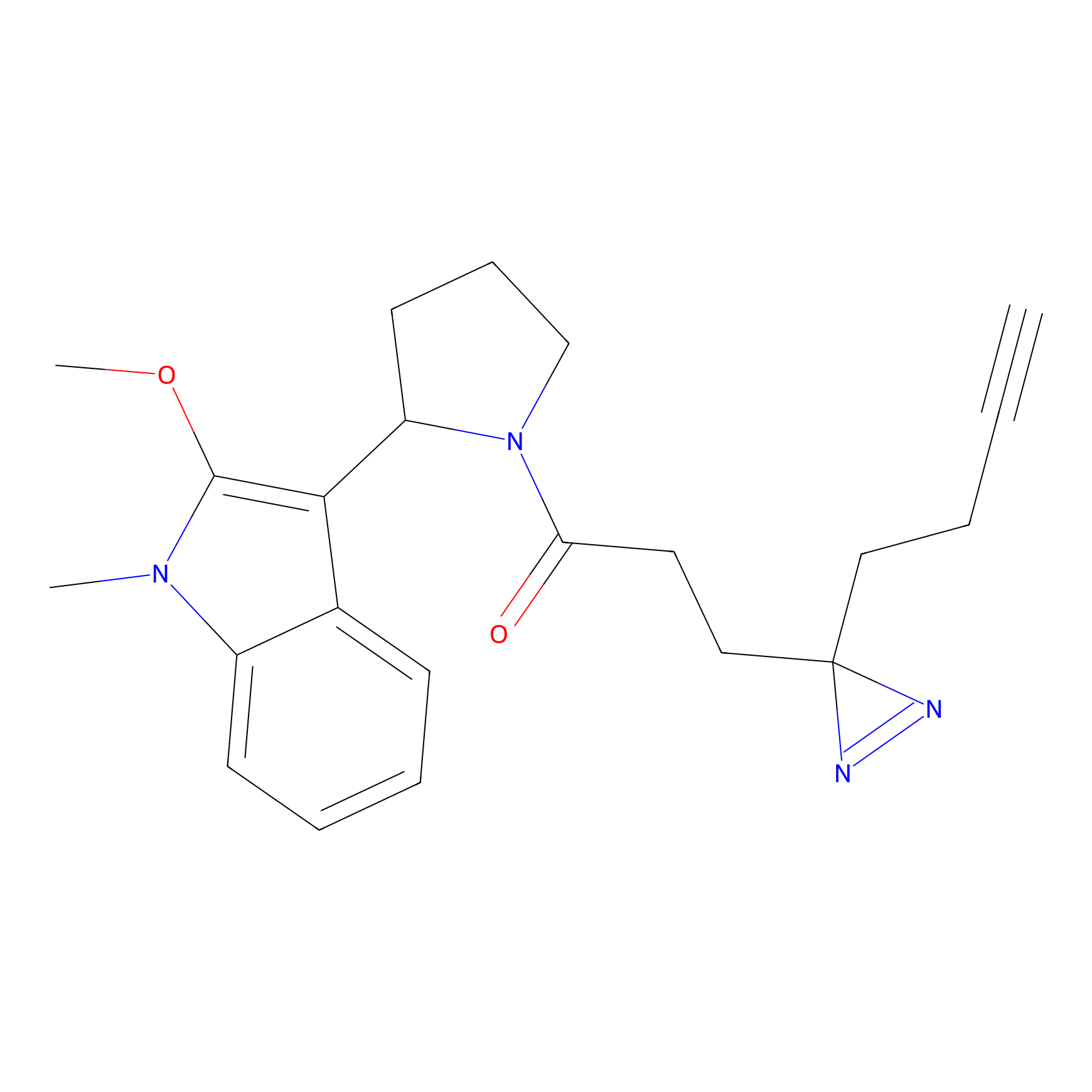
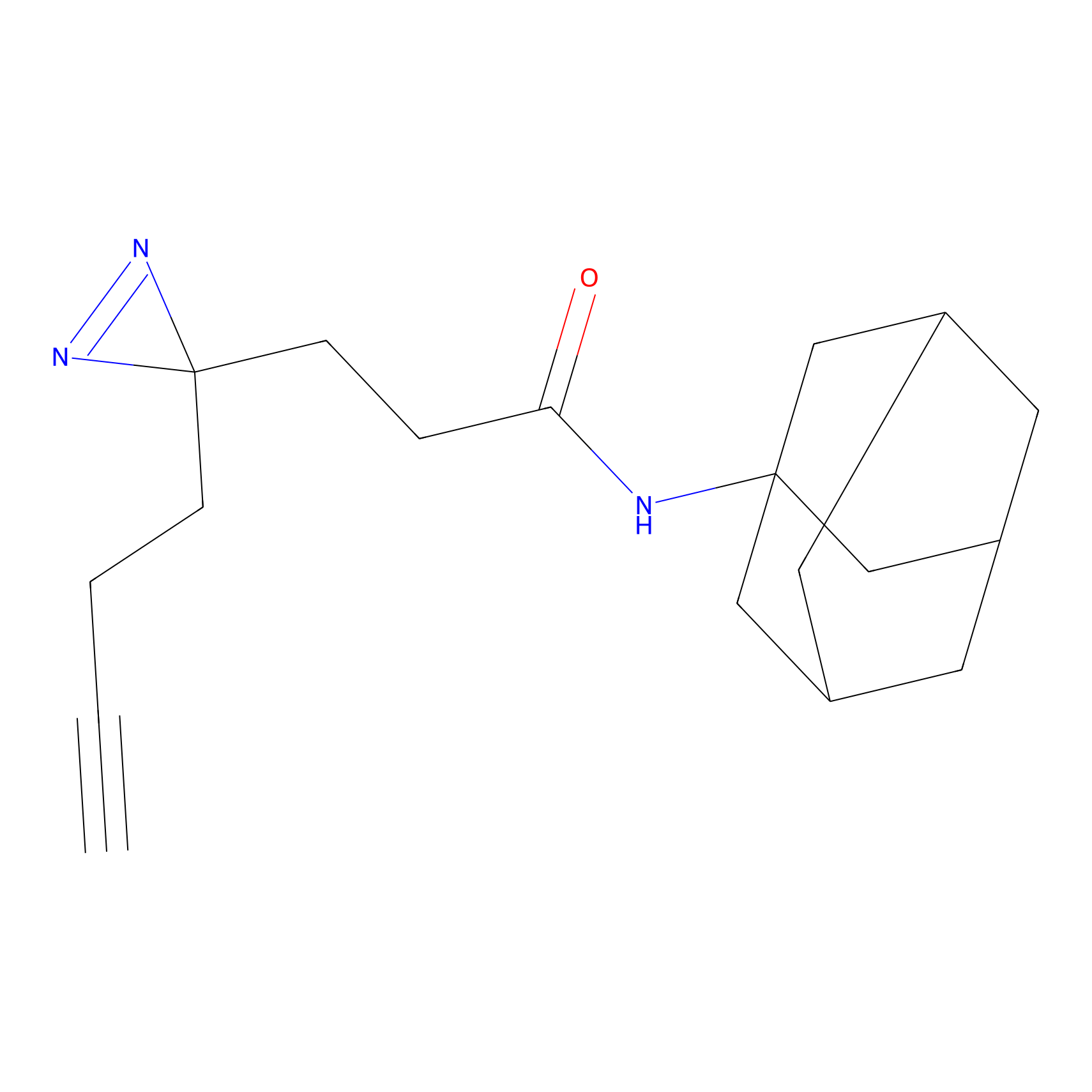
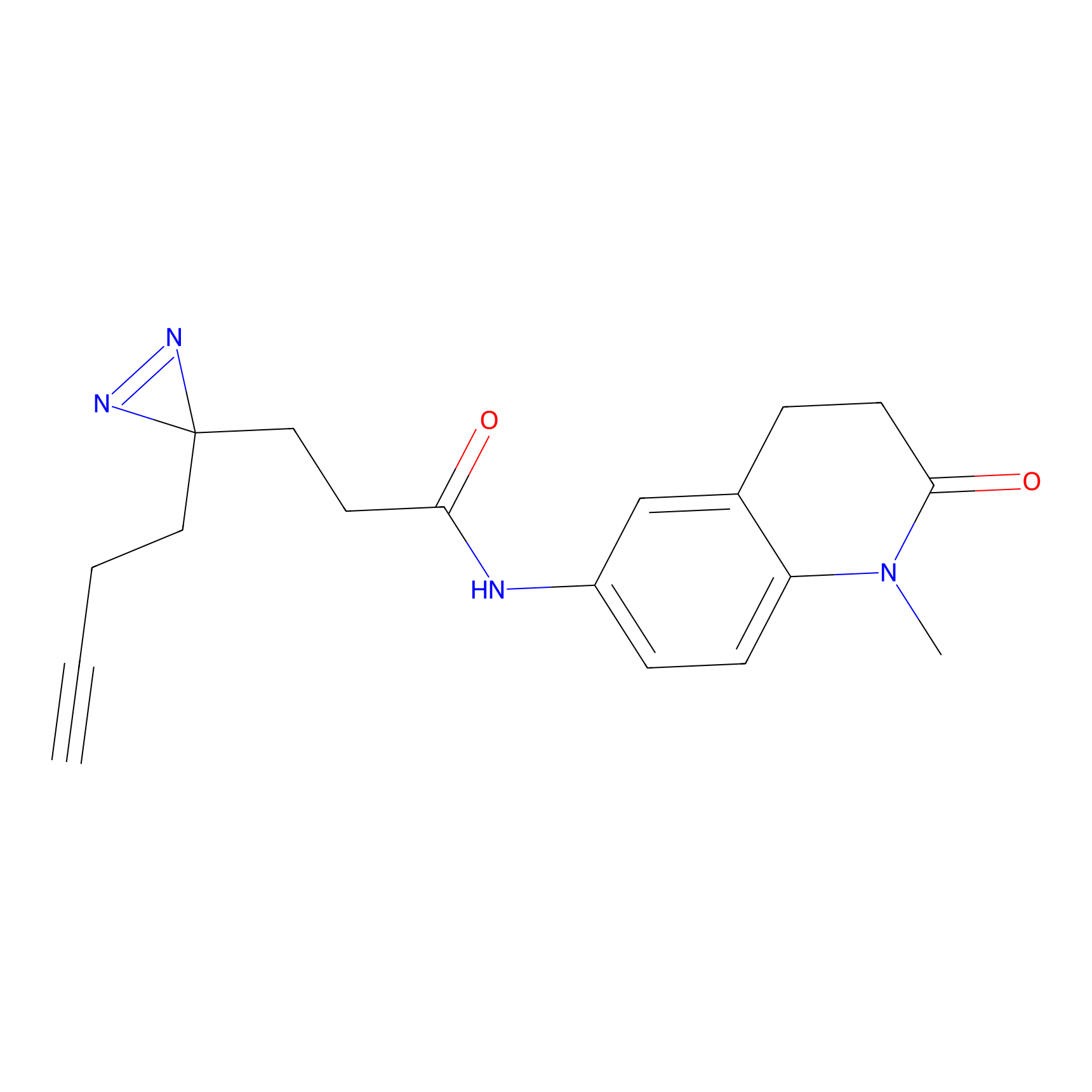
m-APA Probe Info |
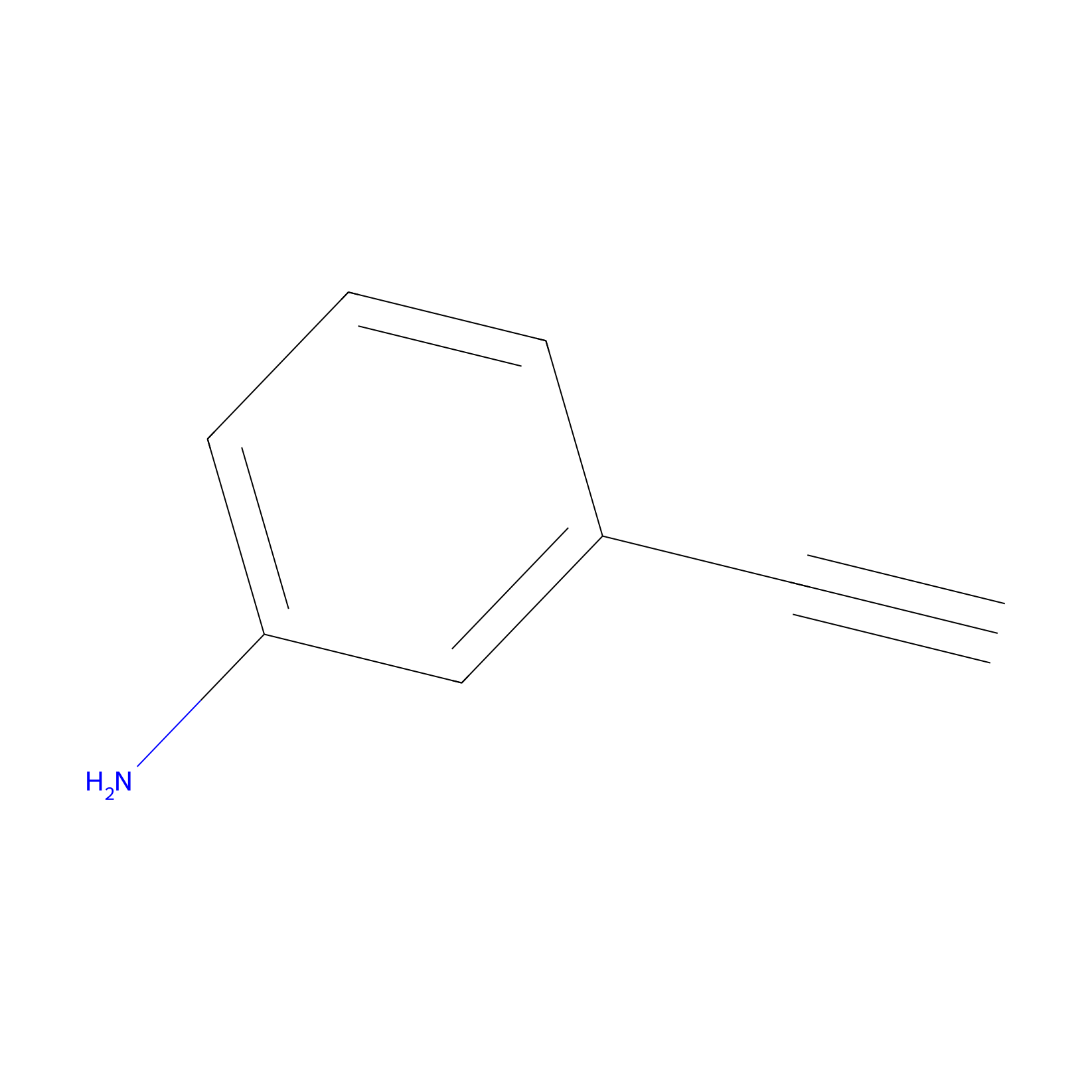 |
15.00 | LDD0402 | [1] | |
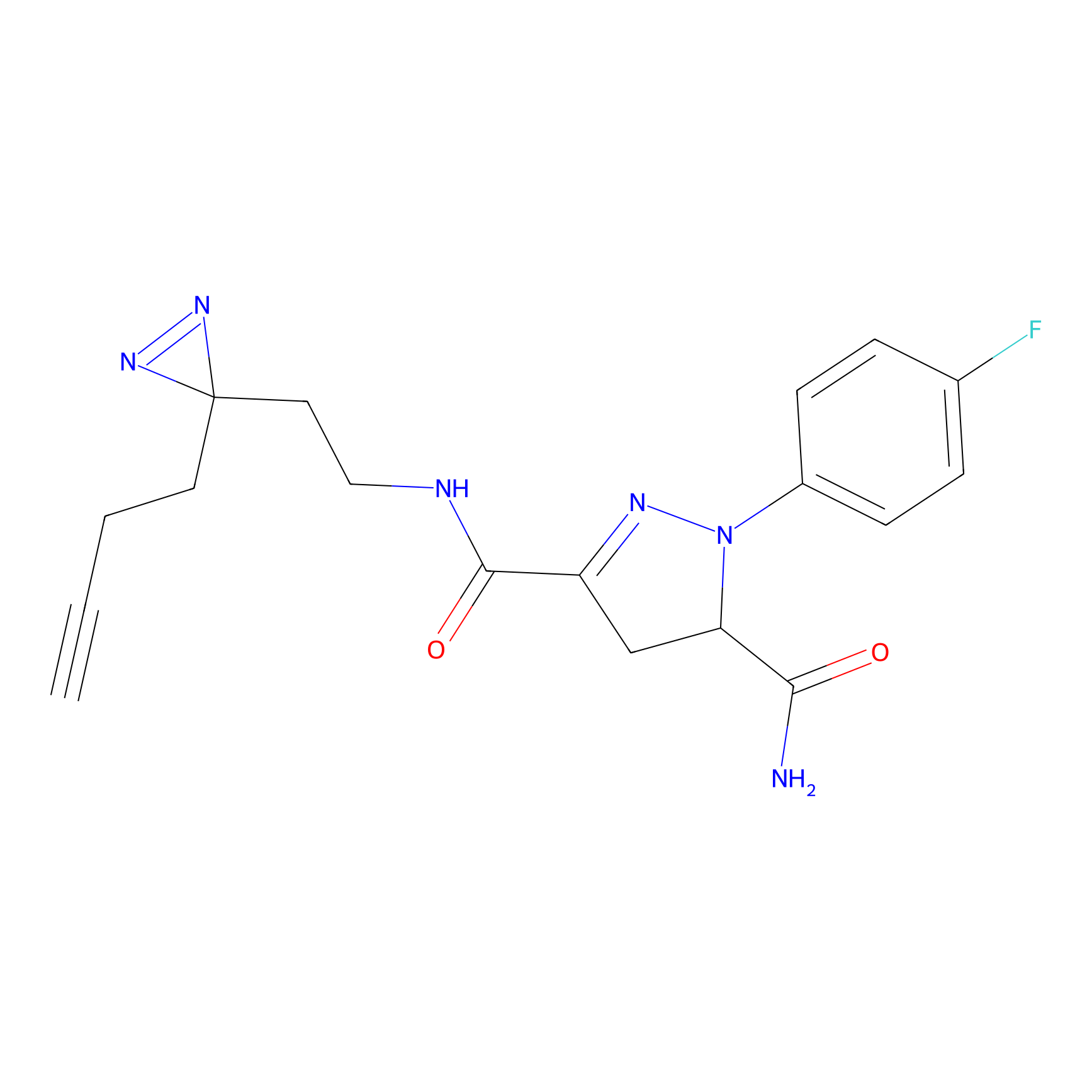
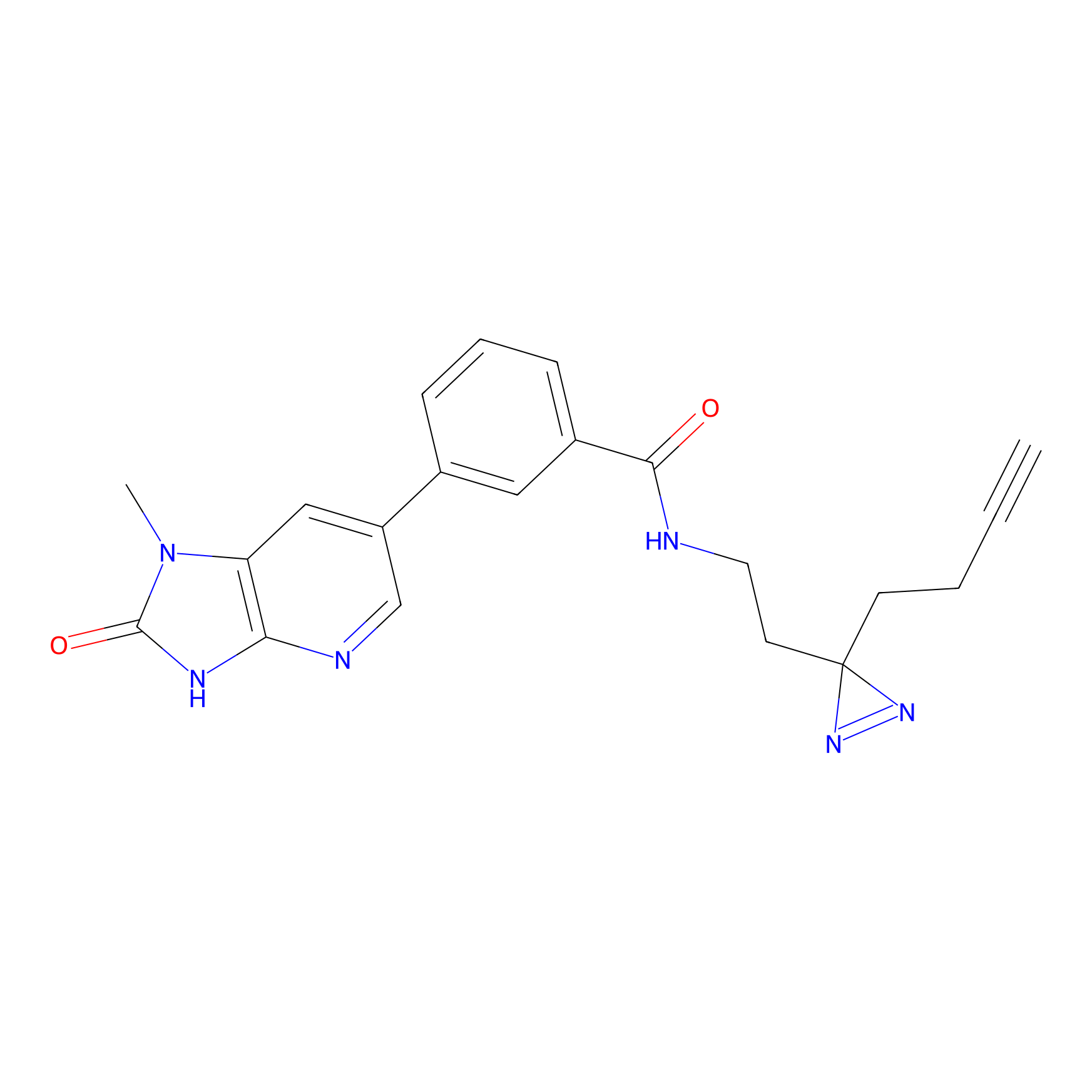
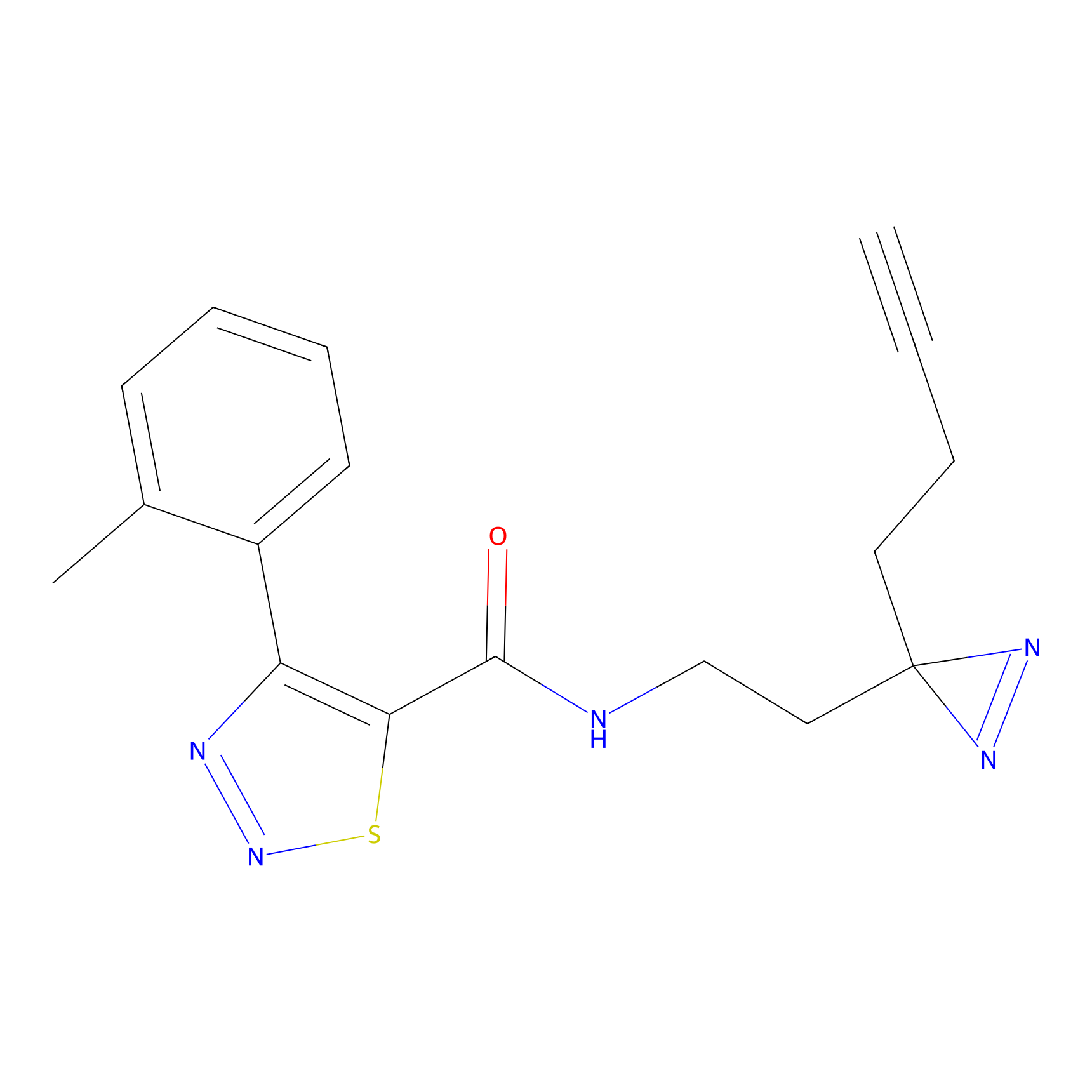
|
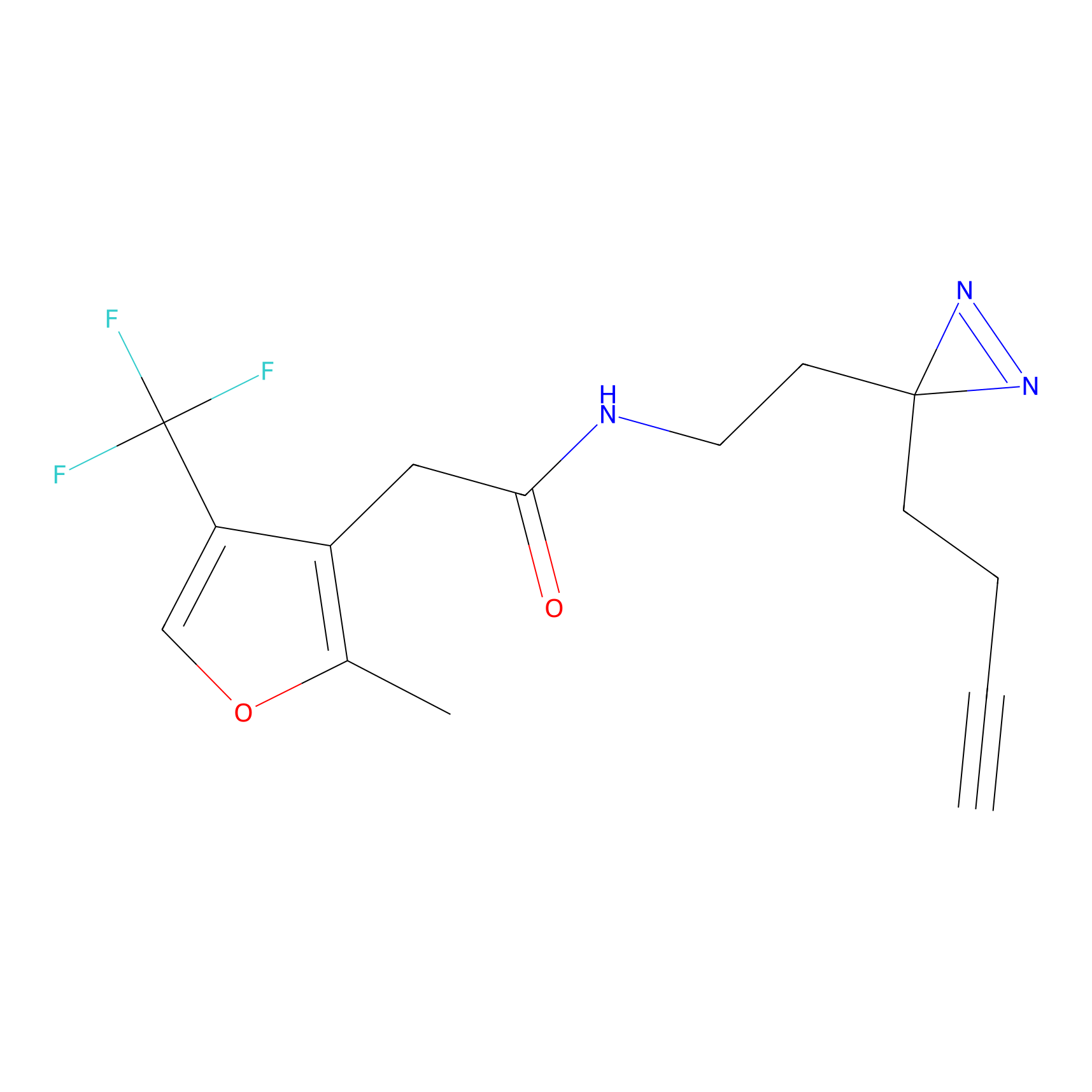
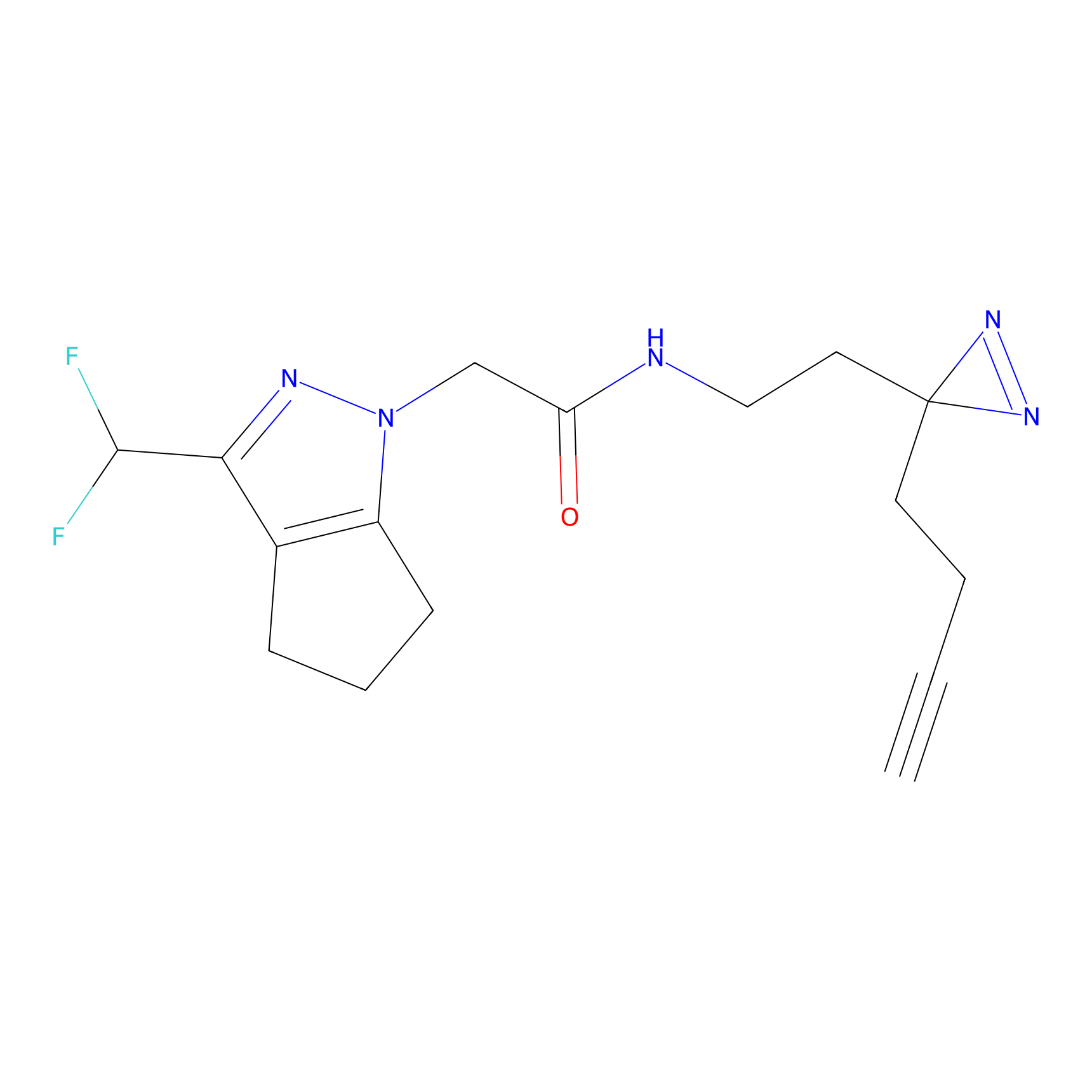
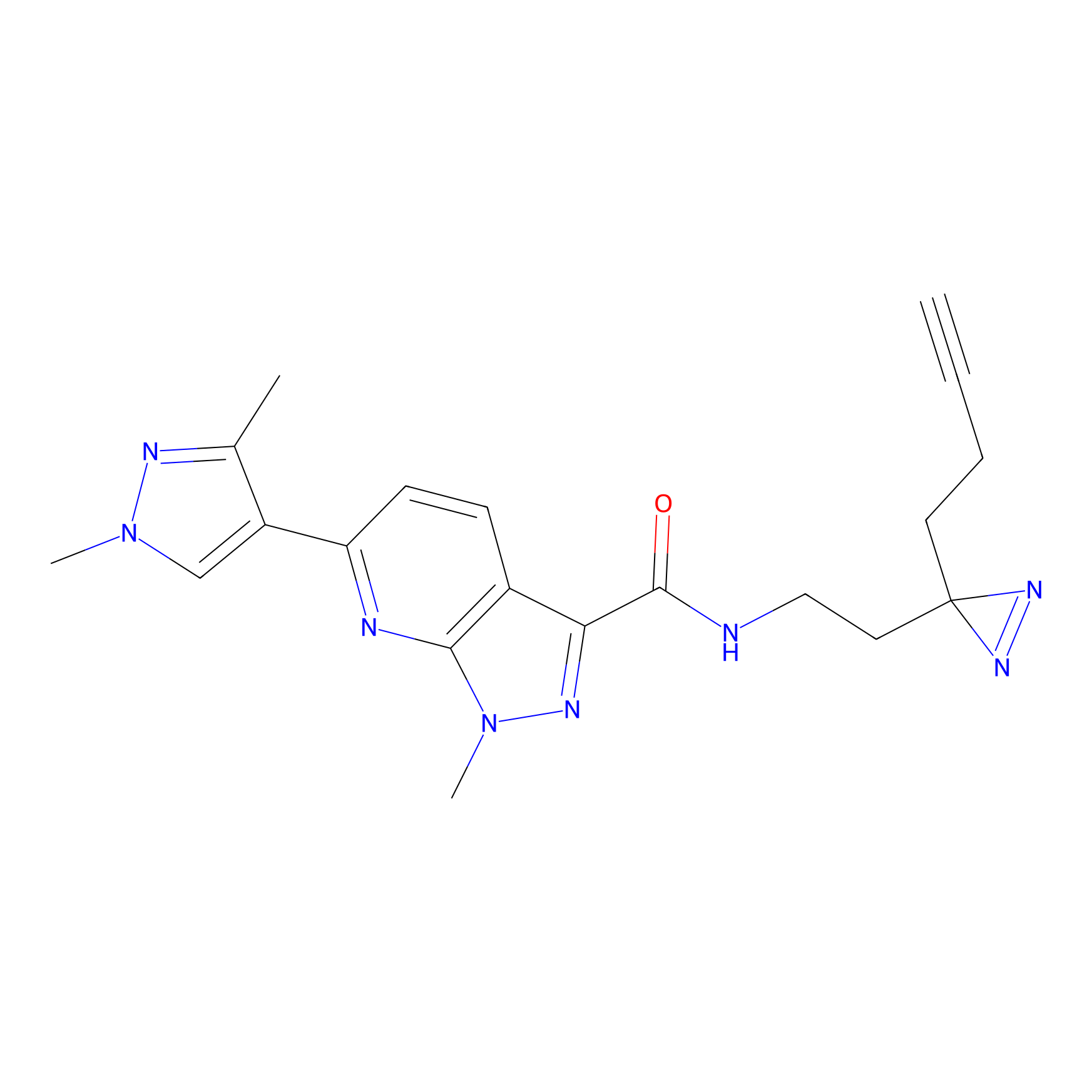
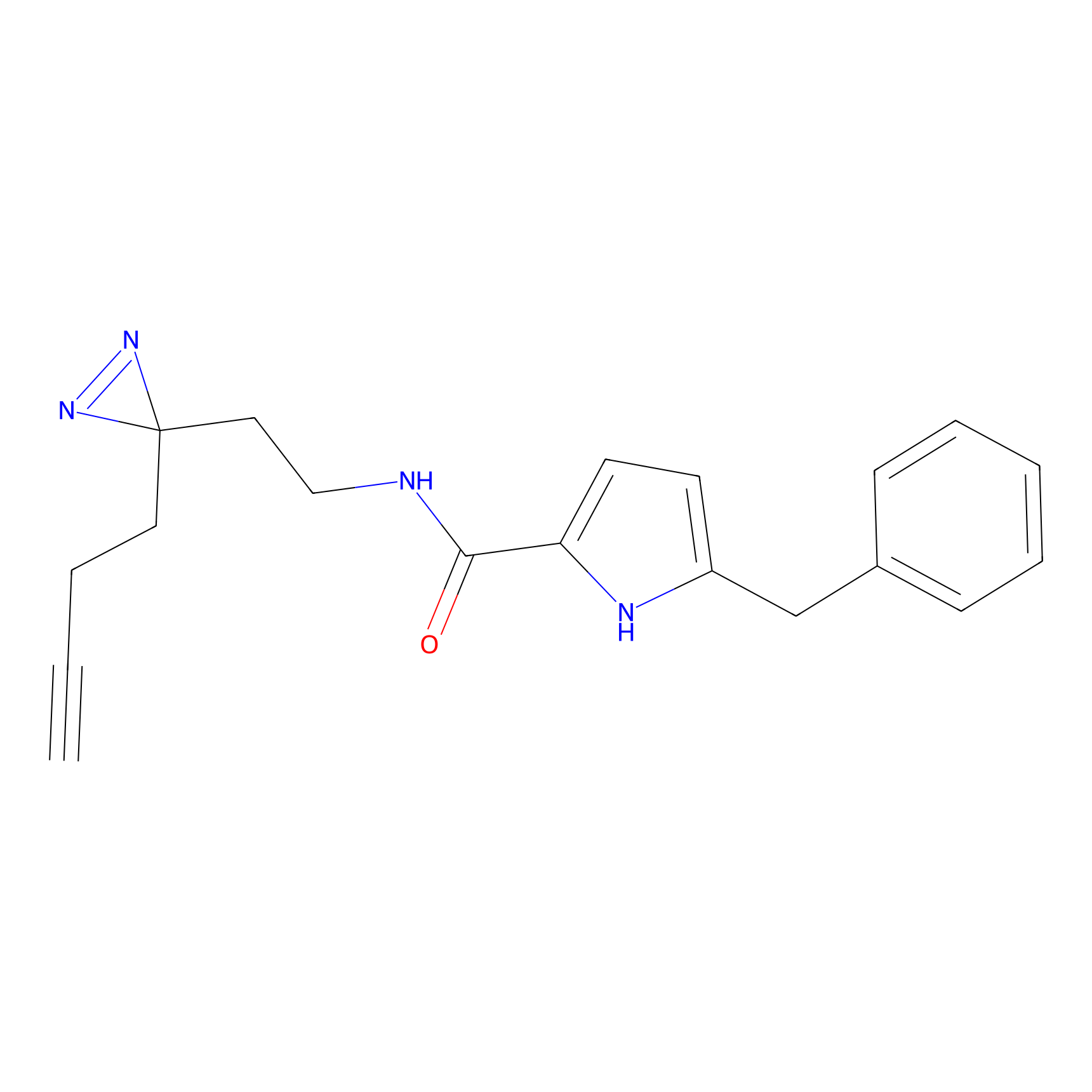
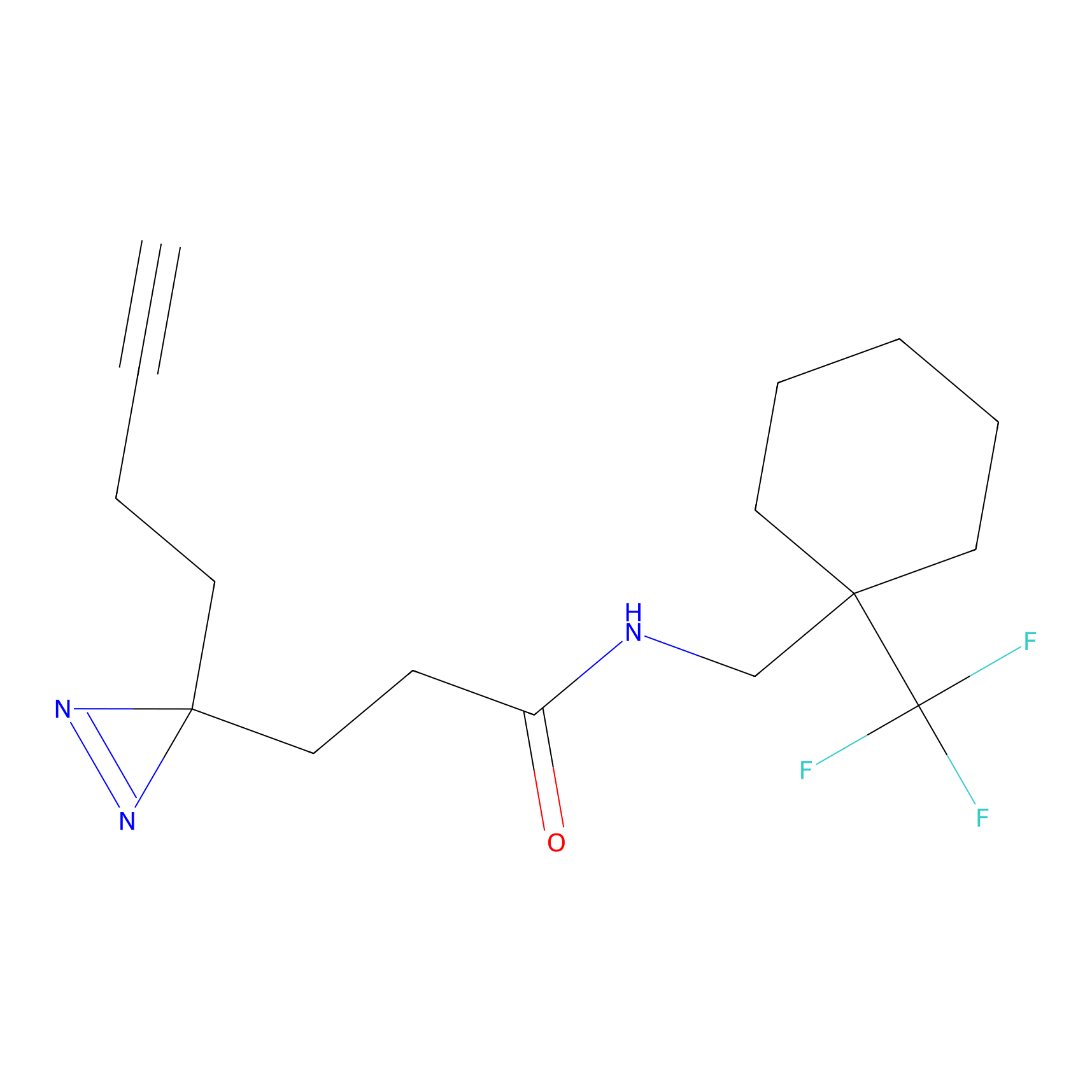
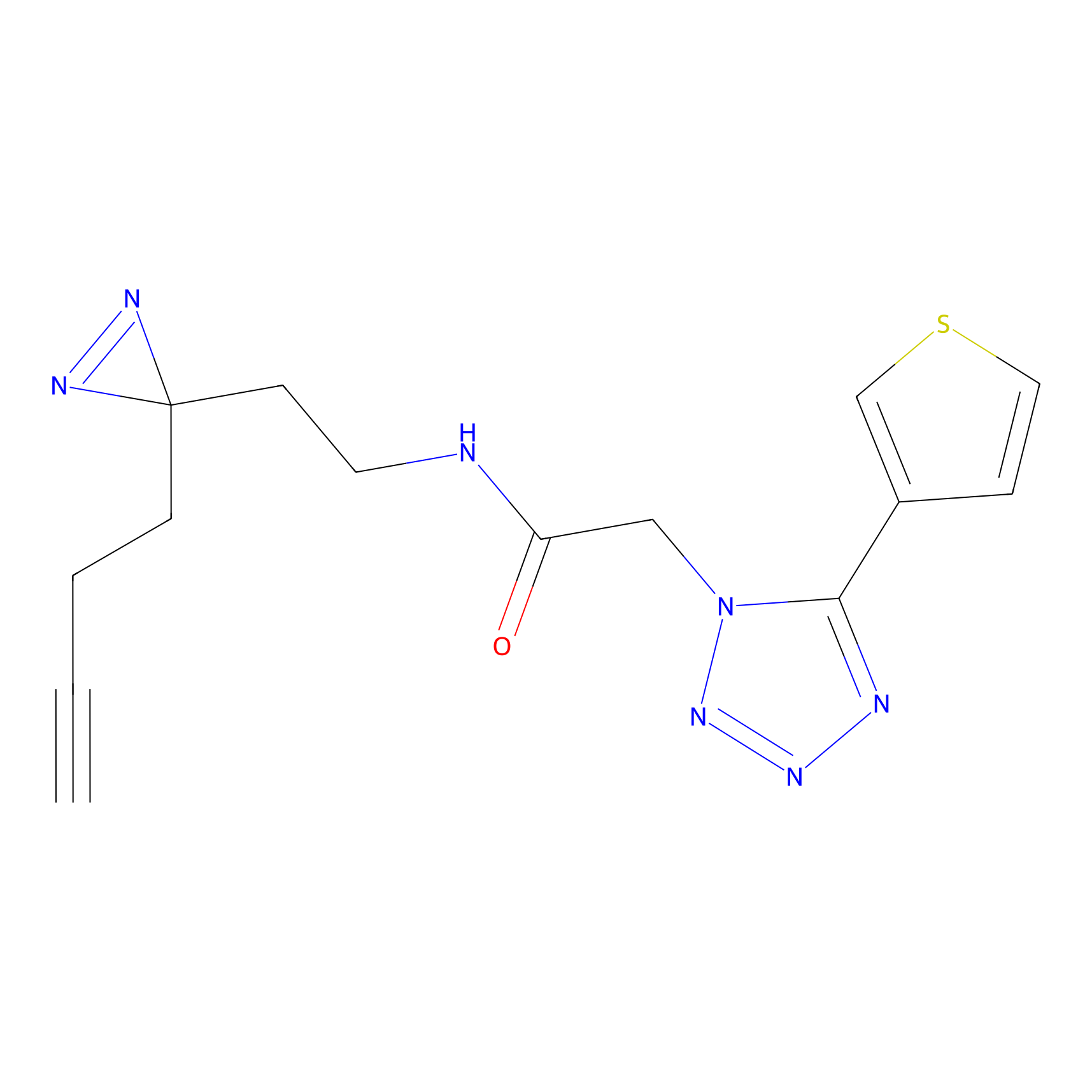
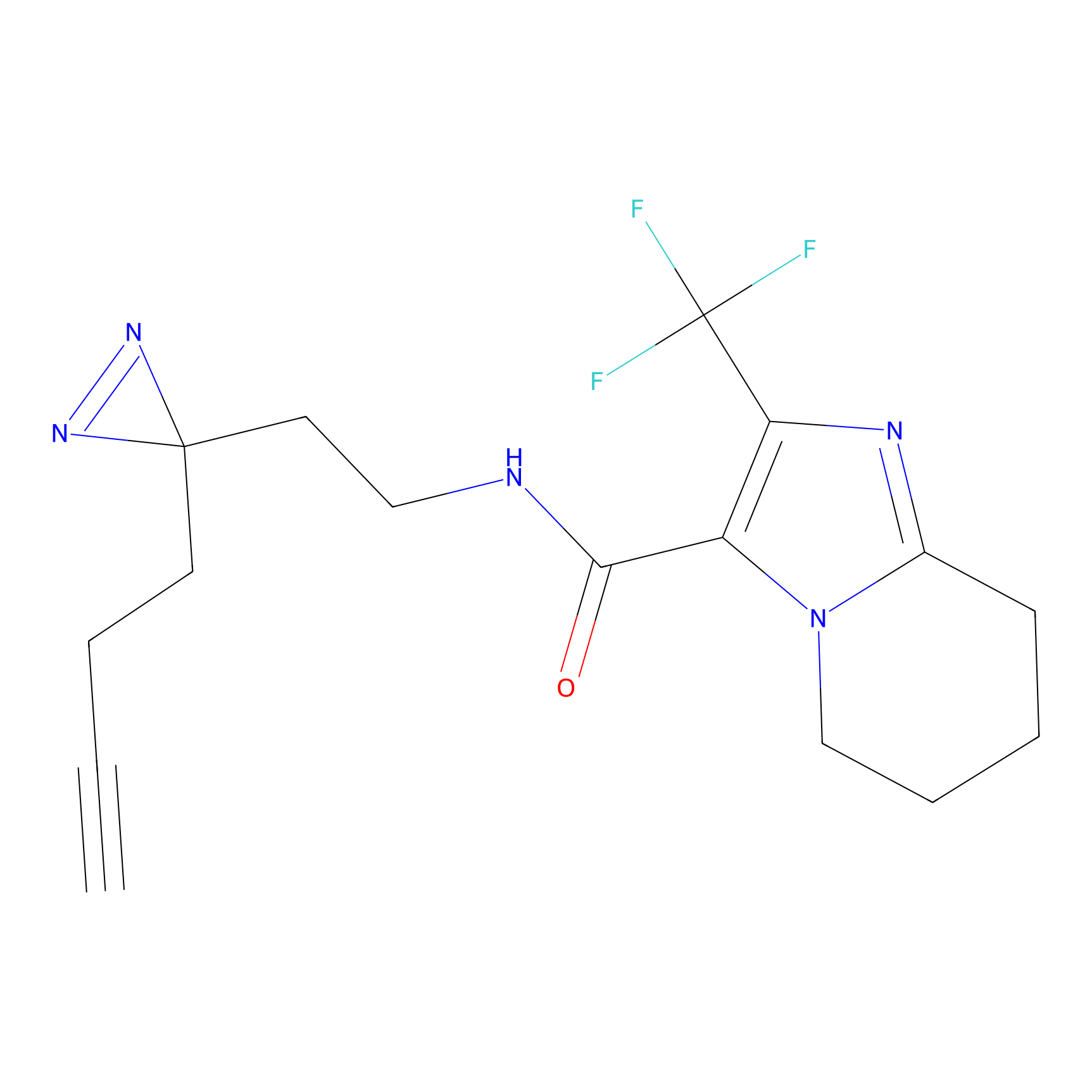
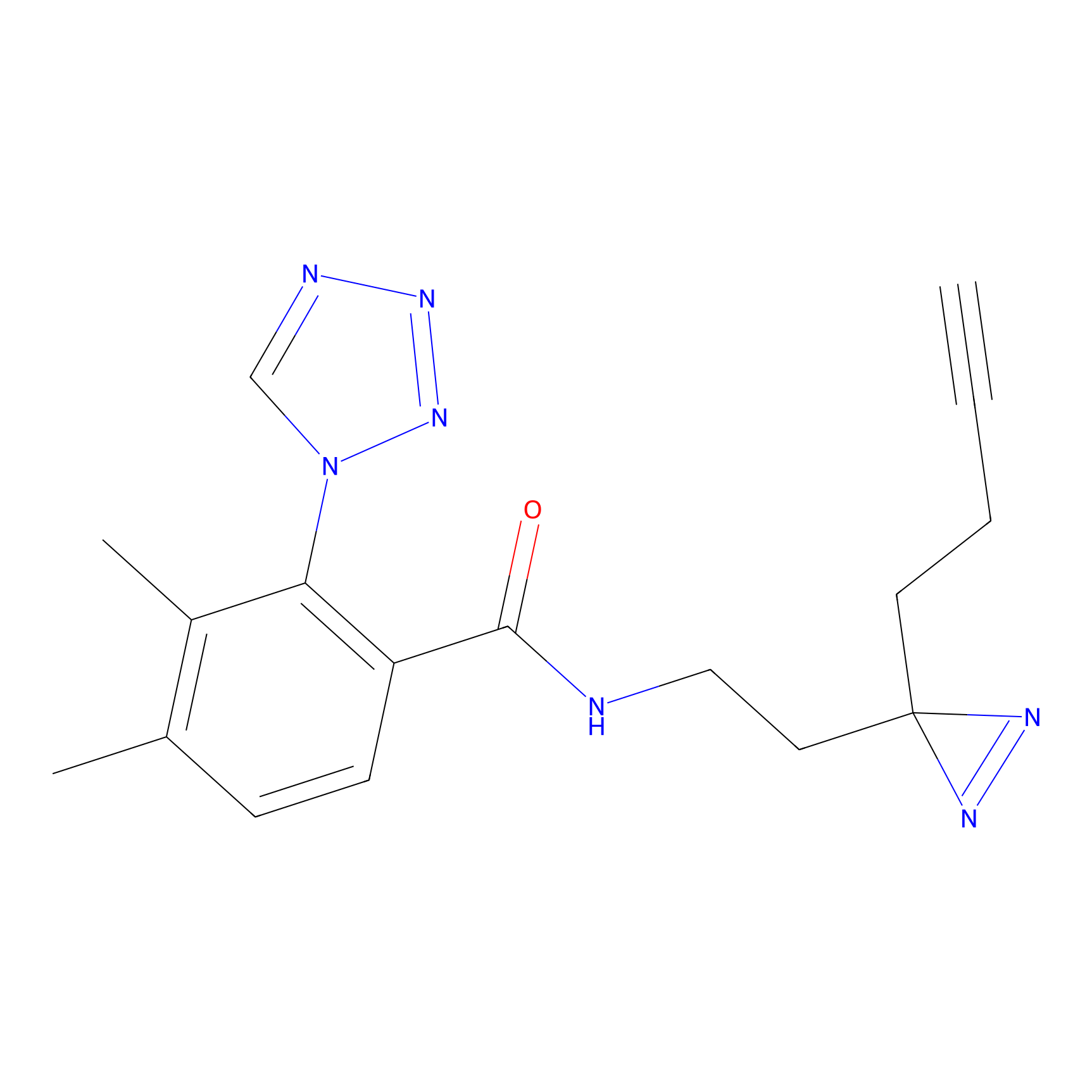
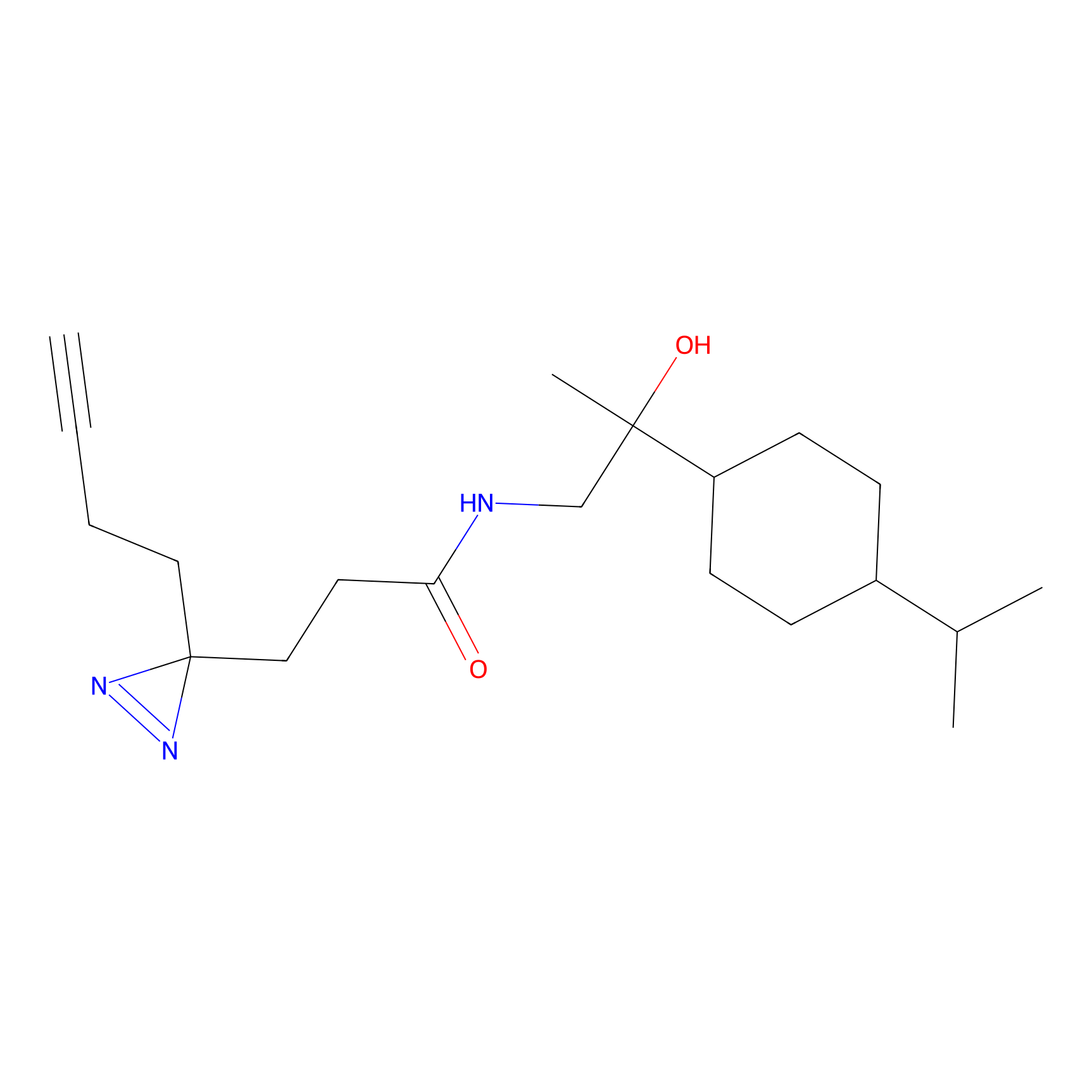
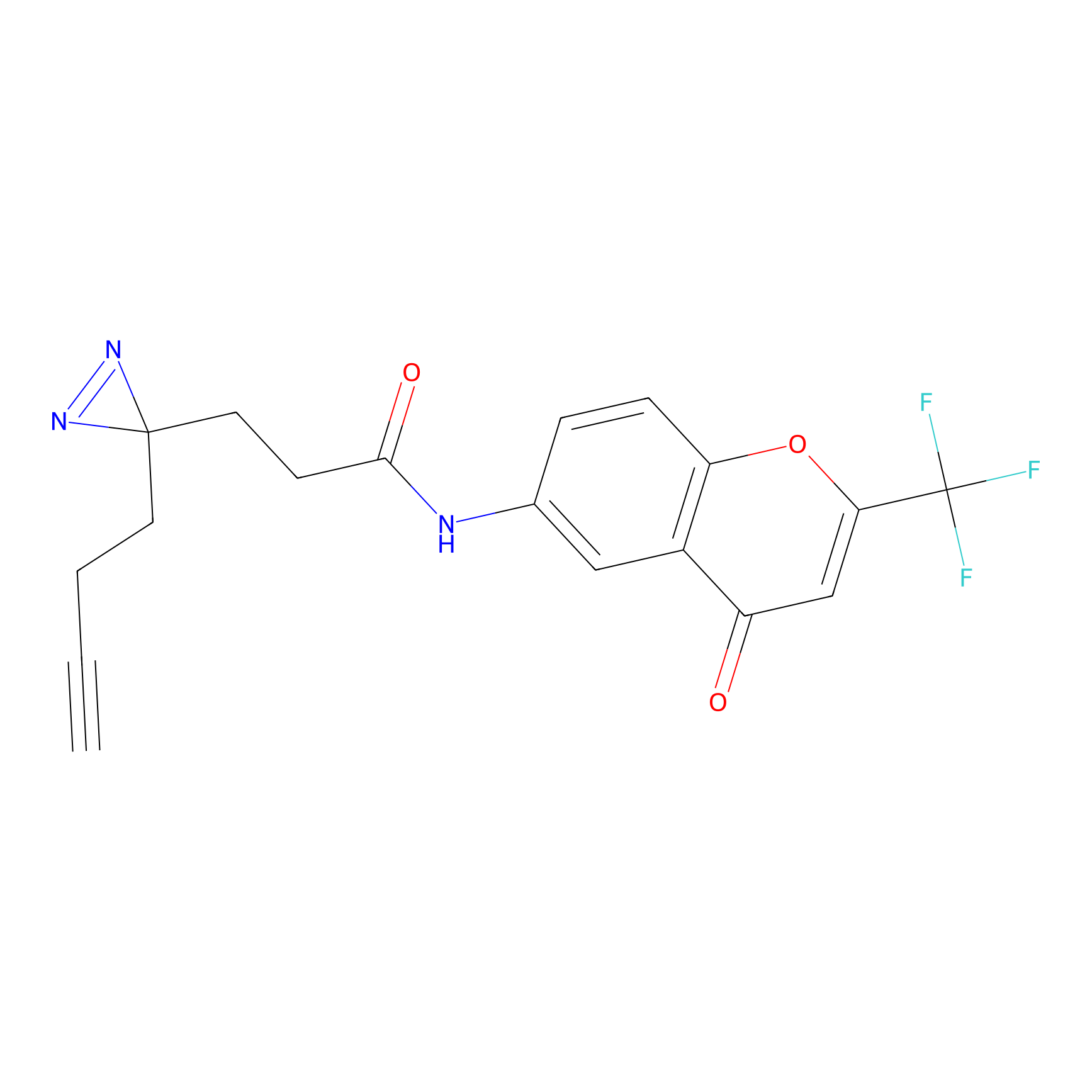
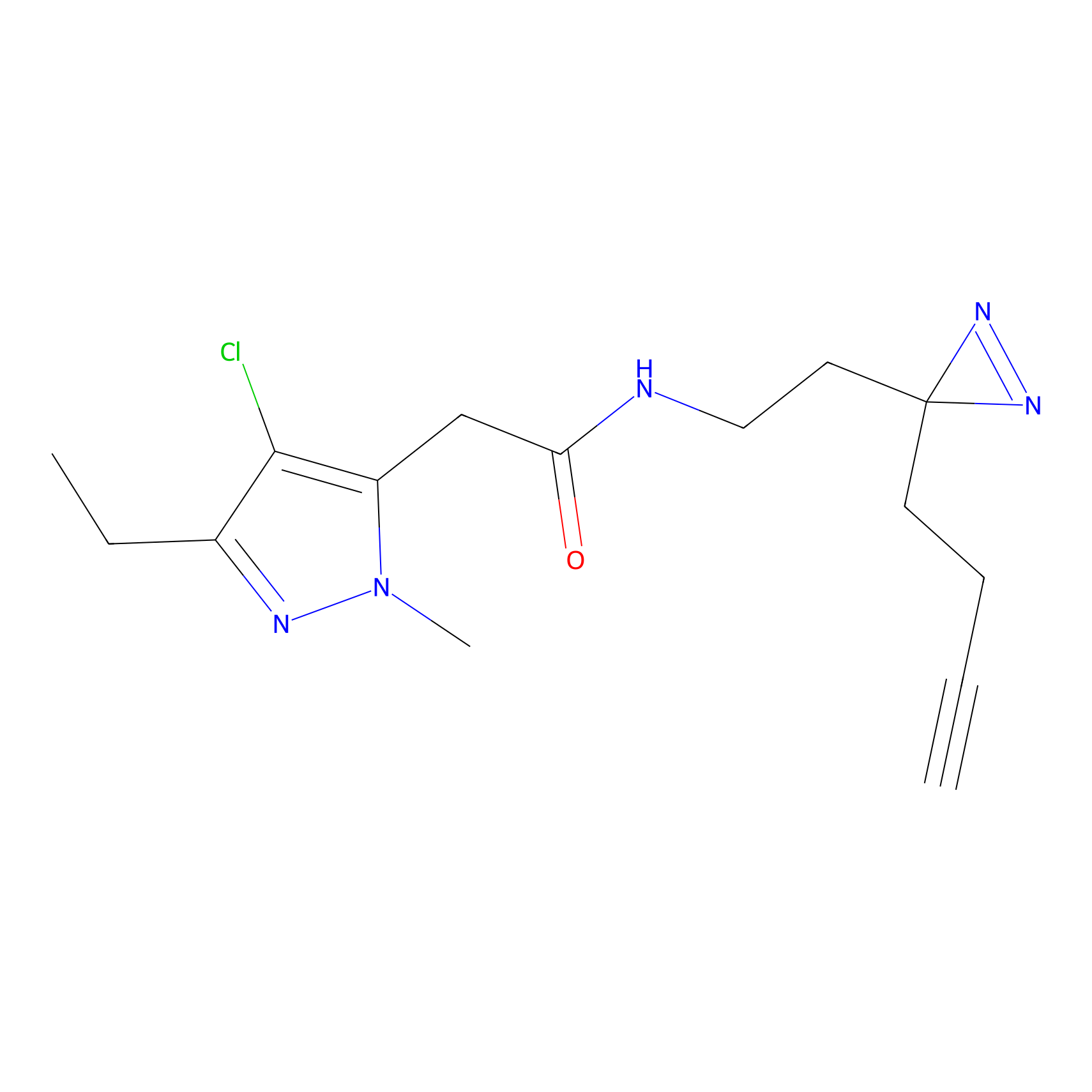
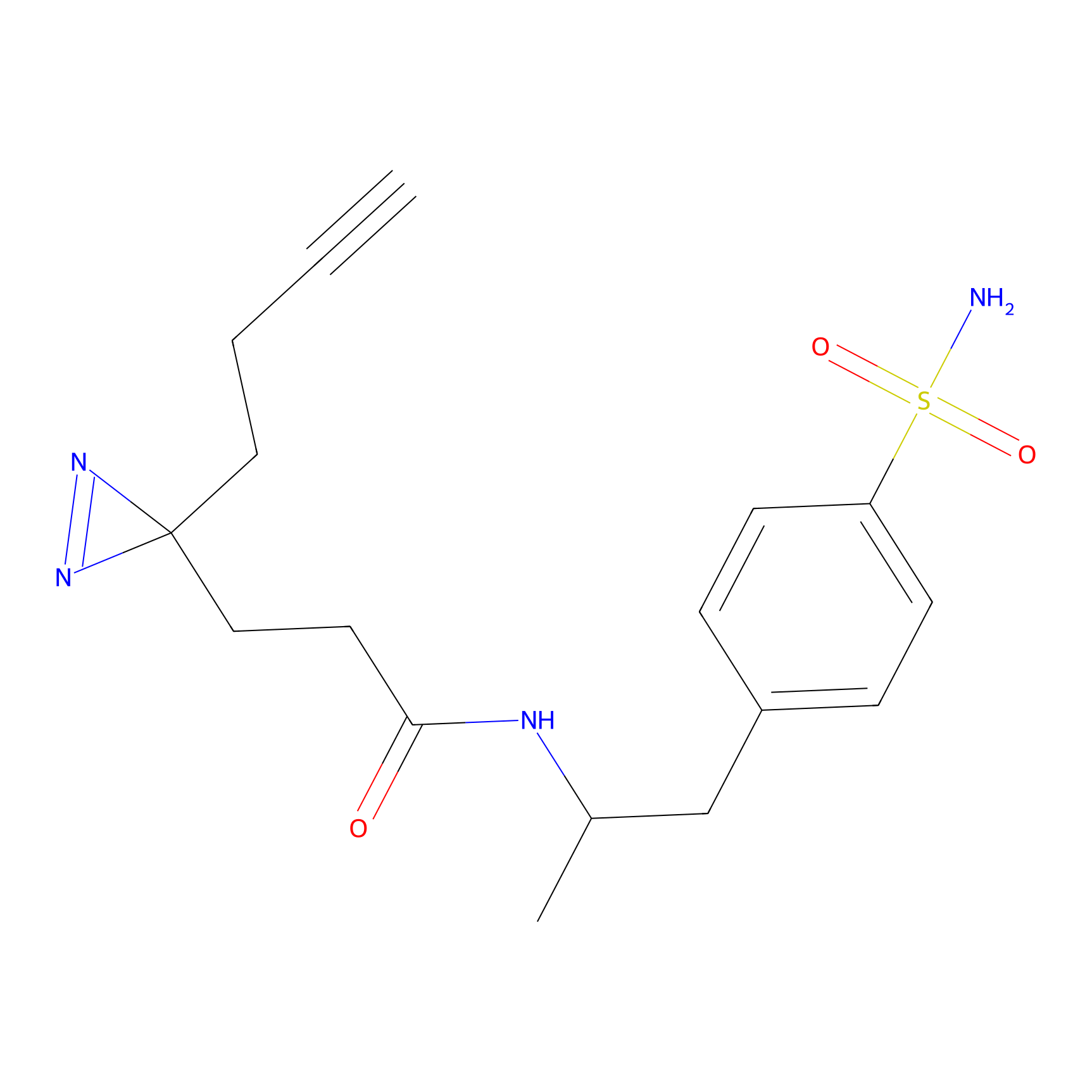
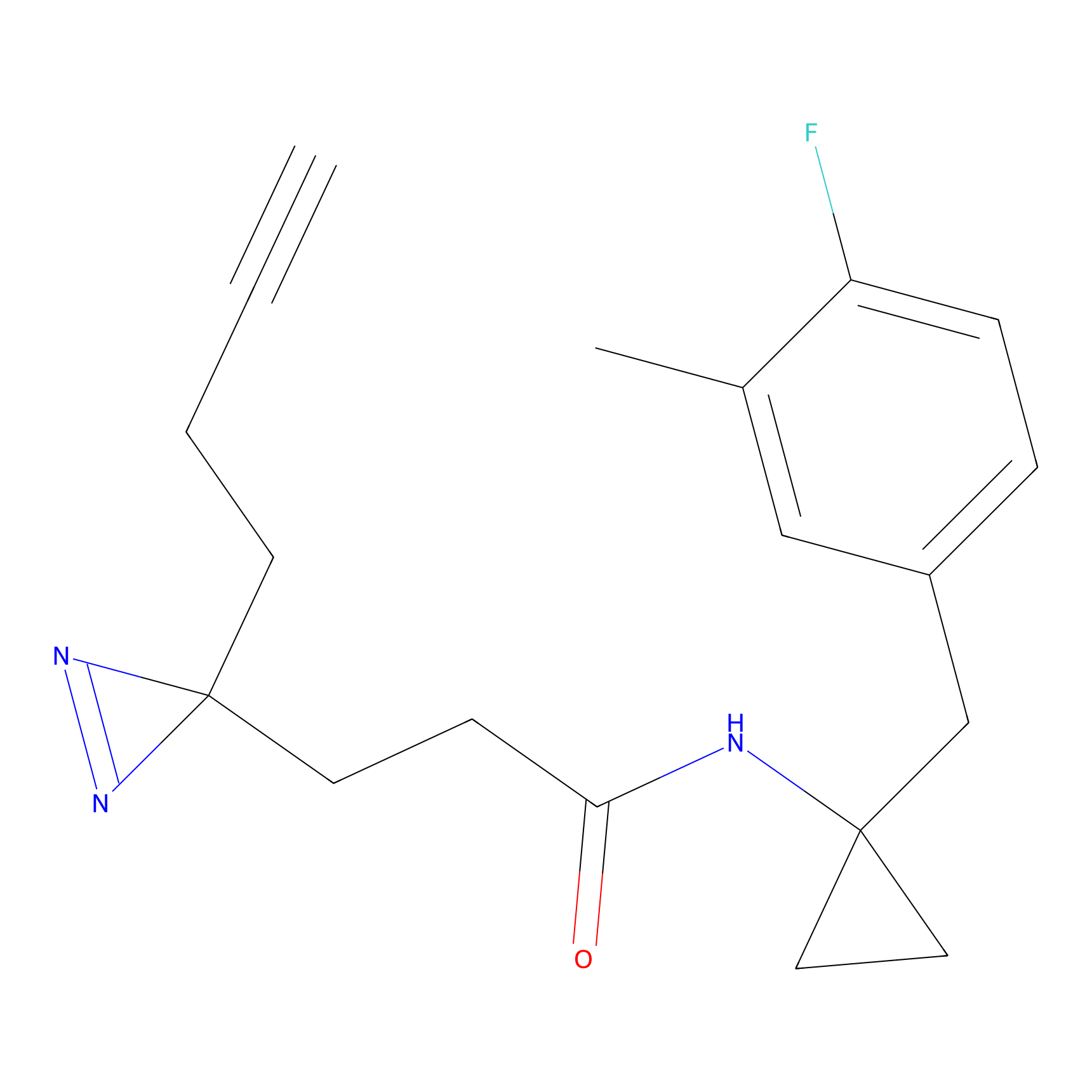
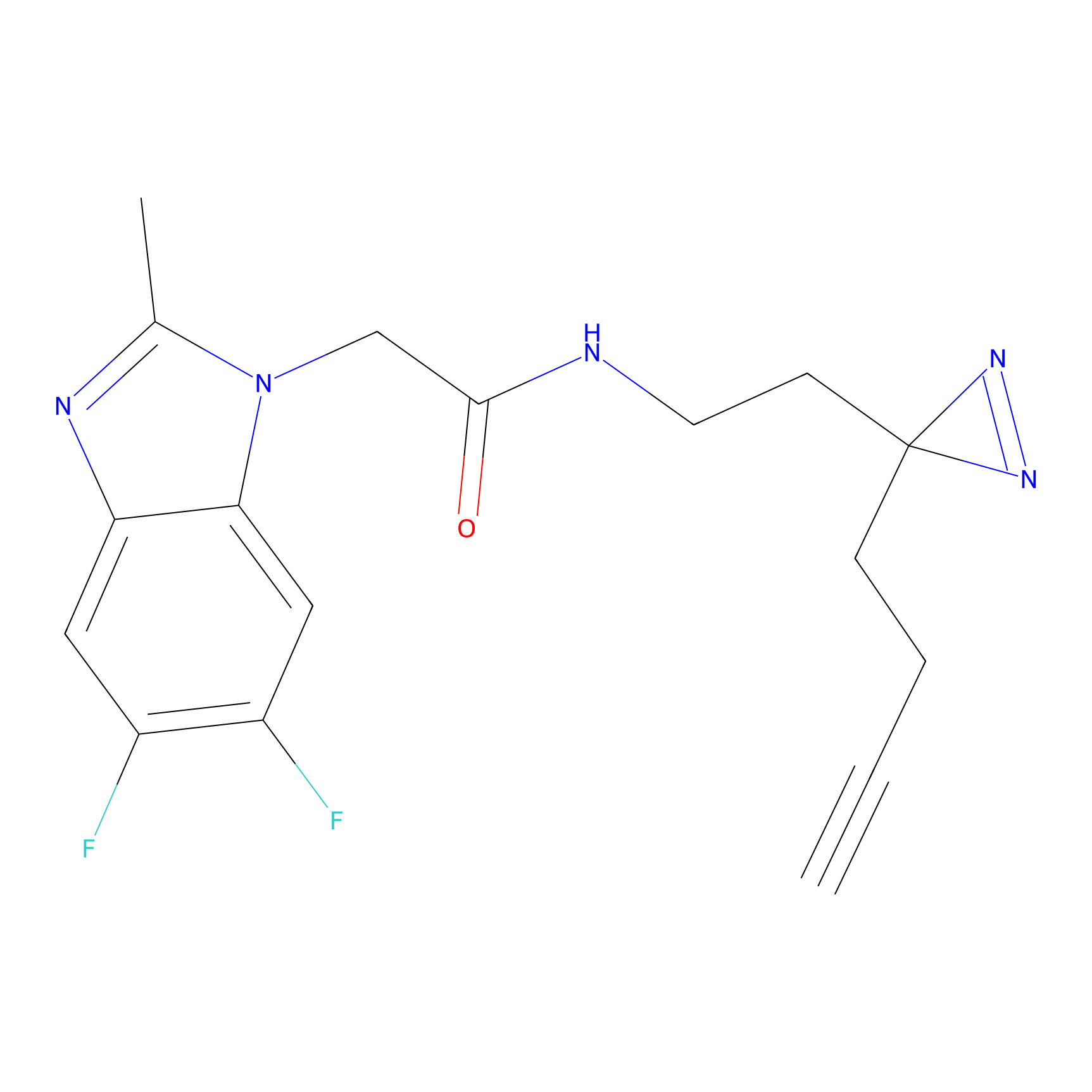
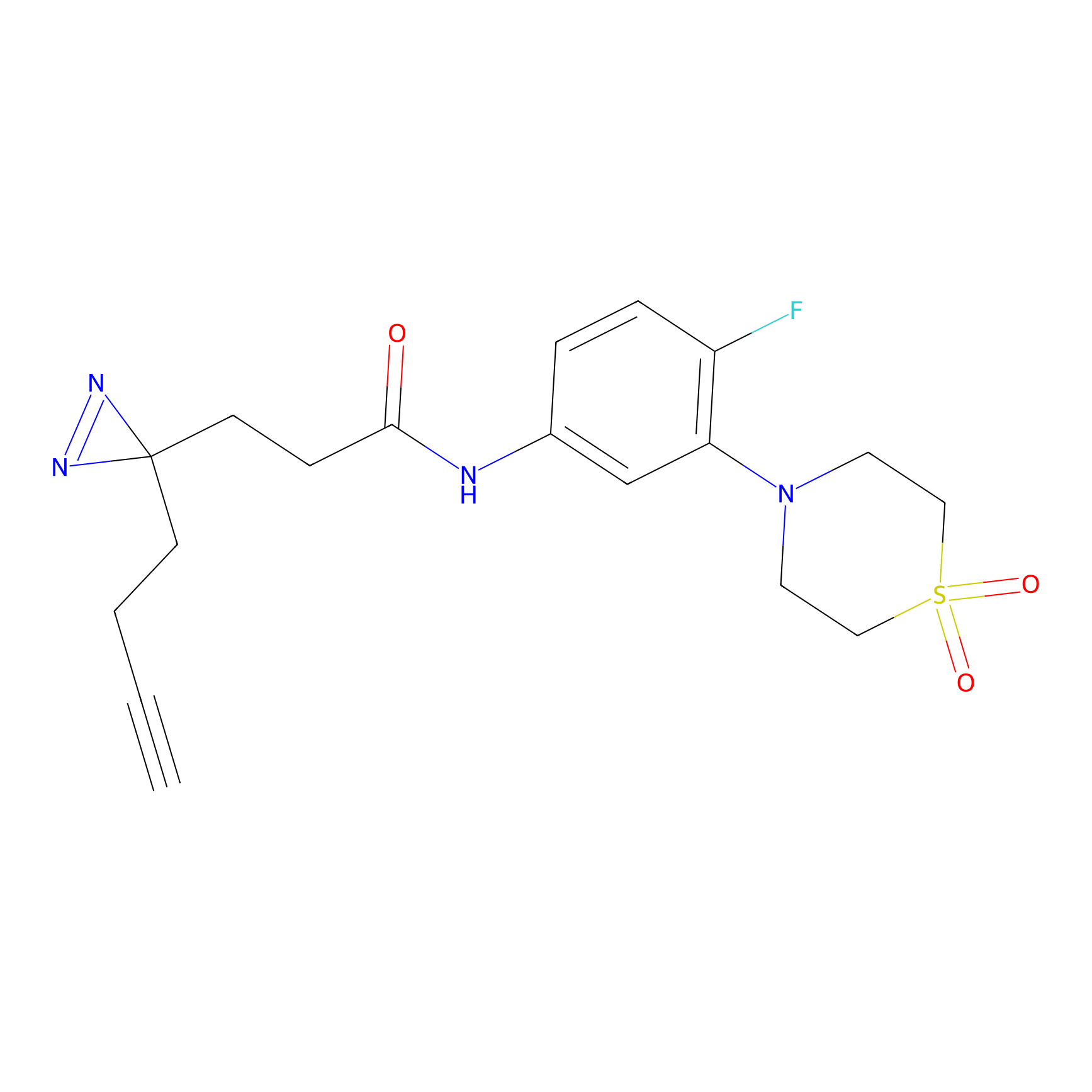
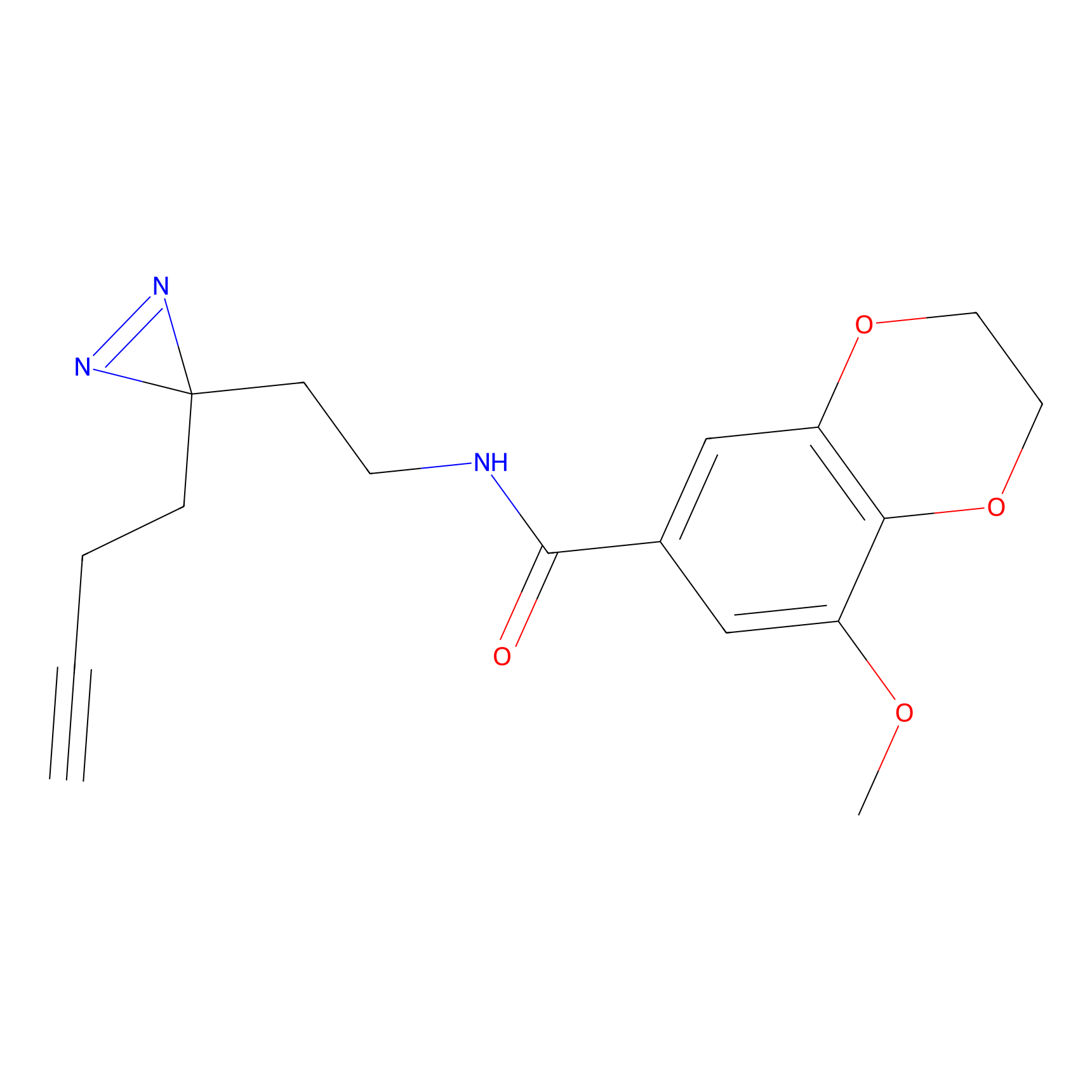
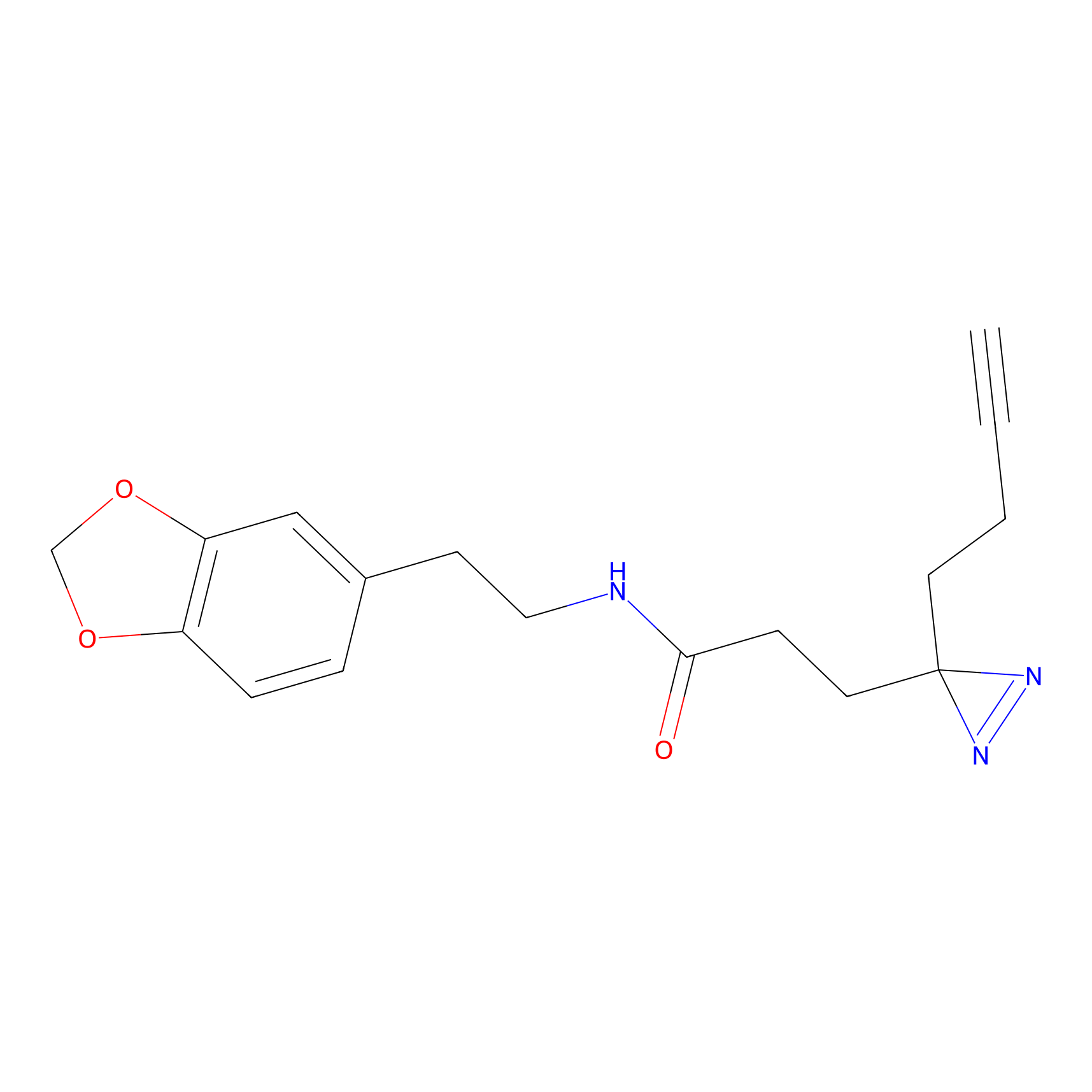
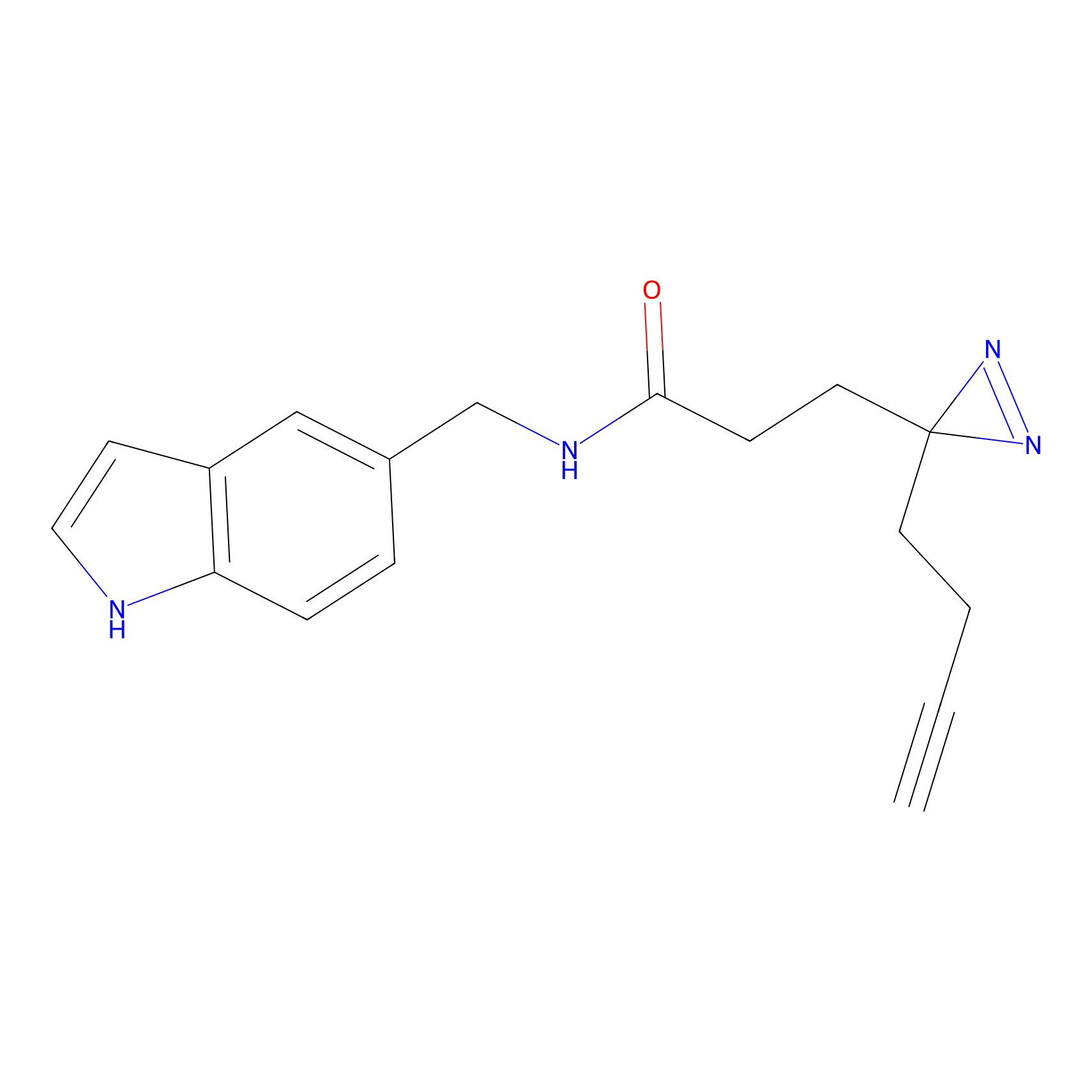
NAIA_5 Probe Info |
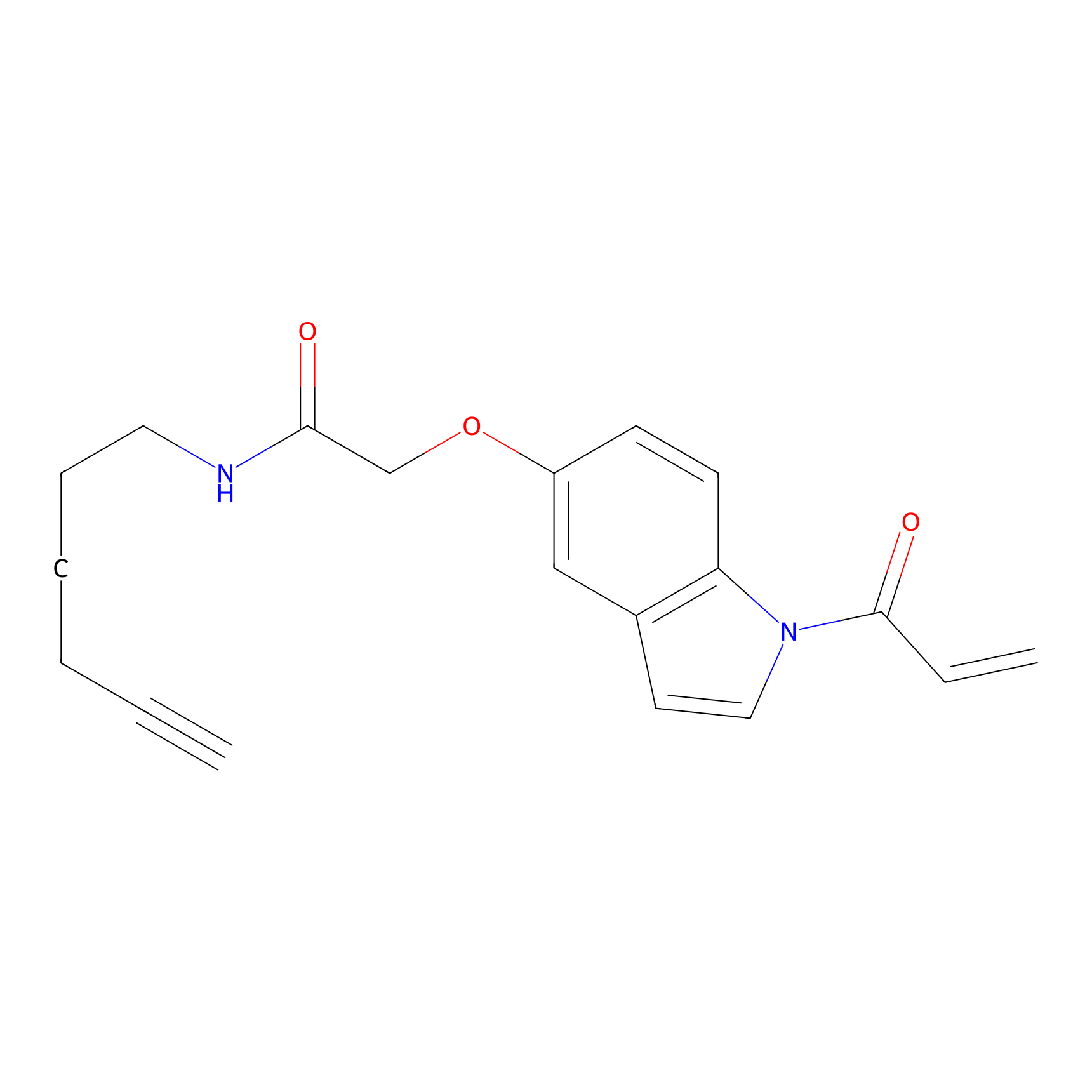 |
C244(2.07) | LDD2227 | [2] | |
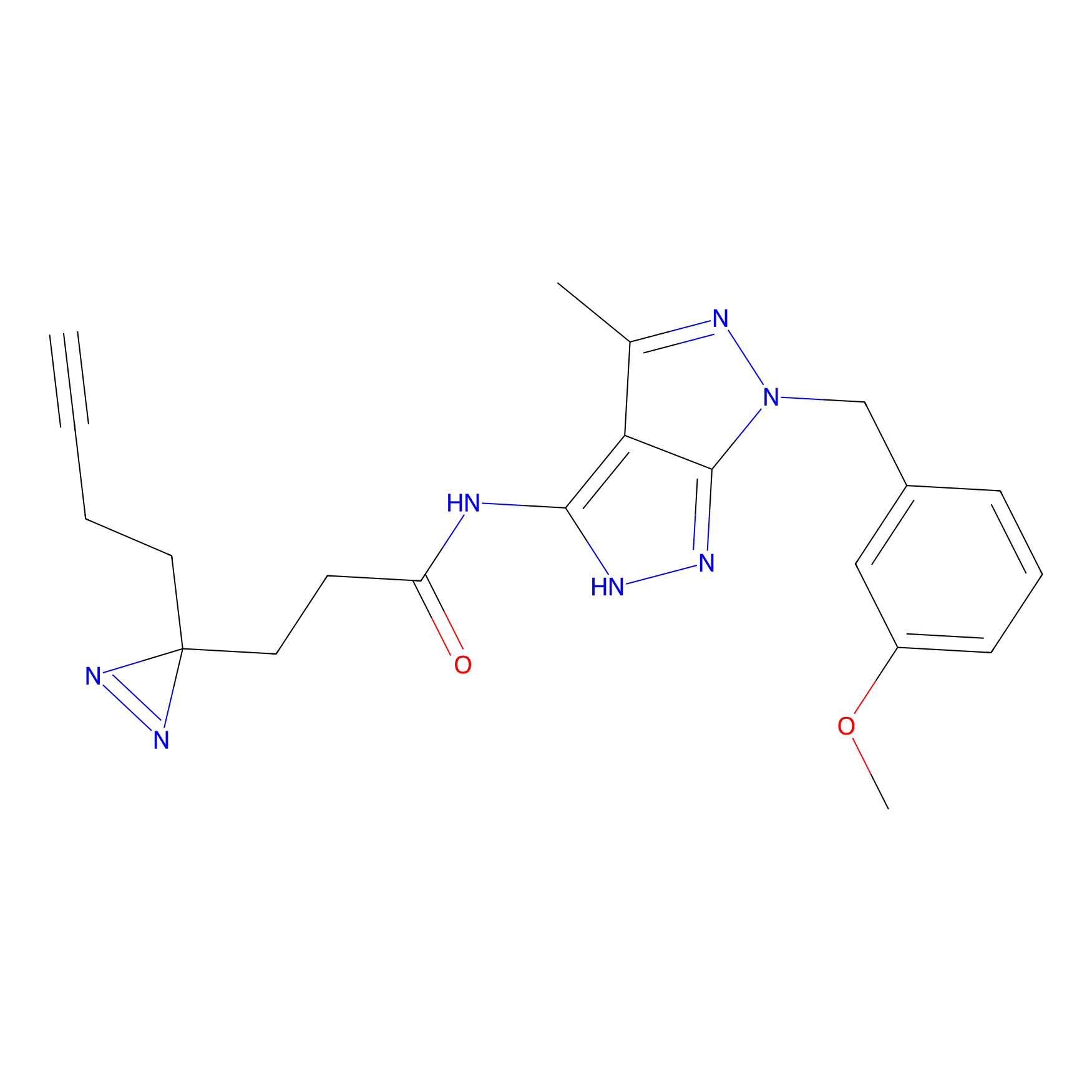
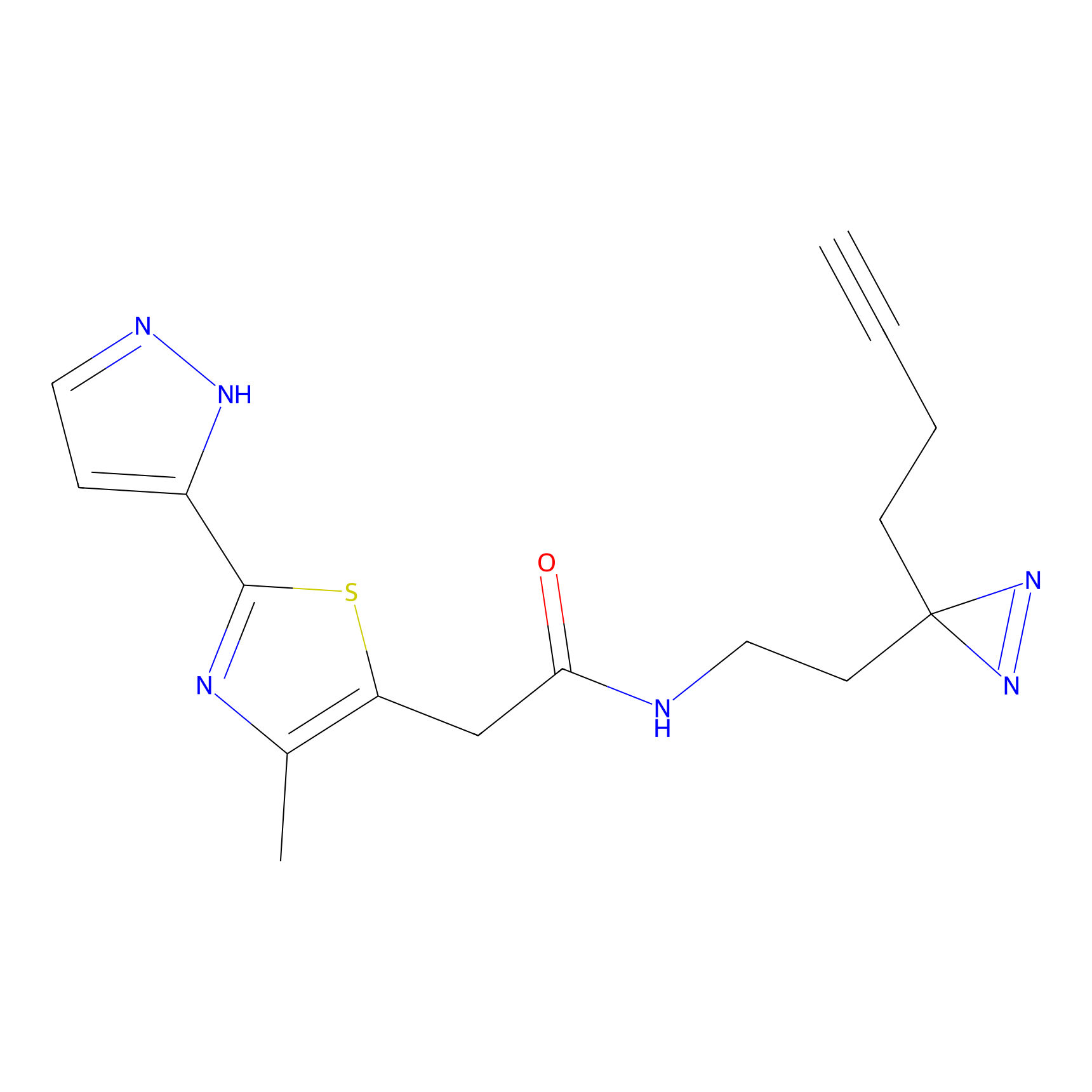
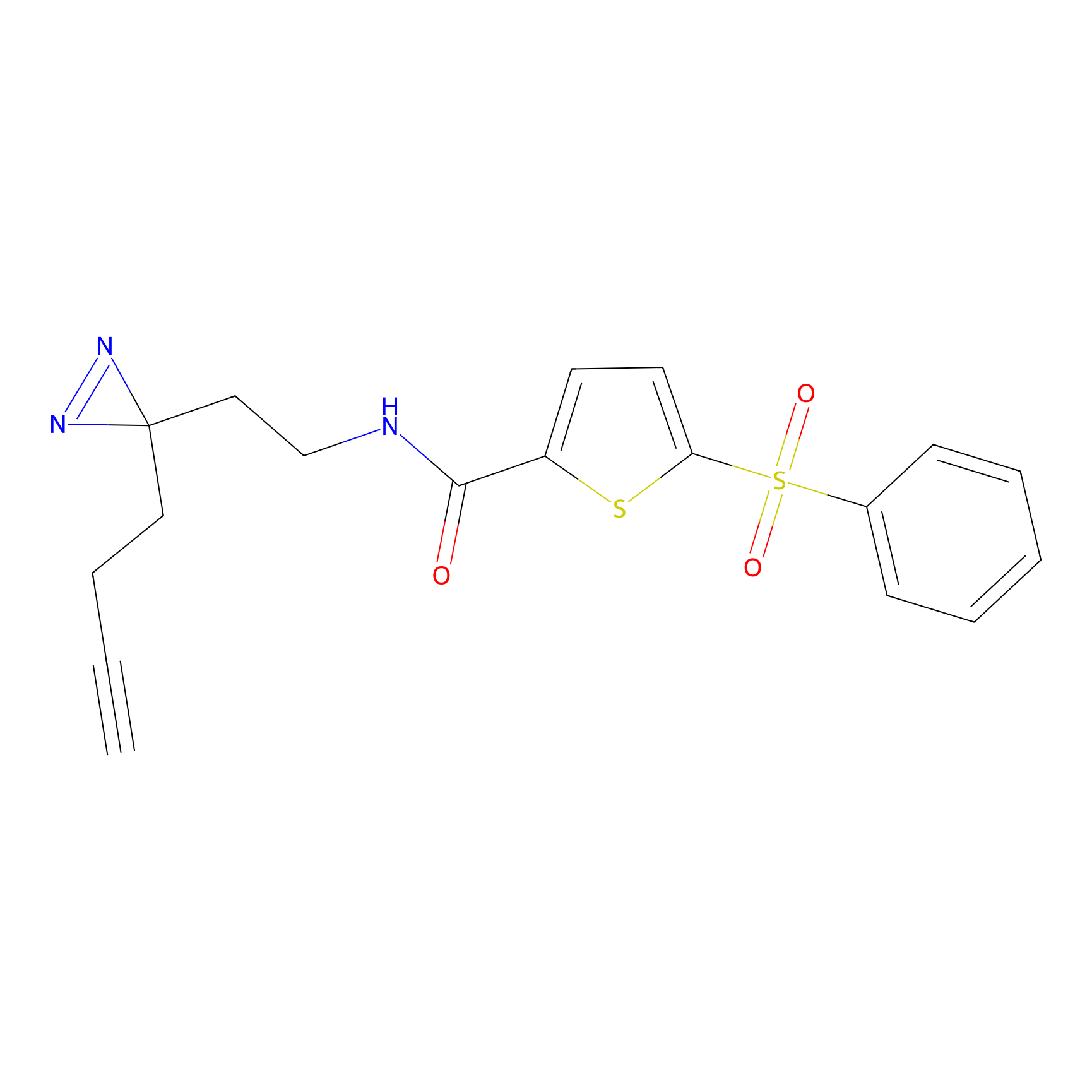
|
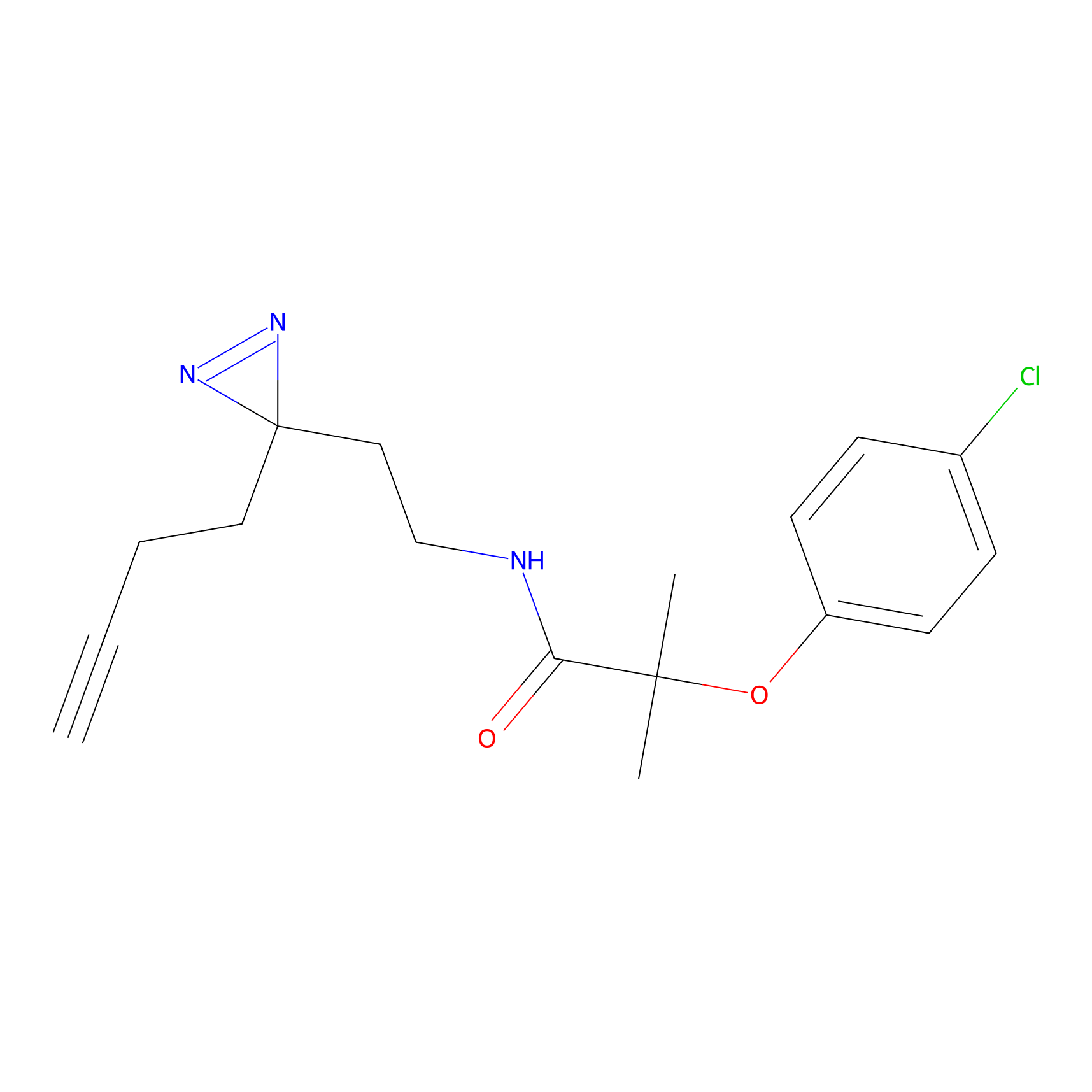
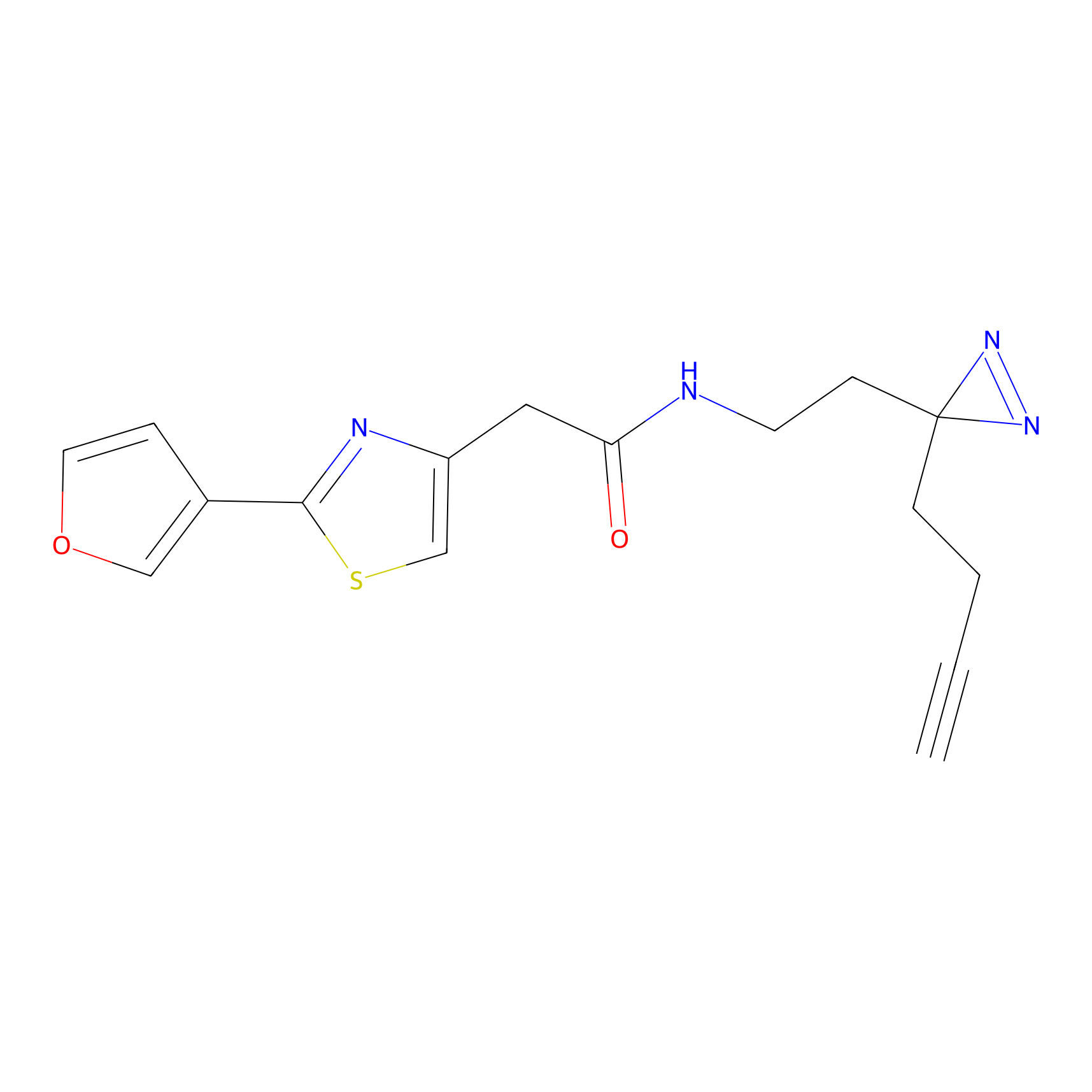
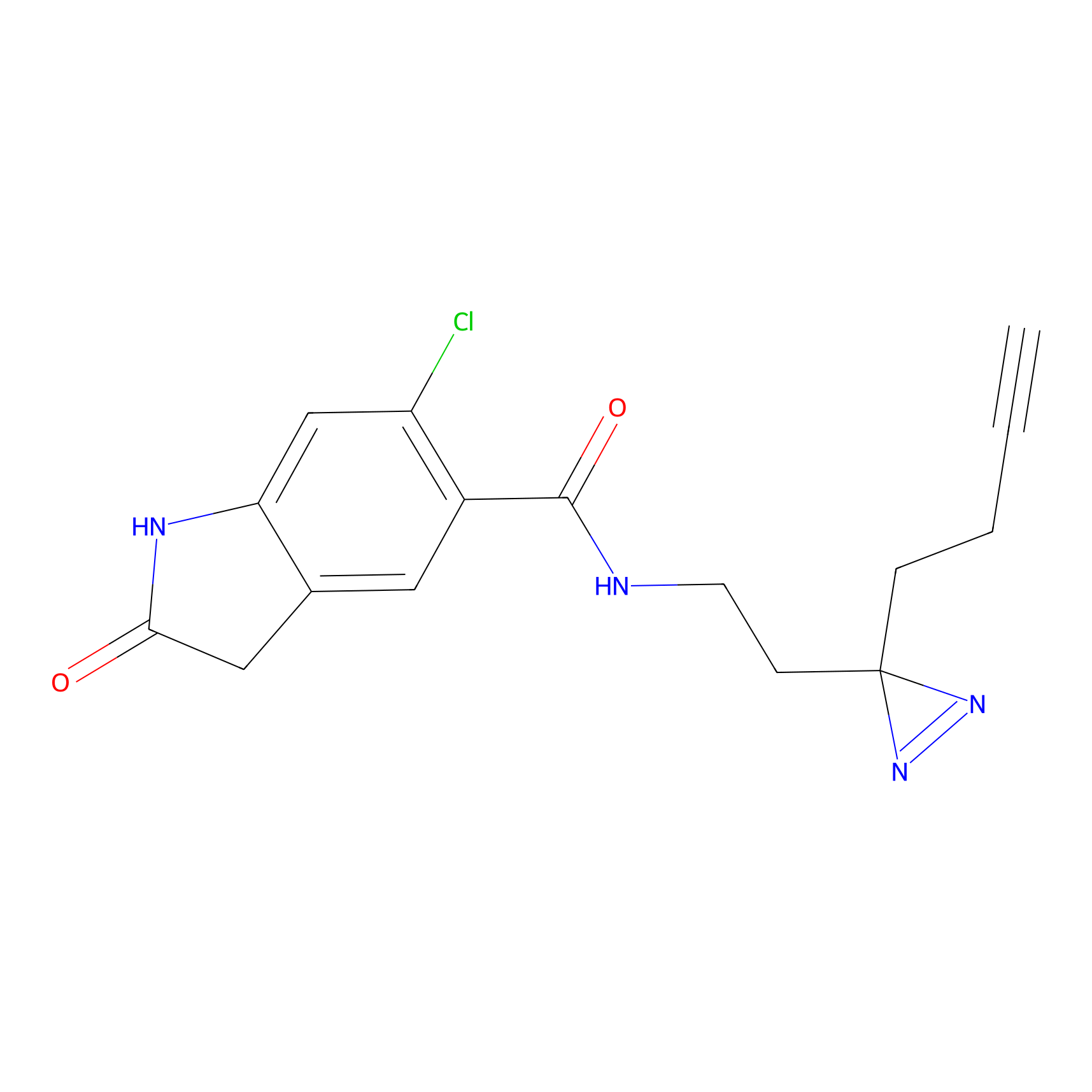
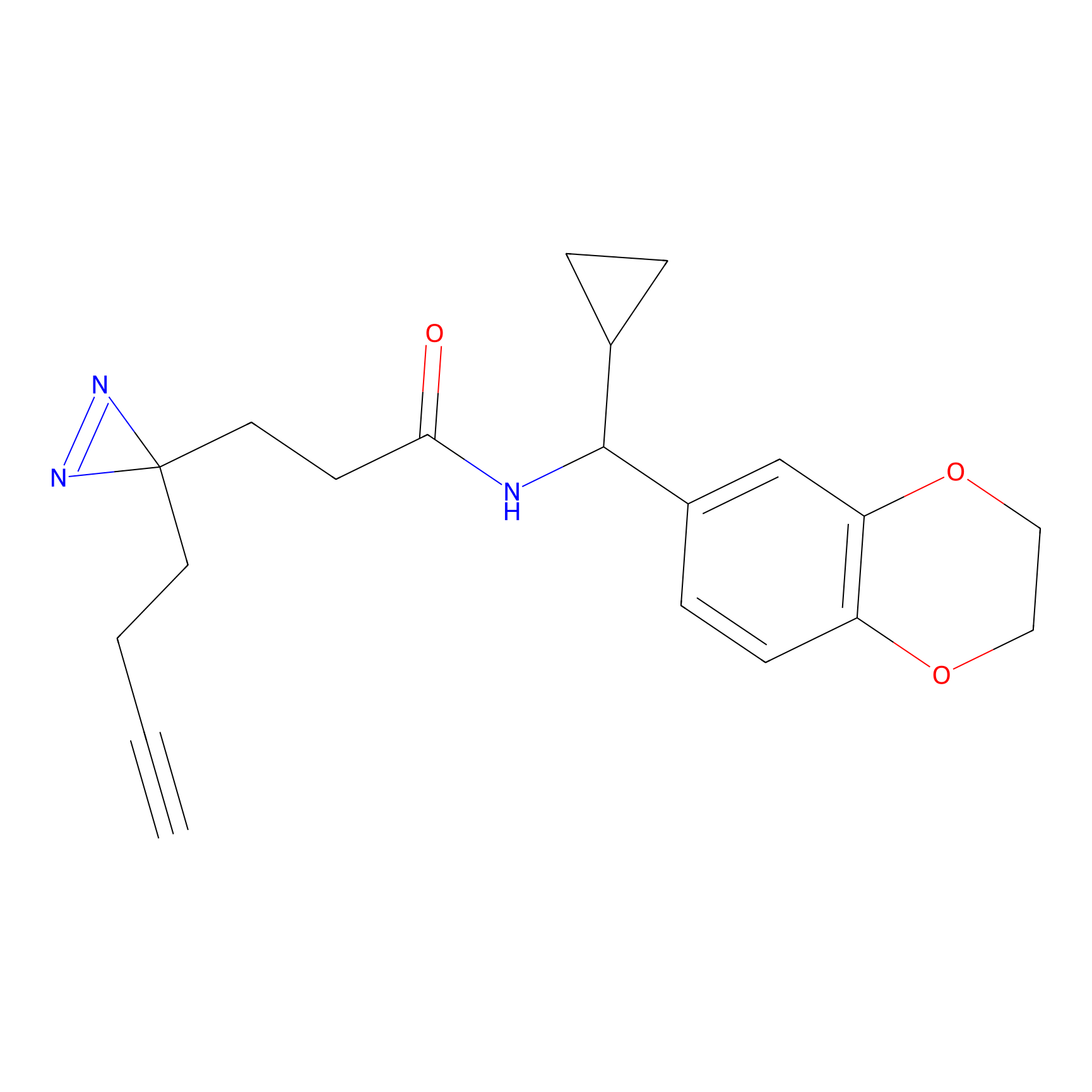
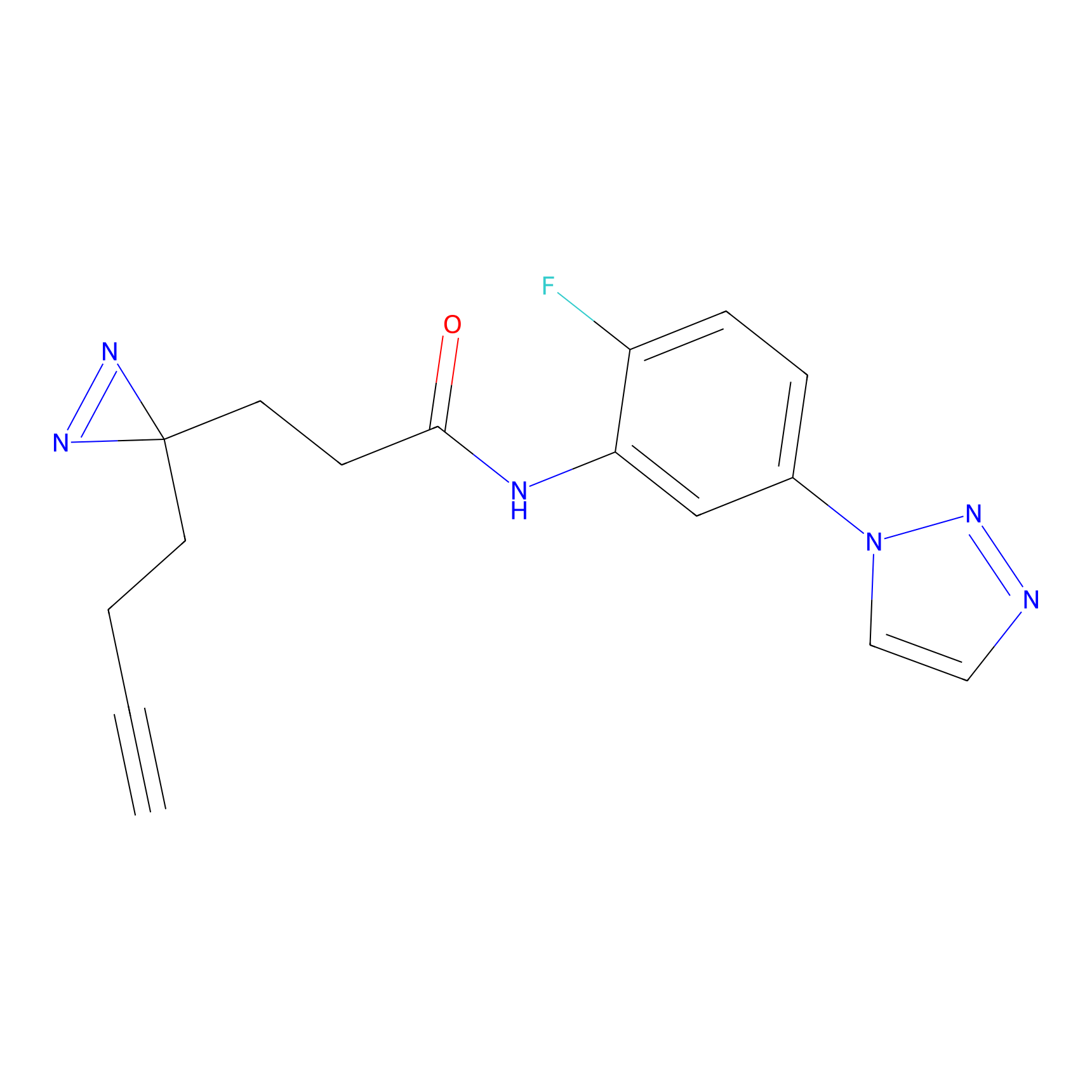
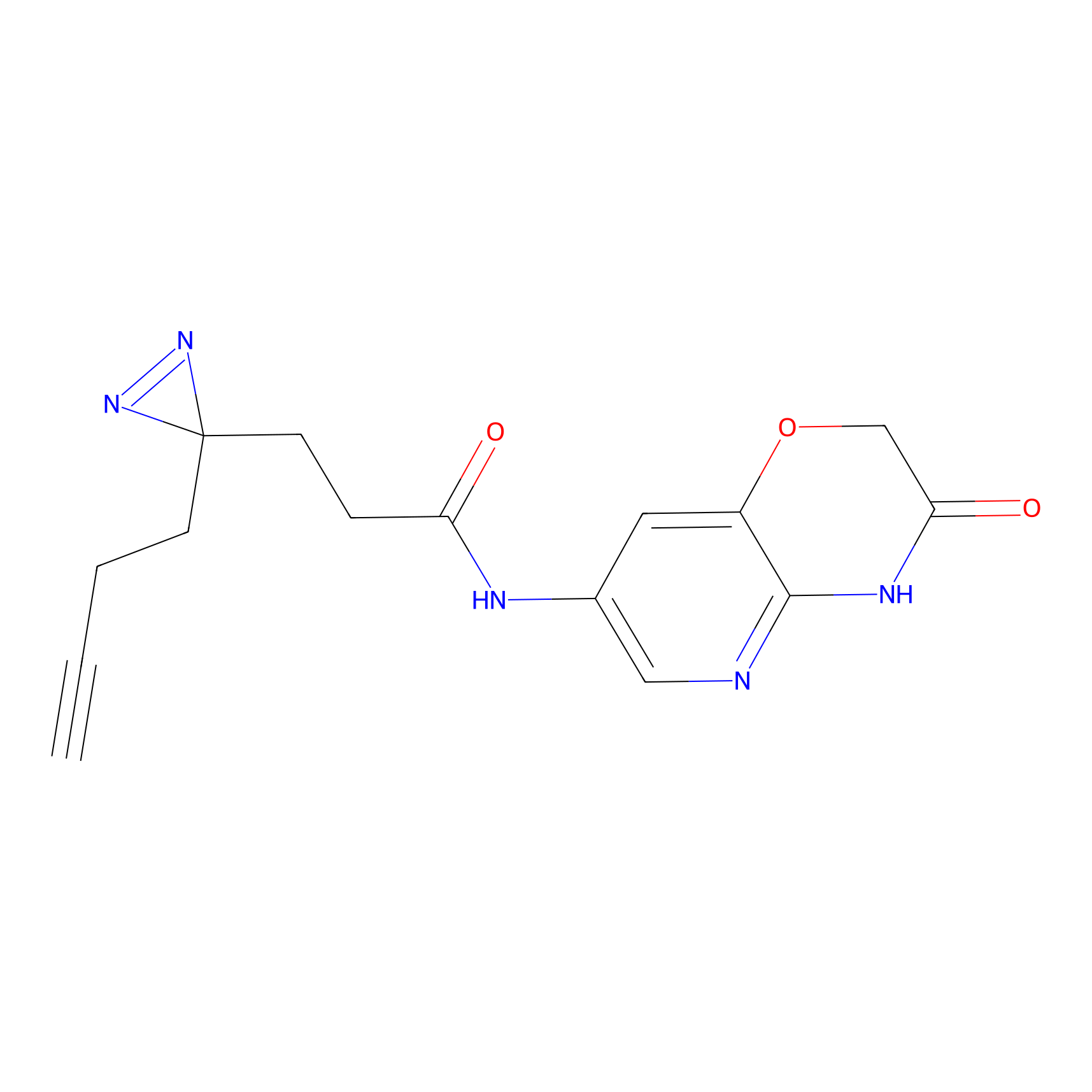
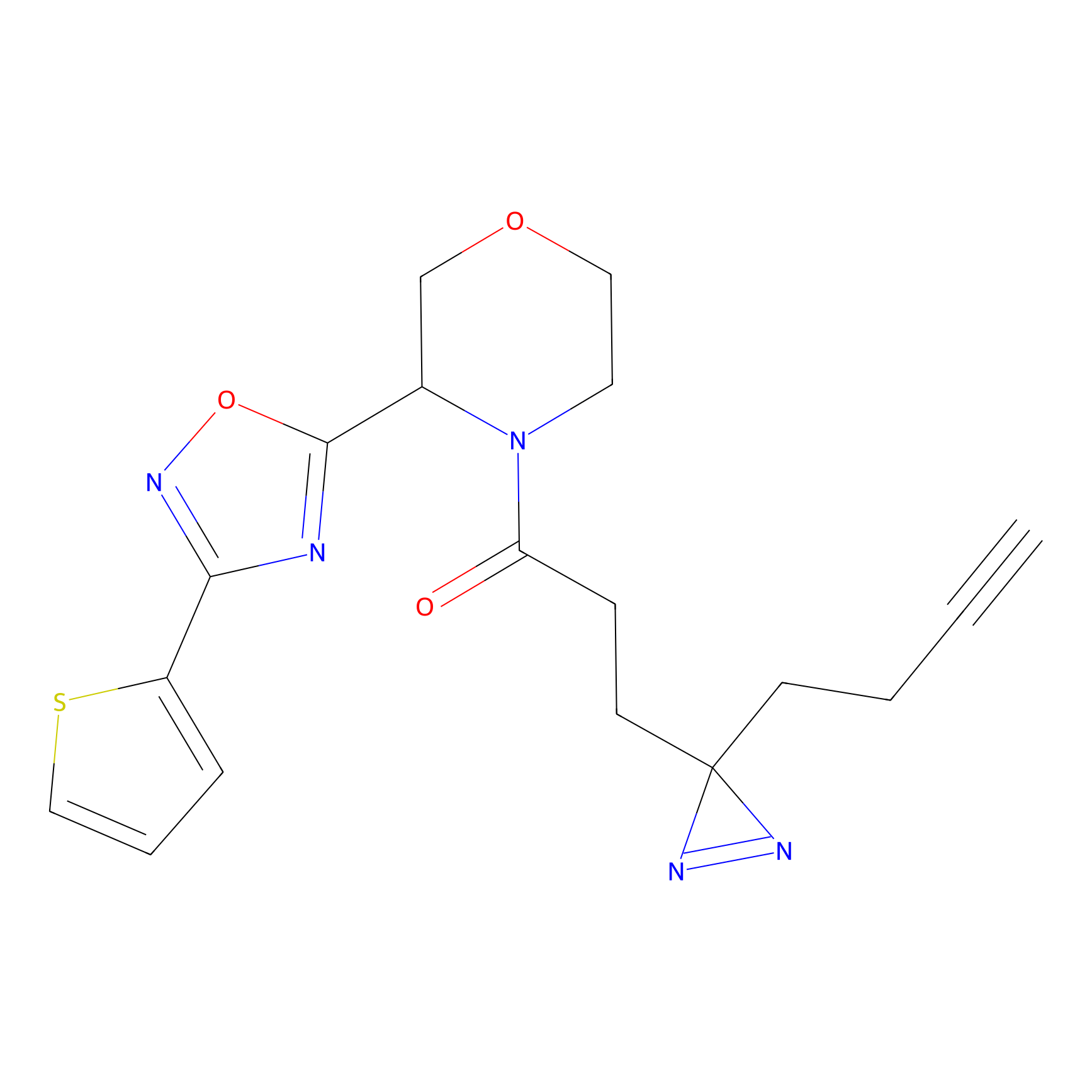
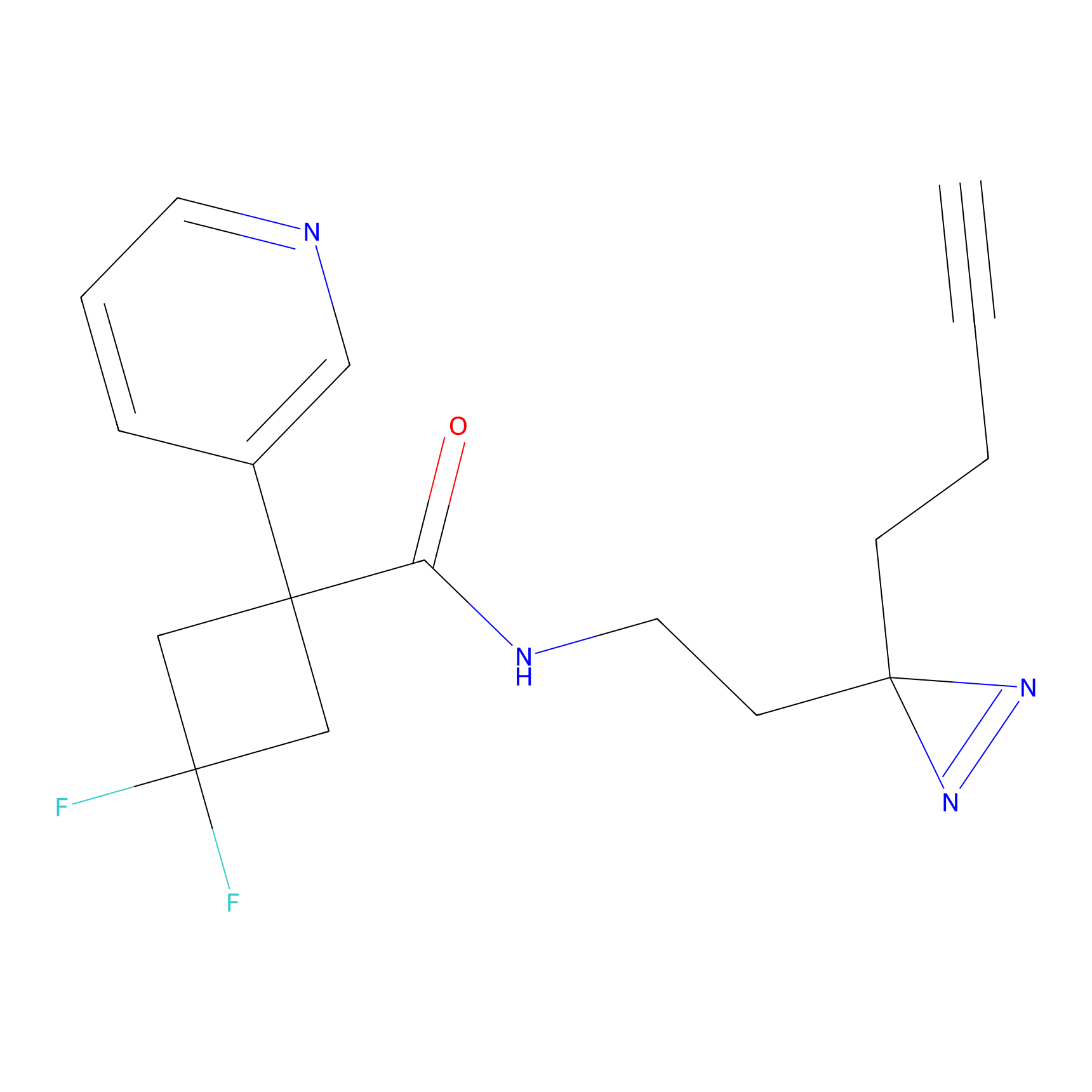
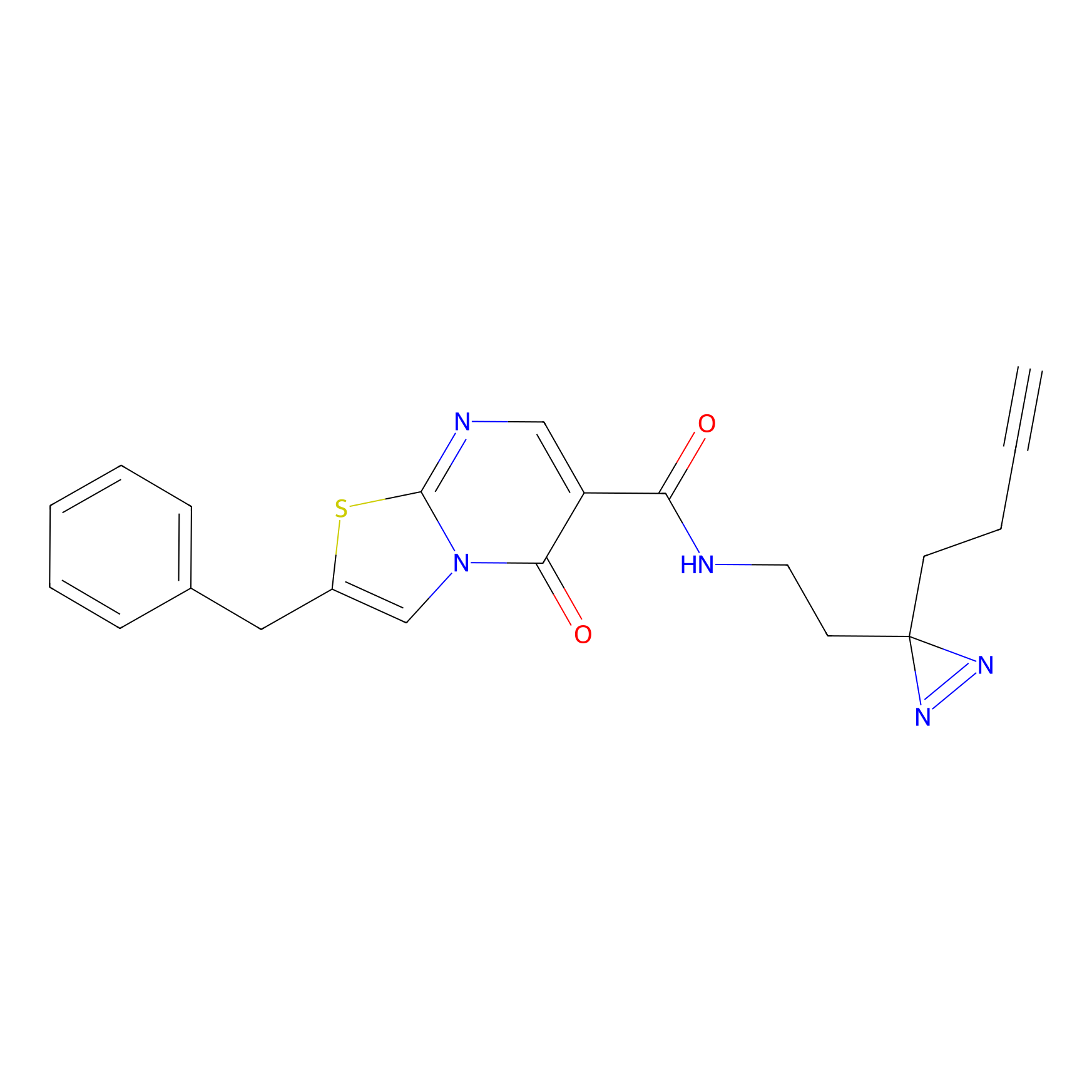
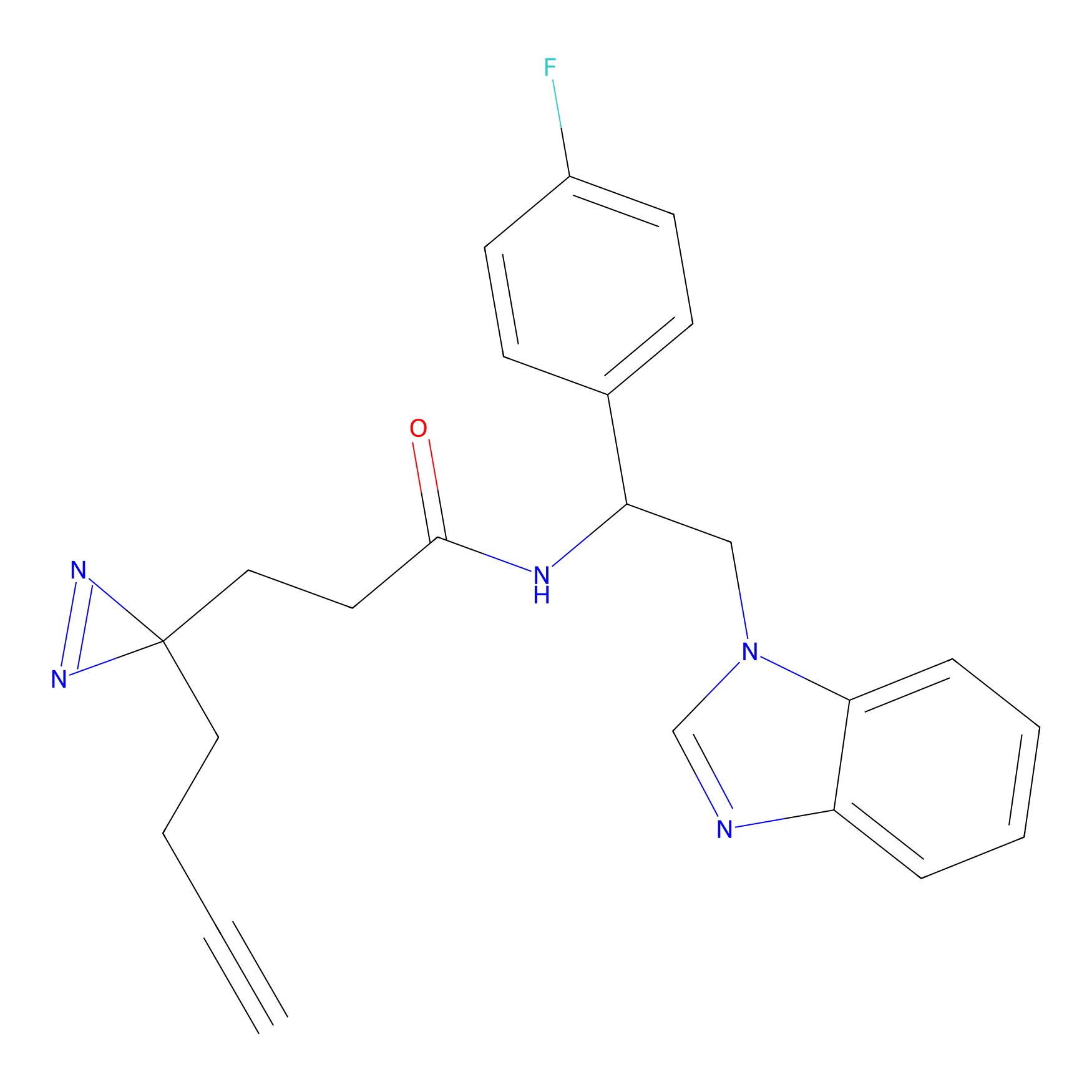
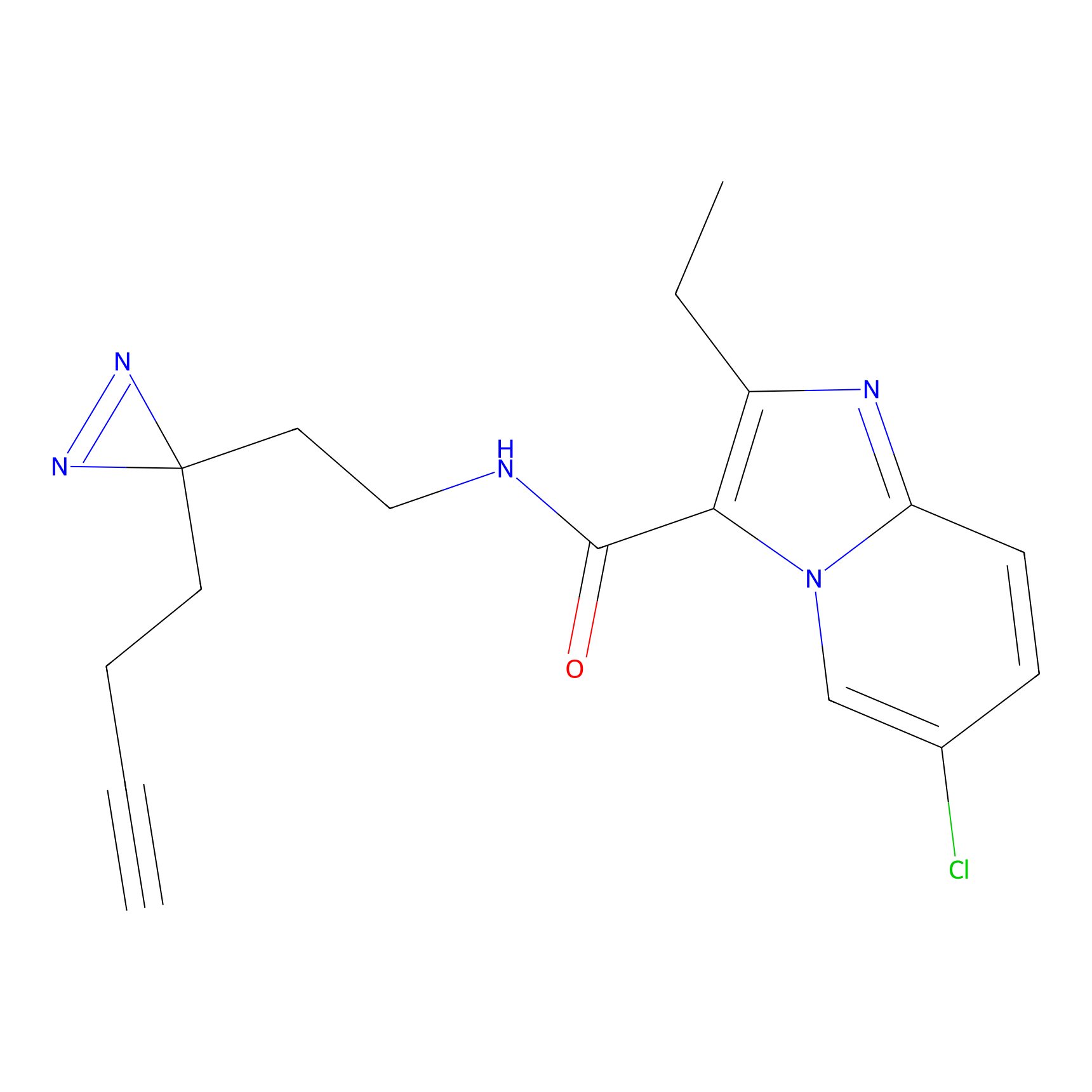
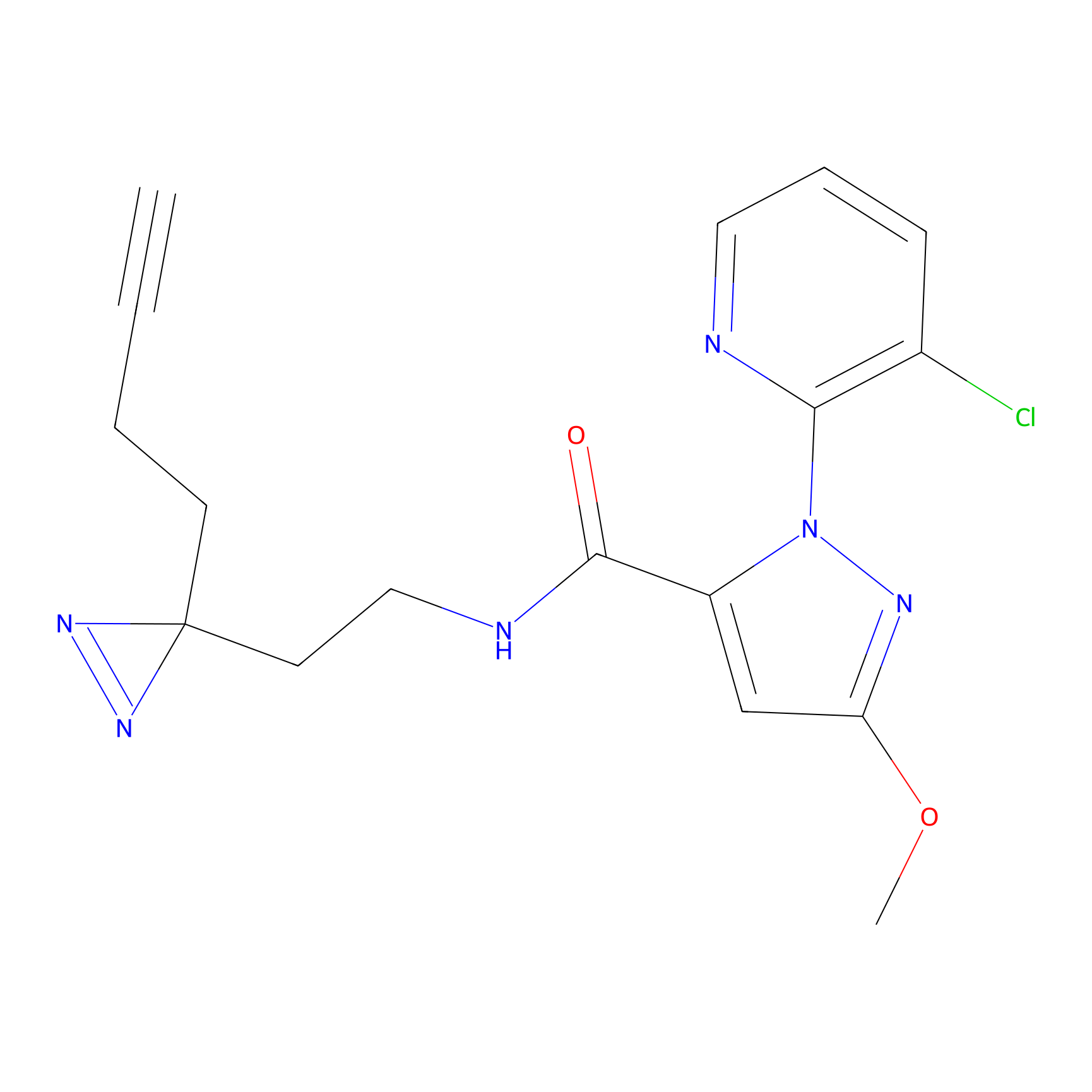
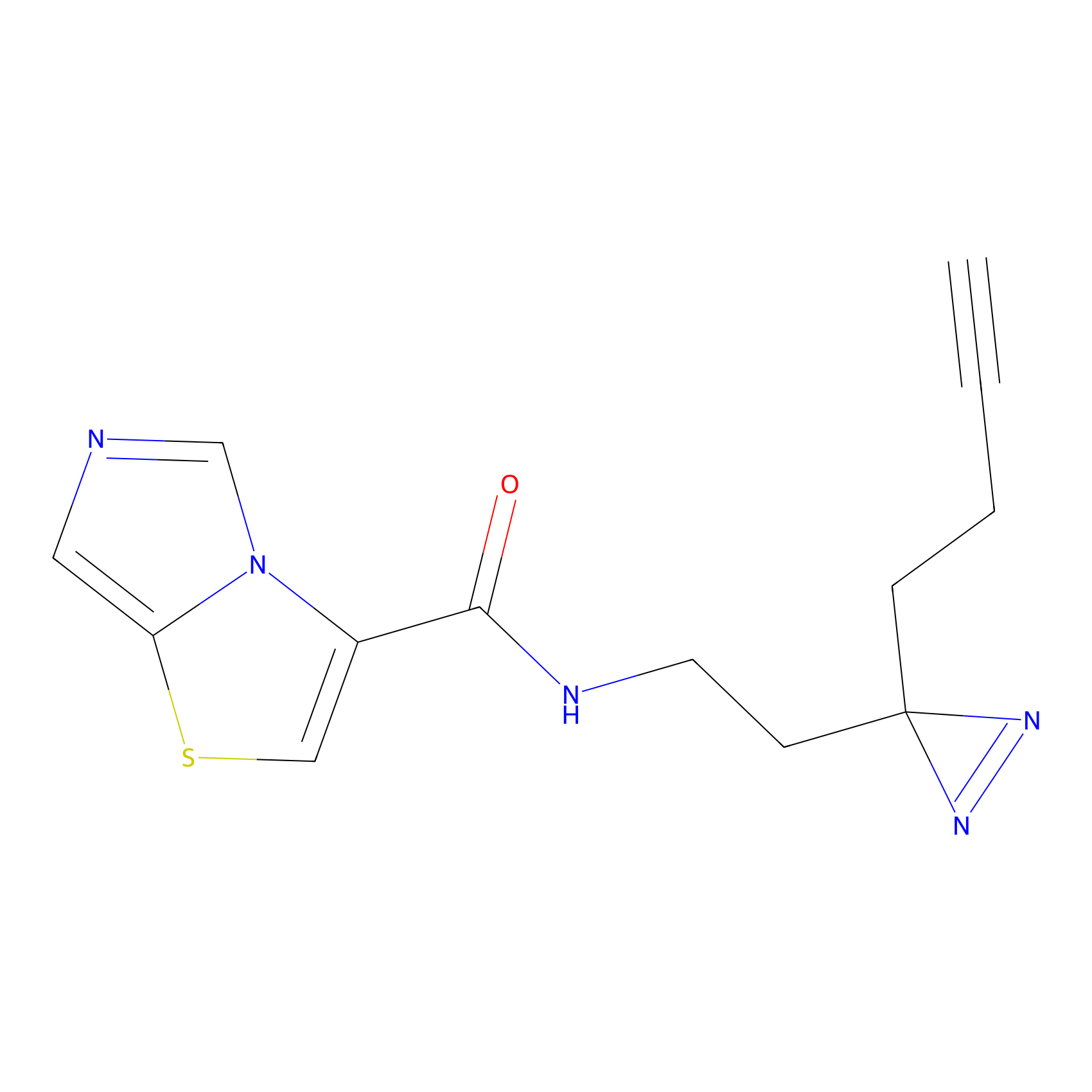
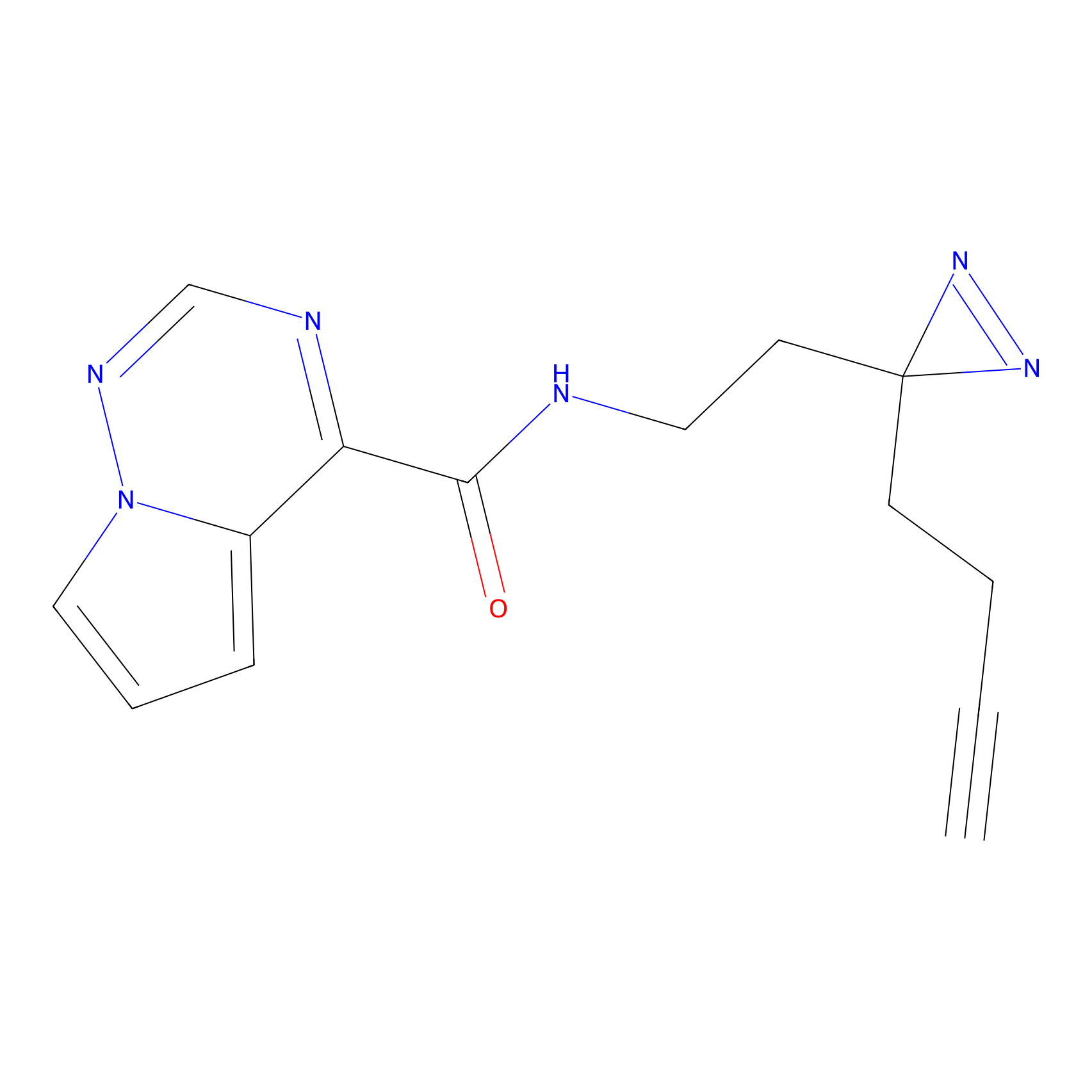
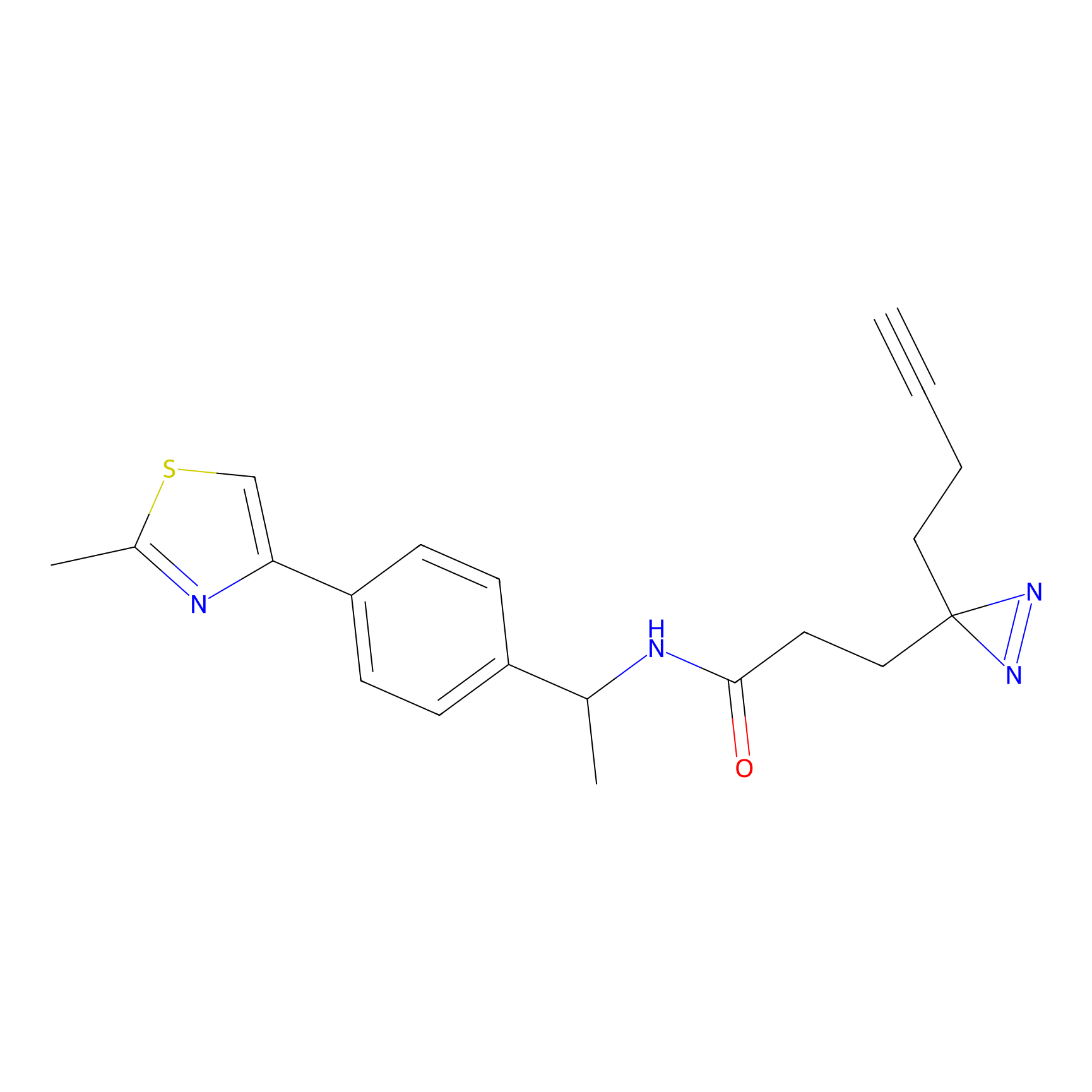
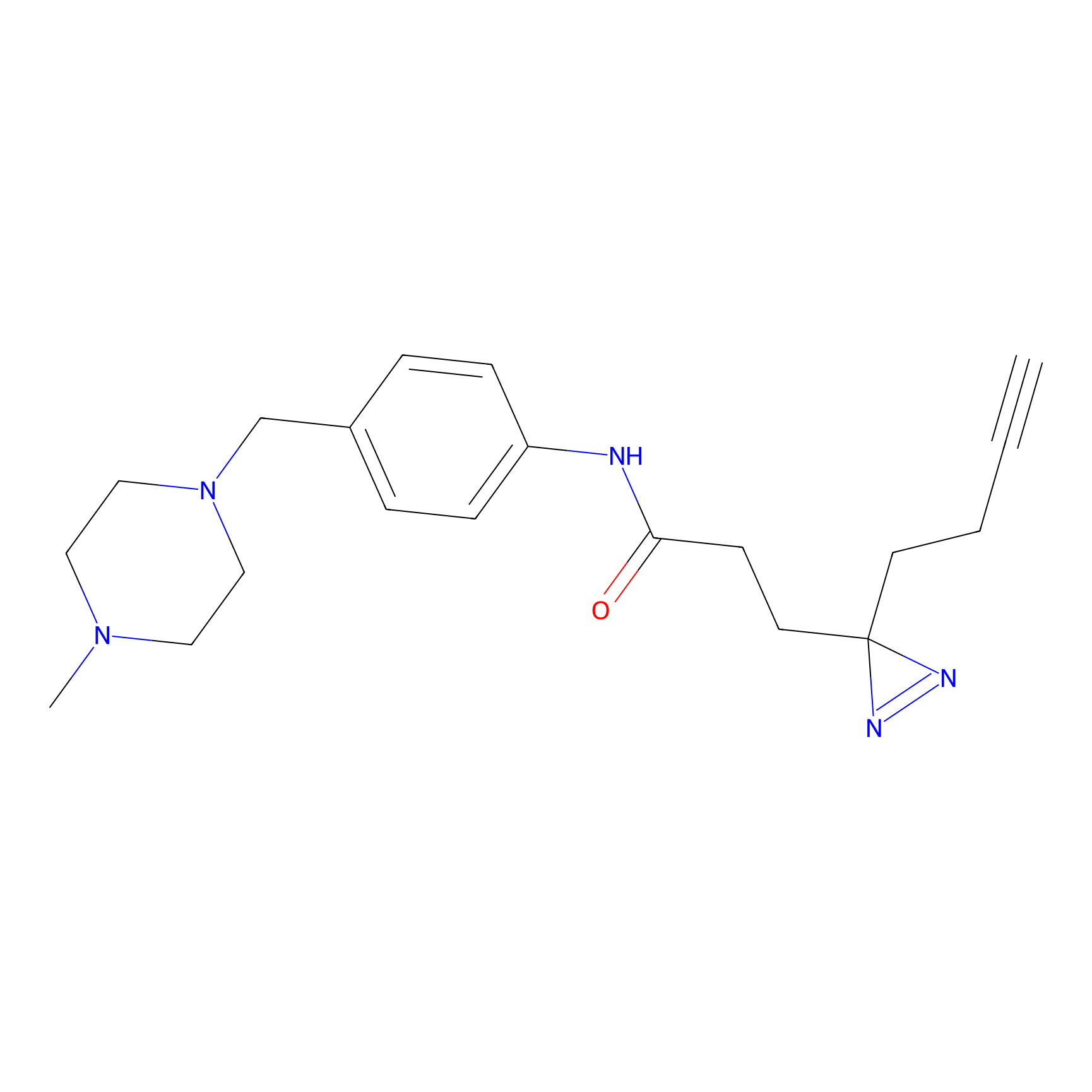
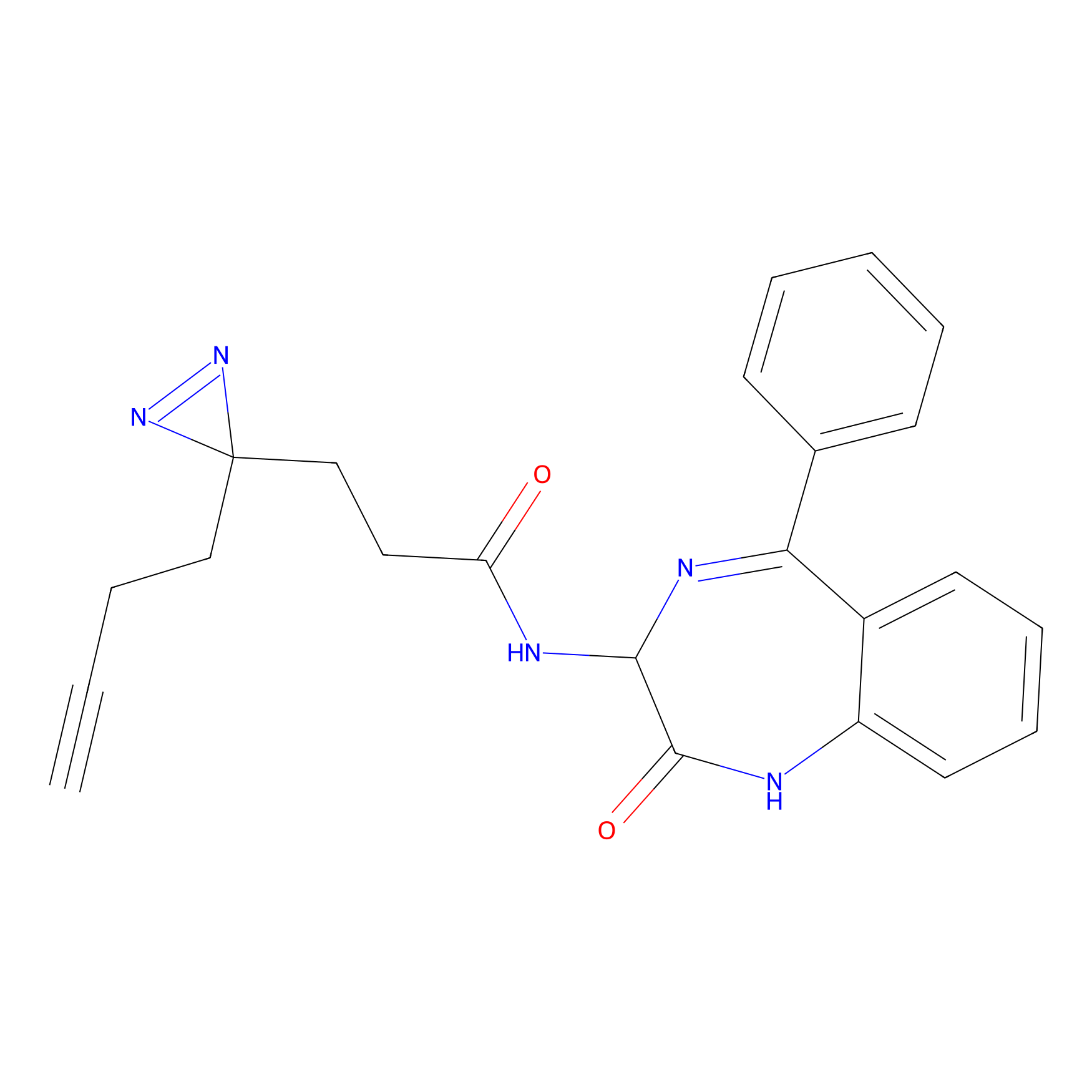
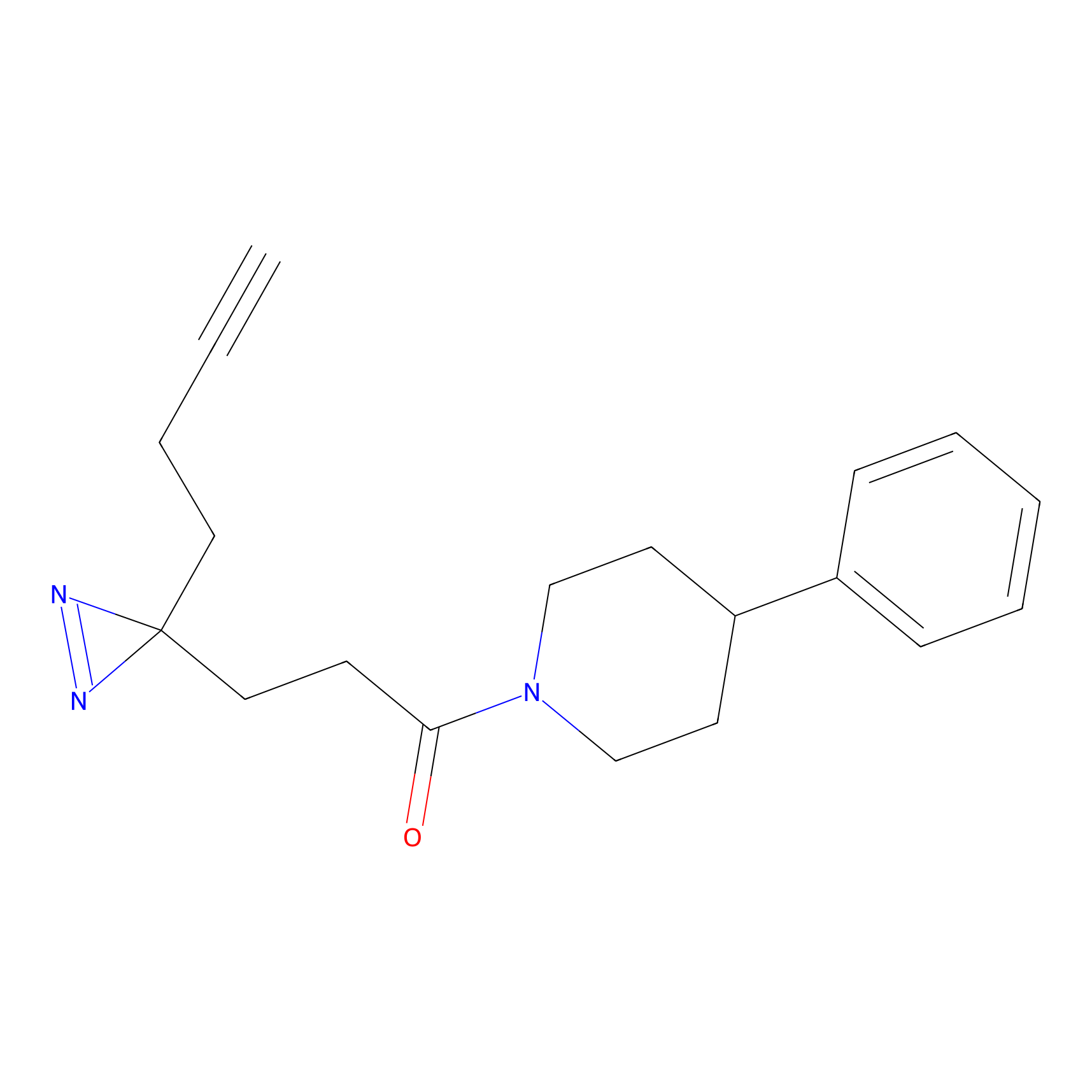
AHL-Pu-1 Probe Info |
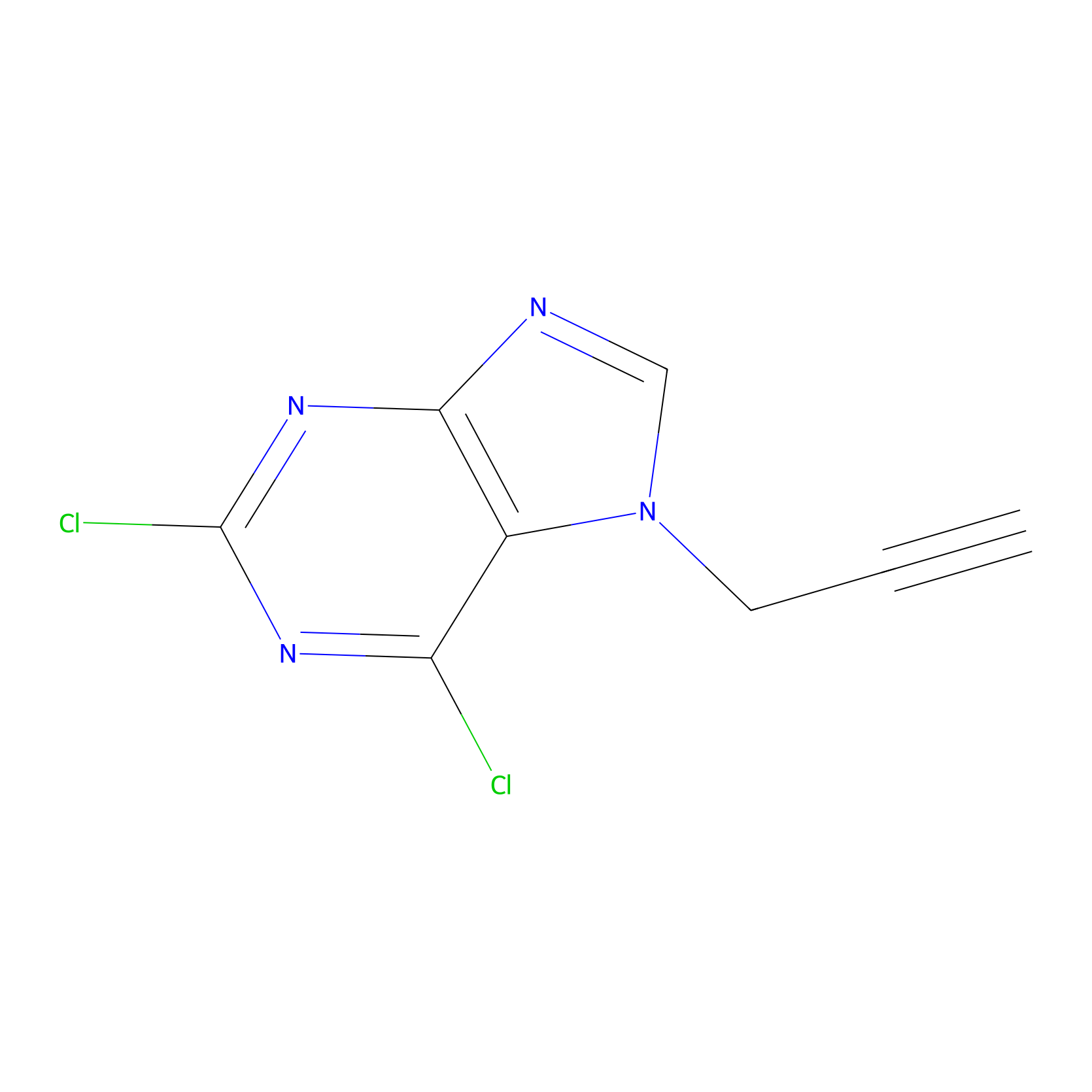 |
C244(2.27) | LDD0169 | [3] | |
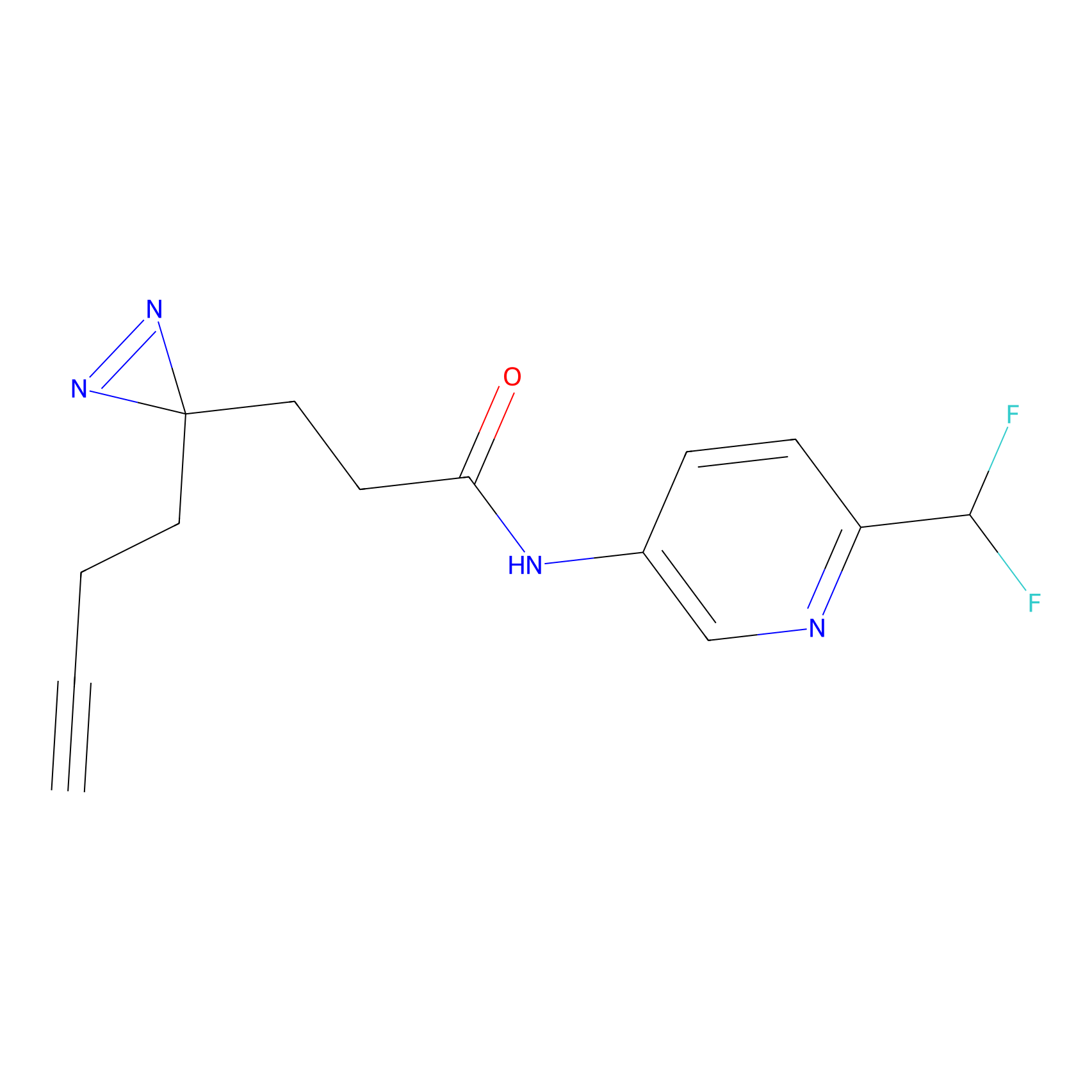
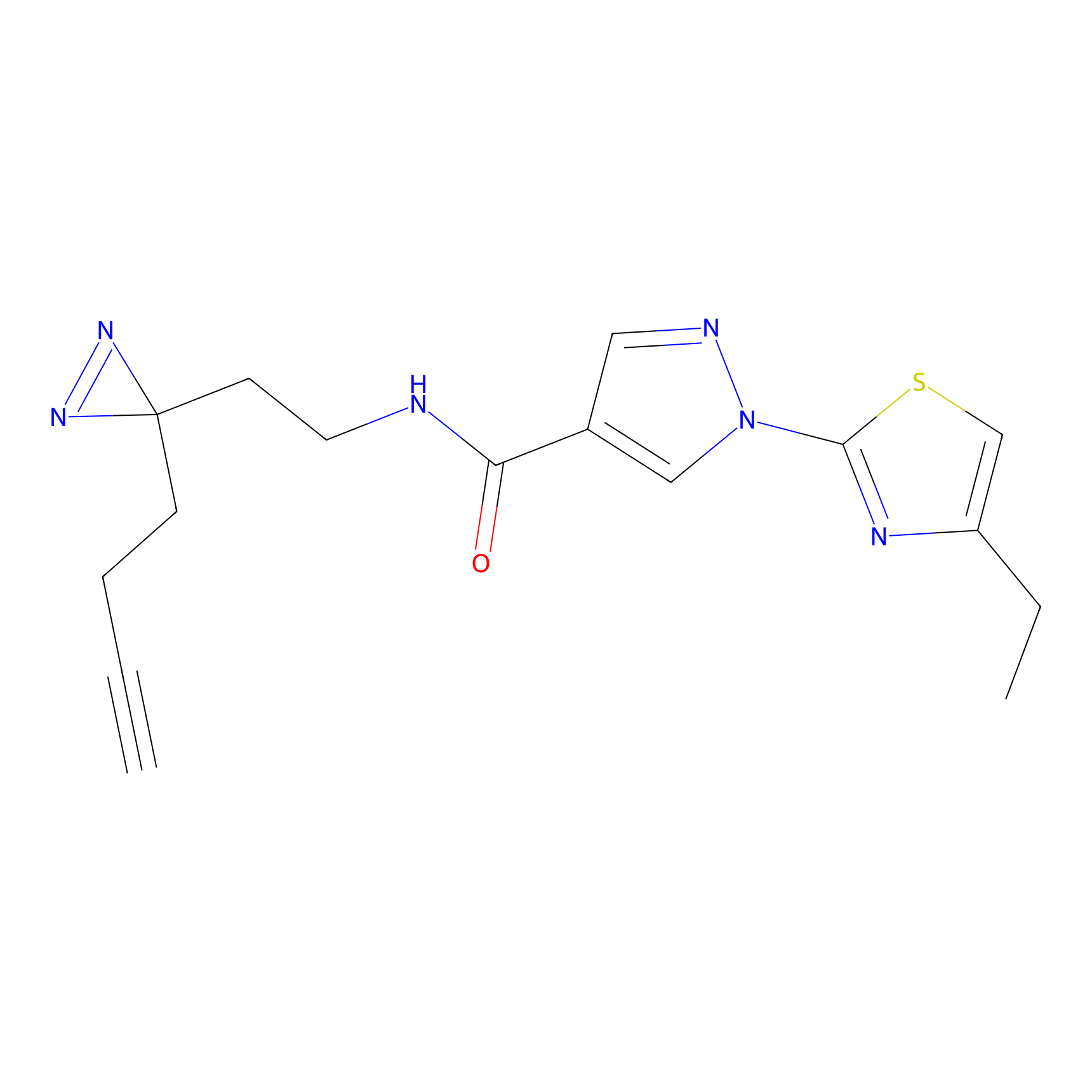
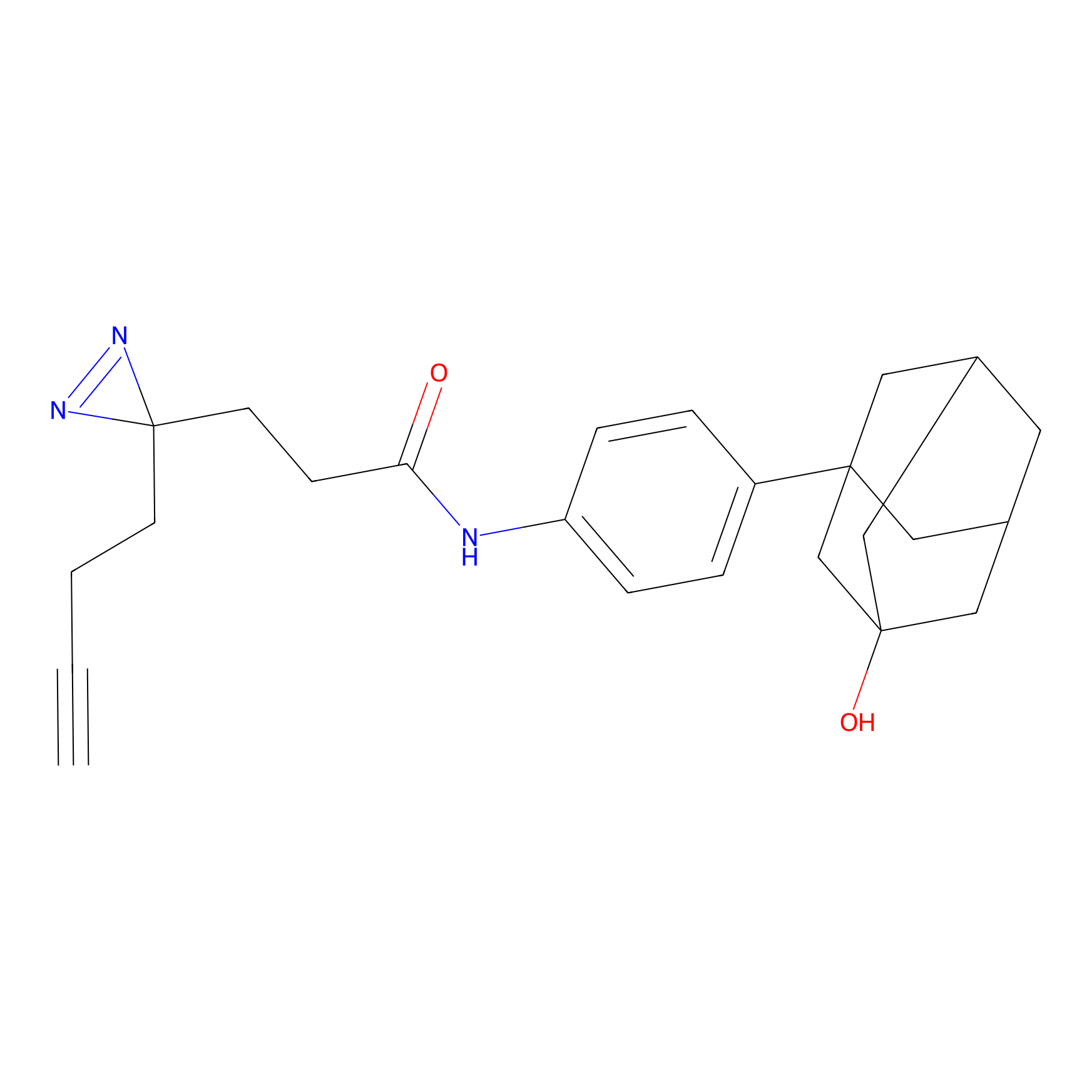
|
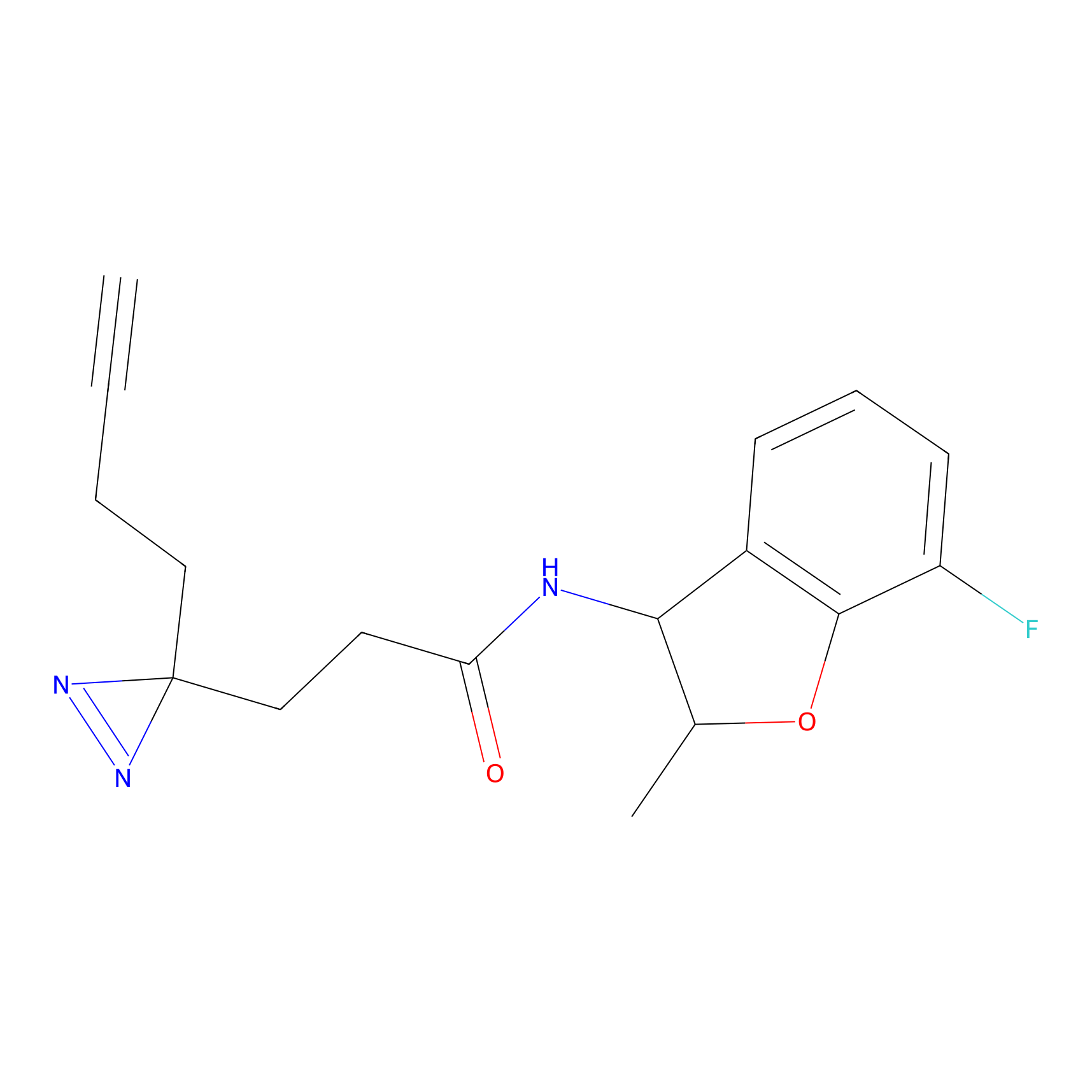
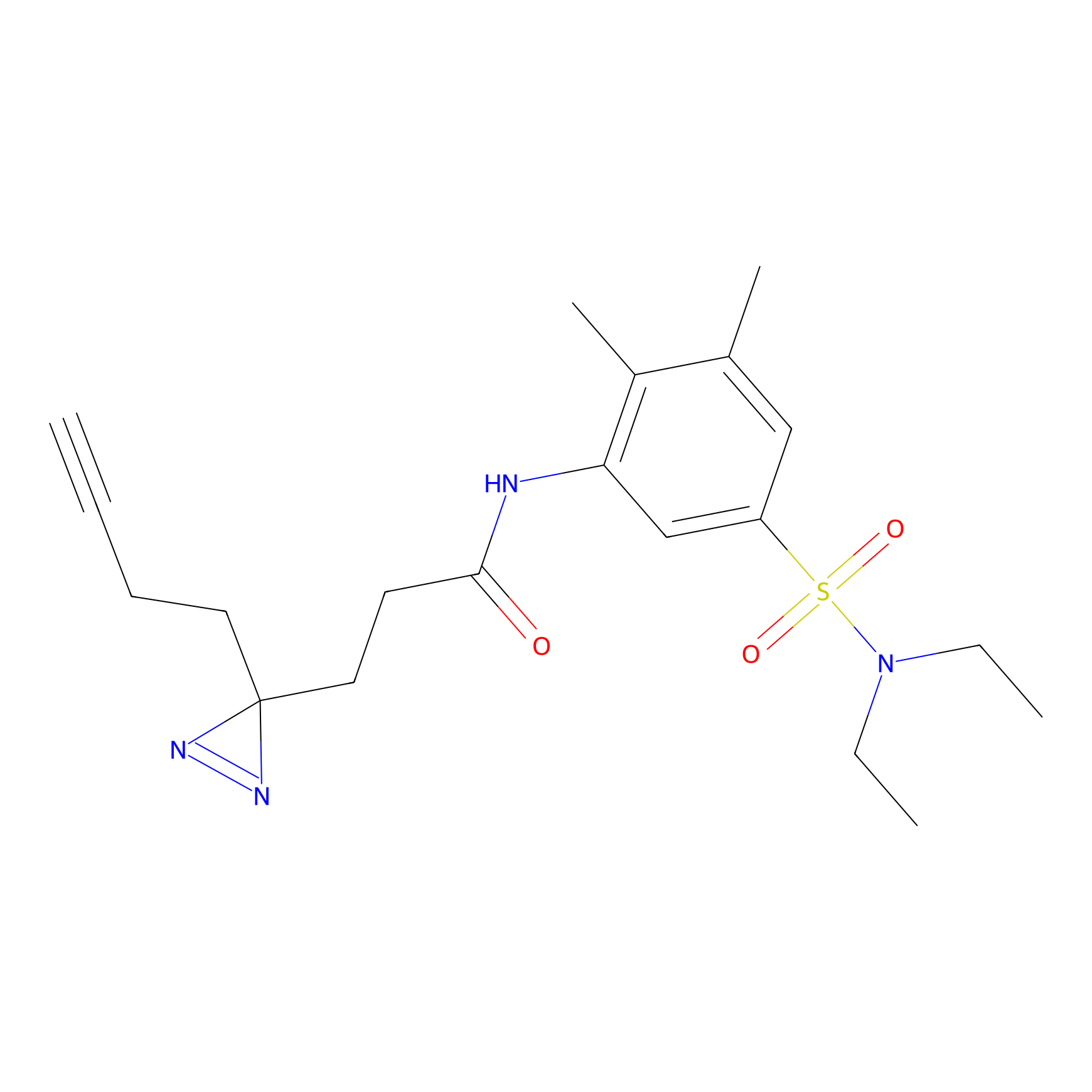
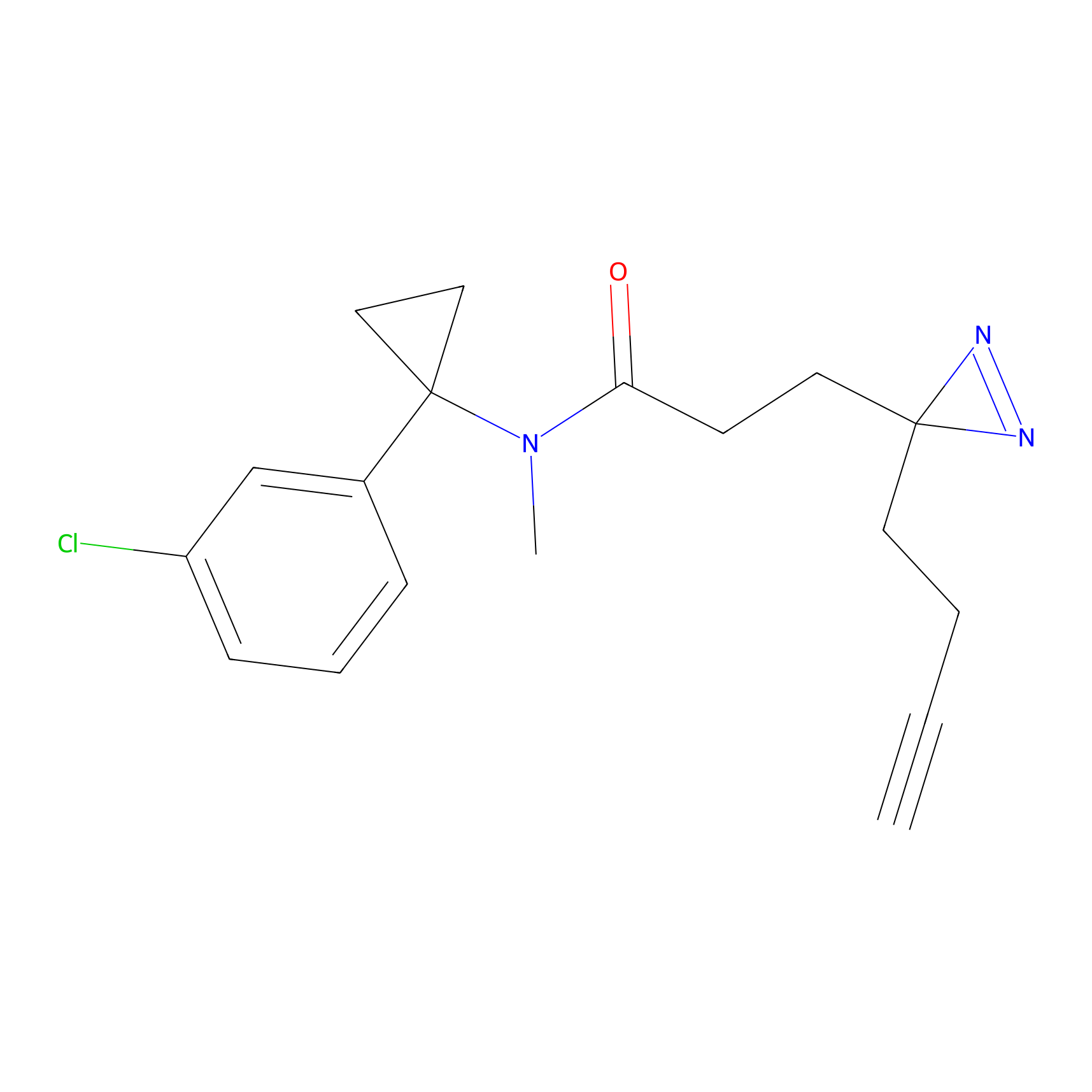
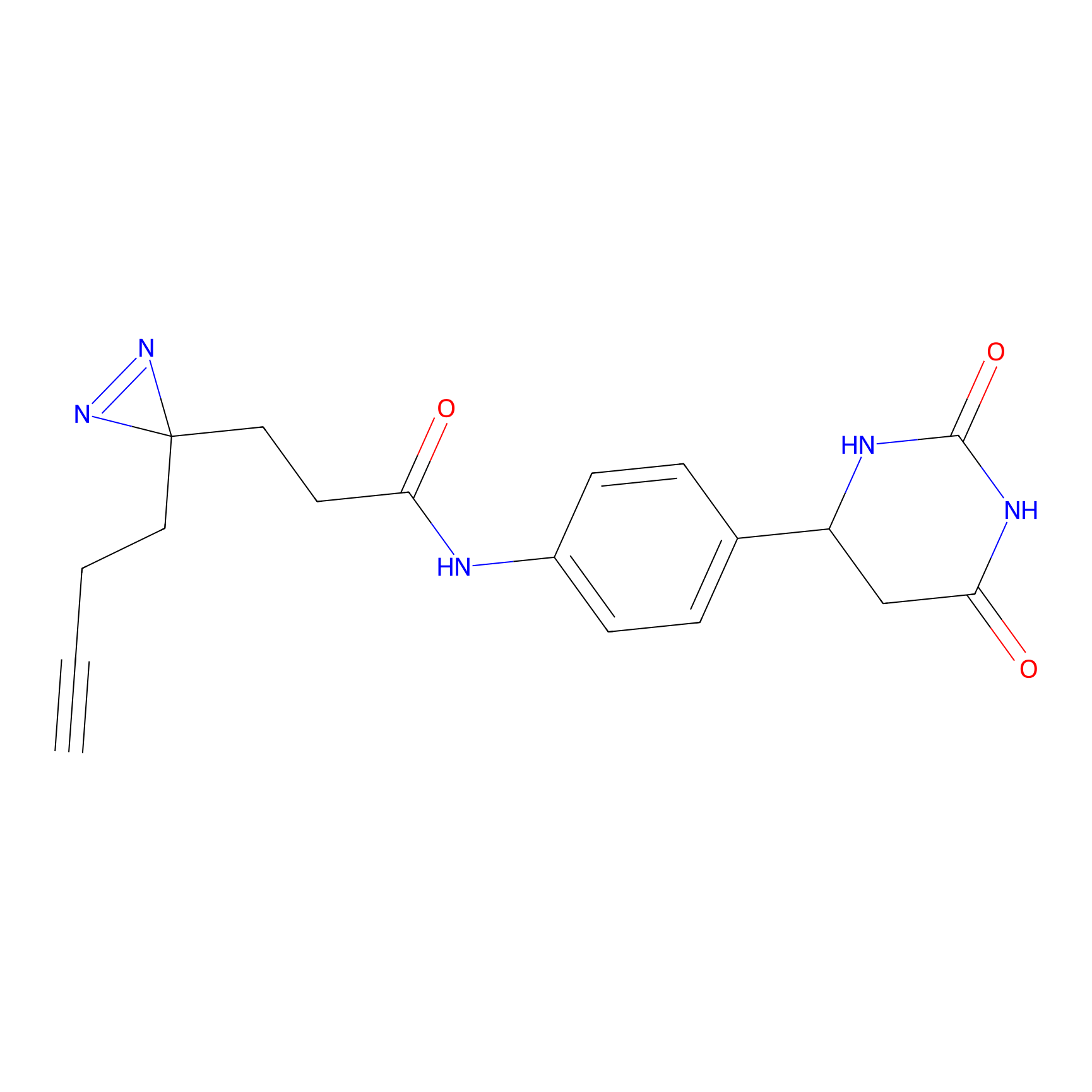
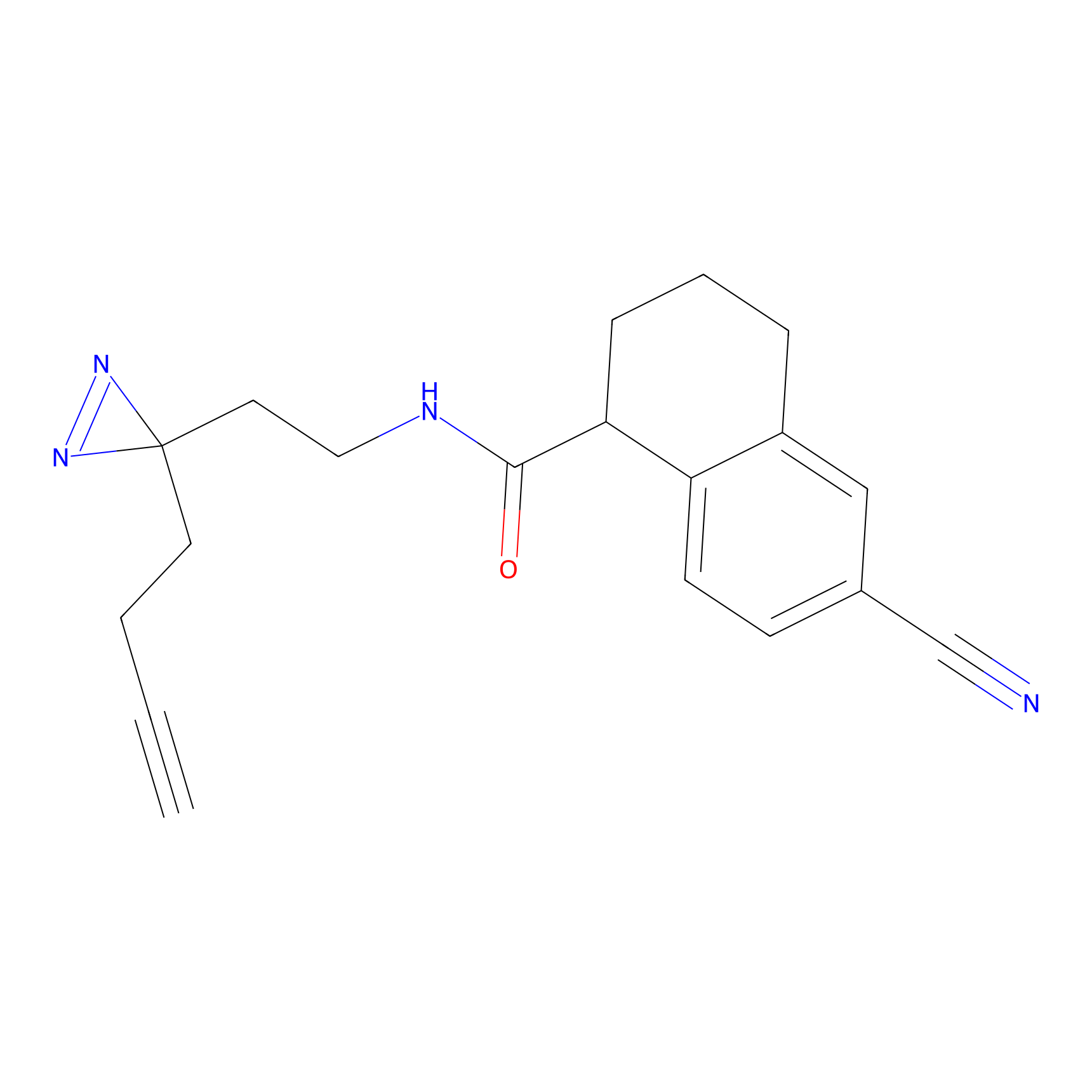
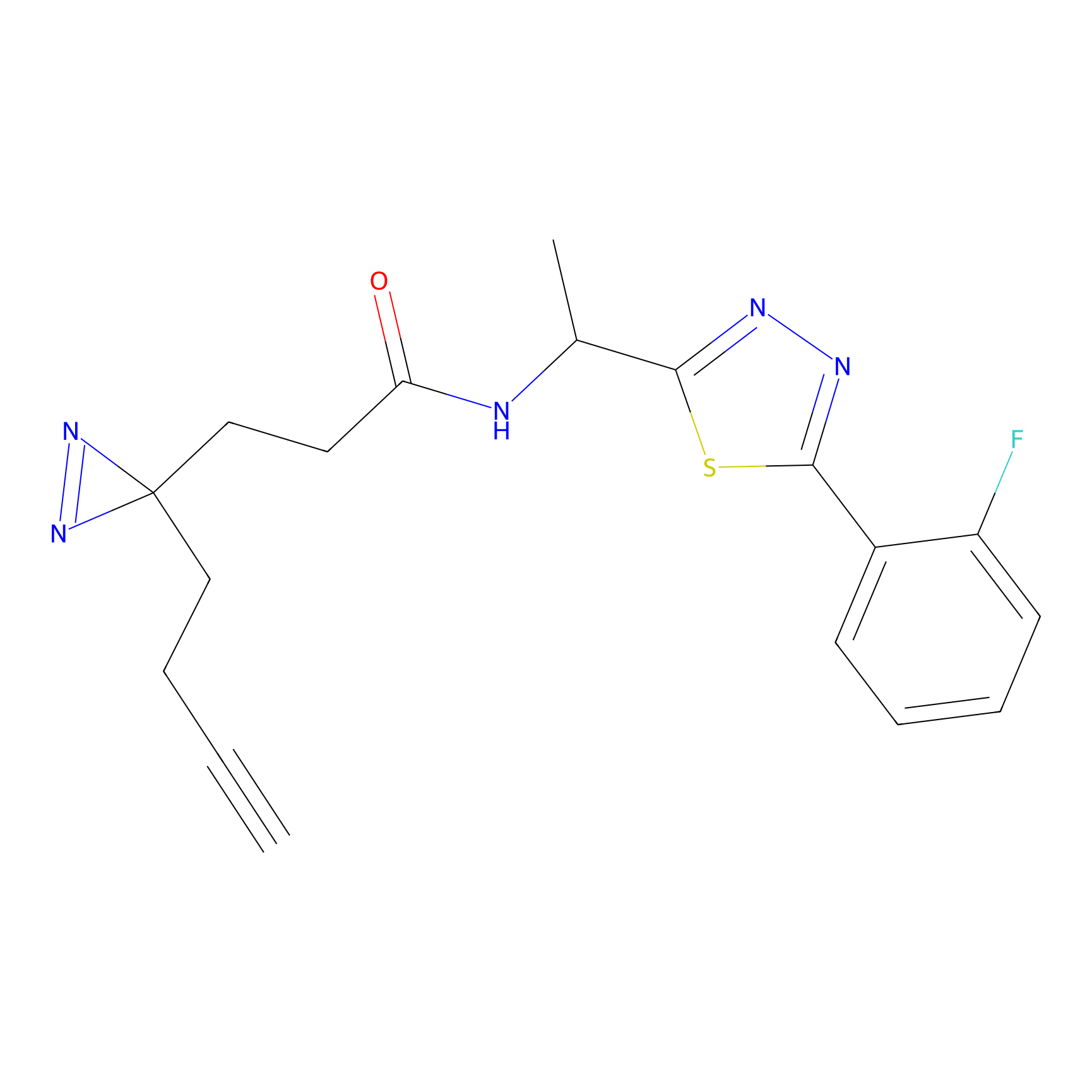
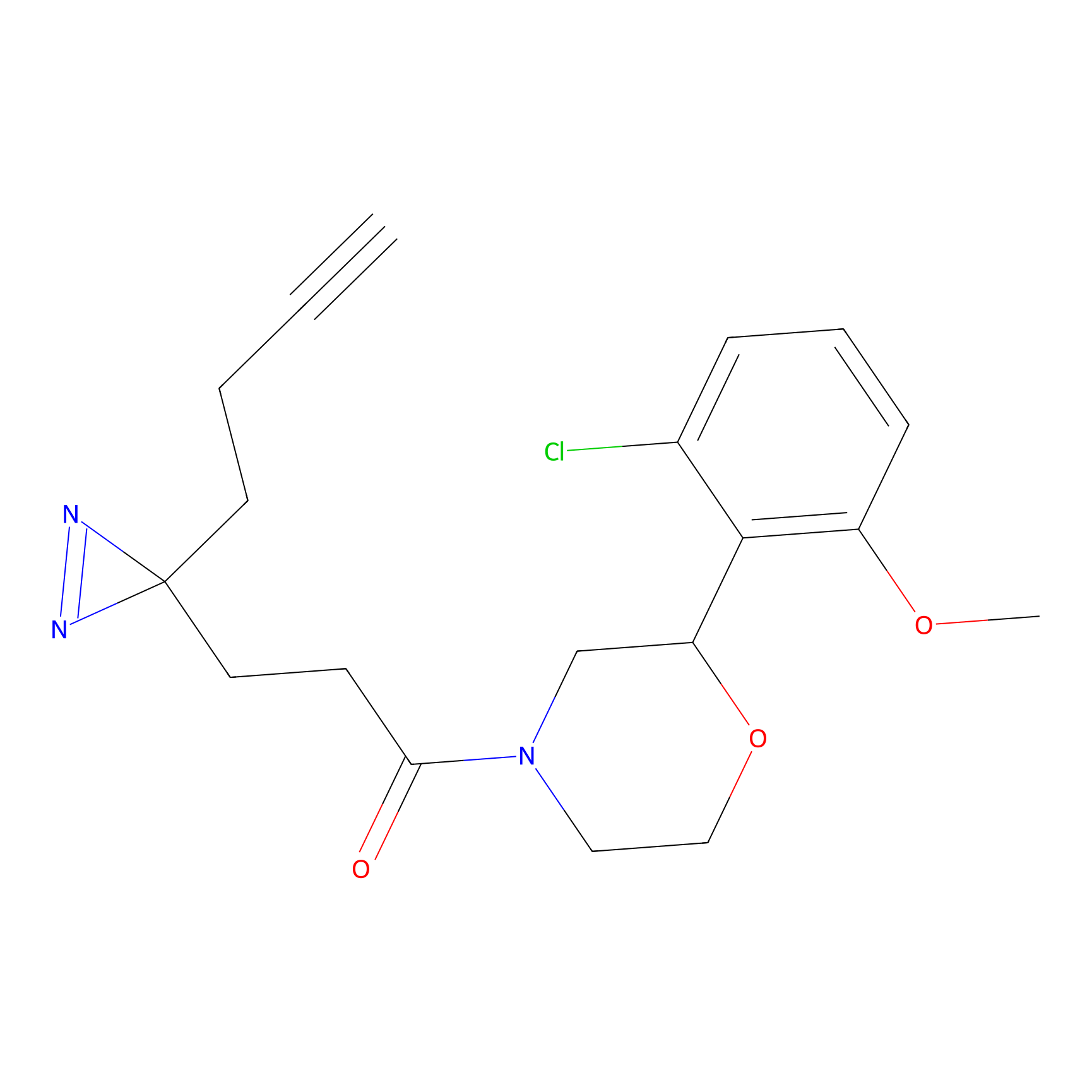
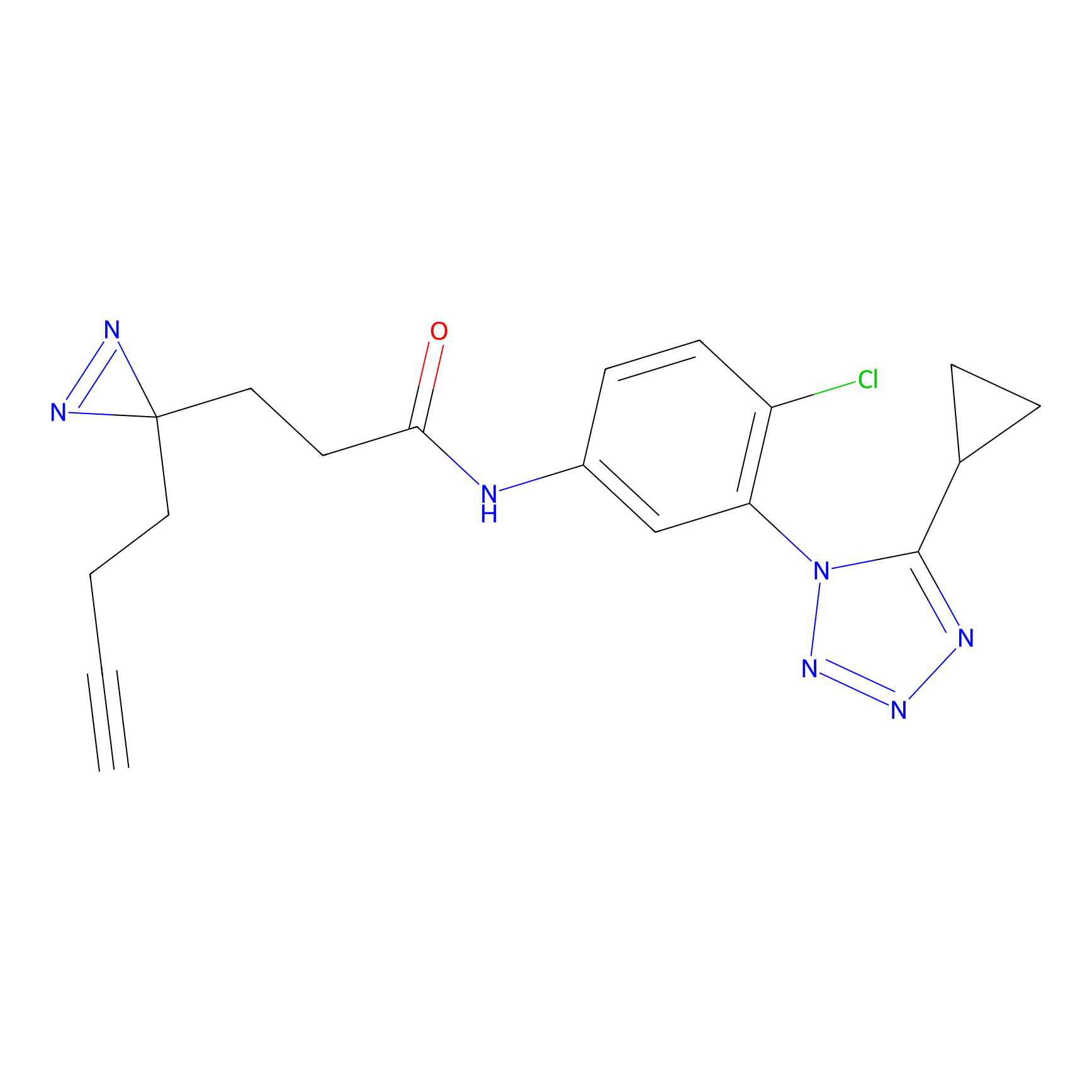
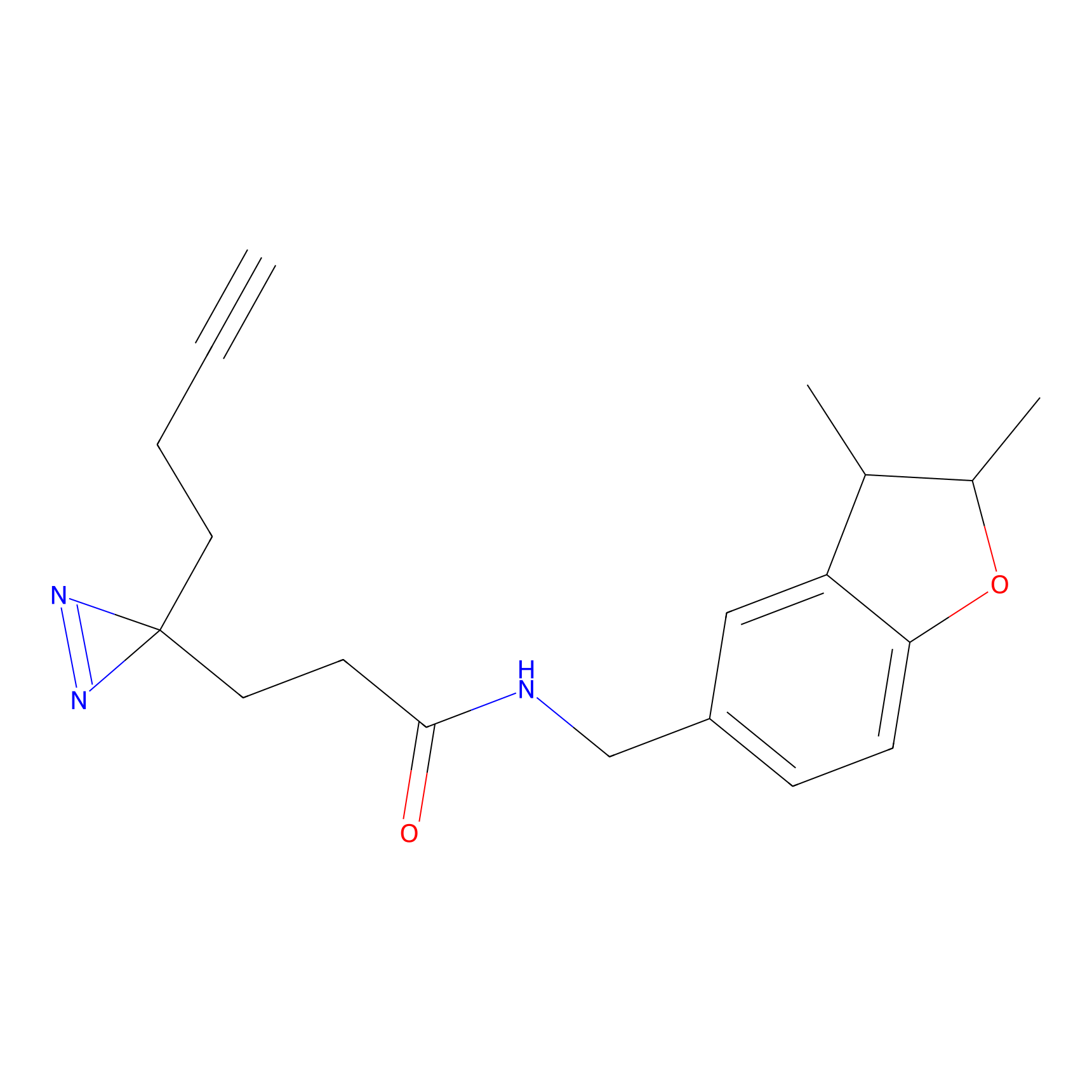
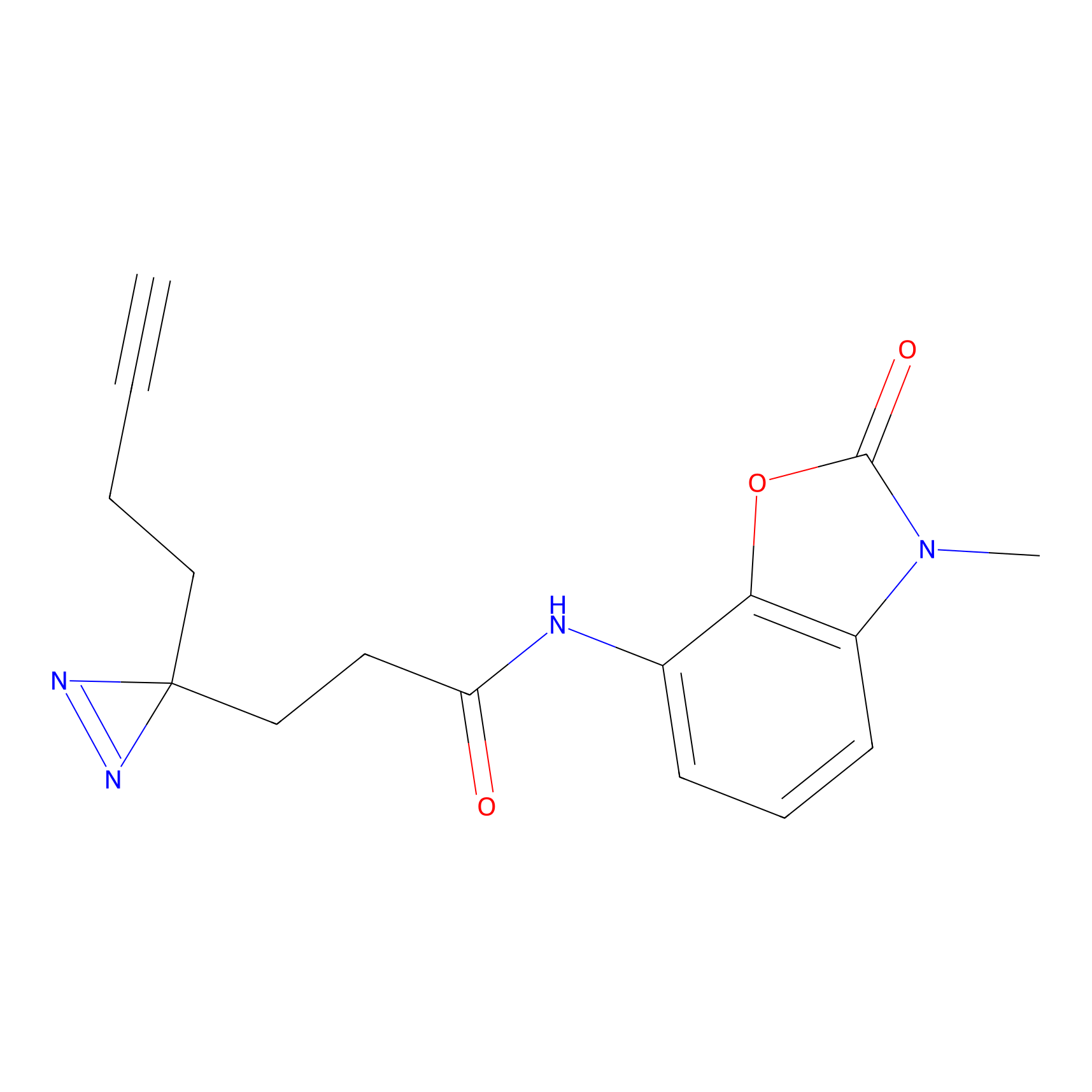
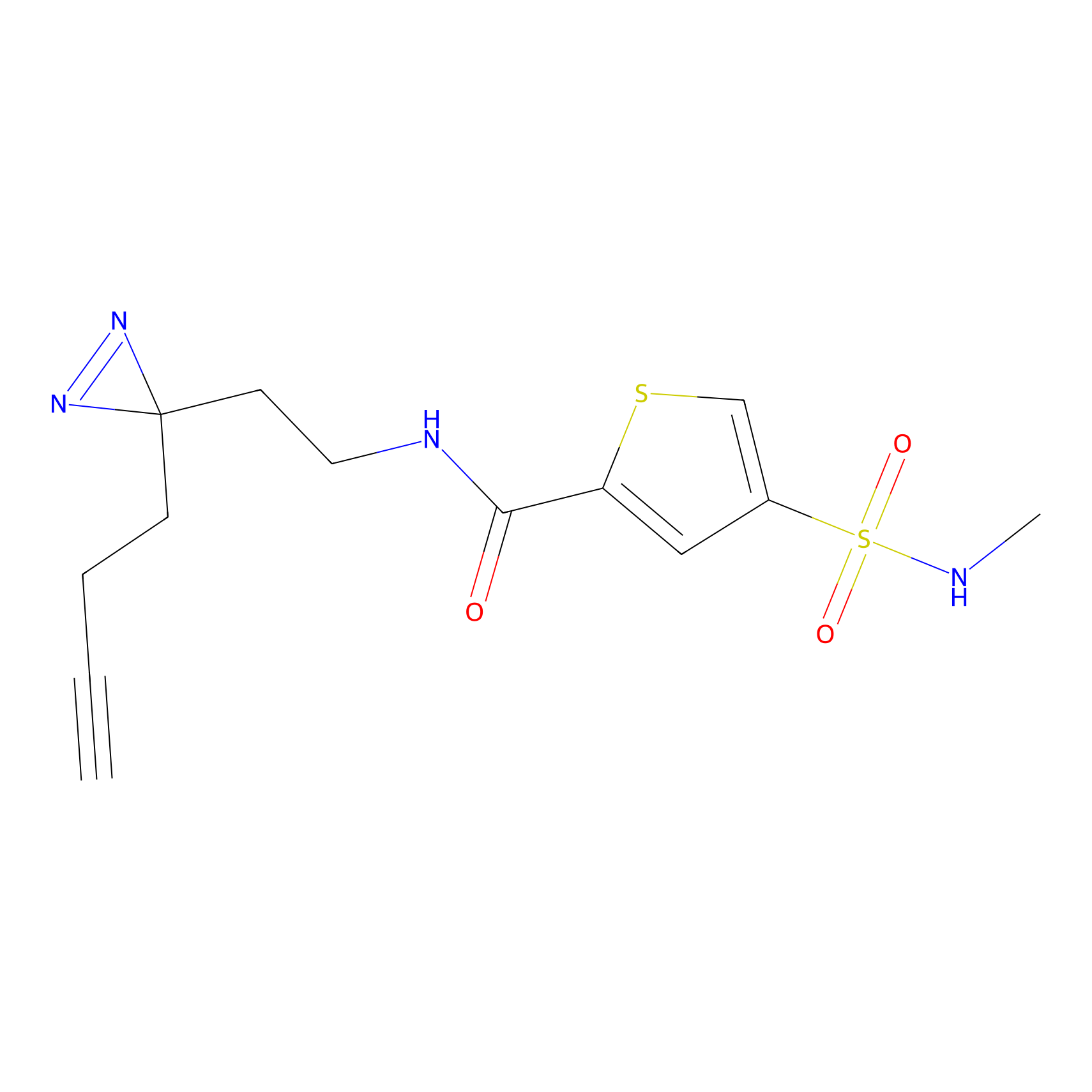
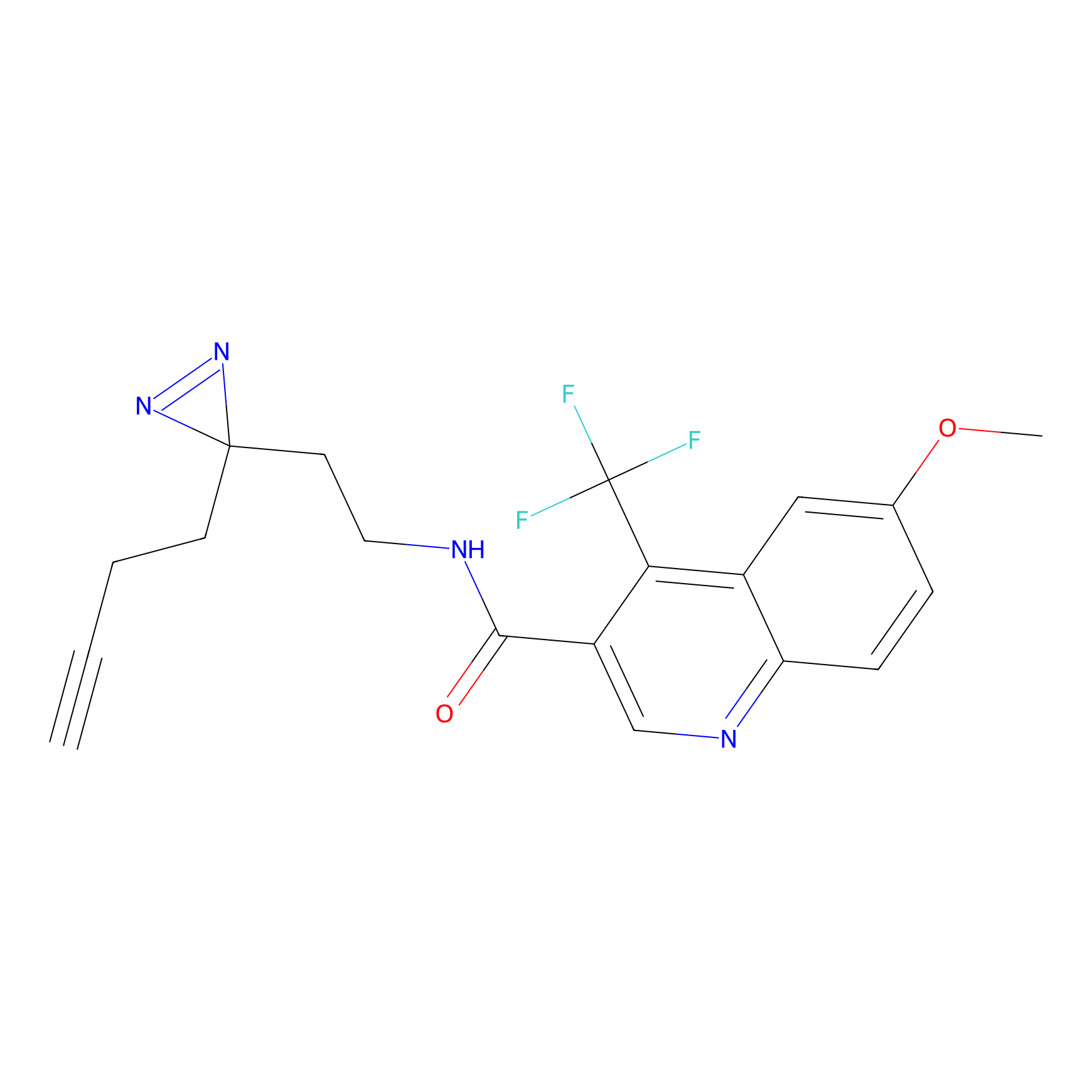
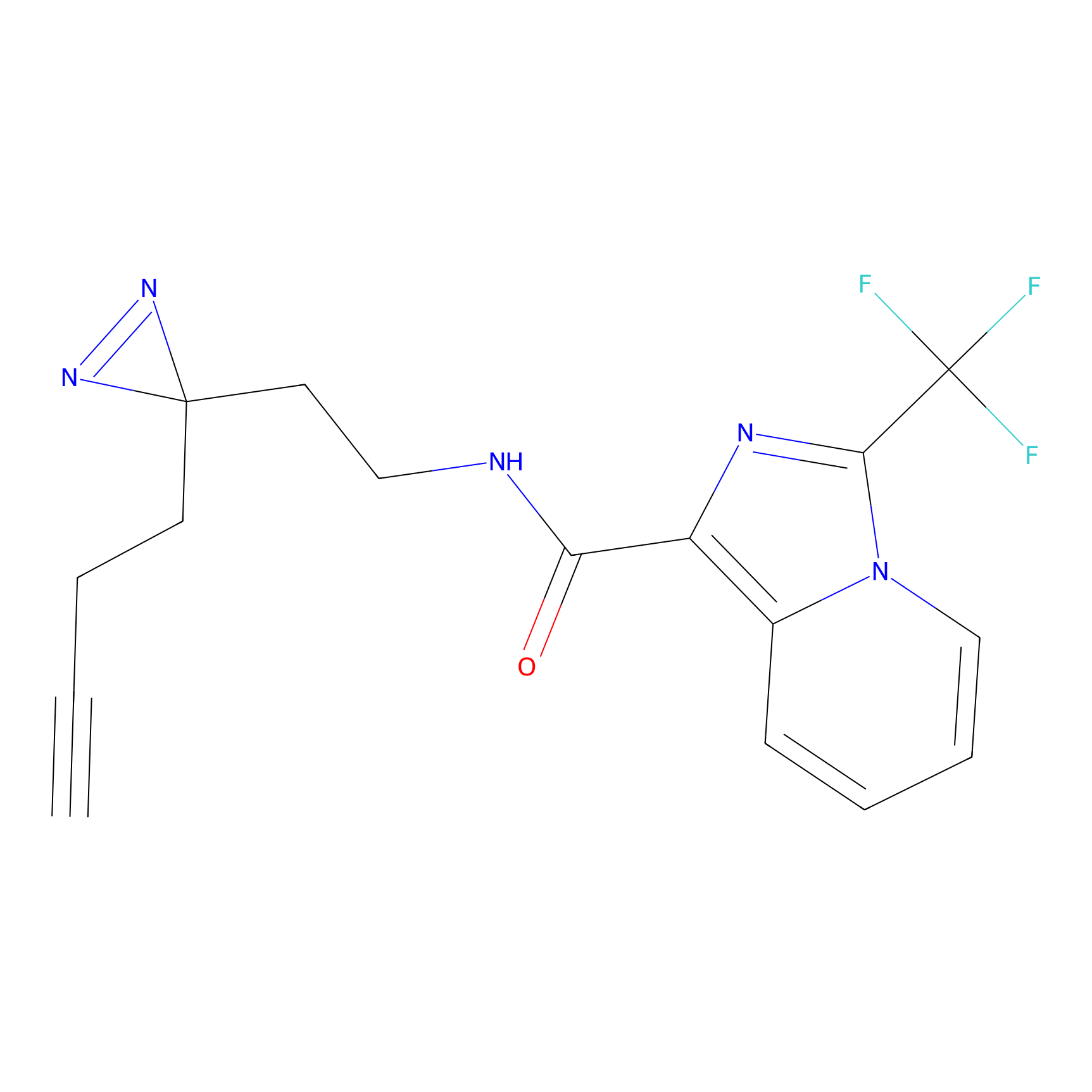
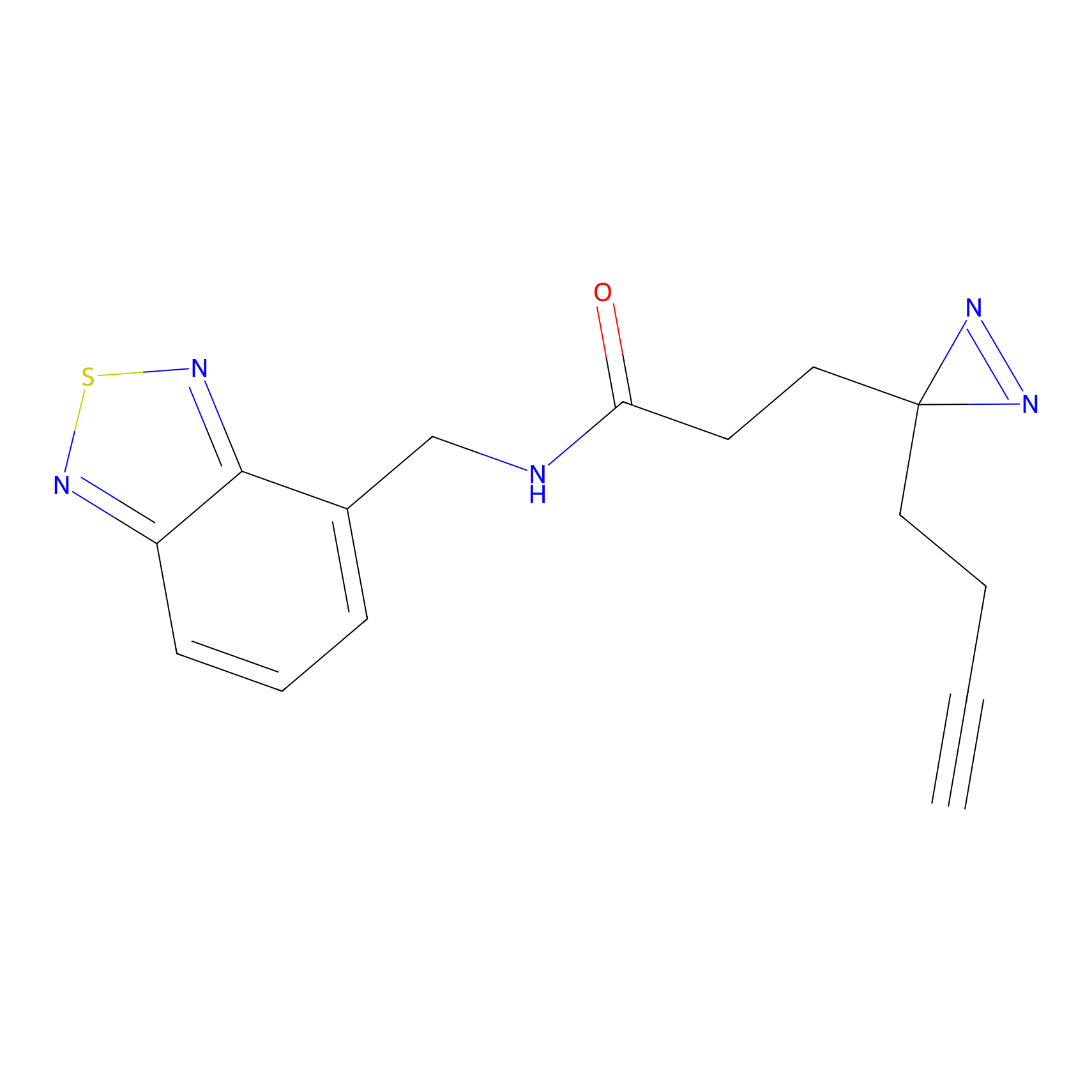
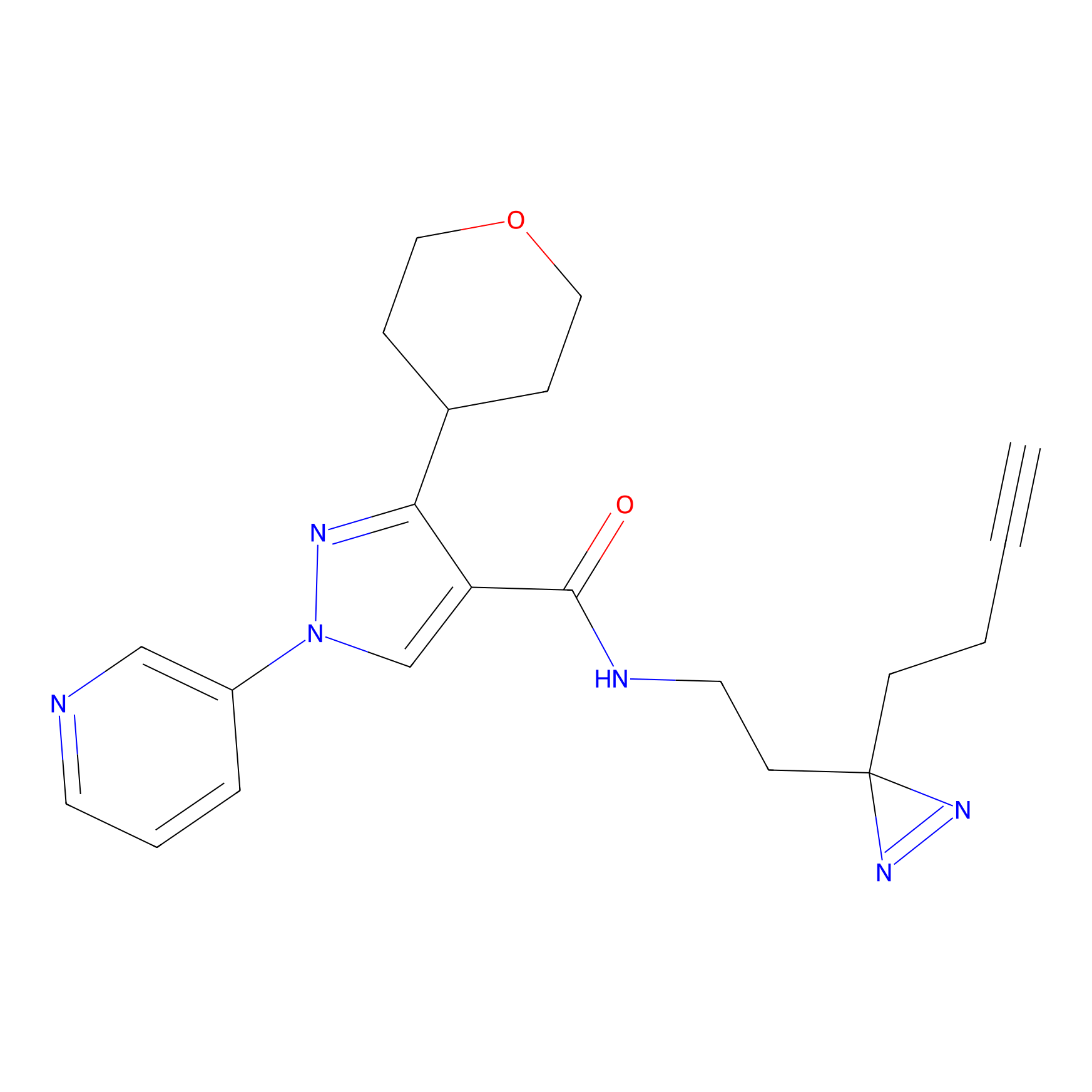
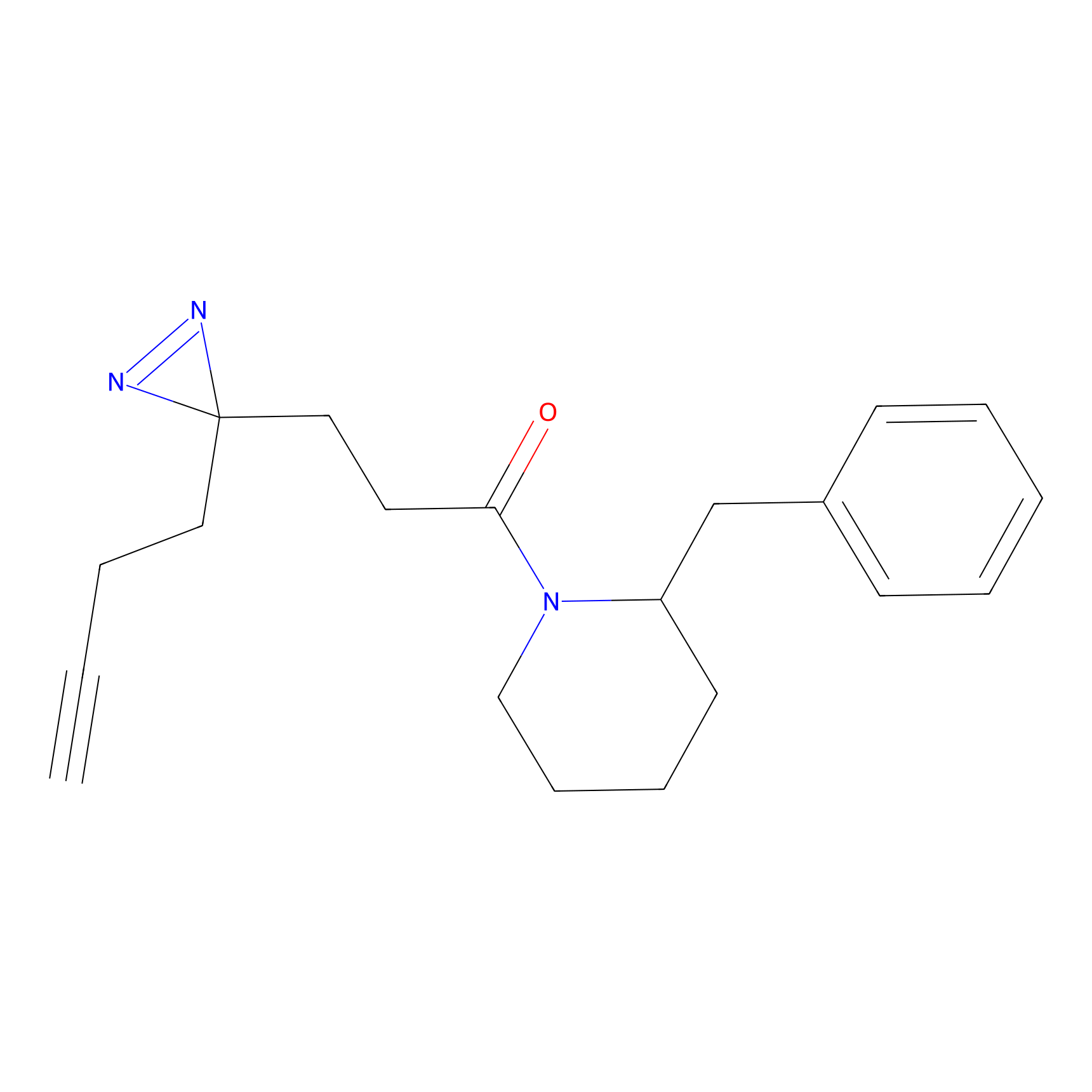
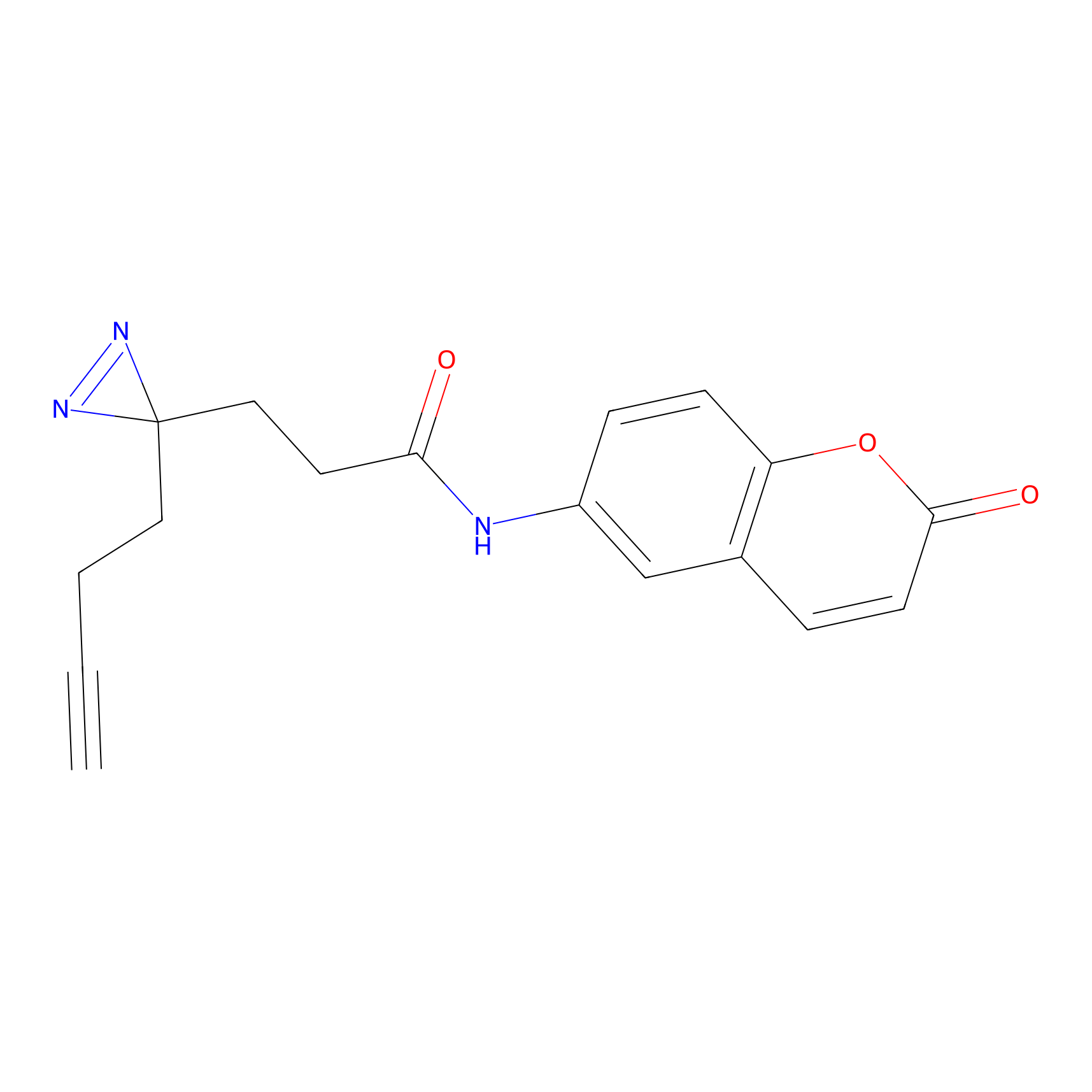
DBIA Probe Info |
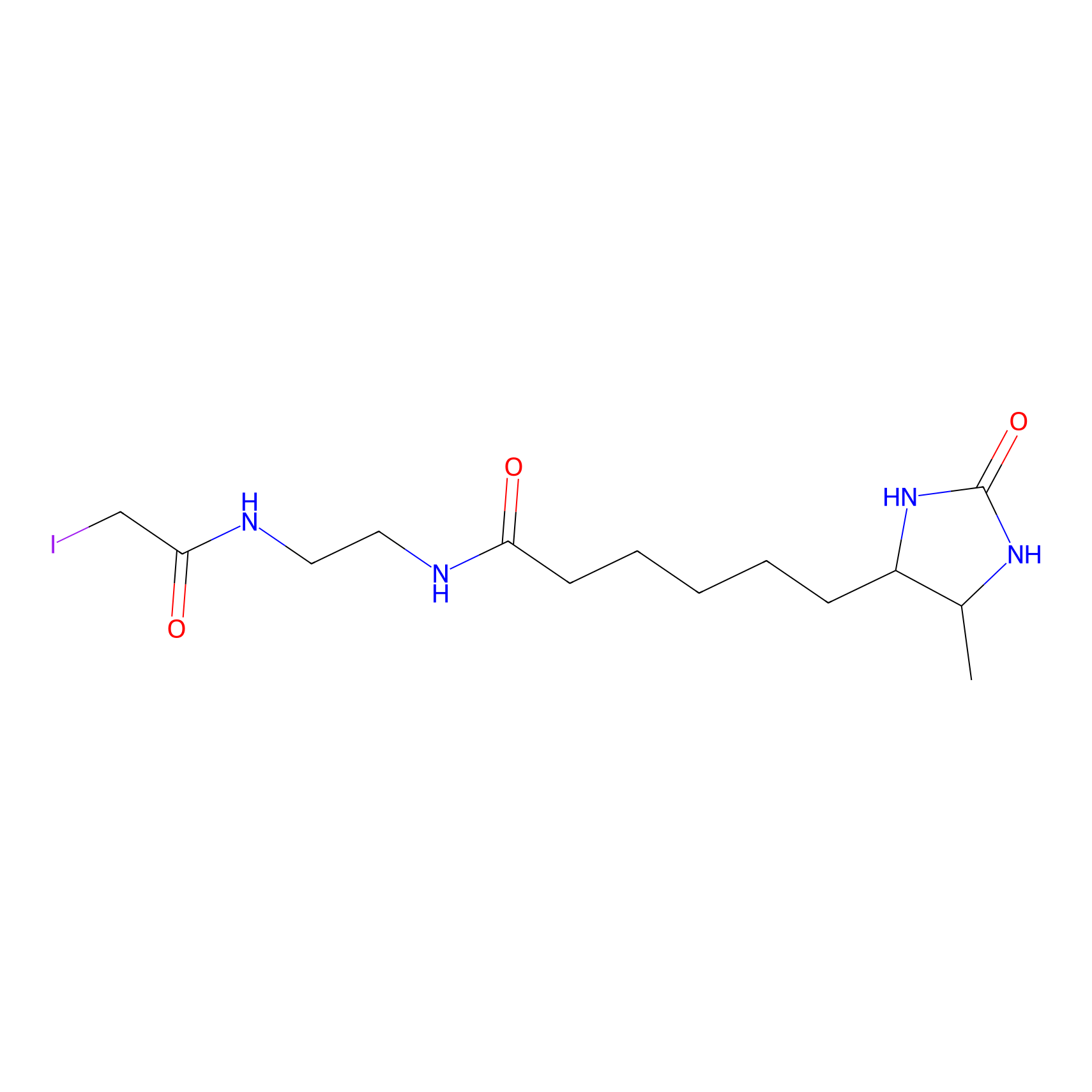 |
C244(1.05) | LDD1492 | [4] | |
|
4-Iodoacetamidophenylacetylene Probe Info |
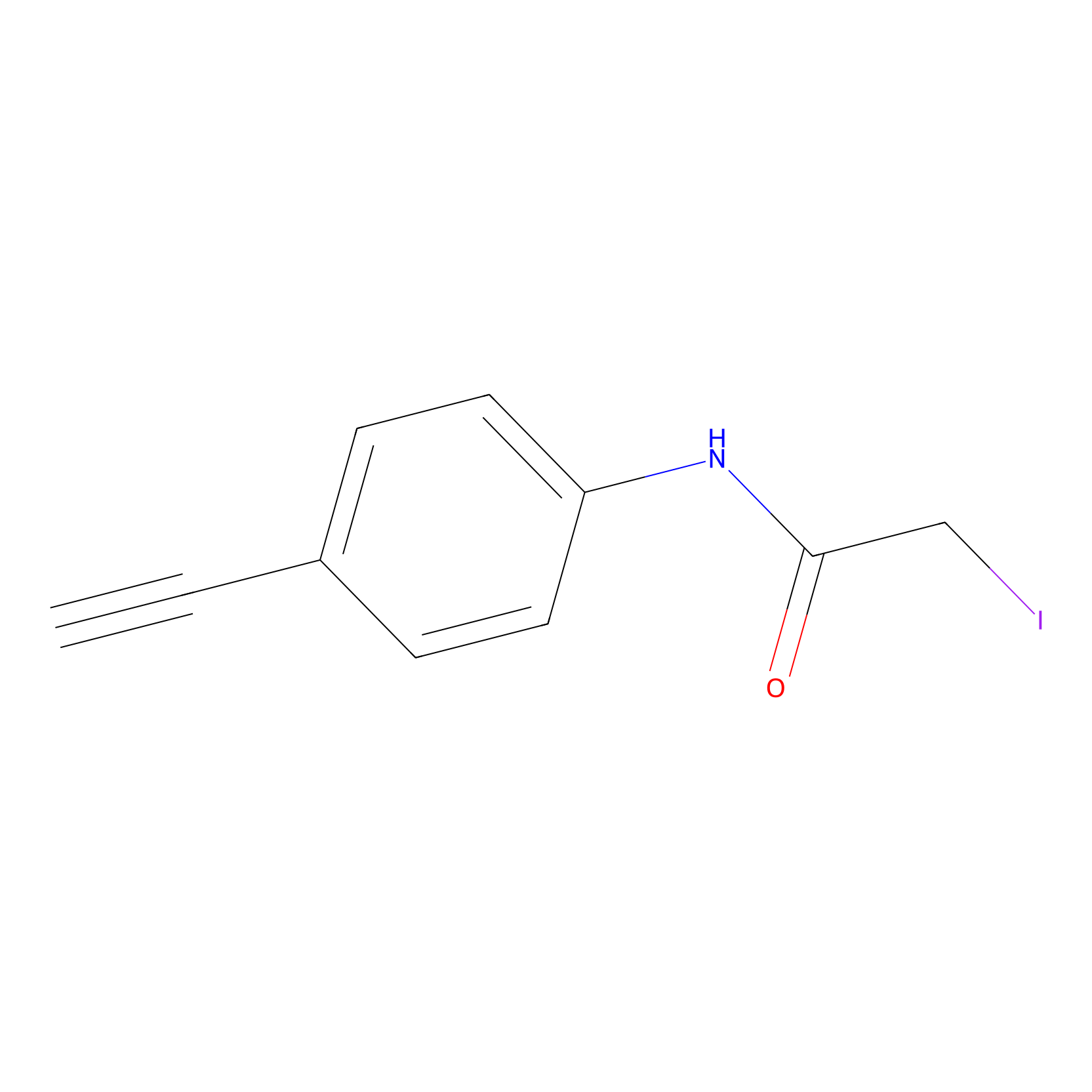 |
N.A. | LDD0038 | [5] | |
|
IA-alkyne Probe Info |
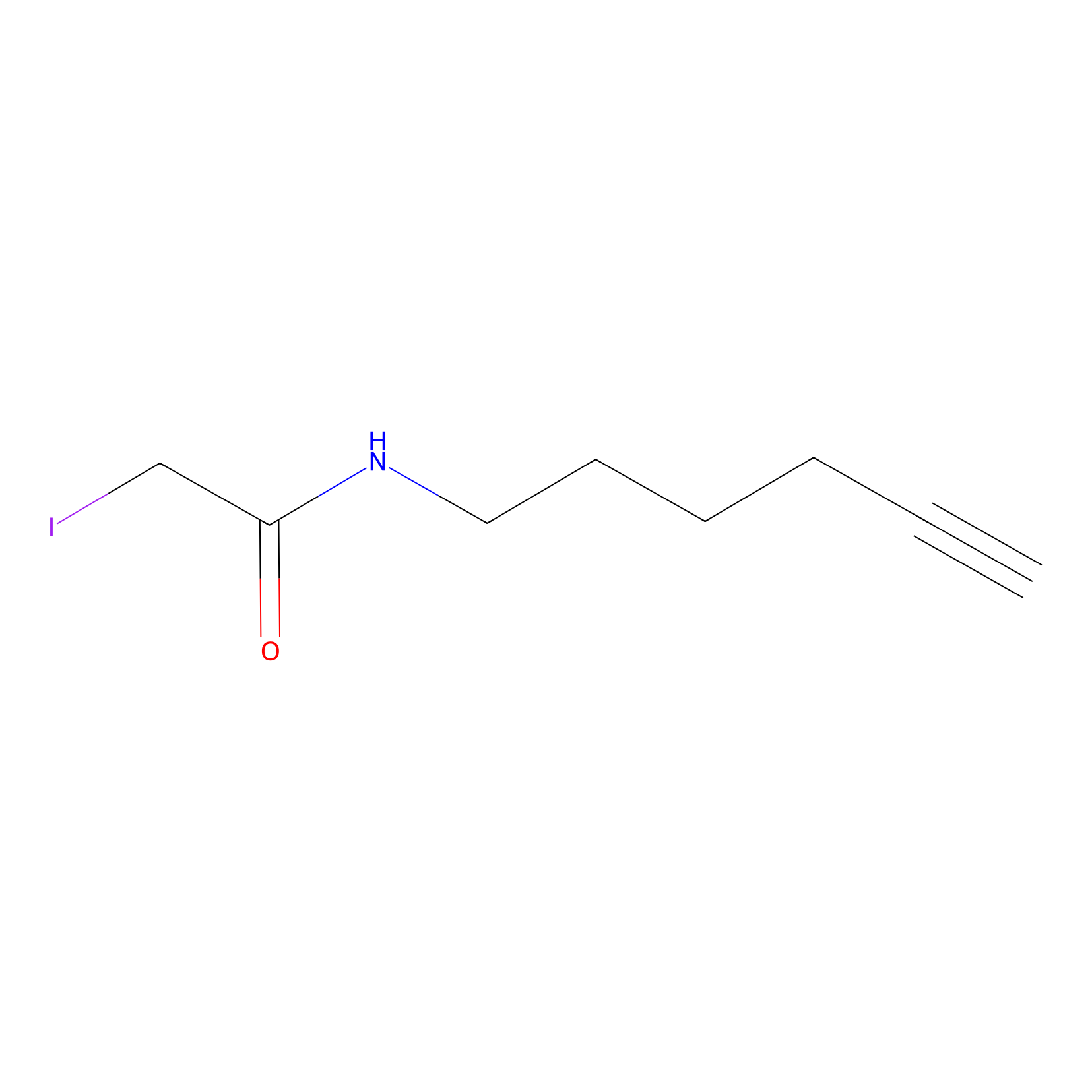 |
N.A. | LDD0036 | [5] | |
|
IPIAA_L Probe Info |
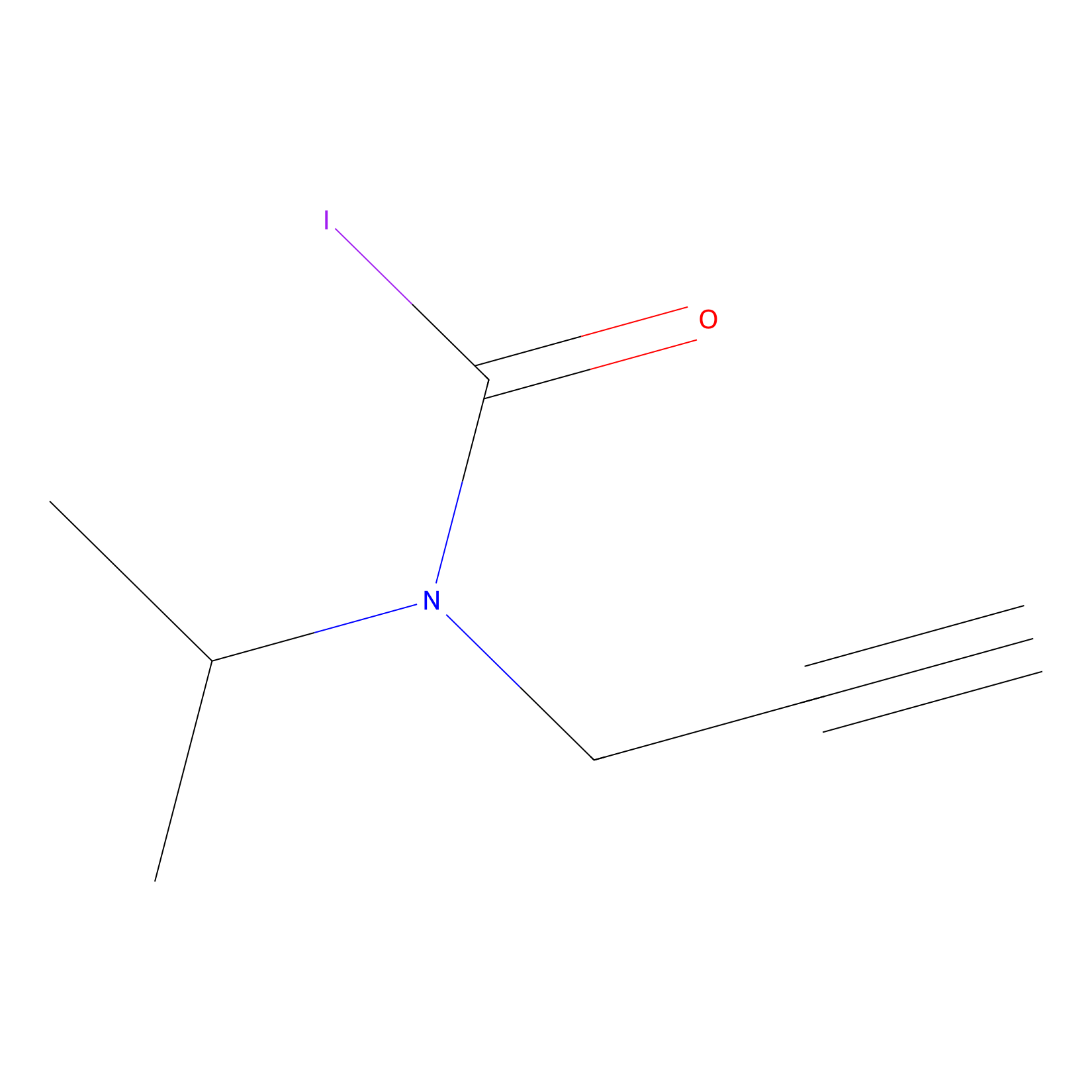 |
N.A. | LDD0031 | [6] | |
|
Lodoacetamide azide Probe Info |
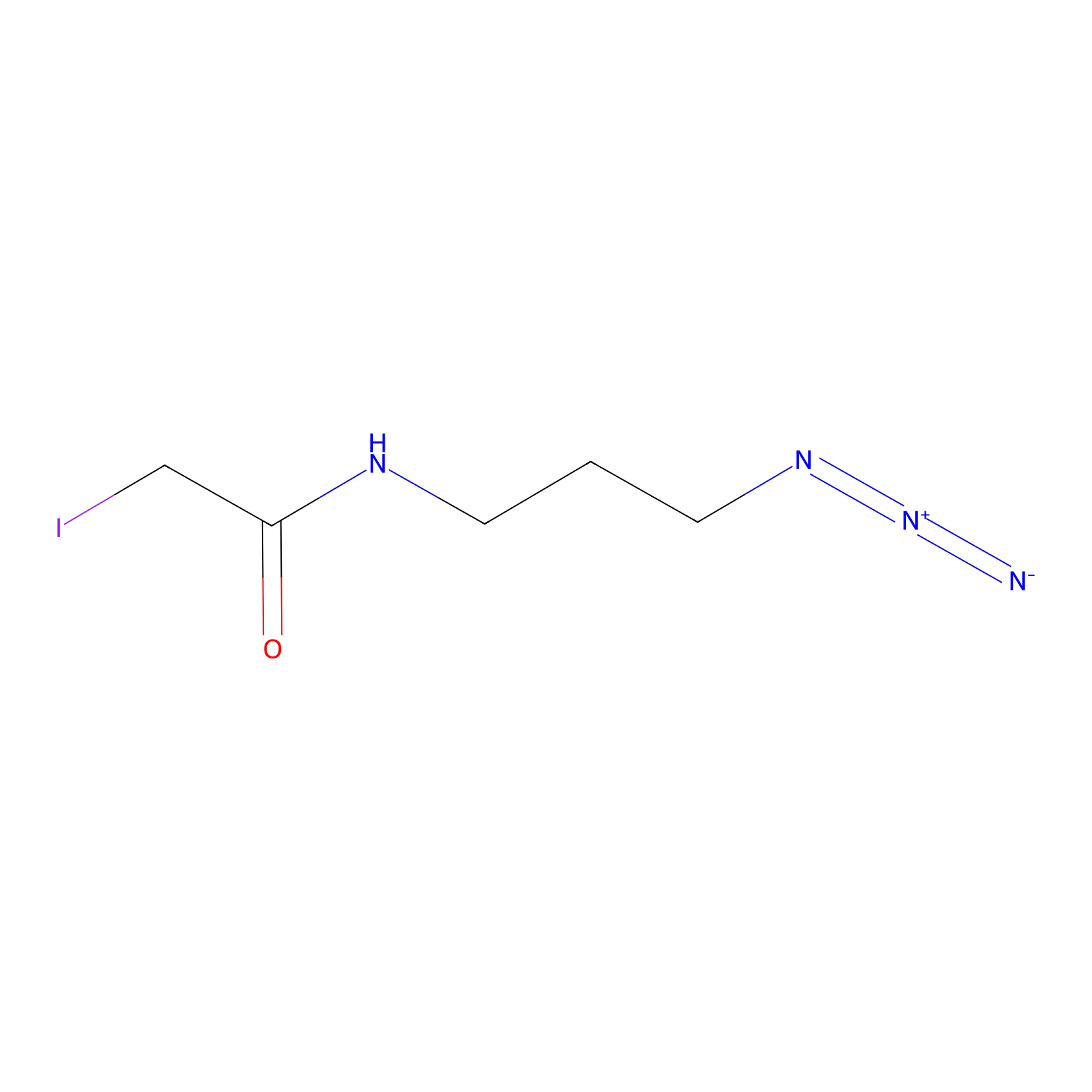 |
N.A. | LDD0037 | [5] | |
|
IPM Probe Info |
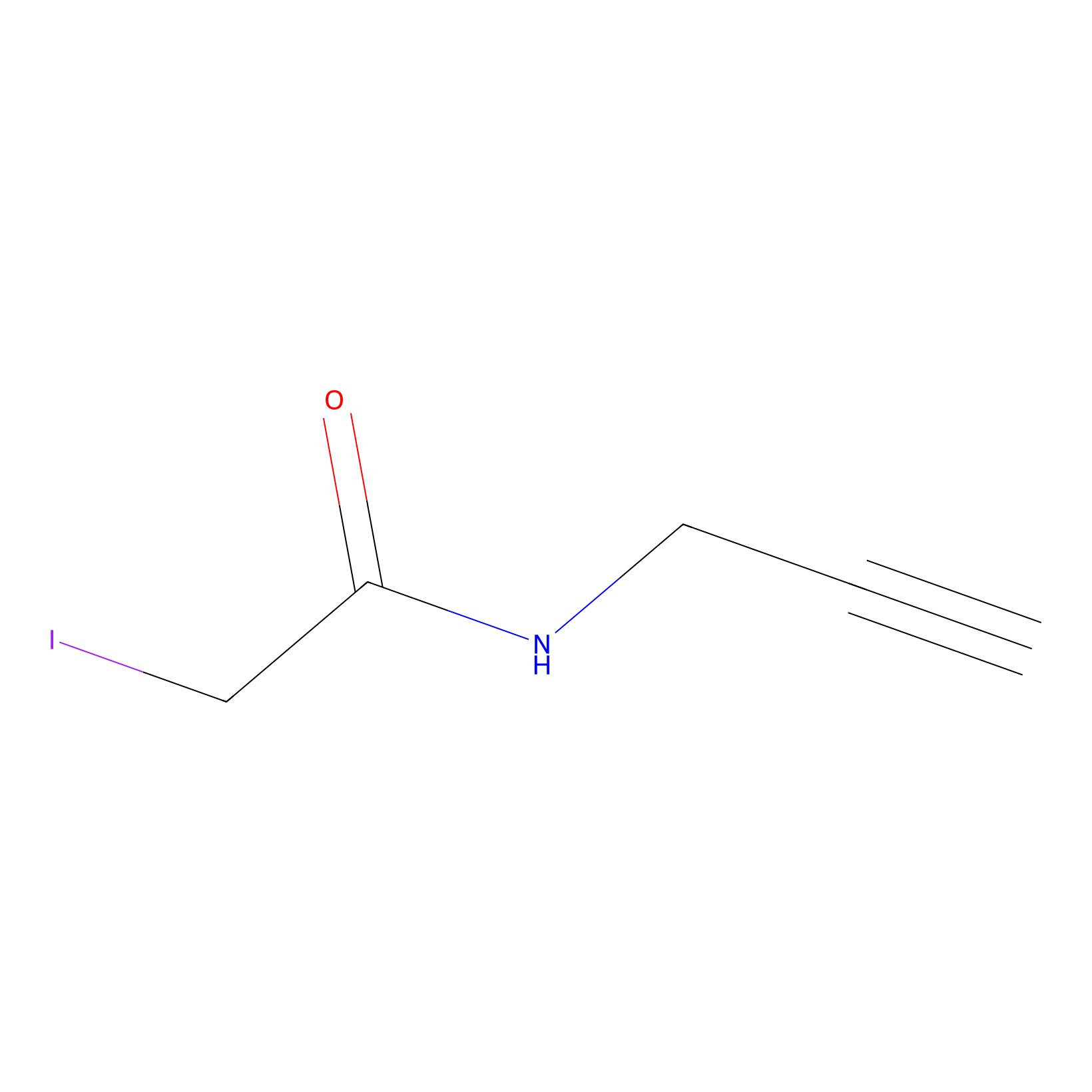 |
N.A. | LDD2156 | [7] | |
|
W1 Probe Info |
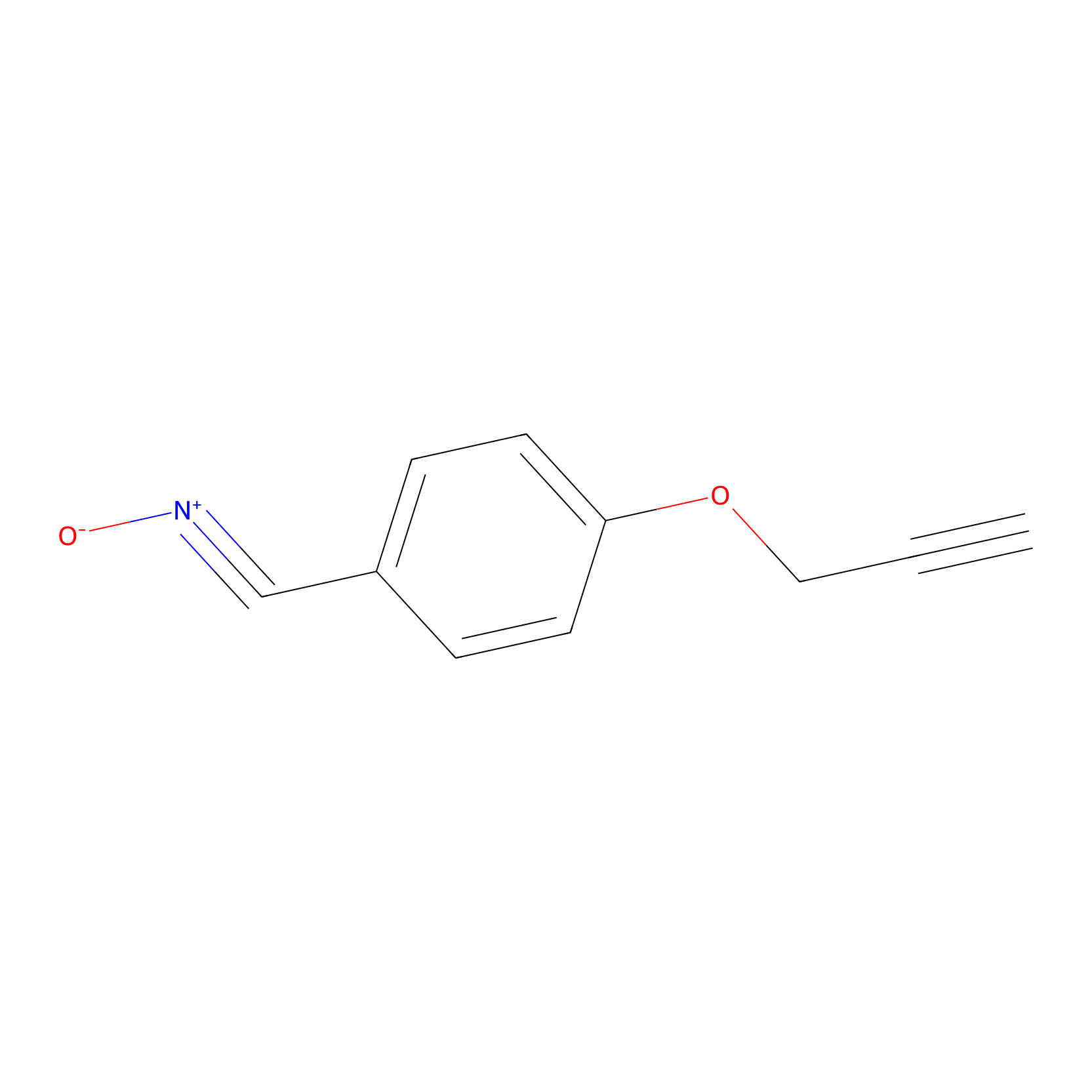 |
N.A. | LDD0236 | [8] | |
|
AOyne Probe Info |
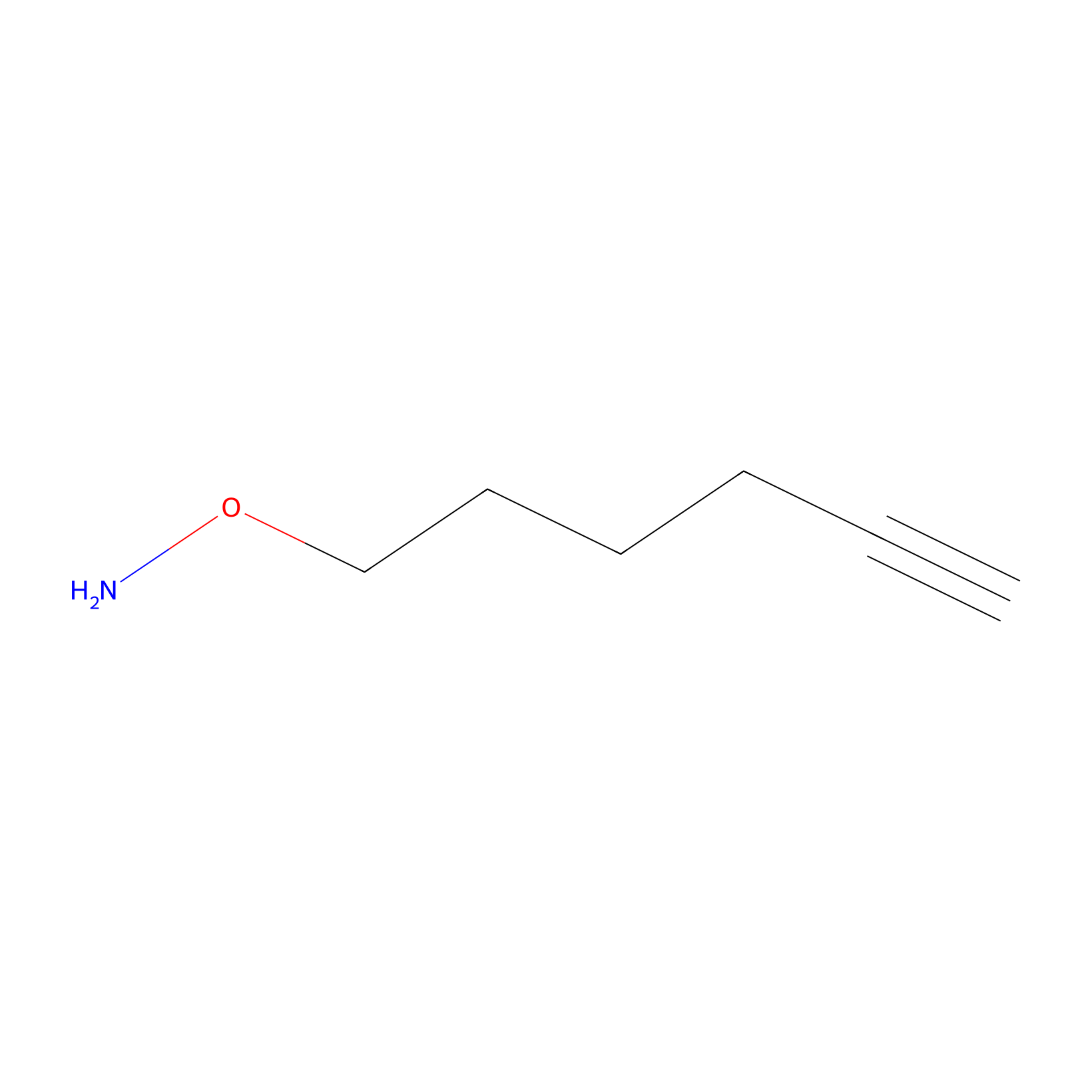 |
12.40 | LDD0443 | [9] | |
PAL-AfBPP Probe
Competitor(s) Related to This Target
| Competitor ID | Name | Cell line | Binding Site(Ratio) | Interaction ID | Ref |
|---|---|---|---|---|---|
| LDCM0026 | 4SU-RNA+native RNA | HEK-293T | C244(2.27) | LDD0169 | [3] |
| LDCM0214 | AC1 | HEK-293T | C244(0.94) | LDD1507 | [4] |
| LDCM0215 | AC10 | HEK-293T | C244(0.98) | LDD1508 | [4] |
| LDCM0226 | AC11 | HEK-293T | C244(1.05) | LDD1509 | [4] |
| LDCM0237 | AC12 | HEK-293T | C244(1.11) | LDD1510 | [4] |
| LDCM0259 | AC14 | HEK-293T | C244(1.04) | LDD1512 | [4] |
| LDCM0270 | AC15 | HEK-293T | C244(1.06) | LDD1513 | [4] |
| LDCM0276 | AC17 | HEK-293T | C244(1.00) | LDD1515 | [4] |
| LDCM0277 | AC18 | HEK-293T | C244(1.04) | LDD1516 | [4] |
| LDCM0278 | AC19 | HEK-293T | C244(0.99) | LDD1517 | [4] |
| LDCM0279 | AC2 | HEK-293T | C244(0.98) | LDD1518 | [4] |
| LDCM0280 | AC20 | HEK-293T | C244(1.06) | LDD1519 | [4] |
| LDCM0281 | AC21 | HEK-293T | C244(0.87) | LDD1520 | [4] |
| LDCM0282 | AC22 | HEK-293T | C244(1.01) | LDD1521 | [4] |
| LDCM0283 | AC23 | HEK-293T | C244(1.02) | LDD1522 | [4] |
| LDCM0284 | AC24 | HEK-293T | C244(0.98) | LDD1523 | [4] |
| LDCM0285 | AC25 | HEK-293T | C244(1.06) | LDD1524 | [4] |
| LDCM0286 | AC26 | HEK-293T | C244(0.94) | LDD1525 | [4] |
| LDCM0287 | AC27 | HEK-293T | C244(1.09) | LDD1526 | [4] |
| LDCM0288 | AC28 | HEK-293T | C244(0.95) | LDD1527 | [4] |
| LDCM0289 | AC29 | HEK-293T | C244(0.99) | LDD1528 | [4] |
| LDCM0290 | AC3 | HEK-293T | C244(1.07) | LDD1529 | [4] |
| LDCM0291 | AC30 | HEK-293T | C244(0.97) | LDD1530 | [4] |
| LDCM0292 | AC31 | HEK-293T | C244(0.95) | LDD1531 | [4] |
| LDCM0293 | AC32 | HEK-293T | C244(1.04) | LDD1532 | [4] |
| LDCM0294 | AC33 | HEK-293T | C244(1.07) | LDD1533 | [4] |
| LDCM0295 | AC34 | HEK-293T | C244(0.99) | LDD1534 | [4] |
| LDCM0296 | AC35 | HEK-293T | C244(1.06) | LDD1535 | [4] |
| LDCM0297 | AC36 | HEK-293T | C244(0.93) | LDD1536 | [4] |
| LDCM0298 | AC37 | HEK-293T | C244(1.01) | LDD1537 | [4] |
| LDCM0299 | AC38 | HEK-293T | C244(1.04) | LDD1538 | [4] |
| LDCM0300 | AC39 | HEK-293T | C244(1.04) | LDD1539 | [4] |
| LDCM0301 | AC4 | HEK-293T | C244(1.00) | LDD1540 | [4] |
| LDCM0302 | AC40 | HEK-293T | C244(1.18) | LDD1541 | [4] |
| LDCM0303 | AC41 | HEK-293T | C244(1.05) | LDD1542 | [4] |
| LDCM0304 | AC42 | HEK-293T | C244(0.90) | LDD1543 | [4] |
| LDCM0305 | AC43 | HEK-293T | C244(1.03) | LDD1544 | [4] |
| LDCM0306 | AC44 | HEK-293T | C244(1.00) | LDD1545 | [4] |
| LDCM0307 | AC45 | HEK-293T | C244(0.94) | LDD1546 | [4] |
| LDCM0308 | AC46 | HEK-293T | C244(0.99) | LDD1547 | [4] |
| LDCM0309 | AC47 | HEK-293T | C244(1.06) | LDD1548 | [4] |
| LDCM0310 | AC48 | HEK-293T | C244(1.11) | LDD1549 | [4] |
| LDCM0311 | AC49 | HEK-293T | C244(1.02) | LDD1550 | [4] |
| LDCM0312 | AC5 | HEK-293T | C244(1.04) | LDD1551 | [4] |
| LDCM0313 | AC50 | HEK-293T | C244(0.99) | LDD1552 | [4] |
| LDCM0314 | AC51 | HEK-293T | C244(0.98) | LDD1553 | [4] |
| LDCM0315 | AC52 | HEK-293T | C244(0.98) | LDD1554 | [4] |
| LDCM0316 | AC53 | HEK-293T | C244(1.02) | LDD1555 | [4] |
| LDCM0317 | AC54 | HEK-293T | C244(0.98) | LDD1556 | [4] |
| LDCM0318 | AC55 | HEK-293T | C244(1.03) | LDD1557 | [4] |
| LDCM0319 | AC56 | HEK-293T | C244(1.20) | LDD1558 | [4] |
| LDCM0320 | AC57 | HEK-293T | C244(1.08) | LDD1559 | [4] |
| LDCM0321 | AC58 | HEK-293T | C244(0.98) | LDD1560 | [4] |
| LDCM0322 | AC59 | HEK-293T | C244(1.05) | LDD1561 | [4] |
| LDCM0323 | AC6 | HEK-293T | C244(0.97) | LDD1562 | [4] |
| LDCM0324 | AC60 | HEK-293T | C244(1.04) | LDD1563 | [4] |
| LDCM0325 | AC61 | HEK-293T | C244(0.93) | LDD1564 | [4] |
| LDCM0326 | AC62 | HEK-293T | C244(0.98) | LDD1565 | [4] |
| LDCM0327 | AC63 | HEK-293T | C244(1.03) | LDD1566 | [4] |
| LDCM0328 | AC64 | HEK-293T | C244(1.01) | LDD1567 | [4] |
| LDCM0334 | AC7 | HEK-293T | C244(1.00) | LDD1568 | [4] |
| LDCM0345 | AC8 | HEK-293T | C244(1.12) | LDD1569 | [4] |
| LDCM0248 | AKOS034007472 | HEK-293T | C244(1.05) | LDD1511 | [4] |
| LDCM0356 | AKOS034007680 | HEK-293T | C244(1.03) | LDD1570 | [4] |
| LDCM0275 | AKOS034007705 | HEK-293T | C244(1.06) | LDD1514 | [4] |
| LDCM0156 | Aniline | NCI-H1299 | 12.24 | LDD0403 | [1] |
| LDCM0632 | CL-Sc | Hep-G2 | C244(2.07) | LDD2227 | [2] |
| LDCM0367 | CL1 | HEK-293T | C244(0.98) | LDD1571 | [4] |
| LDCM0368 | CL10 | HEK-293T | C244(0.90) | LDD1572 | [4] |
| LDCM0369 | CL100 | HEK-293T | C244(1.09) | LDD1573 | [4] |
| LDCM0370 | CL101 | HEK-293T | C244(1.00) | LDD1574 | [4] |
| LDCM0371 | CL102 | HEK-293T | C244(0.89) | LDD1575 | [4] |
| LDCM0372 | CL103 | HEK-293T | C244(1.01) | LDD1576 | [4] |
| LDCM0373 | CL104 | HEK-293T | C244(1.10) | LDD1577 | [4] |
| LDCM0374 | CL105 | HEK-293T | C244(0.83) | LDD1578 | [4] |
| LDCM0375 | CL106 | HEK-293T | C244(0.92) | LDD1579 | [4] |
| LDCM0376 | CL107 | HEK-293T | C244(1.01) | LDD1580 | [4] |
| LDCM0377 | CL108 | HEK-293T | C244(0.98) | LDD1581 | [4] |
| LDCM0378 | CL109 | HEK-293T | C244(1.01) | LDD1582 | [4] |
| LDCM0379 | CL11 | HEK-293T | C244(1.01) | LDD1583 | [4] |
| LDCM0380 | CL110 | HEK-293T | C244(0.90) | LDD1584 | [4] |
| LDCM0381 | CL111 | HEK-293T | C244(0.89) | LDD1585 | [4] |
| LDCM0382 | CL112 | HEK-293T | C244(1.09) | LDD1586 | [4] |
| LDCM0383 | CL113 | HEK-293T | C244(0.99) | LDD1587 | [4] |
| LDCM0384 | CL114 | HEK-293T | C244(0.83) | LDD1588 | [4] |
| LDCM0385 | CL115 | HEK-293T | C244(0.93) | LDD1589 | [4] |
| LDCM0386 | CL116 | HEK-293T | C244(0.98) | LDD1590 | [4] |
| LDCM0387 | CL117 | HEK-293T | C244(0.95) | LDD1591 | [4] |
| LDCM0388 | CL118 | HEK-293T | C244(0.90) | LDD1592 | [4] |
| LDCM0389 | CL119 | HEK-293T | C244(0.98) | LDD1593 | [4] |
| LDCM0390 | CL12 | HEK-293T | C244(1.08) | LDD1594 | [4] |
| LDCM0391 | CL120 | HEK-293T | C244(1.01) | LDD1595 | [4] |
| LDCM0392 | CL121 | HEK-293T | C244(0.94) | LDD1596 | [4] |
| LDCM0393 | CL122 | HEK-293T | C244(1.00) | LDD1597 | [4] |
| LDCM0394 | CL123 | HEK-293T | C244(0.90) | LDD1598 | [4] |
| LDCM0395 | CL124 | HEK-293T | C244(0.95) | LDD1599 | [4] |
| LDCM0396 | CL125 | HEK-293T | C244(1.00) | LDD1600 | [4] |
| LDCM0397 | CL126 | HEK-293T | C244(0.97) | LDD1601 | [4] |
| LDCM0398 | CL127 | HEK-293T | C244(0.94) | LDD1602 | [4] |
| LDCM0399 | CL128 | HEK-293T | C244(0.98) | LDD1603 | [4] |
| LDCM0400 | CL13 | HEK-293T | C244(1.05) | LDD1604 | [4] |
| LDCM0401 | CL14 | HEK-293T | C244(0.95) | LDD1605 | [4] |
| LDCM0402 | CL15 | HEK-293T | C244(0.95) | LDD1606 | [4] |
| LDCM0403 | CL16 | HEK-293T | C244(1.04) | LDD1607 | [4] |
| LDCM0404 | CL17 | HEK-293T | C244(0.97) | LDD1608 | [4] |
| LDCM0405 | CL18 | HEK-293T | C244(1.14) | LDD1609 | [4] |
| LDCM0406 | CL19 | HEK-293T | C244(1.12) | LDD1610 | [4] |
| LDCM0407 | CL2 | HEK-293T | C244(0.96) | LDD1611 | [4] |
| LDCM0408 | CL20 | HEK-293T | C244(1.12) | LDD1612 | [4] |
| LDCM0409 | CL21 | HEK-293T | C244(0.90) | LDD1613 | [4] |
| LDCM0410 | CL22 | HEK-293T | C244(1.07) | LDD1614 | [4] |
| LDCM0411 | CL23 | HEK-293T | C244(1.09) | LDD1615 | [4] |
| LDCM0412 | CL24 | HEK-293T | C244(1.25) | LDD1616 | [4] |
| LDCM0413 | CL25 | HEK-293T | C244(0.96) | LDD1617 | [4] |
| LDCM0414 | CL26 | HEK-293T | C244(1.02) | LDD1618 | [4] |
| LDCM0415 | CL27 | HEK-293T | C244(1.04) | LDD1619 | [4] |
| LDCM0416 | CL28 | HEK-293T | C244(1.15) | LDD1620 | [4] |
| LDCM0417 | CL29 | HEK-293T | C244(1.11) | LDD1621 | [4] |
| LDCM0418 | CL3 | HEK-293T | C244(0.94) | LDD1622 | [4] |
| LDCM0419 | CL30 | HEK-293T | C244(1.03) | LDD1623 | [4] |
| LDCM0420 | CL31 | HEK-293T | C244(1.00) | LDD1624 | [4] |
| LDCM0421 | CL32 | HEK-293T | C244(1.06) | LDD1625 | [4] |
| LDCM0422 | CL33 | HEK-293T | C244(1.04) | LDD1626 | [4] |
| LDCM0423 | CL34 | HEK-293T | C244(0.98) | LDD1627 | [4] |
| LDCM0424 | CL35 | HEK-293T | C244(1.08) | LDD1628 | [4] |
| LDCM0425 | CL36 | HEK-293T | C244(1.16) | LDD1629 | [4] |
| LDCM0426 | CL37 | HEK-293T | C244(0.96) | LDD1630 | [4] |
| LDCM0428 | CL39 | HEK-293T | C244(0.98) | LDD1632 | [4] |
| LDCM0429 | CL4 | HEK-293T | C244(1.00) | LDD1633 | [4] |
| LDCM0430 | CL40 | HEK-293T | C244(1.17) | LDD1634 | [4] |
| LDCM0431 | CL41 | HEK-293T | C244(1.02) | LDD1635 | [4] |
| LDCM0432 | CL42 | HEK-293T | C244(0.99) | LDD1636 | [4] |
| LDCM0433 | CL43 | HEK-293T | C244(1.06) | LDD1637 | [4] |
| LDCM0434 | CL44 | HEK-293T | C244(1.06) | LDD1638 | [4] |
| LDCM0435 | CL45 | HEK-293T | C244(1.00) | LDD1639 | [4] |
| LDCM0436 | CL46 | HEK-293T | C244(0.98) | LDD1640 | [4] |
| LDCM0437 | CL47 | HEK-293T | C244(0.98) | LDD1641 | [4] |
| LDCM0438 | CL48 | HEK-293T | C244(1.05) | LDD1642 | [4] |
| LDCM0439 | CL49 | HEK-293T | C244(1.00) | LDD1643 | [4] |
| LDCM0440 | CL5 | HEK-293T | C244(1.07) | LDD1644 | [4] |
| LDCM0441 | CL50 | HEK-293T | C244(0.92) | LDD1645 | [4] |
| LDCM0443 | CL52 | HEK-293T | C244(1.06) | LDD1646 | [4] |
| LDCM0444 | CL53 | HEK-293T | C244(0.94) | LDD1647 | [4] |
| LDCM0445 | CL54 | HEK-293T | C244(0.89) | LDD1648 | [4] |
| LDCM0446 | CL55 | HEK-293T | C244(1.12) | LDD1649 | [4] |
| LDCM0447 | CL56 | HEK-293T | C244(1.09) | LDD1650 | [4] |
| LDCM0448 | CL57 | HEK-293T | C244(0.92) | LDD1651 | [4] |
| LDCM0449 | CL58 | HEK-293T | C244(0.97) | LDD1652 | [4] |
| LDCM0450 | CL59 | HEK-293T | C244(1.03) | LDD1653 | [4] |
| LDCM0451 | CL6 | HEK-293T | C244(1.04) | LDD1654 | [4] |
| LDCM0452 | CL60 | HEK-293T | C244(1.11) | LDD1655 | [4] |
| LDCM0453 | CL61 | HEK-293T | C244(1.00) | LDD1656 | [4] |
| LDCM0454 | CL62 | HEK-293T | C244(1.04) | LDD1657 | [4] |
| LDCM0455 | CL63 | HEK-293T | C244(1.01) | LDD1658 | [4] |
| LDCM0456 | CL64 | HEK-293T | C244(0.97) | LDD1659 | [4] |
| LDCM0457 | CL65 | HEK-293T | C244(1.00) | LDD1660 | [4] |
| LDCM0458 | CL66 | HEK-293T | C244(1.02) | LDD1661 | [4] |
| LDCM0459 | CL67 | HEK-293T | C244(1.08) | LDD1662 | [4] |
| LDCM0460 | CL68 | HEK-293T | C244(1.13) | LDD1663 | [4] |
| LDCM0461 | CL69 | HEK-293T | C244(0.92) | LDD1664 | [4] |
| LDCM0462 | CL7 | HEK-293T | C244(1.06) | LDD1665 | [4] |
| LDCM0463 | CL70 | HEK-293T | C244(1.01) | LDD1666 | [4] |
| LDCM0464 | CL71 | HEK-293T | C244(1.10) | LDD1667 | [4] |
| LDCM0465 | CL72 | HEK-293T | C244(1.13) | LDD1668 | [4] |
| LDCM0466 | CL73 | HEK-293T | C244(0.92) | LDD1669 | [4] |
| LDCM0467 | CL74 | HEK-293T | C244(0.97) | LDD1670 | [4] |
| LDCM0469 | CL76 | HEK-293T | C244(0.94) | LDD1672 | [4] |
| LDCM0470 | CL77 | HEK-293T | C244(0.95) | LDD1673 | [4] |
| LDCM0471 | CL78 | HEK-293T | C244(1.04) | LDD1674 | [4] |
| LDCM0472 | CL79 | HEK-293T | C244(1.07) | LDD1675 | [4] |
| LDCM0473 | CL8 | HEK-293T | C244(0.81) | LDD1676 | [4] |
| LDCM0474 | CL80 | HEK-293T | C244(1.18) | LDD1677 | [4] |
| LDCM0475 | CL81 | HEK-293T | C244(0.89) | LDD1678 | [4] |
| LDCM0476 | CL82 | HEK-293T | C244(1.01) | LDD1679 | [4] |
| LDCM0477 | CL83 | HEK-293T | C244(0.95) | LDD1680 | [4] |
| LDCM0478 | CL84 | HEK-293T | C244(1.06) | LDD1681 | [4] |
| LDCM0479 | CL85 | HEK-293T | C244(0.90) | LDD1682 | [4] |
| LDCM0480 | CL86 | HEK-293T | C244(0.89) | LDD1683 | [4] |
| LDCM0481 | CL87 | HEK-293T | C244(0.95) | LDD1684 | [4] |
| LDCM0482 | CL88 | HEK-293T | C244(0.96) | LDD1685 | [4] |
| LDCM0483 | CL89 | HEK-293T | C244(1.11) | LDD1686 | [4] |
| LDCM0484 | CL9 | HEK-293T | C244(0.90) | LDD1687 | [4] |
| LDCM0485 | CL90 | HEK-293T | C244(0.89) | LDD1688 | [4] |
| LDCM0486 | CL91 | HEK-293T | C244(0.97) | LDD1689 | [4] |
| LDCM0487 | CL92 | HEK-293T | C244(0.92) | LDD1690 | [4] |
| LDCM0488 | CL93 | HEK-293T | C244(0.90) | LDD1691 | [4] |
| LDCM0489 | CL94 | HEK-293T | C244(0.98) | LDD1692 | [4] |
| LDCM0490 | CL95 | HEK-293T | C244(0.88) | LDD1693 | [4] |
| LDCM0491 | CL96 | HEK-293T | C244(1.16) | LDD1694 | [4] |
| LDCM0492 | CL97 | HEK-293T | C244(0.97) | LDD1695 | [4] |
| LDCM0493 | CL98 | HEK-293T | C244(0.99) | LDD1696 | [4] |
| LDCM0494 | CL99 | HEK-293T | C244(0.93) | LDD1697 | [4] |
| LDCM0495 | E2913 | HEK-293T | C244(0.99) | LDD1698 | [4] |
| LDCM0468 | Fragment33 | HEK-293T | C244(0.99) | LDD1671 | [4] |
| LDCM0427 | Fragment51 | HEK-293T | C244(0.98) | LDD1631 | [4] |
| LDCM0022 | KB02 | HEK-293T | C244(1.05) | LDD1492 | [4] |
| LDCM0023 | KB03 | HEK-293T | C244(1.09) | LDD1497 | [4] |
| LDCM0024 | KB05 | HEK-293T | C244(1.00) | LDD1502 | [4] |
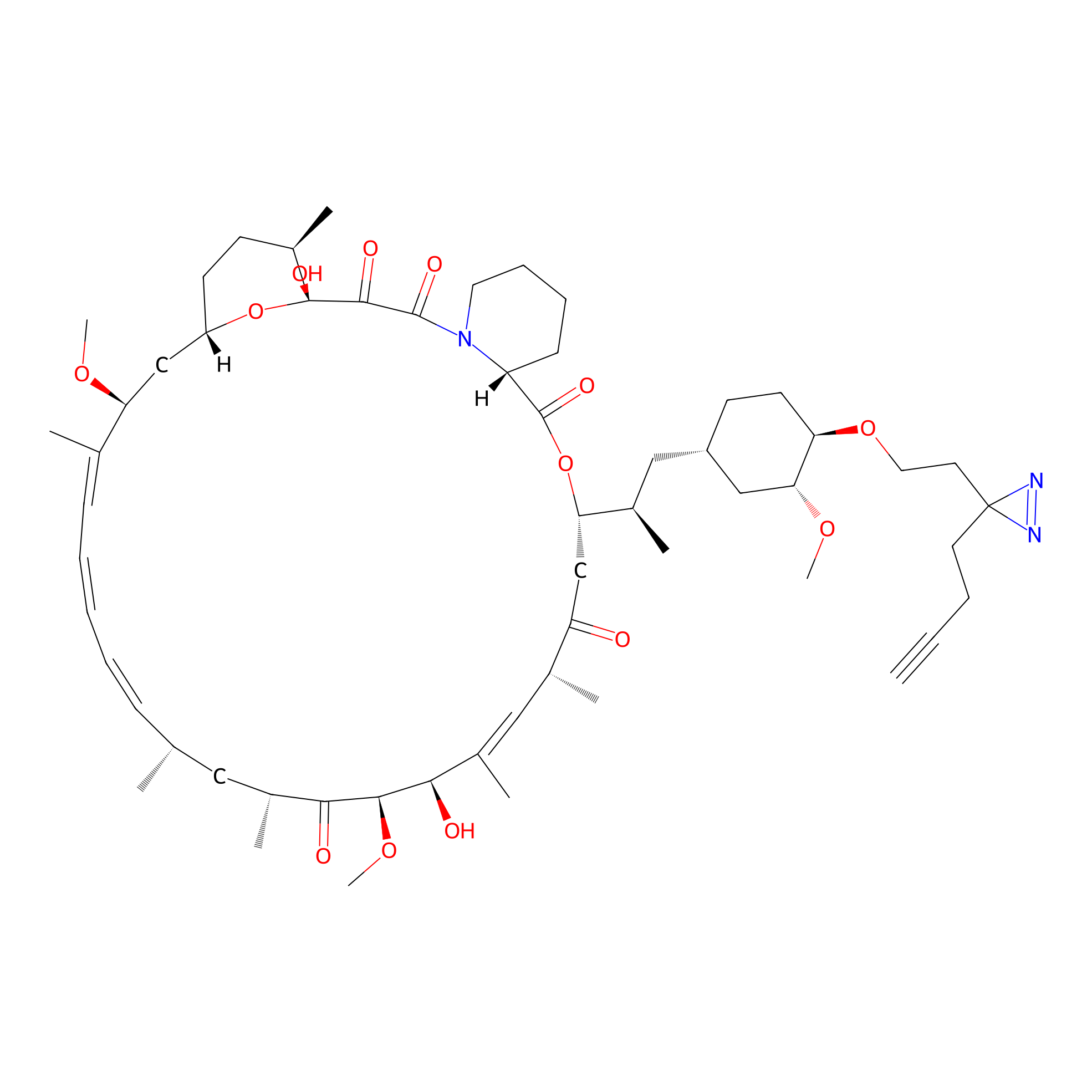
| LDCM0090 | Rapamycin | JHH-7 | 4.06 | LDD0213 | [12] |
The Interaction Atlas With This Target
The Protein(s) Related To This Target
Other
References